
PODSTAWY BIOCHEMII DLA OCHRONY ŚRODOWISKA
Nukleotydy i kwasy nukleinowe
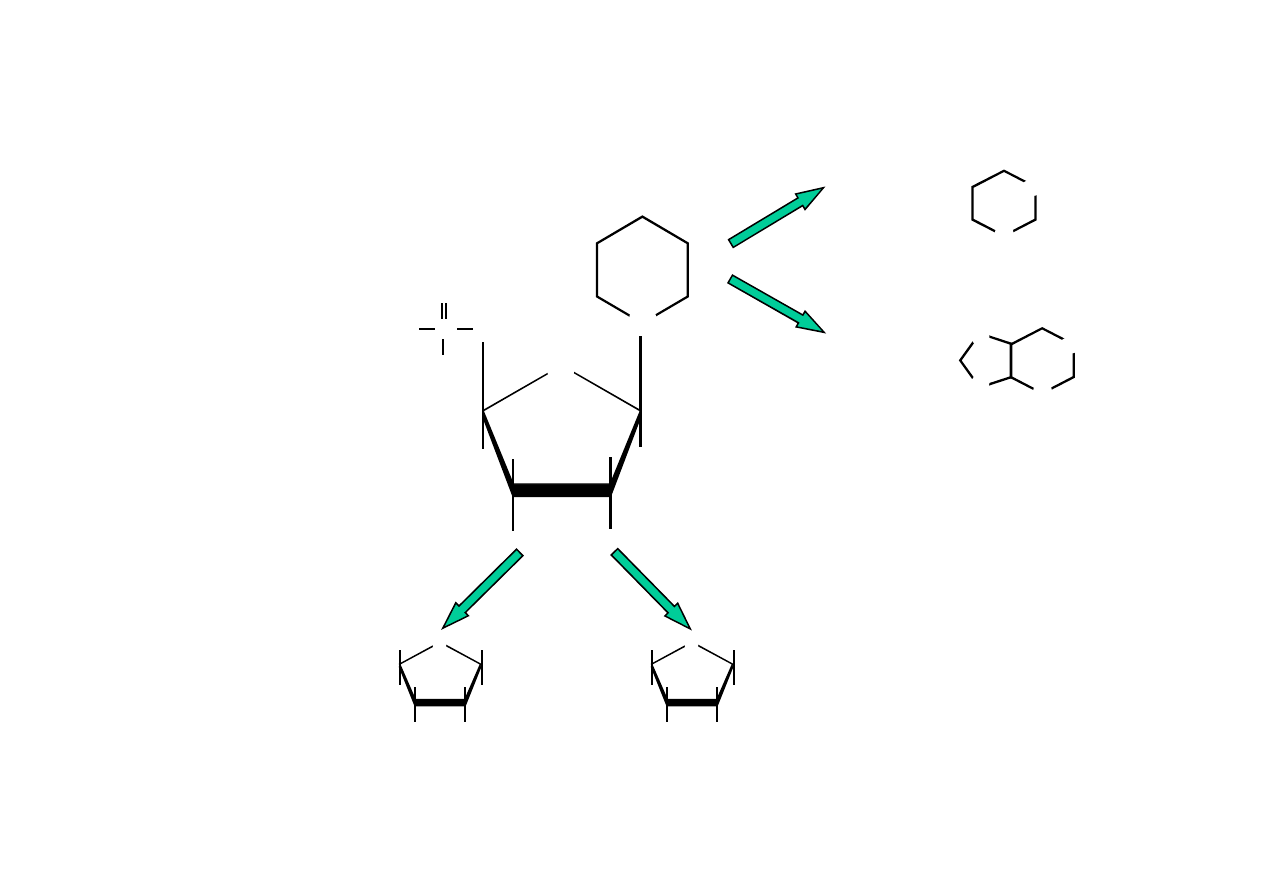
N
1
2
3
4
5
6
N
PIRYMIDYNA
cytozyna
tymina
uracyl
N
N
1
2
3
4
5
6
N
N
7
8
9
PURYNA
adenina
guanina
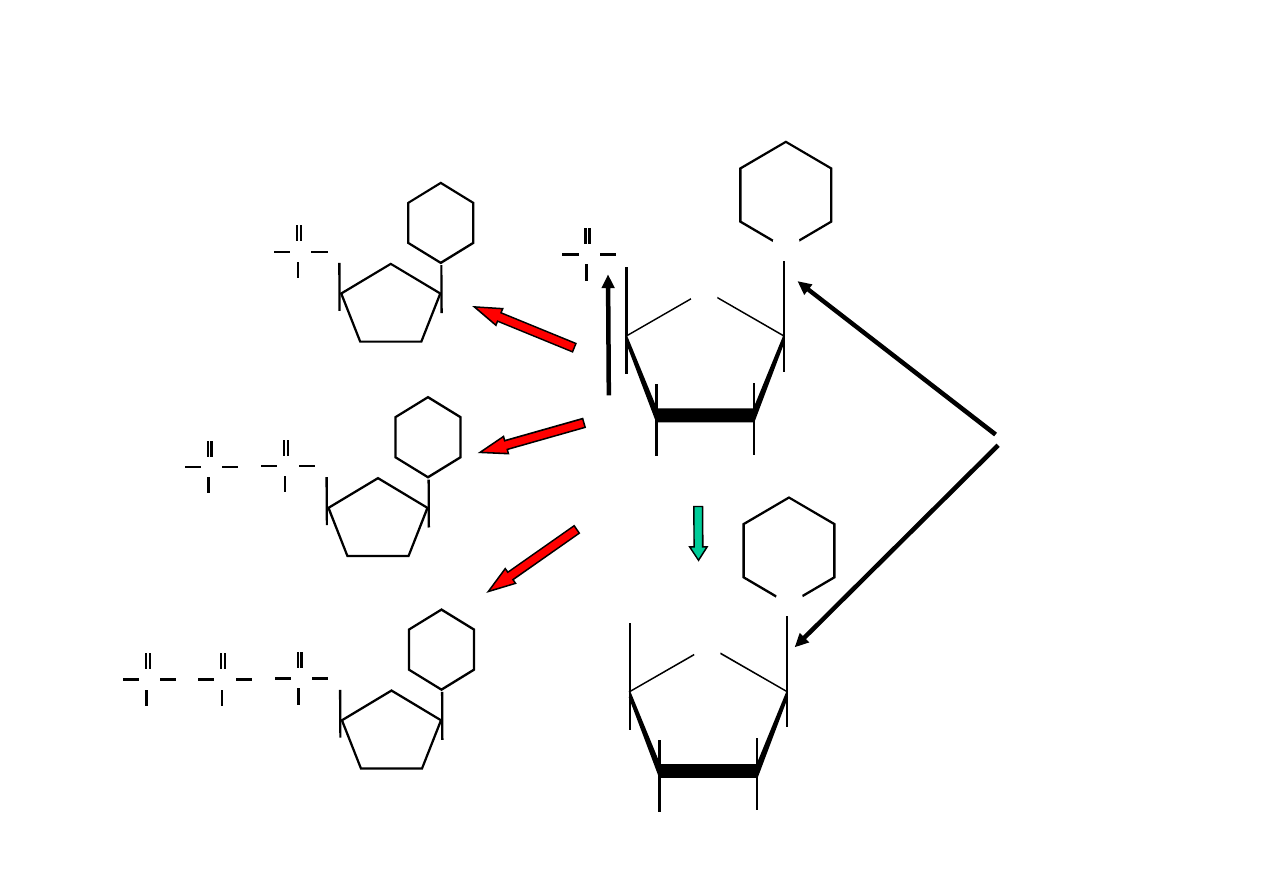
O
N
zasada
cukier
1’
4’
5’
CH
2
P
O
O
-
O
-
reszta kwasu
ortofosforowego
Nukleotydy
OH
OH
H
O
H
H
H
OH
HOCH
2
H
OH
H
O
H
H
H
OH
HOCH
2
ryboza
2-deoksyryboza
2’
3’
O
N
1’
2’
3’
4’
5’
CH
2
P
O
O
-
O
-
CH
2
P
O
O
-
O
-
mono
foforan nukleozydu
O
O
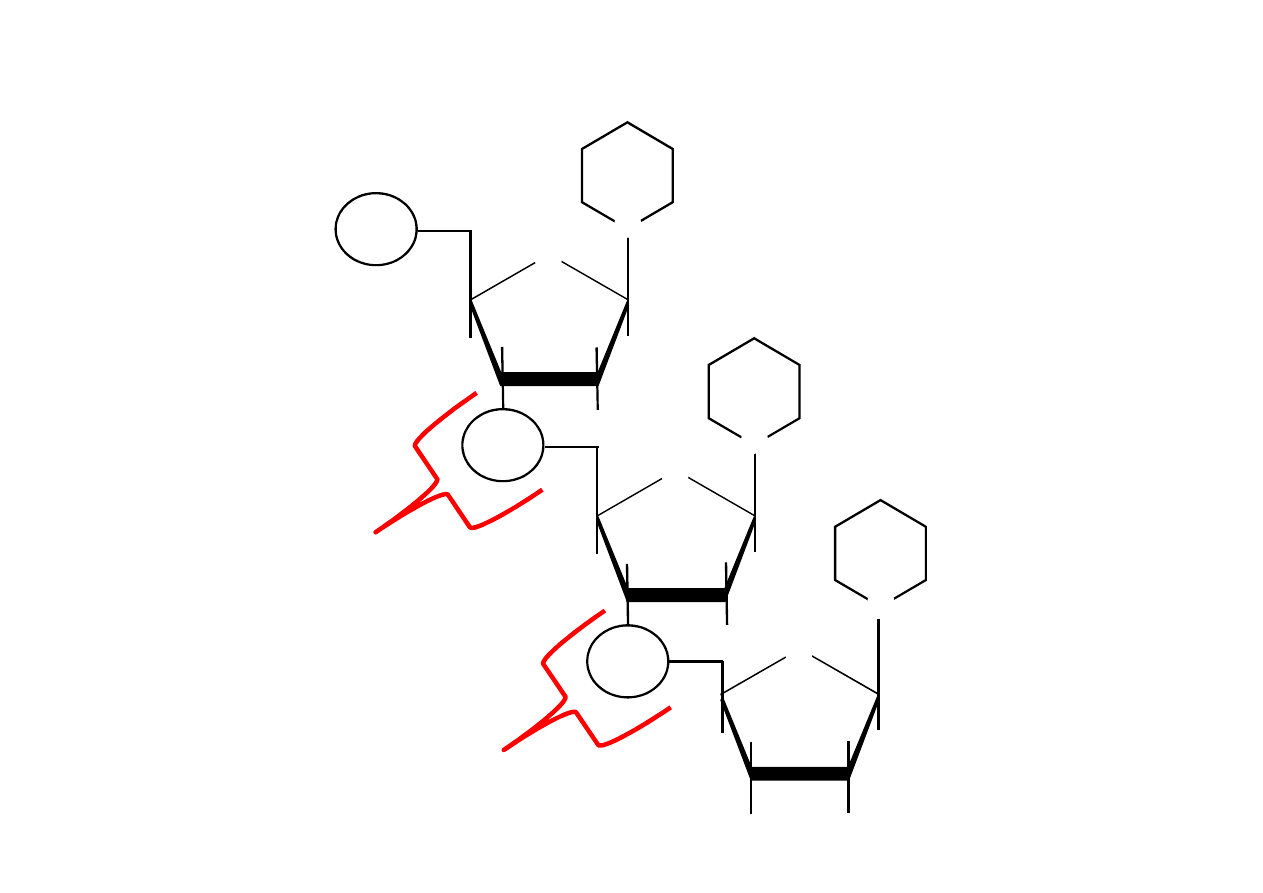
Nukleotydy
wiązanie
N-glikozydowe
wiązanie
estrowe
nukleotyd
O
N
1’
2’
3’
4’
5’
CH
2
OH
nukleozyd
CH
2
P
O
O
-
P
O
-
O
-
di
foforan nukleozydu
CH
2
P
O
O
O
-
P
O
O
-
O
P
O
O
-
O
-
tri
foforan nukleozydu
N-glikozydowe
estrowe
koniec 5’ łańcucha
O
cukier
1’
2’
3’
4’
5’
fosforan
N
zasada
5’
fosforan
N
zasada
Fragment łańcucha kwasu nukleinowego
koniec 3’ łańcucha
N
O
zasada
cukier
1’
2’
3’
4’
5’
O
cukier
1’
2’
3’
4’
5’
fosforan
fosforan
N
wiązanie
fosfodiestrowe
wiązanie
fosfodiestrowe
OH

Kwasy nukleinowe
DNA
kwas rybonukleinowy
bierze udział
kwas deoksyrybonukleinowy
RNA
bierze udział
w rozkodowywaniu
informacji zawartej w DNA
koduje informację o budowie
i działaniu całego organizmu
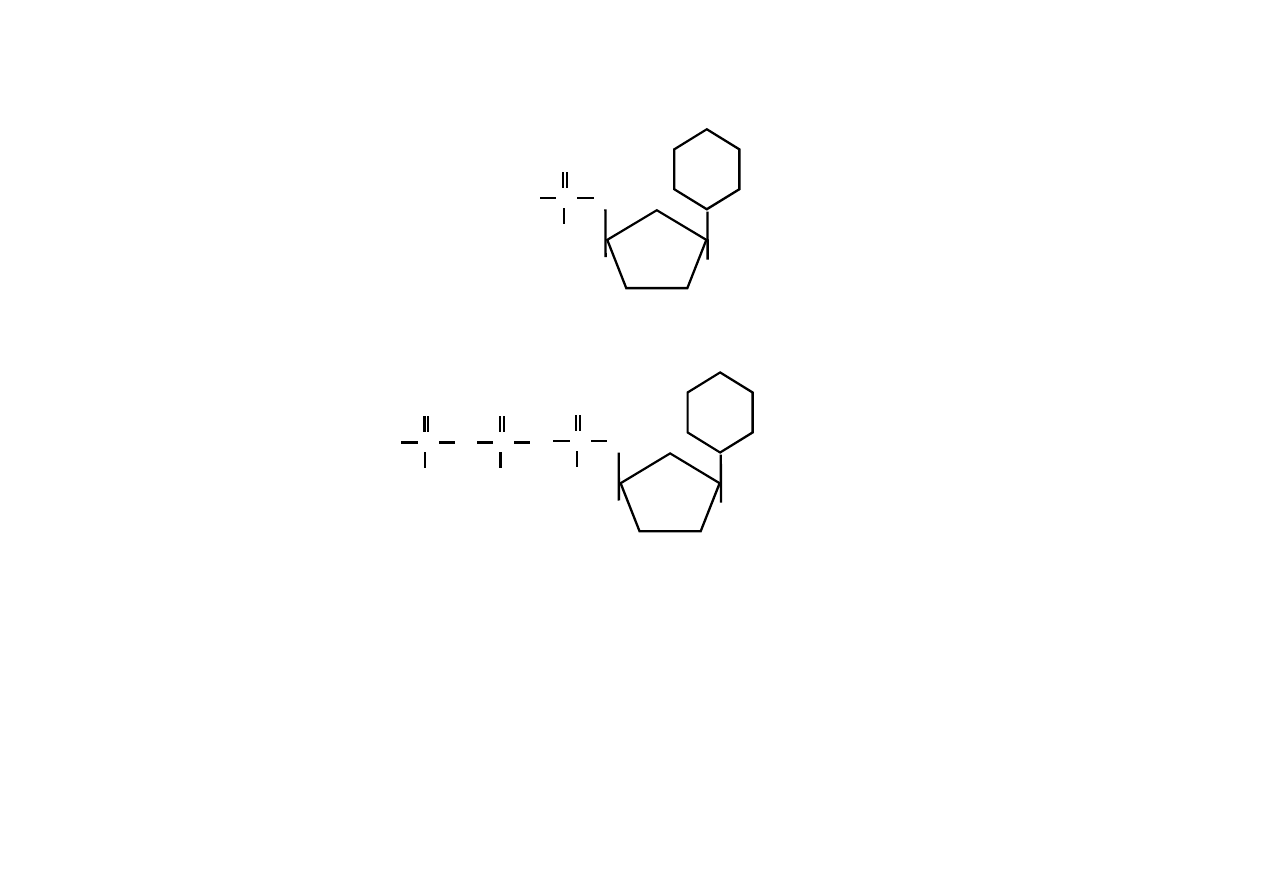
CH
2
P
O
O
-
O
-
mono
foforan nukleozydu
CH
2
P
O
O
P
O
O
P
O
O
-
CH
2
P
O
O
-
P
O
-
O
P
O
-
O
tri
foforan nukleozydu
DNA dATP, dGTP, dCTP,
dTTP
RNA ATP, GTP, CTP,
UTP
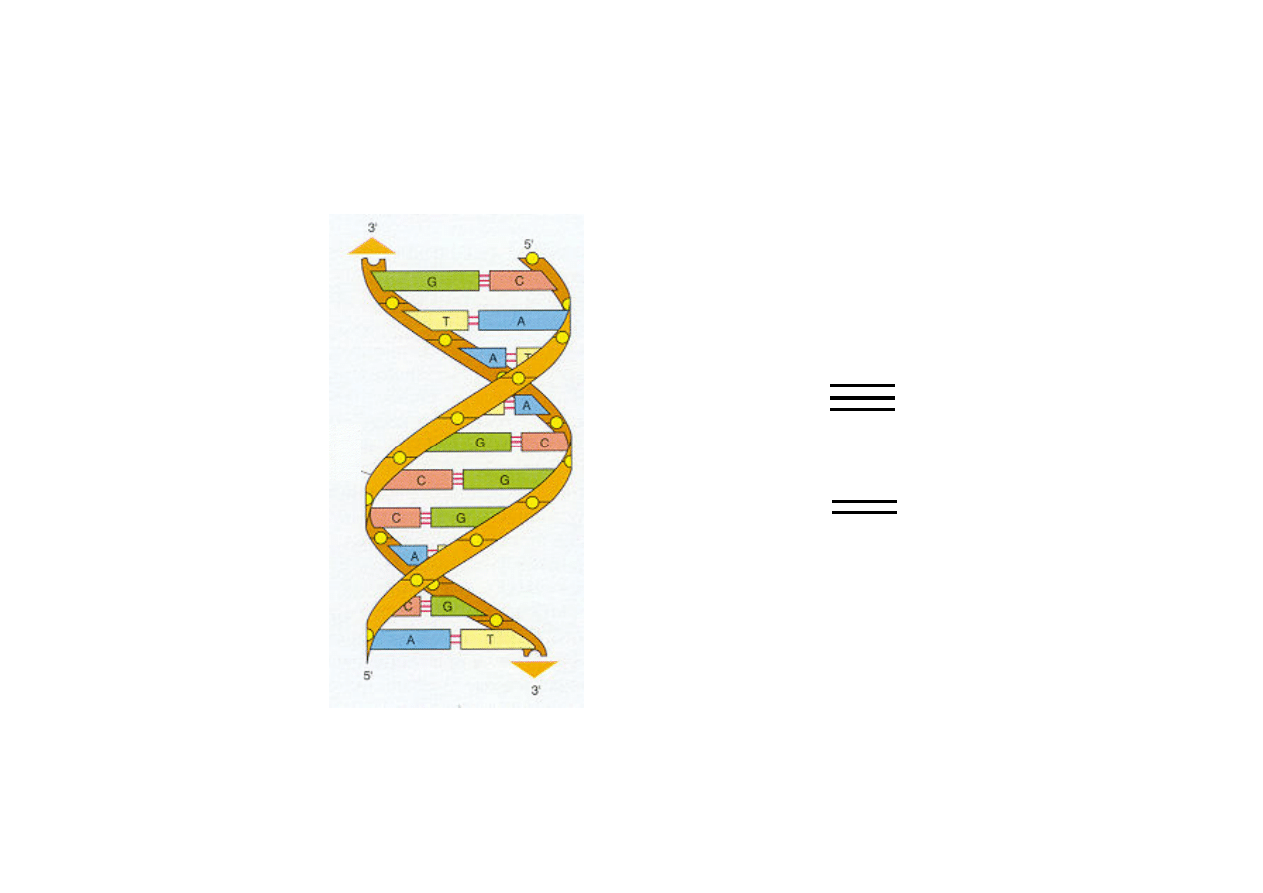
Podwójna helisa DNA
G C
komplementarne parowanie zasad
A T

5’ GGTACCAATTCAAAGCTTATG 3’
3’ CCATGGTTAAGTTTCGAATAC 5’
Enzymy restrykcyjne
Alu I
AG
↓
↓
↓
↓
CT
TC
↑
↑
↑
↑
GA
5’
CT
TATG 3’
3’
GA
ATAC 5’
5’ GGTACCAATTCAA
AG
3’
3’ CCATGGTTAAGTT
TC
5’
Kpn I
GGTAC
↓
↓
↓
↓
C
C
↑
↑
↑
↑
CATGG
5’
GGTAC
3’
3’
C
5’
5’
C
AATTCAAAGCTTATG 3’
3’
CATGG
TTAAGTTTCGAATAC 5’
Hind III
A
↓
↓
↓
↓
AGCTT
TTCGA
↑
↑
↑
↑
A
5’ GGTACCAATTCA
A
3’
3’ CCATGGTTAAGT
TTCGA
5’
5’
AGCTT
ATG 3’
3’
A
TAC 5’
5’ GGTACCAATTCA
A
||||||||||||||||||||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
5’ GGTACCAATTCA
A
||||||||||||||||||||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
Hind III
A
↓
↓
↓
↓
AGCTT
TTCGA
↑
↑
↑
↑
A
Hind III
5’ GGTACCAATTCA
A
3’
5’
AGCTT
ATG 3’
5’ GGTACCAATTCA
A
3’
|||||||||||||
5’
AGCTT
ATG 3’
|||
5’ GGTACCAATTCA
A
|||||||||||||
||||
|||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
|||||||||||||
3’ CCATGGTTAAGT
TTCGA
5’
5’
AGCTT
ATG 3’
|||
3’
A
TAC 5’
|||||||||||||
3’ CCATGGTTAAGT
TTCGA
5’
|||
3’
A
TAC 5’
ligaza
wektor
trawienie enzymem restrykcyjnym
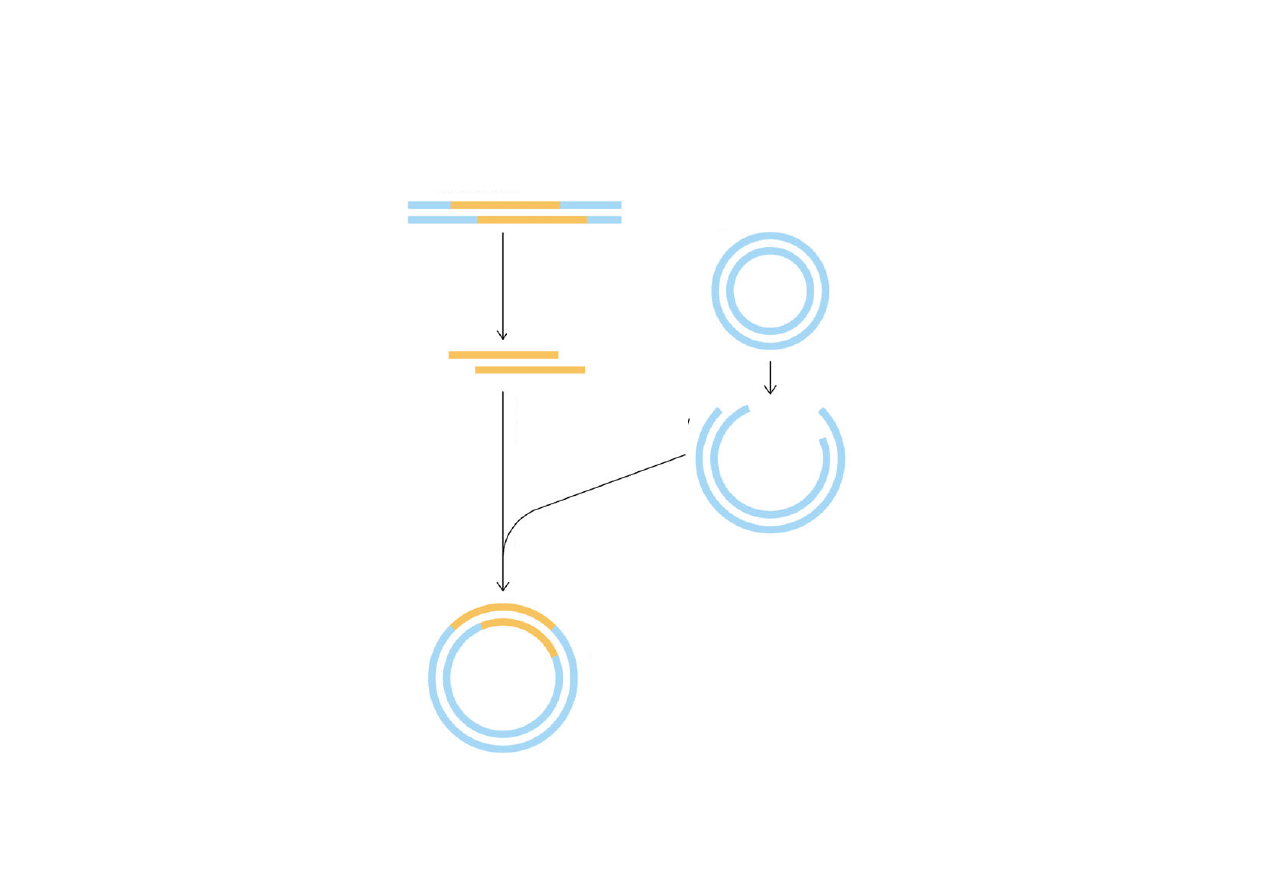
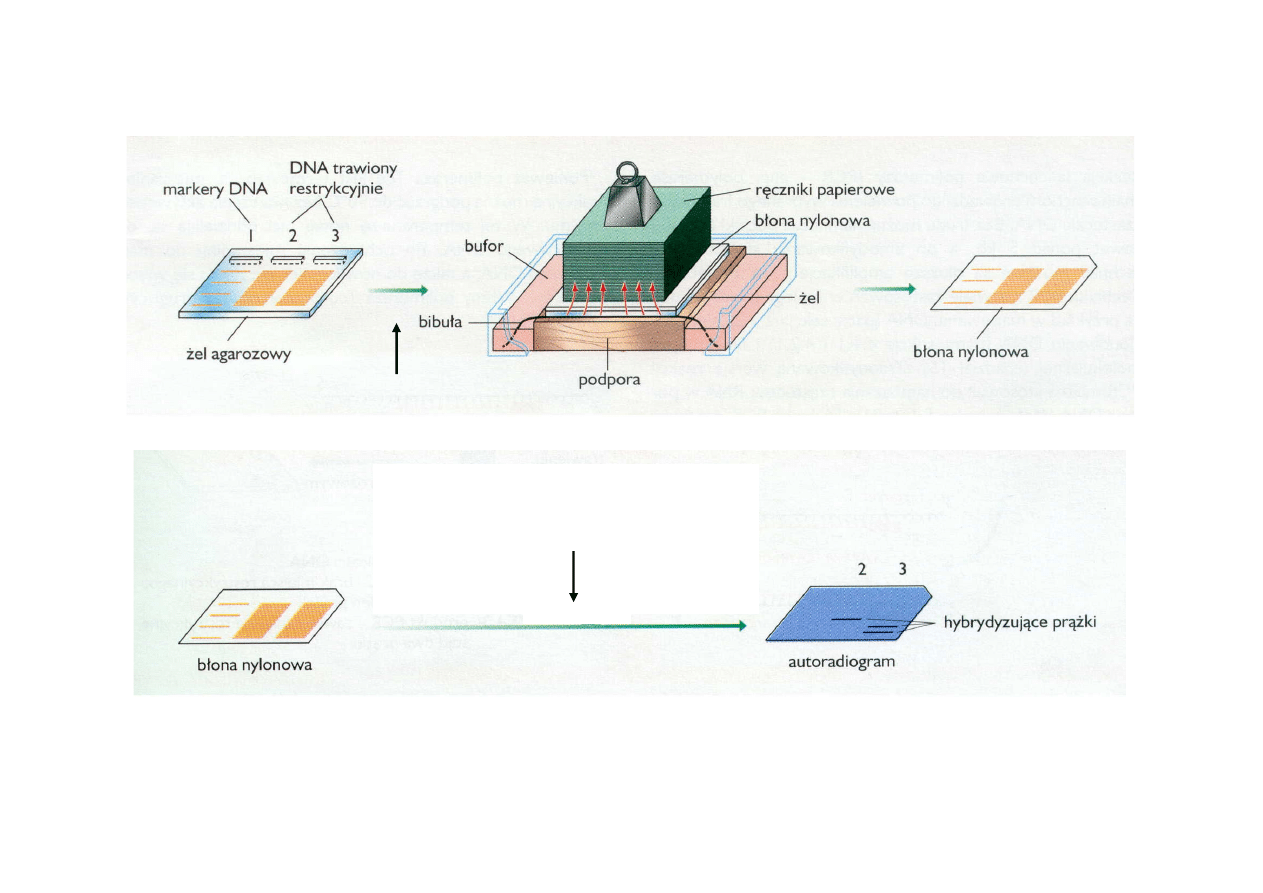
Klonowanie DNA
DNA zawierający wstawkę,
którą chcemy przeklonować
trawienie
enzymem
restrykcyjnym
wstawka
ligacja wektora
ze wstawką
plazmid ze wstawką gotowy
do wprowadzenia do bakterii
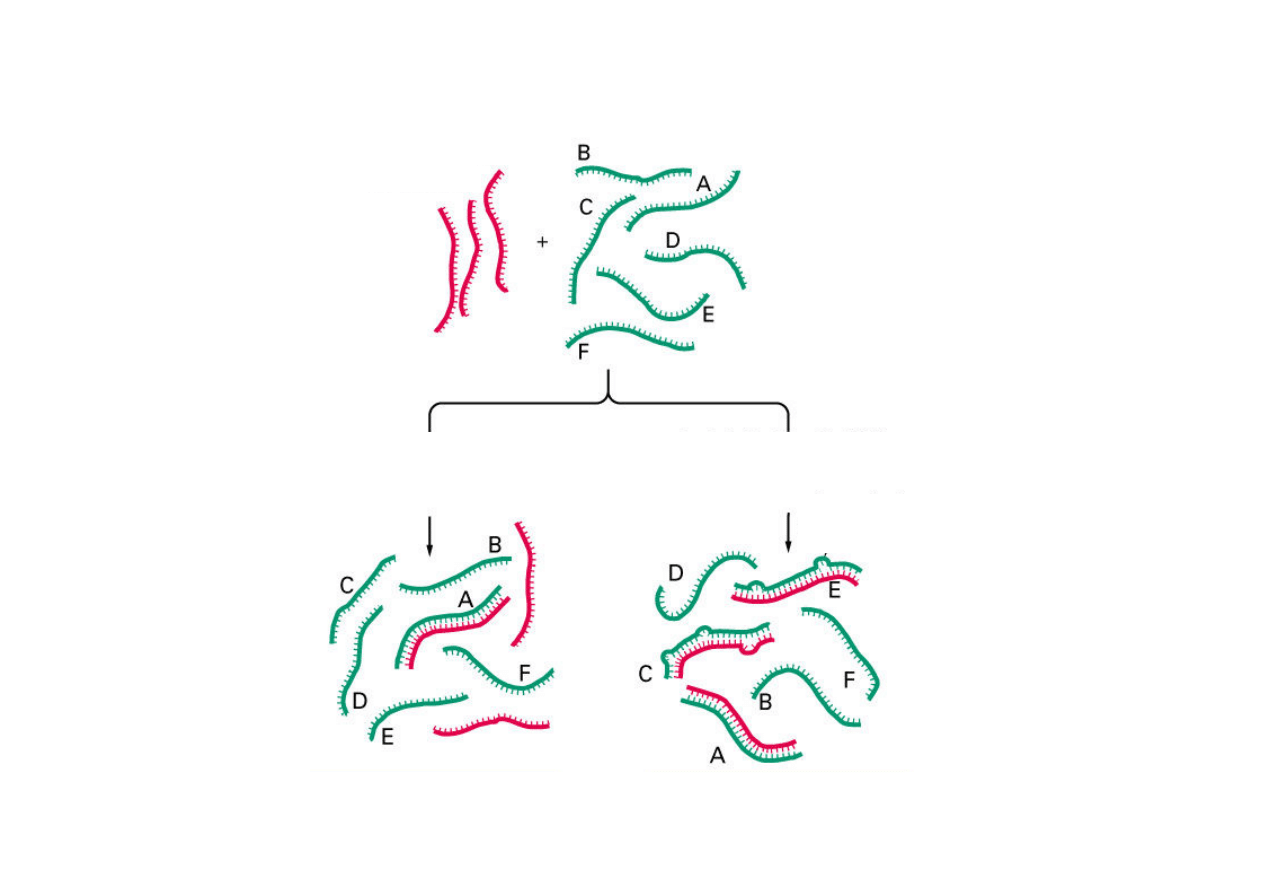
mieszanina
jednoniciowych
cząsteczek DNA
jednoniciowa wyznakowana
sonda komplementarna
do cząsteczki A
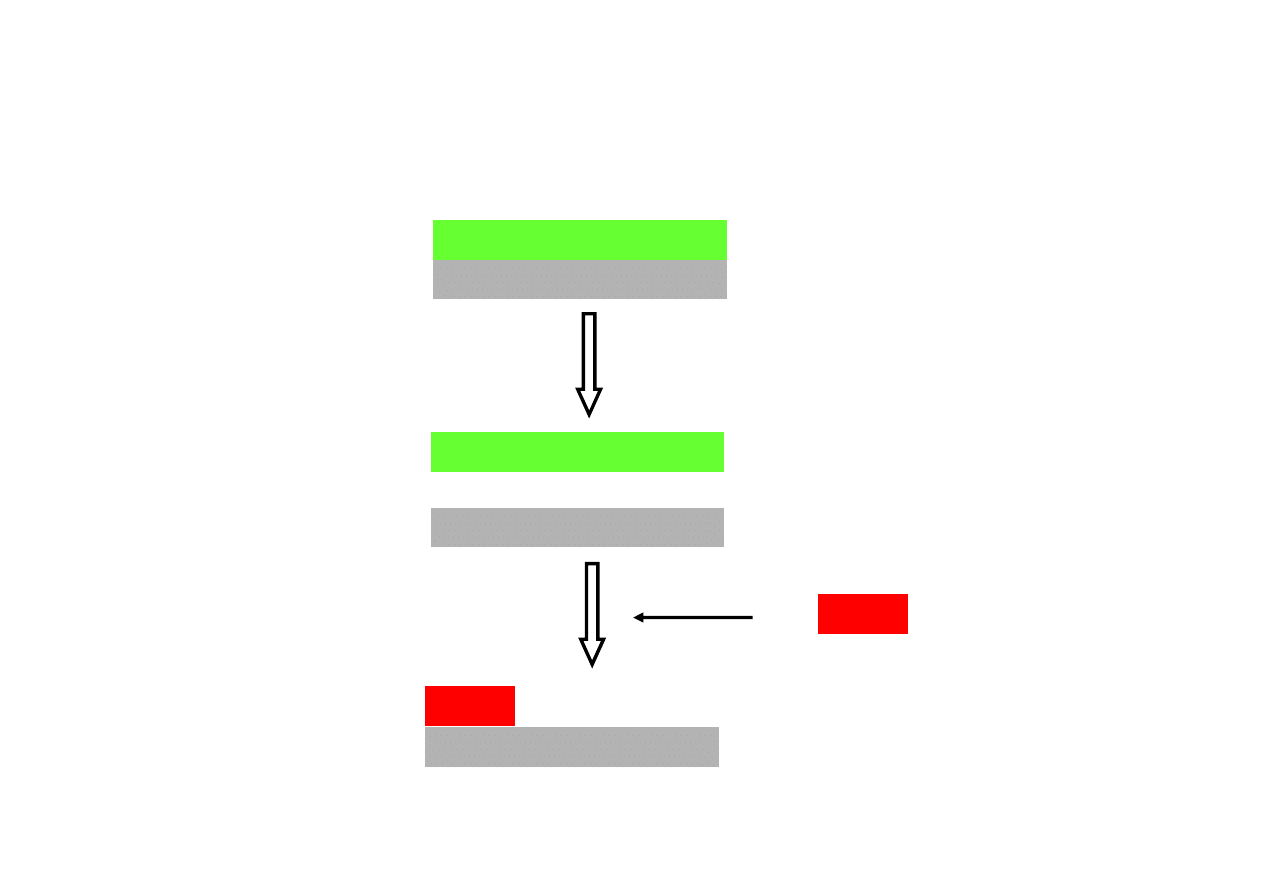
Hybrydyzacja
hybrydyzacja
w warunkach ostrych
hybrydyzacja
w warunkach łagodnych
tylko cząsteczka A tworzy
stabilną dwuniciową cząsteczkę
cząsteczki A, C i E tworzą
stabilne dwuniciowe cząsteczki
Hybrydyzacja
denaturacja DNA w celu
uzyskania jednoniciowych cząsteczek
inkubacja błony z jednoniciową
wyznakowaną radioaktywnie sondą DNA
Sekwencjonowanie DNA
denaturacja
dwuniciowy DNA
GCATATGTCAGTCCAG
CGTATACAGTCAGGTC
5’
3’
3’
5’
GCATATGTCAGTCCAG
GCAT
CGTATACAGTCAGGTC
5’
3’
5’
3’
jednoniciowy DNA
(matryca do sekwencjonowania)
GCATATGTCAGTCCAG
CGTATACAGTCAGGTC
5’
3’
3’
5’
GCAT
5’
3’
przyłączenie znakowanego startera
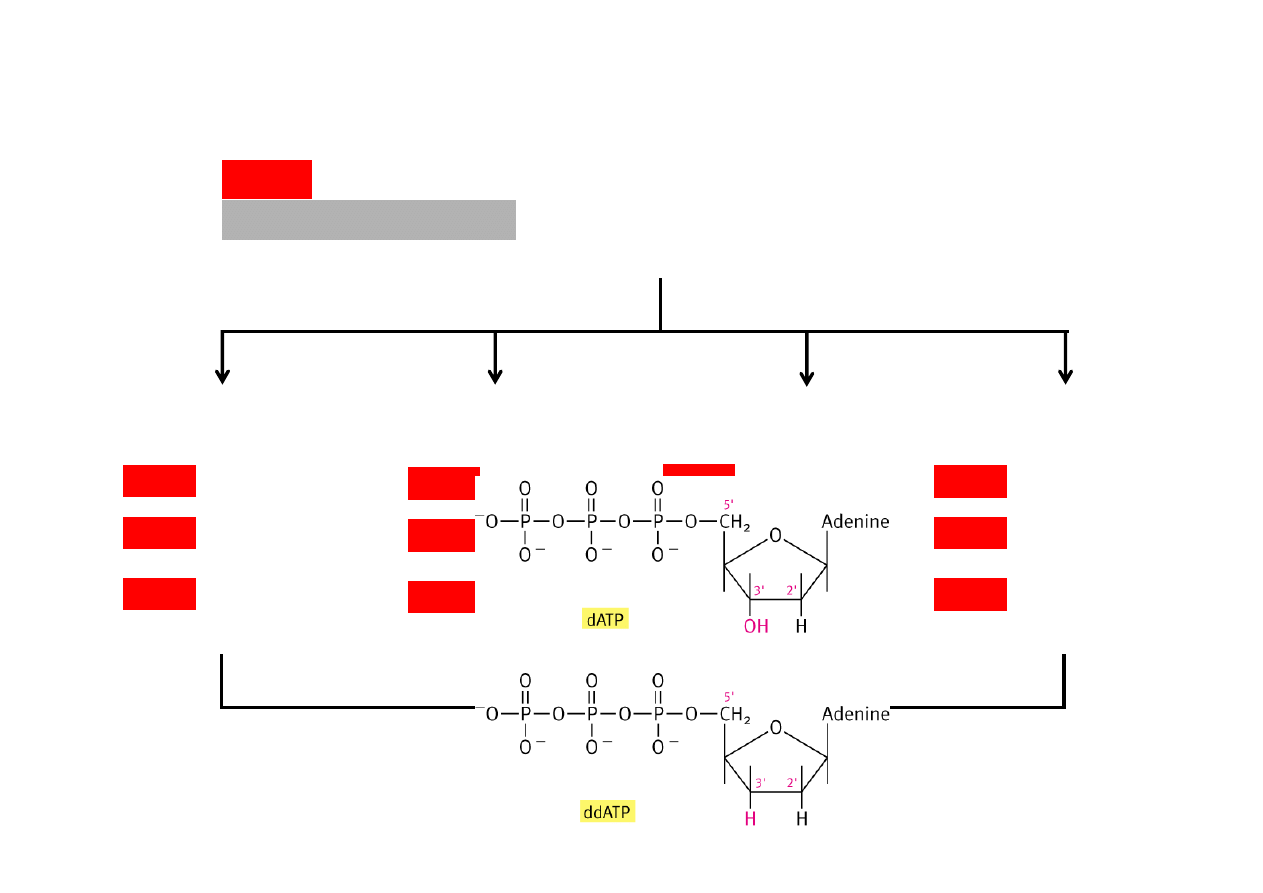
Sekwencjonowanie DNA
GCAT
CGTATACAGTCAGGTC
5’
3’
5’
3’
dATP, dTTP, dCTP, dGTP
+
ddATP
ddGTP
ddCTP
ddTTP
polimeraza DNA
polimeraza DNA
polimeraza DNA
polimeraza DNA
polimeraza DNA
GCAT
GCAT
GCAT
A
ATGTC
A
ATGTCAGTCC
A
GCAT
GCAT
GCAT
A
T
ATG
T
ATGTCAG
T
GCAT
GCAT
GCAT
ATGT
C
ATGTCAGT
C
ATGTCAGTC
C
GCAT
GCAT
GCAT
AT
G
ATGTCA
G
ATGTCAGTCC
G
elektroforeza w żelu poliakrylamidowym
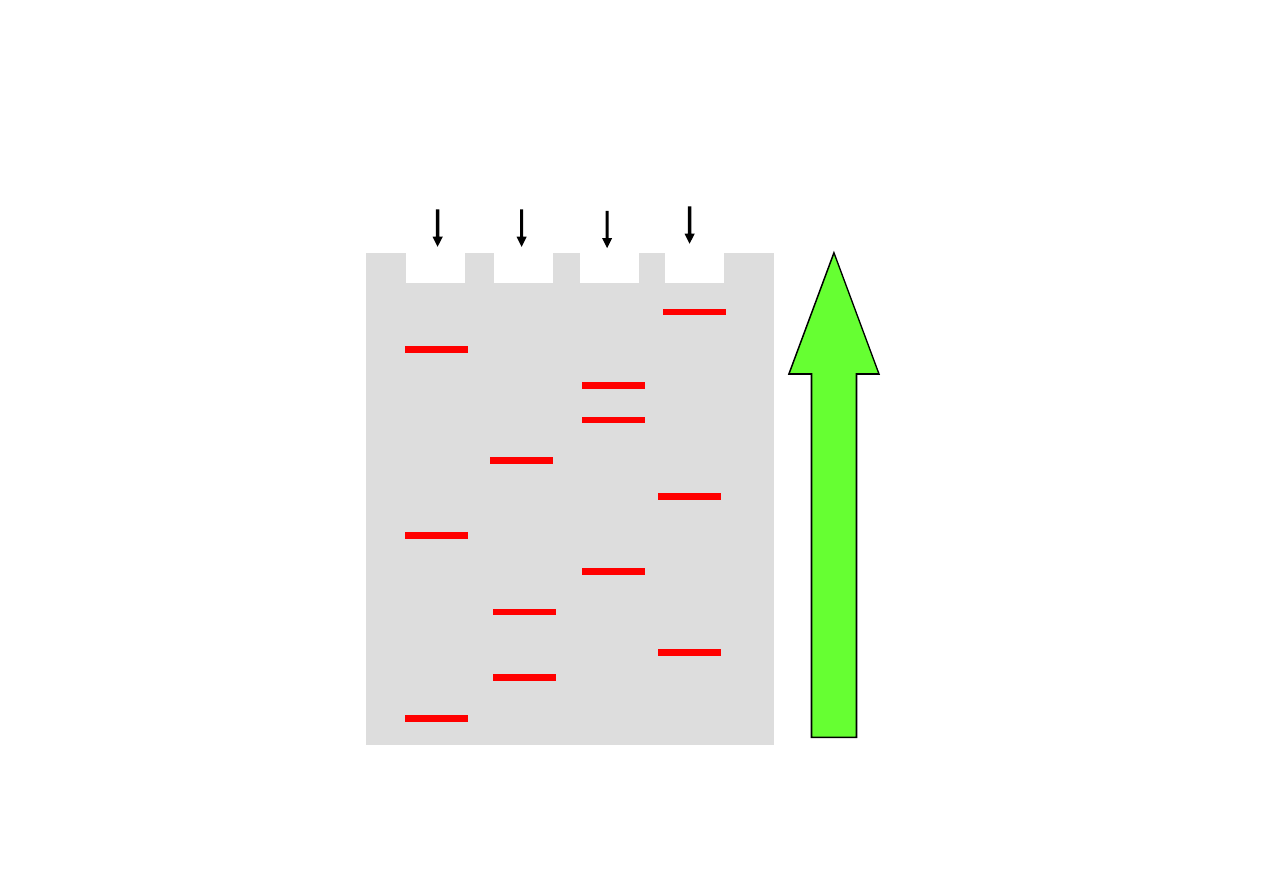
Sekwencjonowanie DNA
ddATP ddTTP ddCTP ddGTP
G
A
C
C
T
3’
A T C G
T
G
A
C
T
G
T
A
5’
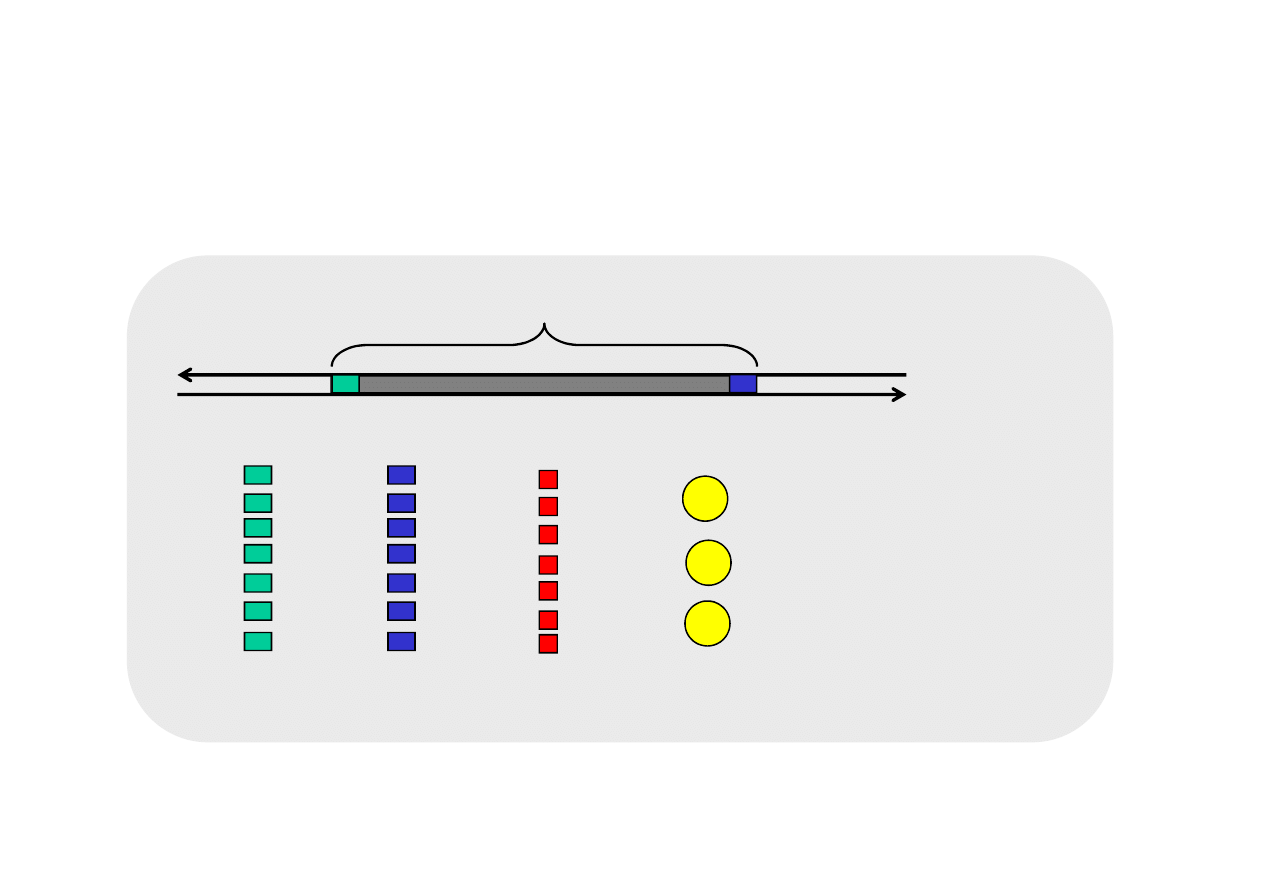
Reakcja PCR
mieszanina reakcyjna
dwuniciowa
matryca DNA
sekwencja docelowa
bufor
dNTP
starter1
starter2
polimeraza Taq

Cykl syntezy DNA w reakcji PCR
etap 1 - denaturacja matrycy
etap 2 - przyłączenie starterów
etap 2 - przyłączenie starterów
etap 3 - wydłużanie starterów
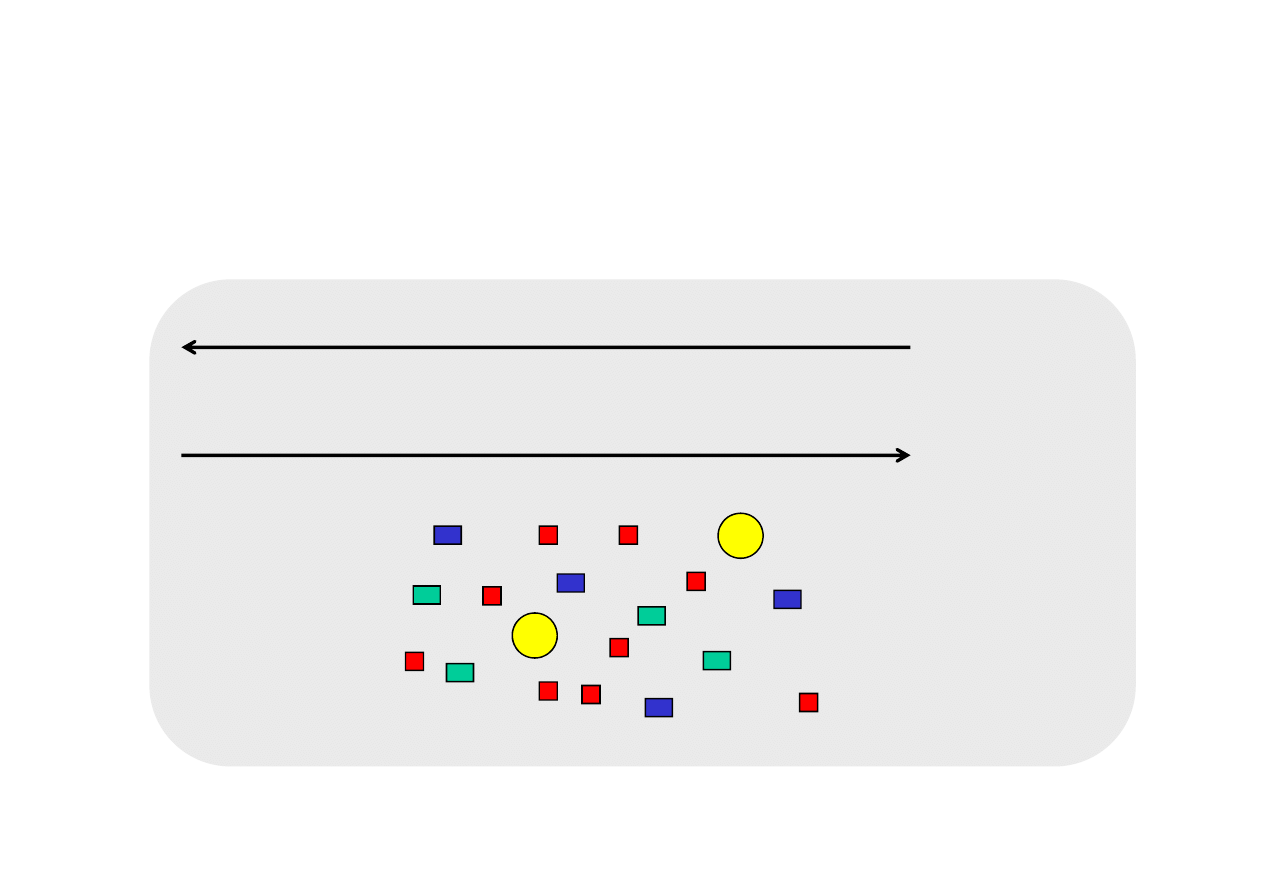
CYKL 1
denaturacja 94°C
jednoniciowa
matryca DNA
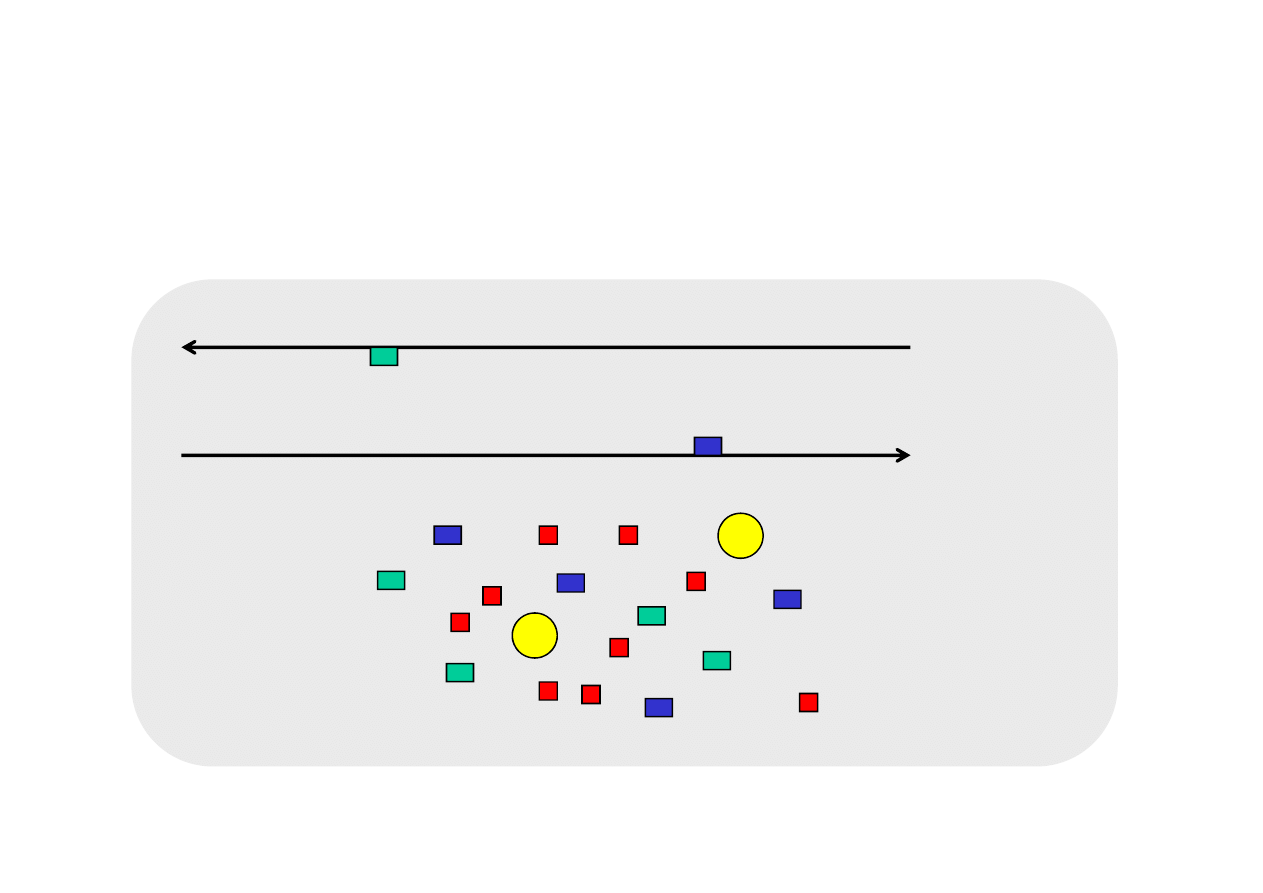
CYKL 1
przyłączenie startera do matrycy 40-65°C
starter 1
starter 2
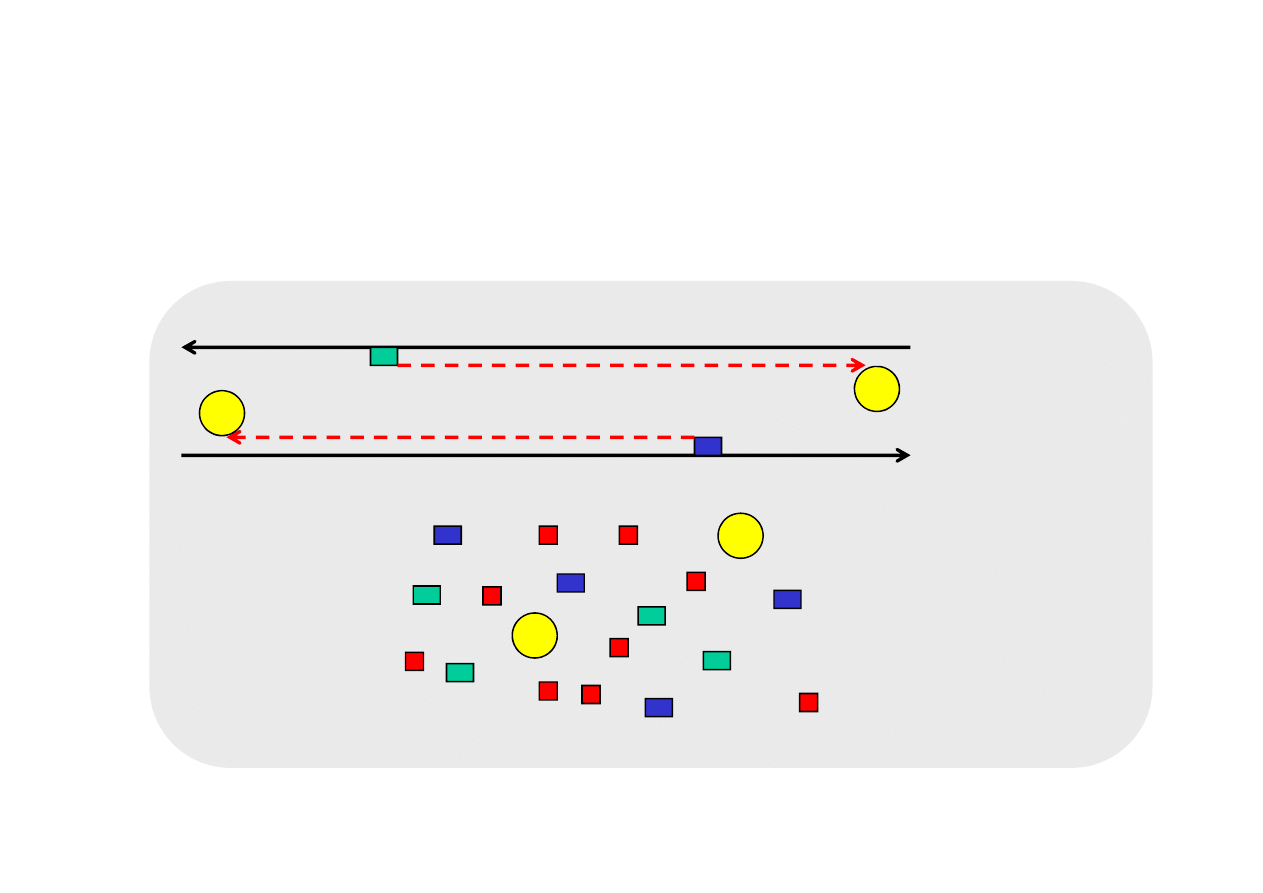
CYKL 1
wydłużanie startera 72°C
starter 1
starter 2
powstają cząsteczki
dłuższe od sekwencji
docelowej
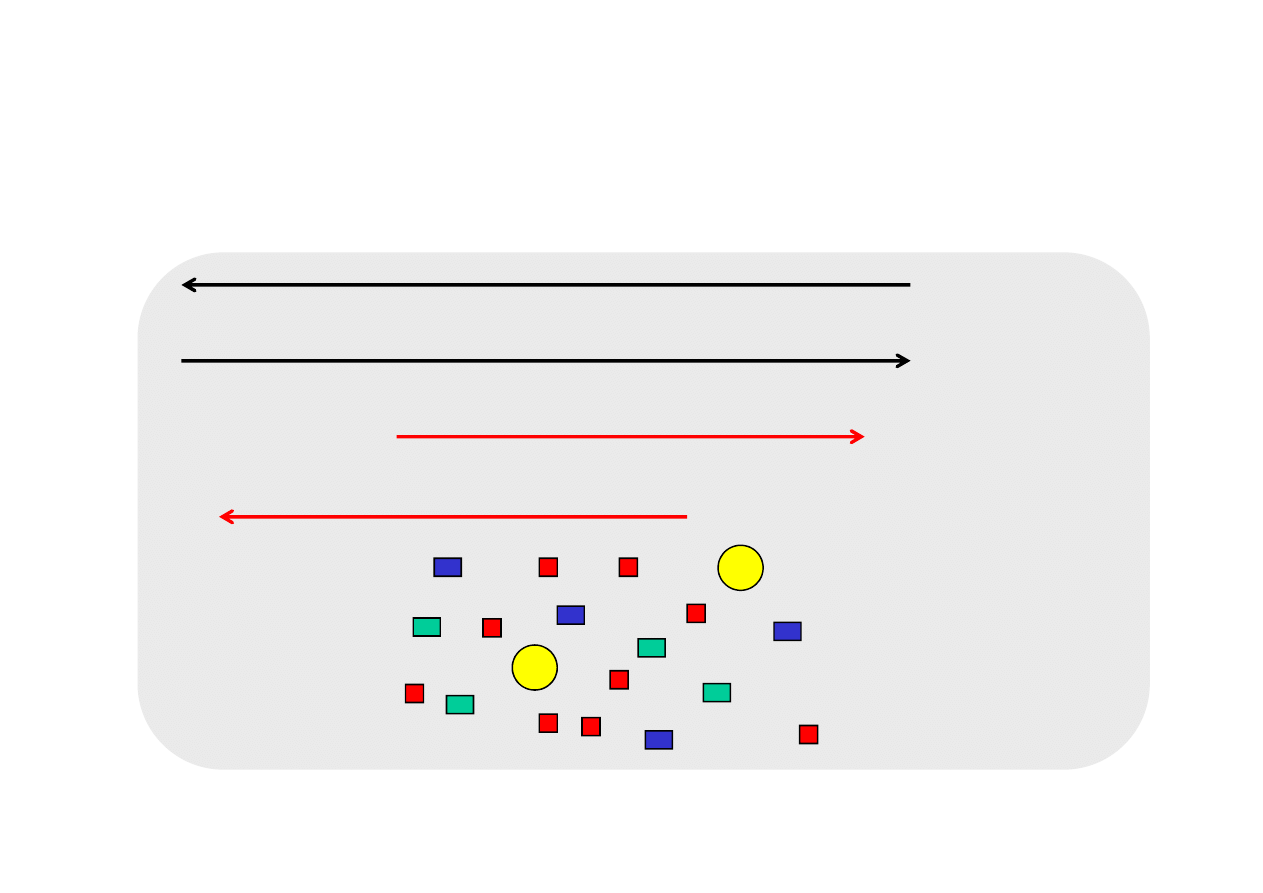
CYKL 2
denaturacja matrycy 94°C
matryca
DNA, który powstał
w cyklu 1
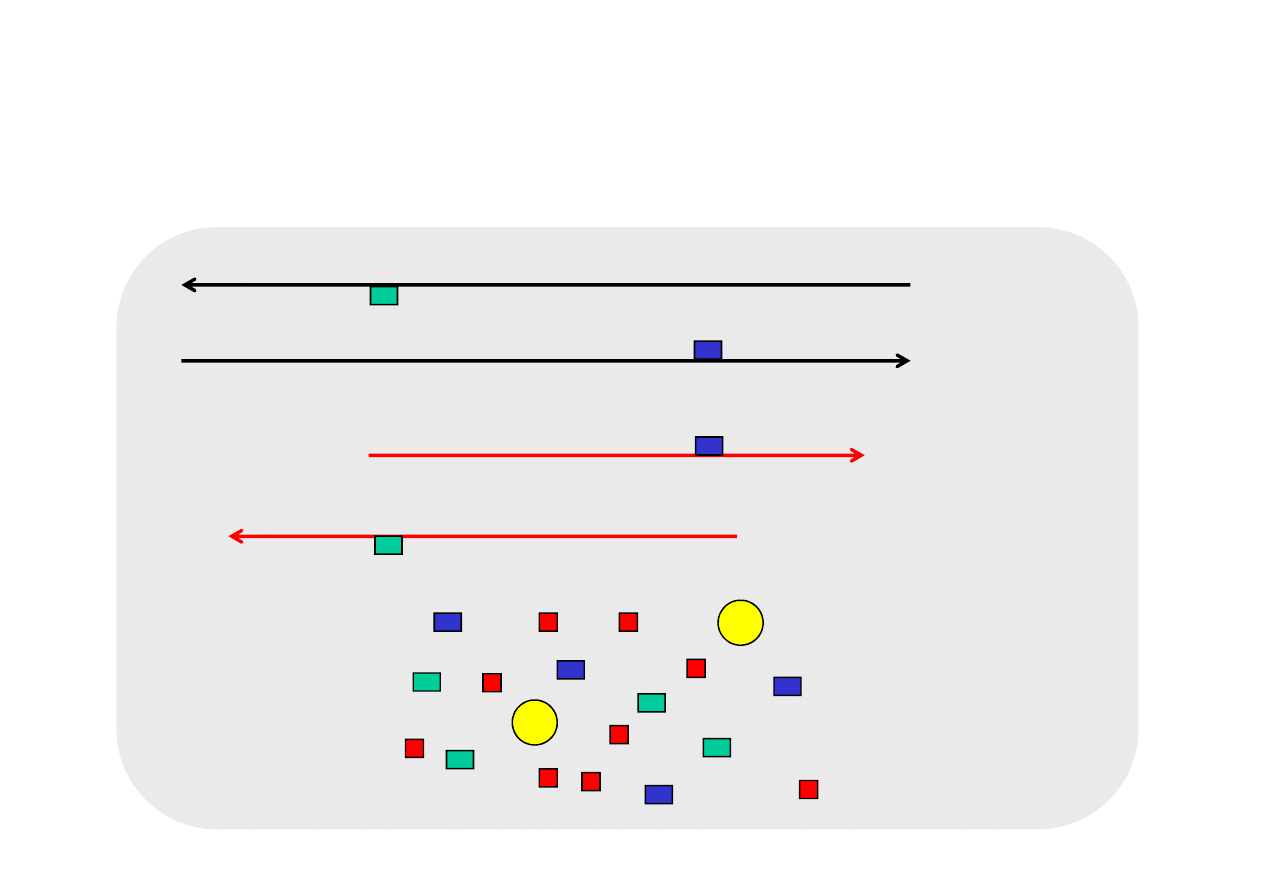
CYKL 2
przyłączenie startera do matrycy 40-65°C
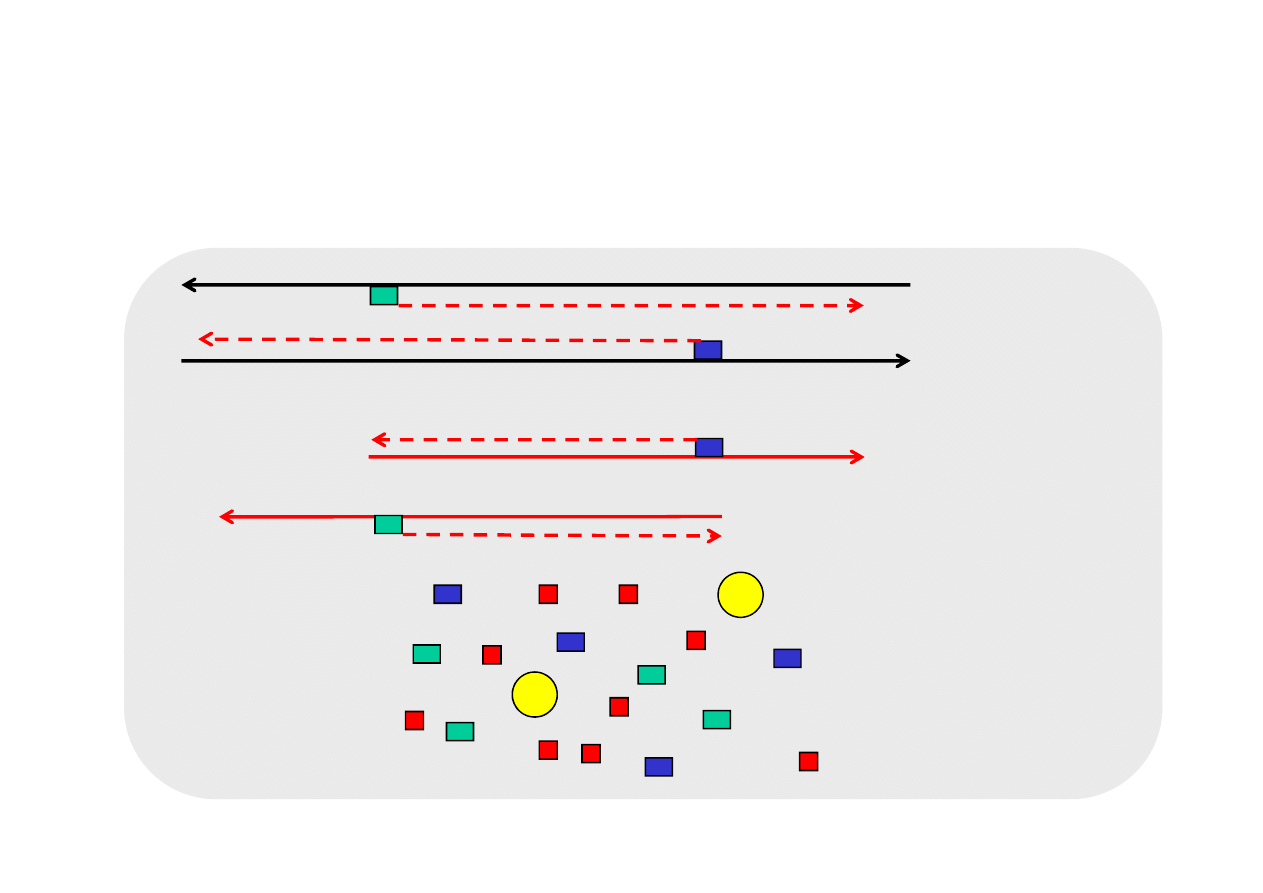
CYKL 2
wydłużanie starterów 72°C
powstają cząsteczki
dłuższe od sekwencji
docelowej
powstają cząsteczki
o długości sekwencji
docelowej
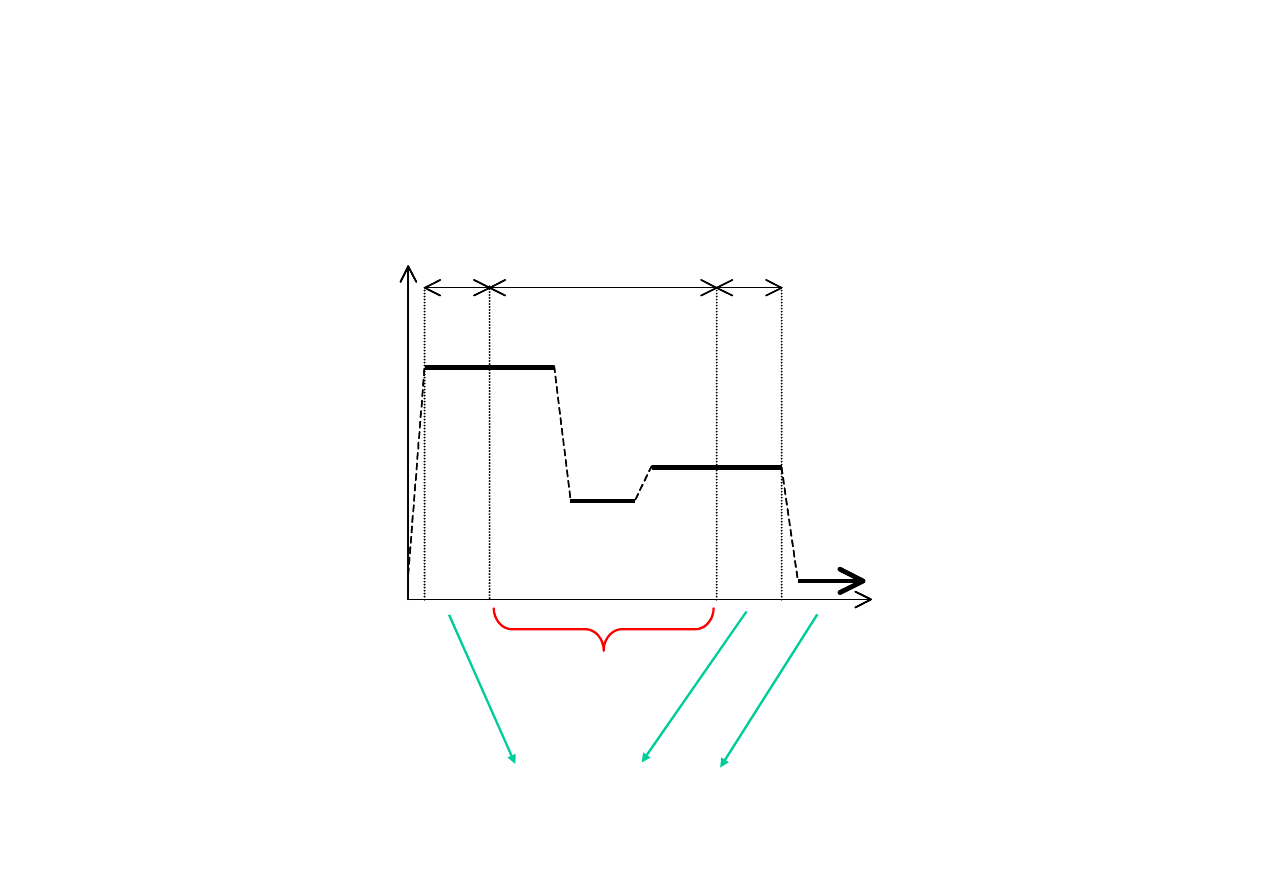
94°C
15s
94°C
3 min
te
m
p
er
at
u
ra
15x
1x
1x
94°C
15s
94°C
3 min
te
m
p
er
at
u
ra
15x
1x
1x
Profil termiczny reakcji PCR
65°C
15s
72°C
30s
czas
72°C
30s
4°C
±∞
65°C
15s
72°C
30s
czas
72°C
30s
4°C
±∞
cykl
podstawowy
cykle dodatkowe
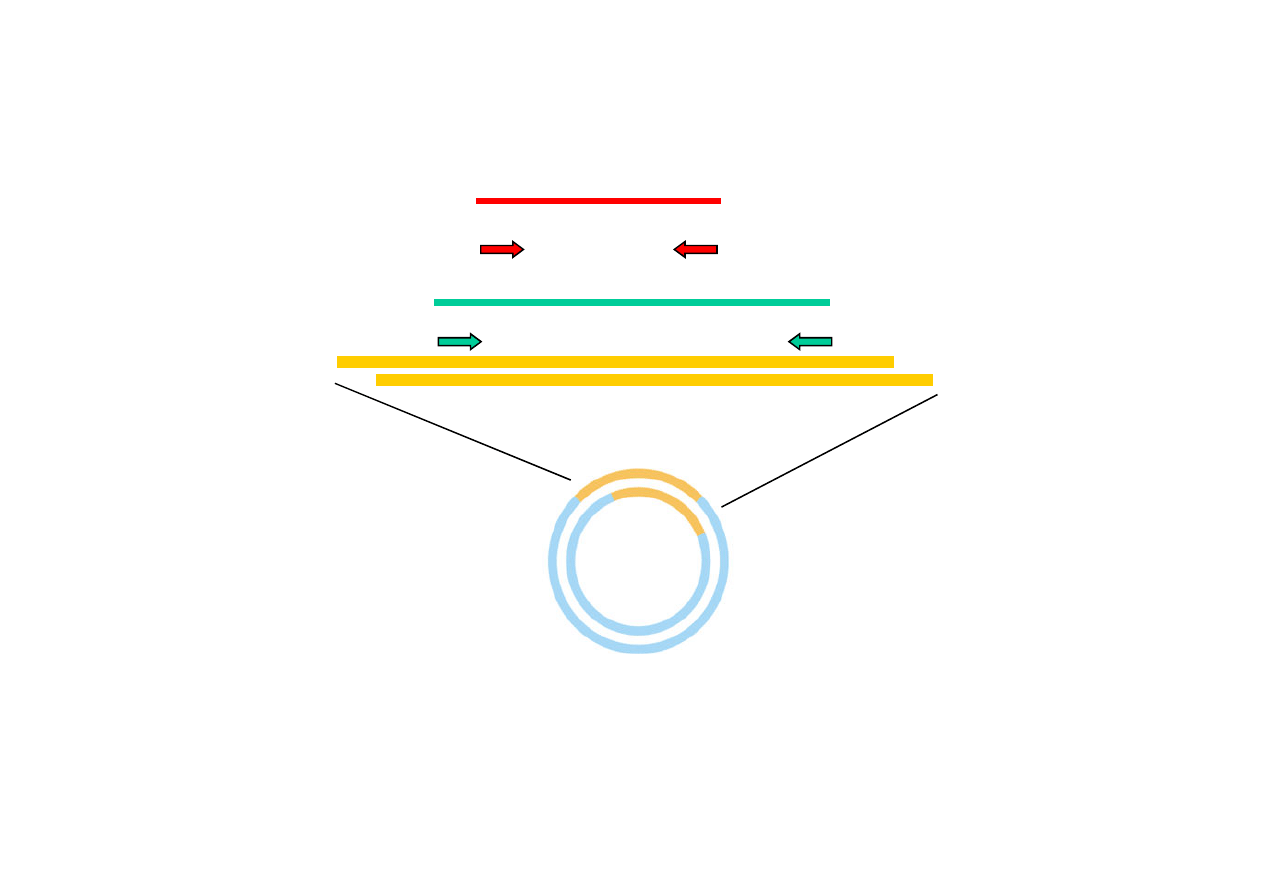
A1
A2
B2
B1
0,6 kb
0,35 kb
Część praktyczna
plazmid pUC18/cDNAPPRas2
ze wstawką 0,8 kp
mieszanina A:
startery A1 i A2
mieszanina B:
startery B1 i B2
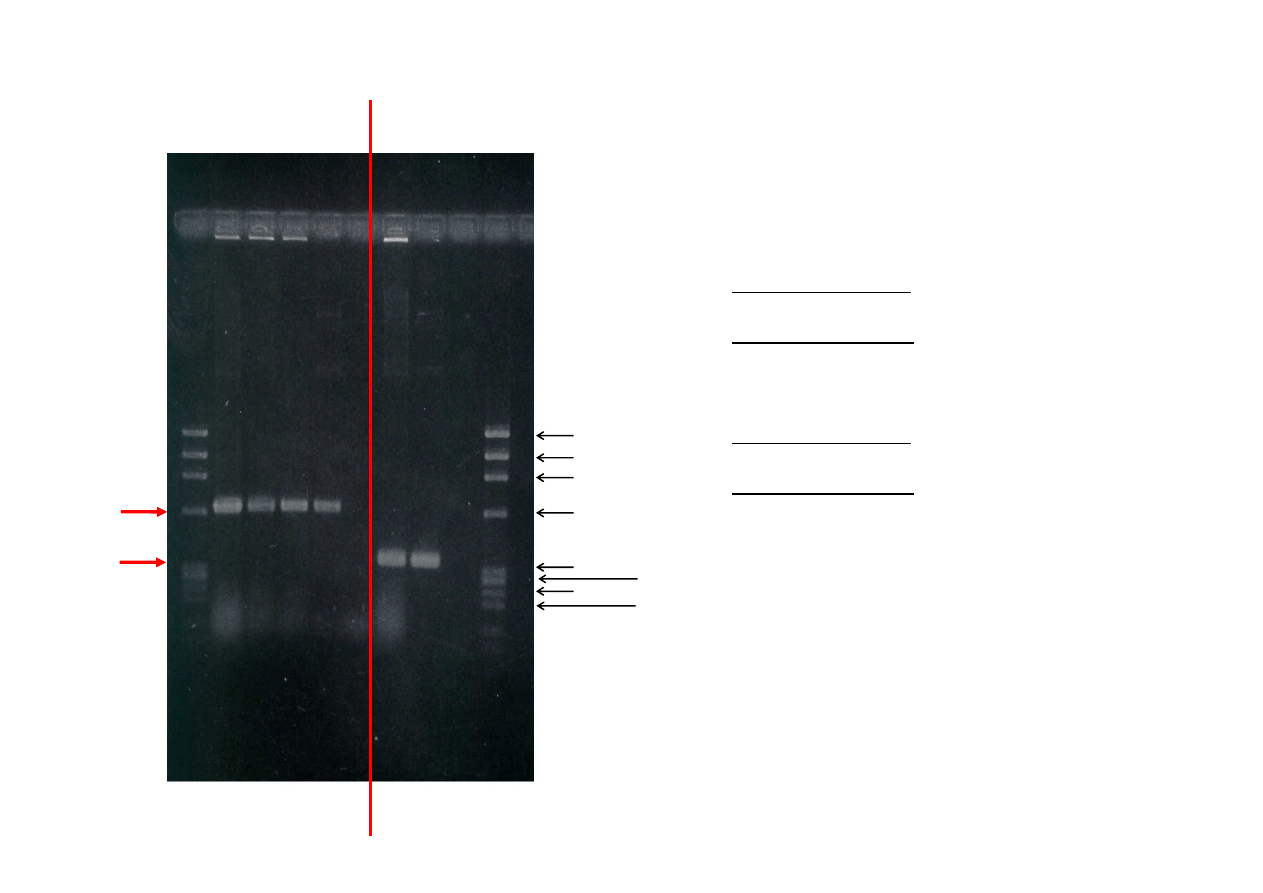
1 - wzorce wielkości
2, 3, 4 - produkty reakcji A, w której jako matrycy
użyto lizatu bakterii)
5 - kontrola pozytywna, czyli produkty reakcji A,
w której jako matrycy użyto czystego plazmidu
6 - kontrola negatywna, czyli produkty reakcji A,
do której nie dodano żadnej matrycy
7 - produkty reakcji B, w której jako matrycy
użyto lizatu bakterii)
8 - kontrola pozytywna, czyli produkty reakcji B,
1 2 3 4 5 6 7 8 9 10
1,35 kb
A
B
w której jako matrycy użyto czystego plazmidu
9 - kontrola negatywna, czyli produkty reakcji B,
do której nie dodano żadnej matrycy
10 - wzorce wielkości
1,08 kb
0,87 kb
0,6 kb
0,28 kb
0,23 kb
0,31kb
0,6 kb
0,35 kb
0,19 kb
Wyszukiwarka
Podobne podstrony:
nukleotydy i kwasy nukleinowe
Nukleotydy i kwasy nukleinowe
kwasy nukleinowe
13 Kwasy nukleinowe
wpływ leków na kwasy nukleinowe
kwasy nukleinowe
Biochmia 12 Kwasy nukleinowe
BW13 KWASY NUKLEINOWE id 95709 Nieznany
pkt1 kwasy nukleinowe-biochemia, Biochemia, Zagadnienia na kolokwia
kwasy nuklein
sprawozdanie kwasy nukleinowe
wszystko wyjście kwasy nukleinowe
kwasy nukleinowe opracowanie
Kwasy nukleinowe
KWASY NUKLEINOWE I STRUKTURA
więcej podobnych podstron