
2. Technika PCR
Skład mieszaniny:
- bufor reakcyjny ( zawierający min. MgCl2 )
- mieszanina primerów
- trójfosforany dNTP
(adeniny, cytozyny, guaniny i tyminy)
- termostabilna polimeraza DNA Taq
- (Thermophilus aquaticus)
- próbka matrycowego DNA

1
. Sekwencje cp DNA
•
rbcL
- gen o długości ~1500 bp
kodujący
•
dużą podjednostkę karboksylazy
•
rybulozo-1,5-bifosforanu (RUBISCO ),
•
takich jak rodzina, rząd czy klasa
•
kluczowego enzymu w fotosyntezie;
•
ze względu na pełnioną funkcję gen
•
ten jest niezwykle silnie
konserwowany
•
ewolucyjnie; używany jest w
filogenezie
•
na wyższych poziomach
taksonomicznych,

2. Sekwencje jądrowe
• nrDNA
- nrDNA stanowią duże rodzinę genów
•
jądrowych kodujących rybosomalne białka oraz
•
rRNA (budujące podjednostki rybosomu); najczęściej
•
używanym markerem nrDNA są regiony ITS
•
(Internal Transcribed Spacer, wewnętrzny
•
transkrybowany przerywnik); są to dwie sekwencje
•
niekodujące rozdzielające geny dla małej i dużej
•
podjednostki rybosomu; transkrypty ITS biorą udział
•
w dojrzewaniu rybosomu, nie są jednak włączane w
•
jego strukturę; brak znaczącej funkcjonalności
•
warunkuje szybkie tempo ewolucji tych sekwencji
•
przezco używane są one w filogenezie na niskich
•
poziomach taksonomicznych, takich jak rodzaj,
•
gatunek a nawet populacja.
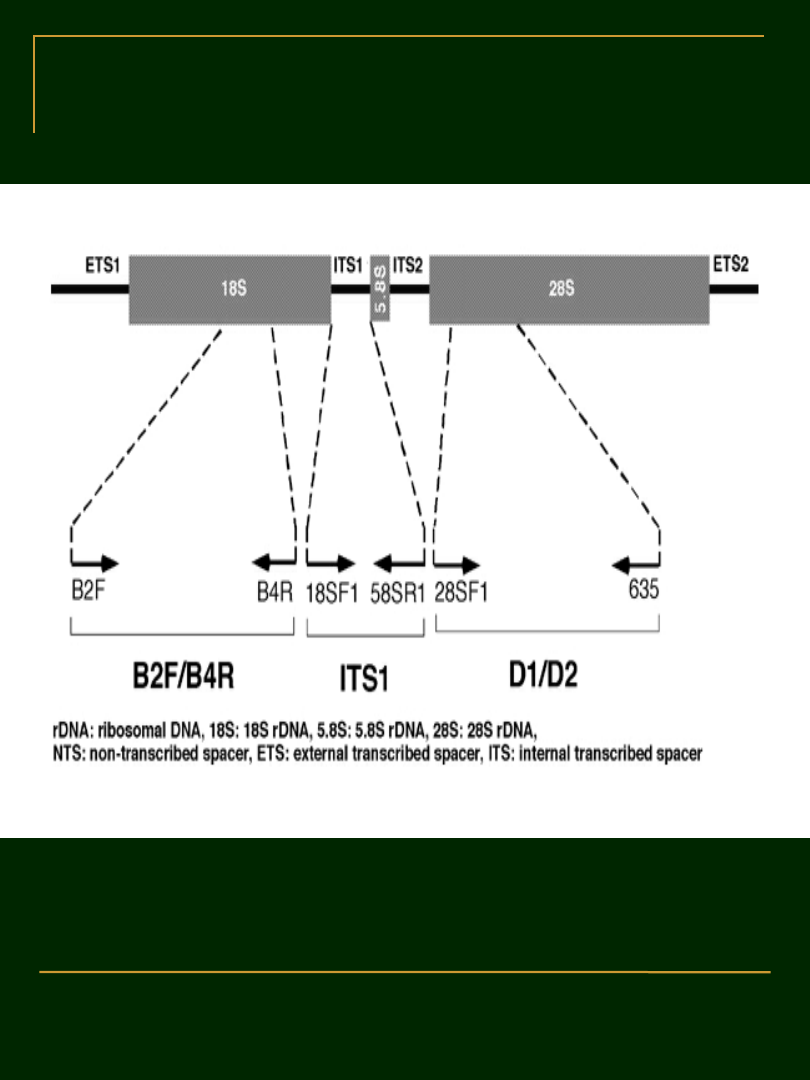
ryc. jednostka rDNA
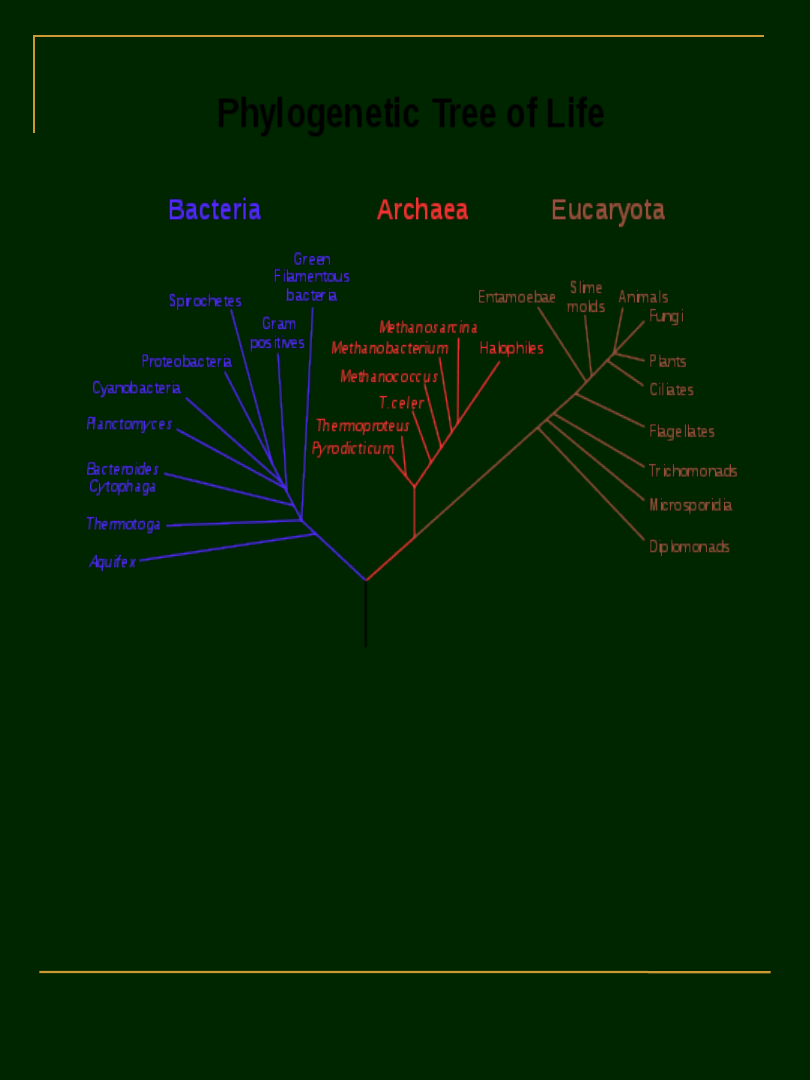
The molecular "Tree of Life" consists of three
domains derived from 16S rDNA genetic data. 16S
rDNA is the gene that codes for ribosomal RNA, a key
part of cellular reproduction. Eukarya includes plants,
animals, and fungi.
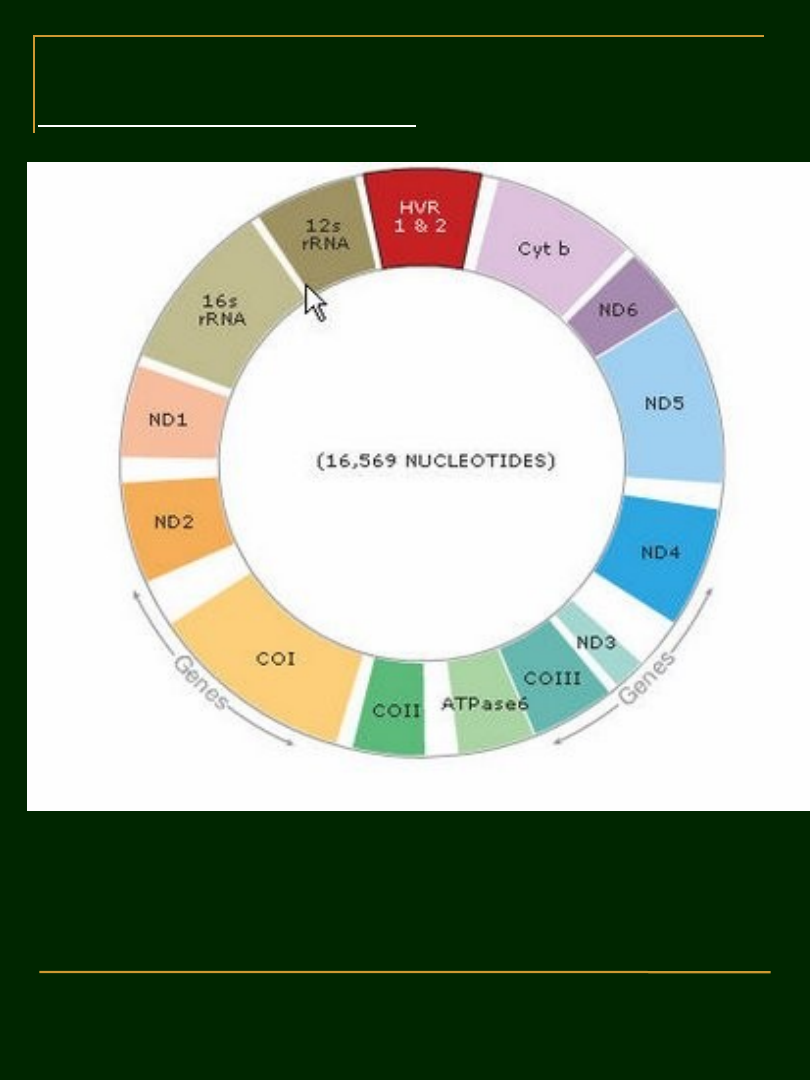
3. Sekwencje mtDNA
• geny mtDNA były dotychczas szerzej
• wykorzystywane w systematyce zwierząt,
• niemniej genom mitochondrialny roślin
• również zawiera sekwencje potencjalnie
• użyteczne w badaniach filogenetycznych;
• dwie dotychczas poznane to
coxl
i
atpA
• należące do genów konserwatywnych;
• ich bardzo wolne tempo ewolucji pozwala
• na wykorzystanie w badaniach na poziomach
• rodziny, rzędu czy klasy.
Palmer JD, Herbon LA.
J Mol Evol. 1988 Dec-1989 Feb;28(1-2):87-97.

Uproszczony schemat procesu badawczego z
zastosowaniem reakcji PCR
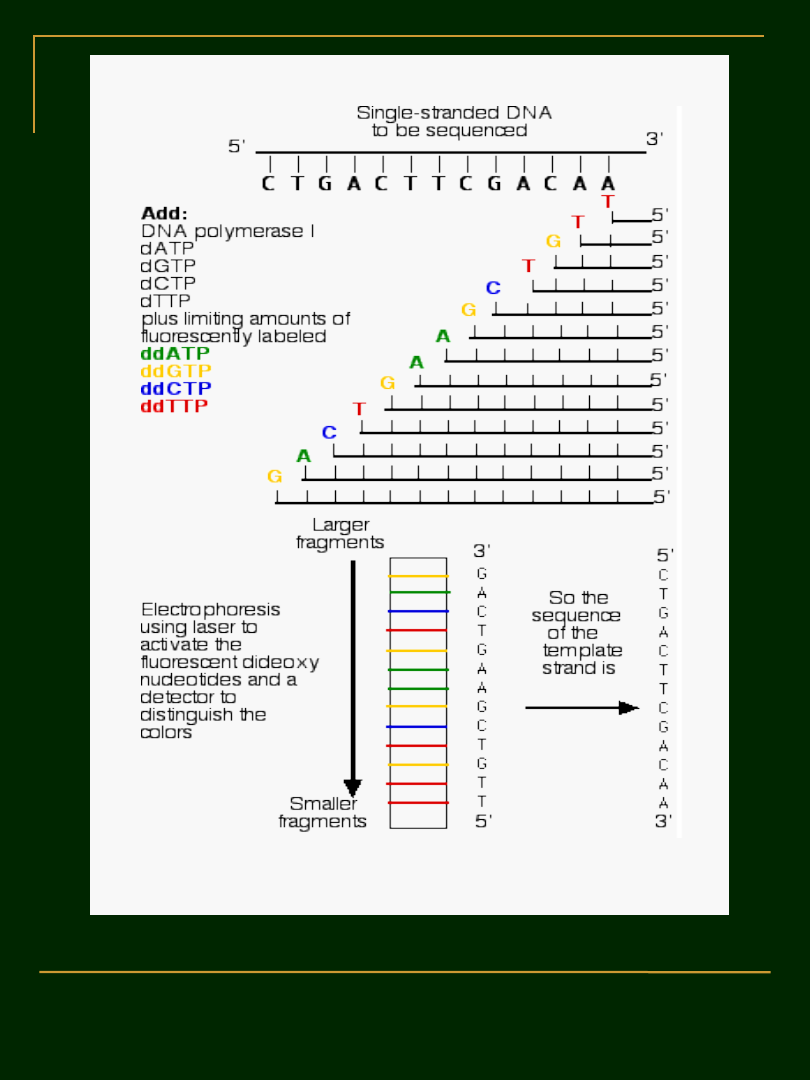

Dopasowanie – PAUP, Clustal X,
Seaview
A
naliza
filogenetyczna
W analizie filogenetycznej wykorzystuje się zestawienie
sekwencji dopasowanych (multiple sequence alignment).
Poszczególne pozycje dopasowania są określane jako
miejsca (site), które są odpowiednikiem cech, natomiast
zasada zajmująca dane miejsce jest zwana stanem cechy
(character state). Aby właściwie oszacować stopień
pokrewieństwa
należy
porównać
ze
sobą
cechy
homologiczne. W tym celu w procesie dopasowania
sekwencji wstawia się przerwy (gaps), które są
odpowiednikiem insercji lub delecji (tzw. indeli, indels).
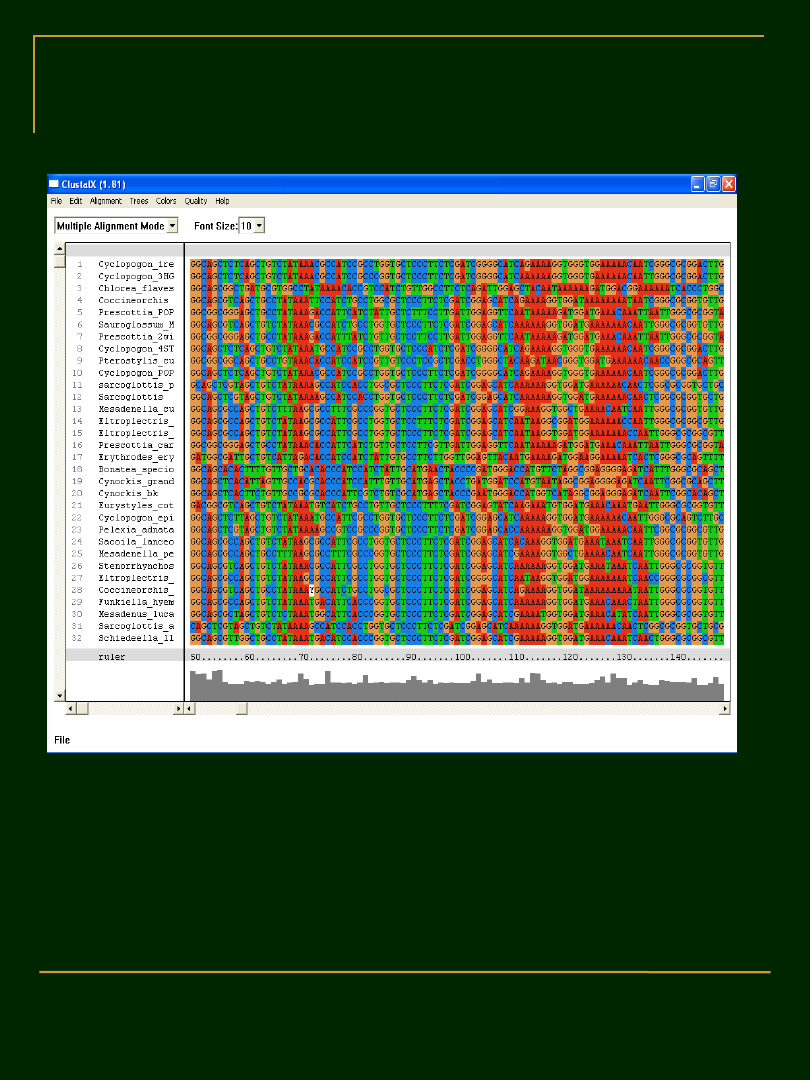
Sekwencje DNA przed dopasowaniem (alignment)
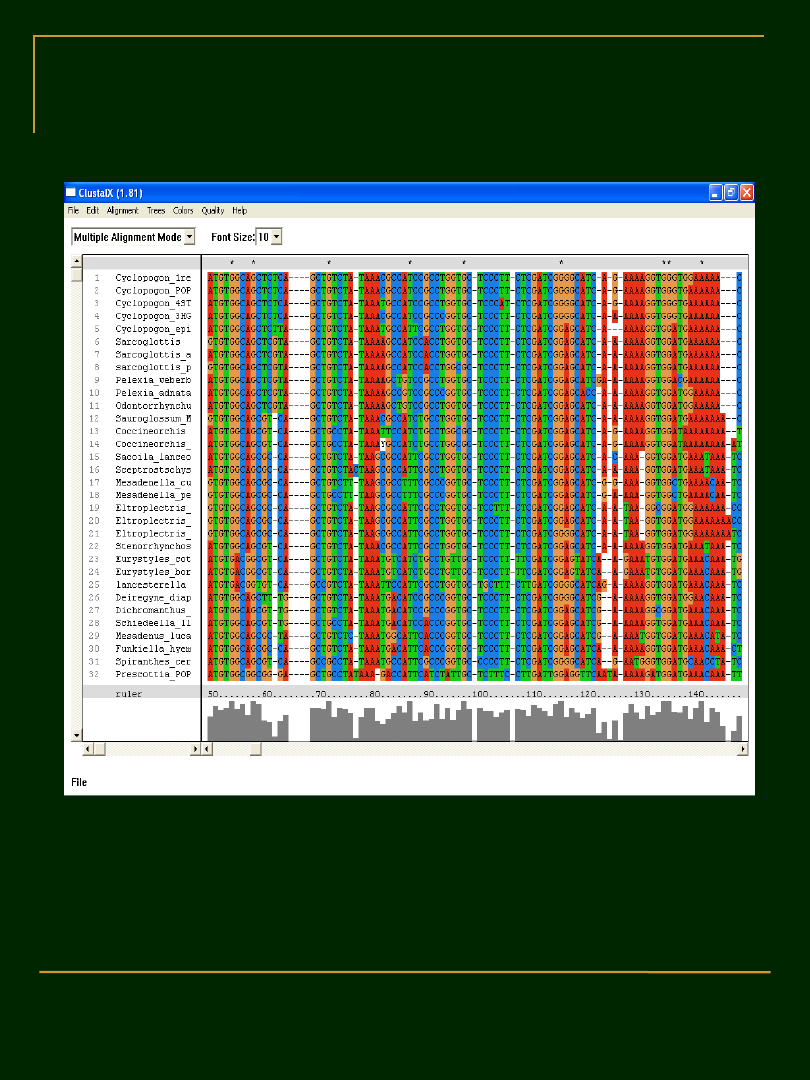
Sekwencje DNA po dopasowaniu (alignment)

Analiza
Filogenetyczna
Model substytucji DNA –
Modeltest 3.7
Konstrukcja drzewa –
PAUP i
MrBayes
Metodę parsymonii
Największej wiarygodności
Analiza bayesowska
Metoda minimalnych odległości
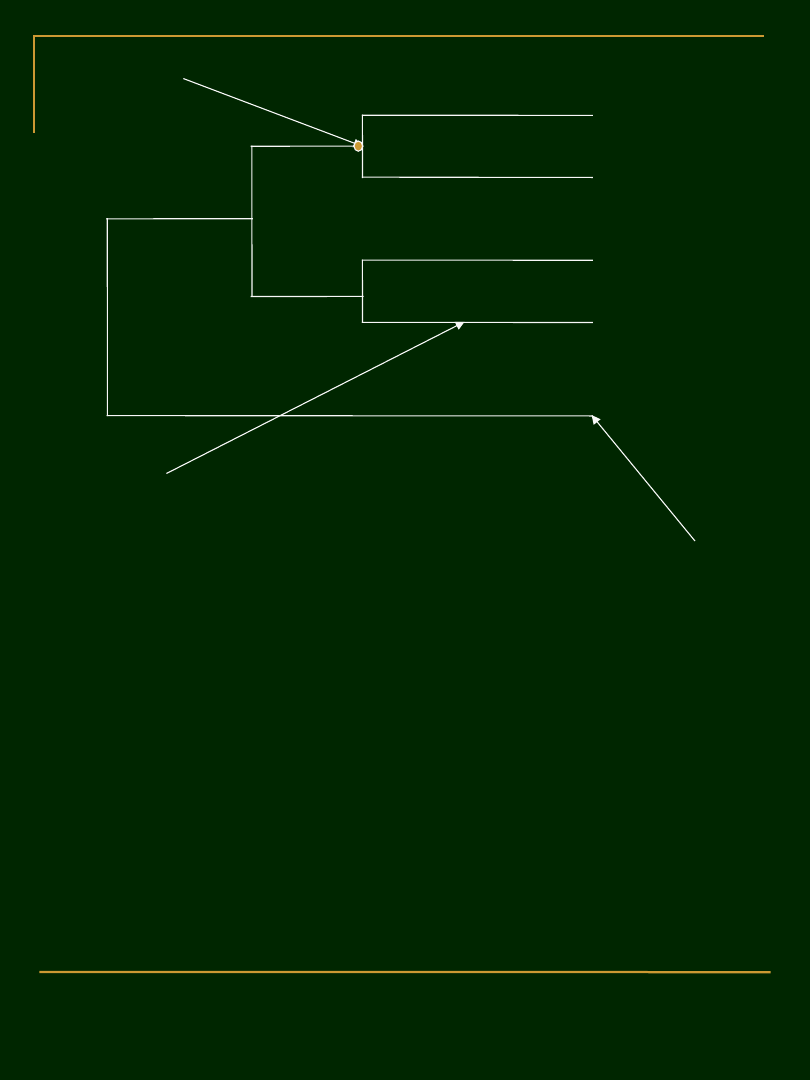
Ryc. Sposób obrazowania powiązań filogenetycznych w
postaci niezakorzenionego drzewa reprezentowanego
przez pięć taksonów (A-E).
A
B
C
D
E
Gałąź
Węzeł wewnętrzny
Węzeł
zewnętrzny
Związki wynikające z analizy filogenetycznej są najczęściej
przedstawiane w postaci drzewa filogenetycznego składającego
z gałęzi, przedzielonych i zakończonych węzłami. Węzły na
szczytach gałęzi reprezentują analizowane taksony, natomiast
wewnętrzne węzły są miejscem rozwidlania gałęzi i odpowiadają
hipotetycznym przodkom. W przypadku molekularnej analizy
filogenetycznej węzły odpowiadają sekwencjom kwasów
nukleinowych lub białek.
Cyclopogon lindleyanus
Cyclopogon pamii
Cyclopogon
sp. 28 07
Eltroplectris 28 07
Pelexia nigrescens
Odontorrhynchus variabilis
Sauroglossum aurantiacum
Sarcoglottis neglecta
Sauroglossum elatum
Coccineorchis sp.
Mesadenella cuspidata
Eltroplectris roseoalba
Skeptrostachys sp.
Mesadenella 28.07
Stenorrhynchos speciosum
Burnsbaloghia diaphana
Stenorrhynchos aurantiacus
Schiedeella llaveana
Mesadenus lucayanus
Spiranthes cernua
Funkiella hyemalis
Lankesterella gnomus
Lankesterella sp.
Lankesterella orthantha
Eurystyles 8215
Eurystyles s.n.
Eurystyles sp.
Eurystyles cotyledon
Prescottia tubulosa
Coccineorchis sp.07
Cranichis ciliilabia
Altensteinia fimbriata
Manniella gustavi
Aspidogyne pumila
Pachyplectron arifolium
Pterostylis curta
Chloraea flavescens
Cynorkis grandiflora
Benthamia latifolia
Coleoglossum viride
2
6
20
6
3
9
1
5
1
2
14
4
3
17
7
1
5
1
14
8
7
10
13
3
19
3
2
3
3
2
4
0
9
7
6
13
5
2
2
3
5
3
10
10
18
21
18
28
12
2
0
14
33
2
3
0
1
2
3
32
9
22
31
15
18
39
18
50
42
25
8
33
46
7
20
27
19
Cyclopogoninae
Spiranthinae
Stenorrhynchidinae
Prescottinae
Manniellinae
Pachyplectroninae
Cranichideae
Goodyereae
Thelymitroideae
Orchidoideae
91
100
59
91
63
58
64
70
70
100
100
86
93
100
66
87
86
79
97
71
60
68
80
99
100
71
99
85
59
100
100
matK
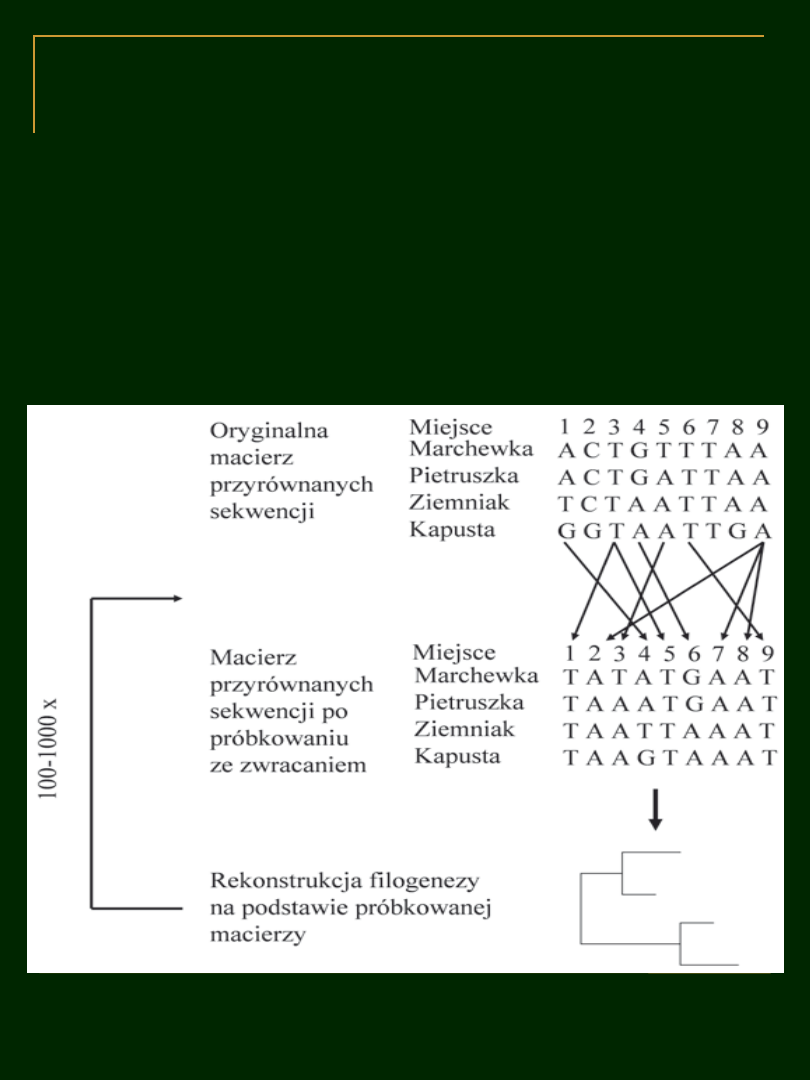
OCENA WIARYGODNOŚCI DRZEWA
Ostatnim etapem każdej analizy jest ocena wiarygodności
otrzymanych
wyników,
która
jest
miarą
prawdopodobieństwa, że taksony danego kladu zawsze
do niego należą. Istnieje kilka metod oceniających
wiarygodność poszczególnych kladów na drzewie, jednak
najpowszechniej używane to testy oceniające drzewo na
podstawie
ponownie
pobieranych
próbek
z
obserwowanych danych (m.in. metoda nieparametryczna
bootstrap)
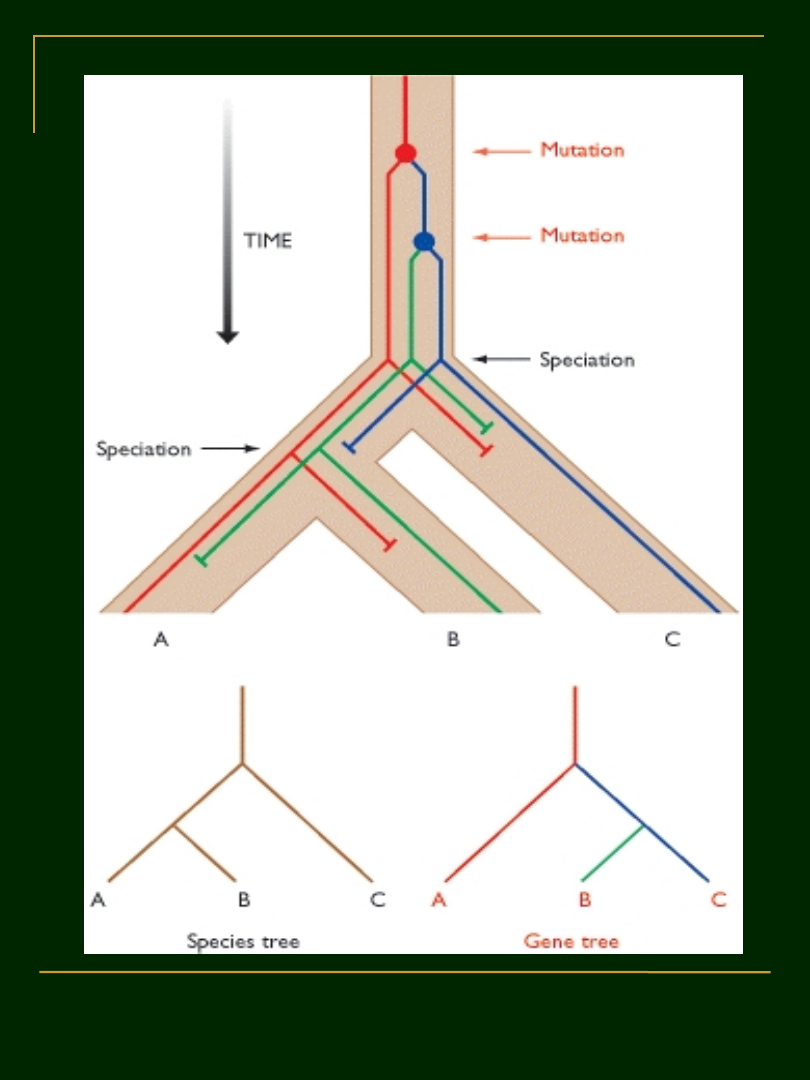
Drzewo gatunku a drzewo
genu
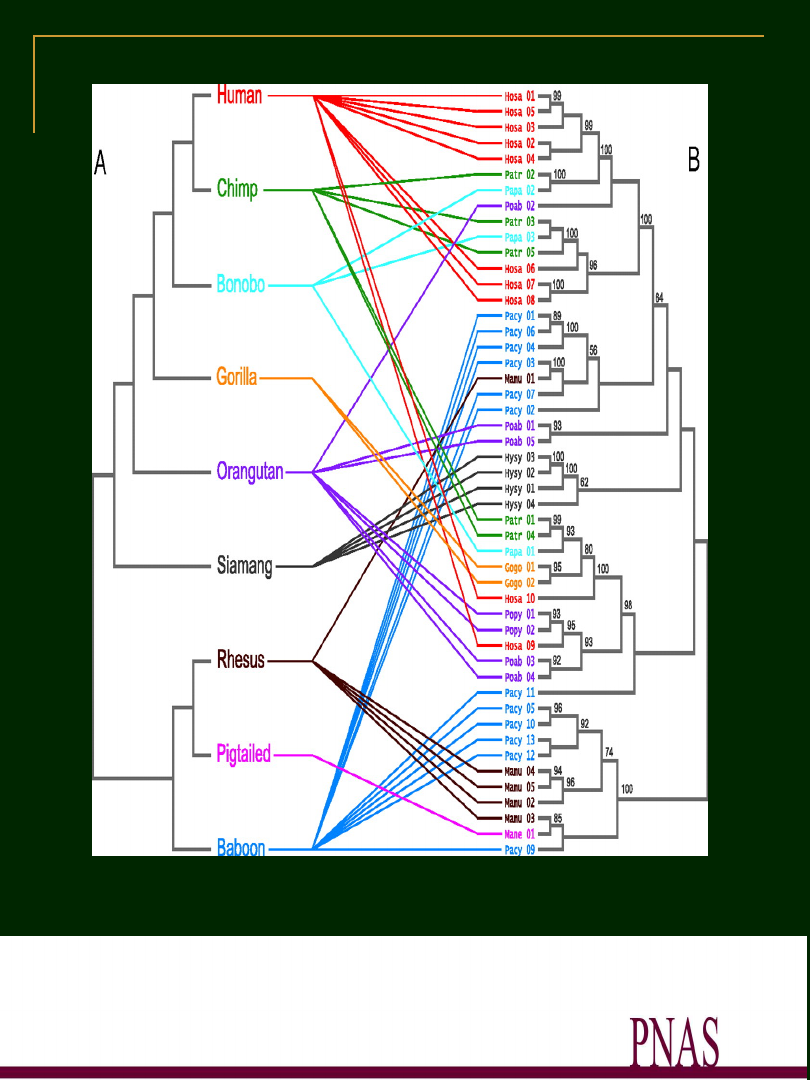
Incongruity of primate species tree and DQA1
promoter region gene tree.
Loisel D A et al. PNAS
2006;103:16331-16336
©2006 by National Academy of Sciences
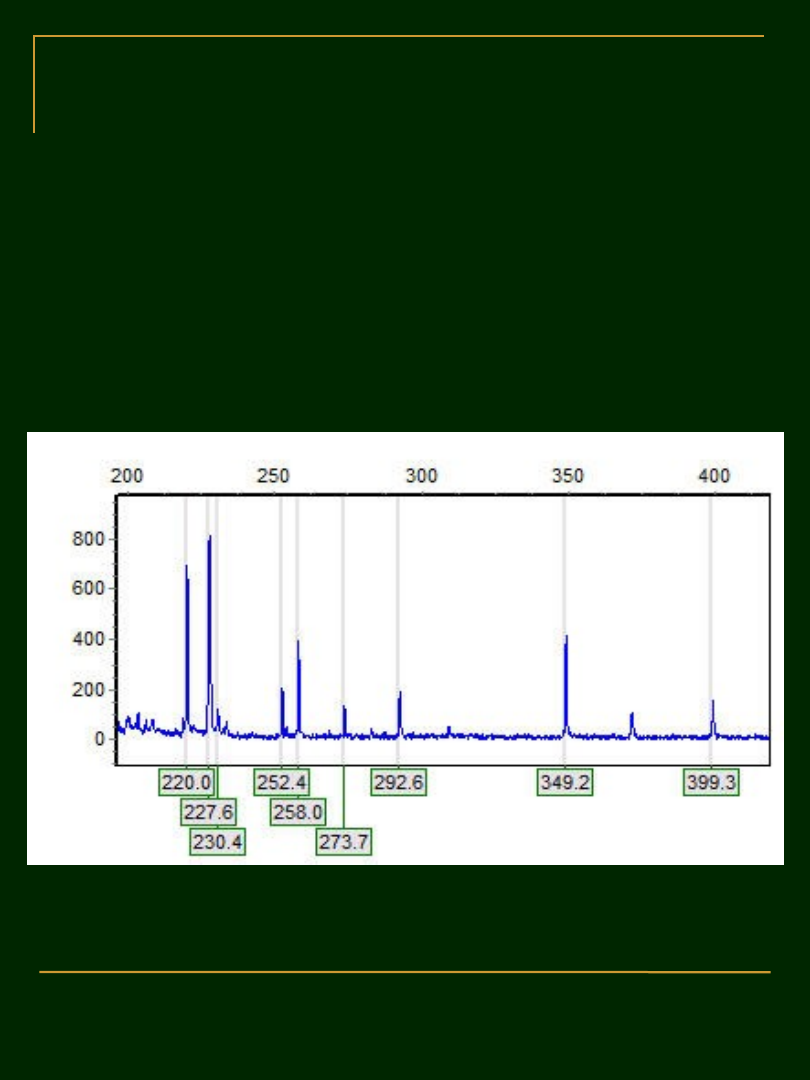
AFLP
– AMPLIFIED FRAGMENTS OF
LENGTH POLYMORPHISM
Polimorfizm długości fragmentów
amplifikowanych
METODY OPARTE NA REAKCJI PCR
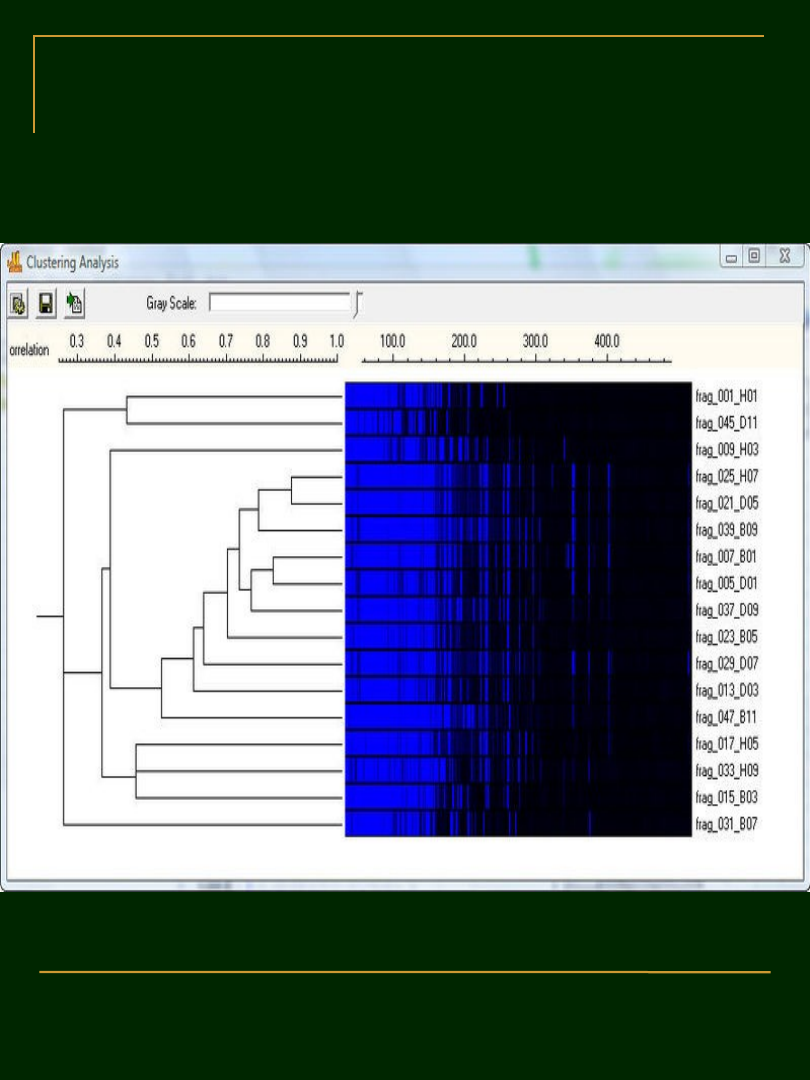
ANALIZA AFLP

(mikrosatelity lub SSR –
Simple Sequence Repeats)
sekwencje zawierające od
10 – 50 powtórzeń motywu o długości
do 6 par zasad. ...
Jednonukleotydowe SSR (A)8
AAAAAAAA
Dwunukleotydowe SSR (GT)6 GTGTGTGTGTGT
Trzynukleotydowe SSR (CTG)4 CTGCTGCTGCTG
Tetranukleotydowe SSR (ACTC)4
ACTCACTCACTCACTC

–
Homozygotyczne
…
CGTAGCCTTGCATCCTT
CTCTCTCTCTCTCT
ATCGGTACTACGT
GG…
…
CGTAGCCTTGCATCCTT
CTCTCTCTCTCTCT
ATCGGTACTACGT
GG…
5’ flanking region microsatellite locus
3’ flanking region
–
Heterozygotyczne
…CGTAGCCTTGCATCCTT
CTCTCTCTCTCTCT
ATCGGTACTACGTGG…
…
CGTAGCCTTGCATCCTT
CTCTCTCTCTCTCTCTCT
ATCGGTACTACGTG
G…
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
Wyszukiwarka
Podobne podstrony:
Wykłady Medycyna Molekularna I, Farmacja, III rok farmacji, Biologia molekularna
BOTANIKA FARMACEUTYCZNA WYKLAD 1 id 92256 (2)
WYKŁAD Z BIOLOGI MOLEKULARNEJ
Vanilla planifolia, Farmacja, Botanika farmaceutyczna
wykład 4 - 23.10.2008, FARMACJA, ROK 5, TPL 3, Zachomikowane
wykład 2 - 09.10.2008, FARMACJA, ROK 5, TPL 3, Zachomikowane
anat.roślin, Farmacja, Botanika farmaceutyczna
EGZAMIN 2010, farmacja, BOTANIKA FARMACEUTYCZNA, Egzamin
WYKŁADY Biologia Molekularna 14
wykład 8 - 19.02.2008, FARMACJA, ROK 5, TPL 3, Zachomikowane
wyklad inzynieria molekularna
opisy roślin, Farmacja, Botanika farmaceutyczna
Botanika, farmacja, BOTANIKA FARMACEUTYCZNA, Koła
więcej podobnych podstron