Łańcuch oddechowy
Łańcuch przenośników elektronów zlokalizowanych w obrębie wewnętrznej błony mitochondrialnej, zbudowany z szeregu białek stanowiących kolejne przenośniki elektronów. Element umożliwiający przeprowadzenie fosforylacji oksydacyjnej. Układ przenośników elektronów w mitochondrium składa się z :
FMN i FAD
ubichinon
NAD i NADP
W oddychaniu komórkowym uczestniczą NAD - dwunukleotyd nikotynamido-adeninowy, NADP - fosforan dwunukleotydu nikotynamido-adeninowego i FAD - dwunukleotyd flawino-adeninowy. Dwoma pierwotnymi akceptorami są NAD i NADP. Grupą czynną obu nukleotydów jest niacyna (witamina PP). Pierścień niacyny przyjmuje od cząsteczki podlegającej odwodorowaniu dwa jony wodoru oraz dwa elektrony i uwalniając jeden proton staje się formą zredukowaną - NADH. Trzecim pierwotnym akceptorem wodoru jest FAD. W niektórych reakcjach zamiast FAD może działać mononukleotyd flawinowy (FMN, ryboflawino-5'-fosforan). NAD po dodaniu kolejnej grupy fosforanowej -[P] powstaje NADP. Redukcja NAD do NADH
cytochromy b,c1, c, a, a1 - hemoproteidy z centralnie położonym atomem żelaza. Najlepiej poznany został cytochrom c. Jest to białko o masie cząsteczkowej 13000; zawiera barwnik zwany ferroporfiryną.
Para elektronów, przepływając przez układ przenośników, powoduje "przerzucenie" przez błonę 3 par protonów. Powrót tych ostatnich spowoduje powstanie 2 (z FADH2) lub 3 (z NADH2) cząstek ATP.
Budowa mitochondrium :
Kompartment zewnętrzny - element mitochondrium zawarty między błoną zewnętrzną i wewnętrzną. Przez przeniesienie wodoru z NADH2 na FMN powstaje FMNH2 i uwalnia się NAD. Na FMNH2 dochodzi do rozdzielenia ładunków : 2 H+ zostają przerzucone przez błonę do k.z., 2 e- przekazane są na ubichinon, w tym momencie staje się hydrofilny i "wynurza" się na powierzchnię błony (dąży do matriks). Pobiera wówczas protony wodorowe ze środowiska - przechodzi poprzez semichinon do hydrochinonu. Przechodzi do wewnątrz błony, gdzie następuje przerzucenie 2 H+ do k.z. i po jednym e- trafia na kolejne przenośniki : cytochrom b --> cytochrom c1 --> cytochrom c - położony w zewnętrznej części błony, co prowadzi do przeniesienia H+ z matriks do k.z. -->cytochrom a -->cytochrom a3 (powrót do matriks). W k.z. gromadzi się ładunek dodatni - różnica potencjałów elektrycznych sięga 140 mV, a różnica pH 1,5. Protony dążą do powrotu do matriksu w celu osiągnięcia równowagi - jedyna droga przepływu wiedzie przez ATP-azę (w oksysomach). Przepływ pary protonów powoduje syntezę 1 ATP (z ADP i fosforanu).
Syntaza ATP
ATP Synthase
David S. Goodsell, tłum. Filip Gołębiowski
Syntaza ATP jest jednym z cudów molekularnego świata. Syntaza ATP to enzym, molekularny silnik, pompa jonowa i jeszcze jeden molekularny silnik, wszystko złączone razem w jedną zadziwiającą maszynę. Odgrywa niezastąpioną rolę w naszych komórkach, tworząc większość z ATP używanego do zasilania procesów komórkowych. Mechanizm w jaki wykonuje te zadania jest prawdziwą niespodzianką.
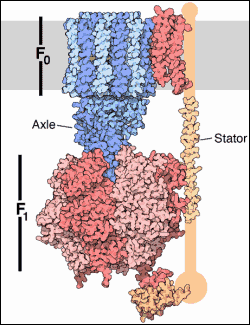
Rotujace motory
W syntezie ATP biorą udział dwa rotujące silniki (motory), każdy zasilany innym paliwem. Ten znajdujący się u góry, nazywany F0, to motor elektryczny. Jest zatopiony w błonie komórkowej (pokazanej tutaj schematycznie jako szary pas) i zasilany przez strumień jonów wodorowych wpoprzek błony. Kołowy rotor (rotująca część silnika - dop.tłum., pokazany na niebiesko) jest obracany kiedy protony przepływają przez motor. Ten rotor jest połączony z drugim motorem, zasilanym przez ATP, określanym jako F1. Motor F1 jest chemicznym silnikiem zasilanym przez ATP. Dwa silniki są połączone przez statecznik (ang. Stator), znajdujący sie po prawej, tak że kiedy obraca się część F0, robi to także F1.
Motor w generator
Więc dlaczego dwa silniki są ze sobą połączone? Sztuczka polega na tym, że jeden silnik może zmusić drugi do obrotu i w ten sposób zmienić go w generator. To właśnie dzieje się w naszych komórkach: silnik F0 wykorzystuje siłę gradientu protonów by zmusić silnik F1 do wytwarzania ATP. W naszych komórkach pożywienie jest rozkładane i wykorzystywane do pompowania jonów wodorowych wpoprzek błony mitochondrialnej. Część F0 syntazay ATP pozwala tym jonom przepływać z powrotem, obracając rotor. Obracający się rotor uruchamia oś (ang. Axle) i silnik F1 staje się generatorem, tworząc ATP podczas obrotów.
Lista części
Duże, skomplikowane molekularne maszyny, jak syntaza ATP, stanowią poważny problem dla biologów strukturalnych, dlatego ich struktury są często wyznaczane w częściach. Obraz pokazany obok jest złożeniem czterech osobnych struktur, łącząc te wyznaczone krystalograficznie i spektroskopią NMR. Silnik F0 jest zawarty w pliku PDB 1c17. Silnik F1 i oś łącząca oba silniki znajduje się w pliku PDB 1e79. Statecznik okazał się być najtrudniejszą do uchwycenia częścią - dwa jego fragmenty pokazane na obrazie pochodzą z plików PDB 2a7u i 1l2p.
Po więcej informacji na temat syntazy ATP z puntu widzenia genomiki zobacz stronę Protein of the Month na stronie European Bioinformatics Institute.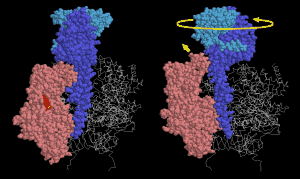
Odkrywanie struktury podjednostki F1
Rekord PDB 1e79 zawiera silnik F1 syntazy ATP. Kiedy działa jak generator, wykorzystuje siłę ruchu rotacyjnego do wytwarzania ATP, a kiedy działa jak silnik (motor) rozkłada ATP by obracać osią w przeciwnym kierunku. Dwoma trudnymi krokami w syntezie ATP są wiązanie ADP i uwalnianie ATP—tworzenie wiązania pomiędzy grupami fosforanowymi nie jest głównym problemem. Podczas obrotu osi, wymusza ona na silniku przyjmowianie trzech odmiennych konformacji, które asystują w powyższych trudnych krokach. Dwa z tych stanów pokazane są na obrazie. Ten po prawej przedstawia konformację, która asystuje podczas wiązania się ADP, a ukazana po prawej pokazuje konformację zmuszoną do otwarcia się by uwolnić ATP. Zauważ jak dziwacznie ukształtowana oś wymusza zmianę konformacji.
Możesz samemu zobaczyć tą strukturę klikając na kod dostępu i wybierając jedną z opcji w panelu 'View Structure'. Środkowa oś w tym pliku składa się z łańcuchów G, H i I. Trzy podjednostki, łańcychy D, E i F w tym pliku, są częściami generującymi ATP - podjednostka E jest pokazana na czerwono po lewej stronie a podjednostka D po prawej stronie. Podjednostki A, B i C są podobne, ale odgrywają strukturalną rolę, utrzymując wszystko na właściwym miejscu. W tym obrazie usunąłem dwie najbliższe podjednostki by prościej ukazać oddziaływanie. 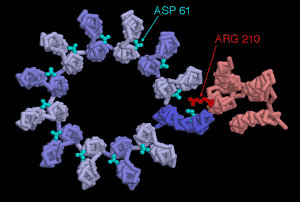
Odkrywanie struktury podjednostki F0
Rekord PDB 1c17 zawiera silnik elektryczny F0. Na tym obrazie, spoglądamy w dół osi obrotu, tak jak byśmy spoglądali w dół z góry pierwszego obrazu. Rotor składa się z 12 identycznych białkowych łańcuchów, tutaj w kolorze niebieskim, zaś pompa jonowa jest pojedynczym łańcuchem, w kolorze czerwonym. Pompa posiada aminokwas argininę, który przekazuje jon wodorowy do asparaginianów w rotorze. Kwas asparaginowy zazwyczaj posiada ujemny ładunek, który jest bardzo niekorzystny w tej sytuacji, ponieważ rotor jest otoczony przez lipidy błony. Dlatego więc rotor obraca się jedynie wtedy, kiedy asparaginiany mają przyłączony atom wodoru neutralizujący ich ładunek. Jony wodorowe przechodzą zawiłą drogę przez silnik F0, uruchamiając podczas tego procesu rotor. Są zbierane przez łańcuch aminokwasów w pompie i przekazywane na argininę. Ten aminokwas przekazuje wodór do rotora, który obraca się dookoła. Potem jony wodorowe są wyładowywane przez inne aminokwasy w pompie i ostatecznie przekazywane na przeciwną stronę błony. Dokładna droga jonów wodorowych poprzez pompę jest ciągle sprawą intensywnych badań.
Te rysunki zostały stworzone programem RasMol. Możesz stworzyć podobne obrazy klikając na kod struktury i wybierając jedną z opcji w panelu 'View Structure'.
Lista wpisów do bazy PDB związanych z syntazą ATP wyznaczona przeszukiwaniem poprzez słowo kluczowe w dniu 1 Grudnia 2005 jest dostępna tutaj. Po więcej informacji na temat syntazy ATP, kliknij tutaj.
Oryginalna podstrona w "Molecule of the Month", by David S. Goodsell: ATP Synthase.
Dodatkowe informacje na temat Syntazy ATP
P. D. Boyer (1997) The ATP Synthase, A Splendid Molecular Machine. Annual Review of Biochemistry 66, 717-749.
G. Oster and H. Wang (1999) ATP Synthase: Two Motors, Two Fuels. Structure 7, R67- R72.
G. Oster and H. Wang (2003) Rotary Protein Motors. Trends in Cell Biology 13, 114- 121.
Wyszukiwarka
Podobne podstrony:
otwarte, ✔ ★Weterynaria, weta, dump
Poprawka, ✔ ★Weterynaria, weta, dump
Teori nadczłowieka, ✔ ★Weterynaria, weta, dump
Mitoza, ✔ ★Weterynaria, weta, dump
Izolacja komórek, ✔ ★Weterynaria, weta, dump
Dokładne odpowiedzi, ✔ ★Weterynaria, weta, dump, biol kom
biologia-WYKLADY wszystkie, ✔ ★Weterynaria, weta, dump
Funkcje mitochondriów, ✔ ★Weterynaria, weta, dump
Genom miochondrialny, ✔ ★Weterynaria, weta, dump
biol-kom, ✔ ★Weterynaria, weta, dump
biolkom ex odp, ✔ ★Weterynaria, weta, dump
kumulacja!V.1.3, ✔ ★Weterynaria, weta, dump, biol kom
BIOLOGIA KOMORKI, ✔ ★Weterynaria, weta, dump, biol kom
więcej podobnych podstron