Markery genetyczne i ich wykorzystanie w hodowli zwierząt (3)

PSS - identyfikacja przyczyny
> Mutacją odpowiedzialną bezpośrednio za wystąpienie objawów PSS okazała się substytucja COT w pozycji 1843 genu receptora rianodyny (RYR1). Należy ona do grupy mutacji zmiany sensu i powoduje zamianę ArgoCys w pozycji 615 łańcucha polipeptydowego. Konsekwencją tej zamiany jest upośledzenie funkcji receptora rianodyny - białka zaangażowanego w transport jonów Ca2* w mięśniach szkieletowych
> Sprawcza mutacja może być wykrywana przy użyciu testu molekularnego RFLP
Technika RFLP
> Technika RFLP polega na restrykcyjnej analizie produktów reakcji PCR
> Obecność polimorfizmu/mutacji skutkuje utworzeniem lub zanikiem miejsca rozpoznawanego przez dany enzym restrykcyjny o trawiąc DNA (produkt PCR) pozyskany od osobnika z mutacją otrzymujemy fragmenty o innej długości niż w wyniku trawienia DNA (produktu PCR) pochodzącego od osobnika bez mutacji. Powstałe fragmenty rozdzielamy w żelu.

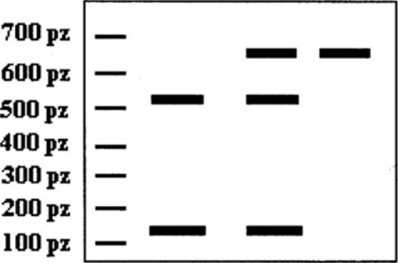
Analiza wyniku w metodzie RFLP.
Od lewej: homozygota niezawierająca miejsca restrykcji, heterozygota, homozygota zawierająca miejsce restrykcji.
Technika RFLP - identyfikacja mutacji w genie RYR1 Sekwencja prawidłowa - trawienie przez enzym restrykcyjny Hhal - produkty trawienia - 524 i 135 pz
5*.....GC
3*.....CG
GC
CG
Sekwencja zawierająca mutację C-T - brak trawienia przez enzym restrykcyjny Hhal - produkt reakcji 659 pz = długość produktu PCR
CC CT TT
Halotanowa grupa sprzężeniowa w chromosomie 6 świni:
GPI - pozycja genu izomerazy fosfoheksozy RYR1 - pozycja genu receptora rianodyny
H - pozycja genów kodujących antygeny układu grupowego krwi H
P02 - pozycja genu enzymu postalbuminy 2
PGD - pozycja genu dehydrogenazy 6-P-glukagonu

GPI

RYRl (H;ili
3
Wyszukiwarka
Podobne podstrony:
Markery genetyczne i ich wykorzystanie w hodowli zwierząt (1) Genetyka zwierząt - ĆwiczeniaMARKĘRY G
Markery genetyczne i ich wykorzystanie w hodowli zwierząt (2) Badania: 1. Polimorf
Scan0006 (34) ZWIERZĘTA MODYFIKOWANE GENETYCZNIE 1. Naukowe wykorzystanie zwierząt
■ Z ŻYCIA UCZELNI Pracownicy Katedry Genetyki i Ogólnej Hodowli Zwierząt przygotowali też warsztaty,
korzyści, wynikających z ich wykorzystania, który będzie regulować te zagadnienia. Zasoby genetyczne
Wstęp Do tej pory postęp w hodowli zwierząt opierał się na wiedzy z zakresu genetyki cech ilościowyc
P1590155 Produkcja i hodowla zwierząt opiera się przede woćci ich rozmnażania. Jeżeli zwierzęta nie
Zakład Genetyki i Podstaw Hodowli Zwierząt12 i 13
Higiena pytania na egzamin (1) Imię i nazwisko specjalność: hodowla zwierząt Data: 1.
10 Poradnik dla nauczycieli technologii informacyjnej od zakresu i stopnia ich wykorzystania, można
więcej podobnych podstron