
Tempo reinicjacji zależy między innymi od tempa
uwalniania RNA pol III z miejsca terminacyjnego.
Odpowiada i reguluje to czynnik białkowy La

Eukariotyczna polimeraza I
Enzym prowadzący 45S pre-rRNA
Wynikiem jego aktywności jest ok. 60%
całkowitej puli jądrowej syntezy RNA
U człowieka występuje ok. 130 aktywnych genów rRNA
Eukariotyczna polimeraza I
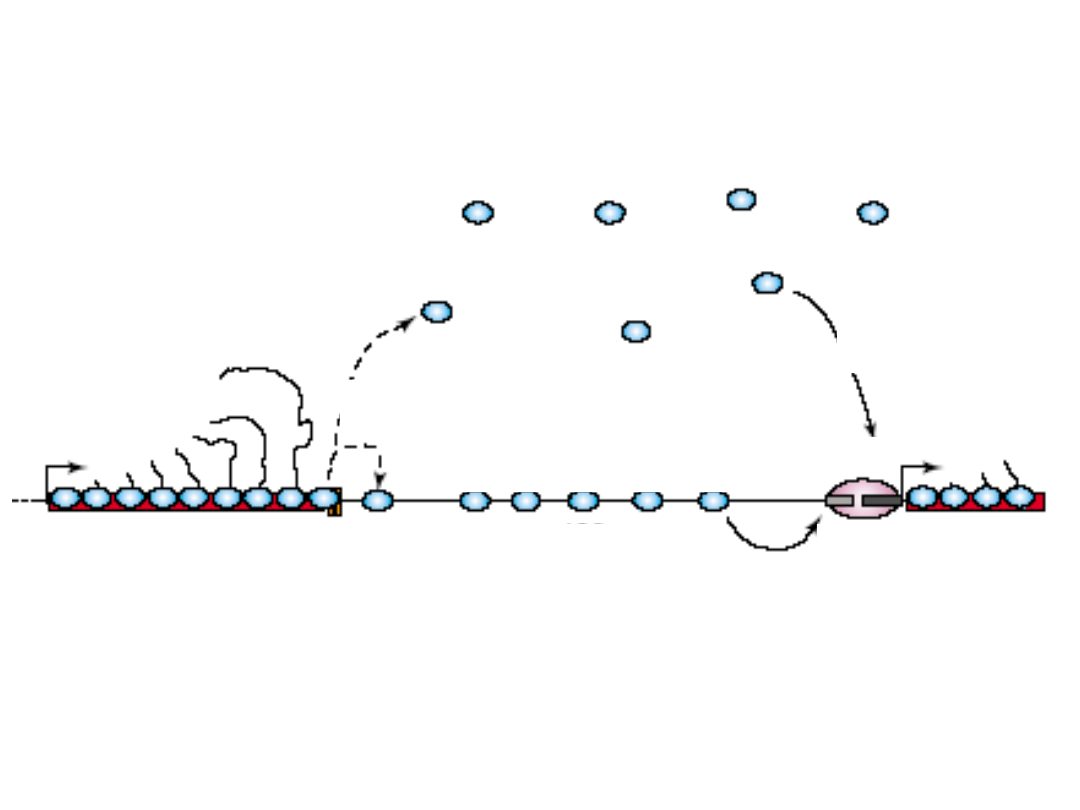
Pre-rRNA
blok transkrypcyjny rDNA
IGS
Promotor
Oddysocjowanie
Pol I i RNA
wolna pula polimerazy I
związana z matrycą
pula polimerazy I
PIK
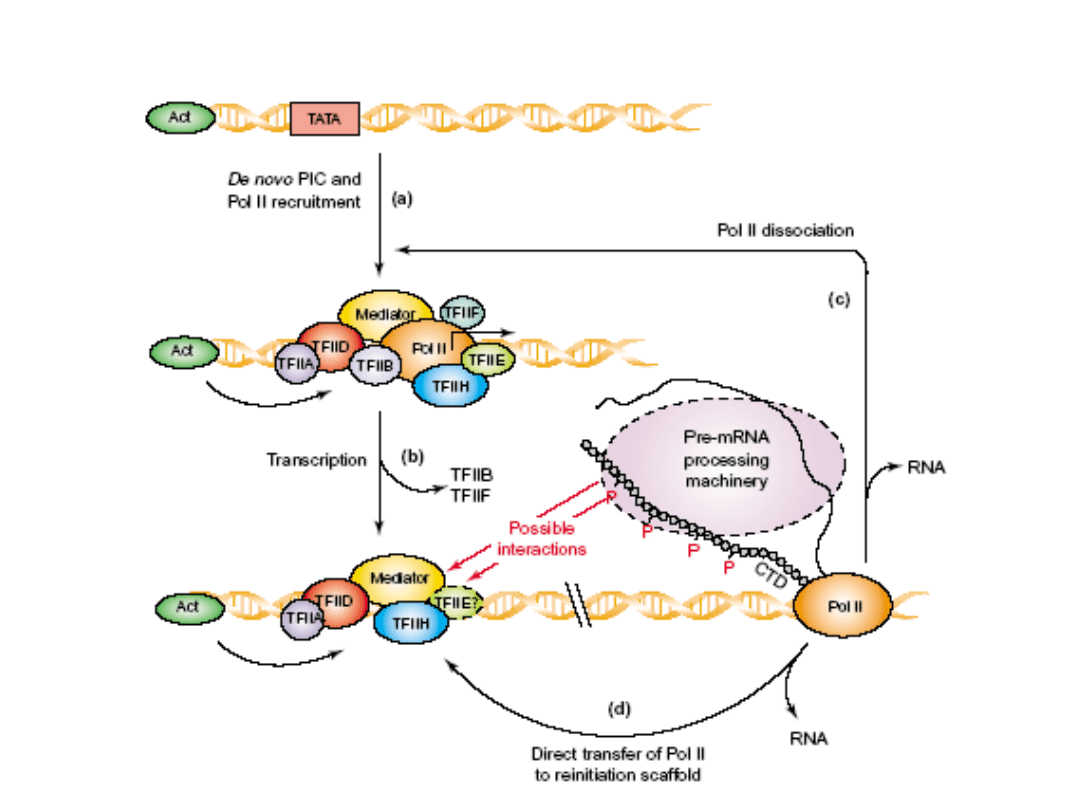
De novo
przyłączenie Pol I
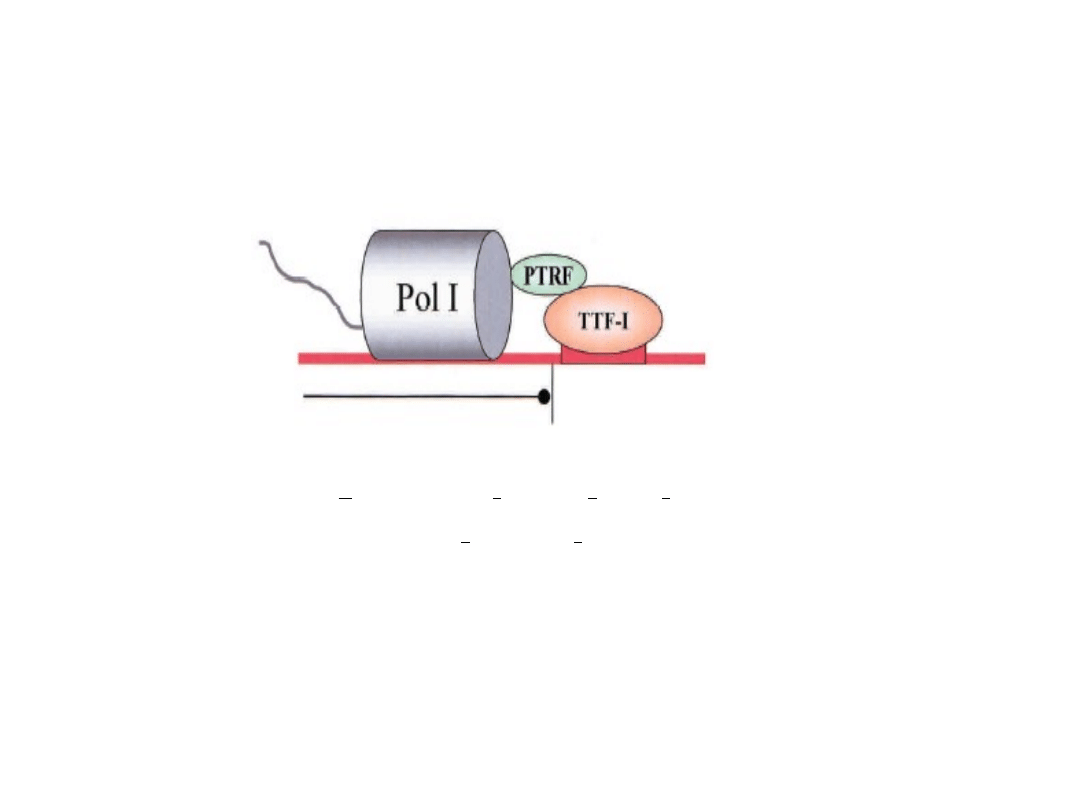
Terminacja transkrypcji
• PTRF (polymerase and transcript release factor)
– czynnik uwalniający polimerazę i transkrypt
• TTF-I (transcription termination factor) czynnik
kończący transkrypcję

Na jednym genie znajduje się jednocześnie
ok. 100-120 Pol I
Tempo syntezy tego enzymu to ok. 40 nukleotydów/s
Inicjacja następuje więc co ok. 3 s

mRNA:
1. geny aktywowane są z różną
częstotliwością od kilku sekund do
kilkunastu godzin
2. rozmiar pre-mRNA: od kilkuset do kilku
milionów par zasad

Co wpływa na tak różny poziom ekspresji (ponad 1000 x) różnych genów?
1. Różnorodność elementów promotorowych.
2. Obecność wielu czynników aktywujących lub
spowalniających sam proces transkrypcji
3. Zjawiska związane z reinicjacją kolejnej rundy transkrypcyjnej
4. Stabilizacja czynników transkrypcyjnych na promotorze.
5. Tempo elongacji (związane z czterema elementami dojrzewania mRNA)
6. Recykling białkowych„ruchomych” elementów systemu: np. polimerazy
Eukariotyczna polimeraza II

Dla genów polimerazy II dodatkowo na tempo reinicjacji
a zatem na poziom ekspresji wpływają takie procesy jak
czapeczkowanie 5’ konca, splicing, a przede wszystkim
poliadenylacja.

Polimeraza bakteryjna
Holoenzym bakteryjnej pol RNA:
Efekt konkurencji czynnika sigma
Czynniki stałe i fakultatywne
Efekt poziomu cz. Sigma na tempo wzrostu bakterii

Regulatory polimeraz:
1. Polimeraza bakteryjna –białko Rap (regulacja tau70)
2. Polimeraza III - białko La
3. Polimeraza I - białko C
4. Polimeraza II - fosforylacja CTD domeny, ubikwitynizacja

Proporcja gen/polimeraza u prokariota i eukariota jest stała !
E. coli ~2600 operonów i ~ 2000 polimeraz
Człowiek ~ 30-40 tyś genów i 40-60 tyś pol II

Mediator i TFIIH obecne w małej ilości kopii ~6000
Czynników „ruchomych” kilka razy
więcej: TFIIB, TFIIF ~ 20 000 kopii
Są w „nadmiarze” i mogą
oddysocjowywać i reasocjować.

80% genów ulega ekspresji na bardzo niskim poziomie: 0.1-1
cząsteczkina komórkę. Gdy zachodzi potrzeba stabilny PIK
pozwala na wysoką ekspresje.
Dla tych genów PIK jest niestabilny i ulega ciągłej dysocjacji i reasocjacji
Alternatywnie – model fabryk transkrypcyjnych: zgrupowanie po
kilka genów i nagromadzenie czynników transkrypcyjnych w
określonej przestrzeni.
Zgrupowanie w genomie różnych genów (średnio ok. 10) o podobnej ekspresji
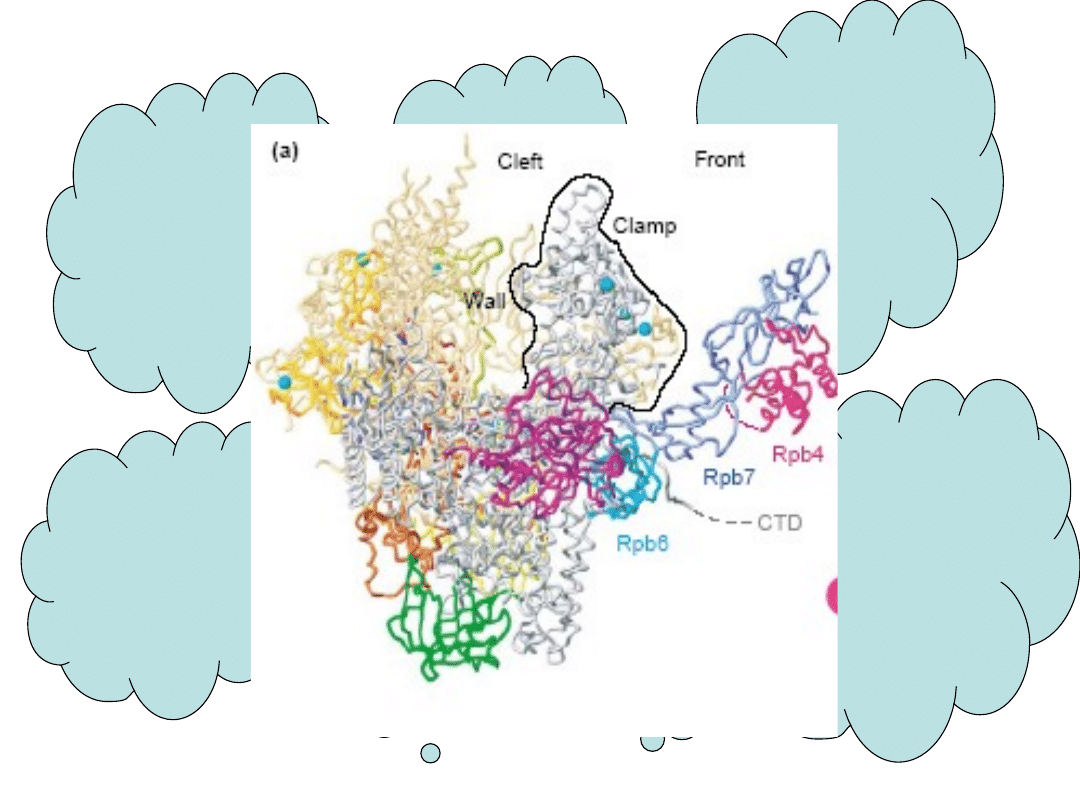
Polimeraza
II:
Kompleks
białkowy
utworzony z
2 dużych
podjednoste
k
RPB1 i
RBP2
i 10
mniejszych
Składa się
z domeny
C-
końcowej
CTD
i
N -
końcowej
Forma aktywna,
hiper-P –
pol II
O
związana z
elongacją
transkrypcji
Forma
nieaktywna,
hypo-P –
pol II
A
związana z
inicjacja
transkrypcji
Stopień
ufosforylow
ania
domeny
CTD
koreluje z
aktywną lub
nieaktywną
jej formą
Domena
CTD
wchodzi w
skład RPB1 i
zawiera
powtórzenia
sekwencji :
Tyr-Ser-Pro-
Thr-Ser-Pro-
Ser
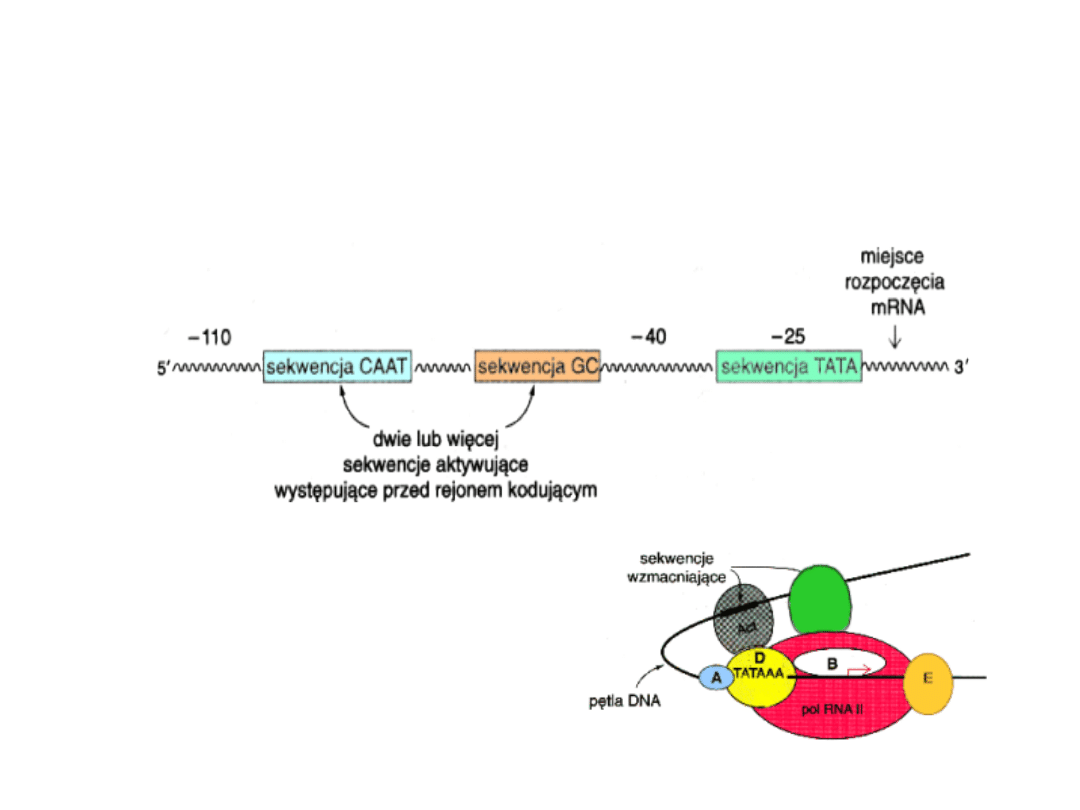
Schemat promotora genu eukariotycznego :
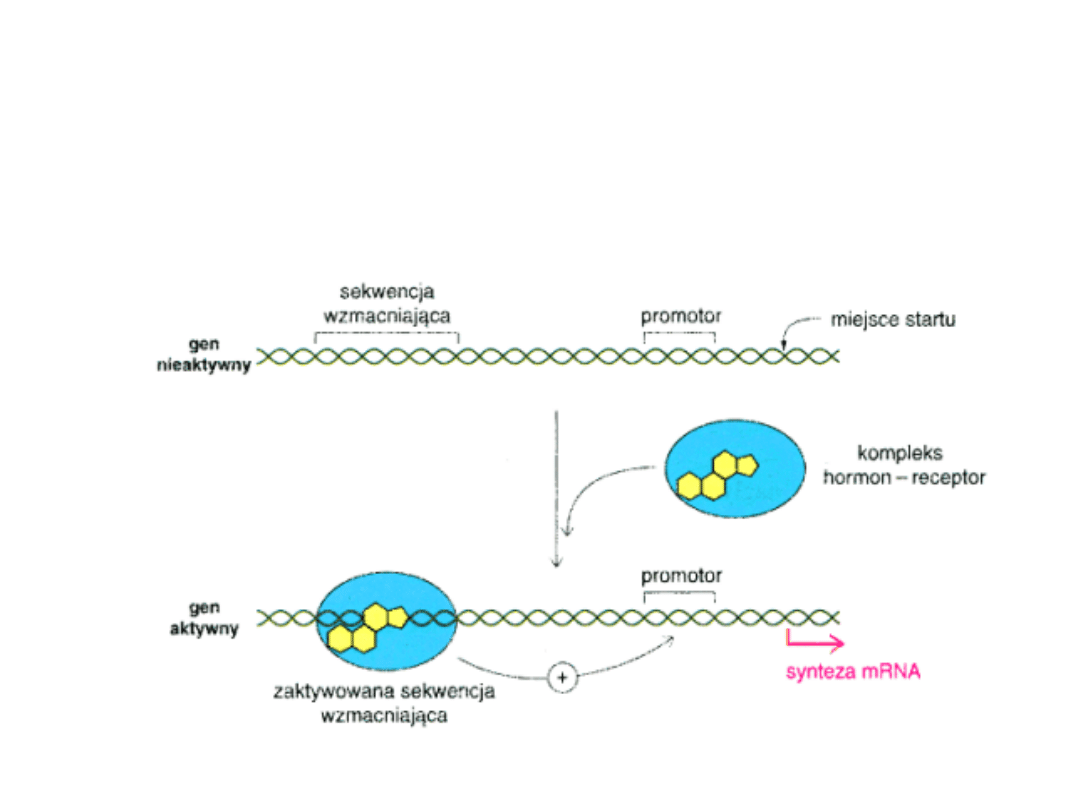
Wiązanie receptora GR do genu :
na przykładzie steroidowy receptor GR <
glukokortykoidowy >
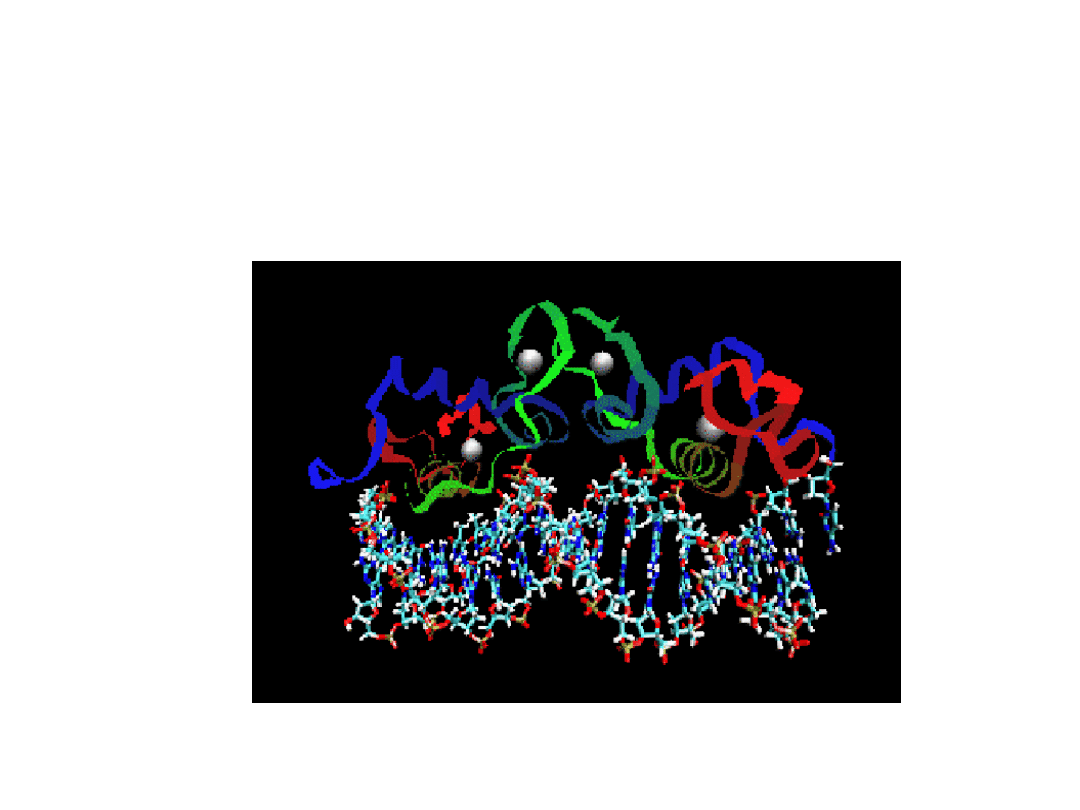
Wiązanie receptorów GR do DNA ; pomagają one regulować
ekspresję genetyczną :

•
Wykorzystano komórki, do których wprowadzono MMTV ze
znakowanymi białkami GFP białka GRIP-1 oraz pol II.
Białka GRIP-1 – glucocorticoid receptor interacting protein-
1
•
Następnie sprawdzano dynamikę zachowań białek GRIP-1,
receptorów GR i pol II pod wpływem hormonu sterydowego
– dex ( dexamethasone )
•
Wykorzystano w tych badaniach metodę FRAP i iFRAP
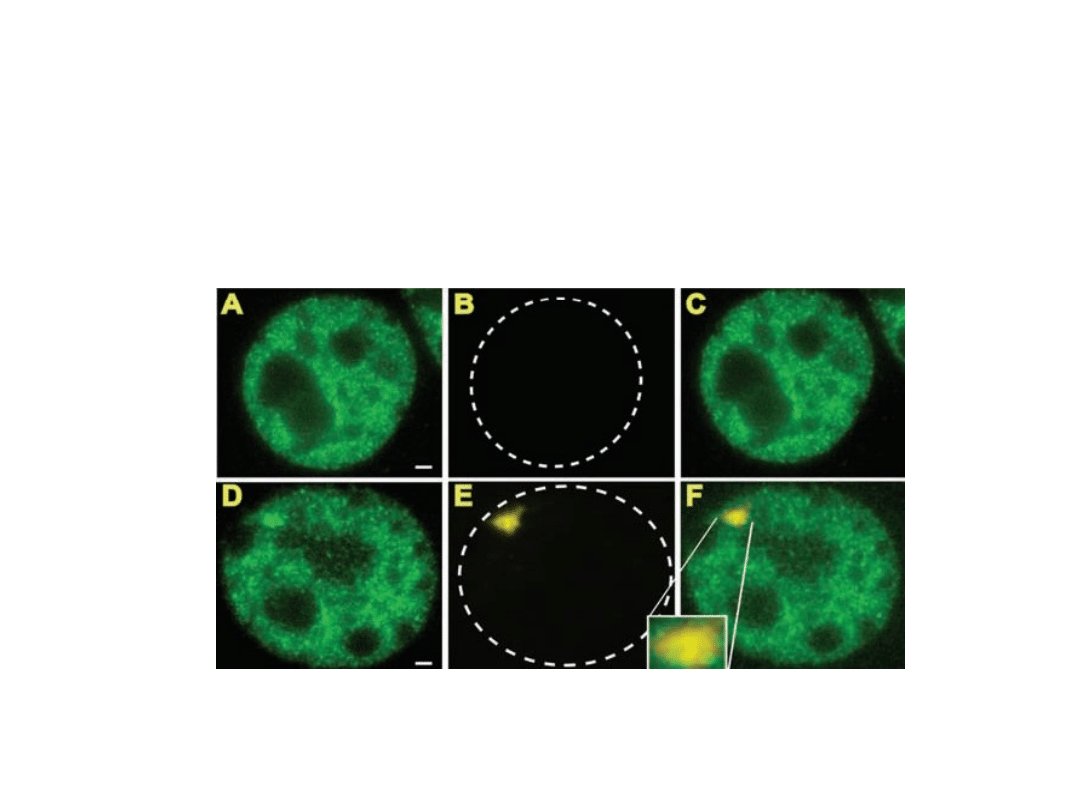
Wykrywanie co–lokalizacji GFP-GRIP-1 z nowopowstałym
transkryptem MMTV ( mouse mammary tumor virus ) metodą
FISH’a :

Analiza dynamiki białek GRIP-1 metodą FRAP w komórkach
MMTV :
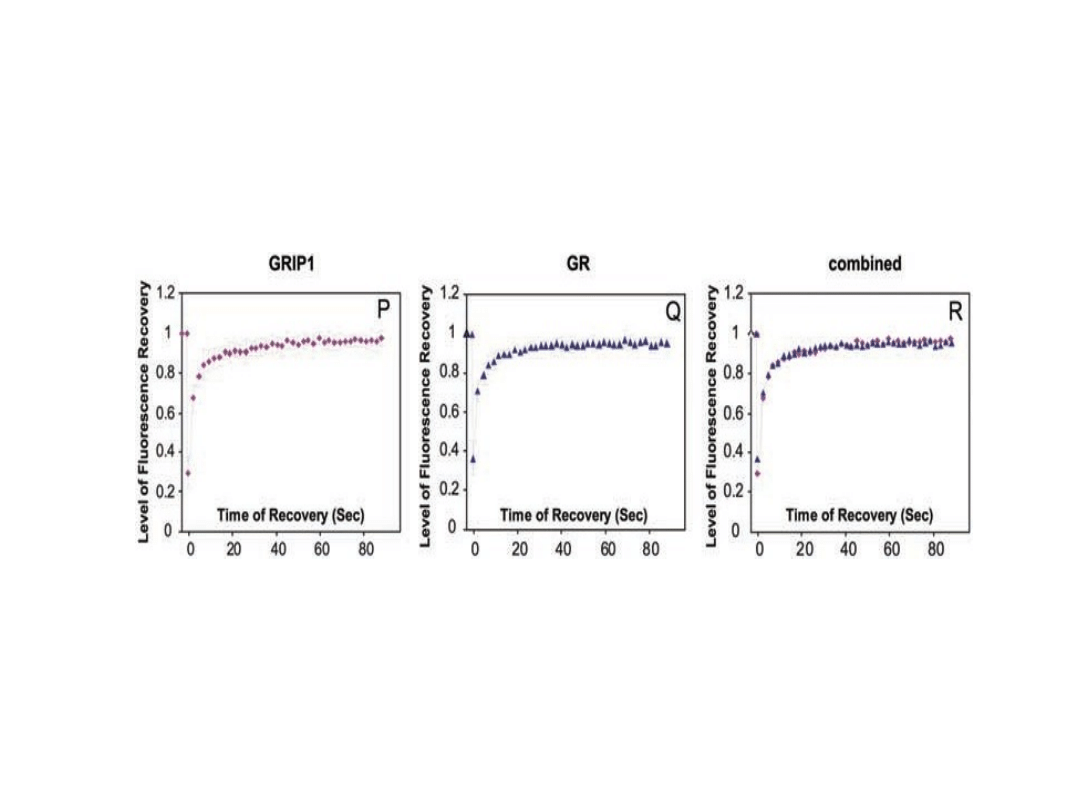
Porównanie dynamiki lokalizacji białek GRIP-1 oraz receptora
GR :

Wnioski :
• Intensywność odzyskiwania świecenia, a tym
samym dynamika zachowania GRIP-1 oraz GR
jest wizualnie nie do odróżnienia
• Białka GRIP-1 nie ograniczają dynamiki
zachowania GR
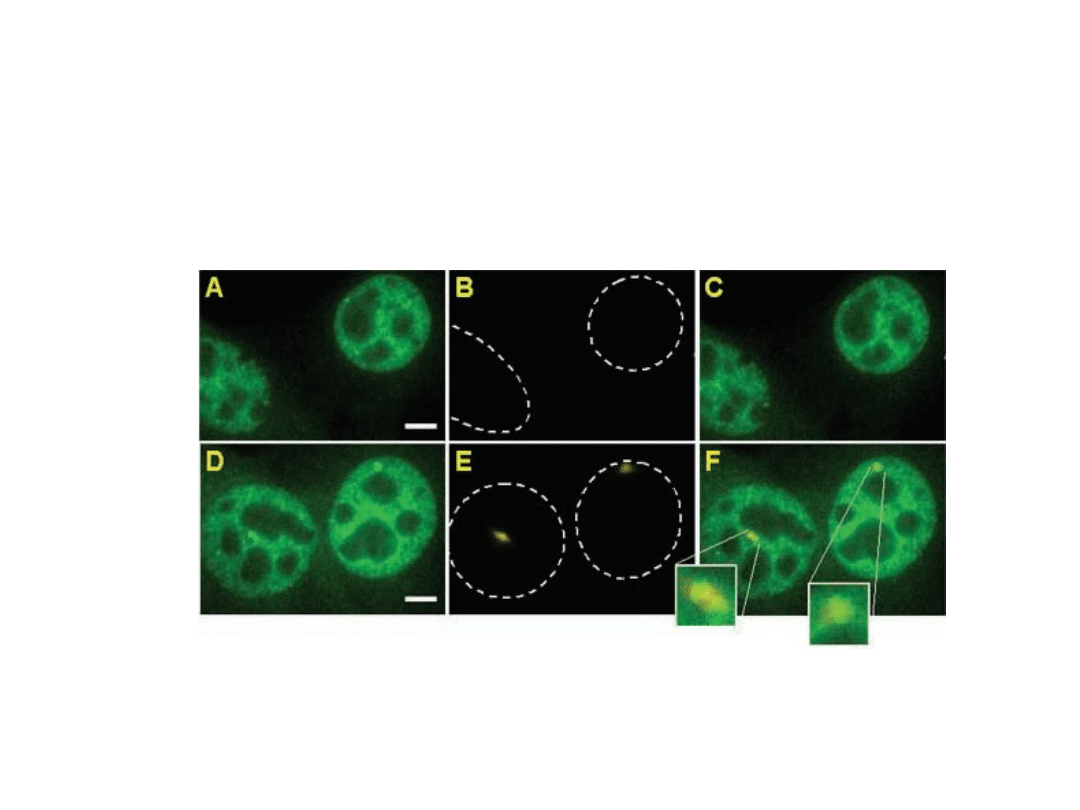
Wyniki co-lokalizacji pol II z nowopowstałym transkryptem
MMTV – z użyciem metody FISH’a :
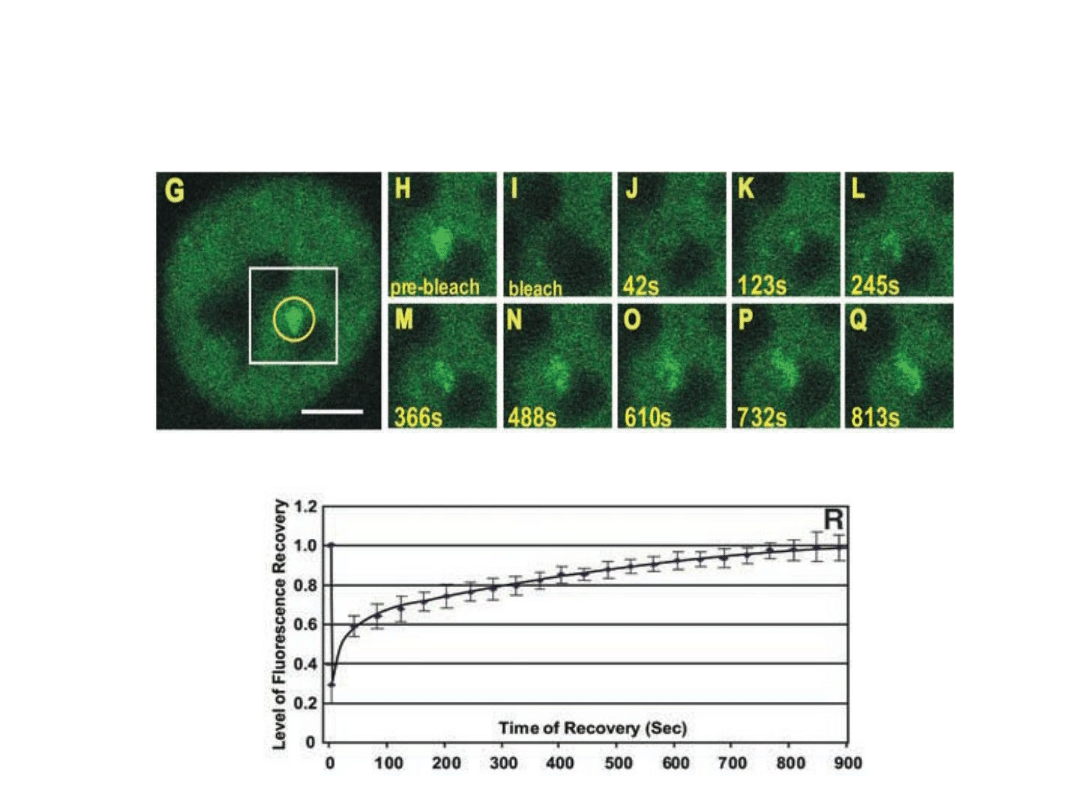
Wyniki analizy dynamiki pol II metodą FRAP :

Wnioski :
• Tempo odzyskania fluorescencji w miejscu
wypalenia, wynikające z wędrówki pol II, różni się
zasadniczo od tempa dynamiki białek GRIP-1 lub
receptora GR
• Czas jaki potrzebuje pol II na przemieszczenie się
do wypalonego miejsca jest o wiele dłuższy niż w
przypadku białek GRIP-1
• Dla białek GRIP-1 czas ten wynosi 5s, natomiast
dla pol II 13 min
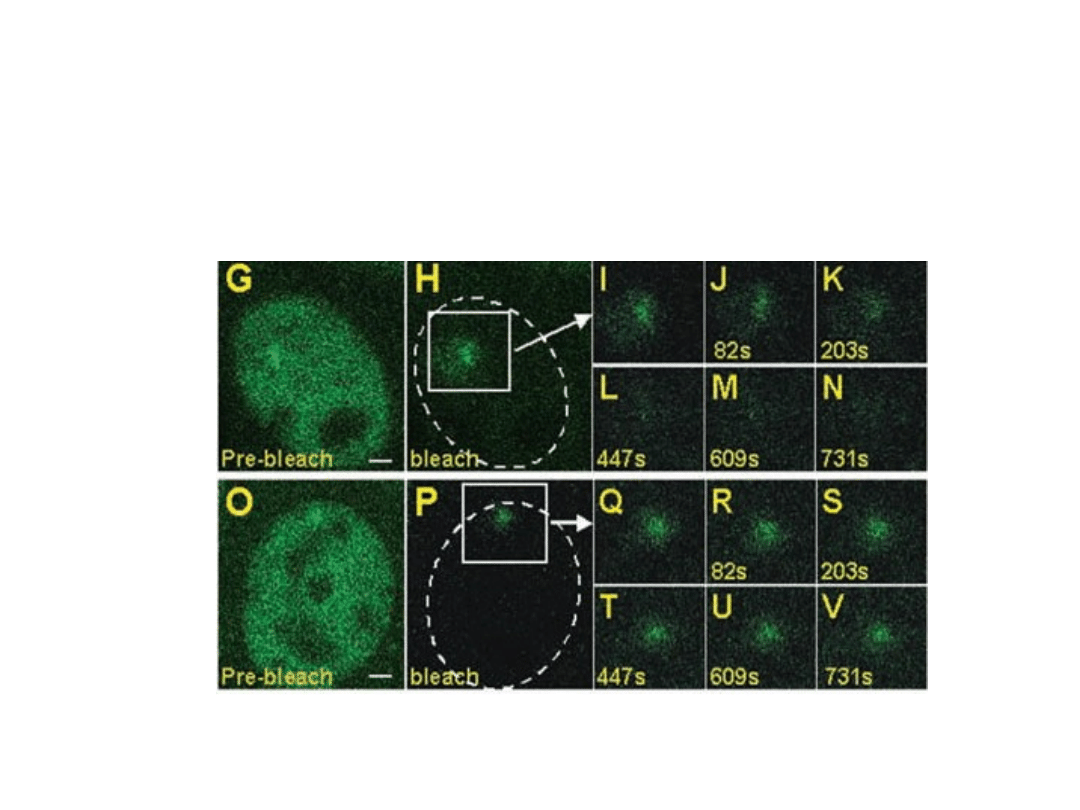
Badanie dynamiki GFP-pol II metoodą iFRAP :
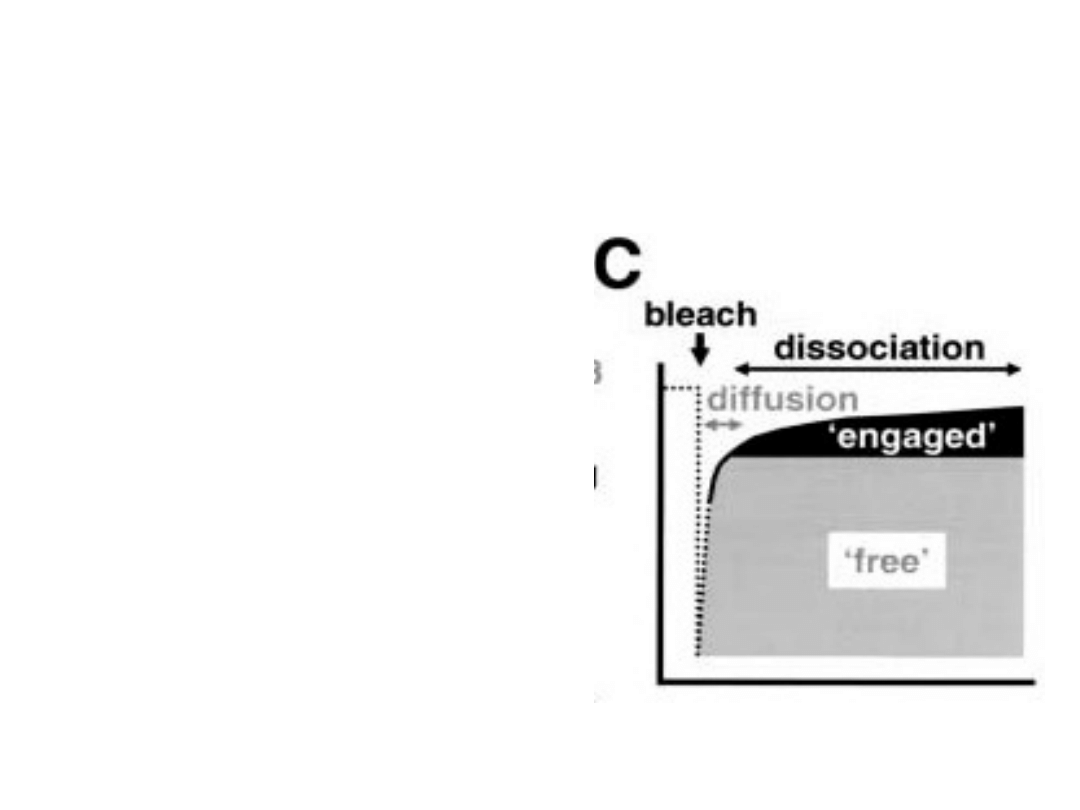
Polimeraza II – frakcje :
•
Z użyciem metody FRAP
stwierdzono istnienie dwóch
frakcji polimerazy II :
Wolna = zaangażowana w
przebieg procesu
transkrypcji
Szybka = mobilna, nie
wykazująca aktywności
transkrypcyjnej
Poza tymi dwiema
frakcjami, które były
znakowane białkami GFP, w
komórkach tych istnieje
również pula polimerazy II
endogennej - nie
znakowanej
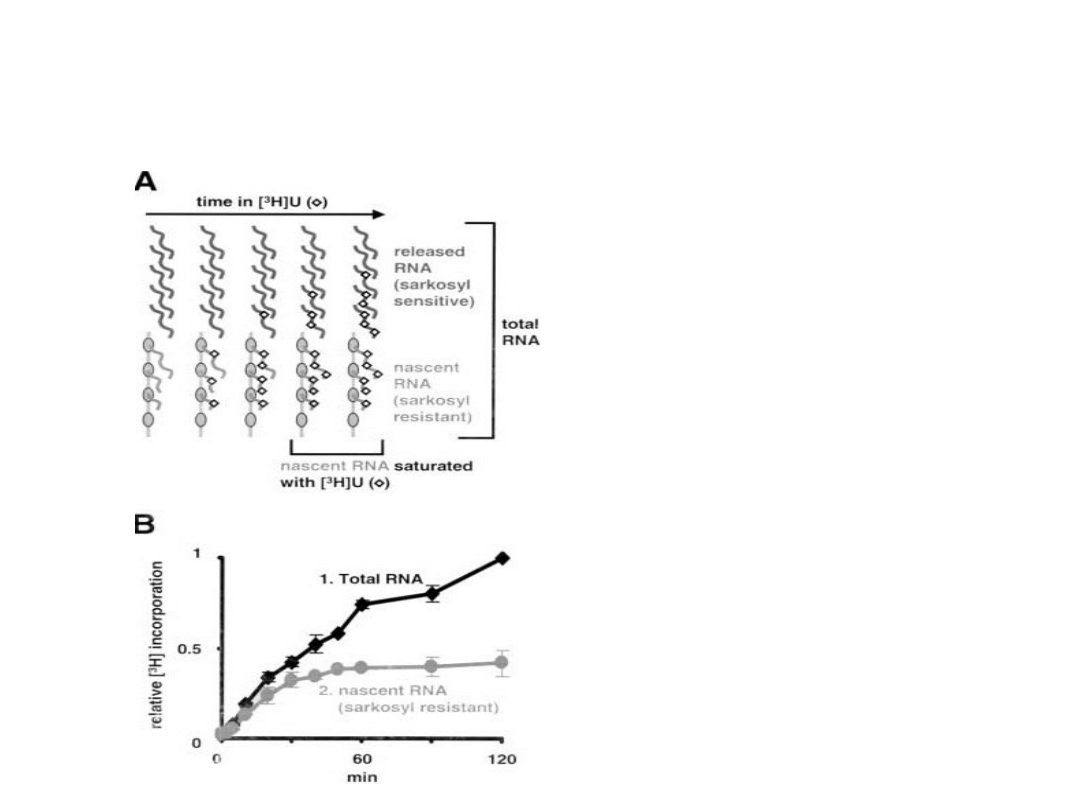
Wpływ sarkosylu na dynamikę endogennej pol II – użycie
metody radiolabeling :
•
Sarkosyl jest to detergent
służący do wyekstrahowania
transkryptu związanego z DNA
•
Nowopowstały transkrypt,
który jest odporny na działanie
sarkostylu początkowo
wykazuje bardzo zbliżoną
kinetykę do kinetyki totalnego
RNA, lecz po pewnym czasie
staje się nasycony
•
Okres półtrwania wynosi 14
min co jest spowodowane
tempem inicjacji i elongacji
przy czym tempo inicjacji i
terminacji muszą być sobie
równe
•
Okres półtrwania pol II na
matrycy wynosi również 14 min
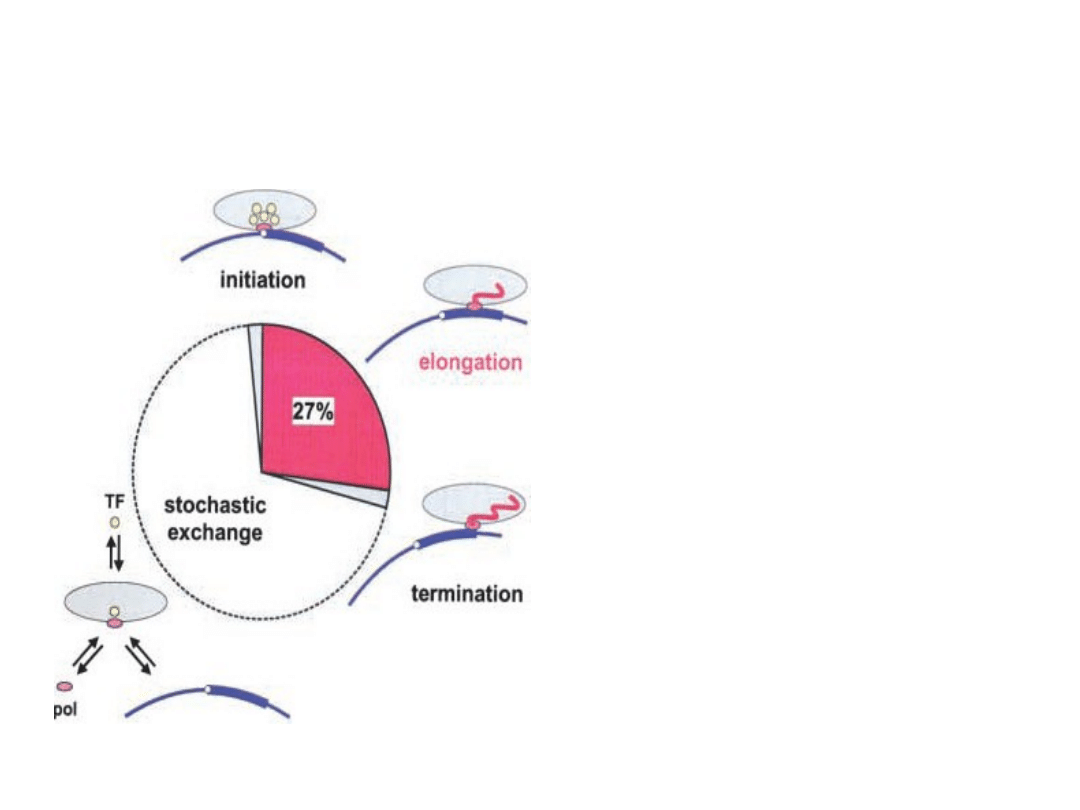
Miejsce polimerazy II w cyklu transkrypcyjnym :
• Fakcja szybka pol II
stanowi 75 % całej
puli plimerazy II,
natomiast pol II
zaangażowana 25 –
27 %.
• Pol II zaangażowana
jest to pol II
elongacyjna

Wnioski:
•
Liczba aktywnych transkrypcyjnie kompleksów rośnie po
dodaniu ligandu – hormonu
•
Jest to naturalna odpowiedz komórki na potraktowanie
jej hormonem niezależnie od poziomu ekspresji pol II
•
Rekrutacja pol II na promotorze jest wprost
proporcjonalna do ubytku polimerazy na promotorze
•
Zastosowanie technik : photobleaching, analizy Fish’a,
metody FRAP, iFRAP, FLIP ujawniły dużą dynamikę białek
wiążących się na promotorze, zarówno GRIP-1, jak i
receptorów GR
•
Dynamika zachowań białek GRIP-1 na matrycy jest
proporcjonalna do dynamiki zachowań receptorów GR, a
tym samym nie są one zdolne do wizualnego odróżnienia

•
Zadziałanie na komórkę hormonem i jego złączenie z
GR, powoduje wzrost aktywności rekrutacji czynników
transkrypcyjnych na promotorze
•
Dynamika zachowań GRIP-1 oraz GR wpływa na
inicjację transkrypcji
•
Pol II i GRIP-1 różnią się tempem dynamiki, co jest
dowodem na wysoko dynamiczną naturę zdarzeń
mających miejsce podczas inicjacji
•
Czas potrzebny do przemieszczenia się białek GRIP-1
do miejsca wypalonego jest równy 5s, natomiast dla pol
II wynosi 13 min, a więc tempo dynamiki białek GRIP-1
jest większe
•
Użycie sarkostylu potwierdziło, że zarówno znakowana
pol II (egzogenna ), jak i endogenna zachowują się
podobnie czyli wykazują zbliżoną dynamikę
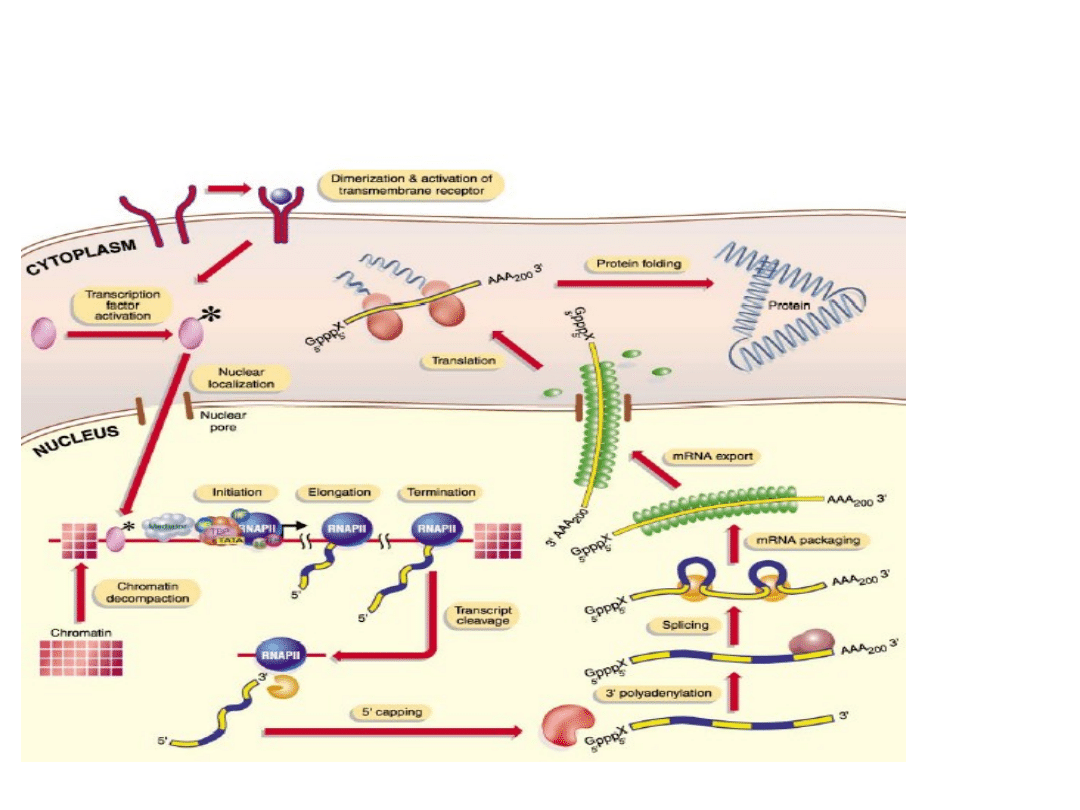
Klasyczny pogląd na ekspresję genów :
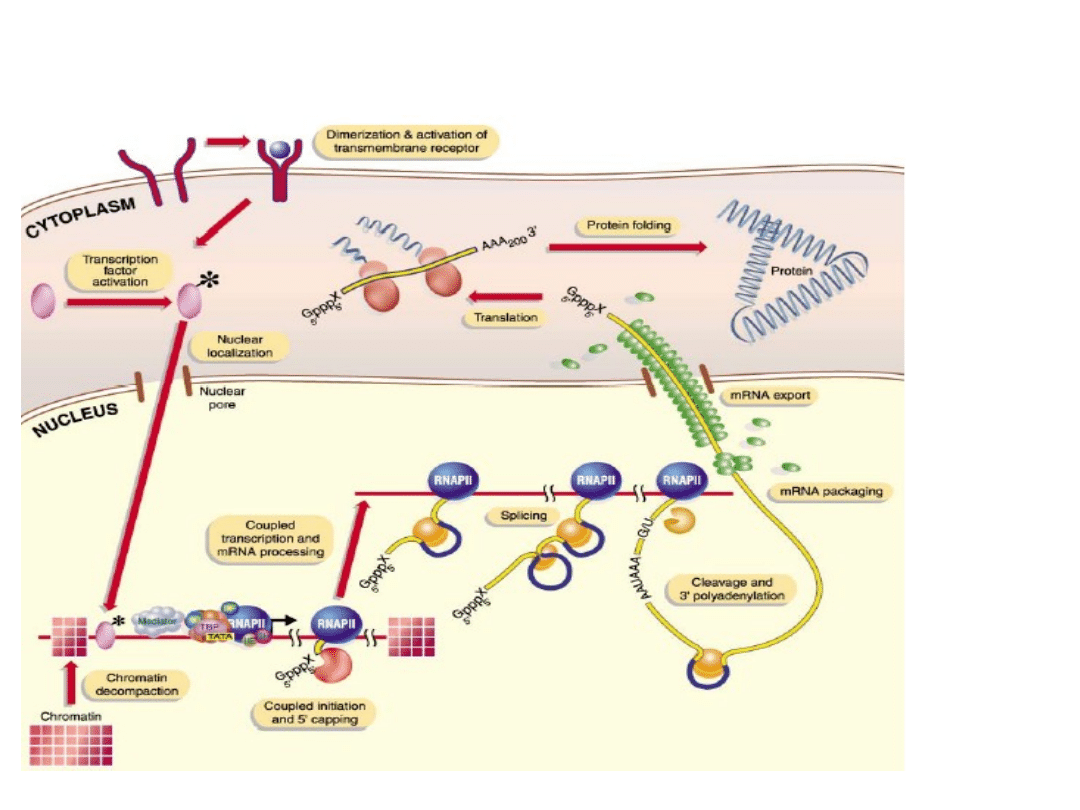
Obecny pogląd na ekspresję genów :
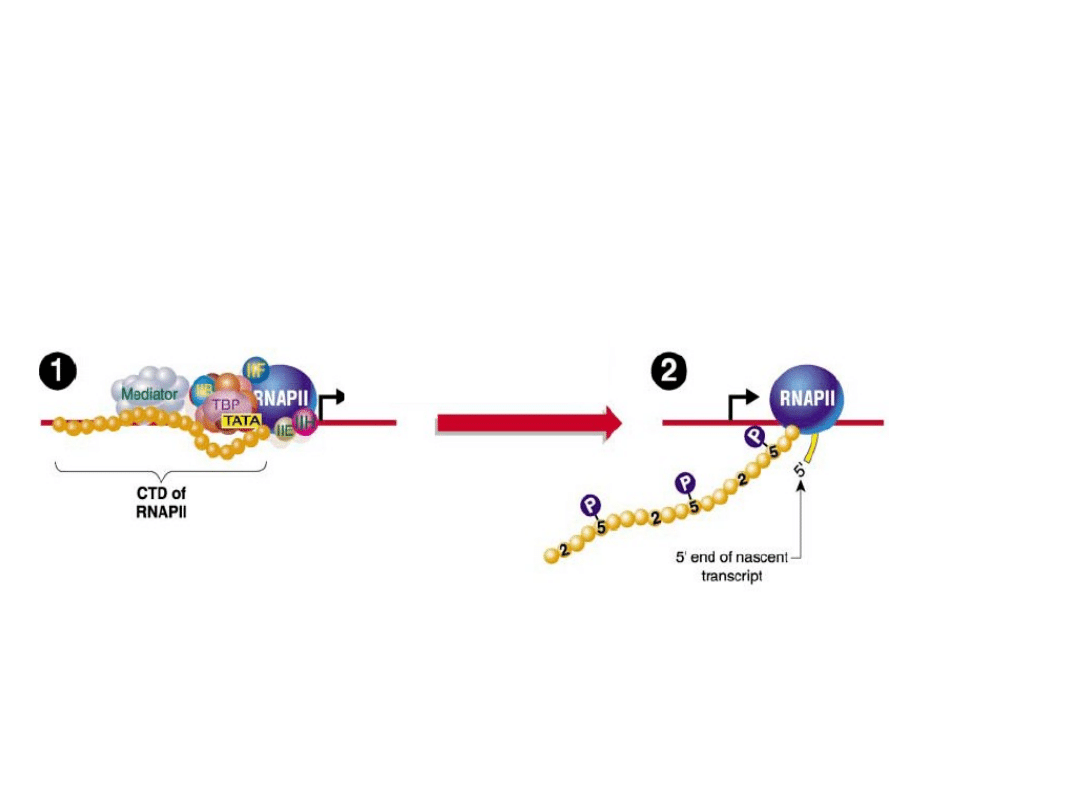
• Elementem integrującym procesy dojrzewania mRNA jest C-końcowa
domena polimerazy II. CTD zawiera wiele reszt aminokwasowych,
które ulegają odwracalnej fosforylacji podczas cyklu transkrypcyjnego.
Pol IIA, forma hypofosforylowana, jest składnikiem kompleksu
preinicjacyjnego, a jej fosforylacja i przekształcenie w
hiperfosforylowaną pol IIO powoduje rozpoczęcie elongacji.
• Tworzenie czapeczki, splicing oraz poliadenylacja mogą
zachodzić także niezależnie od transkrypcji, co zostało
zaobserwowane w eksperymentach in vitro oraz in vivo.
Jednak przebiegające kotranskrypcyjnie zachodzą z
większą specyficznością i wydajnością.
INICJACJA TRANSKRYPCJI
KOMPLEKS INICJACYJNY
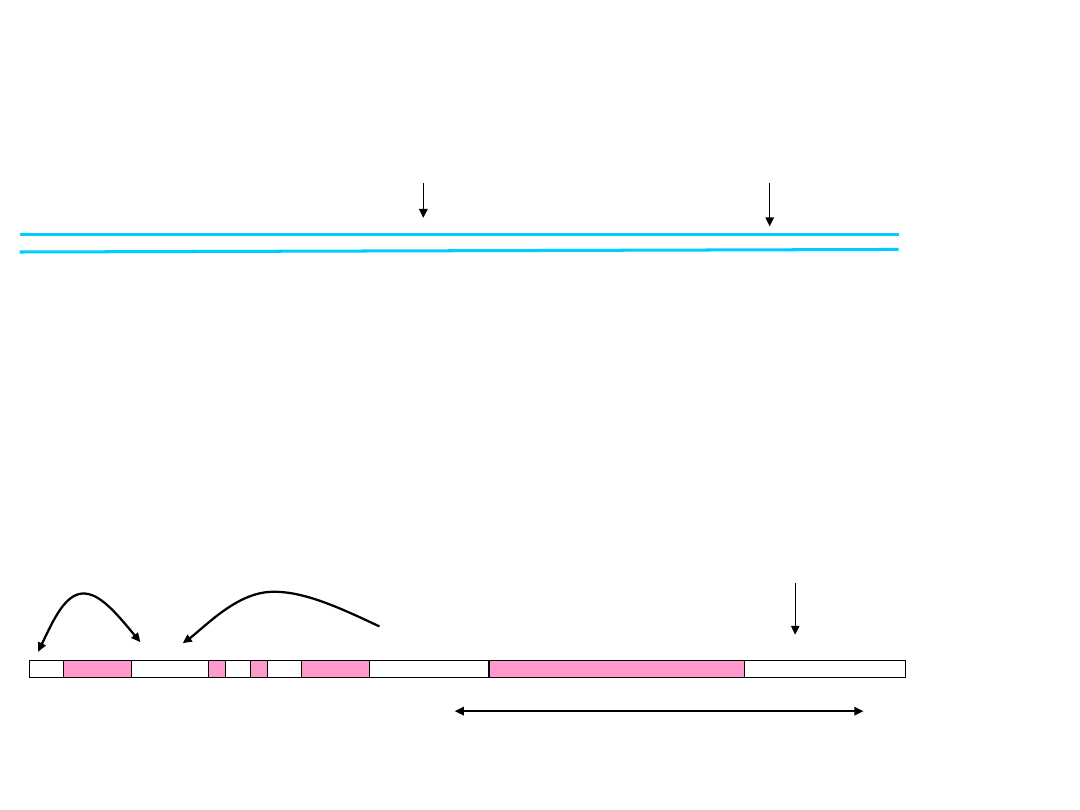
Budowa miejsca terminacji transkrypcji :
AG
AAUAAA
WYZNACZENIE OSTATNIEGO
EGZONU
RNA
SYGNAŁ POLI(A)
MIEJSCE PAUZUJĄCE
TERMINACJA TRANSKRYPCJI
CUGGCGGCGG
UUUUUUUUUUUUUUU
DOJRZEWANIE 3’ KOŃCA
CIĘCIE/POLIADENYLACJA
CA
G/U
AATAAA
DN
A
CA
G/T
MIEJSCE PAUZUJĄCE
TERMINACJA TRANSKRYPCJI
CTGGCGGCGG
TTTTTTTTTTTTTTTTT
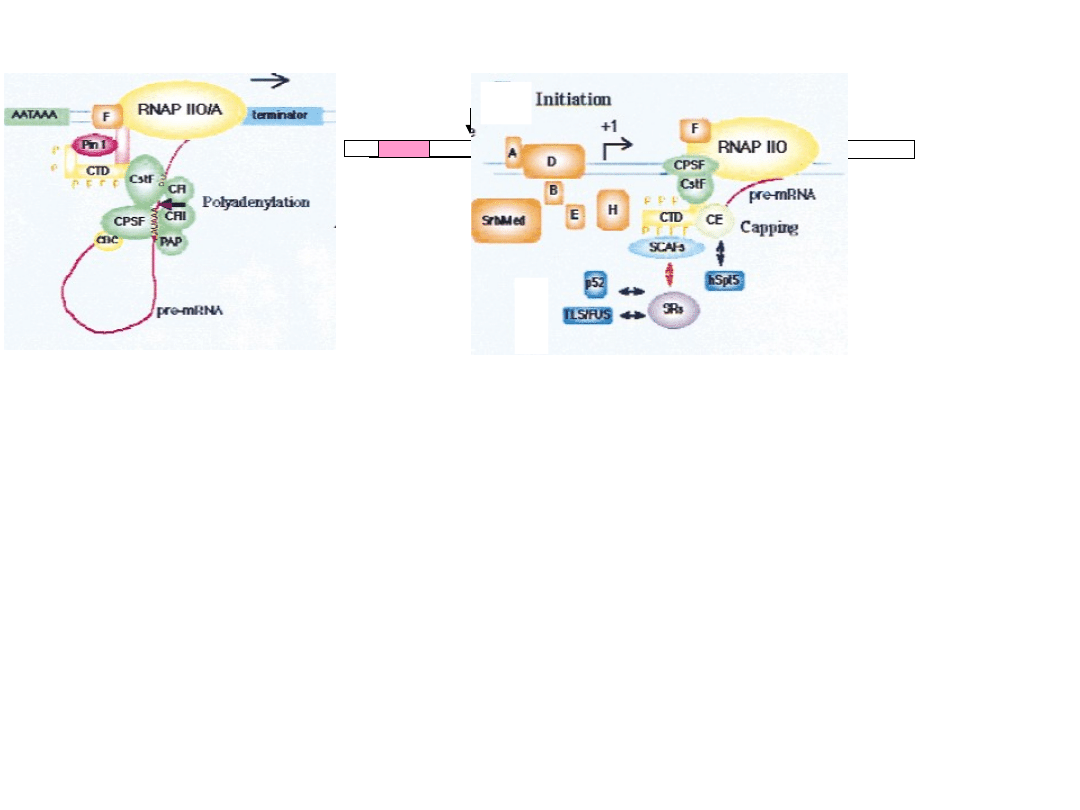
Czynniki tworzące kompleks
poliadenylacyjny :
• CPSF
– czynnik specyficzności cięcia i poliadenylacji - wiąże się z
sekwencją AAUAAA, stymuluje przyłączenie się polimerazy poli(A)
(PAP)
• PAP
– polimeraza poli(A) - niezbędny składnik kompleksu
poliadenylacyjnego
• CstF
– czynnik stymulacji cięcia - wiąże się z sekwencją G/U,
stymuluje przyłączenie się CF I i CF II
• CF I
i
CF II
– czynniki cięcia I i II - tną transkrypt w obrębie
nukleotydu A z dinukleotydu CA
• CTD
– domena C-końcowa polimerazy II - uczestniczy w formowaniu i
funkcjonowaniu stabilnego, aktywnego katalitycznie kompleksu
dojrzewania przez bezpośrednie interakcje z czynnikami poliadenylacji.
AG
AAUAAA
CA
G/U
SYGNAŁ POLI(A)
CIĘCIE
UUUUUUUU
CUGGCGGCGG
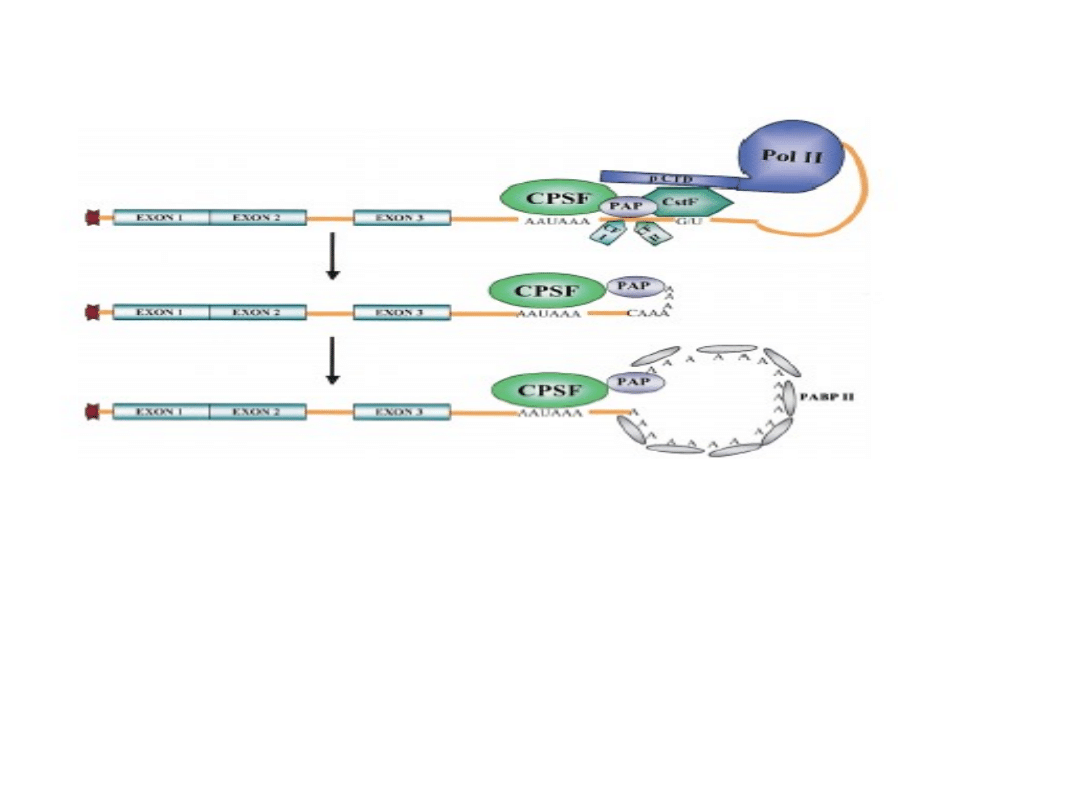
Przebieg poliadenylacji:
• przyłączenie CPSF i CstF do odpowiednich sekwencji
• oddziaływanie w/w czynników powoduje przyłączenie
się PAP i CF I i CF II
• cięcie w obrębie nukleotydu A
• dołączanie ogona poli(A) wspomagane przez PABP II
Terminacja transkrypcji a poliadenylacja :
• Mutacje sekwencji poli(A) pozwoliły na wykrycie związku
poliadenylacji z terminacją transkrypcji.
• Podobny efekt wywołują zmiany w ilości reszt T na końcu 3’
oraz mutacje w miejscu cięcia.
AATAAA
DN
A
CA
G/T
MIEJSCE PAUZUJĄCE
TERMINACJA TRANSKRYPCJI
CTGGCGGCGG
TTTTTTTTTTTTTTT
AAGAAA
CA
G/T
MIEJSCE PAUZUJĄCE
TERMINACJA TRANSKRYPCJI
CTGGCGGCGG
TTTTTTTTTTTTT
MUTACJA
POL
POL

Model terminacji transkrypcji :
• model „torpedy” -
cięcie transkryptu w
obrębie dinukleotydu
CA jest
zasygnalizowane
polimerazie przez
działanie 5’ - 3’
egzonukleazy, które
szybko degraduje
produkt 3’cięcia, co
powoduje, że
polimeraza staje się
skłonna do
oddysocjowania od
matrycy DNA
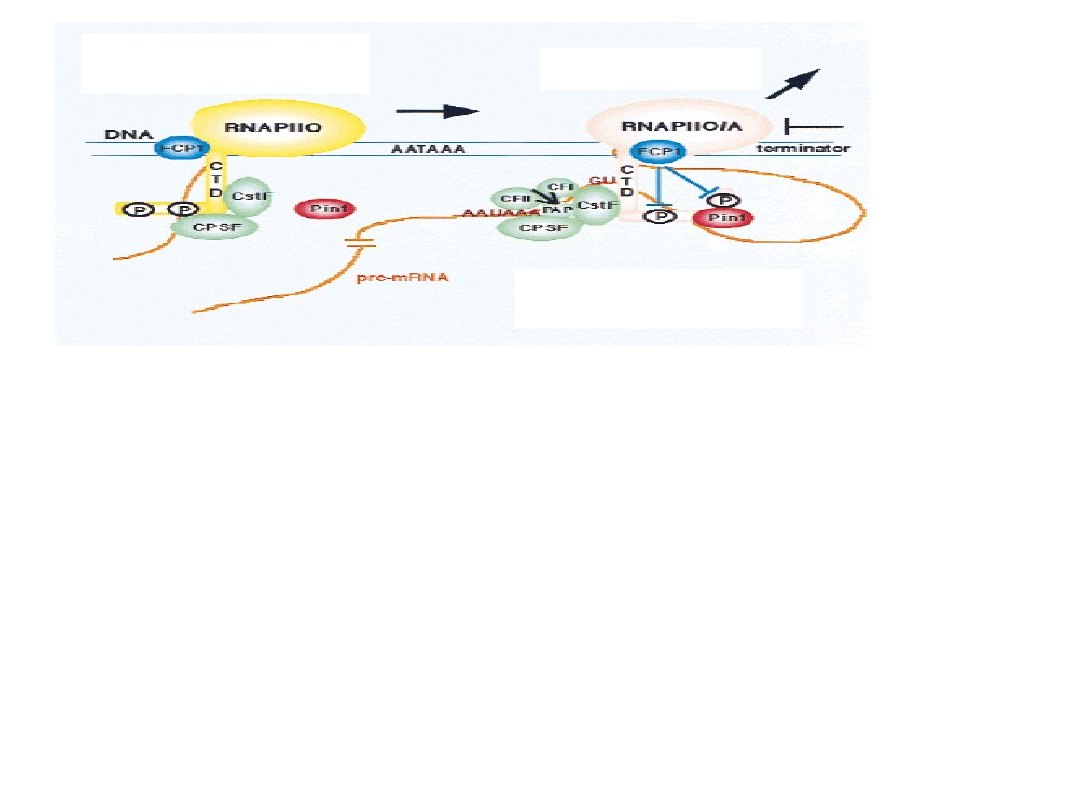
•
Po przejściu pol IIO przez sygnał poli(A), rozpoczyna się tworzenie
kompleksu poliadenylacyjnego. CPSF i CstF opuszczają CTD i
przyłączają się do odpowiednich sekwencji.
•
pol II chwilowo zatrzymuje się w miejscu pauzującym, co daje CTD
więcej czasu na oddziaływanie z pozostałymi czynnikami kompleksu.
Oddziaływanie to jest możliwe dzięki wygięciu transkryptu pre-mRNA
w dużą pętlę.
•
Po całkowitym uformowaniu kompleksu dochodzi do
endonukleolitycznego cięcia i rozpadu kompleksu.
•
Następuje stopniowa defosforylacja CTD, spowalniająca bieg pol II.
Dodatkowo działanie egzonukleazy trawiącej produkt 3’ powoduje
terminację i oddysocjowanie pol II od matrycy.
•
Pozwala to na reinicjację transkrypcji i rozpoczęcie nowego cyklu
transkrypcyjnego.
ELONGACJA
TERMINACJA
miejsce pauzujące
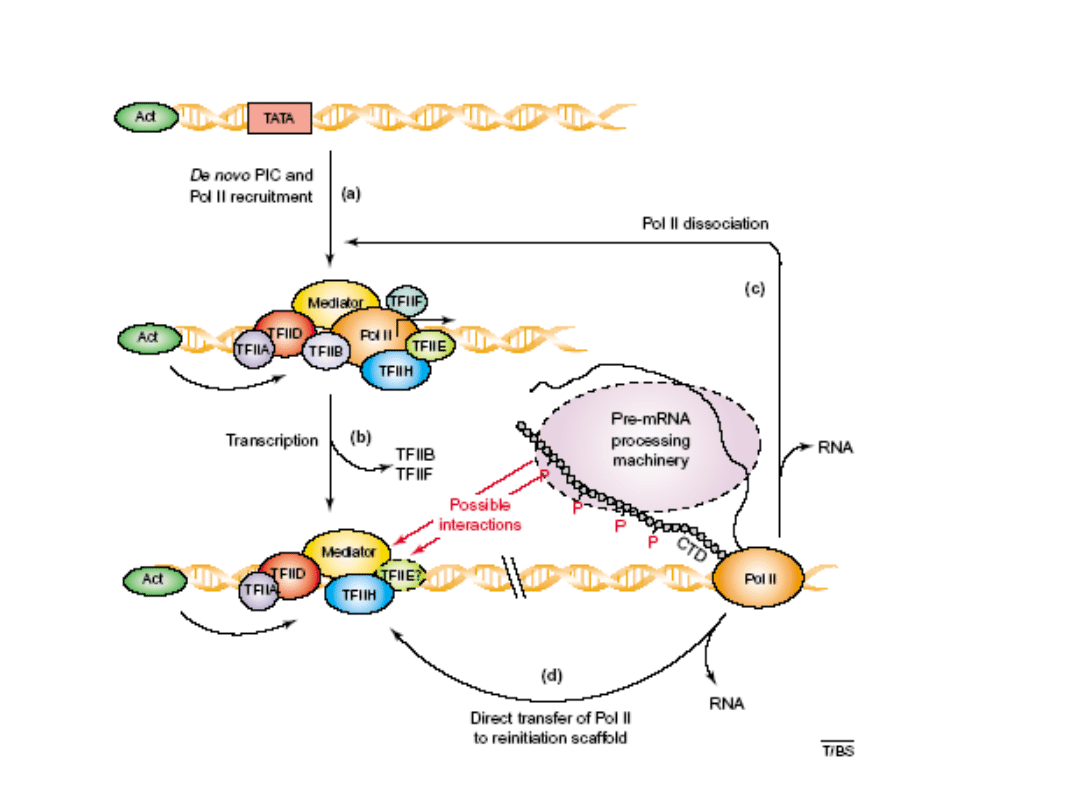
Mechanizm zależnej od białek reinicjacji
transkrypcji :
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
Wyszukiwarka
Podobne podstrony:
Biol kom cz 1
Biol kom cz 4
Biol kom cz੠2
Biol kom cz 5
Biol kom cz 2
Biol kom cz 1
Biol mol kom cz 3
Sp asp proc kom cz VII 2010
Test biol kom, biologia komórki(3)
BIOL.KOM pytania chyba Witaliński, biologia uj, biologia II, biologia komorki, egz
Biol KOM zaliczenie
Organella Biol Kom
Kult uwar kom cz VIII 2010
Kult uwar kom cz II 2010
biol kom wyklad 2103, Chemia środków bioaktywnych (umcs), BIOLOGIA KOMÓRKI
Kultura a kom cz I 2010
Biol KOM zaliczenie id 87300
Pare pytań z egzaminu na biol kom
biol kom pyt 11
więcej podobnych podstron