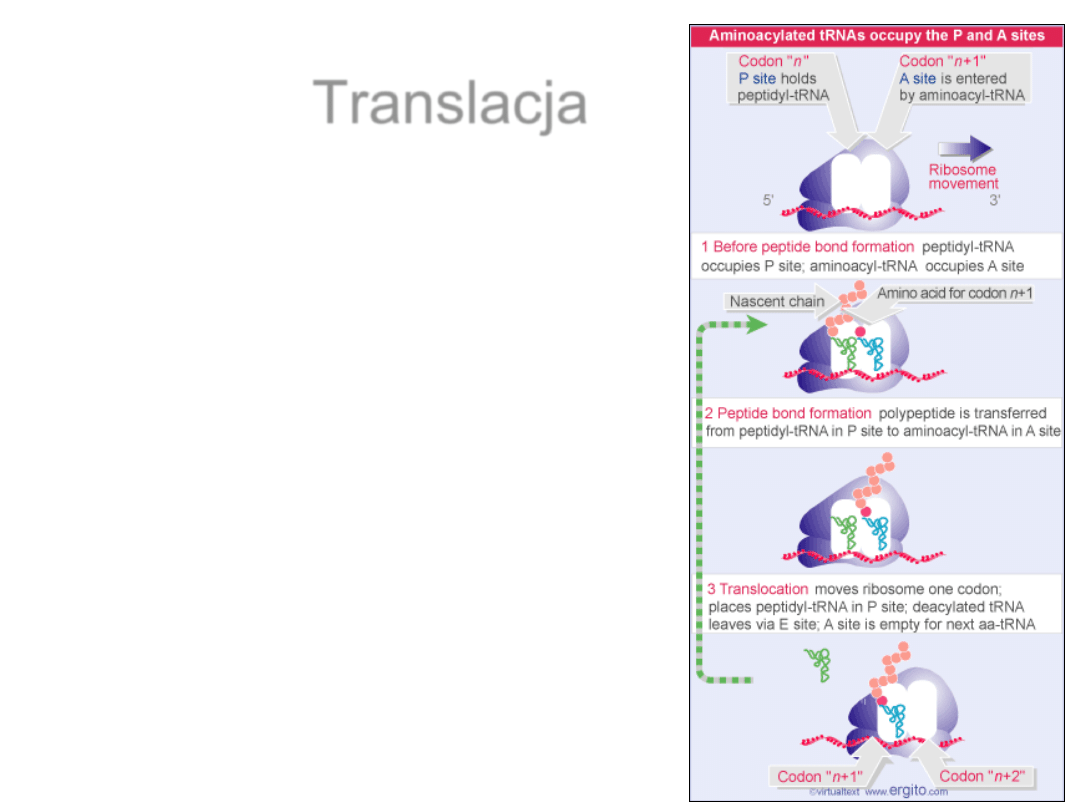
Translacja
• Inicjacja
• Elongacja
• Terminacja

Białka są syntetyzowane w kierunku od końca
aminowego do karboksylowego
Rozmieszczenie 3H-
leucyny w łańcuchach α hemoglobiny
syntetyzowanej po ekspozycji retikulocytów
na działanie trytowanej leucyny
Translacja mRNA
przebiega w kierunku 5’→ 3’
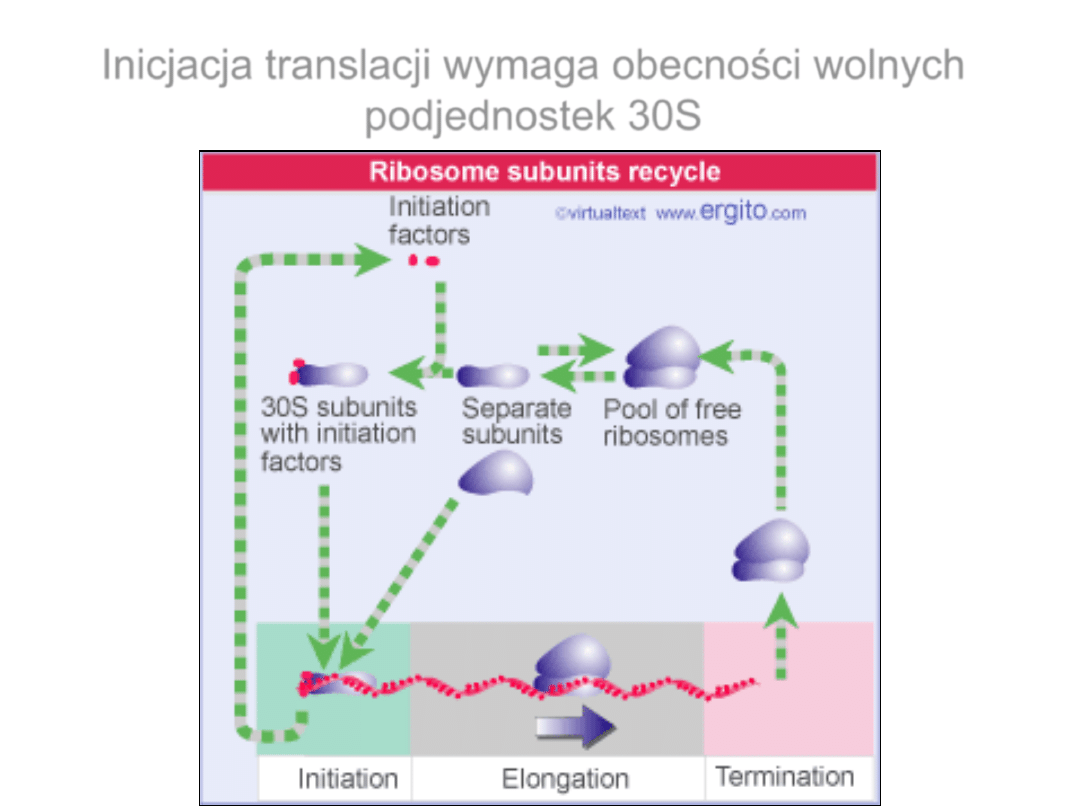
Inicjacja translacji wymaga obecności wolnych
podjednostek 30S
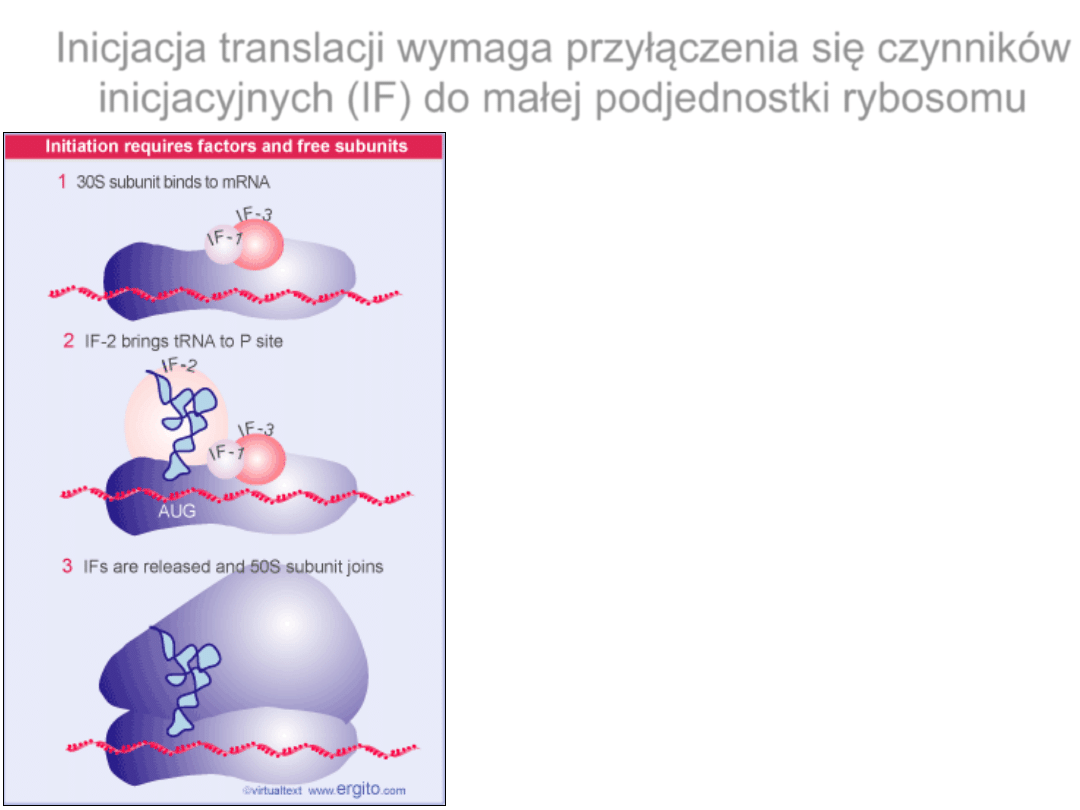
IF-
3 wiąże się do podjednostki 30S i
odpowiada za specyficzne
przyłączanie mRNA.
IF-
2 wiąże specjalny inicjatorowy
tRNA i kontroluje jego przyłączenie w
miejscu P rybosomu.
IF-
1 wiąże się do 30S tylko jako cześć
kompleksu inicjacyjnego. Czynnik ten
wiąże się do miejsca A i zapobiega
związaniu się aminoacylo-tRNA.
Prawdopodobnie przeciwdziała on
także przyłączeniu się podjednostki
50S.
Inicjacja translacji wymaga przyłączenia się czynników
inicjacyjnych (IF) do małej podjednostki rybosomu
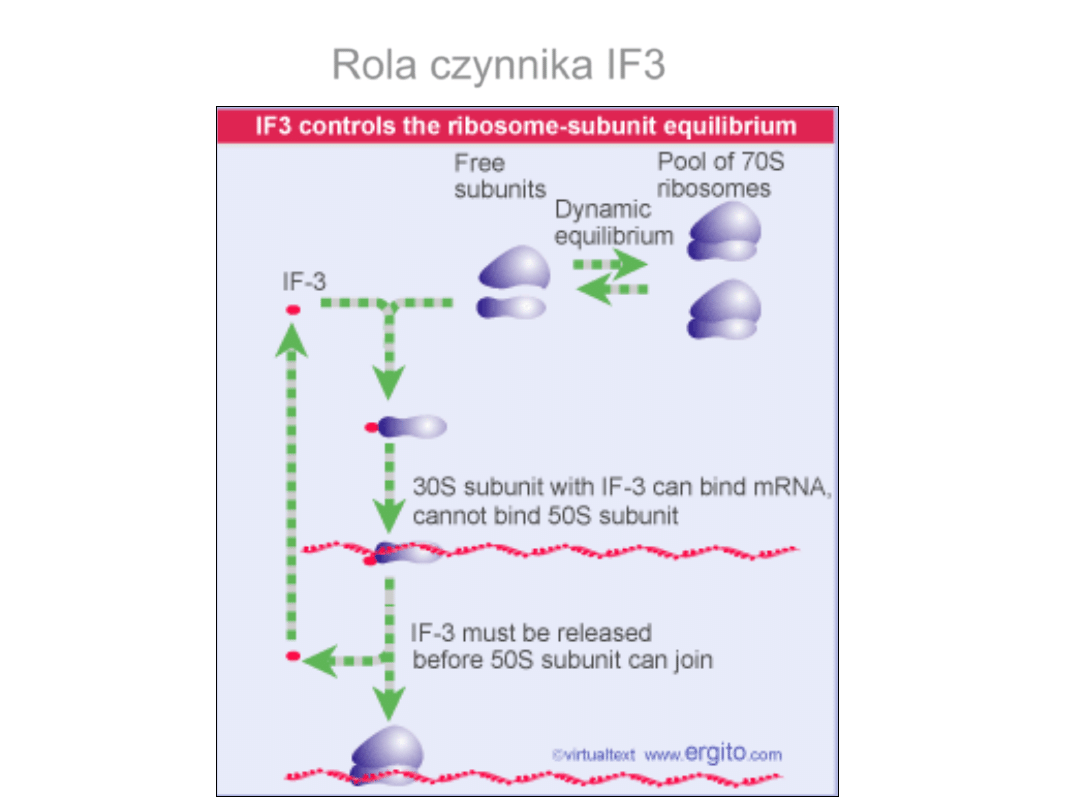
Rola czynnika IF3
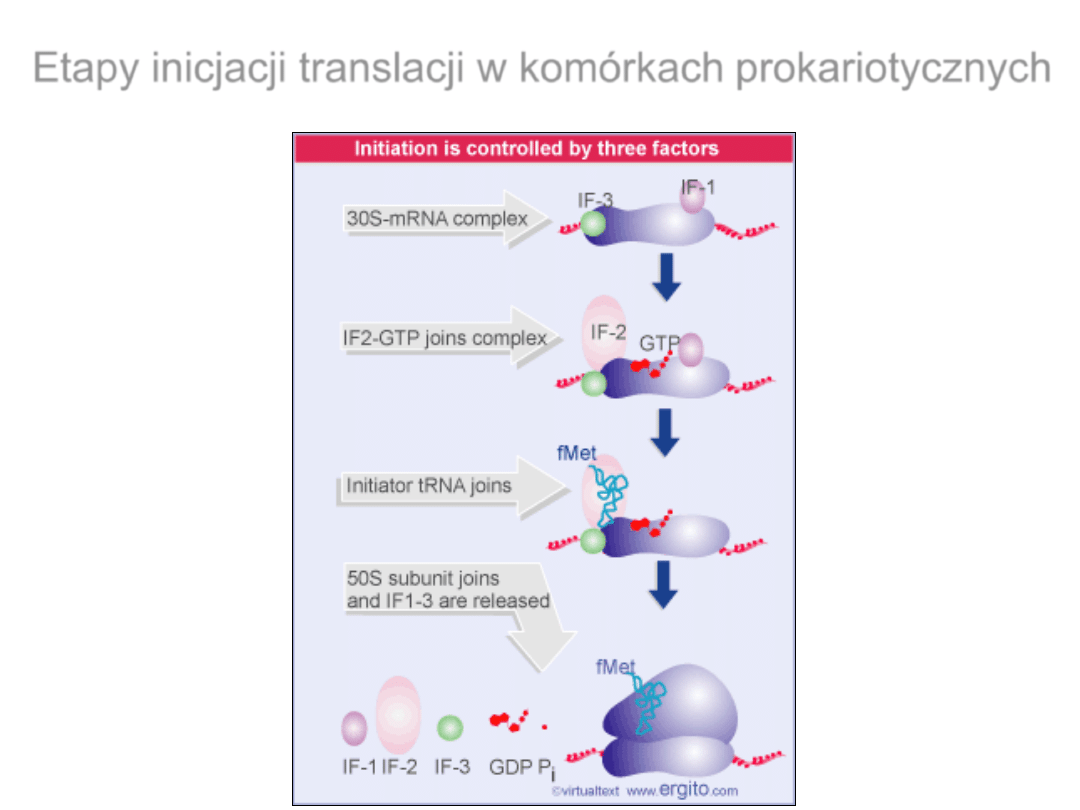
Etapy inicjacji translacji w komórkach prokariotycznych
Kodony
inicjacji:
AUG
Ale u bakterii
może też
występować
GUG lub UUG
tRNA
f
Met
tRNA
m
Met
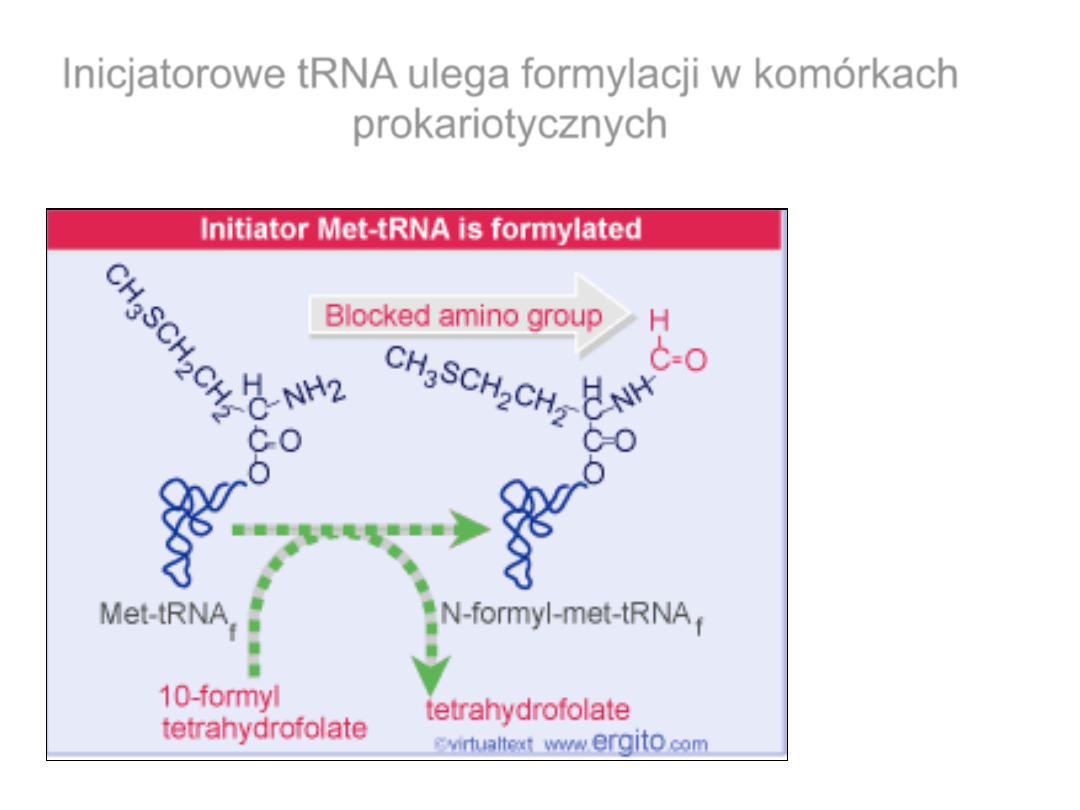
Inicjatorowe tRNA ulega formylacji
w komórkach
prokariotycznych
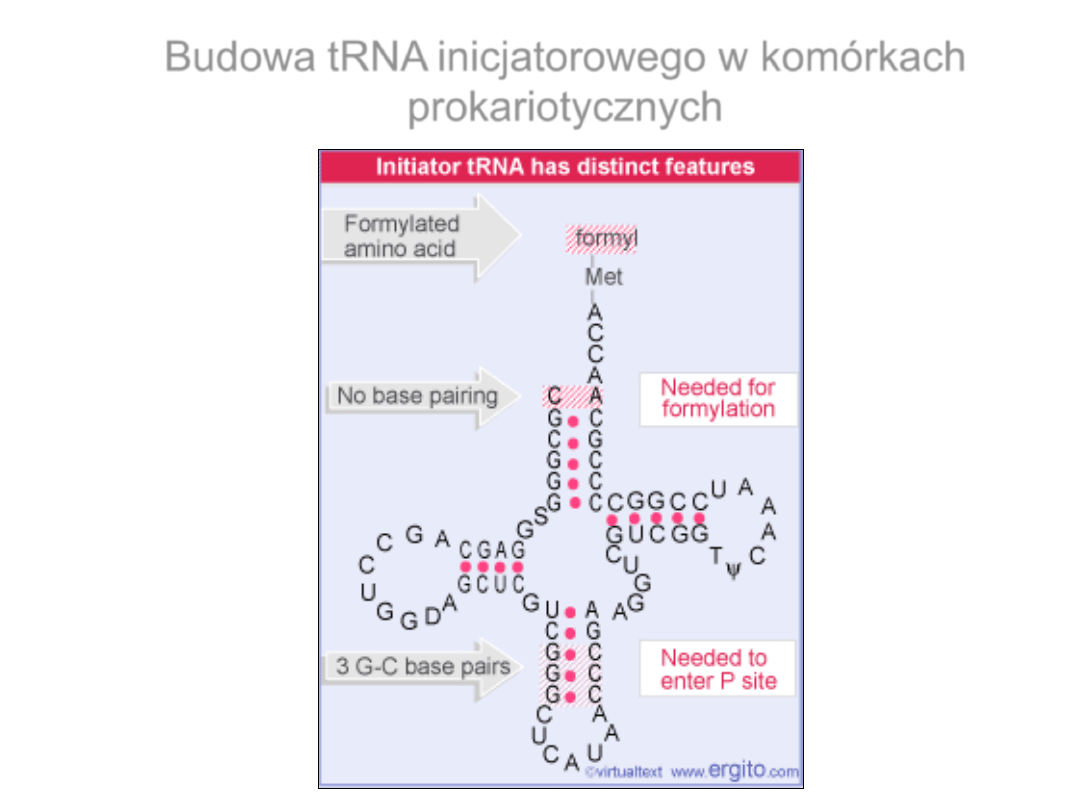
Budowa tRNA inicjatorowego
w komórkach
prokariotycznych
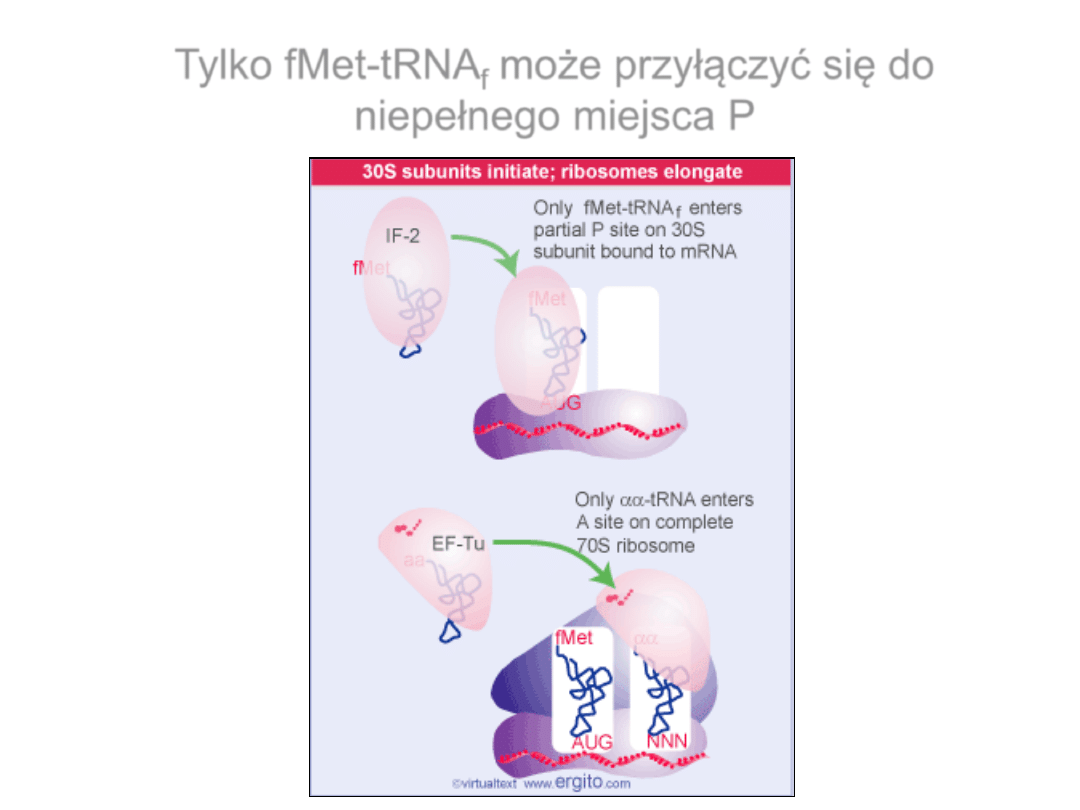
Tylko fMet-tRNA
f
może przyłączyć się do
niepełnego miejsca P
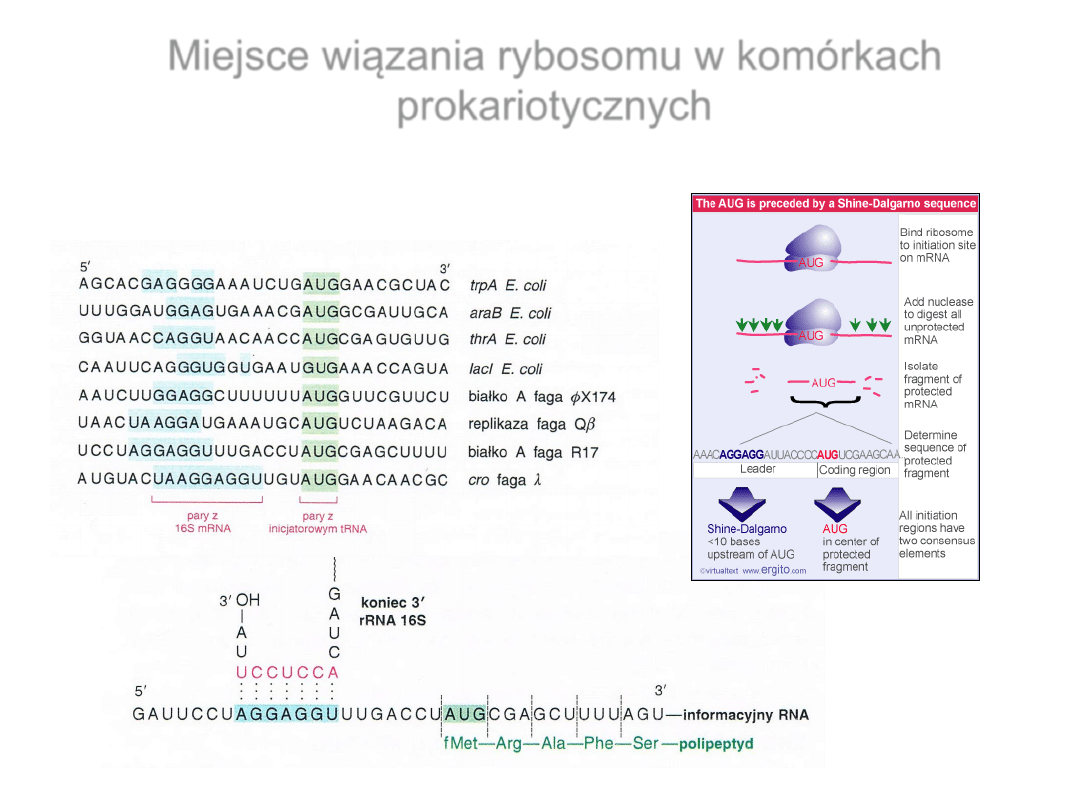
Miejsce wiązania rybosomu w komórkach
prokariotycznych
Sekwencje miejsc inicjacji translacji w niektórych
cząsteczkach RNA bakteryjnych i wirusowych
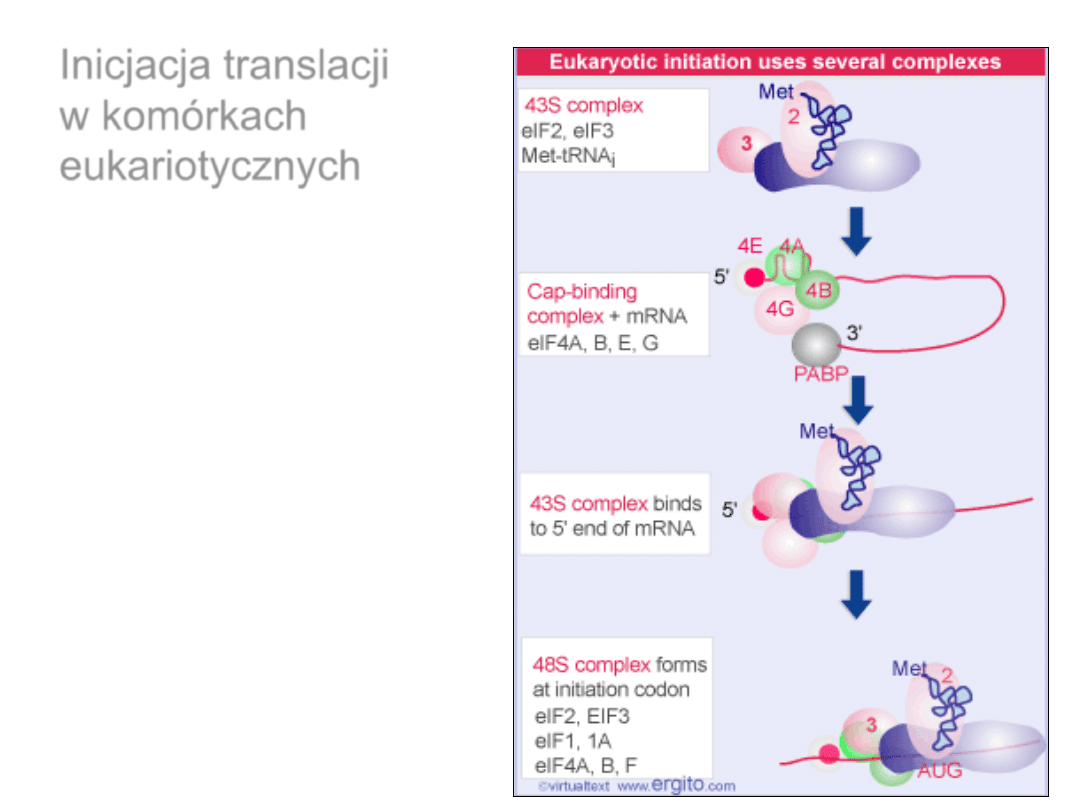
Inicjacja translacji
w komórkach
eukariotycznych
AUG
kodon startu
tRNA
i
Met
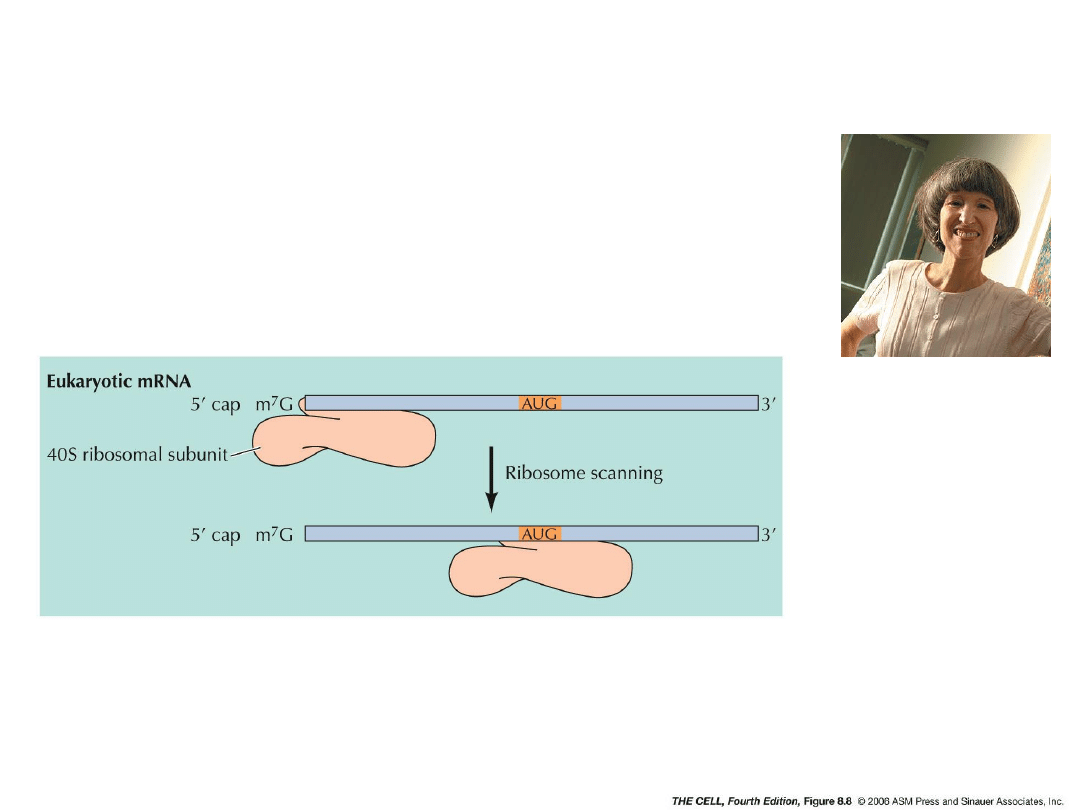
Rozpoznawanie mRNA przez 40S
u eukariontów :
- więcej czynników translacyjnych (eIF)
- brak sekwencji SD
= odnajdywanie kodonu Start przez skaning
tzw. sekwencja Kozak
5’ CC A/G CC
AUG
G 3’
Marilyn Kozak
- podjednostka 40S, zawierająca Met-tRNA
i
Met
wiąże się do miejsca Kap na mRNA i skanuje mRNA aż
znajdzie odpowiednią sekwencję
- wykorzystywana jest energia z hydrolizy ATP

Czynniki inicjacyjne wiążące się do mRNA
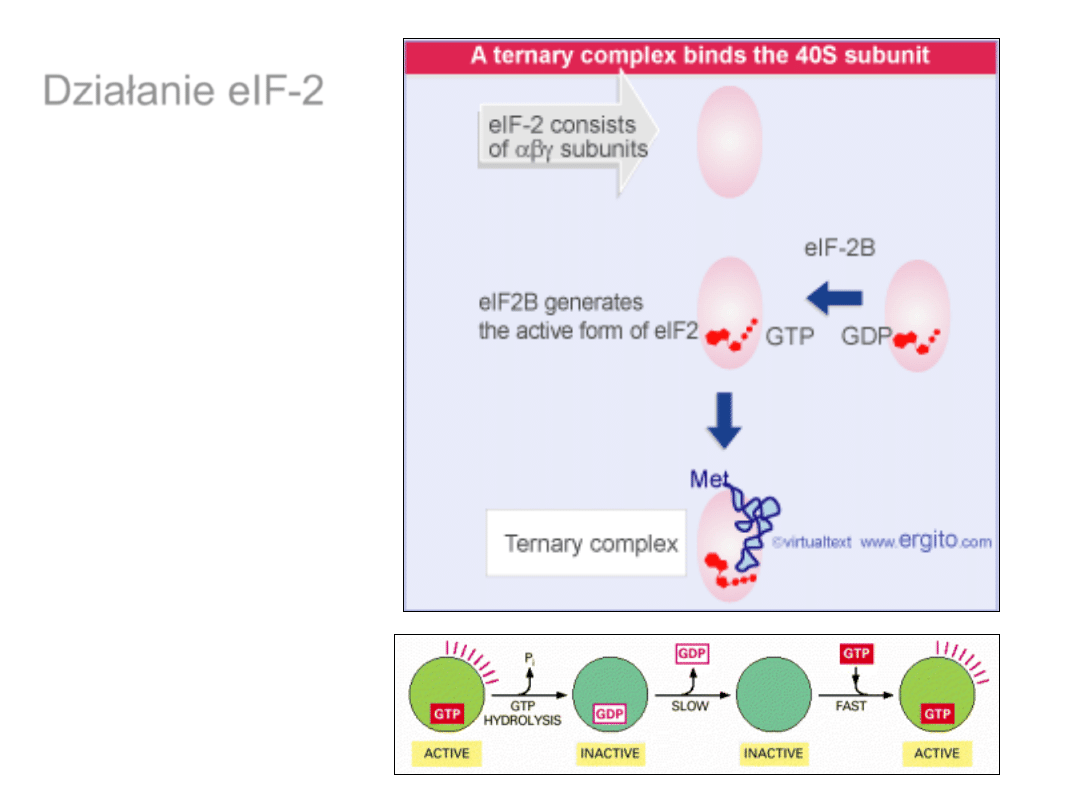
Działanie eIF-2
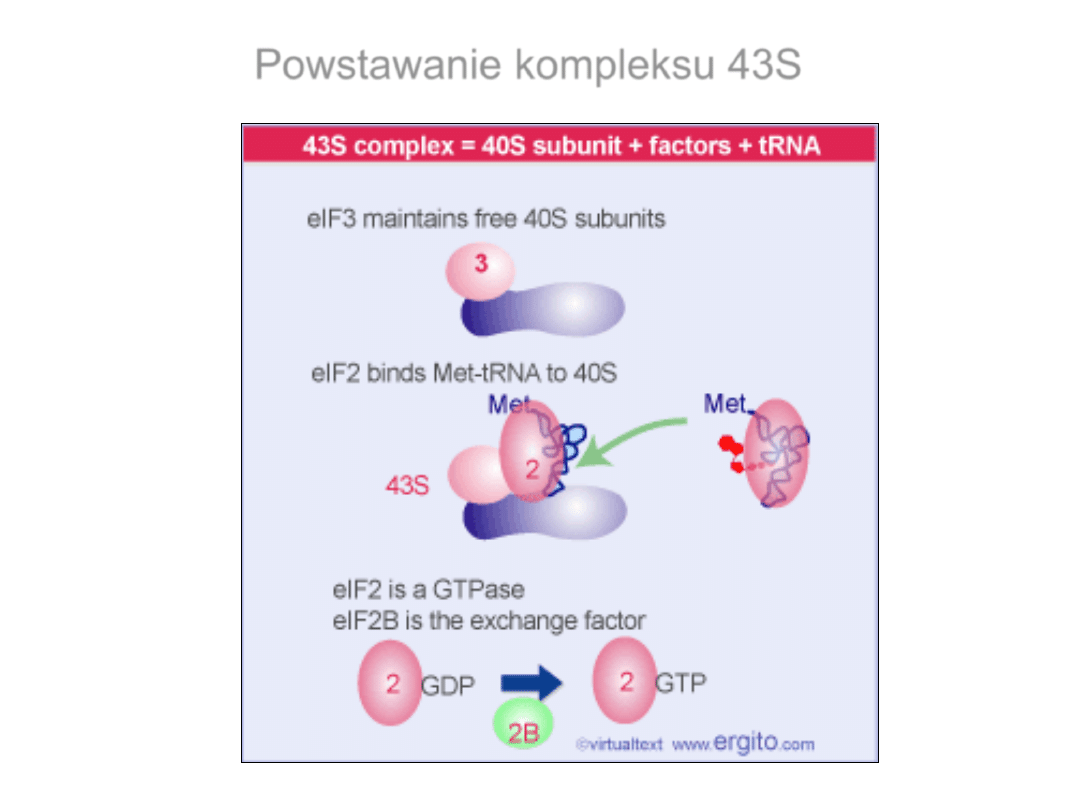
Powstawanie kompleksu 43S
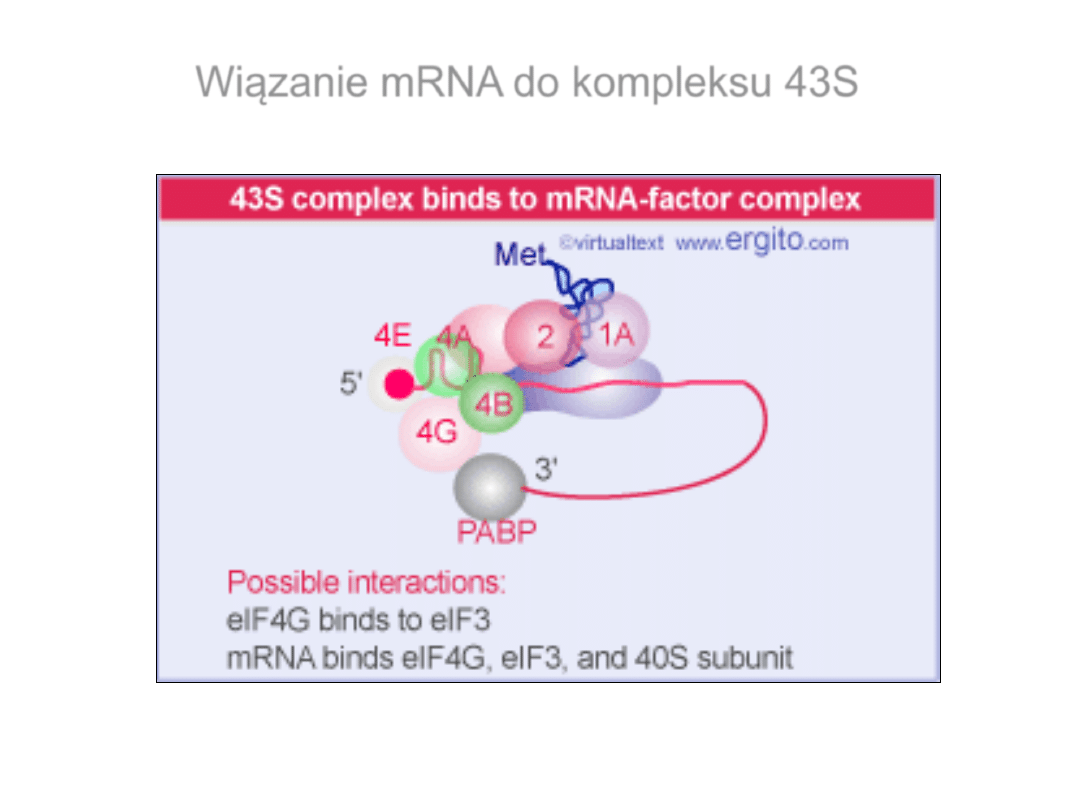
Wiązanie mRNA do kompleksu 43S
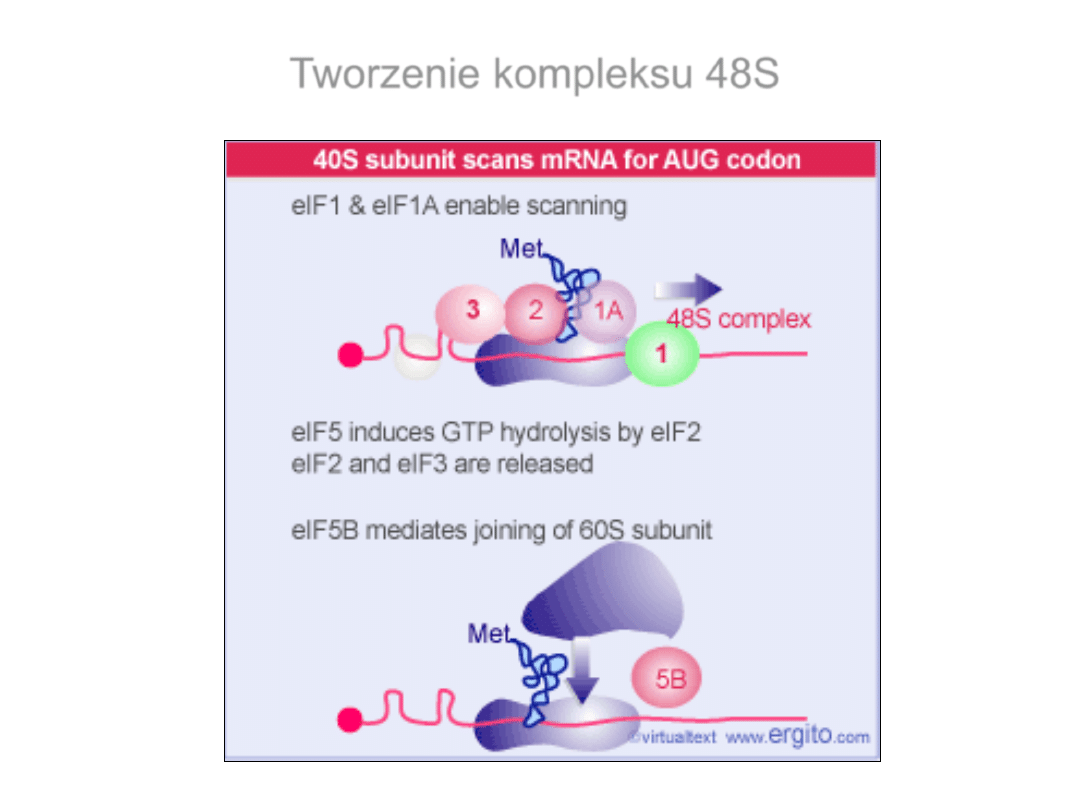
Tworzenie kompleksu 48S

Alternatywny proces translacji
Mechanizm inicjacji
wewnętrznej zależny jest od odcinka
sekwencji,
który
zwany
jest
wewnętrznym
miejscem
oddziaływania z rybosomem (IRES, ang. Internal Ribosome
Entry Site)
Sekwencje takie
występują w szeregu wirusowych RNA, ale
także od IRES zależy translacja niektórych mRNA w komórkach
eukariotycznych
zaangażowanych w:
- cykl
komórkowy
-
apoptozę
-
regulację odpowiedzi na warunki stresowe
-
regulację odpowiedzi na proces nowotworzenia
Z elementami IRES
wiążą się specyficznie białka – czynniki
wiążące się z IRES in trans (ITAF, ang. IRES Trans-Acting
Factors)
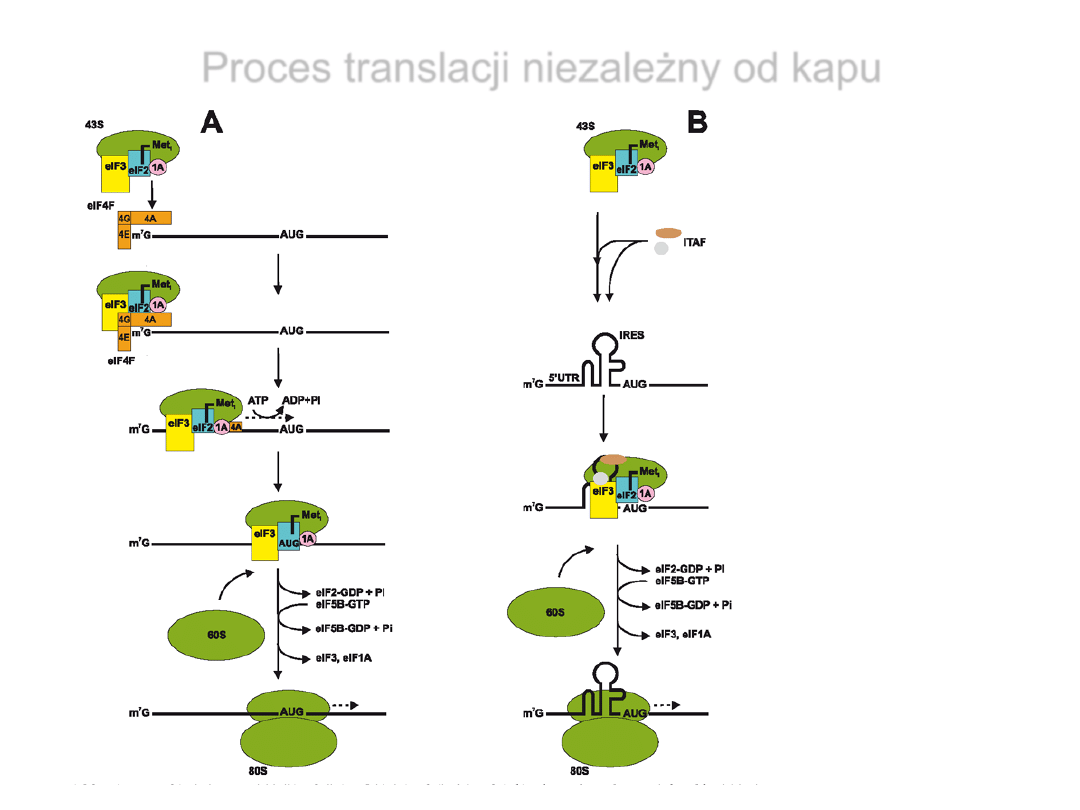
Proces translacji niezależny od kapu
Błaszczyk i wsp., 2007, Postępy Biochemii, 53, 400-409
Strategie wiązania 43S do
IRES:
-poprzez eIF4G i eIF4A
-
bezpośrednie
oddziaływanie stabilizowane
przez eIF3
- bez jakichkolwiek
czynników i tRNA
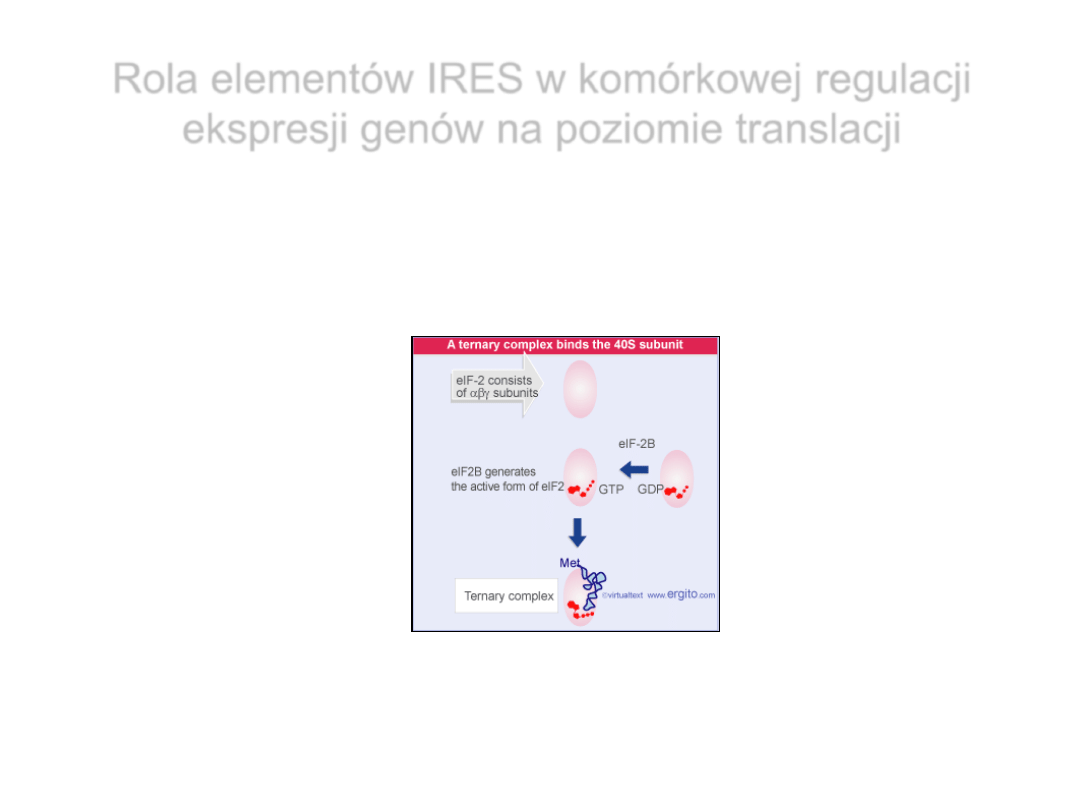
Rola elementów IRES w komórkowej regulacji
ekspresji genów na poziomie translacji
•
Obniżona aktywność czynników:
- eIF4E -
podczas apoptozy oraz przejścia z fazy G2 do mitozy
- eIF2
– warunki stresowe: głód, szok cieplny, promieniowanie UV, hypoksja,
stres endoplazmatyczny
Rożne komórkowe mRNA mają różne wymagania co do poziomu aktywnego
kompleksu eIF2-GTP-Met-tRNA
i
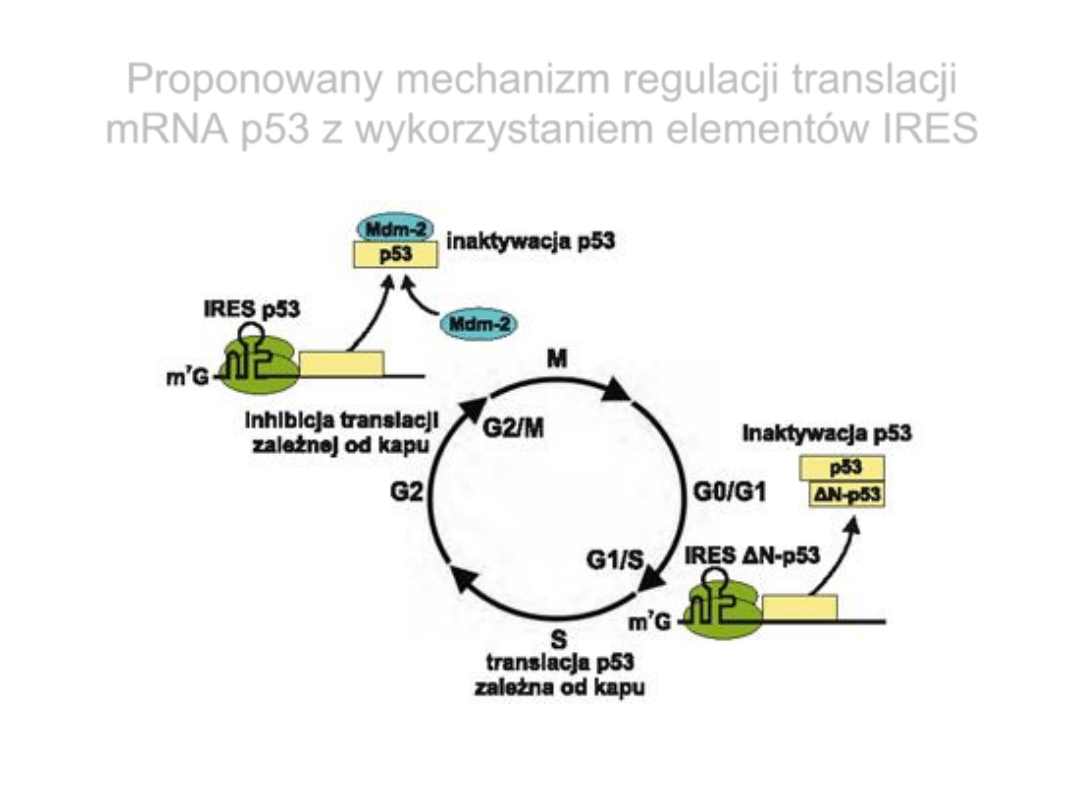
Proponowany mechanizm regulacji translacji
mRNA
p53 z wykorzystaniem elementów IRES
Błaszczyk i wsp., 2007, Postępy Biochemii, 53, 400-409
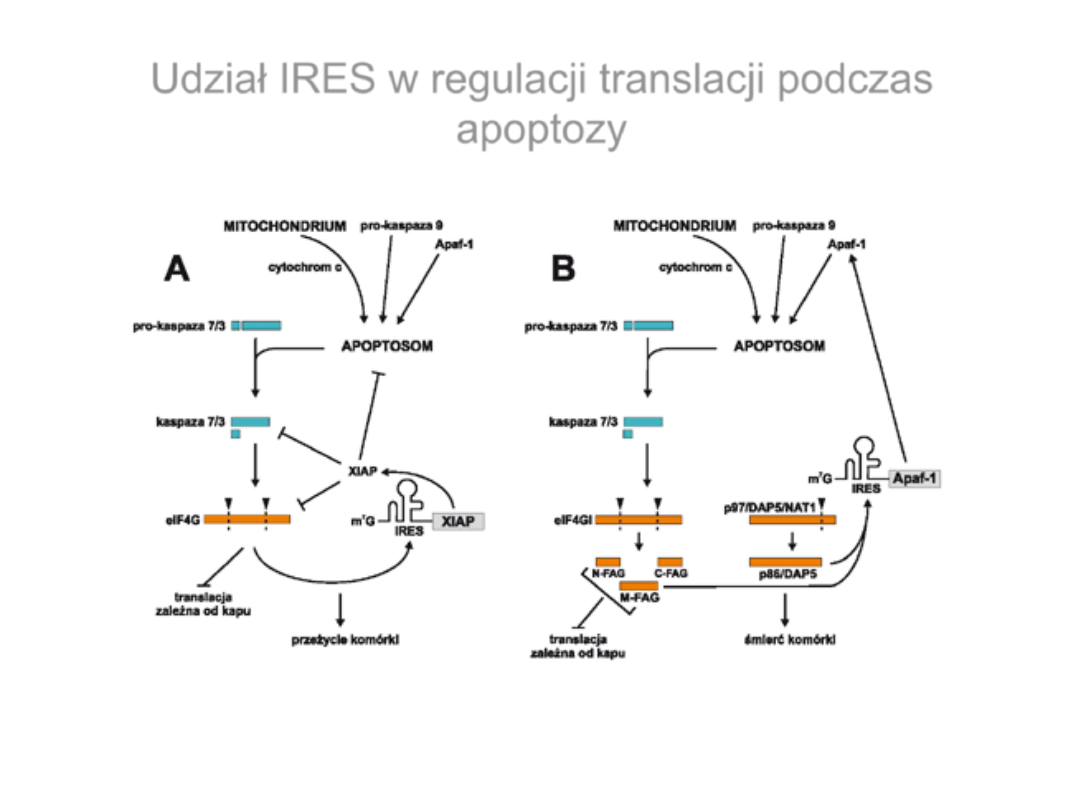
Udział IRES w regulacji translacji podczas
apoptozy
Błaszczyk i wsp., 2007, Postępy Biochemii, 53, 400-409

Udział IRES w regulacji procesu nowotworzenia
•
Istnieje grupa
onkogenów, które ulegają wewnętrznej inicjacji translacji:
członkowie rodziny genów myc, Bag-1, czynniki wzrostu VEGF,FGF-2
•
Szereg
czynników IRES funkcjonuje w warunkach rozwoju nowotworu
•
mRNA czynnika transkrypcyjnego HIF-1 zawiera element IRES,
który
funkcjonuje w warunkach
obniżonego poziomu tlenu w komórce
•
U chorych na szpiczaka plazmocytowego zidentyfikowano
mutację w genie
c-myc
(C→U) w regionie IRES. Mutacje zwiększa sześciokrotnie aktywność
IRES
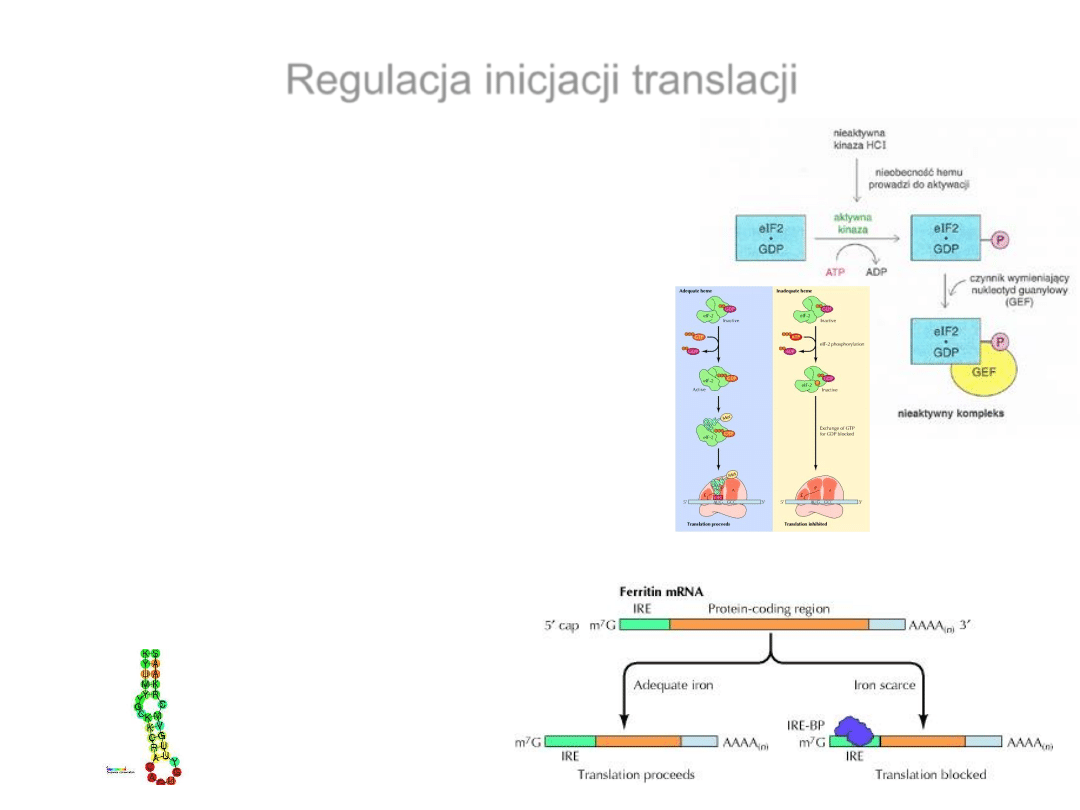
Regulacja inicjacji translacji
•
Regulacja ogólna – dotyczy zasadniczych zmian
w ilości syntetyzowanych białek i oddziałuje na
wszystkie mRNA w podobnym stopniu. Dotyczy
fosforylacji eIF_2, co powoduję represję inicjacji
translacji
•
Regulacja specyficzna-
dotyczy mechanizmów
działających na konkretny transkrypt albo małą
grupę transkryptów. Przykłady:
-
operony kodujące białka rybosomowe E. coli
- mRNA ferrytyny
Iron response element
Wyszukiwarka
Podobne podstrony:
2014 BPEG część 8 regulacja translacji
2014 BPEG część 6 elongacja translacji
2014 BPEG część 7 terminacja translacji
2014 BPEG część 1 mRNA i tRNA
2014 BPEG część 9 fałdowanie i modyfikacje białek
2014 BPEG część 3 syntetazy
2014 BPEG część 2 kod genetyczny
2014 BPEG część 4 rybosomy
Mydło - część tłumaczenie translatorem, Ekologia, Dom bez chemii
BPEG część 10 systemy degradacji i autofagia
Konsumpcja alkoholu i innych środków psychoaktywnych wśród studentów poszczególnych kierunków uniwer
Inzynieria materialowa czesc obliczeniowa, Elektrotechnika AGH, Semestr III zimowy 2013-2014, Inżyni
Podstawy statystyki i ekonometrii 2014 część 1
Inicjatywy lokalne 2014 regulam Nieznany
Podstawy statystyki i ekonometrii 2014 część 2
translacja inicjacja(1)
2014 06 test tech czesc II
egzamin zawodowy MGR 2014 czesc Nieznany
więcej podobnych podstron