
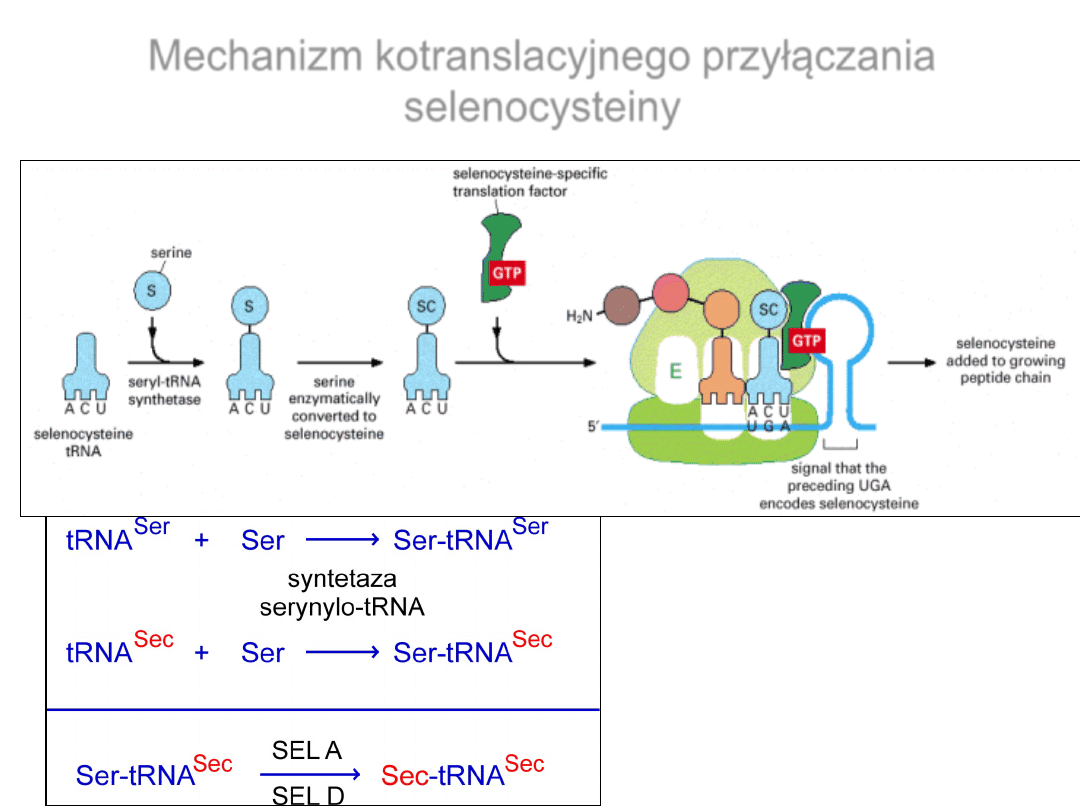
Cysteina H
S
-CH
2
-CH-COO
-
NH
2
Selenocysteina H
Se
-CH
2
-CH-COO
-
NH
2
Kodowanie selenocysteiny (Sec, U)

Kotranslacyjne włączenie selenocysteiny do
łańcucha polipeptydowego występuje u eukariota,
archea i bakterii.
Selenocysteina występuje w centrach aktywnych
enzymów oksydo-redukcyjnych
(np. peroksydazy glutationowej, dejodynazy).
•
Peroksydaza glutationowa redukuje cytotoksyczne nadtlenki.
•
Dejodynaza
redukuje tetrajodotyroninę (tyroksynę) do trójodotyroniny.
Kodowanie selenocysteiny
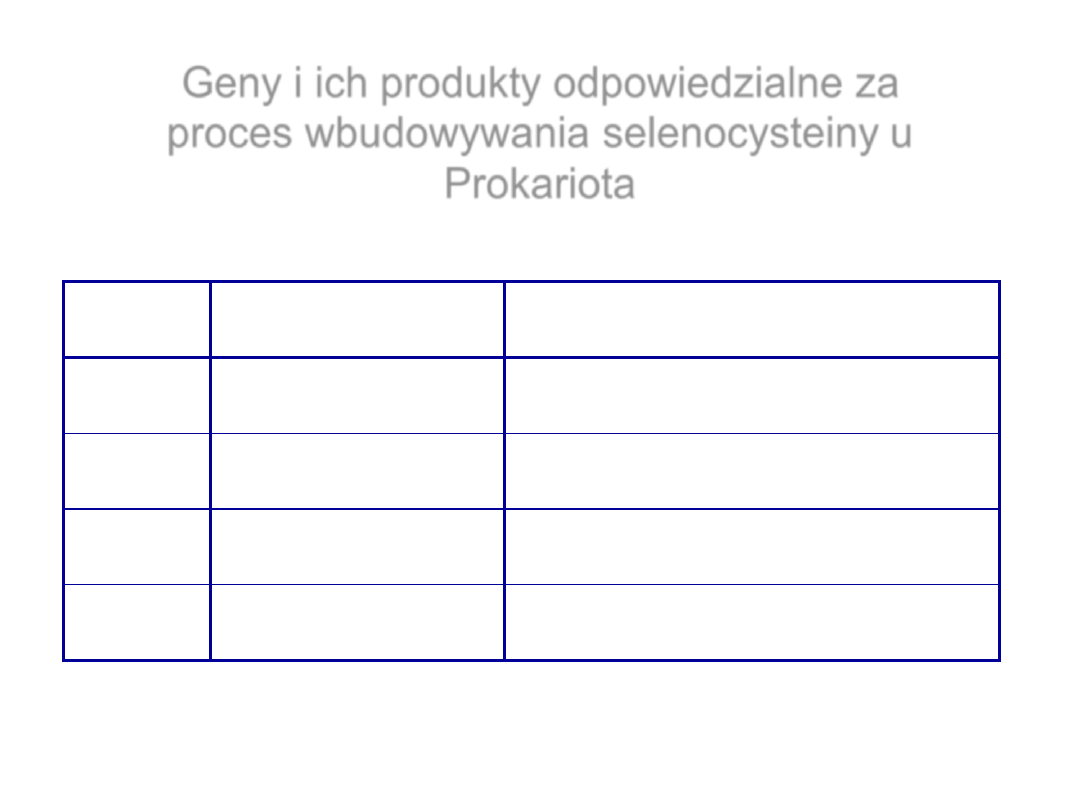
Geny i ich produkty odpowiedzialne za
proces wbudowywania selenocysteiny u
Prokariota
Gen
Produkt
Funkcja
selA
białko SEL A
ser-tRNA
→ sec-tRNA
selB
białko SEL B
czynnik elongacji
selC
tRNA
tRNA
selD
białko SEL D
ser-tRNA
→ sec-tRNA
SelB/ eEFSec
SECIS
Selenocysteine
Insertion Sequence
Mechanizm kotranslacyjnego
przyłączania
selenocysteiny
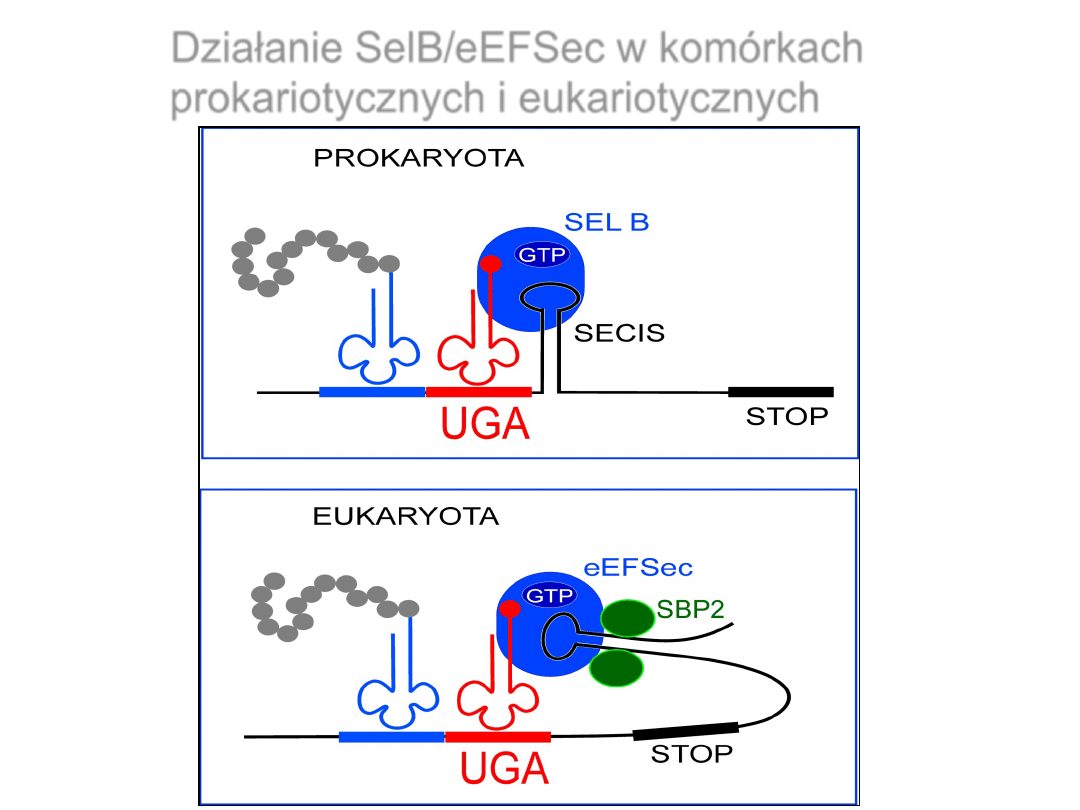
Działanie SelB/eEFSec w komórkach
prokariotycznych i eukariotycznych
Czteroliterowy kodon stop
a wbudowywanie selenocysteiny
E. coli:
UGAU
– wydajny terminator – ma wysokie powinowactwo do RF-2
UGAC
– ma
ło wydajny terminator – ma stosunkowo niskie powinowactwo
do RF-2
Kolejny nukleotyd po sekwencji UGA
sec
UGA
sec
C
EUKARYOTA:
UGA puryna
– wydajny terminator – wysokie powinowactwo do eRF-1.
UGA pirymidyna
– ma
ło wydajny terminator – stosunkowo niskie
powinowactwo do eRF-1.
Wi
ększość enzymów selenowych (ale nie wszystkie!) ma sekwencję UGA
pirymidyna
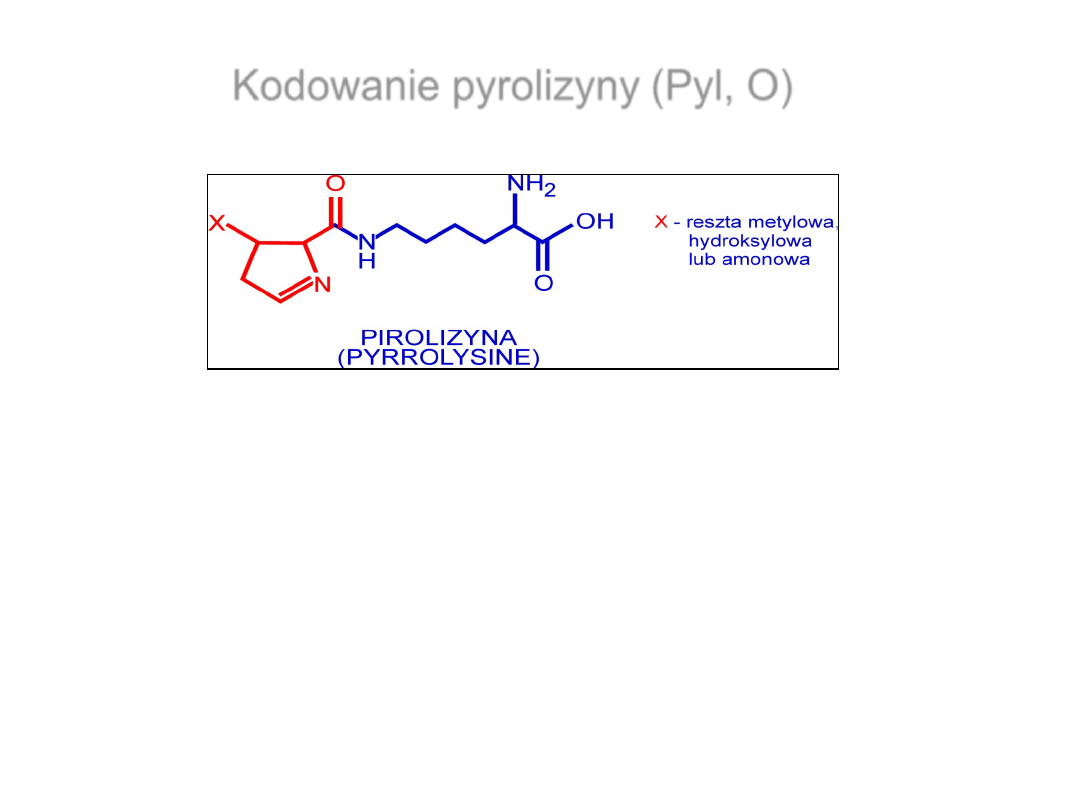
•
Aminokwas występujący w jednym białku: metylotransferazie
metyloaminowej w bakteriach Metanosarcina
należących do
Archebacteria.
•
Kodowany przez
UAG
•
W mRNA tej metylotransferazy znaleziono strukturę podobną
do struktury SECIS. Nazwano ją PYLIS.
Kodowanie pyrolizyny (Pyl, O)

Systemy kontroli jakości mRNA
•
Degradacja mRNA
niosących przedwczesny kodon stop,
nonsense-mediated decay (NMD)
•
Degradacja mRNA bez kodonu stop, non-stop decay
(NSD)
•
Degradacja mRNA
posiadających przeszkodę dla ruchu
rybosomu , no-go decay (NGD)
•
Degradacja
związana
z
nieprawidłową
budową
podjednostki 40S, non-functional 18S-rRNA decay, 18S-
NRD)

mRNA
podlegające procesowi NMD
•
mRNA z PTC (premature terminataion codon) wprowadzonymi przez
alternatywne składnie
•
mRNA selenoprotein, w przypadku których kodon dla selenocysteiny,
przy braku selenu odczytywany jest jako kodon PTC
•
mRNA podlegające nieprecyzyjnemu skanowaniu podczas inicjacji
translacji
•
mRNA posiadajace introny
•
mRNA z przesuniętą ramką odczytu
•
policistronowe mRNA
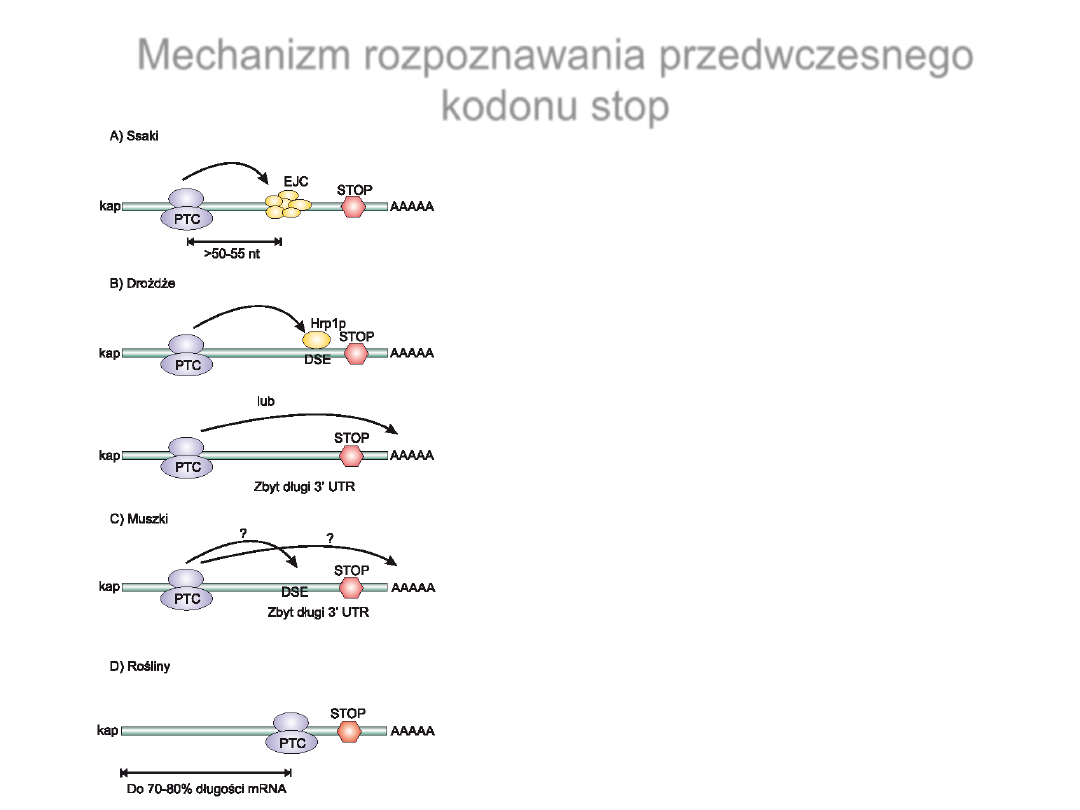
Mechanizm rozpoznawania przedwczesnego
kodonu stop
•
EJC – (exon-exon junction
complex) egzonowy kompleks
łącznikowy
•
DSE (downstream sequence
element): TGYYGATGYYYYY
(Y= T lub C)
Dzikiewicz A., Szweykowska-
Kulińska Z., Postępy Biochemii, 2006, 52,390-397
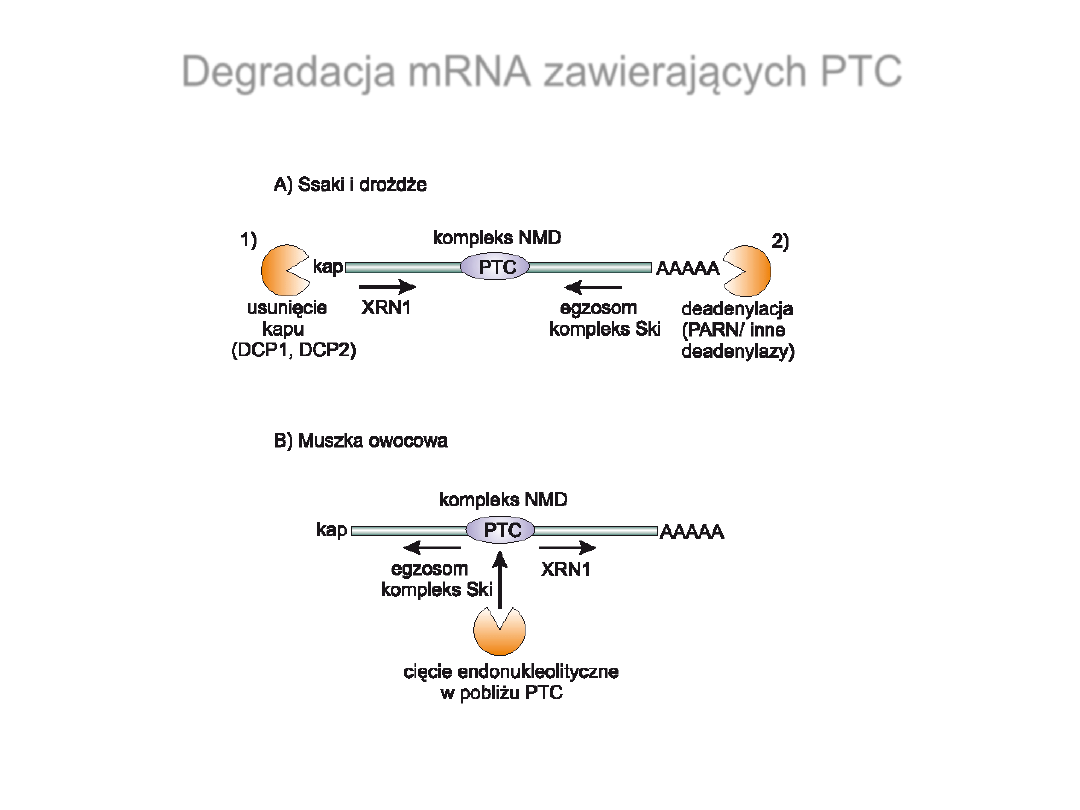
Degradacja mRNA
zawierających PTC
Dzikiewicz A., Szweykowska-
Kulińska Z., Postępy Biochemii, 2006, 52,390-397
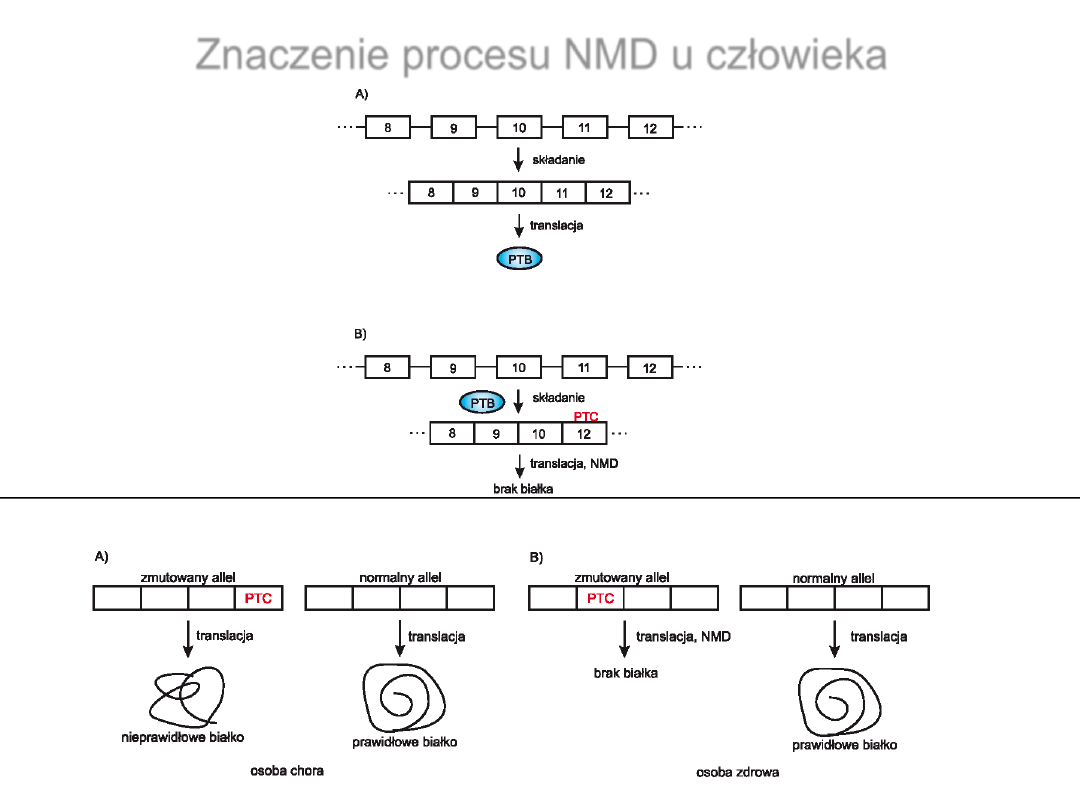
Znaczenie procesu NMD u człowieka
Białko wiążące się z traktem
polipirymidynowym (PTB) reguluje
poziom swojej ekspresji przez
alternatywne składanie pre-mRNA
i NMD
Położnie mutacji wprowadzającej kodon stop ma znaczenie dla fenotypu heterozygot
Dzikiewicz A., Szweykowska-
Kulińska Z., Postępy Biochemii, 2006, 52,390-397
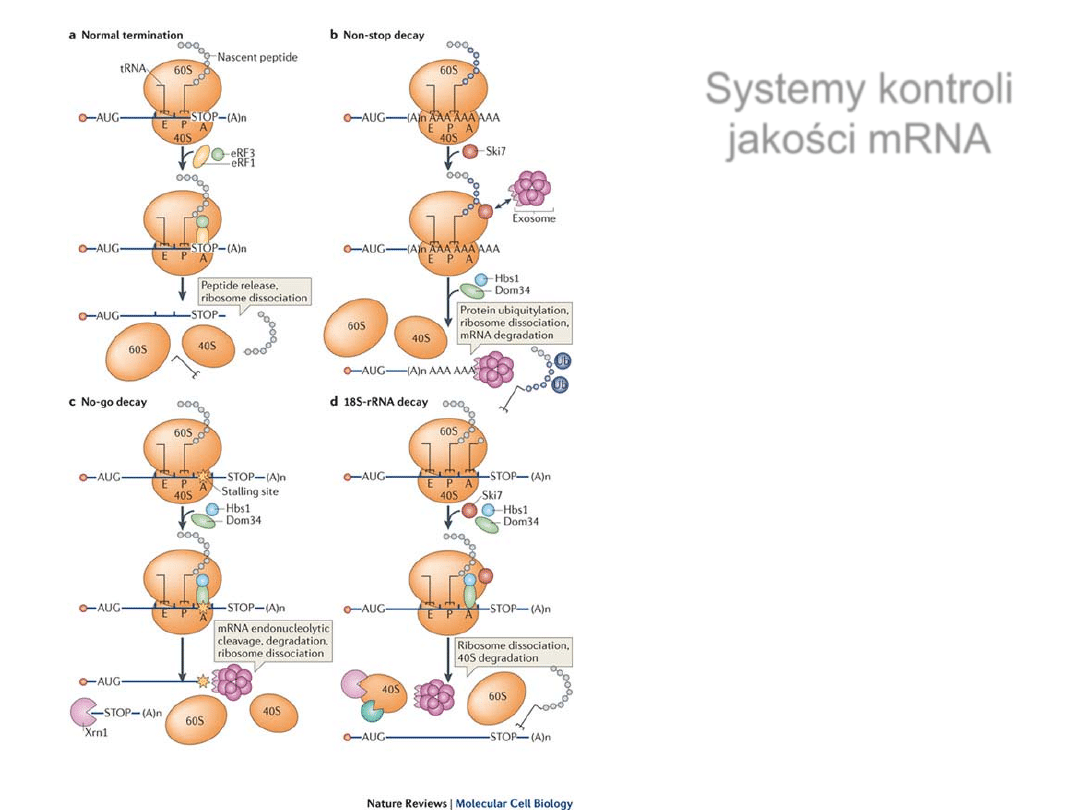
Systemy kontroli
jakości mRNA
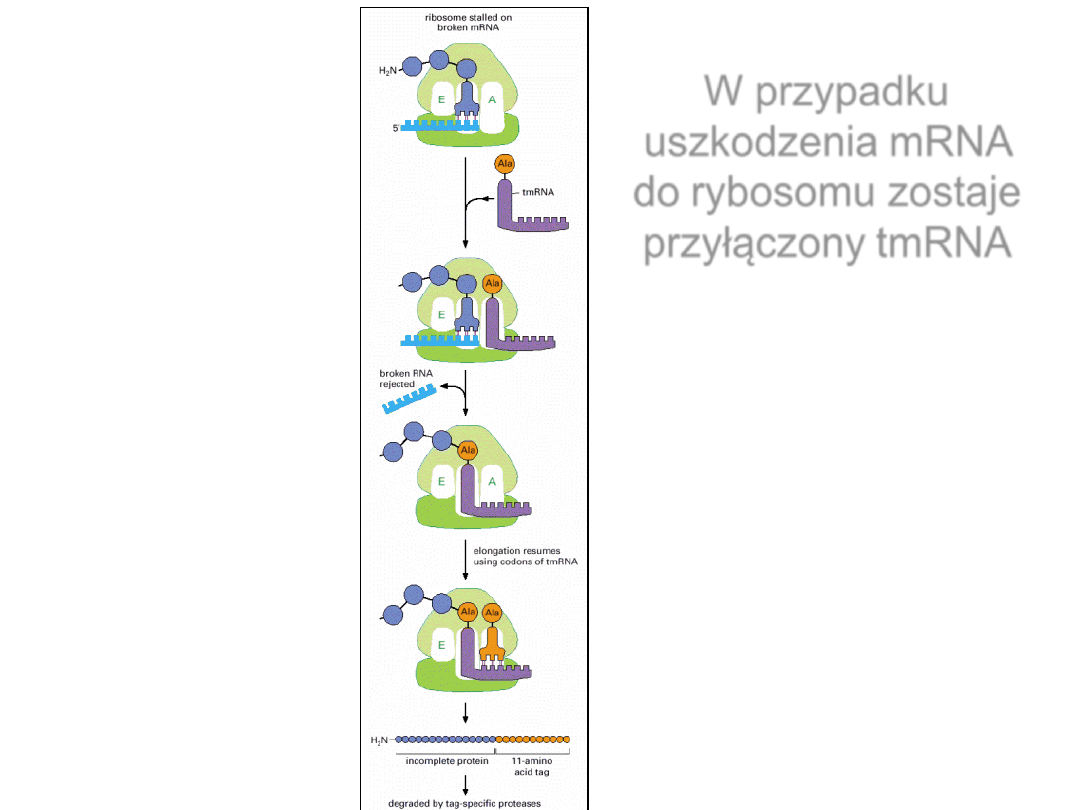
W przypadku
uszkodzenia mRNA
do rybosomu zostaje
przyłączony tmRNA
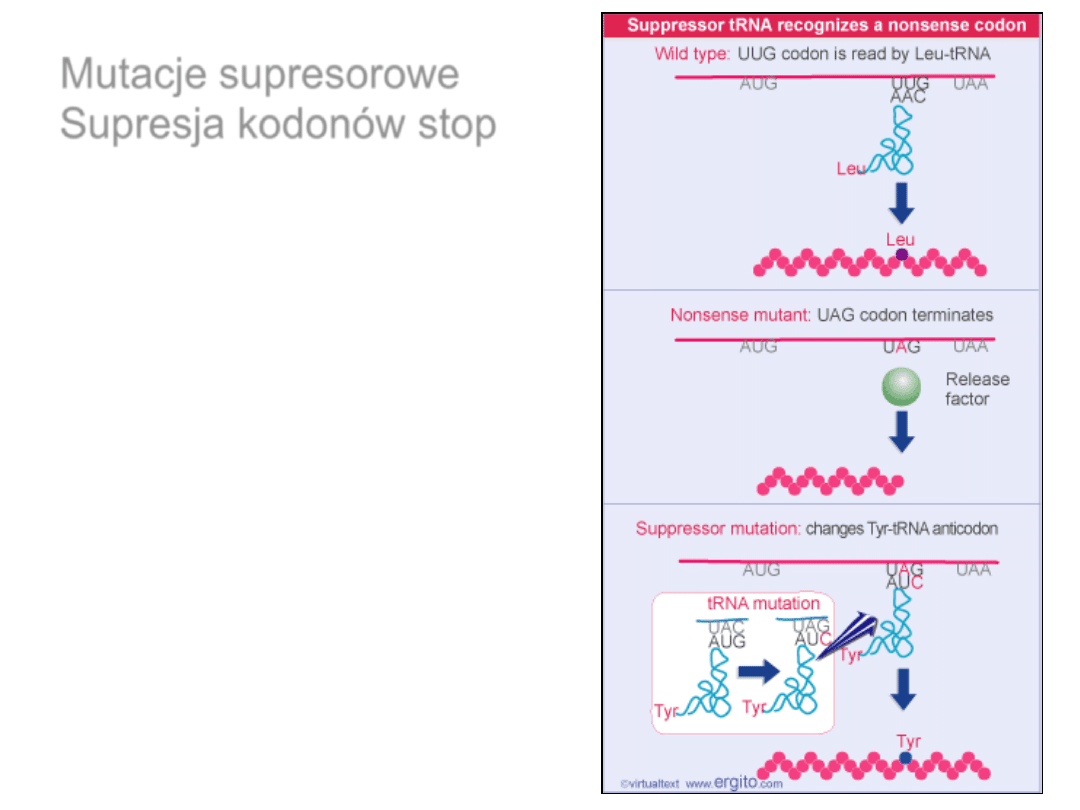
Mutacje supresorowe
Supresja kodonów stop
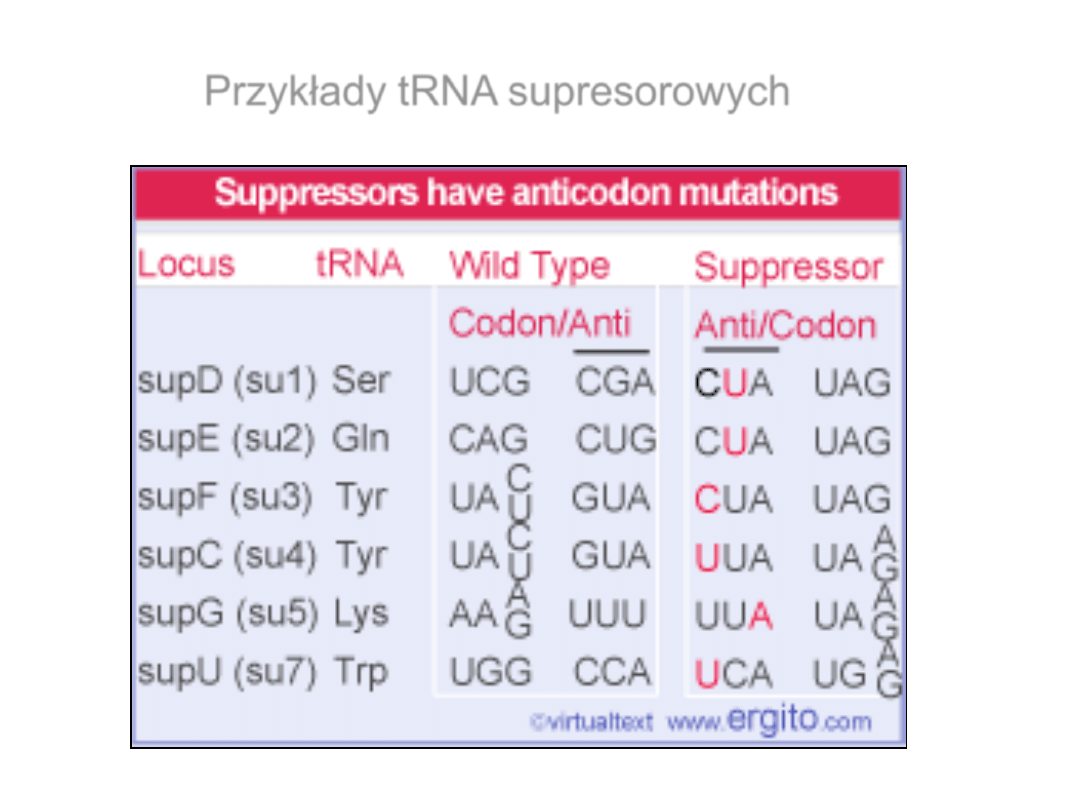
Przykłady tRNA supresorowych
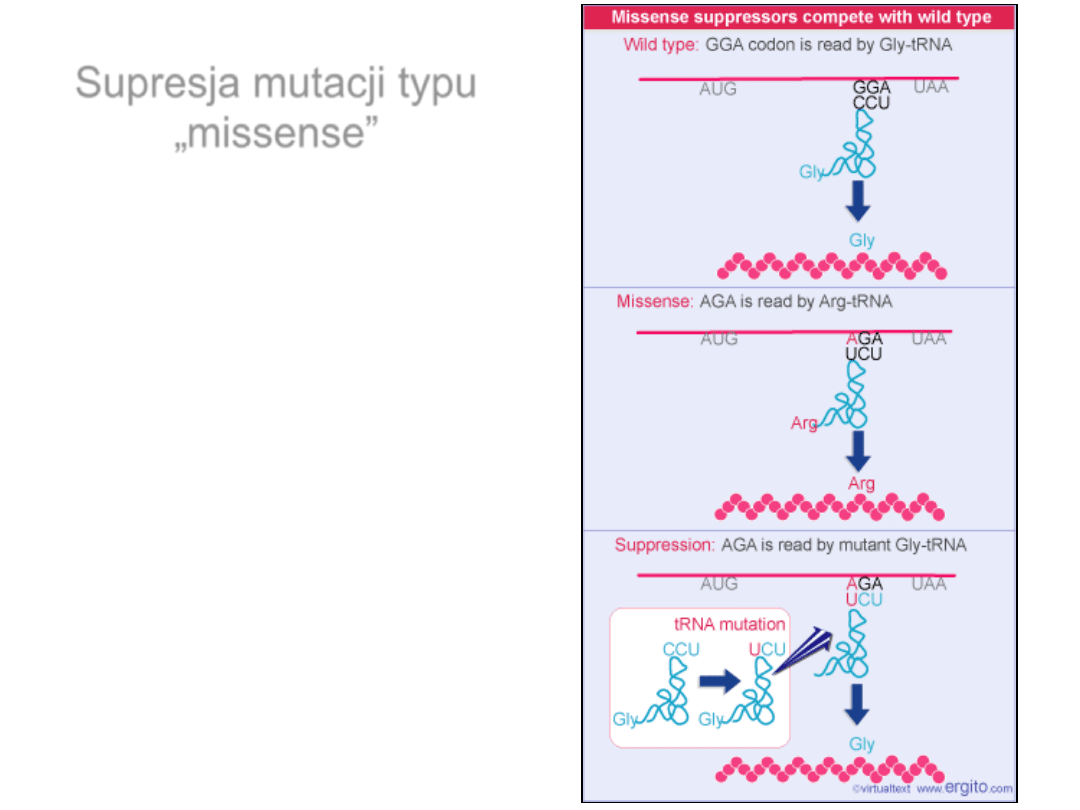
Supresja mutacji typu
„missense”
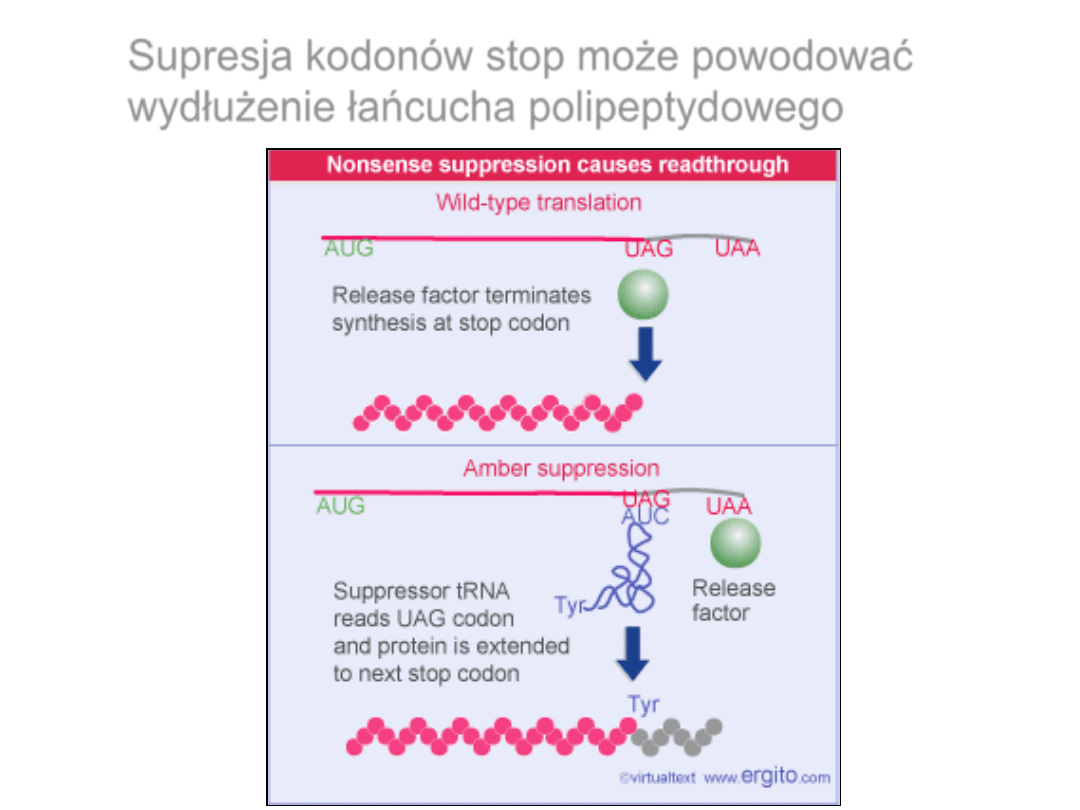
Supresja kodonów stop może powodować
wydłużenie łańcucha polipeptydowego

Supresja mutacji nonsensownych w E.coli
•
Wydajność supresji UAG – 10-50%.
Supresja z taką wydajnością wszystkich kodonów
terminacyjnych byłaby letalna; UAG występuje rzadko.
•
Wydajność supresji UAA najwyżej 10%; UAA jest
najczęściej używanym kodonem terminacyjnym.
•
UGA jest mało efektywnym kodonem terminacyjnym. W
1-3% przypadków jest odczytywany jako UGG (Trp).
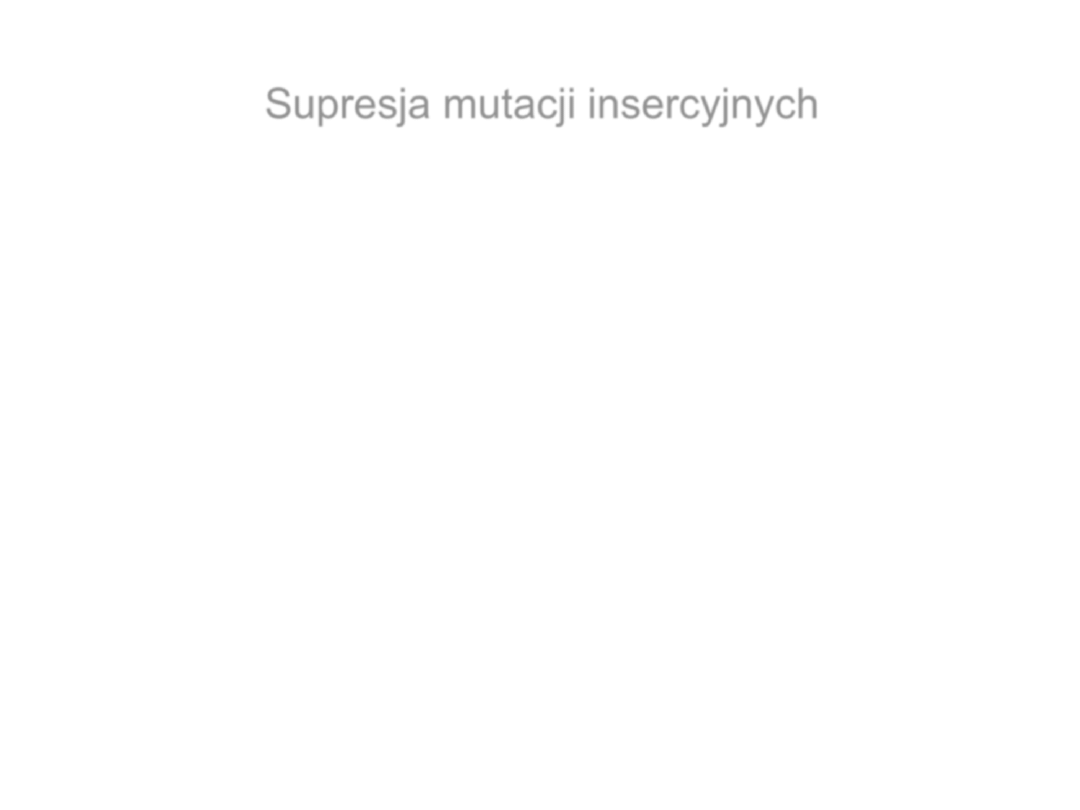
Supresja mutacji insercyjnych
Suf D (S. typhimurium)
Kodon glicyny 5’ GGG 3’
→ 5’ GGGG 3’
Antykodon supresorowego tRNA
Gly
5’CCCC 3’
Suf-
2 (drożdże)
Kodon 5’ CCCU 3’
Antykodon supresorowy tRNA
Pro
5’ AGGG 3’
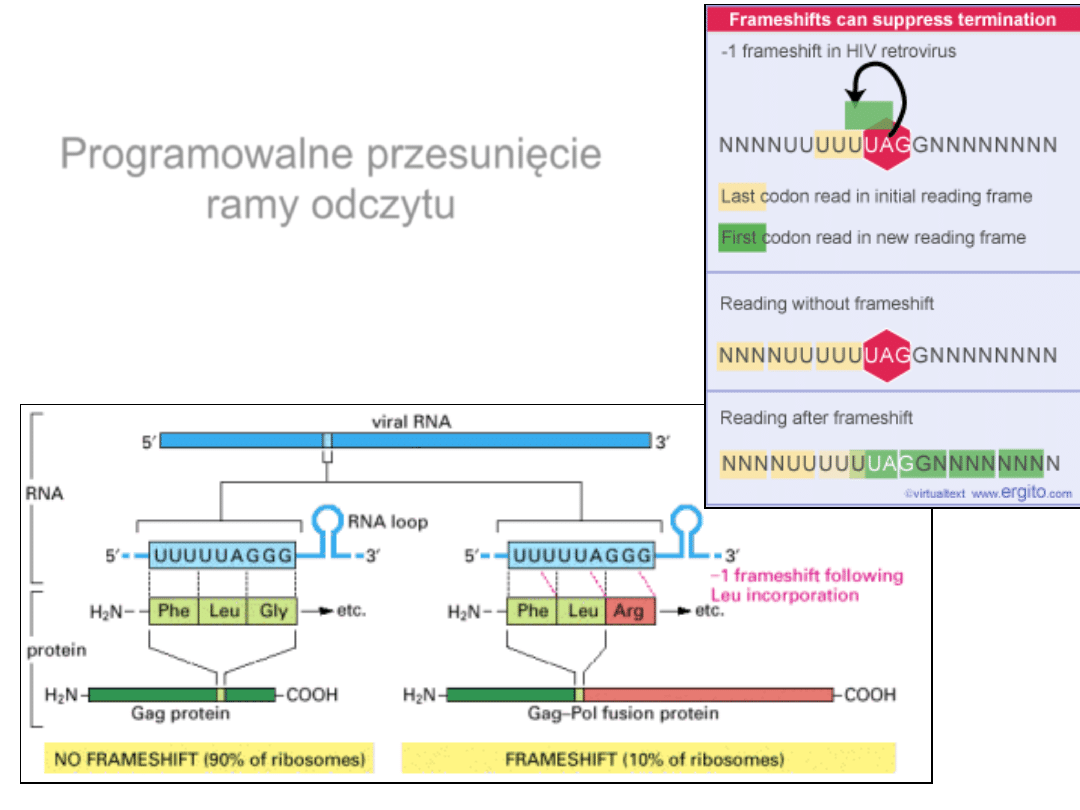
Programowalne przesunięcie
ramy odczytu
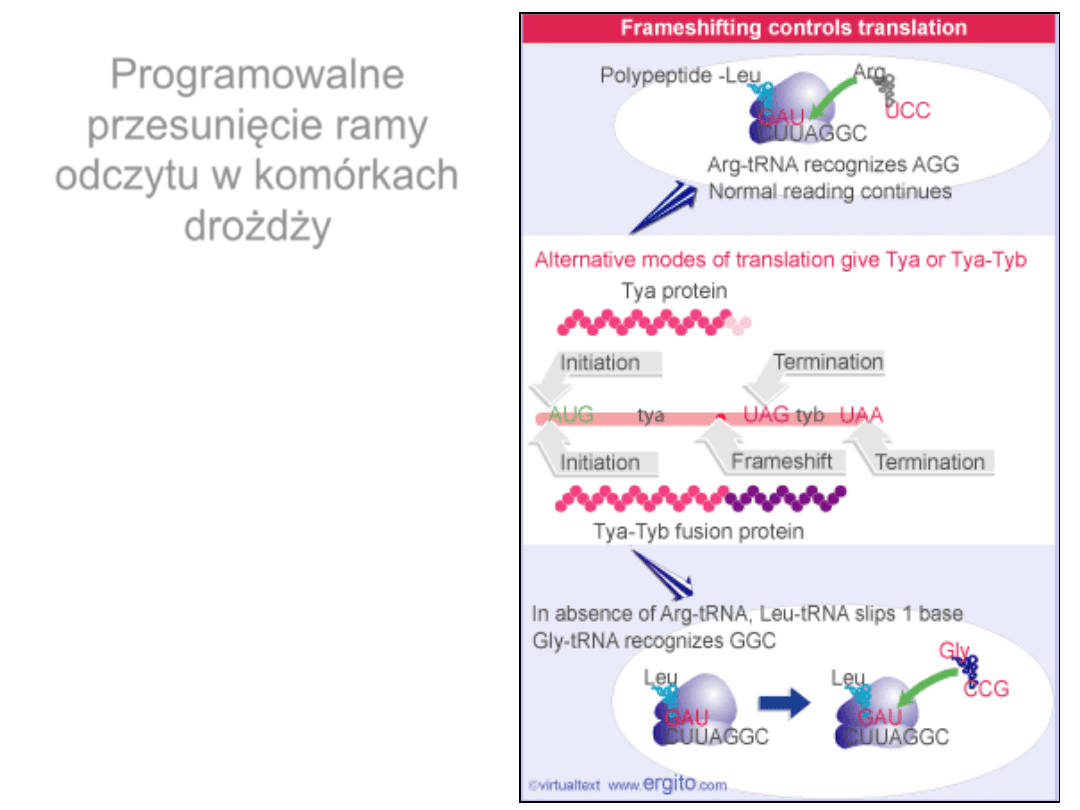
Programowalne
przesunięcie ramy
odczytu w komórkach
drożdży
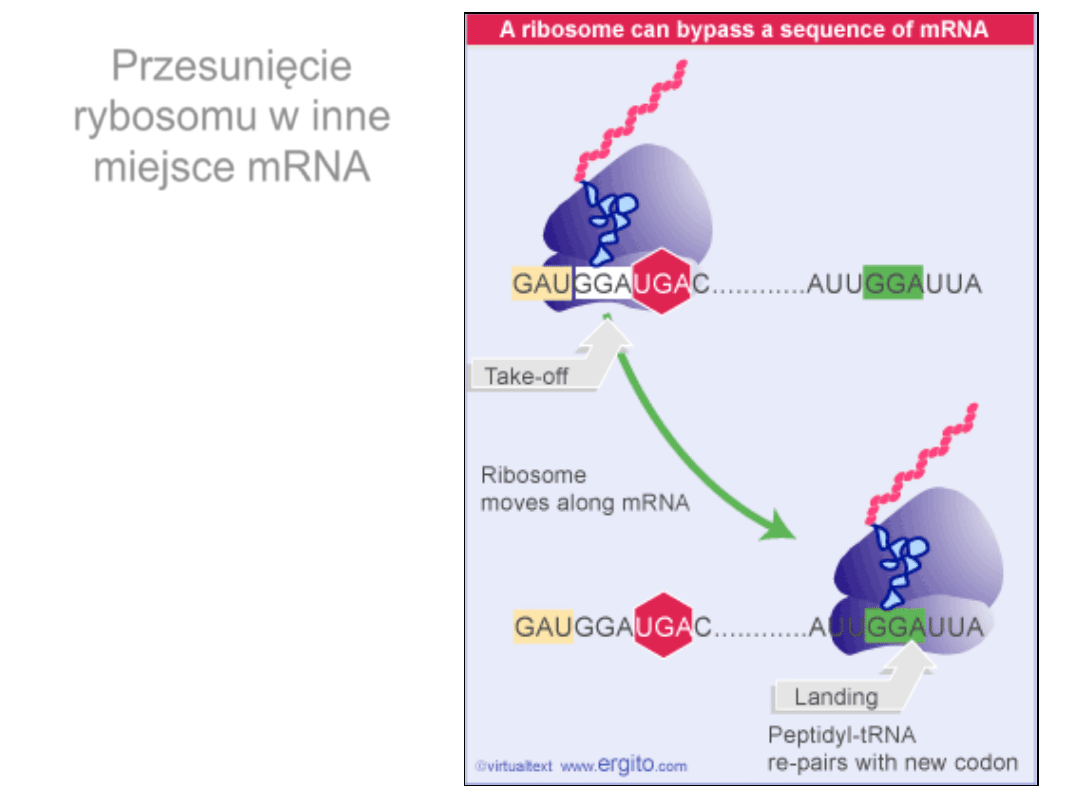
Przesunięcie
rybosomu w inne
miejsce mRNA
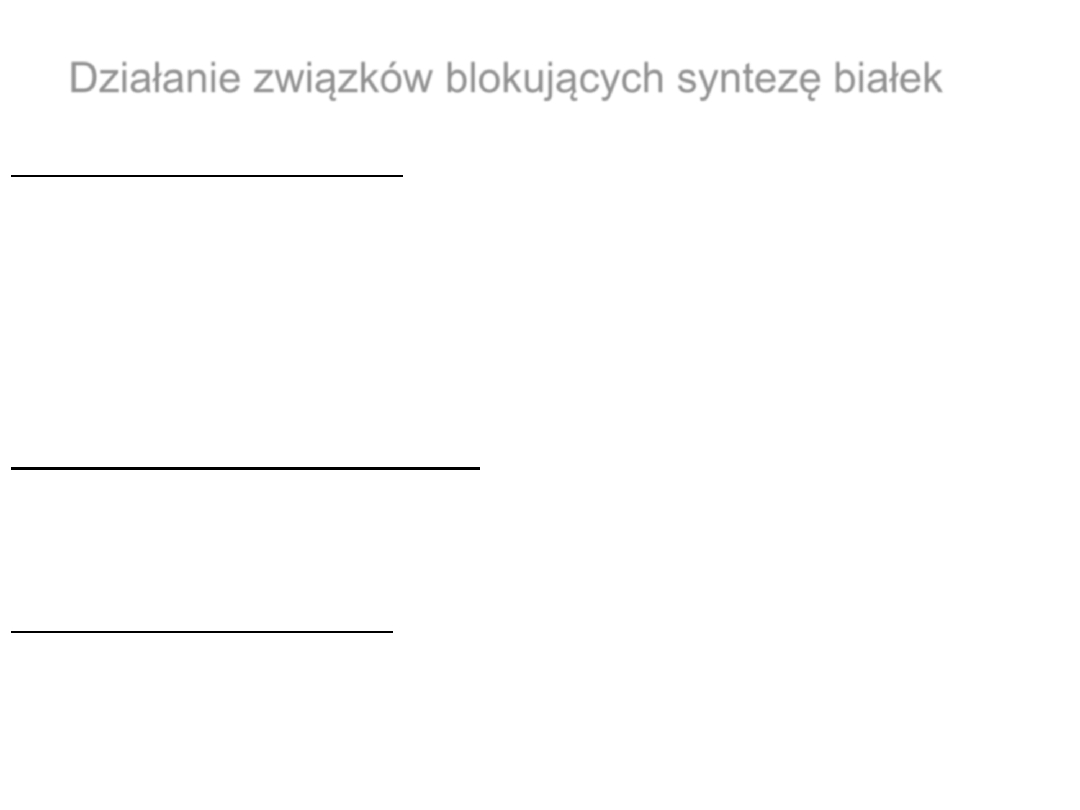
Działanie związków blokujących syntezę białek
Działające tylko na prokariota
Tetracyklina
Blokuje związanie aminoacylo-tRNA do miejsca A rybosomu
Streptomycyna
Blokuje formowanie formylometionylo-tRNA,
też błędne odczytywanie kodonów
Chloramfenikol
Blokuje aktywność transferazy peptydylowej
Erytromycyna
Blokuje translokację rybosmu
Działające na prokariota i eukariota
Puromycyna
Powoduje przedwczesne uwolnienie łańcucha polipeptydowego
Działające tylko na eukariota
Cykloheksamid
Blokuje translokację rybosmu
Anizomycyna
Blokuje aktywność transferazy peptydylowej
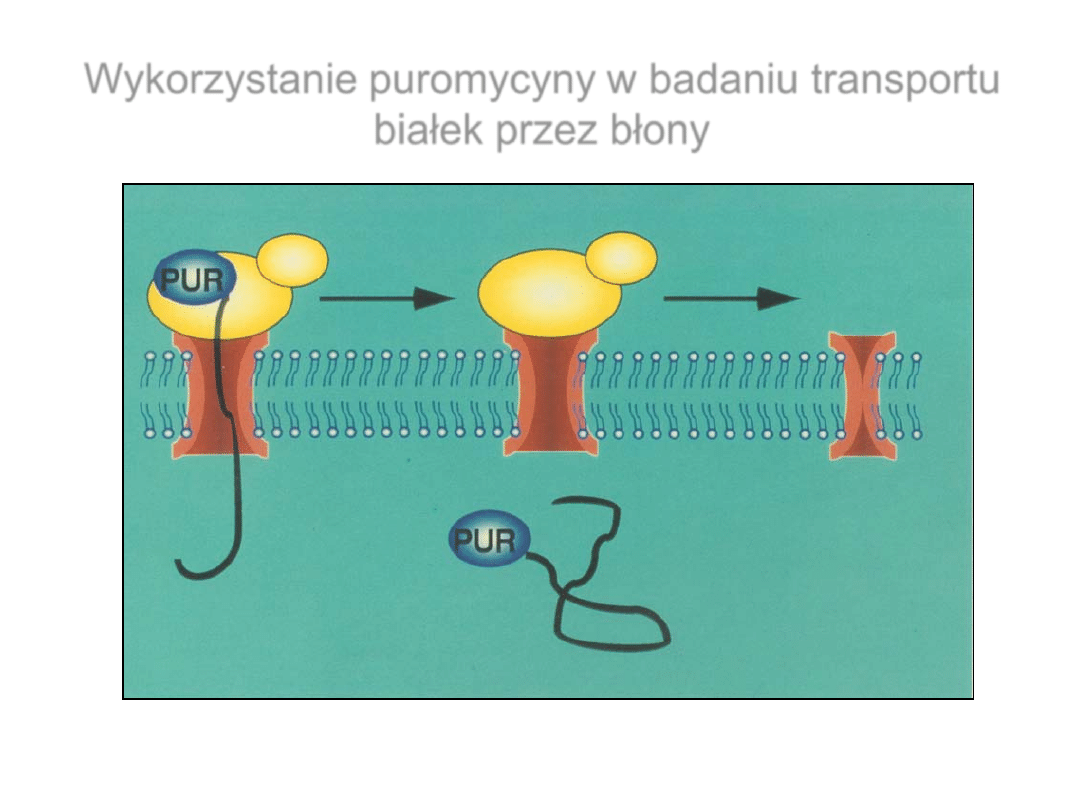
Wykorzystanie puromycyny w badaniu transportu
białek przez błony
Cell (1991) 65

Toksyny wpływające na zahamowanie procesu
translacji
Rodzaj toksyny
Aktywność
enzymatyczna
Cel komórkowy
Toksyna błonicy
(Corynebacterium diphteriae)
transferaza ADP-
rybozylowa
EF-2
Egzotoksyna A
(Pseudomonoas aeruginosa)
transferaza ADP-
rybozylowa
EF-2
Rycyna
(Ricinus communis)
N-glikozydaza
28S rRNA
Toksyny Shiga
Shigella dysenteriae
E.coli. EHEC 0157:H7
N-glikozydaza
28 rRNA
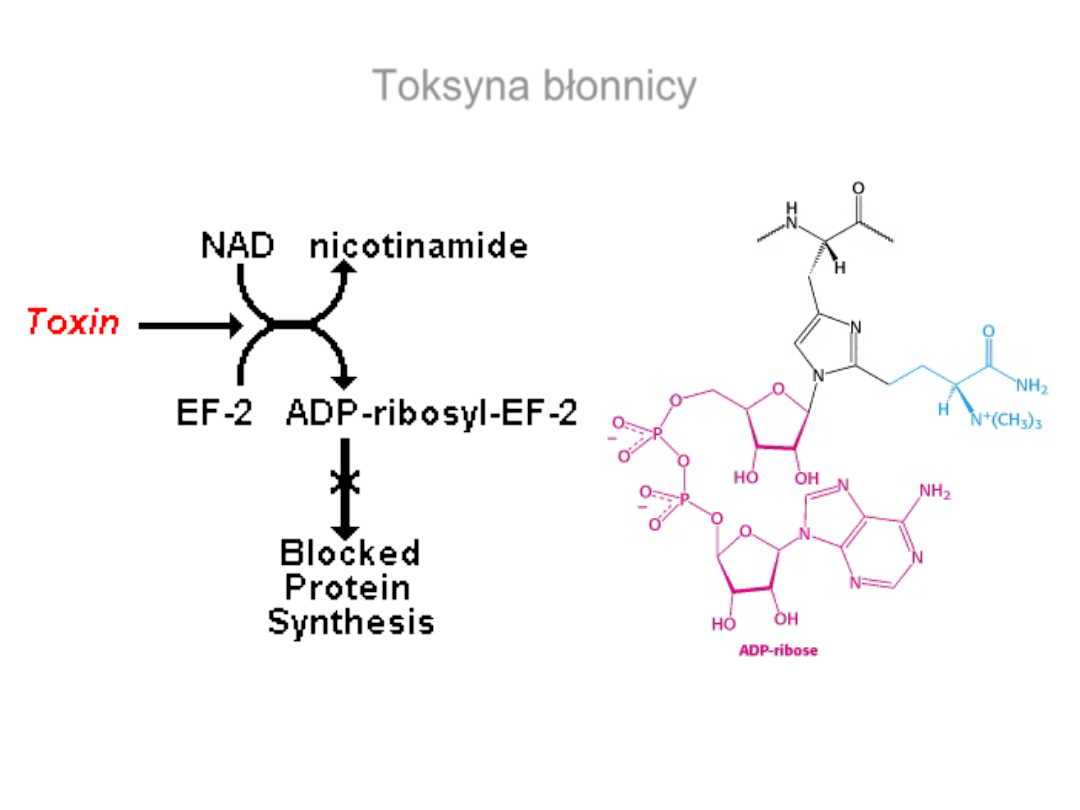
Toksyna
błonnicy
dyftamid
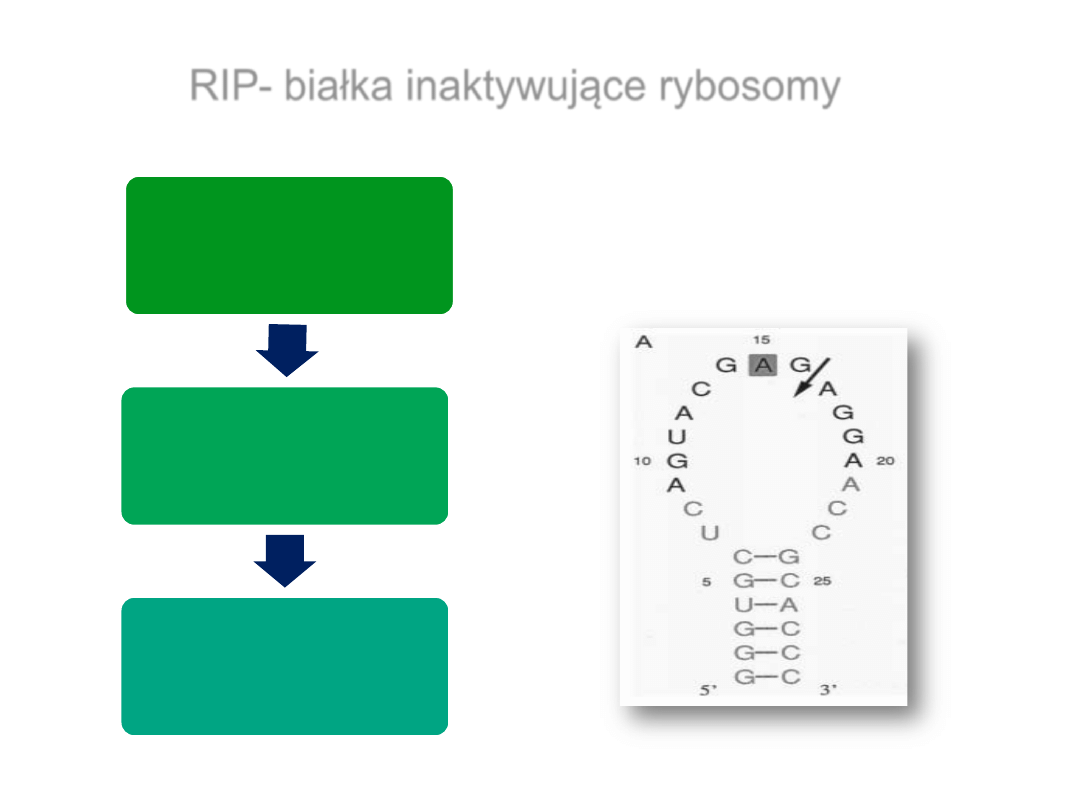
RIP-
białka inaktywujące rybosomy
4324
pętla sarcyny/rycyny 28S rRNA szczura (wg
Szewczak i Moore,1993)
Pętla
sarcyny/rycyny
–
zawiera
miejsce
wiązania
eukariotycznego
czynnika elongacji 1 i 2 (eEF-1 i eEF-2)
Depurynacja w 28S
RNA
Niemożliwe
przyłączenie czynników
translacyjnych do
rybosomu
Zahamowanie translacji
białek
Document Outline
- Slide Number 1
- Slide Number 2
- Slide Number 3
- Slide Number 4
- Slide Number 5
- Slide Number 6
- Slide Number 7
- Systemy kontroli jakości mRNA
- mRNA podlegające procesowi NMD
- Mechanizm rozpoznawania przedwczesnego kodonu stop
- Degradacja mRNA zawierających PTC
- Znaczenie procesu NMD u człowieka
- Systemy kontroli jakości mRNA
- Slide Number 14
- Slide Number 15
- Slide Number 16
- Slide Number 17
- Slide Number 18
- Supresja mutacji nonsensownych w E.coli
- Supresja mutacji insercyjnych
- Programowalne przesunięcie ramy odczytu
- Slide Number 22
- Slide Number 23
- Slide Number 24
- Wykorzystanie puromycyny w badaniu transportu białek przez błony
- Toksyny wpływające na zahamowanie procesu translacji
- Toksyna błonnicy
- RIP- białka inaktywujące rybosomy
Wyszukiwarka
Podobne podstrony:
2014 BPEG część 5 inicjacja translacji
2014 BPEG część 6 elongacja translacji
2014 BPEG część 7 terminacja translacji
2014 BPEG część 1 mRNA i tRNA
2014 BPEG część 9 fałdowanie i modyfikacje białek
2014 BPEG część 3 syntetazy
2014 BPEG część 2 kod genetyczny
2014 BPEG część 4 rybosomy
Podstawy Automatyki Lab 2014 CW3 Badania regulatora dwupołożeniowego
B molekularna wykład regulacja translacji i stabilności mRNA Kopia
Mydło - część tłumaczenie translatorem, Ekologia, Dom bez chemii
BPEG część 10 systemy degradacji i autofagia
33 Przebieg i regulacja procesu translacji
2 regulacja napiecia modelu transformator zaczepy, aaa, studia 22.10.2014, Materiały od Piotra cukro
1 regulacje prawne w zakresie bhp czesc 1
Inzynieria materialowa czesc obliczeniowa, Elektrotechnika AGH, Semestr III zimowy 2013-2014, Inżyni
Enzymy chemiczne regulatory reakcji część II
Komputer w układzie regulacji część I Cw5
więcej podobnych podstron