Wykład Bioinformatyka
Wykład 5, 2008
1
Bioinformatyka
Wykład 5
E. Banachowicz
Zakład Biofizyki Molekularnej
IF UAM
1
http://www.amu.edu.pl/~ewas
Porównywanie sekwencji
• Pierwsze pytanie biologa molekularnego,
kiedy odkryje nową sekwencję:
Czy w bazie sekwencji są już sekwencje podobne
do mojej?
sekwencje są identyczne –
nic nowego….
sekwencja jest podobna (ma „krewnych”)
– nowy członek
znanej rodziny
sekwencja ma kilka podobnych regionów, motywów lub
domen
– można zaproponować funkję
Nie ma znaczącego podobieństwa
– dużo pracy…..
Wykład Bioinformatyka
Wykład 5, 2008
2
Porównywanie sekwencji
• Celem porównania białek jest między
innymi przypisanie informacji znanej dla
jednej cząsteczki drugiej cząsteczce
Algorytmy
• macierze punktowe (DotPlot)
• programowanie dynamiczne
• metody heurystyczne (BLAST, FASTA)
• metody statystyczne (modele Markova,
statystyka Bayesa)
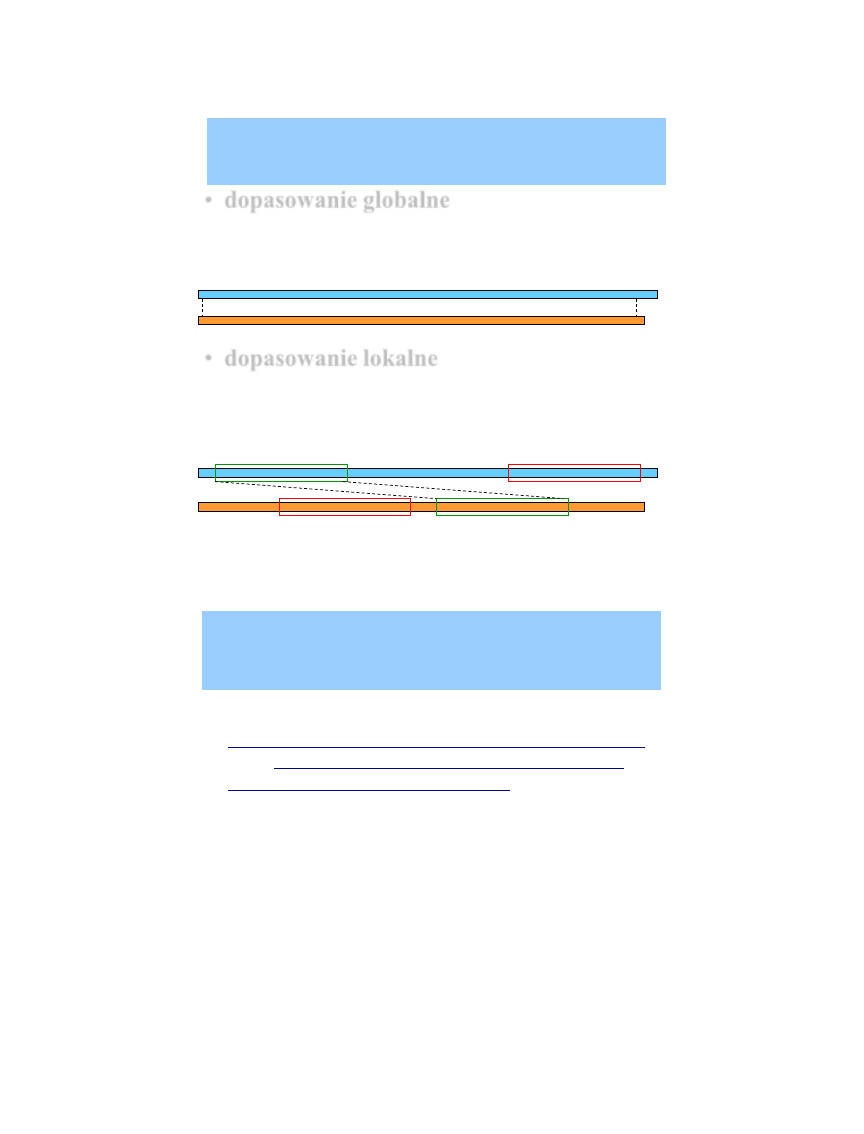
Rodzaje dopasowań
• pokrycie sekwencji
– globalne, lokalne
• liczba sekwencji porównywanych
– para (pairwise sequence alignment)
– więcej niż dwie (multiple sequences alignment)
Wykład Bioinformatyka
Wykład 5, 2008
3
Pokrycie sekwencji
• dopasowanie globalne – dopasowanie
wzdłuż całej sekwencji
(zastosowanie: do białek
składających się z pojedynczej domeny lub
homologicznych słabo zróżnicowanych)
• dopasowanie lokalne – uwzględnia
domenową naturę białek, szuka
subsekwencji (
zastosowanie: do białek
wielodomenowych, mRNA z sekwencją genomową
)
5
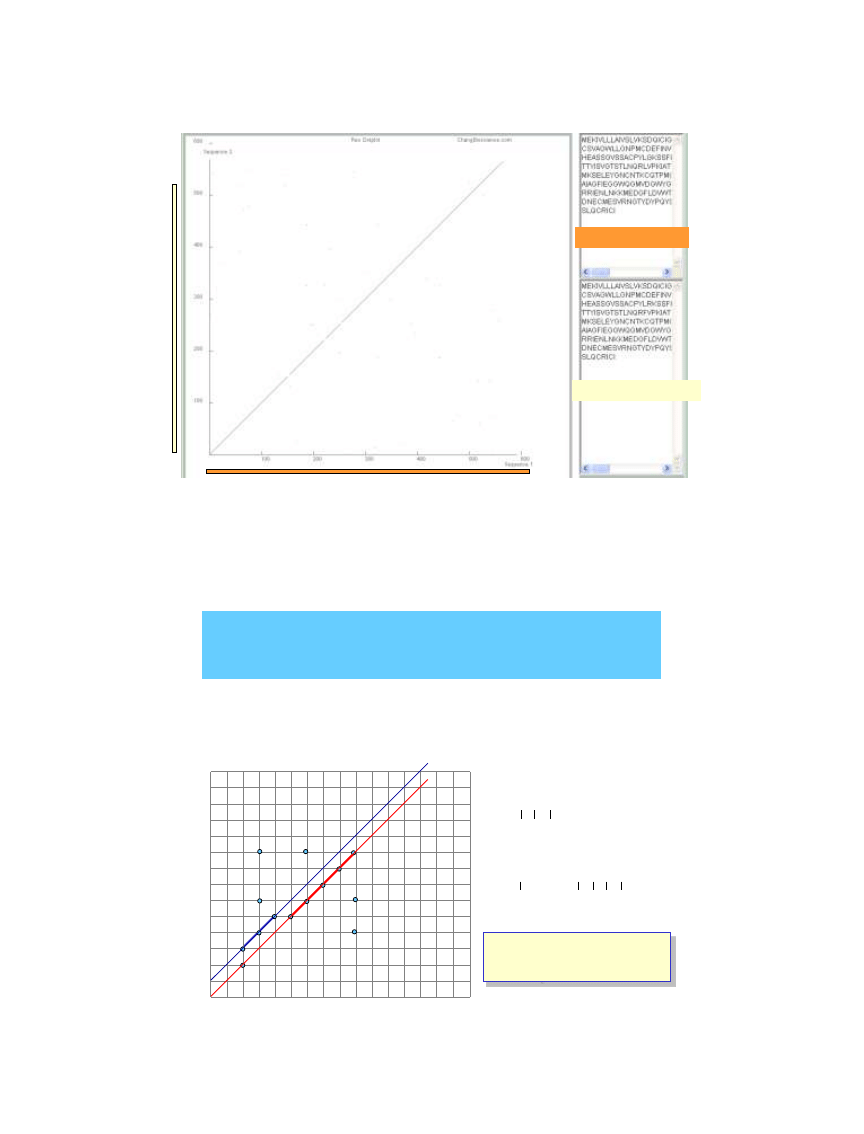
Macierze punktowe (DotPlot)
•
http://www.biophys.uni-duesseldorf.de/local/DOTPLOT/dotplot.html
• Dotter:
http://www.cgb.ki.se/cgb/groups/sonnhammer/Dotter.html
•
http://www.changbioscience.com/res/resd.html
• wykrywają fragmenty powtarzalne i rearanżacje
• opierają się na ocenie wizualnej – czasami skuteczniejsze niż
alignment
Wykład Bioinformatyka
Wykład 5, 2008
4
H5N1 (HA, swan)
H5N1 (HA, chicken)
Dotter
• niektóre układy punktów tworzą ścieżkę
• każda ścieżka odpowiada jednemu dopasowaniu
8
M Y S E Q U E N C E
H
I
S
S
E
Q
E
N
C
E
M Y
S E Q
U E N C E
H I S
S E Q
E N C E
M Y
S
E Q U
E N C E
H I
S
S E Q
E N C E
znaleźć najlepszą
ścieżkę!
Wykład Bioinformatyka
Wykład 5, 2008
5
Liniowe zestawienie dwóch sekwencji
Pairwise Sequence Alignment
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
sekwencja 2
sekwencja 1
ClareSansom
Punktacja dla białek jest bardziej złożona – 20 aminokwasów
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
6
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
ClareSansom
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
7
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
ClareSansom
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
ClareSansom

Wykład Bioinformatyka
Wykład 5, 2008
8
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
Insercja/Delecja => GAPS
zestawienie – jeden-do-jeden
przerwy odzwierciedlają wydarzenia ewolucyjne
(wyzwanie dla programistów)
ClareSansom
ABEERNALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
9
ABEERN-ALEDLAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFGSTOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
10
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFG-STOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFG--STOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
11
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFG---STOUTFAWATERM
Insercja/Delecja => GAPS
ClareSansom
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFG---STOUTFA-WATERM
Insercja/Delecja => GAPS
ClareSansom
Wykład Bioinformatyka
Wykład 5, 2008
12
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAFG---STOUTFA--WATERM
Insercja/Delecja => GAPS
ClareSansom
ABEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAF-G--STOUTFA--WATERM
G<->W = -7
G<->G = +5
Insercja/Delecja => GAPS
ClareSansom

Wykład Bioinformatyka
Wykład 5, 2008
13
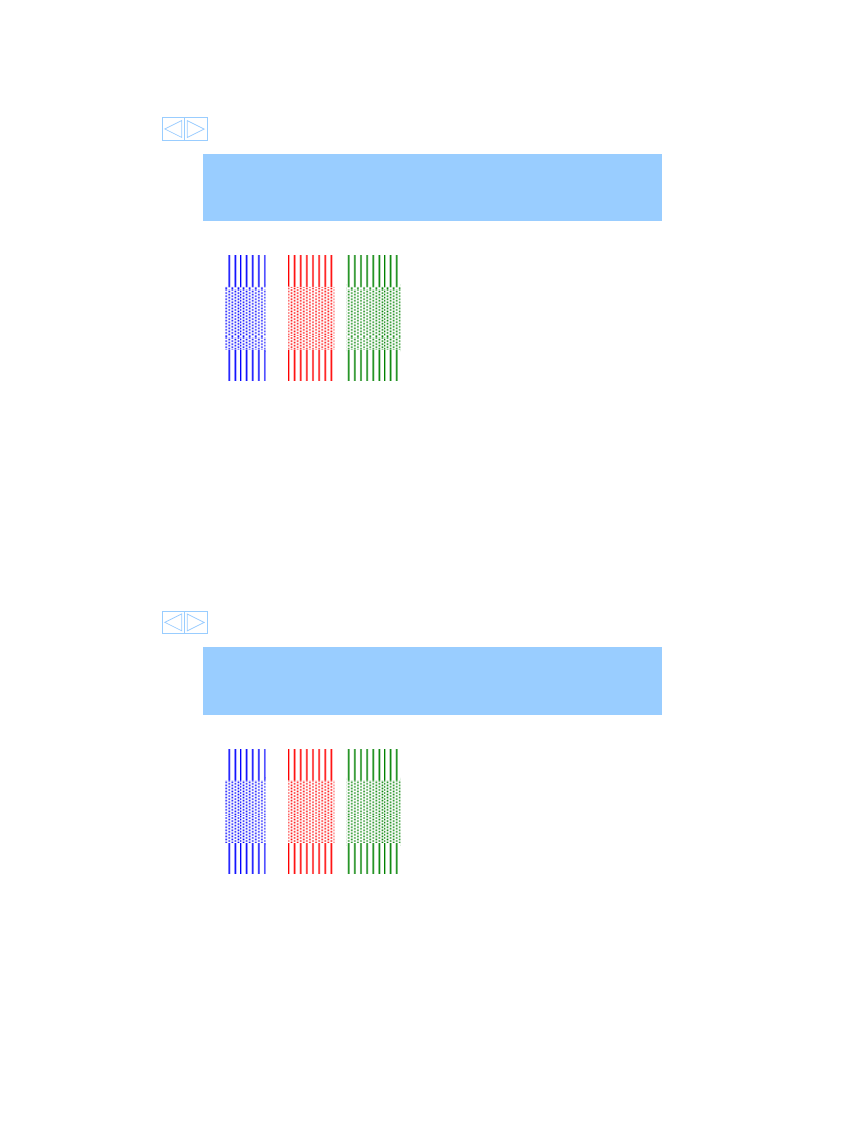
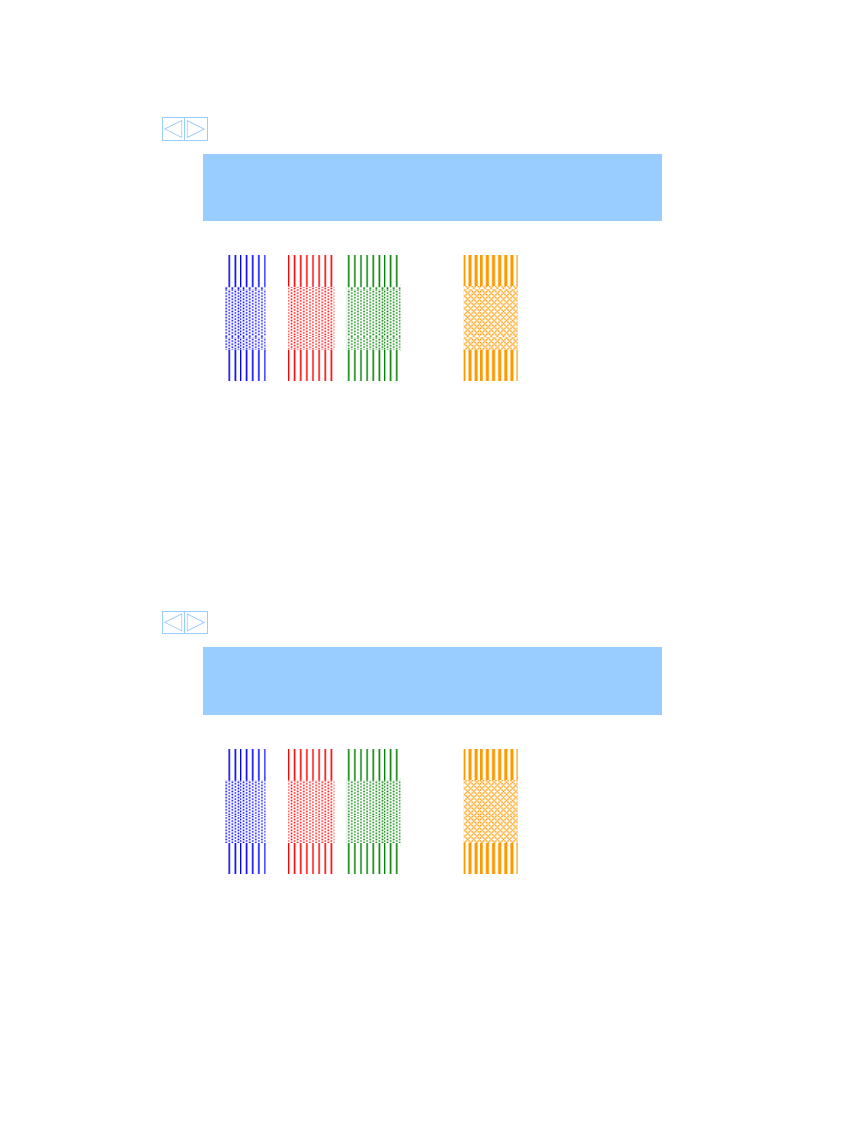
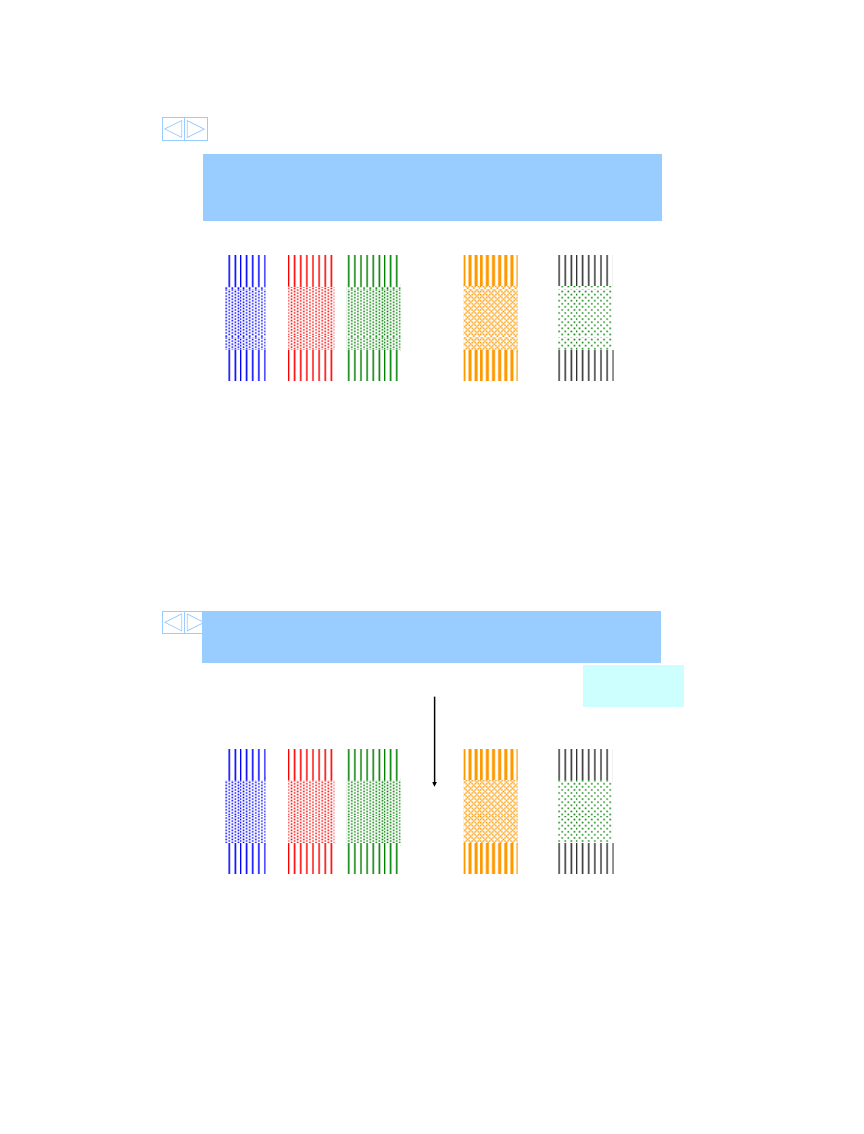
A-BEERN-ALED-LAGERDFWGALSTOUTWRARWATERA
ACBEERGYALEDILAGERAF-G--STOUTFA--WATERM
G<->W = -7
G<->G = +5
A<->C = -2
A<->A = +2
Insercja/Delecja => GAPS
ClareSansom
Insercja/Delecja => GAPS
• Punktownia przerw programowanie
dynamiczne
Wykład Bioinformatyka
Wykład 5, 2008
14
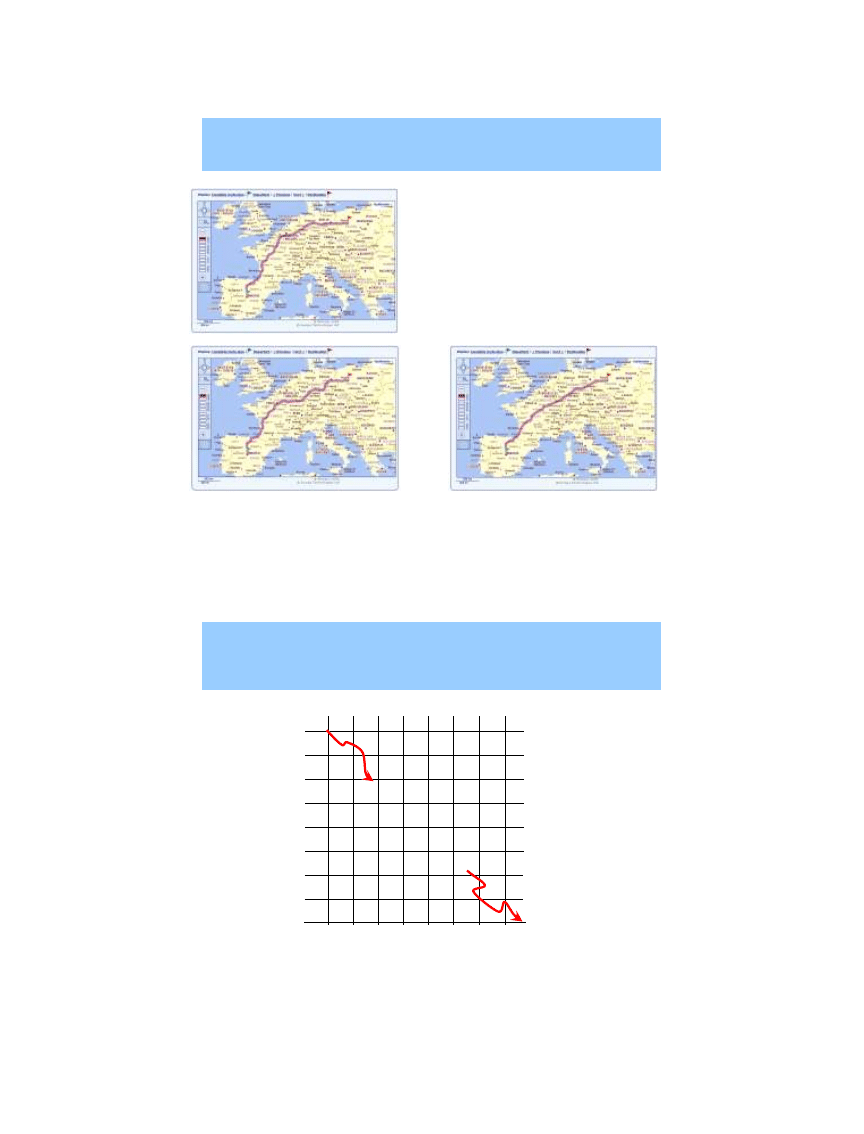
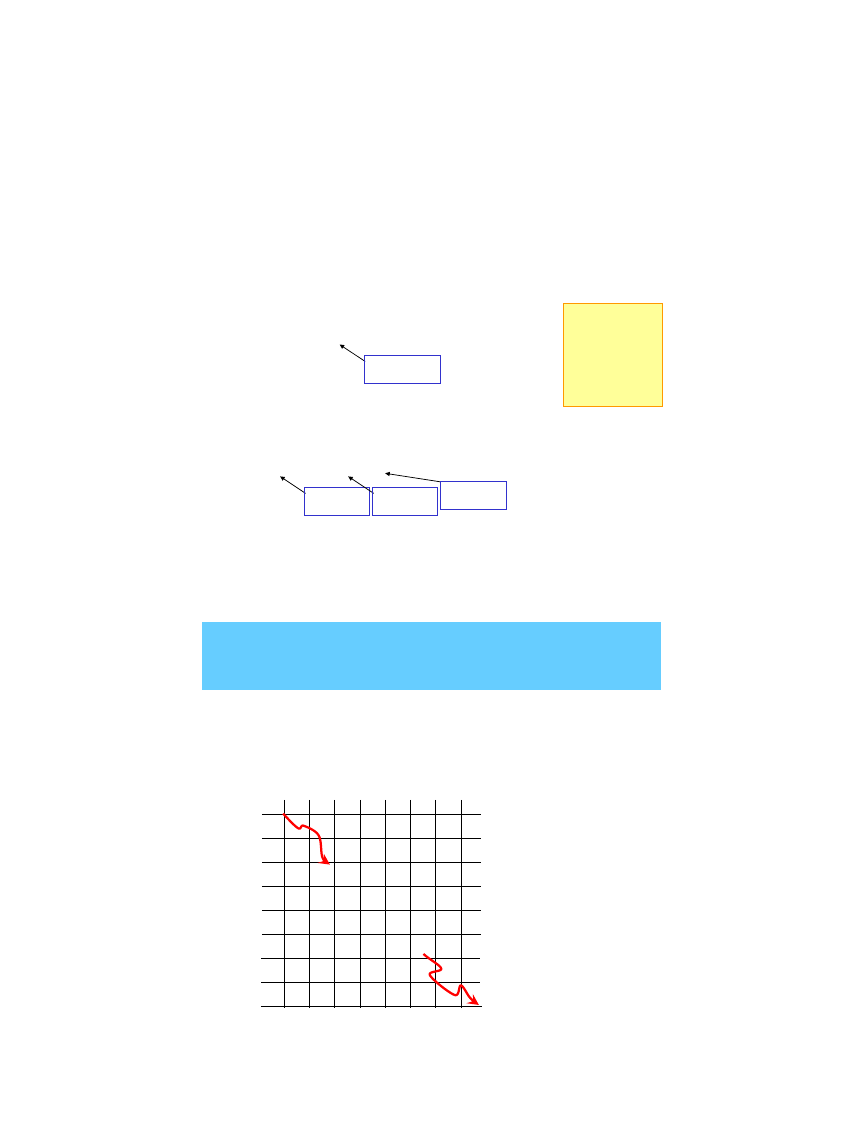
Najlepsza ścieżka
27
optymalna?
najszybsza?
najkrótsza?
Madryt –Poznań
http://www.viamichelin.com
Najlepsza ścieżka?
28
G
A T
T
C T
A
G
A
T
T
A
C
C
A
Wykład Bioinformatyka
Wykład 5, 2008
15
Algorytm Needlemana-Wunscha
• strategia „najlepszej ścieżki” –
programowanie dynamiczne
• przeszukiwanie dotyczy pełnego zakresu
sekwencji (obszaru dopasowania)-
dopasowanie globalne
• każda podścieżka stanowić może fragment
optymalnej ścieżki. Ścieżki szuka się
poszerzając zakres podscieżek.
29
Needlemann, Wunch (1970) J.Mol.Biol. 48, 443-453
30
Sekwencja A
S
ek
we
nc
ja
B
Sekwencja A
Sekwencja A
S
ek
we
nc
ja
B
S
ek
we
nc
ja
B

Wykład Bioinformatyka
Wykład 5, 2008
16
Algorytm Smitha-Watermana
• dopasowanie lokalne
• ścieżka dopasowania nie musi osiągać krawędzi
analizowanej sekwencji
• ścieżka jest lokalnie optymalna jeśli jej
wydłużanie/skracanie nie poprawia obliczonej dla niej
wartości
• system wartościowania dopasowania zaniża wartości w
regionach słabego dopasowania = przerwanie ścieżki
• mogą istnieć ścieżki złożone z kilku połączonych
ścieżek
31
Smith, Waterman (1981) J.Mol.Biol. 147, 195-197
Szukanie wielu dopasowań
-subdopasowania
• Metoda optymalna daje zawsze najlepsze
dopasowanie – nawet jeśli nie ma ono znaczenia
biologicznego
→ znaczących, niezachodzących na siebie
dopasowń lokalnych można naleźć kilka –
subdopasownia
Trzeba szukać więcej niż jednego dopasowania!
(lalign, SIM)
32
Przykład:
•Czynnik krzepnięcia IX (F9, SWISS-PROT P00740)
•Czynnik krzepnięcia XII (F12, SWISS-PROT P00748)

Wykład Bioinformatyka
Wykład 5, 2008
17
Dopasowanie optymalne
i suboptymalne
>sp|P00740|FA9_HUMAN Coagulation factor IX precursor (EC 3.4.21.22)
(Christmas factor) (Plasma thromboplastin component) (PTC) [Contains:
Coagulation factor IXa light chain; Coagulation factor IXa heavy chain] -
Homo sapiens (Human).
MQRVNMIMAESPGLITICLLGYLLSAECTVFLDHENANKILNRPKRYNSGKLEEFVQGNL
ERECMEEKCSFEEAREVFENTERTTEFWKQYVDGDQCESNPCLNGGSCKDDINSYECWCP
FGFEGKNCELDVTCNIKNGRCEQFCKNSADNKVVCSCTEGYRLAENQKSCEPAVPFPCGR
VSVSQTSKLTRAETVFPDVDYVNSTEAETILDNITQSTQSFNDFTRVVGGEDAKPGQFPW
QVVLNGKVDAFCGGSIVNEKWIVTAAHCVETGVKITVVAGEHNIEETEHTEQKRNVIRII
PHHNYNAAINKYNHDIALLELDEPLVLNSYVTPICIADKEYTNIFLKFGSGYVSGWGRVF
HKGRSALVLQYLRVPLVDRATCLRSTKFTIYNNMFCAGFHEGGRDSCQGDSGGPHVTEVE
GTSFLTGIISWGEECAMKGKYGIYTKVSRYVNWIKEKTKLT
>sp|P00748|FA12_HUMAN Coagulation factor XII precursor (EC 3.4.21.38)
(Hageman factor) (HAF) [Contains: Coagulation factor XIIa heavy chain; Beta-
factor XIIa part 1; Beta-factor XIIa part 2; Coagulation factor XIIa light
chain] - Homo sapiens (Human).
MRALLLLGFLLVSLESTLSIPPWEAPKEHKYKAEEHTVVLTVTGEPCHFPFQYHRQLYHK
CTHKGRPGPQPWCATTPNFDQDQRWGYCLEPKKVKDHCSKHSPCQKGGTCVNMPSGPHCL
CPQHLTGNHCQKEKCFEPQLLRFFHKNEIWYRTEQAAVARCQCKGPDAHCQRLASQACRT
NPCLHGGRCLEVEGHRLCHCPVGYTGPFCDVDTKASCYDGRGLSYRGLARTTLSGAPCQP
WASEATYRNVTAEQARNWGLGGHAFCRNPDNDIRPWCFVLNRDRLSWEYCDLAQCQTPTQ
AAPPTPVSPRLHVPLMPAQPAPPKPQPTTRTPPQSQTPGALPAKREQPPSLTRNGPLSCG
QRLRKSLSSMTRVVGGLVALRGAHPYIAALYWGHSFCAGSLIAPCWVLTAAHCLQDRPAP
EDLTVVLGQERRNHSCEPCQTLAVRSYRLHEAFSPVSYQHDLALLRLQEDADGSCALLSP
YVQPVCLPSGAARPSETTLCQVAGWGHQFEGAEEYASFLQEAQVPFLSLERCSAPDVHGS
SILPGMLCAGFLEGGTDACQGDSGGPLVCEDQAAERRLTLQGIISWGSGCGDRNKPGVYT DVAYYLAWIREHTVS
P00740
P00748
Dopasowanie optymalne
i suboptymalne
34
http://www.ch.embnet.org/software/LALIGN_form.html

Wykład Bioinformatyka
Wykład 5, 2008
18
35
oraz dopasowania 2 i 3:
36
http://www.expasy.ch/tools/sim-prot.html
P00740
P00748
Wykład Bioinformatyka
Wykład 5, 2008
19
Wartości substytucji
i kary za przerwy
• schemat wartościowania I:
– (match) dopasowany: +1
– (mismatch) niedopasowany: -1
– (gap) przerwa: -1
(nie-afiniczne kary za przerwy –każda przerwa traktowana jest tak samo)
37
• schemat wartościowania II:
– dopasowany: +1
– niedopasowany: -1
– otwarcie przerwy: G
– przedłużenie przerwy: L
(afiniczne kary za przerwy –kara za otwarcie, kary za przedłużenie )
Punktacja
38
S – punktacja za dopasowanie
Score = Max(S)
ATTGTCAAAGACTTGAGCTGATGTGCAAATT
GGTCAGACATGA-CTGACAAGGGTATCGCCA
obszar dopasowania
dopasowanie
niedopasowanie
przerwa
S =
(dopasowania) -
(niedopasowania) -
(przerwy)
Wykład Bioinformatyka
Wykład 5, 2008
20
Punktowanie przerw
• non-affine model (nieafinicznie): równo
(match:4, mismatch:-3, gap:-4)
• affine model (afinicznie): G + L n
(match:4, mismatch:-3, gap creation:-8, gap:-4)
39
A T G C
A 4 -3 -3 -3
T -3
4 -3 -3
G –3 -3
4 -3
C -3 -3 -3
4
GGTGCCAC-TCCAC-----CTG
AGTGCCACCCCCAATGCCGCTG
-3 4 4 4 4 4 4 4-4-3 4 4 4-3-4-4-4-4-4 4 4 4 :23
-3 4 4 4 4 4 4 412-3 4 4 4-312-4-4-4-4 4 4 4 :7
GGTGCCAC-TCCA---C--CTG
AGTGCCACCCCCAATGCCGCTG
-3 4 4 4 4 4 4 4-4-3 4 4 4-4-4-4 4-4-4 4 4 4 :26
-3 4 4 4 4 4 4 412-3 4 4 412-4-4 412-4 4 4 :2
4 -12
-3 -12
4 -12 4 -12
Programowanie dynamiczne –
najlepsza ścieżka
• schemat wartościowania I:
– (match) dopasowany: +1
– (mismatch) niedopasowany: -1
– (gap) przerwa: -1
(nie-afiniczne kary za przerwy –każda przerwa traktowana jest tak samo)
40
G
A T
T
C T
A
G
A
T
T
A
C
C
A
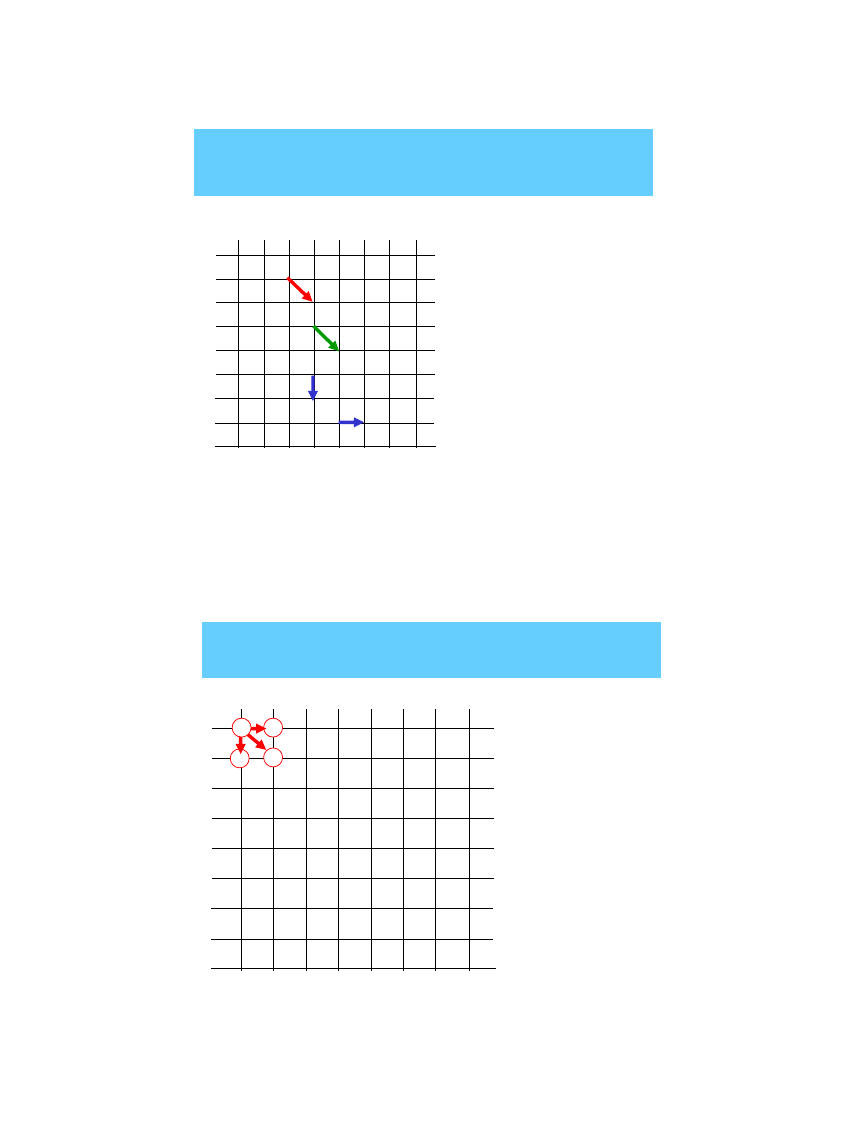
Wykład Bioinformatyka
Wykład 5, 2008
21
Programowanie dynamiczne
zasady:
41
G
A T
T
C T
A
G
A
T
T
A
C
C
A
T
dopasowane z
A
= -1
T
dopasowane z
T
= +1
NULL
dopasowane z
C
= -1
C
dopasowane z
NULL
= -1
Programowanie dynamiczne
42
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
-1
+1
-1
0
Wykład Bioinformatyka
Wykład 5, 2008
22
Programowanie dynamiczne
43
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
-1
0
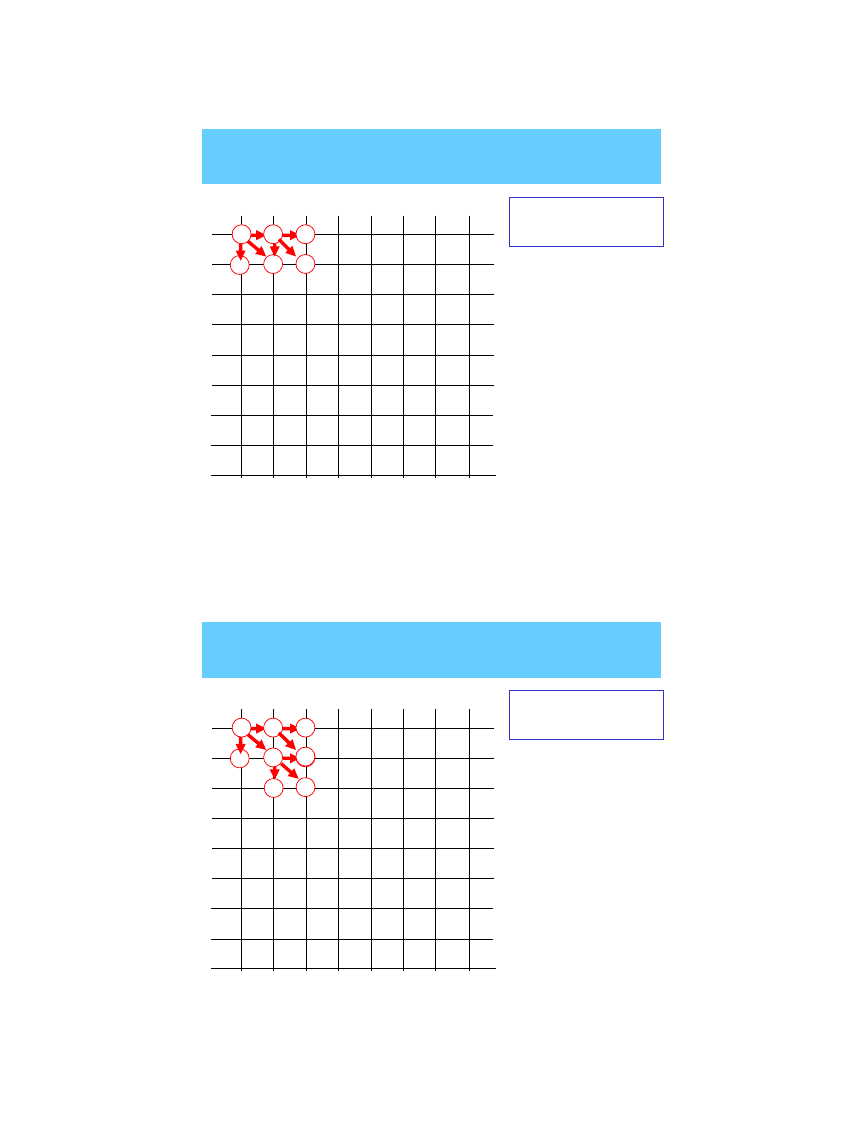
stopniowe
poszerzanie ścieżek
-1
-2
-2
Programowanie dynamiczne
44
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
-1
0
stopniowe
poszerzanie ścieżek
-1
-2
-2
0
+2
0
Wykład Bioinformatyka
Wykład 5, 2008
23
Programowanie dynamiczne
45
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
0
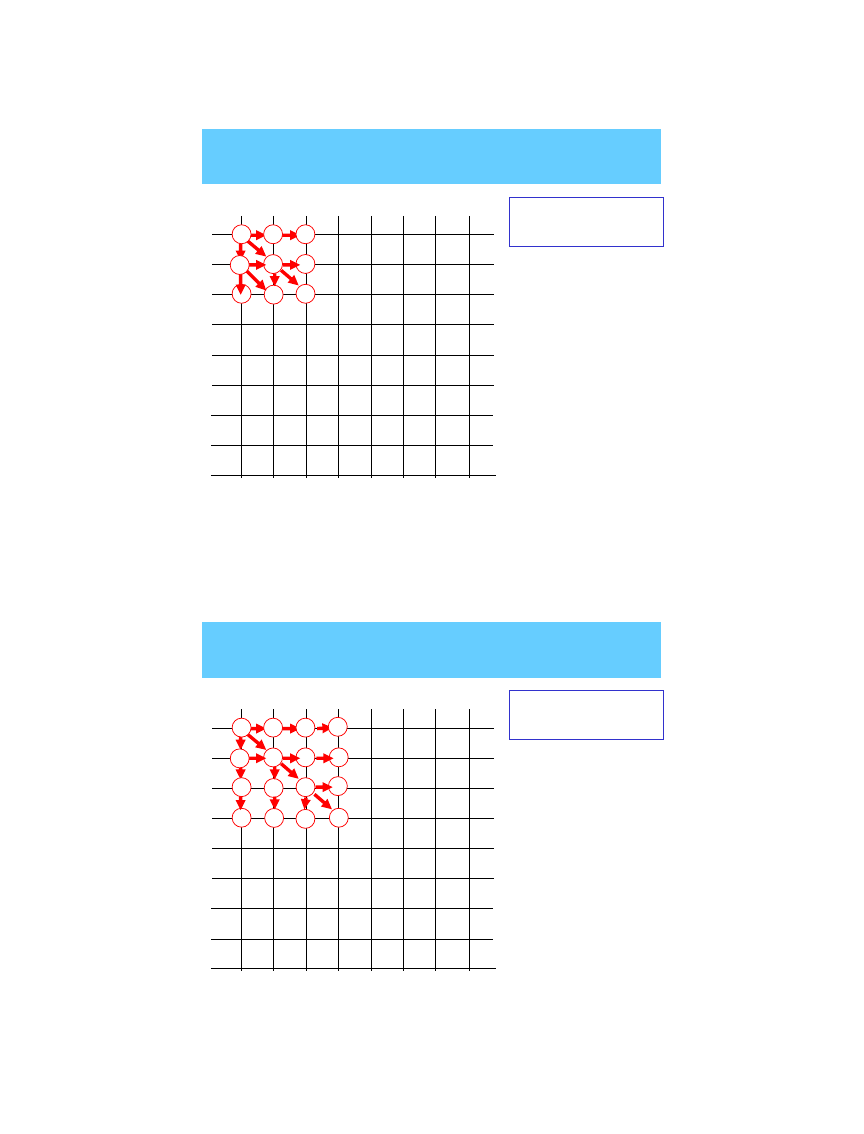
stopniowe
poszerzanie ścieżek
-1
-2
0
0
+2
-2
-1
Programowanie dynamiczne
46
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
0
stopniowe
poszerzanie ścieżek
-1
-2
0
0
+2
-1
-3
-2
-1
-3
-1
+3
+1
+1
Wykład Bioinformatyka
Wykład 5, 2008
24
Programowanie dynamiczne
47
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
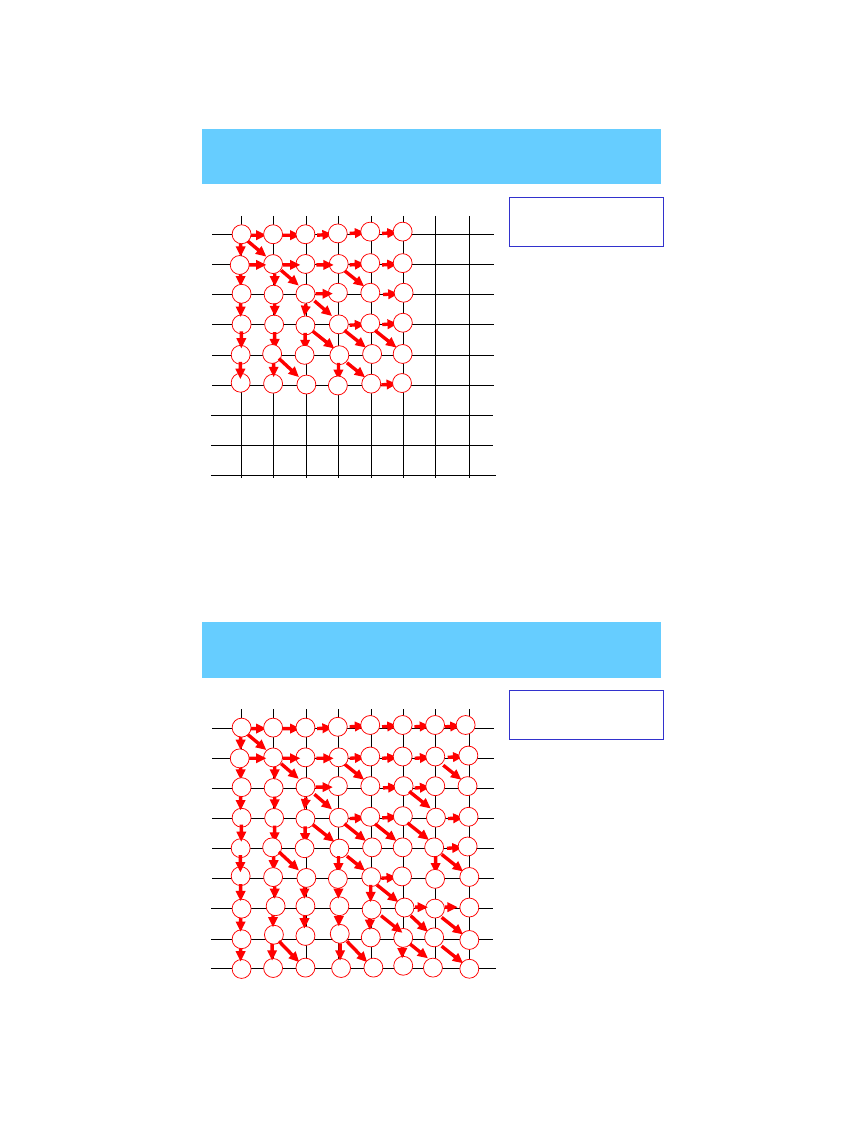
stopniowe
poszerzanie ścieżek
+1
0
-1
-2
0
0
+2
-1
-3
-2
-1
-3
-1
+3
+1
+1
-4
-5
-2
-3
0
-1
+2
+1
+2
+1
+2
+3
+2
+1
-1
-3
-5
-4
-2
0
•wszystkie punkty
musza zostać zbadane
Programowanie dynamiczne
48
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
0
stopniowe
poszerzanie ścieżek
-1
-2
0
0
+2
-1
-3
-2
-1
-3
-1
+3
+1
+1
-4
-5
-2
-3
0
-1
+2
+1
+2
+1
+2
+3
+2
-5
-4
-2
0
-6
-7
-4
-5
-3
-2
-1
0
+2
+3
+1
+1
+2
+2
+4
+4
+3
+3
-8
-7
-6
-6
-5
-4
-3
-4
-3
-2
-1
-2
0
-1
0
+1
+1
+2
+2
+2
+3
Wykład Bioinformatyka
Wykład 5, 2008
25
Programowanie dynamiczne
49
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
+1
0
stopniowe
poszerzanie ścieżek
-1
-2
0
0
+2
-1
-3
-2
-1
-3
-1
+3
+1
+1
-4
-5
-2
-3
0
-1
+2
+1
+2
+1
+2
+3
+2
-5
-4
-2
0
-6
-7
-4
-5
-3
-2
-1
0
+2
+3
+1
+1
+2
+2
+4
+4
+3
+3
-8
-7
-6
-6
-5
-4
-3
-4
-3
-2
-1
-2
0
-1
0
+1
+1
+2
+2
+2
+3
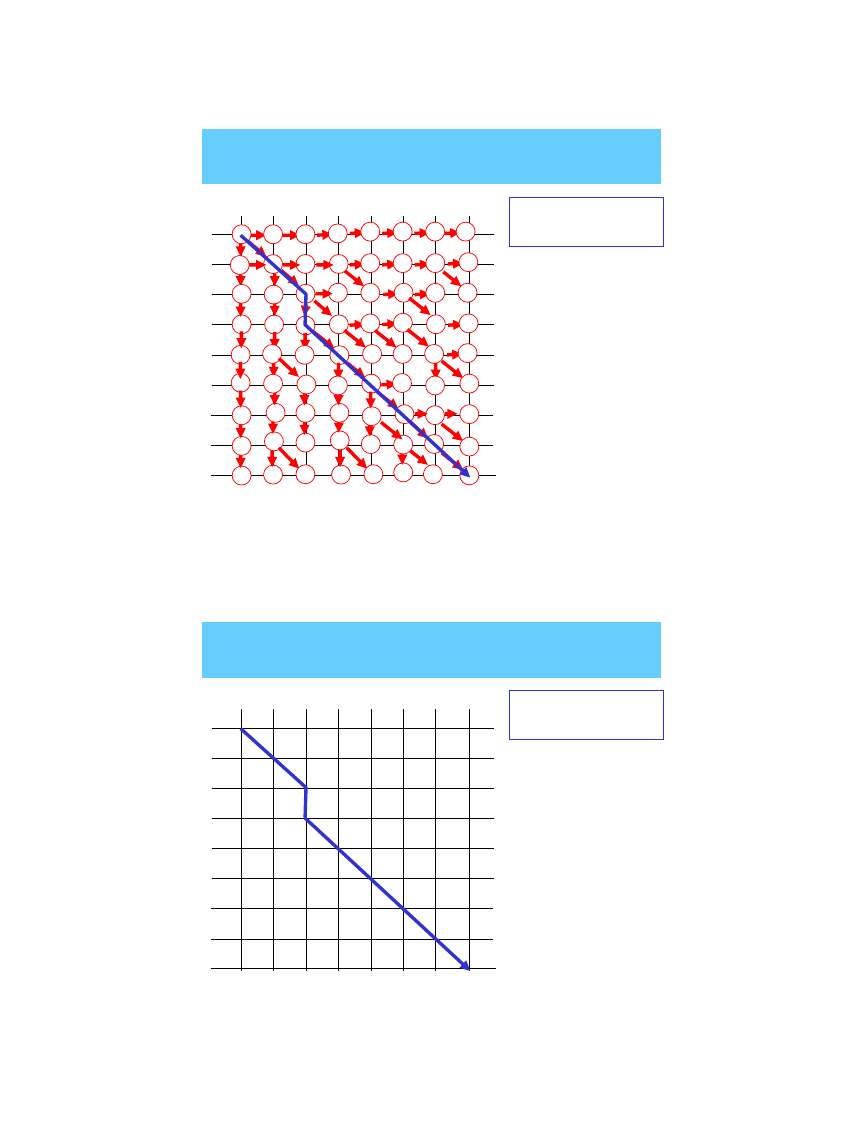
GA-TACTA
GATTACCA
Programowanie dynamiczne
50
G
A
T
A
C
T
A
G
A
T
T
A
C
C
A
stopniowe
poszerzanie ścieżek
GA-TACTA
GATTACCA

Wykład Bioinformatyka
Wykład 5, 2008
26
Statystyczne znaczenie dopasowań
• jaka jest wartość/ istotność dopasowania?
51
Dla dopasowań globalnych:
porównanie obliczonej wartości dla danego dopasowania z
wartościami obliczonymi dla wielu dopasowań
przypadkowych sekwencji o podobnym składzie i długości
Dla dopasowań lokalnych:
podstawą jest rozkład wartości granicznej,
scharakteryzowanej paramerami K i λ
ćwiczenia
52

Wykład Bioinformatyka
Wykład 5, 2008
27
Dopasowanie Globalne
-cząsteczki jednodomenowe
• Adresy:
– Expasy:
– NCBI:
• Narzędzia:
– Bl2seq:
http://www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi
– SIM + LALNVIEW:
http://www.expasy.ch/tools/#align
– LALIGN:
http://www.ch.embnet.org/software/LALIGN_form.html
– Align:
http://www.ebi.ac.uk/emboss/align/index.html
53
Dopasowanie globalne
• Trypsyna: (mysz) P07146, (langusta)
P00765
• Zeta-krystalina (soczewka oka kręgowców)
Q08257, oksydoreduktaza chininowa
(E.Coli) P28304
54
Wykład Bioinformatyka
Wykład 5, 2008
28
DotPlot- Dotter
• Dotter:
http://www.cgb.ki.se/cgb/groups/sonnhammer/Dotter.html
• wykrywają fragmenty powtarzalne i rearanżacje
• opierają się na ocenie wizualnej – czasami skuteczniejszej niż
alignment
• idealny do szukania lokalnego podobieństwa
55
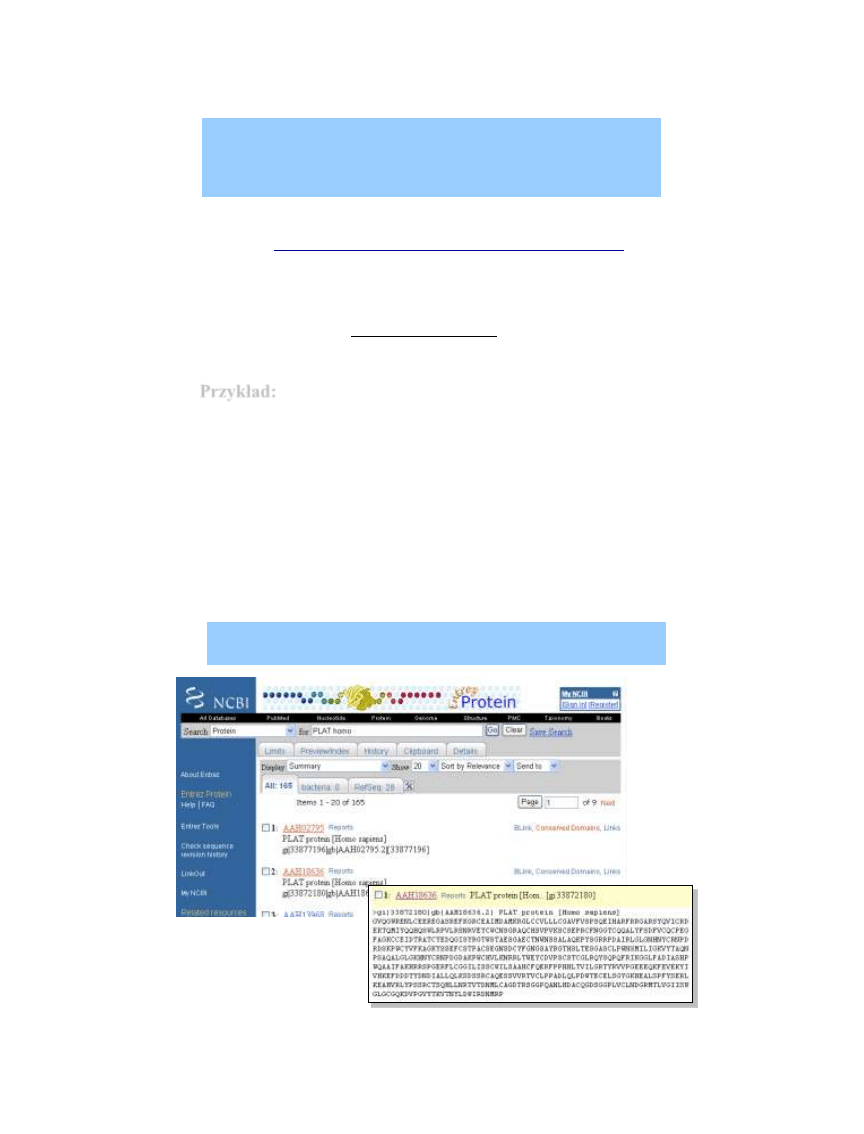
Przykład:
•czynnik krzepnięcia XII (F12): P00748
•aktywator tkankowo specyficznego plazminogenu (PLAT): P00750
(Bioinformatyka. Podręcznik do analizy genów i białek. A.D. Baxevanis, B.F.F. Quellette,
PWN, 2005 )
Dotter - sekwencje
56
Wykład Bioinformatyka
Wykład 5, 2008
29
Dotter - sekwencje
57
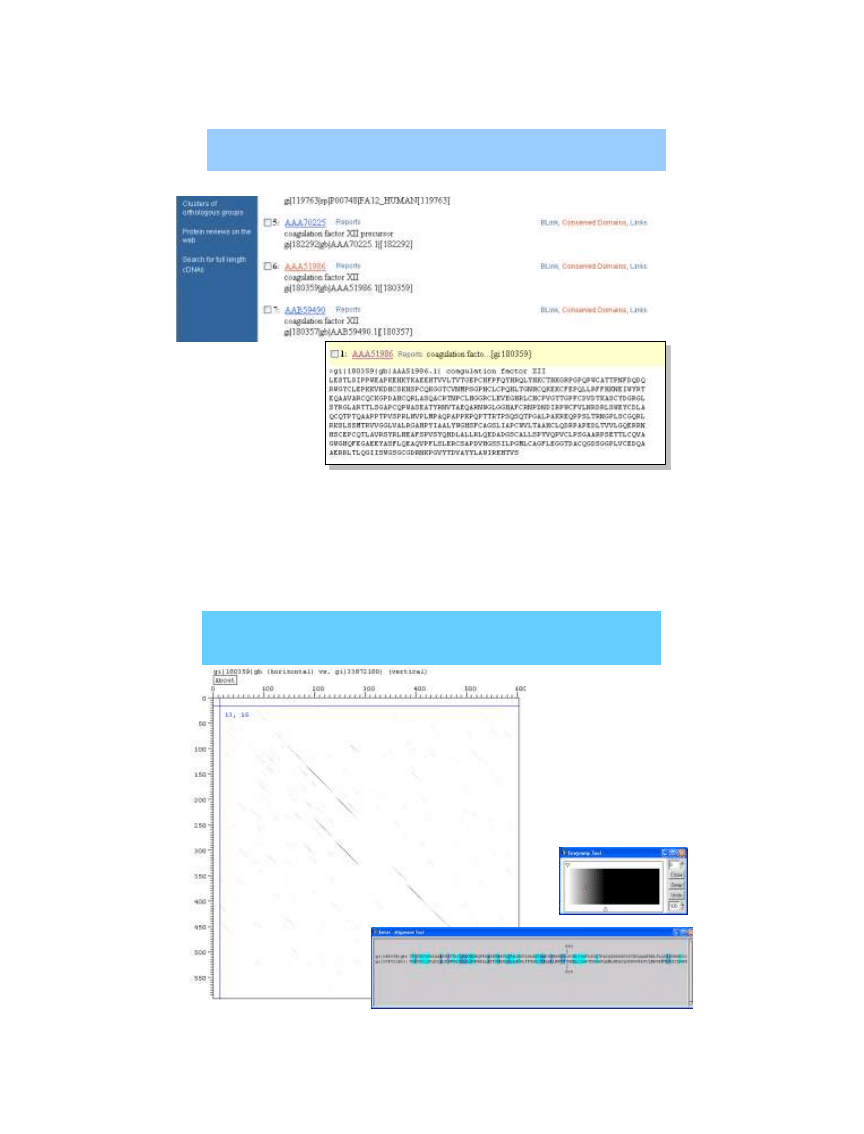
Dotter
58
Wykład Bioinformatyka
Wykład 5, 2008
30
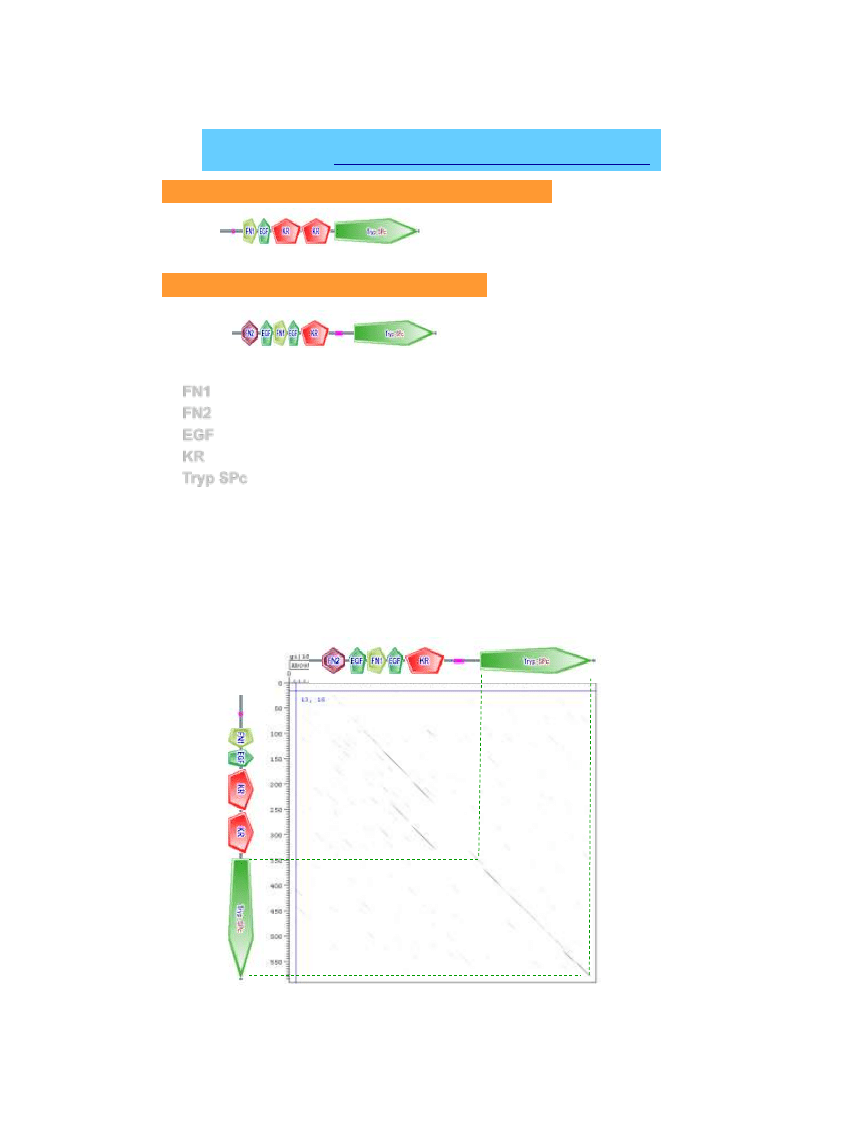
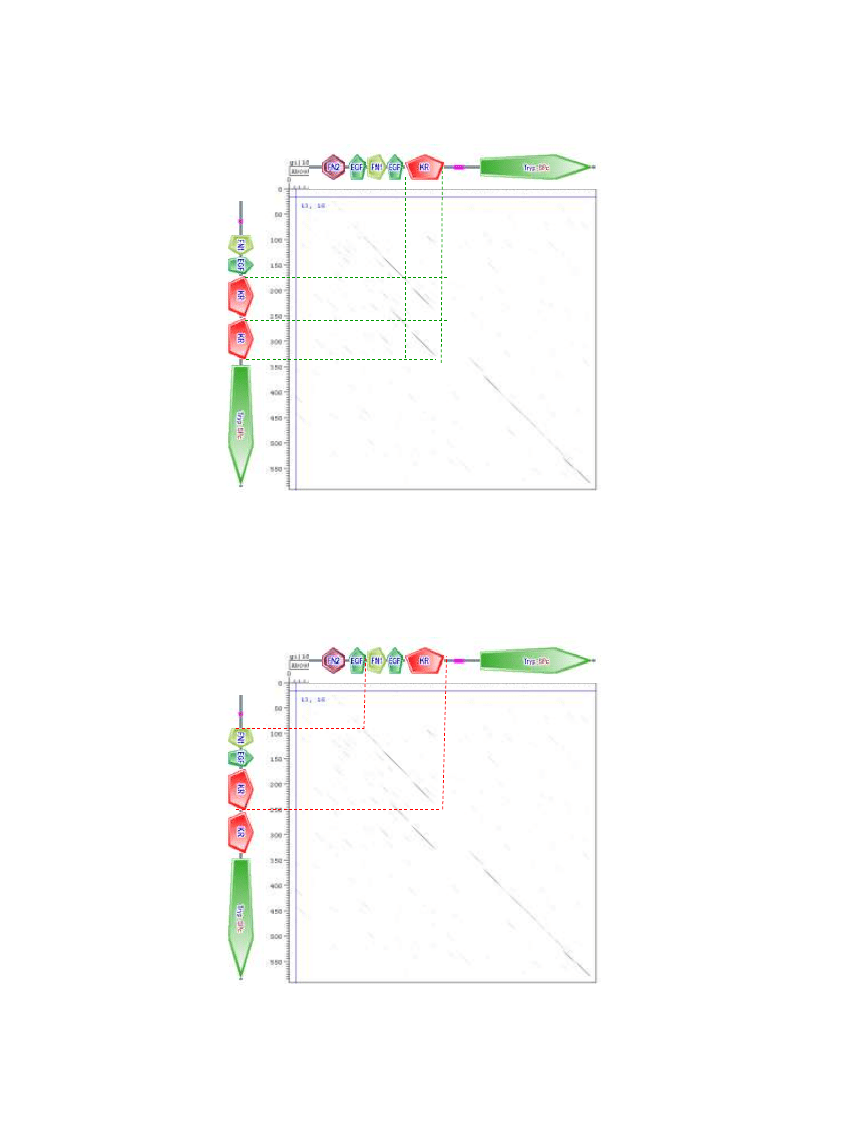
SMART (
http://smart.embl-heidelberg.de/)
59
>gi|33877196|gb|AAH02795.2| PLAT protein [Homo sapiens]
>gi|180359|gb|AAA51986.1| coagulation factor XII
FN1
fibrynonektyna typ I, powtarzalna jednostka
FN2
fibrynonektyna typ II, powtarzalna jednostka
EGF
moduł nabłonkowego czynnika wzrostu
KR
domena typu kringle
Tryp SPc
domena katalityczna zapewniająca aktywność
proteinazy serynowej
60
czynnik krzepnięcia krwi
ak
ty
w
ato
r
tk
an
k
o
w
o
s
p
ec
y
ficzn
eg
o
p
lazm
in
o
g
en
u
Wykład Bioinformatyka
Wykład 5, 2008
31
61
czynnik krzepnięcia krwi
ak
ty
w
ato
r
tk
an
k
o
w
o
s
p
ec
y
ficzn
eg
o
p
lazm
in
o
g
en
u
62
czynnik krzepnięcia krwi
ak
ty
w
ato
r
tk
an
k
o
w
o
s
p
ec
y
ficzn
eg
o
p
lazm
in
o
g
en
u

Wykład Bioinformatyka
Wykład 5, 2008
32
KONIEC
c.d.n. (macierze substytucji, statystyczne znaczenie dopasowań)
63
Wyszukiwarka
Podobne podstrony:
Porównanie technik sekwencyjnych
PORÓWNYWANIE TECHNOLOGII
W5 sII PCR i sekwencjonowanie cz 2
Metodyka harcerska i starszoharcerska porównanie
Porównanie dwóch regionalnych strategii innowacji
19 Teorie porównanie
Metoda symultaniczno sekwencyjna
Wykład VIII Synteza układów sekwencyjnych
KOLOKWIUM 2 zadanie wg Adamczewskiego na porownawczą 97
1F CWICZENIE zadanie wg Adamczewskiego na porownawczą 97id 18959 ppt
Porównanie USB FireWire
Dowody za obiektywno¶ci± ewolucji z zakresu morfologii porównawczej 1 cz
więcej podobnych podstron