
Sekwencje DNA i RNA
PCR i sekwencjonowanie

Reakcja PCR
(Polymerase Chain Reaction)
•Najczulsza
metoda
służąca
do
wykrywania
i powielania fragmentów DNA o
określonej sekwencji.
•Pozwala na wykrycie i amplifikację
pojedynczej
kopii
interesującej
sekwencji
z preparatu DNA całkowitego.

Składniki mieszaniny
reakcyjnej
• Termostabilna polimeraza DNA
• Para syntetycznych
oligonukleotydów jako startery
syntezy DNA
• Trójfosforany deoksynukleotydów
• Kationy dwuwartościowe (Mg
2+
)
• Bufor o pH 8.3-8.8
• Kationy jednowartościowe (K
+
)
• Matryca DNA
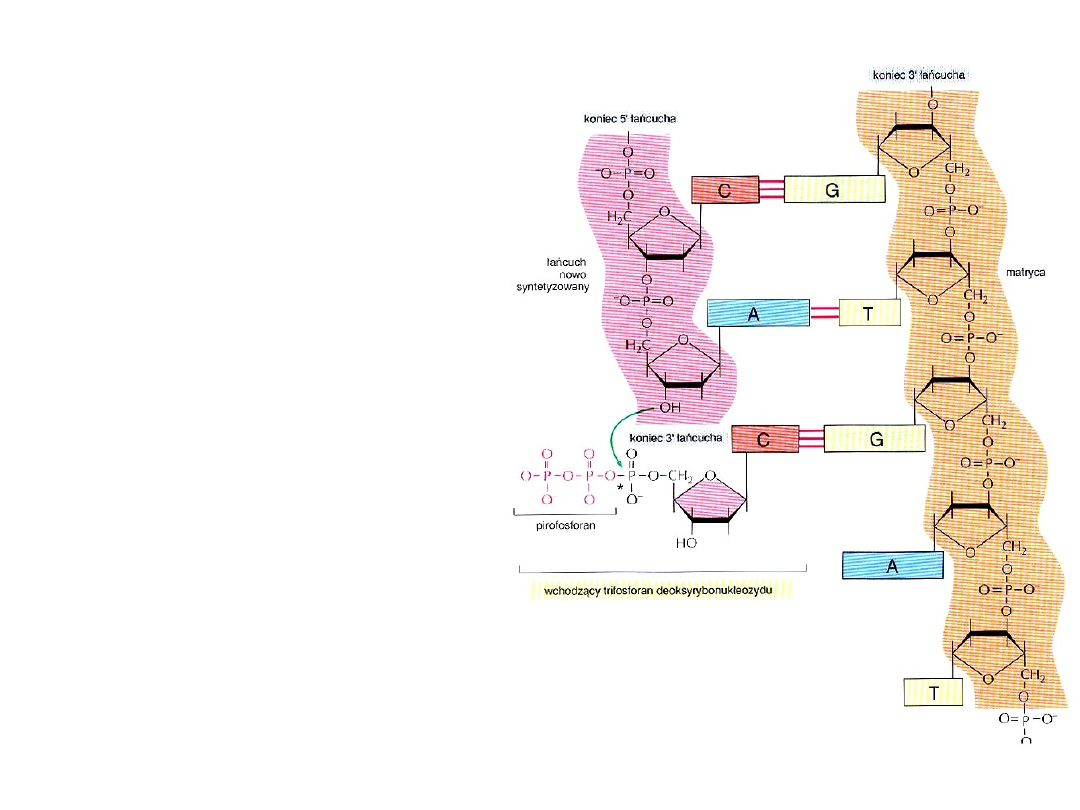
Polimeraza DNA
przyłącza
nukleotydy
do końca 3’ – tzn.
katalizuje
reakcję
wydłużania
w kierunku 5’→3’

Etapy PCR:
• denaturacja 92-96 C
• hybrydyzacja – przyłączenie
primerów do matrycy 54 – 56 C
• elongacja – 72 C
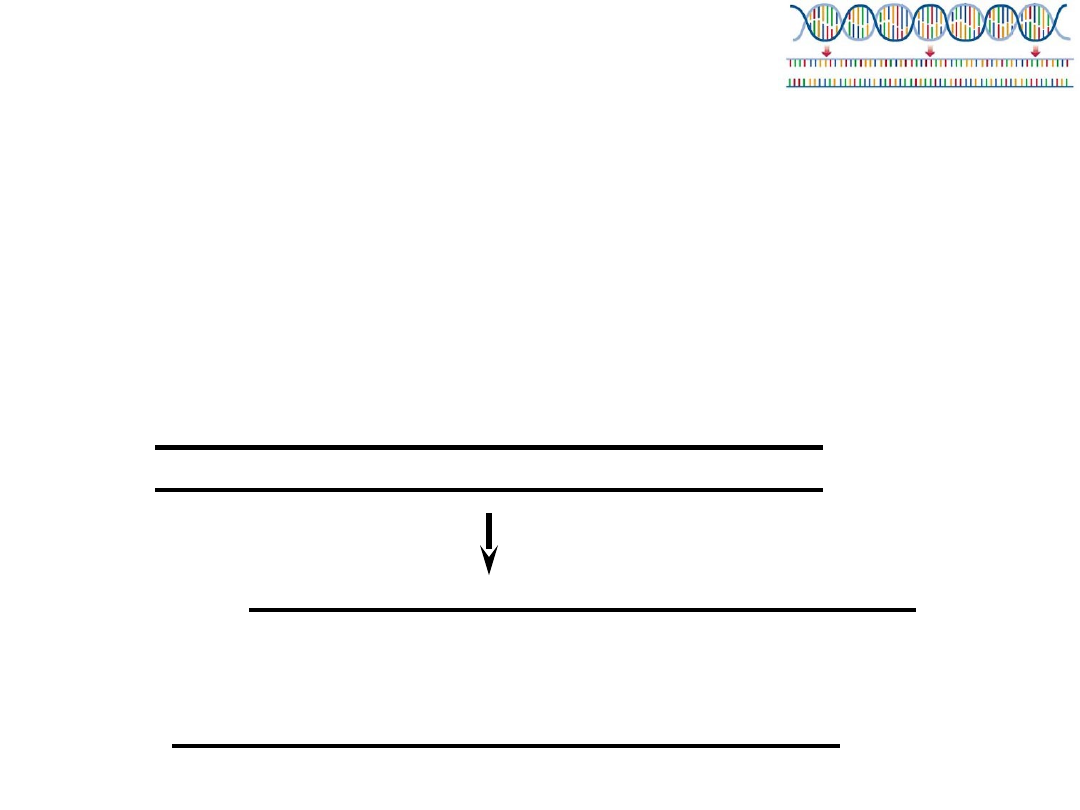
Denaturacja
Denaturacja – rozdzielenie nici DNA pod wpływem
temperatury
Temperatura denaturacji zależy od składu zasad
fragmentu który chcemy powielić i odporności
polimerazy na wysoką temperaturę.
Najpopularniejsza polimeraza używana standartowo w
PCR – Taq wytrzymuje temperaturę 94-95 stopni C.
Przy fragmentach bogatych w GC używa się polimeraz
bardziej opornych na wysoką temperaturę (np. Pwo,
Pfu).
5’
3’
5’
3’
5’
3’
5’
3’
94
0
C
Pierwszy cykl
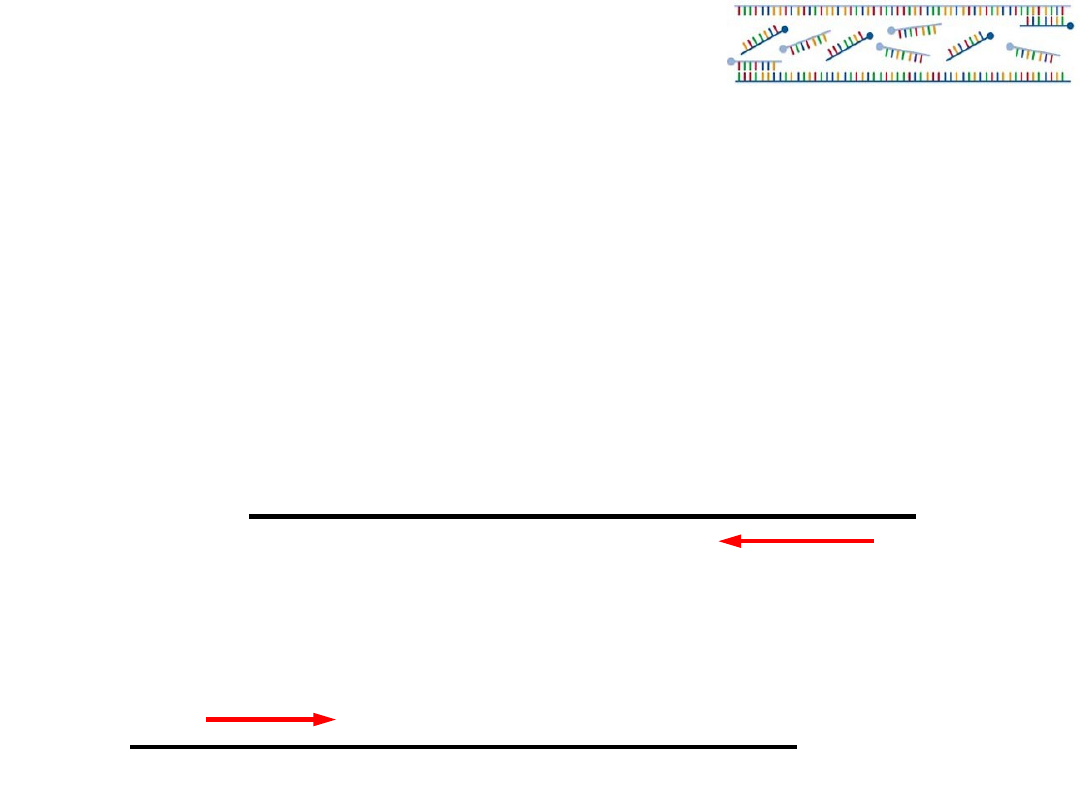
Annealing
(przyłączanie starterów)
Przyłączanie starterów (
Annealing)
Temperatura:
za wysoka – słaba wydajność
za niska - niespecyficzne powielanie
Stosuje się temperaturę około 3-5 stopni poniżej
oszacowanej temperatury topnienia.
Praktyczny wzór na temperaturę topnienia dla starterów
o długości około 20 nukleotydów:
T=2x(A+T) + 4(G+C)
5’
3’
3’
5’
5’
3’
3’
5’
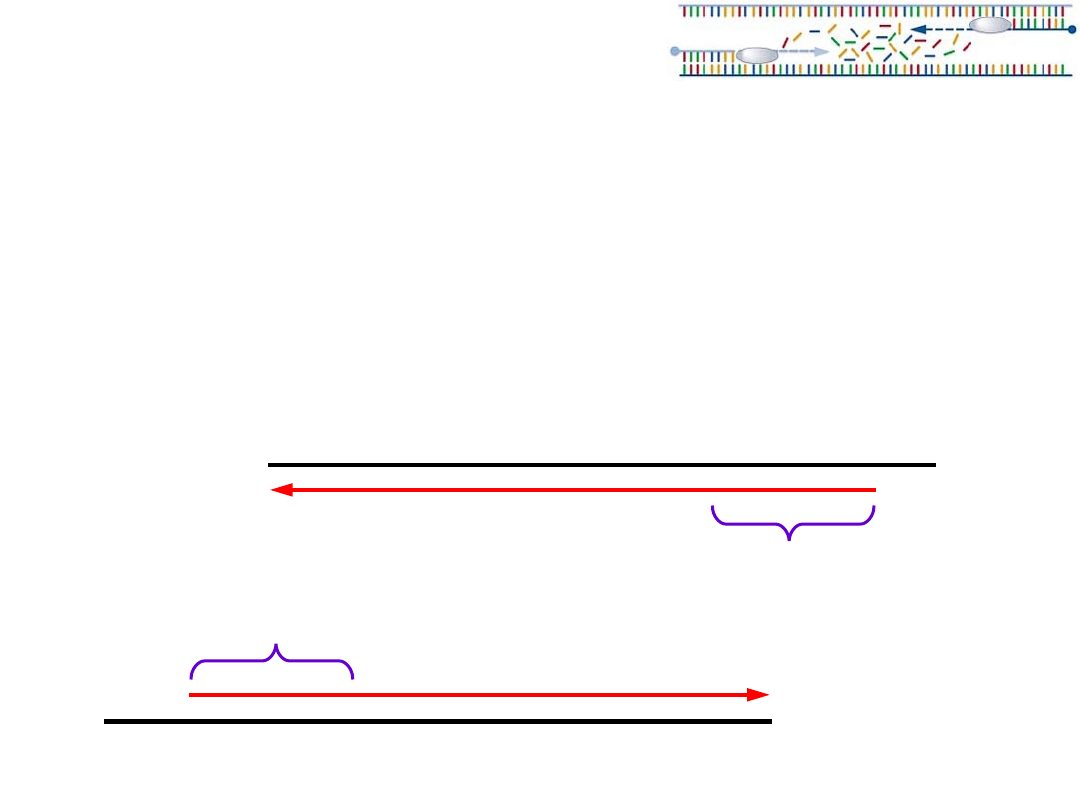
Extension (wydłużanie łańcucha)
Wydłużanie łańcucha (Extension)
Temperatura tej części cyklu zależy od właściwości polimerazy.
Dla polimerazy Taq optymalna jest temperatura 72-78 stopni.
Szybkość reakcji w optymalnych warunkach dla polimerazy Taq
wynosi około 2000 nukleotydów/min.
Do pełnej syntezy badanego fragmentu czas trwania tego etapu
wyznaczamy zakładając połowę optymalnej prędkości.
5’
3’
3’
5’
Sekwencja startera
5’
3’
5’
3’
Sekwencja startera
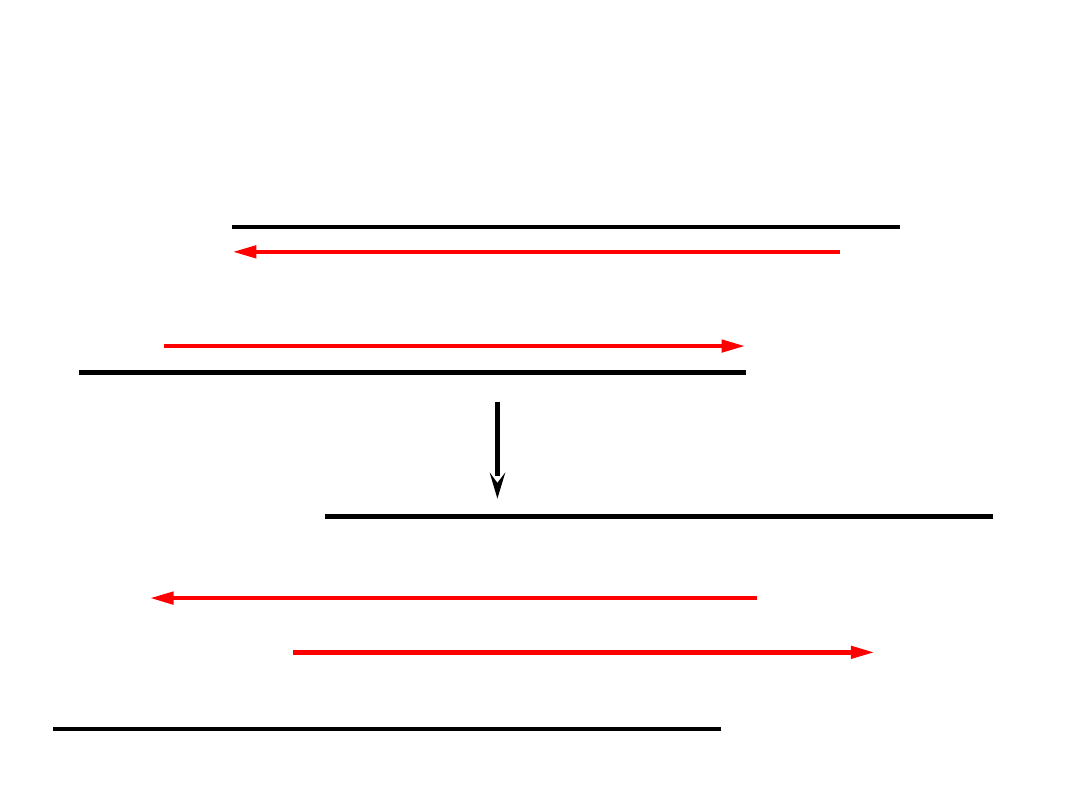
Drugi cykl - denaturacja
5’
3’
5’
3’
3’
5’
5’
3’
94
0
C
5’
3’
3’
5’
5’
3’
5’
3’
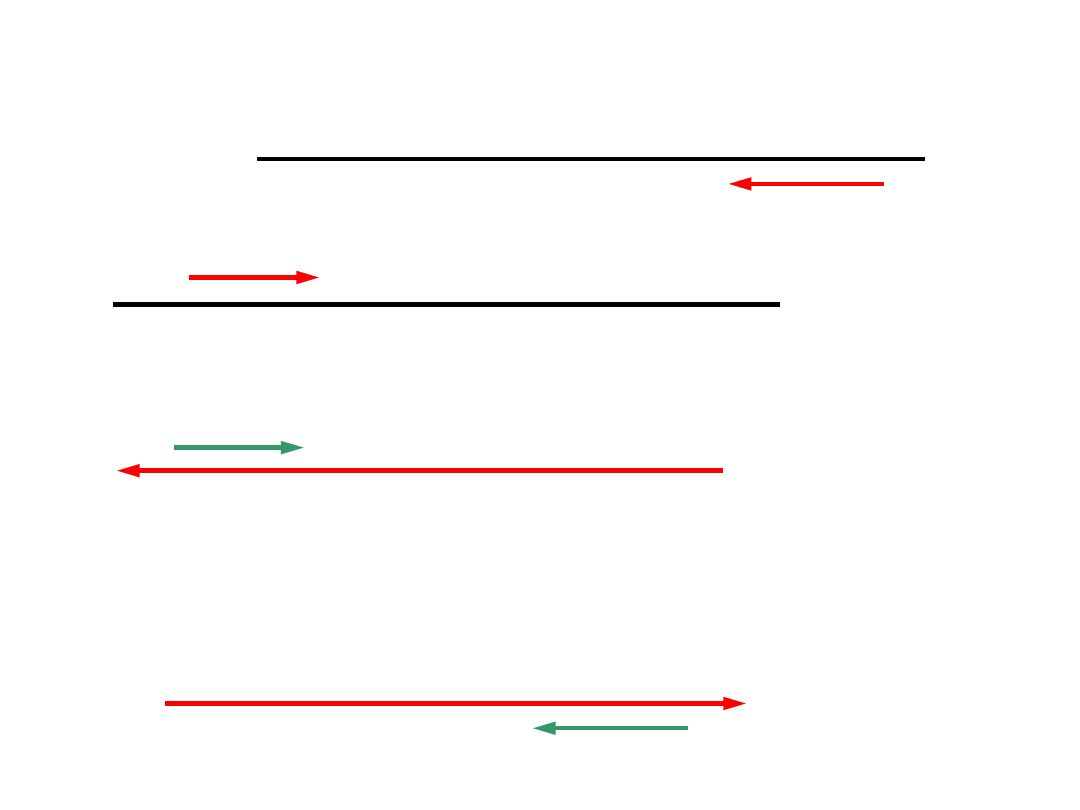
Przyłączanie starterów
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
3’
5’
3’
5’
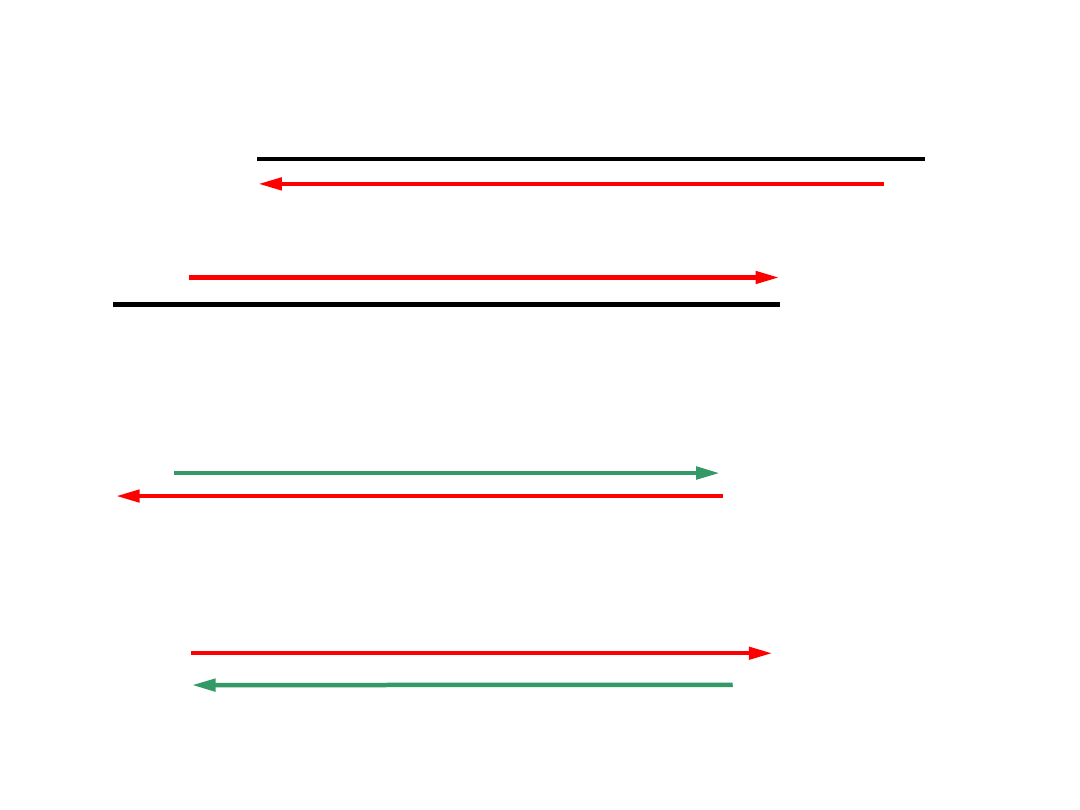
Wydłużanie nici (elongacja)
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
3’
5’
3’
5’
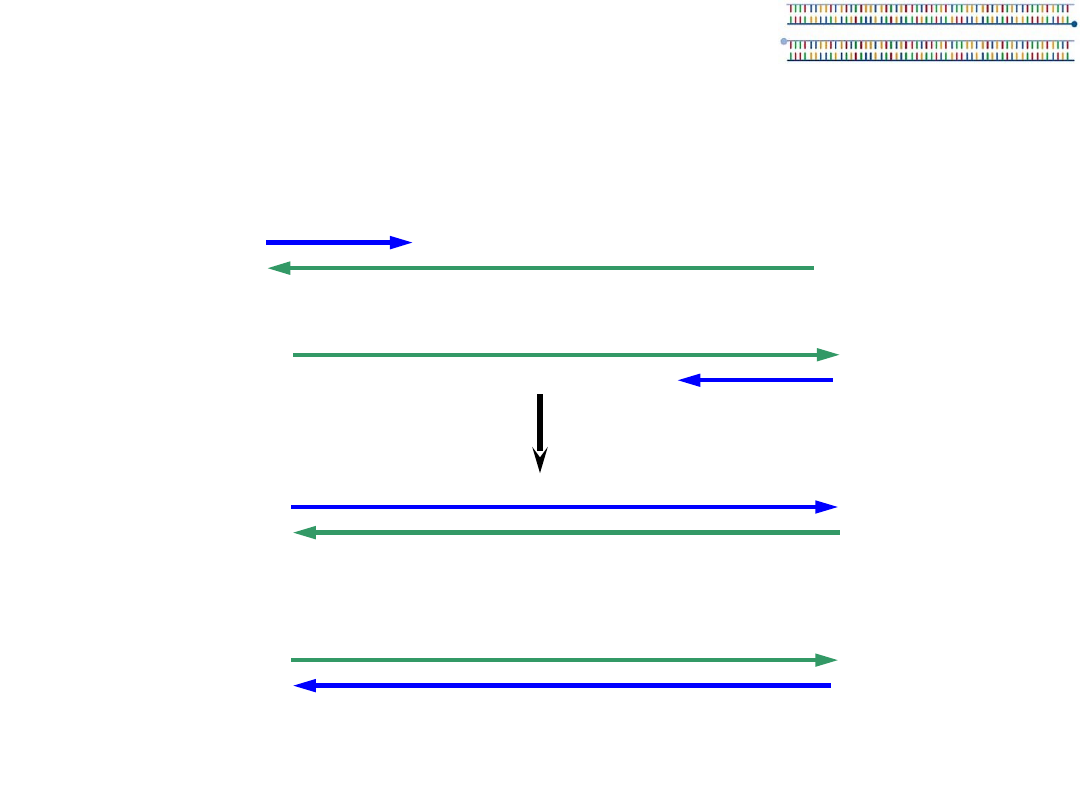
W trzecim cyklu produkowany jest
już właściwy produkt
5’
3’
3’
5’
3’
5’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
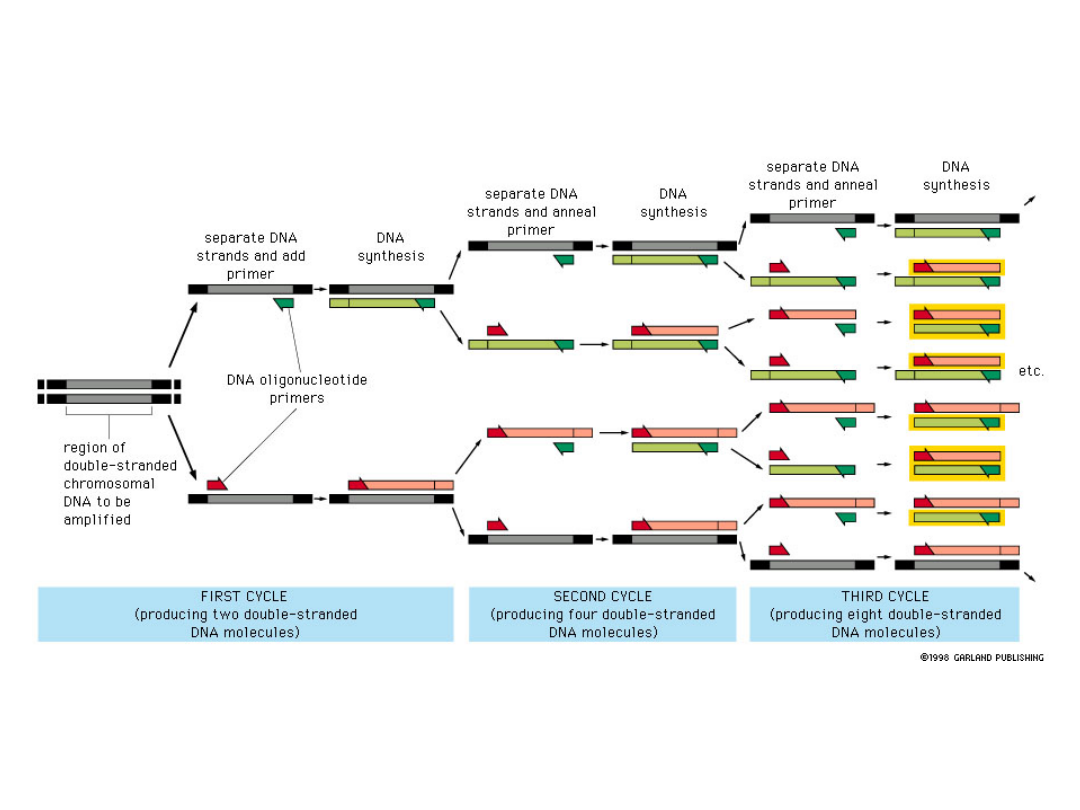
PCR
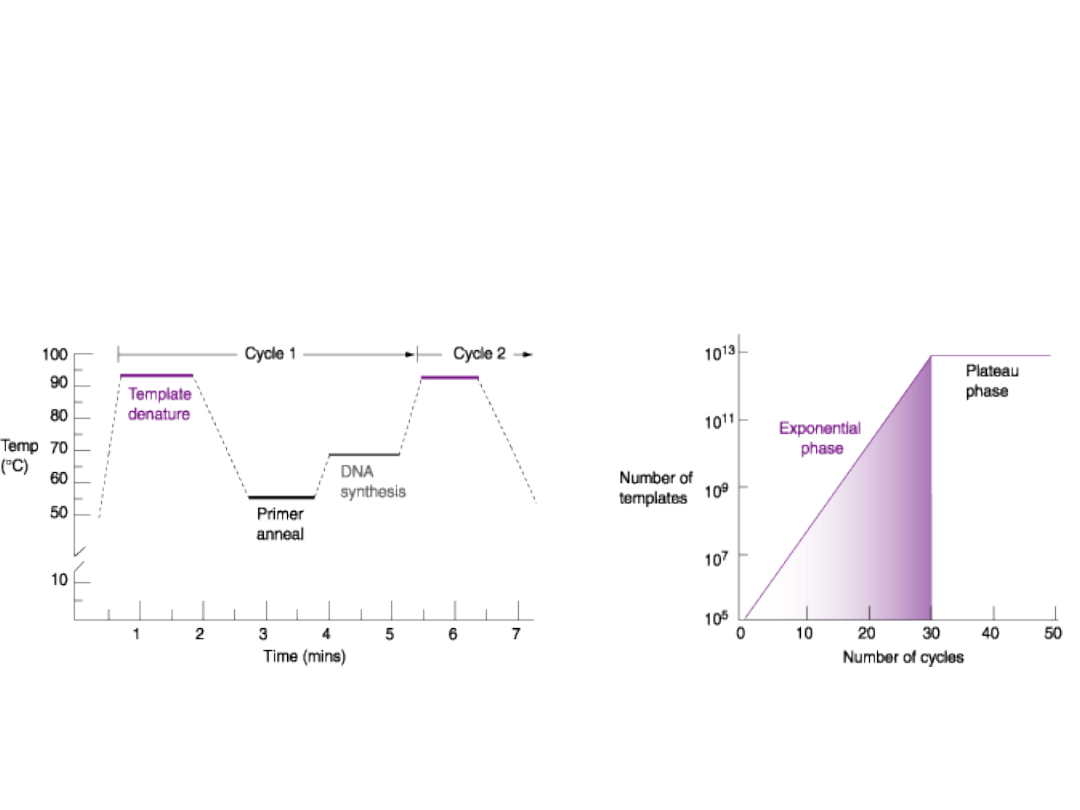
Temperatura jest najważniejszym
fizycznym czynnikiem wpływającym
na wynik w reakcji PCR
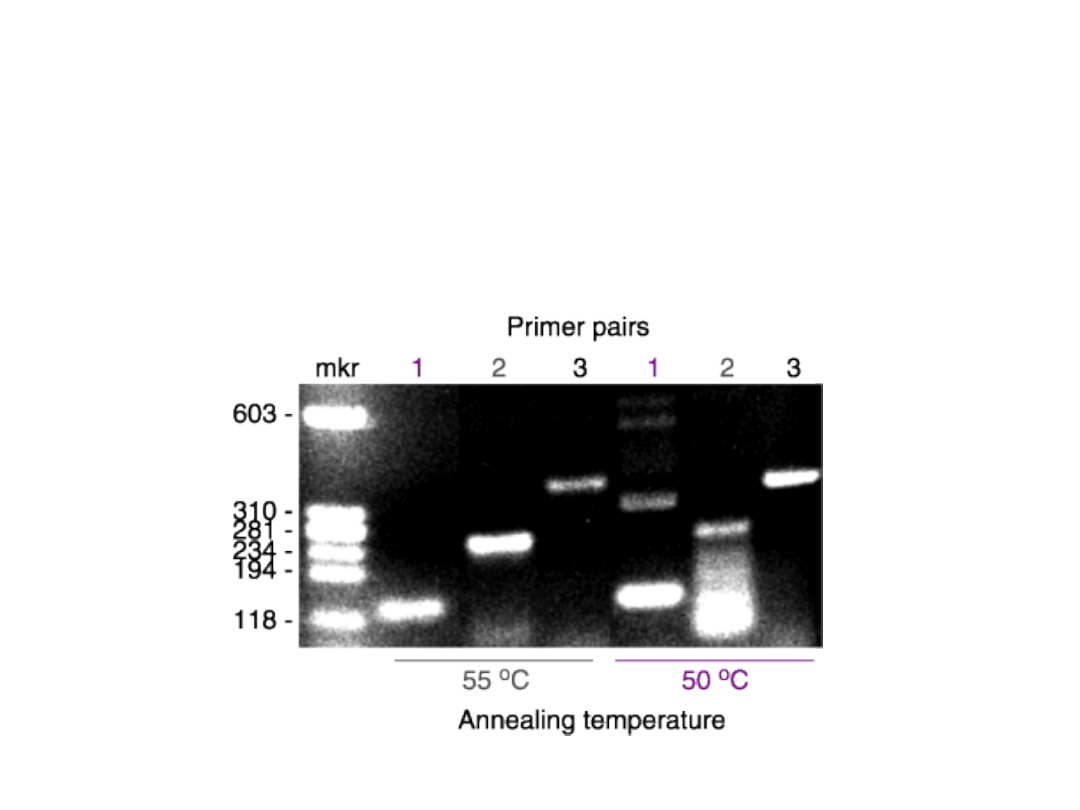
Wynik klasycznego PCR – obraz elektroforetyczny
zamplifikowanego produktu
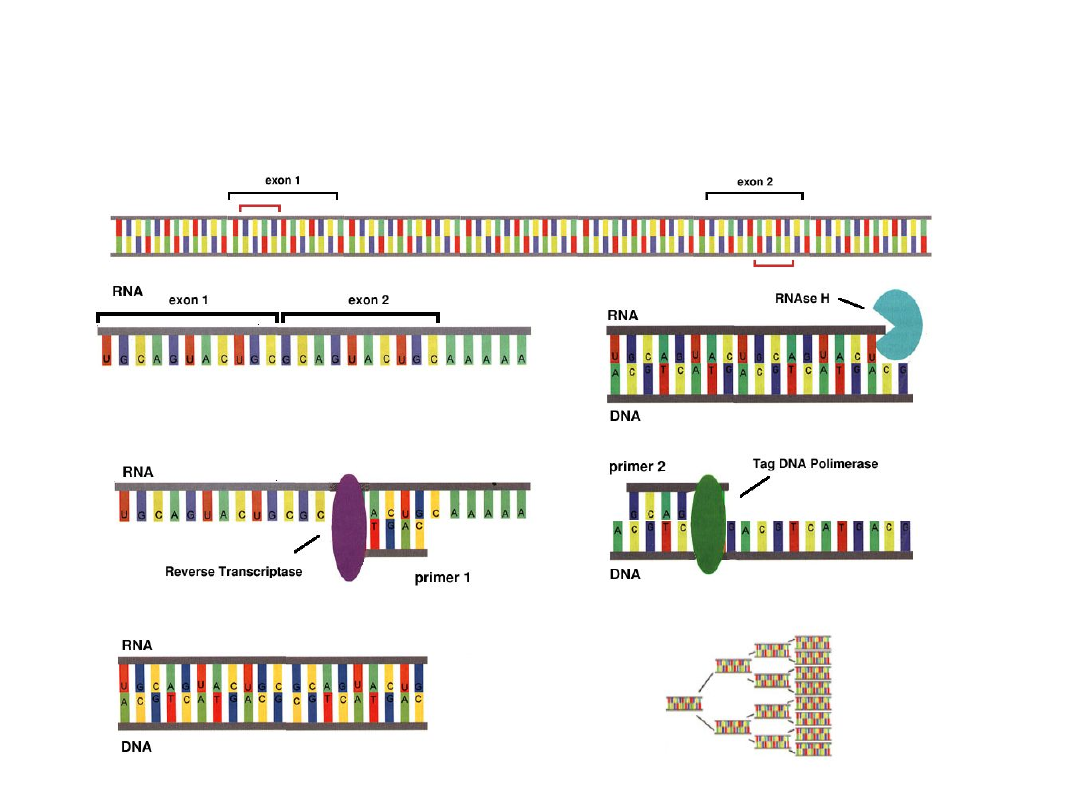
Reverse Transcription PCR

Q-PCR – PCR w czasie rzeczywistym
• Q-PCR
jest
metodą
do
ilościowego
oznaczania kwasów nukleinowych w badanej
próbie.
W przeciwieństwie do konwencjonalnego PCR
, gdzie można oznaczać jedynie obecność lub
brak produktu metoda ilościowego PCR-u
pozwala na określenie jego ilości (względnej
lub bezwzględnej)
• Za pomocą pomiaru fluorescencji w każdym
cyklu reakcji jest możliwe śledzenie jej
przebiegu.
Pierwszy
znaczący
wzrost
fluorescencji jest skorelowany z początkowa
ilością matrycy.

Monitorowanie reakcji real- time
PCR
• Do monitorowania reakcji opracowano
różne interkalujące barwniki: bromek
etydyny, SYBR Green I
• sondy hybrydyzacyjne (TaqMan,
Fluorescence Resonance Energy Transfer,
Molecular Beacons)


SYBR Green I
1. Wady:
Brak specificzności (przyłaczanie do
dimerów primerów lub
niespecyficznych produktów reakcji
PCR)
2. Zalety:
Metoda stosunkowo tania i uniwersalna

Metody real – time PCR oparte
o sondy specyficzne względem
sekwencji

• S
• s
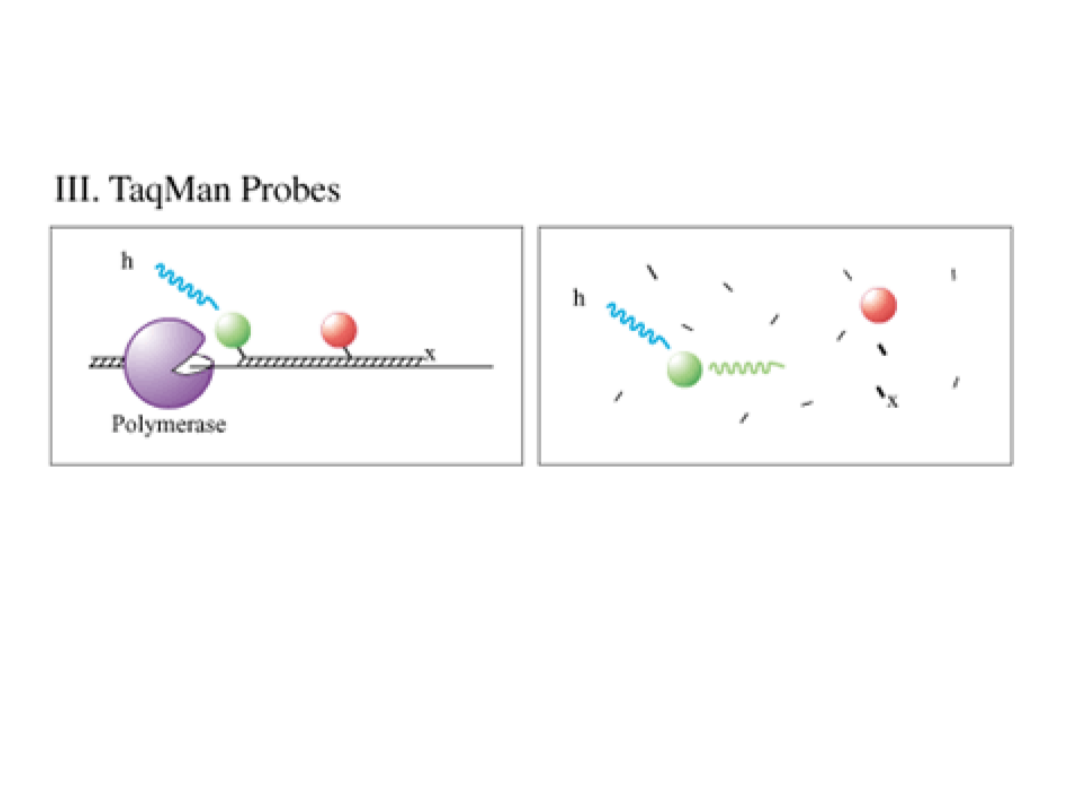
TaqMan
•Koniec 5’- barwnik reporterowy (TET, VIC)
•Koniec 3’-wygaszacz (TAMRA)
•Koniec 3’ zablokowany (fosforylowany)
•Na końcu 5’ nie może być zasady G - znosi
fluorescencję
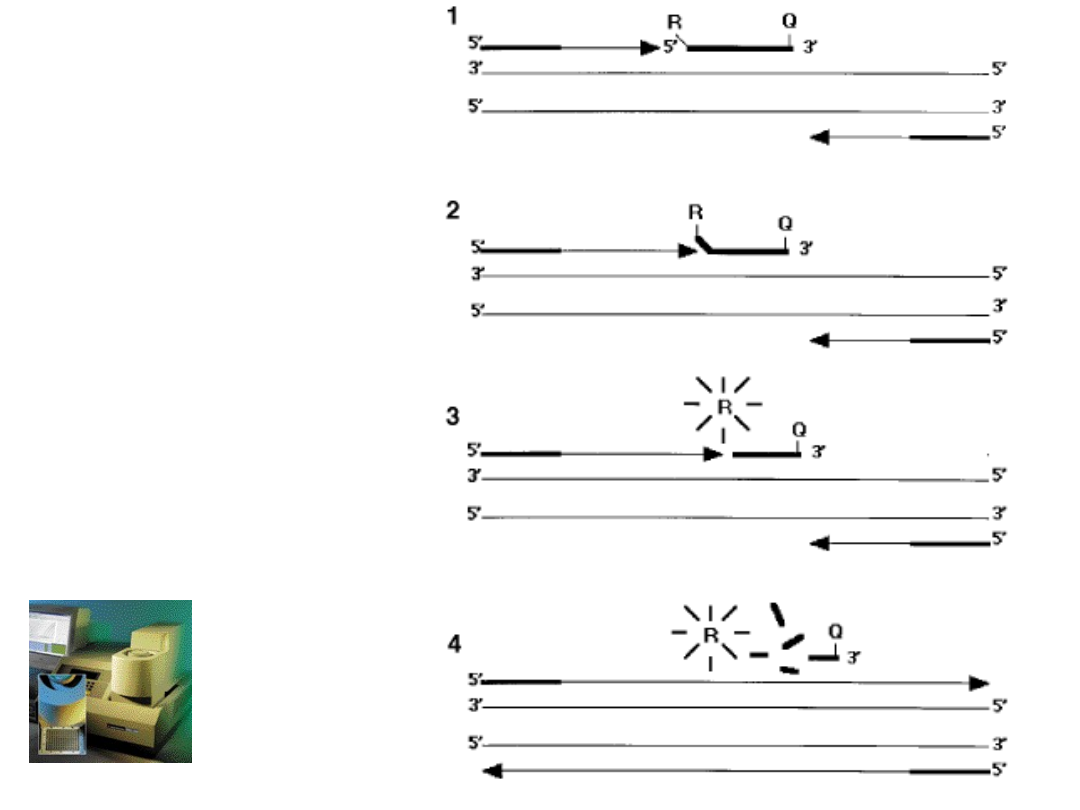
Real Time PCR
(Q-PCR)
Pozwala na śledzenie
akumulacji produktów w
trakcie przebiegu
reakcji PCR

Molecular Beacons
• Mają strukturę szpilki do włosów
• Sekwencja pętli jest komplementarna do
specyficznej matrycy
• Sekwencje pnia są komplementarne względem
siebie
• Końce 5’ i 3’ są kowalencyjnie związane z
fluorochromem i wygaszaczem
• Gdy sonda rozwija się to zaczyna fluoryzować
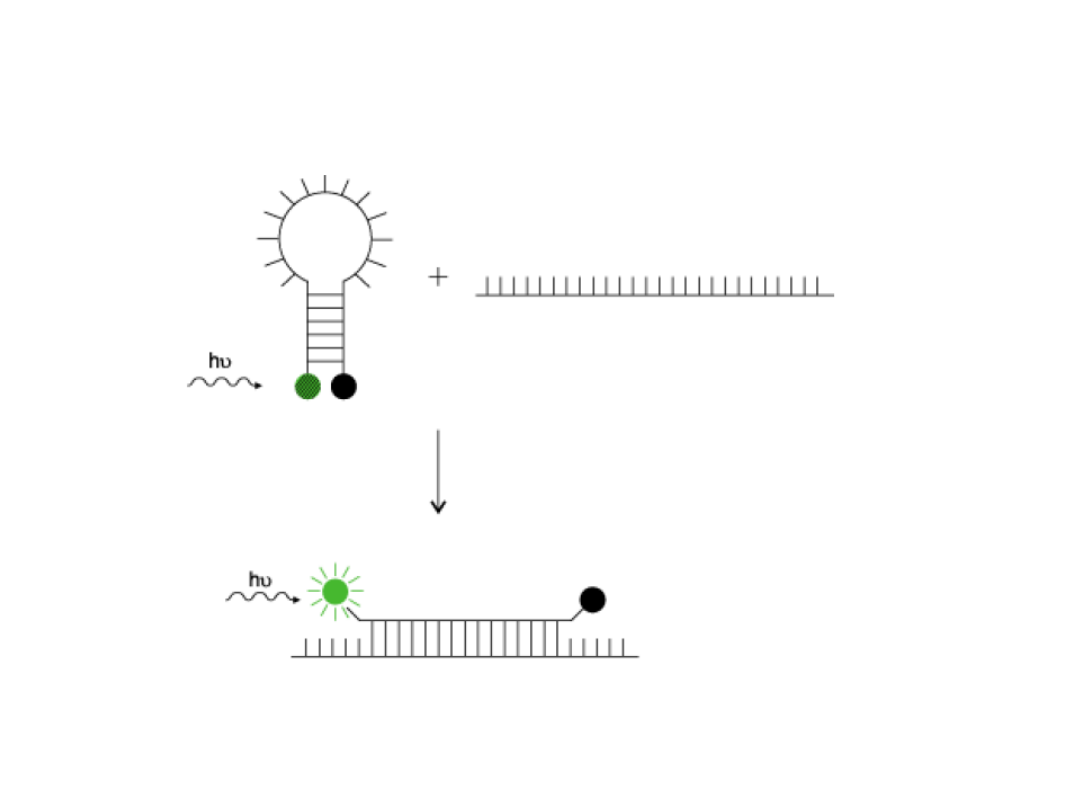
Molecular Beacons
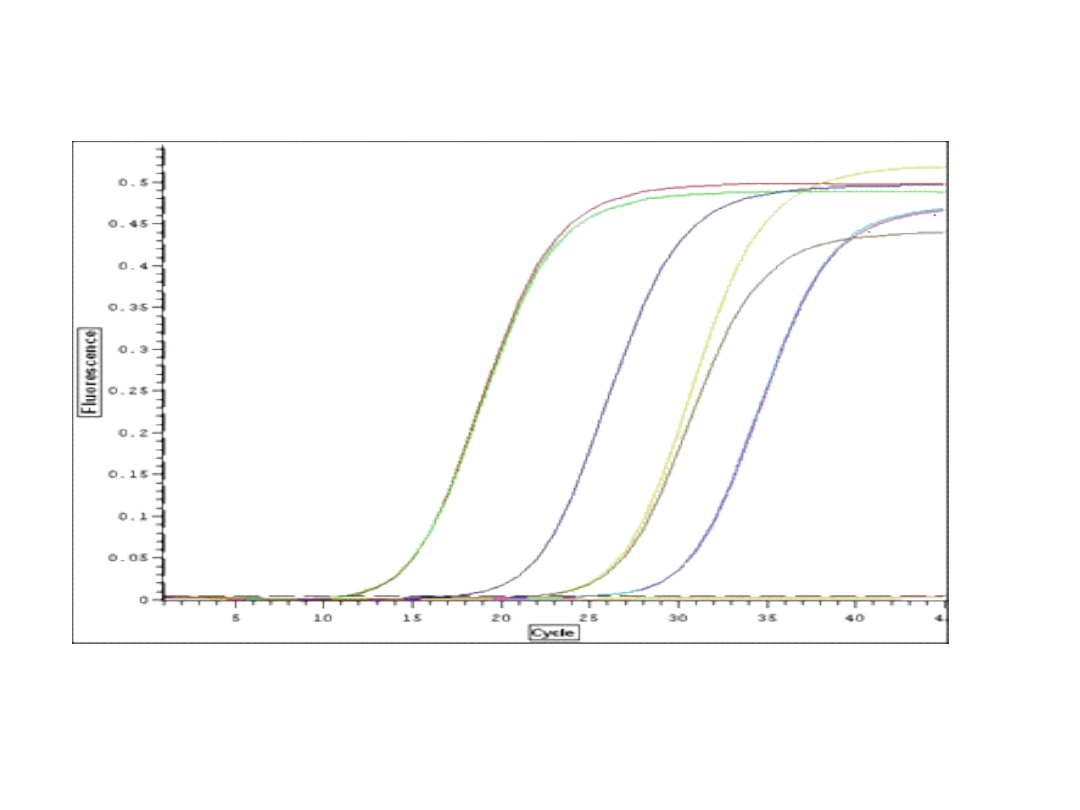
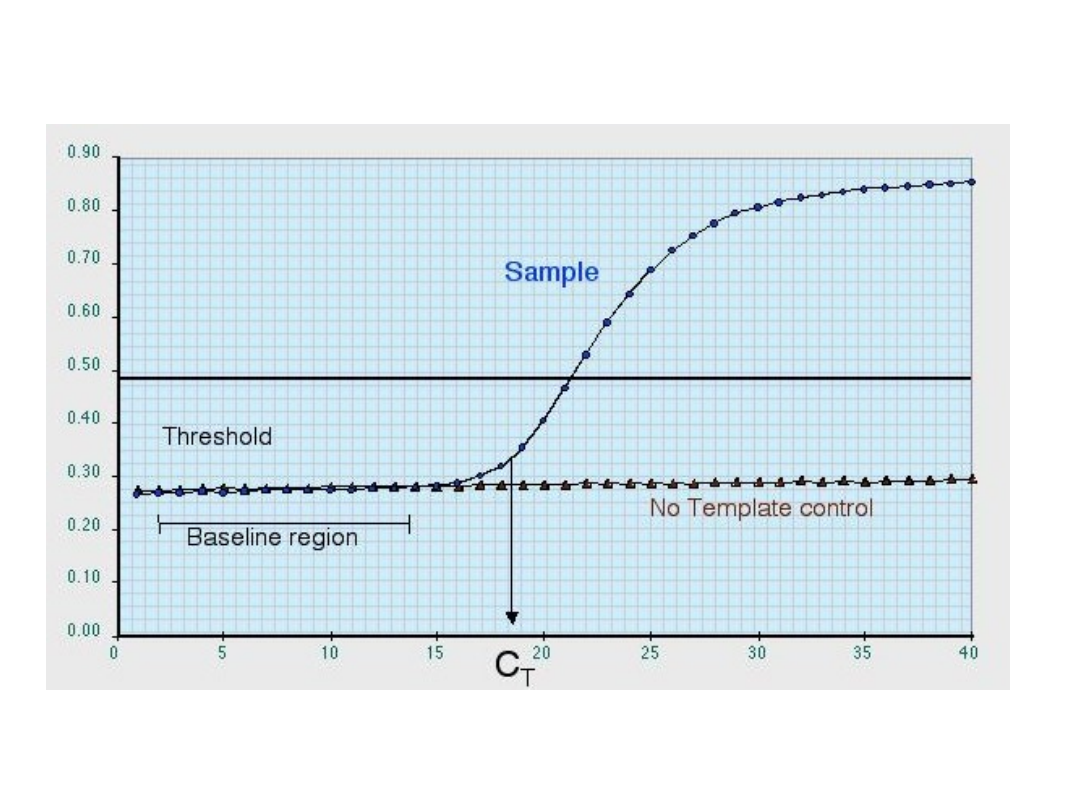
Graficzna analiza wyników reakcji PCR
Po zakończeniu reakcji otrzymujemy tzw. krzywą amplifikacji-obrazująca
przebieg reakcji QRT-PCR, czyli przyrost produktu w czasie kolejnych cykli.
W każdym cyklu dokonywany jest automatyczny pomiar fluorescencji.

Parametry krzywej amplifikacji:
• Linia bazowa (baseline) – w początkowych cyklach reakcji
ilość produktu jest zbyt mała, by obserwować jego
przyrost
• Linia odcięcia (threshold line)
• Ct (Treshold Cycle) – odzwierciedla numer cyklu reakcji
PCR w którym intensywność fluorescencji przekracza
wyznaczoną linię odcięcia.
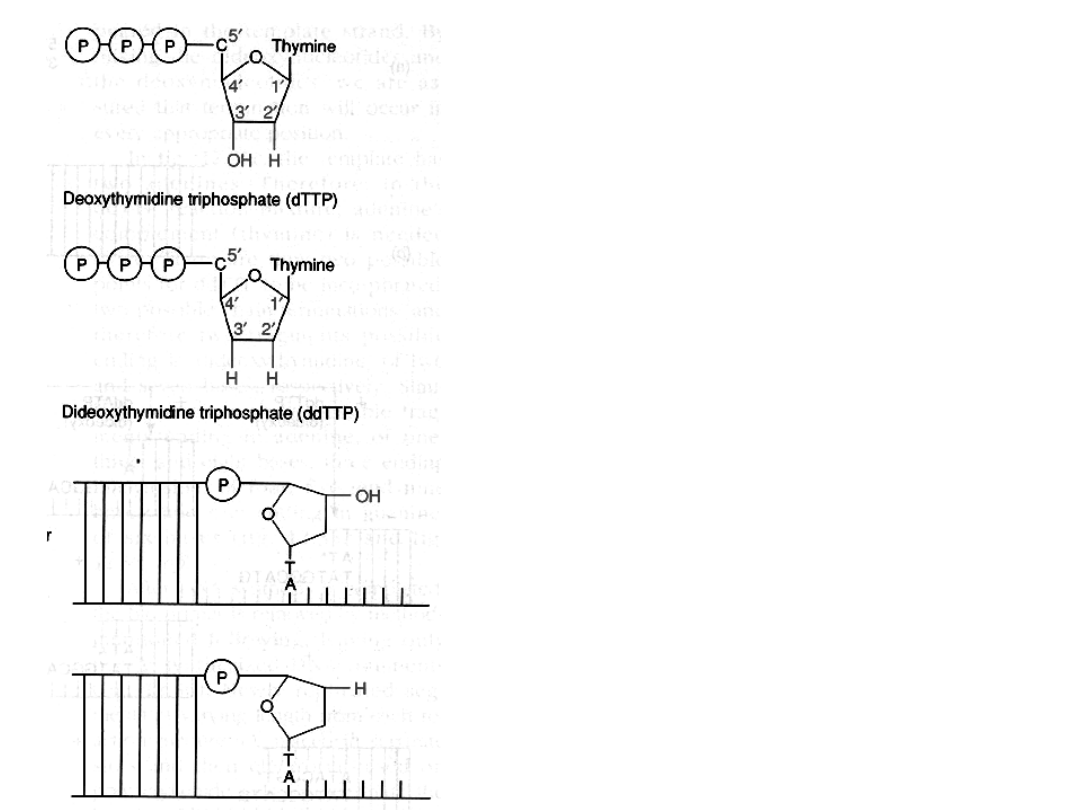
Sekwencjonowanie DNA
(metoda dideoxy-)
•Metoda oparta jest na użyciu
czterech stopujących reakcję
wydłużania łańcucha nukleotydów,
które w pozycji 3’ mają H zamiast OH
(di-deoxy: ddATP, ddGTP, ddCTP,
ddTTP).
•W klasycznej metodzie każdy
nukleotyd dodawany jest oddzielnie
do czterech prowadzonych równolegle
reakcji replikacji DNA.
•Produkty tych czterech reakcji są
rozdzielane metodą elektroforezy w
żelu akrylamidowym
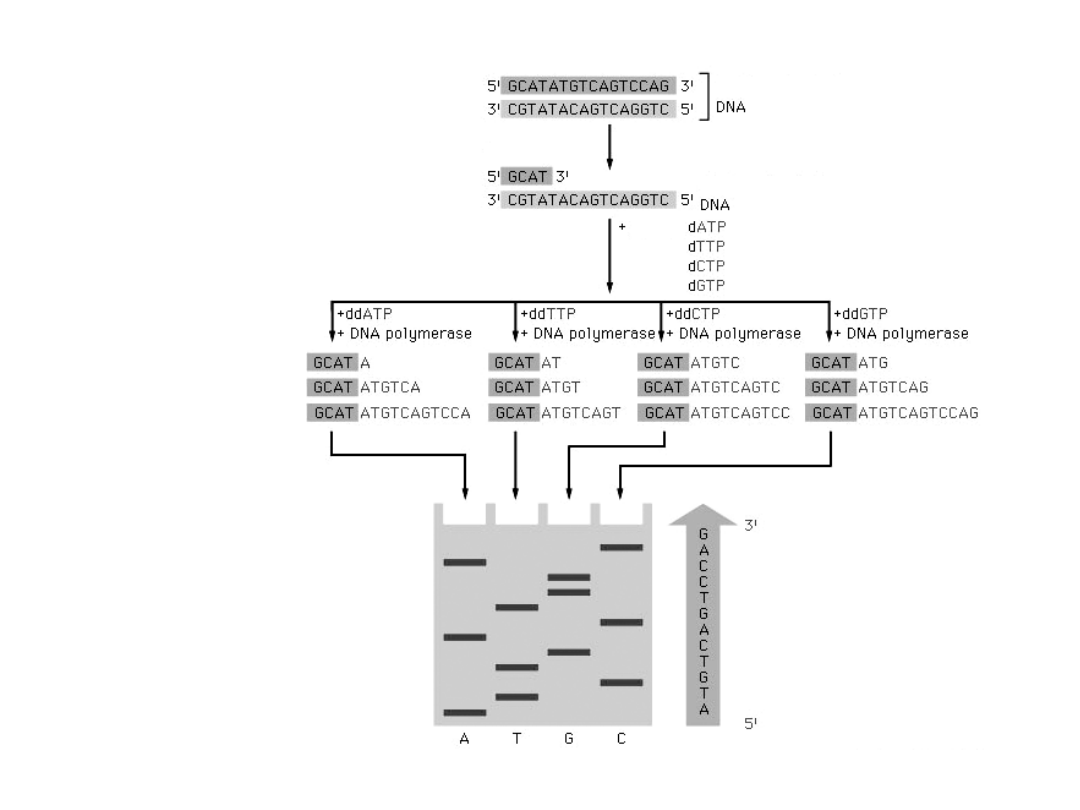
Nukleotydy
prawidłowe w
nadmiarze
Znakowany
radioaktywnie
starter
Dideoxy nukleotydy
Polimeraza DNA
Sekwencjonowanie DNA
(metoda dideoxy-)
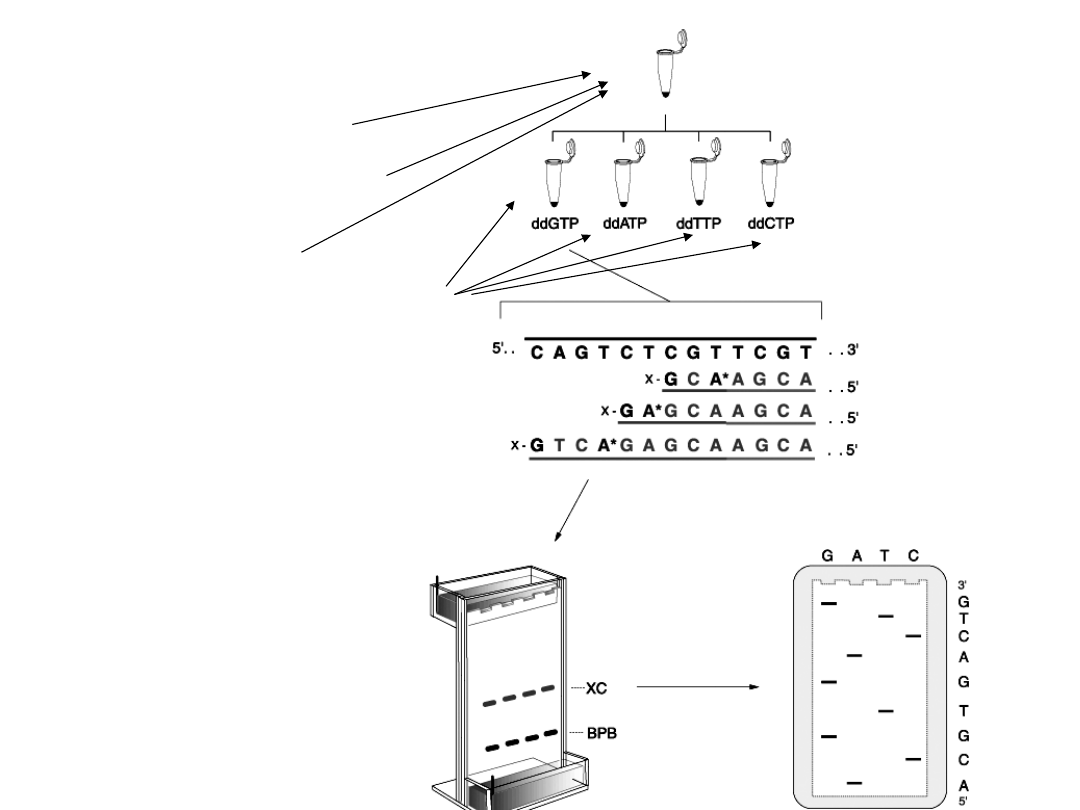
Znakowanie startera
Nukleotydy prawidłowe
Polimeraza DNA
Nukleotydy stopujące reakcję

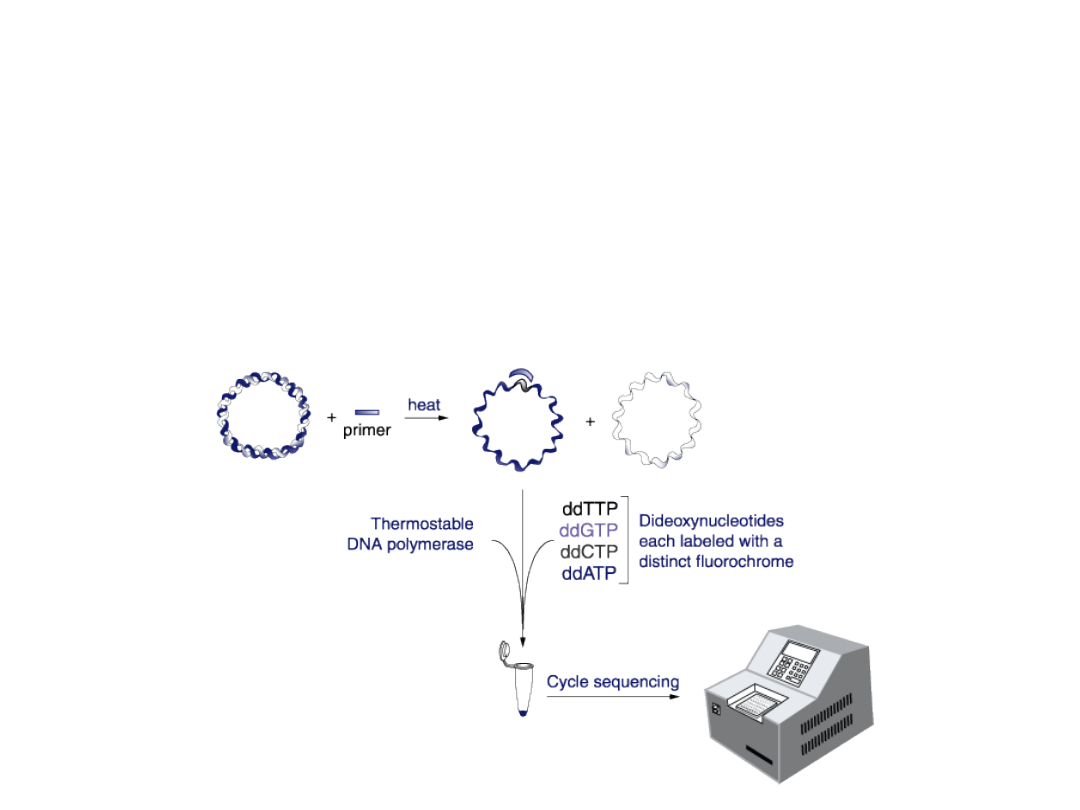
Sekwencjonowanie automatyczne
W systemie tym stosuje się dideoxy nukleotydy znakowane
czterema różnymi znacznikami fluorescencyjnymi. W systemie
stosuje się reakcję PCR startującą od jednego primera i
zatrzymującą się na nukleotydach dideoxy. Wszystkie cztery
znakowane nukleotydy dideoxy są w jednej mieszaninie. Po
reakcji PCR mieszanina nanoszona jest na żel
poliakrylamidowy a reakcja elektroforezy prowadzona jest w
aparacie wyposażonym w detektor.
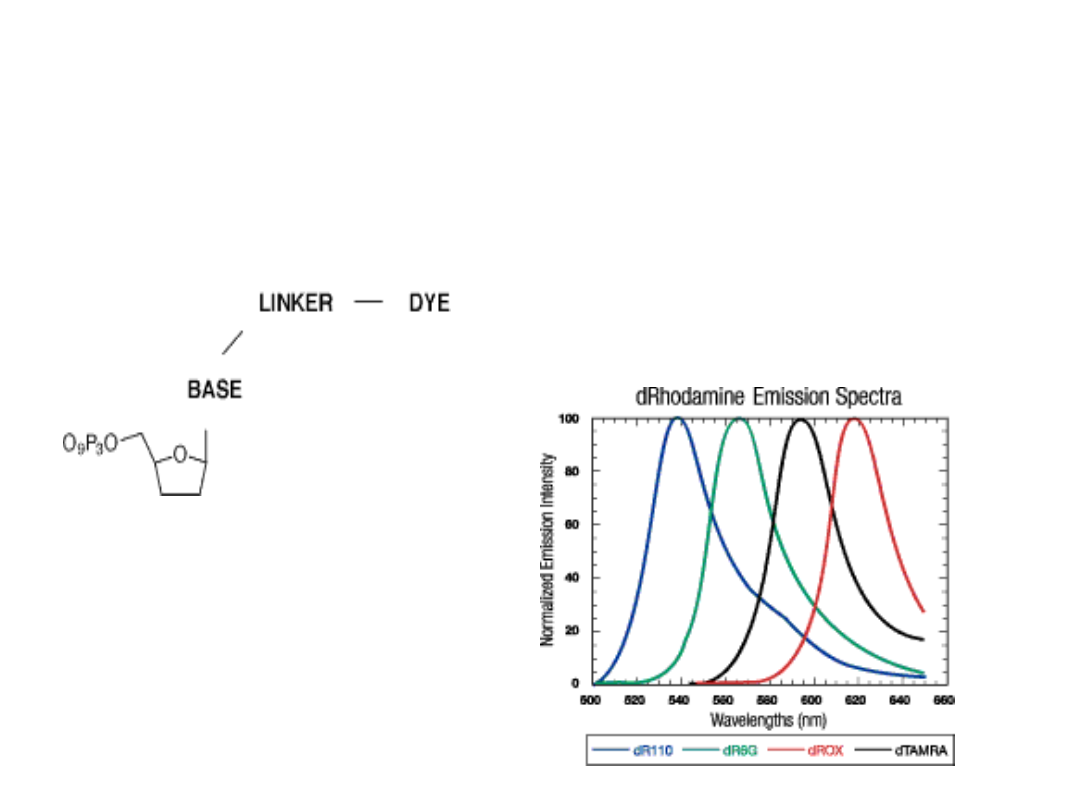
System ABI (Applied Biosystems Incorporated) używa
czterech znakowanych fluorescencyjnymi
barwnikami
ddNTPs
i polimerazy Taq (AmpliTaq).
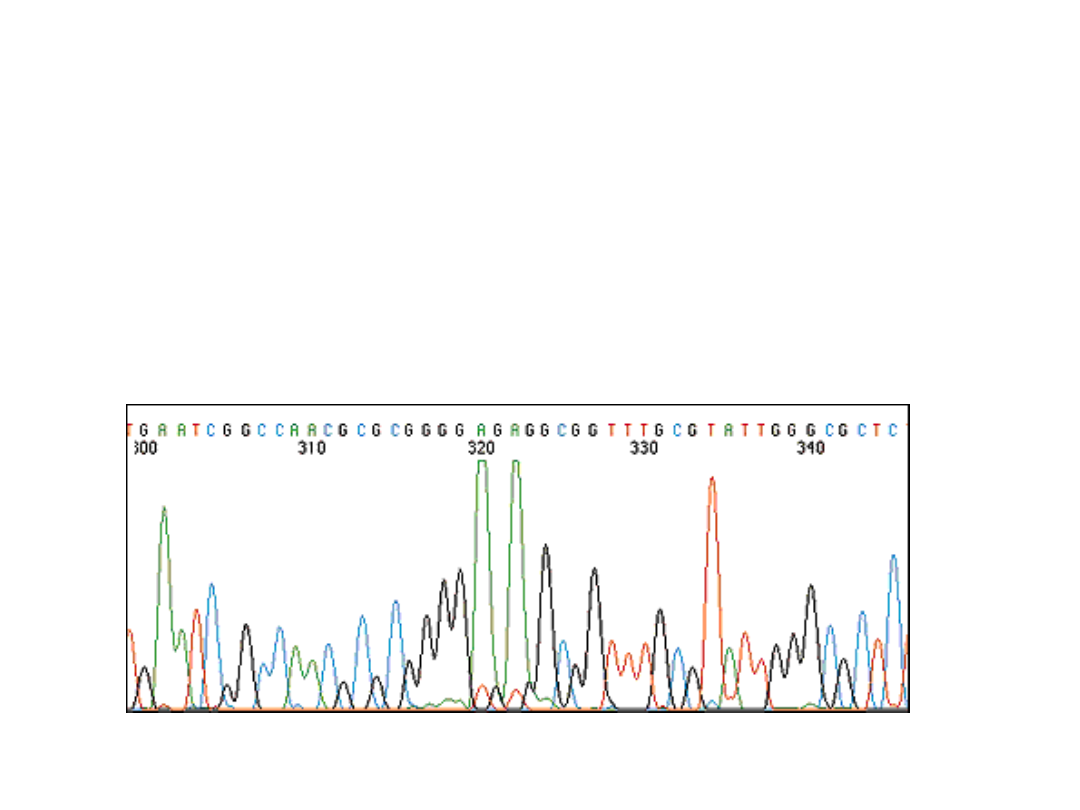
Wzbudzanie fluorochromów i detekcja emisji mają
miejsce w określonej pozycji w pobliżu dolnej części
żelu. Dane są odczytywane na podstawie spektrum
emisyjnego kolejnych pasm mijających detektor w
czasie elektroforezy. Dane podawane są do komputera
podłączonego on-line do urządzenia.

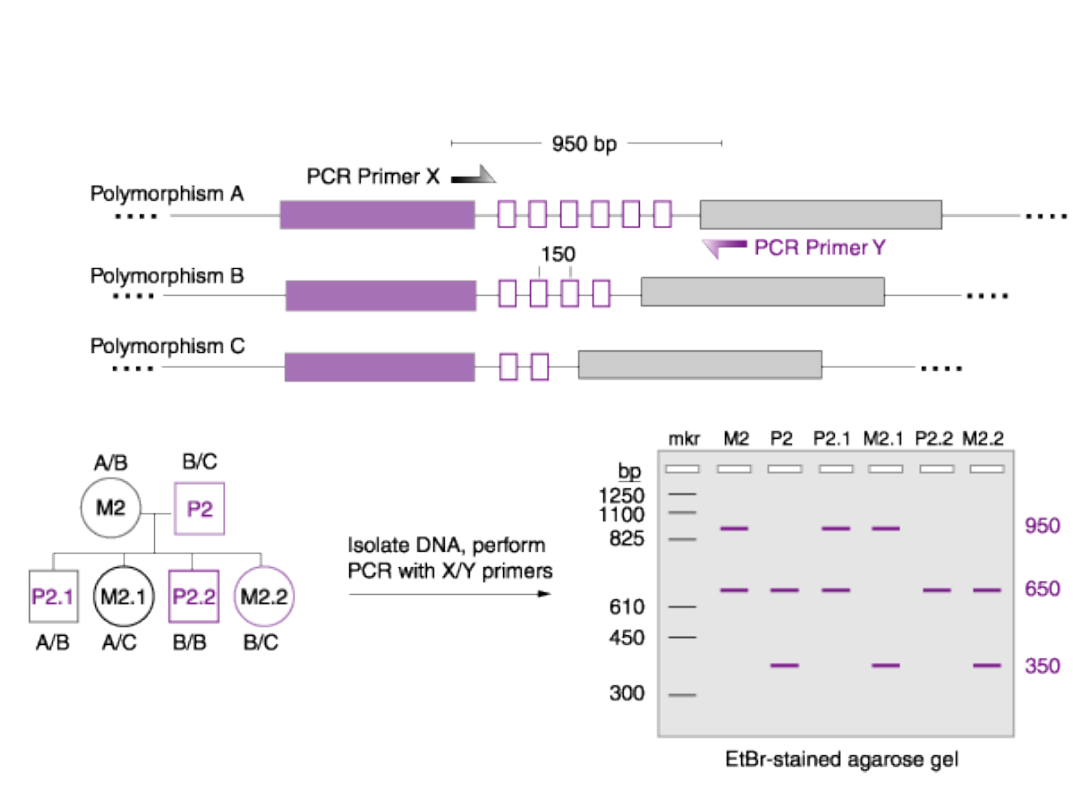
Analiza polimorfizmu powtarzalnych sekwencji DNA
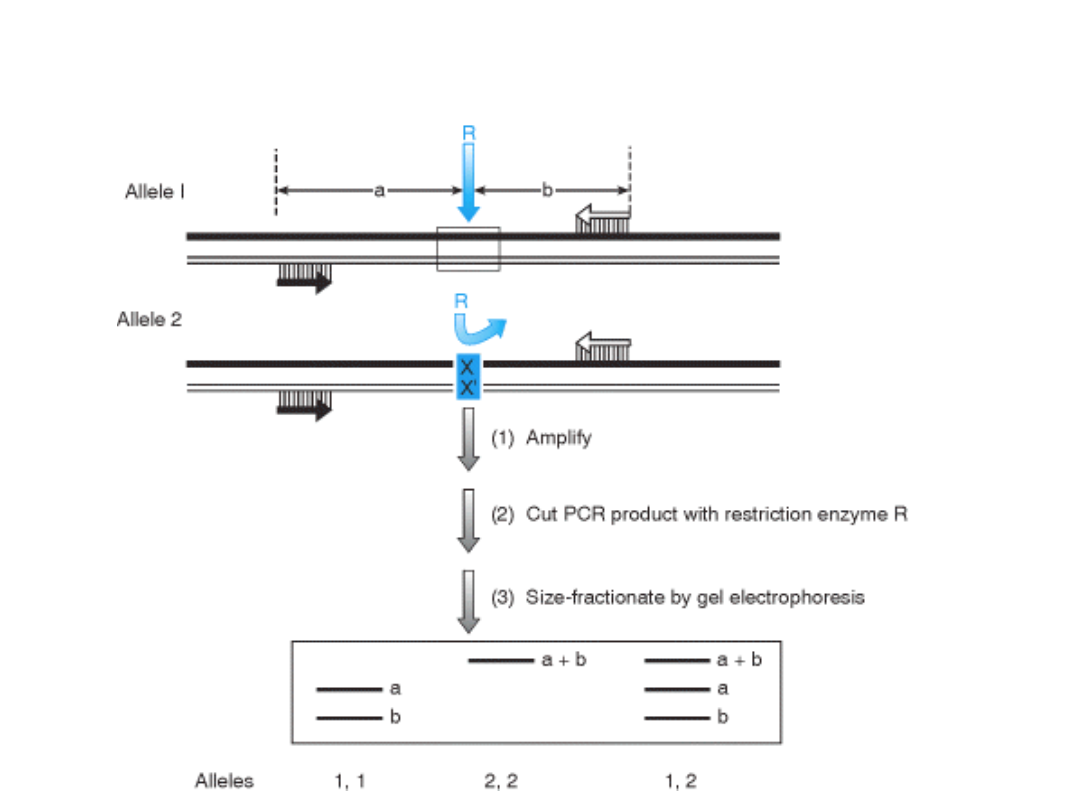
RFLP PCR – polimorfizm długości
fragmentów restrykcyjnych
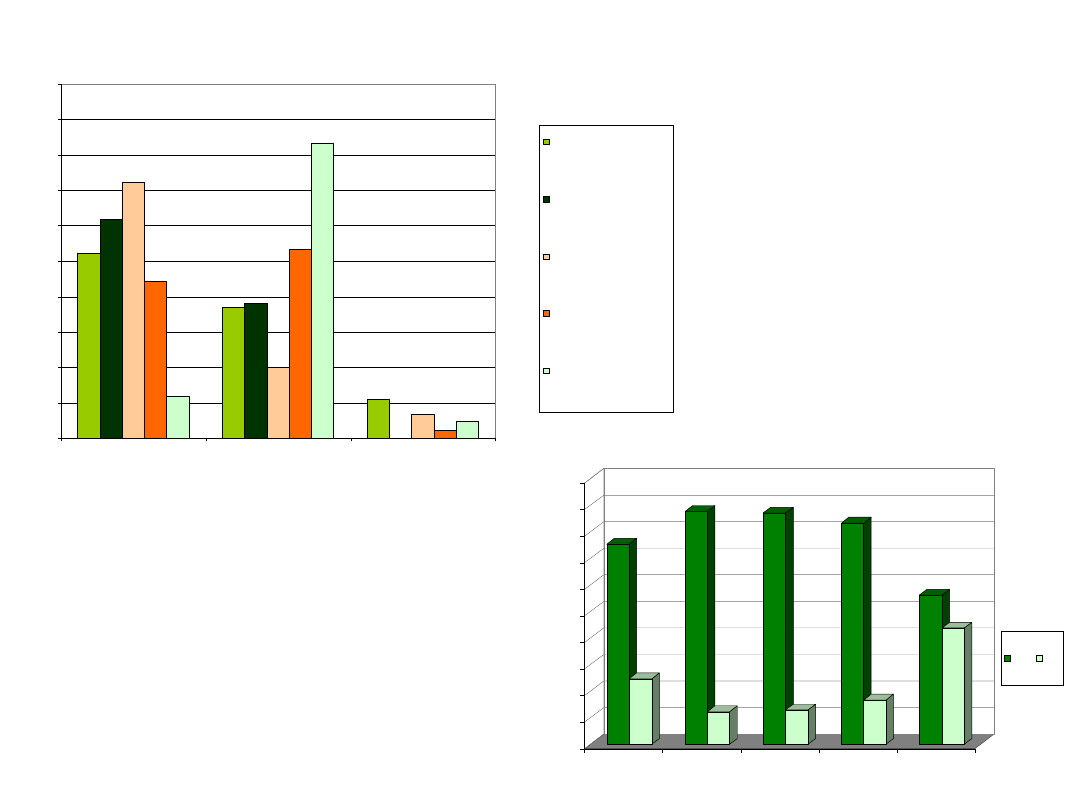
POLIMORFIZM W KODONIE 145 GENU APE1,
ROZKŁAD CZĘSTOŚCI GENOTYPÓW
0
10
20
30
40
50
60
70
80
90
100
TT(Asp/Asp)
TG(Asp/Glu)
GG(Glu/Glu)
GENOTYPY
%
B
A
D
A
N
E
J
P
O
P
U
L
A
C
JI
CHORZY Z
NOWOTWORAMI
REGIONU GŁOWY I SZYI
CHORZY Z RAKIEM PŁUC
CHORZY Z RAKIEM
SZYJ KI MACICY
CHORZY Z RAKIEM
J ELITA GRUBEGO
ZDROWI
0
10
20
30
40
50
60
70
80
90
100
CHORZY NA
NOWOTWORY
REGIONU GŁOWY I
SZYI
CHORZY NA RAKA
PŁUC
CHORZY NA RAKA
SZYJ KI MACICY
CHORZY NA
NOWOTWORY
J ELITA GRUBEGO
ZDROWI
ROZKŁAD CZĘSTOŚCI ALLELI GENU 145 APE 1
Asp
Glu
Porównanie
częstości
genotypów w
stanach
patologicznych
wskazuje na
istnienie korelacji
pomiędzy pewnymi
wariantami
genetycznymi
a chorobami
Pewne allele tego samego
genu mogą być
odpowiedzialne
za wystąpienie stanów
patologicznych lub
skłonności do określonych
patologii
A. Gdowicz 2005

• FRET opiera się na transferze energii od
fluorochromu będącego donorem do
fluorochromu bedącego akceptorem (dwie
sondy)
• Cząsteczki donora i akceptora muszą być w
bliskiej odległości (10-100A)
• Donor wzbudzany jest niebieskim światłem
• Donor znajduje się na końcu 3’-Fluorescyna
• Akceptor na końcu 5’-Red
• Sygnał mierzony jest za pomocą fluorymetru
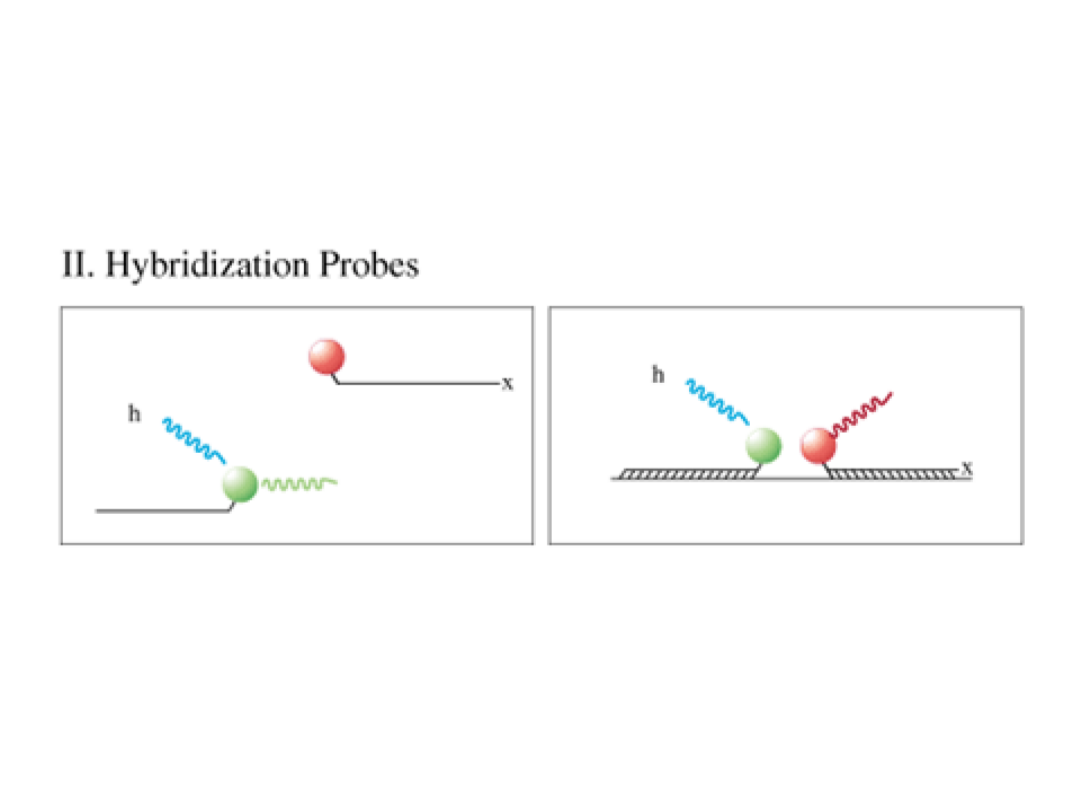
FRET
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
Wyszukiwarka
Podobne podstrony:
W4 PCR, Sekwencjonowanie
BMiGO Wykład 4 PCR, Sekwencjonowanie
Wyklad 4 PCR, Sekwencjonowanie
W4 PCR, Sekwencjonowanie
ortopedia - wykłady, W5, Chirurgia koni, wykład głowa cz.1, 22.03.2011, Chirurgia
Biol kom cz 1
Systemy Baz Danych (cz 1 2)
cukry cz 2 st
wykłady NA TRD (7) 2013 F cz`
JĘCZMIEŃ ZWYCZAJNY cz 4
Sortowanie cz 2 ppt
W5 Zawiesia
SII 17 Technologie mobilne
CYWILNE I HAND CZ 2
motywacja cz 1
więcej podobnych podstron