
AGROBIOTECHNOLOGIA
Ćwiczenia 1
Izolacja plazmidowego DNA z bakterii
Plazmid- dwuniciowe, koliste, pozachromosomowe cząsteczki DNA. Występują w formie superhelikalnej, a ich
rozmiary mieszczą się w zakresie do 200 tys par zasad. Dziedziczenie plazmidów następuje niezależnie od genu
bakteryjnego. Charakterystyczną ich cechą jest zdolność do trwałego utrzymania.
Plazmidy nie kodują funkcji, które byłyby konieczne do życia bakterii, zwiększają natomiast różnorodność
zajmowanych środowisk.
Cechy plazmidu:
-kilka tyś par nukleotydów
-jest kolisty (okrągły)
-dwuniciowa cząsteczka DNA
-na nici zlokalizowane są geny: odporności na antybiotyki
-kom bakteryjna może mieć kilka rodzajów plazmidów
Cechy przenoszone przez plazmidy:
-odpornośc na antybiotyki
-odporność na jony metali ciężkich
-katabolizm toksycznych związków
-chorobotwórczość bakterii
-interakcje z roślinami
Znaczenie roztworów i innych substancji:
-roztwór I- GTE (50mM glukoza, 25mM Tris-HCL (pH=8, 10mM EDTA) komórki bardziej pęcznieją zapewnia
odpowiednie ciśnienie osmotyczne- zwiększa, hamuje ewentualną degradacje DNA (trawienie), przygotowuje
ściany do lizy, pH 7-8.
a) roztwór TRIS, który miał na celu ustabilizowanie pH do ok. 8
b) roztwór EDETA, który ma zdolność do wiązania jonów dwuwartościowych ( nukleazy bez tych jonów nie
tworzą DNA) zmniejszona stabilność ściany komórkowej bakterii spowodowana jest wiązaniem jonów Ca2+
przez EDTA,
- roztworu II, (0,2 M NaOH, 1%SDS), pozwala na utrzymanie pH na poziomie 12. Roztwór powoduje całkowitą
lize komórek bakteryjnych, fragmentacje oraz denaturację chromosomowego DNA bakterii oraz hydrolizę
kwasów rybonukleinowych
- SDS jest silnym detergentem o ph ok. 12, niszczy błony komórkowe i denaturuje białka
-NaOH powoduje denaturację DNA i rozpoczyna hydrolizę RNA
DNA w formie CCC jest odporne na działanie SDS iNaOH
- roztwór III (7,5M octan amonu: CH3COONH3 i ph 5), powoduje obniżenie pH. Powoduje wytrącenie
chromosomowego DNA, wytrącenie białek, SDS i RNA
bromek etydyny- pozwala na obrazowanie prążków DNA w żelu oraz warstw DNA w ultrawirowaniu
jest barwnikiem fluoroscencyjnym, interkalatorem, barwi DNA. Cząsteczki EB wnikają – interkalują – do
wnętrza DNA, pomiędzy pary zasad, dzięki temu cząsteczki DNA są widoczne w UV. Zmniejsza tempo migracji
cząsteczek DNA podczas elektroforezy agarozowej o 15%.
Glukoza-zapewnienie właściwego ciśnienia osmotycznego, sprawia że komórki pęcznieją i są dobrze widoczne
Erenaza-to wyjątkowa nukleaza ale nie wymaga jonów dwuwartościowych

Zadanie
10% SPS
5M NaOH
1% SDS
0,2 M NaOH
∑= 4ml
jeśli są dwa roztwory:
większy roztwór / mniejszy roztwór
10 SDS/1 =
10x
1000:
10x
= 100 – 1000ml
0,4ml –
4ml (tyle co chcemy)
NaOH
5/0,5 =
25x
1000:
25
= 40
40ml – 1000ml
0,16
– 4ml
wynik
0,4ml+0,16+…H2O (uzupełniamy)
∑=4ml
Zadanie (ile agarowy trzeba odważyć, aby uzyskać 1,2g)
Stężenie %
100g – 100ml – 100%
1,2g – 100ml – 1,2%
x – 40ml
Zadanie
Stężenie molowe
Roztwór 3 –
7,5 molowy
octan amonu
Masa molowa 77g/mol
77g – 1mol – 1000ml
x
–
7,5mol
– 1000ml
x
– 1000ml
... – 50ml
Izolacja plazmidowego DNA metody lizy alkalicznej obejmuje 3 etapy:
1. całonocną kulturę bakterii (pożywka LB z …)
2. liza bakterii (zniszczenie błony komórkowej)
3. oczyszczanie materiału biologicznego (oddzielenie białek, RNA, DNA genowego)
Izolacja plazmidowego DNA metodą lizy alkalicznej służy do izolacji DNA z komórek bakteryjnych. Metoda ta
pozwala na otrzymanie dobrze oczyszczonego materiału. W pierwszej kolejności zachodzi liza komórek
bakteryjnych, denaturacja białek, oraz DNA. W drugim etapie "pokomórkowy gruz" jest strącany i w roztworze
pozostają cząsteczki plazmidów. Podczas oddzielania DNA plazmidowego, metoda ta wykorzystuje różnicę w
zdolności do renaturacji plazmidów i chromosomu bakteryjnego. To pierwsze ulega szybkiej renaturacji po
usunięciu czynnika denaturującego i następuje trzecia część procedury.
Otrzymane prążki (od dołu):
1. plazmid, który uległ denaturacji w formie jednoniciowej
2. forma zrelaksowana, którą tworzy dwuniciowe, koliste DNA, które jest nie bardziej rozwinięte niż CCC
3. forma superhelikalna CCC
4. forma liniowa, dwuniciowa plazmidu (powstała na skutek pęknięcia plazmidu)
5. plazmid, który posiada jedną nić przeciętą, która spowalnia migrację prążka w żelu. Obraz przedstawia kilka
prążków o różnej długości, co może być spowodowane różnym miejscem przecięcia jednej nici

Elektroforeza – to wędrówka obdarzonych ładunkiem cząstek (jonów) w polu elektrycznym. Technika
stosowana przy rozdziale, analizie i oczyszczaniu kwasu nukleinowego i białek.
Wyróżniamy żele:
1.agarozowe
2.poliakrylamidowe
-natywne
-denaturujące
Żele agarozowe:
-natywny – agarowa rozpuszcza w buforze, w którym prowadzimy elektroforezę
-denaturujące – agaroza rozpuszczona w buforze z dodatkiem substancji denaturującej
Agaroza – polisacharyd z glonów morskich, jest to frakcja agaru oczyszczana z krasnorostów.
Katoda –
Anoda +
- → + migrują kwasy nukleinowe (mają ładunek - ) do próby dodaje się LB (barwnik obciążający: nadaje próbce
kolor, migruje w stronę anody, zwiększa gęstość)
DNA i RNA mają ładunek – i zgodnie z tym ładunkiem nadawany przez grupy fosforanowe cząstki migrują w
polu elektrycznym w kierunku anody.
DNA na żelu agarowym jest barwione za pomocą bromku etydyny (interkulacje z dwuniciowym DNA lub SYBR
Gold i wizualizowane pod UV albo niebieskie światło). Bromek wnika pomiędzy nicie SYBR Gold mniejsze
ilości DNA możemy zobaczyć.
LB-barwnik obniżający
-zwiększa gęstość
-nadaje kolor
-migruje w stronę anody
Bromek etydyny- pozwala nam zobaczyć DNA , barwnik
ELEKTROFOREZA
Szybkość migracji zależy od wielu czynników:
1.masa cząstkowa DNA lub RNA (im mniejsza masa tym większa migracja)
2.stężenie agarozy (im bardziej stężone to mniejsze pory)
3.konformacji DNA lub RNA (plazmidowe przyjmuje 3 komformacje: superhelikalna CCC, liniową i koliste-
formy te z różną szybkością migrują)
4.stosowanego napięcia (jeśli zbyt wysokie napięcie to ugotujemy żel)
5.składu i siły jonowej buforu (Tris, EDTA, kwas borowy lub octowy, aby doprowadzić do odpowiedniego
pH=8)
Im mniej agarozy tym żel jest rzadszy i ma większe pory i tym większe cząstki mogą przez nie przechodzić.
DNA genomowe, roślinne, duże 0,8-1%
DNA plazmidowe 1,2-2%

ĆWICZENIA 2
Bakteriofagi λ – posiada dwuniciowe liniowy DNA o długości 48,5 kpz. Na końcach cząsteczki znajdują się
sekwencje coś o długości 12pz, które są wzajemnie komplementarne i umożliwiają one cyrkulizację cząsteczki po
tym, jak wniknie ona do komórki bakterii.
Główka
Ogon
↓
Funkcje
lityczne
↓
→
→
cos
{
cos
Sekwencje zbędne można wstawić do
23kpz
Enzymy restrykcyjne
-Bam H I
-Hind III
-Xho I
-Eco R I
enzymy restrykcyjne (kl.2)- podstawowe narzędzie inżynierii genetycznej.
-enzymy produkowane naturalnie przez wiele gatunków bakterii jako mechanizm obronny (restrykcyjno-
modyfikacyjny). Bakteria zabezpiecza swoje DNA metylując je. Dokonują tego metylazy rozpoznające te same
miejsca co odpowiadające im endonukleazy.
-enzymy tną DNA w specyficznych miejscach połączeń A,G,C i T- należą do grupy endonukleaz. Przecinają
wiązanie fosfodiestrowe.
-rozpoznając i przecinając krótkie (4-8nt) palindromowe sekwencje DNA. Sekwencja palindromowa oznacza taką
sekwencje DNA, dla której sekwencja komplementarna jest identyczna (przy założeniu, że obie sekwencje
czytamy z uwzględnieniem polarności nici; zgodnie z przyjętym obyczajem – od końca 5’ do 3’)
-przecięcie w identycznym miejscu obu nici powoduje powstanie tępych końców DNA, inne symetryczne
przecięcia prowadzą do powstania dwóch odpowiadających sobie końców lepkich.
Podział według sekwencji rozpoznawanej:
- czwórkowe – rozpoznają sekwencję DNA złożoną z czterech nukleotydów. Statystycznie w dowolnym DNA
takich miejsc jest dużo – co 256pz. Restryktazy takie mogą strawić DNA na bardzo małe fragmenty
- szóstkowe – rozpoznają sekwencję DNA złożoną z 6 nukleotydów. Dowolne miejsce restrykcyjne złożone jest z
6 nukleotydów występuje statystycznie co ok. 4096 pz w DNA, w którym ilości poszczególnych nukleotydów są
równe
- ósemkowe – stosowane niezbyt często; tną DNA bardzo rzadko
Sposób działania Eco R1 (szóstkowy)
5’ GAATTC 3’
3’ CTTAAG 5’
enzym pozostawia wysunięty koniec 5’
Nomenklatura enzymów restrykcyjnych:
- nazewnictwo opiera się na literowych skrótach
- pierwsza litera pochodzi od rodzaju bakterii
- druga i trzecie od gatunku
- następna litera oznacza szczep lub typ
- numery rzymskie oznaczają kolejne enzymy z danego szczepu lub typu
E
co
R
I
Escherichia
coli

R- szczep
I- pierwszy enzym z tego szczepu
BamHI- pierwszy enzym wyizolowany z Bacillus amyloliquefaciens szczepu H.
Neoschizomery
- enzymy restrykcyjne rozpoznające tę samą sekwencję DNA a przecinające DNA w odmiennych miejscach (np.
SmaI i XmaI)
SmaI
5’ CCC GGG 3’
3’ GGG CCC 5’
XmaI
5’ C CCGGG 3’
3’ G GGCCC 5;
Izoschizomery:
- enzymy pochodzące z różnych organizmów bakteryjnych, ale rozpoznające tą samą sekwencję i przecinające ją
identycznie. (np. SphI i BbuI)
Spoi
5’ GCATG C 3’
3’ C GTACG 5’
BbuI
5’ GCATG C 3’
3’ C GTACG 5’
Jednostki enzymów restrykcyjnych:
* jednostka enzymu restrykcyjnego to taka ilość enzymu, która trawi kompletnie 1 mikrogram DNA faga lambda
(50kb) w ciągu 1 godziny w temperaturze 37oC
* warunki reakcji :
- jony Mg2+ (kofaktor)
- bufor o wymaganym stężeniu NaCl i pH
- 37oC lub inna
-zbyt wysokie ph
-zbyt niska siła jonowa
-stezenie glicerolu>5%
-zbyt wysokie stezenie enzymu w próbce
Dodatkowo może być detergent – Tryton – X- 100 redukuje napięcia powierzchniowe, BSA – albumina z grasicy
cielęcej – zwiększa ogólne stężenie białka.
* przerywanie reakcji:
- niekonieczne
- 20 min. 65oC-80oC
- EDTA pH=8,0 (10mM)
- ekstrakcja fenolem
Warunki trawienia a niespecyficzna aktywność (aktywność starowa- star activity):
- jeżeli przeprowadza się trawienie w warunkach znacznie odbiegających od optymalnych dla danego enzymu, to
często zdarza się że obserwujemy niespecyficzne cięcia.
- enzym rozpoznaje sekwencje różniące się do sekwencji specyficznej (kanonicznej), np. o jedną zasadę
- w przypadku EcoRI, dla którego sekwencją kanoniczną jest GAATTC, takie zmienione przecinane sekwencje to
np.
C
AATTC, GAATT
G
, G
T
ATTC itp.
Modyfikacje końcowe:
-tępych –dołaczanie adaptorów (krótkich fragmentów DNA zakończonych z 1 strony na tępo z drugiej lepką,
specyficznie dla danej restryktyratów lub linkerów|(krótkich fr DNAzawierajacych określone miejsce

restrykcyjne)zakończonych tępo,przed dołączeniem tych ostatnich należy zmetylowac genomowi DNA od
poprzedniego metylaza a po kougacja strawić DNA dana restryktazą uzyskując lepkie końce
- lepkich- które mają cofniętą nić 3’ – można wpełnić przy pomocy fragmentu Klenowa polimerazy DNA Ecoli i
dNTP.
-lepkich, które mają wystającą nić 3’ można wytrącić stosując polimerazę DNA bakteriofagi T4.
-lepkich, które mają cofniętą nić 5’ można je wytępić obcinając jednoniciowy odcinek DNA przy pomocy
nukleozy S1 lub polimerazy 1 E coli wykorzystując jej aktywność egzonukleolityczną 3’->5’
Wykorzystanie mapy restrykcyjne-, czyli obraz cząsteczki z zaznaczonych miejscami rozpoznawalnymi przez
różne ER z wzgl.. odległości między nimi
Porównanie DNA z różnych organizmów:
-izolacja
-izolacja i identyfikacja genów
-rekombinowanie i klonowanie genów
-badanie polimorfizmu miejsc restrykcyjnych (RFLP)
-identyfikacja mutacji, diagnostyka chorób genetycznych
ĆWICZENIA 3
27.03.2010r
PCR - łańcuchowa reakcja polimerazy to technika opublikowana 1 1885 r. obecnie stosuje się głównie
taqpolimeraze PCR jest potrzebne :
1.matryca DNA
2. startery
3.wolne nukleotydy
4.polimeraza(najczęściej taqpolimeraza.)
Etapy PCR:
- denaturacja – rozdzielenie dwuniciowej DNA (94
0
)
- przyłączanie starterów – temp. Topnienia starterów
- wydłużanie starterów (72) – optymalna temp. dla działania polimerazy
Czynniki wpływające na PCR:
-stężenie matrycy
Ilość matrycowego DNA mieści się w zakresie.
Plazmidowy lub fagowy DNA 0,01-1mg.
Genowy DNA 0,1-1mg.
Większą ilość matrycowego DNA, zwiększa ilość niespecyficznych łańcuchów PCR.
……5’ nić nie matrycowa (kodująca, sensowna) 3’…..
3’ ← 5’ primer prawy (reverse)
……3’ nić matrycowa (antysensowna) 5’
5’ → 3’ primer lewy (forward, antysens)
Wolne nukleotydy – muszą być w mieszaninie, polimeraza jest potrzebna – (jest stabilna w wysokiej temp) +
bufor (jony magnezu – są istotne)
1,2% 40ml
1,2g – 100ml – 1,2%
x – 40ml
↓

0,48g – 40ml – 1,2%
Każdy cykl reakcji:
1.termiczna denaturacja powierzchniowego DN (temp 94-95 stC w obecności nadmiaru kazdego z 2 oligo
nukleotydów i 4 dNTP)
2.Hybrydyzacja primerów do sekwencji komplementarnej z matryca w 40-60 st CC przez 20-40 sek
3.wydłużenie startera od końca 3’-OH przez sukcesywne dosyntetyzowanie dNTP w temp 72°C. Proces ten jest
katalizowany przez polimerazę DNA (1min – 1000pz – 1kpz)
Do reakcji PCR potrzebne jest:
1.matryca DNA (lub RNA)
2.startery (dwa różne)
3.wolne nukleotydy (dNTP)
4.polimeraza (najczęściej Tag polimerazę)
5.jony Mg
2+
,Mn
2+
, bufor
Czynniki chemiczne
1.stezenie matrycy -ilość matrycowego DNA mieści się w zakresie;
- plazmidowe lub fagowe DNA 0,001-1ng
- genomowe DNA 0,1 – 1 ug
Większa ilość matrycowego DNA zwiększa ilość niespecyficznych produktów PCR.
Jeśli matrycy jest za mało,to powstanie bardzo słaby prążek.
2.wolne nukleotydy
w mieszaninie replikacyjnej każdego DTP jest równa ilość (zwykle po 0,2mM). Nierówna ilość DTP redukuje
wydajność i dokładność amplifikacji. Optymalne stężenie dNTP zależy od:
-długości amplifikowanych produktów
-stężenie MgCl
2
(Mg
2+
łączy z nukleotydami i daje rozpuszczony kompleks, który jest substratem dla polimerazę,
stężenia starterów)
3.jony dwuwartościowe
Stezenie końcowe 0.5 do 5 mM. Jony dwuwartościowe tworzą nierozpuszczalny kompleks. Jak podwyższymy
steżenie dNTP to steżenie jonów też
Jeżeli próbka DNA zawiera EDTP (np. jeśli DNA zostało zawieszone w buforze TE, a nie w wodzie) to
zawartość jonów magnezu powinna proporcjonalnie wzrosnąć.
Stężenie chlorku magnezu zależy od:
-stężenia dNTP
-ilość EDTA w mieszaninie reakcyjnej)
4.polimeraza Tag . stezenie 0,5 do 2,5 unita. Wyższe steżenie polimerazy powoduje powstawanie
niespecyficznych produktów, a także powoduje niekorzystne zwiększenie lepkości roztworu, bo polimerazę Tag
jak każdy enzym zawieszona jest w gricerolu.
6.bufor
Czynniki fizyczne wpływające na PCR:
1.rodzaj termocyklera (grzanie od góry, dokładne utrzymywanie parametrów, powtarzalność)
2.probówki (cienkościenne do PCR, muszą pasować do aparatu)
Program PCR:
32 c
ykli
2-5min (2°)
94°C
1-60s (30°)
94°C
30-60s (30°)
54°C
30-90s (30°)
77-74°C
5-10min (5°)
72°C
Bez ograniczeń
4°C
Liczba zsyntetyzowanych fragmentów DNA rośnie wykładniczo jak 2n, gdzie n to liczba cykli.
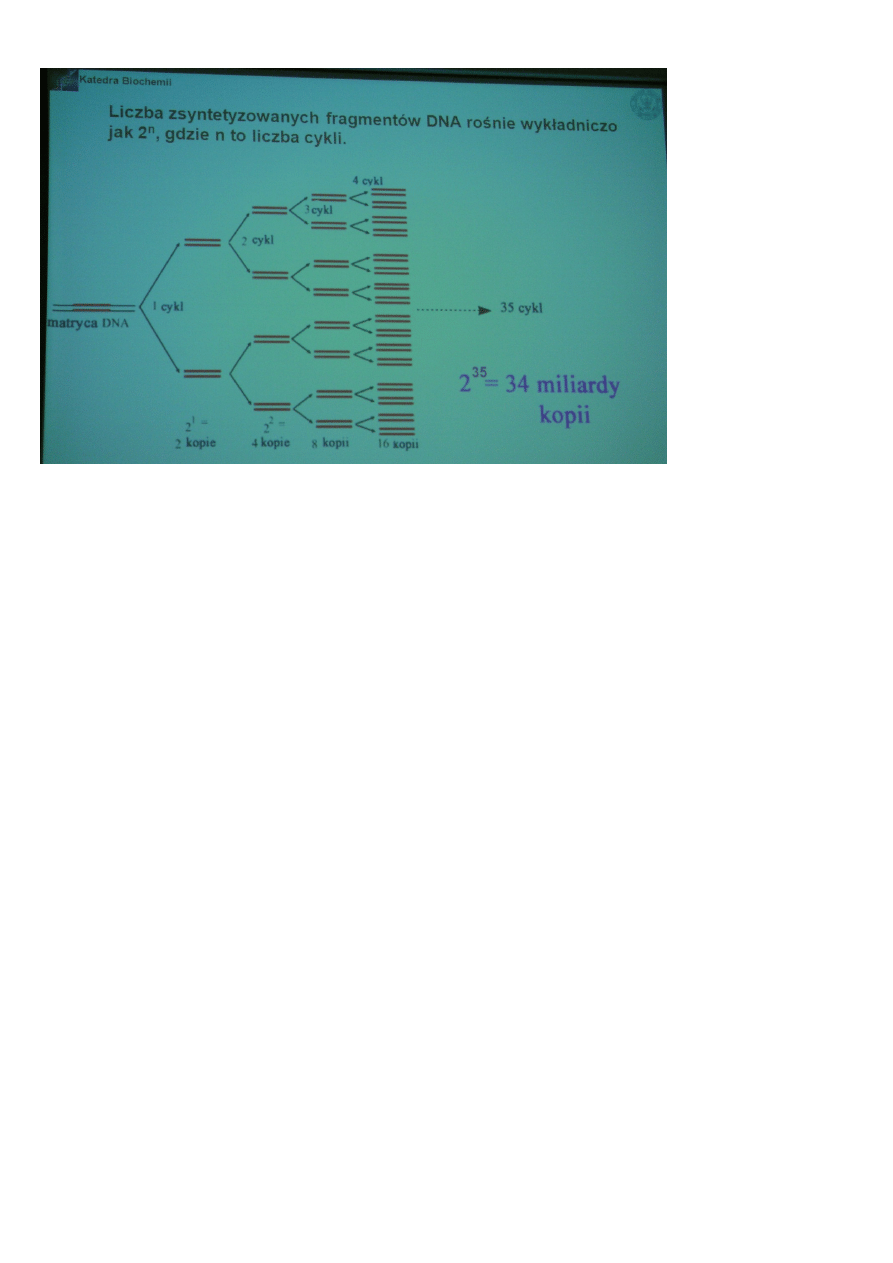
35 cykli przy jednej cząsteczce DNA
2
35
= 34 miliardy kopii
Temperatura topnienia-temp dysocjacji dupleksu starter-matryca
Obliczanie Tm
Tm=[4x(G+C) + 2x(A+T)]
Startery – krótkie fragmenty oligonukleotydowe (16-24pz) komplementarne do matrycy oskrzydlające fragment
DNA, który chcemy amplifikować, najczęściej to ich sekwencja i stężenie decyduje o powodzeniu reakcji
- powinny mieć wyrównaną ilość zasad G/C i A/T
- Tm 50-60oC (temperatura dysocjacji dupleksu starter – matryca oznaczona jako temperatura topnienia
Tm)(Tm=[4x](G+c)+2x(A+T))
- nie powinny zbyt silnie wiązać się na końcu 3’ (unikać CCC lub GGG na końcu 3’):
- specyficzność końca 3’ decyduje o powodzeniu amplifikacji:
- nie powinny tworzyć wewnętrznych struktur 2-rzędowych
- nie mogą komplementować między sobą na końcu 3’
- stężenie 0,1-0,5mikroM (za dużo – pomyłki, za mało- mniej produktu)
- dNTP – wolne nukleotydy (ATP, GTP, CTP, TTP). W mieszaninie reakcyjnej każdego dNTP zwykle po
200mikroM. Nierówna ilość dNTP redukuje wydajność i dokładność amplifikacji. Optymalne stężenie dNTP
zależy od długości amplifikowanego produktu, stężenia MgCl
2
, stężenia starterów.
- dodajemy również MgCl
2+
– zwykle 0,5 do 5 mM. Tworzy kompleks z dNTP dając substrat dla polimerazy.
Podwyższając stężenie nukleotydów należy zwiększać stężenie jonów Mg
2+
. Nukleotydy redukują pulę wolnych
jonów Mg2+ wpływając w ten sposób na aktywność polimerazy i hybrydyzację starterów. Za dużo jonów Mg
2+
zwiększa ilość niespecyficznych produktów PCR i obniża wierność (zgodność) syntezy. Jeżeli próbka DNA
zawiera EDTA zawartość jonów Mg
2+
powinna proporcjonalnie wzrosnąć.
- kolejny odczynnik – polimeraza Taq – zwykle 0,5 – 2,5 unita.
Wewnętrzne struktury starterów:
- spinka do włosów
- self- dimer
- dimer w wyniku połączenia startera, który ma pracować na jednej nici z innym, który ma pracować na drugiej
nici.
Wyszukiwarka
Podobne podstrony:
Ćwiczenia agrobiotech 1 3
sprawozdanie cwiczenie 22, studia, agrobiotechnologie
AGROBIOTECHNOLOGIA ĆWICZENIA
Agrobiotechnologia ćwiczenie 1
Ćwiczenie nr 1 koncowe, studia, agrobiotechnologie
sprawozdanie cwiczenie 2, studia, agrobiotechnologie
METODY BIOTECHNOLOGICZNE W OCHRONIE SRODOWISKA CWICZENIE 1, studia, agrobiotechnologie
Agrobiotechnologia ściąga z ćwiczeń
sprawozdanie cwiczenie 22, studia, agrobiotechnologie
3 ćwiczenia BADANIE asfaltów
Ćwiczenie7
Cwiczenia 2
Ćwiczenia V
więcej podobnych podstron