
Wykorzystanie genetyki
w hodowli zwierząt

• Kontrola pochodzenia
• Możliwość regulacji płci u zwierząt
• Tworzenie genetycznych map markerowych i
ich wykorzystanie w analizie sprzężeń z
genami cech istotnych ekonomicznie
• Wykrywanie genów o dużym efekcie (geny
główne)
i wprowadzanie ich do różnych ras zwierząt
• „Genotypowanie” pod kątem genów cech
istotnych ekonomicznie
• Szacowanie zmienności genetycznej (dystans
genetyczny)
• Szacowanie spokrewnienia między
osobnikami oraz ich homozygotyczności
• Tworzenie szczepów wsobnych zwierząt
laboratoryjnych
i wyspecjalizowanych linii zinbredowanych
zwierząt gospodarskich
• Tworzenie ras zwierząt i linii syntetycznych

• Wykrywanie i ograniczanie
występowania wad wrodzonych
• Wykrywanie aberracji
chromosomowych (badania
cytogenetyczne i molekularna
analiza DNA)
• Diagnostyka molekularna chorób
genetycznych i infekcyjnych
• Poszukiwanie wskaźników
oporności zwierząt na choroby
• Uzyskiwanie zwierząt odpornych na
choroby za pomocą metod
genetycznego doskonalenia

Kontrola pochodzenia – „wczoraj i
dziś”
Grupa krwi
Elektroforetyczne warianty białek surowicy krwi i erytrocytów
(genotypy)
(fenotypy)
A D P Q
TF ALB ES CA PGD PHI PI
źrebię
a/ce cgm/dghmp ac/ad abc/b
HO AB I E FS I SU
klacz
adf/ce dghmp/adl a/ac -/abc
DH B FI E FS I UZ
ogier 1
ce/b cgmr/bcmq ac/d ac/b
DR B ES E DS L ST
ogier 2
a/bc dkl/cgm d/ad -/b
DO A I E F I SU
Bydło i owce grupy krwi
Konie i świnie grupy krwi i polimorficzne białka

Kontrola pochodzenia – „dzisiaj i
jutro”
Wykorzystanie
sekwencji
mikrosatelitarnych
:
• PCR
• hybrydyzacja
Wykorzystanie sekwencji
minisatelitarnych :
• hybrydyzacja
Wykorzystanie polimorfizmu
pojedynczych podstawień - SNP :
• PCR-RFLP
• technika mikromacierzy
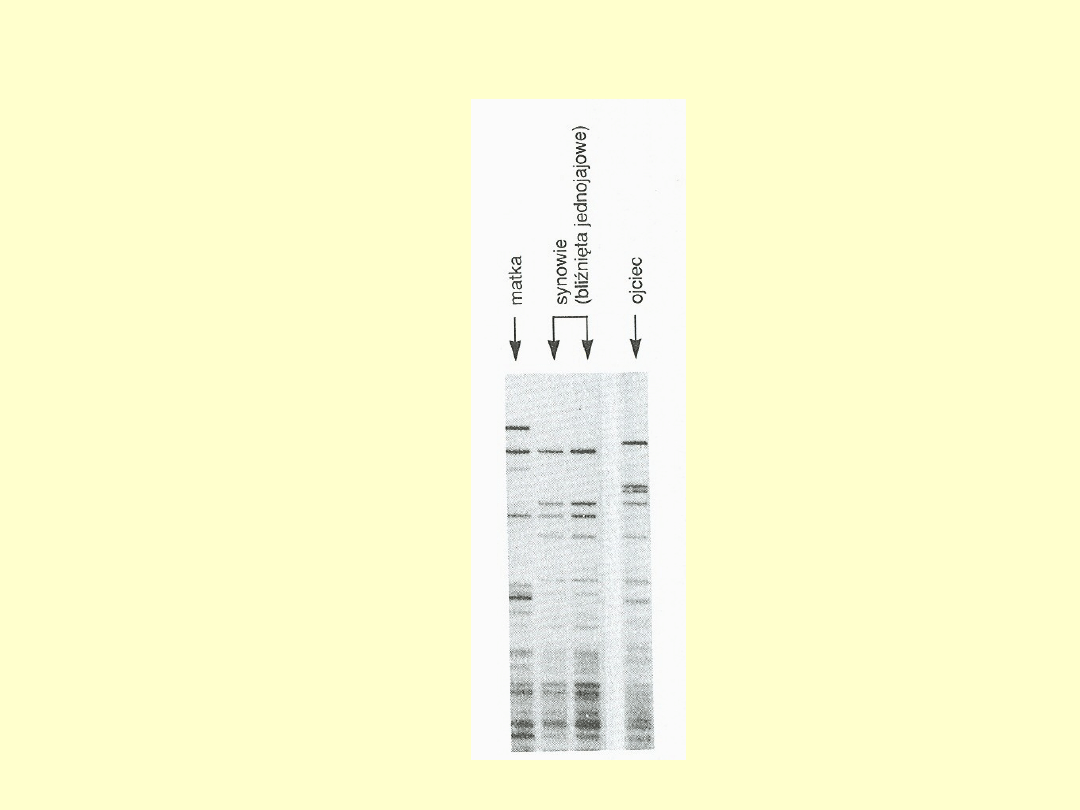
Kontrola pochodzenia

Regulacja płci
2. Określanie płci zarodka
(po zapłodnieniu in vitro lub „wypłukaniu” zarodków
z dróg rodnych
samicy) :
>
metody cytologiczne
>
metody immunologiczne
>
metody molekularne :
1. Segregacja plemników na frakcje męską i żeńską –
cytometr przepływowy
wykorzystanie w zapłodnieniu in vitro
۵
hybrydyzacja
- DNA zarodka z sondą specyficzną dla chromosomu Y
- in situ z sondą specyficzną dla chromosomu Y
(na preparacie mikroskopowym)
۵
PCR
- amplifikacja sekwencji DNA zlokalizowanej w chromosomach X
i Y (gen AMLG;
ZFX– ZFY)
- amplifikacja sekwencji DNA zlokalizowanej w chromosomie Y
(np. gen SRY)

Tworzenie genetycznych map markerowych i ich wykorzystanie
w analizie sprzężeń z genami cech istotnych ekonomicznie
• Mapa fizyczna :
- odległość między loci -
liczba par
zasad [pz] między danymi
loci
• Mapa genetyczna
(
częstość zjawiska crossing
over) :
– odległość
między loci –
wartość
funkcji
Kosambiego
– jednostka odległości : 1
cM (centiMorgan) – jeden
crossing over na 100
mejoz

Nowa generacja map - mapy radiacyjne
Odległość między loci – w cR
Większe nasycenie mapy
loci markerowymi

Przykłady genów o dużym efekcie
(geny główne)
gatune
k
gen główny - cecha
produkcyjna
gen główny
- zdrowotność
Bydło
Owca
Świnia
Koń
Gen -kazeiny – wydajność
białka
w mleku
Gen BMPR-IB – liczba
jagniąt
w miocie
Gen hormonu wzrostu GH
– wydajność
mięsna
????
Gen ITBG2 –
choroba
BLAD
Gen FGFR3 – zespół
„pajęczy”
Gen RYR1 –
gorączka
złośliwa
(syndrom
stresowy)
Gen DNA-PK –
ciężki
złożony brak
odporności (SCID)

Wykrywanie genów o dużym efekcie
(geny główne)
„Genotypowanie” pod kątem genów cech istotnych ekonomicznie
najczęściej PCR i PCR-RFLP
Genotyp w locus -
kazeiny
wariant A i B :
a – marker
b – produkt nie
strawiony
1 – trawienie enzymem
HinfI
2 – trawienie enzymem
HindIII

G
XY
Dr = - ln ----------------
(G
X
G
Y
)
1/2
G
X
= (2n
x
x
i
2
- 1) / (2n
x
- 1)
G
Y
= (2n
y
y
i
2
- 1) / (2n
y
- 1)
G
XY
= x
i
y
i
x
i
i y
i
- frekwencje i-tego allelu w locus
w populacjach X i Y
n
x
i n
y
- liczebność osobników w populacjach X i Y
Szacowanie dystansu genetycznego
(Dr)
na podstawie polimorfizmu sekwencji mikrosatelitarnych
oraz polimorfizmu białek i antygenów erytrocytarnych

Wzrost homozygotyczności – depresja
inbredowa
– wpływ na strukturę genetyczną populacji
Dziedziczenie w pełni addytywne A
1
= 5 A
2
= 2
Genotyp Wartość Frekwencja genotypów
genotypowa P
0
P
1
[F=0,25] P
2
[F=0,375]
-------------------------------------------------------------------------------------------------
A
1
A
1
10
0,25
0,3125
0,34375
A
1
A
2
7
0,50
0,375
0,3125
A
2
A
2
4
0,24
0,3125
0,34375
P0
= 10 x 0,25 + 7 x 0,50 + 4 x 0,25 = 7,0
P1
= 10 x 0,3125 + 7 x 0,375 + 4 x 0,3125 = 7,0
P0
= 10 x 0,34375 + 7 x 0,3125 + 4 x 0,34375 = 7,0

Wzrost homozygotyczności – depresja
inbredowa
– wpływ na strukturę genetyczną populacji
Dziedziczenie – zupełna dominacja A
1
= 5 A
2
= 2
Genotyp Wartość Frekwencja genotypów
genotypowa P
0
P
1
[F=0,25] P
2
[F=0,375]
-------------------------------------------------------------------------------------------------
A
1
A
1
10
0,25
0,3125
0,34375
A
1
A
2
10
0,50
0,375
0,3125
A
2
A
2
4
0,24
0,3125
0,34375
P0
= 10 x 0,25 + 10 x 0,50 + 4 x 0,25 = 8,5
P1
= 10 x 0,3125 + 10 x 0,375 + 4 x 0,3125 = 8,125
P0
= 10 x 0,34375 + 10 x 0,3125 + 4 x 0,34375 = 7,9375

Krzyżowanie linii zinbredowanych efekt heterozji
Heterozja – wybujałość mieszańców - związana
heterozygotycznym stanem genotypu
wskaźnik heterozji :
x
F1
- x
FMP
VR = ----------------- x 100 %
x
FMP
x
FMP
- średnia wartość cechy u rodziców
x
F1
- średnia wartość cechy u potomstwa
Zdolność kombinacyjna (krzyżownicza) linii :
* ogólna (przyczyna - wariancja addytywna)
* specyficzna (przyczyna - wariancja nieaddytywna)
Wpływ heterozji na strukturę genetyczną populacji
Dziedziczenie w pełni addytywne
Genoty
py
Wartość
genotypo
wa
Frekw.
genot.
I
w
populacji
II
A
1
A
1
A
1
A
2
A
2
A
2
10
7
4
0,49
0,42
0,09
0,04
0,32
0,64
p = 0,7
p’ = 0,2
q = 0,3
q’ = 0,8
I
= 8,2
II
= 5,2
= wartość genotypowa x
frekwencja
I
= 10 x 0,49 + 7 x 0,42 + 4 x
0,09 = 8,2
II
=10 x 0,04 + 7 x 0,32 + 4 x
0,64 = 5,2
Po
= 1/2 (8,2 + 5,2) = 6,7

Wpływ heterozji na strukturę genetyczną populacji
Krzyżowanie osobników z populacji I i II
gamety popul. II
gamety
popul. I
A
1
p’=0,2
A
2
q’=0,8
__________________________________________________
A
1
p = 0,7
0,14
0,56
A
2
q = 0,3
0,06
0,24
---------------------------------------------------------------------------
A
1
A
1
0,14
A
1
A
2
0,62
F1
= 10 x 0,14 + 7 x 0,62 + 4 x 0,24 = 6,7
A
2
A
2
0,24
H
F1
=
F1
-
Po
= 0
Frekwencja genów w F
1
Frekwencja genotypów w F
2
A
1
= p
2
+ pq = 0,14 + 0,31= 0,45
A
1
A
1
= p
2
= 0,2025
A
2
= q
2
+ pq = 0,24 + 0,31= 0,55
A
1
A
2
= 2pq = 0,495
A
2
A
2
= q
2
= 0,3025
F2
= 6,7
H
F1
=
F2
-
Po
= 0

Wpływ heterozji na strukturę genetyczną populacji
Dziedziczenie – zupełna dominacja
p = 0,7 p’ = 0,2
q = 0,3 q’ = 0,8
I
= 9,46
II
= 6,16
Po
= 1/2 (9,46 + 6,16) = 7,81
Genoty
py
Wartość
genotypo
wa
Frekw.
genot.
I
w
populacji
II
A
1
A
1
A
1
A
2
A
2
A
2
10
10
4
0,49
0,42
0,09
0,04
0,32
0,64

Pokolenie F
1
powstałe ze skrzyżowania osobników z
populacji I i II
A
1
A
1
0,14
A
1
A
2
0,62
A
2
A
2
0,24
F1
= 10 x 0,14 + 10 x 0,62 + 4 x 0,24 = 8,56
H
F1
=
F1
-
Po
= 8,56 – 7,81 = 0,75
Pokolenie F
2
A
1
A
1
= 0,2025
A
1
A
2
= 0,4950
A
2
A
2
= 0,3025
F2
= 10 x 0,2025 + 10 x 0,4950 + 4 x 0,3025 =
8,185
H
F2
=
F2
-
Po
= 8,185 – 7,81 = 0,375
H
F2
= 1/2
H
F2
Wpływ heterozji na strukturę genetyczną populacji
Dziedziczenie – zupełna dominacja [cd]
Wpływ heterozji na strukturę genetyczną populacji
Dziedziczenie – niezupełna dominacja
Genoty
py
Wartość
genotypo
wa
Frekw.
genot.
I
w
populacji
II
A
1
A
1
A
1
A
2
A
2
A
2
10
9
4
0,49
0,42
0,09
0,04
0,32
0,64
p = 0,7 p’ = 0,2
q = 0,3 q’ = 0,8
I
= 9,06
II
= 5,48
Po
= 1/2 (9,04 + 5,48) = 7,44

Wykrywanie i ograniczanie wad wrodzonych
Wady warunkowane czynnikami
środowiskowymi:
eliminacja osobnika obarczonego wadą
zapewnienie dobrych warunków
środowiskowych,
przede wszystkim ciężarnym samicom
Wady warunkowane genetycznie:
eliminacja osobnika obarczonego wadą
wykrycie nosicielstwa i eliminacja
nosiciela

Genetyczne
uwarunkowanie chorób
dziedzicznych
• mutacje genomowe -
chromosomowe liczbowe (zmiana
liczby chromosomów)
– badania kariotypu
• aberacje chromosomowe (zmiana
struktury chromosomu)
• mutacje genowe (zmiana sekwencji
nukleotydowej genu)
Diagnostyka molekularna chorób genetycznych i infekcyjnych

Wykrywanie aberracji chromosomowych
Wykorzystanie metod cytogenetyki
- barwienie prążkowe chromosomów
- hybrydyzacja in situ
Barwienie prążkowe wskazuje na delecję
w ramieniu p chromosomu

molekularna diagnostyka chorób
genetycznych
PCR – polimerazowa reakcja łańcuchowa
metody z zastosowaniem enzymów
restrykcyjnych - RFLP - Restriction
Fragment Length Polymorphism
hybrydyzacja DNA-DNA
sekwencjonowanie DNA
Opracowanie testu diagnostycznego

Uzyskiwanie zwierząt odpornych na choroby
za pomocą metod genetycznego doskonalenia
selekcja zwierząt w kierunku odporności na
patogeny
(choroby infekcyjne) na podstawie markerów
fenotypowych
odporności/podatności - metody tradycyjne
wykrywanie nosicielstwa zmutowanych alleli i
brakowanie nosicieli
(choroby genetyczne) za pomocą badania
polimorfizmu DNA
- metody genetyki molekularnej
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
Wyszukiwarka
Podobne podstrony:
studenci genetyka w hodowli
Metody inzynierii genetycznej w hodowli zwierzat wyklady(calosc1)
genetyka molekularna i hodowla roślin, W14R03, Wykłady z genetyki i hodowli roślin ozdobnych, Sulech
Zastosowanie genetyki w hodowli, rolnictwie i medycynie
Praktyczne zastosowanie genetyki w hodowli ryb akwariowych cz III
zagadnienia , Ogrodnictwo, Ogrodnictwo UP Wro, ROK II, semestr III, genetyka i hodowla roślin
Kolokwia,egzaminy, Dzienni07, Wyniki z zaliczenia ćwiczeń i wykładów z Genetyki i hodowli roślin ogr
ZNACZENIE GENETYKI W HODOWLI ZWIERZĄT, Biologia, Referaty
Zastosowanie inżynierii genetycznej w hodowli roślin
test-a-egzmain-genetyka, Medycyna roślin sem.3, genetyka i hodowla roślin
studenci genetyczne uwarunkowanie odporno
studenci genetyczne uwarunkowanie odporności
Praktyczne zastosowanie genetyki w hodowli ryb akwariowych cz II
test-b-ezgamin-genetyka, Medycyna roślin sem.3, genetyka i hodowla roślin
Genetyka ogólna dla studentów cz 2
IG.7 - Detekcja zakażeń w hodowlach komórkowych techniką PCR, Genetyka, Inżynieria genetyczna
więcej podobnych podstron