
WYKŁAD VI
WYKŁAD VI
BUDOWA GENÓW
BUDOWA GENÓW

BUDOWA GENÓW
BUDOWA GENÓW
Gen zawiera instrukcję dotyczącą
Gen zawiera instrukcję dotyczącą
syntezy polipeptydu lub cząsteczki
syntezy polipeptydu lub cząsteczki
strukturalnego RNA.
strukturalnego RNA.
W sensie fizycznym jest odcinkiem
W sensie fizycznym jest odcinkiem
DNA o określonej sekwencji
DNA o określonej sekwencji
nukleotydów kodującym sekwencje
nukleotydów kodującym sekwencje
aminokwasów polipeptydu lub tylko
aminokwasów polipeptydu lub tylko
nukleotydów w RNA.
nukleotydów w RNA.

Wielkość genów jest różna i waha się
Wielkość genów jest różna i waha się
od 100pz do kilku milionów pz.
od 100pz do kilku milionów pz.
U bakterii
U bakterii
geny tworzą
geny tworzą
zespoły
zespoły
(
(
operony
operony
),
),
a u Euc.
a u Euc.
większość jest
większość jest
rozproszona lub
rozproszona lub
tworzą tzw.
tworzą tzw.
rodziny
rodziny
wielogenowe
wielogenowe
Rodziny wielogenowe
Rodziny wielogenowe
nie podlegają
nie podlegają
wspólnej regulacji,
wspólnej regulacji,
mogą być proste lub złożone.
mogą być proste lub złożone.

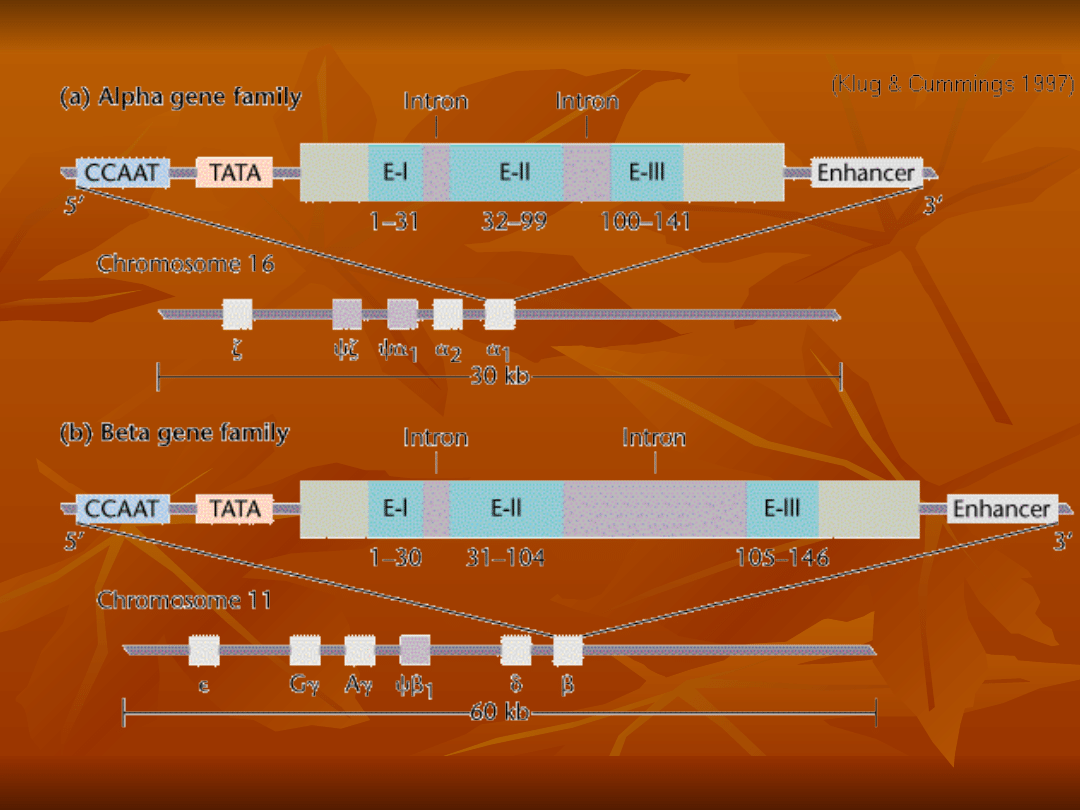
Rodziny wielogenowe u Euc.
Rodziny wielogenowe u Euc.
Proste zawierają geny identyczne
Proste zawierają geny identyczne
Wielogenowe
rodziny
złożone
Wielogenowe
rodziny
złożone
składają się z genów podobnych
składają się z genów podobnych
ale nie identycznych, jak np. geny
ale nie identycznych, jak np. geny
kodujące różne cząsteczki globin
kodujące różne cząsteczki globin
różniące się między sobą tylko
różniące się między sobą tylko
pojedynczym aminokwasem.
pojedynczym aminokwasem.

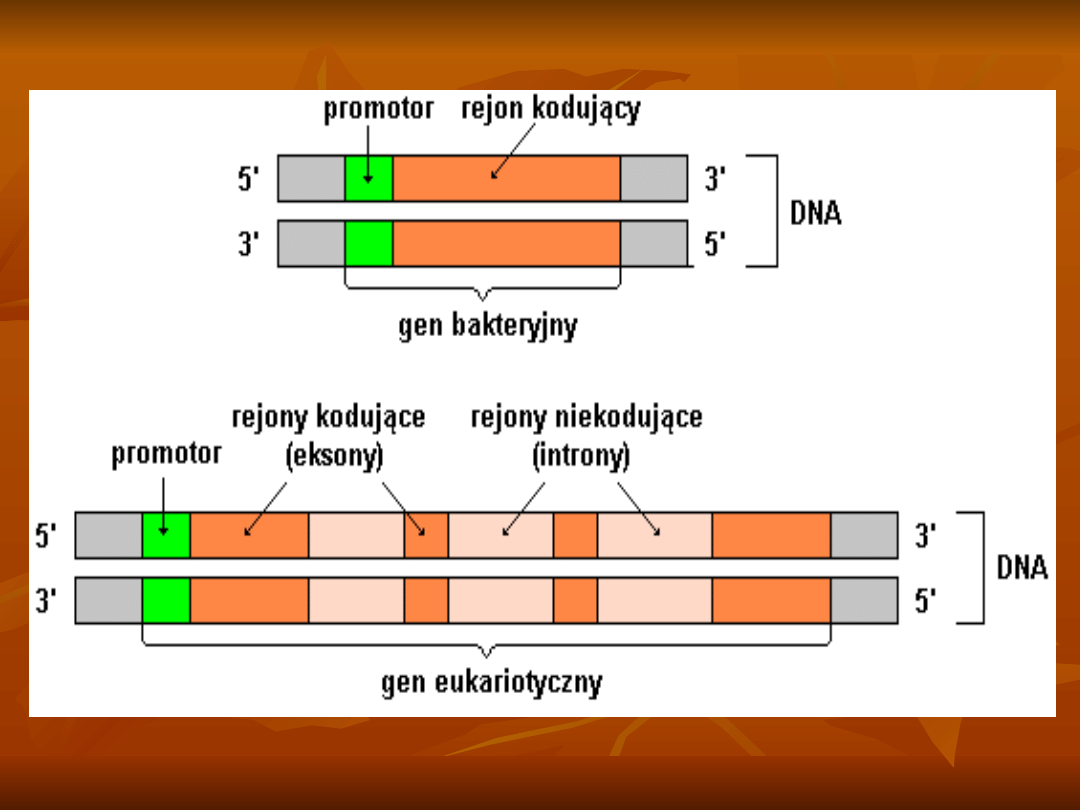
U E. informacja kodująca w
U E. informacja kodująca w
genach
genach
zapisana jest w segmentach sekwencji
zapisana jest w segmentach sekwencji
nukleotydów zwanych
nukleotydów zwanych
eksonami
eksonami
, a
, a
które są porozdzielane od siebie
które są porozdzielane od siebie
sekwencjami niekodującymi –
sekwencjami niekodującymi –
intronami.
intronami.
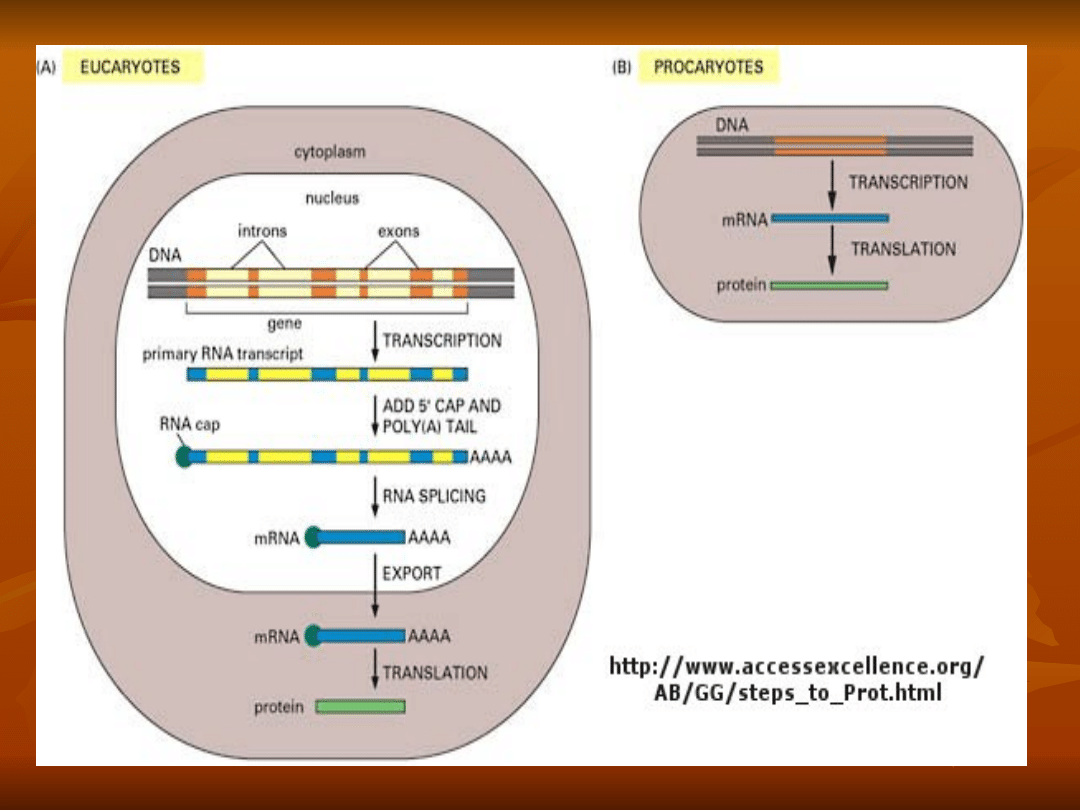
Ekson zawiera sekwencje, które po
Ekson zawiera sekwencje, które po
transkrypcji uczestniczą w translacji,
transkrypcji uczestniczą w translacji,
w intronach sekwencje nie uczestniczą
w intronach sekwencje nie uczestniczą
w translacji, są wycinane po
w translacji, są wycinane po
transkrypcji z pre-mRNA.
transkrypcji z pre-mRNA.
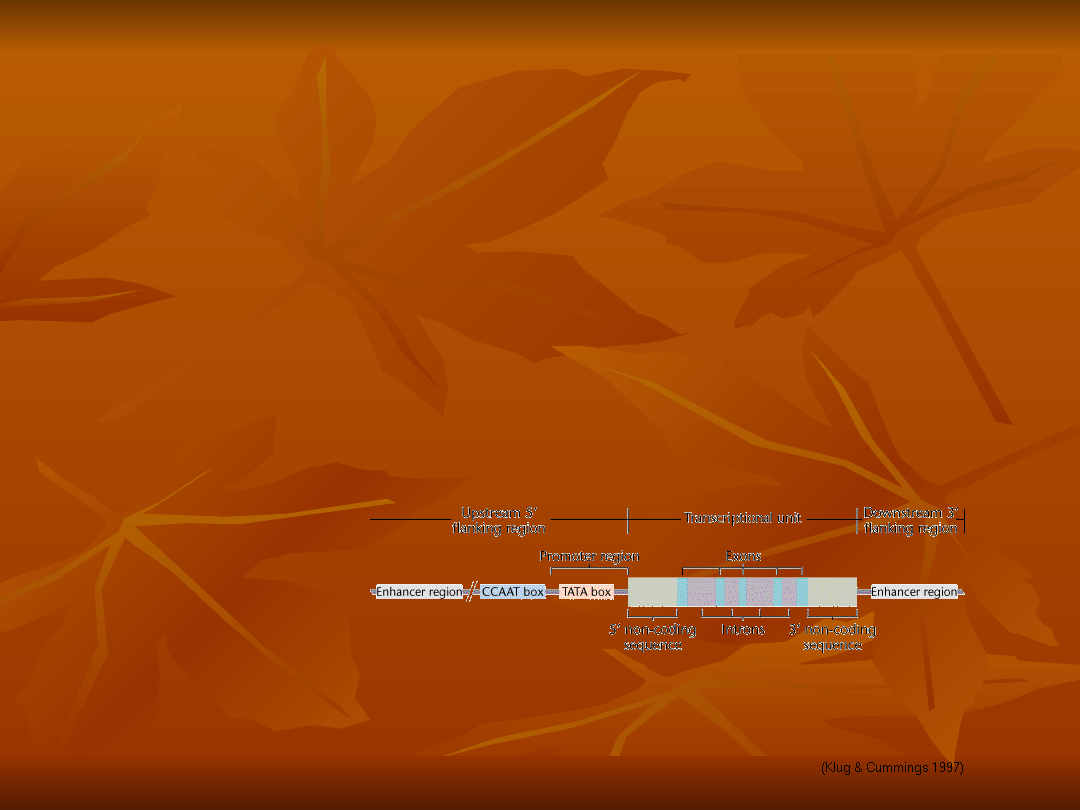
Geny u Eucaryota
Geny u Eucaryota
większość genów ma budowę
większość genów ma budowę
nieciągłą,
nieciągłą,
poza genemi histonów,
poza genemi histonów,
genami białek szoku cieplnego
genami białek szoku cieplnego
i niektórych interferonów.
i niektórych interferonów.

Geny z intronami i egzonami nazywa
Geny z intronami i egzonami nazywa
się mozaikowymi
się mozaikowymi
lub genami nieciągłymi.
lub genami nieciągłymi.
Obecnie przyjmuje się, że mozaikowa
Obecnie przyjmuje się, że mozaikowa
organizacja genów była pierwotna,
organizacja genów była pierwotna,
a ciągłe geny-bezintronowe są
a ciągłe geny-bezintronowe są
zjawiskiem wtórnym.
zjawiskiem wtórnym.
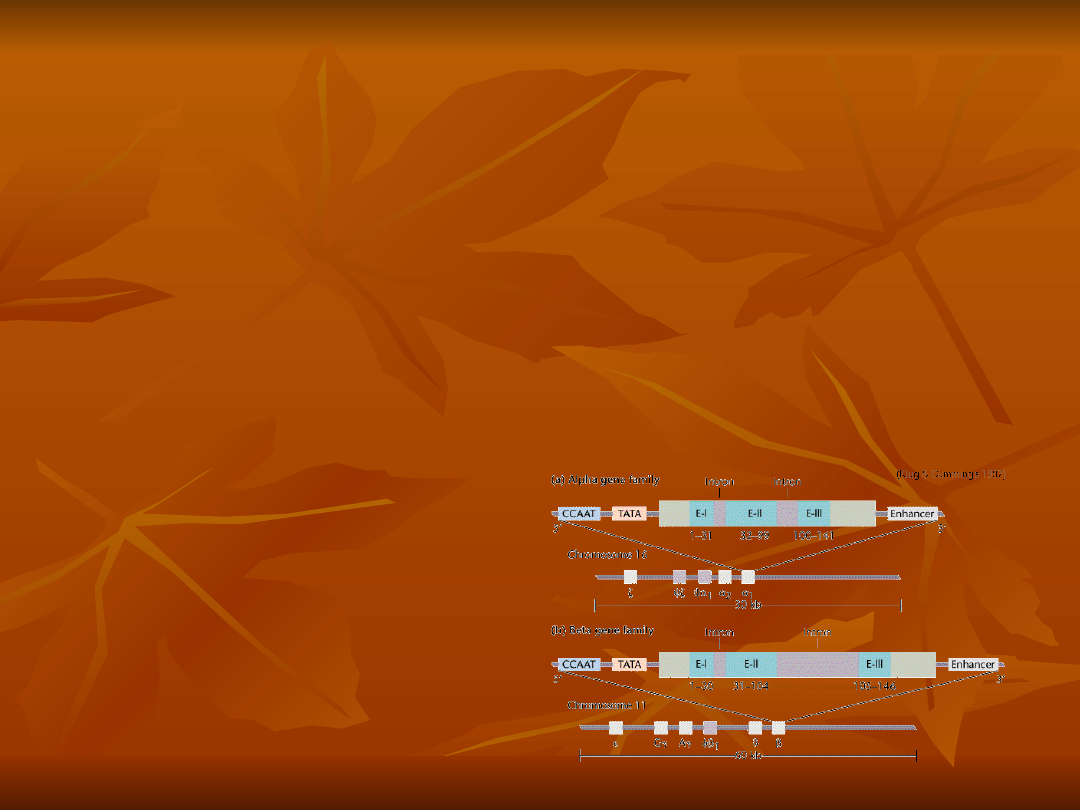
geny u E. i są to tzw. geny podzielone, u P.
– geny ciągłe

Poszczególne geny różnią się:
Poszczególne geny różnią się:
długością – liczbą pz
długością – liczbą pz
liczbą i długością intronów
liczbą i długością intronów
Liczba intronów w różnych genach
Liczba intronów w różnych genach
mieści się w granicach od
mieści się w granicach od
0
0
do
do
ponad 50.
ponad 50.

Najmniejszy gen u człowieka
Najmniejszy gen u człowieka
-
-
z 1 eksonu
z 1 eksonu
liczącego
liczącego
669pz
669pz
koduje białko
koduje białko
,
,
które kieruje
które kieruje
różnicowaniem gonady pierwotnej w
różnicowaniem gonady pierwotnej w
kierunku jąder
kierunku jąder
.
.
w genie kodującym u nas dystrofinę
w genie kodującym u nas dystrofinę
jest ich aż 78 i jest to nasz najdłuższy
jest ich aż 78 i jest to nasz najdłuższy
gen
gen
(
(2,5mln pz koduje 3 685
aminokwasów i stanowi 0,6% dł.
całego genu)
.
.
Geny RNA
Geny RNA
Geny, które kodują cząsteczki
Geny, które kodują cząsteczki
funkcjonalnego RNA (tRNA lub rRNA),
funkcjonalnego RNA (tRNA lub rRNA),
również mogą zawierać introny,
również mogą zawierać introny,
ale przerwy te występują zdecydowanie
ale przerwy te występują zdecydowanie
rzadziej niż w genach kodujących mRNA.
rzadziej niż w genach kodujących mRNA.

W wielu genach ilość DNA w
W wielu genach ilość DNA w
intronach przewyższa ilość DNA
intronach przewyższa ilość DNA
eksonów,
eksonów,
może go być dziesięć razy więcej,
może go być dziesięć razy więcej,
a w niektórych przypadkach nawet
a w niektórych przypadkach nawet
ponad sto razy więcej.
ponad sto razy więcej.
Specyficzna budowa ludzkich
Specyficzna budowa ludzkich
genów sprawia, że dzięki
genów sprawia, że dzięki
wykorzystaniu instrukcji
wykorzystaniu instrukcji
zapisanych w poszczególnych
zapisanych w poszczególnych
eksonach w różny sposób te same
eksonach w różny sposób te same
geny mogą kierować produkcją
geny mogą kierować produkcją
różnych białek.
różnych białek.

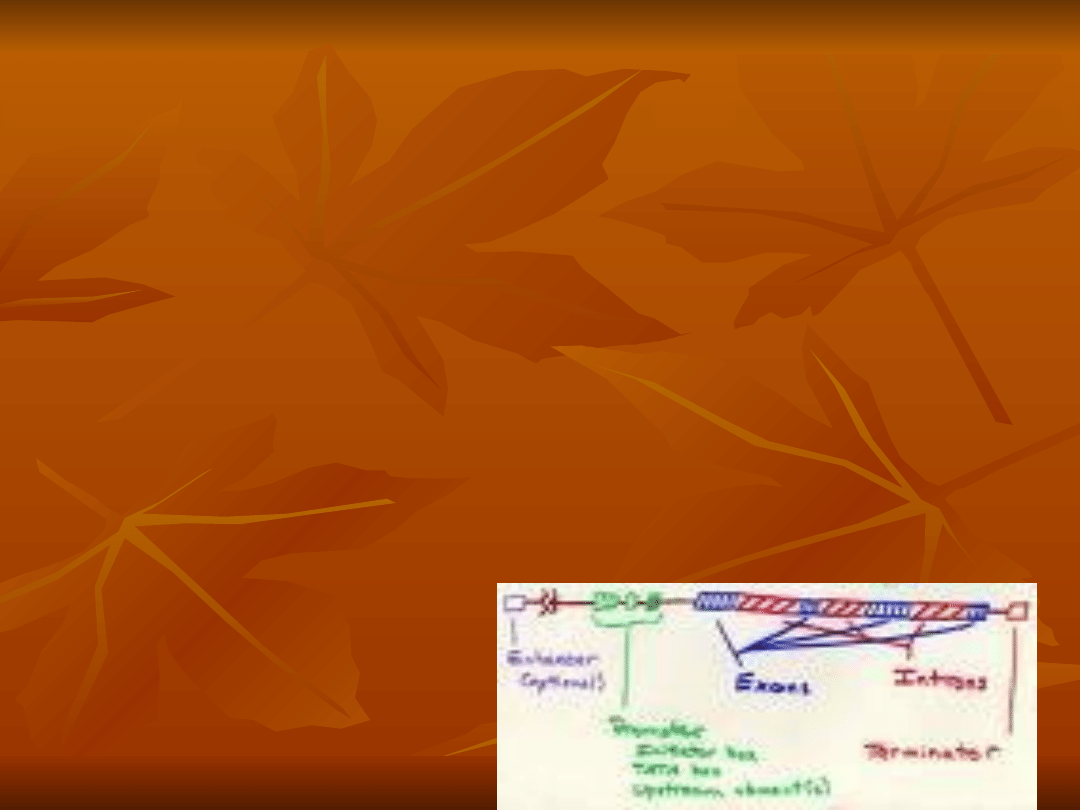
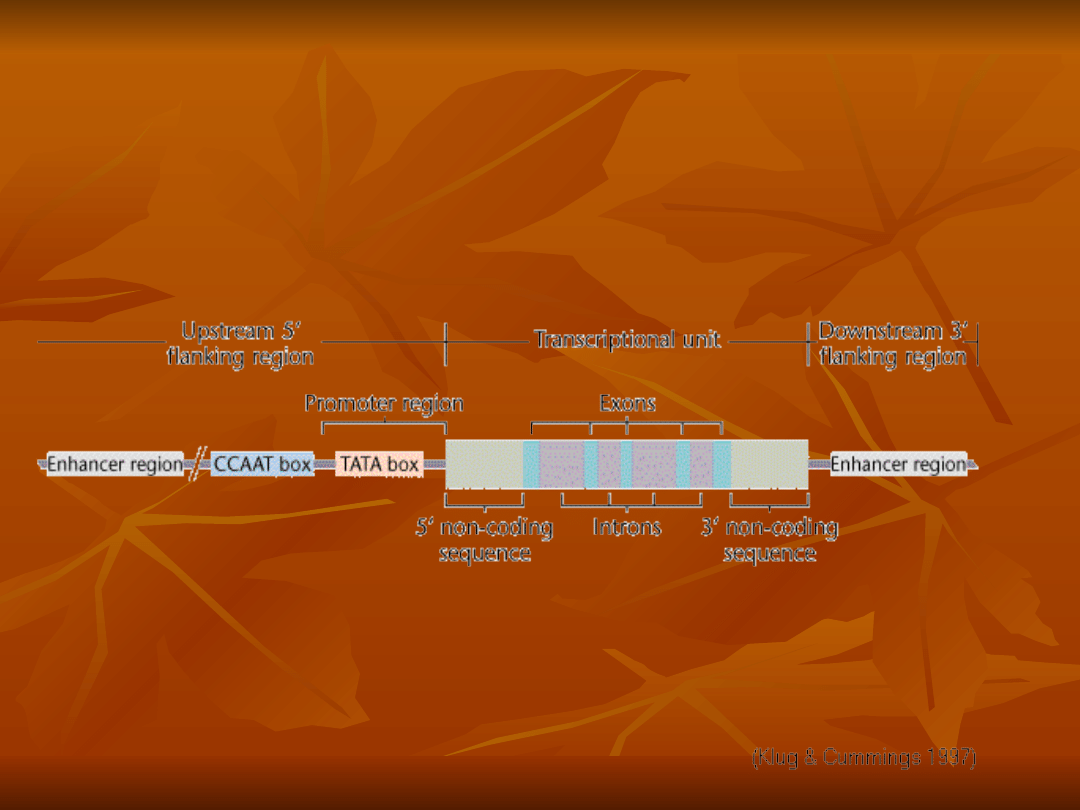
W skład genu wchodzą
W skład genu wchodzą
sekwencje początkowe i
sekwencje początkowe i
końcowe genu,
końcowe genu,
które ulegają transkrypcji, ale nie
które ulegają transkrypcji, ale nie
podlegają
translacji,
w
tym
podlegają
translacji,
w
tym
sekwencje promotorowe,
sekwencje promotorowe,
Czyli
sekwencje
związane
z
Czyli
sekwencje
związane
z
procesem inicjacji transkrypcji genu.
procesem inicjacji transkrypcji genu.
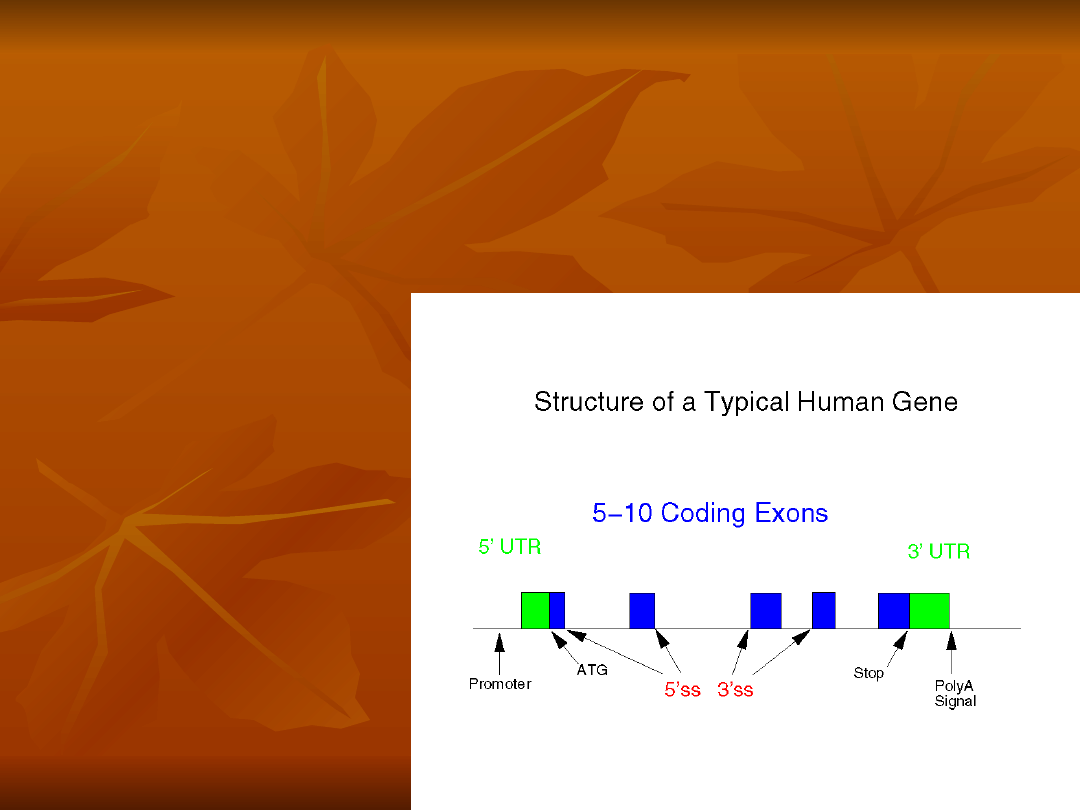
Na końcu 5’ i 3’ znajdują się
Na końcu 5’ i 3’ znajdują się
sekwencje
sekwencje
związane z rozpoczęciem obróbki
związane z rozpoczęciem obróbki
potranskrypcyjnej pre-mRNA
potranskrypcyjnej pre-mRNA
Nazywa się je sekwencjami
Nazywa się je sekwencjami
UTR
UTR
(Untranslations)
(Untranslations)
Ekspresja
genu
jest
ściśle
Ekspresja
genu
jest
ściśle
regulowana
przez
sekwencje
regulowana
przez
sekwencje
położone
powyżej
sekwencji
położone
powyżej
sekwencji
kodującej i jest to:
kodującej i jest to:
miejsce wiązania polimerazy RNA,
miejsce wiązania polimerazy RNA,
miejsce
wiązania
miejsce
wiązania
niezbędnych
niezbędnych
czynników transkrypcyjnych
czynników transkrypcyjnych
oraz miejsce PROMOTORA czyli
oraz miejsce PROMOTORA czyli
inicjacji syntezy cząsteczki RNA
inicjacji syntezy cząsteczki RNA
.
.

Sekwencje promotorowe
Sekwencje promotorowe
Sekwencje te mogą być b. liczne w
Sekwencje te mogą być b. liczne w
niektórych genach,
niektórych genach,
dzieli
się
je
na
dzieli
się
je
na
sekwencje
sekwencje
promotora
podstawowego
promotora
podstawowego
–
–
położone
w
miejscu
składania
położone
w
miejscu
składania
kompleksu inicjacyjnego
kompleksu inicjacyjnego
oraz
oraz
na
na
sekwencje
położone
sekwencje
położone
powyżej promotora podstawowego.
powyżej promotora podstawowego.

Każda z trzech eukariotycznych
Każda z trzech eukariotycznych
polimeraz RNA zależnych od DNA
polimeraz RNA zależnych od DNA
rozpoznaje różne sekwencje
rozpoznaje różne sekwencje
promotorowe
promotorowe
i to właśnie różnice pomiędzy
i to właśnie różnice pomiędzy
promotorami decydują o tym, która
promotorami decydują o tym, która
polimeraza RNA zależna od DNA
polimeraza RNA zależna od DNA
przeprowadza transkrypcję których
przeprowadza transkrypcję których
genów.
genów.
Każda polimeraza RNA wykorzystuje
Każda polimeraza RNA wykorzystuje
promotor innego typu.
promotor innego typu.

U kręgowców wyróżnia się trzy
U kręgowców wyróżnia się trzy
różniące się strukturą typy
różniące się strukturą typy
promotorów:
promotorów:
1.Promotory rozpoznawane przez
1.Promotory rozpoznawane przez
polimerazę RNA I
polimerazę RNA I
składają się z
składają się z
promotora
promotora
podstawowego
podstawowego
,
,
który obejmuje miejsce startu transkrypcji
który obejmuje miejsce startu transkrypcji
położonego między nukleotydami –45 a +20 -
położonego między nukleotydami –45 a +20 -
sekwencje te warunkują zajście transkrypcji
sekwencje te warunkują zajście transkrypcji
oraz
oraz
elementu kontrolnego
elementu kontrolnego
UCE (
UCE (
U
U
pstream
pstream
C
C
ontrol
ontrol
E
E
lement
lement
) znajdującego się +100 pz
) znajdującego się +100 pz
powyżej miejsca startu (czyli przed miejscem
powyżej miejsca startu (czyli przed miejscem
startu)
startu)
Polim RNA I (RNA Pol I) transkrybuje
Polim RNA I (RNA Pol I) transkrybuje
większość genów rRNA, znajduje się w
większość genów rRNA, znajduje się w
jąderku.
jąderku.

2. Promotory rozpoznawane przez
2. Promotory rozpoznawane przez
polimerazę RNA II
polimerazę RNA II
są b. różne i znajdują się w odległości do kilku
są b. różne i znajdują się w odległości do kilku
tys. pz powyżej miejsca startu transkrypcji.
tys. pz powyżej miejsca startu transkrypcji.
Promotor podstawowy składa się z dwóch
Promotor podstawowy składa się z dwóch
segmentów:
segmentów:
z bloku –25 zwanego sekwencją TATA lub
z bloku –25 zwanego sekwencją TATA lub
kasetą TATA i z sekwencji inicjatorowej – Inr
kasetą TATA i z sekwencji inicjatorowej – Inr
Do pełnej aktywności promotora niezbędne są
Do pełnej aktywności promotora niezbędne są
inne sekwencje występujące w rejonie od -40
inne sekwencje występujące w rejonie od -40
do -110.
do -110.
W 56% genów są sekwencje bogate w pary CG
W 56% genów są sekwencje bogate w pary CG
powtarzane
wielokrotnie
i
są
nazywane
powtarzane
wielokrotnie
i
są
nazywane
wysepkami CpG, s
wysepkami CpG, s
ą
ą
ważnym elementem w
ważnym elementem w
inicjacji transkrypcji.
inicjacji transkrypcji.

Sekwencje CCAAAT w odległości 70-90pz
Sekwencje CCAAAT w odległości 70-90pz
od miejsca inicjacji transkrypcji położone
od miejsca inicjacji transkrypcji położone
przed blokiem TATA są również ważne w
przed blokiem TATA są również ważne w
trans
trans
krypcji,
krypcji,
są miejscem wiązania
są miejscem wiązania
innych czynników transkrypcyjnych.
innych czynników transkrypcyjnych.
Polim RNA II (RNA Pol II)
Polim RNA II (RNA Pol II)
transkrybuje wszystkie geny
transkrybuje wszystkie geny
kodujące białka i niektóre geny
kodujące białka i niektóre geny
małych jądrowych RNA (snRNA-
małych jądrowych RNA (snRNA-
small nuclear RNA), znajduje się w
small nuclear RNA), znajduje się w
nukleoplaźmie
nukleoplaźmie

Obszar promotorowy genu znajduje się
na jego 5' końcu i zawiera kilka
istotnych rejonów rozpoznawanych przez
polimerazę RNA oraz czynniki
transkrypcyjne
najbardziej powszechnym jest kaseta
TATA ( tzw. TATA-box ).
Jest to 7-nukleotydowa sekwencja
położona w odległości ok. 25 p.z. od
miejsca startu transkrypcji, która w
pełni prezentuje się następująco: 5'-
TATAAAA -3'.

Obecność kasety TATA, choć niezbędna
w przypadku prawie wszystkich genów;
nie jest wystarczająca, aby z promotora
ruszyła transkrypcja. Kaseta TATA
stanowi tzw. część rdzeniową promotora.
W 56% genów są sekwencje bogate w
W 56% genów są sekwencje bogate w
pary CG powtarzane wielokrotnie i są
pary CG powtarzane wielokrotnie i są
nazywane wysepkami CpG, s
nazywane wysepkami CpG, s
ą
ą
ważnym
ważnym
elementem w inicjacji transkrypcji.
elementem w inicjacji transkrypcji.
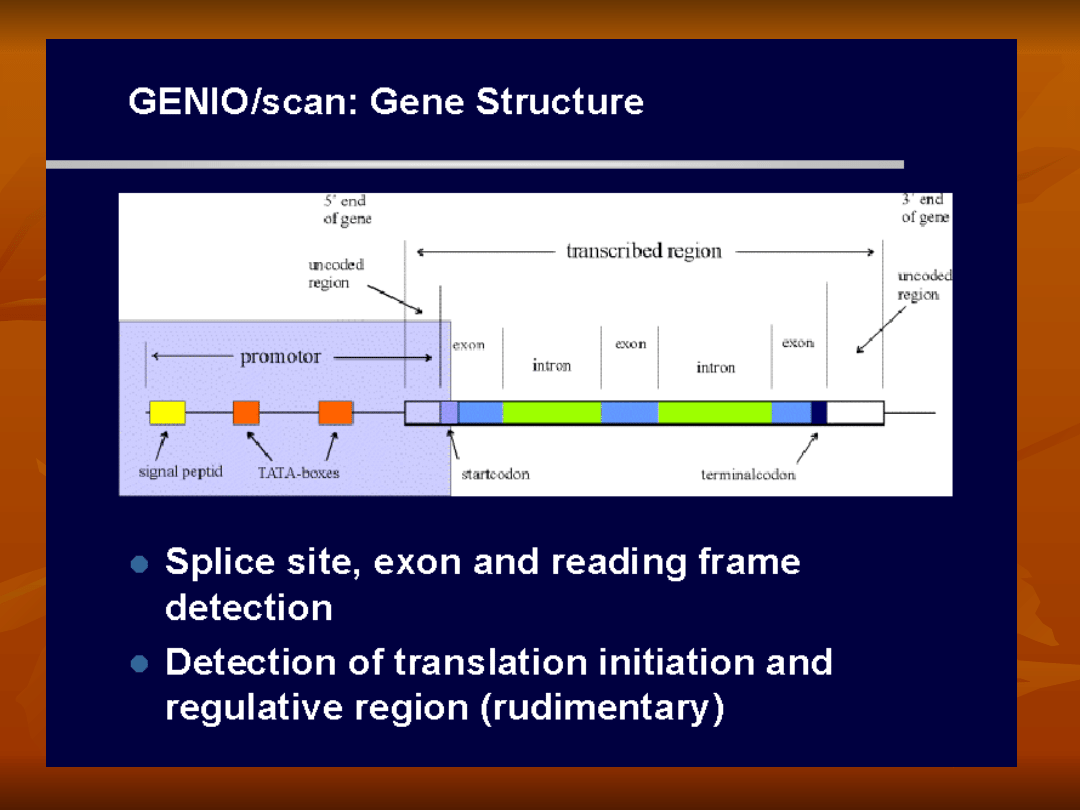

Rejony rozpoznawane przez
Rejony rozpoznawane przez
polimerazę II RNA
polimerazę II RNA

3. Promotory dla
3. Promotory dla
polimerazy RNA
polimerazy RNA
III
III
są nietypowe, gdyż znajdują się
są nietypowe, gdyż znajdują się
wewnątrz genów i znacznie się
wewnątrz genów i znacznie się
różnią
różnią
.
.
I
I
ch element podstawowy to
ch element podstawowy to
sekwencja licząca od 50 do 100pz
sekwencja licząca od 50 do 100pz
podzielone na dwa bloki.
podzielone na dwa bloki.
Polim III (RNA Pol III) transkrybuje
Polim III (RNA Pol III) transkrybuje
geny tRNA, 5S rRNA, kilka snRNA,
geny tRNA, 5S rRNA, kilka snRNA,
znajduje się w nukleoplaźmie
znajduje się w nukleoplaźmie

Wiele
Wiele
genów zawiera
genów zawiera
dodatkowe sekwencje
dodatkowe sekwencje
Są one
Są one
położone w różnych odległościach od
położone w różnych odległościach od
genów, ale mimo nawet znacznej odległości
genów, ale mimo nawet znacznej odległości
gen pozostaje zawsze pod ich kontrolą i są to
gen pozostaje zawsze pod ich kontrolą i są to
:
:
sekwencje
wzmacniające
sekwencje
wzmacniające
ehnacerowe,
ehnacerowe,
które znacznie stymulują transkrypcję i
które znacznie stymulują transkrypcję i
znajdują się poza miejscem promotorowym,
znajdują się poza miejscem promotorowym,
wyciszające
wyciszające
silencerowe,
silencerowe,
które hamują
które hamują
transkrypcję
transkrypcję
.
.
Sekwencje terminacji –
Sekwencje terminacji –
takie znaczenie
takie znaczenie
mają sewkencje palindromowe
mają sewkencje palindromowe

Enhancery
Enhancery
enhancery tkankowo-specyficzne
enhancery tkankowo-specyficzne
wzmagające transkrypcję tylko tam, gdzie
wzmagające transkrypcję tylko tam, gdzie
obecne są białka wiążące się w ich obrębie
obecne są białka wiążące się w ich obrębie
tylko w niektórych komórkach wykazują
tylko w niektórych komórkach wykazują
aktywność wzmacniającą
aktywność wzmacniającą
Możliwość oddziaływań enhancer :
Możliwość oddziaływań enhancer :
promotor mimo odległości pomiędzy nimi
promotor mimo odległości pomiędzy nimi
wynoszącej niekiedy kilka tys. p.z. istnieje
wynoszącej niekiedy kilka tys. p.z. istnieje
dzięki dużej elastyczności nici DNA. Owa
dzięki dużej elastyczności nici DNA. Owa
elastyczność objawia się zdolnością DNA do
elastyczność objawia się zdolnością DNA do
dowolnego wyginania się.
dowolnego wyginania się.
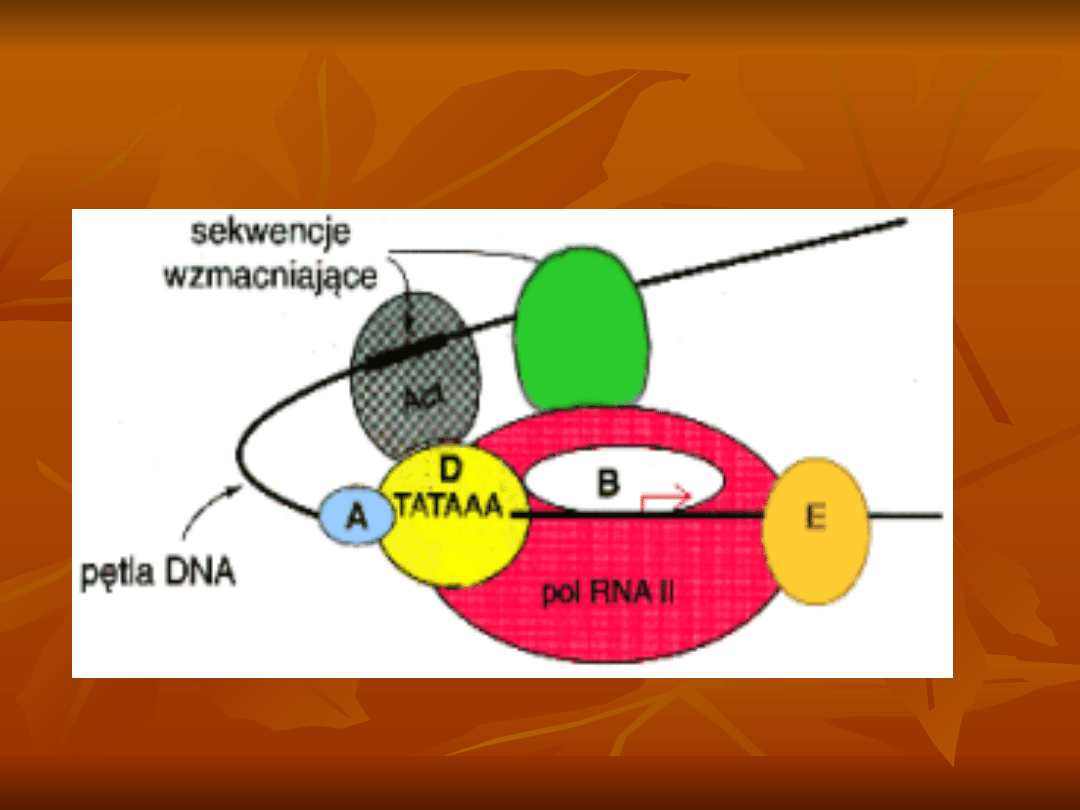
Interakcje enhancer : promotor
Interakcje enhancer : promotor

Silencery - (ang. silence -
Silencery - (ang. silence -
cisza ),
cisza ),
sekwencje służące wyciszeniu aktywności
sekwencje służące wyciszeniu aktywności
promotora; podobnie jak enhancery mogą być w
promotora; podobnie jak enhancery mogą być w
różnym stopniu oddalone od genu w obu
różnym stopniu oddalone od genu w obu
kierunkach, a także występować w jego wnętrzu.
kierunkach, a także występować w jego wnętrzu.
Np. w genach kodujących immunoglobuliny
Np. w genach kodujących immunoglobuliny
(białka syntetyzowane jedynie w limfocytach B)
(białka syntetyzowane jedynie w limfocytach B)
silencer wbudowany jest w obrębie enhancera,
silencer wbudowany jest w obrębie enhancera,
jego znaczenie uwidocznia się w komórkach
jego znaczenie uwidocznia się w komórkach
innych niż limfocyty B. Jest to swoiste
innych niż limfocyty B. Jest to swoiste
zabezpieczenie przed ekspresją genów
zabezpieczenie przed ekspresją genów
immunoglobulin związane z inaktywacją
immunoglobulin związane z inaktywacją
enhancera.
enhancera.
BUDOWA GENÓW
BUDOWA GENÓW

Struktura genu
Struktura genu
prokariotycznego
prokariotycznego
Promotor (przed miejscem inicjacji
Promotor (przed miejscem inicjacji
transkrypcji) zawiera dwie ramki:
transkrypcji) zawiera dwie ramki:
+10 (AT)
+10 (AT)
+35 (TTGACA)
+35 (TTGACA)
Obie ramki są miejscami wiązania
Obie ramki są miejscami wiązania
polimerazy RNA zależnej od DNA.
polimerazy RNA zależnej od DNA.
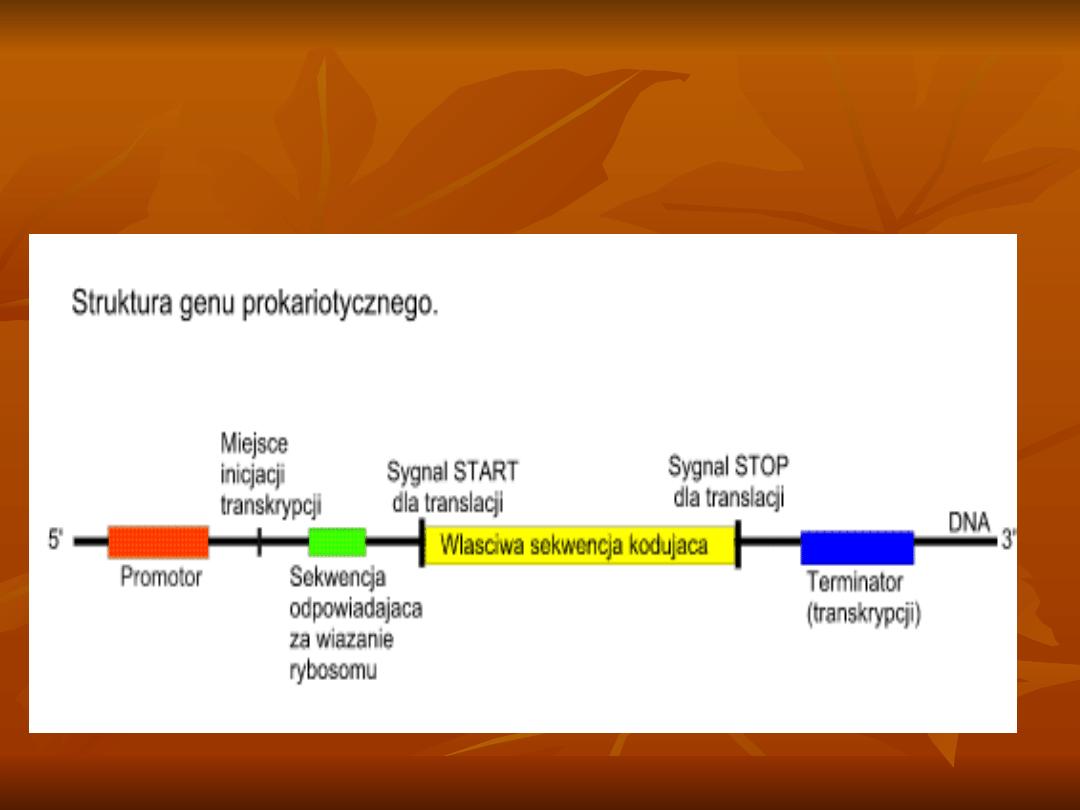
Struktura genu
Struktura genu
prokariotycznego
prokariotycznego


Cistron
jednostka spełniająca funkcję
biochemiczną,
której ekspresja prowadzi do
powstania pojedynczego łańcucha
polipeptydowego
składa się z kilku tysięcy par
nukleotydów, może zostać
podzielony na mniejsze części.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
Wyszukiwarka
Podobne podstrony:
6 Wykład VIII Budowa genów
6 Wykład VI Budowa genów
6 Wykład VI Budowa genów
8 Wykład VIII Ekspresja genów II
Prawo medyczne wykład VIII Obowiązek ratowania życia
Turystyka, wykład VIII, Agroturystyka
Wykład VIII Synteza układów sekwencyjnych
Wykład VIII 03 04 2012
pdf wykład 02 budowa materii, podstawowe prawa chemiczne 2014
Wykład VIII, Studia Biologia, Mikrobiologia, wykłady z ogólnej
prawo administracyjne-wyklad VIII, prawo administracyjne(1)
Wykład VIII Leki stosowane w?rmatologii
WSTĘP DO HISTORII KULTURY STAROPOLSKIEJ, WYKŁAD VIII,$ 11 10
WYKŁAD VIII
WYKŁAD VIII i IX
Wykład VIII( 11 2012
biol.medyczna, wykład - Ogólna budowa komórki, Ogólna budowa komórki
Wyklad VIII - zadania, Wykład III
Wykład VIII& 11 2013
więcej podobnych podstron