
Najpierw Eucaryota
Pochodzenie genów kodujących peptydy
1. Fragmenty kodujące peptydy, są to „introny”
genów kodujących w świecie RNA małe
funkcjonalne cząsteczki RNA typu tRNA.
2. Replikacja i transkrypcja RNA początkowo
zachodziły równolegle i na 1 matrycy
3. tRNA jako primery transkrypcji i promotory
replikacji
4. Telomeraza jako relikt pre-replikazy RNA na
RNA
5. pre-rybosom – transkrypcja „rybogenów, pre-
telomeraza – replikacja RNA

Najpierw Eucaryota
Pierwotne geny kodujące peptydy
1. Białka o funkcjach uniwersalnych i mało specyficznych,
niekatalityczne
2. Białka o stosunkowo małej złożoności strukturalnej,
wystarczającej do pełnienia funkcji metabolicznych
3. Białka, których introny genów kodują funkcjonalne RNA
•
RBP
–
białka (niektóre) rybosomalne (rekrutacja białek poźniejszych)
–
Sm proteins (białka spliceosomów)
–
RNA-aza P
•
HSP
–
chaperoniny (to też białka HSP)
Białka te stopniowo przejmowały niektóre funkcje
rybozymów, eliminowały konieczność wysokich stężeń
jonów (np. Mg), a każde zwiekszenie specyficzności i
szybkości replikacji/transkrypcji RNA natychmiast było
podchwytywane przez dobór naturalny z powodu
nieliniowości hipercykli.
W końcu pojawiła się możliwość redukcji RN. Istniejące
telomerazy i odwrotne transkryptazy wyprodukowały z
nich stabilne DNA, które mogło potencjalnie „odtwarzać”
takie cząsteczki białek i RNA w dowolnym czasie.

Najpierw Eucaryota
Pierwotne geny kodujące peptydy
Widać, że w pierwotnych genach
„białkowych” sens (znaczenie) pojawia
się co najmniej równolegle, o ile nie
wyprzedza kodowania konkretnego
denotatu – konkretnego białka. Nie tylko
działały sekwencje zbliżone w obrębie
kwazigatunku, ale również pierwotne
kodowanie nie musiało być idealnie
jednoznaczne, wobec pełnienia wielu
nieostro określonych funkcji przez białka.
Najpierw Eucaryota
Pierwotne geny kodujące peptydy
• Powstanie DNA to zatem tylko przepisanie
informacji zawartej w pierwotnym RNA na
trwalszy nośnik, umożliwjający odczytanie
(wielokrotne) w późniejszym czasie.
• Wynikałoby z tego:
replikacja RNA na RNA-> odwrotna
transkrypcja (RNA->DNA)->transkrypcja
(DNA->RNA)->replikacja (DNA-> DNA)
białka
DNARNA
• Powstanie DNA to zatem tylko przepisanie
informacji zawartej w pierwotnym RNA na
trwalszy nośnik, umożliwiający odczytanie
(wielokrotne) w późniejszym czasie.
• Wynikałoby z tego:
replikacja RNA na RNA-> odwrotna
transkrypcja (RNA->DNA)->transkrypcja
(DNA->RNA)->replikacja (DNA-> DNA)
białka
DNARNA
• Powstanie DNA to zatem tylko przepisanie
informacji zawartej w pierwotnym RNA na
trwalszy nośnik, umożliwiający odczytanie
(wielokrotne) w późniejszym czasie.
• Wynikałoby z tego:
replikacja RNA na RNA-> odwrotna
transkrypcja (RNA->DNA)->transkrypcja
(DNA->RNA)->replikacja (DNA-> DNA)
białka
DNARNA
• Powstanie DNA to zatem tylko przepisanie
informacji zawartej w pierwotnym RNA na
trwalszy nośnik, umożliwiający odczytanie
(wielokrotne) w późniejszym czasie.
• Wynikałoby z tego:
replikacja RNA na RNA-> odwrotna
transkrypcja (RNA->DNA)->transkrypcja
(DNA->RNA)->replikacja (DNA-> DNA)
białka
DNARNA

Fragmenty z Ricoeura
„Tekst” = tekst pisany
• „Tekst jest niemy.”
• „Tekst jest podobny do zapisu muzycznego, a
czytelnik - do dyrygenta orkiestry, który postępuje
zgodnie z instrukcjami partytury.”
• „(…) rozumieć, to (…) wytworzyć nowe zdarzenie
wychodząc od tekstu, w którym zdarzenie
pierwotne zostało zobiektywizowane.”
• „Tekst jako całość (…) może być oglądany z
rozmaitych stron, lecz nigdy ze wszystkich stron
jednocześnie. (…) Zawsze jest możliwe odniesienie
tego samego zdania w rozmaity sposób do tych
lub innych zdań uznanych za kluczowe w tekście.”
Wiele z tych myśli można odnieść do sposobu
funkcjonowania DNA („tekstu pisanego”) i RNA
(jako odpowiednika „mowy”).

Uczciwość wymaga…
• Nie da się jednak (niestety ) ukryć, że
wszelkie spekulacje co do mechanizmu
powstania pierwszego faktycznego,
konkretnego zapisu struktury
pierwszorzędowej białka w postaci ciągu
dezoksyrybonukleotydów, mają bardzo nikłe
podstawy eksperymentalne. My tu wciąż
mówimy o powstaniu pierwszych białek w
ogóle, pierwszych genów białkowych w
ogóle…
• Ale takie są właśnie bezdroża teorii ewolucji

Najpierw Eucaryota
Fakty wskazujące na pierwotny charakter
genomu eukariotycznego
1.Liczniejsze relikty „świata RNA”. Trudno sobie
wyobrazić, by ewolucja „wsteczna” dotycząca
wielu ważnych procesów metabolicznych, była
możliwa
2.Obróbka mRNA i rRNA u Procaryota szybsza,
wydajniejsza i (bo) bardziej „białkowa”
3.=> Brak wytłumaczenia innej genezy
spliceosomu
4.=> Brak wytłumaczenia innej genezy
telomerazy
5.=> haploidalność jako cecha wyspecjalizowana,
konsekwencja podniesienia wydajności
replikacji
(pierwotne genomy były nawet do 200n)

Fakty wskazujące na pierwotny charakter genomu eukariotycznego

Najpierw Eucaryota
Pochodzenie genomu Prokaryota
1.Powstanie kolistego dsDNA
plazmidu
2.Mechanizm przeniesienia
informacji
z genomu linearnego na
kolisty
3.Przyczyny preferencji genomu
kolistego nad linearnym (i dalsza
ewolucja – informacji)
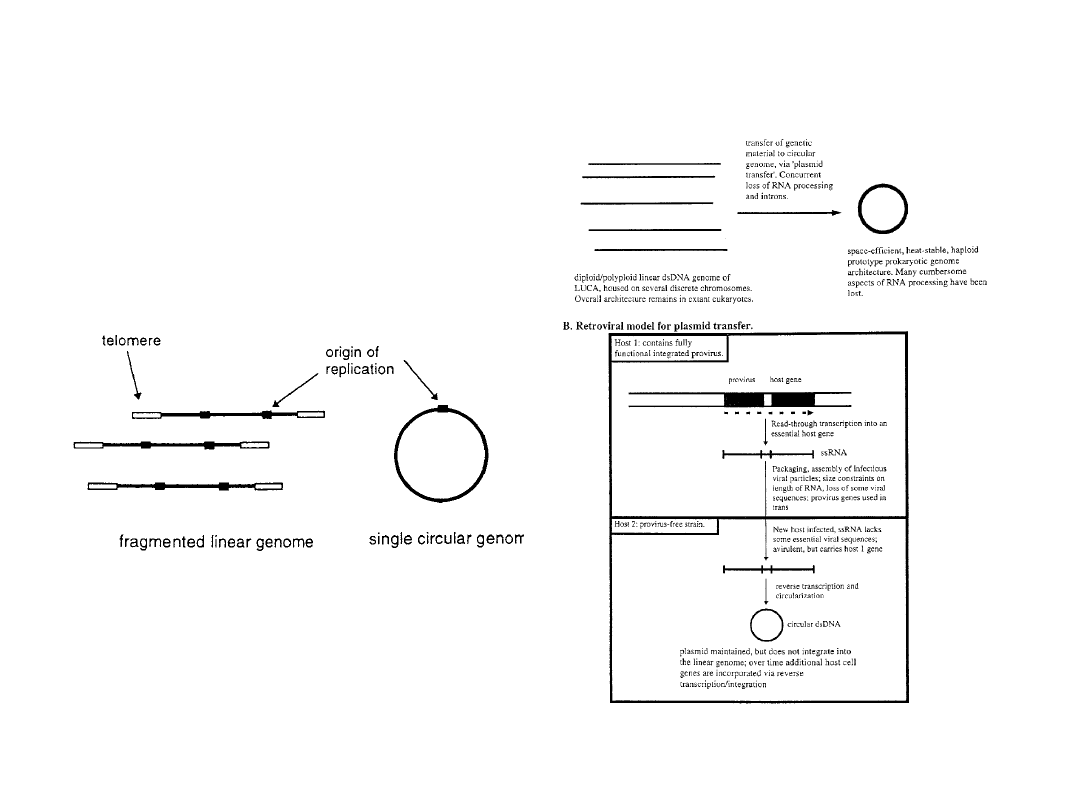
Pochodzenie genomu Prokaryota

Pochodzenie genomu Prokaryota
1.Odwrotna transkryptaza – forma telomerazy
(mowa o funkcjach, a nie o współczesnych
białkach) relikt ze świata RNA
2.Retrowirusy – model takiego procesu (genomy
kodują RT), mogły powstać już wtedy (choć
prawdopodobne, że powstały bardzo
niedawno – u kręgowców)
3.Dalszy przebieg ewolucji – konsekwencja tego
faktu, powtórzenia i silna presja selekcyjna w
środowiskach o wysokiej temperaturze:
powstanie operonów, utrata wielu funkcji RNA,
specjalizacja genomu i metabolizmu

1. Brak termofilnych eukariontów
2. ssRNA termoniestabilne -> specjalizacja, skrócenie i redukcja
procesów obróbki rRNA, przejęcie części z nich przez
wyspecjalizowane białka
3. silniejsza stabilizacja RNA przez białka (RNA musi istnieć nawet u
org. termofilnych) => wymusza ewolucję
4. Przyspieszenie procesów replikacji, transkrypcji i translacji,
zmniejszenie rybosomów (!)
5. Brak splicingu, redukcja funkcji komponenty RNA snRNA i snoRNA
na rzecz białek, obecność sekwencji kodującej 3’ terminalne CCA w
genach tRNA (np.. nukleaza Z z
Thermotoga maritima)
6. Termoniestabilność Gln i Asn (i innych amidów); nietypowa
produkcja tRNA
Gln
poprzez przyłączanie Glu i transaminację tRNA
Glu
(z wyjątkiem bakterii G-)
7. dcDNA jest bardziej termostabilne niż dsDNA.
8. Odwrotna giraza (kombinacja helikazy i topoizomerazy I) zwiększa
jeszcze termostabilność dcDNA poprzez dodatkowe
skomplikowanie struktury trzeciorzędowej (niezbędna do replikacji
dla termofilów).
9. optimum termiczne dla RNA-azy P u E. coli około 60
○
C
,
u termofilów
około 70
○
C…
…potem Prokaryota
hipoteza termoredukcji

hipoteza termoredukcji
ewolucja funkcji snoRNA

…potem
Prokaryota
strategia r
dN/dt=rN(1-N/K)
– r- maksymalna wewnetrzna predkość wzrostu
populacji, K – „bezpieczna gęstość” populacji
selekcja typu r – charakterystyczna dla bardzo
niestabilnych środowisk, gdzie przeżycie gatunku nie
zależy bardzo od przystosowania, lecz od płodności i
tempa namnażania:
małe, szybko rozmnażające się, szybko ewoluujące
(zmienne) organizmy.
szybki wzrost
szybka replikacja w trudnych warunkach
konieczność zachowania wierności replikacji
zmniejszenie genomu i uproszczenie budowy
rozproszenie puli genowej pomiędzy większą liczbę
osobników – transfer horyzontalny informacji
zwiększenie tempa mutacji
rozproszenie genomu pomiędzy różne cząsteczki DNA

Wartość teorii: weryfikacja
eksperymentalna
1. zbadanie RNAazy MRP u pierwotniaków (Microsporidia,
Giardia, Entamoeba)– obecność, wielkość i zawartość RNA
2. Telomeraza u roślin
3. Funkcje RNA w miejscu katalitycznym telomerazy
4. poszukiwania genów kodujących małe cząsteczki RNA w
intronach i badanie ich ewolucji (intron-first?)
5. filogenetyczna dystrybucja ARS dla Glutaminy (czy tylko u
Eukaryota)?
6. Badanie czynników selekcyjnych powodujących transfer
fragmentów genomu gospodarza do genomów retrowirusów
7. badanie metabolizmu/informacji genetycznej) organizmów
ewoluujących w innych ekstremalnych środowiskach (pH,
siła jonowa, środowiska oligotroficzne

Literatura
1.
A. Rzhetsky & F.J. Ayala (1999) „The enigma of intron
origins”, Cell Mol Life Sci, 55: 3-6
2.
A.M. Poole, D.C. Jeffares & D. Penny (1998) „The Path
from the RNA World”, J Molec Evol, 46: 1-18
3.
D.C. Jeffares, A.M. Poole & D. Penny (1998) „Relics from the
RNA World”, J Molec Evol, 46: 18-36
4.
T. Schultz, E. Samoylova,W. Radloff, I.V. Hertel, A.L.
Sobolewski,W. Domcke (2004) „Efficient Deactivation of a
ModelBase Pair via Excited-State Hydrogen Transfer”,
Science, 306: 1765-8.
5.
M.C.Y. Chang, C.S. Yee, J.A. Stubbe & D.G. Nocera (2004)
„Turning on ribonucleotide reductase by light-initiatedamino
acid radical generation” Proc Natl Aacad Sci U S A, 101:
6882–7
6.
E.R. Pianka „Ekologia ewolucyjna” PWN, Warszawa 1981
7.
P. Ricoeur „Język, tekst, interpretacja” Wybór pism, PIW,
Warszawa 1989,

Pytanie na kolejny wykład
Co tak naprawdę koduje DNA i czy
naprawdę „przeżywają” geny
zawierające „najlepszą”
informację genetyczną?

Od pierwotnego genomu DNA
do współczesnych genomów
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
Wyszukiwarka
Podobne podstrony:
Miliony cząsteczek podobnych do DNA i RNA
od relatywizmu do prawdy
od 33 do 46
od 24 do 32
Ewolucja techniki sekcyjnej – od Virchowa do Virtopsy®
Od zera do milionera
OD BABILONII DO HISZPANII
Od złotówki do stówki
Moje dziecko rysuje Rozwój twórczości plastycznej dziecka od urodzenia do końca 6 roku życia
Zagadnienia z botaniki pytania od 30 do 38, Botanika
Dziecko poznaje smaki - żywienie niemowląt, Dziecko, Żywienie niemowląt, żywienie dzieci (od noworod
Od zera do gier kodera6
03 Od krzaczka do krzaczka
Od marzen do realizacji fragment id 330850
od zera do ecedeela cz 2 (2)
Dajczak W Od retoryki do argumentacji prawniczej
Optymalizacja dostaw od producent%F3w do hurtowni
Pierwszy rok dziecka rozwój czesc II od urodzenia do 6 do 12 m cy
więcej podobnych podstron