Regulacja ekspresji
genów u
organizmów
eukariotycznych.
III.
Genetyczna regulacja morfogenezy
u roślin
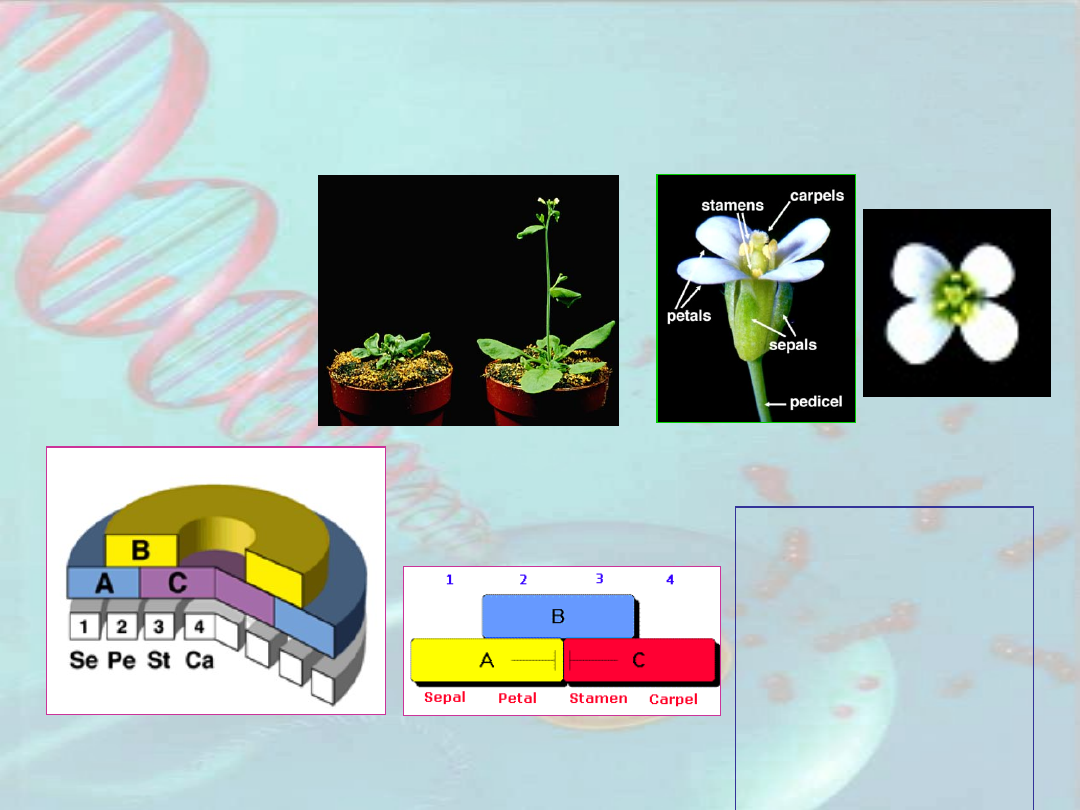
Geny odpowiedzialne za tożsamość
organów kwiatowych u A. thaliana
Morfologia kwiatu
dzikiego
A
- APETALA1
(AP1),
APETALA2
(AP2)
B
- APETALA3
(AP3),
PISTILLATA
(PI)
C
- AGAMOUS
(AG)
Gen lfy jest
konieczny dla
rozwoju
merystemu
kwiatowego
Sepal
– działki kielicha;
petal
– płatki korony;
stamen
– pręcikowie;
carpel
- słupkowie
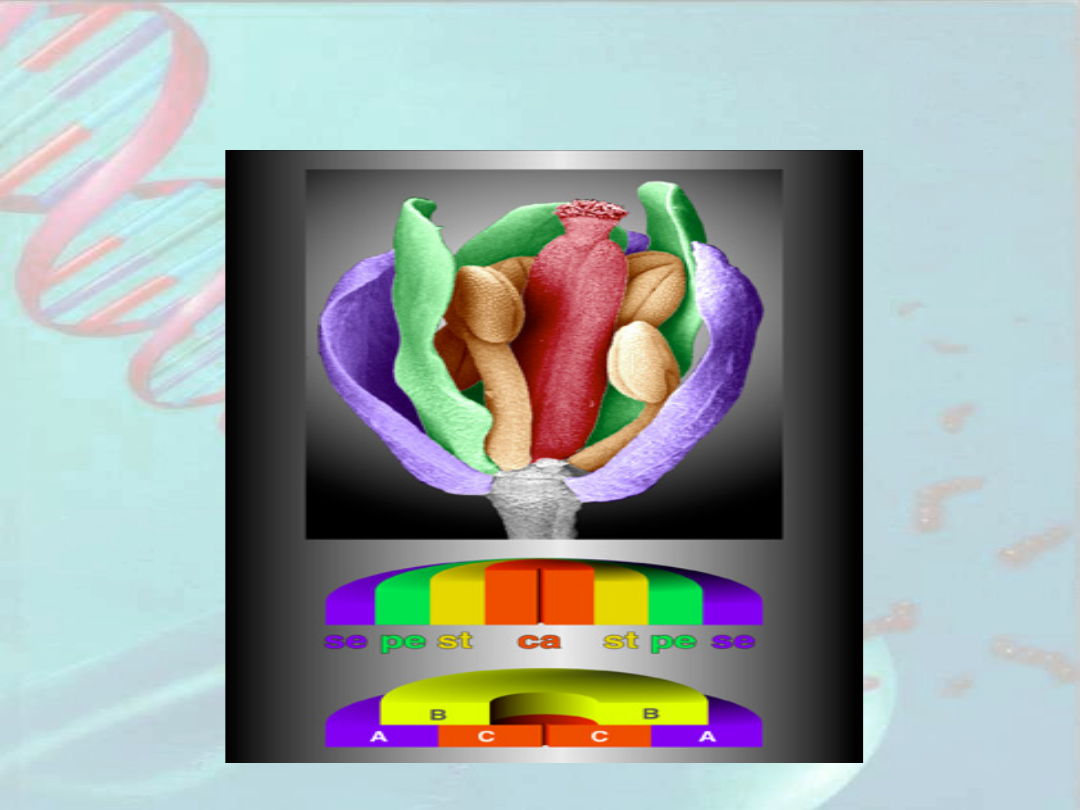
Geny odpowiedzialne za tożsamość
organów kwiatowych u A. thaliana

okółek
1
okółek 2 okółek 3 okółek 4
typ
dziki
działki
kielich
a
płatki
korony
pręciko
wie
słupkow
ie
typ 1
słupko
wie
pręciko
wie
pręciko
wie
słupkow
ie
typ 2
działki
kielich
a
płatki
korony
płatki
korony
działki
kielicha
typ 3
działki
kielich
a
działki
kielicha
słupkow
ie
słupkow
ie
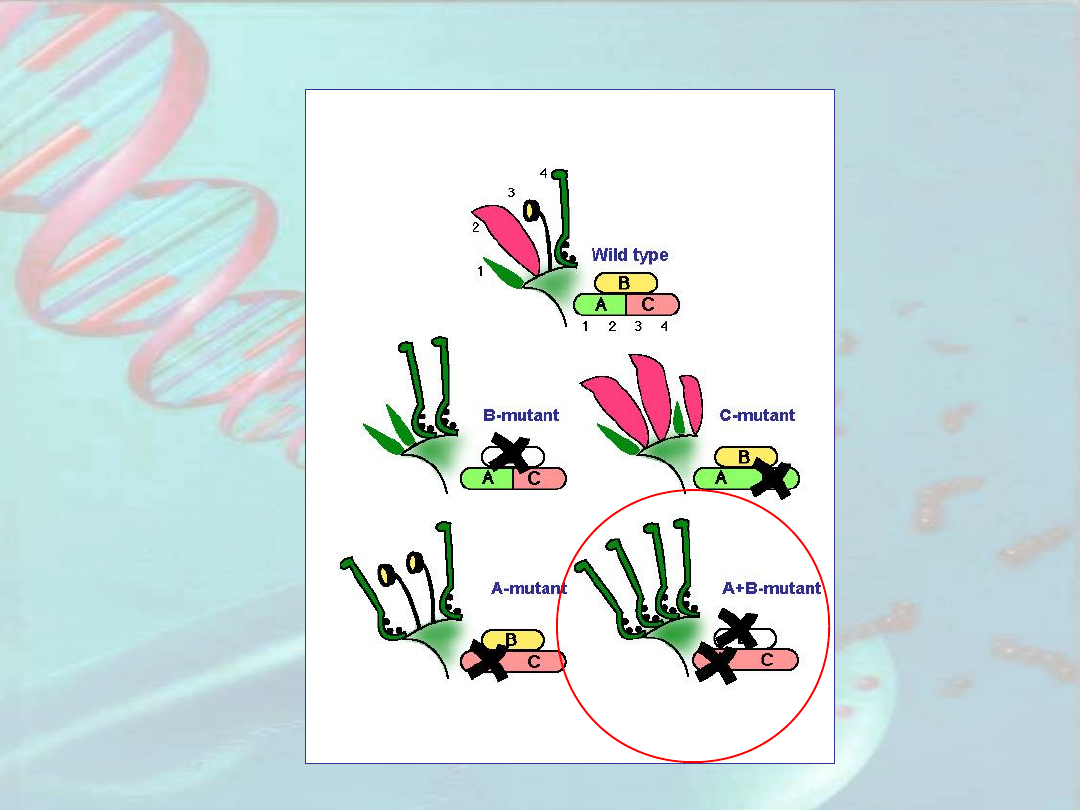
Mutacje genów tożsamości organów kwiatowych
u A. thaliana
Mutacje genów tożsamości organów
kwiatowych u A. thaliana
apetala
2
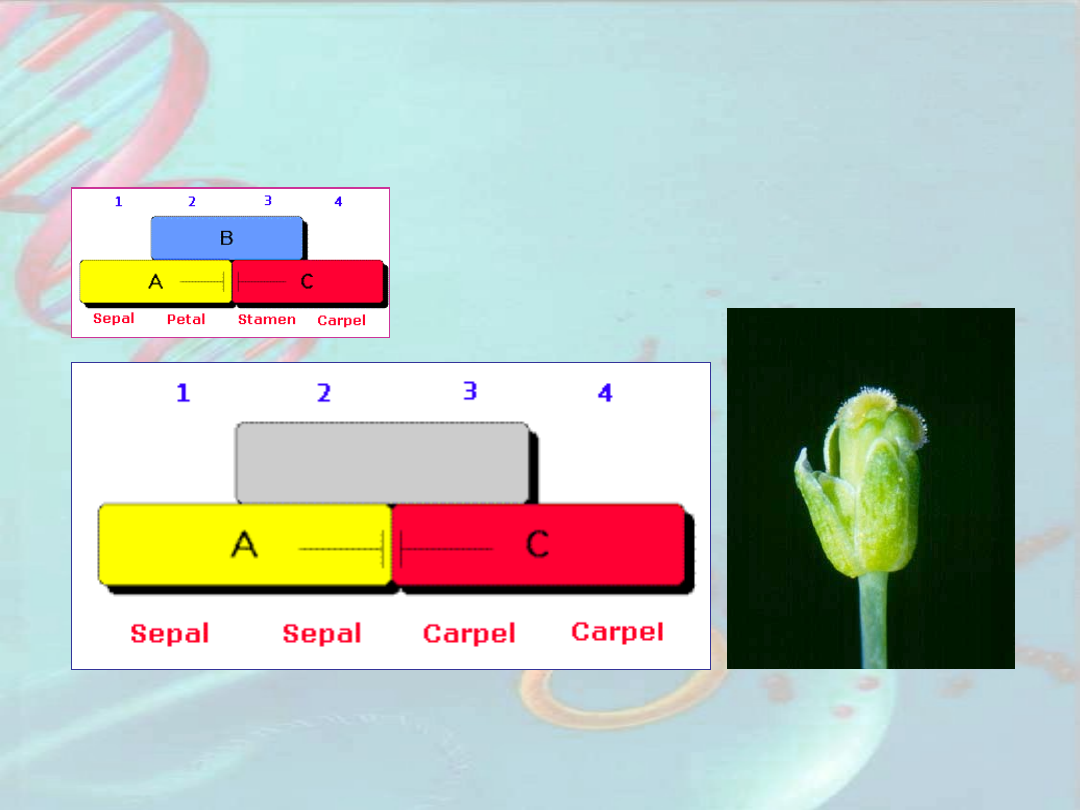
Mutacje genów tożsamości organów
kwiatowych u A. thaliana
pistila
ta
Mutacje genów tożsamości organów
kwiatowych u A. thaliana
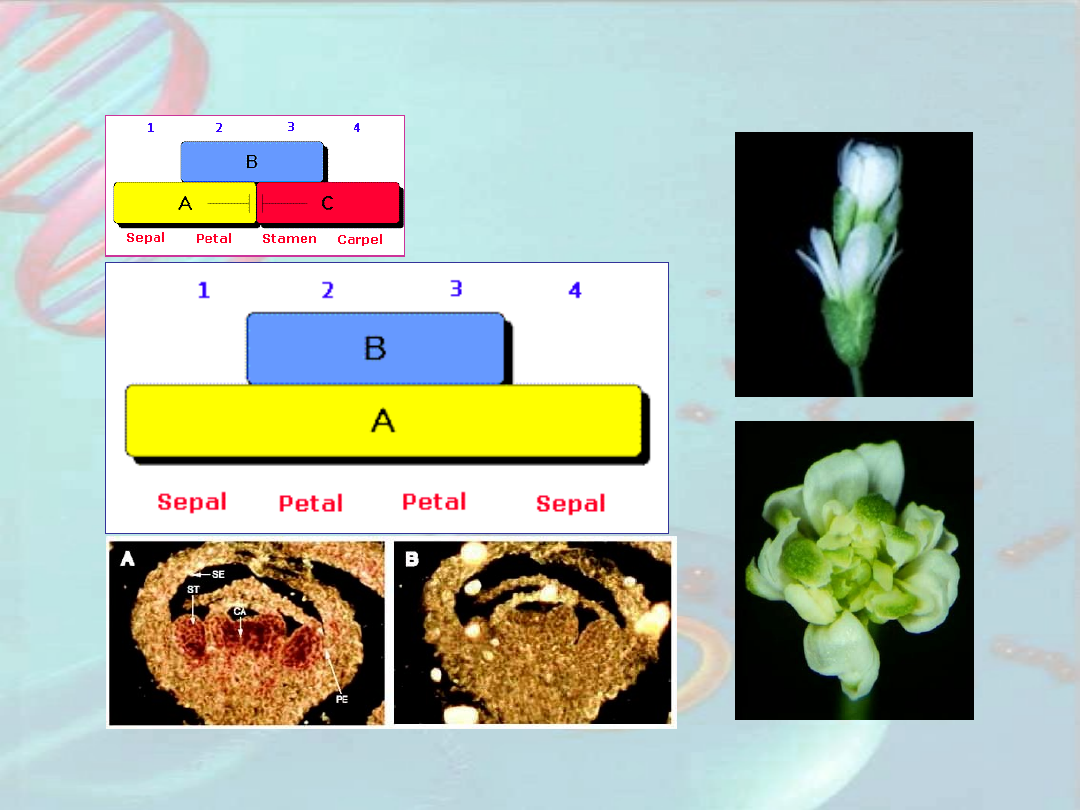
Mutant
agamous
Typ
dziki

Fenotypy mutantów
Arabidopsis
Sepal
– działki kielicha;
petal
– płatki korony;
stamen
– pręcikowie;
carpel
- słupkowie
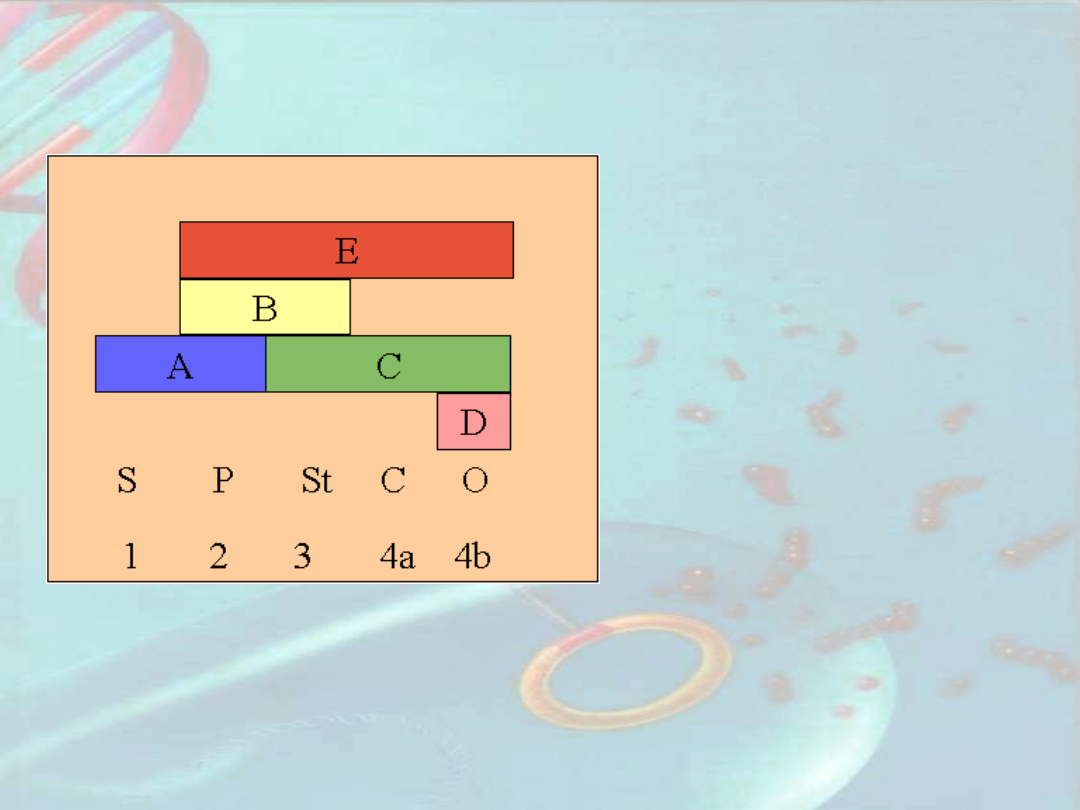
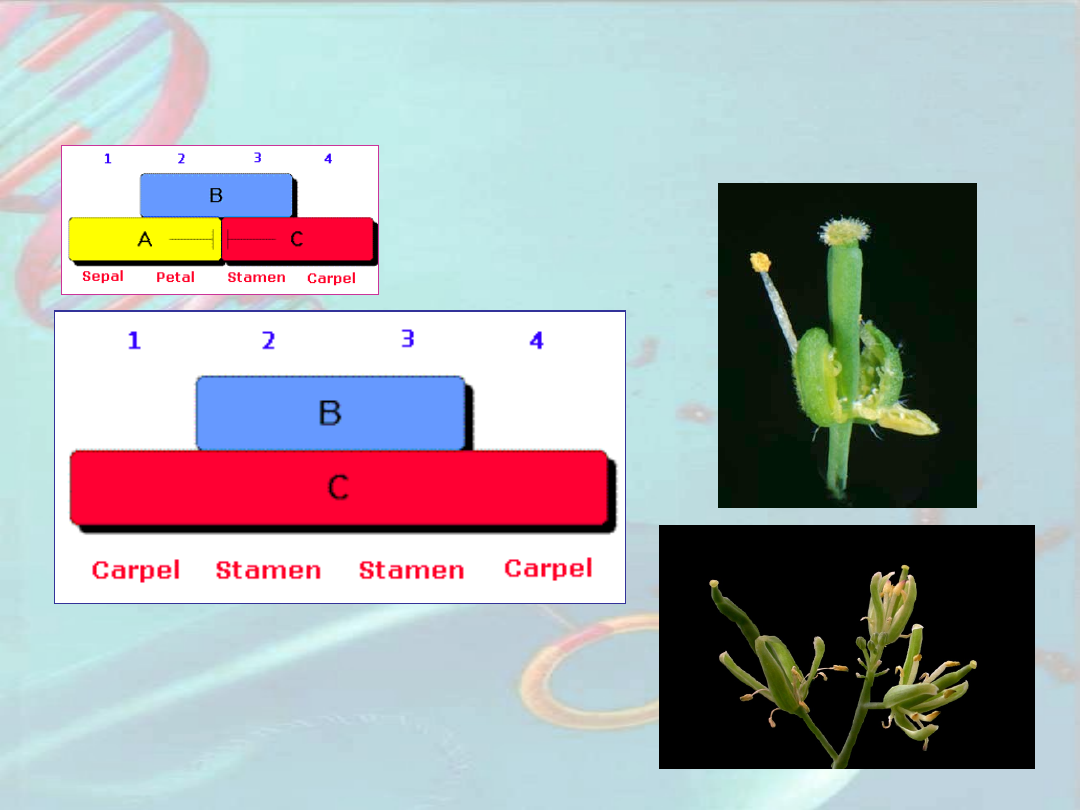
Model
ABCDE
S - sepal
– działki kielicha;
P - petal
– płatki korony;
S - stamen
– pręcikowie;
C - carpel
– słupkowie
O – ovule, ovary
– zalążek,
zalążnia
Geny D – seedstick, shatterproof
Geny E – sepallata 1, 2, 3, 4, 5

Mutacje genów tożsamości organów kwiatowych
u petunii
FBP1 (grupa B, podobny do PI)
FBP2 (grupa E, podobny do SEP)
FBP6 (grupa C, podobny do AG)
FBP7, 11 (grupa D)
http://www.dbp.science.ru.nl/our%20research.htm
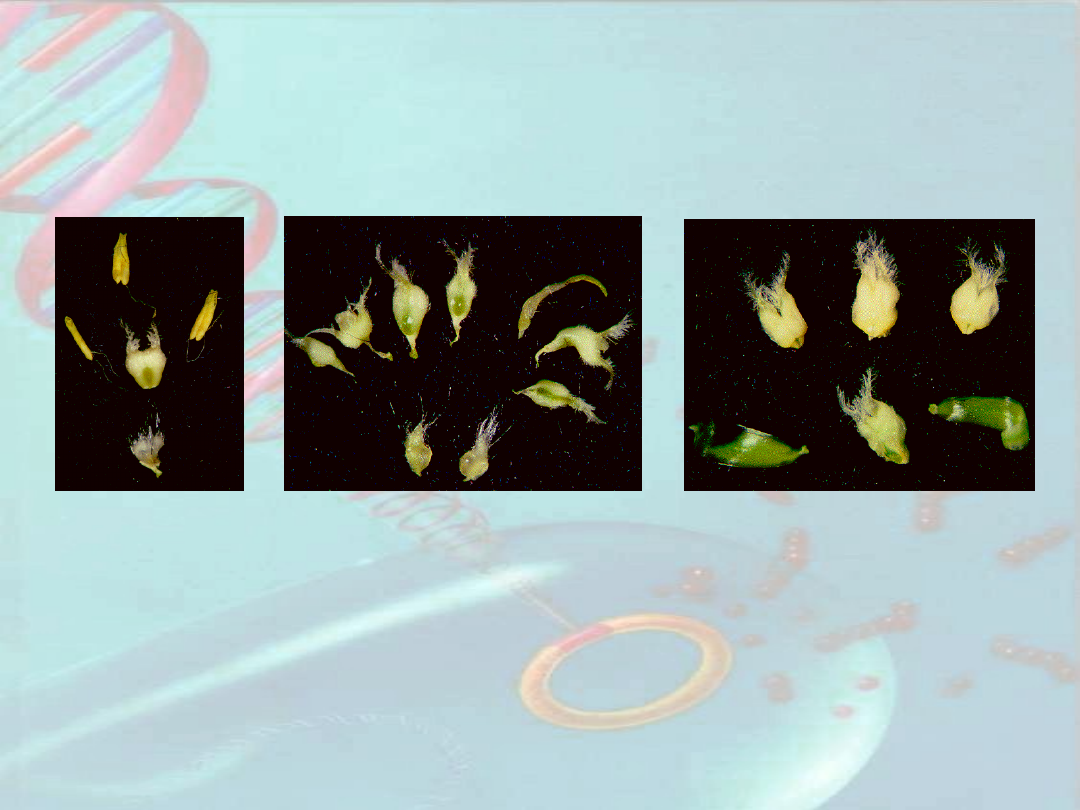
Kwiat
jęczmienia, typ
dziki
Mutant mo,,a –
zredukowana liczba
pylników, więcej słupków i
znamion, wydłużone
znamiona
Mutant mo,,b –
zredukowana liczba
pylników, więcej słupków,
wydłużone lodikule (odp.
płatków korony)
Mutacje genów tożsamości organów kwiatowych
u jęczmienia
Kandydat: gen
pJS18-2
, podobny do innych genów z
rodziny MADS

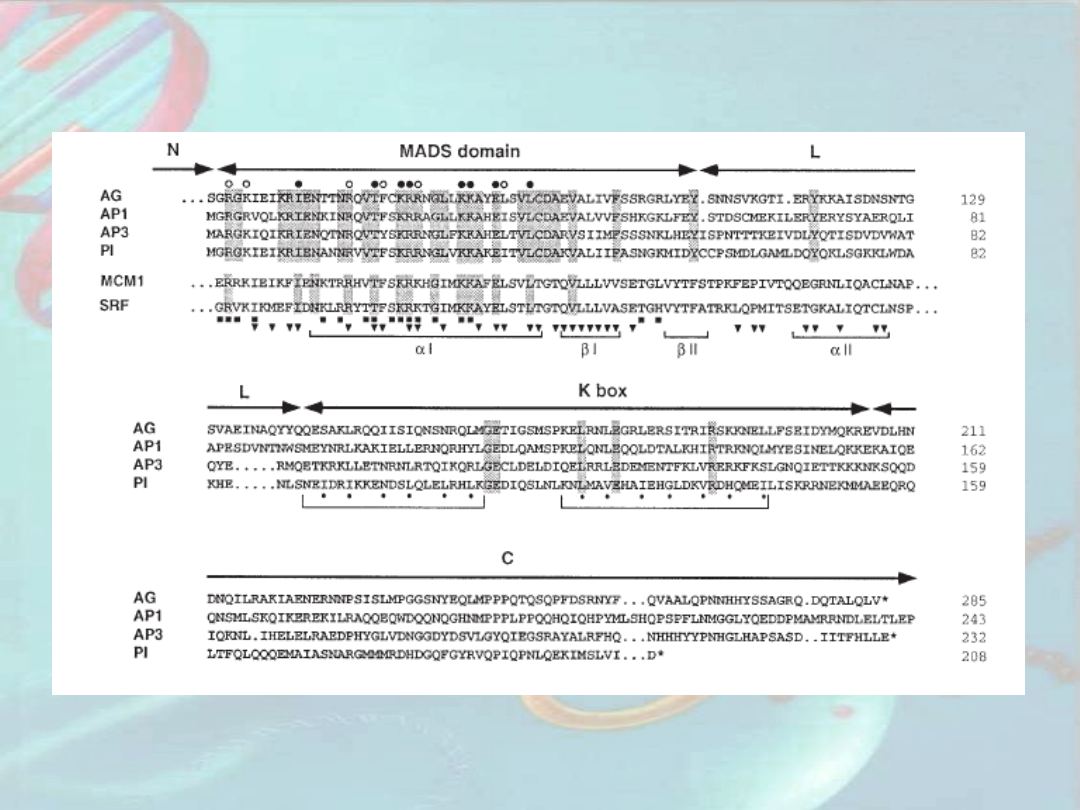
Rodzina genów MADS
Geny tożsamości organów kwiatowych u rzodkiewnika:
AP1, AP2, AP3, PI, AG
, wyżlinu:
DEF (DEFICIENS), GLO
(GLOBOSA)
oraz wiele innych roślinnych i zwierzęcych
tworzą rodzinę genów MADS; kodują białka regulatorowe
–
regulatory transkrypcji
(TF – transcriptional factor)
posiadające domenę wiążącą MADS (MADS-box) na N-
terminalnym końcu
M
– MCM1 (drożdże)
A
– AG (rzodkiewnik)
D
– DEF (wyżlin)
S
– SRF (człowiek)
Mutant def u
wyżlinu
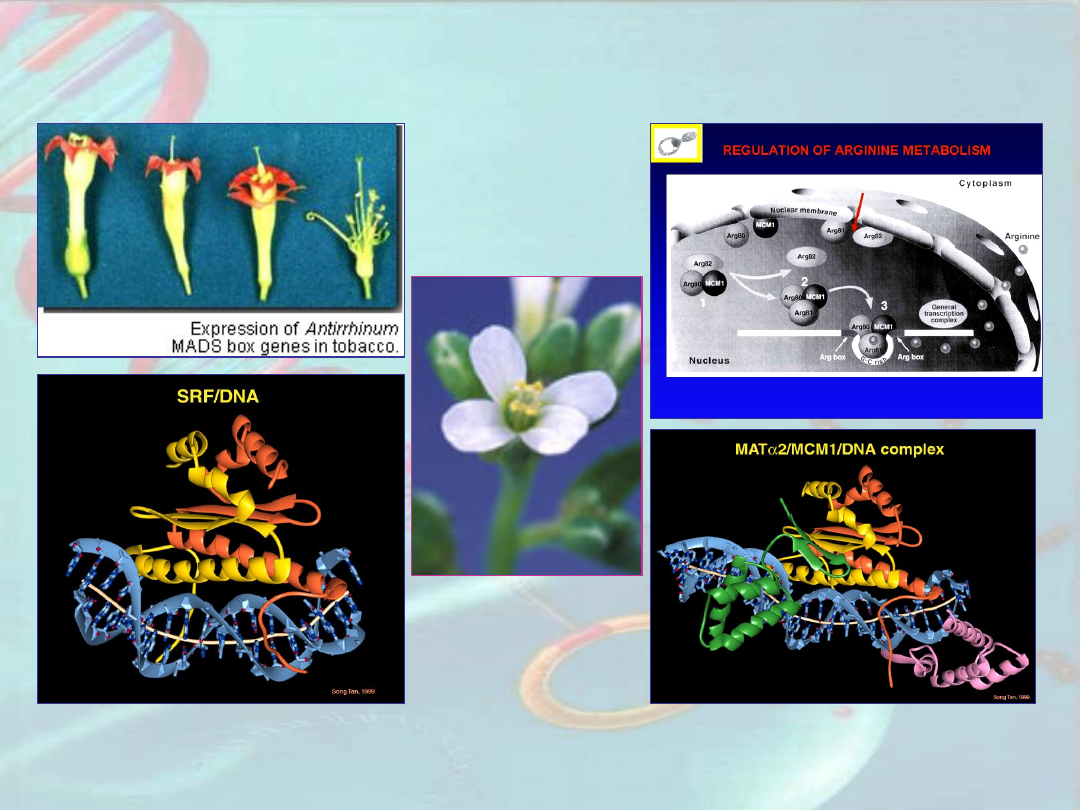
Rodzina genów MADS
rzodkiew
nik
drożd
że
człowiek
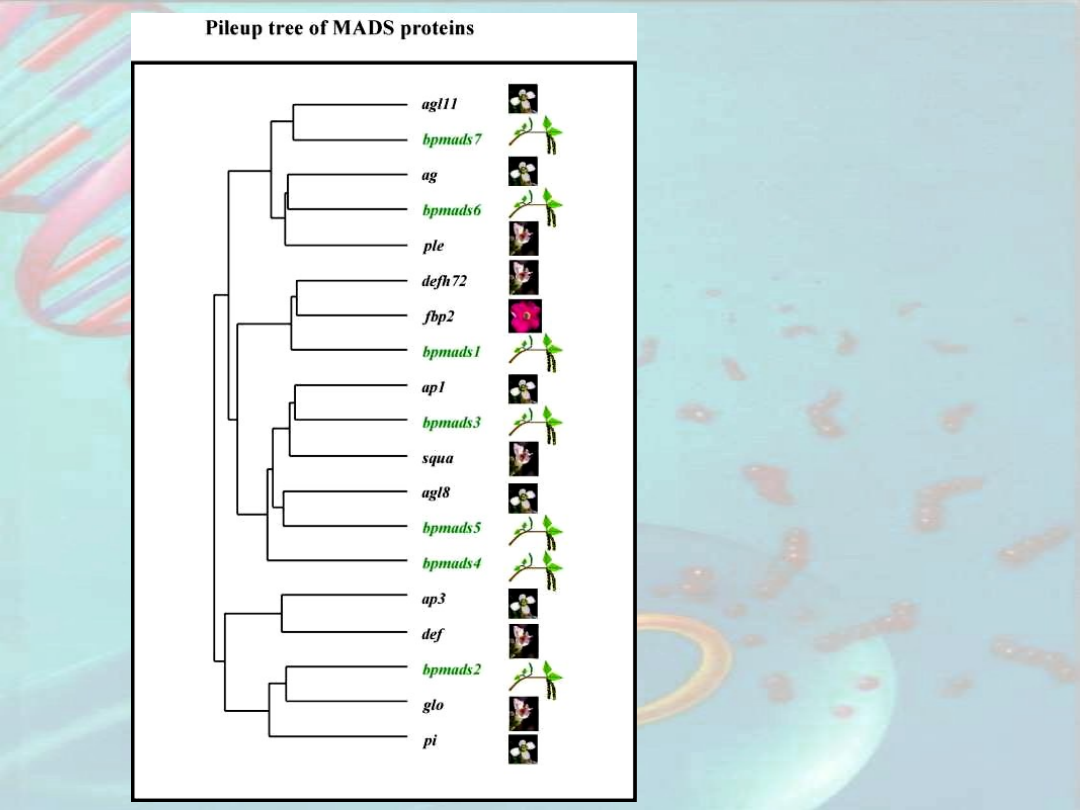
Podobieństwo genów
MADS u:
•rzodkiewnika
•leszczyny
•wyżlinu


„K-
box”
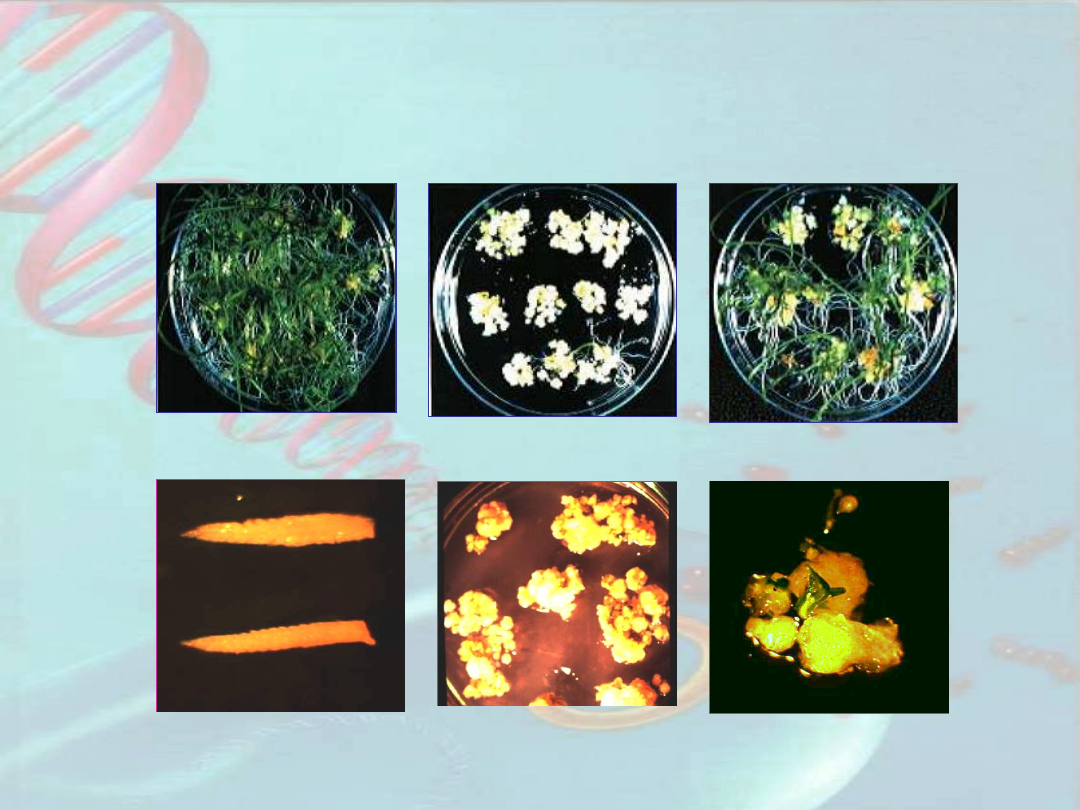
Genetyczne uwarunkowania reakcji w
warunkach in vitro
Jęczmień – niedojrzałe
zarodki
Żyto – niedojrzałe
kwiatostany


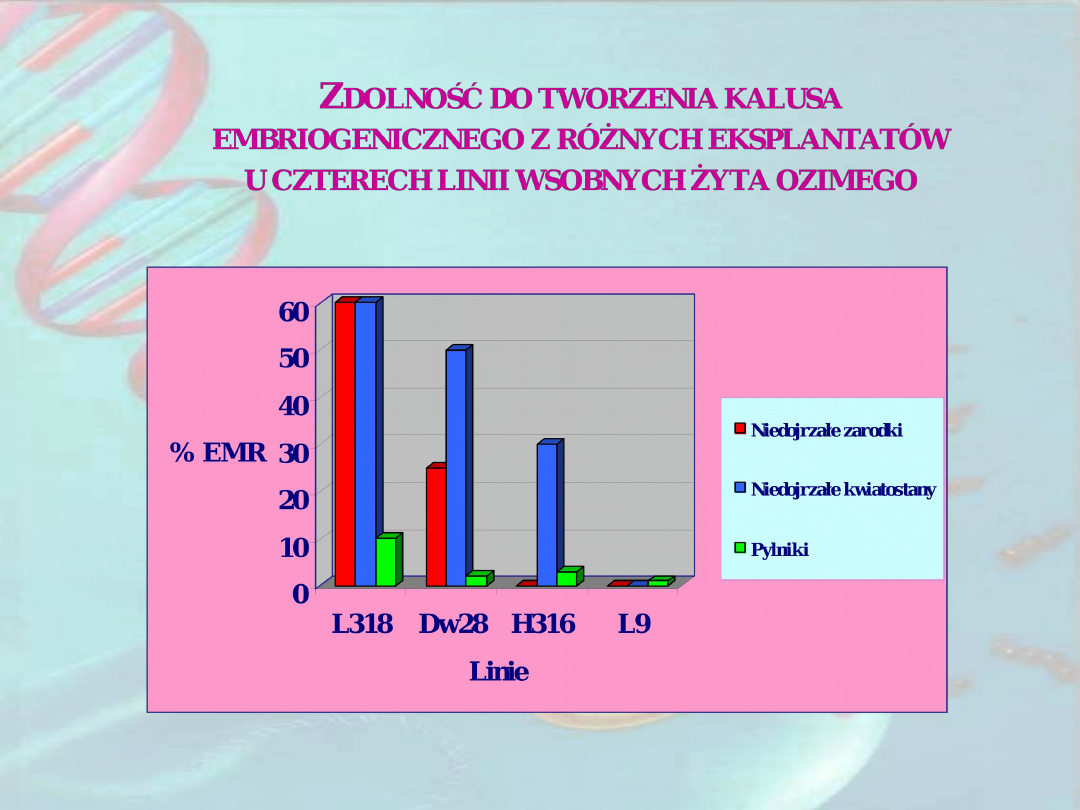
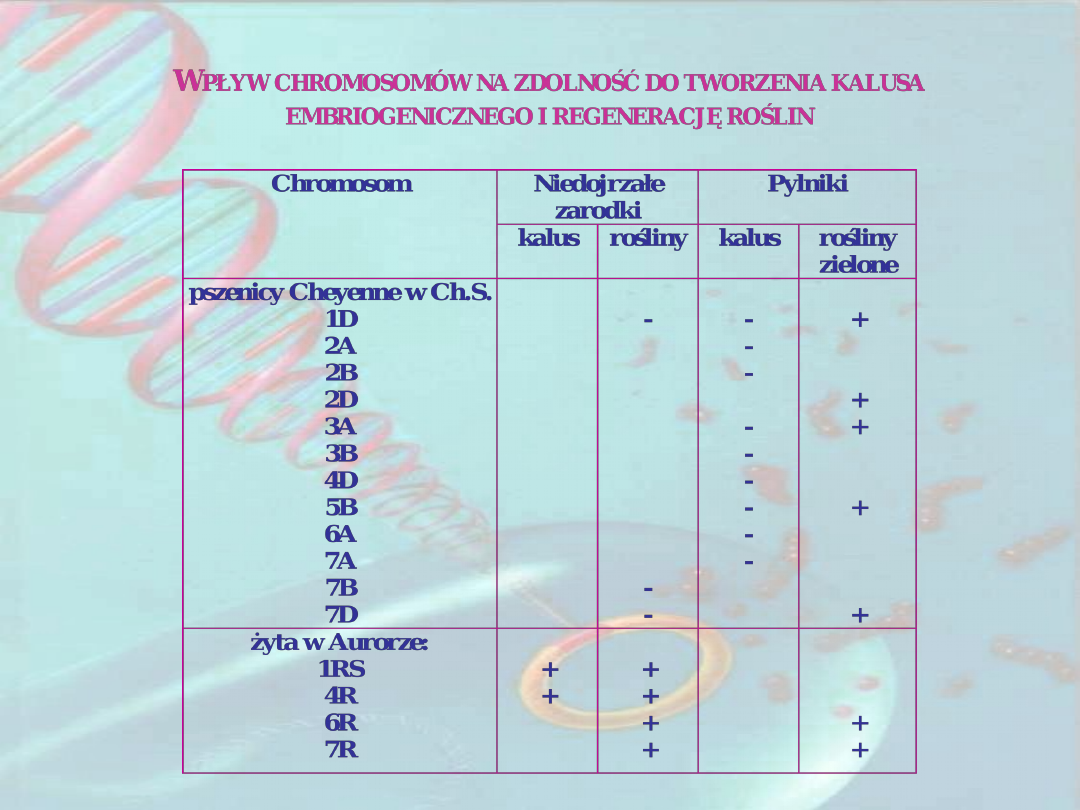

Loci cech ilościowych
kontrolujących zdolność do
regeneracji roślin in vitro
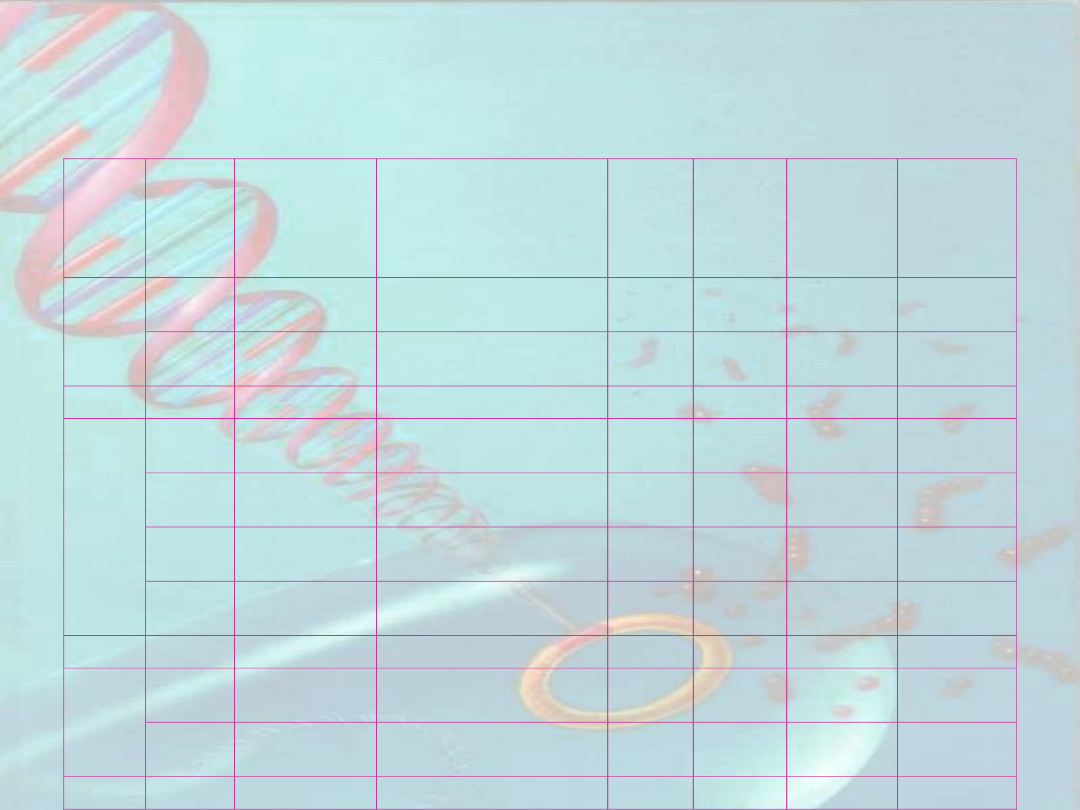
Cech
a
QTL
Chromoso
m
Przedział (długość
[cM])
Pozycj
a QTL
Wartoś
ć LOD
Efekt
addytyw
ny
%
wyjaśnia
nej
zmiennoś
ci
ECI
eci-1
5R
WMS6 – S32_900
(6.8)
0.0
3.59
-31.309
20.8
eci-2
6R
WRM229 –
SCM0068 (55.8)
4.0
3.23
-31.223
22.1
Σ
4.81
26.2
ESE
ese-1
1R
SCM0177 – S10_900
(24.3)
18.0
3.78
16.929
28.4
ese-2
4R
SCM0139 –
RMS1117 (14,4)
6.0
2.33
18.211
21.5
ese-3
5R
SCM109 – RMS1044
(35.7)
10.0
2.55
17.453
24.2
ese-4
6R
S_400 – SCM0078
(38.4)
28.0
3.64
10.581
41.6
Σ
7.69
69.1
ICI
ici-1
4R
SCM0047 –
SCM0139 (3.2)
0.0
2.32
8.029
11.4
ici-2
7R
M539_138 –
PB19_968 (27.1)
22.0
3.67
11.008
20.6
Σ
4.98
24.4
ISE
ise-2
7R
S45_550 – S45_520
(5.3)
0.0
2.40
-9.203
10.8
Charakterystyka QTLi kontrolujących reakcję niedojrzałych
zarodków (ECI, ESE) i niedojrzałych kwiatostanów (ICI,
ISE) żyta

7R
SCM0136
21.4
S45_550
5.0
S45_520
5.0
1.6
SCM0050
5.0
M1S39_222
6.5
RMS1012
3.3 RMS1018
1.7 SCM0063
2.2 REMS1135b
5.7
M3S38_130
14.8
OPN1_667
15.4
REMS1187
17.7
M5S39_138
22.3
PB19_968
28.8
M1S39_155
21.8
REMS1138
SCM86
0.7
SCM0041
ISE
ICI
ESE
ECI
6R
M1S29_198
11.9
SCM180
16.3
SCM0135
10.5
0.7
M3S39_190
16.7
WRM229
40.7
SCM0068
19.3
REMS1160
12.3
SCM0082
19.4
Q8_805
34.1
SCM2
6.3
S3_400
30.0
SCM0078
11.7
M1S29_145
RMS1121
SCM0168
5.9
5R
SCM0060
38.3
SCM268
14.3
SCM138
2.2
SCM0159
5.8
SCM109
28.2
RMS1044
27.7
RMS1115
4.6
8.9
SCM0152
11.5
D16_820
26.4
SCM0164
6.2
REMS1186
2.9
REMS1266
1.5
SCM0029
6.9
SCM0151
23.3
WMS6
6.4
S32_900
20.3
SCM0172
10.3
SCM0179
9.1
M5S38_100
SCM0077
REMS1132
SCM0098
RMS1063
1R
S44_750
10.3
S44_700
5.9
RE1135c
3.9
M5S38_146
4.6
SCM0107
3.1
SCM9
5.8
SCM0177
20.3
S10_900
SCM0021
4R
2R
SCM0099
REMS1130
S17_1000
19.8
E4_820
10.9
REMS1261
5.1
SCM75
9.6
RMS1042
5.1
SCM43
4.0
SCM0023
0.7
M5S39_185
SCM0091
SCM0087
SCM0095
S3_950
21.7
S42_650
11.5
REMS1277
10.9
M3S38_210
15.8
S10_750
9.2
M5S39_260
20.6
M5S38_110
12.1
4.9
WMS2
2.9
SCM206
1.7
5.0 SCM0084
3.3 SCM0162
5.1 F17_850
7.1
F17_1700
20.8
S27_850
10.6
SCM0112
3R
19.6
12.8
SCM101
SCM0047
3.1
SCM0139
RMS1117
2.3
WRM230
Mapa sprzężeń markerów molekularnych i zlokalizowane
na niej QTLe kontrolujące reakcję niedojrzałych zarodków
(ECI, ESE)
i niedojrzałych kwiatostanów (ICI, ISE) żyta
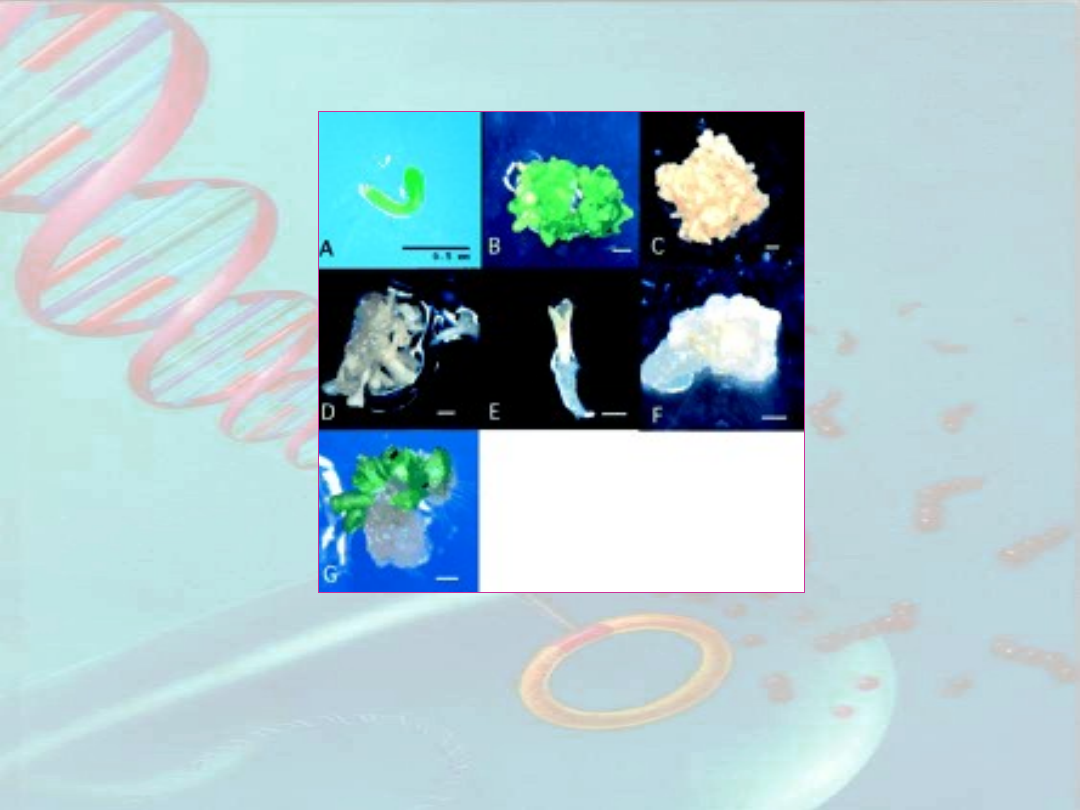
(A) Niedojrzały zarodek somatyczny (B) pierwotne
zarodki somatyczne (SE) (C) kalus embriogeniczny (D, E)
wtórne zarodki (F) kalus embriogeniczny o niskiej
kompetencji (G) trzeciorzędowe zarodki somatyczne
Somatyczna embriogeneza u
rzodkiewnika
Journal of Experimental Botany, Vol. 53, No. 374, pp. 1575-1580
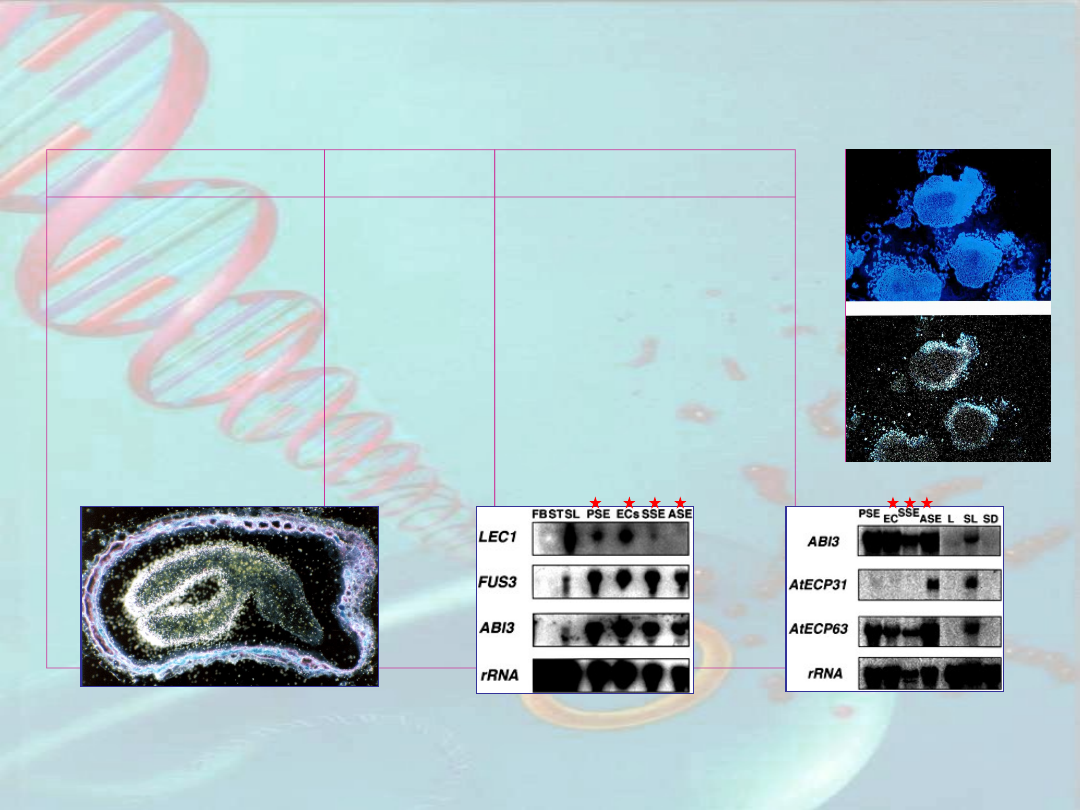
Geny ulegające ekspresji podczas
somatycznej embriogenezy
Gen
Gatunek Funkcja (białko)
SERK
LEC1, LEC2,
LEC3
WUS
FUS
CUS
ABI3
rzodkiew
nik
rzodkiew
nik
kukurydz
a
rzodkiew
nik
ogórek
rzodkiew
nik
Białko
receptorowe
regulator
transkrypcji
regulator
transkrypcji
regulator
transkrypcji
regulator
transkrypcji
regulator
transkrypcji
LEC1,
rzodkiewnik
CUS1,
ogórek
Ekspresja niektórych genów podczas somatycznej
embriogenezy
FB
- Pąki kwiatowe,
ST
– pędy,
SL
– zielone łuszczyny),
PSEs
– pierwotne zarodki
somatyczne,
ECs
– kalus embriogeniczny,
SSE
– wtórne zarodki somatyczne,
wtórne zarodki somatyczne traktowane ABA

Fenotypowe efekty nadekspresji
genu BBM u Arabidopsis i
Brassica
Plant Cell. 2002 August; 14(8): 1737–1749

http://www.salk.edu
http://www.biologie.uni-hamburg.de
http://biology.kenyon.edu/courses
http://www.arabidopsis.com
http://datf.cbi.pku.edu.cn
http://honeybee.helsinki.fi/MMSBL/Gerberalab
Plant Cell Preview (PDF)
A Gene Regulatory Network Model for
Cell-Fate Determination during
Arabidopsis thaliana...
Espinosa-Soto et al. Plant Cell.2004; 0:
104021725
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
Wyszukiwarka
Podobne podstrony:
wykład 6 fizj roślin, biologia, fizjologia roślin
wyklad 4 fizj roślin, biologia, fizjologia roślin
Wykład 3 ZATRUCIA ROŚLINAMI I GRZYBAMI
wyklady tech roślinna (ziemniak)
Wykład Chemoprevemtion Rosliny Kapustne
wykład 1 systematyka rośliny
Wykład7 morfogeneza zwierząt
WYKLAD 6 Leki roslinne stosowane w leczeniu chorob zoladka, jelit
WYKŁAD 6 Leki roślinne stosowane w leczeniu chorób żołądka, jelit
Zagad.Biologia 2011, I rok, Fizjologia roślin - wykłady, Fizjologia roślin
Wykłady - lek roślinny(2), studia -farmacja gumed, rok V, lek roślinny
Wykłady Fizjo Roślin
wykład 6 fizj roślin, biologia, fizjologia roślin
wyklad 4 fizj roślin, biologia, fizjologia roślin
Wykład 3 ZATRUCIA ROŚLINAMI I GRZYBAMI
Wykłady lek roślinny(1)
Wykład 2 Fizjologia roślin
więcej podobnych podstron