
Struktura i funkcje
jądra komórkowego i jąderka
Strukturalne uwarunkowania procesów
zachodzących w jądrze komórkowym
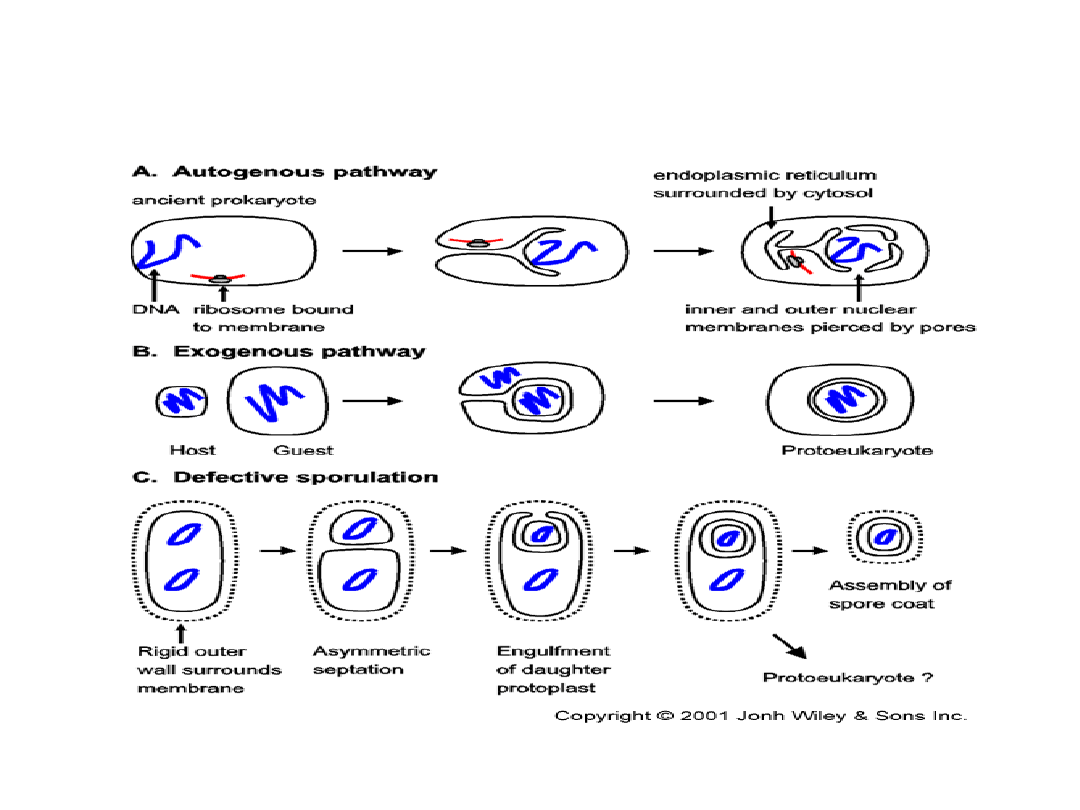
Hipotezy dotyczące ewolucji jądra i błony jądrowej

Składniki jądra komórkowego - DNA
cechy chemiczne
i morfologiczne
lokalizacja/funkcja
satelitarny
- wysoki stopień kondensacji
- tandemowy układ krótkich,
powtarzających się sekwencji
heterochromatyna
nie podlega
transkrypcji
rodzaj
umiarkowanie
powtarzalny
- rozmaity stopień kondensacji
- rozmaita długość sekwencji
odcinki pomiędzy
sekwencjami unikalnymi,
przerywniki
Regulacja aktywności genów
rDNA
- nieznaczny stopień kondensacji
- znaczna powtarzalność sekwencji
przewężenia wtórne
chromosomów,
czasami kariolimfa
ulega transkrypcji na pre-rybosomy,
nie podlega translacji
inne DNA
- nieznaczny stopień kondensacji,
(luźne fibryle chromatynowe)
lub okresowo skondensowane
- unikalne sekwencje nukleotydów
euchromatyna
ulega transkrypcji,
niektóre rodzaje RNA (mRNA)
podlegają translacji

wielkość
genomu
% repetytywnego
DNA
muszka owocowa
organizm
Powtarzające się (repetytywne) DNA
% kodujących
sekwencji DNA
nicień C. elegans
pies domowy
100 MB
175 MB
2.4 GB
mysz
2.5 GB
człowiek
2.9 GB
16.5
14.0
<10
33.7 - ~57
31
1.45
40
1.40
>50
1.20
ryż
466 MB
42
11.8
kukurydza
2.5 GB
77
1
hipoteza „informatyczna”
DNA służy do przechowywania informacji:
pamięć stała – sekwencje kodujące DNA, informacja stabilna i przekazywana kolejnym
pokoleniom komórek
pamięć średnio-terminowa – za pomocą trwałych kompleksów z RNA i białkami oraz modyfikacji
chemicznych DNA (metylacja, acetylacja), może być przekazywana potomnym komórkowm
pamięć krótko-terminowa – dynamicznie powstające kompleksy z RNA i białkami, regulowane
w czasie cyklu komórkowego i w odpowiedzi na warunki środowiska, informacja nie jest
przekazywana komórkom potomnym

wielkość
genomu
ilość genów
muszka owocowa
organizm
nicień C. elegans
100 MB
175 MB
mysz
2.5 GB
człowiek
2.9 GB
18 424
13 601
30 000
25-35 000
u człowieka:
ok. 25 tys. genów
100-200 tys. rodzajów m-RNA
1-3 mln różnych białek
tylko
ok. 2% transkryptów cRNA koduje białka
98% transkryptów ncRNA powstaje na podstawie
tzw. strukturalnego DNA (niekodujące sekwencje)
im bardziej skomplikowany organizm tym większa ilość strukturalnego DNA
dlaczego?
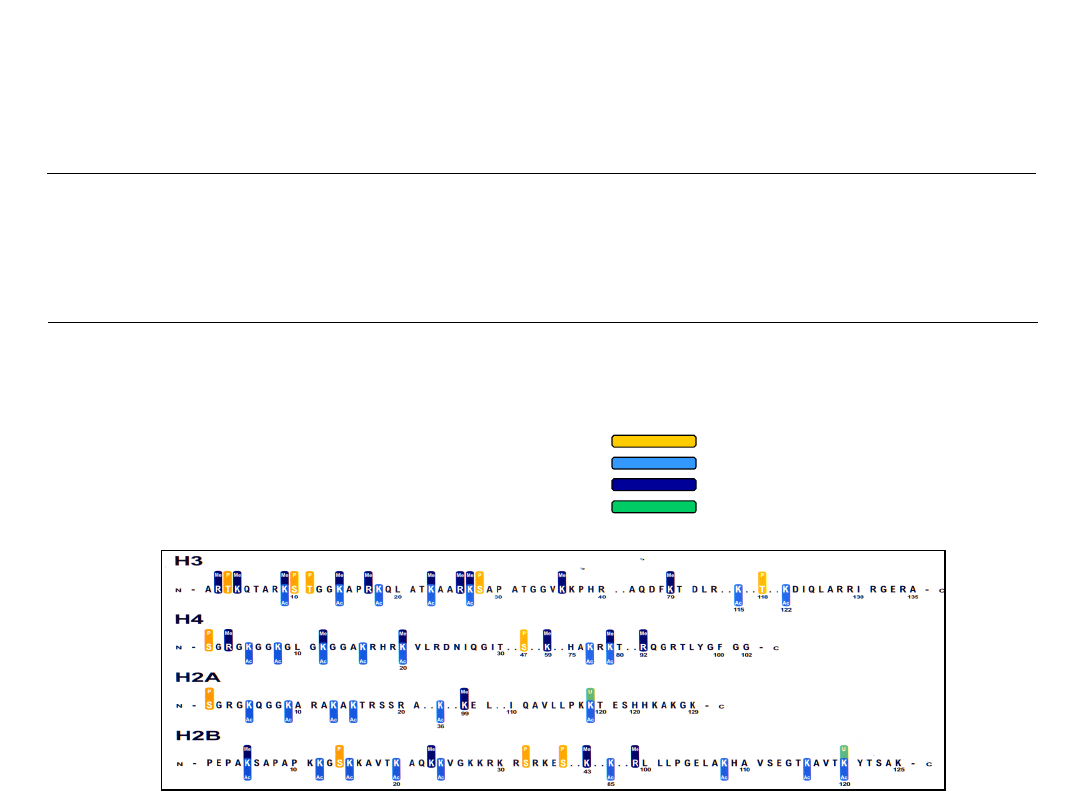
Składniki jądra komórkowego - białka
zawartość [%]
masa cząsteczkowa
[kDa]
klasy histonów
argininy
lizyny
H1
1.8
26.8
19.5-21
H2A
6.4
16.0
14.5
H2B
9.3
10.8
13.8
H3
13.3
9.6
15.3
H4
13.7
9.8
13.0
białka histonowe
post-translacyjne modyfikacje białek histonowych
kod histonowy – wzór modyfikacji post-translacyjnych obecnych w białkach histonowych
fosforylacja
acetylacja
metylacja
ubikwitynacja
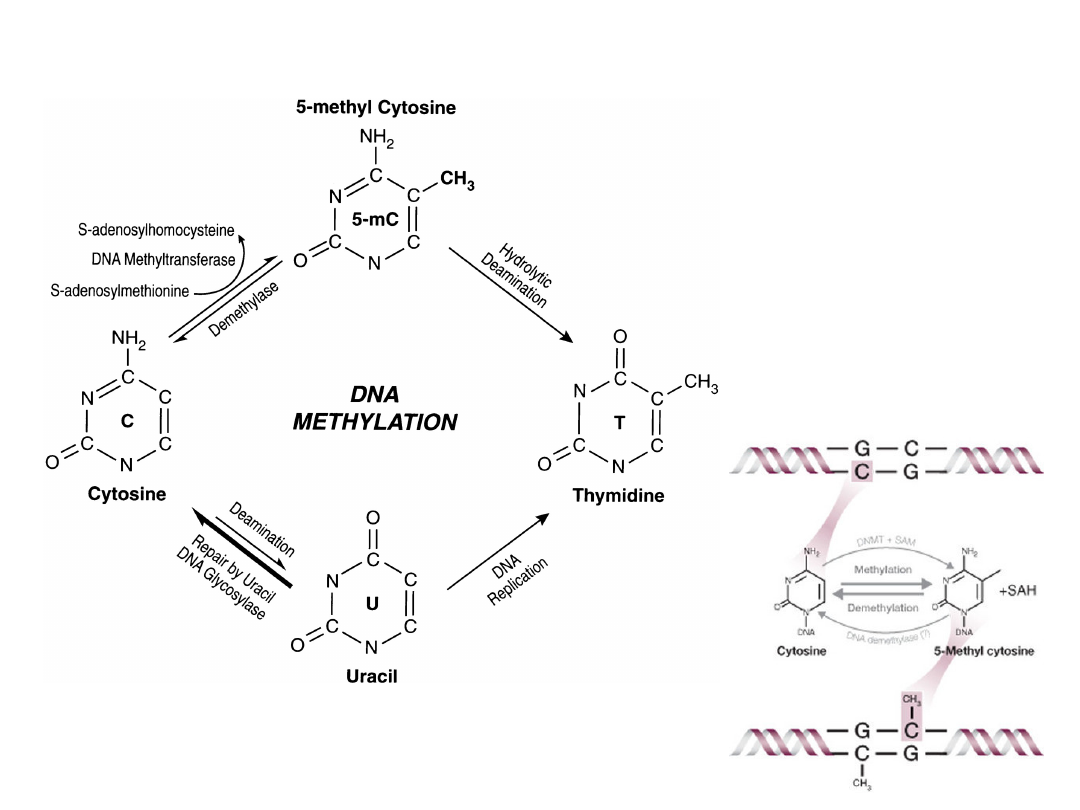
Reszty cytozyny w DNA mogą być metylowane
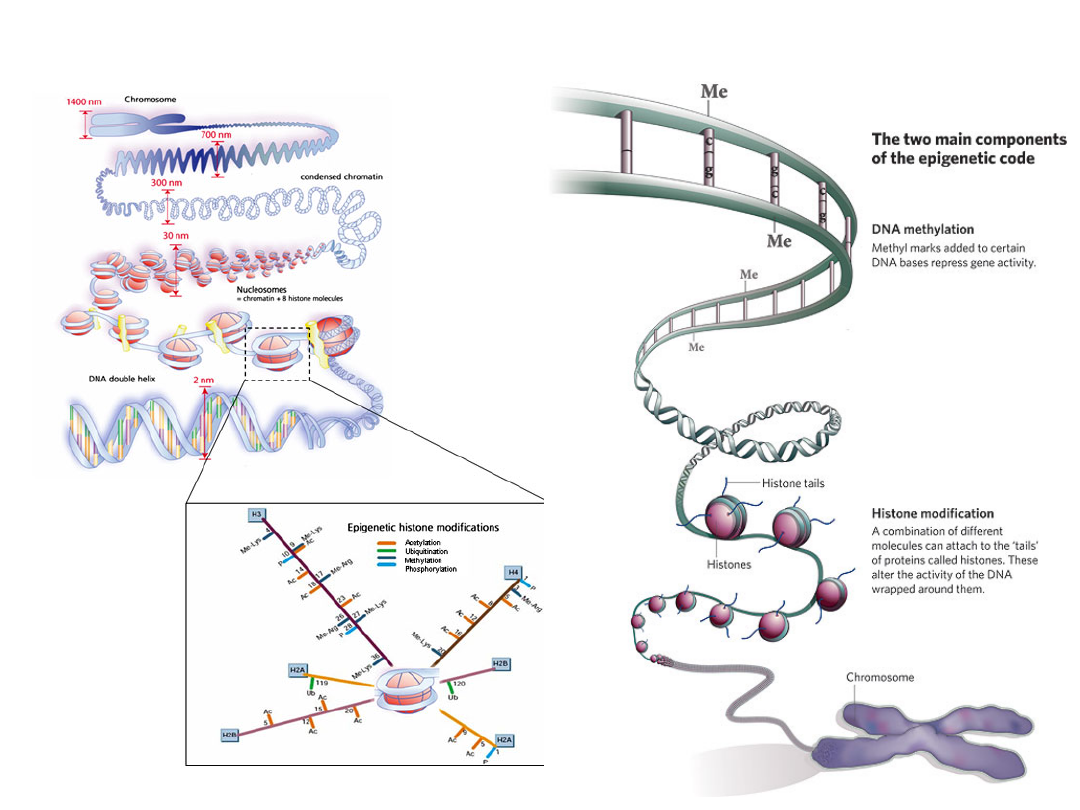
Składniki kodu epigenetycznego

Zmiany w metylacji cytozyn i modyfikacje histonów
reguluje profil ekspresji genów

białka niehistonowe
enzymatyczne
polimerazy DNA, RNA, poli-A
terminalne nukleotydylotransferazy
ligazy, N-glikozydazy
kinazy polinukleotydowe
topoizomerazy I, II ( i )
regulatorowe
proteazy, kinazy, fosfatazy
metylotransferazy/demetylazy
acetylotransferazy/deacetylazy
syntaza poli(ADP)rybozy
glikohydrolaza poli(ADP)rybozylowa
strukturalne
białka HMG (ang. high mobility group)
niektóre białka enzymatyczne i regulatorowe
Składniki jądra komórkowego - białka
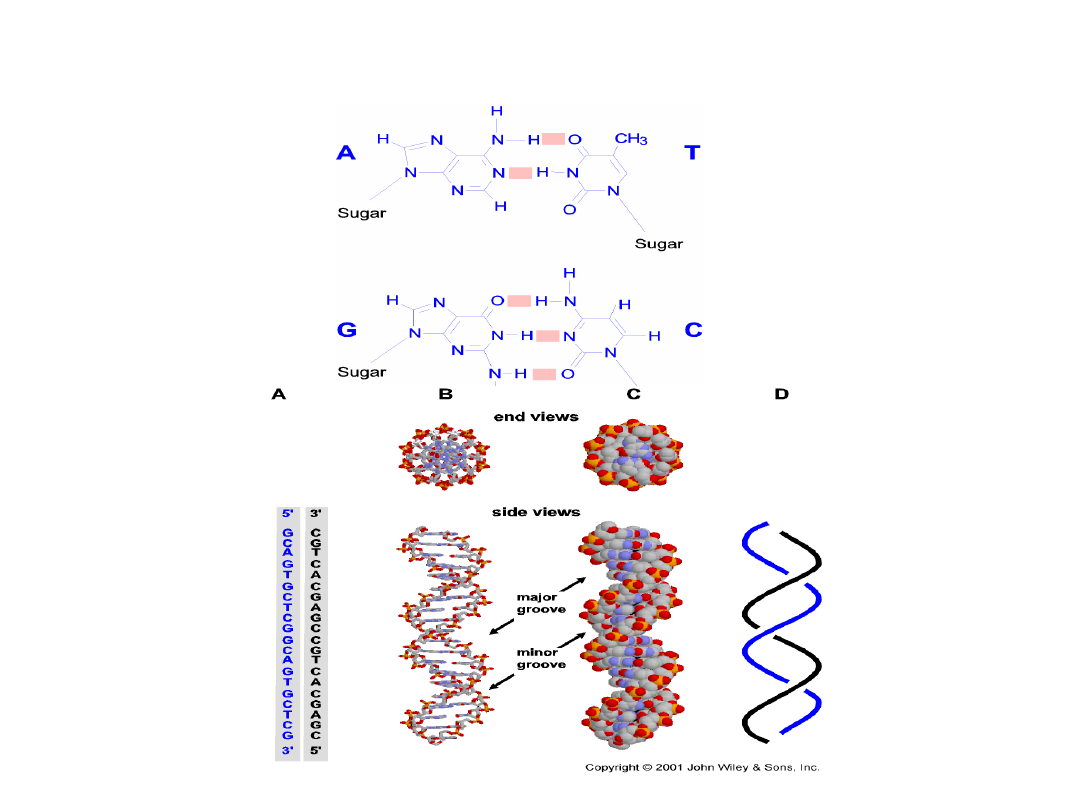
Struktura DNA
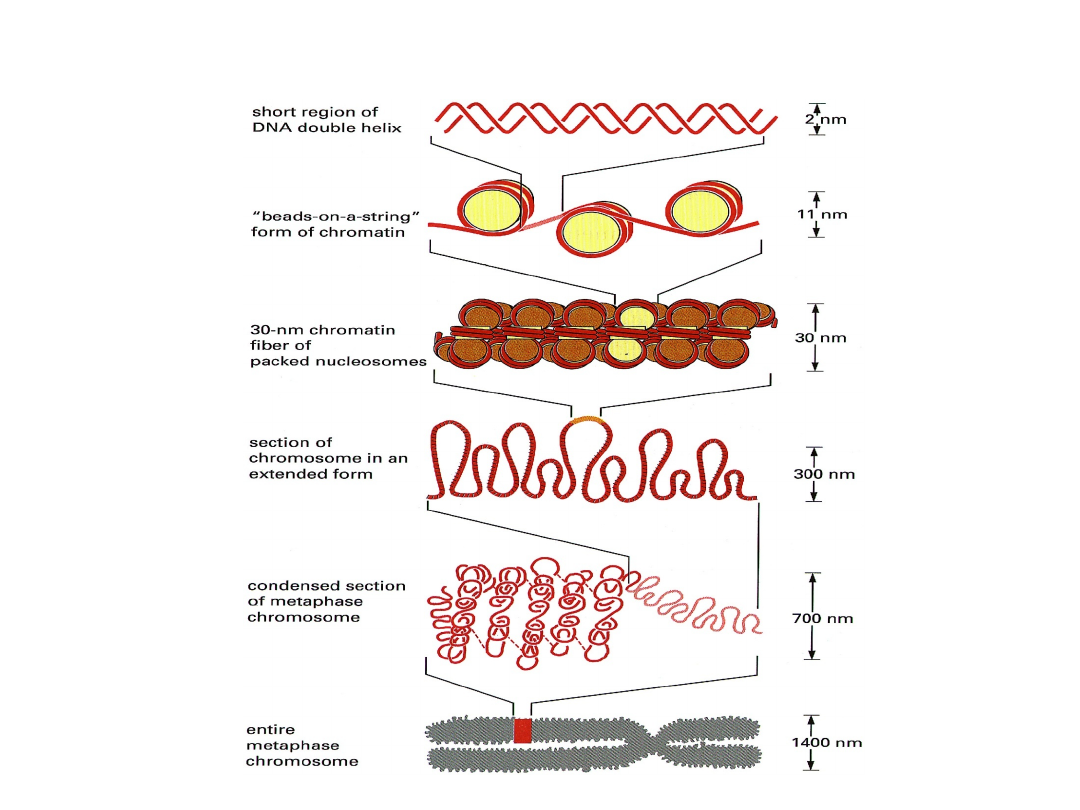
Poziomy organizacji chromatyny

Poziomy organizacji chromatyny w jądrze komórkowym

Struktura typowego chromosomu metafazalnego
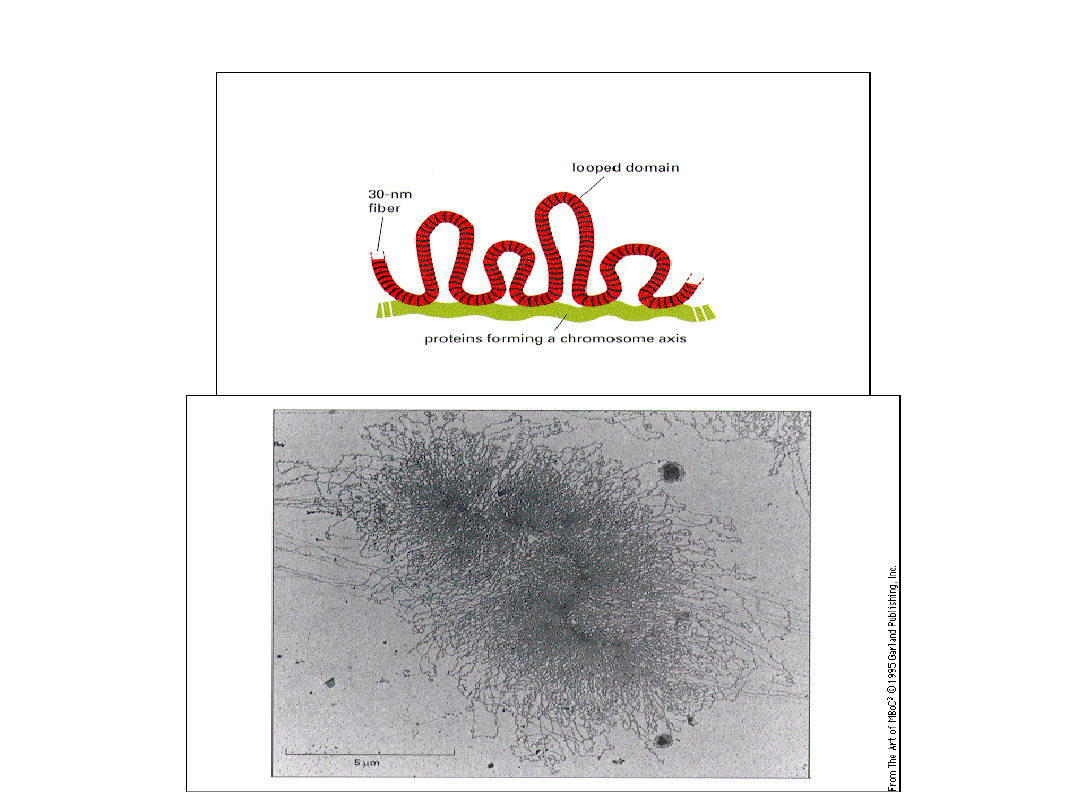
Struktura chromosomu
30 nm filamenty
60000 pętli, każda 5-150 kpz
(średnio 60 kpz)
ok. 1000-3000 miejsc replikacji
Poziomy organizacji DNA w jądrze
helisa DNA
pętla DNA
nukleosom
„chmura”
chromatyny
„obszar”
chromosomalny
1 m
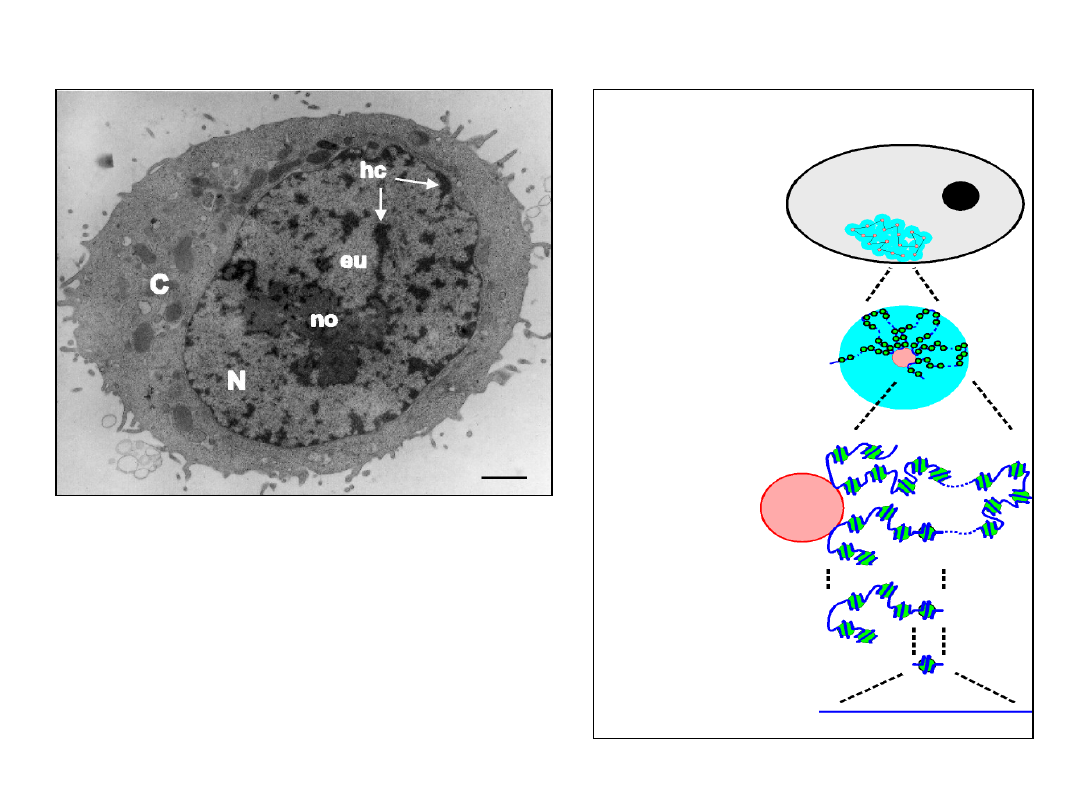
Organizacja DNA w jądrze
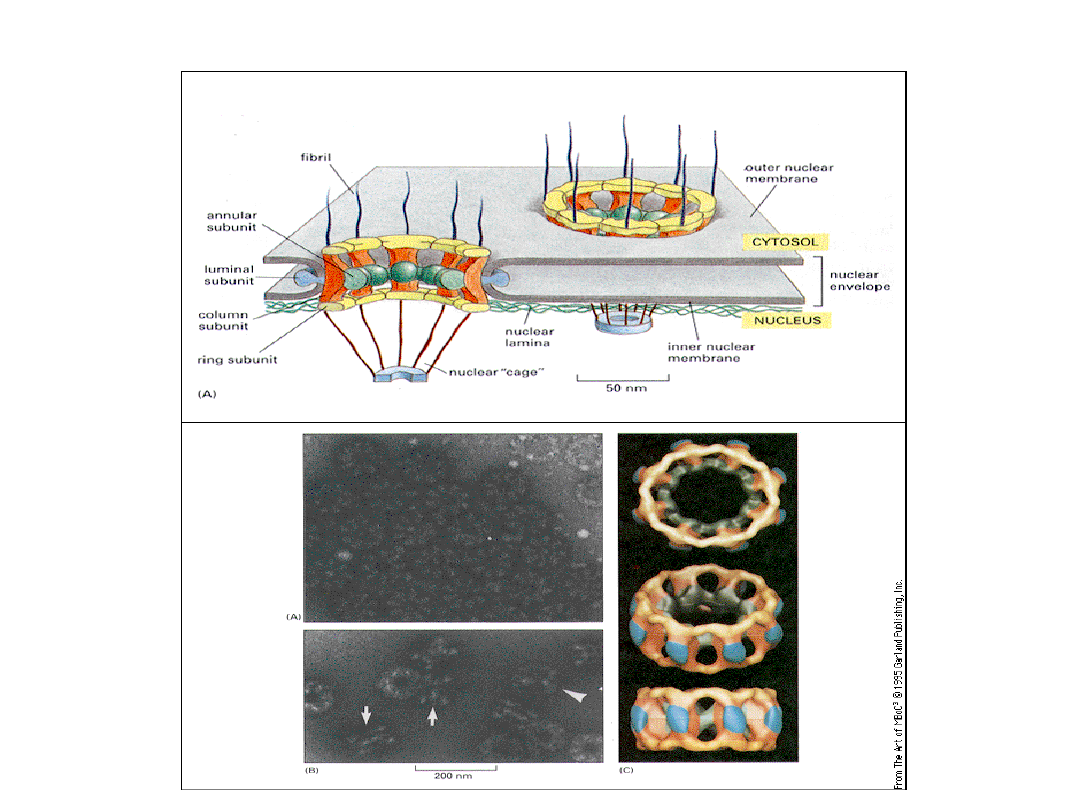
Organizacja strukturalna por jądrowych
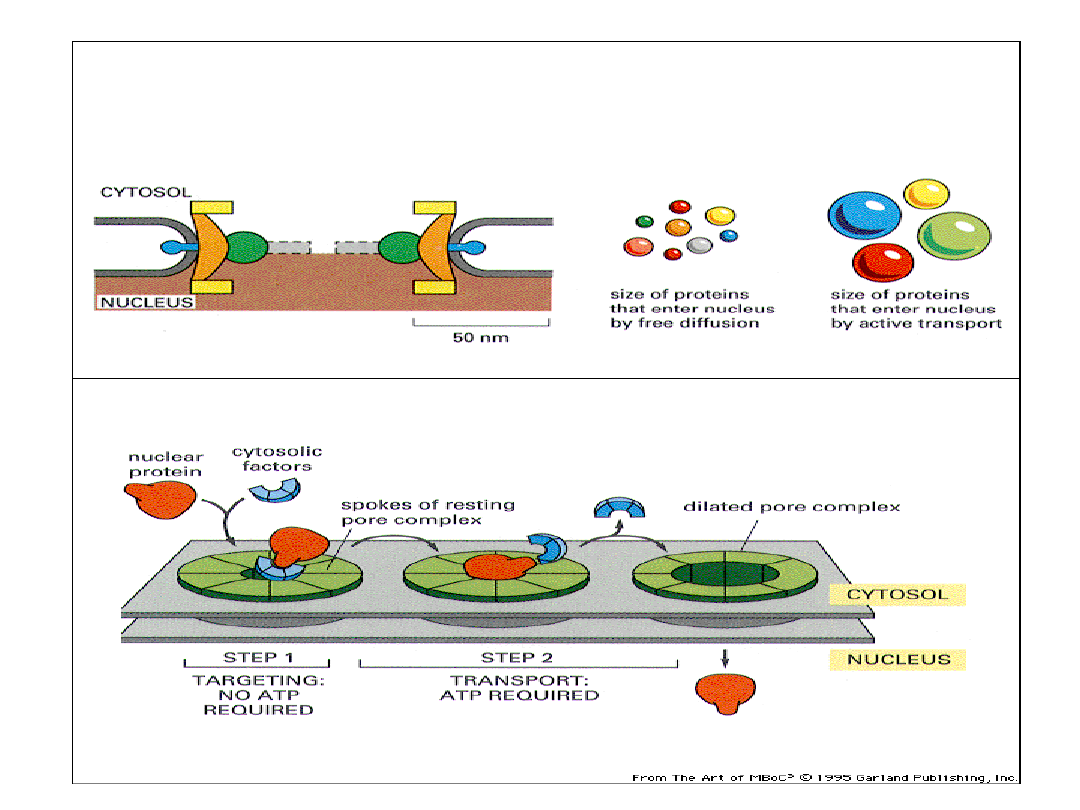
Schemat transportu przez pory jądrowe
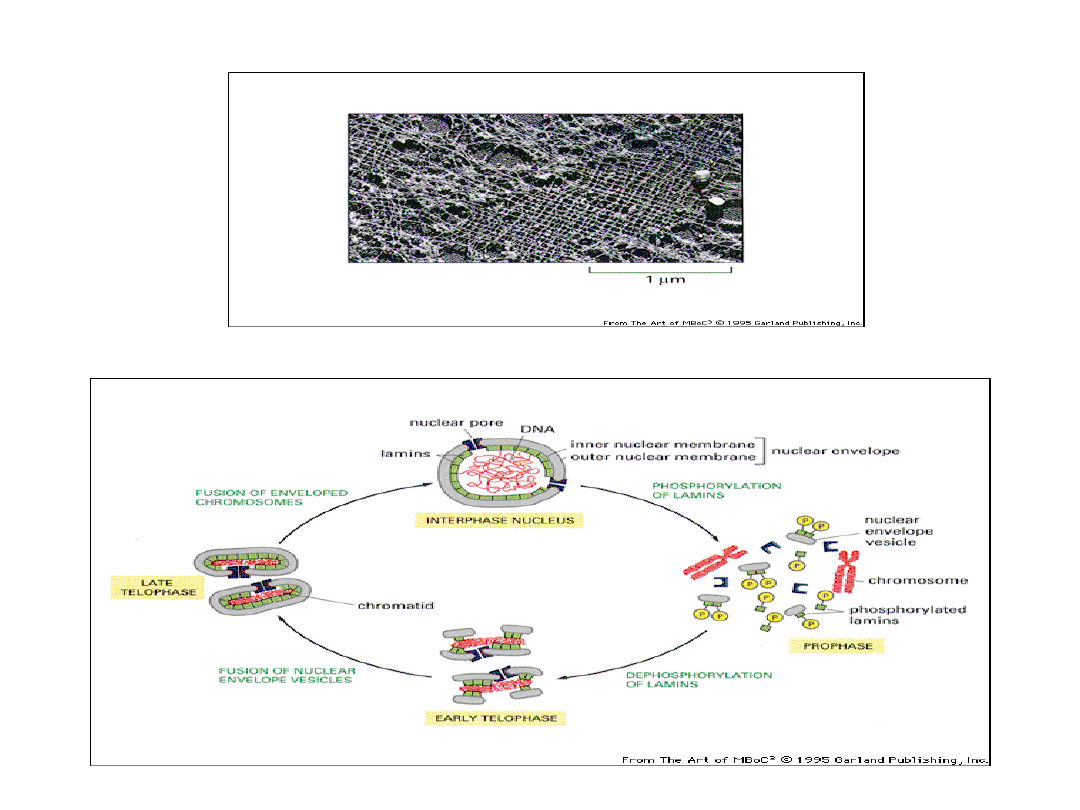
Siatka lamin
Struktura błony jądrowej rozpada się i odtwarza się
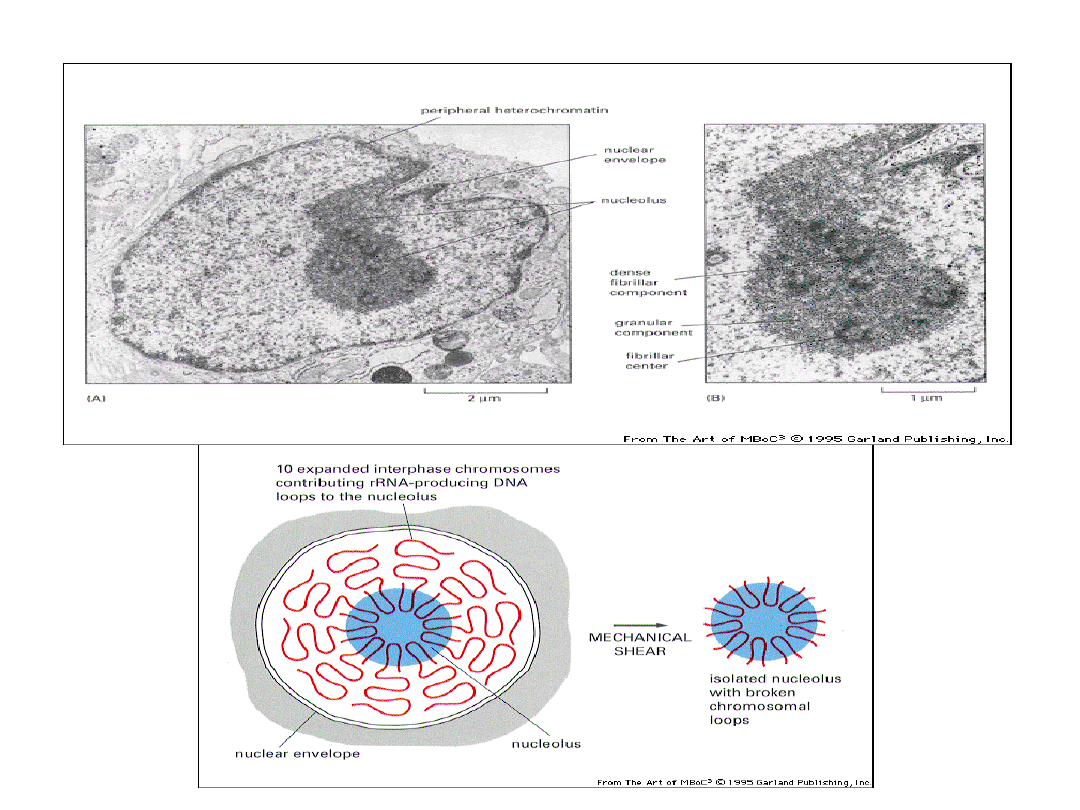
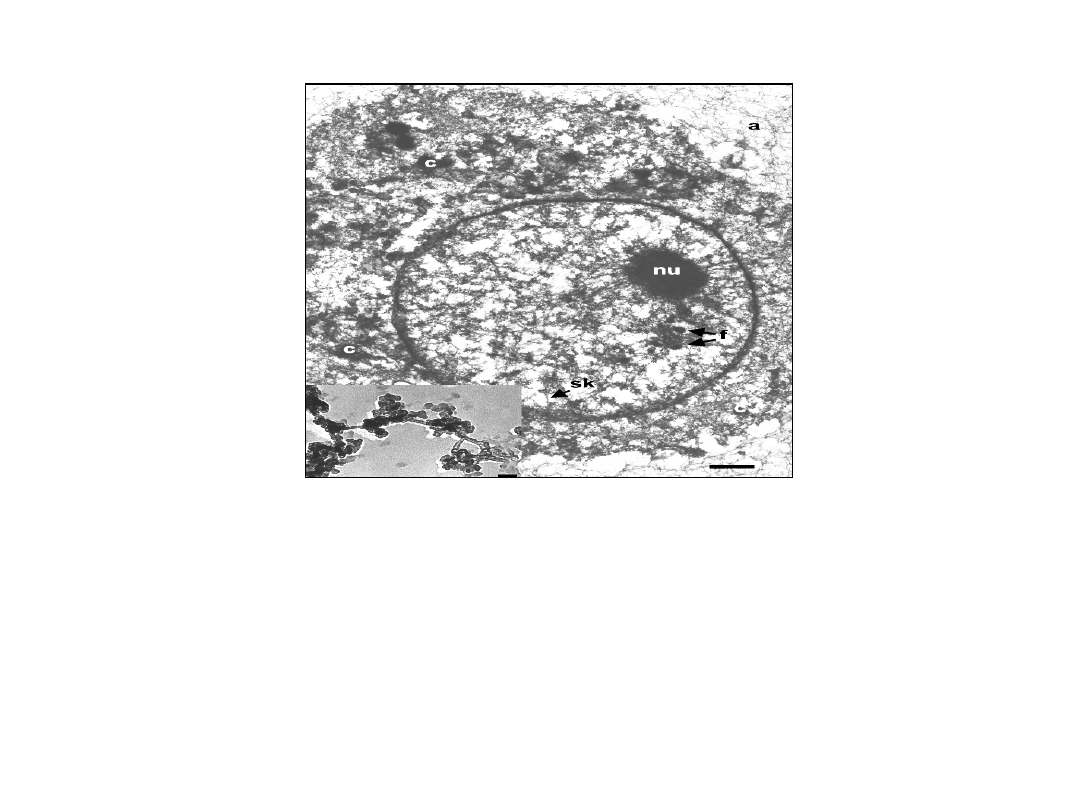
Struktura jąderka
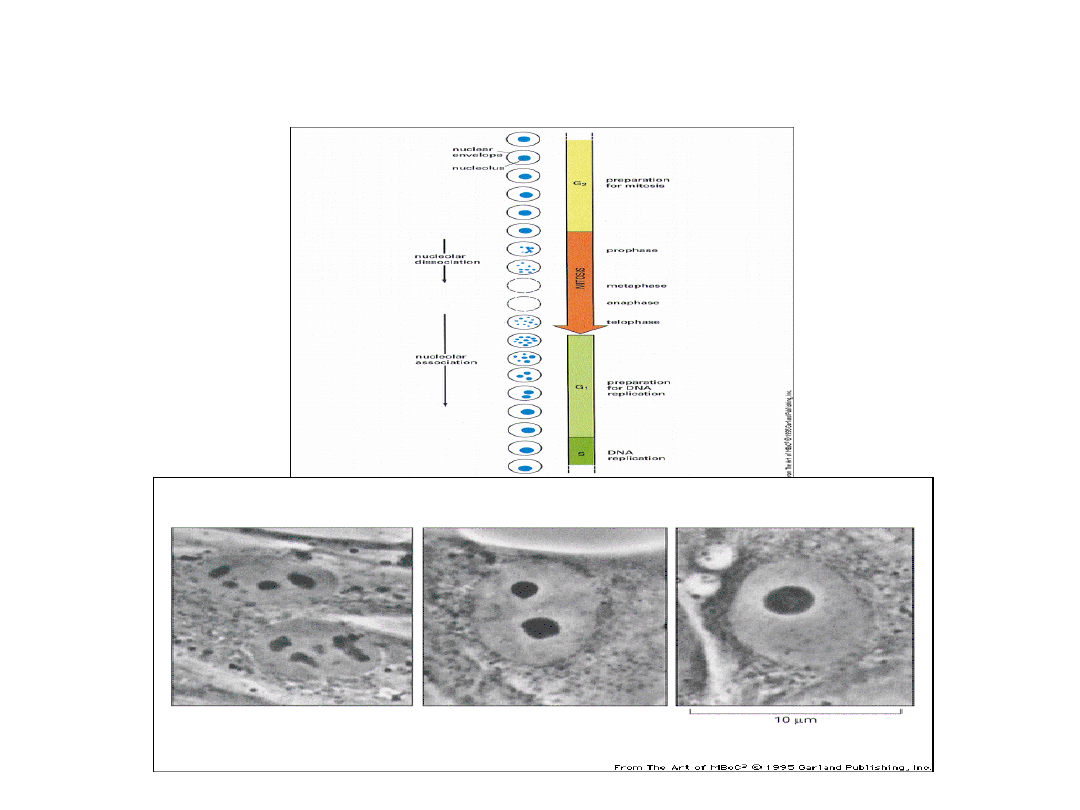
Struktura jąderka zmienia się w czasie
cyklu komórkowego
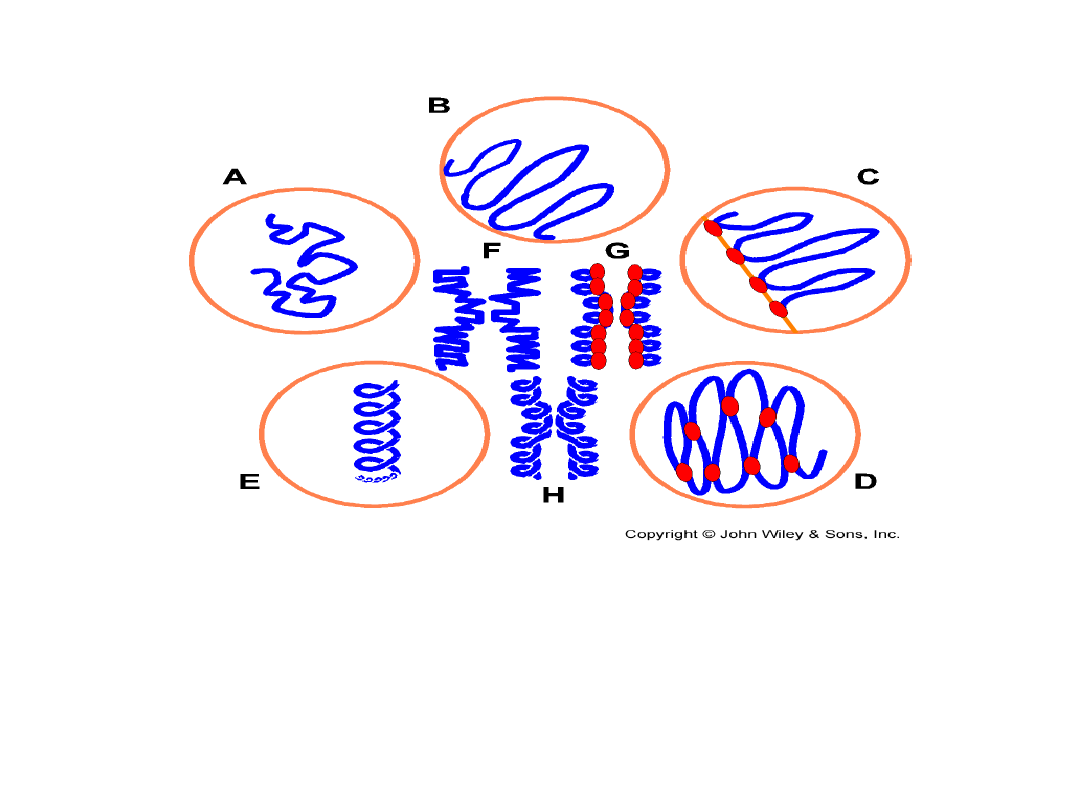
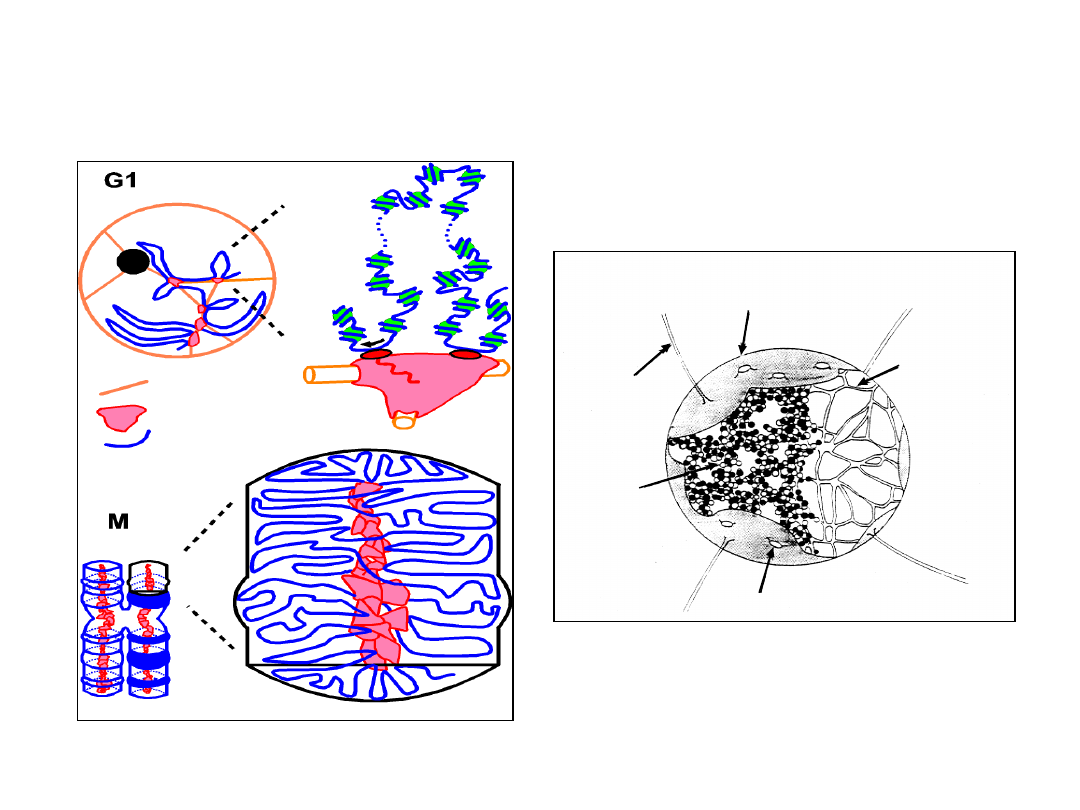
Modele struktury chromosomu
A
pętla chromatyny
B
pętla chromatyny związana
z siatką lamin
C
pętla chromatyny związana
ze szkieletem jądrowym
D
pętla chromatyny związana
z białkami
E-H
tworzenie struktury chromosomu mitotycznego

- tzw. spagetti model
- struktura oparta na macierzy (szkielecie) jądrowej
- nowy model oparty na teorii koloidów
Wewnątrz jądra jest bardzo wysokie stężenie makrocząsteczek
100-200 mg/ml (najwyższe około 1000 mg/ml)
W tych warunkach aktywność chemiczna jest bardzo wysoka
np.
[A]+[B] [AB]
Faworyzowane jest tworzenie kompleksu białko-białko
(a nie tylko równowaga między wolnymi i związanymi cząsteczkami)
Niektóre struktury w jądrze przypominają właściwościami ciekłe kryształy
np. jąderko, ciałka PML itp.
Tworzenie struktury chromosomu może być faworyzowane w wyniku
efektu ‘załoczenia’ cząsteczek i występowania efektu wypychania (crowding efect)
Problem wolnych (niezwiązanych jonów i wody)
Modele struktury jądra komórkowego
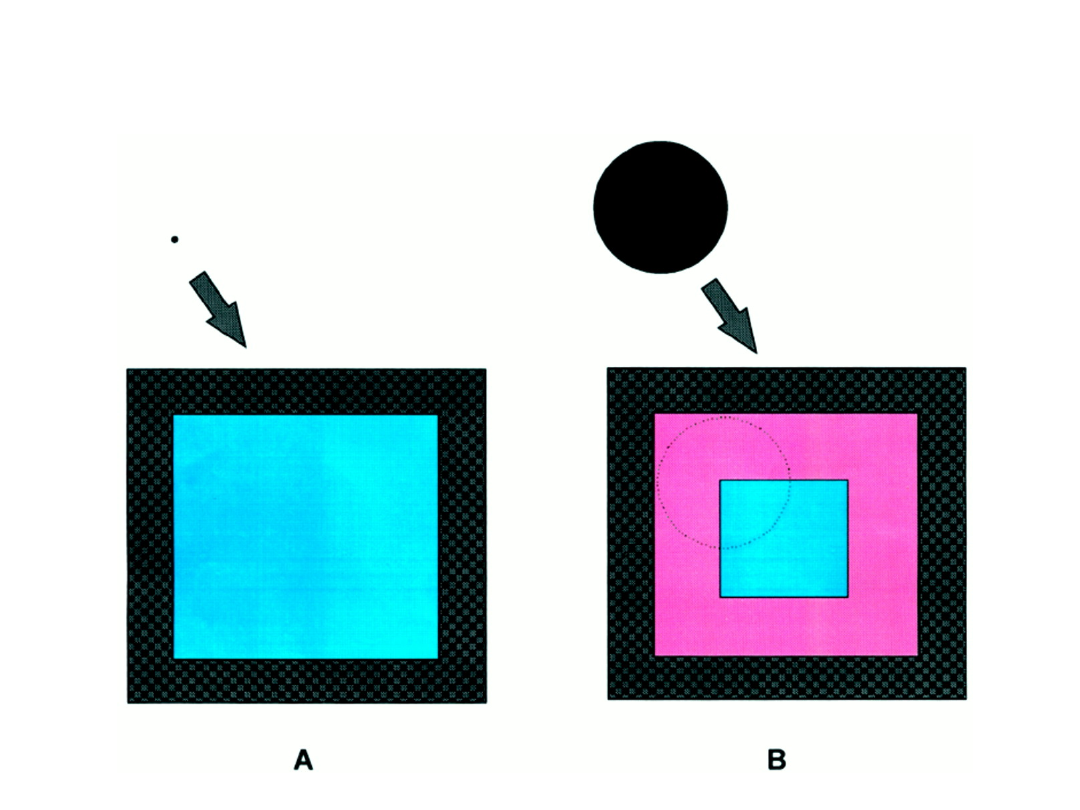
Objętość wykluczona (różowy) i dostępna (niebieski)
w kwadratowej ‘klatce’ dla cząsteczek o różnej wielkości
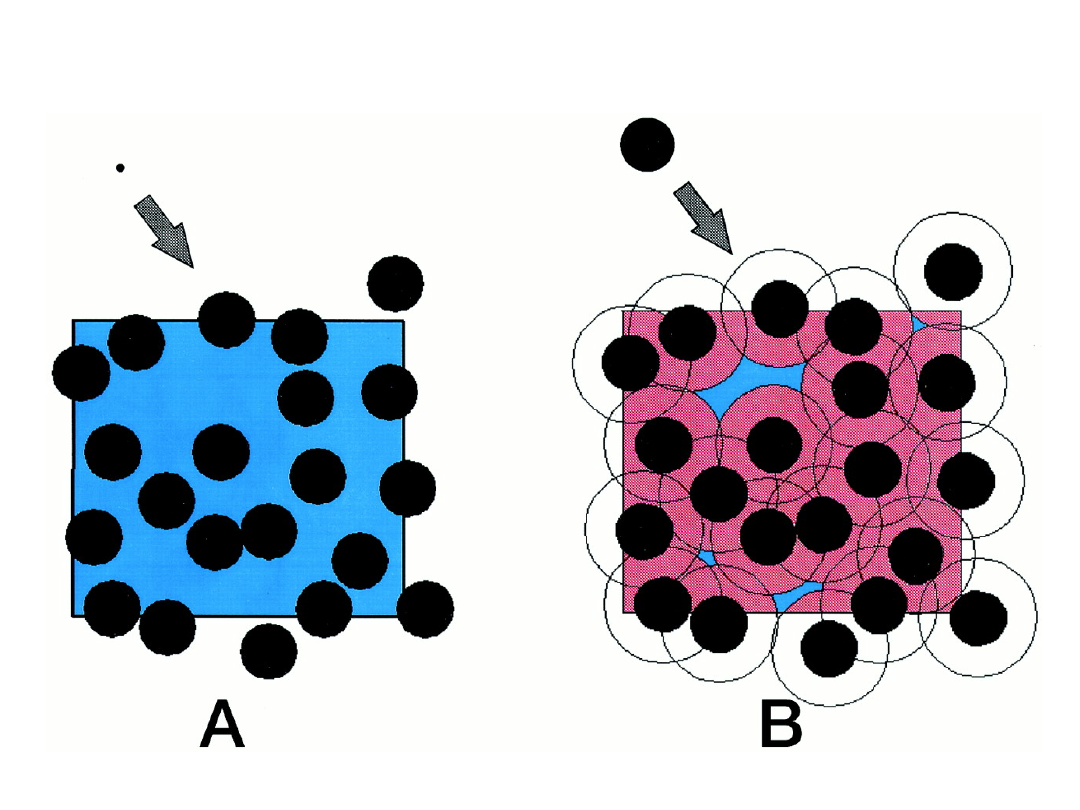
Objętość wykluczona (różowy i czarny) oraz dostępna
(niebieski)
dla sferycznych cząsteczek o różnej wielkości
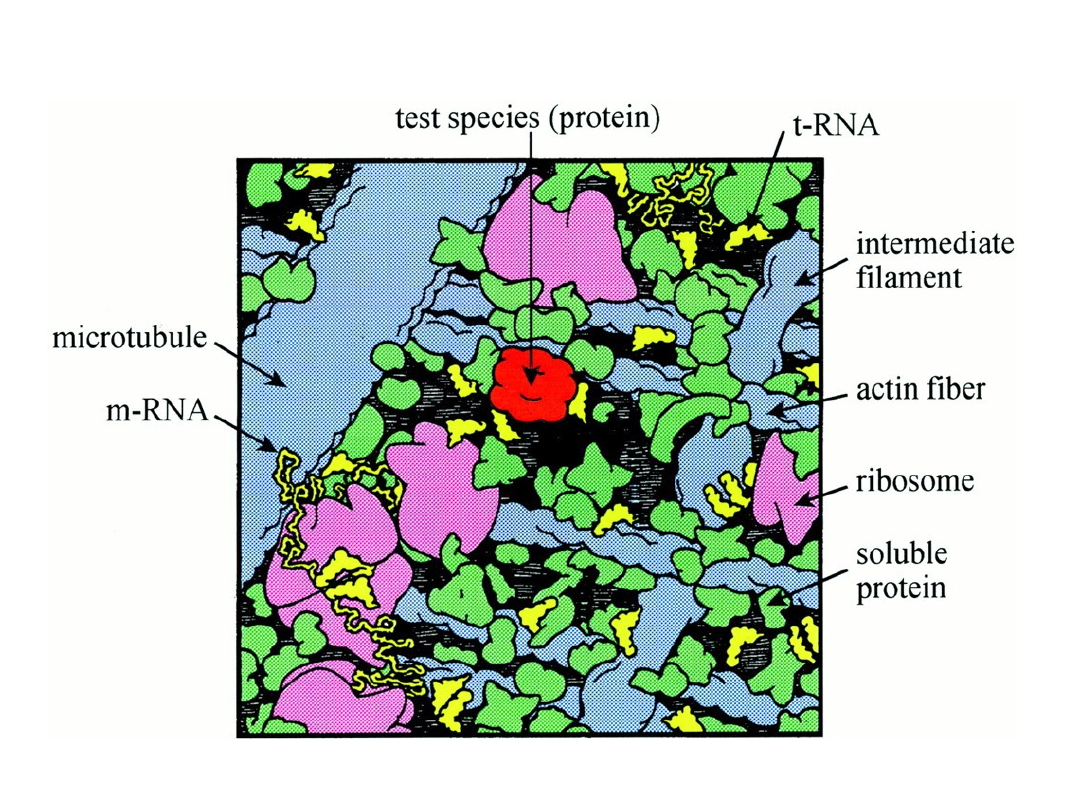
Model cytoplazmy pod powiększeniem ok.
1,000,000 razy
Minton A P J. Biol. Chem. 2001;276:10577-10580
Odkryta i opatentowana w 1948
I.B. Zbarskii i współpracownicy
Instytut Biologii Rozwoju, Moskwa
„residual nuclear protein fraction”
Powtórnie odkryta w 1974
R. Berezney i D.S. Coffey
„the nuclear matrix”
Macierz jądrowa

trawienie niespecyficznymi nukleazami np. DNAza I (i RNAza A)
ekstrakcja buforem z niejonowymi detergentami
ekstrakcja roztworami chlorku sodu 0.15-2 M
ekstrakcja roztworem 0.25 M siarczanu amonu
izolacja jąder komórkowych
komórka
białka
73-98%
(10-12% ogólnej zawartości białek jądrowych)
DNA
< 1%
(1-3% DNA)
RNA
0.05-1.2%
(20-30% jądrowego RNA)
fosfolipidy
0.5-7%
(2-9% fosfolipidów frakcji jądrowej)
skład macierzy bardzo zależy od sposobu jej otrzymywania !
etap tzw „stabilizacji”- jony Cu
2+
, ogrzewanie, traktowanie czynnikami stabilizującymi
mostki dwusiarczkowe
ekstrakcja dijodosalicylanem litu 25 mM (detergent)
Izolacja macierzy jądrowej
Skład macierzy jądrowej

laminy A-C
NuMA, dezmina
DNA topoizomeraza II
HMG1,2
SATB1
nukleolina
mt p53
Rb
kinaza kazeinowa 2
Białka niehistonowe, wykazujące wysokie powinowactwo do DNA:
białka blaszki jądrowej
resztkowe struktury jąderka
sieci wewnątrzjądrowej
rusztowania chromosomów (chromosome scaffold)
białka cytoszkieletu np. aktyna
Białka macierzy jądrowej
Zawartość zależy od sposobu izolacji macierzy:
hnRNA
snRNA (U1-U6 snRNA)
ślady rRNA
ok. 25% jądrowego RNA związane jest w macierzy jądrowej
RNA w macierzy jądrowej
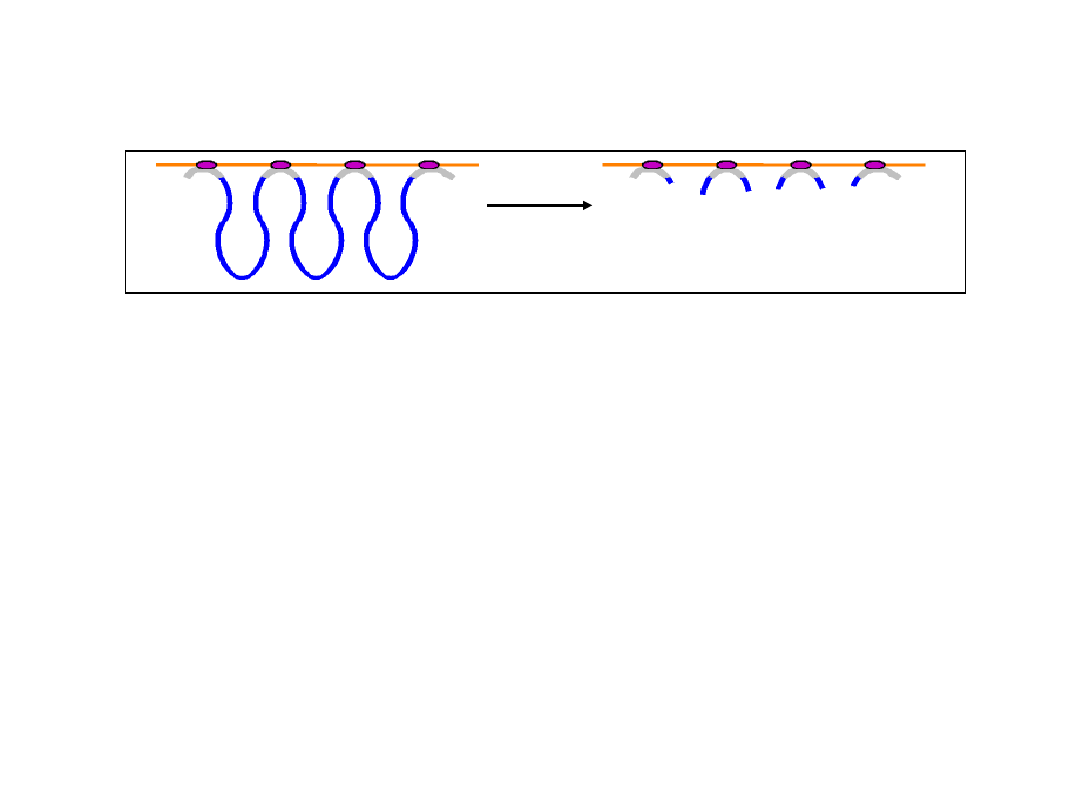
ang. MAR, matrix associated regions, SAR, scaffold-associated regions
300-1000 pz, bogate w pary AT (średnio 70%)
nie ma sekwencji consensus, konstytutywne i funkcjonalne
przykład:
mozaika motywów TAAT, ATTA, TAAAT, ATTTTA i TAAAAT
również TGTTTTG, TTTTGGGG
polimorficzne: palindromiczne, Z-DNA
cechy strukturalne MAR:
zwiększona podatność na zaginanie DNA
zdolność do przyjmowania struktury
jednoniciowej (otwieranie struktury)
DNAza I
Rejony MAR w macierzy jądrowej
fabryka/chromomer
chromatyna/DNA
szkielet jądrowy
filamenty
pośrednie
pora jądrowa
filament
podstawowy
macierz
jądrowa
siatka lamin
Struktura chromosomu i jądra komórkowego
a macierz jądrowa

regulacja rozmieszczenia DNA w jądrze
porządkowanie struktury jądra i procesów w nim zachodzących
tworzenie continuum mechanicznego
z cytoszkieletem
Fukcjonalne znaczenie MAR
znajdują się w pobliżu lub zawierają rejony regulatorowe DNA
np. początki replikacji
rejony MAR mogą zwiększać ponad 1000-krotnie ekspresję obcych genów
w komórkach transfekowanych
(zniesienie efektu pozycyjnego?)
macierz
pętla
nić 30 nm
Funkcje macierzy jądrowej
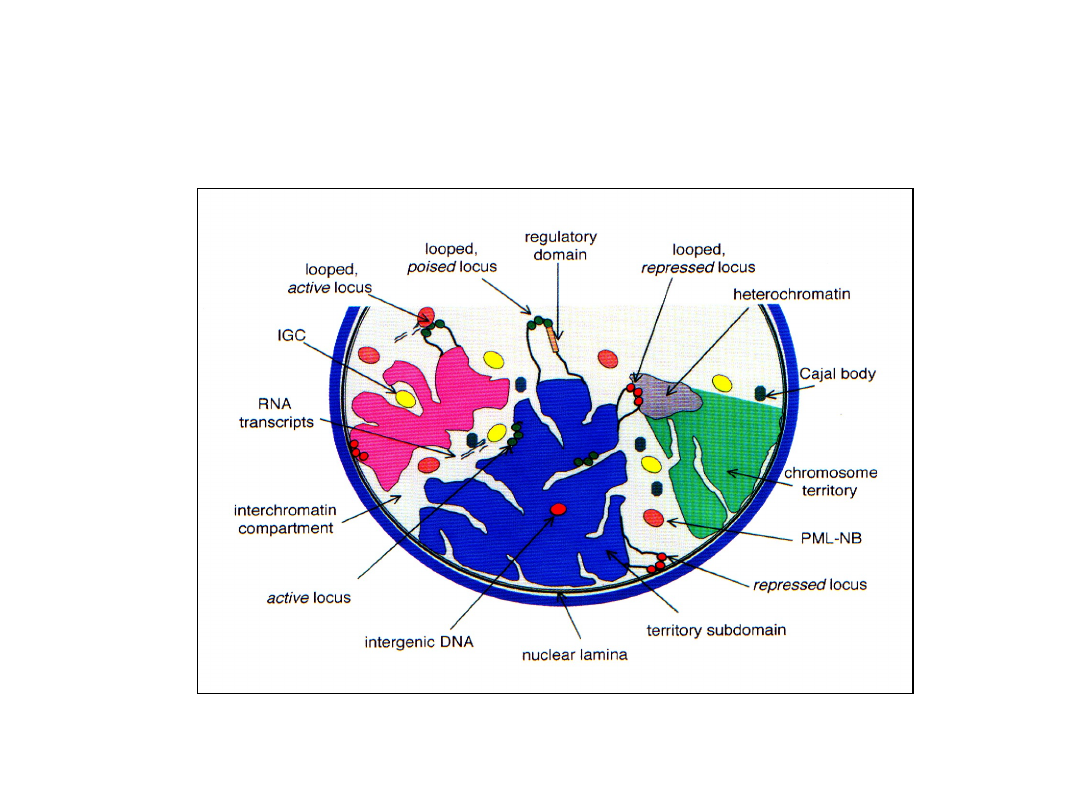
Model struktury jądra
umiejscowiony czy rozproszony?
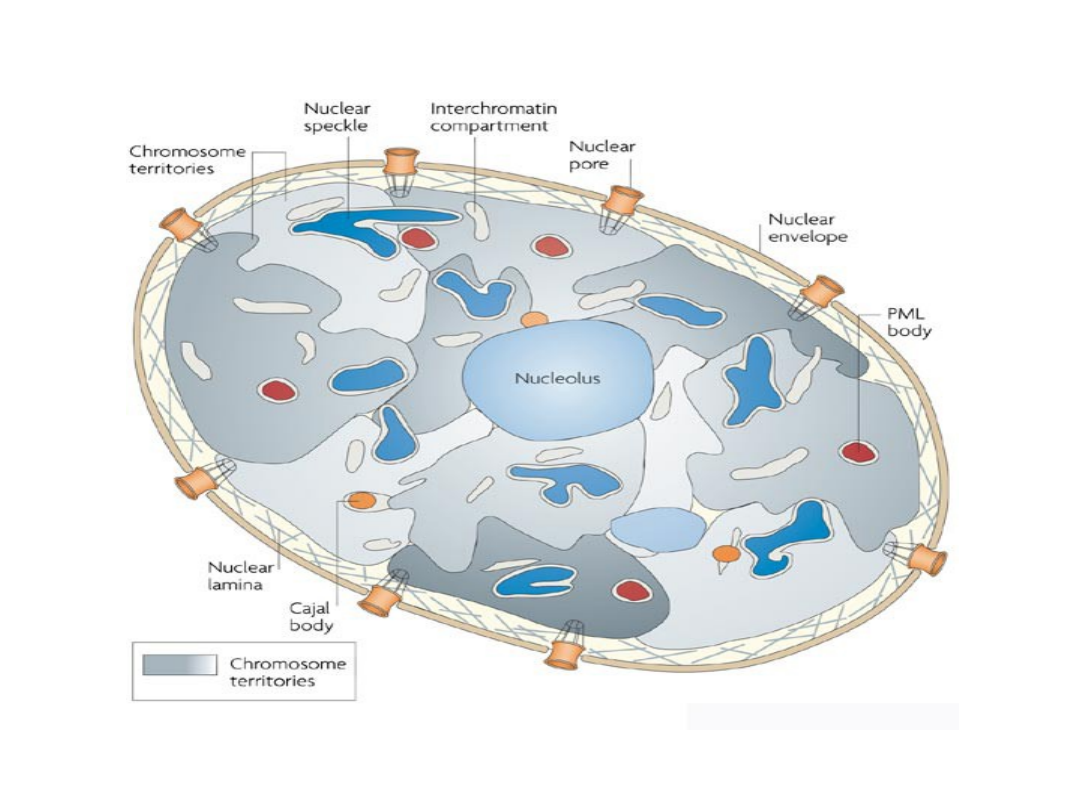
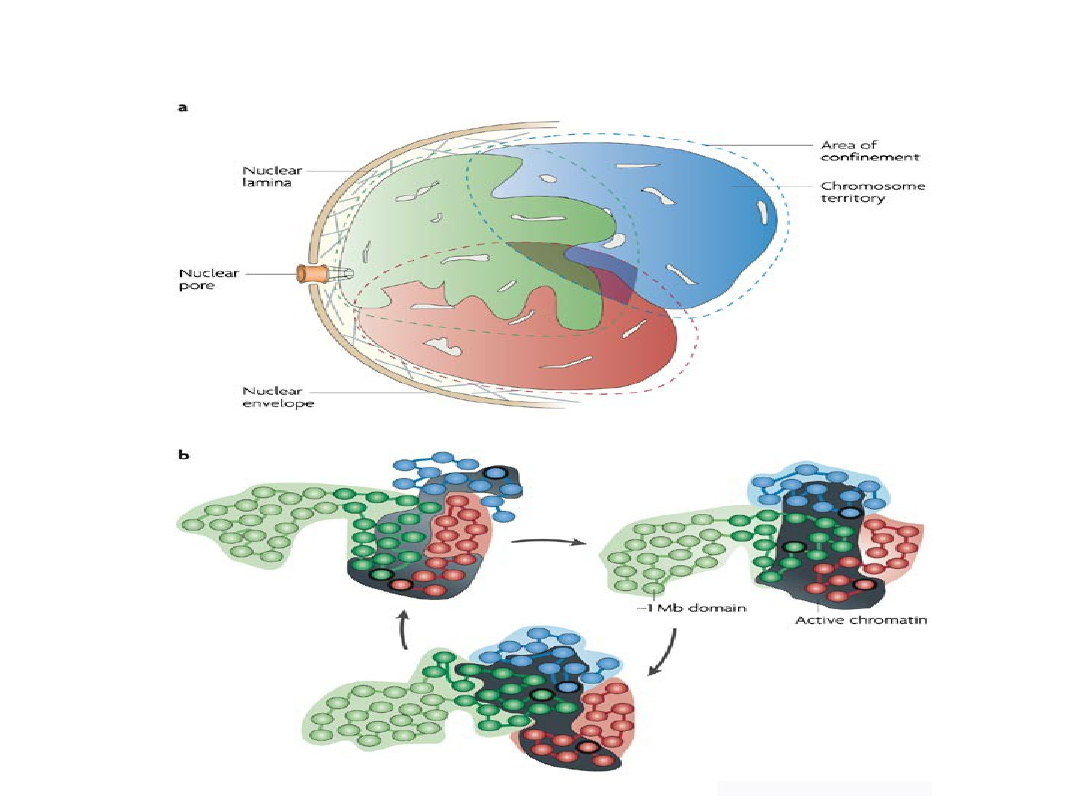
Model umiejscowiony struktury jądra wskazuje na
możliwość zachodzenia/regulacji procesów jądrowych
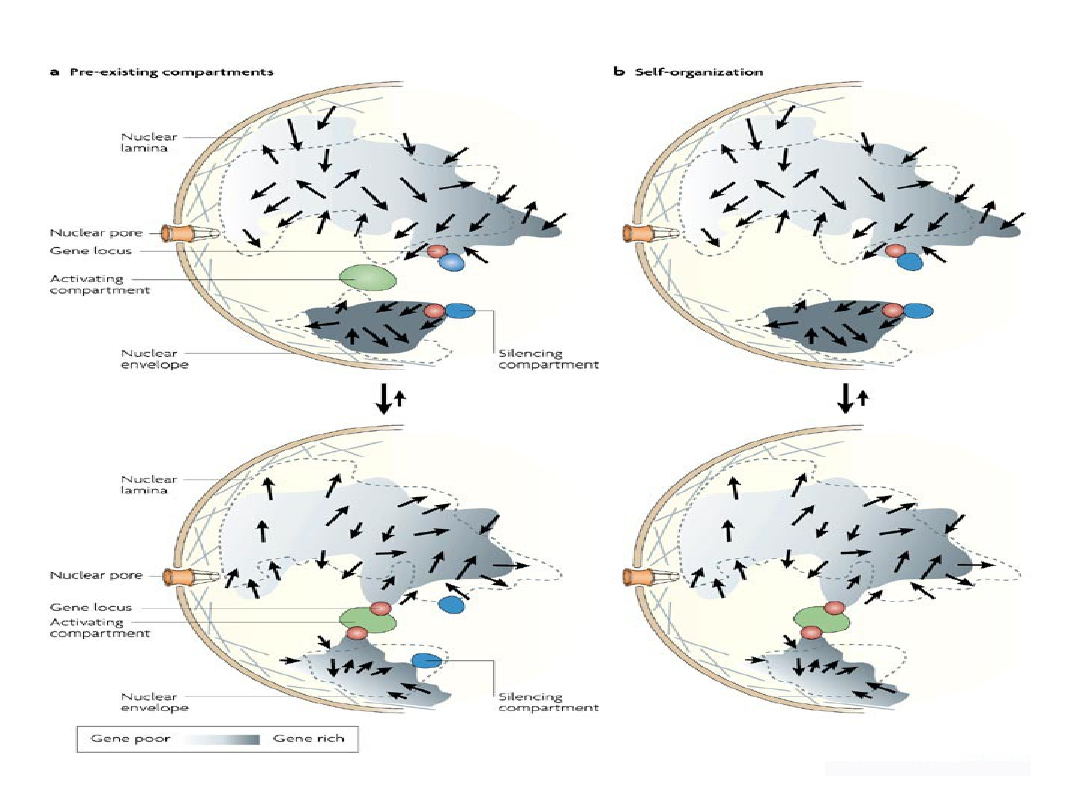

Główne różnice pomiędzy organizacją genomu
u Prokaryota i Eukaryota
prokaryota
eukaryota
wielkość genomu
600 kb - 9.5 Mb
3 Mb - 140 000 Mb
średnia wielkość
/ilość genu(ów)
950 bp/4300
2500 bp/19 000
horyzontalne
przenoszenie genów
częste
b. rzadkie
częstość sekwencji
niekodujących
rzadkie
b. częste
występowanie
intronów
rzadkie
powszechne
występowanie
powtórzeń genów
rzadkie
powszechne
haploidia
haploidia
/poliploidia
ploidalność
liczba
chromosomów
1
>1
heterozygotyczność
nie
tak

Strukturalne uwarunkowania procesów
zachodzących w jądrze komórkowym
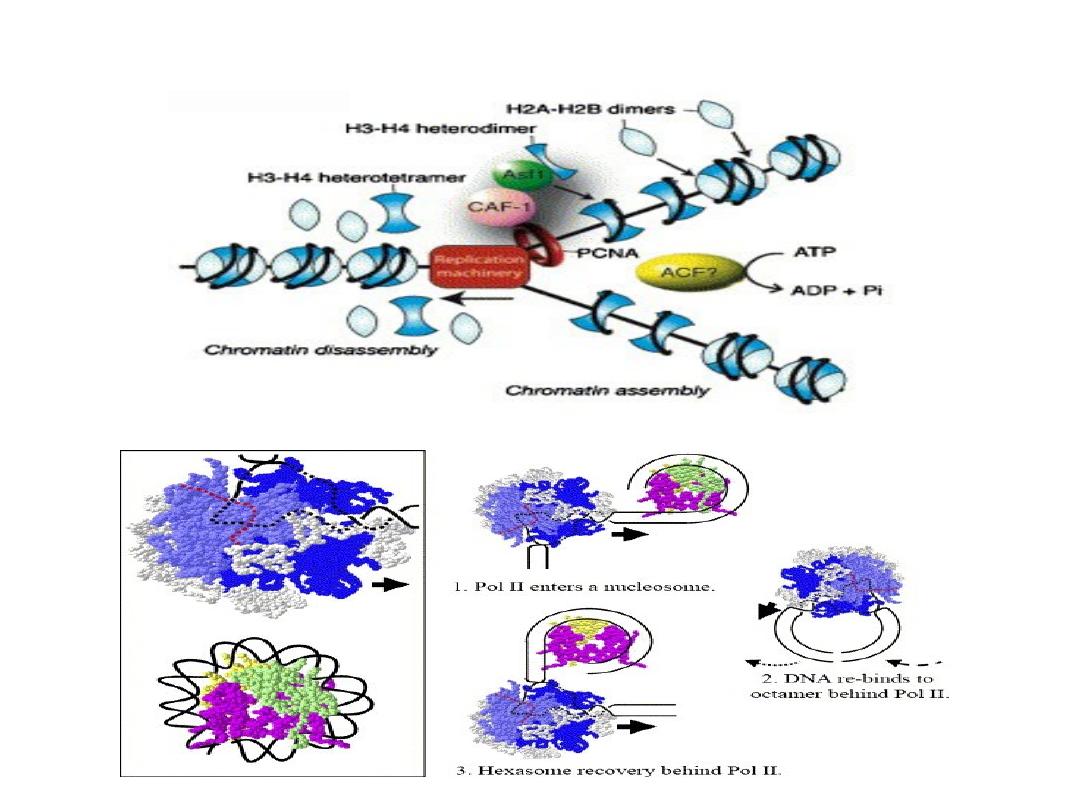
W czasie transkrypcji i replikacji DNA
struktura chromatyny musi się zmieniać
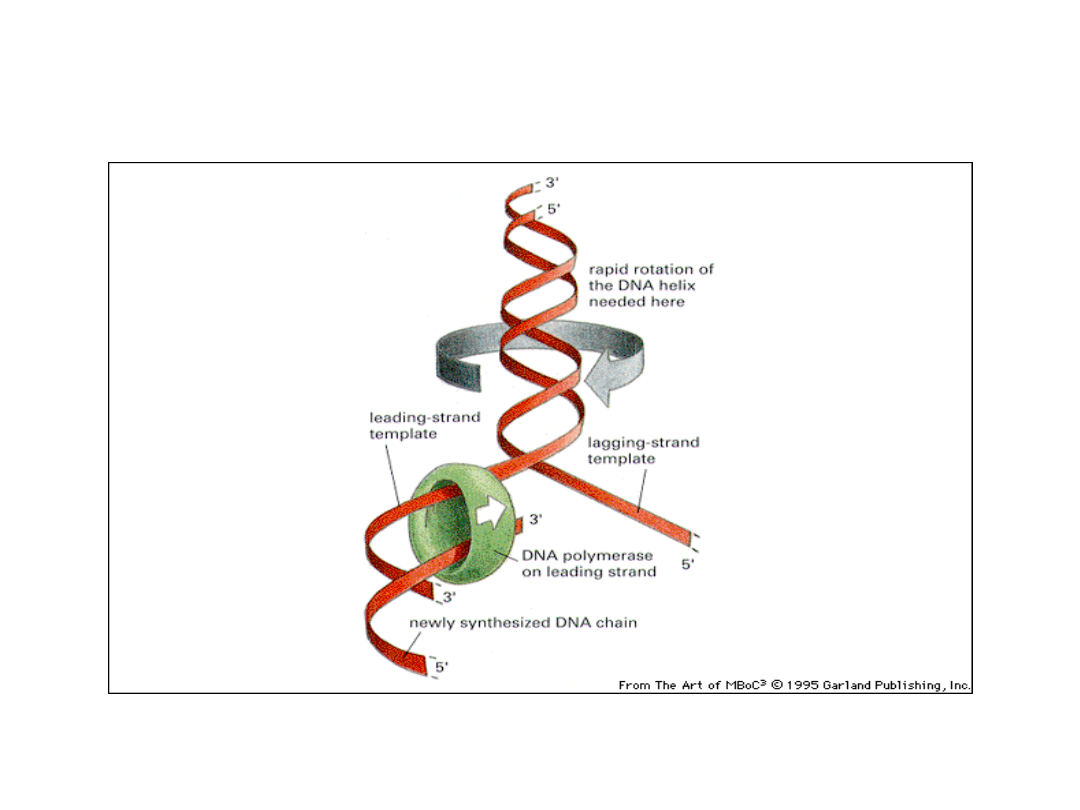
W czasie replikacji DNA powinno się obracać
aby znieść wzrastającą ilość nadskręceń
topologia DNA regulowana jest przez enzymy – DNA topoizomerazy
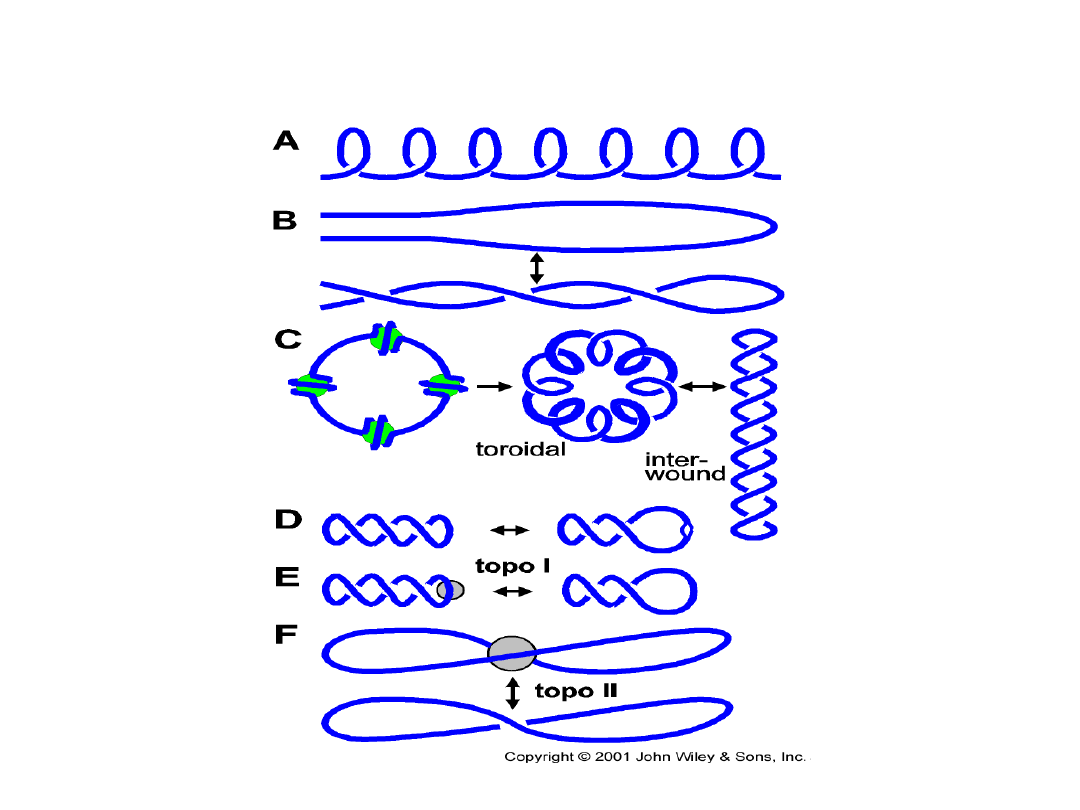
Problemy topologiczne w DNA
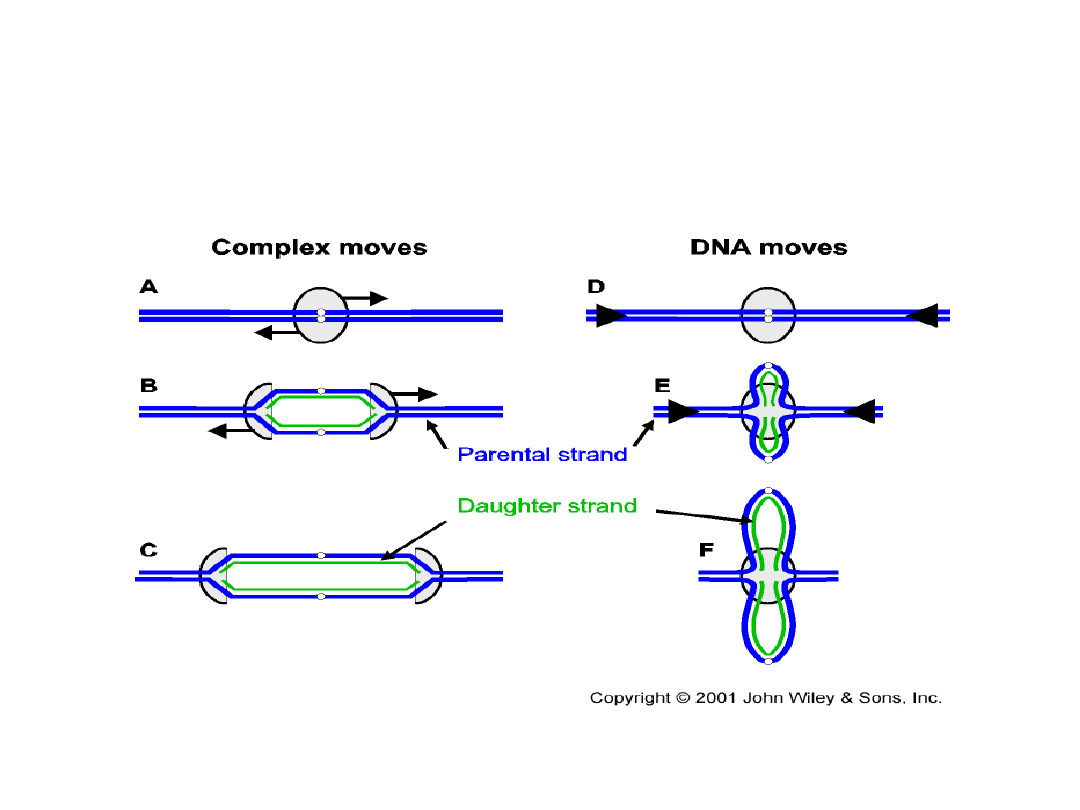
Co porusza się w czasie replikacji :
kompleks polimerazy DNA czy samo DNA ?
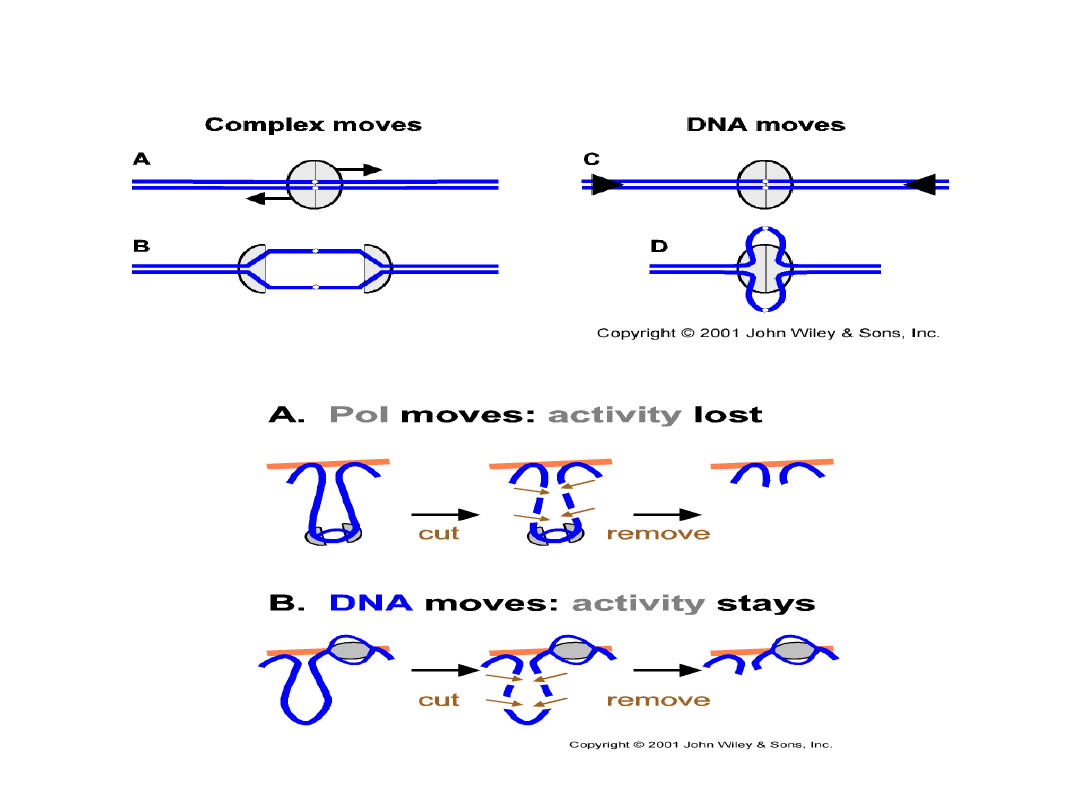
dowód 1: system replikacyjny wirusa SV40 (antygen T)
dowód 2:
Istnienie kompleksów replikacyjnych
wykazano również w komórkach
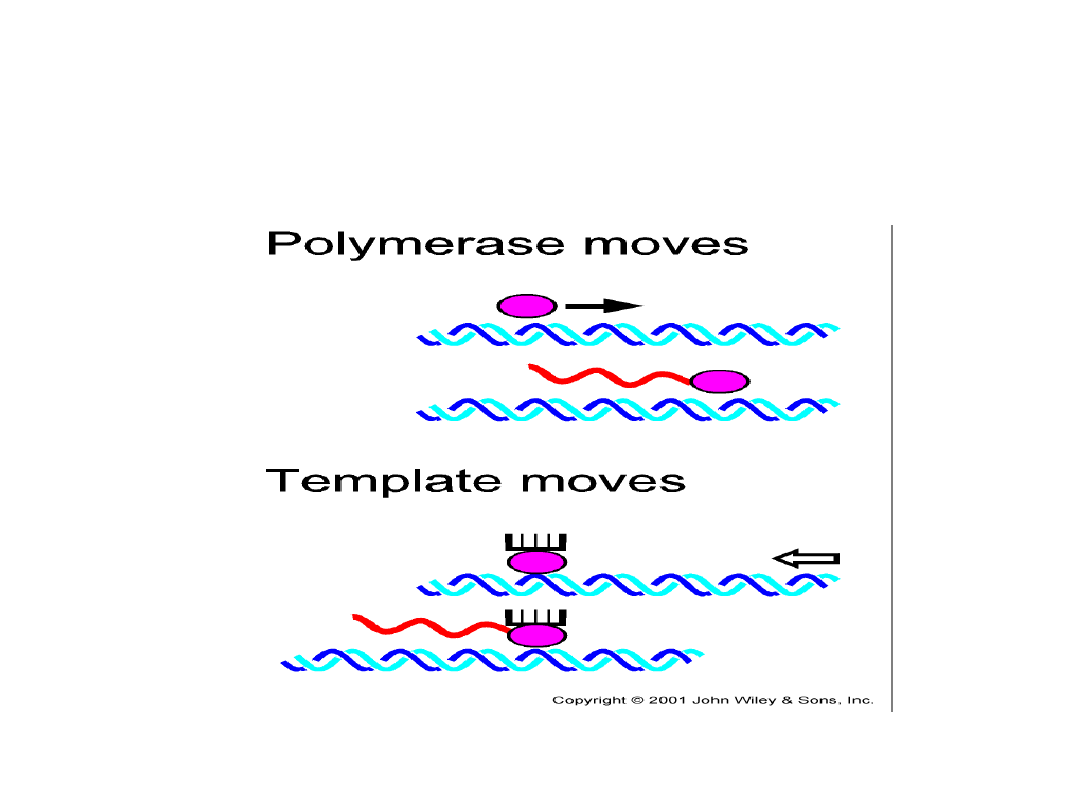
Podobne pytania stawiano
nt. kompleksu transkrypcyjnego
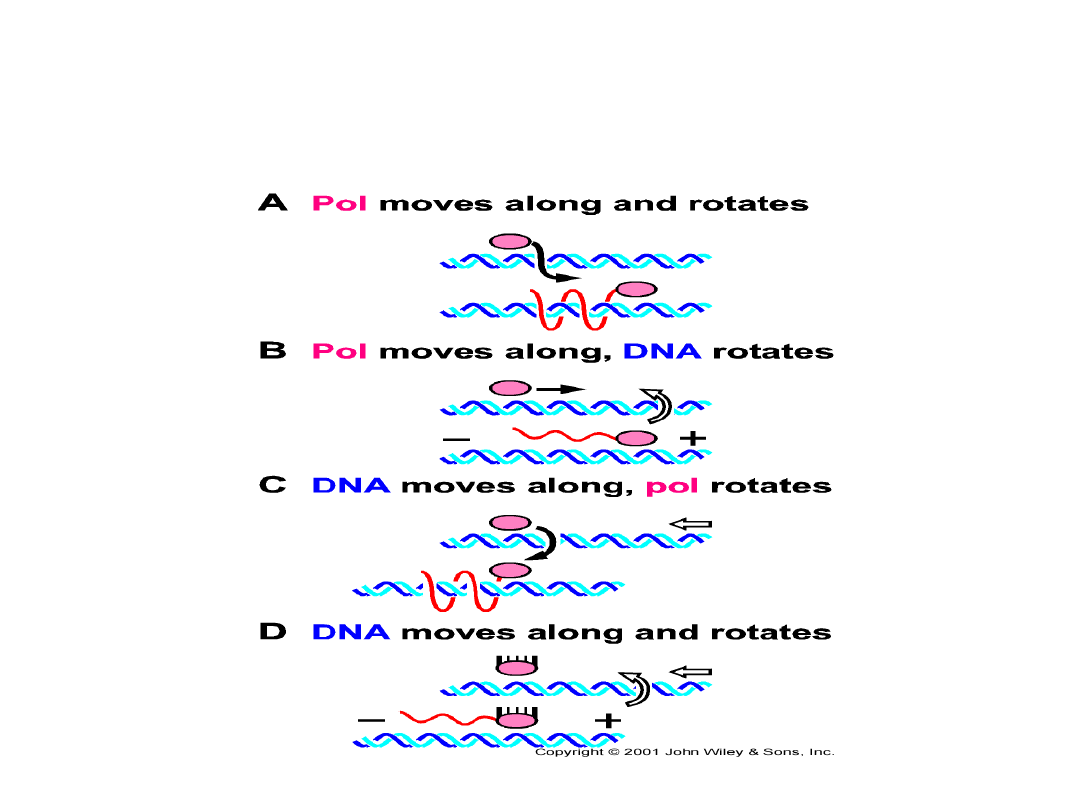
Podczas procesu transkrypcji polimeraza RNA
i/lub matryca DNA muszą wykonywać
bardziej skomplikowane ruchy względem siebie
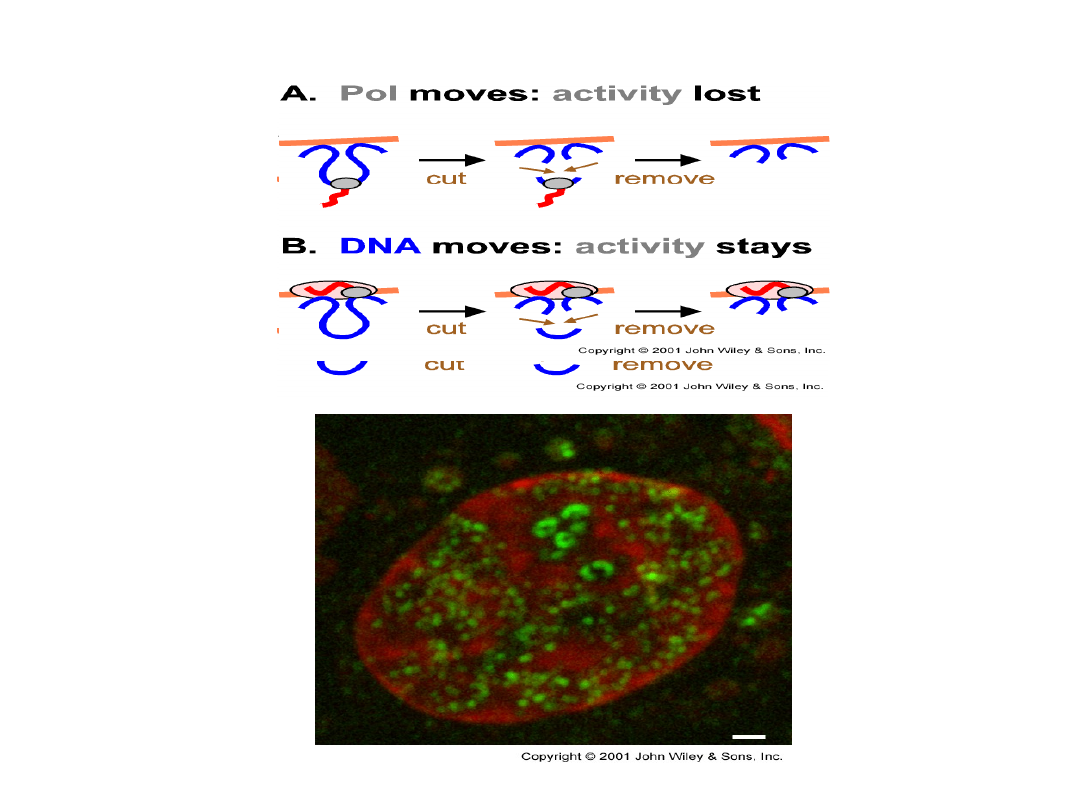
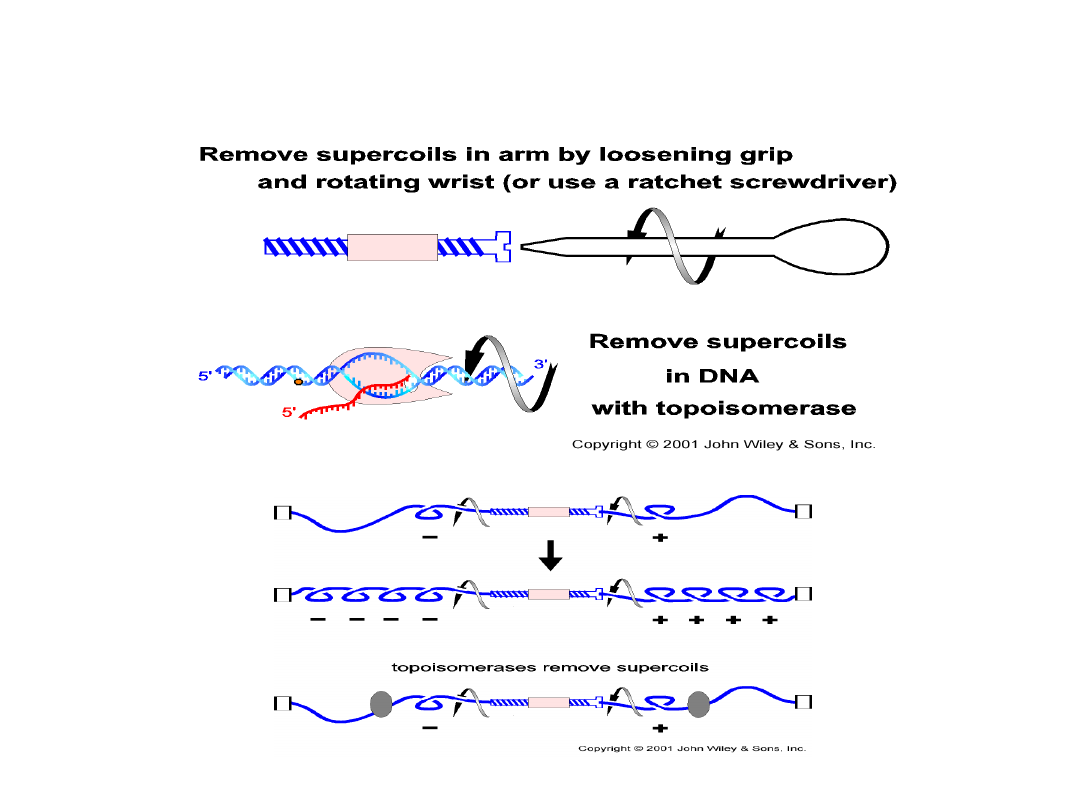
Istnieje analogia w ruchu kompleksu transkrypcyjnego
i układu śruba-nakrętka
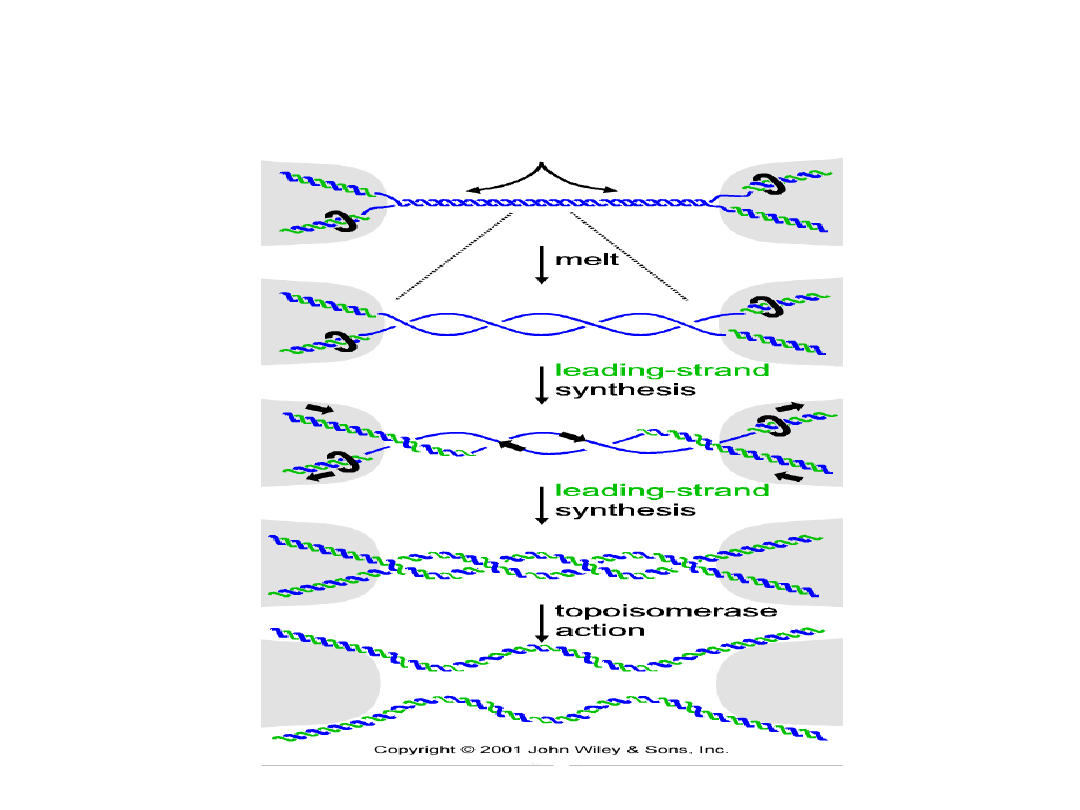
Pozostaje jeszcze problem replikacji miejsc
gdzie spotykają się kompleksy replikacyjne

Problem replikacji końców liniowych cząsteczek DNA
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
Wyszukiwarka
Podobne podstrony:
W4 Struktura jadra
W3 Struktura jadra (asus Komputer's conflicted copy 2012 05 23)
w4 progr strukt
52+55 struktura nablonka plemnikotworczego cewek nasiennych jadra
6 W4 prorozwojowe struktury spoleczne
W4 Proces wytwórczy oprogramowania
STRUKTURA TRENINGU
W4 2010
30 Struktury zaleznosci miedzy wskaznikami zrow rozw K Chmura
Statystyka SUM w4
w4 3
rodzaje struktur rynkowych 2
Struktura regionalna
więcej podobnych podstron