
Replikacja, naprawa i
rekombinacja DNA u
eukariontów
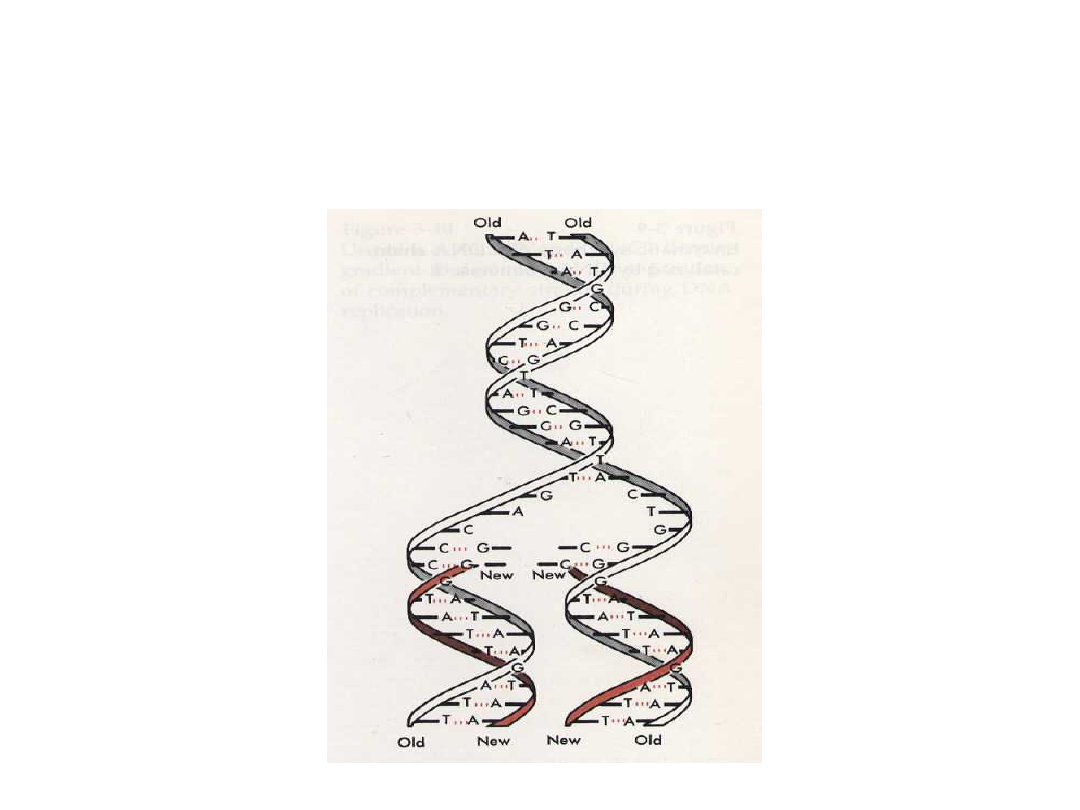
Zasada replikacji DNA
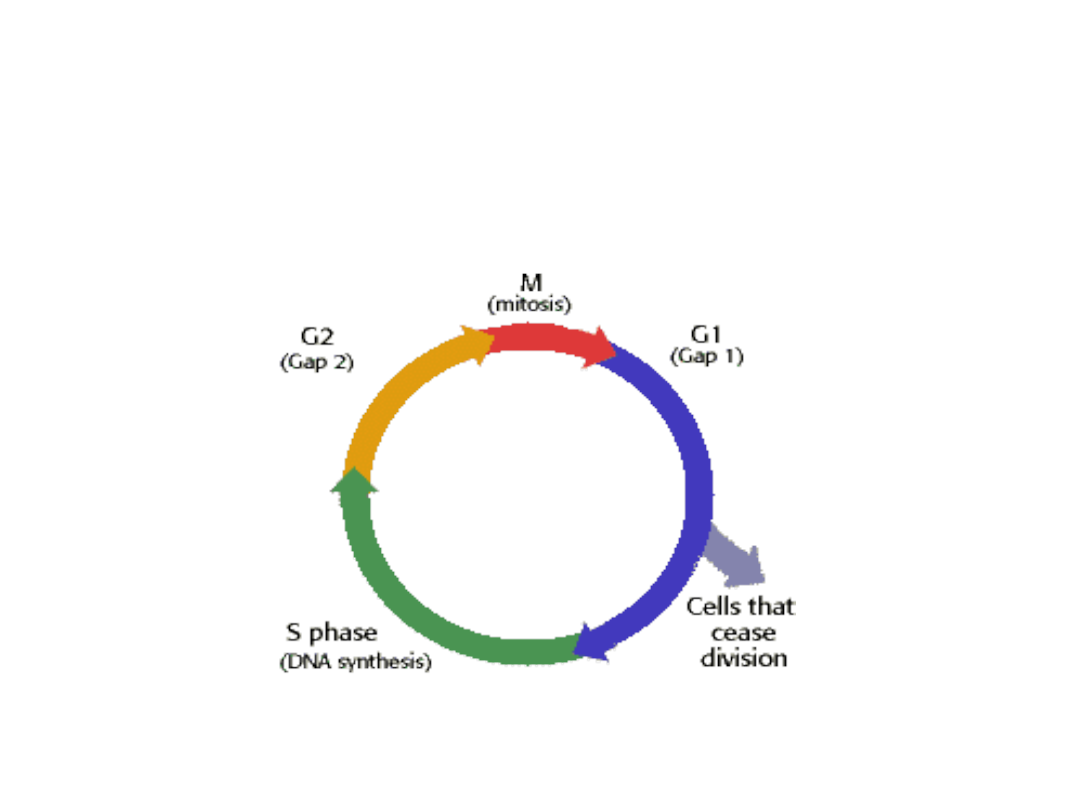
Replikacja DNA jest elementem cyklu komórkowego
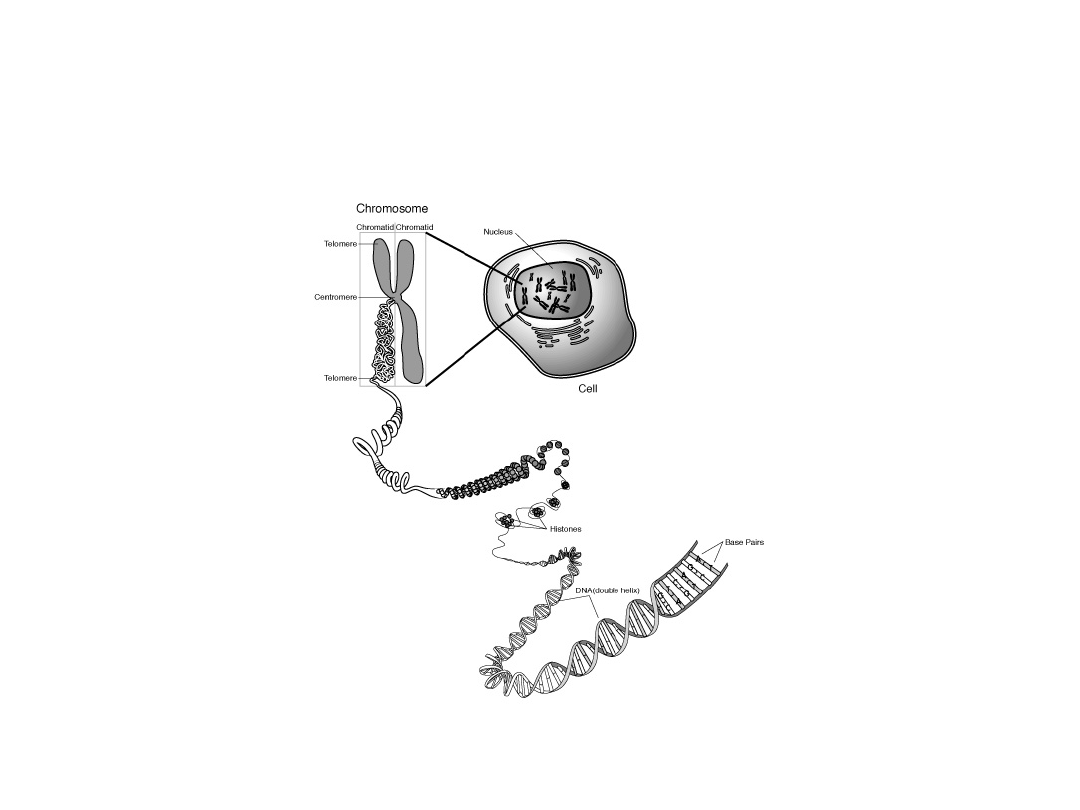
U eukariontów DNA występuje w kompleksie
zwanym chromatyną

Replikacja u eukariontów
• Inicjacja, elongacja, terminacja
• Problem końców chromosomów
• Jądro - organelle
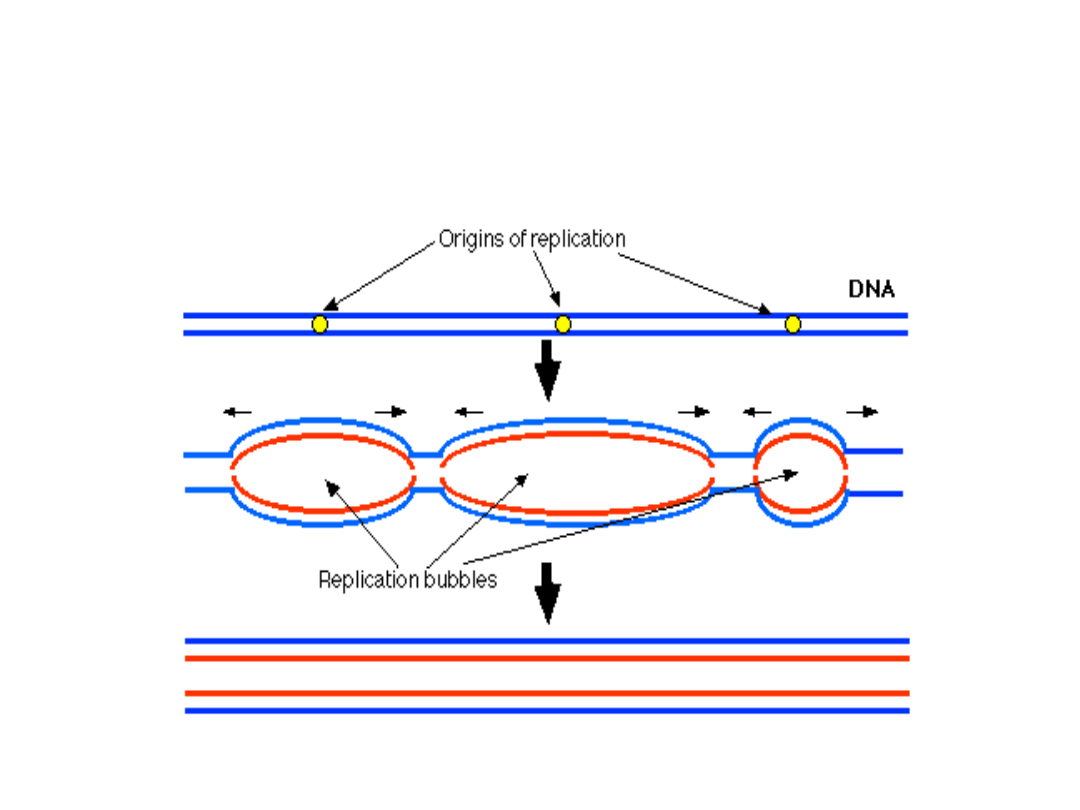
Inicjacja
(replikacja zaczyna się jednocześnie w wielu
miejscach)

ORI (Origins of replication)
• ARS (Autonomously Replicating Sequence) u drożdży
- Specyficzna sekwencja 200 pz jest minimalną sekwencja
wymaganą do inicjacji replikacji chromosomowego DNA.
U ssaków inicjacja (ORI) obejmuje sekwencję 10 000 pz
U roślin sekwencje ORI nie są zidentyfikowane
W chromosomach istnieje wiele potencjalnych ORI, ale nie
wszystkie funkcjonują w każdej komórce
Fragment DNA replikowany z jednego ORI nosi nazwę replikonu (
u roślin długość replikonu to przeciętnie 50-70 kb)
Nie wszystkie ORI startują w tym samym momencie, jednak
porządek ich uruchamiania jest w komórkach ściśle
kontrolowany i zależy od stanu kondensacji chromatyny w
danym miejscu.

Kontrola inicjacji
• Licencjonowanie (kontrola pozytywna) –
zapewnia, że
chromosomy będą się replikować tylko wtedy, gdy w sposób
prawidłowy przejdą przez mitozę i znajda się w komórce
potomnej
.
• Aby nastąpiła inicjacja, do ORI musi się przyłączyć
Kompleks Rozpoznający Origin (ORC – Origin Recognition
Complex) i dodatkowe białka (czynniki licencjonujące Cdc-6
i Cdt-1) umożliwiające ścisłe pokrycie sąsiadującego DNA
białkami MCM (Minichromosome Maintenance). Tylko DNA
pokryty białkami MCM może być replikowany. Białka MCM
są usuwane przez przesuwające się widełki replikacyjne.
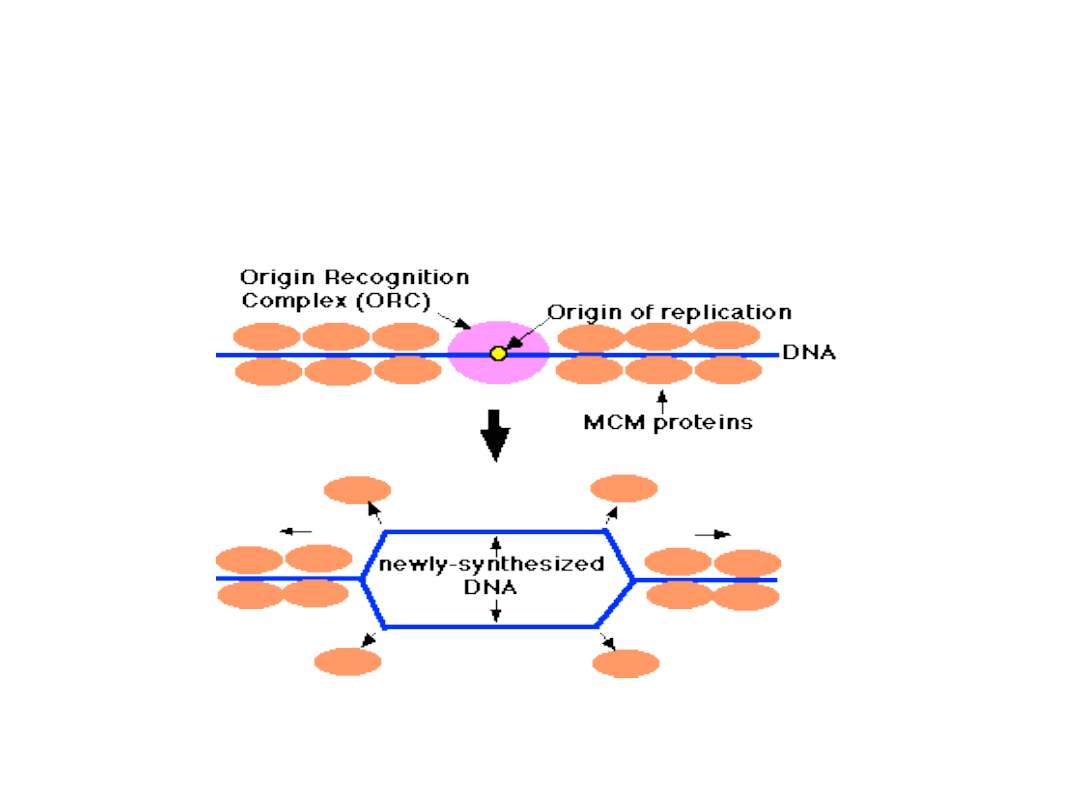
Licencjonowanie w ORI

Negatywna kontrola inicjacji - Geminina
• Geminina – białko występujące w komórkach w
fazie G2
• Przeciwdziała przyłączaniu się białek MCM do
świeżo zreplikowanego DNA (zablokowanie
czynnika Cdt-1)
• Jest degradowana po zakończeniu mitozy
• Gemininy nie wykryto w drożdżach i roślinach!
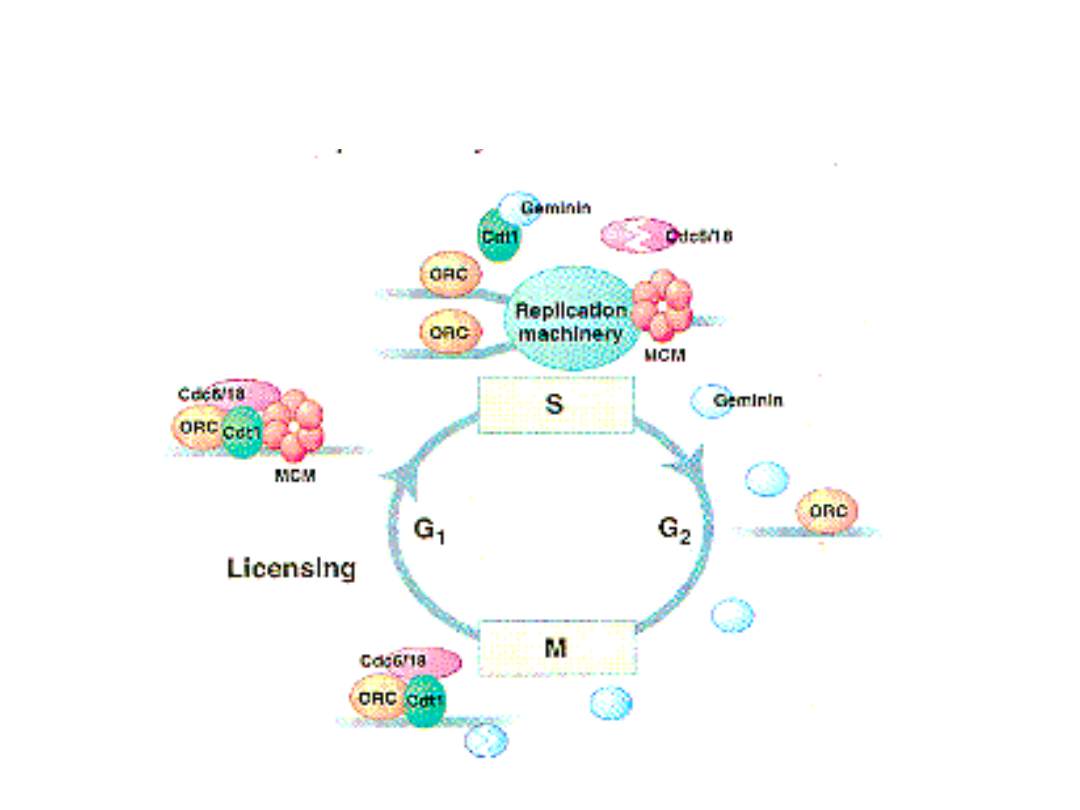
Kontrola inicjacji w cyklu komórkowym

Elongacja
• Kompleks wielo enzymatyczny zawierający polimerazę DNA
katalizuje przyłączanie deoksyrybonukleotydów do 3’ końca
DNA lub RNA przyłączonego do nici matrycowej DNA.
• Synteza DNA idzie w kierunku
5’ – 3
’, a matryca jest
odczytywana w kierunku
3’-5’.
• Polimeraza DNA może dodawać nukleotydy tylko do już
istniejącego fragmentu kwasu nukleinowego (primera).
• Polimeraza DNA jest nie aktywna w nieobecności primera z
wolną grupą 3’ OH, związanego poprzez wiązania wodorowe
z matrycą.
• Primery są syntetyzowane przez specyficzną polimerazę
rybonukleotydową zwaną DNA primazą. Inicjuje ona syntezę
primera rozpoczynając od rybonukleotydu purynowego.
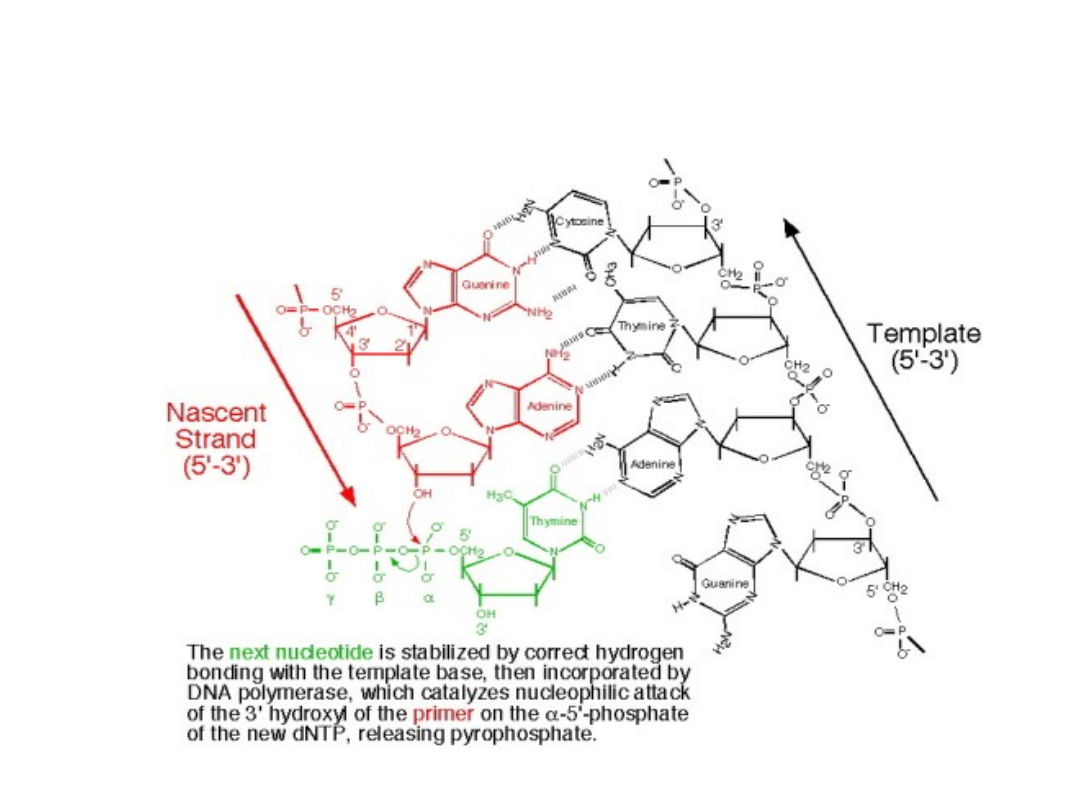
Przyłączanie deoksyrybonukleotydów przez polimerazę DNA

Elongacja - cd
• Ze względu na asymetrię widełek replikacyjnych
*(kierunki!) synteza DNA jest w połowie nieciągła. Na nici
wiodącej (jeden primer) dodawanie nukleotydów odbywa
się w sposób ciągły. Na nici opóźnionej synteza odbywa się
w formie krótkich fragmentów Okazaki, z których każdy
wymaga swojego primera.
• Wytworzenie ciągłej cząsteczki na matrycy nici opóźnionej
wymaga systemu naprawy DNA zawierającego specyficzną
rybonukleazę – RNazę H, który usuwa primery RNA i
zastępuje je fragmentami DNA. Inny enzym – ligaza DNA
łączy koniec 3’ nowego fragmentu DNA z 5’ końcem
fragmentu DNA poniżej.
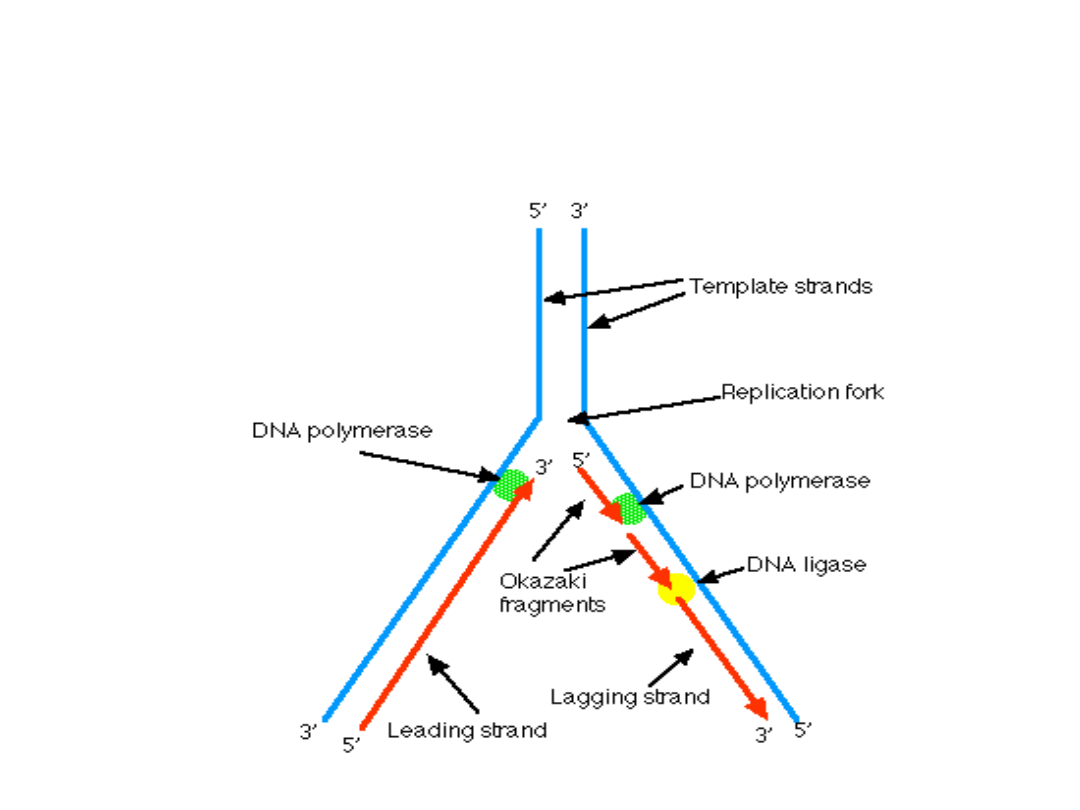
Elongacja - widełki replikacyjne

Elongacja - cd
• W jądrze eukariontów występują trzy główne polimerazy
DNA – α, δ i ε.
• α – głównie funkcja primazy
• δ i ε – synteza DNA na nici prowadzącej i opóźnionej.
• Helikaza DNA (występuje w kompleksie z pol δ i ε ) rozplata
dwuniciowy DNA u nasady widełek.
• Topoizomerazy DNA usuwają napięcia torsyjne powstające
w wyniku rozplatania (są przyłączone przed widełkami).
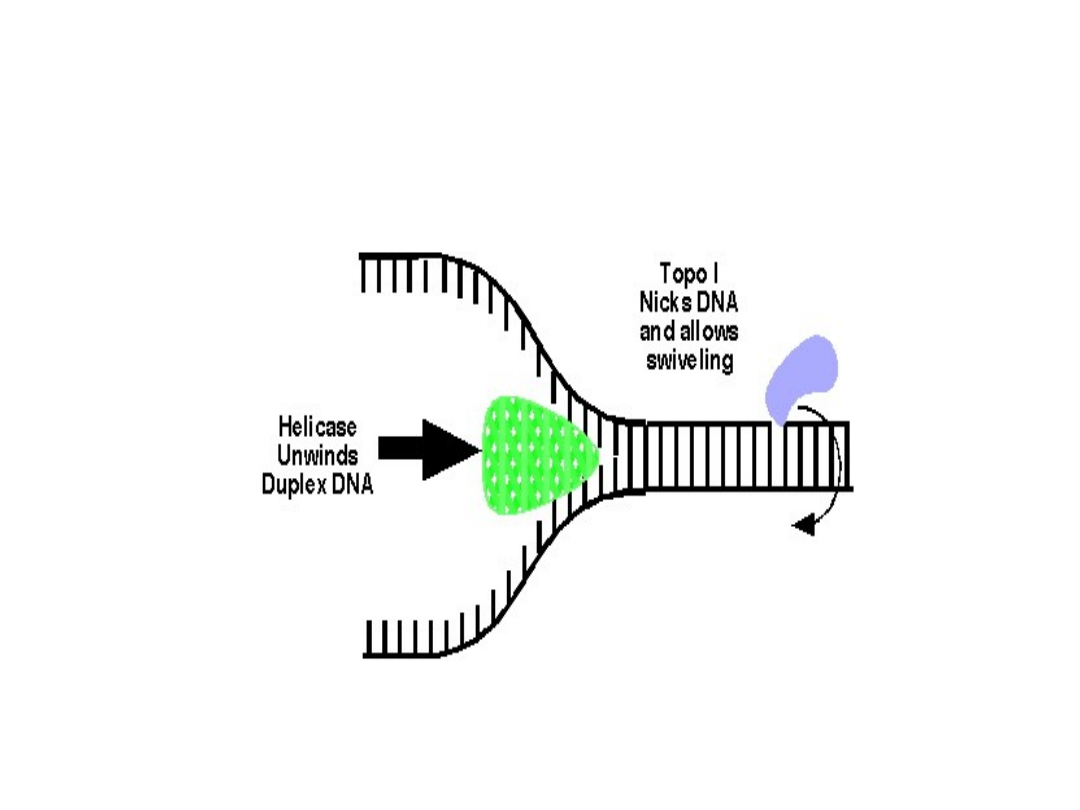
Elongacja - cd

Wierność replikacji
• Wysoka wierność replikacji (śr. 1 błąd na 10
9
zreplikowanych pz).
• Polimerazy DNA są enzymami z funkcją autokorety
(proofreading), które usuwają własne błędy podczas
replikacji.
• Kluczowe dla autokorekty są ich aktywności 3’-5’
egzonukleazy, dzięki którym usuwają źle sparowane
nukleotydy od 3’ końca nowo zsyntetyzowanego fragmentu
DNA. Specjalny system naprawy uzupełnia następnie
brakujące fragmenty nici.
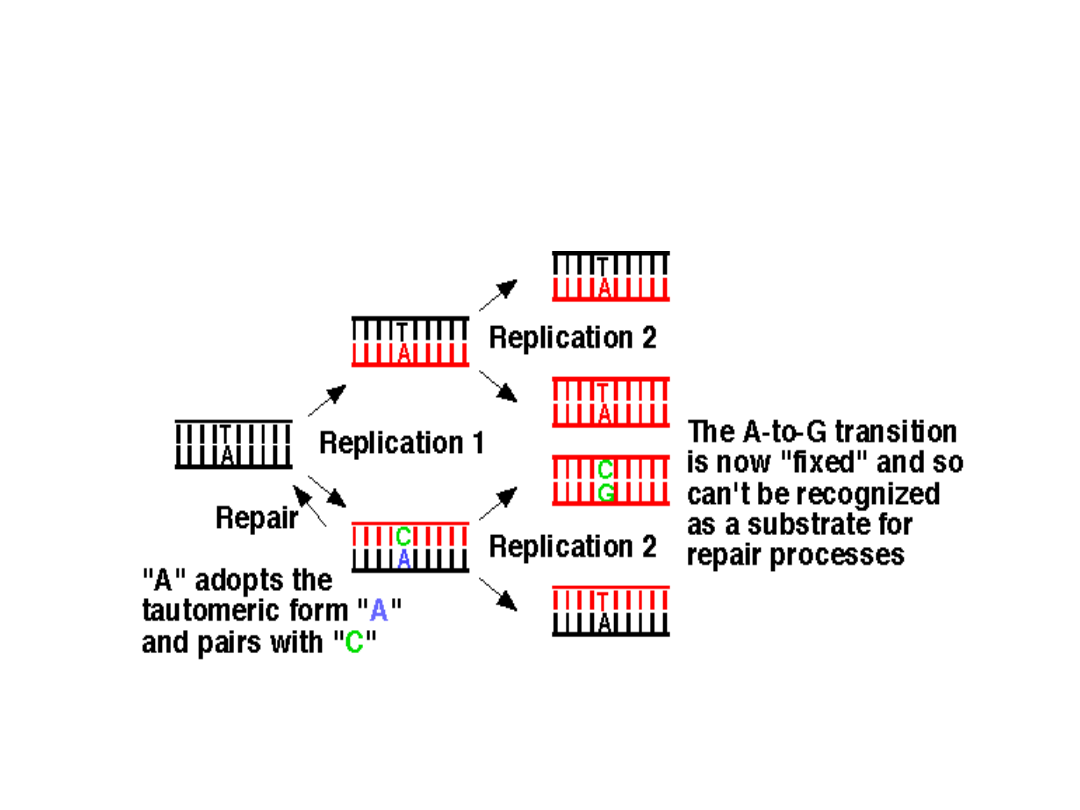
Niektórych błędów nie da się skorygować

Terminacja replikacji
• Terminacja następuje w miejscach, w których
spotykają się nowo syntetyzowane nici DNA
powstałe z dwóch sąsiadujących miejsc ORI.
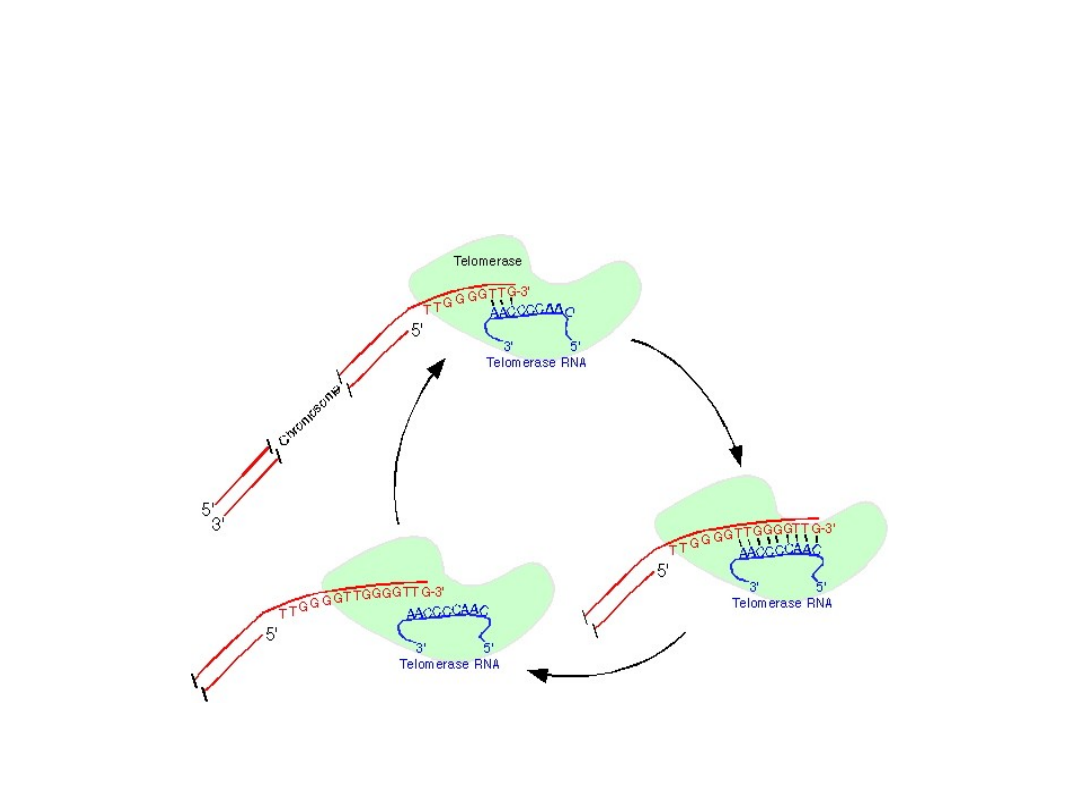
Aktywność telomerazowa -replikacja końców
chromosomów (telomerów)
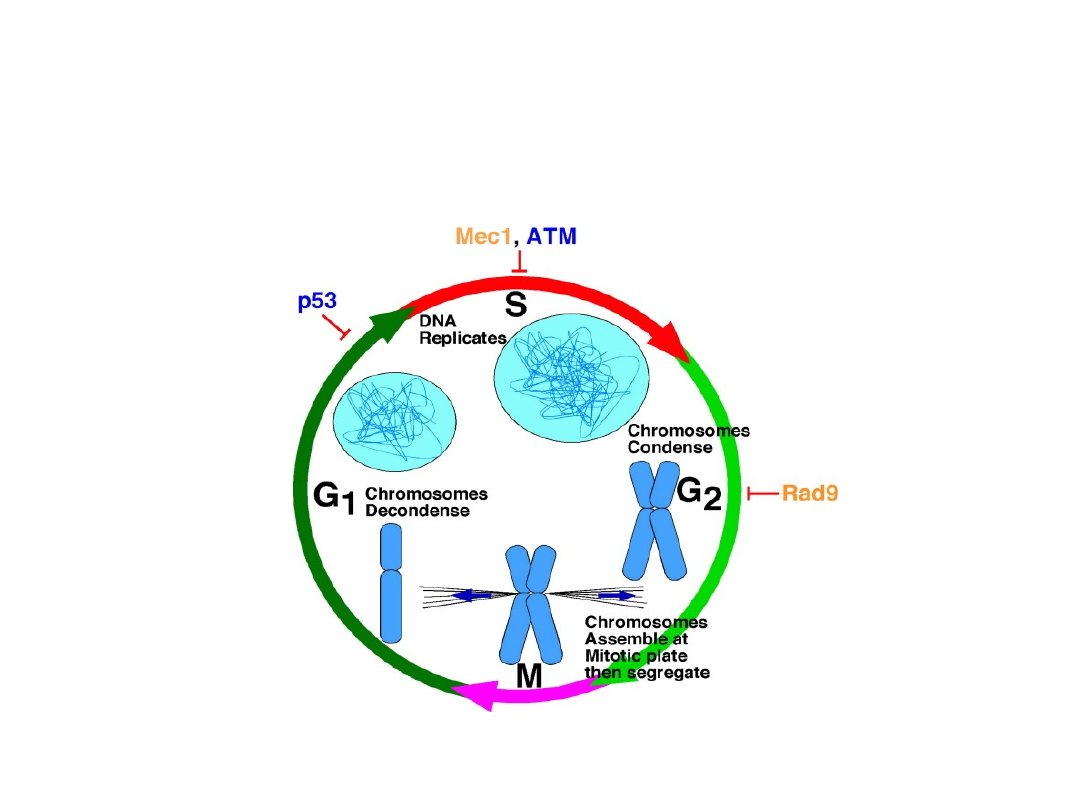
Punkty kontrolne (checkpoints) w cyklu komórkowym
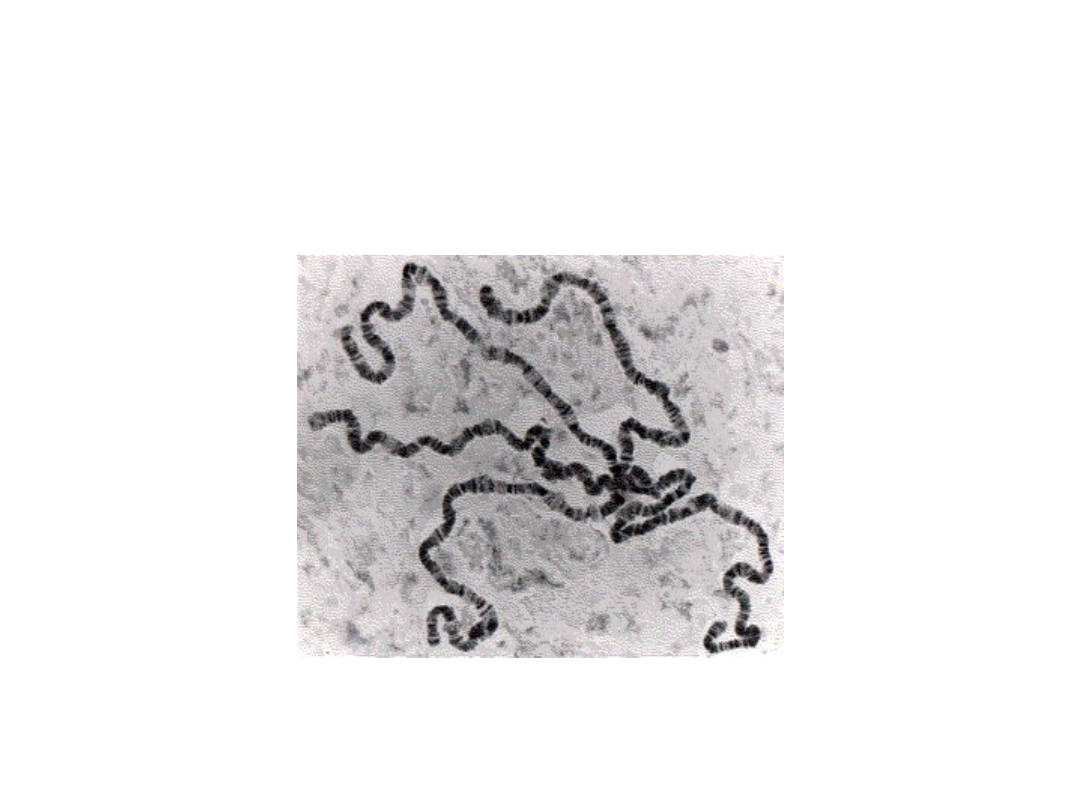
Chromosomy politeniczne (Drosophila – 10 rund replikacji bez
rozdzielania cząsteczek = 2048 cząsteczek ułożonych obok siebie)
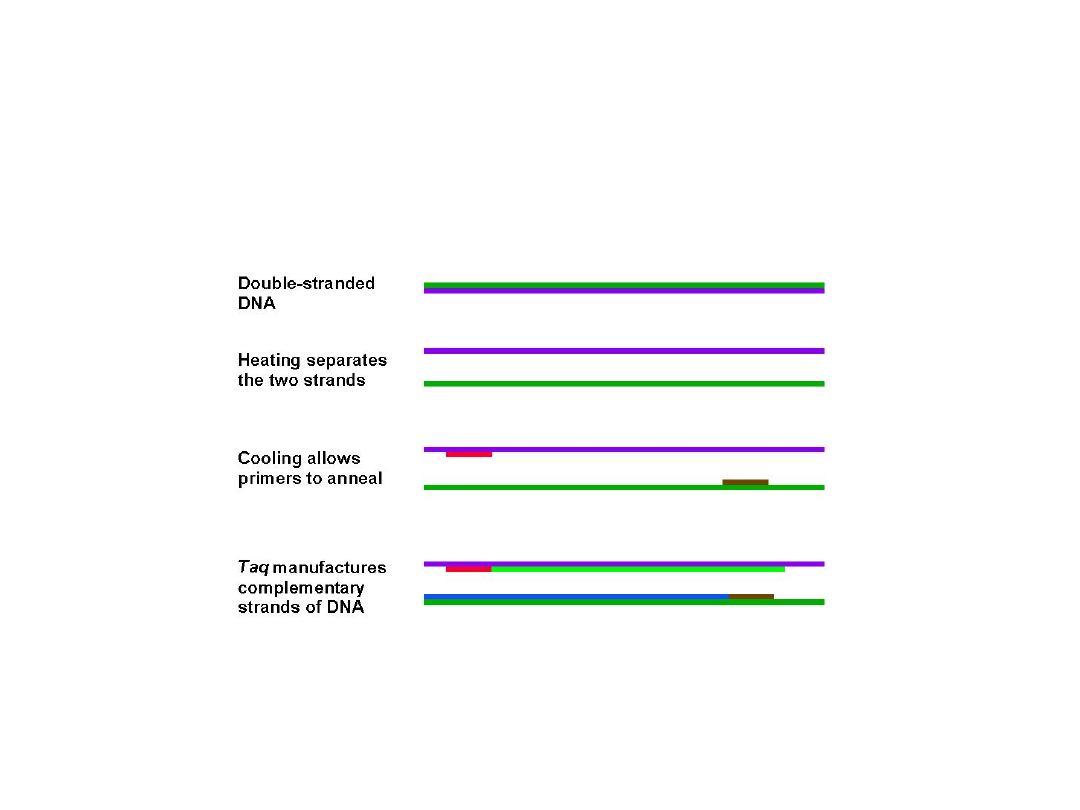
Łańcuchowa reakcja polimerazy (PCR)

Produkty PCR (polimeraza Taq)

Przyczyny uszkodzeń DNA i ich efekty
• Tlen, wolne rodniki
• UV
• Związki alkilujące
• Spontaniczna deaminacja (C
do U)
• Przerwanie łańcucha
• Modyfikacje chemiczne zasad
• włączanie niesparowanych
zasad w trakcie replikacji
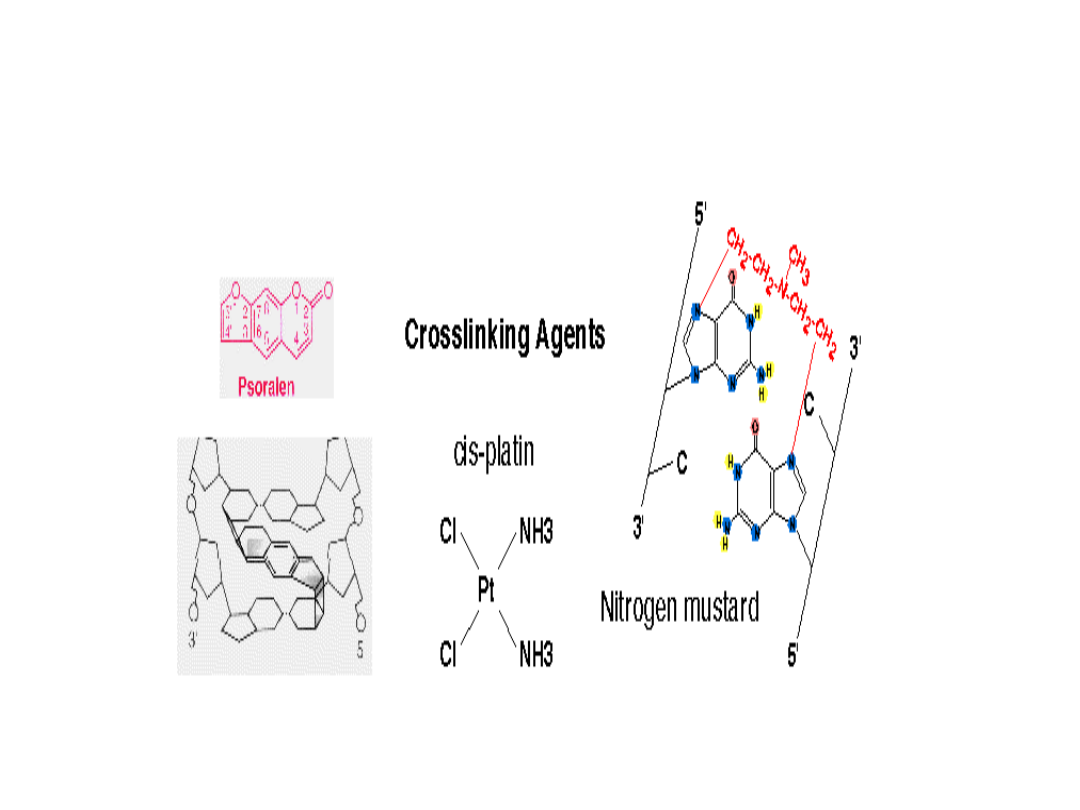
Uszkodzenia DNA - przykłady

Systemy naprawy DNA
• Wycięcie nukleotydów
• (dimery pirymidyn, aberracje
struktury)
• Naprawa błędnie sparowanych
nukleotydów (mylnie sparowane
zasady)
• Naprawa przez wycięcie zasad
(nietypowe – hipoksantyna,
uracyl-, zalkilowane zasady)
• Naprawa bezpośrednia
(metyloguanina, dimery
pirymidyn)
• System helikazy XPA
(Xerdoerma pigmentosum)
• Homologi bakteryjnych białek
typu Mut
• Glikozylaza DNA, polimeraza δ
• Guanino-6-metylotransferaza

Rekombinacja DNA
• Gra ważną rolę w podziale mejotycznym komórek
(zapewnia zróżnicowanie genetyczne gamet) i, na dłuższą
metę - w ewolucji (rearanżacje sekwencji DNA umożliwiają
nowe kombinacje sekwencji, które mogą generować nowe
rodzaje RNA i białek, wpływając na fenotyp).
• Mechanizmy rekombinacji są powiązane ściśle z
mechanizmami replikacji i naprawy
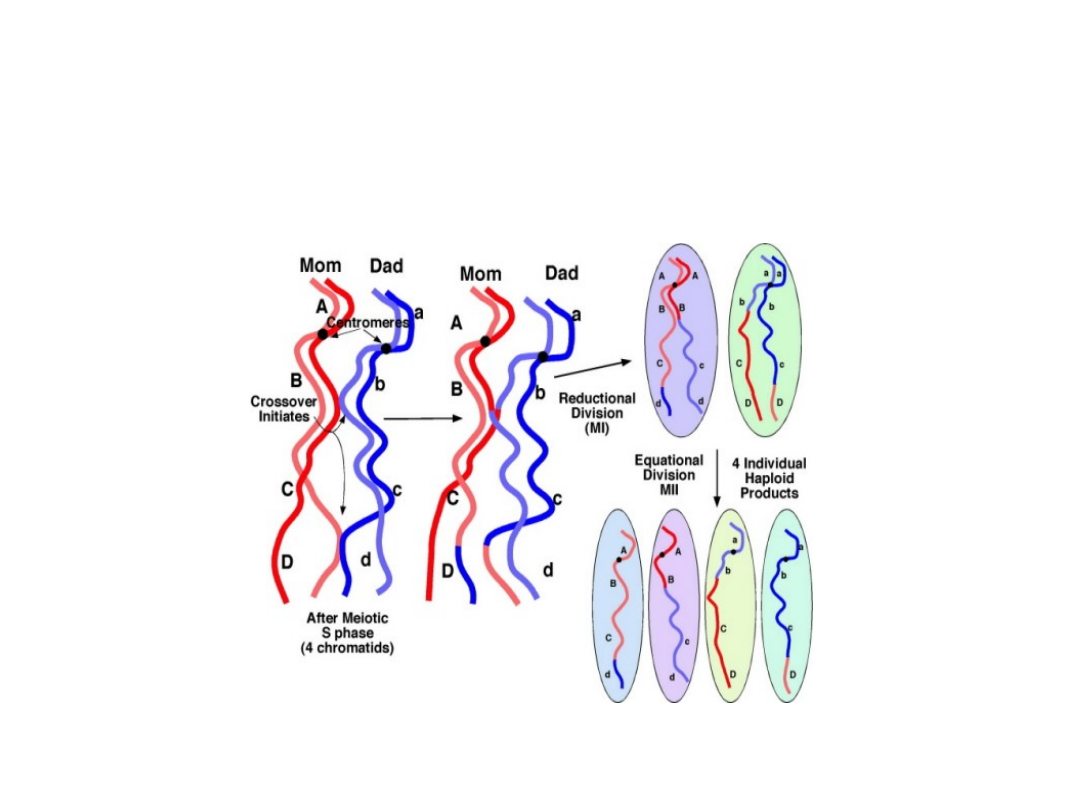
Rekombinacja w podziałach mejotycznych

Rodzaje rekombinacji
• Rekombinacja homologiczna występuje pomiędzy długimi
sekwencjami, które zawierają rejony w dużym stopniu do siebie
podobne (np. rekombionacja mejotyczna). Wymaga białek typu
RecA; katalizują one reakcję przeniesienia nici, która umożliwia
jednoniciowemu fragmentowi wniknięcie w strukturę
dwuniciową w rejonie homologii (powstaje przejściowa
struktura trójniciowa).
• Rekombinacja miejscowo specyficzna (np. rearanżacja genów
immunoglobulin) występuje w specyficznych loci, nie wymaga
długich rejonów homologii ani białek typu RecA. Wymaga
białka rekombinazy (integrazy) i krótkich sekwencji
palindroomowych w DNA donorowym i akceptorowym.
• Rekombinacja nieuprawniona może zachodzić w obecności
krótkich rejonów homologicznych (udział polimerazy RNA), a
także przy braku jakiejkolwiek homologii (np. integracja do
genomów roślin) (udział gyrazy, tj. topoizomerazy II).
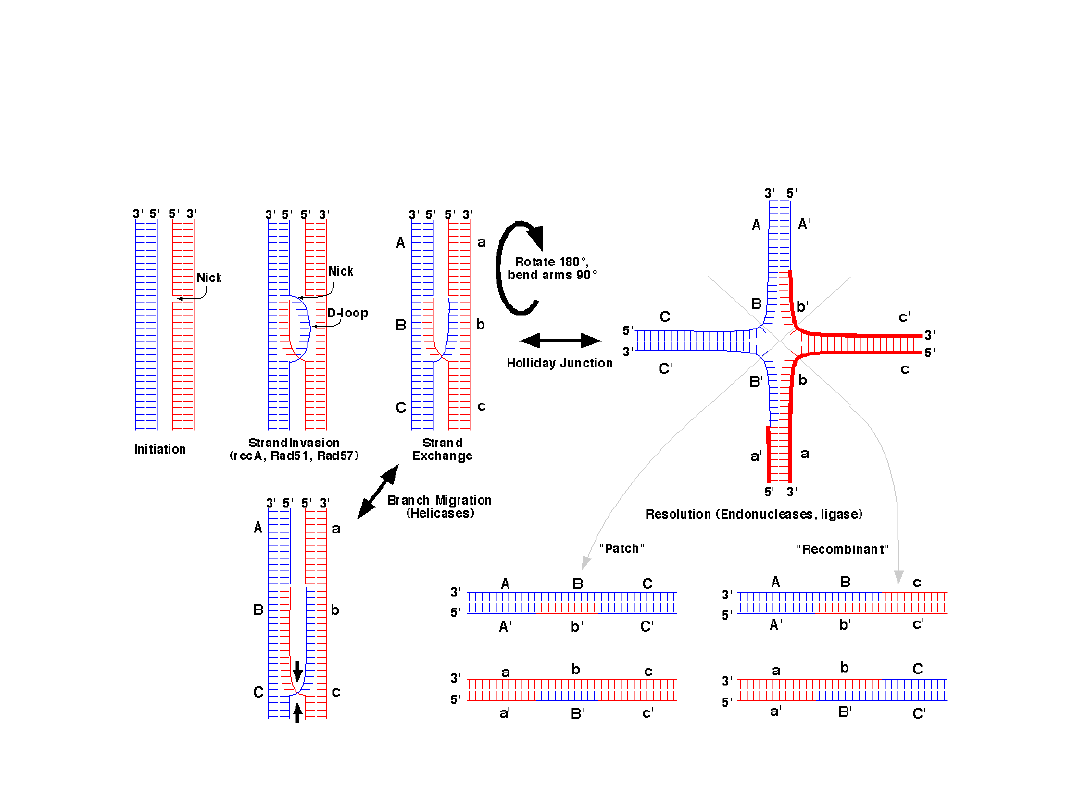
Struktura Hollidaya – etap pośredni w rekombinacji
homologicznej
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
Wyszukiwarka
Podobne podstrony:
Replikacja, naprawa i rekombinacja DNA u eukariontów 2009
10 Replikacja, naprawa i rekombinacja DNA
Ożyhar, Biologia molekularna,Replikacja naprawa i rekombinacjaDNA
Wykład 12 Replikacja i naprawa DNA
Ściąga Naprawa DNa, Operony, Koniugacja, Rekombinacja
Replikacja DNA i choroby związane
3 ogolny schemat replikacji i onkogeza DNA wirusowa
Materiał genetyczny, mutacje, systemy naprawy DNA, test Amesa
Izolacja DNA z komórek prokariotycznych i eukariotycznych
3 Systemy naprawcze w DNA
09 Replikacja DNA u?kterii
Uszkodzenia i naprawa DNA obieralny prof E Pastwa
nowotwory,DNA,RNA,replikacja
biologia, Replikacja DNA, Zjawisko transkrypcji to zjawisko powstawania rodzajów RNA ( t, m, r, )
Materiał genetyczny, mutacje, systemy naprawy DNA,
biologia, NAPRAWA DNA, Mechanizmy naprawy oksydacyjnych uszkodzeń DNA
Replikacja DNA
więcej podobnych podstron