
1
Wykład 8
Zmienność DNA, mutacje
Mutacje
Mutacja
(de Vries, 1909)
– nagła, skokowa
zmiana w materiale genetycznym organizmu,
mutacje mogą być dziedziczone (zachodzące w
gametach, germinalne) lub nie dziedziczone
(somatyczne).
Fenotypowe skutki mutacji somatycznych:
- autosomalnych, recesywnych
-
autosomalnych dominujących
-
sprzężonych z chromosomem X
Mutacje – podział
1
mutacja genowa
– powstająca wskutek zmiany
genu w jego nowy allel
mutacja chromosomowa strukturalna
– zmiana
struktury chromosomu
mutacja liczbowa
– powstająca wskutek zmiany
liczby chromosomów
mutacja genomowa
– zwielokrotnienie
haploidalnego zestawu chromosomów
Mutacje – podział
2
Mutacja spontaniczna
– nie posiada żadnej
znanej przyczyny, nie jest z nią związane
działanie żadnych czynników, zachodzi
przypadkowo. Stosunkowo mała częstość
występowania (u E. coli od 10
-5
do 10
-10
na
replikację).
Mutacja indukowana
– powstająca wskutek
działania naturalnego lub sztucznego czynnika.
Mutacja adaptacyjna
– doświadczenie Luria-
Delbr
ück: obecność bakteriofaga T1 nie indukuje
mutacji adaptacyjnych w hodowli E. coli (1943).
Mutacje – podział
3
Mutacja utraty funkcji
– niezależnie od zakresu
zmiany prowadzi do utraty funkcji genu.
Całkowita utrata funkcji – mutacja zerowa (null).
Mutacja uzyskania funkcji
– produkt genu
uzyskuje nowe funkcje lub zwiększa
dotychczasową.
Mutacja neutralna
– nie zmienia funkcji genu.
Mutacje genowe
Mutacja punktowa
– zmiana sekwencji DNA obejmująca jedną (lub
kilka) pz.
Substytucja
– zastąpienie starego nukleotydu nowym, tranzycja
(puryna
– puryna, pirymidyna – pirymidyna), transwersja (puryna –
pirymidyna, pirymidyna
– puryna).
Delecja
– wypadnięcie jednej (lub więcej) pary nukleotydów z
sekwencji DNA.
Insercja
– wstawienie jednej (lub więcej) pary nukleotydów do
sekwencji DNA.
Delecja i insercja zmieniają ramkę odczytu i mogą prowadzić do
poważnych zmian w kodowanych białkach.
Skutkiem mutacji genowej może być zmiana sensu kodonu (zmiana
sekwencji aminokwasów), mutacja niema (tryplet synonimiczny) lub
powstanie kodonu nonsensownego (przerwanie syntezy białka).
2
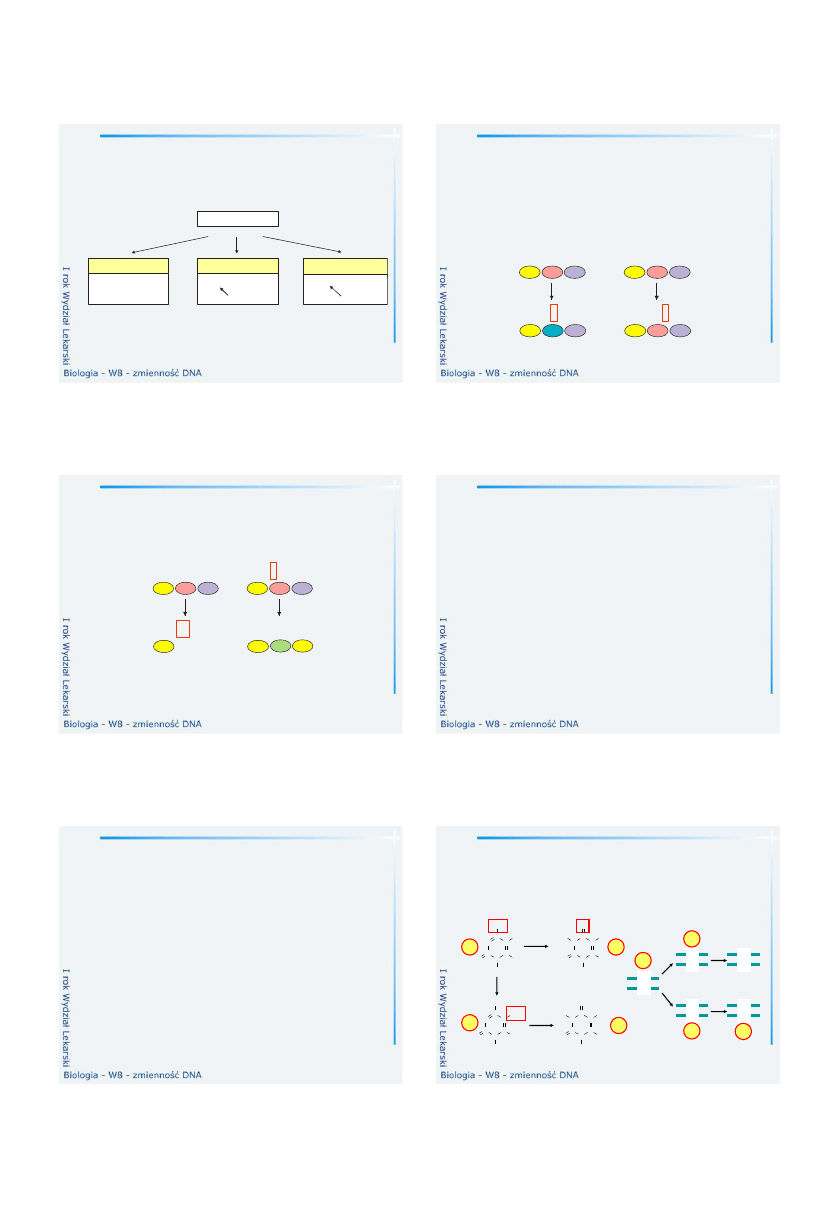
Mutacje genowe – przykład
TAK JAN PIŁ TEN SOK
Delecja
utrata J
TAK ANP IŁT ENS OK
Insercja
wstawienie M
TAK J A NPI ŁTE NSO K
M
Substytucja
TAK AN PIŁ TEN SOK
P
TAK JAN PIŁ TEN S K
A
TAK JA PIŁ TEN SOK
Ś
Mutacje punktowe
1
Substytucja może zmienić sens kodonu lub nie.
Mutacje nieme nie zmieniają sekwencji
aminokwasów w produkowanym białku.
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
transwersja
tranzycja
T
A
T
A
T
A
T
A
T
A
T
A
T
A
T
A
T
A
T
A
A
T
Leu
Leu
Leu
Leu
Ile
Ile
Ile
Asn
Pro
Pro
Pro
Pro
A
T
A
T
A
T
A
T
T
A
T
A
Mutacje punktowe
2
Zmiana ramki odczytu przez insercję lub delecję może
znacząco zmienić sekwencję produkowanego białka.
Wynikiem mutacji genowych u człowieka są choroby
monogenowe: mukowiscydoza, fenyloketonuria,
alkaptonuria, albinizm, retinoblastoma, achondroplazja,
hemoglobinopatie.
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
C
G
insercja
delecja
T
A
T
A
T
A
STOP
T
A
T
A
T
A
T
A
T
A
T
A
T
A
A
T
Leu
Leu
Leu
Leu
Ile
Ile
Ser
Pro
Pro
A
T
A
T
A
T
T
A
T
A
T
A
C
G
C
G
T
A
Leu
Czynniki uszkadzające DNA
Czynniki endogenne:
•
depurynacja, depirymidynacja
•
formy tautomeryczne
•
wolne rodniki
•
metylacja nieenzymatyczna
•
błędy replikacji
•
transpozony
Czynniki egzogenne:
•
biologiczne (wirusy)
•
chemiczne (produkty pirolizy, leki, kwas azotawy, pestycydy, dym
tytoniowy, mykotoksyny, aminy aromatyczne, węglowodory
policykliczne)
•
fizyczne (promieniowanie kosmiczne, jonizujące, UV)
Chemiczne czynniki mutagenne
•
kwas azotawy (HNO
2
)
•
hydroksylamina
•
związki alkilujące (sulfonian
dietylowy, sulfonian
etylometylowy, iperyt
azotowy)
•
analogi zasad azotowych (2-
aminopuryna, 5-bromouracyl)
•
wolne rodniki tlenowe
•
nadtlenki
•
policykliczne węglowodory
aromatyczne
Leki:
• busulfan
• cyklofosamid
• aminopteryna
• azatioperyna (Imuran)
• ametopteryna
(Methotrexat)
• winkrystyna (Oncovin)
• aktynomycyna
• daunomycyna
Występujące w produktach spożywczych: produkty pirolizy
aminokwasów, mykotoksyny, środki konserwujące (np. azotyn
sodowy).
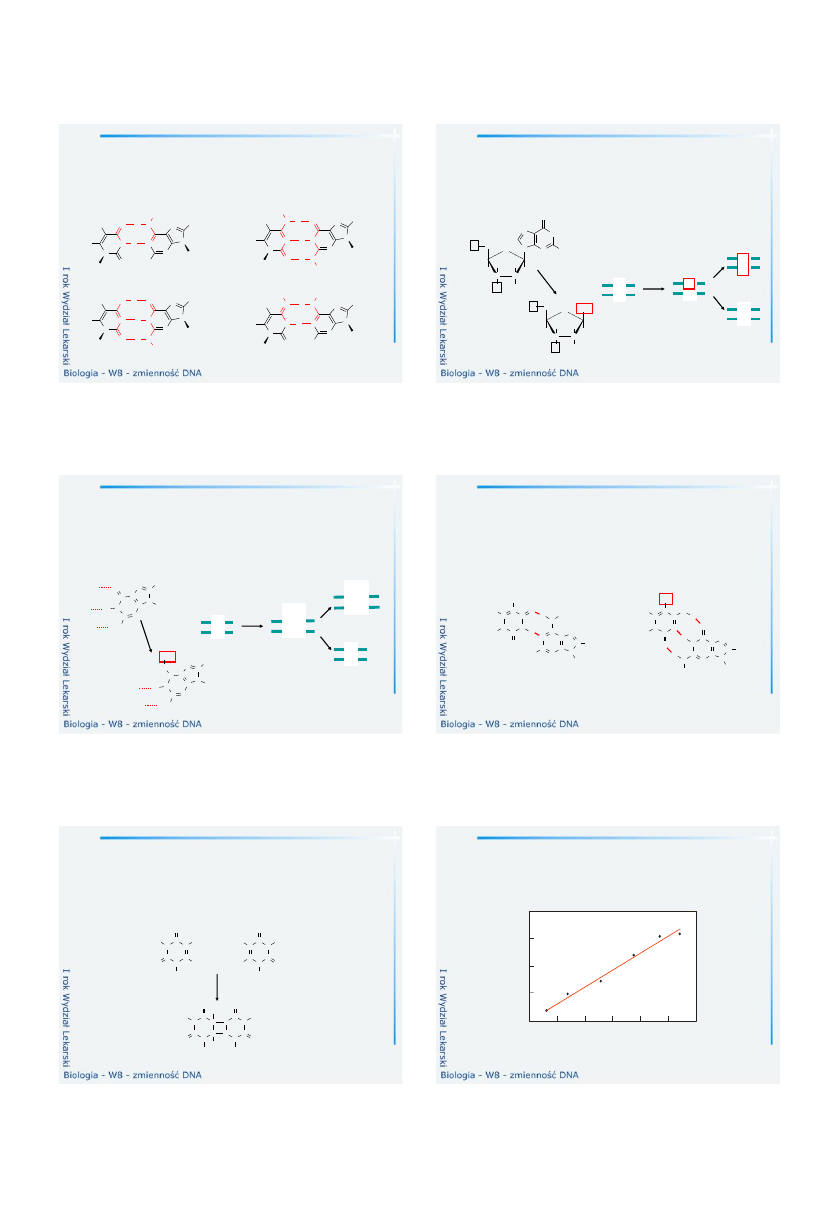
Modyfikacje zasad – mutacje
1
Deaminacja
– usunięcie grupy aminowej z cytozyny, adeniny lub
guaniny daje zasadę o odmiennej możliwości parowania (działanie
np. kwasu azotawego, nitrozamidu).
N
C
N
C
C
C
O
NH
2
H
H
H
N
C
N
C
C
C
O
O
H
H
H
H
deaminacja
nitrozamid
N
C
N
C
C
C
O
O
CH
3
H
H
H
cytozyna
uracyl
tymina
N
C
N
C
C
C
O
NH
2
CH
3
H
H
5-metoksycytozyna
metylacja
deaminacja
1
4
3
2
C
G
U
G
C
G
T
A
1
2
3
4
T
A
3
Modyfikacje zasad – mutacje
2
Przesunięcie tautomeryczne
– formy tautomeryczne zasad
azotowych mogą tworzyć nietypowe (niehomologiczne) połączenia z
innymi zasadami: A=
C
lub
T
G
N
N
C
H
3
O
O
H
H
N
N
N
N
H
N
O
H
H
H
Tymina (enol)
Guanina (keto)
N
N
C
H
3
O
H
O
H
N
N
N
N
H
H
N
H
H
Tymina (keto)
Adenina (amino)
N
N
H
N
O
H
H
H
N
N
N
N
H
H
N
H
H
Cytozyna (imino)
Adenina (amino)
N
N
H
N
O
H
H
H
N
N
N
N
H
N
O
H
H
H
Cytozyna (amino)
Guanina (keto)
Modyfikacje zasad – mutacje
3
Depurynacja
– usunięcie grupy purynowej z adeniny lub guaniny
może zajść na skutek zmian temperatury.
Przy replikacji
depurynacja prowadzi do delecji.
G
C
G
C
C
delecja
N
N
N
N
O
C
C
H
OH
H
P
H
H
CH
2
P
NH
2
O
OH
O
C
C
H
OH
H
P
H
H
CH
2
P
depurynacja
Modyfikacje zasad – mutacje
4
Alkilacja
– przyłączenie do grup aminowych lub ketonowych danej
cząsteczki grupy metylowej lub etylowej. Czynnikami alkilującymi są
między innymi: etylonitrozomocznik, sulfonian etylometanowy,
dimetylonitrozamina.
G
C
G
C
CH
3
G
C
alkilacja
C
C
N
C
N
N
C N
C
H
O
N
H
2
H
H
C
C
N
C
N
N
C N
C
H
O
N
H
2
H
H
CH
3
CH
3
G
T
Modyfikacje zasad – mutacje
5
Analogi zasad nukleotydowych
– cząsteczki puryn lub pirymidyn o
własnościach zbliżonych do zasad azotowych DNA. 5-bromouracyl
(BrU) jest analogiem tyminy ale obecność bromu umożliwia wiązanie
BrU również z guaniną.
N
C
N
C
C
C
O
O
CH
3
H
H
H
C
C
N
C
N
N
C
N
C
N
H
H
H
H
H
N
C
N
C
C
C
O
O
Br
H
H
H
C
C
N
C
N
N
C
N
C
H
O
N
H
H
H
H
tymina
adenina
BrU
guanina
Modyfikacje zasad – mutacje
6
Dimery cyklobutylowe
– działanie promieniowania UV może
powodować powstawanie kowalencyjnych wiązań pomiędzy
sąsiadującymi cząsteczkami np. tyminy. Zmniejszenie odległości
pomiędzy nukleotydami prowadzić może do delecji przy replikacji.
tymina
N
C
N
C
C
C
O
O
CH
3
H
H
H
N
C
N
C
C
C
O
O
C
H
3
H
H
H
N
C
N
C
C
C
O
O
CH
3
H
H
H
N
C
N
C
C
C
O
O
H
H
tymina
UV
Modyfikacje zasad – mutacje
7
Działanie promieniowania jonizującego (np. promieni Roentgena)
powoduje mutacje, których częstotliwość zależy od dawki
promieniowania.
dawka promieniowania X [R]
1000
2000
3000
4000
5000
%
s
p
rz
ęż
o
n
y
c
h
z
X
m
u
ta
c
ji
le
ta
ln
y
c
h
10
5
15
wg. Concepts of genetics, Pearson Int.Edition, 2009

4
Transpozycja DNA
1
Transpozycja
– przemieszczenie fragmentów DNA (transpozonów)
pomiędzy różnymi pozycjami wewnątrz genomu.
Transpozycja zachodzi rzadko
– raz na około 10
3
– 10
4
podziałów
komórkowych.
miejsce docelowe
T
A
T
A
C
G
G
C
T
A
5’
3’
3’
5’
sekwencja insercyjna
T
A
T
A
C
G
G
C
T
A
T
A
T
A
C
G
G
C
T
A
Transpozycja DNA
2
Transpozycja replikacyjna i niereplikacyjna.
5’
3’
3’
5’
5’
3’
3’
5’
transpozycja replikacyjna
dawca
biorca
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
transpozycja niereplikacyjna
Mutacje chromosomowe
(strukturalne) 1
Inwersja
– odwrócenie fragmentu chromosomu o 180°.
Inwersja obejmująca centromer –
pericentryczna
(eucentryczna).
Inwersja nie obejmująca centromeru –
paracentryczna
(acentryczna).
Zapis: inv(9)(p11q12)
a
a
a
a
b
b
b
b
c
c
c
c
o
180
o
180
Mutacje chromosomowe
(strukturalne) 2
Translokacja
– przemieszczenie fragmentu chromosomu
w obrębie tego samego lub innego chromosomu.
Translokacja wzajemna
– wymiana pomiędzy
chromosomami niehomologicznymi.
Zapis: t(1;11)(q42.1;q14.3)
translokacja
wzajemna
5p
5q
15p
15q
chromosomy
5 pary
chromosomy
15 pary
Mutacje chromosomowe
(strukturalne) 3
Translokacja zrównoważona
– całkowita wymiana
pomiędzy chromosomami, nie ma dodatkowych lub
brakujących genów.
Translokacja niezrównoważona
– wymiana niecałkowita,
pojawiają się dodatkowe lub brakujące geny.
5q
1 5q
1 5q
15p
15p
ro zdział
naprzem ienny
rozdział
przyległy 2
rozdział
przyl egł y 1
prawidło wa zrównoważona
niezrównoważona
5q
5 p
5p
Mutacje chromosomowe
(strukturalne) 4
Translokacja robertsonowska (fuzja centryczna)
–
łączenie się całych (lub prawie całych) ramion długich
chromosomów akrocentrycznych lub telocentrycznych i
utrata ramion krótkich. W fuzjach najczęściej uczestniczą
chromosomy 14 i 21.
tra nslokacja
robe rtsono wska
14
14
t(14q21q)
2 1
21
nosiciel
t(14q21q)
mutacje
letalne
gamety nosiciela
zespół
Downa

5
Mutacje chromosomowe
(strukturalne) 5
Duplikacja
– podwojenie tych samych odcinków
chromosomów.
a
b
a
b
a
b
Mutacje chromosomowe
(strukturalne) 6
Delecja
– utrata odcinka chromosomu (terminalna i
interstycjalna). Miejsca łamliwe występują w
chromosomach 2 (2q13), X (Xq27), 6, 9, 12, 20.
Zapis: del(6p15.2)
a
a
b
c
c
Mutacje chromosomowe
(strukturalne) 7
Insercja
– włączenie fragmentu innego
chromosomu.
Zapis: ins(12;6)(q24.2;q23q25)
ins(12;6)(q20;q35)
12
6
Mutacje chromosomowe
(strukturalne) 5
Chromosom kolisty
– powstaje w wyniku pęknięcia i
połączenia końców chromosomu (u człowieka 4, 13, 18
oraz X).
Izochromosom
– powstaje wskutek nieprawidłowego,
poprzecznego podziału centromeru chromosomu
metafazowego. Mogą powstawać z autosomów oraz
chromosomu X.
p
p
p
q
q
q
po replikacji
Mutacje chromosomowe
(liczbowe) 1
Mutacje ilościowe w większości przypadków powstają na skutek
nierozdzielenia się (nondysjunkcji) par chromosomów
homologicznych w czasie podziałów. Mutacje ilościowe dzieli się na
dwie klasy:
•
aneuploidie
(zmiana ilości chromosomów danego rodzaju)
•
poliplidie
(zmiana ilości całych zestawów chromosomów)
Przyczyny powstawania aneuploidii:
• utrata centromeru
• nondysjunkcja
• translokacja Robersonowska
Przykłady:
Nullisomia (2n-2), monosomia (2n-1) z. Turnera (45,X), trisomia
(2n+1) z. Downa (47,XX+21)(47,XY+21), z. Patau (47,XX+13),
(47,XY+13), z. Klinefeltera (47,XXY), tetrasomia (2n+2)
Mozaikowatość: np. (46, XX/47,XX+21).
Mutacje chromosomowe
(ilościowe) 2
Euploidie
(poliploidie)
– zwiększenie haploidalnego
zestawu chromosomów (3n, 4n, 5n itd).
Np. triploidia
– 69,XXX lub 69,XXY etc.
Autopoliploidie
– garnitur chromosomów zwiększony jest
o ten sam zestaw chromosomów (AABB + AABB =
AAAABBBB). Autopoliploidie u człowieka są letalne i
prowadzą do poronień.
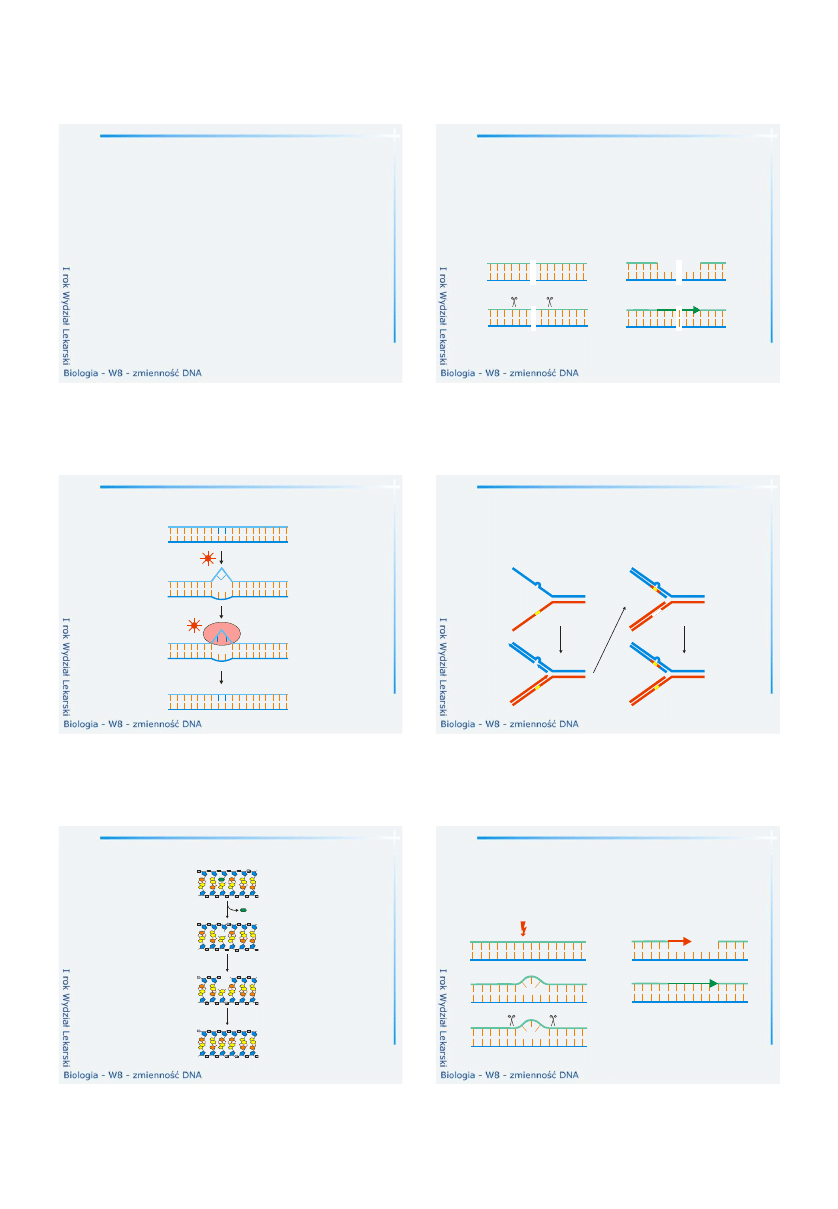
Allopoliploidie
– występowanie dwóch lub więcej
niehomologicznych zespołów chromosomów. Występują u
mieszańców międzygatunkowych (nie możliwych u
człowieka) AABB + CCDD = ABBCCDD (np. pszen-żyto)
6
Naprawa DNA
Mechanizmy naprawy przeciwdziałają zmianom DNA.
Zmiany DNA:
• jednoniciowe
• dwuniciowe
Naprawa zmian jednoniciowych:
• kontrola poprawności replikacji
• naprawa braku sparowania
• naprawa fotoreaktywacyjna
• naprawa przez wycięcie (zasady lub nukleotydu)
Naprawa zmian dwuniciowych :
• rekombinacja homologiczna
• rekombinacja niehomologiczna
Naprawa braku sparowania
Błędy pozostające po replikacji powodują brak sparowania.
Stara i nowa nić odróżniane są poprzez różne wzory metylacji.
Wykrywanie i wycinanie - hMSH1, hMLH1, hMSH2 (MutH, MutL,
MutS u E. coli).
Zastąpienie wyciętego fragmentu – polimeraza i ligaza.
5’
5’
3’
3’
T
G
T
G
G
C
G
Fotoreaktywacja
T
T
A A
T
T
A A
T
T
A A
PRE
T
T
A A
5’
3’
5’
3’
5’
3’
5’
3’
UV
niebieskie
Poreplikacyjna naprawa DNA
Podczas replikacji zniszczony fragment jest pomijany. Po
replikacji ten fragment ulega rekombinacji a ubytek w
drugiej nici jest uzupełniany przez polimerazę i ligazę.
nić wiodąca
nić opóźniona
uszkodzenie
Naprawa – wycięcie zasady
5’
3’
5’
3’
5’
3’
5’
3’
glikozylaza DNA
AP site
endonukleaza AP
polimeraza DNA
+
ligaza DNA
Naprawa – wycięcie nukleotydu
Naprawa przez wycięcie nukleotydów
– zniszczenia DNA jest
wykrywane dzięki zmianom w konfiguracji trójwymiarowej. Nici są
rozdzielane i stabilizowane. Wielkość wycinanego fragmentu jest
różna u Prokaryota (3’-5-D-8-5’) i Eukaryota (3’-5-D-21(3)-5’).
czynnik uszkadzający (np. UV)
5’
5’
3’
3’
wykrycie uszkodzenia
wycięcie
polimeryzacja
ligacja (zszycie)

7
Rekombinacja homologiczna
1
Rekombinacja homologiczna
–
proces zachodzący podczas
crossing-
over jest również
używany do naprawy
dwuniciowych uszkodzeń DNA.
Naprawa odbywa się w późnej
fazie S/G2.
W
modelu Holliday’a
rekombinacja odbywa się
pomiędzy dwoma przerwami w
pojedynczych niciach dupleksu
DNA.
Migracja rozgałęzienia
wytwarza heterodupleks DNA.
5’
3’
5’
3’
czynnik uszkadzający (np. UV)
rozpoznanie uszkodzenia,
wycięcie końców 5’
5’
3’
5’
3’
koniec 3’ łączy się z homologicznym
regionem chromatydy siostrzanej
5’
3’
5’
3’
5’
3’
5’
3’
synteza brakującego odcinka DNA
5’
3’
5’
3’
5’
3’
5’
3’
rozkład heterodupleksu,
odtworzenie drugiej nici DNA
5’
3’
5’
3’
Rekombinacja homologiczna
2
Alternatywny model naprawy
dwuniciowego uszkodzenia
DNA.
W tym przypadku formowane
są dwa połączenia Holliday’a.
połączenie Holliday’a
połączenie Holliday’a
Scalanie niehomologicznych
końców DNA
Mechanizm naprawy DNA
wykorzystywany głównie
przez wielokomórkowe
organizmy eukariotyczne.
5’
3’
5’
3’
czynnik uszkadzający (np. UV)
przyłączenie heterodimerów
Ku70/80
5’
3’
5’
3’
rekrutacja DNA-PKcs
5’
3’
5’
3’
utworzenie kompleksu
naprawczego
5’
3’
5’
3’
5’
3’
5’
3’
naprawa DNA
Odpowiedź SOS
Odpowiedź SOS uruchamiana jest u bakterii (np. E. coli)
w przypadku wystąpienia rozległych uszkodzeń DNA
uniemożliwiających replikację.
Komórki indukują ekspresję ponad 20 genów, które
wymuszają replikację pomimo uszkodzeń.
Włączenie mechanizmu SOS zatrzymuje podział komórki.
Podstawowymi genami biorącymi udział w odpowiedzi
SOS są: LexA, RecA,
l
.
Wyszukiwarka
Podobne podstrony:
Biologia W4 2014
Spisane pytania biologia molekularna 2014, UG, MOLEKUŁY
Biologia W7 2014
Biologia W2 2014
Biologia W5 2014
Podstawy biologiczne - wykłady 2014
Biologia W4 2014
Biologia W10 2014
Biologia W9 2014
Biologia EGZAMIN 2014
arkusz i odpowiedzi matura probna biologia rozszerzona operon 2013 2014
rodzaje mat biolog1 25 02 2014
WYTYCZNE DO ĆWICZEŃ Z BOTANIKI SYSTEMATYCZNEJ UP 2014, biologia, Biologia I rok, Botanika systematyc
cwiczenie 1 oksydoreduktazy i transferazy wykrywanie aktywnosci enzymow w materiale biologicznym 05
2014 biologia maj odp
Botanika systematyczna program ćwiczeń 2014, biologia, Biologia I rok, Botanika systematyczna
więcej podobnych podstron