1.Wyjaśnij zależność między liczbą par zasad tworzących sekwencję palindromową i odległością pomiędzy miejscami restrykcyjnymi w DNA.
Liczba par zasad w miejscu restrykcyjnym determinuje odległość między miejscami restrykcyjnymi.
Średnia odległość między miejscami restrykcyjnymi = 4n
n- liczba zasad w miejscu restrykcyjnym
Sekwencja palindromowa w genetyce oznacza taką sekwencję DNA dla której sekwencja komplementarna jest identyczna.
5' A A T T 3' lub 5' A G G C C T 3'
3' T T A A 5' 3' T C C G G A 5'
Sekwencje palindormowe są często miejscem działania enzymów restrykcyjnych co czynie je bardzo interesującymi dla inżynierii genetycznej
Sekwencje restrykcyjne to krótkie, palindromowe fragmenty DNA rozpoznawane przez
enzymy restrykcyjne. Endonukleazy rozpoznają z reguły sekwencje o długości 4 do 8 par zasad,
a produkty ich trawienia maja zakończenia w postaci jednoniciowego ogona na końcu 3` lub
5` („lepkie” końce). „Lepkie” końce umożliwiają łączenie fragmentów DNA pochodzących
z różnych źródeł. Zatem jest to stosunkowo prosty sposób na wbudowanie fragmentów DNA
do plazmidów lub DNA innych wektorów.
RESTRYKTAZY - tną DNA tworząc powtarzalny komplet fragmentów. Restryktazy (inaczej enzymy restrykcyjne , endonukleazy restrykcyjne) - to enzymy izolowane z bakterii , zdolne do rozpoznawania specyficznych sekwencji w DNA (z reguły są to sekwencje palindromowe) i do przecinania dwuniciowej cząsteczki DNA w ściśle określonym miejscu , w obrębie lub okolicy sekwencji rozpoznawanej. Otrzymywane fragmenty DNA nie są losowe a w każdym prążku na żelu znajdują się cząsteczki DNA o identycznej sekwencji nukleotydowej. Z reguły różne enzymy rozpoznają odmienne sekwencje DNA. Istnieją jednak wyjątki - tzw. izoschizomery - enzymy izolowane z różnych organizmów ale rozpoznające te same sekwencje. Zdarza się także , że dwa enzymy wytwarzają takie same lepkie końce , mimo rozpoznawania różnych sekwencji DNA. Umożliwia to klonowanie DNA strawionego jednym enzymem w wektorze strawionym innym , dającym takie same lepkie końce.
Jednostka enzymu restrykcyjnego to taka jego ilość , która trawi kompletnie 1mg DNA faga l (około 50 kb) w czasie 1 godz. w temperaturze 37°C.
Podział restryktaz według rodzaju wytwarzanych końców :
tępe końce - nici rozcięte naprzeciwko siebie - wszystkie nukleotydy są sparowane z komplementarnymi nukleotydami na przeciwnym łańcuchu
lepkie końce - 3` lub 5` ssDNA (jednoniciowe) ogony na obu końcach utworzone przez niesymetryczne cięcie, komplementarne do podobnych tworzonych w innych cząsteczkach DNA przez te same enzymy restrykcyjne niezależnie od źródła DNA, co pozwala na ligowanie DNA nawet bardzo różniących się gatunków czyli formowanie chimerycznych molekuł.
Podział restryktaz według sekwencji rozpoznawanej :
czwórkowe , rozpoznają sekwencję DNA złożoną z czterech nukleotydów. Statystycznie w dowolnym DNA takich miejsc jest dużo - co 256 bp. Restryktazy takie mogą strawić DNA na bardzo małe kawałki.
szóstkowe , rozpoznają sekwencję DNA złożoną z sześciu nukleotydów. Dowolne miejsce restrykcyjne złożone z sześciu nukleotydów występuje statystycznie co około 4096 bp w DNA , w którym ilości poszczególnych nukleotydów są równe. Proporcje te zmieniają się w zależności od organizmu , dlatego dobór enzymu szóstkowego i warunki należy ustalić eksperymentalnie.
ósemkowe , stosowane niezbyt często. Tną DNA bardzo rzadko.
Zastosowania :
Konstrukcja map restrykcyjnych.
Klonowanie i obróbka DNA.
Badanie polimorfizmu miejsc restrykcyjnych (RFLP)
Wprowadzenie DNA do genomu innego źródła
LIGAZY - trwale łączą pocięte fragmenty.
Ligazy DNA katalizują formowanie wiązań fosfodiestrowych pomiędzy końcem hydroksylowym 3` a końcem fosforowym 5` DNA. Pozwala to na reperowanie jednoniciowych przerw w dupleksie DNA , łączenie fragmentów restrykcyjnych posiadających homologiczne lepkie czy też nawet tępe końce.
Dwa najczęściej stosowane enzymy to ligaza DNA E.coli i ligaza DNA z faga T4. Tylko ta ostatnia jest w stanie wydajnie łączyć tępe końce, nawet w normalnych warunkach reakcji. Wadą jest jej mniejsza specyficzność rozpoznawania struktury końców, co daje większe tło w doświadczeniach spowodowane nieprawidłowymi ligacjami.
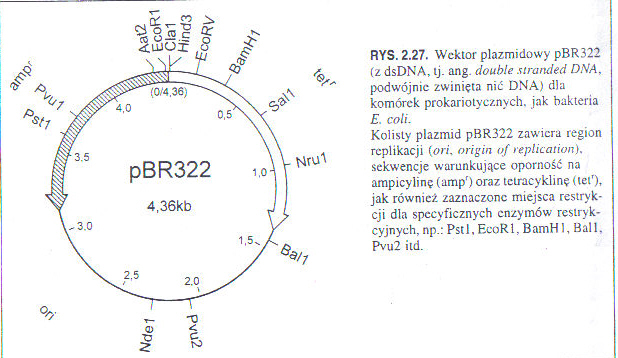
2.Omów cechy wektorów na przykładzie wektora plazmidowego pBR322
Wektory to przenośniki genów (w tym sztucznie skonstruowane, zwane kasetami ekspresyjnymi) są cząsteczkami najczęściej kolistego DNA i są odpowiednio dobrane do gospodarza tj. zapewniają integrację z gnomem biorcy oraz możliwość namnażania się w odpowiedniej ilości kopii. Najczęściej stosowanymi genami dla komórek prokariotycznych są wektory:
plazmidowe (np. wektor dla Escherichia coli - pUC19 lub pBR322)
fagowe ( bakteriofagi lambda)
hybrydy (tzw. kosmity)
W przypadku komórek eukariotycznych np. drożdży stosuje się:
wektory integracyjne
replikatywne
episomalne
Wektory plazmidowe są oparte na plazmidach, czyli są to koliste cząsteczki DNA zdolne do autonomicznej replikacji w komórkach gospodarza - niezależnej od replikacji chromosomów, pełnią funkcje pomocniczych chromosomów. Dla potrzeb inżynierii genetycznej dzieli się plazmidy na nieekspresyjne i ekspresyjne, czyli umożliwiające ekspresję wklonowanego do wektora genu.
Wektory plazmidowe odznaczają się:
możliwie małą liczbą miejsc cięcia przez restryktazy (najlepiej jednym) - przy czym z reguły dany plazmid posiada kilka takich miejsc dla kilku różnych enzymów.
wielkość naturalnych plazmidów mieści się w zakresie od 200 do 100 000 pz, (par zasad) zaś wielkość konstruowanych metodami inżynierii genetycznej rekombinowanych wektorów plazmidowych wynosi na ogół ok. 10 000 pz (par zasad).
są nośnikami genów o informacji służącej inaktywacji antybiotyków, syntezy toksyn i rozkładania innych naturalnych związków np. alkaloidów.
zawierają region replikacji - umożliwiający replikację wektora w komórkach gospodarza
Wektor plazmidowy pBR322:
zawiera miejsca restrykcji dla specyficznych enzymów restrykcyjnych np. Pst1, EcoR1, BamH1, Bal1 itd.
wielkość 4,36 kb (par zasad)
zawiera sekwencje warunkujące oporność na ampicylinę (amp') oraz tetracyklinę (tet')
zawiera region replikacji (ori)
3.Omów podstawy metody klonowania cDNA
technika klonowania komplementarnego DNA (cDNA), jest podstawą w izolacji i analizie genów eukariontów, polega ona na :
izolacji z komórek eukariontów RNA - z ogonami poli (A) jest izolowany z ogólnego RNA z wykorzystaniem sond oligo (dł) metodą chromatografi kolumnowej
ogon poli A- jest to dodawany szereg nukleotydów adeninowych zwanych fragmentem poliadenylowym lub poli-A do końca 3' cząsteczki mRNA. mRNA u eukariotów powstaje podczas transkrypcji jako heterogenne hnRNA (pre-mRNA), a następnie ulega obróbce posttranskrypcyjnej, podczas której dodawana jest czapeczka, wycinane są introny, i dodawany jest ogon poli-A. Zabieg taki ma na celu zabezpieczenie cząsteczki mRNA eukariontów przed degradacją zanim zdąży opuścić jądro komórkowe.
polimeraza DNA zależna od RNA (odwrotna transkryptaza) jest wykorzystywana do budowy pierwotnej nici DNA na primerze oligo(dł), tworzy hybryd mRNA- DNA. Następnie jest trawiony przez RNAazę, która wycina odcinki i tworzy luki w nici mRNA.
Odwrotna transkryptaza- enzym, który składa poszczególne deoksynukleotydy w nić DNA, wykorzystując nić RNA jako matrycę. W wyniku odwrotnej transkrypcji powstaje pojedyncza nić DNA będąca kopią RNA; sekwencja nukleotydowa DNA jest komplementarna do sekwencji RNA. Tak uzyskany DNA nosi nazwę cDNA.
Druga nić DNA jest składana przez polimerazę DNA wykorzystującą jako matrycę pierwszą nić cDNA. Dwuniciowy cDNA można już sklonować. Jakkolwiek bezpośrednie określanie sekwencji RNA jest niewdzięczne i uciążliwe, jednak dzięki przekształceniu RNA w sklonowany cDNA analiza sekwencji i funkcji RNA staje się prosta.
Naturalną formą wzajemnych oddziaływań kwasów nukleinowych jest tzw. hybrydyzacja, czyli tworzenie struktur dwu lub więcej niciowych, będących wynikiem parowania zasad purynowych i pirymidynowych (T lub U z A, C z G). Proces ten zachodzi z bardzo wysoką specyficznością. To ostatnie wykorzystano do wykrywania w mieszaninach DNA lub RNA określonych sekwencji przy pomocy tzw. oligonukleotydowych sond molekularnych. Oligonukleotydowe sondy molekularne są to oligomery o zdefiniowanych (najczęściej programowanych) sekwencjach, które są komplementarne do sekwencji adresowanej (poszukiwanej). W odpowiednio dobranych warunkach sonda hybrydyzuje jedynie z sekwencją komplementarną, pomimo obecności w badanym materiale wielu (często kilku lub kilkudziesięciu milionów) fragmentów o wysokiej homologii do sekwencji poszukiwanej.
polimeraza DNA z E.coli buduje fragment okazaki, a następnie wypełnia luki, usuwa kawałki RNA i powstaje druga nić DNA
dwuniciowy DNA jest klonowany w odpowiednim wektorze
Dlaczego biblioteki cDNA mają duże znaczenie?
Biblioteki genowe- odcinki genomu odpowiedzialne za konkretne geny danego odcinka. Biblioteka cDNA- mieszana populacja rekombinantów, zbiór genów uzyskanych np. metodą hybrydyzacji sondy, reprezentuje geny ulegające ekspresji. Poddając analizie zgromadzone w takiej bibliotece cząsteczki DNA można stwierdzić, które geny są aktywne w danym typie komórek lub tkanek.
Poszczególne klony cDNA można izolować i określać ich własności zwykle w taki sam sposób, jak klony genomowego DNA. Jakkolwiek bezpośrednie określanie sekwencji RNA jest niewdzięczne i uciążliwe, jednak dzięki przekształceniu RNA w sklonowany cDNA analiza sekwencji i funkcji RNA staje się prosta.
4.Zasada metod PCR i przykłady ich praktycznego stosowania
a) Metoda PCR (polymerase chain reaction)jest to reakcja łańcuchowa polimerazy, umozliwiająca przeprowadzenie selektywnej amplifikacji (namnażania) in vitro poszczególnych fragmentów DNA bez potrzeby jego klonowania. Do przeprowadzenia tej reakcji potrzeba:
wzorcowy jednoniciowy DNA
dwa startery (primery) krótkie o sekwencji 20-nukleotydów komplementarne do znanych sekwencji końcowych wzorcowego DNA
deoksynukleinowe trifosforany- dNTPs
polimeraza DNA.
Reakcja ta polega na przeprowadzeniu wielu cykli (20-30) rund w ciągu 2-3h. Każdy cykl obejmuje:
termiczną denaturację (temp. 92-96°C) wzorcowego dsDNA ( dwuniciowy DNA) do ssDNA (jednonicowy DNA);
przyłączanie starterów ( tworzenie wiązań wodorowych) do wzorcowego ssDNA (temp. 40-60°C), czyli denaturację;
wydłużanie (polimeryzacja) DNA przy końcach 3'-OH w temp. 72°C w wyniku sukcesywnego przyłączania dNTPs za pomocą termo stabilnej polimerazy DNA( np. polimerazy Taq).
Startery dozowane w nadmiarze w stosunku do powielanego DNA powodują powielanie DNA do 105 razy. Powielone DNA można identyfikować, poddać analizie restrykcyjnej lub nieradioaktywnej hybrydyzacji oraz detekcji.
b)Zastosowanie:
laboratoria genetyczne do prac badawczych;
wykrywanie chorobotwórczych mutacji;
sekwencjonowanie DNA;
mutageneza In vitro;
identyfikacja genów;
badanie ekspresji genów;
diagnostyka drobnoustrojów, chorób dziedzicznych;
w sadownictwie;
w kryminalistyce ( otrzymywanie odcisku palca)
5.Podaj przykłady zastosowań genetycznie modyfikowanych mikroorganizmów, roślin i zwierząt
Mikroorganizmy:
Synteza hormonów- hormon wzrostu
Synteza enzymów- insulina, chymotrypsyna
Produkcja leków
Biodegradacja odpadów
Rośliny:
Odporność na herbicydy- chemiczne środki ochrony roślin, środki chwastobójcze (najczęściej na RoundUpReady-RR)- soja (najczęściej uprawiana roślina transgeniczna), kukurydzę, rzepak, tytoń, pomidory. Modyfikacja dająca oporność na RoundUpReady zostaje uzyskana albo po przez wprowadzenie do rośliny genu kodującego syntazę EPSPS nie wrażliwą na herbicyd, albo poprzez wprowadzenie genu odpowiedzialnego za powstanie enzymu GOX (oksydoreduktazy glifosatu), który rozkłada aktywny składnik RoundUp'u - glifosat.
Odporność na choroby- powodowane przez pleśnie, grzyby i wirusy- wprowadzenie transgenu kodującego enzymy - hitynaza lub glukanaza, które niszczą ich ścianę komórkową lub kodujący osmotynę - białko wiążące się z błoną komórkową powodując jej zniszczenie.
Odporność na wirusy uzyskuje się poprzez wprowadzenie do rośliny genów białek płaszcza (kapsydu) danego wirusa, a także jego enzymów: replikazy, proteazy - pojawienie się tych białek powoduje to, iż późniejsza infekcja tym wirusem jest znacznie słabsza lub skutki choroby pojawiają się z dużym opóźnieniem.
Np. tytoń odporny na wirusa mozaiki tytoniowej (TMV), ogórka na wirusa mozaiki ogórka, kalafiora na wirusa mozaiki kalafiora.
Odporność na owady- szkodniki- Gen odpowiedzialny za taką odporność - gen Bt - uzyskuje się z bakterii glebowej Bacillus thuringensis. Gen ten koduje specyficzne białko - Cry 9C- które jest toksyczne dla owadów, bezpieczne dla ludzi i zwierząt. Np. ziemniak StarLink odporny na stonkę, bawełna, kapusta, pomidory, oraz przede wszystkim kukurydza - kukurydza StarLink wprowadzenie genu Bt (MON 810) oraz żepaku.
Odporność na nie korzystne warunki środowiska (tolerancja stresu)-odporność mróz, wysoką i niską temperaturę, suszę, i zasolenie gleby, nadmiar promieniowania, zanieczyszczenia środowiska, głównie szkodliwe metale w glebie,
Ekspresja fermaceutyków- naturalny bioreaktor np. modyfikowana sałata produkująca szczepionkę na zapalenie wątroby typu B i innych farmaceutyków
Poprawa cech jakościowych (wartości żywieniowej) roślin-
pomidory transgeniczny Flavr Savr- zmniejszenie aktywności genu odpowiadającego za procesy dojrzewania i mięknięcie owoców- trwałość w czasie przechowywania
ziemniaki ze zwiększonej ilości skrobii
złoty ryż- wszczepienie genu powoduje zwiększenie produkcji beta- karotenu
pszenica o zwiększonej zawartością glutenu
intensyfikacja koloru roślin ozdobnych
zmiana tekstury, zabarwienia, zapachu
produkcja białek odżywczych, większej zawartości mikroelementów, usuwanie substancji alergennych,
nadające lepszy smak i intensywniejszy aromat, zwiększenie zawartości kofeiny- kawa,
kawa bez kofeiny (obniżenie o 70%)
Zwierzęta:
uzyskanie pożądanych cech hodowlanych
szybciej rosnące zwierzęta i ryby- karpie, łososie, świnie, króliki i owce- wszczepienie hormonu wzrost
lepsza jakość mleka i mięsa
krowy dające więcej mleka i o specyficznym składzie- więcej beta- i kappa- kazeiny- więcej i szybsza produkcja sera
odporność na choroby
wełna toksyczna dla moli i nie kurcząca się w praniu
kozy produkujące włókno podobne do pajęczyny
modyfikacja świń jako dawcy narządów- transgeniczny knurek TG 1154
modele stanów chorobowych
świecące ryby- jako wskaźnik czystości wody
transgeniczne koty dla alergików
wykorzystanie
kóz, krów i owiec jako bioreaktory- do produkcji białek, enzymów i innych substancji wykorzystywanych w przemyśle farmaceutycznym np. antytrombina, antytrypsyna, erytropoetyna , laktoferyna.
króliki- produkcja interleukin, czynnika IGF1, ludzkiego hormon wzrostu, alfa-glukozydaza, czy białka C
6.Omów konstrukcję i warunki hodowli szczepu bakteryjnego lub grzybowego zdolnego do nadprodukcji chymozyny cielęcej lub insuliny ludzkiej
a)produkcja chymozyny EC 3.4.23.4 renina
Chymozyna- enzym powodujący ścinanie się skrzepu mleka, powoduje powstawanie skrzepu kazeinowego.
Początkowo otrzymywany z żołądków cieląt. Lecz te źródła są nie stabilne, różna aktywność i czystość tego enzymu. Enzym ten może być też wydobywany z rośliny owadożernym np. rosiczka jest to jednak chymozyna roślinna.
Pierwszym genetycznie modyfikowanych mikroorganizmów produkującym chymozynę był Mucor niehen. Na skalę przemysłową produkuje się rekombinowaną chymozynę otrzymujemy przy użyciu Eschericha coli z wołowym genem produkcji chymozyny jest to chymozyna A. Inne mikroorganizmy to Kluyeromyces lactis oraz Aspergillus niger var avamori- chymozyna B.
Warunki produkcji:
produkcja ma trzy etapy (namnażanie komórek zrekombinowanych, fermentacja inakulacyjna oraz fermentacja wstępna)
odpowiednie napowietrzenie
warunki aseptyczne (kontrola pH i T)
b)produkcja insuliny
Insulina jest polipeptydem z grupy albumin składającym się z dwóch łańcuchów , krótszego A zbudowanego z 21 aminokwasów oraz dłuższego B zawierającego 30 aminokwasów, połaczonym dwoma mostkami dwusiarczkowymi. Brak insuliny lub jej niedobór jest przyczyną poważnych zaburzeń przemiany sacharydów ,a w konsekwencji także białek i tłuszczów. Insulina zmniejsza stężenie glukozy w surowicy krwi i nasila jej spalanie , wzmaga syntezę glikogenu i peptydów w wątrobie , a także hamuje glukoneogenezę z aminokwasów i intensyfikuje syntezę wolnych kwasów tłuszczowych.
Do niedawna insulinę otrzymywano metodą ekstrakcji z trzustek zwierzęcych lub metoda pół syntetyczną polegająca na enzymatycznym przekształceniu insuliny wieprzowej w insulinie ludzką, poprzez wymianę końcowego aminokwasu -alaniny - łańcucha B insuliny wieprzowej w położeniu 30 na L-treoninę.
Metoda inżynierii genetycznej uzyskano insulinę humanizowaną określaną jako insulinę ludzką rekombinowaną przez biosyntezę w komórkach E. Coli lub S. Cerevisiae , które są odpowiednio transformowane
dzięki wprowadzeniu genu proinsuliny ludzkiej lub też genów A i B insuliny ludzkiej. Z proinsuliny insulina jest otrzynywana po enzymatycznym (z udziałem trypsyny i karboksypeptydazy) odszczepieniu łańcucha C.
w drugim rozwiązaniu skonstruowano dwa szczepy mikroorganizmów wytwarzających odpowiednio łańcuchy A i B , które należy przeprowadzić w S-sulfonowe pochodne a następnie połączyć metodami chemicznymi (tioliza i utlenianie) w cząsteczkę aktywnego hormonu.
Bardzo ważnym procesem technologicznym jest oczyszczanie insuliny co pozwala na zapewnienie właściwej jakości preparatu oraz jego pełnej aktywności fizjologicznej. Po oddzieleniu biomasy metoda wirowania lub filtracji pożywka jest zobojętniana i zagęszczana w próżni, a surowa insulina wysalana w punkcie izoelektrycznym i krystalizowana w z dodatkiem soli cynku. Preparat po kilkukrotnej krystalizacji zawiera 80-90% insuliny. Dalsze oczyszczanie odbywa się z udziałem metod chromatograficznych, sączenia molekularnego oraz elektroforezy.
Szczegóły produkcji z E.coli
Do otrzymywania rekombinowanje insuliny zastosowano szczep E. Coli transformowany plazmidem z fragmentem DNA kodującym zmodyfikowany prekursor ludzkiej insuliny składający się z łancucha B i dipeptydu łączącego łańcuch A insuliny.
Sekwencja DNA kodująca prekursor jest dołączona do zmodyfikowanego fragmentu genu ludzkiej dysmutazy.
Biomasa komórek E. Coli zawierająca białko dyfuzyjne -mini-pre-mini-proinsulinę -syntetyzowane w postaci nierozpuszczalnych ciałek inkluzyjnych jest oddzielana od pożwyki metoda wirowania lub filtrowania, a następnie dezintegrowana metoda enzymatyczną.
Wyizolowane białko fuzyjne jest rozpuszczane w celu odtworzenia przestrzennej konformacji charakterystycznej dla natywnego białka. Z odpowiednio zmodyfikowanego chemicznie białka fuzyjnego po enzymatycznej konwersji, w czasie której zostaje oddzielony peptyd dysmutazy i peptyd A insuliny, otrzymuje się preparat różniący się od insliny ludzkiej obecnością dodatkowych aminokwaśów na C-końcu łancucha B.
Po oczyszczeniu metoda chromatografii kolumnowej i enzymatycznym usunięciu reszt dodatkowych aminokwasów rekombinowany hormon jest następnie strącany, oczyszczany metodą wysokosprawnej chromatografii cieczowej, krystalizowany i suszony pod próżnią w warunkach zapewniających apyrogenność i czystość mikrobiologiczna leku. Otrzymywany preparat ma czystośc 98-99%
c) warunki hodowli
Źródło N, C, S ,P, składników odżywczych, witamin i wody
Zawartość tlenu
pH
temp
ciśnienie osmotyczne
7.Porównaj metody mikro- i ultrafiltracji oraz podaj przykłady ich zastosowań w biotechnologii Żywności
Mikro- i ultrafiltracja - ogólna zasada działania..
Zasadą mikrofiltracji i ultrafiltracji jest fizyczna separacja. Wielkość porów membrany określa stopień, do którego rozpuszczona substancja stała, mętność lub mikroorganizmy są usuwane.Substancje, których cząsteczki są większe niż pory membrany są całkowicie zatrzymywane na filtrze. Substancje, których cząsteczki są mniejsze niż pory membrany są częściowo usuwane, w zależności od budowy warstwy "odrzucającej" membrany. Mikrofiltracja oraz ultrafiltracja to procesy zależne od ciśnienia, dzięki którym usuwane są z wody rozpuszczone substancje stałe oraz inne substancje, lecz w mniejszym stopniu niż w procesie nanofiltracji czy odwróconej osmozy.
b) Mikrofiltracja.
W procesie mikrofiltracji używane są membrany o wielkości porów 0.1-10 µm. Przez membrany przechodzą do supernatantu białka, bimolekuły i niewykorzystane składniki. Zanieczyszczenie wirusami jest częściowo usuwane w procesie, mimo że wirusy są mniejsze niż pory membrany mikrofiltracji. Wynika to z faktu, że wirusy mogą przylegać do biofilmów bakterii. Mikrofiltracja nie usuwa substancji rozpuszczonych takich jak np. białka. Mikrofiltracja nie wymaga stosowania dużych ciśnień - już przy niewielkim (0,1-0,3 MPa) przepływ jest duży.
Przykłady zastosowań mikrofiltracji:
· Sterylizacja na zimno w procesie produkcji napojów i w przemyśle farmaceutycznym
· Klarowanie soków, wina oraz piwa
· Separacja bakterii z wody (oczyszczanie biologiczne wody)
· Oczyszczanie ścieków
· Separacja emulsji olej/woda
· Oczyszczanie wstępne wody przed procesami nanofiltracji i osmozy odwróconej
· Separacja typu ciecz/substancja stała w przemyśle farmaceutycznym i produkcji żywności
c) Ultrafiltracja
Jest to proces filtracji z użyciem sit molekularnych, membran i wszelkich materiałów porowatych. Pory membran ultrafiltracji zatrzymują cząsteczki o wielkości 0.001 - 0.1 µm. Masa cząstki powyżej 10kDa. Proces ten umożliwia rozseparowanie roztworów rzeczywistych oraz mieszanin gazów na poszczególne związki chemiczne, jony, pierwiastki. Ultrafiltracja umożliwia także całkowite usunięcie wirusów. Wymaga jednak ona dużych ciśnień oraz jest czasochłonna.
Przykłady zastosowań ultrafiltracji:
· Przemysł mleczarski (mleko, ser)
· Przemysł spożywczy (białka)
· Przemysł metaliczny (separacja emulsji typu olej/ woda, farby)
· Przemysł tekstylny
8.Jak z ekstraktu tkankowego wydzielić ogólny mRNA stosując metodę chromatografii powinowactwa ?
a)chromatografia
Sprzęt- kolumna wypełniona nośnikiem (żywicą) oraz podstawa (saport)
Zasada rozdziału- na podstawie różnicy masy (sita molekularne), ładunku (elucja gradientem soli) lub oddziaływań biologiczne
b)chromatografia powinowactwa to skuteczna i powszechnie stosowana metoda do izolacji białek. Do różnorodnych procesów biotechnologicznych, a także wielu analiz biochemicznych trzeba użyć substancji biochemicznie czynnych o najwyższym stopniu czynności. Spełnienie tego warunku jest niezwykle trudne, czasami wręcz niemożliwe, zwłaszcza gdy substancje wyodrębnia się z zastosowaniem tradycyjnych metod preparowanych. W technice tej wykorzystuje się duże powinowactwo białek do specyficznych grup chemicznych. Chromatografia powinowactwa może być skutecznie stosowana do izolacji:
czynników transkrypcyjnych
białek regulujących ekspresje genów
enzymów
przeciwciał
hormony
poprzez zdolność do wiązania białek ze specyficznymi sekwencjami DNA.
Przebieg metody:
mieszaninę białka przesącza się przez kolumnę wypełnioną nośnikiem, do którego zostały przyłączone specyficzne sekwencje DNA.
Białka wykazujące duże powinowactwo do tych sekwencji będą zatrzymywane na kolumnie.
W tym przypadku czynnik transkrypcyjny jest uwalniany przez przemycie kolumny roztworem o dużym stężeniu soli.
Chromatografia powinowactwa, ze względu na swe unikatowe właściwości, pozwala znacznie uprościć procedurę izolowania wybranej substancji jednocześnie zachowując jej aktywność biologiczną. Jest to szczególny typ chromatografii adsorbcyjnej, w której wykorzystuje się wzajemne powinowactwo danej substancji. Jedna z nich, chemicznie związana z nierozpuszczalnym nośnikiem i zwana ligandem specyficznie absorbuje druga substancję izolując ja z mieszaniny innych związków. Oddziaływanie substancji rozpuszczonej w fazie ruchomej z unieruchomionym ligandem może mieć różny charakter. Chromatografie powinowactwa przeprowadza się w dwóch etapach.
izolowanie plazminogenu z osocza krwi z użyciem żelu Sepharose ze związana lizyną polega, na tym, że przez kolumnę zrównoważoną odpowiednim buforem przepuszcza się rozcieńczone buforem osocze. Dochodzi wówczas do specyficznej sorpcji przepływającego plazminogenu na unieruchomionej lizynie. Po dokładnym odmyciu nieswoiście związanych białek i innych składników osocza
w drugim etapie następuje rozszczepienie kompleksu lizyna- plazminogen pod wpływem kwasu E(epsilon)-aminokapronowego, użytego do elucji plazminogenu. Jest to typowy przykład swoistej elucji substancji z sorbntu, ponieważ zarówno lizyna, jak i kwas E(epsilon)-aminokapronowy są inhibitorami plazminy, powstałej przez aktywacje plazminogenu i wiążą się z tym samym miejsce w cząsteczce enzymu. Związki izolowane metoda chromatografii powinowactwa można eulowac również w niespecyficzny sposób za pomoca buforów o niskim lub wysokim pH, roztworów o dużej sile jonowej, czy związków rozrywających wiązania wodorowe. Zastosowanie zarówno specyficznych , jak i niespecyficznych eluentów pozwala uzyskać czysty preparat izolowanej substancji(w tym wypadku mRNA). W obu przypadkach należy wymienić bufor w drodze dializy lub filtracji żelowej.
Czasem dzięki tej metodzie izoluje się także witaminy, glutation oraz kwasy nukleinowe.
9.Omów główne kierunki biokonwersji skrobi w przemyśle syropiarskim i gorzelniczym
a) skrobia
Skrobia składa się z podjednostek glukozy połączonej wiązaniami α-1,4-glikozydowymi i w miejscach rozgałęzień wiązaniami α-1,6-glikozydowymi.
b) biokonwersja
Biokonwersja skrobi w przemyśle syropiarskim i gorzelniczym polega na hydrolizie skrobi. Działając odpowiednimi enzymami dochodzi do hydrolizy wiązań np.
α-amylaza działa w przypadkowy sposób na wiązania α-1,4-( w przemyśle wykorzystuje się głównie α-amylazy pochodzenia bakteryjnego- termostabilna, upłynnia skrobię i pochodzenia grzybowego - mniej odporna na temperaturę, odporna na zmiany pH)
glukoamylaza na wiązania α-1,4- i z mniejszą aktywnością na wiązania α-1,6-glikozydowe odszczepiając od nieredukującego końca cząsteczki skrobi glukozę,
β-amylaza działa na wiązania α-1,4- odszczepiając cząsteczki maltozy (również od nieredukującego końca cząsteczki skrobi).
pullulanaza lub izoamylaza- hydrolizujące wiązania α-1,6-glikozydowe (w miejscach rozgałęzień)
α-amylaza bakteryjna- termostabilna, upłynnia skrobię
c) przemysł gorzelniczy
Celem biokonwersji skrobi jest hydroliza skrobii do cukrów fermentujących a następnie produkcji alkoholu konsumpcyjnego (nieodwodniony) i bioetanolu (odwodniony). [lub też etanolu i etanolu nie odwodnionego]
Przed zacieraniam surowiec skrobiowy jest przygotowywany po przez parowanie (w parnikach temperatura i wysokie ciśnienie), dzięki temu uwalnia się skrobia z surowca, skrobi kleikuje i przechodzi w stan rozpuszczalny- proces ten ułatwia działanie enzymów amylolitycznych.
Zacieranie- dodaje się preparaty enzymatyczne (α-amylazy i glukoamylazy pochodzenia mikrobiologicznego), lub rozdrobniony słód jęczmienny (np. α-amylazę, β-amylazę). Zacieranie odbywa się w odpowiedniej dla działania enzymów temperaturze i pH. Podczas zacierania zachodzą procesy enzymatyczne, a najważniejszy (proces enzymatyczny) to hydroliza skrobi, czyli w tym wypadku scukrzanie skrobi do cukrów fermentujących pod wpływem dodanych enzymów.
Można też produkować alekohol z celulozy przy pomocy endo i egzo celulazy
d) przemysł syropiarski- przemysł syropów skrobiowych
dotyczy procesów enzymatycznych tj. hydroliza skrobi, czy też konwersja glukozy do fruktozy.
dobrane parametry hydrolizy (głownie czas konwersji, ale również temperatura i pH) pozwalają osiągnąć pożądany stopień scukrzenia (stopień hydrolizy).
Przykłady
Syrop glukozowy otrzymuje się poprzez możliwie jak największą depolimeryzacje skrobi (niemal całkowite scukrzenie skrobi), (glukoamylaza γ- amylaza+ pullanaza (1,6 wiązania))
syropy wysokoscukrzone,
syropy maltozowe (α-amylaza grzybowa)
syropy wysokomaltozowe
maltodekstryny- teksturotwórczymi dodatkami do żywności (α-amylaza bakteryjna)
maltooligosacharydy mające właściwości prebiotyczne
syropów wysokofruktozowych- substancje słodzące, również dla diabetyków (izomeraza glukozowa powoduje Glu Fru)
te produkty o wysokiej zawartości cukrów redukujących mają szerokie zastosowanie jako substancje słodzące, środki pomocnicze w procesach fermentacyjnych i wiele innych. Produkcja syropów fruktozowych opiera się na enzymatycznej konwersji glukozy do fruktozy.
10.Omów bioprocesy stosowane podczas przemysłowej produkcji aminokwasów egzogennych
Aminokwasy egzogenne u człowieka dorosłego (w nawiasach nazwy angielskie i ich oficjalne skróty) to:
walina (valine, Val)
U dziecka należy jeszcze wymienić dwa aminokwasy:
W latach 50. opracowano liczne technologie produkcji aminokwasów metodą syntezy enzymatycznej i biosyntezy mikrobiologicznej (gł. L-lizynę).
a)synteza enzymatyczna (droższa)
Dobierając odpowiednie enzymy można produkować L-aminokwasy z mieszaniny racemicznej D,L-prekursora. Przykładowo:
z D,L-α-amino-ε-koprolaktamu, otrzymanego metodą syntezy chemicznej, przy użyciu enzymu L-hydrolazy z drożdży Cryptococcus, Candida lub Trichosporon
racemaza z bakterii Achromobacter, Flavobacterium przekształca pozostałą formę D-kaprolaktamu w formę L-, z której ponownie przy użyciu L-hydrolazy otrzymujemy L-lizynę.
z mieszaniny N-acetyl-D,L-aminokwasów, używając aminoacylazy z A.oryzae, otrzymuje się L-aminokwasy, a pozostałą formę D-prekursora poddaje się racemizacji.
Ponieważ w procesie syntezy enzymatycznej używa się prekursorów i enzymów o dużej czystości (drogie), zastosowanie immobilizowanych enzymów umożliwia znaczne obniżenie kosztów enzymów, automatyzację procesu, pełniejsze wykorzystanie prekursorów, co w konsekwencji może obniżyć koszty produkcji danego aminokwasu o kilkadziesiąt procent.
b) biosynteza mikrobiologiczna- znacznie tańsza ze względu na fakt, że składniki pożywki są zazwyczaj tanie, proces przebiega w niższej temperaturze i uzyskuje się wyłącznie L-aminokwasy. W wyniku selekcji drobnoustrojów można uzyskać organizmy przydatne do przemysłowej produkcji aminokwasów. Jednocześnie przez dobór warunków hodowli, a zwłaszcza mutagenizację drobnoustrojów metodami fizycznymi, chemicznymi lub inżynierii genetycznej, można uzyskać organizmy zdolne do nadprodukcji danego aminokwasu.
Mutagenizacja umożliwia uzyskanie tzw. auksotroficznych mutantów tj. drobnoustrojów z rozkojarzonym systemem regulacji, powodujących nadprodukcję danego aminokwasu ze związku chemicznego, który jest prekursorem dla zahamowanej biosyntezy innych aminokwasów. Aminokwas, którego biosynteza została zahamowana powinien być dodawany do pożywki. Mutagenizacja wywołuje zmiany kierunków metabolicznych w wyniku zmiany aktywności określonych enzymów, co powoduje zahamowanie biosyntezy niektórych aminokwasów, a nadprodukcję pożądanego. Mutacja genu regulatorowego może spowodować stałe ograniczenie syntezy określonych enzymów uczestniczących w syntezie danego aminokwasu, a mutacja genu strukturalnego powoduje, że drobnoustrój staje się niewrażliwy na nadmiar danego aminokwasu i nie ogranicza jego syntezy.
dodanie do pożywki określonego prekursora np. dodanie do pożywki L-homoseryny zwiększa biosyntezę L-treoniny przez Bacillus subtilis lub Proteus rettgeri.
dodaje się związki powodujące zwiększenie przepuszczalności błony cytoplazmatycznej, a tym samym nadprodukcję pożądanego aminokwasu.
c)przykłady aminokwasów, mikroorganizmów i zastosowanie
alanina- Pseudomonas damuhae- wzmacniacz smaku, piekarstwo
kwas asparaginowy- E.coli
L-cysteina- Enterobacter aerogenes- chleb
Kwas glutaminowy- Corynobacterium glutaminum- wzmacniacz smaku
Treonina- Brevibacterium, E. coli
d)L-lizyna
L-lizyna determinuje wartość biologiczną białka, a zwłaszcza zwiększa wykorzystanie białek roślinnych zwartych w paszy. Do produkcji L-lizyny w skali przemysłowej używa się bakterii z rodzaju
Corynebacterium
Brevibacterium lactofermenum H15.
Również drożdże są zdolne do syntezy L-lizyny, jednak nie używa się ich w przemysłowej produkcji tego aminokwasu.
Przebieg biosyntezy L-lizyny przez drobnoustroje przedstawia się następująco. Szczawiooctan z cyklu kwasów trikarboksylowych może przez aminotransferazę glutaminianową ulec przemianie w L-asparaginian, z którego kinaza asparaginowa katalizuje powstanie semialdehydu beta-asparaginowego. Dehydrogenaza homoserynowa z semialdehydu beta-asparaginowego jest pierwszym enzymem szlaku warunkującym powstanie L-homoseryny, a następnie L-metioniny, L-treoniny lub L-izoleucyny. Enzymem decydującym o syntezie L-lizyny z semialdehydu jest syntetaza dihydrodipikolinianowa.
kinaza
Szczawioocntan L-asparaginian asparaginowa semialdehydu beta-
dehydrogenaza syntetaza
asparaginowego homoserynowa L-homoseryna dihydropikolinianowa L-lizyna
Aktywność kinazy asparaginowej można w 90% zahamować nadmiarem L-lizyny i L-treoniny, natomiast L-izoleucyna i L-walina zwiększają jej aktywność o 50%, znosząc jednocześnie hamujące działanie L-lizyny i L-treoniny.
Aktywność dehydrogenazy homoserynowej jest kilkunastokrotnie większa od aktywności syntetazy dihydrodipikolianowej, co preferuje syntezę L-metioniny, L-treoniny i L-izoleucyny. Dlatego w biosyntezie mikrobiologicznej L-lizyny używa się mutantów auksotroficznych, które nie syntetyzują dehydrogenazy homoserynowej lub mutantów regulatorowych, których kinaza asparaginowa jest niewrażliwa na obecność analogu L-lizyny, tj. S-(2-aminoetylu)-L-cysteiny i L-treoniny. Najkorzystniej jest używać drobnoustrojów wykazujących te dwa rodzaje mutacji.
Źródłem węgla w hodowli bakterii może być: melasa z buraka cukrowego, melasa z trzciny cukrowej, glukoza, alkohol etylowy, kwas octowy, n-parafiny, a źródłem azotu: sole amonowe, hydrolizat białek sojowych. Do pożywki dodaje się L-homoserynę lub L-treoninę i L-metioninę. Aby ograniczyć syntezę kwasu L-glutaminowego, do pożywki dodaje się ponad 30 mikrogramów / dm3 biotyny. Jeżeli składnikiem pożywki jest melasa z trzciny cukrowej, to biotyny się nie dodaje. Kwasowość pożywki (pH 6,9-7) utrzymuje się używając amoniaku. Czas napowietrzanej hodowli w temperaturze 28 - 33 stopni Celsjusza wynosi do 48 h. Wydajność syntezy L-lizyny w zależności od użytego drobnoustroju i źródła węgla w pożywce np.:
C. glutamicum, glukoza - 39 g/dm3 ;
B. flavum, kwas octowy - 75 g/dm3.
Po zakończeniu hodowli płyn pohodowlany, zawierający oprócz L-lizyny wiele innych cennych składników, zagęszcza się do 40% s.s., otrzymując preparat paszowy L-lizyny. W celu otrzymania koncentratu L-lizyny oczyszczony płyn pohodowlany zakwasza się kwasem solnym, otrzymując chlorowodorek L-lizyny, który adsorbuje się na kationowej kolumnie jonowymiennej. L-lizynę wymywa się amoniakiem i ponownie zakwasza kwasem solnym. Po zagęszczeniu roztwór suszy się, uzyskując 70-proc. koncentrat L-lizyny lub poddaje krystalizacji i po rozpuszczeniu oraz ponownej krystalizacji otrzymuje się 97 - 98-proc. koncentrat.
e)Treonina
Do produkcji treoniny używa się specjalnych szczepów Escherichia coli, które są przykładem racjonalnego projektowania organizmów używanych w przemyśle. Niestety E. coli posiada dużo bardziej skomplikowany szlak syntezy treoniny niż C. glutamicum i podlega on regulacji na wielu etapach.
szlak syntezy treoniny Escherichia coli:
hamowany na zasadzie ujemnego sprzężenia zwrotnego przez treoninę, lizynę, metioninę i izoleucynę.
część enzymów zaangażowanych w ten szlak jest kodowanych przez geny w jednym operonie.
Na jego początku znajduje się peptyd z powtarzającymi się treoninami i izoleucynami. W przypadku dużej dostępności tych aminokwasów translacja peptydu zachodzi szybko i w konsekwencji ulega przedwczesnej terminacji.
W przeciwnym razie, gdy panuje niedostatek izoleucyny i treoniny, mRNA jest przepisywane wolniej co skutkuje efektywną syntezą enzymów.
Oprócz samego procesu syntezy u E. coli występuje też szereg innych mechanizmów regulacji, takich jak: enzymy rozkładające treoninę czy systemy jej transportu.
Przy tak skomplikowanym systemie regulacji zdecydowano się zastosować selekcję szczepów pod względem oporności na analog treoniny. Poskutkowało to powstaniem bakterii częściowo niewrażliwych na hamowanie syntezy tego aminokwasu. Podobnie poczyniono przy selekcji auksotrofów izoleucynowych. Ponadto wprowadzono do bakterii wielokopijne plazmidy z operonem treoninowym co poskutkowało zwiększeniem wydajności produkcji.
Racemizacja, proces powstawania racematu dzięki przekształceniu jednego z enancjomerów lub nierównocząsteczkowej mieszaniny obu enancjomerów. Racemizacja może być wywołana ogrzewaniem lub reakcją chemiczną.
Racemat, odmiana racemiczna, mieszanina równomolowych ilości enancjomerów. Nie wykazuje aktywności optycznej, ponieważ skręcalność obydwu enancjomerów jest wzajemnie równoważona. Racemat można rozdzielić na enancjomery za pomocą optycznie aktywnych reagentów (wykorzystując różnice w rozpuszczalności powstałych izomerów), metodami chromatograficznymi, lub biologicznymi (wykorzystując bakterie, pleśnie). Przykładem racematu jest kwas mlekowy fermentacyjny.
Enancjomery, antymery, izomery zwierciadlane, dawniej zwane antypodami optycznymi, obiekty występujące w dwóch postaciach, które są odbiciami lustrzanymi, nie nakładalnymi na siebie. Enancjomerami mogą być kryształy (np. odmiana β kwarcu) lub cząsteczki stereoizomerów (np. kwas D-winowy i kwas L-winowy). Skręcają one płaszczyznę polaryzacji o ten sam kąt, lecz w przeciwnych kierunkach (odmiany lewoskrętne oznaczane są symbolem (-), prawoskrętne (+)).
23
mRNA
DNA
Wiązania wodorowe
hybryd mRNA- DNA
Wycinanie odcinków przez mRNAazę
Wiązania wodorowe
DNA
mRNA
Aminotransferaza glutaminowa
Wyszukiwarka
Podobne podstrony:
Pytania biotechnologia
Pytania biotechnologia
Kol1 zebrane pytania, Biotechnologia, II rok, Biochemia, Kolokwia
Egzamin - zebrane pytania, Biotechnologia, II rok, Mikrobiologia
PYTANIA, biotechnologia UP Wrocław losowe pierdoły, Biochemia
mikroby pytania, Biotechnologia, Semestr IV, Mikro zwierz
pytanianaegzamin, Biotech PWr I stopien, chemia fiz wykład Komorowski
pytania 1, biotechnologia Sem 5 Olsztyn, III rok, III rok BARDZO DOBRE !!!!
pytania z biotechnologii
biochemia pytania biotechnologia
pytania biotechnologia 25str, biotechnologia
pytania biotechnologia 10str, biotechnologia
Pytania biotechnologia
pytania biotechnol
więcej podobnych podstron