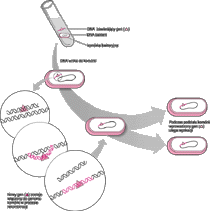
Transformacja Escherichia coli plazmidowym DNA
Transformacja
Transformacja bakteryjna |
|
ransformacja bakteryjna polega na zmianie genomu komórkowego na skutek pobrania cząsteczek DNA z pożywki.
Transformacja bakterii: DNA wprowadza do komórki nową informację genetyczną
Metodę tę zastosowano przy pierwszych udanych próbach wprowadzenia obcego genu do bakterii. Właśnie to odkrycie stanowiło dowód, że DNA jest nośnikiem informacji genetycznej. W praktyce, gdy populacja komórek z pewnym defektem genetycznym (na przykład niezdolnością do syntezy aminokwasu tryptofanu, metabolizowania cukru laktozy czy też syntezy jednej z cząsteczek występujących na powierzchni komórki) są wystawiane na działanie DNA wydzielonego z komórek, mogących wykonywać tę funkcję, niektóre z komórek uzyskują zdolności charakterystyczne dla dawcy DNA. Transformacja taka jest trwała i dziedziczona przez komórki potomne, ponieważ nowo nabyty funkcjonalny gen staje się częścią genomowego DNA komórek biorcy. Transformacja bakterii okazała się niemal nieprzydatna w początkowych fazach badań nad genetyką Prokaryota dla kilku przyczyn, które są w tym miejscu mało istotne. Obecnie proces transformacji nabrał wielkiego znaczenia i jest kluczowym etapem wielu doświadczeń ze zrekombinowanym DNA. Termin „transformacja” stosuje się powszechnie dla określenia trwałej zmiany zapisu genetycznego komórki i jego wpływu na cechy komórki tak u Prokaryota, jak i Eukaryota. Opracowano techniki ułatwiające wprowadzanie plazmidowego DNA do określonych komórek bakteryjnych bezpośrednio z otaczającego je roztworu. Dysponując takimi metodami, można pojedyncze komórki transformować zrekombinowanym plazmidowym DNA, a następnie wyhodować pojedyncze kolonie zawierające nowe sklonowane cząsteczki zrekombinowanego DNA. Należy zaznaczyć, że ponieważ każda komórka gospodarza pobiera jeden plazmid, każda kolonia ma tylko jeden rodzaj zrekombinowanego plazmidu. Ponieważ wiele wektorów plazmidowych ma zaledwie kilka tysięcy par zasad, wzbogacenie ich w nowy gen, fragment eukariotycznego DNA jest jeszcze znaczniejsze niż w przypadku wektorów fagowych. Plazmidy były pierwszymi wektorami, jakie wykorzystano do molekularnego klonowania DNA w bakteriach. |
Wybrane fragmenty pochodzą z książki
Język genów autorstwa Paul'a Berg'a i Maxine Singer
Za transformację odpowiedzialne są :
sygnałowe sekwencje pobierania (ang. uptake-signal sequences) na chromosomie w wielu kopiach np. u Haemophilus influenzae i Nesseria gonorrhoeae
obecność receptorów na powierzchni np. Bacillus subtilis, Streptococcus pneumoniae komórki;
Kompetencja jest to zdolność do wiązania i pobierania DNA
Kompetencja może być konstytutywna (np. Nesseria gonorrhoeae) lub indukowana:
przez warunki wpływające hamująco na wzrost np. kompetencje wzmaga wysoki poziom cAMP
warunki żywieniowe (np. Bacillus subtilis, Streptococcus pneumoniae )
gęstość populacji bakteryjnej tzw. Quorum sensing, czyli regulacja przez „wyczuwanie liczebności”
u Streptococcus pneumoniae kompetencja jest także związana z procesem transbłonowego transportu wapnia
Wysoka wydajność naturalnej transformacji występuje tylko u kilku bakterii: Acinetobacter, Azotobacter, Bacillus, Streptococcus, Haemophilus, Nesseria, Thermus
Wiele prokariotów jest słabo transformowalna bądź wcale w naturalnych warunkach
Kompetencja indukowalna laboratoryjnie
Elektroporacja w polu elektrycznym - ok. 16kV/cm przez ułamek sekundy - transformacja E.coli przez plazmidowy DNA
Przygotowanie komórek kompetentnych.
Hodowlę nocną szczepu E.coli DH5α i TOP10F' odmładza się w pożywce LB (1:50) przez wytrząsanie w 37ºC do uzyskania OD600= 0,4.
Hodowlę odwirowuje się (5 tys. obr/ min., 15 min.)Osad zawiesza w 50 ml zimnego
0,1M CaCl2 i pozostawia na 30 minut w lodzie. Zawiesinę bakterii osadza się przez wirowanie
(5 tys. obr./ min., 10 min.,4ºC).
Osad bakteryjny zawiesza się w 100 ml mieszaniny 0,1M CaCl2 z 10% glicerolem i inkubuje w
lodzie przez 30 minut. Komórki kompetentne przechowuje się w 70ºC.
Przebieg transformacji
Do zawiesiny komórek kompetentnych dodać DNA (odpowiednio do zaleceń), zamieszać i inkubować w lodzie przez 30 min.
Umieścić zawiesinę bakterii z DNA w łaźni wodnej o temp. 42ºC na 2 min.
Dodać 1 ml pożywki LB ogrzanej do temp. 37ºC i inkubować 30 min, w celu ekspresji oporności na antybiotyk. Mieszaninę transformacyjną wysiać na płytki po 0.1 ml i wetrzeć głaszczką. Równocześnie nastawić kontrolę czystości szczepu tj. komórki bez DNA plazmidowego.
Materiały:
TOP10F'
lacIq Tn 10 (TetR), mcrAΔ(mrr- hsd RMS- mcr BC) φ80' lacZΔ15, ΔlacX74 deo R recA 1 ava D139 Δ (ava - leu) F697 galU galK rspL (strR) end A1nup G
E.coli DH5α (F- φ 80▲lacZM15) recA1 endA1gyrA96thi1 hsdR17 (r-,m-) supE44relA1 deoR▲(lacZYA-argF)U169
Plazmidy pUC19(2683 pz), pACYC pBADDraD (~5000pz), pCC90DraCmut (~12 000 pz).
Komórki kompetentne szczepów E.coli DH5α oraz TOP10F'
Płytki z antybiotykami: LA z Ampicyliną
Płytki z LA
Lód
TE
Ćwiczenie 1.
Komórki kompetentne szczepów bakteryjnych DH5α oraz TOP10F' - badanie podatności szczepów na transformację plazmidem pUC19 (stężony [260 ng/μl] i rozcieńczony 10-1 [26 ng/μl]). Wysiać na płytki mieszaniny transformacyjne po 0.1 ml.
Kontrola biorcy - wysiać po 0.1 ml bakterii z próby kontrolnej (bez DNA) na podłoże z antybiotykiem;
Obliczyć liczbę transformantów w obu przypadkach. Który ze szczepów jest bardziej podatny na transformację.
Ćwiczenie 2
Badanie wpływu wielkości plazmidu na wydajność transformacji
Plazmidy natywne stężone i rozcieńczone 10-1: pUC19 (2683 pz), pACYC pBADDraD (5000pz), pCC90DraCmut (12 000 pz).
pUC19 stężony 260 ng/ μl; pACYCpBADDraD 280 ng/ μl; pCC90DraCmut 250 ng/ μl
Do jednej probówki dodać 100μl komórek kompetentnych TOP10F' i plazmidowe DNA (1 μl )stężone; do drugiej probówki dodać 100μl komórek kompetentnych TOP10F' i plazmidowe DNA rozcieńczone. Dla plazmidu pCC90DraCmut zrobić również transformację większym stężeniem tj. dodać10 μl plazmidu o stężeniu 0.77μg/ μl (jedno na całą grupę). Na płytki wysiewać po 0.1 ml mieszaniny transformacyjnej.
Ćwiczenie 3
Obliczanie wydajności transformacji
Dla TOP10F' + pUC19 obliczyć wydajność transformacji, w tym celu na płytki z LA+Ap wysiać po 0.1 ml mieszaniny transformacyjnej bez rozcieńczania i rozcieńczoną 10-1, 10-2
Policzyć wyrosłe kolonie, określić częstość transformacji komórek plazmidowym DNA.
Wydajność transformacji = miano transformantów/miano biorcy x 100%
Kontrola miana biorcy: komórki kompetentne rozcieńczyć (0.1 ml komórek + 0.9 ml soli fizjologicznej) 10 -6, 10 -7, 10 -8, 10-9 i wysiać po 0.1 ml na płytki bez antybiotyku. Obliczyć miano biorcy.
Ćwiczenie 4
Badanie wpływu stężenia DNA na wydajność transformacji; do transformacji użyć różne stężenia DNA:
bez rozcieńczenia, rozcieńczone 10-1, 10-2, 10-3, 10-4, 10-5, 10-6 i zbadać czy ma ono wpływ na wydajność transformacji. Obliczyć liczbę transformantów w stosunku do stężenia DNA użytego do transformacji. Narysować wykres, w którym momencie stężenie DNA nie będzie miało już wpływu na wydajność transformacji. Patrz wykres przykładowy poniżej.
Figure 9-12. The genetic transfer of streptomycin-resistance (strr) to streptomycin-sensitivity (strs) cells of E. coli. The recovery of strr transformants among strs cells depends on the concentration of strr DNA. (From G. S. Stent and R. Calendar, Molecular Genetics, 2d ed. Copyright © 1978 by W. H. Freeman and Company, New Yor
Ćwiczenie 5
Badanie wpływu konformacji plazmidu na wydajność transformacji: plazmid pUC19 trawiony do formy liniowej (260 ng); do transformacji brać 10 μl mieszaniny restrykcyjnej Wysiać 100 μl mieszaniny transformacyjnej.
Porównać wydajność transformacji w przypadku formy CCC (z poprzedniego ćwiczenia) i formy L.
Wyszukiwarka
Podobne podstrony:
T7 Transformacja układu odniesienia
11 BIOCHEMIA horyzontalny transfer genów
Transformacje91
5 Algorytmy wyznaczania dyskretnej transformaty Fouriera (CPS)
Weterynaria sądowa
inspekacja weterynaryjna
11Tor z transformatoramiid 13123 ppt
Transformacje2
20 H16 POST TRANSFUSION COMPLICATIONS KD 1st part PL
Immunologia Transfuzjologiczna1[1]
3 Rodzaje jednorodnych transformacji stosowanych w kinematy
Transfer sk adki US
Badanie transformatora
Efficient VLSI architectures for the biorthogonal wavelet transform by filter bank and lifting sc
6 Miedzynarodowy transfer wyklad 11 04 2012 id 43355
500 GB Free Transferu
Ćwiczenie T1 Transformator trójfazowy, t1 f
więcej podobnych podstron