Genetyka zwierz
ą
t
Hodowla zwierz
ą
t II rok
Wykład 11 (15.12.2009 – 5.01.2010)
Markery genetyczne w hodowli zwierz
ą
t
dr hab. Dorota Cie
ś
lak, prof. nadzw
Marker genetyczny - definicja
dowolna cecha jako
ś
ciowa, uwarunkowana w sposób prosty
łatwa do kontroli i przydatna do celów analizy genetycznej

3
Marker genetyczny - charakterystyka
Polimorfizm
– im wi
ę
ksza liczba alleli w locus danego
markera tym wi
ę
ksza jego przydatno
ść
do analizy (układ
homozygotyczny – stwierdzenie zaj
ś
cia c/o jest niemo
ż
liwe)
Wysoki udział osobników heterozygotycznych
– je
ż
eli
dominuje jeden z wielu alleli w danym locus
(np. frekwencja powy
ż
ej 0.9) wówczas mimo du
ż
ego
polimorfizmu locus to jest mało informatywne
Mutacje
– cz
ę
sto
ść
mutacji w locus markera powinna by
ć
bardzo niska;
Rodzaje
markerów
genetycznych
klasa I
sekwencje koduj
ą
ce –
analiza produktów genów
lub sekwencji DNA genów
klasa II
sekwencje niekoduj
ą
ce –
analiza sekwencji DNA
głównie powtarzalne sekwencje
mikrosatelitarne
, w mniejszym
stopniu minisatelitarne

klasa I
sekwencje koduj
ą
ce –
analiza sekwencji DNA genów
lub produktów genów

Markery genetyczne klasy I (sekwencje
koduj
ą
ce = geny)
– antygeny erytrocytarne (grupy krwi)
– polimorficzne białka (surowicy krwi, mleka,
jaja itp.)
– antygeny głównego kompleksu zgodno
ś
ci
tkankowej

Grupy krwi
• W te
ś
cie serologicznym wykrywana jest
determinanta antygenowa
– surowica testowa zawiera specyficzne
przeciwciało rozpoznaj
ą
ce dany antygen
– reakcja „antygen - przeciwciało” aglutynacja
lub hemoliza
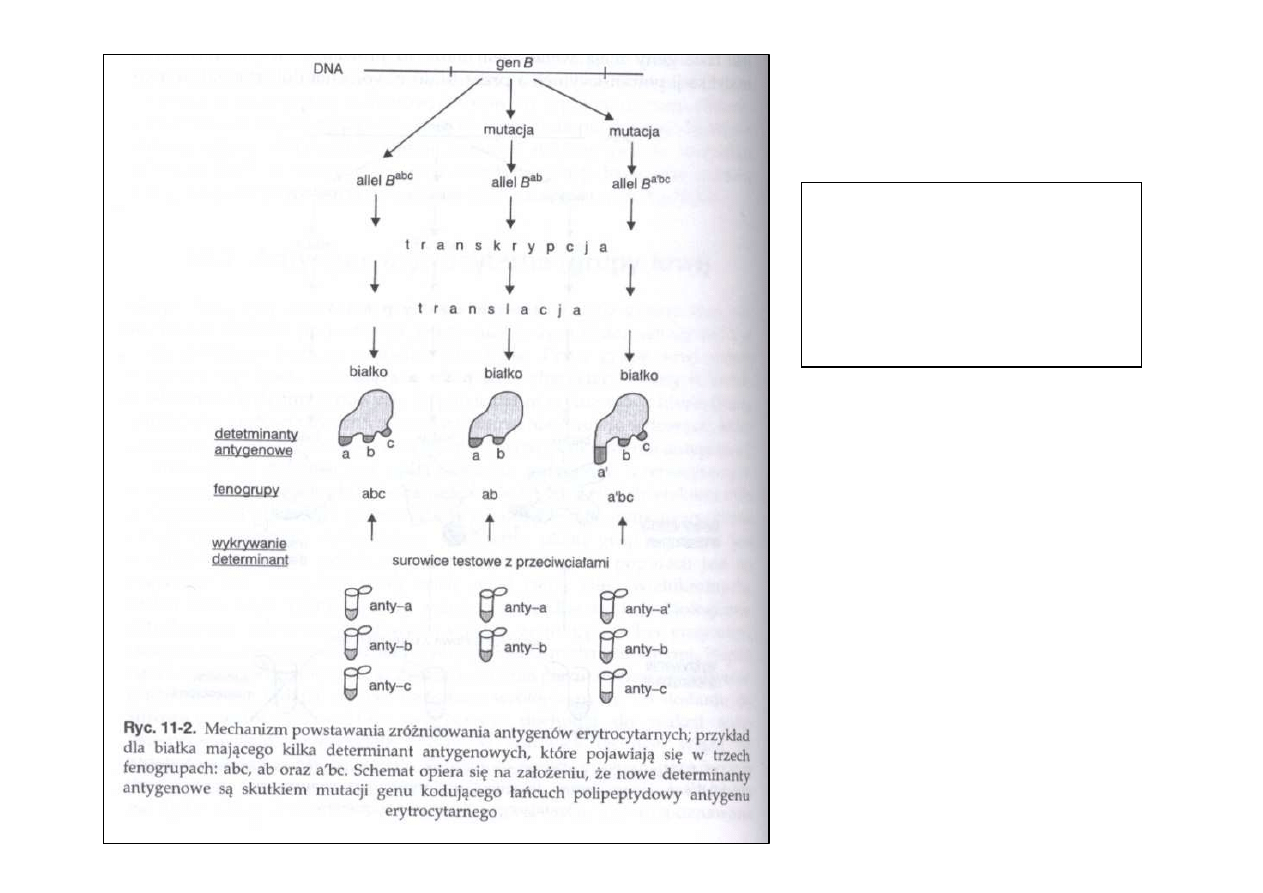
Genetyczne
podło
ż
e
polimorfizmu
erytrocytarnego
Charon &
Ś
wito
ń
ski 2004

• białko mo
ż
e mie
ć
jedn
ą
lub wi
ę
cej determinant
antygenowych
• ró
ż
ne formy determinant antygenowych s
ą
wynikiem
mutacji punktowych w genach je koduj
ą
cych
• allel mo
ż
e by
ć
odpowiedzialny za powstanie białka o
kilku determinantach antygenowych

Układy grupowe krwi
zwierz
ą
t gospodarskich
• Bydło
11
83
1122
•
Ś
winia
15
76
76
• Ko
ń
7
34
56
• Kura domowa
13
Liczba
Układów
antygenów
alleli

Białka surowicy krwi, erytrocytów,
leukocytów
• druga co do wielko
ś
ci grupa markerów klasy I
• polimorfizm wynika ze zmienno
ś
ci form strukturalnych
białek (mutacje punktowe w genie koduj
ą
cym białko lub
enzym odpowiedzialny za modyfikacje potranslacyjne)
• najbardziej polimorficzne białka np. inhibitor
α
-proteaz
konia – 20 alleli

Polimorfizm markerów genetycznych – metody
detekcji
• Grupy krwi
– test serologiczny (surowice testowe)
• Polimorfizm białek
– elektroforeza
• Polimorfizm DNA
– elektroforeza (sekwencje mikrosatelitarne)
– hybrydyzacja ze znakowan
ą
sond
ą
(sekwencje minisatelitarne - tzw. „odcisk
palca DNA”)
Polimorfizm
białek mleka
Kazeiny
np. w locus
α
s1 wykryto
5 alleli
białko serwatki
β
-laktoglobulina
DNA
–
analiza
niezale
ż
na od
płci, wieku,
stanu
fizjologicznego
białko –
analiza mleka
(wył
ą
cznie u
samic podczas
laktacji)
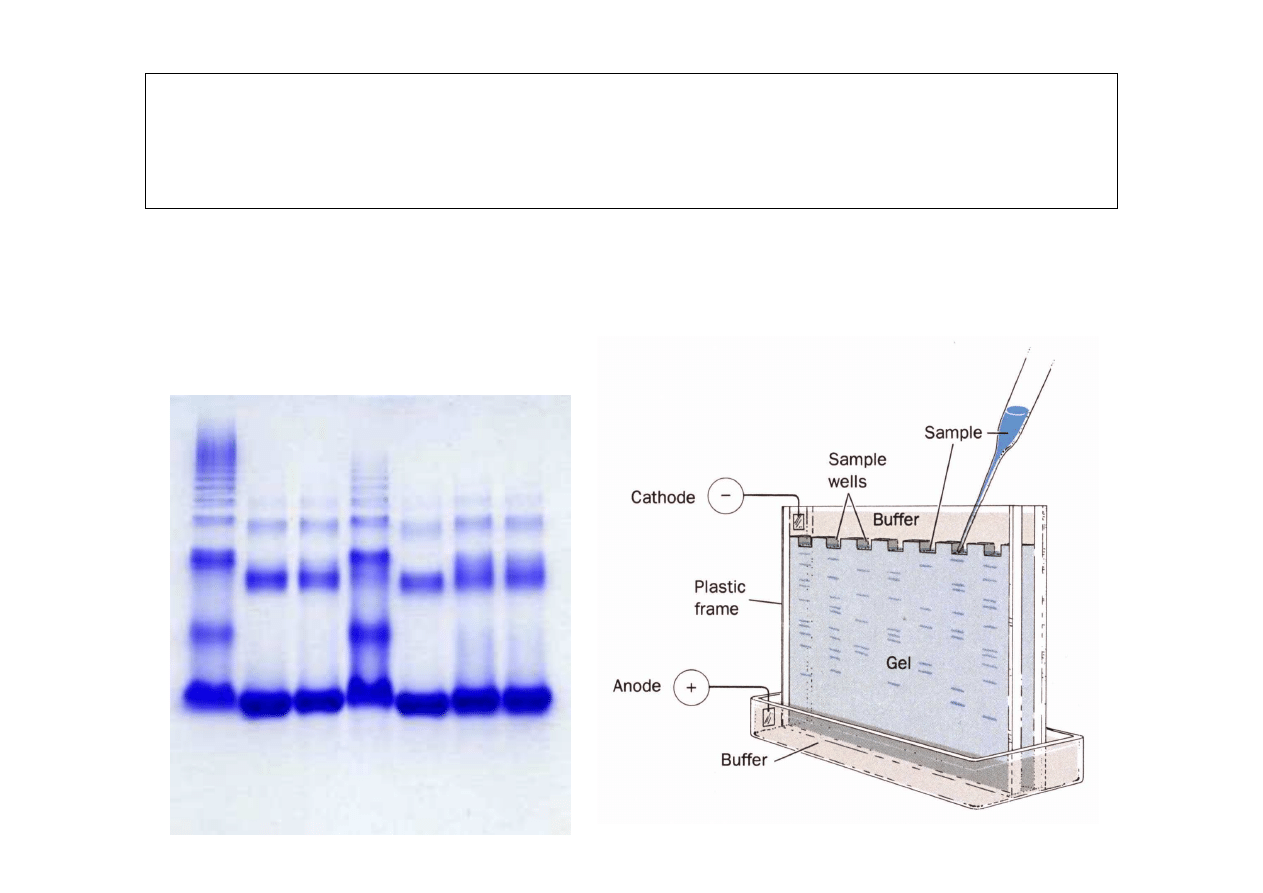
Identyfikacja polimorfizmu białek przy pomocy
elektroforezy
•
rozdział w stałym polu elektrycznym
•
szybko
ść
przemieszczania zale
ż
y od masy, konformacji
przestrzennej i ładunku

Polimorfizm nukleotydów sekwencji
koduj
ą
cych – genów (markery I klasy)
zmiany pojedynczych nukleotydów -
SNP
(single
nucleotide polymorphism) wykrywane przy u
ż
yciu
endonukleaz – enzymów restrykcyjnych technik
ą
RFLP (polimorfizm długo
ś
ci fragmentów
restrykcyjnych)

RFLP
(polimorfizm długo
ś
ci fragmentów restrykcyjnych)
• Zasada:
– specyficzno
ść
trawienia przez enzym restrykcyjny zale
ż
y
od tego, czy w danej sekwencji nukleotydów nie wyst
ą
piła
mutacja
• Cel:
– identyfikacja mutacji punktowej w obr
ę
bie sekwencji
rozpoznawanej przez okre
ś
lony enzym restrykcyjny
• Etapy:
– amplifikacja fragmentu DNA (technika PCR)
– trawienie DNA wybranym enzymem restrykcyjnym
– elektroforeza fragmentów DNA

Enzymy restrykcyjne (endonukleazy)
Pochodz
ą
z komórek bakteryjnych, w których
słu
żą
do obrony przed obcym DNA (np.
bakteriofagów)
Rozcinaj
ą
cz
ą
steczk
ę
DNA po rozpoznaniu
charakterystycznej dla danego enzymu
sekwencji nukleotydów

Trawienie DNA przez endonukleazy
enzym rozpoznaje sekwencj
ę
CGCG i rozcina j
ą
mi
ę
dzy
zasadami G i C
np. enzym Tha I
sekwencja niezmieniona – enzym rozpoznaje sekwencj
ę
i tnie ła
ń
cuch
5` …CGCG…. 3`
5`..CG CG..3`
3` …GCGC…. 5`
3`..GC GC..5`
sekwencja zmutowana – enzym nie rozpoznaje sekwencji, brak ci
ę
cia
5` …CG
A
G…. 3`
3` …GCGC…. 5`

Mutacja punktowa (zmiana sekwencji nukleotydów)
mo
ż
e spowodowa
ć
1.
zanik istniej
ą
cego miejsca restrykcyjnego
(rozpoznawanego wcze
ś
niej przez dany enzym)
2. pojawienie si
ę
nowego miejsca restrykcyjnego
W zale
ż
no
ś
ci od sytuacji powstaje nowy układ mo
ż
liwy
do identyfikacji przy u
ż
yciu enzymu restrykcyjnego, a co
wa
ż
niejsze ró
ż
nicuj
ą
cy osobniki w populacji
Rozdzia
ł
elektroforetyczny produktu PCR (659 pz) dla
genu RYR1; M – marker wielko
ś
ci GeneRuler™ 50 bp
DNA
600pz
M
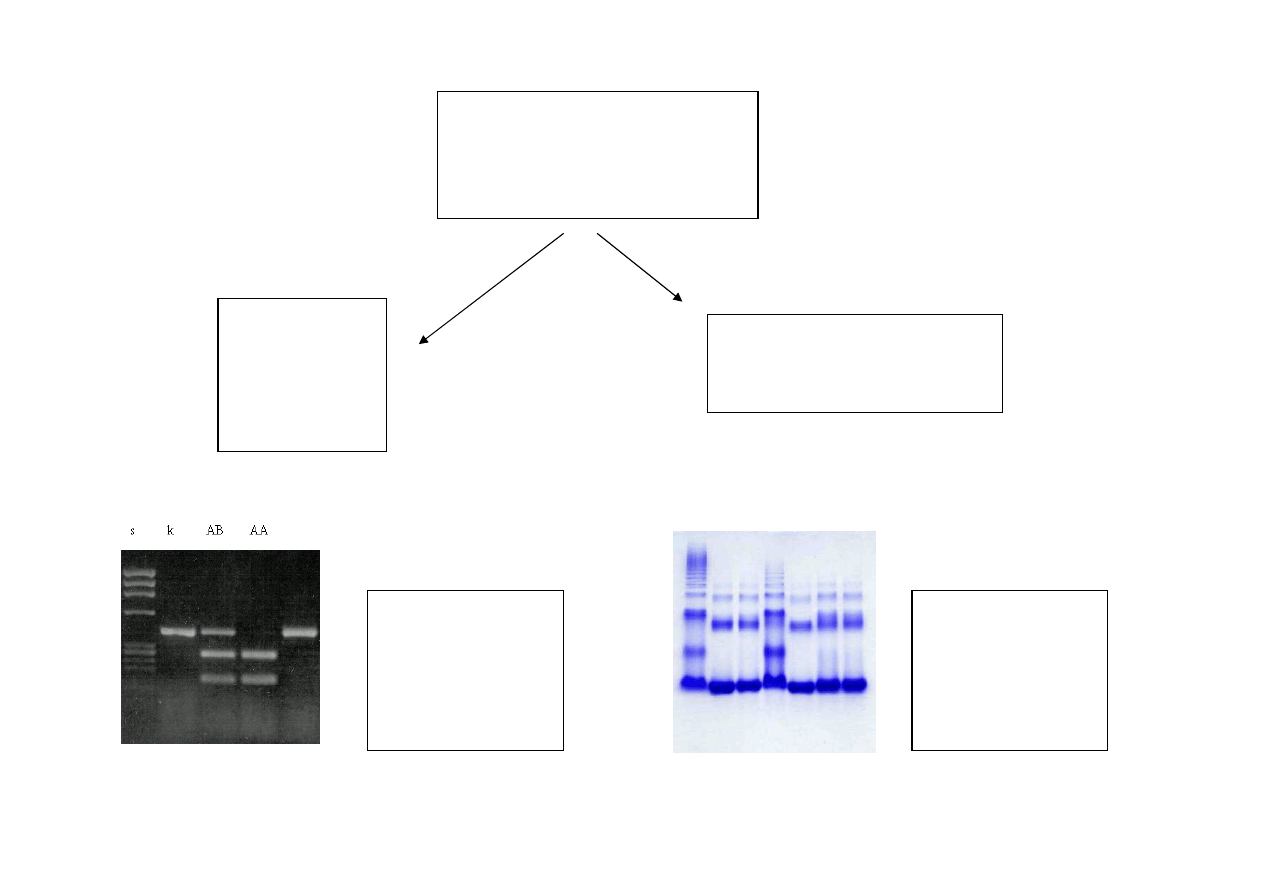
Test molekularny (PCR-RFLP)
Analiza restrykcyjna produktów PCR dla genu receptora
rianodyny (
ś
winia domowa)
M – marker wielko
ś
ci DNA GeneRuler™ 50 bp.
Ś
cie
ż
ki 1 – produkt PCR (659
pz), 2 i 7 – genotyp CC (524 pz, 135 pz), 2 – genotyp CT (524 pz, 358 pz, 166
pz, 135 pz), 4, 5 i 6 – genotyp TT (358 pz, 166 pz, 135 pz.(Enzym
restrykcyjny – Alw 21I)
600pz
M
1
2
3
4
5
6
7
524pz
358pz
166pz
135pz

klasa II
sekwencje niekoduj
ą
ce –
głównie powtarzalne
sekwencje
mikrosatelitarne
, w
mniejszym stopniu
minisatelitarne
analiza sekwencji DNA

Polimorfizm (wielopostaciowo
ść
)
sekwencji niekoduj
ą
cych - zmienna
liczba powtórze
ń
tandemowych
(markery klasy II)

Polimorfizm sekwencji powtarzaj
ą
cych si
ę
tandemowo
Sekwencje powtarzaj
ą
ce si
ę
tandemowo
(wielokrotne powtórzenie okre
ś
lonego motywu – sekwencji
DNA, jeden za drugim, w ró
ż
nych miejscach genomu)
– sekwencje
mikrosatelitarne (do 10 nukleotydów
)
– sekwencje
minisatelitarne (> 10 nukleotydów
)

Sekwencje
minisatelitarne
- odcisk
palca DNA (DNA fingerprint)
Polimorfizm jest wykrywany metod
ą
hybrydyzacji
– izolacja całkowitego DNA z komórki
– trawienie genomowego DNA wybranym enzymem restrykcyjnym
– elektroforeza
– hybrydyzacja DNA z wyznakowan
ą
sond
ą
dla danej sekwencji
– detekcja obrazu na kliszy radiograficznej
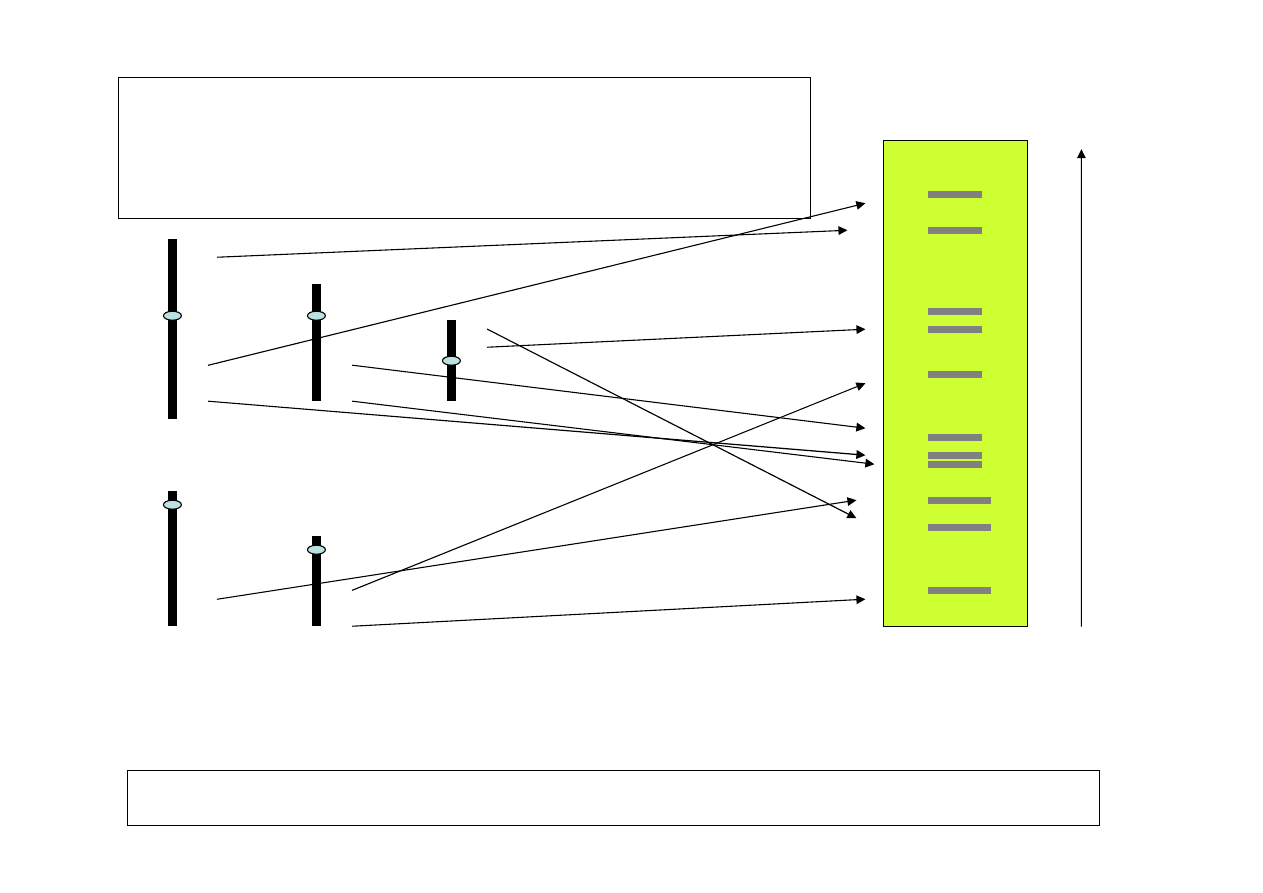
*
sekwencja minisatelitarna : np. GGGAGGTGGGCAGGAGG
*
*
*
*
*
+
_
*
Polimorfizm sekwencji minisatelitarnych -
równoczesna analiza wielu loci - „odcisk
palca DNA”
Elektroforeza DNA + hybrydyzacja z sond
ą
*
*
*
*
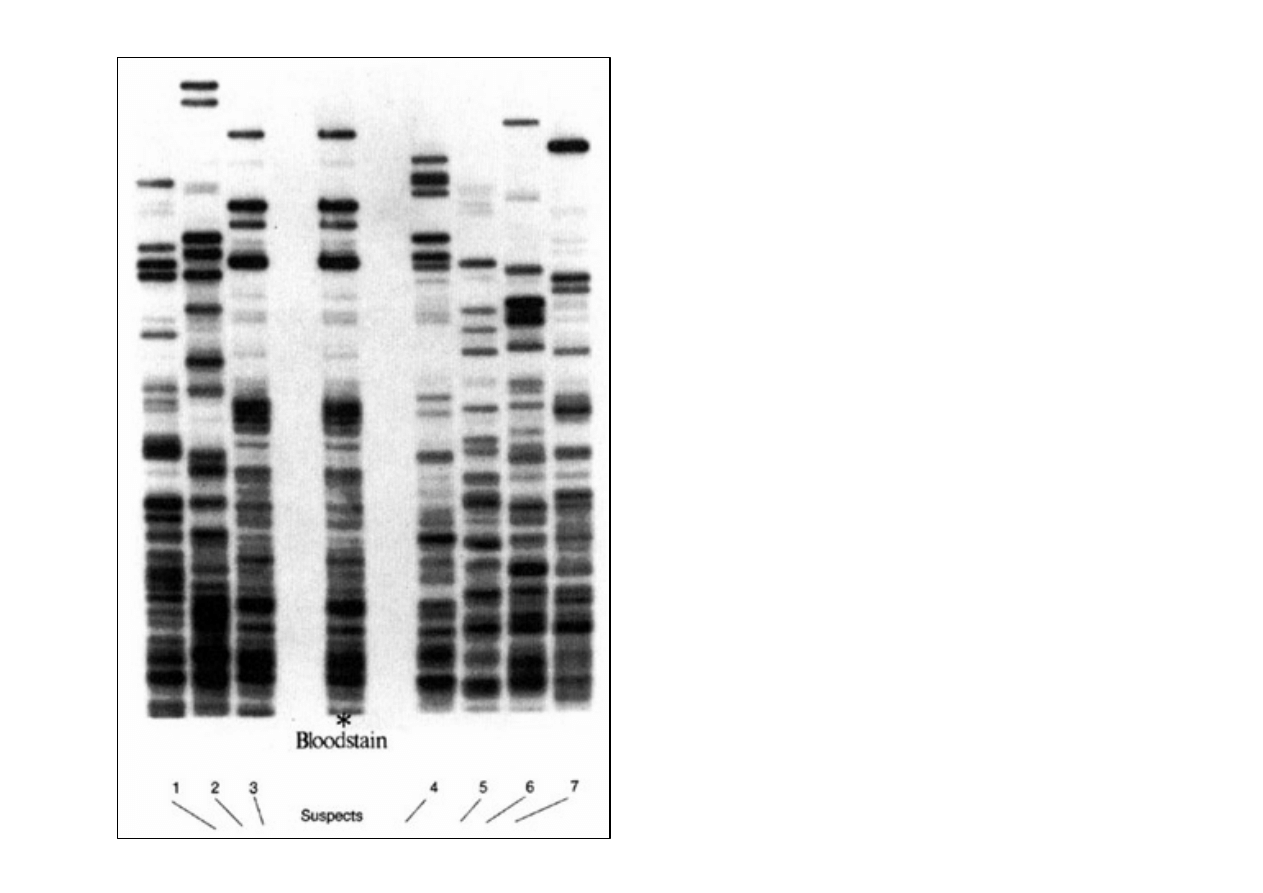
Identyfikacja
ś
ladów
biologicznych
Dowód: plama krwi na
miejscu przest
ę
pstwa
(
bloodstain
)
1-6 profil DNA sze
ś
ciu
podejrzanych

Sekwencje
mikrosatelitarne
• Tandemowe
powtórzenia kilku
nukleotydów (najcz
ęś
ciej 2 - 4)
5’
CACACA
CACACACACA 3’
3’ GTGTGTGTGTGTGTGT 5’
Allel A (CA)
8
5’ CACACACACACACACACACA 3’
3’ GTGTGTGTGTGTGTGTGTGT 5’
Allel B (CA)
10

Sekwencje mikrosatelitarne
• Wyst
ę
puj
ą
w wielu miejscach genomu (w
intronach, eksonach i promotorach genów)
• odró
ż
niane s
ą
od siebie dzi
ę
ki sekwencjom
„flankuj
ą
cym” (ograniczaj
ą
cym), które maj
ą
unikalny układ nukleotydów

Sekwencje flankuj
ą
ce (organiczaj
ą
ce)
sekwencje mikrosatelitarne
…….AACGTTGGATC
CACACACACACACACACACA
TGGTCAAGGGTTAA……

Sekwencje mikrosatelitarne
• najliczniejsza grupa markerów klasy II (wysoki
stopie
ń
polimorfizmu i równomierne rozproszenie
w genomie)
• polimorfizm identyfikuje si
ę
poprzez amplifikacj
ę
odcinka DNA metod
ą
PCR i odczyt wyniku na
ż
elu lub poprzez elektroforez
ę
(zazwyczaj
prowadzona w automatycznym sekwenatorze)

Markery genetyczne w hodowli
zwierz
ą
t

Wykorzystanie markerów
genetycznych w hodowli zwierz
ą
t
• Kontrola pochodzenia
• Identyfikacja markerów sprz
ęż
onych z genami
kontroluj
ą
cymi istotne cechy hodowlane
(markerowe mapy genomu)
• Ocena zmienno
ś
ci genetycznej wewn
ą
trz i mi
ę
dzy
populacjami (dystans genetyczny, poziom hetero-
i homozygotyczno
ś
ci, zmiany zachodz
ą
ce w
strukturze genetycznej populacji)

Kontrola pochodzenia przy pomocy
markerów genetycznych
w fenotypie/genotypie potomka musz
ą
wyst
ą
pi
ć
warianty/alele pochodz
ą
ce
od rodziców, niezgodno
ść
cho
ć
by w jednym locus czyni układ niemo
ż
liwym

Kontrola pochodzenia przy pomocy
markerów genetycznych
• Ustalenie fenotypu osobnika i jego domniemanych
rodziców (układy grupowe krwi z wieloma
antygenami np. B u bydła – 40 antygenów)
• Okre
ś
lenie (na ile to mo
ż
liwe) genotypu osobników
– trudne w przypadku grup krwi (fenogrupy)
– łatwe w przypadku polimorfizmu białek i
sekwencji mikrosatelitarnych DNA
• Porównanie fenotypu i genotypu osobnika z
fenotypami i genotypami domniemanych rodziców
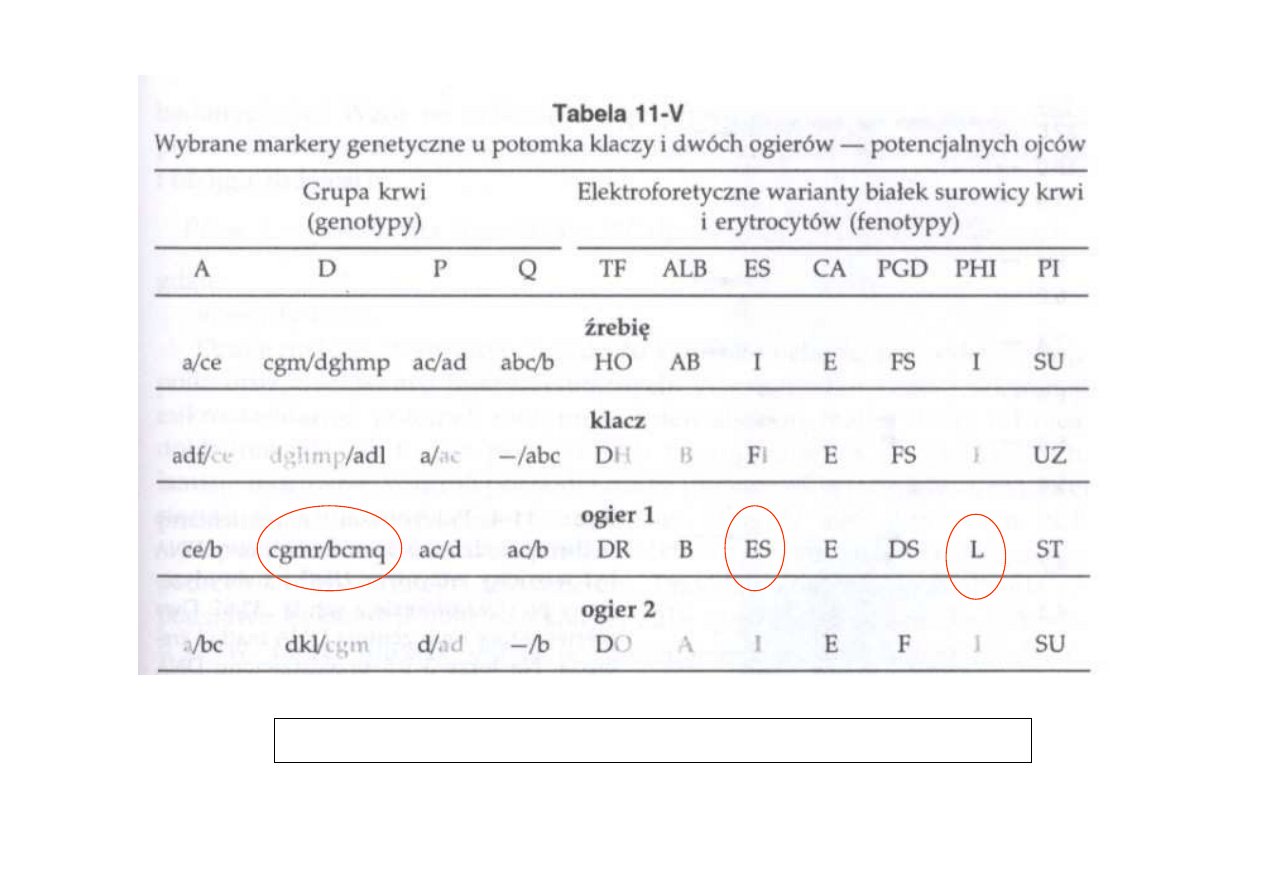
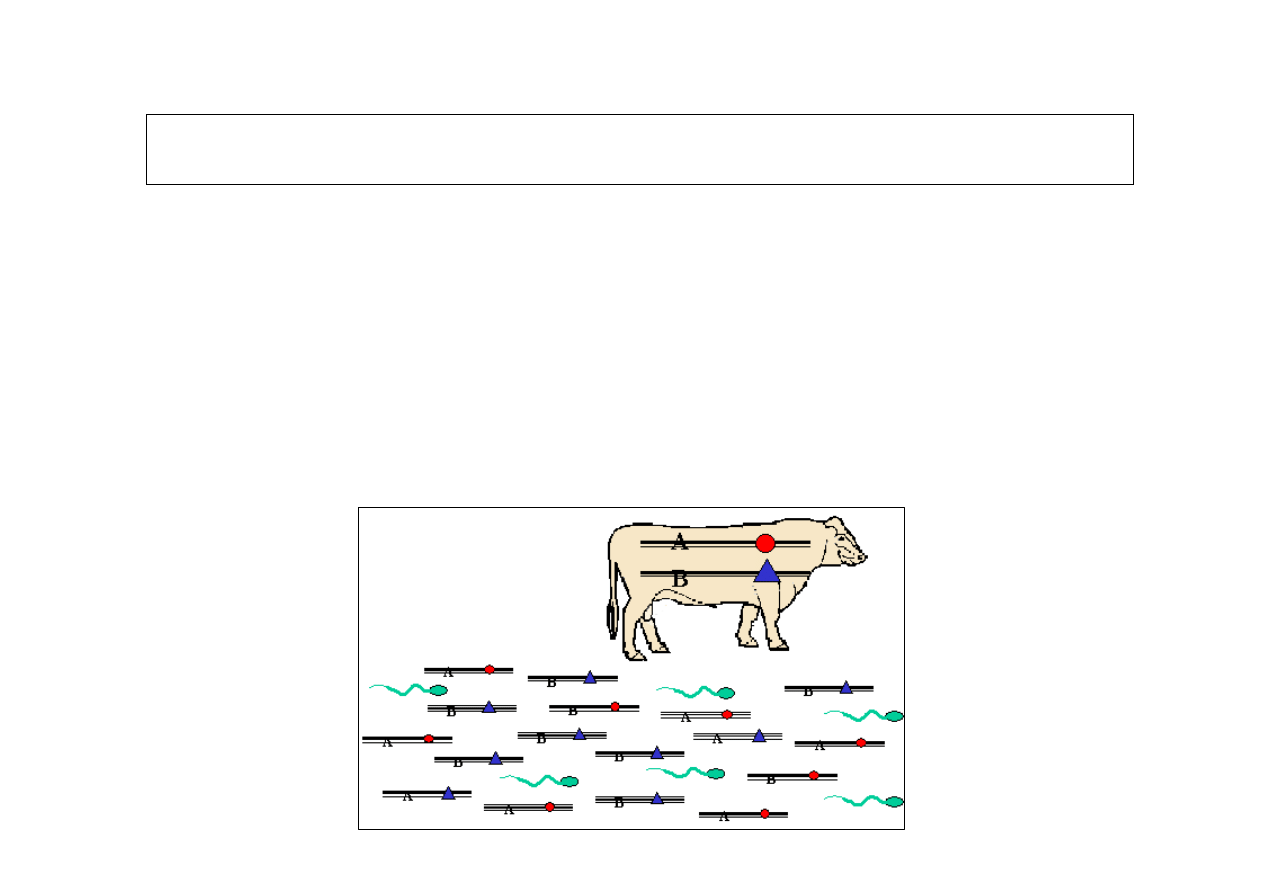
Przykładowe zestawienie danych dot kontroli pochodzenia
Charon & Switonski 2004

Kontrola pochodzenia przy pomocy
markerów genetycznych
• Wykluczenie pochodzenia
:
– je
ś
li w genotypie (fenotypie) potomka wyst
ą
pi
ą
inne allele (lub antygeny - fenotyp) ni
ż
stwierdzone u domniemanych rodziców
• Wyst
ą
pienie zgodno
ś
ci genotypu
(fenotypu) osobnika
z genotypem (fenotypem) domniemanych rodziców
pozwala stwierdzi
ć
,
ż
e
nie ma podstaw do
wykluczenia pochodzenia
z prawdopodobie
ń
stwem:
– ponad 99% - je
ś
li badano ok. 10 polimorficznych
loci mikrosatelitarnych
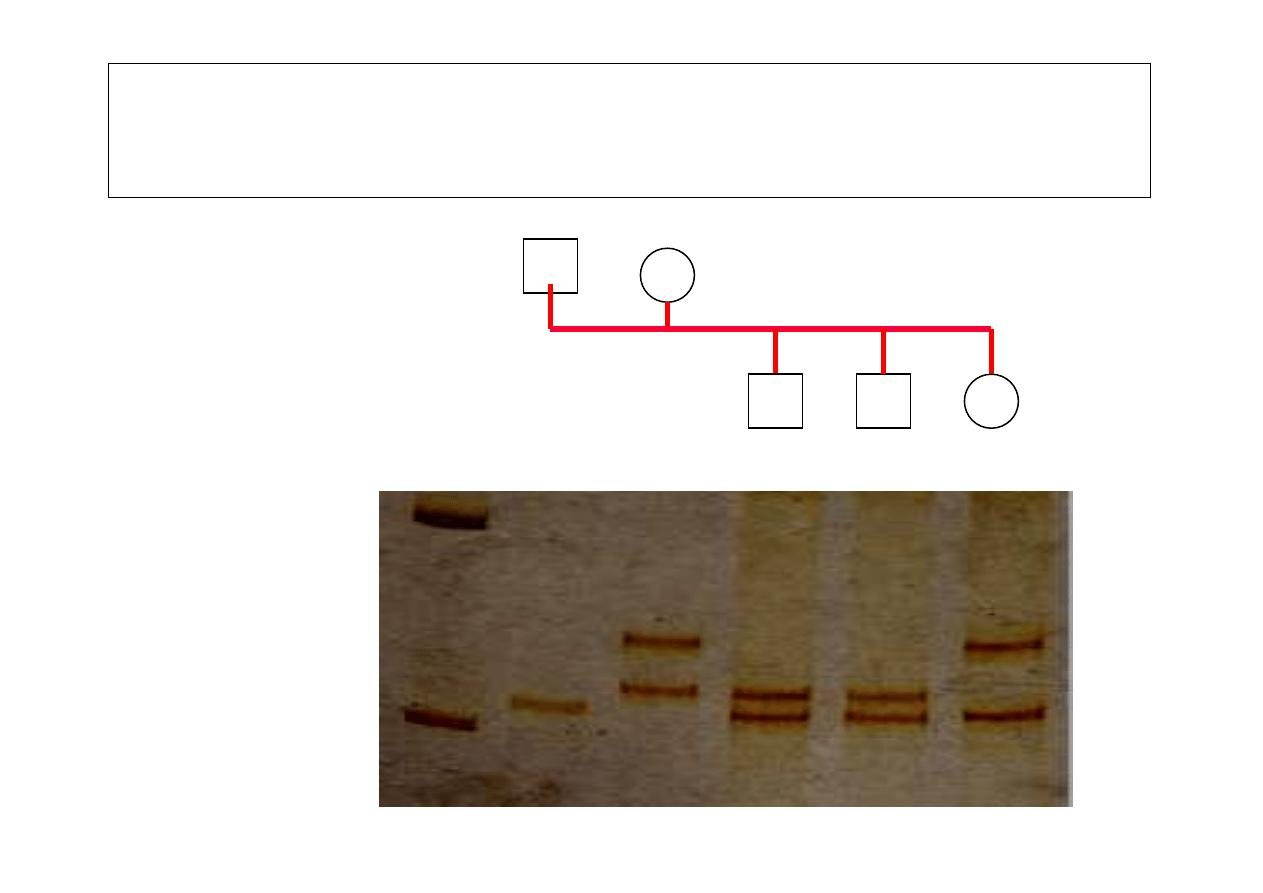
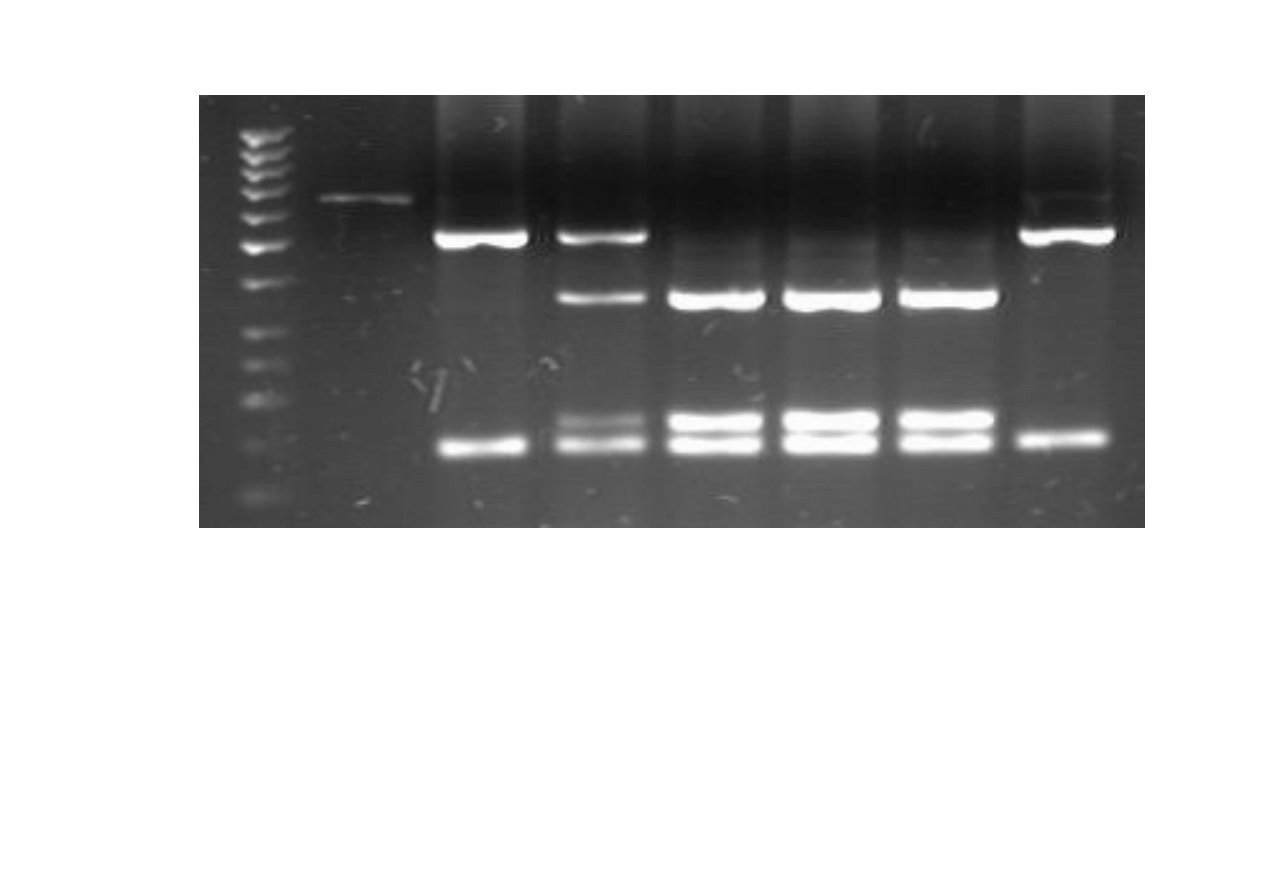
Analiza dziedziczenia alleli sekwencji mikrosatelitarnej
(pozycja 2319) w rodzinie lisa pospolitego
(elektroforeza w
ż
elu poliakrylamidowym)
339pz
220pz
marker
DNA
AA BC AB AB
AC
M
3 allele:
A - 235 pz
B - 243pz
C - 263pz
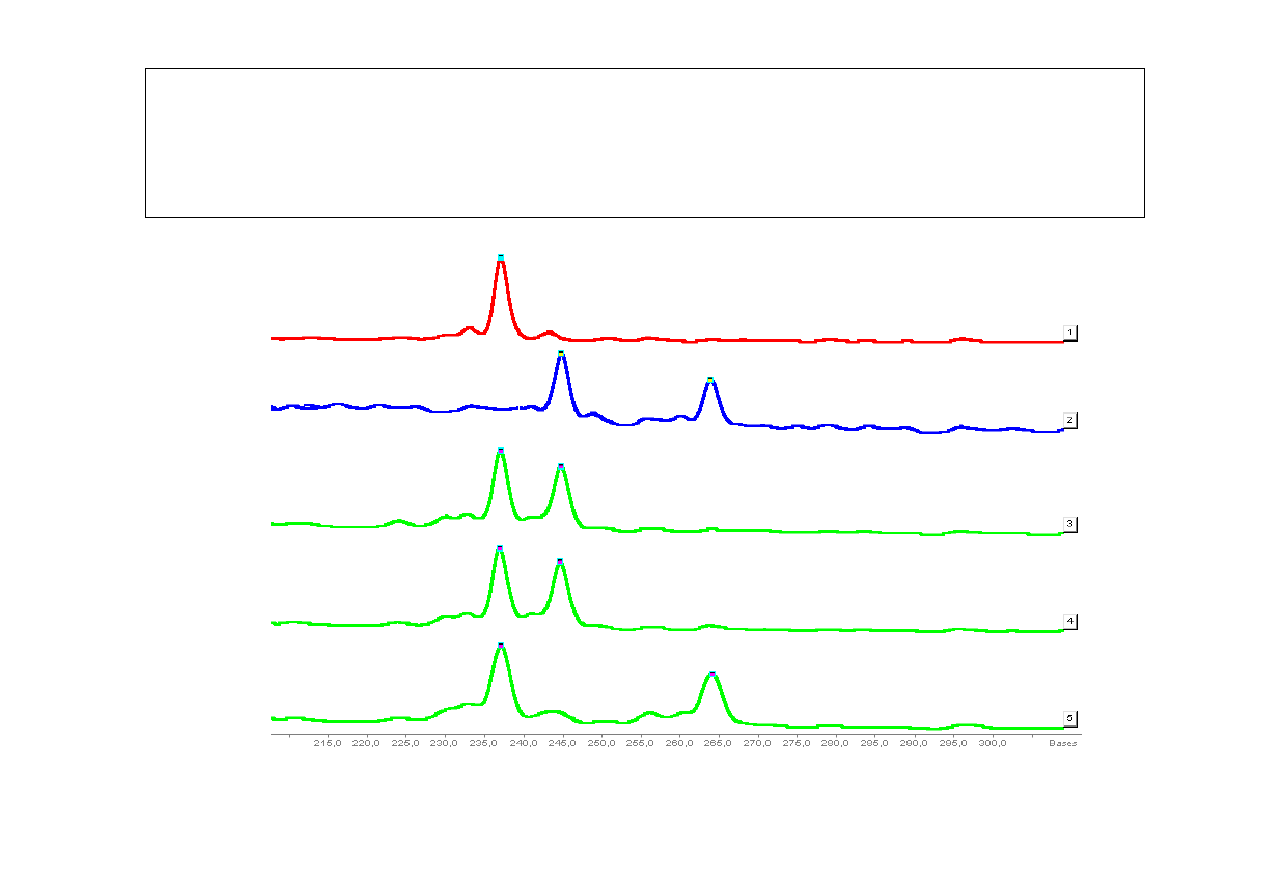
AA
BC
AB
AB
AC
Dziedziczenie alleli sekwencji mikrosatelitarnej
2319 w rodzinie lisa pospolitego
(analiza w automatycznym sekwenatorze)
Wyszukiwarka
Podobne podstrony:
markery genetyczne
Wykorzystanie markerow genetycznych
markery genetyczne
Markery genetyczne
MARKER GENETYCZNY, MARKER GENETYCZNY - dziedziczna cecha genetyczna z łatwo rozpoznawalnymi allelami
markery genetyczne
markery genetyczne
sprawozdanie z genetyki 11, żywienie człowieka i ocena żywności, semestr 4, genetyka
egzamin (11), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
Wykład 11 - Podstawowe pojęcia i terminy genetyczne, Pielęgniarstwo licencjat AWF, Genetyka
inzynieria genetyczna 11
11 Patobiochemia nowotworow markery nowotworowe
WYKŁADY GENETYKA Genetyka diagnostyka pl. 21.11.09r
Ćwiczenie 11, I semestr biotech, genetyka
więcej podobnych podstron