
MARKERY
GENETYCZNE
Marker
genetyczny-
gen
występujący w postaci przynajmniej
dwóch
łatwo
rozróżnialnych
alleli,
którego dziedziczenie można śledzić w
potomstwie krzyżówki, co umożliwia
ustalenie
jego
pozycji
na
mapie
genetycznej.
Markery
genetyczne
dziedziczą się zgodnie z prawami
Mendla.

Klasy markerów:
klasa pierwsza obejmuje klasyczne markery, czyli
sekwencje kodujące- geny. Polimorfizm tych markerów
wykrywany
jest
poprzez
analizę
produktów
genów( metody serologiczne i technika elektroforezy
białek) lub badanie DNA tych genów( metody RFLP,
SSCP). Do pierwszej klasy należą:
Antygeny erytrocytarne (grupy krwi)
Białka surowicy krwi, erytrocytów i leukocytów
Allotypy immunoglobulin i lipoprotein
Białka mleka
klasa druga markerów to sekwencje niekodujące;
wśród nich najważniejsze miejsce zajmują tandemowo
powtarzające się sekwencje mikrosatelitarne, a w
mniejszym stopniu sekwencje minisatelitarne. Ich
identyfikacja prowadzona jest wyłącznie na drodze
elektroforezy.

Oprócz wymienionych wyżej dwóch klas
markerów
genetycznych
można
wyróżnić
jeszcze
grupę
markerów
związanych
z
polimorfizmem chromosomowym. Polimorfizm
chromosomowy dotyczy przede wszystkim
wielkości
bloków
heterochromatyny
konstytutywnej
oraz
obszarów
jąderkotwórczych.

Pierwszymi stosowanymi
markerami były geny
W
pierwszych
mapach
genetycznych,
konstruowanych w pierwszych dekadach XX w. dla
organizmów takich jak muszka owocowa, jako
markerów używano genów. Aby być użytecznym do
analiz genetycznych, gen musi występować w co
najmniej dwóch formach, czyli allelach, każdej
odpowiadającej za inny fenotyp. Na początku
jedynymi genami nadającymi się do badania były
takie które odpowiadały fenotypom rozróżnialnym
wzrokowo. Genetycy szybko jednak zdali sobie
sprawę, że w ten sposób można badać dziedziczenie
zaledwie ograniczonej liczby fenotypów nadających
się do obserwacji wzrokowej. Aby stworzyć bardziej
ogólne mapy genów, konieczne stało się znalezienie
cech lepiej rozróżnialnych i mniej złożonych niż
nadające się do obserwacji wzrokowej. Odpowiedzią
było wykorzystanie biochemii do rozróżniania
fenotypów.

Szczególnie ważne okazało się to u dwóch typów
organizmów-
mikroorganizmów
i
ludzi.
Mikroorganizmy maja niewiele cech dających się
obserwować wzrokowo, więc mapowanie genów tych
organizmów musi się opierać na fenotypach
biochemicznych. Ludzie także mają widoczne cechy
obserwowalne wzrokowo, ale już od lat dwudziestych
badano fenotypy biochemiczne uzyskiwane przez
typowanie krwi.

Klasa pierwsza markerów
Antygeny erytrocytarne
Grupy krwi były najwcześniej wykorzystane w analizie
genetycznej, np. dotyczącej kontroli pochodzenia. Do
pionierów tych badań należy zaliczyć m.in. polskiego
badacza Ludwika Hirszfelda. Przez grupę krwi należy
rozumieć typ krwi, cechujący się obecnością
charakterystycznych
białek,
o
właściwościach
antygenowych,
na
powierzchni
erytrocytów.
Identyfikacja antygenów erytrocytarnych wymaga
zastosowania surowic testowych, które zawierają
swoiste przeciwciała skierowane przeciw konkretnemu
antygenowi
.

Białka surowicy krwi, erytrocytów i
leukocytów
Są drugą, po grupach krwi, pod względem liczebności grupą
markerów genetycznych klasy pierwszej. Poszczególne warianty
tych białek są określane za pomocą technik elektroforetycznych
(np. elektroforeza w żelu poliakryloamidowym lub elektroforeza
dwuwymiarowa czy rozdział białek w gradiencie pH).
Identyfikacja wariantów tych białek musi być potwierdzona w
teście porównawczym. Na przykład u koni testy takie
wykonywane są dla 16 systemów białek.
Polimorfizm białek surowicy krwi wynika ze zmienności ich form
strukturalnych, będących skutkiem mutacji punktowych w
genie kontrolującym syntezę danego białka lub genach
kodujących
enzymy
odpowiedzialne
za
modyfikacje
potranslacyjną . Spośród ponad 25 białek najbardziej
polimorficzne są albumina, inchybitor α-proteaz i transferyna.
Spośród białek i enzymów erytrocytarnych szczególnie
interesujące są deaminaza adenozyny i hemoglobina.

Allotypy immunoglobulin i
lipoprotein
Są to ugrupowania chemiczne (najczęściej jeden lub kilka
aminokwasów) występujące na cząsteczkach białek i
różnicujące osobniki tego samego gatunku. U większości
gatunków zwierząt gospodarskich poznane allotypy
zlokalizowane są przede wszystkim na α-, β-, γ-globulinach.
Stopień polimorfizmu allotypowego jest różny zależnie od
gatunku. U świń zidentyfikowano dotychczas 16 allotypów
we frakcji β-globulin, 14 allotypów dla γ-globulin i 3 dla α-
globulin. Natomiast u owiec 11 antygenowych markerów
cząsteczek α-globulin, 10 determinant antygenowych we
frakcji β-globulin oraz 7 frakcji γ-globulin.

Białka mleka
Głównymi białkami mleka są kazeiny α
S1
, α
S2
, β i κ oraz białko
serwatkowe- β-laktoglobulina. U bydła loci kazein znajdują się
obok siebie w 6 chromosomie i zajmują razem odcinek DNA
długości ok. 200 kpz (tysiący par zasad). Genotyp zwierzęcia w
loci kontrolujących białka mleka można określać za pomocą
metody elektroforezy stosowanej zarówno w przypadku
rozdziału białek (żel skrobiowy lub poliakryloamidowy), jak i
analizy w poziomie DNA (żel agarozowy lub poliakryloamidowy).
Określenie genotypu na podstawie badania białka jest możliwe
tylko u samic będących w okresie laktacji. Natomiast analiza
polimorfizmu
genów
kodujących
białka
może
być
przeprowadzona u osobników obu płci w każdym wieku.

Geny są bardzo użytecznymi markerami, ale
nie idealnymi. Problemem, szczególnie w
przypadku większych genomów, jak genomy
kręgowców i roślin kwiatowych, jest to, że mapa
oparta całkowicie na genach nie jest zbyt
szczegółowa, gdyż w genomach eukariotów geny
są poprzedzielane dużymi przerwami. Co więcej,
zaledwie część całej liczby genów występuje w
łatwych do odróżnienia formach allelicznych.
Mapy genów nie są wiec zbyt dokładne.
Mapowane cechy niebędące genami nazywane są
markerami DNA. Tak jak markery genowe, aby
były użyteczne, markery DNA muszą występować
w przynajmniej dwóch formach allelicznych. Trzy
typy cech sekwencji DNA odpowiadają temu
wymogowi:
Polimorfizmy
długości
fragmentów
restrykcyjnych (RFLP)
Polimorfizmy długości prostych sekwencji
(SSLP)
Polimorfizmy punktowe (SNP)

POLIMORFIZMY DŁUGOŚCI
FRAGMENTÓW
RESTRYKCYJNYCH
RFLP był pierwszym badanym typem markerów
DNA. RFLP uwarunkowany jest różnicami w
sekwencji nukleotydów w obrębie genu. Mutacje
te mogą powodować powstanie lub likwidację
istniejącego
miejsca
cięcia
dla
enzymu
restrykcyjnego. Enzymy restrykcyjne rozpoznają
specyficzne dla nich sekwencje nukleotydowe i
przecinają
DNA
w
określonym
miejscu.
Polimorfizm fragmentów restrykcyjnych DNA
znajduje zastosowanie w testach identyfikacji
nosicielstwa określonego allelu danego genu.
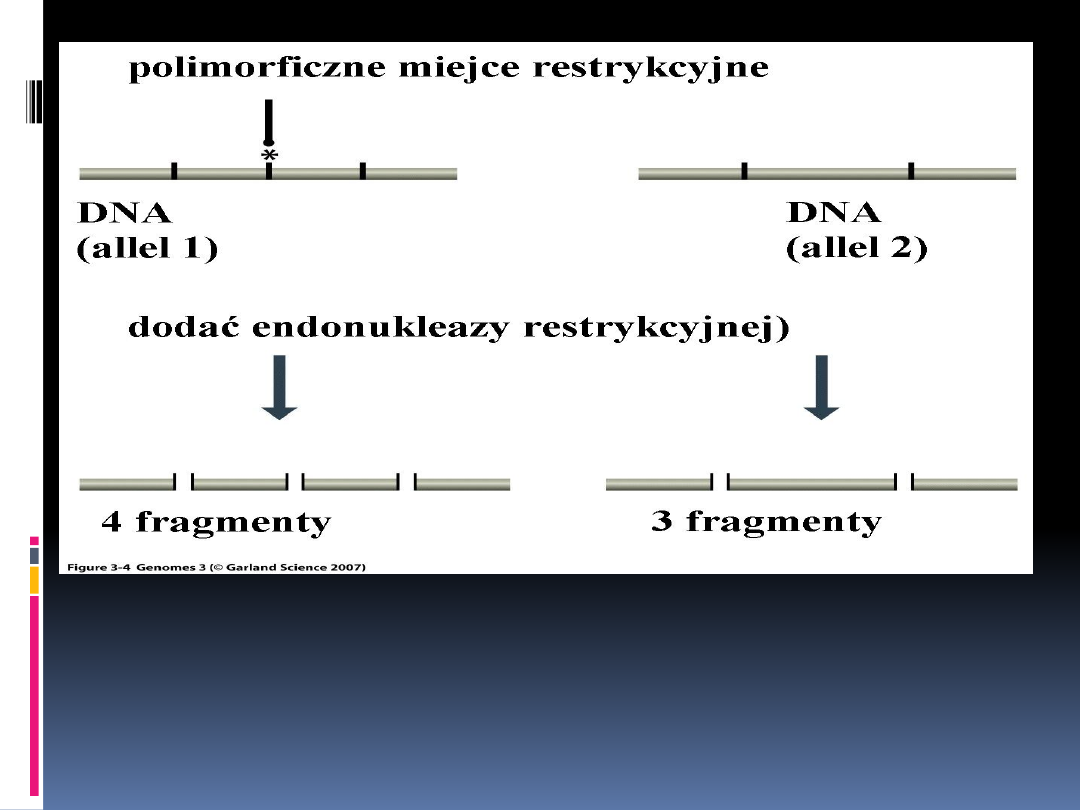
Polimorfizm długości fragmentów restrykcyjnych
Cząsteczka DNA po lewej ma polimorficzne miejsce
restrykcyjne (oznaczone gwiazdką) nieobecne w cząsteczce
po prawej. RFLP ujawnia się po działaniu enzymem
restrykcyjnym, ponieważ jedna cząsteczka jest cięta na cztery
fragmenty, a druga na trzy.

Metody oznaczania
RFLP
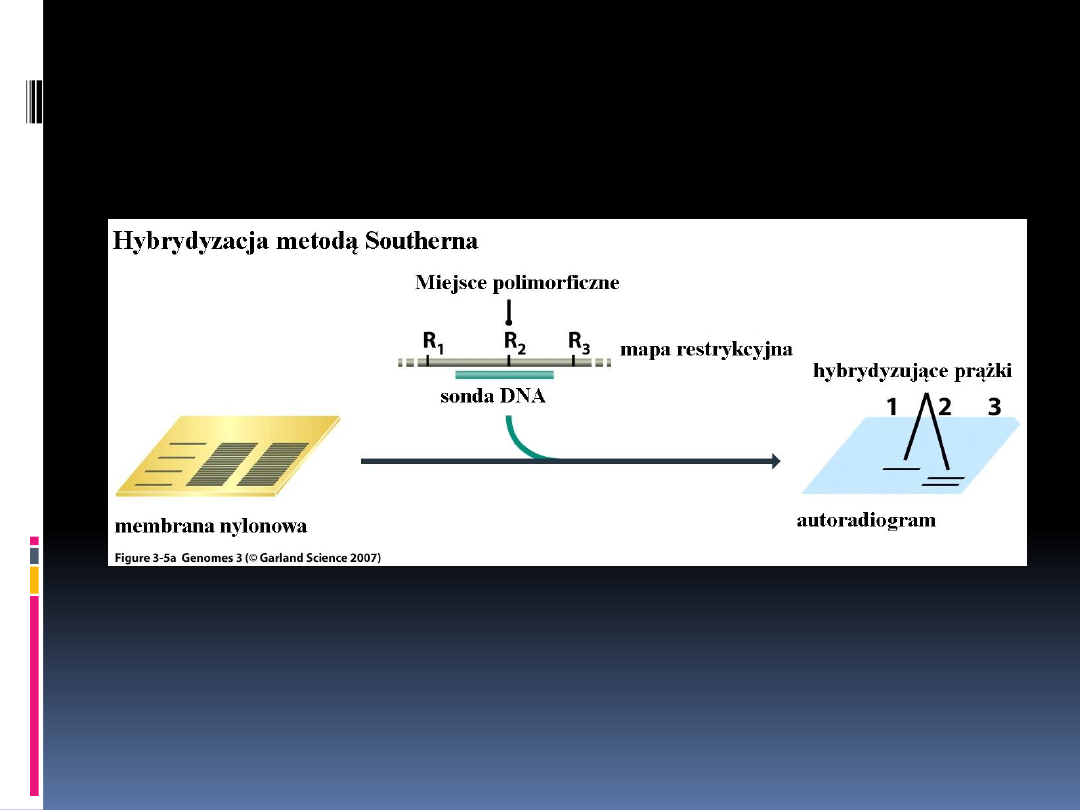
a) Hybrydyzacja metodą
Southerna
DNA trawi się odpowiednim enzymem restrykcyjnym i
rozdziela w żelu agarozowym. Smugę fragmentów
restrykcyjnych przenosi się na membranę nylonową i
hybrydyzuje z sondą – fragmentem DNA obejmującym
polimorficzne miejsce restrykcyjne.
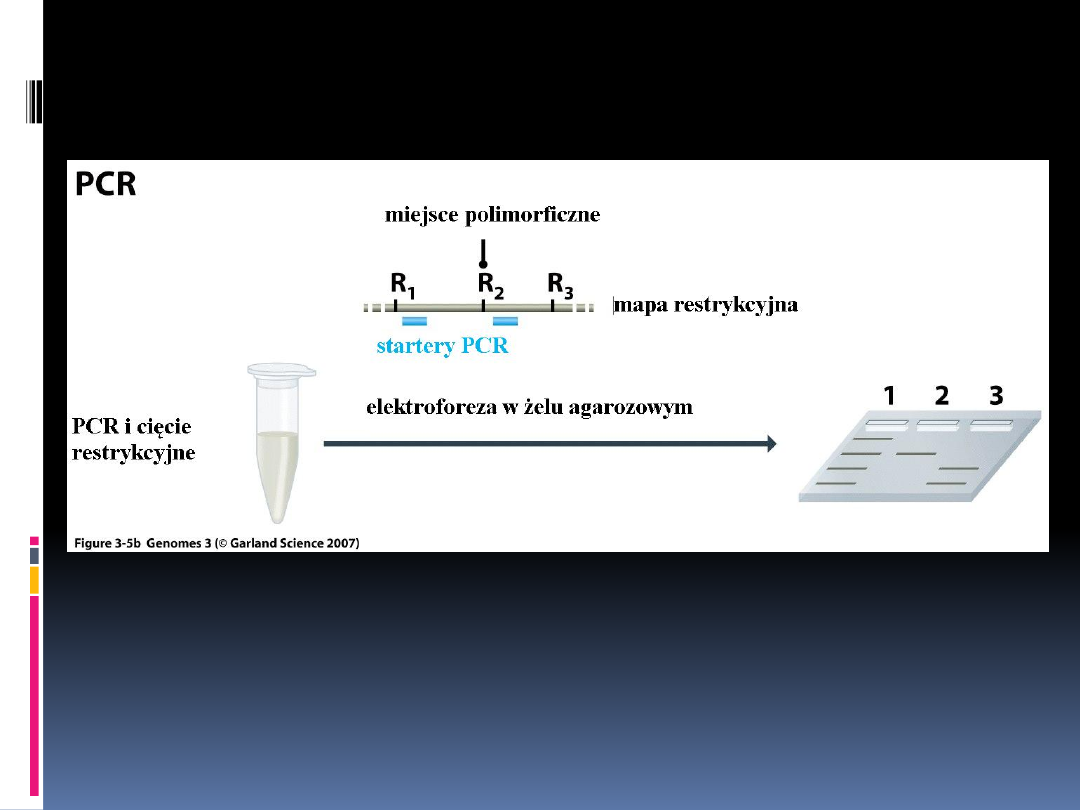
b) Technika PCR
Stosuje się startery, które przyłączają się w miejscach
otaczających polimorficzne miejsca restrykcyjne z obu stron.
Po reakcji PCR produkt trawi się odpowiednim enzymem
restrykcyjnym, a następnie analizuje się przez elektroforezę
w żelu agarozowym. Jeśli nie ma tego miejsca, na żelu
agarozowym widać jeden prążek, jeśli miejsce jest obecne,
widać dwa prążki.

POLIMORFIZMY DŁUGOŚCI
PROSTYCH SEKWENCJI
SSLP są szeregami powtórzeń sekwencji, które
przejawiają różnice długości; różnymi allelami
zawierającymi
różną
liczbę
jednostek
powtarzalnych. W odróżnieniu od RFLP, SSLP
może być wieloallelowe. Istnieją dwa typy
SSLP:
Minisatelity znane także jako zmienna liczba
powtórzeń tandemowych, w których jednostka
powtarzalna ma do 25 bp długości.
Mikrosatelity (proste powtórzenia tandemowe – STR)
których powtórzeniami są znacznie krótsze sekwencje,
zwykle do 13 bp.
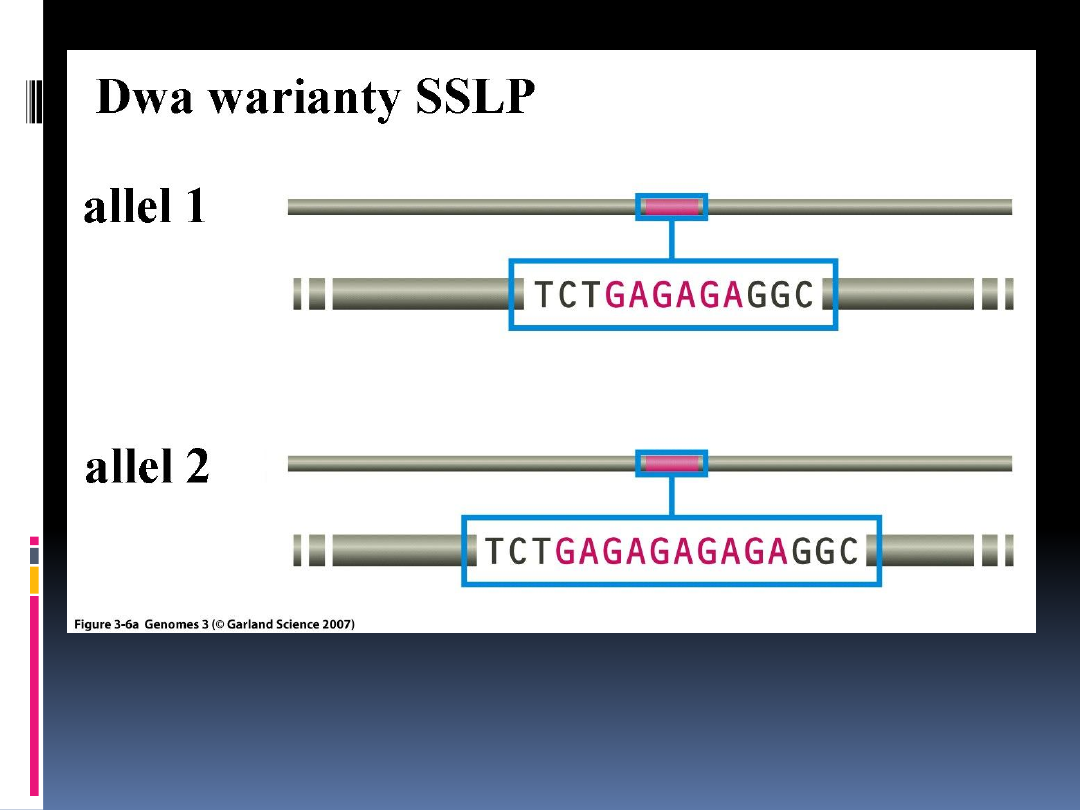
Dwa allele krótkiego powtórzenia tandemowego (STR),
zwanego także mikrosatelitą. W allelu 1 motyw „GA” jest
powtórzony trzy razy, a w allelu 2 pięć razy.
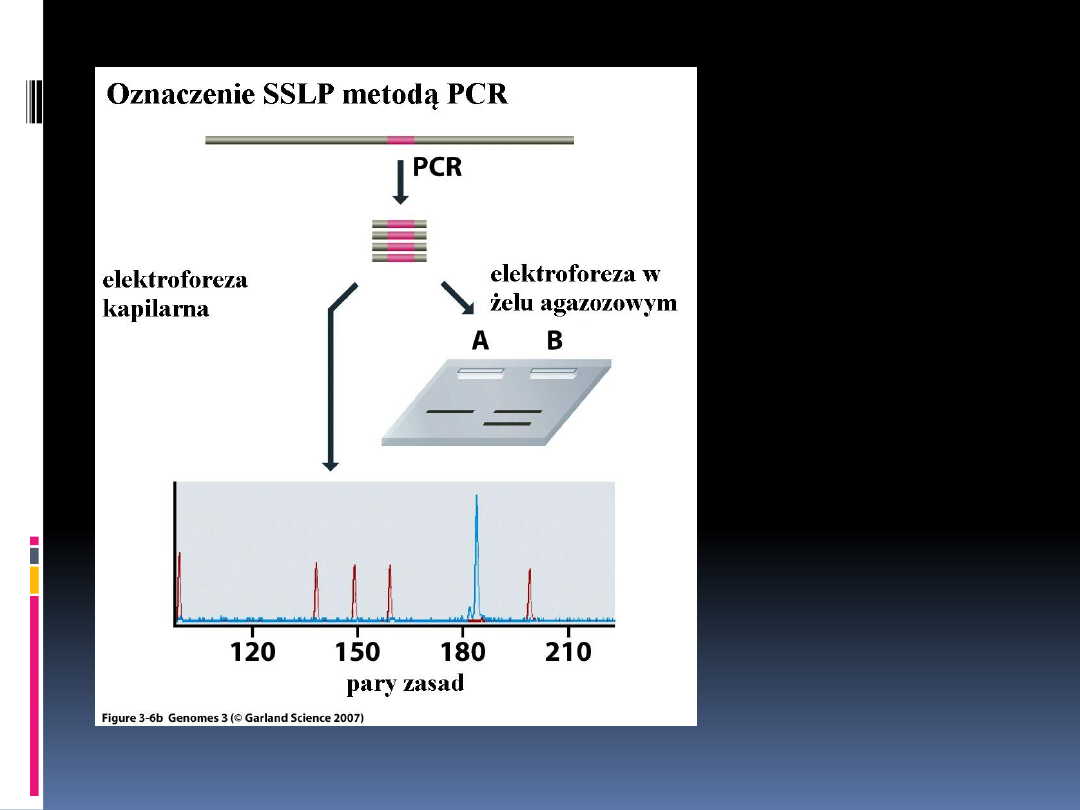
STR
i
obszar
otaczającej
go
sekwencji
amplifikuje się, a
rozmiar
badanego
produktu
identyfikuję
się
przez elektroforezę
w żelu agarozowym
lub
elektroforezę
kapilarną. W żelu
agarozowym ścieżka
A zawiera produkt
PCR, a ścieżka P –
markery
DNA.
Wyniki elektroforezy
kapilarnej
przedstawiono jako
elektroforetogram,
na którym położenie
szczytu
wskazuje
rozmiar
produktu
PCR.

Polimorfizm punktowy
SNP – polimorfizm ten związany jest z
występowaniem
pojedynczych
zmian
sekwencji nukleotydów w homologicznych
sekwencjach. Mutacja punktowa prowadzi
do powstania miejsca polimorficznego
typu SNP, jeśli każdy z alleli występuje w
populacji z częstością nie mniejszą niż 1
%. Zaletą polimorfizmu SNP jest jego
powszechność w genomach różnych
gatunków.
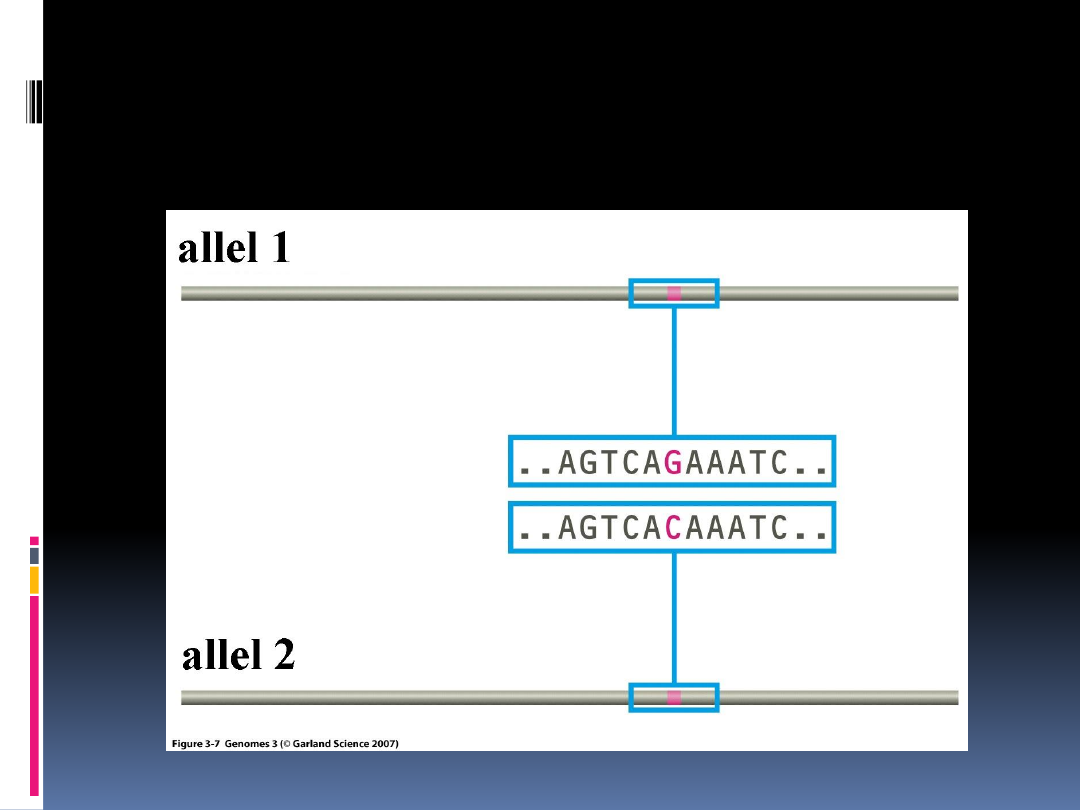
Polimorfizm punktowy (SNP)

Zastosowanie markerów w
hodowli zwierząt
Kontrola pochodzenia
Ocena wartości genetycznej zwierząt
pod katem cech użytkowych i selekcji
pośredniej, tzw. MAS
Ocena
zmienności
genetycznej
wewnątrz i między populacjami oraz
szacowaniu dystansu genetycznego
Badania nad odpornością/ podatnością
zwierząt na różne choroby i zaburzenia
genetyczne.

Kontrola pochodzenia
Prowadzona jest na podstawie układów grupowych i
polimorficznych białek. Podstawą tej kontroli jest to, że
potomek dziedziczy jeden allel z każdego locus od ojca,
drugi od matki. Zatem w przypadku grup krwi nie może
on mieć antygenu erytrocytarnego, którego nie
stwierdzono u jednego z rodziców. Do kontroli
pochodzenia najlepsze są układy grupowe z dużą liczba
antygenów:
• bydło – grupa B – 40 antygenów, C- 12 , S- 8
• świnie – grupa E – 17 antygenów, M- 12, L- 12
• konie – grupa D – 17 antygenów, A- 7, P- 4, Q-3
Wyróżniamy dwie podstawowe metody stosowane do
kontroli
pochodzenia;
jedna
jest
oparta
na
polimorfizmie minisatelitarnym, druga na polimorfizmie
mikrosatelitarnym.

Ocena wartości genetycznej
zwierząt pod katem cech
użytkowych i selekcji pośredniej,
tzw. MAS.
W metodzie tej kryterium wyboru
zwierzęcia jest jego genotyp w locus
markera
genetycznego
wykazującego
asocjację
z
doskonaloną
cechą
użytkową.
Metoda ta daje możliwość określenia
genotypu bardzo młodych zwierząt.

Ocena zmienności genetycznej
wewnątrz i między populacjami
oraz szacowaniu dystansu
genetycznego.
Szacowanie dystansu genetycznego i wybór ras,
które cechuje duża zmienność genetyczna, służy
zachowaniu biologicznej różnorodności zwierząt
gospodarskich.
Z
kolei
ocena
zmienności
genetycznej między populacjami ułatwia wybór
optymalnego
wariantu
krzyżowania,
a
w
krzyżowaniu towarowym uzyskanie maksymalnego
efektu
heterozji,
ujawniającego
głównie
zwiększeniem
wydajności
cech
użytkowych
zwierząt.

Badania nad odpornością/
podatnością zwierząt na różne
choroby i zaburzenia genetyczne
Dzięki tym badaniom nosiciele
niekorzystnych alleli mogą być
wykrywani, zanim wystąpią objawy
chorobowe. Szczególne znaczenie
mają badanie związku między
antygenami/
genami
głównego
układu zgodności tkankowej (MHC) a
występowaniem chorób zwłaszcza
infekcyjnych.

DZIĘKUJE
BIBLIOGRAFIA
1. T.A. Brown: Genomy, Warszawa 2009
2. Krystyna M. Charon: Genetyka zwierząt,
Warszawa 2004
Document Outline
- Slide 1
- Slide 2
- Klasy markerów:
- Slide 4
- Pierwszymi stosowanymi markerami były geny
- Slide 6
- Klasa pierwsza markerów
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- POLIMORFIZMY DŁUGOŚCI FRAGMENTÓW RESTRYKCYJNYCH
- Slide 13
- Metody oznaczania RFLP
- a) Hybrydyzacja metodą Southerna
- b) Technika PCR
- POLIMORFIZMY DŁUGOŚCI PROSTYCH SEKWENCJI
- Slide 18
- Slide 19
- Polimorfizm punktowy
- Polimorfizm punktowy (SNP)
- Zastosowanie markerów w hodowli zwierząt
- Kontrola pochodzenia
- Slide 24
- Slide 25
- Slide 26
- DZIĘKUJE
Wyszukiwarka
Podobne podstrony:
markery genetyczne
11 markery genetyczneid 12288
Wykorzystanie markerow genetycznych
markery genetyczne
Markery genetyczne
MARKER GENETYCZNY, MARKER GENETYCZNY - dziedziczna cecha genetyczna z łatwo rozpoznawalnymi allelami
markery genetyczne
11 markery genetyczneid 12288
Biotechnologia -W, Markery, Inżynieria genetyczna - zespół technik pozwalających na badanie procesów
Wybrane markery chorb nowotworowych
Seminarium3 Inne zaburzenia genetyczne
Genetyka regulacja funkcji genow
Analiza genetyczna w medycynie sądowej
03 PODSTAWY GENETYKI
więcej podobnych podstron