Wykład 4
1
POZYSKIWANIE SZCZEPÓW MIKROORGANIZMÓW O
ZNACZENIU PRZEMYSŁOWYM cz. 2
Mikrobiologia Przemysłowa
1
Proces pozyskiwania mikroorganizmów do zastosowań w
przemyśle obejmuje następujące po sobie etapy
1. Pobranie próby ze środowiska naturalnego
2. Hodowla wzbogacająca
3. Test selekcyjny
4. Testy fermentacyjne
5. Identyfikacja gatunkowa wybranego mikroorganizmu
2

Wykład 4
2
5. Identyfikacja taksonomiczna
mikroorganizmów
Identyfikacja taksonomiczna mikroorganizmu -
określenie
przynależności badanego organizmu do odpowiedniej
jednostki taksonomicznej,
najczęściej gatunku
3
Drzewo życia – czyli filogenetyczny podział organizmów
planety Ziemia
FILOGENETYKA -
dyscyplina biologii
zajmująca się odtwarzaniem dróg rozwoju
rodowego
poszczególnych grup organizmów, zarówno żyjących współcześnie, jak
i w epokach minionych
4

Wykład 4
3
Drzewo życia – czyli filogenetyczny podział
organizmów planety Ziemia
Drzewo filogenetyczne zbudowane jest w oparciu o taksony
Takson - grupa organizmów na tyle do siebie podobnych, że można ją wyróżnić i
zaklasyfikować do kategorii systematycznej np. klasy, rodziny, rodzaju, gatunku
Podstawową jednostką klasyfikacji biologicznej jest gatunek
System klasyfikacji musi być oparty na cechach występujących we wszystkich
żywych organizmach
5
Identyfikacja taksonomiczna
mikroorganizmów
Gatunek (w ujęciu mikrobiologicznym) - populacja mikroorganizmów wykazujących
wysoki stopień podobieństwa w ściśle określonym zakresie cech, a różniących się
od innych gatunków tego samego rodzaju;
Szczep – grupa drobnoustrojów potomnych jednej komórki
Szczep referencyjny – izolat danego gatunku opisany jako pierwszy i najlepiej
scharakteryzowany (wzorzec)
6

Wykład 4
4
Identyfikacja taksonomiczna bakterii
Gatunek
to grupa szczepów, w tym szczep referencyjny, wykazujących co najmniej 70%
homologii pełnego genomowego DNA (hybrydyzacja DNA-DNA)
grupa szczepów różniących się zawartością G+C w genomowym DNA nie więcej
niż o 3% molowe
nG + nC
% G+C = x 100%
nA + nT + nC + nG
przy czym tylko te cechy nie mogą decydować o przynależności do gatunku
W obrębie rodzaju różnice w %mol G+C nie mogą przekroczyć 10%
7
Cechy umożliwiające identyfikację mikroorganizmów
prokariotycznych - metody konwencjonalne
morfologia
skład chemiczny ściany komórkowej
obecność inkluzji komórkowych i substancji zapasowych
zdolność do tworzenia pigmentów
sposób odżywiania
wymagania pokarmowe
źródła C, N, S
skład jakościowy i ilościowy produktów fermentacji
wymagania tlenowe
zakres temperatur i pH wzrostu
wrażliwość na antybiotyki
patogenność
zależności symbiotyczne z innymi organizmami
środowisko występowania
obecność określonych antygenów (charakterystyka immunologiczna)
8

Wykład 4
5
Cechy umożliwiające identyfikację grzybów - metody
konwencjonalne
Sposób generatywnego rozmnażania się
W identyfikacji drożdży rozpatruje się ponadto:
Cechy morfologiczne komórki
Sposób rozmnażania wegetatywnego
Cechy hodowlane populacji na pożywkach płynnych i stałych
Pigmentację
Zdolność tworzenia wegetatywnych spor, pseudogrzybni i grzybni
Cechy biochemiczne (synteza ureazy, asymilacja i fermentacja różnych
źródeł węgla i azotu)
Osmotolerancyjność i osmofilność
Budowa ściany komórkowej, zawartość glukanów, mannanów i chityny
Ekologia
Patogenność
Podstawą identyfikacji grzybów strzępkowych jest:
Morfologia i cechy makroskopowe grzybni oraz cechy biochemiczne
9
Obserwacja komórek bakterii
•
Obserwacje mikroskopowe żywych bakterii nie pozwalają na rozpoznanie kształtów i
szczegółów struktur komórkowych. Kształt bakterii obserwuje się zwykle po ich
wybarwieniu.
•
Do celów diagnostycznych stosuje się barwienie różnicujące, metodę Grama,
która jest podstawą podziału bakterii na dwie grupy o odmiennych cechach
fizjologicznych i biochemicznych.
10
Wykład 4
6
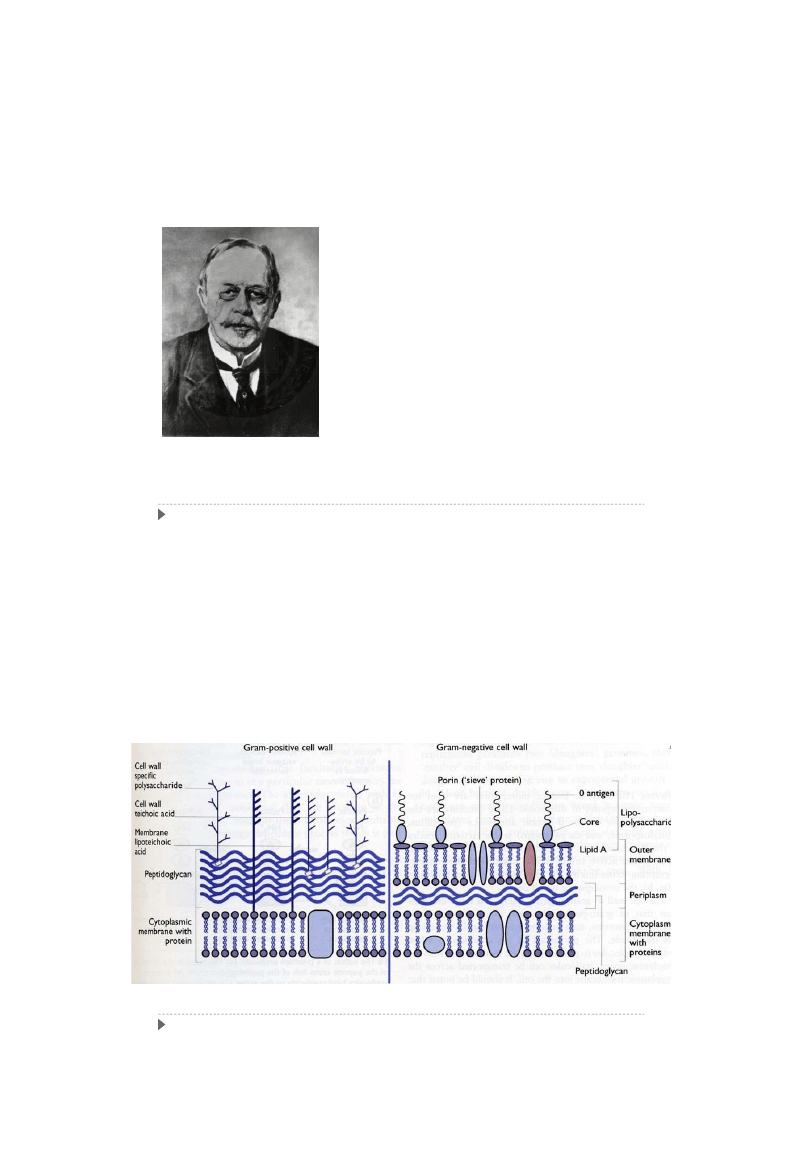
Hans Christian Gram
(13.09.1853 – 14.11.1938)
Duński farmakolog i lekarz. W
roku 1884 opublikował autorską
metodę barwienia bakterii.
Dokonał przełomowego odkrycia
przyczyniającego się do rozwoju
mikrobiologii
11
Budowa ściany komórkowej bakterii G+ i G-
12
Wykład 4
7
13
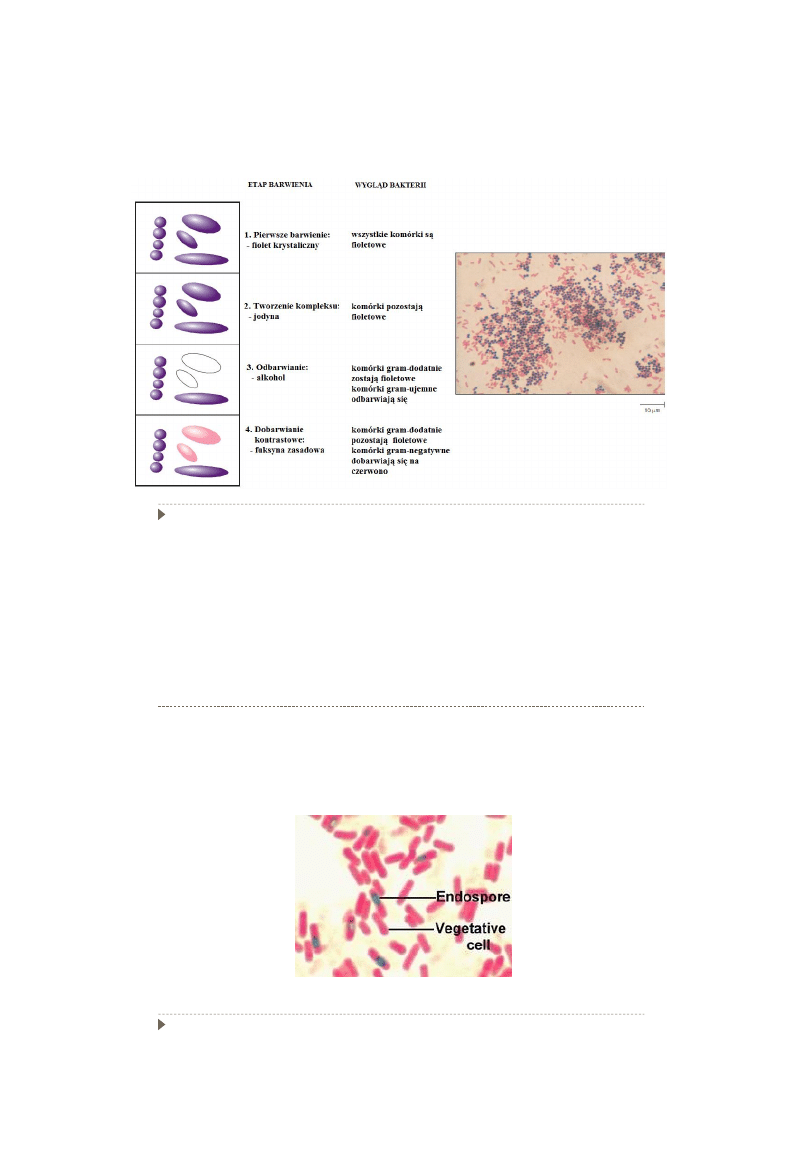
Obserwacja przetrwalników bakteryjnych
Metoda Schaeffera-Fultona
Barwienie przetrwalników zielenią malachitową na gorąco
Dobarwianie komórek wegetatywnych fuksyną zasadową
Bacillus subtilis
14

Wykład 4
8
Metody identyfikacji mikroorganizmów
Podział metod identyfikacji mikroorganizmów:
biochemiczne
biofizyczne
biologii molekularnej
immunochemiczne
15
I. Metody biochemiczne
Polegają na określeniu zdolności mikroorganizmów do asymilacji, fermentacji
lub rozkładu określonych związków chemicznych
cechy biochemiczne określa się na podstawie reakcji chemicznych
zachodzących w odpowiednio skomponowanych pożywkach wzrostowych
wyniki testów biochemicznych odczytuje się makroskopowo
wzrost lub jego brak
zmiana zabarwienia pożywki
reakcja barwna po wprowadzeniu odczynnika reagującego z wytwarzanym metabolitem
wytworzenie gazu
16
Wykład 4
9
Metody biochemiczne
Poszczególne taksony
mikroorganizmów posiadają
charakterystyczne dla nich szlaki
enzymatyczne
17
Metody biochemiczne
Obecnie stosuje się zestawy miniprobówek lub płytek zawierających odwodnione
podłoża z ewentualnym indykatorem.
Czysta kultura o określonej gęstości jest zawieszana w rozcieńczalniku, a następnie
taka sama objętość mieszana jest z podłożem.
18

Wykład 4
10
Testy biochemiczne
INKUBACJA
(18 – 48 h)
Zawieszenie
Naniesienie
Interpretacja wyników w teście API (bioMerieux)
Identyfikacja ponad 550 gatunków bakterii (głównie
chorobotwórczych) oraz drożdży
19
Testy biochemiczne
Istnieją również wersje zautomatyzowane, gdzie wynik w postaci odczytu
densytometrycznego zawiesiny jest analizowany przez odpowiedni program.
Wynik testu może być również wprowadzany w postaci numerycznej do programu,
który analizuje dane i generuje listę możliwych gatunków wraz z %
prawdopodobieństwa identyfikacji.
20

Wykład 4
11
Testy biochemiczne
Przykładem takiego uniwersalnego testu biochemicznego do identyfikacji
mikroorganizmów (bakterii, drożdży i grzybów) jest system „Biolog" firmy „Biolog
Inc", który służy do identyfikacji ponad 2500 gatunków drobnoustrojów.
96 studzienek
Forma mikropłytki umożliwia uzyskanie charakterystycznego obrazu testu
unikatowego dla gatunku drobnoustroju
21
II. Metody biofizyczne
Umożliwiają identyfikację taksonomiczną mikroorganizmów poprzez oznaczanie
produktów ich metabolizmu lub wybranych związków wchodzących w skład
struktur komórkowych
Techniki elektroforetyczne
Techniki oparte na chromatografii gazowej
22
Wykład 4
12
Identyfikacja drobnoustrojów za pomocą
technik elektroforetycznych
Identyfikacja drobnoustrojów za pomocą technik elektroforetycznych oparta jest
na analizie profili białkowych
skład ilościowy i jakościowy wszystkich białek komórkowych, który jest
charakterystyczny dla danej jednostki taksonomicznej
Sposób postępowania:
izolacja białek komórkowych
rozdział elektroforetyczny w żelu poliakrylamidowym
barwienie rozdzielonych białek, co umożliwia uzyskanie obrazu profili
polipeptydowych charakterystycznych dla danego gatunku mikroorganizmu
porównanie z profilami białkowymi bazy danych
Metoda pozwalająca określić przynależność gatunkową mikroorganizmu
23
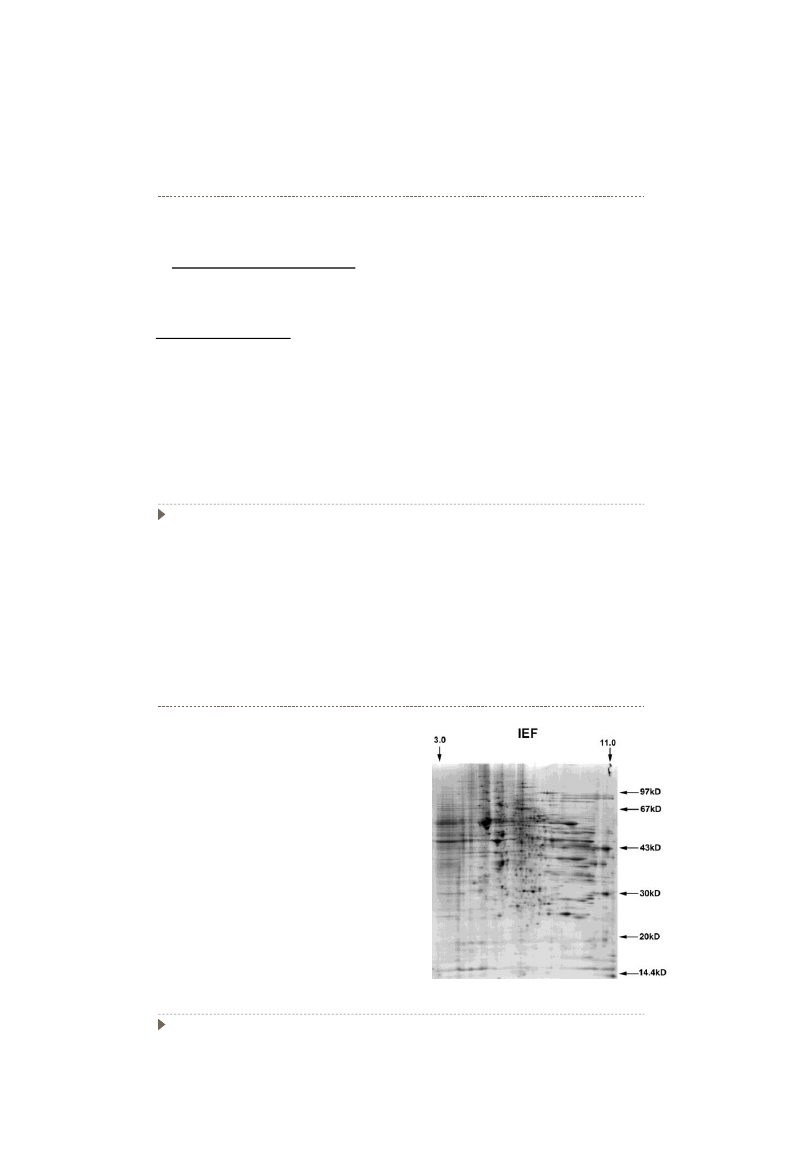
Identyfikacja drobnoustrojów za pomocą
technik elektroforetycznych
Elektroforeza dwukierunkowa
Przed identyfikacją białek często stosuje
się inkubację hodowli w pożywce
zawierającej marker metaboliczny, np.
znakowaną
radioaktywnie siarkę
, która
zostaje wbudowana do białek komórek
badanego szczepu.
Po rozdziale obraz żelu jest odczytywany
(skaner promieniowania β lub
densytometr), a następnie porównywany
przez odpowiedni program z bazą danych
.
24

Wykład 4
13
Identyfikacja mikroorganizmów w oparciu
o chromatografię gazową
Analiza profili kwasów tłuszczowych pochodzących z lipidów ściany
komórkowej
skład kwasów tłuszczowych jest unikalny i charakterystyczny dla każdego gatunku
mikroorganizmu przy zachowaniu kontrolowanych warunków hodowli
zarówno ilościowy, jak i jakościowy skład ściany komórkowej bakterii kodowany jest przez
DNA genomowe i nie ma na niego wpływu informacja genetyczna zawarta w plazmidach
Kwasy tłuszczowe ściany komórkowej mogą być analizowane:
jakościowo – obecność specyficznych kwasów tłuszczowych
ilościowo – określenie zawartości poszczególnych kwasów tłuszczowych
25
Identyfikacja mikroorganizmów w oparciu
o chromatografię gazową
Sposób postępowania:
Komórki czystej kultury identyfikowanych bakterii poddaje się saponifikacji (hydroliza
triglicerydów) i uwolnieniu kwasów tłuszczowych
Następnie prowadzi się metylowanie kwasów oraz ich ekstrakcję z fazy wodnej w celu
zwiększenia lotności, po czym przeprowadza rozdział metodą chromatografii gazowej
Wyniki są analizowane przez komputer i porównywane z bazą profili chromatograficznych
charakterystycznych dla poszczególnych gatunków drobnoustrojów
Metoda pozwalająca określić
przynależność gatunkową mikroorganizmu
26
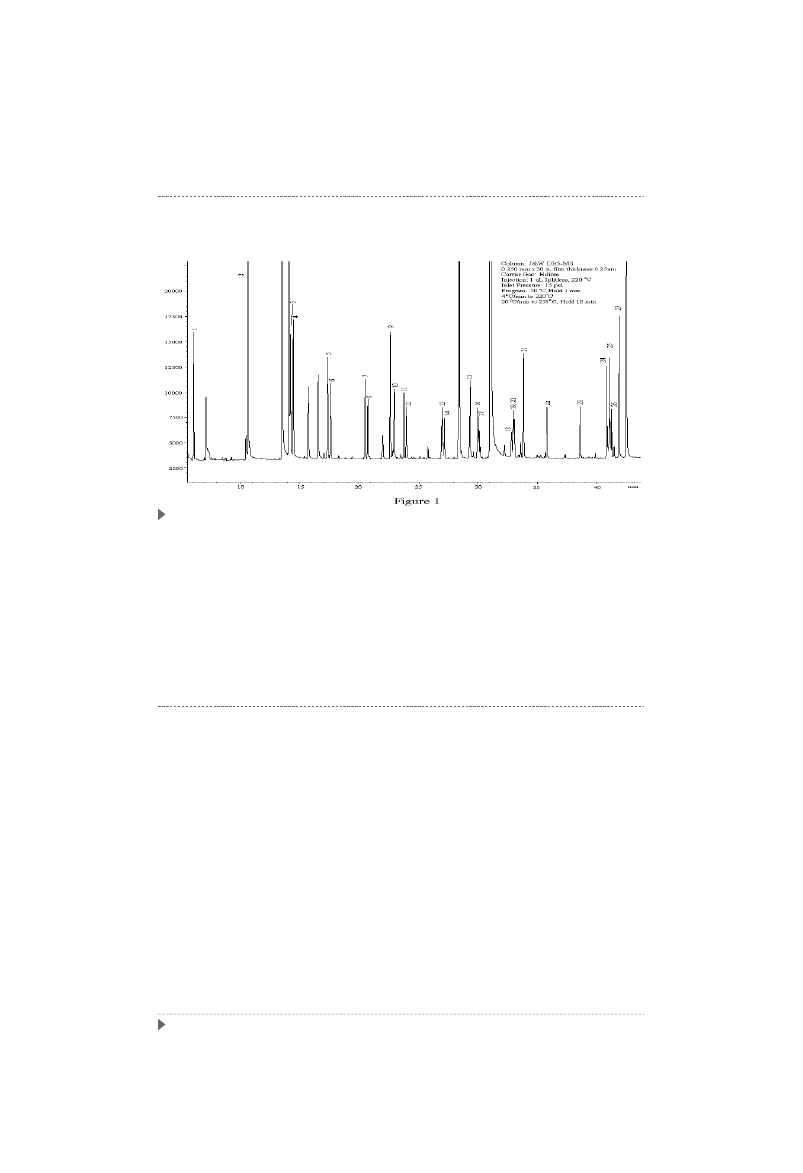
Wykład 4
14
Identyfikacja mikroorganizmów w oparciu o
chromatografię gazową
Poszczególne piki są identyfikowane w oparciu o wzorce kwasów tłuszczowych.
27
Identyfikacja mikroorganizmów w oparciu o
FTIR
Spektroskopia w podczerwieni z transformacją Fouriera (FTIR)
Uzyskuje się widma w podczerwieni komórek poddanych
dezintegracji, które są odzwierciedleniem ich składu chemicznego –
obraz zależny od:
rodzaju białek,
kwasów nukleinowych,
elementów błony plazmatycznej i ściany komórkowej
Unikatowy rozkład widma dla gatunku jest porównywany z bazą
szczepów wzorcowych o znanej przynależności systematycznej.
28

Wykład 4
15
III. Metody biologii molekularnej
Oparte na analizie kwasów nukleinowych
ilościowej (% molowy GC)
jakościowej (homologia)
Kariotypowanie elektroforetyczne
Analiza polimorfizmu miejsc restrykcyjnych RFLP
Techniki wykorzystujące łańcuchową reakcję polimerazy PCR
Analiza polimorfizmu fragmentów zamplifikowanych AFLP
Metody analizy porównawczej rRNA (16S i 18S)
Sekwencjonowanie fragmentów lub całych genomów
29
DNA – cel molekularny
Jako standardowy cel molekularny wykorzystywany w metodach biologii molekularnej,
które służą identyfikacji rodzajowej mikroorganizmów, wykorzystuje się geny
należące do tzw. rDNA:
16S rDNA
dla organizmów prokariotycznych
18S rDNA
dla organizmów eukariotycznych
Molekularna miara stopnia pokrewieństwa
– sekwencja DNA wspólnych
genów – genów kodujących rybosomalny RNA
(Carl Woese)
30

Wykład 4
16
Molekularna miara stopnia pokrewieństwa
Porównanie sekwencji genów rRNA z różnych organizmów
Organizm I ACTGCATTA
C
GCCTTAAGAGGCTCT
Organizm II ACTGCATTA
G
GCCTTAAGAGGCTCT
Organizm III ACTGCAT
A
A
T
GC
ACA
A
T
GAGGCTCT
Sekwencja
zmienna
Sekwencja
zakonserwowana
Sekwencja
zakonserwowana
Organizm I
Organizm II
Organizm III
31
Metody biologii molekularnej
Metody identyfikacji oparte na badaniu homologii fragmentów
kwasów nukleinowych
Ponieważ sekwencja nukleotydów w łańcuchu kwasu nukleinowego jest
specyficzna, charakterystyczna dla danego gatunku organizmu, określenie
stopnia podobieństwa fragmentu DNA lub RNA badanego mikroorganizmu, w
odniesieniu do wzorca wiadomego pochodzenia, umożliwia jego identyfikację
taksonomiczną
Metody hybrydyzacyjne
Metody oparte o technikę PCR i jej modyfikacje
32

Wykład 4
17
Metody hybrydyzacyjne
Polegają na zastosowaniu tzw. sond genetycznych
sondy są to krótkie jednoniciowe fragmenty DNA, zawierające sekwencje unikalne
dla danego organizmu
kwas nukleinowy pełniący rolę sondy genetycznej jest znakowany
radioaktywnym fosforem [32P] lub wodorem [3H]
barwnikiem fluorescencyjnym
Enzymem (peroksydaza)
Pozwala to na określenie stopnia hybrydyzacji sondy z badanym DNA lub RNA
33
Metody hybrydyzacyjne
Najpopularniejsze systemy detekcji wykorzystują sondy DNA komplementarne do
charakterystycznych dla identyfikowanego mikroorganizmu sekwencji rybosomalnego
RNA (rRNA)
Wykorzystanie rRNA zwiększa czułość oznaczenia, gdyż występuje on w komórce
bakteryjnej w dużej liczbie kopii (od 1 000 do 10 000)
Inną zaletą tego rozwiązania jest dostępność informacji odnośnie sekwencji rRNA
pochodzących od różnych, często bardzo blisko genetycznie spokrewnionych
mikroorganizmów, dzięki czemu możliwe jest otrzymywanie sond o bardzo wysokiej
specyficzności
34
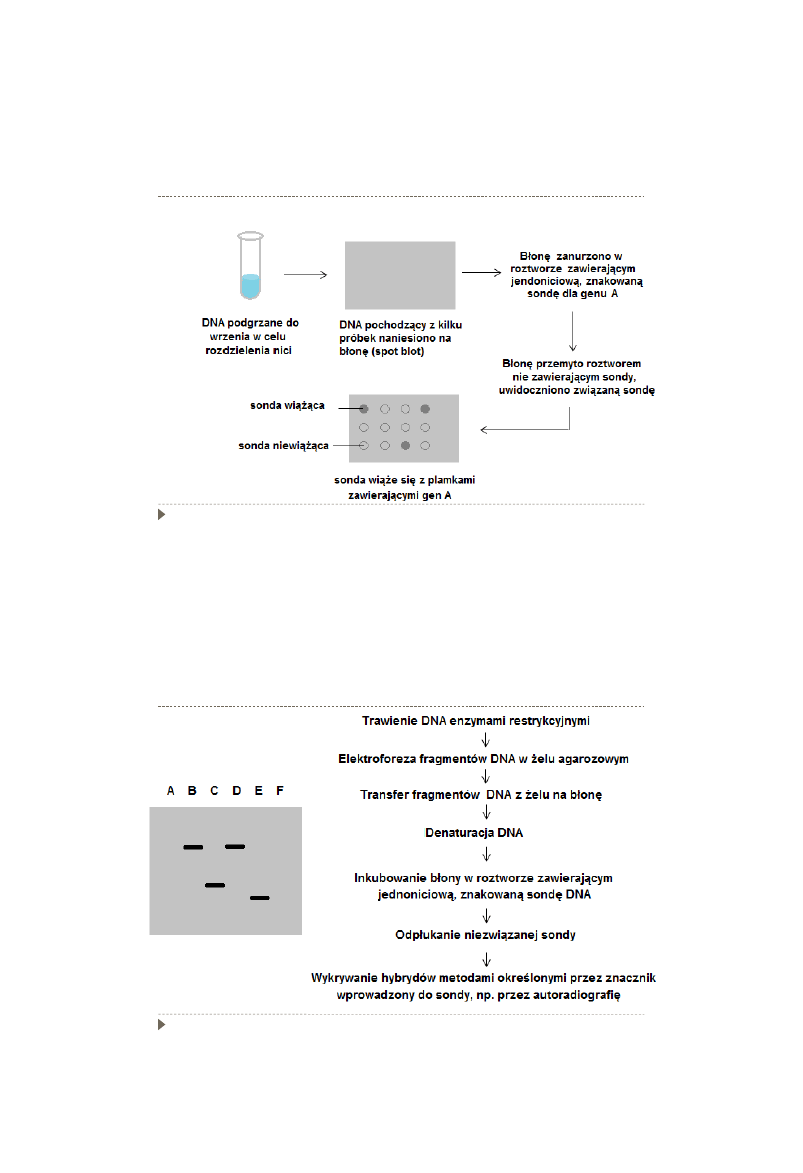
Wykład 4
18
Metody hybrydyzacyjne
Dot blot
35
Metody hybrydyzacyjne
Southern blotting
36
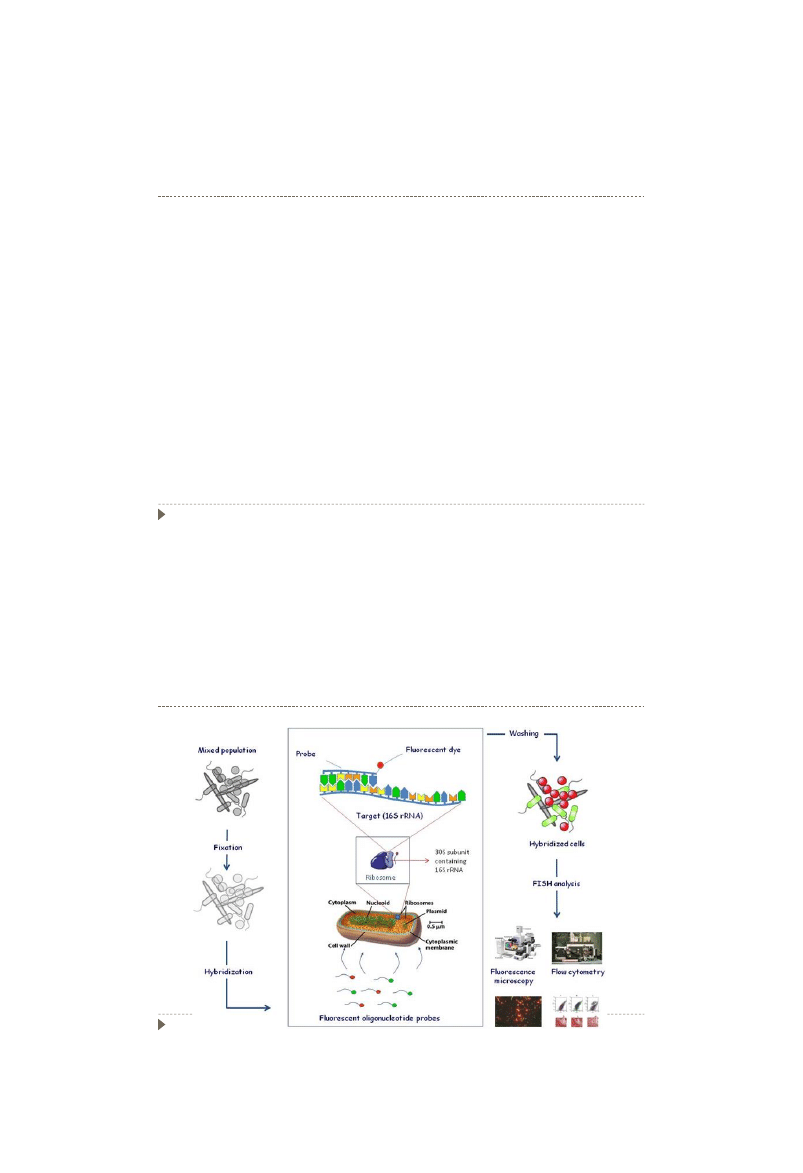
Wykład 4
19
Metody hybrydyzacyjne
homologia kwasów nukleinowych powyżej 70% świadczy o przynależności
organizmu do określonego gatunku
stopień hybrydyzacji powyżej 20% wskazuje na zgodność identyfikowanego
drobnoustroju z danym rodzajem
hybrydyzacja od 1 do 5% może zachodzić między DNA lub RNA organizmów
niespokrewnionych
37
FISH – hybrydyzacja fluorescencyjna in situ
Umożliwia identyfikację mikroorganizmów widzianych pod mikroskopem
38
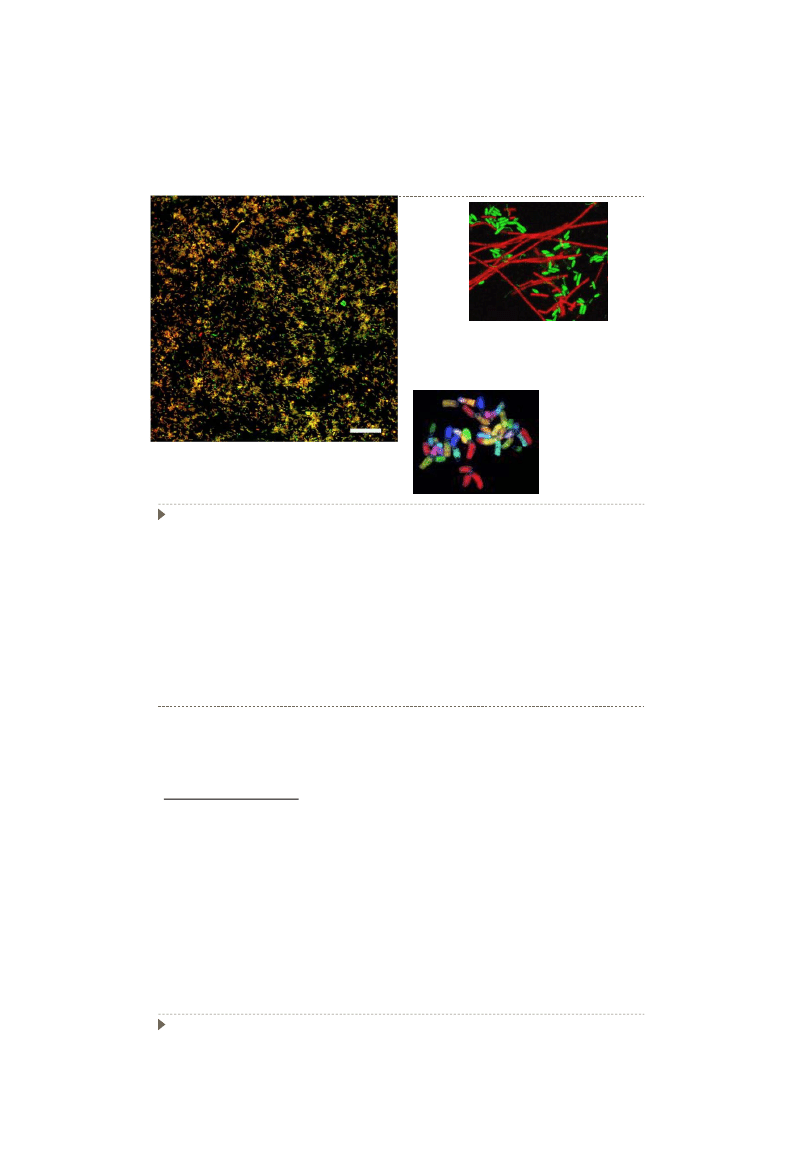
Wykład 4
20
FISH – hybrydyzacja fluorescencyjna in situ
Wynik FISH dla
mieszaniny wybranych
bakterii z rodzaju
Bacillus, Escherichia,
Pseudomanas, Shigella
Wyznaczone bakterie (
żółte/pomarańczowe
) i
mikroorganizmy nie będące bakteriami (
zielone
)
pobrane z powierzchni glonów Ulva sp.
Wynik FISH dla mikroorganizmów z
domen Archaea (
czerwone
) i
Bacteria (
zielone
)
39
Metody wykorzystujące technikę PCR
Technika PCR (Polymerase Chain Reaction) - enzymatyczna amplifikacja
(zwielokrotnienie) in vitro specyficznej sekwencji nukleotydów
Sposób postępowania:
amplifikacja fragmentu DNA
sekwencjonowanie
porównanie wyników z danymi zamieszczonymi w banku genów
5% różnica w sekwencji wystarczy dla wyodrębnienia gatunku
Specyficzne fragmenty uzyskane w wyniku reakcji PCR mogą być również
wykorzystane w metodzie hybrydyzacji.
40
Wykład 4
21
Metody wykorzystujące technikę PCR
badanie mieszanej populacji mikroorganizmów
41
P
C
R
Sekwencjonowanie
DNA
Izolacja DNA
agctgatcgtagtctgt
aggctggtgtaccctg
atgcttgtatgttaaaag
ctagatgctgagtgagt
cgtagtggatcgatgct
gagtcgtaggctggct
Analiza
Sekwencji
42
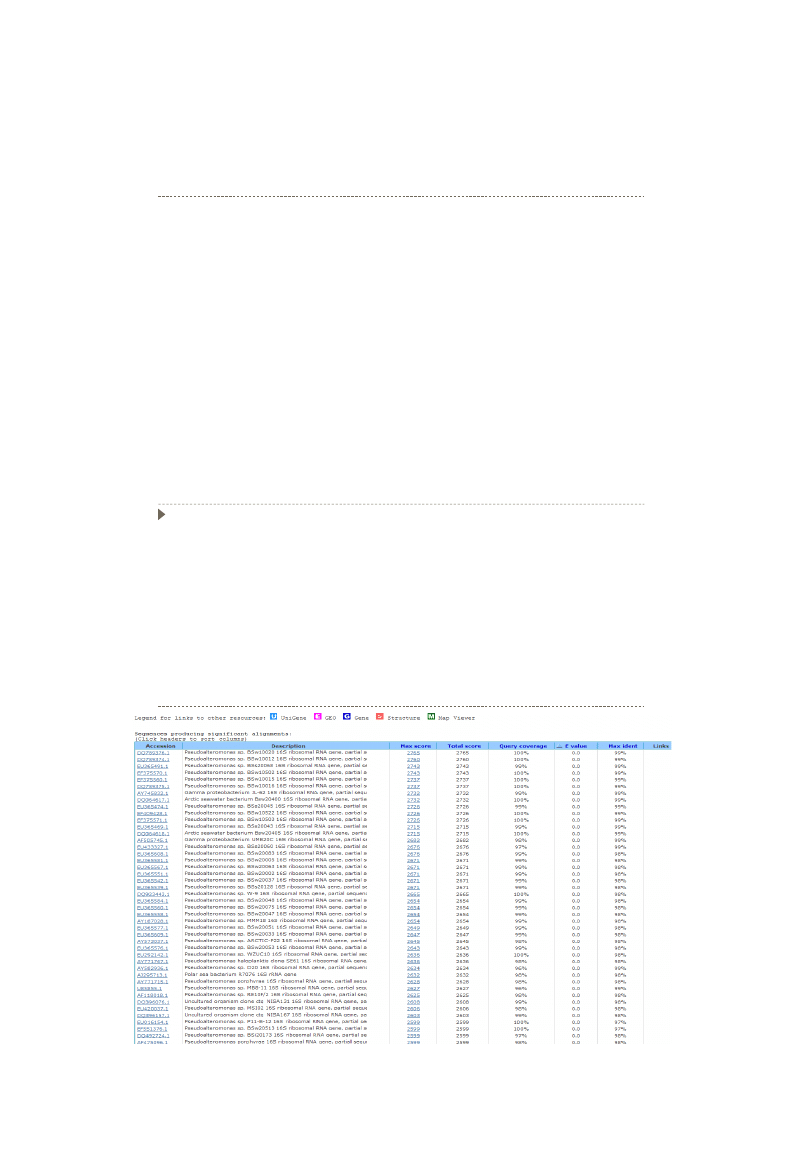
Wykład 4
22
sekwencjonowanie
Nowe metody sekwencjonowania typu Next-Generation Sequencing (NGS)
umożliwiają odczyt do kilku mld pz
Rozwój metod bioinformatycznych umożliwia coraz szybszą i efektywniejszą analizę
takiej ilości danych, dzięki czemu możliwe jest porównywanie coraz większej ilości
fragmentów DNA, a nawet całych genomów badanych mikroorganizmów
43
Analiza sekwencji
44

Wykład 4
23
Metody wykorzystujące technikę PCR
Geny kodujące białka bakteryjne
białko szoku termicznego Hsp 70
białko szoku termicznego Hsp 60
syntaza asparaginylo-tRNA
syntaza alanylo-tRNA
dehydrogenaza glutaminianowa
hydrolaza pirofosforanu
podjednostka
β polimerazy RNA (RpoB)
podjednostka
β’ polimerazy RNA (RpoC)
czynniki elongacyjne: EF 1
α
/Tu, EF-Tu, Ef-G/2
białka rybosomowe: L2, L5, L11, L14, L15, L22, L23, S5, S12
gyrazy
45
IV. Metody immunochemiczne
Stosowane do szybkiego wykrywania mikroorganizmów patogennych w
-
Medycynie
-
Przemyśle spożywczym (Sanepid) i farmaceutycznym
Oparte są na specyficznej reakcji zachodzącej pomiędzy przeciwciałem i
antygenem
46
Wykład 4
24
Metody immunochemiczne
Antygenem mogą być:
Komórki mikroorganizmów lub ich elementy – antygen cząstkowy
Specyficzne metabolity mikroorganizmów, np. toksyny
Przeciwciała:
Białka z grupy immunoglobulin biorące udział w reakcjach odpornościowych
wyższych organizmów eukariotycznych
47
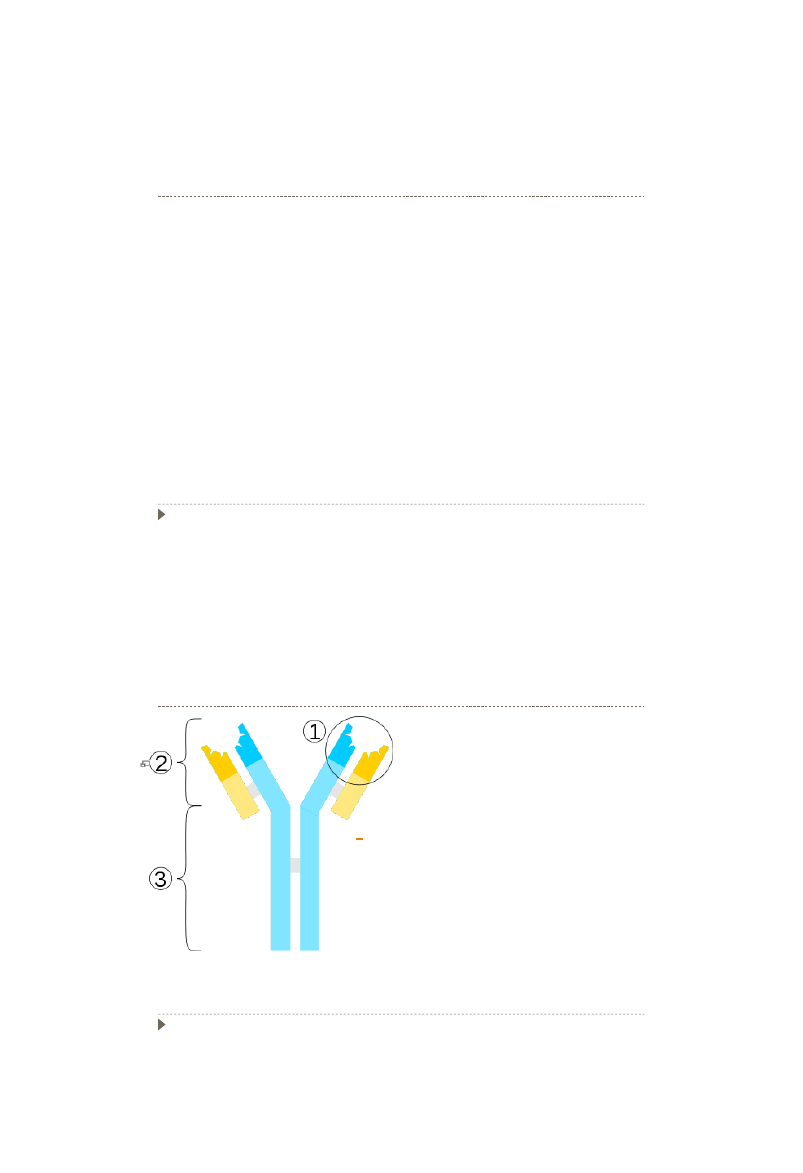
Przeciwciało
Schemat przeciwciała:
1. fragment
wiążący antygen
2. region Fab
3. region Fc
niebieskie
– łańcuchy ciężkie
żółte – łańcuchy lekkie
ciemnoniebieskie
/
ciemnożółte
– regiony zmienne
jasnoniebieskie
/
jasnożółte
– regiony stałe
szare
–
mostki disiarczkowe
48

Wykład 4
25
Metody immunochemiczne
W identyfikacji mikroorganizmów najczęściej stosuje się:
Testy aglutynacji
Metody radioimmunologiczne (RIA)
Metody immunoenzymatyczne (EIA)
Metody immunofluorescencyjne (FIA)
49
Test aglutynacji
Umożliwia identyfikację gatunków bakterii i grzybów strzępkowych na podstawie analizy antygenów
ścian komórkowych, komórek lub specyficznych toksyn wytwarzanych przez komórki.
W celu wzmocnienia efektu stosowane są
cząsteczki lateksu, które opłaszczane są
przeciwciałem. W obecności specyficznego
antygenu zachodzi reakcja immunologiczna
wynikiem czego jest aglutynacja (zlepianie
się)
cząstek
lateksu
opłaszczonych
przeciwciałem.
50

Wykład 4
26
Metody immunochemiczne
Reakcja immunologiczna wykrywana jest poprzez specyficzne
znakowanie samego przeciwciała:
fluorochromami (np. fluoresceiną, rodaminą, fikoerytryną, fikocyjaniną),
radioizotopem
enzymem (peroksydaza chrzanowa, fosfataza alkaliczna, β-galaktozydaza)
51
Metody immunochemiczne
Znakowanie przeciwciała enzymem umożliwia wykrycie reakcji immunologicznej po
dodaniu kompleksu chromogenu z substratem enzymu.
Zmiana barwy środowiska na skutek utworzenia barwnego produktu pozwala na
detekcję nawet bardzo małych ilości antygenu.
52
Wykład 4
27
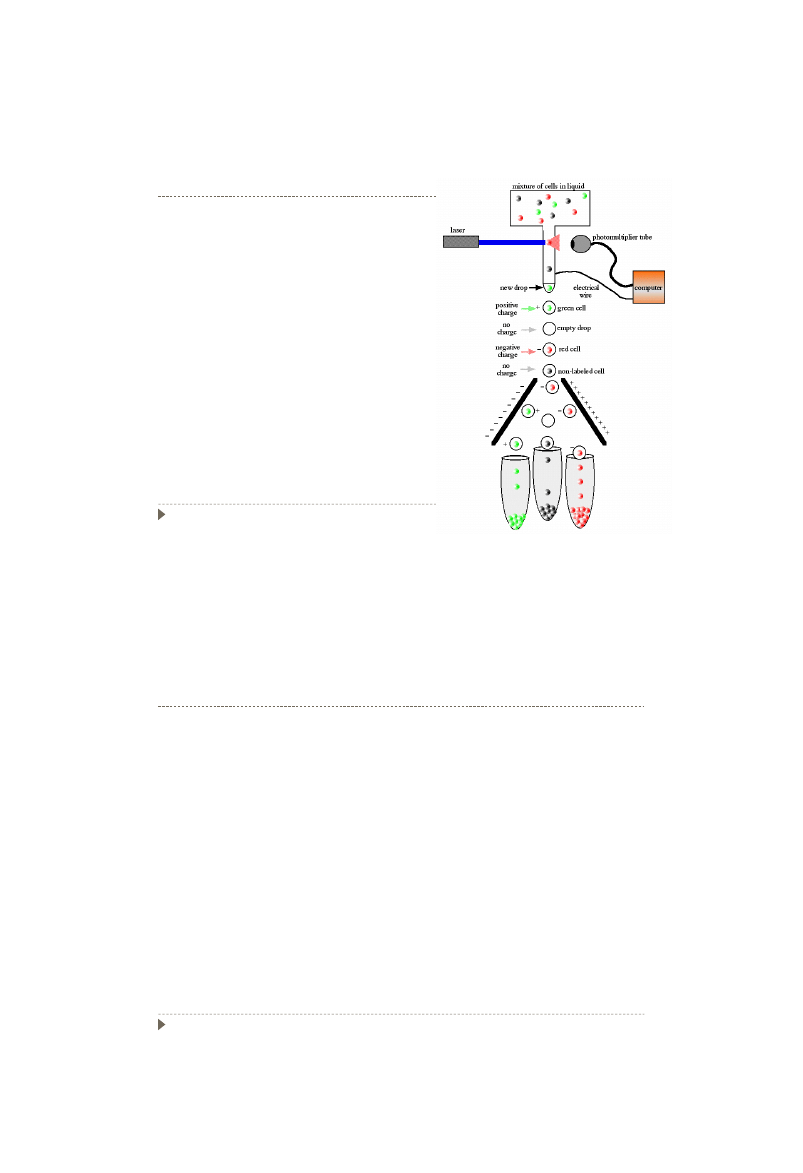
Metody immunofluorescencyjne - FACS
Komórki
znakowane
markerem
fluorescencyjnym
Rozdział
przy
użyciu
cytometru
przepływowego (fluorescence-activated cell
sorter)
–
aktywacja
fluorescencji
przeciwciała przy użyciu lasera
Sortowanie w polu elektrycznym komórek z
różnym znacznikiem
Istotny
jest
dobór
odpowiedniego
przeciwciała
53
Metody immunoenzymatyczne - ELISA
ang. Enzyme Linked Immunosorbent Assay
Wyróżnia się 3 odmiany:
Bezpośrednia współzawodnicząca
Pośrednia współzawodnicząca
Kanapkowa
54
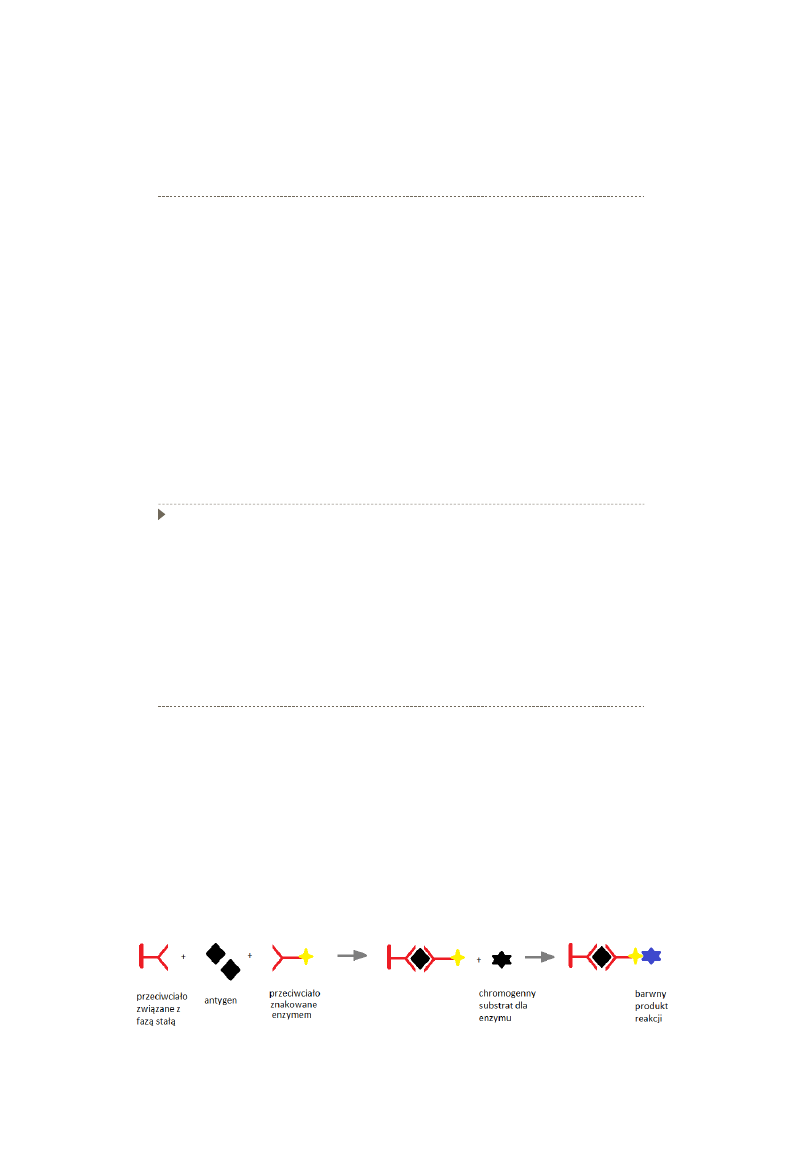
Wykład 4
28
Metody immunoenzymatyczne - ELISA
Metoda współzawodnicząca:
Specyficzne przeciwciało wiązane jest ze stałą fazą (np. powierzchnia mikropłytki
Dodanie określonej ilości antygenu oznakowanego enzymem oraz próba badana
(antygen nieoznakowany - nie połączony z enzymem)
Współzawodnictwo (kompetycja) o miejsce wiązania pomiędzy antygenem
oznakowanym i nieoznakowanym
Ilość oznakowanego antygenu związanego z przeciwciałem jest odwrotnie proporcjonalna do
zawartości antygenu w próbie badanej
.
Odmiana bezpośrednia – stosowane są oczyszczone, oznakowane przeciwciała
specyficzne dla antygenu – wykrywany jest antygen
Odmiana pośrednia – nieoznakowane przeciwciało specyficzne dla antygenu, a następnie
użycie wtórnego przeciwciała, połączonego z enzymem
55
Metody immunoenzymatyczne - ELISA
Metoda kanapkowa:
Antygen z badanej próby jest unieruchomiony pomiędzy dwoma warstwami przeciwciał
Przeciwciało specyficzne dla antygenu związane jest z fazą stałą
Jeżeli w badanej próbie jest antygen – wiąże się on z przeciwciałem i zostaje
unieruchomiony na nośniku. Niezwiązane antygeny są wymywane.
Druga porcja specyficznych przeciwciał jest wyznakowana enzymatycznie.
Barwny kompleks powstaje w wyniku reakcji immunoenzymatycznej
56
Wykład 4
29
Metody immunoenzymatyczne - ELISA
57
Metody immunochromatograficzne
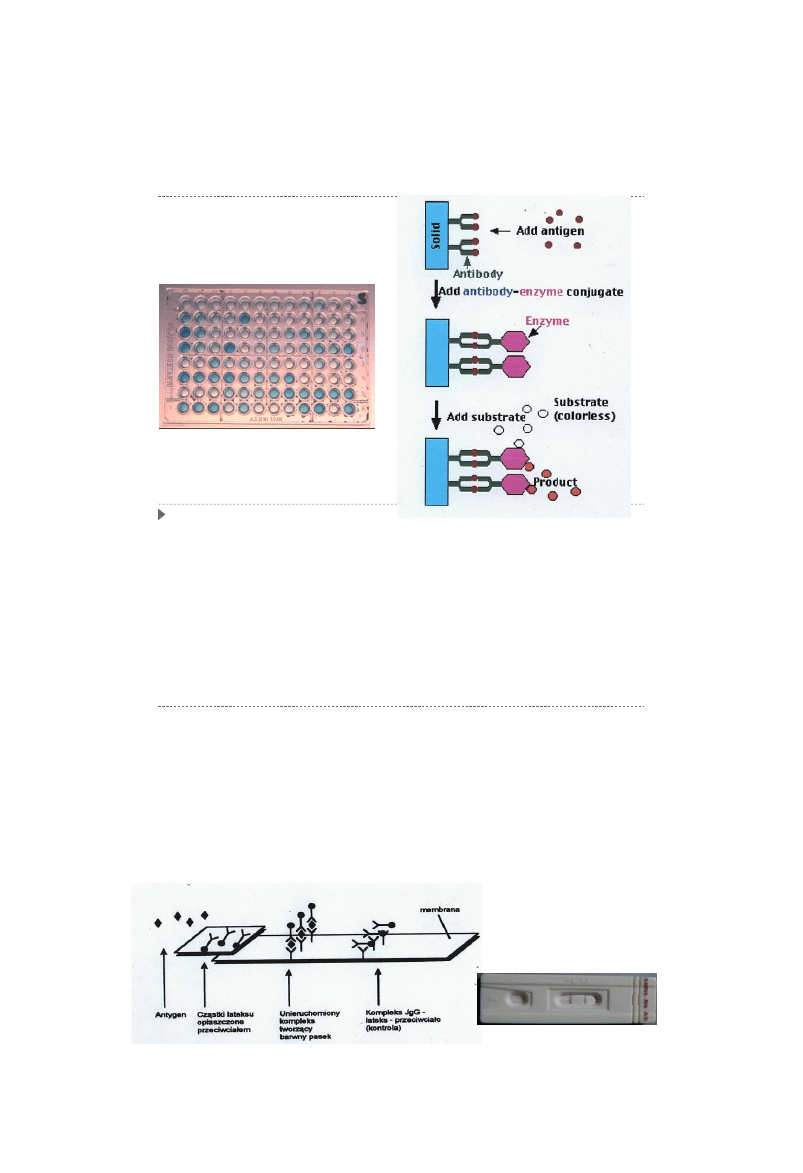
Test kanapkowy ELISA
Zabarwione cząstki lateksu opłaszczone przeciwciałem po wprowadzeniu antygenu z badanym
materiałem migrują wzdłuż membrany.
Kompleks antygen-przeciwciał jest unieruchamiany na pewnej wysokości wskutek reakcji
antygenu z unieruchomionym przeciwciałem monoklonalnym tworząc barwny pasek.
Niezwiązany barwny lateks połączony z przeciwciałami migruje dalej, napotykając na linię
utworzoną z IgG i tworzy pasek kontrolny.
58
Wyszukiwarka
Podobne podstrony:
MP 3 Pozyskiwanie szczepów mikroorganizmów o znaczeniu przemysłowym cz 1a
Pozyskiwanie szczepów mikroorganizmów o znaczeniu przemysłowym
MP 5 Doskonalenie cech produkcyjnych mikroorganizmów o znaczeniu przemysłowym cz 1
MP 5 Doskonalenie cech produkcyjnych mikroorganizmów o znaczeniu przemysłowym cz 1
MP 7 Metody przechowywania szczepów mikroorganizmów przemysłowych
POZYSKIWANIE SZCZEPÓW PRZEMYSŁOWYCH
MP 15 Zastosowanie mikroorganizmów w przemyśle chemicznym
MP 14 Zastosowanie mikroorganizmów w przemyśle farmaceutycznym
Mackiewicz, Szczepkowska Szczes Znaczenie otoczenia w wychowani
MIKROORGANIZMY UŻYTECZNE PRZEMYSŁOWO
karty cz 2a
03 K Blanchard cz 2a Wykresy do Rozdz 5
Ochrona własności przemysłowej cz 4
Ochrona własności przemysłowej cz 5
gdańsk5 pozyskiwanie szczepów
L Gorniak PS13 KONSPEKT cz 2a Konstrukty osobiste G Kelly, Uniwersytet Ekonomiczny w Krakowie - STOS
przemys� cz 4
więcej podobnych podstron