1
Replikacja DNA
Proces enzymatycznej syntezy nici DNA komplementarnych do istniejącej w komórce
cząsteczki DNA - stanowiącej matrycę dla tej reakcji.
W efekcie procesu replikacji ilość DNA w komórce ulega podwojeniu.
MATRYCOWA NIĆ DNA
POLIMERAZA DNA
wolna grupa hydroksylowa 3’
5’
3’
5’
SUBSTRATY
– 5’ TRIFOSFORANY NUKLEOZYDÓW
ELEMENTY NIEZBĘDNE DLA PRZEBIEGU PROCESU REPLIKACJI DNA
OH
OH
O H
O H
O H
O H
2
OH
HO
OH
OH
OH
OH
3’
5’
5’
3’
5’
3’
5’
3’
5’
3’
5’
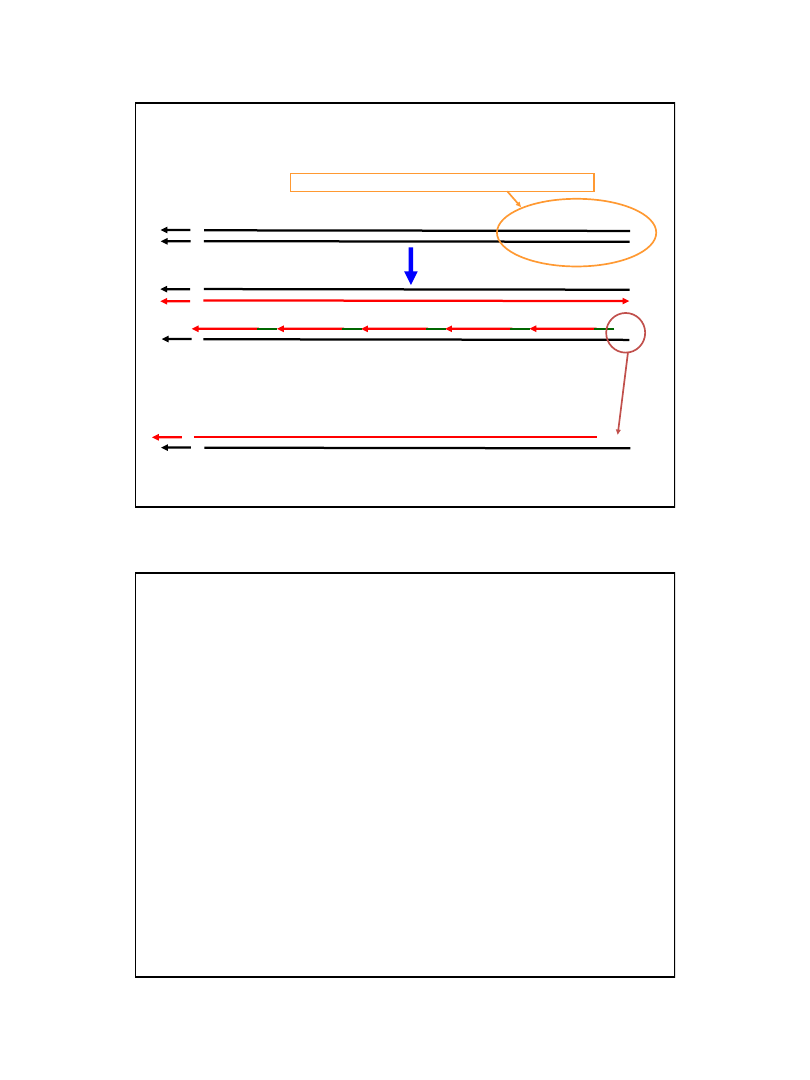
nić opóźniona
nić wiodąca
fragmenty Okazaki
kierunek przemieszczania
widełek replikacyjnych
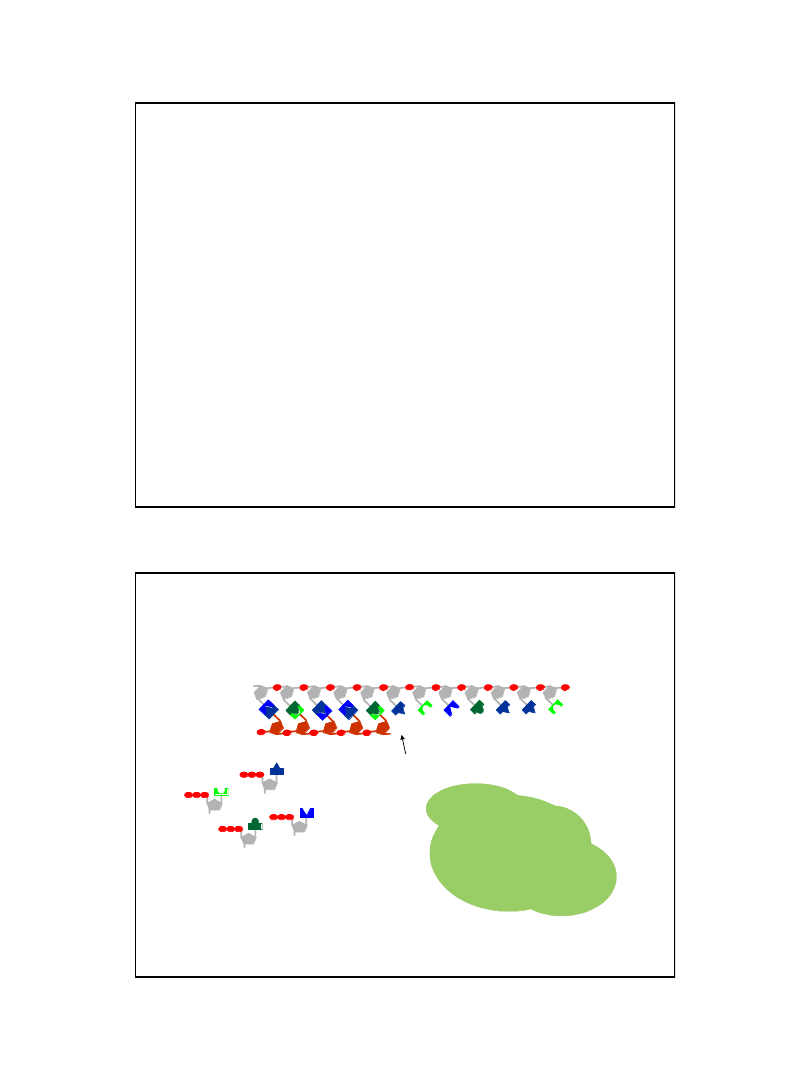
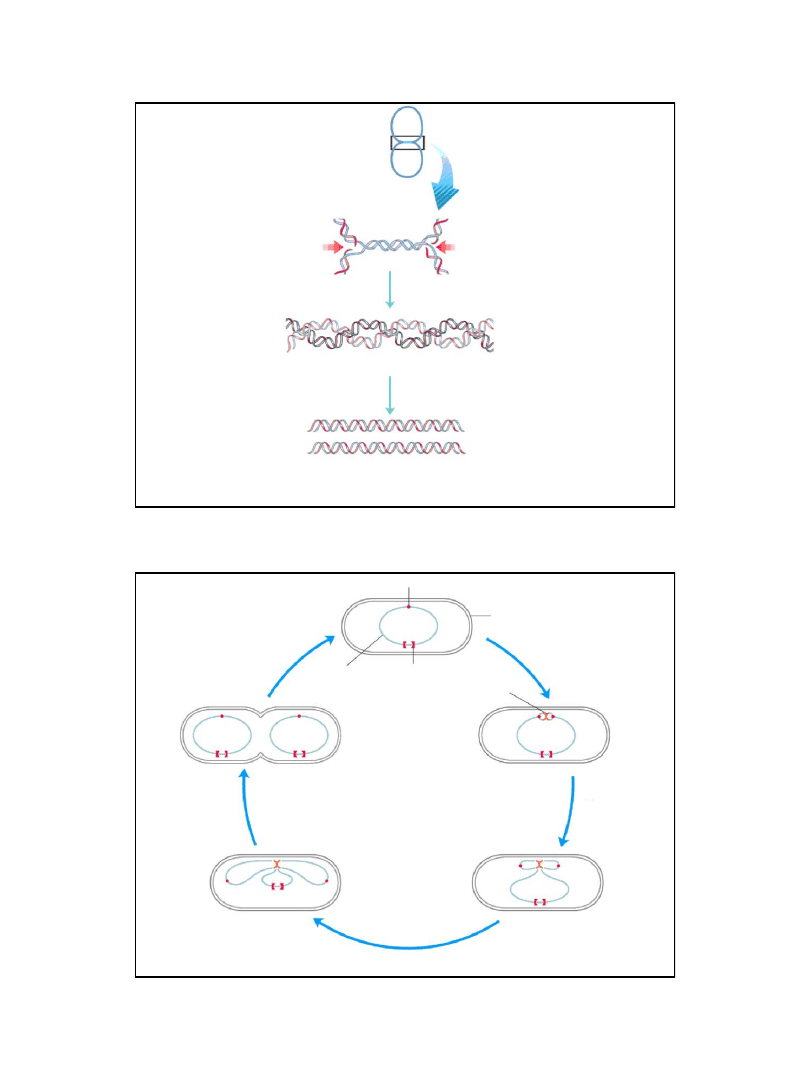
Struktura widełek replikacyjnych
Jedna z potomnych nici DNA jest syntetyzowana w sposób ciągły – w kierunku zgodnym
z przemieszczaniem się widełek replikacyjnych. Nazywamy ją nicią wiodącą (leading strand).
Druga z potomnych nici DNA
– zwana nicią opóźnioną (lagging strand) jest syntetyzowana
w postaci krótkich odcinków zwanych fragmentami Okazaki.
1
2
3
3
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
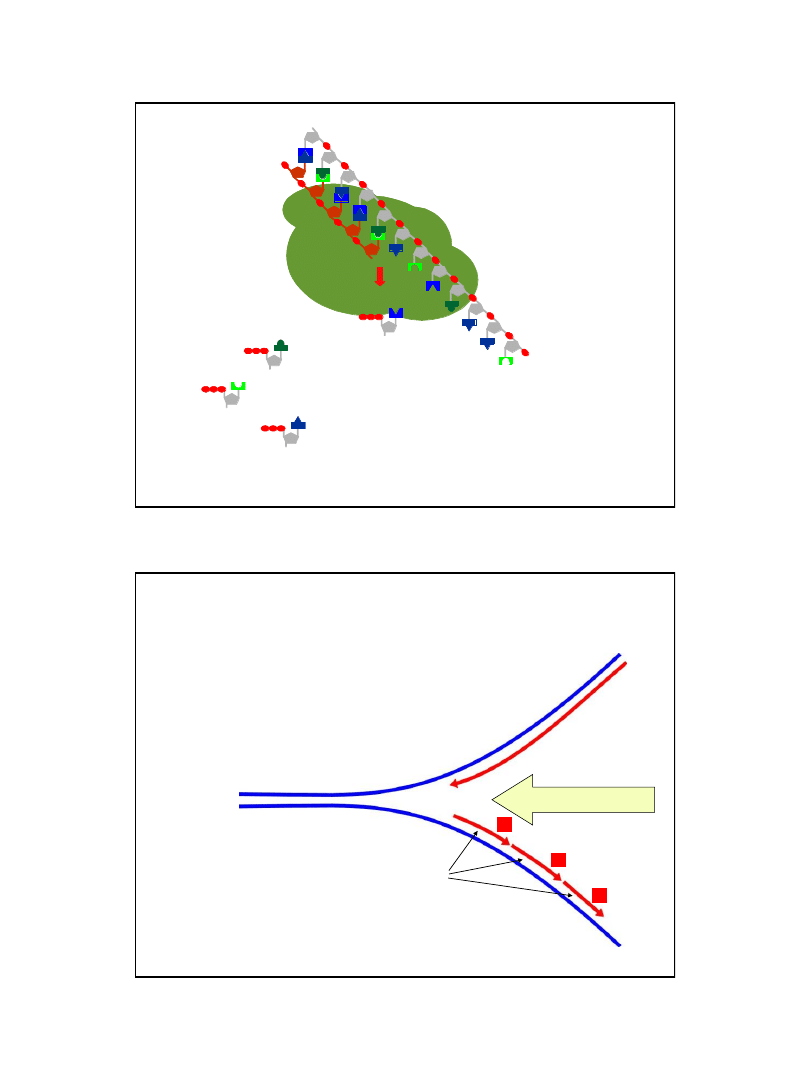
DNA wyizolowany z komórek kultury Escherichia coli utrzymywanej przez
wiele pokoleń w obecności
[
15
N]
NH4Cl, jako jedynego źródła azotu,
poddano wirowaniu w gradiencie gęstości CsCl
Komórki przeniesiono do pożywki zawierającej wyłącznie
14
N
DNA wyizolowano po czasie odpowiadającym jednemu
podziałowi komórkowemu
.... oraz po czasie odpowiadającym dwom podziałom
komórkowym
[
15
N]
DNA
[
15
N]
/
[
14
N]
DNA
[
14
N]
DNA
[
15
N]
/
[
14
N]
DNA
Doświadczenie Meselsona i Stahla (1957)
Dowód doświadczalny na semikonserwatywność procesu replikacji DNA
wg. Alberts B., Johnson A., Lewis J., Raff M., Roberts K., Walter P., 2002,
Molecular Biology of the Cell, 4th. Ed. Garland Publishing Inc.
75
100/0
25
50
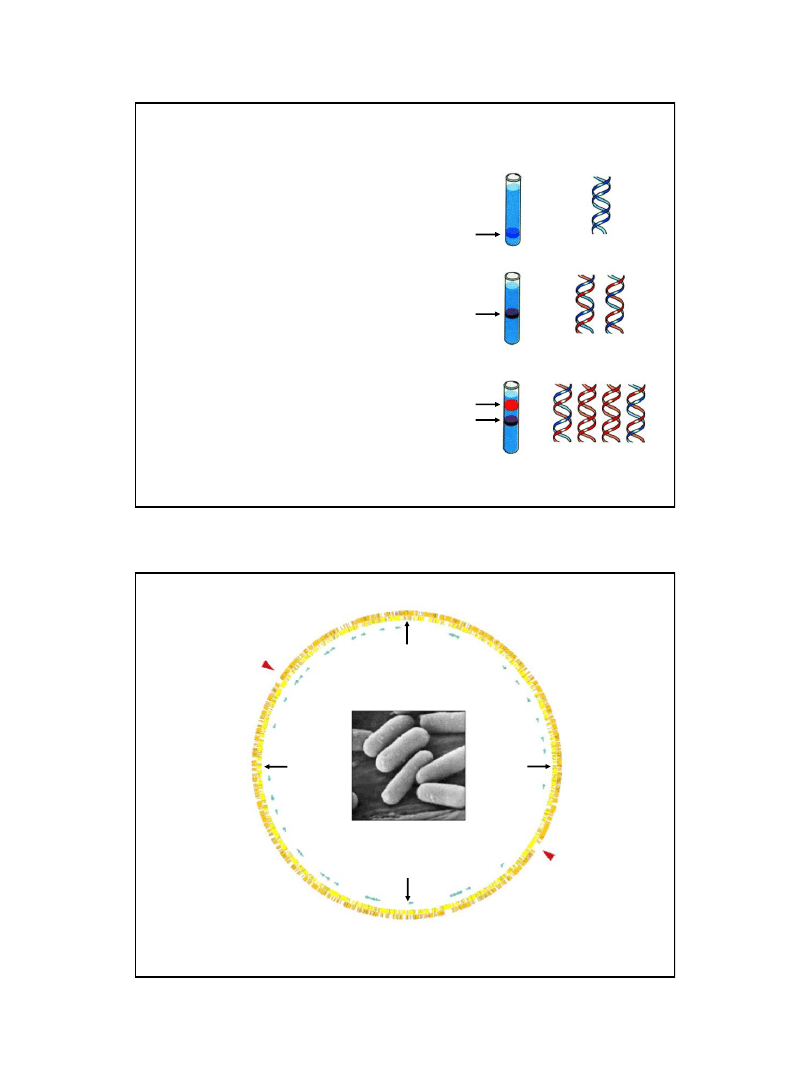
oriC
miejsce inicjacji
replikacji
sekwencje
terminacyjne
Mapa genetyczna Escherichia coli
Escherichia coli K12
4
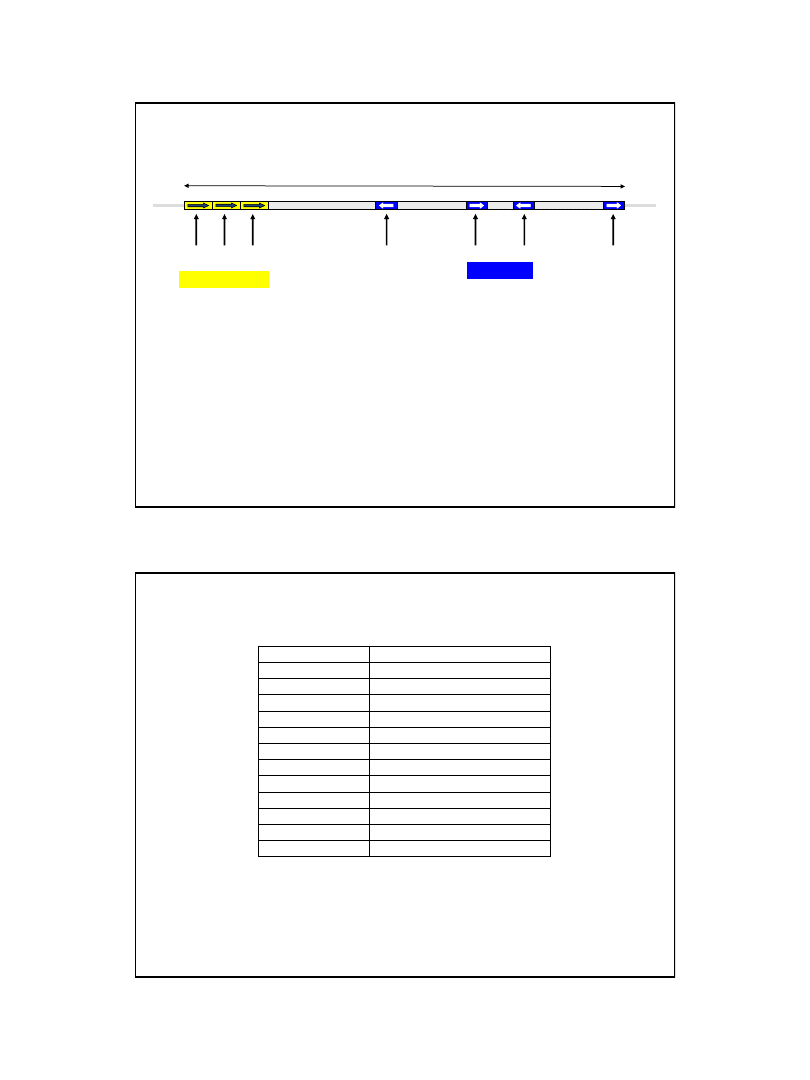
Struktura miejsca startu replikacji (oriC) chromosomu Escherichia coli
GATCTNTTNTTTT
TTATCCACA
Trzy tandemowe powtórzenia
sekwencji 13 pz.
Miejsca wiązania białka DnaA cztery sekwencje po 9 pz.
ok. 245 pz.
nazwa grupy genów*
Objaśnienie nazwy - funkcja
mut
mutageneza
dna
replikacja DNA
pol
polimeraza DNA
rpo
polimeraza RNA
uvr
odporność na promieniowanie UV
rec
rekombinacja DNA
ter
miejsce terminacji (zakończenia) replikacji
ori
miejsce startu replikacji
dam
metylacja nukleotydów adeninowych
lig
ligaza DNA
cou
oporność na kumerycynę**
nal
oporność na kwas nalidyksowy**
*
w nazewnictwie genów (i produktów ich ekspresji) przyjęto konwencję, którą ilustruje następujący przykład:
dnaC -
gen (sekwencja DNA) kodujący białko DnaC,
recA -
gen kodujący białko RecA i.t.d.
**
kumerycyna (coumericin) i kwas nalidyksowy (nalidixic acid) są antybiotykami blokującymi replikację DNA
poprzez wiązanie się z podjednostkami gyrazy DNA
Niektóre grupy genów Escherichia coli uczestniczące w procesach
związanych z metabolizmem DNA
5
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
cztery powtórzenia
sekwencji 9 pz.
Trzy powtórzenia
sekwencji 13 pz.
DnaA
Hu
+
DnaB
DnaC
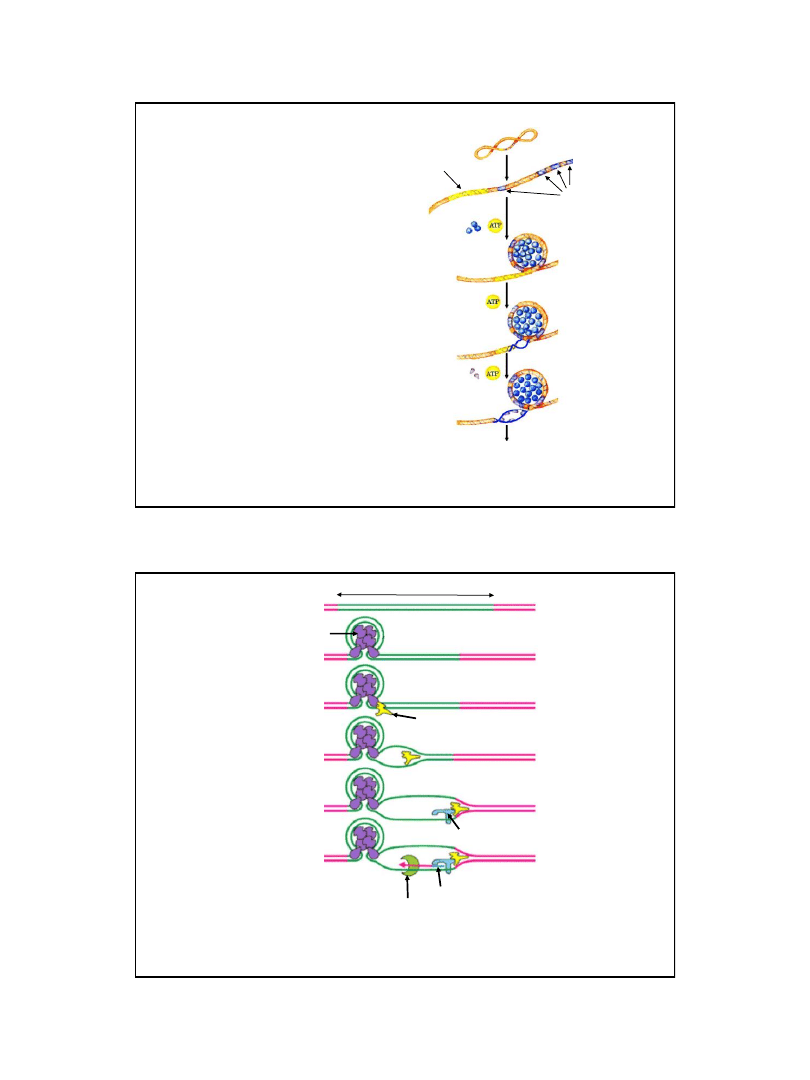
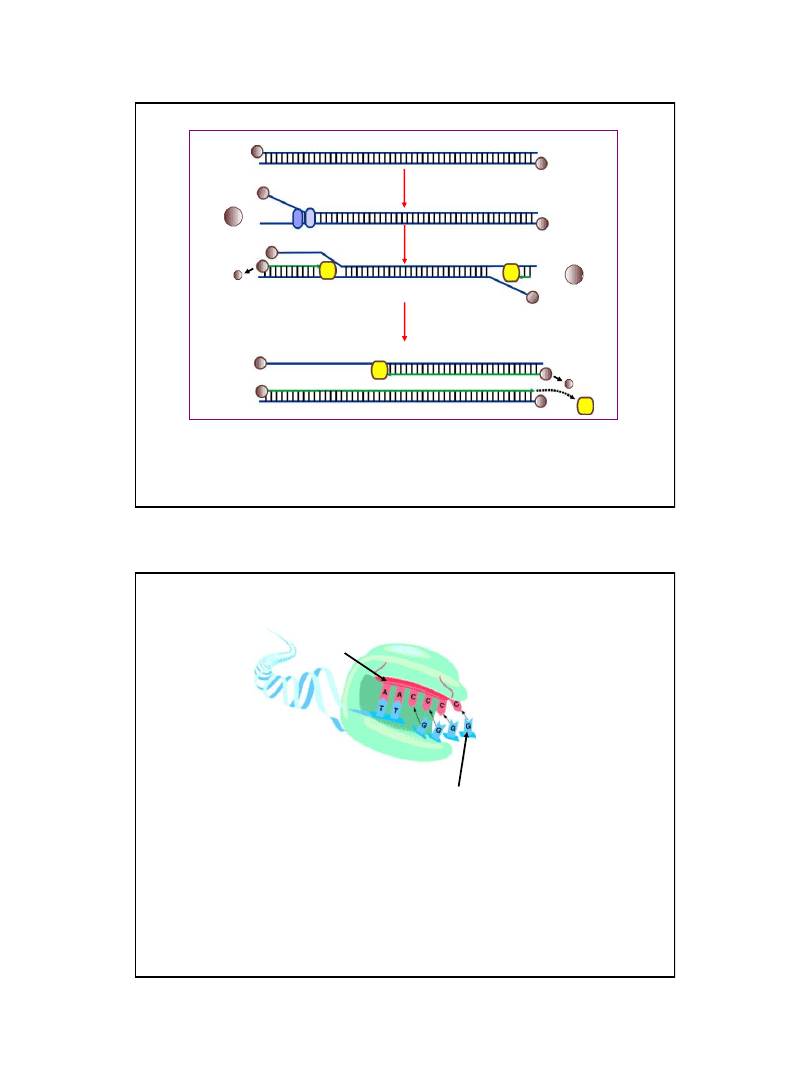
Synteza starterów i zainicjowanie replikacji
Kompleks
ok. 20 cząsteczek białka DnaA wiąże się z powtarzalnymi
sekwencjami 9 pz. w obrębie ori C.
W obecności ATP, oraz białek HU (a także najprawdopodobniej białek FIS
i IHF
), cząsteczka DNA zagina się oplatając agregat białek DnaA.
Naprężenie powstające w cząsteczce DNA powoduje zrywanie wiązań
wodorowych w obrębie powtarzających się sekwencji 13 pz.
Do kompleksu otwartego przyłącza się helikaza (białko DnaB) występująca
w połączeniu z białkiem DnaC w postaci podwójnego heksameru (DnaB
6
x DnaC
6
)
Białko DnaC reaguje z DnaA uwalniając aktywną formę helikazy(DnaB
6
).
Helikazy rozwijają nici DNA wypierając białko DnaA.
Rozdzielone nici są stabilizowane przez białka SSB.
Miejsce inicjacji replikacji (ori
C) ulega aktywacji przez transkrypcję
promotora gid
A, w wyniku której następuje zwinięcie odcinka DNA
w dodatkowe ujemne superskręty – niezbędne do otwarcia
dwuniciowej struktury helisy
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
oriC
polimeraza DNA
białka DnaA
helikaza (DnaB)
primaza (DnaG)
starter (RNA)
Inicjacja replikacji w miejscu oriC chromosomu E. coli (2)
6
Białka niezbędne dla zainicjowania replikacji w miejscu oriC Escherichia coli
Białko
masa
cząst.
l. podj.
Funkcja
DnaA
50 000
1
Otwiera dupleks DNA w specyficznych miejscach
regionu oriC
DnaB (helikaza)
300 000
6 (ident.)
Rozkręca dupleks DNA
DnaC
29 000
1
Jest wymagane dla wiązania DnaB w miejscu startu
replikacji
HU
19 000
2
Białko podobne do histonu, stymuluje inicjację replikacji
Primaza (DnaG)
60 000
1
Syntetyzuje starter RNA
SSB
75 600
4 (ident.)
Wiąże się z pojedynczą nicią DNA
Polimeraza RNA
454 000
6
Umożliwia pojawienie się aktywności DnaA
Topoizomeraza II
(giraza DNA)
400 000
4
Usuwa naprężenia powstałe po rozkręceniu DNA
z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
primaza
5’
3’
5’
3’
5’
3’
HO
-
HO
-
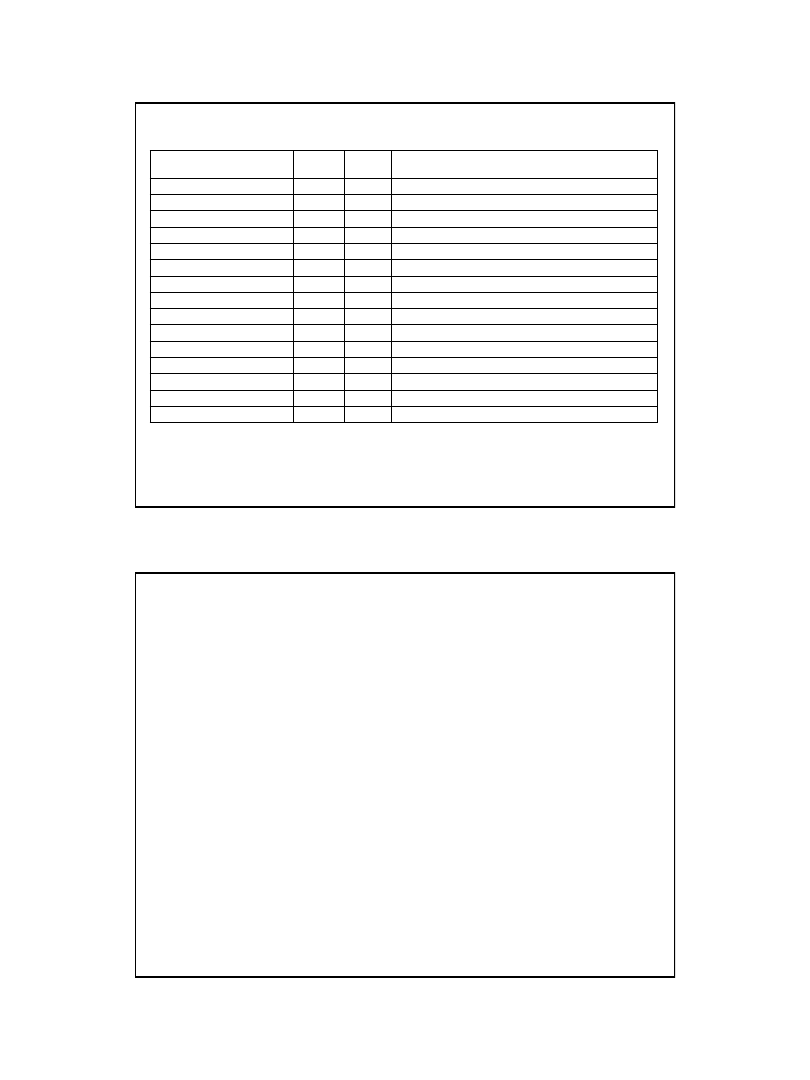
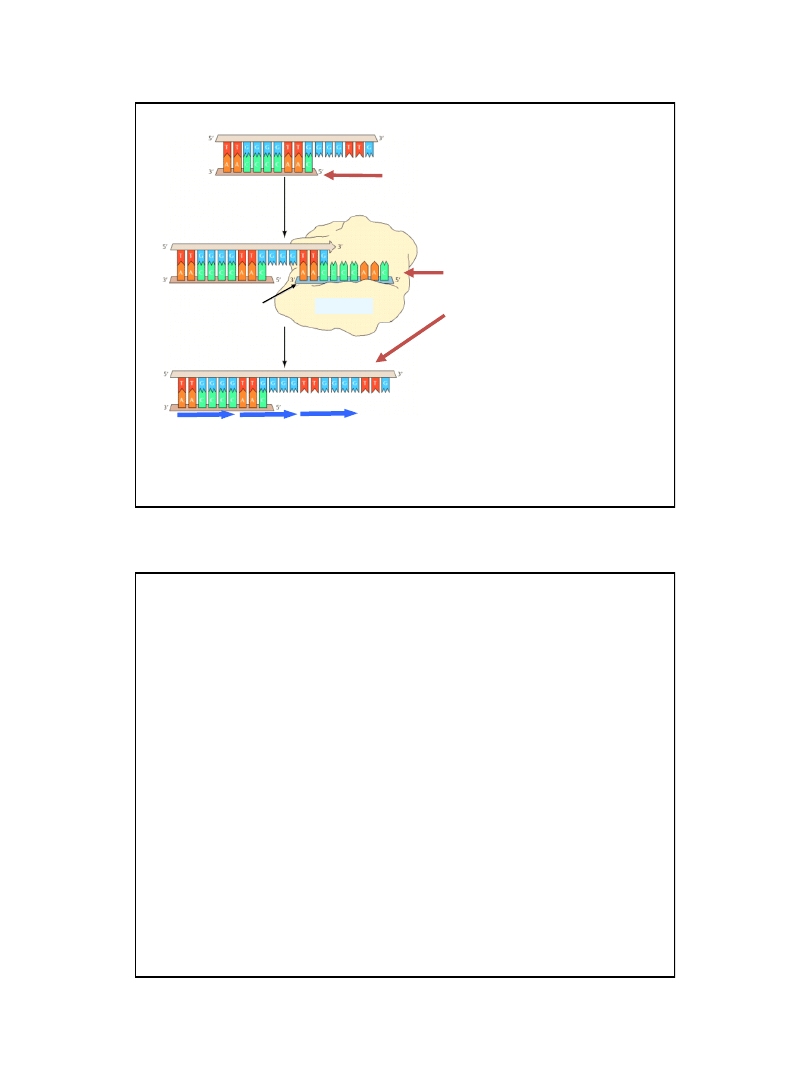
Przebieg reakcji syntezy starterów dla procesu replikacji DNA
reakcja jest katalizowana przez enzym zwany
primazą DNA
Primaza
Specyficzna polimeraza RNA katalizująca syntezę oligonukleotydów RNA na matrycy DNA
w miejscu startu replikacji i później, w czasie przesuwania się widełek replikacyjnych.
Oligonukleotydy te są starterami rozpoznawanymi przez polimerazę III DNA w czasie replikacji
(zarówno nici wiodącej, jak i fragmentów Okazaki nici opóźnionej).
Primaza (E. coli
) jest enzymem monomerycznym o m. cząst. 60 000Da.
Syntetyzowane przez nią oligonukleotydy RNA (startery) mają in vivo długość 11 ± 1 pz.
Primaza włączona jest w strukturę primosomu.
7
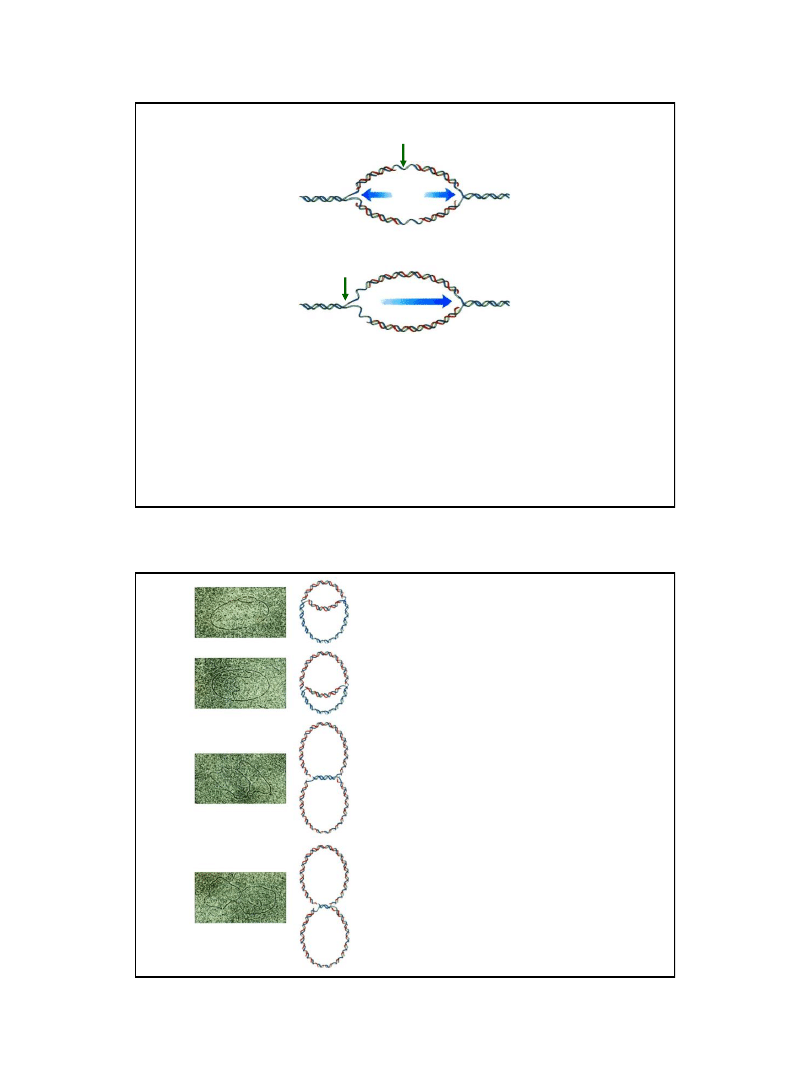
Replikacja może przebiegać dwukierunkowo lub jednokierunkowo
W większości przypadków obserwujemy replikację dwukierunkową:
- replikacja typu
θ chromosomów bakteryjnych,
- replikacja eukariotycznego DNA.
Niektóre replikony ulegają replikacji przebiegającej jednokierunkowo:
- replikacja typu
ζ (forma obracającego się koła) - DNA niektórych bakteriofagów,
-
replikacja typu powiększającej się pętli (D-loop, R-loop) - plazmidowe DNA,
mitochondrialne DNA
Replikacja jednokierunkowa
miejsce startu
replikacji
Replikacja dwukierunkowa
miejsce startu
replikacji
kierunek ruchu
widełek replikacyjnych
kierunek ruchu
widełek replikacyjnych
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
Replikacja kolistego chromosomu bakteryjnego (E. coli)
powoduje powstanie struktury przypominającej kształtem
grecką literę theta (Θ).
Ruch widełek replikacyjnych jest dwukierunkowy -
poruszają się w przeciwnych kierunkach.

8
Ryc. z: Lewin B., (2000), GenesVII,
Oxford University Press.
Mikrofotografia elektronowa kompleksu inicjującego
powstającego w miejscu oriC chromosomu E. coli. Kompleks
uwidoczniono stosując przeciwciała specyficzne wobec
białek DnaB (helikaza).
fot. Barbara Furnell
przed rozpoczęciem replikacji
w 1 minutę po inicjacji replikacji
Mikrofotografia elektronowa kompleksu inicjującego
powstającego w miejscu oriC chromosomu E. coli. Kompleks
uwidoczniono stosując przeciwciała specyficzne wobec
białek DnaB (helikaza).
Replikacja typu
θ
(chromosomu Escherichia coli)
Nowe nici DNA („oczko” replikacyjne) można rozpoznać po tym, że są to fragmenty cząsteczki
o identycznej długości – jeżeli zmierzyć odległość pomiędzy widełkami replikacyjnymi
Ryc. z: Lewin B., (2000), Genes VII, Oxford University Press
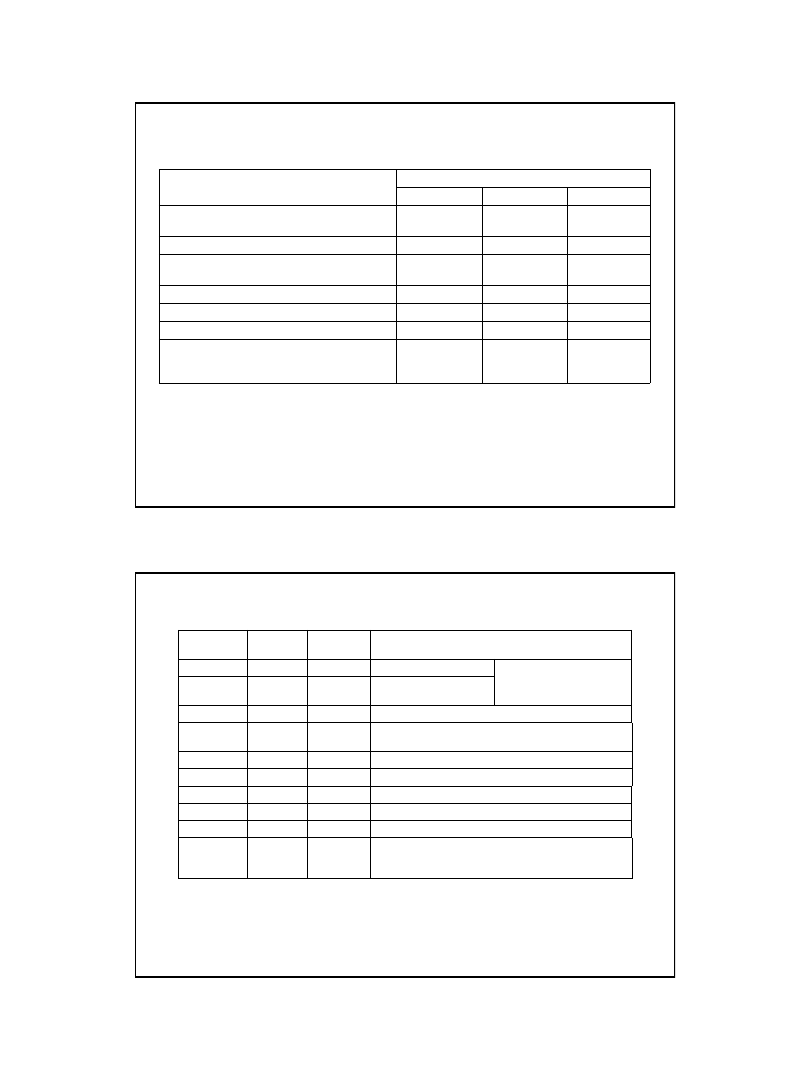
9
Porównanie polimeraz DNA z Escherichia coli
Polimeraza DNA
I
II
III
Gen strukturalny
(podjednostki o aktywności polimerazowej)
polA
polB (dnaA)
polC (dnaE)
Liczba podjednostek
1
> 4
> 10
Masa cząsteczkowa
(podjednostki o aktywności polimerazowej)
103 000
88 000
~900 000
aktywność 3‘ → 5' egzonukleazowa
Tak
Tak
Tak
Aktywność 5‘ → 3' egzonukleazowa
Tak
Nie
Nie
Szybkość polimeryzacji (nukleot./s)
16 - 20
~ 7
250 - 1 000
Zdolność polimeryzacji
(liczba nukleotydów przyłączanych przed
dysocjacją enzymu i matrycy)
3 - 200
10 000
500 000
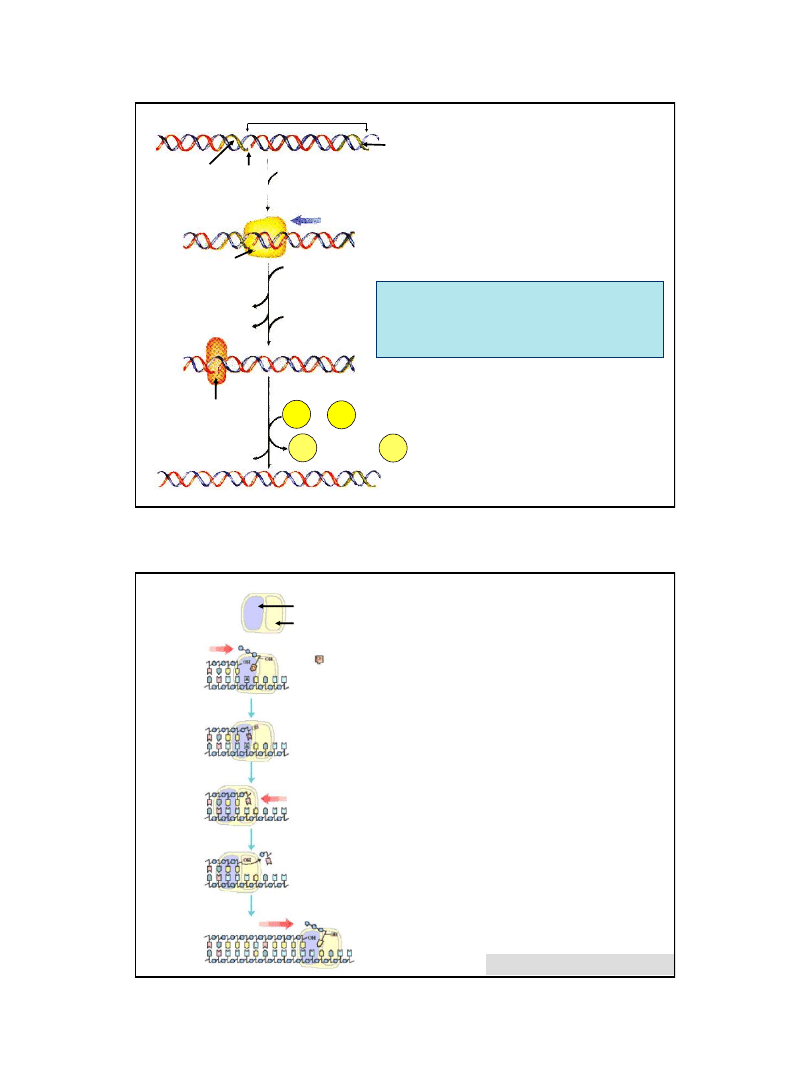
Podjednostki polimerazy III DNA z Escherichia coli
Podjednost
ka
Masa
cząst.
Gen
Funkcja
α (alfa)
132 000
pol
C (
dna
E)
aktywność polimerazowa
podjednostki rdzenia
polimerazy
ε (epsilon)
27 000
dna
Q
(
mut
D)
egzonukleaza 3'→ 5'
(aktywność korekcyjna)
θ (theta)
10 000
hol
E
-
1
τ (tau)
71 000
dna
X
stabilizacja wiązania z matrycą; dimeryzacja rdzenia
enzymu
γ (gamma)
52 000
dna
X
2
podwyższanie zdolności polimweryzacji
δ (delta)
35 000
hol
A
podwyższanie zdolności polimweryzacji
δ'
33 000
hol
B
-
χ (chi)
15 000
hol
C
-
ψ (psi)
12 000
hol
D
-
β (beta)
37 000
dna
N
ATPaza wymagana dla optymalnej zdolności
polimeryzacji
– funkcjonuje, jako „klamra” utrzymująca
holoenzym polimerazy III na matrycy DNA
1
znak "-
" umieszczony w tej rubryce oznacza, że funkcja podjednostki nie została
jednoznacznie zdefiniowana
2
podjednostka γ jest kodowana przez część genu dla podjednostki η na drodze przesunięcia
fazowego w czasie translacji, które prowadzi do przedwczesnej jej terminacji.
10
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
polimeraza III DNA na nici opóźnionej
kończy syntezę kolejnego fragmentu Okazaki
polimeraza III DNA na nici wiodącej
białka SSB
matryca dla nici opóźnionej
helikaza
primaza
starter (RNA)
synteza następnego fragmentu Okazaki
rozpocznie się w tym miejscu
fragment Okazaki
nić wiodąca
matryca dla nici wiodącej
gyraza DNA
(topoizomeraza II)
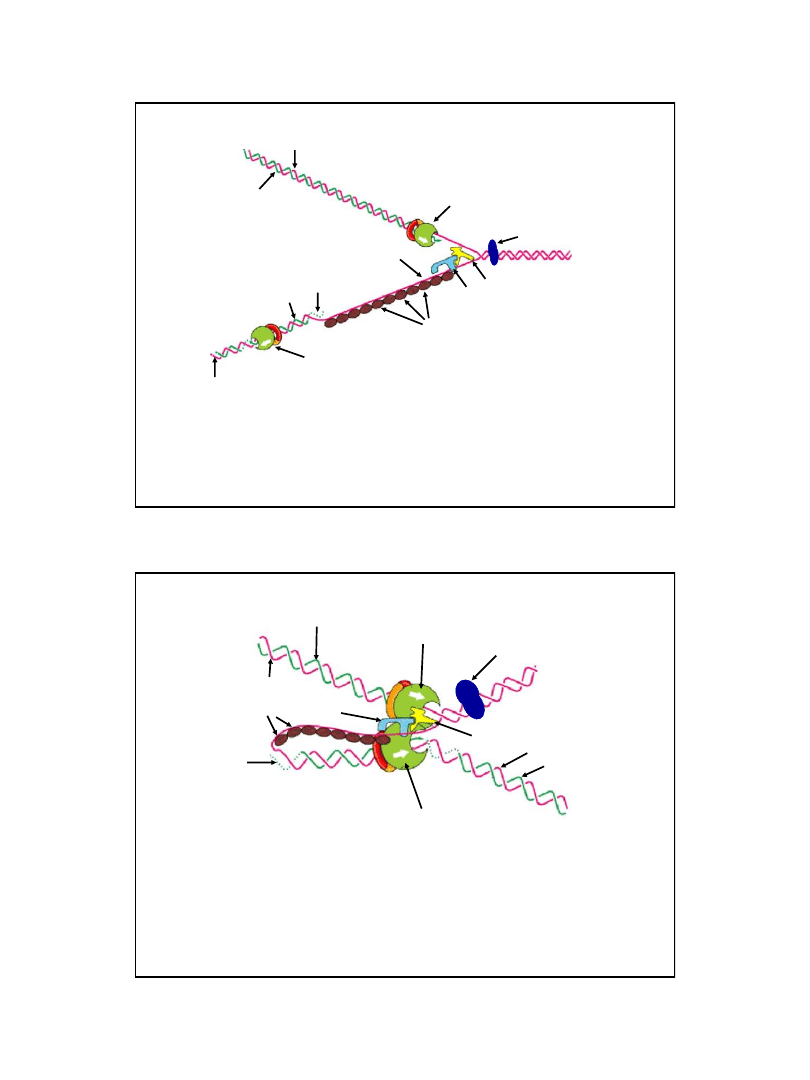
Struktura widełek replikacyjnych Escherichia coli
Zaznaczono główne białka funkcjonujące w ich obrębie
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
polimeraza III DNA
na nici wiodącej
matryca dla nici wiodącej
nić wiodąca
matryca dla nici opóźnionej
polimeraza III DNA
na nici opóźnionej
białka SSB
helikaza
primaza
starter (RNA)
fragment Okazaki
gyraza DNA
(topoizomeraza II)
Funkcjonowanie „dimeru” polimerazy III DNA w czasie replikacji
chromosomu bakteryjnego Escherichia coli
11
PAS
n (PriA)
n (PriB)
n’’ (PriC)
i (DnaT)
primaza (DnaG)
primaza
helikaza
primosom
Kompleks białkowy uczestniczący w inicjacji replikacji chromosomu bakteryjnego.
W jego skład (Escherichia coli) wchodzą białka: i (DnaT), n (PriB), n' (PriA), n'' (PriC) rozpoznające
sekwencje PAS (primosome assembly site -
miejsca składania primosomu), do których przyłączają się
pozostale białka kompleksu (primosomu):
DnaC, (DnaB)6 -
główna helikaza replikacyjna i jako ostatnie - DnaG (primaza).
Primosom
Ryz. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
białka SSB
polimeraza DNA
kooperatywne wiązanie białek SSB z łańcuchem DNA
uniemożliwia powstanie struktur dwuniciowych
Funkcja białek SSB (single-strand binding proteins) w replikacji DNA Escherichia coli
12
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
polimeraza III DNA na nici wiodącej
nić wiodąca (nowa nić DNA)
matryca dla nici opóźnionej
matryca dla nici wiodącej
5’
5’
3’
5’
3’
3’
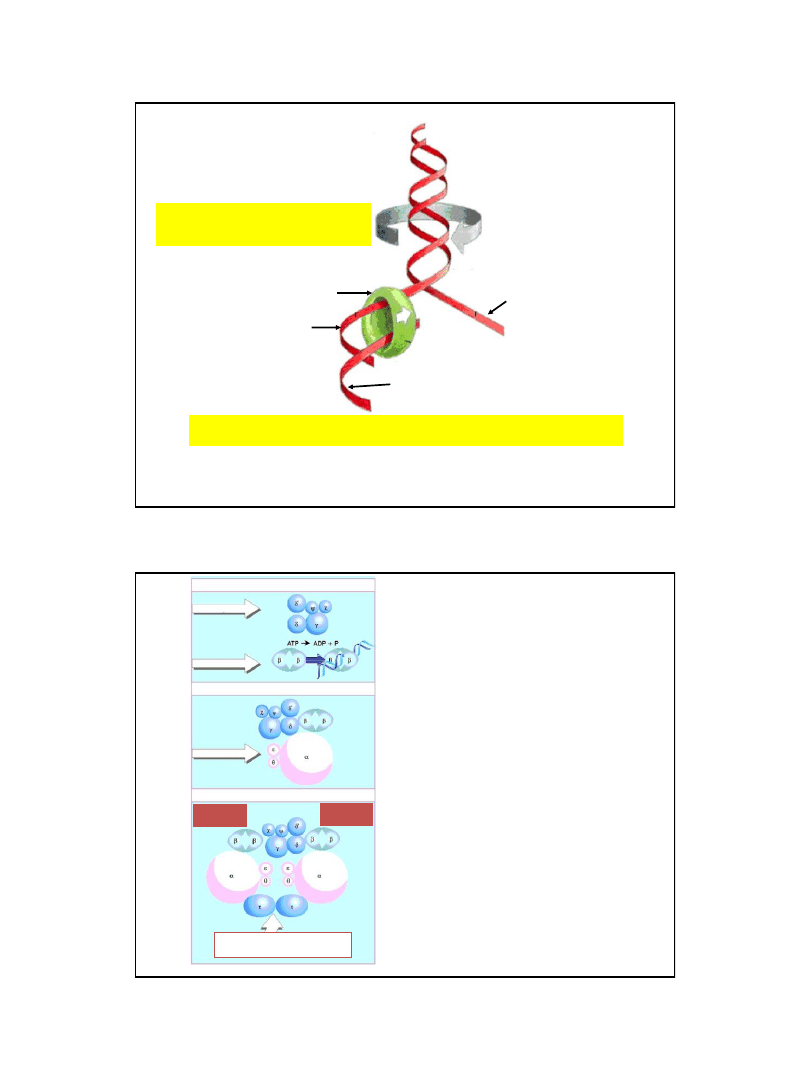
Podczas replikacji pojawia się problem
związany ze skęcaniem się rodzicielskiej
cząsteczki DNA
przy szybkości replikacji wynoszącej 500 nt./s, rodzicielski (matrycowy) DNA, przed
widełkami replikacyjnymi, powinien obracać się z szybkością 50 obrotów na sekundę
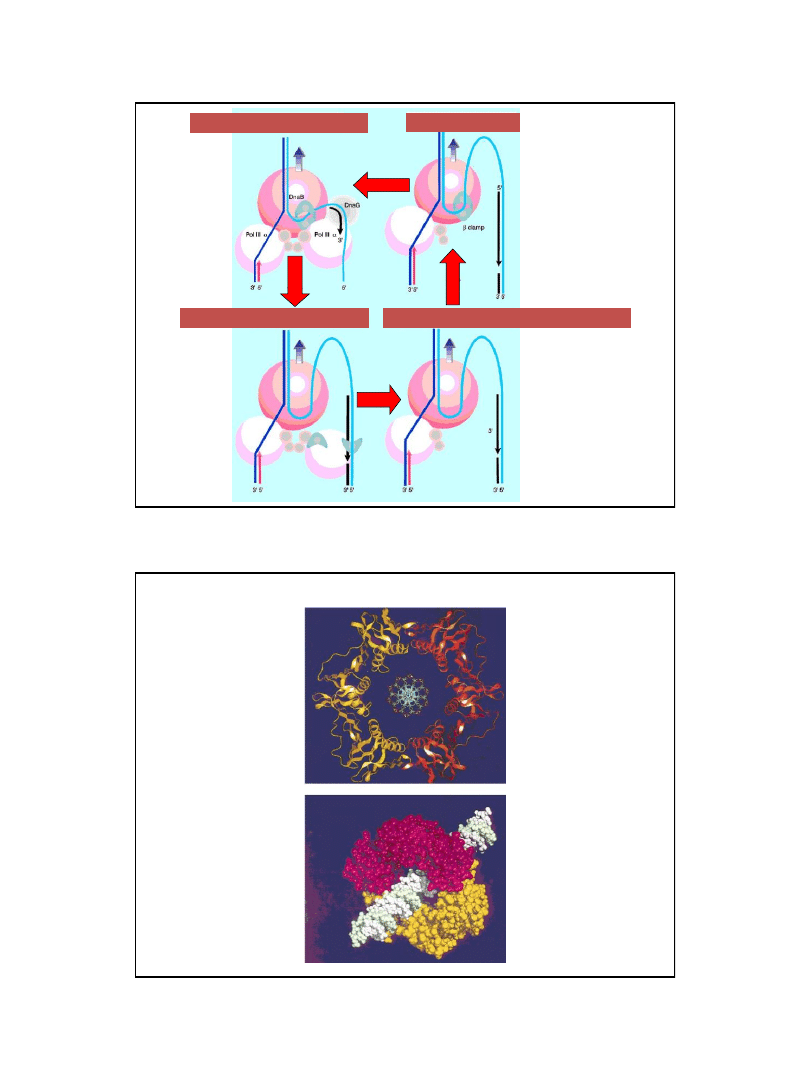
Kompleks tworzący „klamrę” hydrolizuje ATP
Kompleks
„Klamra”
Przyłącza się rdzeń polimerazy
Rdzeń
Przyłączają się
τ
oraz drugi rdze
ń
synteza nici
wiodącej
synteza nici
opóźnionej
Podjednostki
τ
stabilizują strukturę
„dimeru” polimerazy
Powstawanie holoenzymu polimerazy III DNA
Escherichia coli
w postaci „dimeru” prowadzącego
skoordynowana replikację nici wiodącej
i fragmentu Okazaki
Ryc. z: Lewin B., (2000), GenesVII,
Oxford University Press.
13
Ryc. z: Lewin B., (2000), GenesVII,
Oxford University Press.
1.
Inicjacja syntezy fragmentu Okazaki
2.
Terminacja syntezy fragmentu Okazaki
3.
Dysocjacja jednego z rdzeni Polimerazy III i klamry
β
4.
Reasocjacja klamry
β
„Klamra” utworzona przez podjednostki β
polimerazy III DNA utrzymująca holoenzym na cząsteczce DNA
Ryc. z: Lewin B., (2000), GenesVII,
Oxford University Press.
14
Białka Escherichia coli funkcjonujące w regionie widełek replikacyjnych
Białko
masa
cząst.
l. podj.
Funkcja
SSB
75 600
4 (ident.)
Wiąże się z pojedyńczą nicią DNA
białko i (DnaT)
66 000
3
Składnik primosomu
białko n
28 000
2
Tworzenie i funkcja primosomu
białko n'
76 000
1
Składnik primosomu
białko n''
17 000
1
Składnik primosomu
DnaC
29 000
1
Składnik primosomu
DnaB (helikaza)
300 000
6 (ident.)
Rozkręca dupleks DNA, Składnik primosomu
Primaza (DnaG)
60 000
1
Synteza startera RNA, składnik primosomu
polimeraza III DNA
900 000
2x10
Synteza łańcucha DNA
polimeraza I DNA
103 000
1
Wypełnianie przerw w łańcuchu DNA, wycinanie starterów RNA
Ligaza DNA
74 000
1
Ligacja DNA
Topoizomeraza II (giraza DNA)
400 000
4
Superskręcanie DNA
Rep (helikaza)
65 000
1
Rozkręcanie dupleksu DNA
Helikaza II DNA
75 000
1
Rozkręcanie dupleksu DNA
Topoizomeraza I DNA
100 000
4
Usuwa negatywne superskręty
Włączenie niewłaściwego nukleotydu – błąd w Escherichia coli zdarza się z częstością:
1 raz na 10
9
- 10
10
nukleotydów
Wielkość chromosomu E. coli 4.7 x 10
6
pz.
↓
Błąd występuje
1 raz na 1 000 - 10 000 replikacji
Dokładność procesu replikacji
15
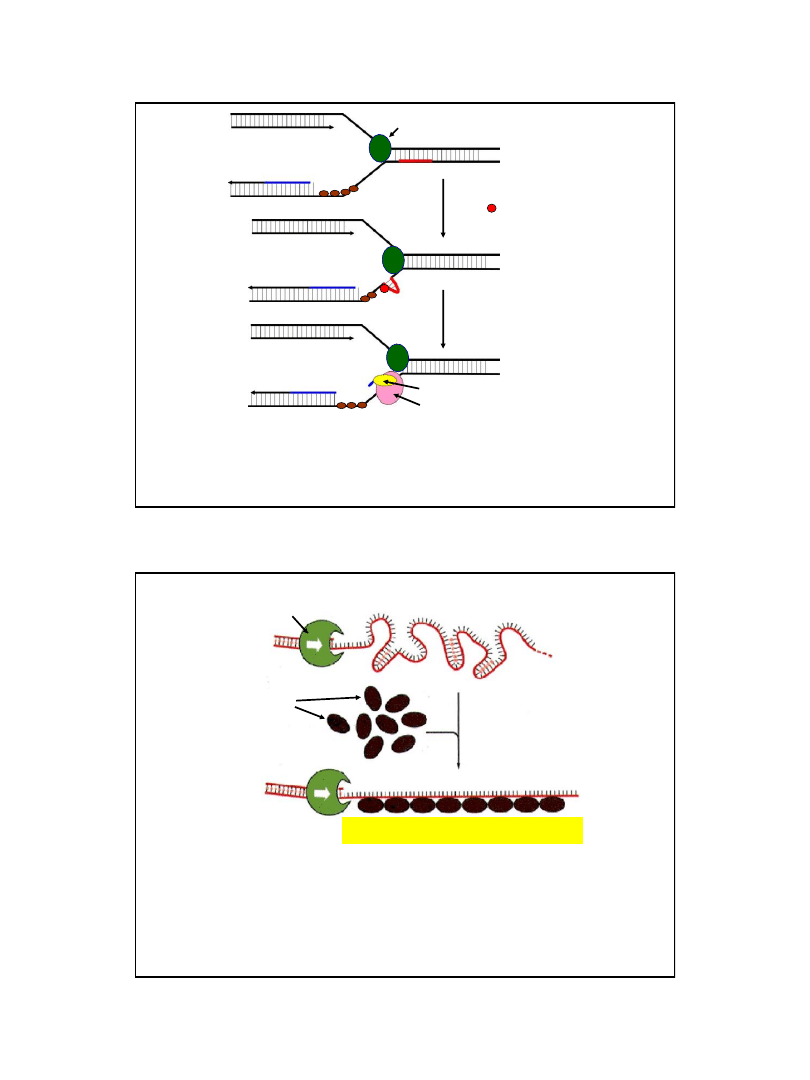
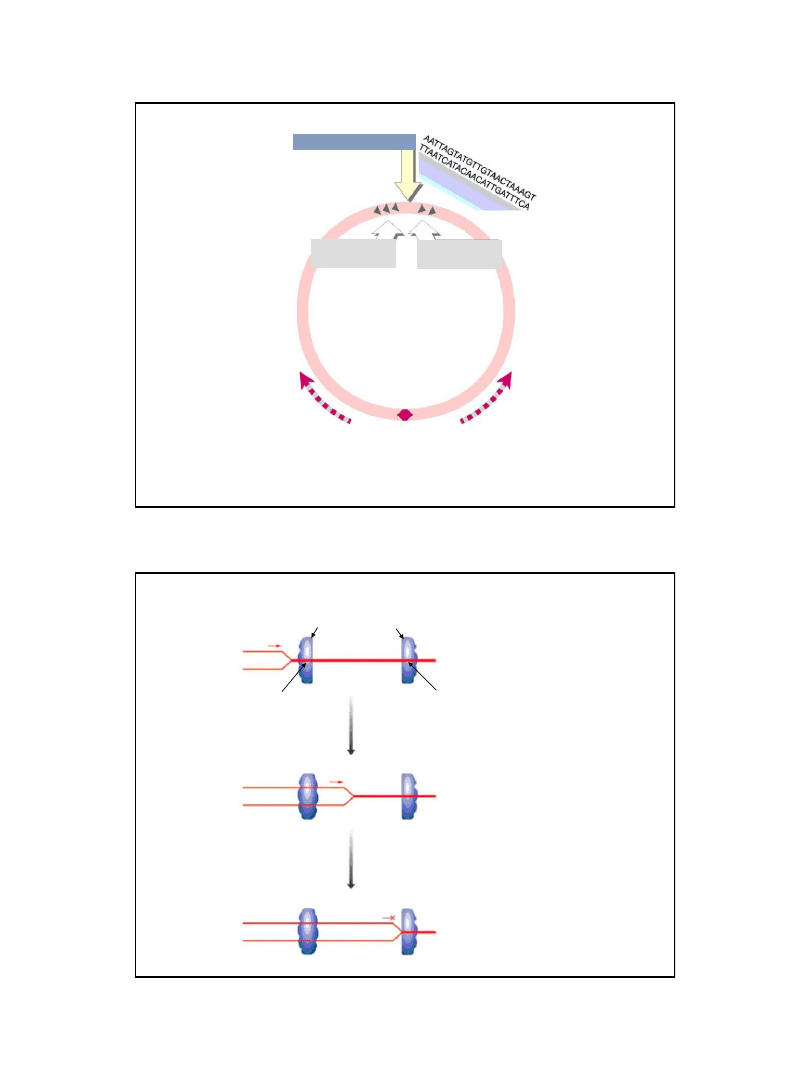
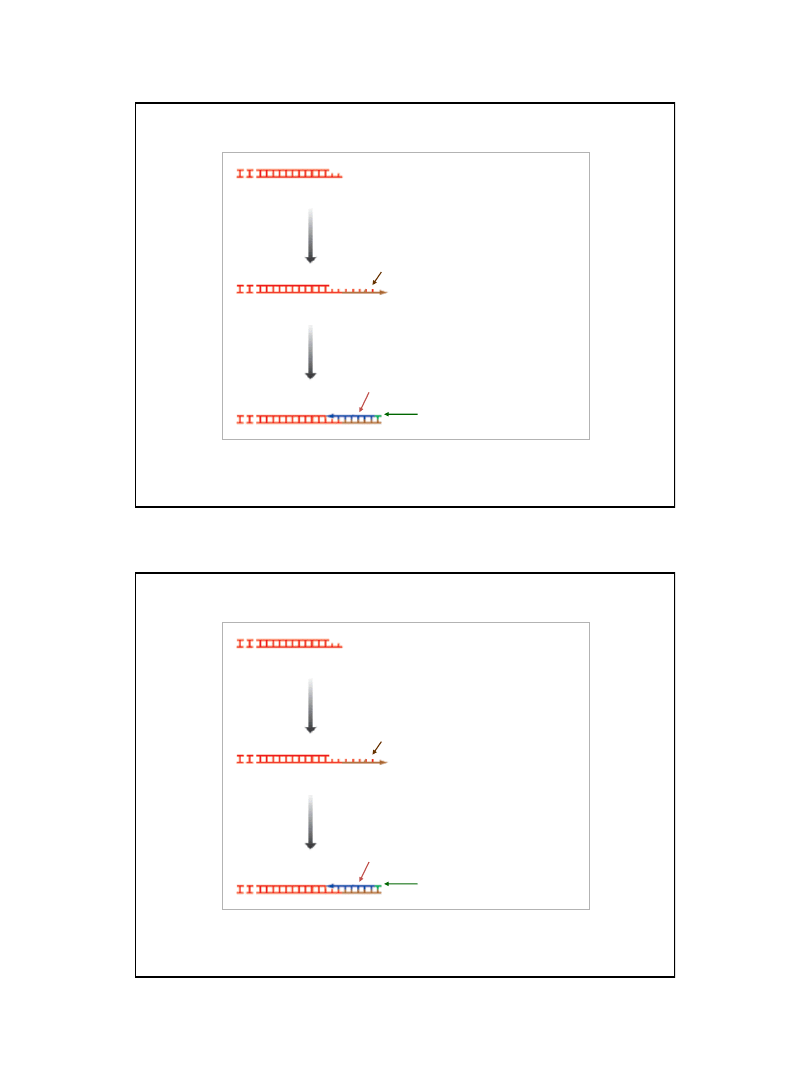
Polimeraza I DNA
starter (RNA)
przerwa
(nick)
5’
3’
starter (RNA)
dATP, dGTP, dCTP, dTTP
AMP, UMP, CMP, GMP
ligaza DNA
fragment Okazaki
ATP lub NAD+
AMP + PPi lub NMN
ligaza DNA
Polimeraza I DNA
Polimeraza I DNA
ligaza DNA
Zakończenie syntezy nici opóźnionej DNA
Usuwanie odcinków starterowych RNA (polimeraza I DNA)
oraz tworzenie wiązań fosfodiestrowych pomiędzy
fragmentami Okazaki (ligaza DNA)
Ryc. z: Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
domena o aktywności
polimerazowej (5’→3’)
domena o aktywości
3’→5’ egzonukleazowej
5’
3’
– symbol oznacza rzadką tautomeryczną formę cytozyny (C*),
która może zastąpić T. Zdarza się jej włączenie w nową nić DNA
Zanim polimeraza przesunie się wzdłuż nici matrycowej, zwykle następuje
tautomeryczne przekształcenie C*→C. Parowanie C – A nie jest możliwe.
Niesparowany koniec 3’-OH nowej nici DNA uniemożliwia elongację
łańcucha DNA. Polimeraza przesuwa się wstecz. Niesparowany nukleotyd
znajduje się teraz w obszarze domeny 3’→5’ egzonukleazowej.
Niesparowany nukleotyd zostaje usunięty
Polimeraza I DNA może kontynuować polimeryzację
(syntezę uzupełniającą)
Nie zaznaczono domeny o aktywności
5’ → 3’ egzonukleazowej
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
16
Domeny strukturalne i funkcjonalne polimerazy I DNA Escherichia coli
domena N-
końcowa
odpowiada za aktywność
egzonukleazową 5’→3’
dwie domeny strukturalne tworzące
łącznie tzw.
duży fragment
lub
–
fragment Klenowa
odpowiadają za aktywność
polimerazową 5’ → 3’
egzonukleazową 3’ → 5’
68 000 Da
ok. 35 000 Da
Cały enzym - 103 000 Da
Ryc. z: Lewin B., (2000), Genes VII, Oxford University Press
Mechanizm działania ligazy DNA
Enzym +
ATP
lub enzym +
NAD
Enzym-
AMP
Adenina-Ryboza-
+ AMP
17
oriC
widełki
replikacyjne
2
widełki
replikacyjne
1
terE,D,A
zatrzymują widełki
1
terC,B
zatrzymują widełki
2
Ryc. z: Lewin B., (2000), Genes VII, Oxford University Press
miejsce spotkania widełek
białka
Tus
wiążą się asymetrycznie
z sekwencjami
ter
ter
ter
widełki replikacyjne mogą przechodzić poprzez
cząsteczkę białka Tus w jednej z orientacji...
... lecz są zatrzymywane przez białko Tus dołączone
w orientacji przeciwnej
W terminacji replikacji u Escherichia coli uczestniczą białka Tus
Ryc. z: Brown T.A., 2002, Genomes, 2nd ed. , BIOS Scientific Publishers Ltd.
18
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
topoizomeraza IV
widełki replikacyjne 1
widełki replikacyjne 2
zakończenie procesu replikacji
rozdzielone chromosomy potomne
połączone chromosomy potomne
Zakończenie procesu replikacji
chromosomu bakteryjnego
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
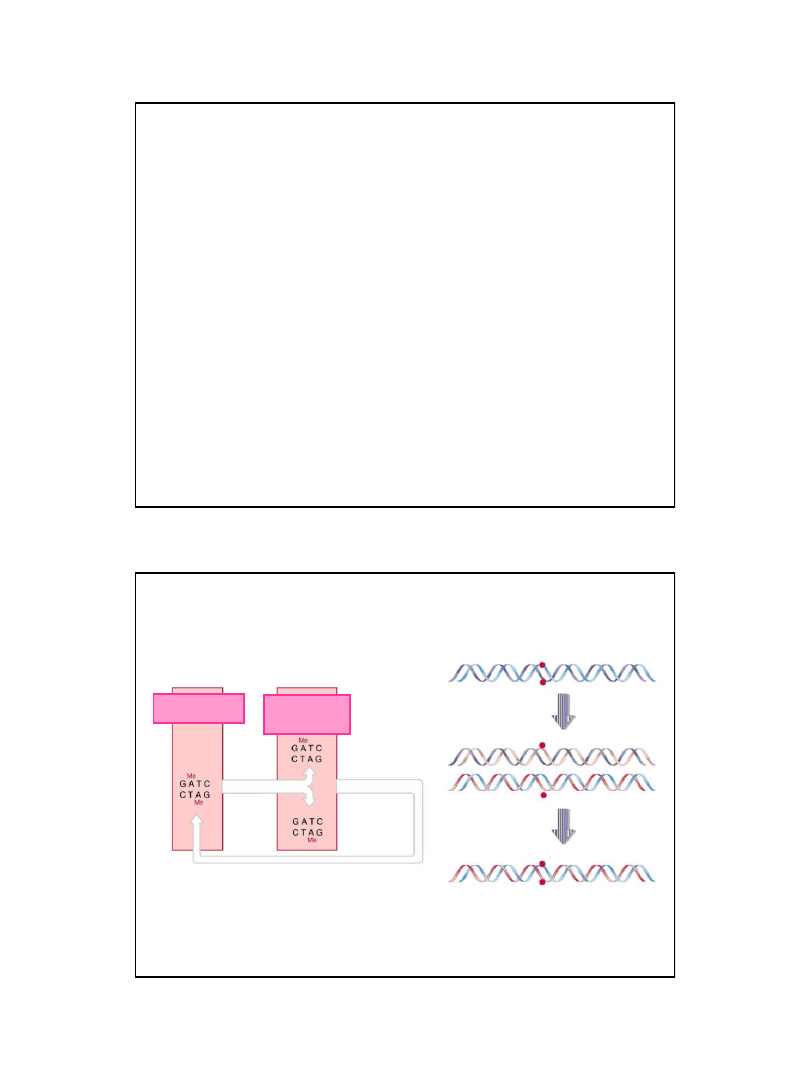
oriC
terminator
replikacji
(ter)
chromosom
komórka bakterii
podział komórki
początek replikacji
replisom
rozdzielenie
miejsc oriC
komórka wydłuża się wraz
z postępem replikacji
rozdział
chromosomów
potomnych
Replikacja DNA i podział komórki E. coli
19
1. Kluczową rolę w regulacji procesu inicjacji replikacji odgrywa transkrypcja RNA – a zatem pośrednio
mechanizmy regulujące ten proces.
a. Zdarzeniem zawsze poprzedzającym replikacj jest transkrypcja sekwencji DNA rozpoczynająca się
na promotorze gidA
. Proces ten wywołuje powstanie dodatkowych negatywnych superskrętów
umożliwiających rozplecenie dwuniciowej cząsteczki DNA.
b. Poziom transkrypcji
genów kodujących białka uczestniczące w procesie inicjacji replikacji decyduje
o powstaniu warunków dla zajścia tego zdarzenia.
2. Sygnałem dla startu procesu replikacji jest nagromadzenie się "inicjatora". Rolę tę najprawdopodobniej
spełnia białko DnaA. Białko DnaA musi wystąpić w formie zaktywowanej (w postaci kompleksu z ATP).
Kompleks DnaA-
ADP jest nieaktywny w inicjacji replikacji. Aktywacja DnaA (zastąpienie ADP przez ATP)
jest blokowane przez wiążące się z kompleksem DnaA-ADP białko IciA (inhibitor of chromosomal initiation).
3. Bezpośrednie sterowanie procesem replikacji następuje na drodze metylacji specyficznych
sekwencji sygnałowych DNA występujących w obrębie oriC.
Regulacja procesu inicjacji replikacji w komórkach Escherichia coli
Ryc. z: Lewin B., (2000), Genes VII,
Oxford University Press.
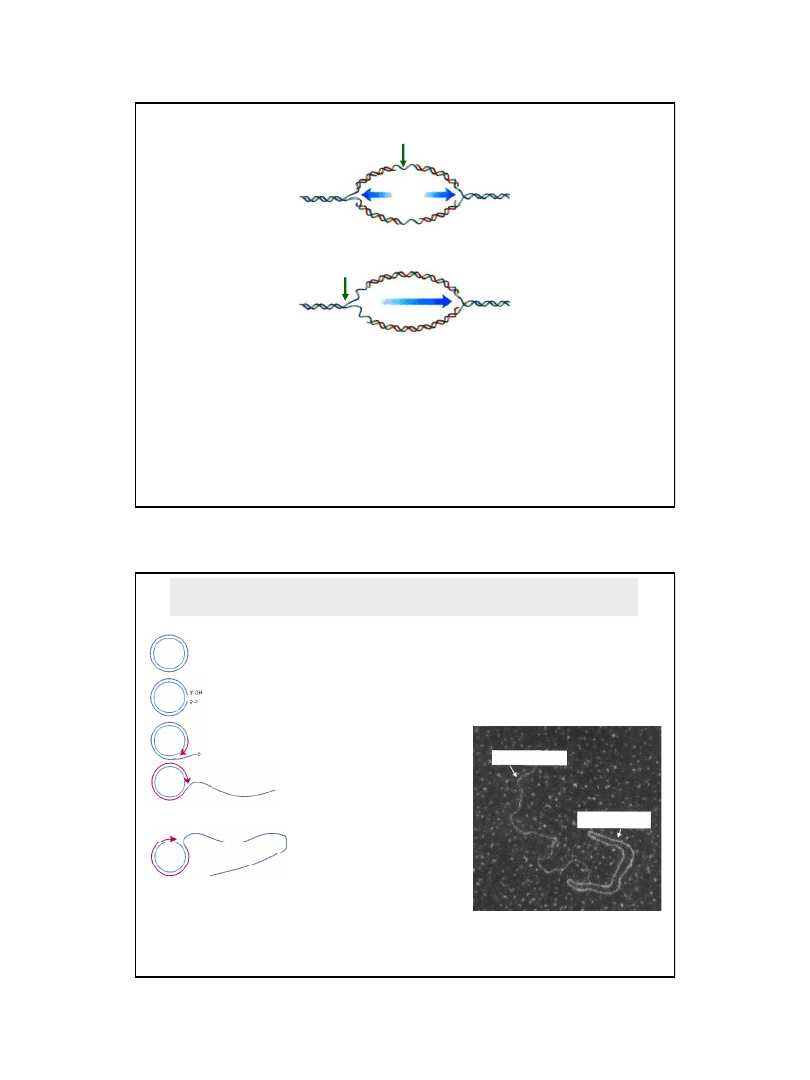
metylaza Dam
replikacja DNA
zmetylowany DNA
miejsca oriC
Hemi-
metylowany DNA
nowych oriC
Replikacja DNA
Metylaza Dam
Aktywne miejsce oriC
Aktywne miejsce oriC
Nieaktywne miejsca oriC
Metylacja DNA w miejscu ori
C może wpływać na jego aktywność
Aktywne w procesie inicjacji replikacji jest tylko w pełni zmetylowane miejsce oriC
20
Replikacja może przebiegać dwukierunkowo lub jednokierunkowo
W większości przypadków obserwujemy replikację dwukierunkową:
- replikacja typu
θ chromosomów bakteryjnych,
- replikacja eukariotycznego DNA.
Niektóre replikony ulegają replikacji przebiegającej jednokierunkowo:
- replikacja typu
ζ (forma obracającego się koła) - DNA niektórych bakteriofagów,
-
replikacja typu powiększającej się pętli (D-loop, R-loop) - plazmidowe DNA,
mitochondrialne DNA
Replikacja jednokierunkowa
miejsce startu
replikacji
Replikacja dwukierunkowa
miejsce startu
replikacji
kierunek ruchu
widełek replikacyjnych
kierunek ruchu
widełek replikacyjnych
wg. Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
Ryc. z: Lewin B., (2000), Genes VII, Oxford University Press
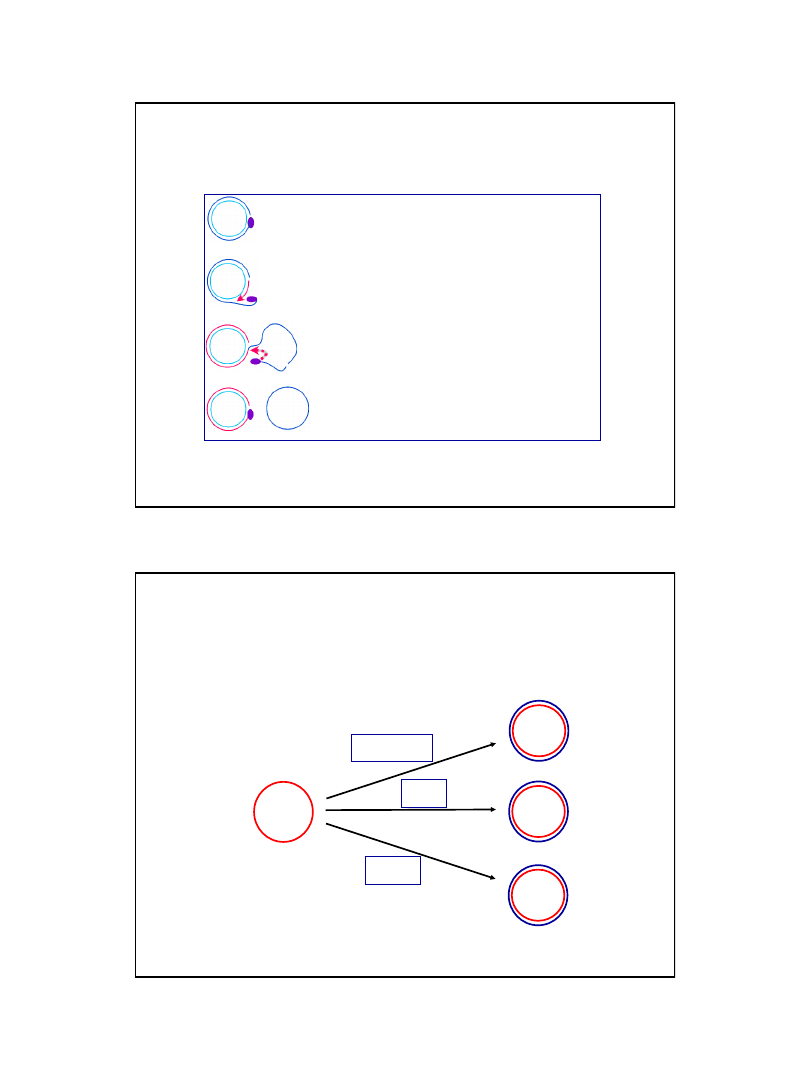
Matrycą jest kolista dwuniciowa cząsteczka DNA
Inicjacja replikacji polega na przecięciu jednej nici w miejscu ori
Elongacja nowej nici DNA zastępuje (wypiera)
jedną ze „starych” nici
w wyniku trwającej elongacji wypierana nić daje
początek multimerycznej, potomnej cząsteczce DNA
Replikacja przebiegająca wg. modelu „obracającego się koła” (rolling circle replication)
zwana również replikacją typu
ζ
kolista dwuniciowa
matryca
fot. David Dressler
liniowy „ogon”
21
nić
-
nić +
białko A przecina nić + w miejscu ori
i wiąże sie z końcem 5’
replikacja typu „obracającego się koła”(ζ)
na matrycy nici
-
po pelnej rundzie replikacyjnej białko A rozpoznaje ponownie
miejsce ori
przecina powstającą nić + i wiąże się z nowym końcem 5’
Jednocześnie przenosi uwolniony 5’-P na powstającą 3’-OH
uwolniona nić + (kompletna kopia komplementarna nici
-
)
tworzy kolistą cząsteczkę formy infekcyjnej DNA bakteriofaga
nić
-
nić +
nić +
Replikacja typu
ζ, jako mechanizm syntezy jednoniciowych, kolistych cząsteczek DNA
formy infekcyjnej bakteriofaga
ΦX 174
Ryc. z: Lewin B., (2000), GenesVII, Oxford University Press
PRZYKŁAD:
nić +
np. bakteriofagi:
M13
G4
ΦX 174
genom formy infekcyjnej (
IF
)
jednoniciowa, kolista
cząsteczka DNA
nić –
nić +
nić –
nić +
forma replikacyjna (
RF
)
dwuniciowa, kolista
cząsteczka DNA
M13
G4
ΦX 174
nić –
nić +
SSB
polimeraza RNA
SSB
primaza
SSB
primosom
Przekształcenie jednoniciowej formy infekcyjnej bakteriofagów w dwuniciową formę replikacyjną
może być inicjowane przez różne elementy bakteryjnego systemu replikacyjnego
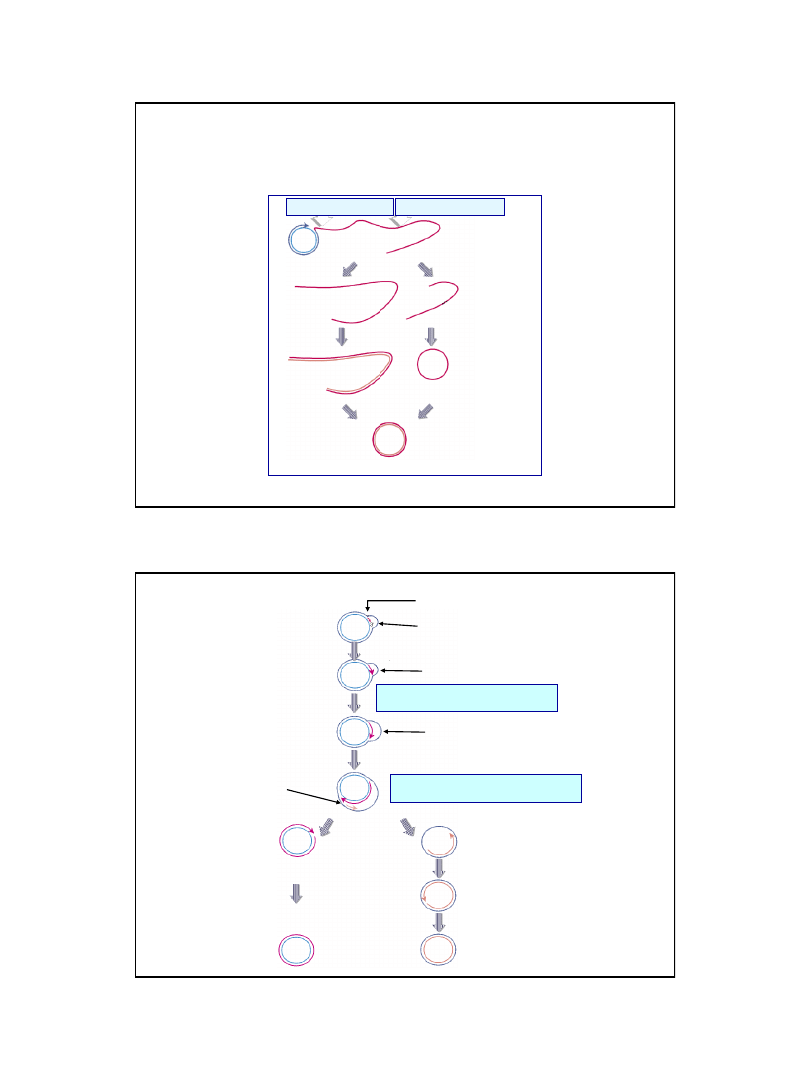
22
Ryc. z: Lewin B., (2000), GenesVII, Oxford University Press
odcinanie monomerów
odcinanie multimerów
kolista cząsteczka jednoniciowa
jednoniciowy multimer
dwuniciowy multimer
kolista cząsteczka dwuniciowa
jednoniciowy monomer
Replikacja typu
ζ może być kończona na różne sposoby właściwe danej cząsteczce DNA
(w sposób charakterystyczny dla danego bakteriofaga lub wirusa)
Synteza nici komplementarnej może rozpoczynać się jeszcze przed odcięciem
monomeru lub multimeru
Replikacja wg. modelu przemieszczającej się pętli (D-loop replication)
Przerwy w nowych niciach
zostają zamknięte przez ligazę
nić L
nić H
inicjowanie syntezy RNA w miejscu ori
dla replikacji nici L na nici H
cięcie RNA generuje 3’OH startera
synteza DNA
synteza nowej nici L tworzy pętlę przez
wypieranie rodzicielskiej nici L
pętla D (D loop – displacement loop)
poszerza się
ori dla replikacji nici H
na nici L
Gdy pętla D przechodzi przez ori na nici L
rozpoczyna się replikacja nowej nici H
Zakończenie replikacji
nowej nici L
uwalnia genomy
potomne
po zakończeniu replikacji powstaje dwuniciowa
kolista cząsteczka potomna DNA
wg. Lewin B., (2000), Genes VII, Oxford University Press
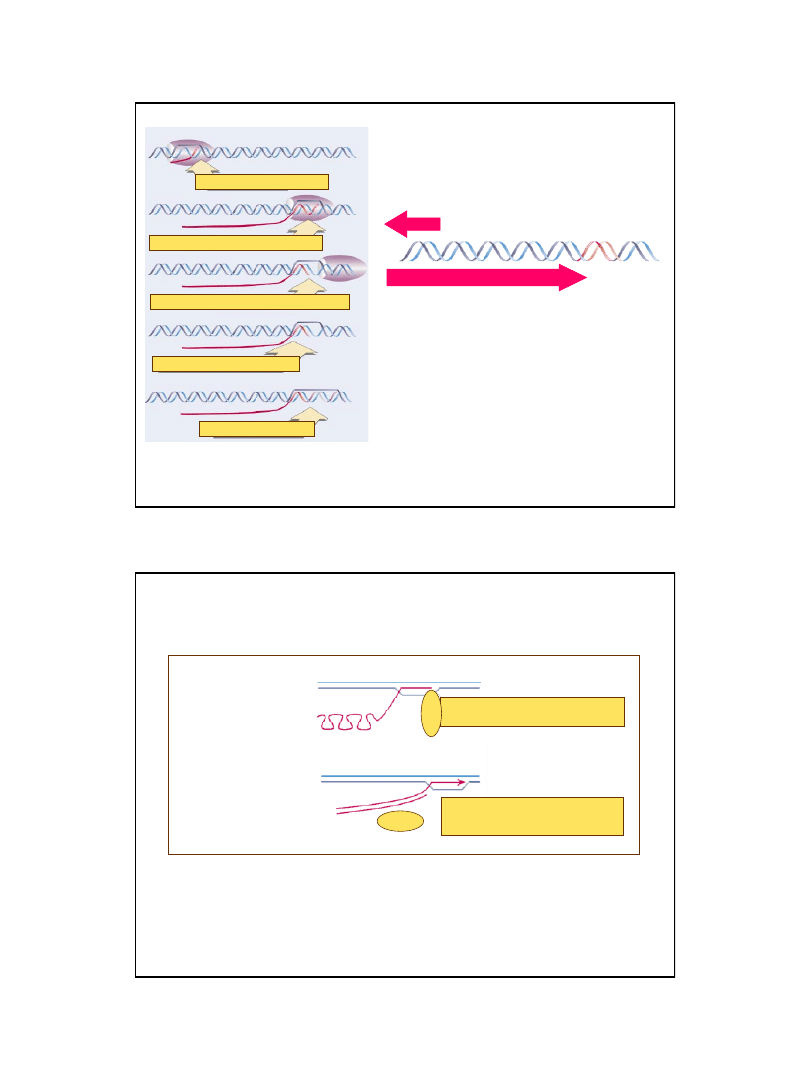
23
inicjacja transkrypcji RNA startera
Polimeraza RNA przechodzi poza miejsce ori
RNAza H przecina RNA generując wolną grupę 3’ OH
Rozpoczyna się synteza DNA
utrzymuje się trwały hybryd DNA-RNA
Starterowy RNA (555 nt.)
RNA I
108 nt.
Sekwencja RNA I jest Komplementarna do końca 5’ RNA starterowego
wg. Lewin B., (2000), GenesVII, Oxford University Press
Replikacja DNA replikonów ColE1 jest inicjowana
przez cięcie starterowego RNA
RNA I pełni funkcję regulacyjną
RNA I nieobecny
RNA I obecny
tworzy oddziałuje
z komplementarną sekwencją
starterowego RNA
RNAza H przecina starterowy RNA
pojawia się 3’-OH
→
inicjacja replikacji
RNAza H nie może przeciąć startera
transkrypcja jest kontynuowana,
replikacja nie może zostać zainicjowana
RNA I reguluje replikację replikonu ColE1
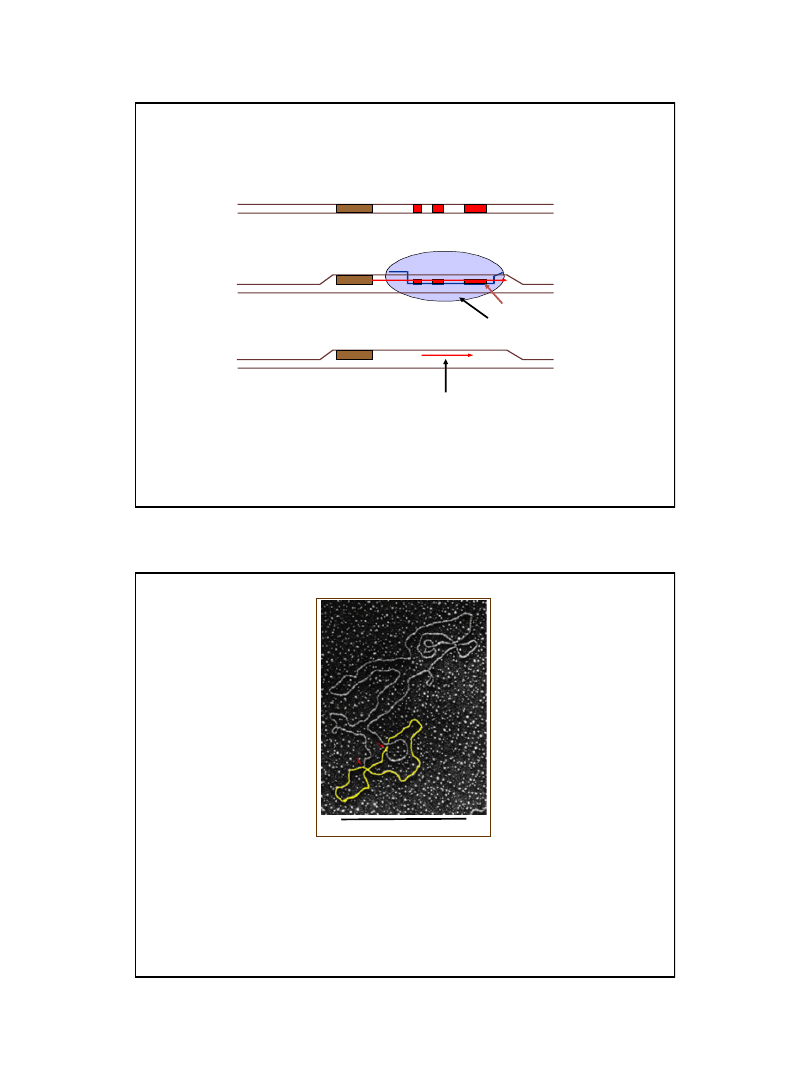
24
Inicjacja replikacji zwierzęcego DNA mitochondrialnego
promotor
nici L
sekwencje CSB
III
II
I
nić L
nić H
nić L
nić H
nić L
nić H
specyficzna nukleaza
mitochondrialna
RNA (136 nt.)
Działanie specyficznej nukleazy generuje powstanie
startera komplementarnego do nici H
wg. Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
1
μm
Obraz ultramikroskopowy cząsteczki zwierzęcego mitochondrialnego DNA
Kolorem żółtem zaznaczono zreplikowany DNA
Replikacja przebiega zgodnie z modelem przemieszczającej się pętli,
katalizuje ją polimeraza
γ
DNA
Fot. D. Clayton
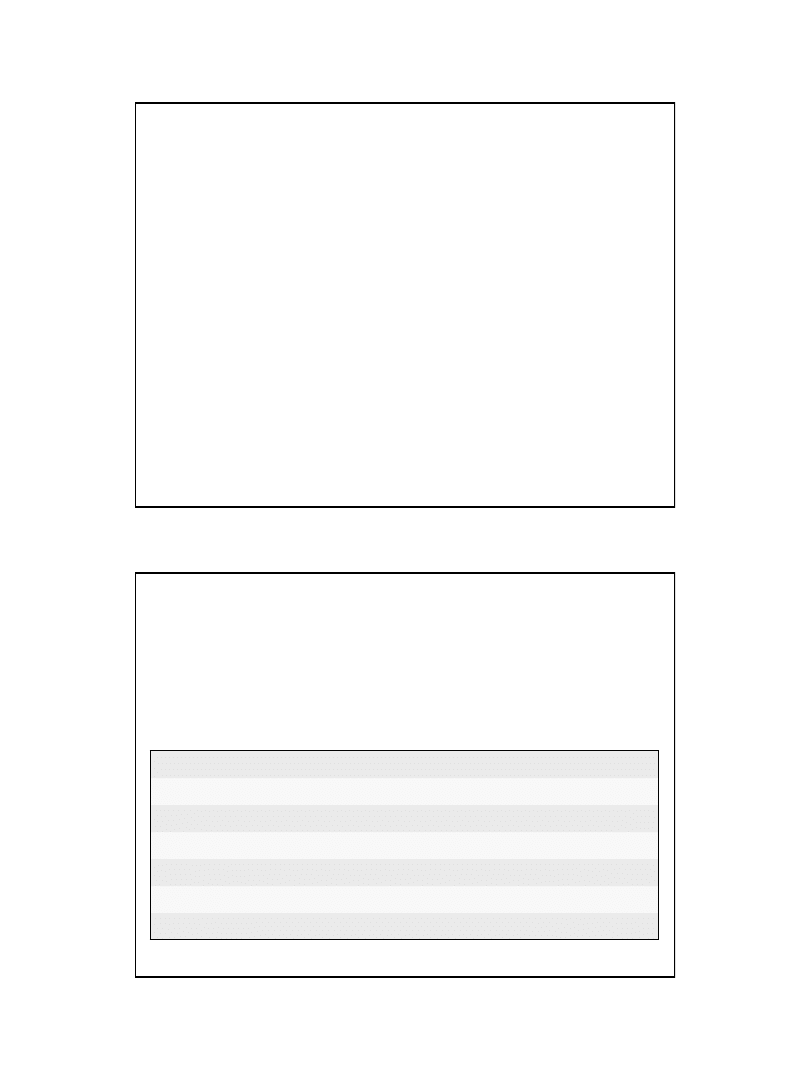
25
Średnia wielkość chromosomu ludzkiego - ok 1,5 x 10
8
pz.
Szybkość replikacji DNA eukariotycznego -
50 pz./s
czas potrzebny dla replikacji DNA jednego chromosomu ludzkiego:
1,5 x 10
8
pz. / 50 nt/s = 3,0 x 10
6
s (ok. 800 godzin)
Szybkość replikacji DNA
Escherichia coli - ok. 500-800 pz./s
Eukaryota - ok. 50 pz./s
Czy cząsteczka eukariotycznego DNA zawiera więcej niż jedno miejsce startu replikacji?
Wniosek:
Cząsteczka DNA w komórce eukariotycznej musi zawierć wiele miejsc startu replikacji (origins)
Cząsteczka DNA w komórce eukariotycznej obejmuje wiele
replikonów
.
Replikonem
nazywamy odcinek DNA ulegający replikacji zainicjowanej w jednym miejscu startu (origin)
i przebiegającej jako ciągły proces aż do momentu zakończenia syntezy nici DNA komplementarnych
do tego odcinka.
Dany replikon zawsze zawiera jedno miejsce startu replikacji (origin). Może także (Prokaryota) zawierać
sygnały kończące replikację (sekwencje decydujące o terminacji replikacji).
Replikon
Organizm
Liczba
replikonów
średnia długość
replikonu [kpz.]
Szybkość ruchu widełek
replikacyjnych [pz./min]
Bakterie (Escherichia coli)
1
4 200
50 000
Drożdże (Saccharomyces cerevisiae)
500
40
3600
Owady (Drosophila melanogaster)
3 500
40
2 600
Płazy (Xenopus laevis)
15 000
200
500
Ssaki (Mus musculus)
25 000
150
2 200
Rośliny(Vicia faba)
35 000
300
Liczba i szybkość replikacji replikonów różnych organizmów

26
Mikrofotografia elektronowa fragmentu chromosomu Drosophila
ulegającego replikacji
(z: Kreigstein H.J., Hogness D.S., 1974, Proc. Natl. Acad. Sci. USA, 71:136.)
Mikrofotografia elektronowa fragmentu chromatyny
ulegającego replikacji w jądrze komórki zarodka Drosophila melanogaster
z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994, Molecular Biology of the Cell,
3rd Ed. Garland Publishing Inc.
Strzałki wskazują położenie pęcherzyków replikacyjnych.
W tkankach embrionalnych miejsca startu replikacji położone są bardzo blisko siebie.
W przedstawionym przykładzie potrzeba tylko ok. 1 minuty aby zakończyć replikację
fragmentu DNA widocznego na zdjęciu. Podziały komórkowe w tkance tego typu mogą
zachodzić nawet co 10 minut.
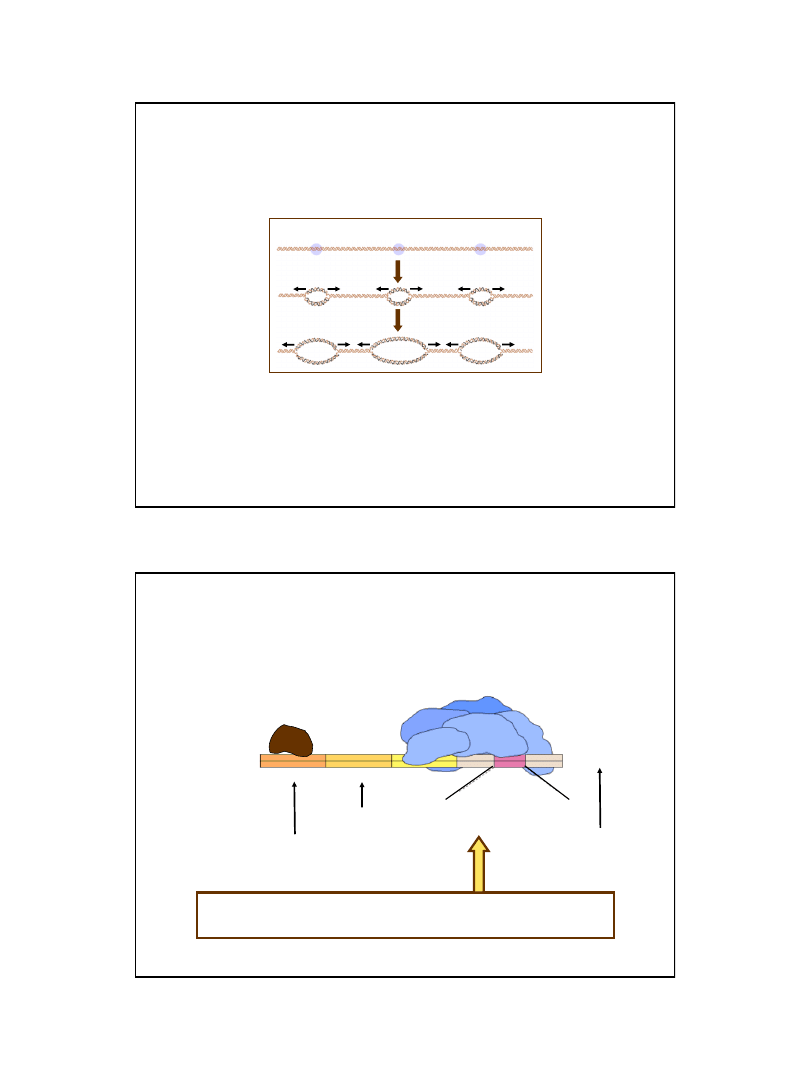
27
ori
ori
ori
inicjacja replikacji
Cząsteczki eukariotycznych DNA zawierają wiele miejsc inicjacji replikacji
ARS
(
A
utonomously
R
eplicating
S
equences) 100
– 200 pz. – wystepują w obszarach międzygenowych
Inicjacja replikacji w komórkach drożdży
Rolę sekwencji inicjacyjnych replikacji (ori) pełnią sekwencje ARS występujące w liczbie 400 -500
w 12 chromosomach Saccharomycesw cerevisiae.
Ich liczba jest zgodna z liczbą replikonów
drożdżowych, która została określona metodami autoradiograficznymi.
(A/T)TTTA(T/C)(A/G)TTT(A/T)
ACS
ORC
B1
DNA
B3
B2
11 pz.
12 pz.
miejsce rozdzielania się
nici DNA
do elementu B3
przyłącza się czynnik
transkrypcyjny
w niektórych ARS
czynniki transkrypcyjne
przyłączają się również
po przeciwnej stronie
elementu ACS
Dołączenie ORC do sekwencji ACS i B1 powoduje owijanie się helisy wokół ORC
połączone z jej silnym zaginaniem co wywołuje w cząsteczce DNA naprężenia
powodujące z kolei rozdzielanie się nici w obszarze B2
TF
ACS
(
A
RS
C
onsensus
S
equence) 11 pz.
ORC
(
O
rigin
R
eplication
C
omplex
) 6 (8) polipeptydów
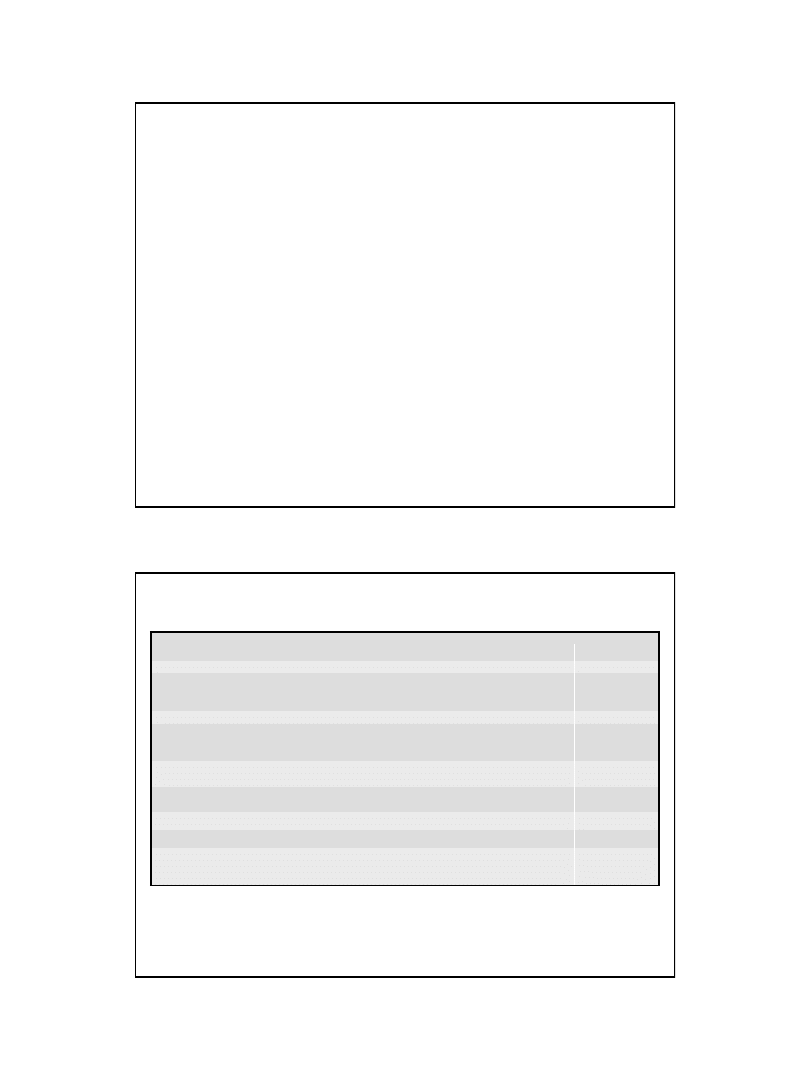
28
W komórkach wyższych eukariontów nie udało się, jak dotąd wykryć sekwencji
odpowiadających ARS (miejsc inicjacji replikacji – ori)
Wiadome już, że określenie: miejsce inicjacji replikacji, w chromosomie eukariotycznym
należy zastąpić określeniem strefa inicjacyjna.
Strefa inicjacyjna
jest to odcinek DNA o długości do 10 000 pz., zlokalizowany w obszarze
międzygenowym, zawierający wiele sekwencji, w obrębie których może być inicjowana replikacja.
Replikacja w obrębie danej strefy inicjacyjnej rozpoczyna się zawsze w jednym miejscu obejmującym
odcinek DNA o długości 200 - 500 pz., choć odcinki te mogą być różne w czasie różnych rund
replikacyjnych.
Strefa inicjacyjna pozostaje pod kontrolą białek strukturalnych chromatyny działających jak represory
replikacji.
Rolę aktywatorów replikacji spelniają najprawdopodobniej białka struktur jądrowych (matriks),
wspomagane przez czynniki transkrypcyjne.
Wiadomo również, że istotną rolę w regulacji cyklu komórkowego (w tym również inicjacji replikacji
spełniają kinazy białkowe i współdziałające z nimi białka zwane cyklinami. Centralną rolę pełni
najprawdopodobniej 34 kDa kinaza cdc2
– produkt genu cdc2 (cell-division-cycle gene).
Pojawienie się specyficznych kinaz – zjawisko promujące fosforylacje różnorodnych białek może
mieć wielorakie i ważkie konsekwencje np.:
-
Fosforylacja lamin → rozpad otoczki jądrowej,
-
Fosforylacja histonu H1 → kondensacja chromatyny.
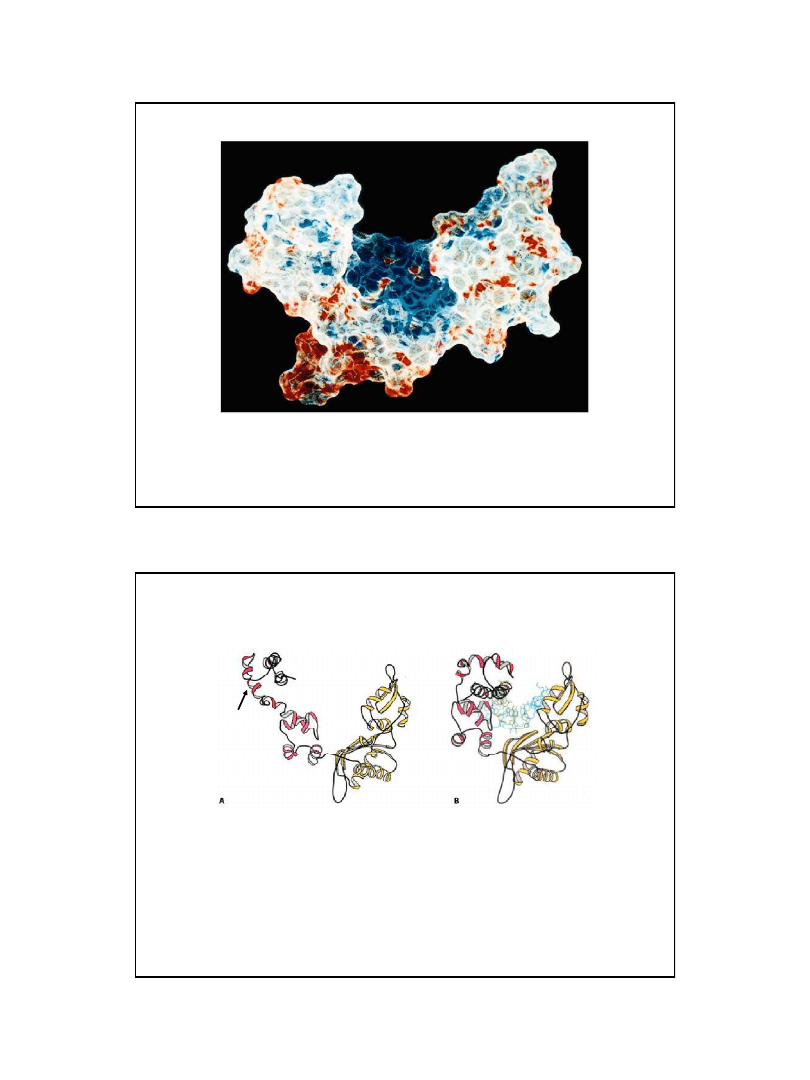
Polimerazy DNA
α
δ
ε
β
γ
Miejsce działania:
jądro
jądro
jądro
jądro
mitochondria
Funkcja:
replikacja,
synteza
starterów
replikacja
replikacja
naprawa DNA
replikacja
mitochondrial-
nego DNA
Wielkość [kDa]:
>250
170
265
40
125
Podjednostki
katalityczne:
165-180
125
215
40
125
towarzyszące: 70, 58, 48
48
55
-
35, 47
Aktywność egzonukleazy
3’→5’
brak
obecna
obecna
brak
obecna
Procesywność
niska
zależna od
PCNA
wysoka
niska
wysoka
Aktywność primazy
obecna
brak
brak
brak
brak
Wierność replikacji
wysoka
wysoka
wysoka
niska
wysoka
Polarność helikazy lub
ATPazy stymulującej
aktywność
3’→5’
5’→ 3’
3’→5’
-
-
Polimerazy DNA występujące w komórce eukariotycznej
29
Struktura domeny katalitycznej polimerazy DNA
β szczura
na podstawie analizy dyfrakcji rtg
Z. Hostomsky, La Jolla, Ca (www.mun.ca)
Ryc. z: Stryer L., Biochemia, Przekład IV wyd., PWN Warszawa 1997.
Struktura eukariotycznej polimerazy DNA
β
Struktura trzeciorzędowa enzymu przypomina polimerazę I DNA z Escherichia coli
domena 8 kDa

30
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed., Sunderland (MA)
PCNA
Polimeraza
Rola białek wspomagających replikację w komórkach ssaków
Białko RCF jest ATPazą wiążącą się w miejscu połączenia startera (RNA)
z nicią matrycy DNA. Podjednostki PCNA wiążą się z tak powstałym kompleksem.
Polimeraza DNA
δ
(
ε
?) wiąże się z kompleksem PCNA-DNA
Struktura PCNA
PCNA
(Proliferating Cell Nuclear Antigen
) jest odpowiednikiem „klamry” tworzonej
w czasie replikacji DNA Escherichia coli przez podjednostki
β jej polimerazy III DNA
Ryc. Z: Krishna T.S., Kong X.P., Gary S.,Burgers P.M., Kuriyan J., 1994,
Cell 79: 1233
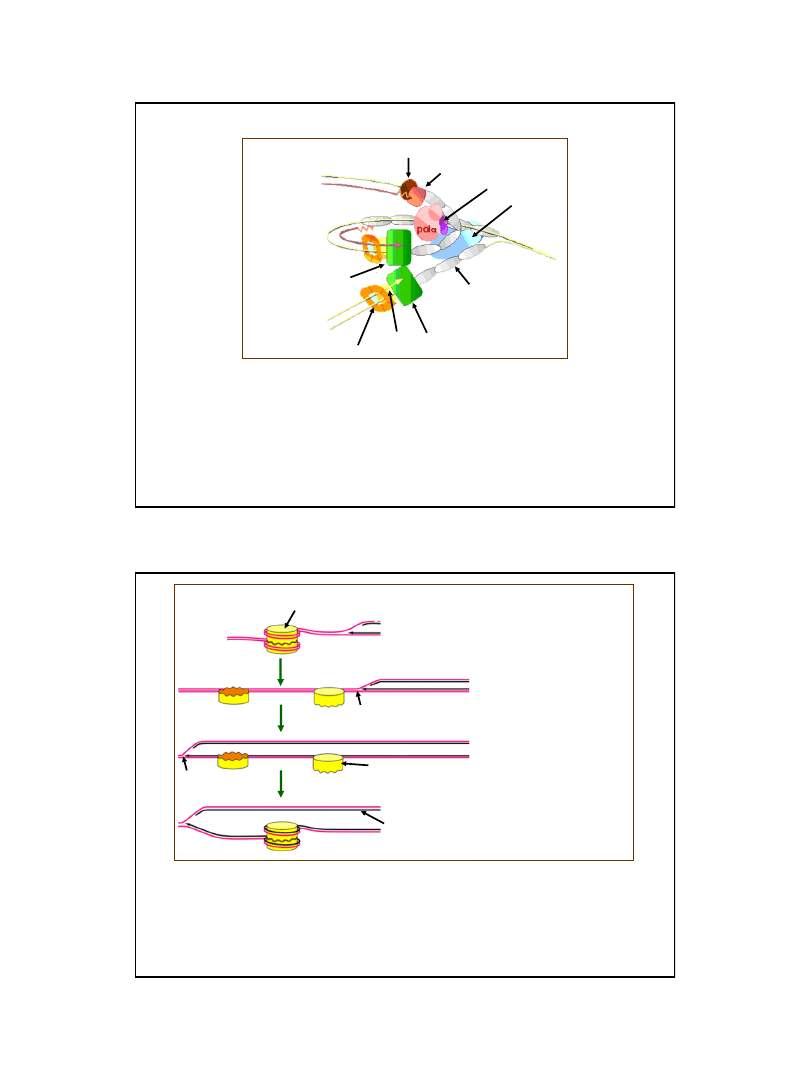
31
5’
3’
5’
3’
Synteza nici wiodącej
pol. DNA
δ
(
ε
?)
Synteza nici opóźnionej
pol. DNA
ε
(
δ
?) + pol.
α
RPA
RNAza H
Ligaza I DNA
helikaza
Primaza
PCNA
RFC
Struktura replisomu w komórce eukariotycznej
Ryc. z: www.mun.ca
-
Funkcje bakteryjnych białek SSB pełnią białka RPA
-
Rolę primosomu spełnia polimeraza DNA α, z którą związane są podjednostki primazy,
-
Funkcję bakteryjnej polimerazy I DNA pełni RNAza H (wycinanie starterów),
oraz najprawdopodobniej polimeraza DNA
β (synteza uzupełniająca) – nie umieszczone
jej na rycinie,
-
Nie jest znany eukariotyczny czynnik odpowiadający z „dimeryzację” holoenzymu
(odpowiednik podjednostek
η polimerazy III DNA z E. coli.
rdzeń nukleosomu
(oktamer histonowy)
widełki replikacyjne
widełki replikacyjne
nukleosom rozwija się, tetramery histonowe
(połówki rdzenia) oddzielają się od siebie
odtwarza się struktura nukleosomu
obie połówki rdzenia pozostają związane z potomną
cząsteczką DNA, w której skład wchodzi nić wiodaca
do potomnej cząsteczki DNA zawierającej nić opóźnioną
zostaną dołączone rdzenie nukleosomów utworzone
przez histony syntetyzowane de novo
wg. Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
Nukleosomy w czasie replikacji
jakkolwiek przebieg mechanizmu dysocjacji i reasocjacji nukleosomów nie został ostatecznie ustalony,
to jednak wiadomo już, że „stare” histony znajduje się po replikacji na tej potomnej cząsteczce DNA,
w której nowa nić była syntetyzowana, jako nić wiodąca.
32
5’
3’
5’
3’
5’
3’
5’
3’
5’
3’
5’
3’
5’
3’
5’
3’
Koniec liniowej cząsteczki DNA tworzącej indywidualny chromosom
REPLIKACJA
w tym końcu chromosomu, jedna z potomnych cząsteczek DNA ma krótszą nić o orientacji 5’→3’
RNA
RNA
RNA
RNA
RNA
Problem replikacji końców liniowych cząsteczek DNA
1. Przekształcenie liniowego replikonu w kolistą cząsteczkę DNA, lub w multimeryczną sekwencję
liniową (bakteriofagi T4, λ).
2. Najprostszym rozwiązaniem jest wykorzystanie grupy OH seryny białka związanego z 5' końcową
cytozyną końca liniowej cząsteczki DNA, jako miejsca dołączenia pierwszego nukleotydu w syntezie
5' → 3' nowej nici (adenowirus, bakteriofag θ29, RNA wirusa polio).
3. Wytworzenie niezwykłej struktury przestrzennej - np. pętli - struktury szpilki do włosów (hairpin).
Praktycznie oznacza to brak wolnego końca nici 3' (replikacja liniowego DNA mitochondrialnego
Paramecium).
4. Końce liniowych cząsteczek DNA mogą nie być precyzyjnie wyznaczone. Liczba kopii krótkich
sekwencji tandemowo powtórzonych w telomerach chromosomów eukariotycznych może być
zmienna.
O strukturze telomerów decyduje enzym telomeraza, co czyni replikację do samego końca 3‘ matrycy
-
zbędną.
Możliwe rozwiązania problemu replikacji końców replikonów liniowych
33
-Ser-C-OH
HO-C-Ser-
55 kDa
3’
5’
3’
5’
3’
5’
80 kDa
3’
3’
3’
5’
5’
5’
3’
5’
5’
55 kDa
80 kDa
25 kDa
białko 55 kDa
25 kDa
białko 80 kDa
ATP, dNTP,
polimeraza DNA
Czynniki komórkowe NF I, NF II, NF III
helikaza, topoizomeraza
Replikacja liniowych cząsteczek DNA genomu adenowirusa
DNA
Telomeraza
matrycowa cząsteczka
RNA
– element
telomerazy
dołączane deoksynukleotydy są
komplementarne do matrycowej
cząsteczki RNA telomerazy
Ryc. z: Griffiths A.J.F., Miller J.H., Suzuki D.T., Lewontin R.C., Gelbart W.M. (1999)
Introduction to Genetic Analysis. 7th ed., W. H. Freeman & Co, New York.
Częścią telomerazy jest krótka cząsteczka RNA funkcjonująca, jako matryca
w procesie wydłużania końca 3’ podwójnej helisy DNA.
Powtórzenia TTGGGG są charakterystyczne dla Tetrahymena
34
telomeraza
RNA telomerazy
na matrycy jest RNA telomerazy
zachodzi odwrotna transkrypcja
– dołączanie deoksynukleotydów
do końca nici wiodącej
Proces ten jest powtarzany wielokrotnie
co prowadzi do wydłużenia końca 3’ przez
dodawanie tandemowych powtórzeń sekwencji
telomerowych. Kiedy długość dodanej sekwencji
umożliwi inicjację fragmentu Okazaki, nastąpi
wydłużenie nici opóźnionej (koniec 5’ chromosomu)
synteza nici opóźnionej DNA nie może być
kontynuowana przy końcu chromosomu
Synteza telomerów
Sekwencje tandemowych powtórzeń tworzących telomery
różnych gatunków organizmów
Tetrahymena, Paramecium CCCCAA
Stylonychia, Oxytrichia
CCCCAAAA
Trypanosoma
CCCCTA
Saccharomyces
C
2-3
A(CA)
1-3
Arabidopsis
CCCTAAA
Homo sapiens
CCCTAAA
5’ → 3’
35
Telomeraza wydłuża wystający koniec 3’
Nowy DNA
Gdy nowy fragment cząsteczki ma wystarczająca długość,
może zostać zainicjowana synteza nowego fragmentu Okazaki
starter
fragment Okazaki
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
Telomeraza zabezpiecza liniowy chromosom przed skracaniem
podczas kolejnych rund replikacyjnych
Telomeraza wydłuża wystający koniec 3’
Nowy DNA
Gdy nowy fragment cząsteczki ma wystarczająca długość,
może zostać zainicjowana synteza nowego fragmentu Okazaki
starter
fragment Okazaki
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
Telomeraza zabezpiecza liniowy chromosom przed skracaniem
podczas kolejnych rund replikacyjnych
36
Transkrypcja
Proces enzymatycznej syntezy RNA na matrycy DNA
►
Inicjacja procesu transkrypcji zachodzi w ściśle określonych regionach cząsteczki DNA
zwanych promotorami
►
Inicjacja transkrypcji jest etapem o najważniejszym znaczeniu dla regulacji ekspresji
informacji genetycznej
►
Regulacja procesu transkrypcji zachodzi zasadniczo na etapie jej inicjacji
znamy przypadki regulacji na drodze przedwczesnej terminacji transkrypcji)
►
Transkrypcja jest katalizowana przez polimerazy RNA
►
Substratami dla syntezy RNA są trifosforany nukleozydów (ATP, UTP, GTP i CTP)
►
Dołączenie pierwszego nukleotydu RNA nie wymaga obecności startera
►
Matrycą dla syntezy RNA jest zwykle tylko jedna nić DNA (znane są stosunkowo
nieliczne wyjątki od tej reguły)
Promotor
Element regulatorowy genu
– sekwencja nie ulegająca transkrypcji lecz decydująca
o jej inicjacji. Zawiera miejsca wiązania polimerazy RNA i czynników transkrypcyjnych.
5’
GGTAAGCGCCAGTCACCTAGTAGATGACATCCA
3’
3’ CCATTCGCGGTCAGTGGATCATCTACTGTAGGT 5’
5’
GGUAAGCGCCAGUCACCUAGUAGAUGACAUCCA
3’
nić kodująca DNA (lub nić +)
nić matrycowa DNA (lub nić -)
transkrypt - RNA
TRANSKRYPCJA
Nazewnictwo nici w ulegającym transkrypcji odcinku DNA
37
5’
3’
3’
5’
5’
3’
5’
3’
5’
3’
5’
3’
3’
5’
DNA
RNA
RNA
RNA
RNA
RNA
W różnych regionach cząsteczki DNA dana nić może być nicią kodującą (+) lub matrycową (-)
Informacja genetyczna przenoszona przez DNA adenowirusa jest kodowana
przez obie nici jego genomu (36 kpz.)
Ten sam fragment danej nici DNA może być w różnym czasie nicią kodującą lub matrycową
38
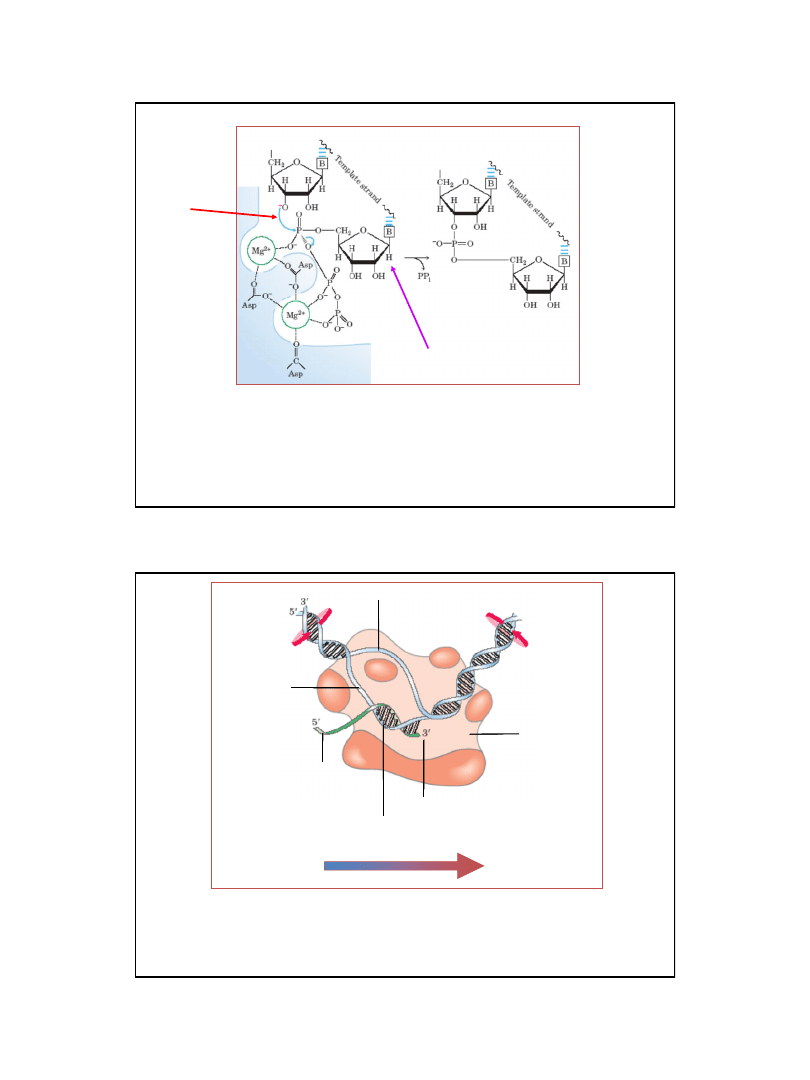
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Przebieg reakcji dołączani nukleotydu w procesie transkrypcji
zaznaczono rolę Asp460, Asp462 i Asp464 podjednostki β polimerazy RNA
Escherichia coli. Jeden jon Mg
2+
wspomaga atak nukleofilowy 3’OH,
drugi ułatwia odłączenie pirofosforanu
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
RNA
(transkrypt)
hybryd DNA-RNA
miejsce aktywne
kanał NTP
nić matrycowa
nić kodująca
skręcanie DNA
rozkręcanie DNA
kierunek transkrypcji
Struktura pęcherzyka transkrypcyjnego
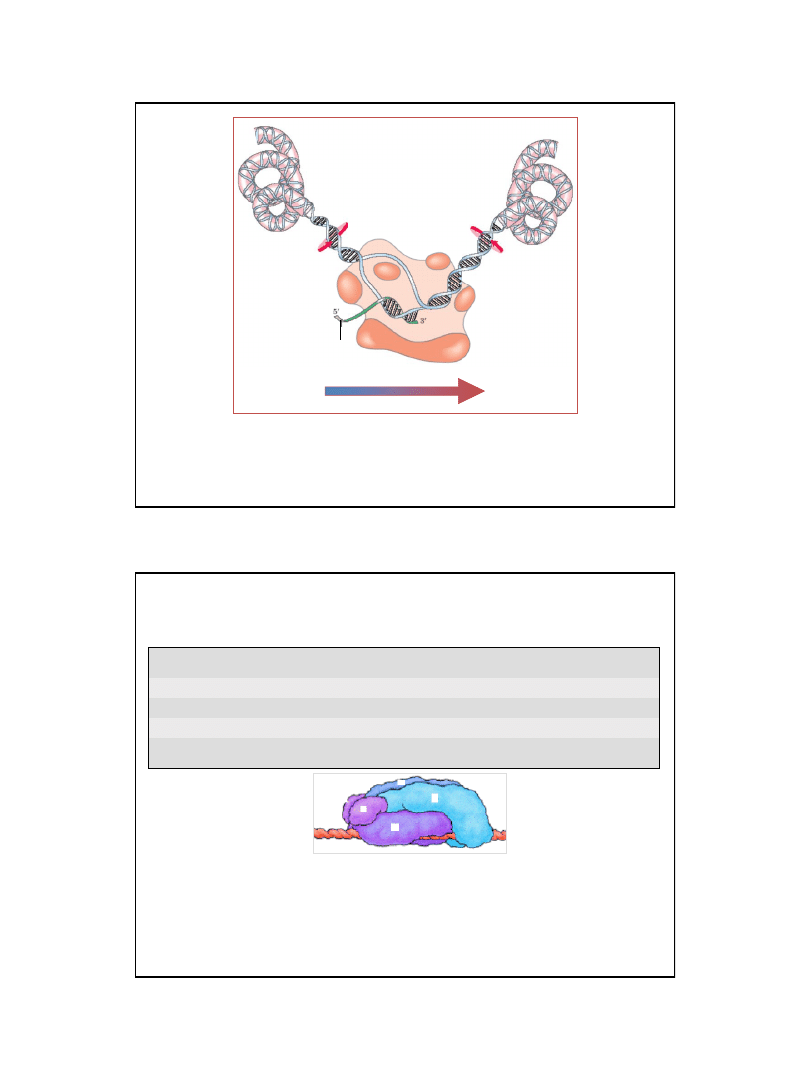
39
Superskręty pozytywne
Superskręty negatywne
kierunek transkrypcji
RNA
(transkrypt)
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
W czasie przemieszczania się pęcherzyka transkrypcyjnego
powstają, kompensujące się wzajemnie, superskręty DNA
Podjednostki
α, β i β' mają podobną wielkość i strukturę w komórkach różnych gatunków bakterii.
Podjednostki
ζ (czynniki ζ) są zróżnicowane.
Podjednostka
Gen kodujący
podjednostkę
Masa
cząst. [D]
Liczba
podj.
Lokalizacja
Funkcja
α
rpoA
ok. 40 000
2
rdzeń
asocjacja podjednostek
β
rpoB
ok. 155 000
1
rdzeń
wiązanie nukleotydów
β'
rpoC
ok. 160 000
1
rdzeń
wiązaniematrycy
ζ
rpoD
32 000 -
- 92 000
1
czynnik
ζ
wiązanie promotora
Struktura polimeraz RNA Eubacteria
podjednostki
α - odpowiadają za stabilność struktury czwartorzędowej polimerazy, uczestniczą
w rozpoznaniu sekwencji promotorowych,
podjednostka
β - wiąże substrat (NTP) - najprawdopodobniej obejmuje centrum katalityczne – miejsce
tworzenia wiązania fosfodiestrowego,
podjednostka
β' - wiąże nić matrycową DNA
Podjednostki
ζ - łącząc się z rdzeniem (2αββ'), tworząc holoenzym, nadaje mu zdolność rozpoznawania
i specyficznego wiązania się z sekwencjami promotorowymi.
α
σ
α
β’
β
40
α
β’
σ
ω
β
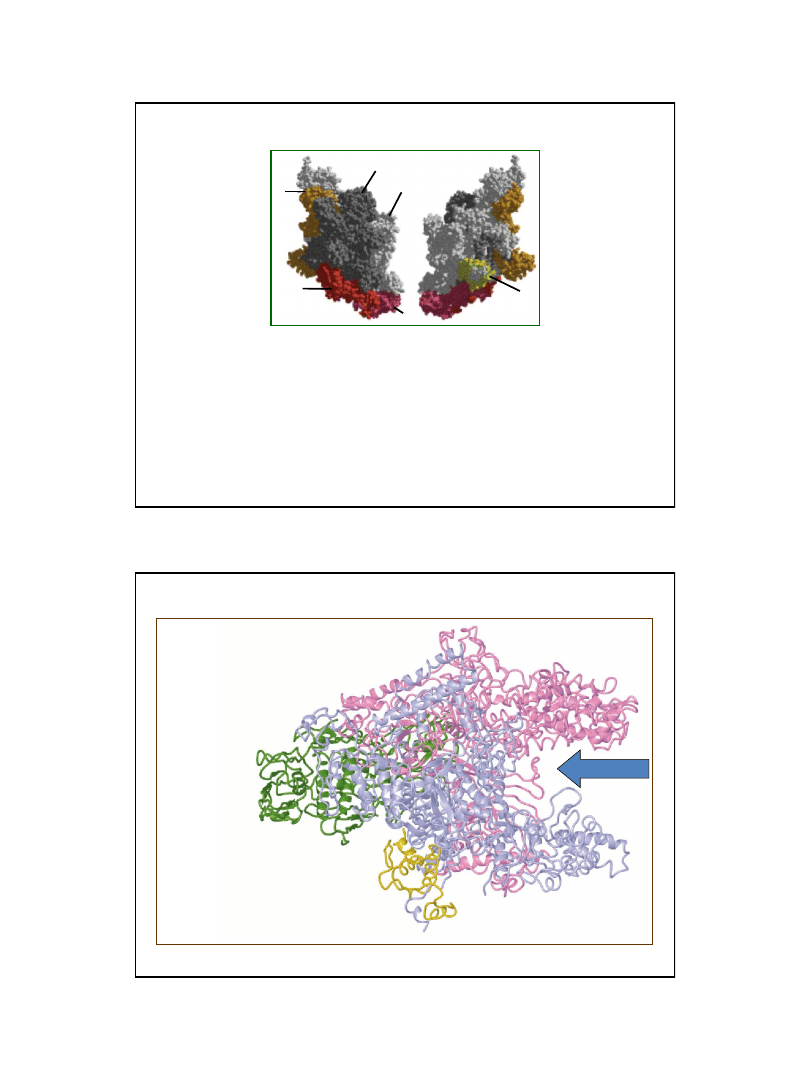
α
Struktura przestrzenna holoenzymu polimerazy RNA z Thermus aquaticus
jest bardzo podobna do struktury polimerazy RNA Escherichia coli
podjednostka
β
podjednostka
β’
podjednostki
α
podjednostka
ω
miejsce aktywne
Struktura rdzenia polimerazy RNA z Thermus aquaticus
Miejsce aktywne może pomieścić 16 pz. DNA
41
α
α
β
β’
σ
α
α
β
β’
σ
α
α
β
β’
σ
σ
promotor
10
4
razy
10
3
razy
ok.
10
7
razy
T
1/2
= ok. 60 min.
T
1/2
= ok. 1 sekunda
T
1/2
– kilka godzin
Podjednostka
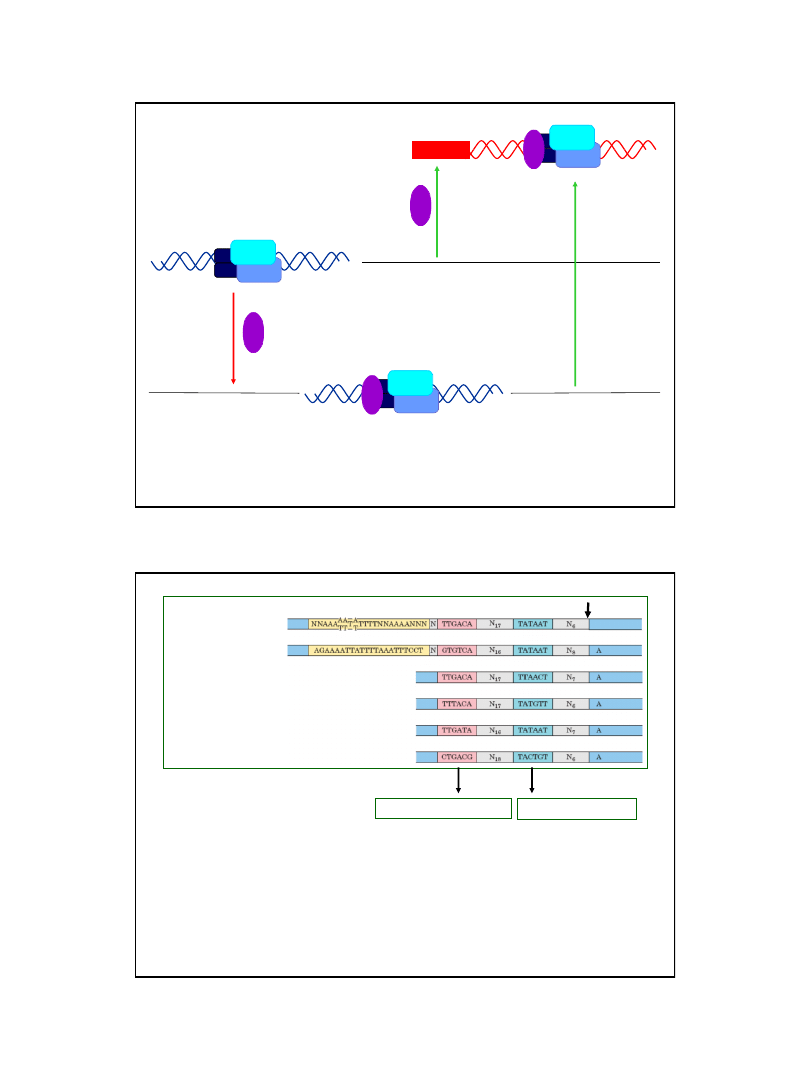
ζ
bakteryjnej polimerazy RNA decyduje o specyficzności wiązania
holoenzymu do sekwencji promotorowej
Sekwencja konsensusowa
lac
trp
araBAD
recA
rrnB P1
+1
region -10
region -35
element UP
T
80
A
95
t
45
A
60
a
50
T
96
T
82
T
84
G
78
A
65
C
54
a
45
region -10
sekwencja Pribnowa
Pribnow box
sekwencja -35
Struktura niektórych promotorów Escherichia coli rozpoznawanych
przez polimerazę RNA z podjednostka ζ
70
42
Zdolność polimerazy RNA z Eschrerichia coli do inicjowania transkrypcji w promotorach, o różnych
sekwencjach regionów – 10 i – 35 wskazuje na relatywnie niską specyficzność enzymu (jeżeli przyjąć
za punkt odniesienia np. specyficzność endonukleaz restrykcyjnych).
Jednocześnie obserwuje się zróżnicowaną wydajność transkrypcji genów pozostających pod kontrolą
różnych promotorów. W zależności od względnej częstości inicjacji transkrypcji zachodzących w ich obrębie,
promotory dzielimy na „silne” i „słabe”.
Wydajność transkrypcji kontrolowanej przez różne promotory zależy od powinowactwa polimerazy RNA
do ich sekwencji.
Dowodem na fundamentalną rolę sekwencji elementów – 10 i – 35 w wyznaczaniu aktywności promotora
mogą być doświadczenia polegające na wymianie nukleotydów – wprowadzaniu mutacji do tych
fragmentów DNA
region
– 10
region
– 35
TATAAT
TTGACA
17
± 1 pz.
TATGTT
TTTACA
AA
→ wzmocnienie promotora
sekwencja konsensusowa promotorów
σ
70
sekwencja słabego promotora Lac
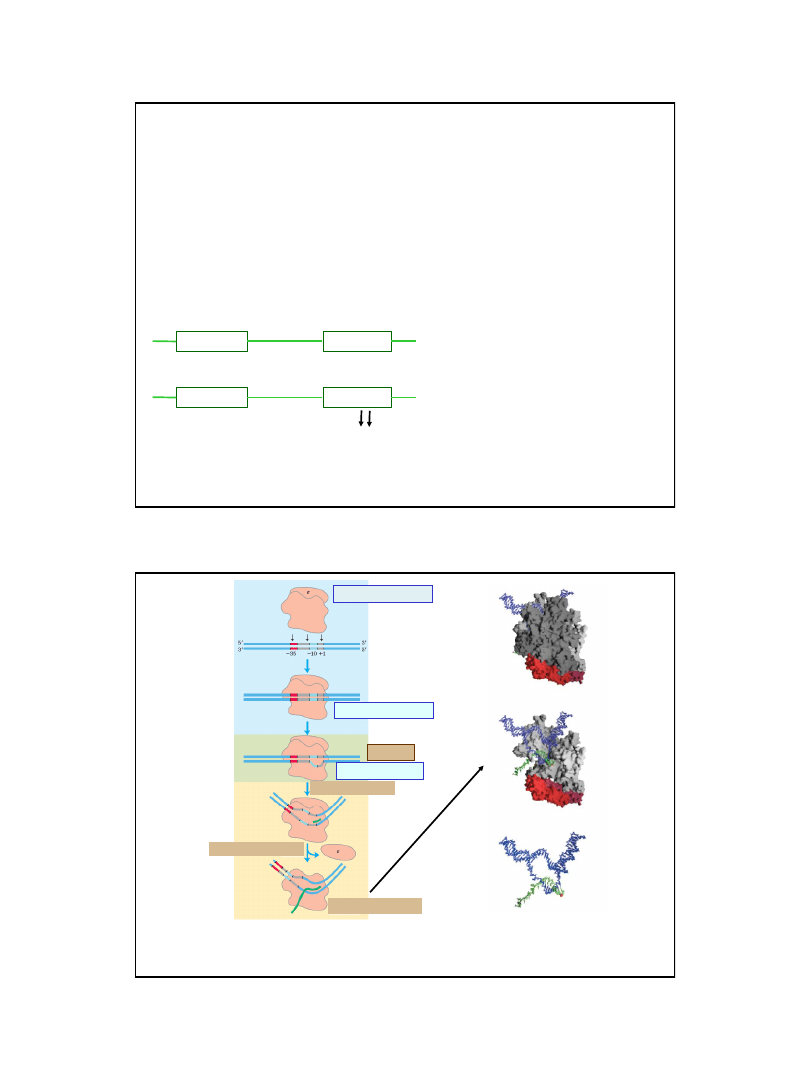
Wiązanie polimerazy
Inicjacja
kompleks zamknięty
kompleks otwarty
Inicjacja transkrypcji
postać elongacyjna
opuszczenie promotora
Wiązanie polimerazy i inicjacja transkrypcji w Escherichia coli
podjednostka
ζ opuszcza kompleks (holoenzym polimerazy RNA) po włączeniu pierwszych 8-9
nukleotydów. Etap elongacji jest katalizowany przez rdzeń polimerazy
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry. 4th Ed. W.H. Freeman & Co.; New York
β
β’
α
α

43
łącząc się z rdzeniem (2αββ'), tworząc holoenzym, nadaje mu zdolność rozpoznawania i
specyficznego wiązania się z sekwencjami promotorowymi.
Określony typ podjednostki ζ jest właściwy dla danego gatunku bakterii, lecz również dla danej klasy
genów.
ζ
70
-
dominujący typ podjednostki w Escherichia coli (90% w standardowych warunkach wzrostu),
ζ
32
-
podjednostka polimerazy RNA rozpoznającej promotory genów szoku cieplnego w Escherichia coli
ζ
24
-
w połączeniu z rdzeniem enzymu warunkuje transkrypcję genów htrA i rpoH
ζ
28
-
uczestniczy w strukturze holoenzymu polimerazy RNA prowadzącego transkrypcję genów
odpowiedzialnych za powstawanie rzęsek i chemotaksje
ζ
54
-
występuje w polimerazach Escherichia coli i Salmonella typhimurium tworząc holoenzym
przepisujący sekwencje genów uczestniczących w asymilacji amoniaku.
Występuje również w polimerazach prowadzących transkrypcję genów nif w Klebsiella pneumoniae,
genów vir Pseudomonas syringae, oraz genów metabolizmu wodoru w komórkach Alcaligenes
eutrophus
ζ
43
-
podstawowy typ podjednostki w komórkach Bacillus subtilis w fazie wegetatywnej wzrostu kultury
ζ
26
-
podjednostka rozpoznająca promotory genów ulegalących transkrypcji w okresie pośrednim
ζ
13,24
-
podjednostka uczestnicząca w inicjacji transkrypcji z promotorów genów późnych Bacillus subtilis
(okres sporulacji)
Wszystkie wymienione powyżej typy podjednostek zawierają regiony o budowie homologicznej (ζ
70
i
ζ
43
mają ich cztery, mniejsze - trzy lub mniej).
Podjednostka
ζ
54
ma budowę całkowicie odmienną.
Podjednostki
ζ
prokariotycznych polimeraz RNA
Gen kodujący
podjedn.
ζ
Podjednostka
sekwencja
-35
sekwencja
-27
fragm.
oddz.
sekwencja
-10
rpoD
ζ
70
(
ζ
D
)
TTGACA
-
N
17
TATAAT
rpoH (htpD)
ζ
32
(
ζ
H
)
TNTCNCCCTTGAA
-
N
13-15
CCCCATTTA
rpoE
ζ
24
(ζ
E
)
GAACTT
-
N
16
TCTGA
rpoF (flaI ?)
ζ
28
(ζ
F
)
TAAA
-
N
15
GCCGATAA
rpoS (katF)
ζ (ζ
S
)
CTGCAA
-
N
16-20
CGGCNAGTA
rpoN (glnF, ntrA)
ζ
54
(ζ
N
)
-
CTGGYAYR
N
4
TTGCA
N - nukleotyd dowolny; Y - nukleotyd pirymidynowy; R - nukleotyd purynowy
W komórce Escherichia coli występuje około 7000 cząsteczek polimerazy RNA.
Z tego 2 000 -
5 000 jest zaangażowanych w procesie transkrypcji.
Porównanie struktury promotorów Escherichia coli rozpoznawanych
przez polimerazy RNA związane z różnymi podjednostkami ζ
44
Porównanie wielkości, struktury i właściwości polimerazy RNA z Escherichia coli
i polimerazy RNA bakteriofaga T7
Dlaczego duży, multimeryczny kompleks enzymatyczny jest wykorzystywany w reakcji,
którą z większą szybkością może katalizować pięciokrotnie mniejszy enzym monomeryczny?
1.
Polimerazy fagowe (np. T7, T3) wykazują specyficzność wyłącznie w stosunku do kilku promotorów
fagowych. Ich aktywność praktycznie nie podlega regulacji.
2. Polimeraza RNA z E. coli
rozpoznaje w sposób specyficzny różnorodne promotory co najmniej
1000 jednostek transkrypcyjnych.
3.
Rozpoznanie niektórych z nich wymaga udziału specyficznych podjednostek ζ lub dodatkowych
czynników białkowych.
4. Polimeraza RNA z E. coli
może współdziałać z induktorami i represorami.
Złożoność strukturalna polimerazy RNA z E. coli jest niezbędna dla jej oddziaływać z licznymi
czynnikami regulatorowymi, a nie dla realizacji jej funkcji katalitycznej - biosyntezy RNA!
Polimeraza RNA z Escherichia coli
Pięć polipeptydów (2αββ’ζ) – łącznie ok 480 kDa
Szybkość polimeryzacji RNA - 40-50 nukleotydów/s
α
α
β
β’
σ
Polimeraza RNA bakteriofaga T7
Jeden polipeptyd
– ok. 100 kDa
Szybkość polimeryzacji RNA - ok. 200 nukleotydów/s
W komórkach prokariontów procesy transkrypcji i translacji zachodzą w tym samym czasie
Promotor
podjednostka
ζ
Terminator
białka
mRNA
podjednostki
rybosomalne
holoenzym
polimerazy RNA
rdzeń polimerazy RNA
mRNA
fot. Oscar Miller

45
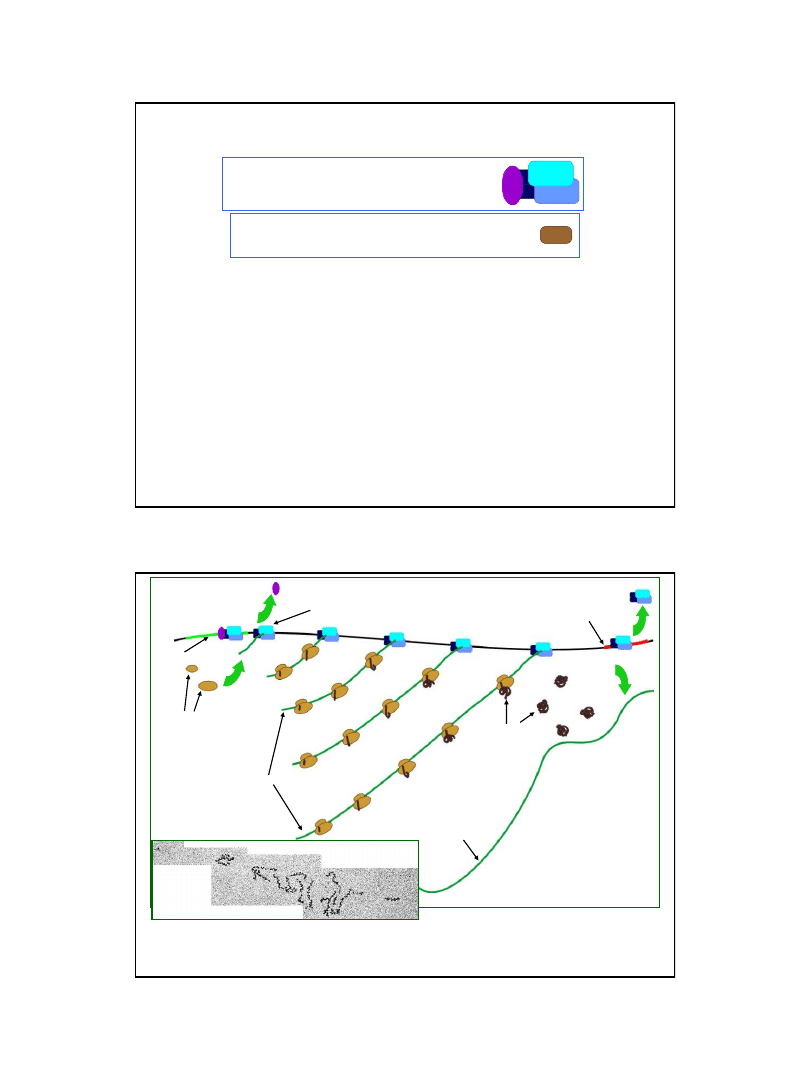
Terminacja transkrypcji u Escherichia coli
Terminacja transkrypcji polega na uwolnieniu powstającego transkryptu z hybrydu: RNA – nić matrycowa DNA,
w obrębie rdzenia polimerazy RNA.
Polimeraza zatrzymuje się przejściowo po napotkaniu niektórych sekwencji DNA.
W przypadku, gdy sekwencje te wykazują pewne określone cechy, transkrypcja nie może być kontynuowana –
następuje jej terminacja.
W Escherichia coli
znane są dwa typy sekwencji terminujących – terminatorów:
1. Terminatory niezależne od białka rho (
ρ
).
Są to palindromowe sekwencje (15-25 pz.) bogate w pary G-C, za którymi wystepują, co najmniej
3 (często 6) nukleotydy adenylowe (na nici matrycowej DNA).
Powstający w rezultacie ich transkrypcji RNA tworzy strukturę typu spinki do włosów (hairpin), za którą
występują powtórzenia nukleotydów urydylowych (oligoU). Spośród wszystkich hybrydów DNA-RNA
hybryd składający się z par rU-dA wymaga najmniejszej energii rozdzielenia nici.
2. Sekwencje rut (rho utilization).
Są to sekwencje bogate w CA (w obrębie powstającego transkryptu RNA) a ubogie w G, co uniemożliwia
tworzenie struktur drugorzędowych. Nie określa się sekwencji zgodnej miejsca rut – sekwencje te
nie wykazują podobieństwa.
Z sekwencjami rut
zlokalizowanymi w obrębie jednoniciowych fragmentow DNA wiąże sie białko
ρ
(rho).
3’
5’
struktura drugorzędowa
typu spinki do włosów
(hairpin) RNA
sekwencja palindromowa
RNA
DNA
3’
5’
3’
5’
3’
5’
Powstawanie struktury drugorzędowej RNA typu spinki do włosów (hairpin)
Przykład z: Horton H.R., Moran L.A., Ochs R.S., Rawn D.J., K Gray Scrimgeour K.G, 2003, Principles of Biochemistry ,
Pearson Higher Education; Prentice-Hall, Inc. ISBN: 0130926434
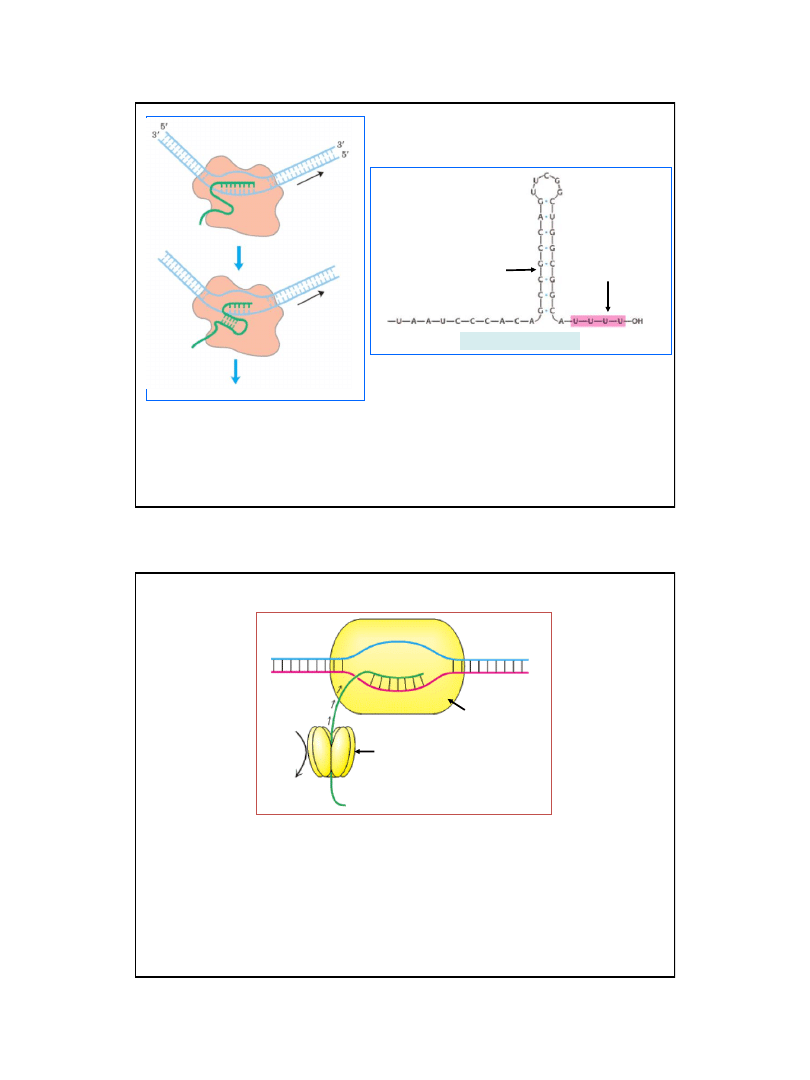
46
Zatrzymanie
polimerazy RNA
Izomeryzacja kompleksu
Terminacja transkrypcji
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Model niezależnej od
ρ
terminacji transkrypcji genów prokariotycznych
5’
3’
Terminator transkrypcji
Struktura typu spinki
do włosów (hairpin)
bogata w pary GC
powtórzenie co najmniej
3 nukleotydów U
Białko
ρ
ppp
5’
polimeraza RNA
ADP + PPi
ATP
Terminacja transkrypcji z udziałem białka
ρ
w Escherichia coli
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry.
5ed., W.H. Freeman and Co., New York
47
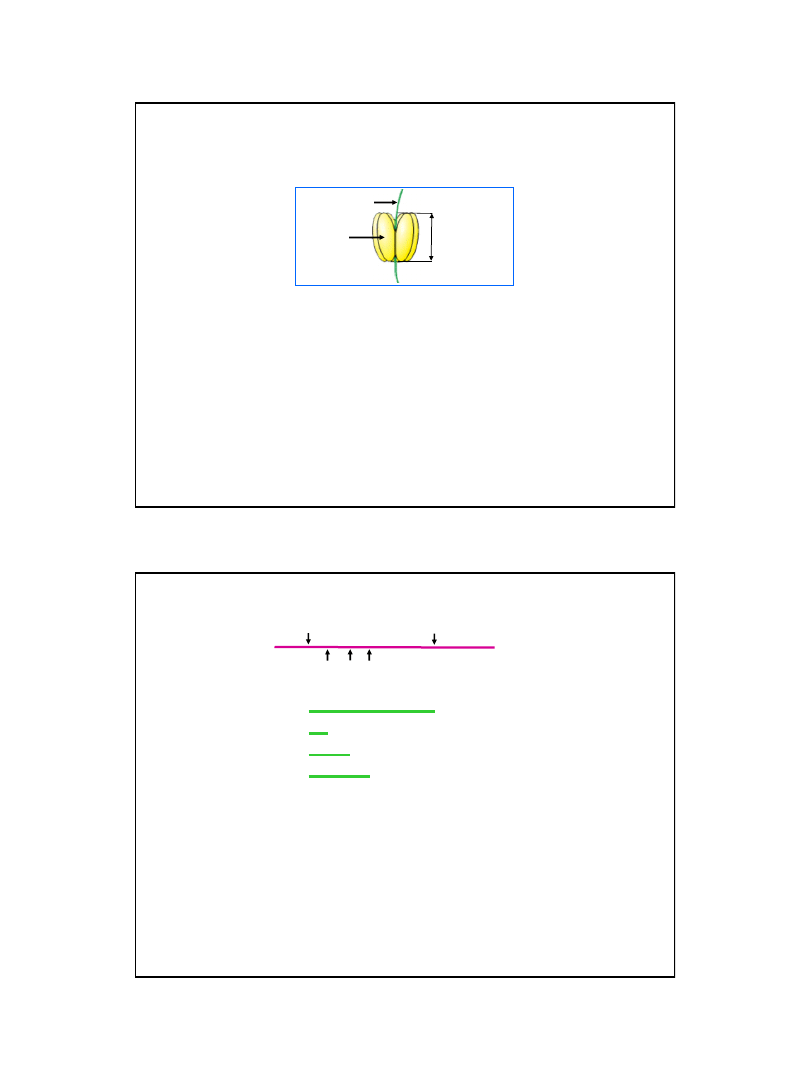
Białko
ρ
(rho)
► Funkcjonuje jako heksamer – zbudowane jest z sześciu identycznych podjednostek
► Specyficznie wiąże się z jednoniciowym RNA
► W obecności jednoniciowego RNA (lecz nie dwuniciowego, lub hybrydu DNA-RNA)
wykazuje aktywność ATP-azy
białko
ρ
RNA
odcinek RNA
długości 72 nt.
miejsca wiązania
ρ
- sekwencje rut (rho-utilization)
Inicjacja
Terminacja bez udziału
ρ
RNA
(transkrypty)
matryca DNA
brak
ρ
→ transkrypt 23S
5’
3’
ρ
obecny od początku transkrypcji → transkrypt 10S
ρ
dodany po 30 sekundach
→ transkrypt 13S
ρ
dodany po 2 minutach
→ transkrypt 17S
5’
3’
5’
3’
5’
3’
Przykład z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry,
5th ed., W.H. Freeman and Co., New York
Dowód doświadczalny na istnienie dwóch typów terminatorów transkrypcji
w obrębie genów prokariotycznych
Transkrypcję in vitro prowadzono w nieobecności białka
ρ
lub dodając je
w różnym czasie od rozpoczęcia reakcjijej inicjacji
Wyszukiwarka
Podobne podstrony:
BKNB 1213 04
03 Sejsmika04 plytkieid 4624 ppt
03 Odświeżanie pamięci DRAMid 4244 ppt
podrecznik 2 18 03 05
od Elwiry, prawo gospodarcze 03
Probl inter i kard 06'03
TT Sem III 14 03
03 skąd Państwo ma pieniądze podatki zus nfzid 4477 ppt
03 PODSTAWY GENETYKI
Wyklad 2 TM 07 03 09
03 RYTMY BIOLOGICZNE CZŁOWIEKAid 4197 ppt
Rada Ministrow oficjalna 97 03 (2)
Sys Inf 03 Manning w 06
KOMPLEKSY POLAKOW wykl 29 03 2012
więcej podobnych podstron