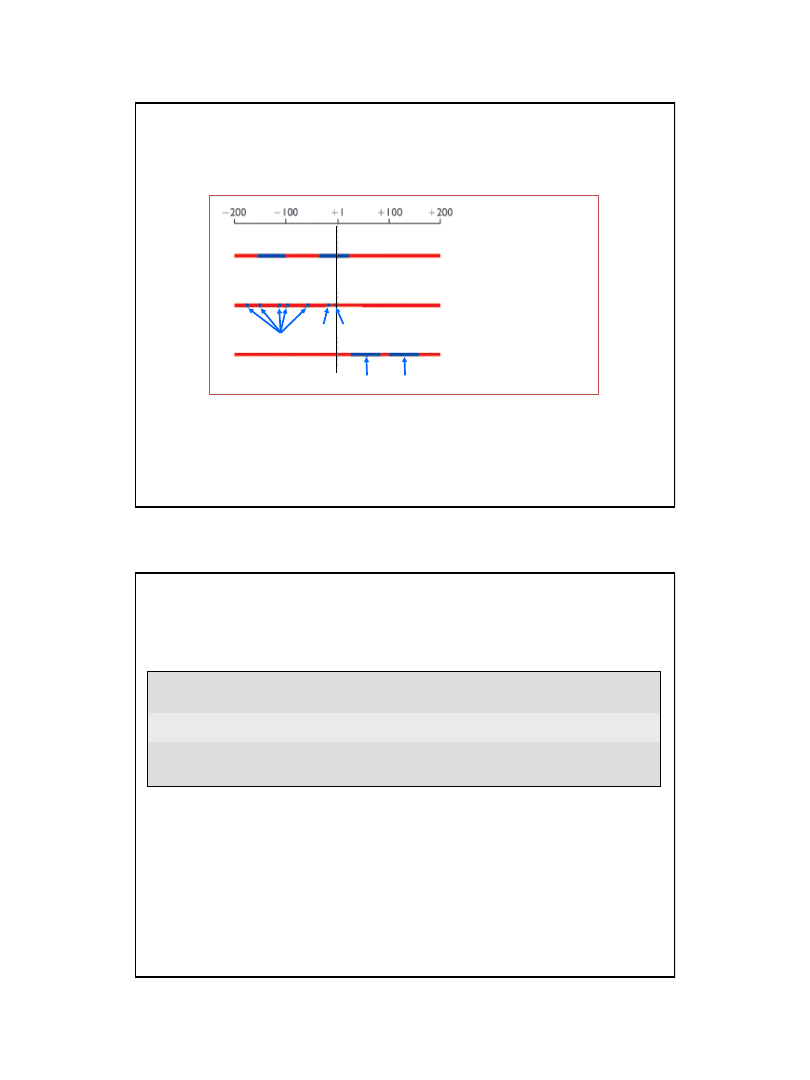
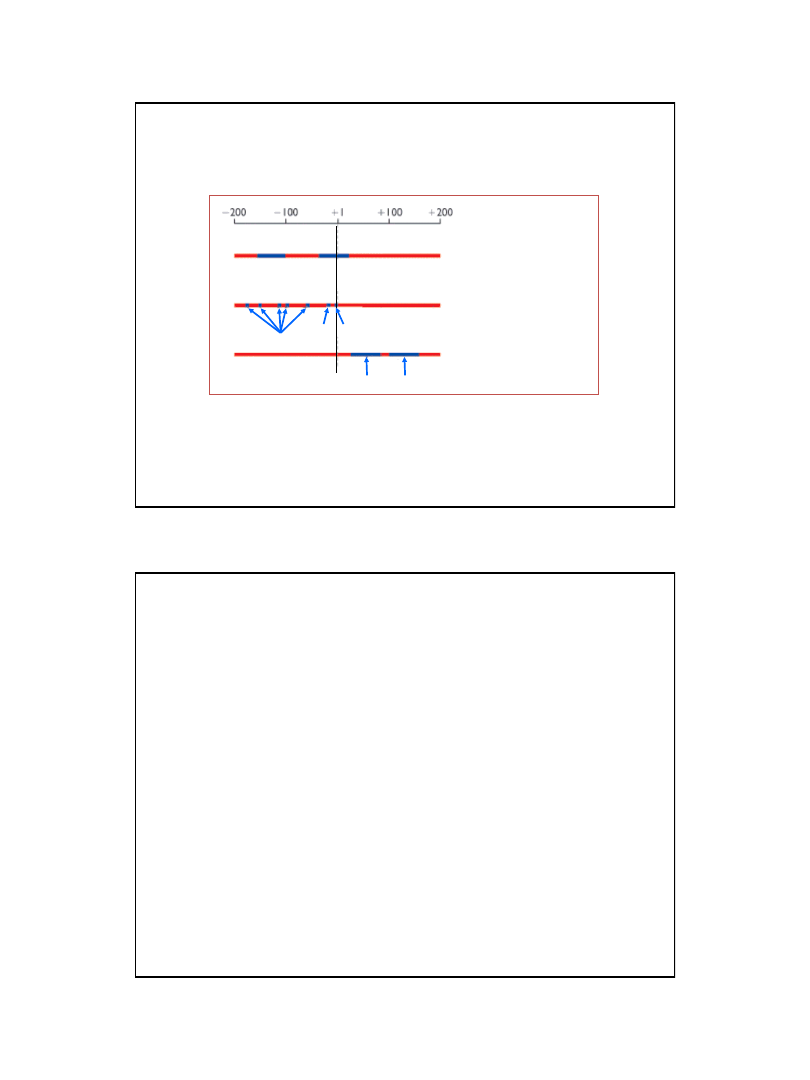
1
5’
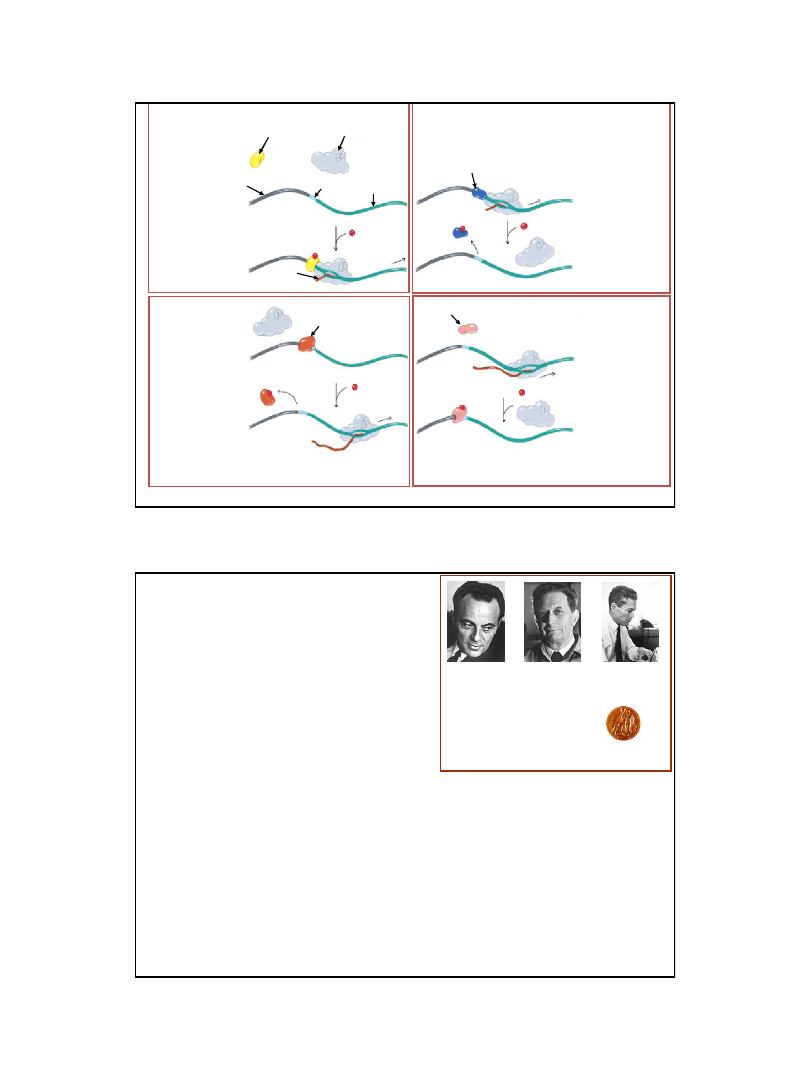
Aktywator
Polimeraza RNA
DNA
Promotor Gen
Ligand
mRNA
5’
Ligand
Represor
Ligand
5’
Aktywator
Represor
Ligand
Represor
uniemożliwia
transkrypcję
Aktywator po przyłączeniu
ligandu stymuluje
transkrypcję
Aktywator stymuluje transkrypcję;
W nieobecności ligandu represor
nie wiąże sie z DNA
Represja zachodzi wyłącznie
w obecności ligandu (korepresora)
Dołączenie ligandu
(induktora) inaktywuje
represor i umożliwia transkrypcję
Po przyłączeniu ligandu
aktywator traci
aktywność
Ryc. z: Horton H.R., Moran L.A., Ochs R.S., Rawn D.J., K Gray Scrimgeour K.G, 2003, Principles of Biochemistry ,
Pearson Higher Education; Prentice-Hall, Inc. ISBN: 0130926434
Nagroda Nobla
1965
w Dziedzinie Fizjologii i Medycyny
Fran
çois Jacob
Jacques Monod
Andr
é Lwoff
Instytut Pasteura, Paryż
Zespół składający się z bakteryjnych genów strukturalnych kontrolowanych przez wspólne
elementy regulatorowe (promotor, operator) i czynnik regulatorowy (represor lub aktywator)
kodowany przez swoisty dla danego operonu gen regulatorowy.
Geny strukturalne zorganizowane w dany operon kodują z reguły enzymy zaangażowane w
realizację skoordynowanych reakcji (szlaku metabolicznego).
Operon
2
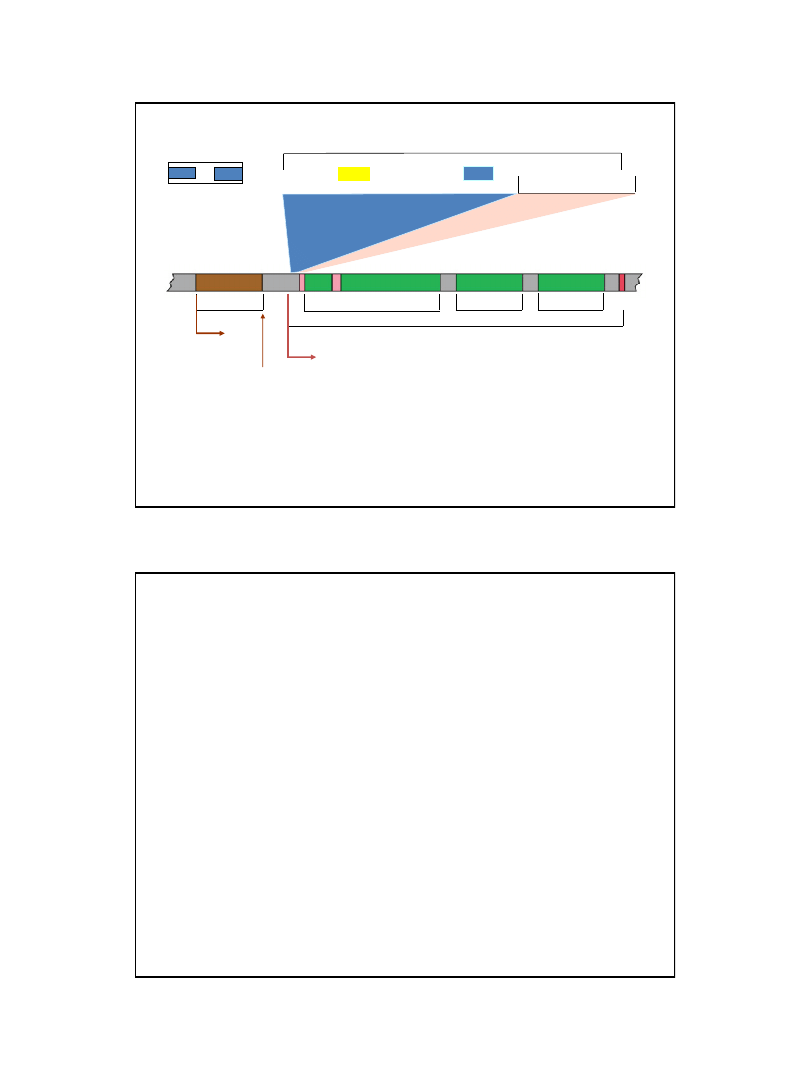
operator (O
1
)
ATTAATGTGAGTTAGCTCACTCATTAGGCACCCCAGGCTTTACACTTTATGCTTCCGGCTCGTATGTTGTGTGGAATTGTGAGCGGATAACAATTTCACAC
-10
miejsce wiązania
białka CAP
region wiązania polimerazy RNA
-35
P
lac
operon lac
lacZ
lacA
lacY
lacI
t
O
2
O
1
P
I
52
pz.
64
pz.
Struktura operonu laktozowego Escherichia coli
lacI
– represor
lacZ
– β-galaktozydaza
lacY
– permeaza laktozy
lacA
– transacetylaza
poz.
– 84
Region DNA oddziałujący z represorem i w ten sposób kontrolujący ekspresję przyległego
genu lub grupy genów. Operator jest zwykle jednym z elementów regulatorowych operonu.
Operator
W operonie laktozowym występują trzy potencjalne miejsca wiązania represora – operatory
w pozycjach:
– 82
,
+ 11
(O
1
) i
+ 412
(O
2
).
Do zablokowania transkrypcji wystarczy przyłączenie się dimeru represora do operatora + 11 (O
1
)
3
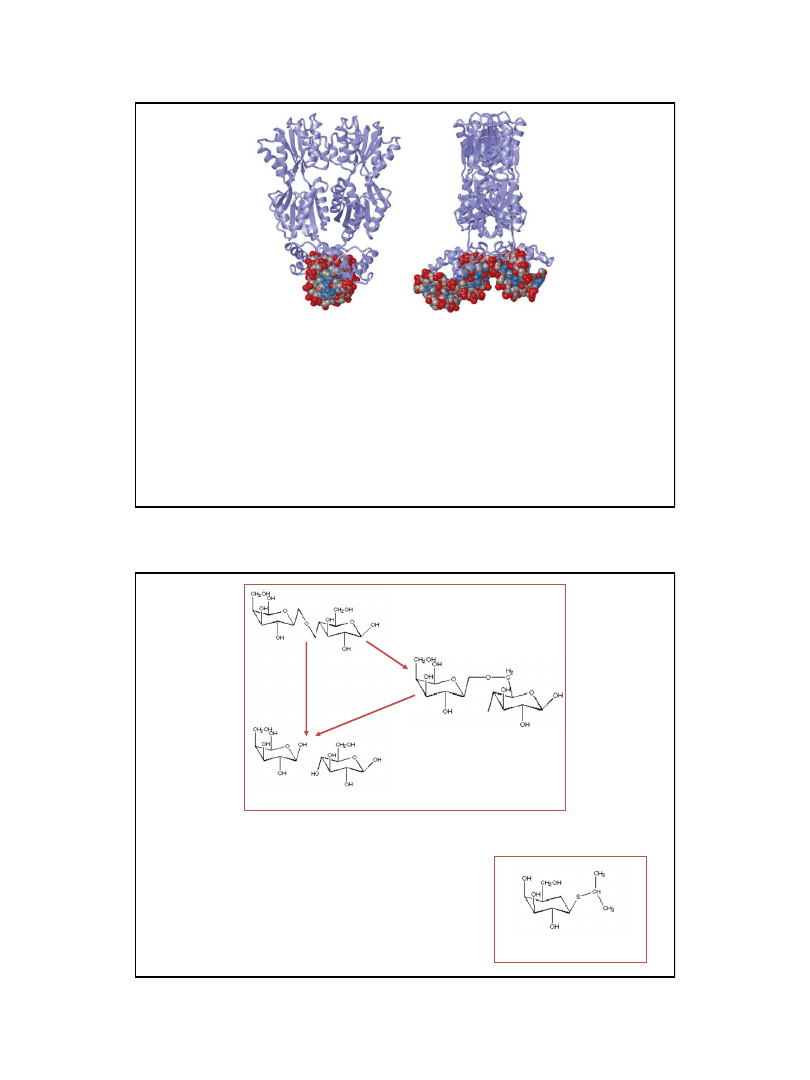
Struktura dimeru represora lac z Escherichia coli
Represor lac z Escherichia coli wiąże się z sekwencją operatora w postaci dimeru.
Może tworzyć tetramery wiążąc jednocześnie dwie sekwencje operatorowe
Białko o c. cząst. ok 37 kDa – produkt genu lacI
W dzikich szczepach E. coli występuje w liczbie ok. 10 cząsteczek na komórkę
(co stanowi 0,001% wszystkich białek E. coli).
Z operatorem wiąże się 4 x 10
6
silniej niż z innymi sekwencjami DNA.
Stała dysocjacji kompleksu represor-operator wynosi 10
-13
M
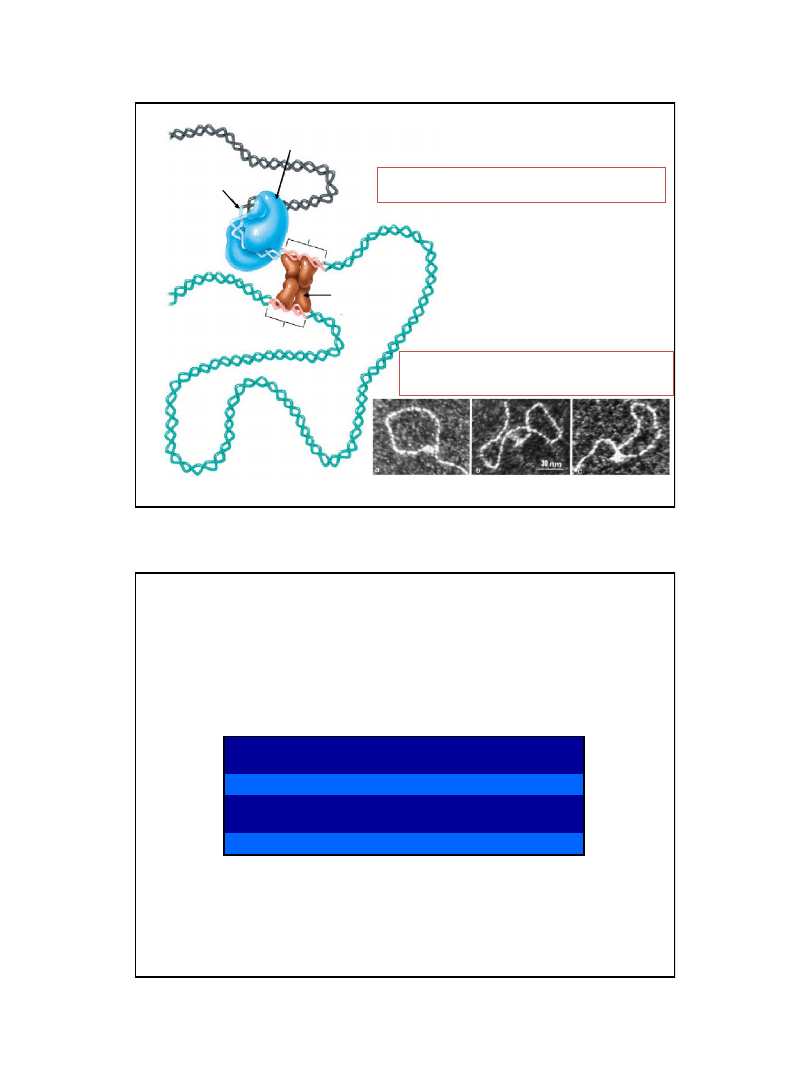
IPTG
(izopropylo-
β-tiogalaktozyd)
allolaktoza
β-galaktozydaza
laktoza
glukoza
galaktoza
silnym, syntetycznym, nie ulegającym
metabolizmowi induktorem jest IPTG
Rozkład laktozy w komórkach Escherichia coli
allolaktoza jest induktorem operonu lac,
słabszym induktorem
jest również laktoza
4
Ryc. z: Horton H.R., Moran L.A., Ochs R.S., Rawn D.J., K Gray Scrimgeour K.G, 2003, Principles of Biochemistry ,
Pearson Higher Education; Prentice-Hall, Inc. ISBN: 0130926434
represor lac
O
1
O
2
Promotor
Polimeraza RNA
Wiązanie tetrameru represora lac z Escherichia coli
do operatorów O
1
i O
2
w nieobecności laktozy
Tworzenie pętli DNA przez jednoczesne wiązanie sie
represora do dwóch operatorów odległych o 535 pz.
zastosowano syntetyczny fragment DNA
Wydajna transkrypcja operonu laktozowego zachodzi jedynie w sytuacji,
gdy w komórce stężenie laktozy jest wysokie, zaś stężenie glukozy jest niskie
(w wyniku czego wzrasta stężenie cAMP)
Regulacja operonu laktozowego
rodzaj cukru w pożywce
relatywny poziom ekspresji
β-galaktozydazy
glukoza
1
glukoza
+
laktoza
50
laktoza
2500
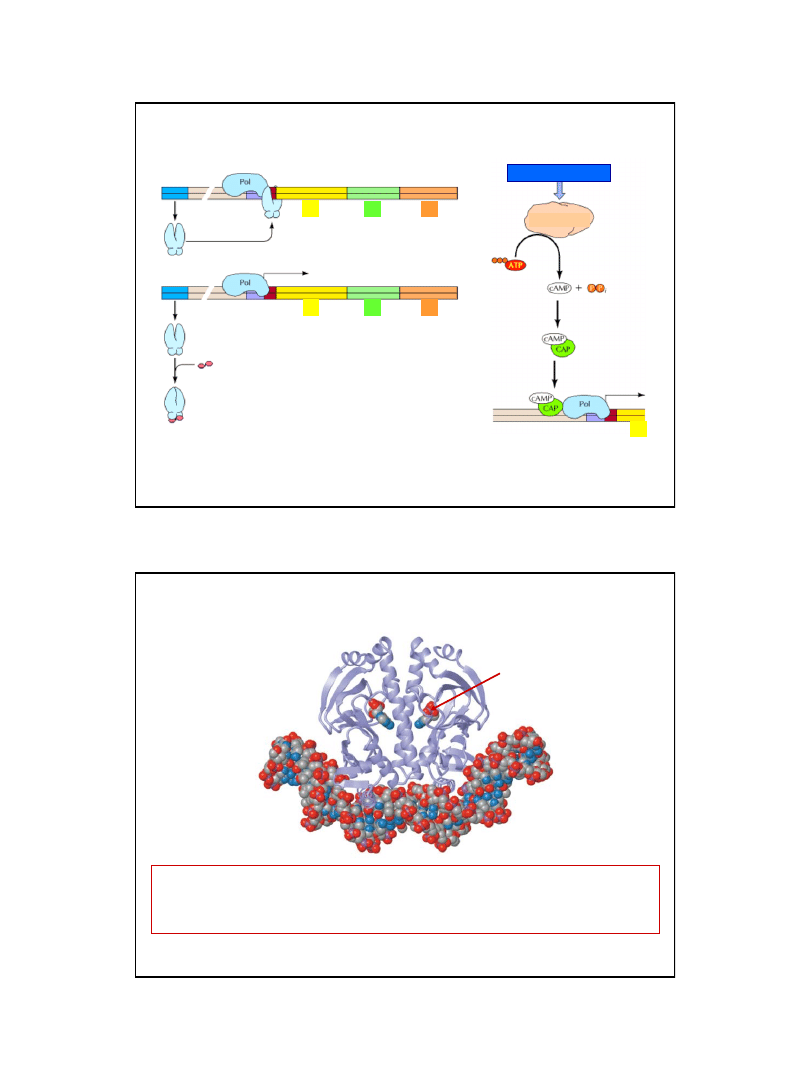
5
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
Transkrypcja jest zablokowana
Laktoza
(allolaktoza)
Represor
nieaktywny
Represor
3’
5’
mRNA lac
3’
5’
3’
5’
3’
5’
I
I
O
P
Z
Y
A
O
P
Z
Y
A
Niski poziom glukozy
cyklaza
adenylowa
P
O
Z
O aktywności operonu laktozowego
decyduje stężenie laktozy....
.... i glukozy
Homodimer (2 x 22 kDa), którego podjednostki zawierają domenę wiążącą DNA, oraz miejsce
wiązania cAMP.
Białko CAP może wiązać się ze specyficznymi odcinkami DNA tylko wówczas, jeżeli przedtem
przyłączy cząsteczkę
cAMP
.
Kompleks cAMP-
CAP może powodować 50 krotne zwiększenie wydajności transkrypcji.
Białko CAP (catabolite gene activator protein)
zwane również: CRP (cAMP receptor protein)
cAMP
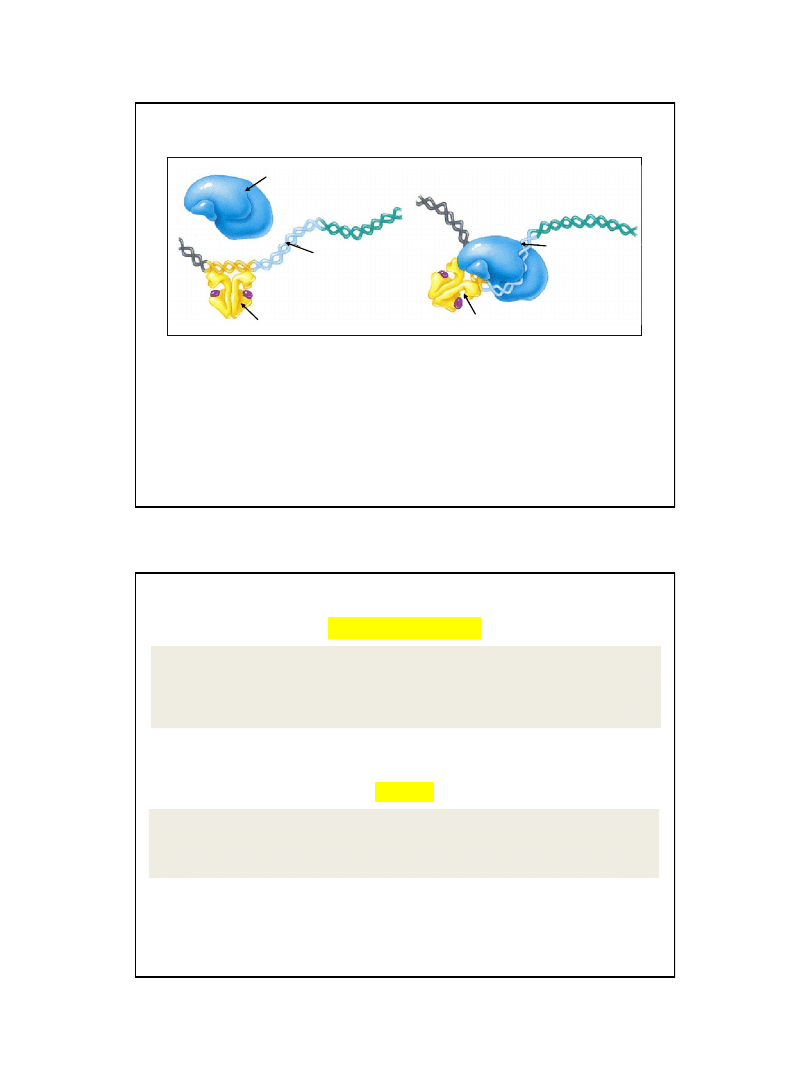
6
Holoenzym polimerazy RNA
dimer CAP-cAMP
dimer CAP-cAMP
Promotor
Promotor
Wiązanie dimeru białka CAP w pobliżu promotora wspomaga tworzenie się
inicjacyjnego kompleksu otwartego a w konsekwencji zwiększa wydajność transkrypcji
inicjowanej przez np. słaby promotor lac
Represja kataboliczna
Represją kataboliczna nazywamy hamujący wpływ glukozy, jako najefektywniejszego substratu
energetycznego, na katabolizm innych cukrowców.
Obecność glukozy w środowisku powoduje obniżenie stężenia enzymów katabolicznych (np. β-
galaktozydaza, galaktokinaza, izomeraza arabinozowa, tryptofanaza) w komórkach Escherichia coli.
Sygnałem molekularnym wysokiego stężenia glukozy jest obniżenie stężenia cAMP.
Poziom cAMP wpływa na ekspresję niektórych genów za pośrednictwem białka CAP
Regulonem nazywamy zespół operonów wykorzystujących wspólny mechanizm regulujący ich
ekspresję.
Regulonem jest np. grupa operonów katabolicznych regulowanych przez białko CAP i cAMP.
Operony danego regulonu nie muszą być (i najczęściej nie są) powiązane strukturalnie – mogą być
zlokalizowane w różnych regionach chromosomu bakteryjnego
Regulon
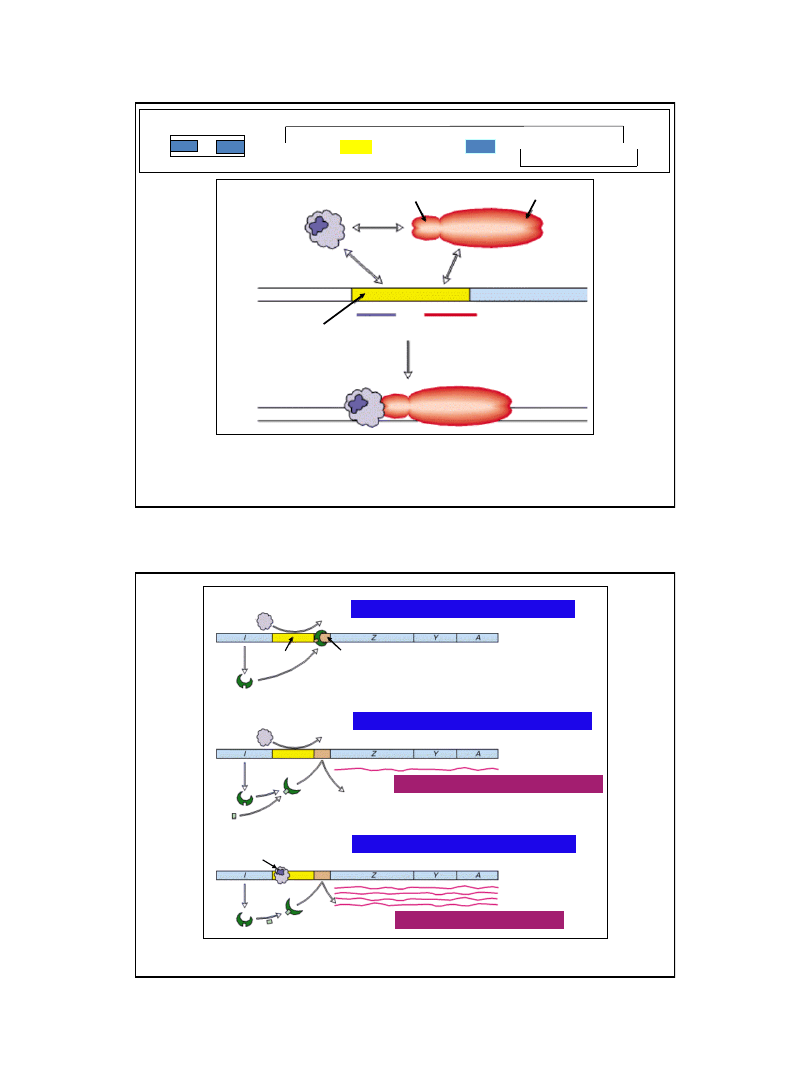
7
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E., 1999,
Molecular Cell Biology, W. H. Freeman & Co.; New York
cAMP-CAP
polimeraza RNA
podjednostka
α
region regulatorowy
operonu laktozowego
miejsce
CAP
miejsce
polimerazy
– 84
– 50
+ 1
Jednoczesne kooperatywne wiązanie białka CAP i polimerazy RNA ze specyficznymi
sekwencjami w obrębie regionu regulatorowego zwiększa wydajność inicjacji transkrypcji
operator (O
1
)
ATTAATGTGAGTTAGCTCACTCATTAGGCACCCCAGGCTTTACACTTTATGCTTCCGGCTCGTATGTTGTGTGGAATTGTGAGCGGATAACAATTTCACAC
-10
miejsce wiązania
białka CAP
region wiązania polimerazy RNA
-35
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E., 1999,
Molecular Cell Biology, W. H. Freeman & Co.; New York
białko CAP
Promotor
kompleks
induktor-represor
cAMP
Laktoza
(induktor)
Operator
Represor
białko CAP
Glukoza obecna (mało cAMP); brak laktozy
Glukoza obecna (mało cAMP); laktoza obecna
Brak glukozy (dużo cAMP); laktoza obecna
niska wydajność transkrypcji mRNA lac
wydajna transkrypcja mRNA lac
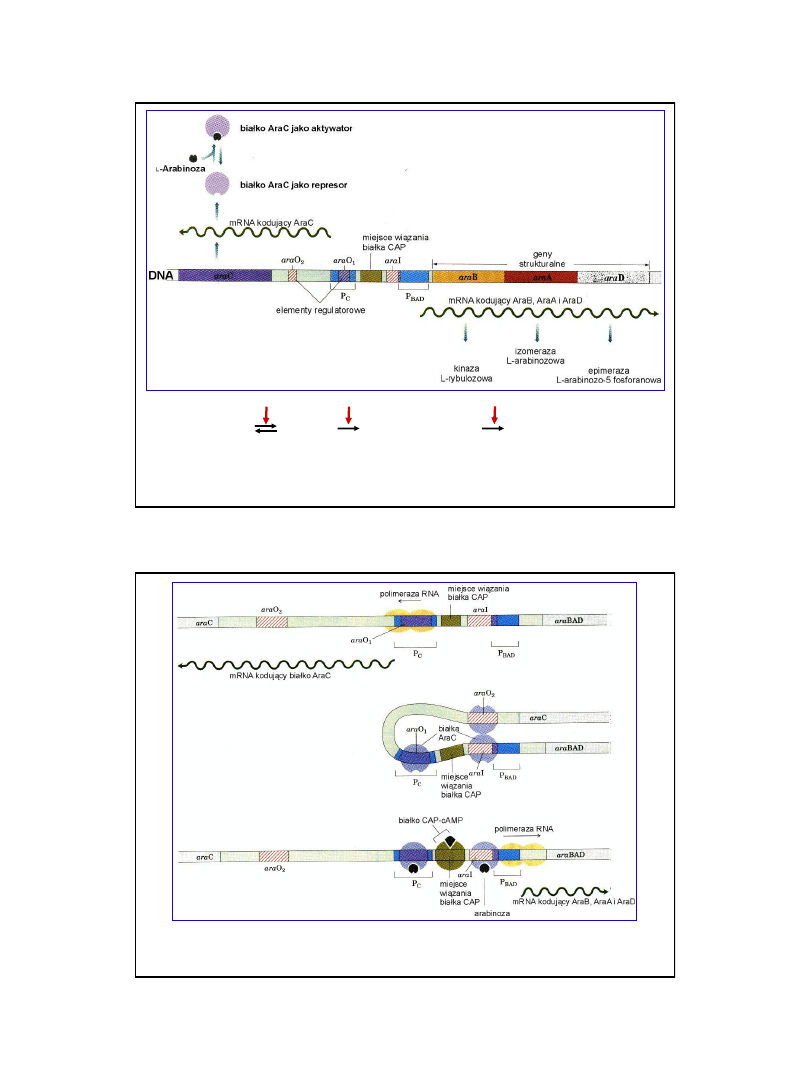
8
Struktura operonu arabinozowego
Ryc. z: Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
kinaza
izomeraza
epimeraza
L-arabinoza L-rybuloza L-rybulozo-5-fosforan D-ksylulozo-5-fosforan
Regulacja operonu arabinozowego
Ryc. z: Lehninger A. L., Nelson D. L., Cox M. M., 1993,
Principles of Biochemistry, 2nd Ed. Worth Publ.Inc.
9
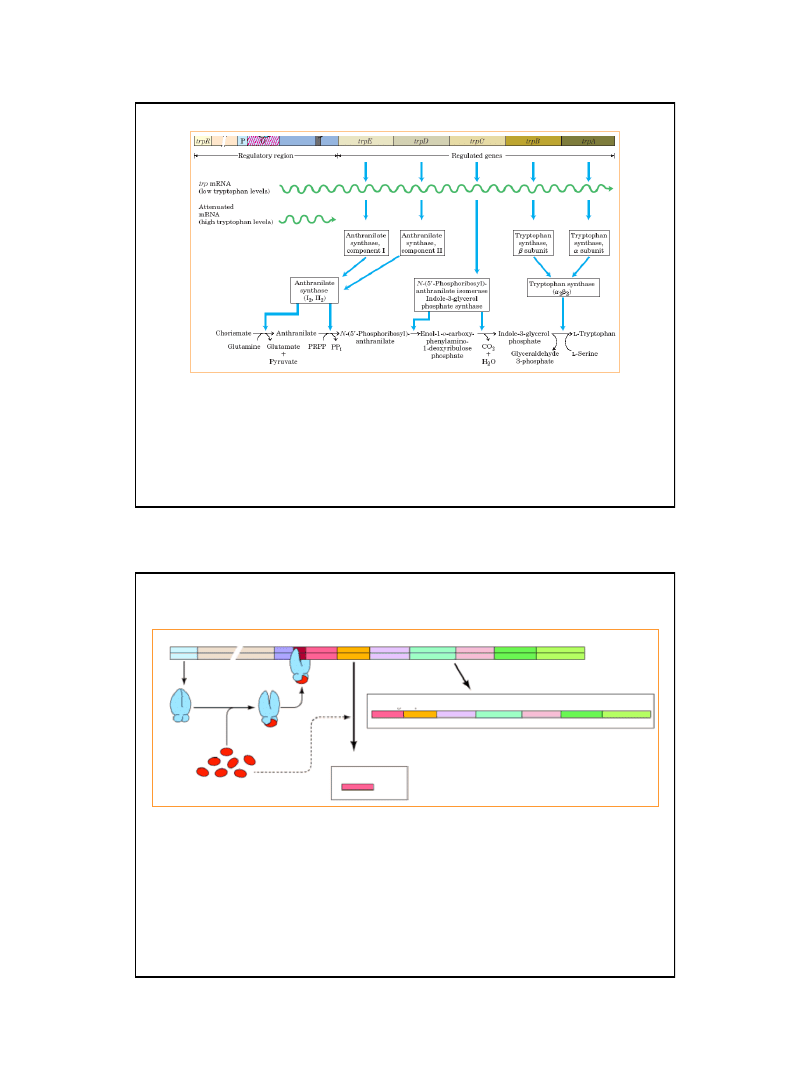
Funkcje białek kodowanych przez poszczególne geny operonu trp
z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
Represor
aktywny
Tryptofan
(korepresor)
Represor
nieaktywny
trpR
trpA
trpB
trpC
trpD
trpE
P O
Att
L
RNA po atenuacji
kompletny mRNA operonu trp
mało tryptofanu
dużo tryptofanu
5’
3’
3’
5’
Regulacja operonu trp
przez represję
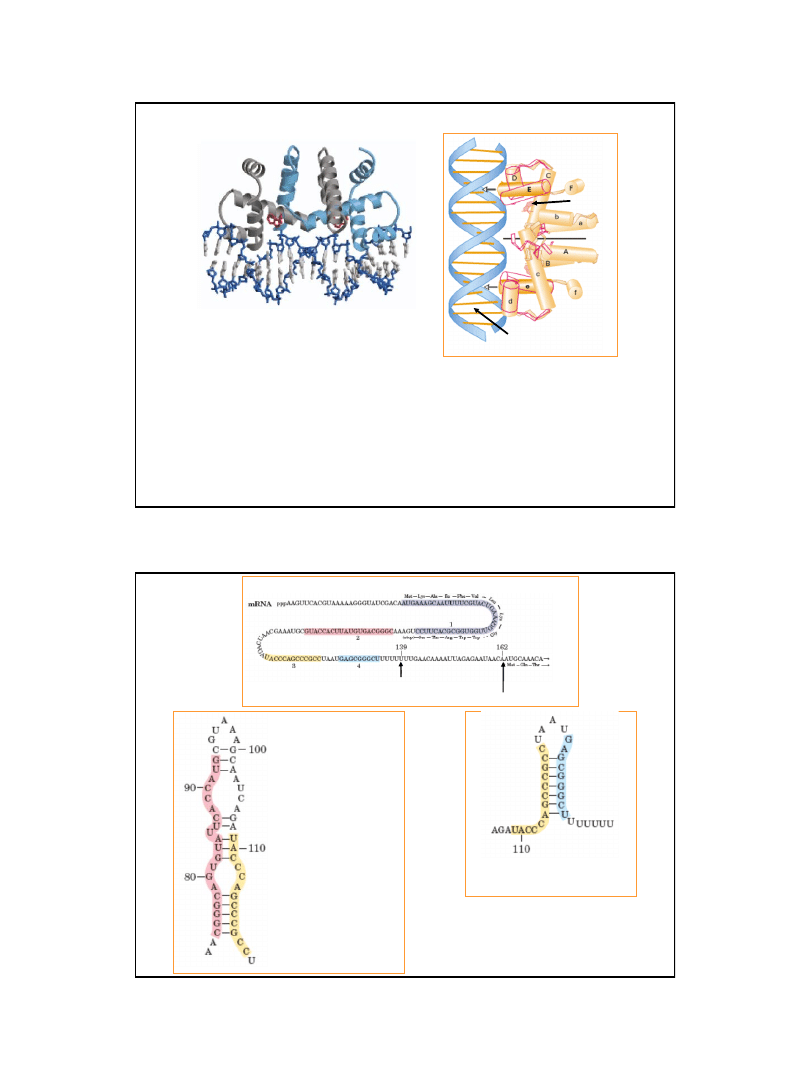
10
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P.,
Baltimore D., Darnell J. E., 1999, Molecular Cell
Biology, W. H. Freeman & Co.; New York
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of
Biochemistry. 4th Ed. W.H. Freeman & Co.; New York
Struktura dimeru represora trp
związanego (w obecności tryptofanu)
z operatorem operonu tryptofanowego
tryptofan
DNA regionu
operatora trp
koniec odcinka liderowego mRNA
miejsce atenuacji
Białko TrpE
Peptyd liderowy
Parowanie regionów 3 i 4
przy wysokim stężeniu tryptofanu
ATENUATOR
Elementy zaangażowane w proces atenuacji
operonu trp
Parowanie regionów 2 i 3
przy niskim stężeniu tryptofanu
wg. Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
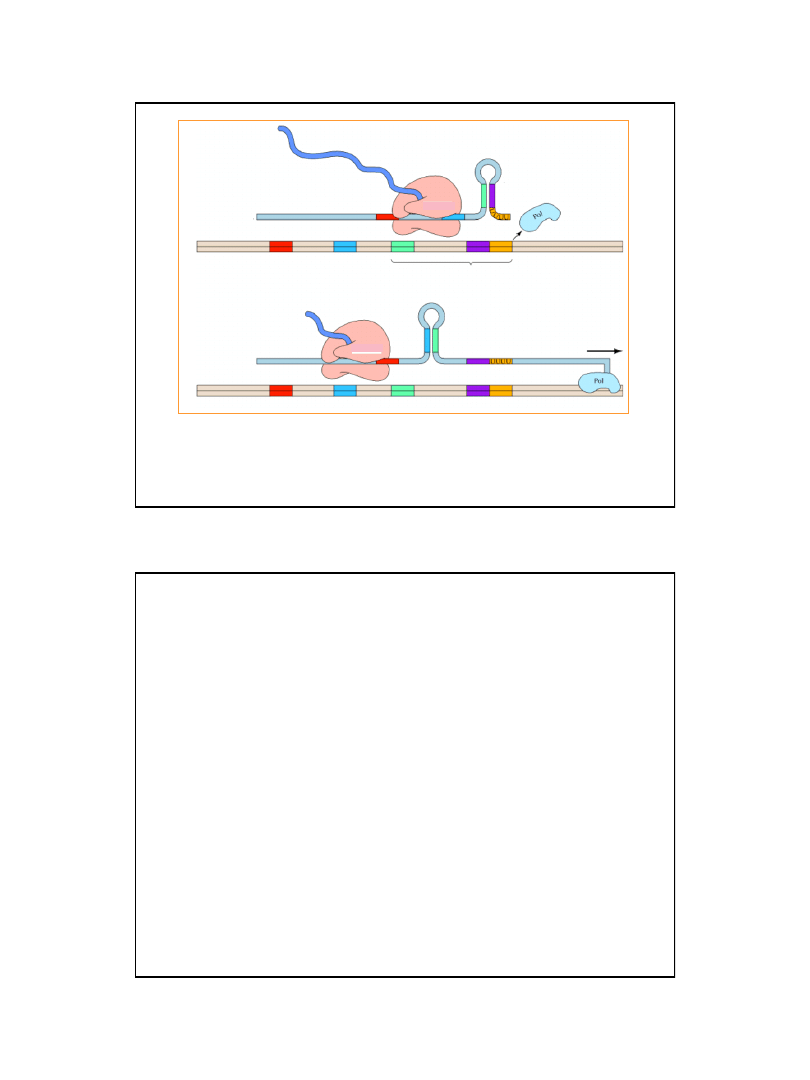
11
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
Rybosom
Rybosom
Atenuator
1
2
3
4
RNA
1
2
3
4
1
2
3
4
1
2
3
4
RNA
Rybosom przemieszcza się
bez przeskód i wchodzi na
region 2
Niskie stężenie tryptofanu
Wysokie stężenie tryptofanu
Rybosom zatrzymuje się
w regionie 1 zawierającym
dwa następujące po sobie
kodony Trp
Regulacja operonu trp
przez atenuację
Zjawisko kontrolowanej, przedwczesnej terminacji transkrypcji wywołane obecnością warunkowego
terminatora
– atenuatora zlokalizowanego zwykle pomiędzy operatorem a pierwszym genem strukturalnym
operonu.
Dla zajścia atenuacji niezbędna jest translacja odcinka liderowego mRNA (obejmującego atenuator). W
zależności od jej przebiegu powstaje, lub nie powstaje drugorzędowa struktura mRNA blokująca dalszą
transkrypcję.
Zjawisko atenuacji jako mechanizm regulacyjny występuje w przypadku operonów kodujących geny
biosyntezy aminokwasów.
Atenuacja
Efektywność regulacji przez represję - 70 x
Efektywność atenuacji 8-10 x
łączna efektywność obu mechanizmów 560 - 700 x obniżona ekspresja genów biosyntezy tryptofanu
Atenuacja jest dodatkowym mechanizmem regulatorowym
uzupełniającym działanie represora tryptofanowego
W przypadku niektórych operonów biosyntezy aminokwasów
atenucja jest JEDYNYM mechanizmem regulacyjnym
12
Thr
Thr
Thr
Thr
Thr
Thr
Thr
Thr
5’
AUG AAA CGC AUU AGC ACC ACC AUU ACC ACC ACC AUC ACC AUU ACC ACA
3’
Met
Lys
Arg
Ser
Ile
Ile
Ile
Ile
Phe
Phe
Phe
Phe
Phe
Phe
Phe
5’
AUG AAA CAC AUA CCG UUU UUC UUC GCA UUC UUU UUU ACC UUC CCC UGA
3’
Met
Lys
Ile
His
Pro
Pro
Ala
Thr
Stop
His
His
His
His
His
His
His
5’
AUG ACA CGC GUU CAA UUU AAA CAC CAC CAC CAC CAC CAC CAC CCU GAC
3’
Met
Lys
Arg
Pro
Thr
Val
Gln
Phe
Asp
Sekwencje aminokwasowe peptydów liderowych i kodujące je sekwencje mRNA
niektórych operonów ulegających atenuacji:
treoninowego
fenyloalaninowego
histydynowego
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E.,
1999, Molecular Cell Biology, W. H. Freeman & Co.; New York
– 140
– 108
Enhancer
glnA
σ
54
polimeraza RNA
NtrB
NtrC
aktywne
nieaktywne
kompleks otwarty
kompleks zamknięty
ADP + P
i
ATP
zgninanie DNA
Aktywacja polimerazy RNA współdziałającej
z podjednostką σ
54
związanej z promotorem
genu glnA (syntetaza glutaminowa)
Polimeraza może wiązać się z promotorem, lecz
nie może zainicjować transkrypcji (przejść w postać
kompleksu otwartego) dopóki nie zostanie uaktywniona
przez kontakt z tetramerem białka NtrC utworzonym
w specyficznym miejscu wiązania.
Białko NtrC pozostaje nieaktywne dopóki nie zostanie
ufosforylowane przez specyficzną kinazę NtrB aktywną
wyłącznie przy niskim poziomie glutaminy
13
operon
β
operon
α
operon str
operon S10
operon sps
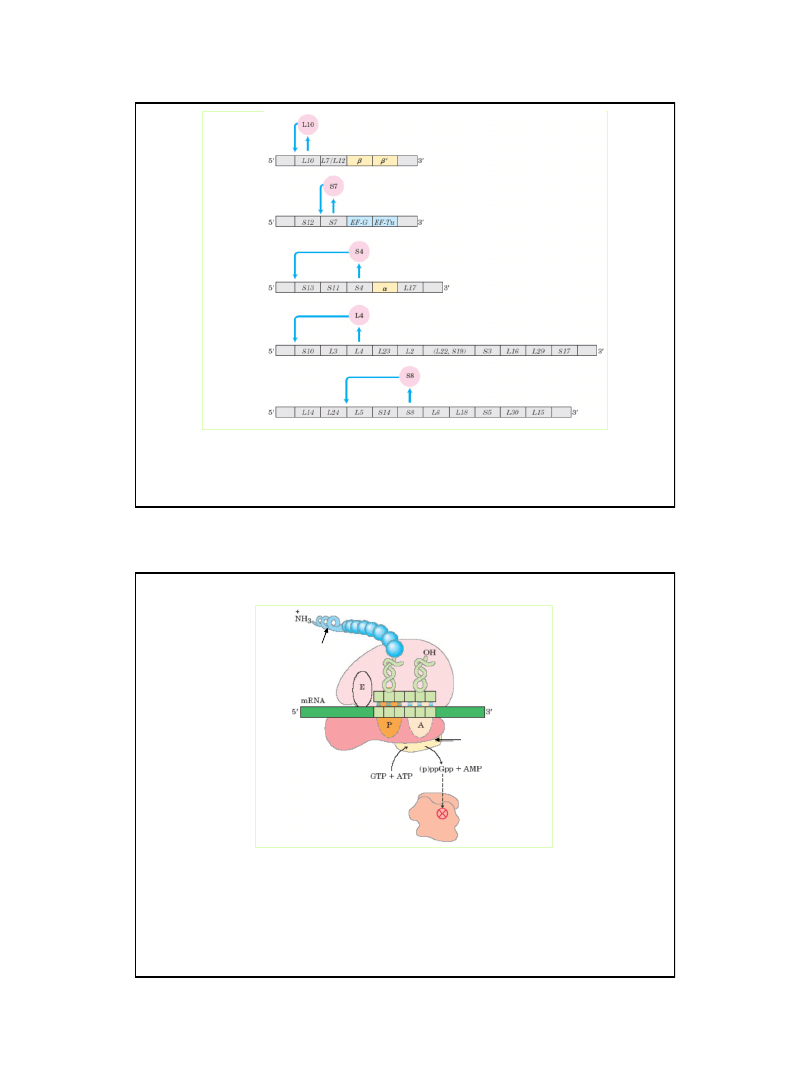
Regulacja operonów kodujących białka rybosomalne Escherichia coli
W co najmniej 20 operonach kodujących 52 białka rybosomalne nie ma niezależnych
sygnałów startu translacji. Jeżeli brakuje rRNA, wskazane białka wiążą się z mRNA
i blokują translację – a w konsekwencji transkrypcję całego operonu.
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
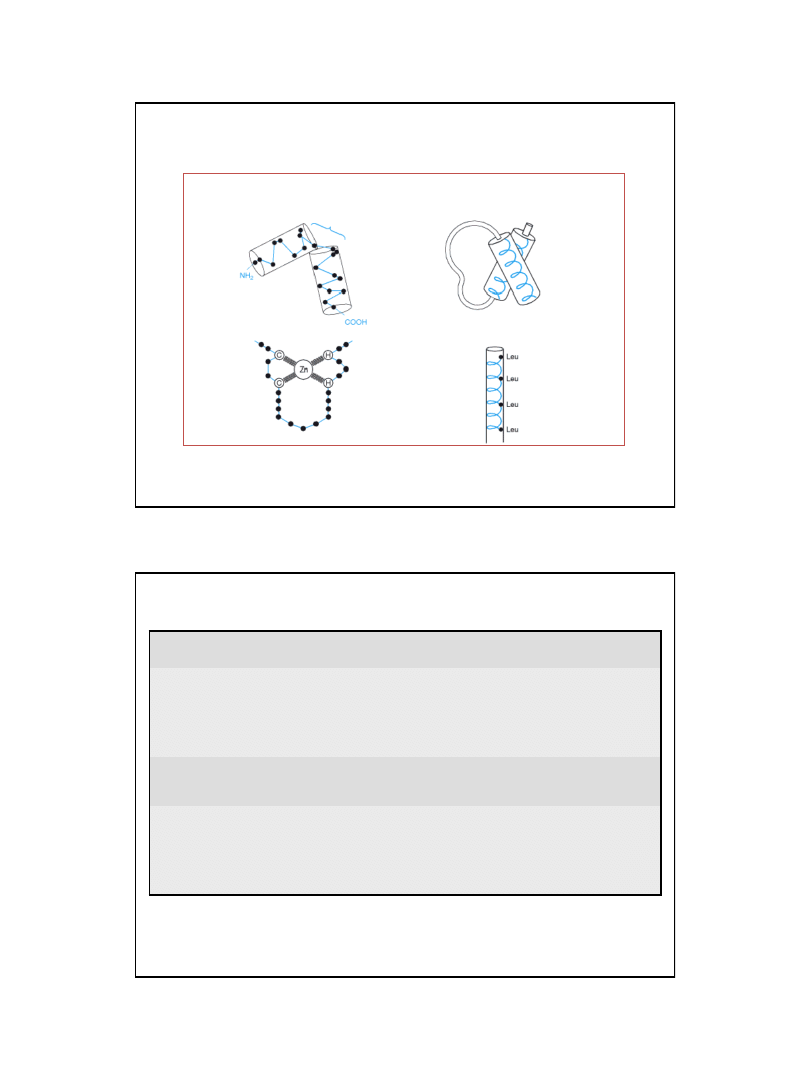
czynnik odpowiedzi
ścisłej (białko RelA)
polimeraza RNA
rosnący łańcuch
polipeptydowy
Odpowiedź ścisła
– mechanizm regulacyjny uruchamiany w odpowiedzi na głód aminokwasowy
Czynnik odpowiedzi ścisłej przyłącza się do rybosomu gdy w miejscu A znajdzie się nieacylowany tRNA.
w kompleksie z rybosomem jest zdolny do syntezy ppGpp.
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
14
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
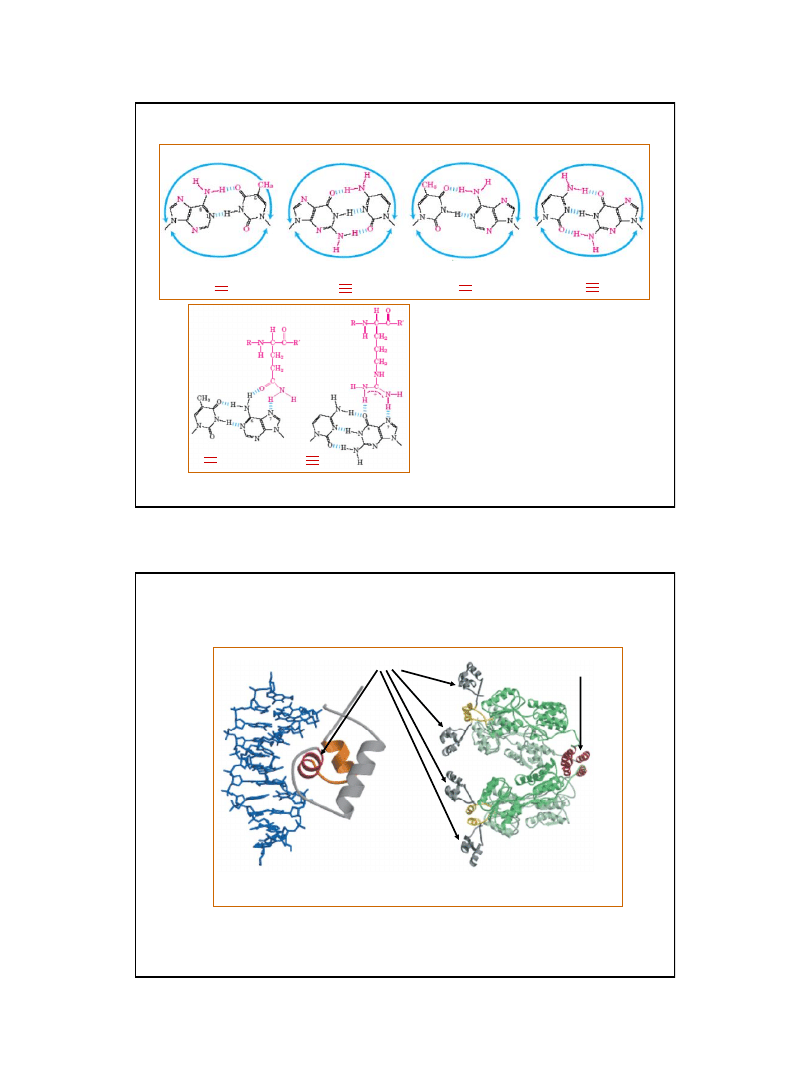
Niektóre motywy strukturalne występujące w białkach wiążących DNA
helisa
α
helisa
α
helisa
α
zwrot
helisa
α
helisa
α
pętla
HLH (helix-loop-helix)
Helisa
– pętla - helisa
HTH (helix-turn- helix)
Helisa
– zwrot - helisa
Zinc finger
Palec cynkowy
Leucine zipper
Suwak leucynowy
Klasyfikacja czynników transkrypcyjnych na podstawie budowy ich domeny wiążącej DNA
Białka zawierające motyw
helisa -
skręt – helisa (HTH)
większość białek regulatorowych u bakterii,
białka homeotyczne u eukariontów
Białka zawierające motywy
palców cynkowych C
2
H
2
(zinc fingers)
TFIIIA
Zif268
SP-1
areA
GATA1
w różnych białkach stwierdzono występowanie
od 1 do 37 palców cynkowych
Białka zawierające
ugrupowania cynkowe (C
4
)
(zinc clusters)
receptory
jądrowe
Białka zawierające motyw
suwaka leucynowego
(leucine zipper)
GCN4
CREB
AP-1
Jun
Fos
zawsze występują w postaci dimerów niektóre
tworzą homodimery, inne heterodimery
15
wg.: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
mniejsza bruzda
większa bruzda
większa bruzda
większa bruzda
większa bruzda
mniejsza bruzda
mniejsza bruzda
mniejsza bruzda
C G
T A
A T
G C
Grupy funkcyjne, które mogą być rozpoznawane przez białka wiążące DNA
w obszarze większej i mniejszej bruzdy zaznaczono kolorem czerwonym
T A
C G
Arginina
Glutamina
(lub asparagina)
Przykłady specyficznych oddziaływań
reszt aminokwasowych z grupami
funkcyjnymi par zasad
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Motyw Helisa
– Zwrot – Helisa (Domena HTH)
Domena HTH represora
laktozowego
Tetramer represora
laktozowego
helisy odpowiedzialne
za wiązanie DNA
helisy odpowiedzialne
za tetrameryzację represora
16
Struktura dimeru represora lac z Escherichia coli
Represor lac z Escherichia coli wiąże się z sekwencją operatora w postaci dimeru.
Może tworzyć tetramery wiążąc jednocześnie dwie sekwencje operatorowe
Białko o c. cząst. ok 37 kDa – produkt genu lacI
W dzikich szczepach E. coli występuje w liczbie ok. 10 cząsteczek na komórkę
(co stanowi 0,001% wszystkich białek E. coli).
Z operatorem wiąże się 4 x 10
6
silniej niż z innymi sekwencjami DNA.
Stała dysocjacji kompleksu represor-operator wynosi 10
-13
M
Homodimer (2 x 22 kDa), którego podjednostki zawierają domenę wiążącą DNA, oraz miejsce
wiązania cAMP.
Białko CAP może wiązać się ze specyficznymi odcinkami DNA tylko wówczas, jeżeli przedtem
przyłączy cząsteczkę
cAMP
.
Kompleks cAMP-
CAP może powodować 50 krotne zwiększenie wydajności transkrypcji.
Białko CAP (catabolite gene activator protein)
zwane również: CRP (cAMP receptor protein)
cAMP
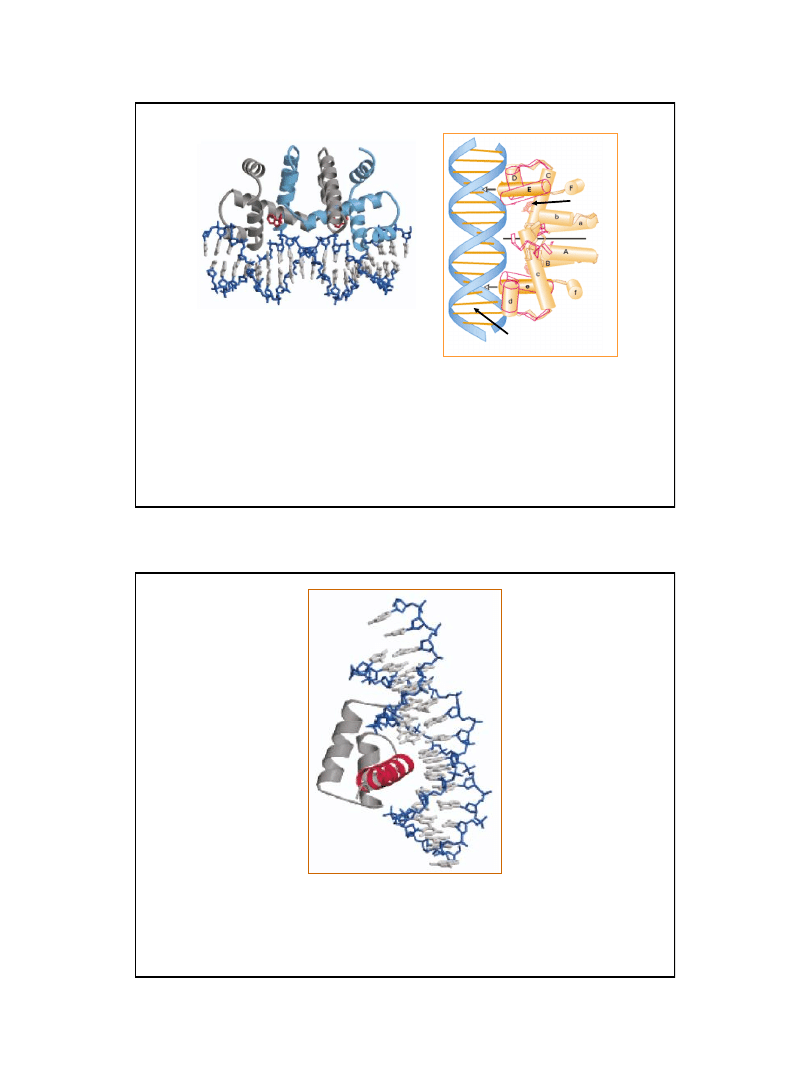
17
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P.,
Baltimore D., Darnell J. E., 1999, Molecular Cell
Biology, W. H. Freeman & Co.; New York
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of
Biochemistry. 4th Ed. W.H. Freeman & Co.; New York
Struktura dimeru represora trp
związanego (w obecności tryptofanu)
z operatorem operonu tryptofanowego
tryptofan
DNA regionu
operatora trp
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Homeodomena
zaznaczono na czerwono helisa stabilizowana przez dwie pozostałe oddziałuje z grupami funkcyjnymi
w obrębie większej bruzdy DNA.
Przedstawiona struktura jest częścią (domeną) znacznie większego białka Ultrabithorax (Ubx)
zaangażowanego w regulację rozwoju embrionalnego Drosophila
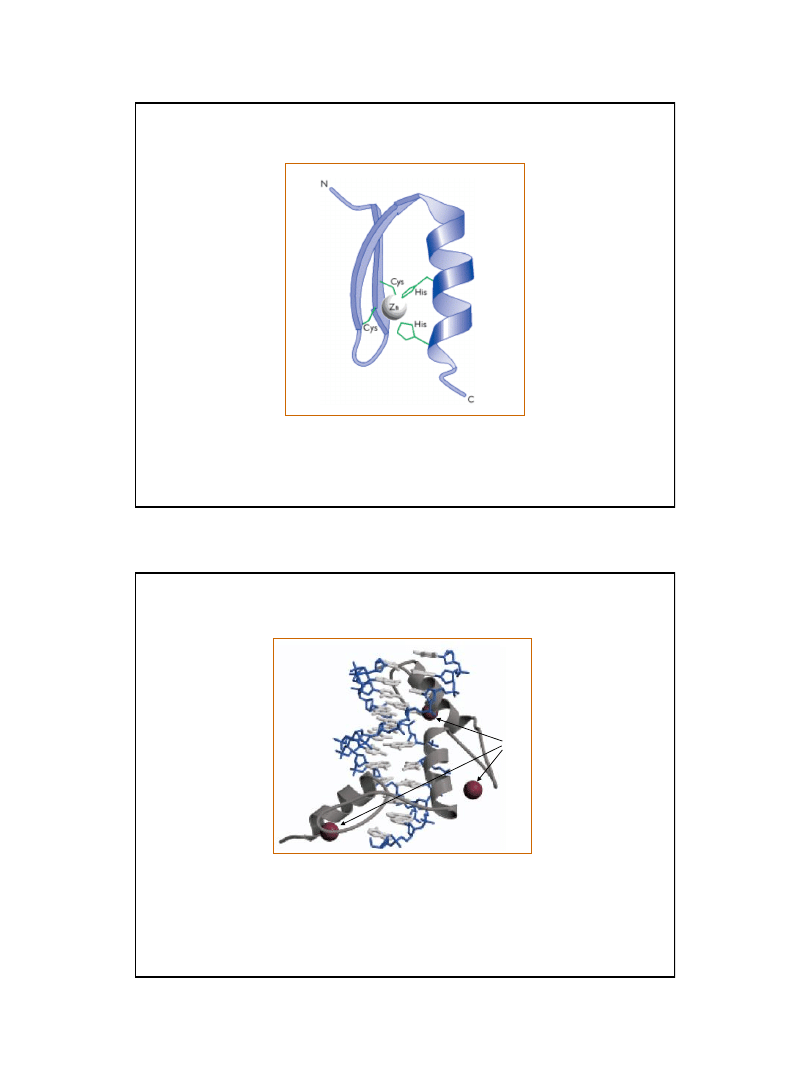
18
Motyw palca cynkowego typu C
2
H
2
helisa
α
struktura
β
Ryc. z: Brown T.A., 2002, Genomes, 2nd ed. , BIOS Scientific Publishers Ltd.
fragment białka Swi5
(S. cerevisiae)
trzy palce cynkowe (kolor szary)
– fragment białka regulatorowego Zif268
Zn
2+
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Palce cynkowe
Zinc fingers
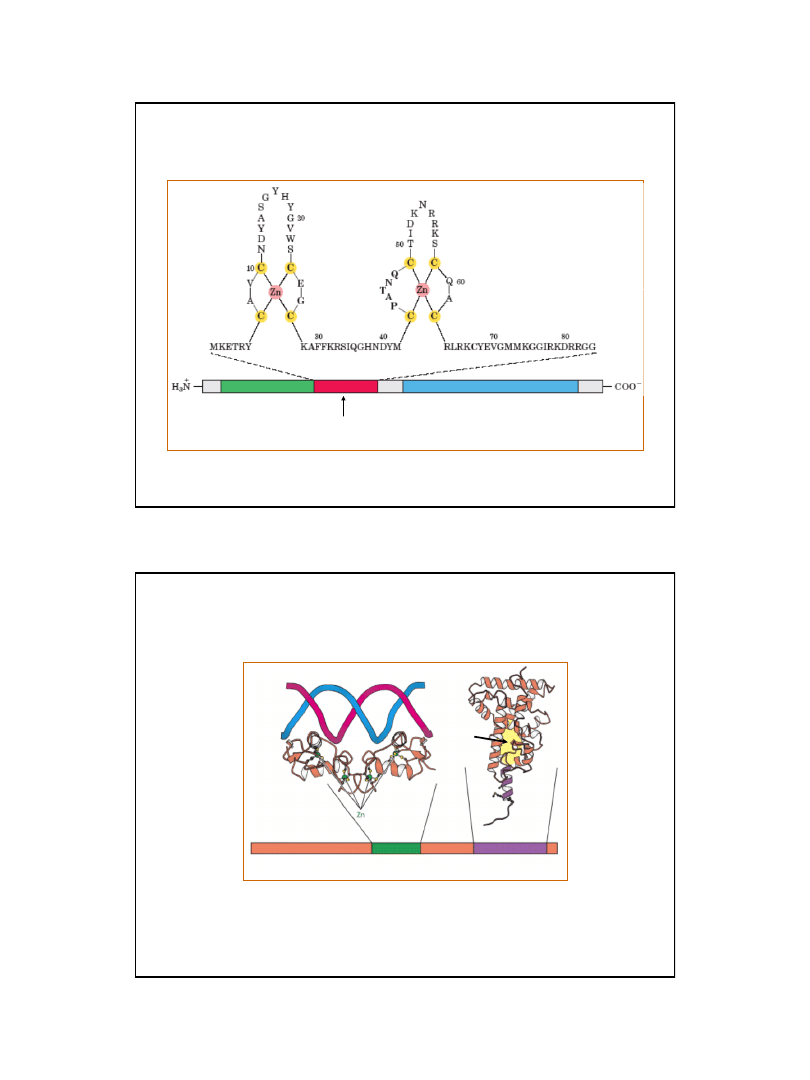
19
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Struktura typowego receptora hormonów steroidowych
Zawiera motywy palców cynkowych typu C
4
(zwanych czasem „ugrupowaniami cynkowymi” – zinc clusters)
region wiązania DNA
(wysoce zachowawczy)
domena wiążąca hormon
(zmienna)
domena aktywująca
transkrypcję (zmienna)
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry 5ed., W.H. Freeman and Co., New York
Region
wiązania DNA
domena wiążąca hormon
ligand
Struktura domenowa jądrowego receptora hormonów
przedstawiono strukturę monomeru, choć regułą jest dimeryczna struktura receptorów tego typu
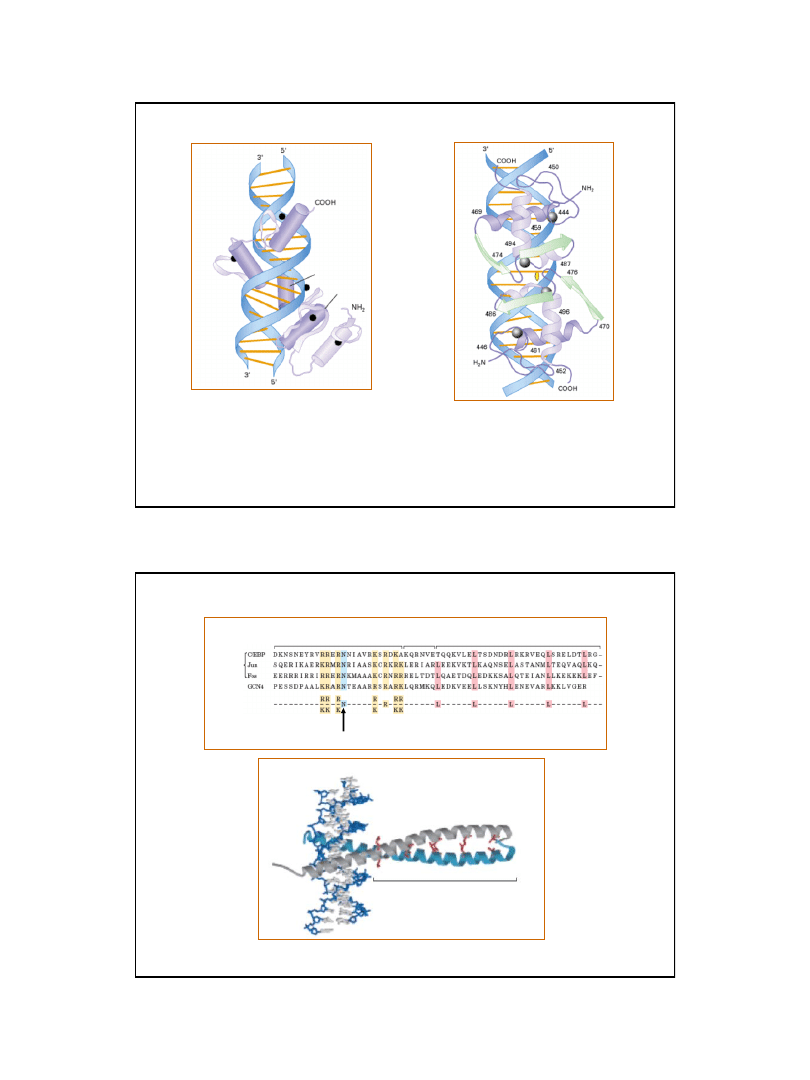
20
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E., 1999,
Molecular Cell Biology, W. H. Freeman & Co.; New York
Przykłady białek zawierających motywy palców cynkowych
palec 2
palec 3
palec 1
palec 4
palec 5
Monomeryczne bialko GL1 (H. sapiens) zawiera
pięć motywów palców cynkowych typu C
2
H
2
Receptor glukokortykoidów – homodimeryczne
bialko zawierające palce cynkowe typu C
4
Ssaki
Drożdże
sekwencja
konsensusowa
Region wiążący DNA
Suwak leucynowy
Łącznik
(6 aa.)
Asparagina
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Motyw suwaka leucynowego (leucine zipper)
Suwak leucynowy
fragment białka GCN4 (helisy
tworzące suwak leucynowy)

21
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Motyw Helisa
– Pętla – Helisa (HLH)
(helix-loop-helix)
Leu
Leu
Dimer czynnika transkrypcyjnego Max (H. sapiens)
– jedną z podjednostek
zaznaczono kolorem, druga jest szara
Polimerazy RNA organizmów eukariotycznych
Polimeraza I
transkrypcja: 5,8S rRNA, 18S rRNA,
28S rRNA
niewrażliwa na α-amanitynę
Polimeraza II
transkrypcja: mRNA, większość snRNA wrażliwa na α-amanitynę
Polimeraza III
transkrypcja: 5S rRNA, tRNA, U6
snRNA, snoRNA scRNA np.(7SL RNA)
wrażliwa na podwyższone
stężenie α-amanityny
22
UCE
Polimeraza I RNA
Polimeraza II RNA
Polimeraza III RNA (5S rRNA)
Promotor wewnętrzny
– 45 + 20
– 25 Inr
elementy kontrolujące
Porównanie struktury promotorów rozpoznawanych przez eukariotyczne polimerazy RNA
Polimerazy RNA organizmów eukariotycznych
Polimeraza I
transkrypcja: 5,8S rRNA, 18S rRNA,
28S rRNA
niewrażliwa na α-amanitynę
Polimeraza II
transkrypcja: mRNA, większość snRNA wrażliwa na α-amanitynę
Polimeraza III
transkrypcja: 5S rRNA, tRNA, U6
snRNA, snoRNA scRNA np.(7SL RNA)
wrażliwa na podwyższone
stężenie α-amanityny
23
UCE
Polimeraza I RNA
Polimeraza II RNA
Polimeraza III RNA (5S rRNA)
Promotor wewnętrzny
– 45 + 20
– 25 Inr
elementy kontrolujące
Porównanie struktury promotorów rozpoznawanych przez eukariotyczne polimerazy RNA
Geny kodujące białka regulujące ekspresję innych genów nazywamy genami
regulatorowymi lub regulatorami.
Białkowe produkty ich ekspresji to czynniki transkrypcyjne - induktory lub represory, ogólnie
nazywane
czynnikami działającymi w układzie trans (trans - acting factors).
Fragment DNA nie kodujący żadnego produktu, lecz wpływający na ekspresję innej sekwencji
DNA (genu), z którym jest fizycznie powiązany (jest elementem tej samej cząsteczki DNA co
gen) nazywamy
sekwencją regulatorową lub elementem działającym w układzie cis (cis -
acting element).
Przykładami elementów działających w układzie cis są promotory i terminatory transkrypcji.
Czynniki działające w układzie trans (białka) działają przez wiązanie się z elementami cis
(sekwencje DNA)
Geny kodujące polipeptydy lub stabilne cząsteczki RNA (np. tRNA, rRNA)
GENY REGULATOROWE, CZYNNIKI trans, ELEMENTY cis
GENY STRUKTURALNE

24
GENY ULEGAJĄCE EKSPRESJI
W SPOSÓB CIĄŁY
(konstytutywny)
GENY, KTÓRYCH EKSPRESJA JEST REGULOWANA
Geny indukowane
Geny ulegające represji
Produkty ich ekspresji są niezbędne dla
funkcjonowania
każdej komórki
danego organizmu
Produkty ich ekspresji są niezbędne
tylko w niektórych typach komórek,
lub tylko w pewnych warunkach
Produkty ich ekspresji są niezbędne
tylko w niektórych typach komórek,
lub tylko w pewnych warunkach
Transkrypcja może zostać włączona
pod wpływem aktywatorów
oddziałujących z sygnałami
molekularnymi.
Transkrypcja może zostać wyłączona
pod wpływem represorów. Włączenie
transkrypcji wymaga usunięcia
represora.
Sygnał molekularny może powodować
w pewnych wypadkach dołączenie
aktywatora, w innych jego dysocjację
Sygnał molekularny może powodować
w pewnych wypadkach dołączenie
represora, w innych jego dysocjację
Kodują (na przykład):
Kodują (na przykład):
Kodują (na przykład):
białka rybosomalne, enzymy cyklu
Krebsa i innych przemian
metabolicznych o podstawowym
znaczeniu
białka naprawiające uszkodzenia DNA,
białka specyficzne dla danego typu
typu komórek, itp.
enzymy biosyntezy tryptofanu u bakterii
W komórkach eukariontów ich
promotory
nie zawierają sekwencji
TATA; dla ich transkrypcji zasadniczo
wystarcza obecność sekwencji bogatej
w pary GC wiążącej czynnik
transkrypcyjny SP1
Klasa obejmuje większość genów
eukariotycznych. Ich promotory
zawierają sekwencję TATA oraz
różnorodne sekwencje regulatorowe
wiążące różne czynniki transkrypcyjne.
Sekwencje regulatorowe mogą być
zlokalizowane również poza regionem
promotorowym, często w znacznej
odległości od miejsca startu
transkrypcji.
Klasyfikacja genów danego organizmu pod względem regulacji ich ekspresji
Porównanie mechanizmów odpowiedzialnych za regulację ekspresji genów
Organizmy prokariotyczne
Organizmy eukariotyczne
Holoenzym polimerazy RNA jest zdolny do
rozplecenia podwójnej helisy DNA w miejscu
startu transkrypcji (przejścia od kompleksu
promotorowego zamkniętego do kompleksu
promotorowego otwartego)
Polimerazy RNA
nie są zdolne do transkrybowania
DNA bez udziału dodatkowych czynników białkowych.
Z miejsca startu transkrypcji muszą zostać usunięte
nukleosomy
Geny eukariotyczne są w większości nieaktywne,
dopóki nie zostaną włączone w swoisty dla siebie
sposób
Ekspresja genu może być skutecznie
kontrolowana przez jedno, lub dwa
białka
regulatorowe
Większość genów jest kontrolowana przez liczne
białka, wiążące się do kilku sekwencji regulatorowych i
tworzące między sobą kombinatoryczne połączenia
Geny są zorganizowane w kilkugenowe jednostki
podlegające wspólnej regulacji (operony)
transkrybowane w postaci policistronowego
mRNA
kodującego kilka białek.
Pojedynczy sygnał molekularny może włączać
lub wyłączać jednocześnie wszystkie geny
wchodzące w skład danego operonu.
Geny
nie są zorganizowane w zespoły podlegające
wspólnemu elementowi regulatorowemu.
Monocistronowość eukariotycznego mRNA
wymusza indywidualną regulację każdego genu
strukturalnego.
Dany sygnał molekularny może włączać zestawy
genów, o ile sekwencje regulatorowe, podlegające
aktywowaniu przez ten sygnał, występują w każdym
z elementów tego zestawu.
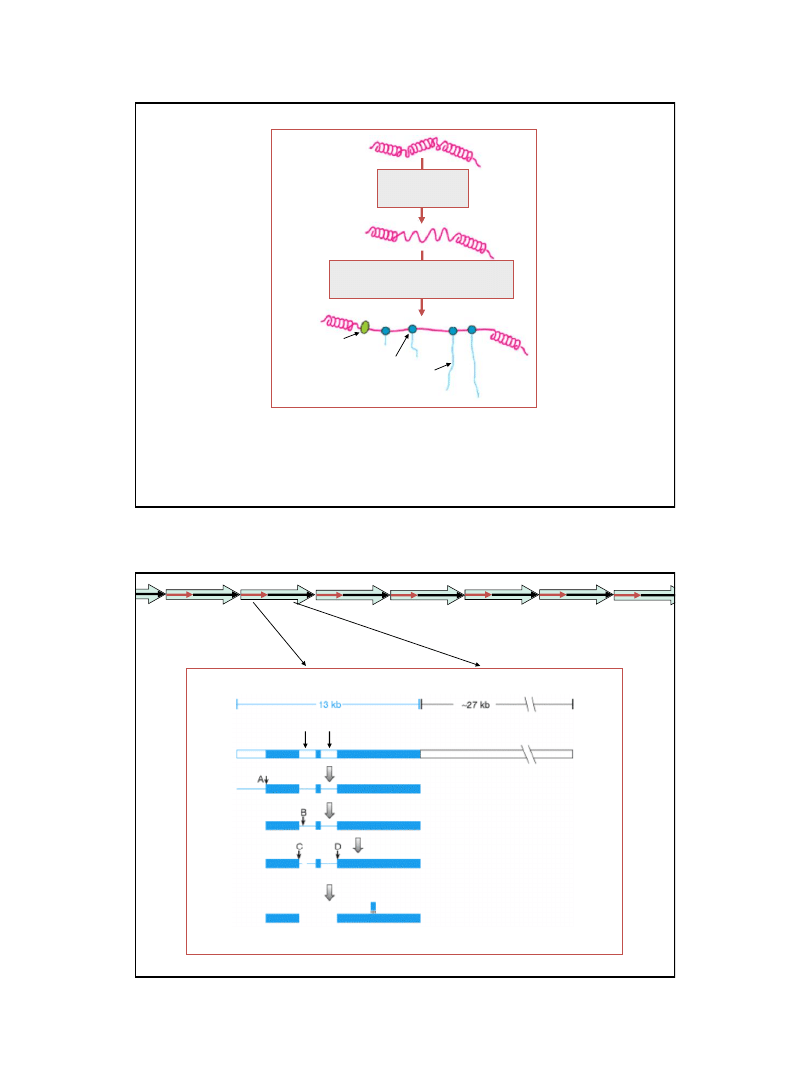
25
ETAP II
Dołączenie czynników transkrypcyjnych
i inicjacja transkrypcji genu
ETAP I
Zmiana struktury
chromatyny
RNA
kompleks białek
inicjujących
transkrypcję
polimeraza RNA
Główne etapy regulacji ekspresji genów organizmów eukariotycznych
sekwencja rDNA ulegająca
transkrypcji
sekwencja międzygenowa
(nie ulega transkrypcji)
– NTS
28S
ITS1 ITS2
18S
5,8S
ETS
20S
45S
32S
DNA
18S
Transkrypt
pierwotny
41S
5,8S
28S
A
– D – miejsca cięcia prekursora przez
specyficzne endonukleazy
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
ok. 40 kpz.
26
Liczba kopii genów rRNA i tRNA w genomach różnych organizmów eukariotycznych
Gatunek
Geny 18S/5,8S/28S
rRNA
Geny 5S
rRNA
Geny tRNA
Drożdże (S. cerevisiae)
140
140
250
Dictyostelium discoideum
180
180
?
Drosophila melanogaster
(X)
(Y)
250
150
165
850
Człowiek
280
2 000
1 300
Xenopus laevis
450
24 000
1 150
Dla porównania: W komórkach Escherichia coli jest 7 kopii zespołu genów kodujących prekursory
5S-16S-
23S rRNA, oraz 60 kopii genów tRNA.
Ryc. z: Lewin B., (2000), GenesVII, Oxford University Press
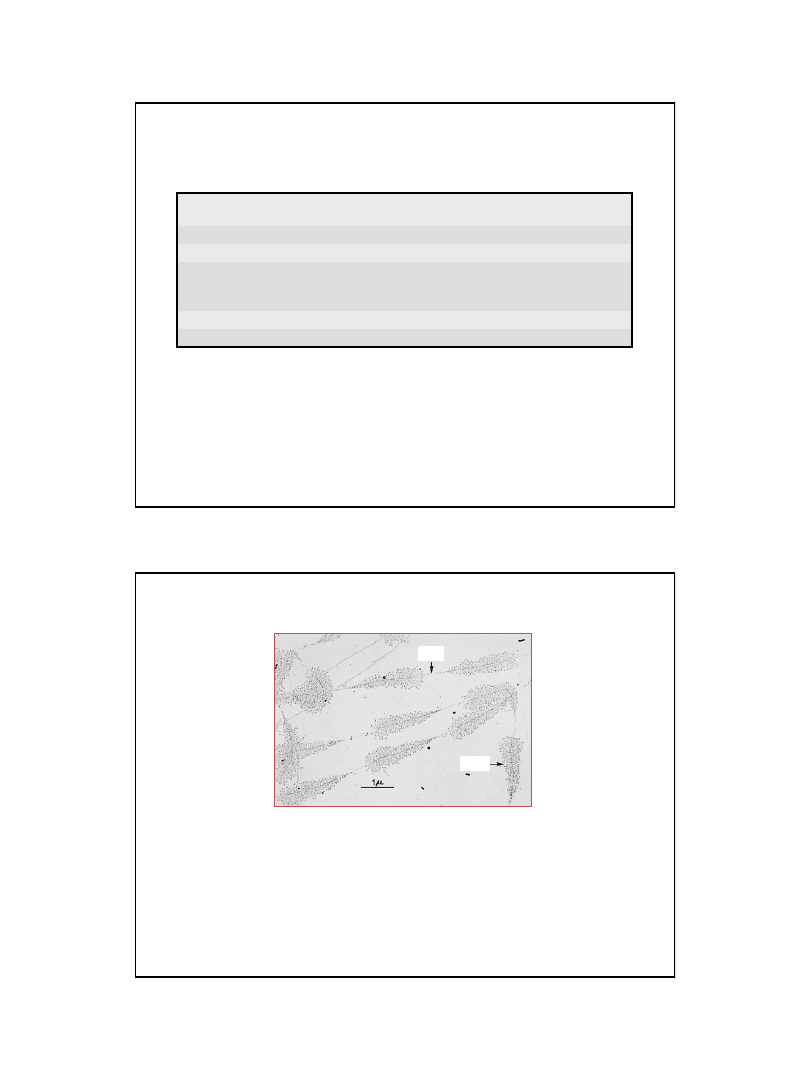
NTS
rRNA
fot. Oscar Miller
Mikrofotografia elektronowa ulegających transkrypcji tandemowych powtórzeń rDNA
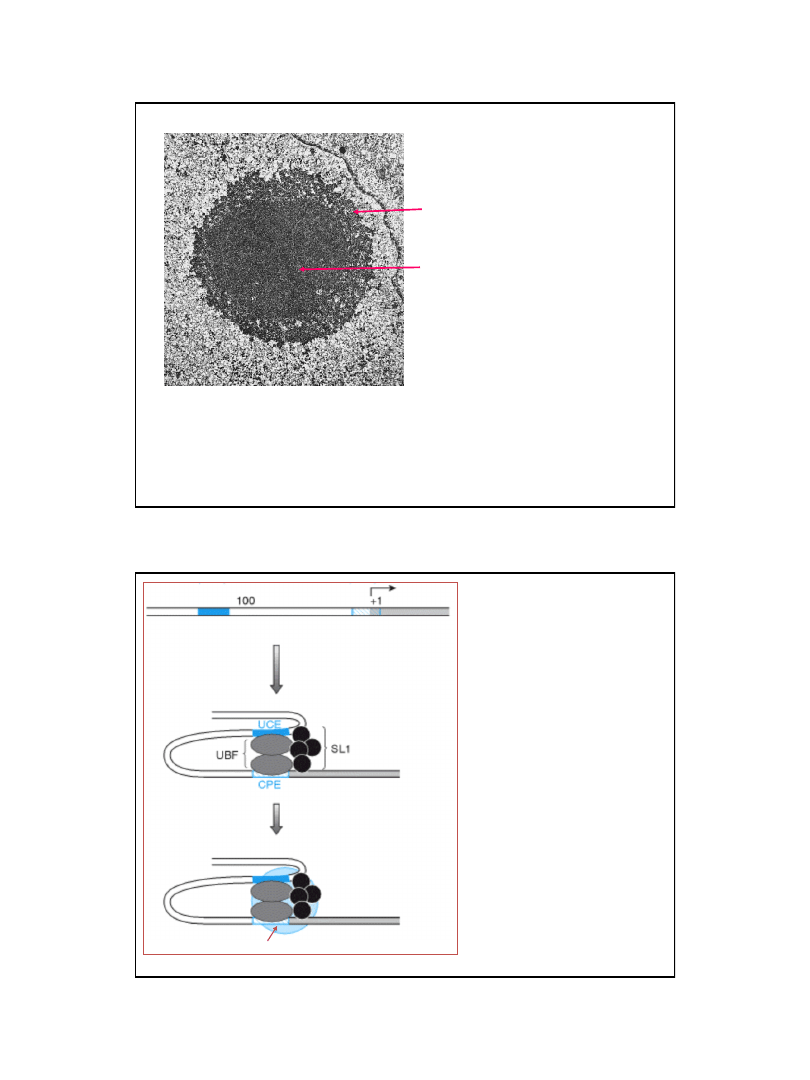
27
Mikrofotografia elektronowa jąderka Notopthalmus viridescens (Amphibia)
fot. Oscar Miller
granularna „kora” jąderka – obszar, w którym
powstają podjednostki rybosomalne
rdzeń jąderka – obszar, w którym
zachodzi transkrypcja rDNA
Ryc. z: Lewin B., (2000), GenesVII, Oxford University Press
CPE
UCE
związanie czynników UBF i SL1
umożliwia przyłączenie
polimerazy I RNA
Polimeraza I RNA
UCE
– upstream control element
CPE
– core promotor element
SL1
– selectivity factor 1
UBF
– upstream binding factor
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
Struktura promotora rozpoznawanego
przez polimerazę I RNA
28
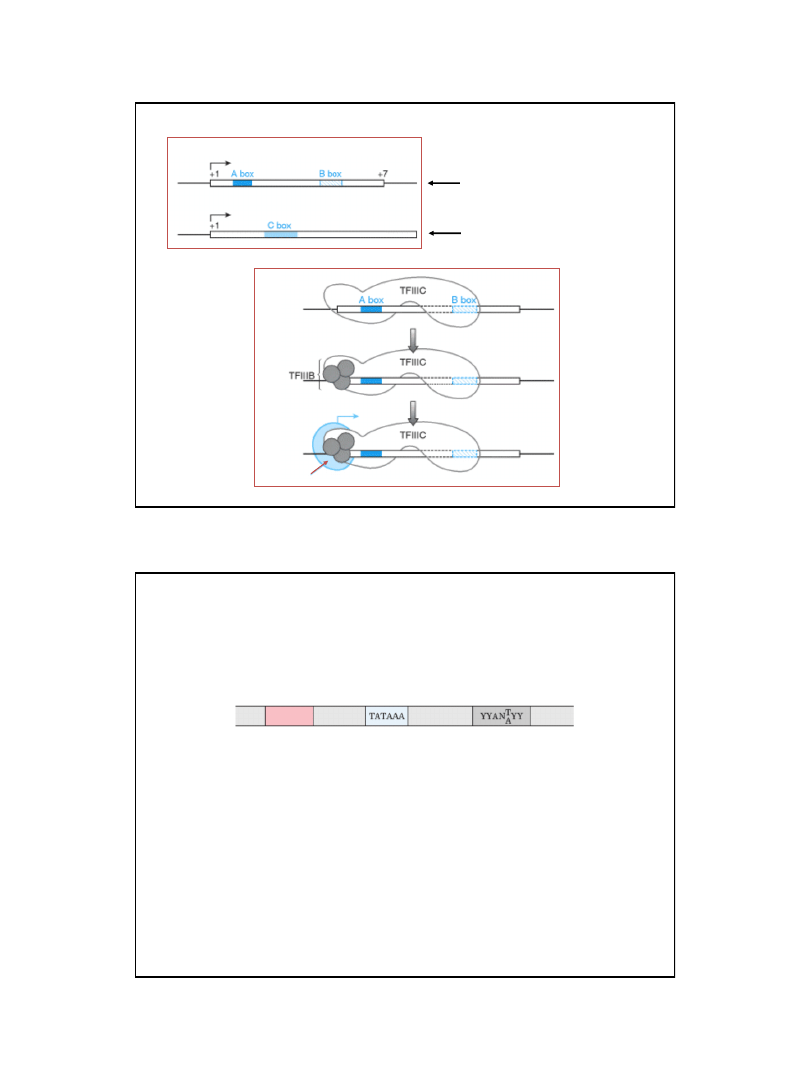
polimeraza III RNA
Promotory genów 5S rRNA i tRNA (rozpoznawane przez polimerazę III RNA)
są zlokalizowane w obrębie sekwencji kodującej
Inicjacja transkrypcji genu tRNA
Promotory genów 5S rRNA
Promotory genów tRNA
TFIIIC, TFIIIB, polimeraza III RNA
TFIIIA, TFIIIB, TFIIIC, polimeraza III RNA
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
+1
– 30
3’
5’
Różne sekwencje
regulatorowe
sekwencja TATA
(kaseta TATA
– TATA box)
Inr
Topografia promotorów genów transkrybowanych przez polimerazę II RNA
organizmów eukariotycznych
29
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
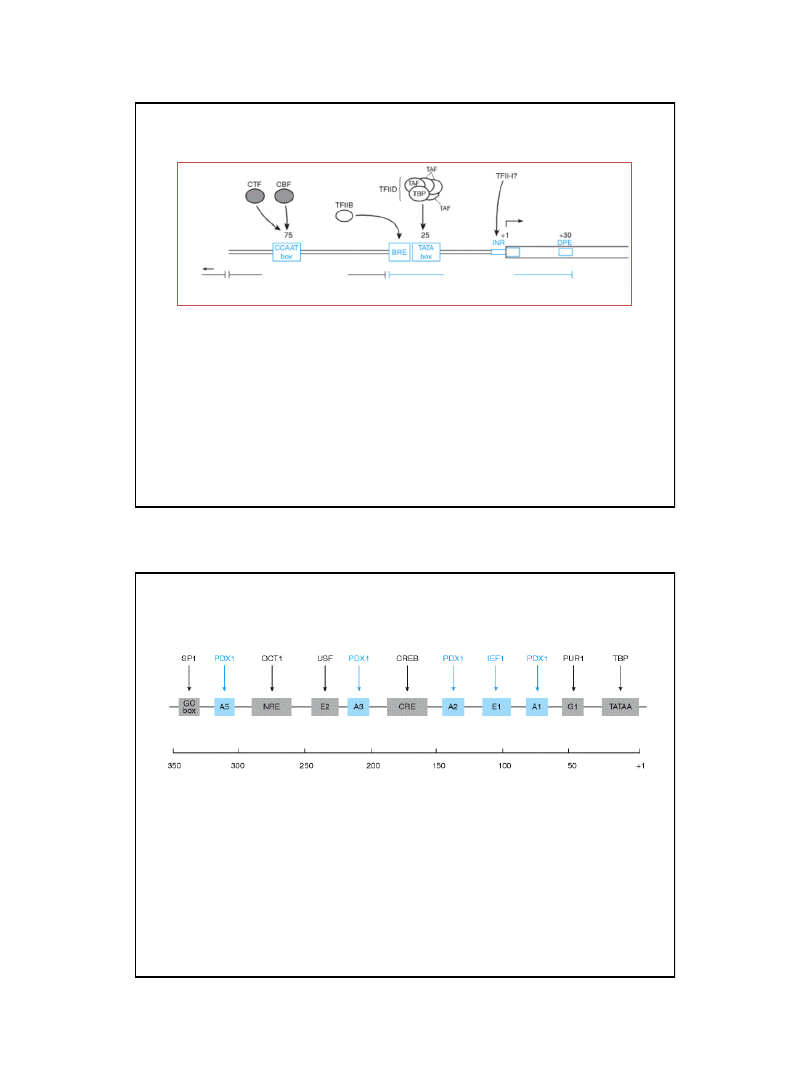
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
rdzeń promotora
region proksymalny
promotora
– 200 do – 50
region
dystalny
promotora
BRE
– TFIIB recogniction element
DPE
– downstream promoter element
CTF
– CCAAT-binding transcription factor
CBF
– CCAAT box-binding factor
TBP
– TATA box-binding protein
TAF
– TBP-associated factors
Przykładowa struktura promotora rozponawanego przez polimerazę II RNA
u wyższych eukariontów
Nie wszystkie zaznaczone elementy występują w promotorach wszystkich genów.
Promotory genów ulegających konstytutywnej ekspresji nie zawierają elementu TATA,
dla ich ekspresji wystarcza obecność elementu GC (nie zaznaczono na rysunku)
Ryc. z: Strachan T., Read A. P., 1999, Human Molecular Genetics 2,
2nd ed., BIOS Scientific Publishers, Ltd., Oxford.
Promotor ludzkiego genu insuliny zawiera różnorodne elementy rozpoznawane
przez
powszechne
i
tkankowo-specyficzne
czynniki transkrypcyjne
30
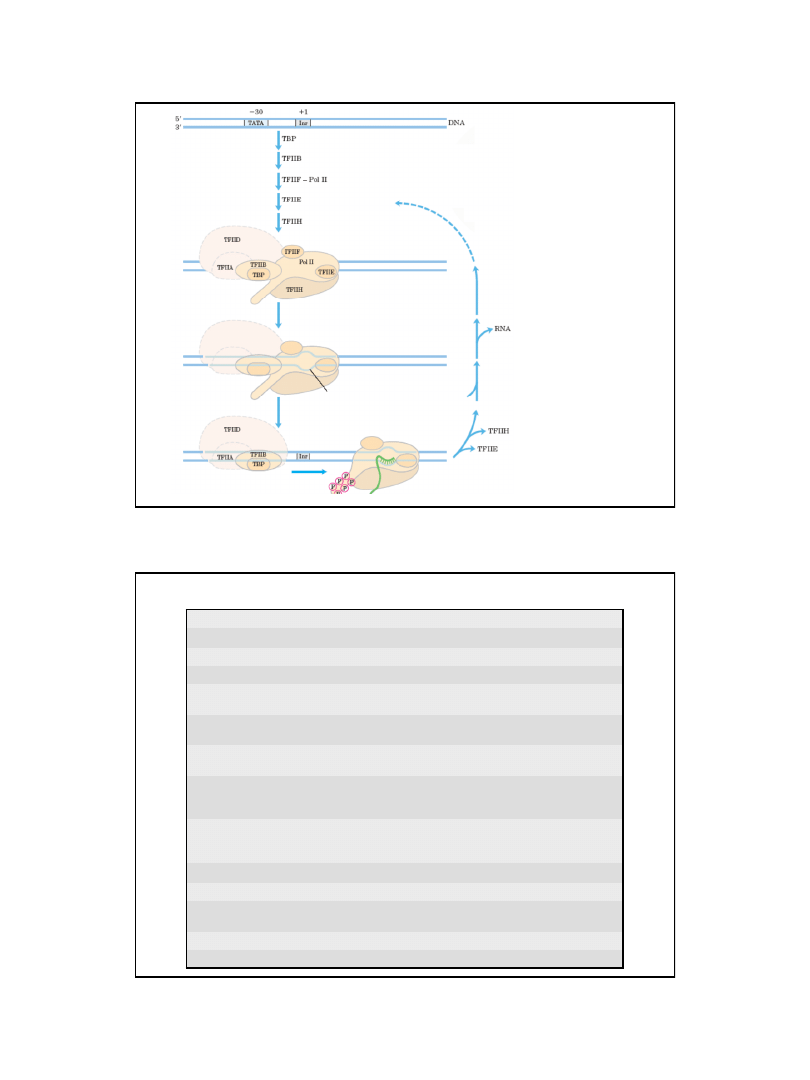
(lub TFIID i/lub TFIIA)
Kompleks zamknięty
Kompleks otwarty
rozdzielenie nici
czynniki
elongacyjne
odłączenie i defosforylacja
polimerazy II RNA
elongacja
terminacja
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of
Biochemistry. 4th Ed. W.H. Freeman & Co.; New York
Białko
l. podj.
M
r
podj.
Funkcja(e)
Inicjacja
Pol. II RNA
12
10 000 - 220 000
Synteza RNA
TBP
1
38 000
Rozpoznanie elementu TATA
TFIIA
3
12-,19-, 35 000
Stabilizuje wiązanie TFIIB i TBP do
promotora
TFIIB
1
35 000
Wiąże się z TBP, powoduje przyłączenie się
kompleksu polimeraza II RNA
– TFIIF
TFIIE
2
34 000, 57 000
Promuje przyłączenie TFIIH, ma aktywność
ATP-azy i helikazy
TFIIF
2
30 000, 74 000
Silnie wiąże się z polimerazą II i z TFIIB,
zapobiega wiązaniu się polimerazy II do
niespecyficznych sekwencji DNA
TFIIH
12
35 000 - 89 000
Rozdziela nici DNA (helikaza); fosforyluje
polimerazę II RNA (w obrębie CTD
największej podjednostki)
Elongacja
zapobiegają zatrzymaniu lub przerwaniu transkrypcji
ELL
1
80 000
p-TEFb
2
43 000, 124 000
fosforyluje polimerazę II RNA (w obrębie
CTD)
SII
1
38 000
SIII (elongina)
3
15-, 18-, 110 000
Czynniki białkowe niezbędne do inicjacji i elongacji transkrypcji kontrolowanej
przez promotory polimerazy II RNA eukariontów
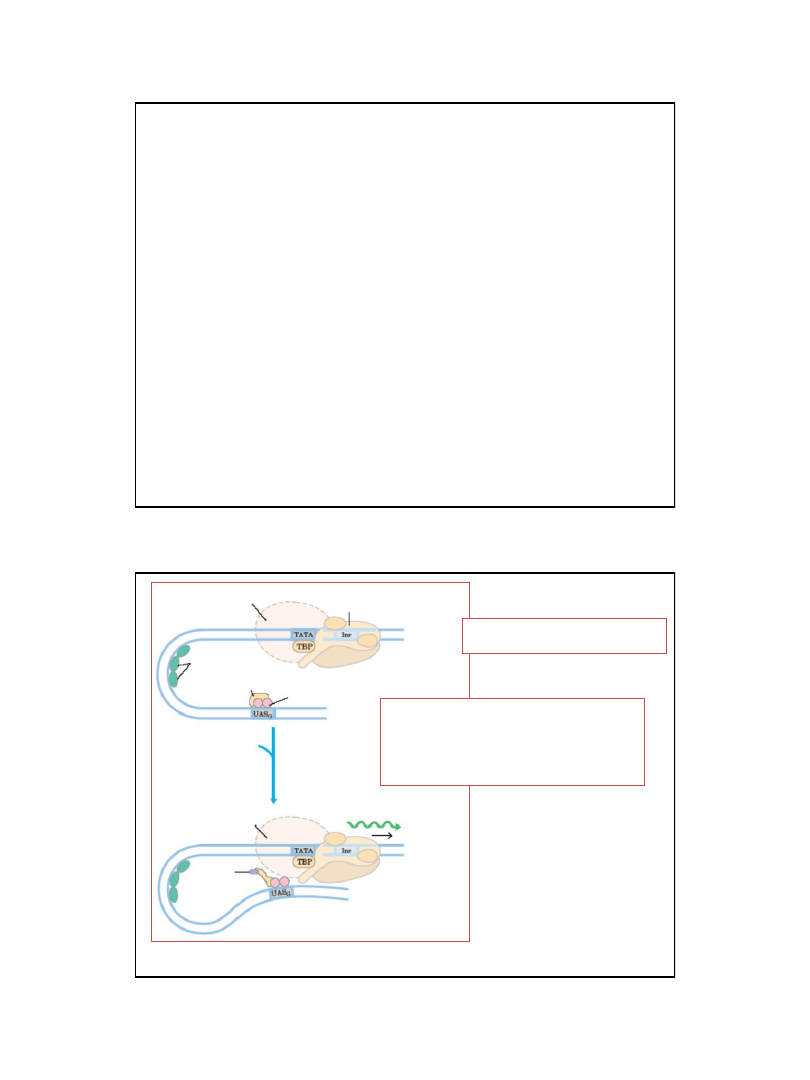
31
Typ elementów działających w układzie cis (sekwencje DNA) zwiększających wykorzystanie
promotorów niektórych genów (wzmacniających transkrypcję). Enhancery mogą funkcjonować
niezależnie od swojej orientacji i położenia względem genu (powyżej lub poniżej), na który
oddziałują.
Z enhancerami oddziałują czynniki transkrypcyjne - niektóre z nich mogą być takie same jak te,
który wiążą się z promotorem.
Uważa się, że działanie wzmacniające enhancerów wynika z bezpośredniego oddziaływania
związanych z nimi czynników transkrypcyjnych z czynnikami związanymi z promotorem.
Interakcja tych dwóch zestawów czynników jest warunkiem wydajnej transkrypcji.
Sekwencja wzmacniająca transkrypcję
(enhancer)
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Kompleks pośredniczący
TFIID lub Mediator
Kompleks polimerazy II RNA
białka HMG
Gal80p
Gal4p
Gal3p
Gal3p
+
galaktoza
Kompleks pośredniczący
Regulacja transkrypcji genów
metabolizmu galaktozy w drożdżach
Gal4p
jest transaktywatorem wiążącym się z elementem
regulatorowym UAS
G
występującym w regionach
promotorowych genów metabolizmu galaktozy
Gal80p
jest inhibitorem transkrypcji uniemożliwiającym
jej aktywację przez Gal4p
Gal3p
jest induktorem, który po związaniu
galaktozy
uzyskuje zdolność wypierania Gal80p z kompleksu z Gal4p
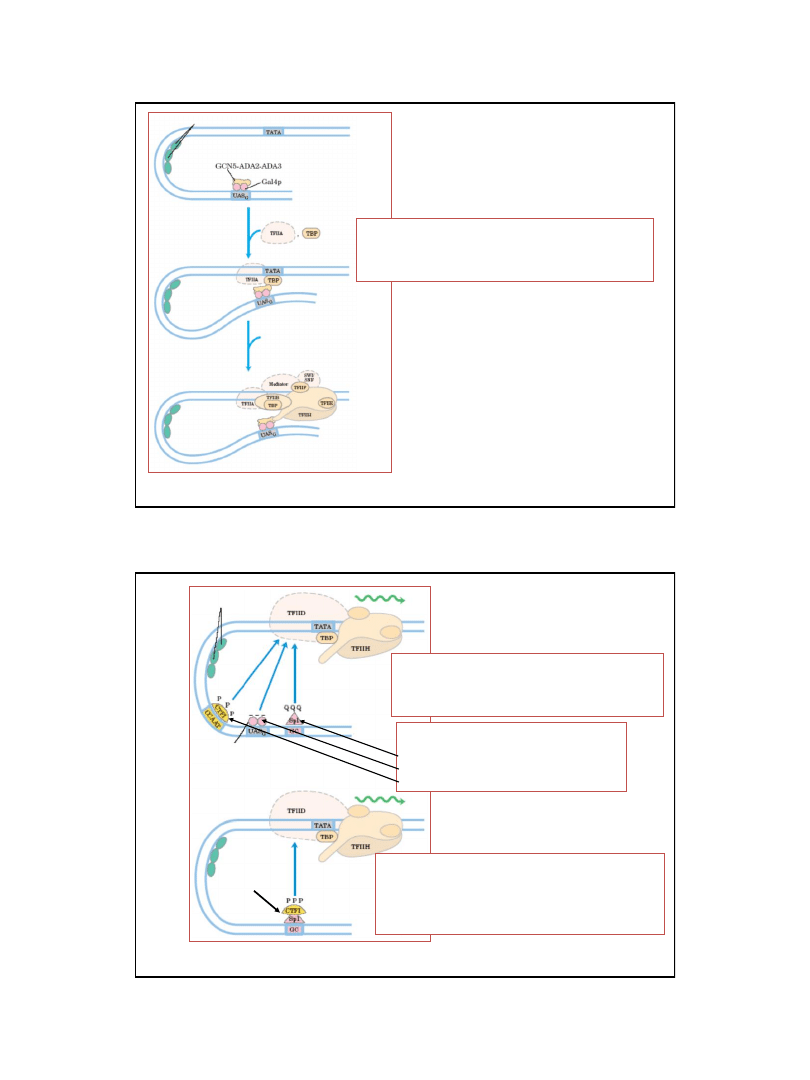
32
Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry. 4th Ed.
W.H. Freeman & Co.; New York
Kompleks polimerazy II RNA
białka HMG
Związanie się transaktywatorów z elementami
regulatorowymi może być warunkiem niezbędnym
dla powstania głównego kompleksu transkrypcyjnego
i przyłączenia polimerazy II RNA
białka HMG
Gal4p
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Typowe transaktywatory wiążące się z DNA
zawierają domenę wiążącą DNA i domenę
aktywacyjną odpowiedzialną za oddziaływanie
z koaktywatorami (takimi jak TFIID lub Mediator)
Domeny aktywacyjne często wykazują
charakterystyczne cechy strukturalne, np. :
Sp1
– domena bogata w glutaminę,
Gal4p
– domena kwasowa
CTF1
– domena aktywacyjna bogata w prolinę,
Transaktywatory mają często budowę modułową
białko chimeryczne zawierające domenę wiążącą DNA
z Sp1 i domenę aktywacyjną z CTF1 może aktywować
transkrypcję o ile w regionie promotora znajduje się element
GC rozpoznawany przez Sp1 (sekwencja konsensusowa
GGGCGG)
białko
chimeryczne
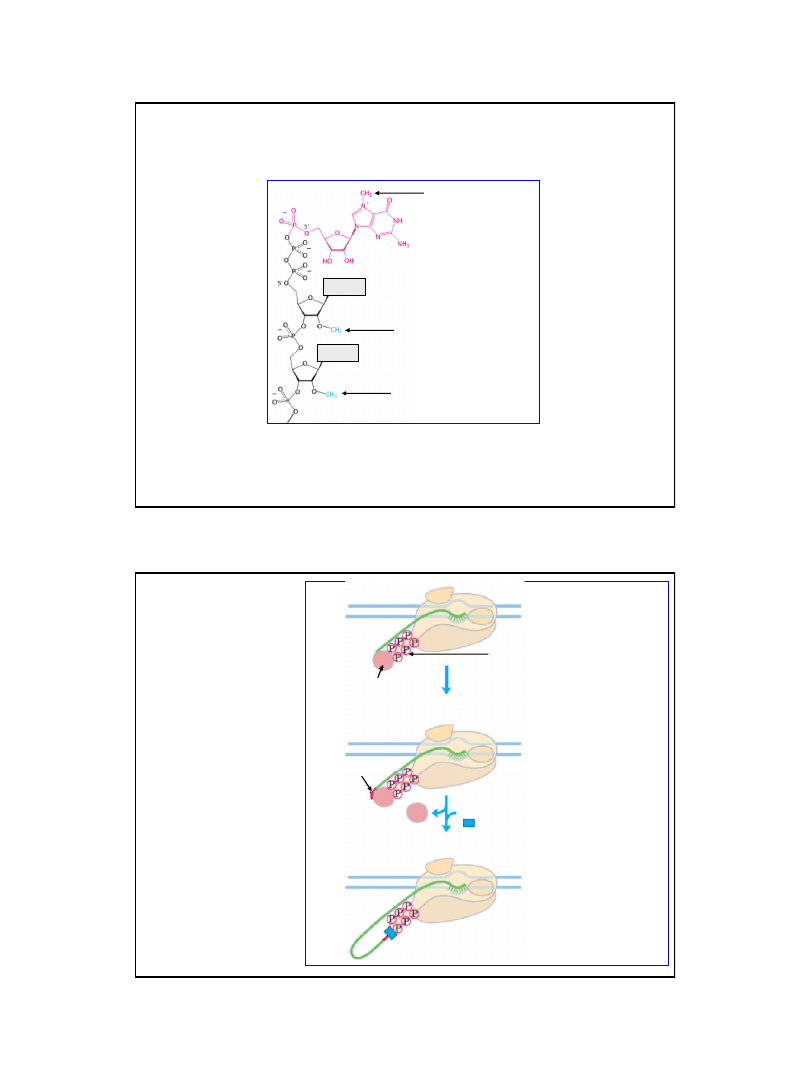
33
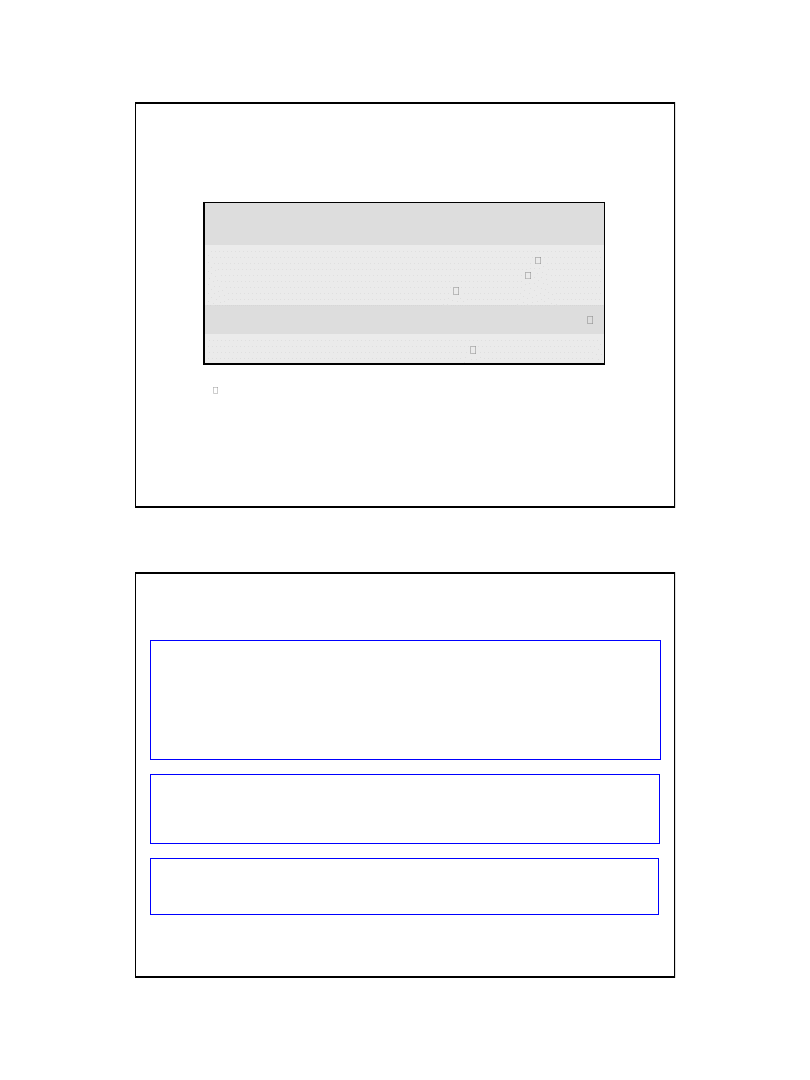
Struktura końca 5’ cząsteczek eukariotycznych mRNA
„Cap” - czapeczka
zasada
zasada
Grupa metylowa
w strukturach cap 0, 1 i 2
Grupa metylowa
w strukturze cap 2
Grupa metylowa
w strukturach cap 1 i 2
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry 5ed.,
W.H. Freeman and Co., New York
Kompleks
syntetyzujący Cap
CBC
Cap
region CTD
(carboxyl-terminal domain)
największej podjednostki
polimerazy II RNA
Cap pozostaje związany z domeną CTD za pośrednictwem
kompleksu CBC (Cap binding complex)
Ryc. z: Nelson D. L., Cox M.M., 2004,
Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Biosynteza cap
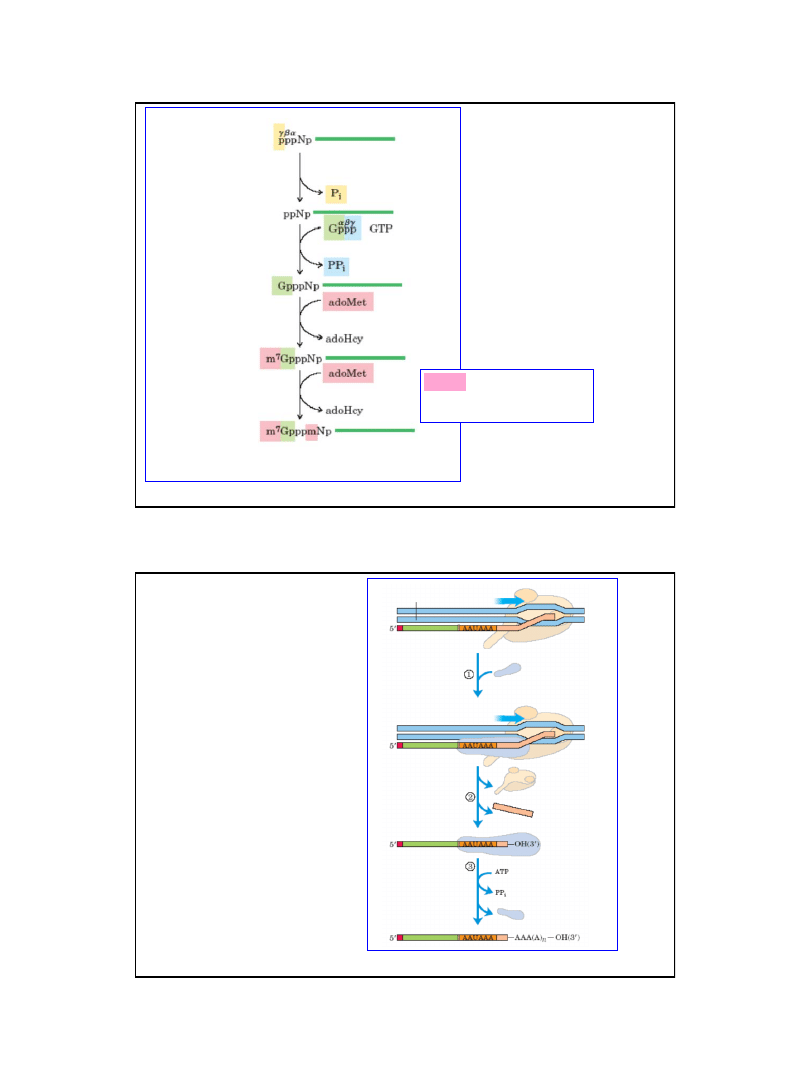
34
„Dojrzały” koniec 5’ pre-mRNA
– z przyłączoną strukturą cap)
fosfohydrolaza
guanylilotransferaza
guanino-7-metylotransferaza
2’-O-metylotransferaza
koniec 5’ pre-mRNA po transkrypcji
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
adoMet
– S-adenozylometionina
(donor grupy metylowej)
adoHcy
– S-adenozylohomocysteina
Mechanizm dołączania struktury „cap”
do końca 5’ pre-mRNA
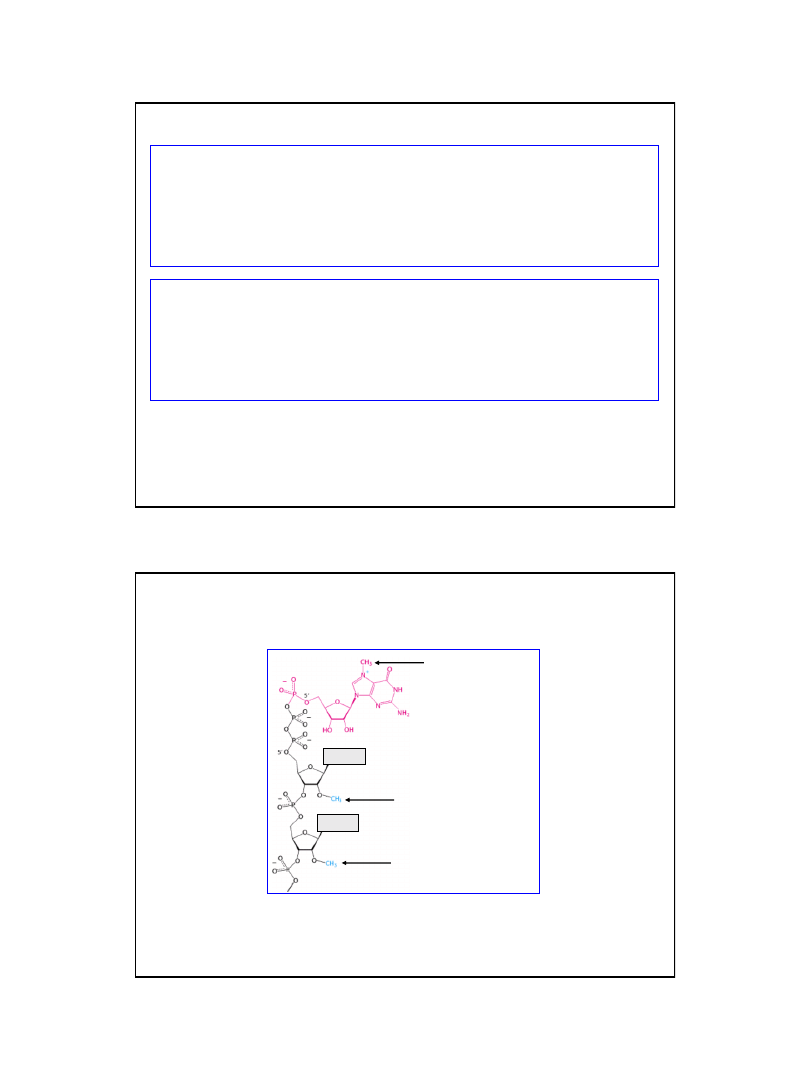
Polimeraza II RNA
Nić matrycowa DNA
Cap
RNA
Kompleks
enzymatyczny
endonukleaza
polimeraza
poliadenylanowa
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Poliadenylacja eukariotycznych
pre-mRNA
35
Sygnały poliadenylacji pre-mRNA w komórkach Eukaryota
1
przedstawiono sekwencje najwyższej zgodności
2
-
miejsce przyłączenia pierwszego nukleotydu A (miejsce poliadenylacji)
3
dla roślin podano nie sekwenckję najwyższej zgodności, lecz najlepiej poznany sygna
poliadenylacji mRNA wirusa mozaiki kalafiora (CaMV)
Organizm
Sekwencja wyznaczająca miejsce
poliadenylacji
1
Drożdże
(Saccharomyces cerevisiae)
UAG-UAUGU-UUU-10N-
2
lub UACAUA-100N-
lub -50N-UAUAUA
Rośliny
3
UUAGUAUGUAUUUGUA-13N-AAUAAA-14N-
Ssaki
AUAAA-10-30N- -10N-(GU)n lub (U)n
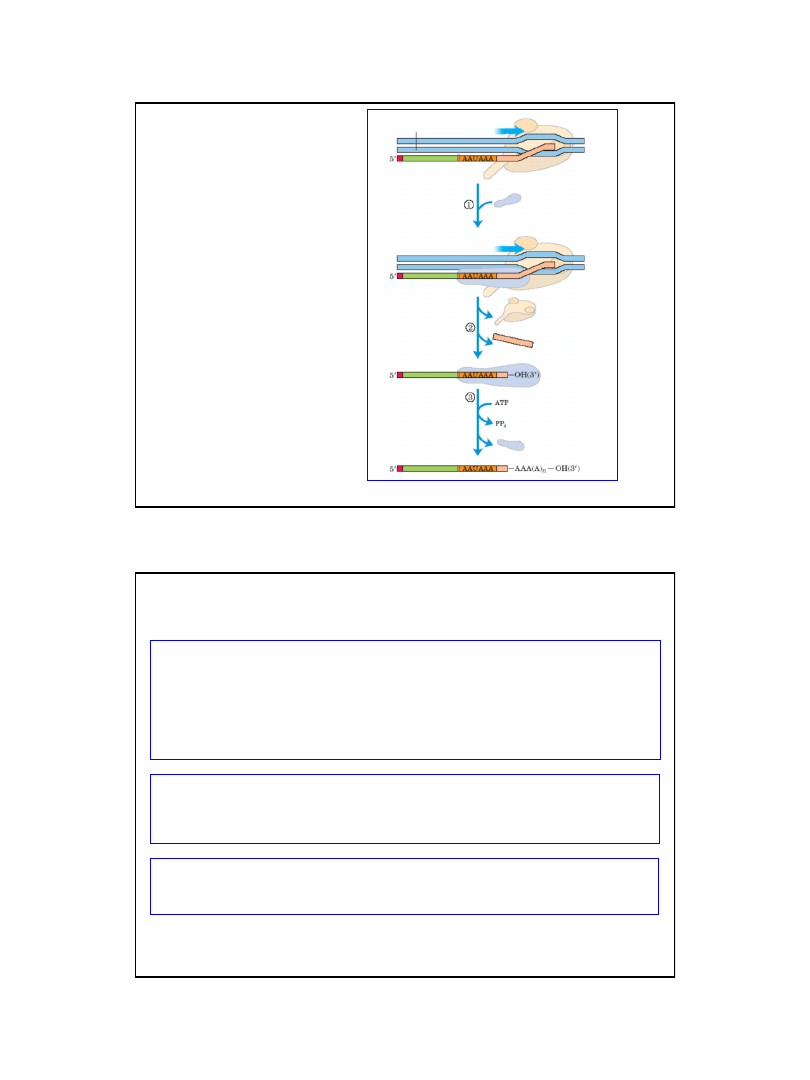
Nie ma uniwersalnego mechanizmu wycinania intronów
z transkryptów genów nieciągłych
Wycinanie intronów z jądrowego mRNA wyższych eukariotów jest realizowane przez system
rozpoznający krótkie sekwencje konsensusowe w miejscach połączeń ekson - intron, oraz tzw. miejsce
rozgałęzienia w obrębie intronu.
w reakcji uczestniczą kompleksy snRNA z białkami snRNP, które łącząc się z podlegającą obróbce częścią
cząsteczki mRNA, tworzą spliceosom
W obrębie spliceosomu następują reakcje transestryfikacji prowadzące do połączenia końców 3' i 5'
sąsiednich eksonów, zaś wycięty intron jest uwalniany w postaci pętli (zwanej czasem lassem - ang. lariat).
Wycinanie niektórych intronów jest autonomiczną właściwościa samego RNA.
Znamy dwa typy intronów zdolne do autokatalicznego usuwania sie z prekursorów RNA: Introny grupy I,
oraz Introny grupy II.
Autokatalityczne właściwości RNA moga są obserwowane również podczas cięcia RNA wiroidów, oraz w
przypadku rybonukleazy P
W przeciwieństwie do powyższych mechanizmów wycinania intronów, które zachodzą na drodze reakcji
transestryfikacji
, wycinanie intronów z prekursorów tRNA (np. w drożdżach, ok. 40 z ok. 400 genów tRNA
zawiera introny o długości 14-46 nt.) przebiega jako następujące po sobie reakcje cięcia i ligacji
fragmentów RNA.
36
Introny grupy I
-
wycinane przez dwukrotną reakcję transestryfikacji
-
w pierwszej reakcji uczestniczy guanozyna (pochodzenia zewnętrznego) jako grupa atakująca
(nukleofilowa)
-
w niektórych wypadkach wymagają obecności białek wspomagających proces (maturazy)
-
występują w genach mRNA, rRNA i tRNA genomu mitochondrialnego, chloroplastowego i jądrowego
różnych eukariontów, lecz nie stwierdzono ich obecności w genach kręgowców
-
występowanie stosunkowo nieregularne: niektóre gatunki Tetrahymena mają intron grupy I w genach 26S
rRNA, w genach innych gatunków tego Rodzaju intronu nie ma.
Introny grupy II
-
występują dwa typy intronów tej grupy (IIA i IIB) różniące się niektórymi nukleotydami sekwencji
konsensusowych
-
wycinane przez dwukrotną reakcję transestryfikacji
-
grupą atakującą w pierwszej reakcji jest grupa 2' adenozyny w domenie VI
-
samowycinanie jest możliwe w warunkach niefizjologicznych (in vitro). In vivo w procesie uczestniczą
białka, jak maturaza kodowane przez ORF w obrębie intronu, lub białka kodowane przez genom jądrowy
-
wszystkie znane introny grupy II napotkano w genomie organellowym eukariontów, stanowią one
większość intronów w roślinnym mitochondrialnym DNA
Struktura końca 5’ cząsteczek eukariotycznych mRNA
„Cap” - czapeczka
zasada
zasada
Grupa metylowa
w strukturach cap 0, 1 i 2
Grupa metylowa
w strukturze cap 2
Grupa metylowa
w strukturach cap 1 i 2
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry 5ed.,
W.H. Freeman and Co., New York
37
Polimeraza II RNA
Nić matrycowa DNA
Cap
RNA
Kompleks
enzymatyczny
endonukleaza
polimeraza
poliadenylanowa
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Poliadenylacja eukariotycznych
pre-mRNA
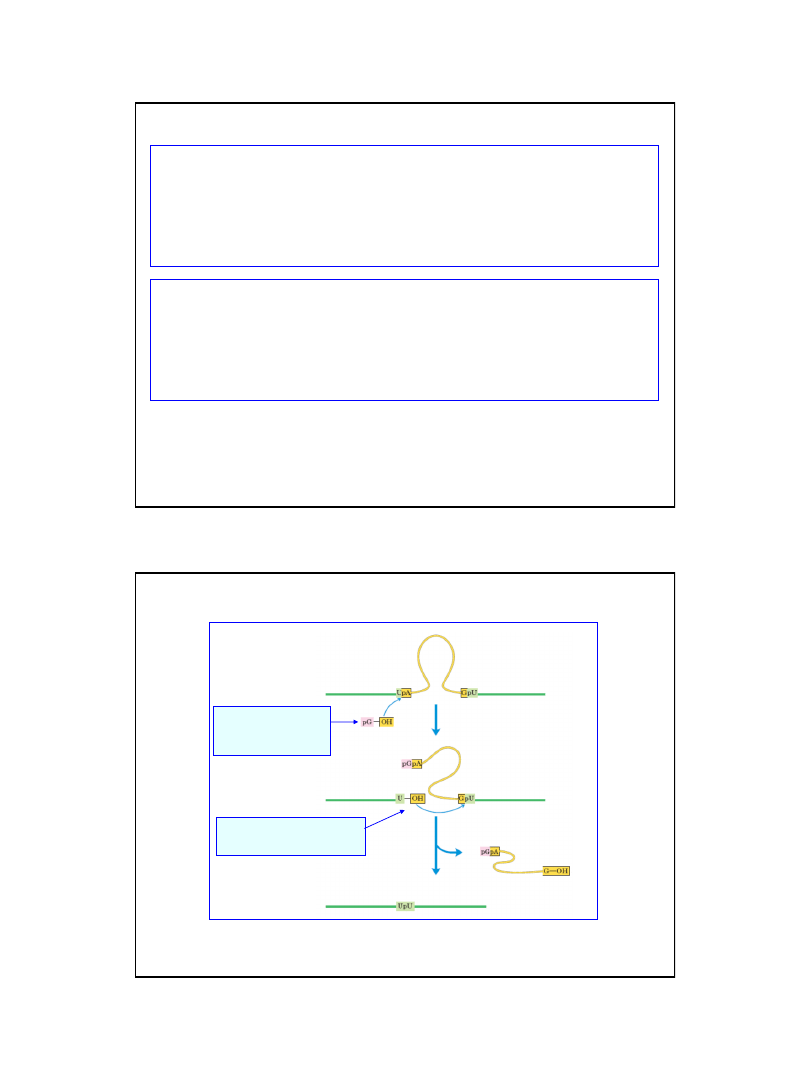
Nie ma uniwersalnego mechanizmu wycinania intronów
z transkryptów genów nieciągłych
Wycinanie intronów z jądrowego mRNA wyższych eukariotów jest realizowane przez system
rozpoznający krótkie sekwencje konsensusowe w miejscach połączeń ekson - intron, oraz tzw. miejsce
rozgałęzienia w obrębie intronu.
w reakcji uczestniczą kompleksy snRNA z białkami snRNP, które łącząc się z podlegającą obróbce częścią
cząsteczki mRNA, tworzą spliceosom
W obrębie spliceosomu następują reakcje transestryfikacji prowadzące do połączenia końców 3' i 5'
sąsiednich eksonów, zaś wycięty intron jest uwalniany w postaci pętli (zwanej czasem lassem - ang. lariat).
Wycinanie niektórych intronów jest autonomiczną właściwościa samego RNA.
Znamy dwa typy intronów zdolne do autokatalicznego usuwania sie z prekursorów RNA: Introny grupy I,
oraz Introny grupy II.
Autokatalityczne właściwości RNA moga są obserwowane również podczas cięcia RNA wiroidów, oraz w
przypadku rybonukleazy P
W przeciwieństwie do powyższych mechanizmów wycinania intronów, które zachodzą na drodze reakcji
transestryfikacji
, wycinanie intronów z prekursorów tRNA (np. w drożdżach, ok. 40 z ok. 400 genów tRNA
zawiera introny o długości 14-46 nt.) przebiega jako następujące po sobie reakcje cięcia i ligacji
fragmentów RNA.
38
Introny grupy I
-
wycinane przez dwukrotną reakcję transestryfikacji
-
w pierwszej reakcji uczestniczy guanozyna (pochodzenia zewnętrznego) jako grupa atakująca
(nukleofilowa)
-
w niektórych wypadkach wymagają obecności białek wspomagających proces (maturazy)
-
występują w genach mRNA, rRNA i tRNA genomu mitochondrialnego, chloroplastowego i jądrowego
różnych eukariontów, lecz nie stwierdzono ich obecności w genach kręgowców
-
występowanie stosunkowo nieregularne: niektóre gatunki Tetrahymena mają intron grupy I w genach 26S
rRNA, w genach innych gatunków tego Rodzaju intronu nie ma.
Introny grupy II
-
występują dwa typy intronów tej grupy (IIA i IIB) różniące się niektórymi nukleotydami sekwencji
konsensusowych
-
wycinane przez dwukrotną reakcję transestryfikacji
-
grupą atakującą w pierwszej reakcji jest grupa 2' adenozyny w domenie VI
-
samowycinanie jest możliwe w warunkach niefizjologicznych (in vitro). In vivo w procesie uczestniczą
białka, jak maturaza kodowane przez ORF w obrębie intronu, lub białka kodowane przez genom jądrowy
-
wszystkie znane introny grupy II napotkano w genomie organellowym eukariontów, stanowią one
większość intronów w roślinnym mitochondrialnym DNA
Mechanizm wycinania intronów grupy I
Ekson 3’
Intron
Transkrypt pierwotny
RNA po usunięciu intronu
Intermediat
Ekson 5’
5’
3’
5’
3’
5’
3’
5’
3’
Grupa 3’ OH guanozyny
działa jako nukleofil
atakując miejsce
splicingowe 5’
Grupa 3’ OH intronu 5’ staje się
nukleofilem odpowiedzialnym
za drugą transestryfikację
Wycięty intron
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
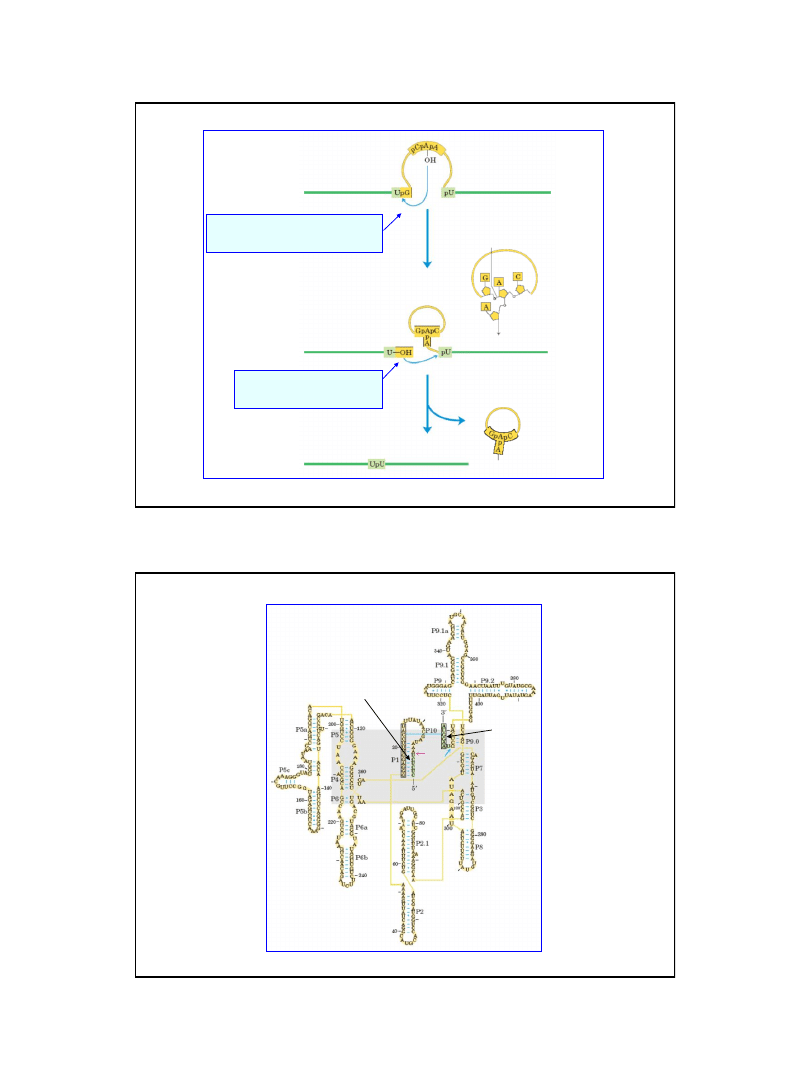
39
Intron
Transkrypt pierwotny
RNA po usunięciu intronu
Intermediat
Wycięty intron
5’
3’
5’
3’
5’
3’
OH (3’)
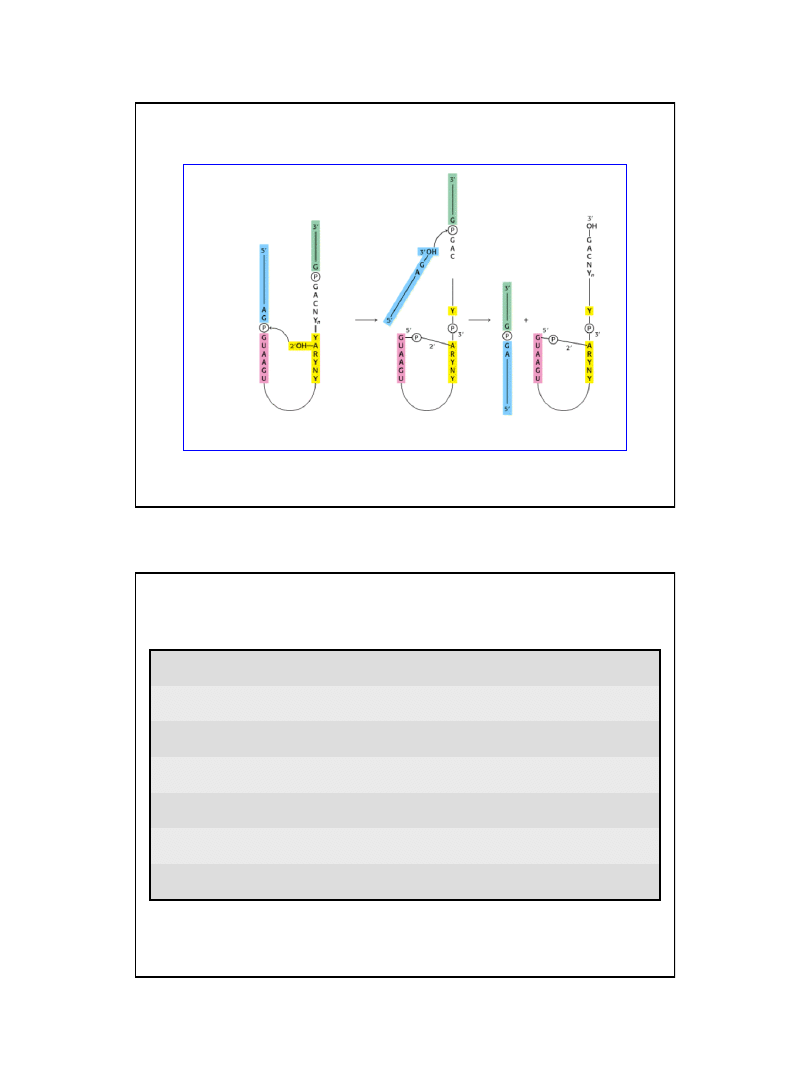
Grupa 3’ OH intronu 5’ staje się
nukleofilem odpowiedzialnym
za drugą transestryfikację
Grupa 2’ specyficznej adenozyny
intronu działa, jako nukleofil atakujący
miejsce splicingowe 5’
wiązanie fosfodiestrowe 2’, 5’
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Mechanizm wycinania intronów grupy II
Struktura drugorzędowa intronu grupy I
Ekson 3’
Ekson 5’
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
(z genu rRNA Tetrahymena)
40
cząsteczki snRNA
w spliceosomie
Intron grupy II
pre-mRNA
Dwukrotna transestryfikacja prowadząca do wycięcia intronu
wynika z autokatalitycznych właściwości RNA.
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E.,
1999, Molecular Cell Biology, W. H. Freeman & Co.; New York
Czynnikiem odpowiadającym za przestrzenne ułożenie reagujących ze sobą
fragmentów RNA jest w przypadku intronów – struktura trzeciorzędowa intronu,
zaś w przypadku intronów pre-mRNA – struktura przestrzenna kompleksu tworzonego
przez spliceosom, z którą odpowiadają sekwencje snRNA tworzące poszczególne
cząsteczki snRNP (U1, U2, U4, U5 i U6).
Elementy typowego intronu eukariotycznych, pre-
mRNA pełniące
funkcje w procesie wycinania intronów (splicing)
Miejsce
rozgałęzienia
Ekson 5’
Ekson 3’
Miejsc połączenia
intron-
ekson 5’
Miejsc połączenia
intron-
ekson 3’
41
Wycinanie intronów i łączenie eksonów (splicing) zachodzi
na drodze dwóch reakcji transestryfikacji
Intron uwolniony w
postaci pętli (lariat)
Ekson 1
Ekson 2
Miejsce
rozgałęzienia
Miejsce
splicingowe 5’
Miejsce
splicingowe 3’
Prekursor
Intermediat
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002), Biochemistry 5ed., W.H. Freeman and Co., New York
Sekwencje nukleotydowe niektórych miejsc połączeń intron - ekson w różnych genach
Intron
Ekson
Intron
Ekson
drugi genu owoalbuminy
UAAG
GUGAGC***************UUACAG
GUUG
trzeci genu owoalbuminy
UCAG
GUACAG***************AUUCAG
UCUG
pierwszy genu β-globiny
GCAG
GUUGGU***************CCUUAG
GCUG
drugi genu β-globin
CAGG
GUGAGU***************CCACAG
UCUC
pierwszy genu immunoglobuliny λ1
UCAG
GUCAGC***************UUGCAG
GGGC
genu wczesnego antygenuT SV 40
UAAG
GUAAAU***************UUUUAG
AUUC
42
Splicing pre-
mRNA zachodzący
z udziałem snRNP
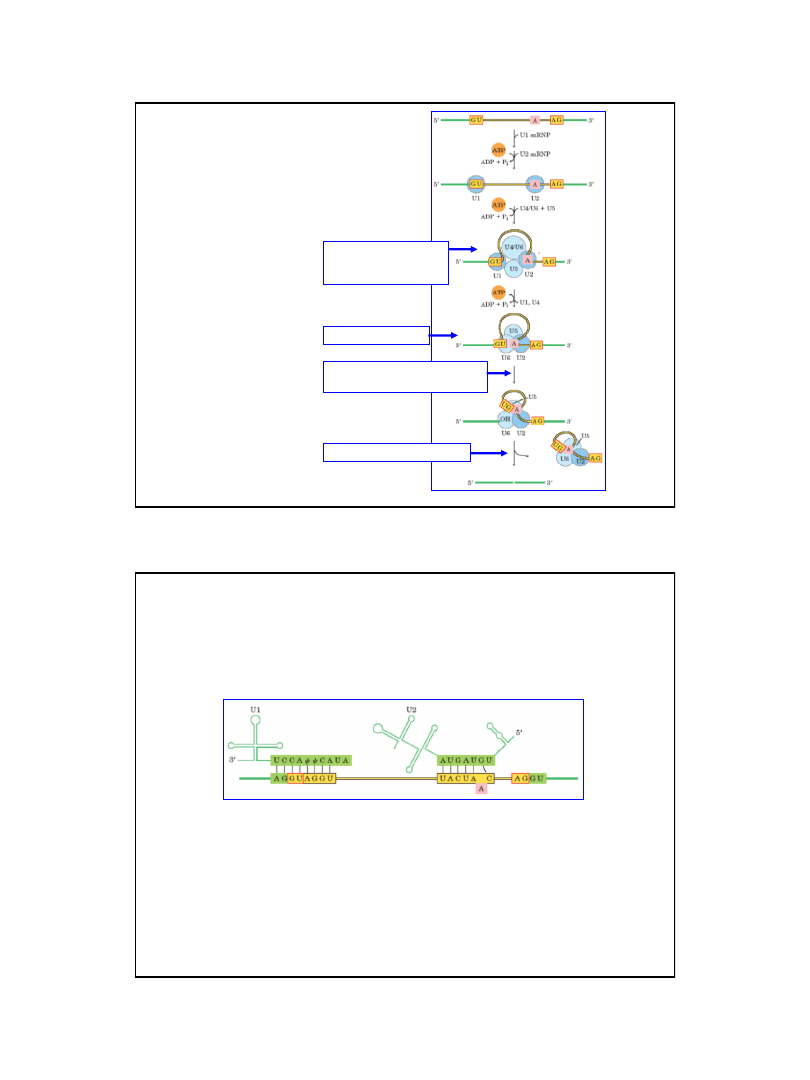
Powstawanie i funkcja spliceosomu
Nieaktywny spliceosom
Aktywność U6
jest blokowana przez U4
Aktywny spliceosom
zachodzi reakcja transestryfikacji
powstawanie struktury lariat
Uwolnienie wyciętego intronu
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger
Principles of Biochemistry 4th Ed. W.H. Freeman & Co.; New York
Ekson 5’
Ekson 3’
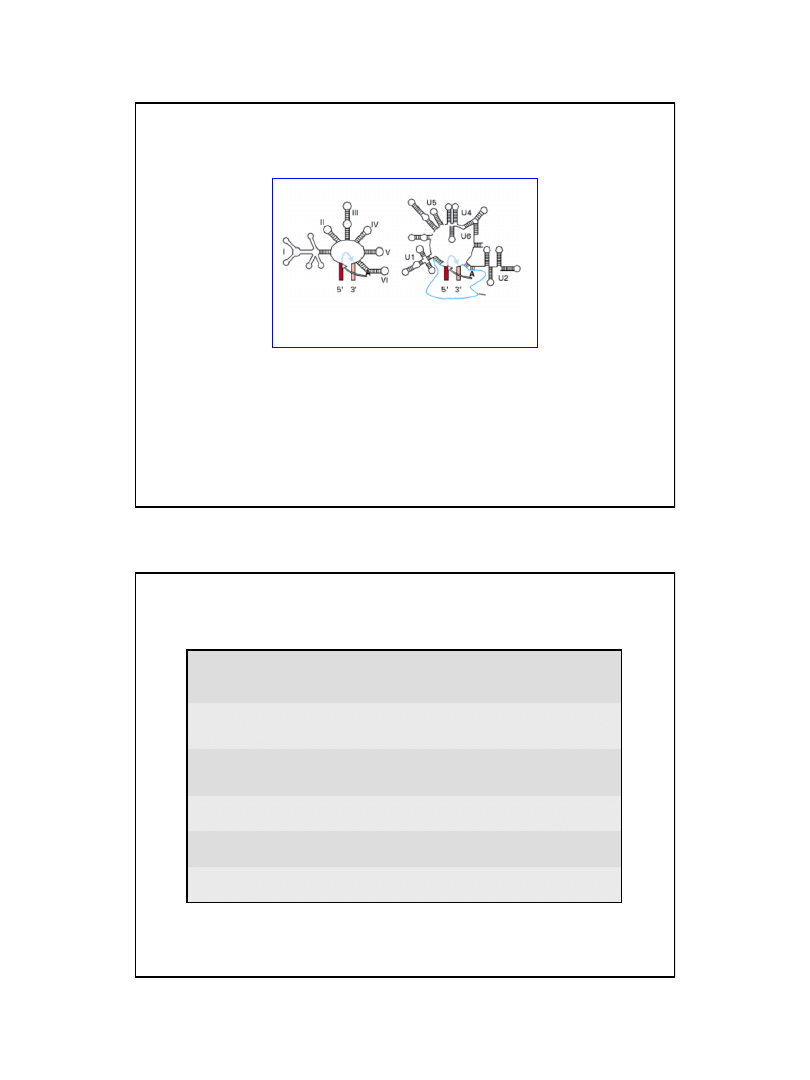
Za rozpoznanie połączeń intron exon oraz miejsca rozgałęzienia odpowiadają snRNA
stanowiące zrąb snRNA
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
43
cząsteczki snRNA
w spliceosomie
Intron grupy II
pre-mRNA
Dwukrotna transestryfikacja prowadząca do wycięcia intronu
wynika z autokatalitycznych właściwości RNA.
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E.,
1999, Molecular Cell Biology, W. H. Freeman & Co.; New York
Czynnikiem odpowiadającym za przestrzenne ułożenie reagujących ze sobą
fragmentów RNA jest w przypadku intronów – struktura trzeciorzędowa intronu,
zaś w przypadku intronów pre-mRNA – struktura przestrzenna kompleksu tworzonego
przez spliceosom, z którą odpowiadają sekwencje snRNA tworzące poszczególne
cząsteczki snRNP (U1, U2, U4, U5 i U6).
Struktury niskocząsteczkowych jądrowych rybonukleoproteidów
(snRNP)
uczestniczących w splicingu pre-mRNA
snRNP
wielkość snRNA
[nt]
Funkcja w splicingu
U1
165
wiąże się do połączenia intron-ekson (najpierw 5',
następnie 3')
U2
185
wiąże się z miejscem rozgałęzienia, tworzy część
centrum katalitycznego
U5
116
wiąże się z połączeniem intron-ekson po stronie 5'
U4
145
maskuje aktywność katalityczną U6
U6
106
katalizuje splicing
44
Porównanie mechanizmów wycinania różnych intronów
przebiegającego poprzez dwie reakcje transestryfikacji
Introny pre-mRNA wycinane
z udziałem spliceosomu
Introny ulegające autokatalicznemu wycinaniu
grupa I
grupa II
Ryc. z: Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. E.,
1999, Molecular Cell Biology, W. H. Freeman & Co.; New York
KOD GENETYCZNY
Zasada zapisu sekwencji aminokwasowych białek w genomach organizmów żywych, w postaci
następujących po sobie trójek zasad sekwencji DNA (i w konsekwencji także mRNA).
Trójki zasad (triplety) zwane są kodonami.
UUU
Phe
UCU
Ser
UAU
Tyr
UGU
Cys
UUC
UCC
UAC
UGC
UUA
Leu
UCA
UAA
STOP
UGA
STOP
UUG
UCG
UAG
UGG
Trp
CUU
CCU
Pro
CAU
His
CGU
Arg
CUC
CCC
CAC
CGC
CUA
CCA
CAA
Gln
CGA
CUG
CCG
CAG
CGG
AUU
Ile
ACU
Thr
AAU
Asn
AGU
Ser
AUC
ACC
AAC
AGC
AUA
ACA
AAA
Lys
AGA
Arg
AUG
Met
ACG
AAG
AGG
GUU
Val
GCU
Ala
GAU
Asp
GGU
Gly
GUC
GCC
GAC
GGC
GUA
GCA
GAA
Glu
GGA
GUG
GCG
GAG
GGG
45
KOD GENETYCZNY
nie jest w pełni uniwersalny
strzałkami zaznaczono kodony, które w mitochondriach człowieka
mają odmienny sens (podany w nawiasie)
UUU
Phe
UCU
Ser
UAU
Tyr
UGU
Cys
UUC
UCC
UAC
UGC
UUA
Leu
UCA
UAA
STOP
UGA
STOP
(Trp)
UUG
UCG
UAG
UGG
Trp
CUU
CCU
Pro
CAU
His
CGU
Arg
CUC
CCC
CAC
CGC
CUA
CCA
CAA
Gln
CGA
CUG
CCG
CAG
CGG
AUU
Ile
(Met)
ACU
Thr
AAU
Asn
AGU
Ser
AUC
ACC
AAC
AGC
AUA
ACA
AAA
Lys
AGA
Arg
(STOP)
AUG
Met
ACG
AAG
AGG
GUU
Val
GCU
Ala
GAU
Asp
GGU
Gly
GUC
GCC
GAC
GGC
GUA
GCA
GAA
Glu
GGA
GUG
GCG
GAG
GGG
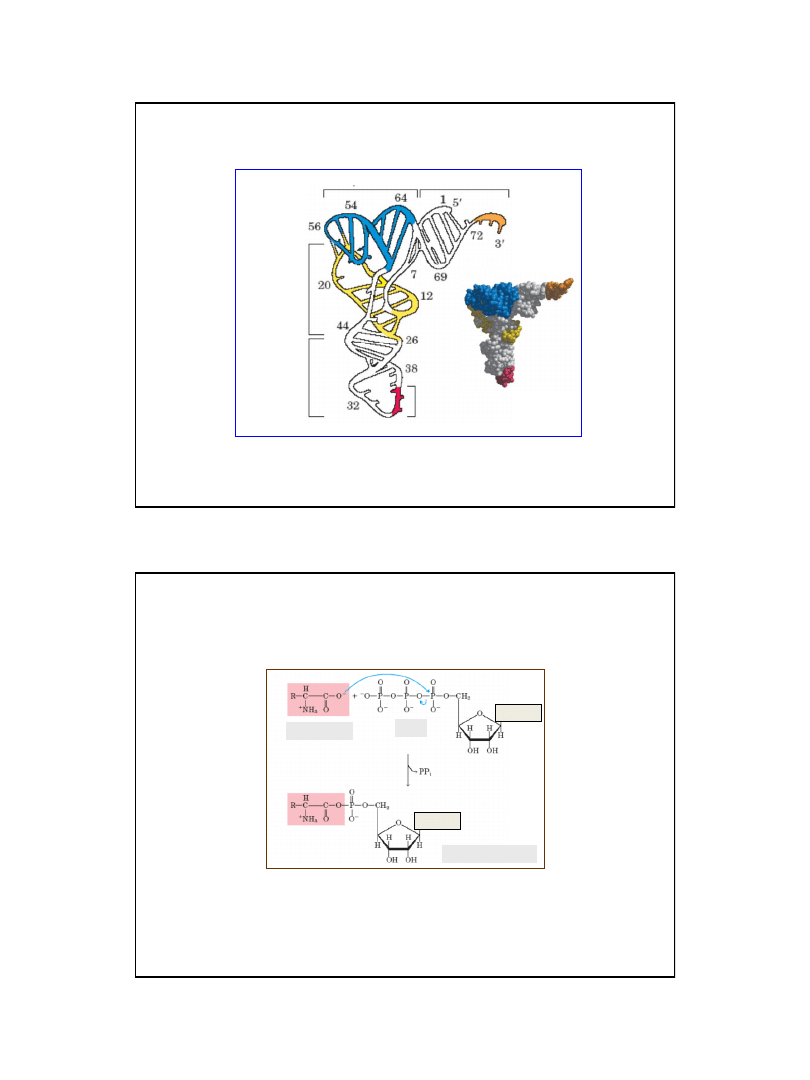
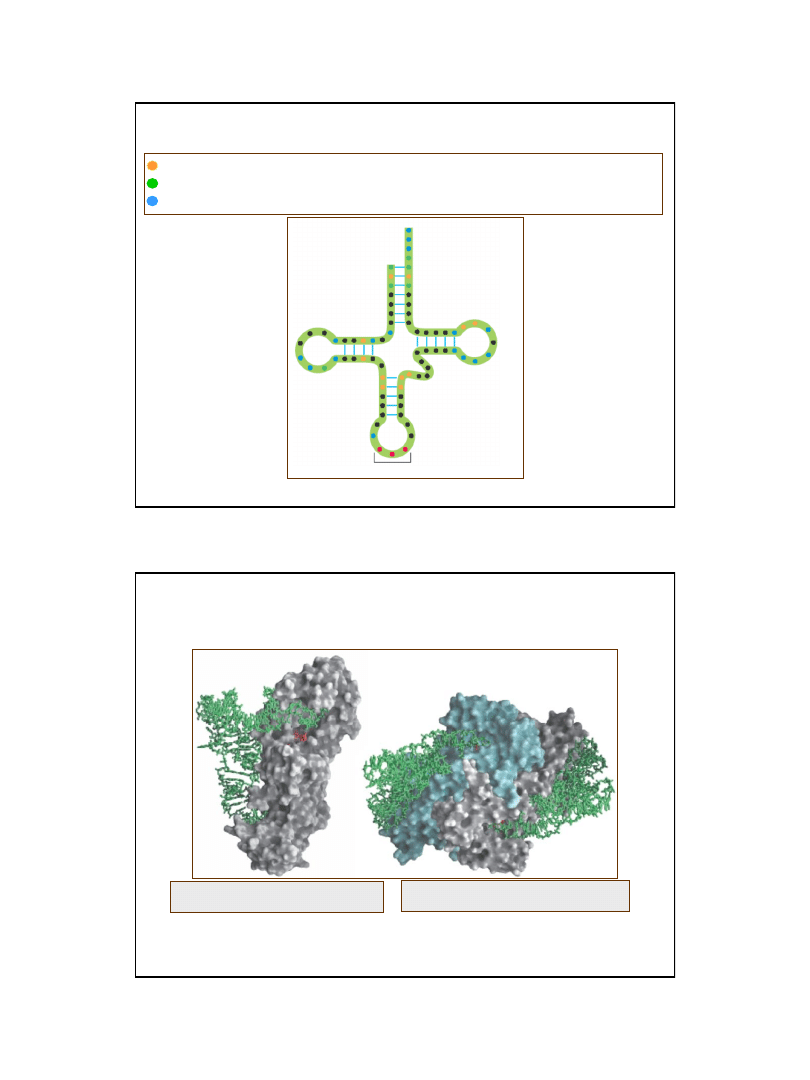
ramię dodatkowe
ramię
akceptorowe
pętla TψC
pętla antykodonowa
antykodon
pętla DHU
Struktura drugorzędowa tRNA
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Cechy wspólne wszystkich poznanych tRNA
-
Długość 73-93 rybonukleotydy (ok. 25 kDa),
-
Zawierają nietypowe (modyfikowane) zasady - zwykle 7-15 na cząsteczkę,
-
Koniec 5' zawsze jest fosforylowany, nukleotydem końcowym jest zwykle pG,
- Koniec 3' zawsze stanowi sekwencja 5'CCA3',
-
mniej więcej połowa nukleotydów jest sparowana i tworzy struktury dwuniciowe,
-
Pętla antykodonowa zawiera sekwencję:
5 '- pirymidyna - pirymidyna - X - Y - Z - zmod. puryna - zasada zmienna - 3'
46
pętla DHU
pętla TψC
pętla
antykodonowa
ramię akceptorowe
antykodon
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Struktura trzeciporzędowa tRNA
Phe
z drożdży
adenina
adenina
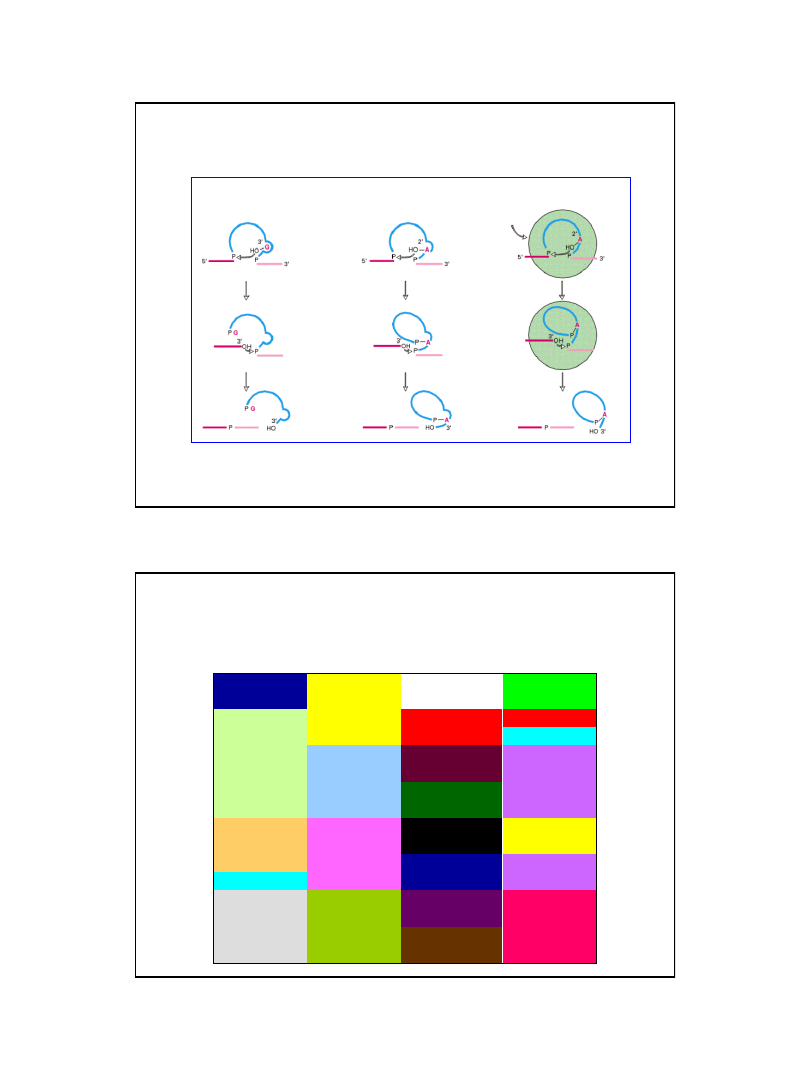
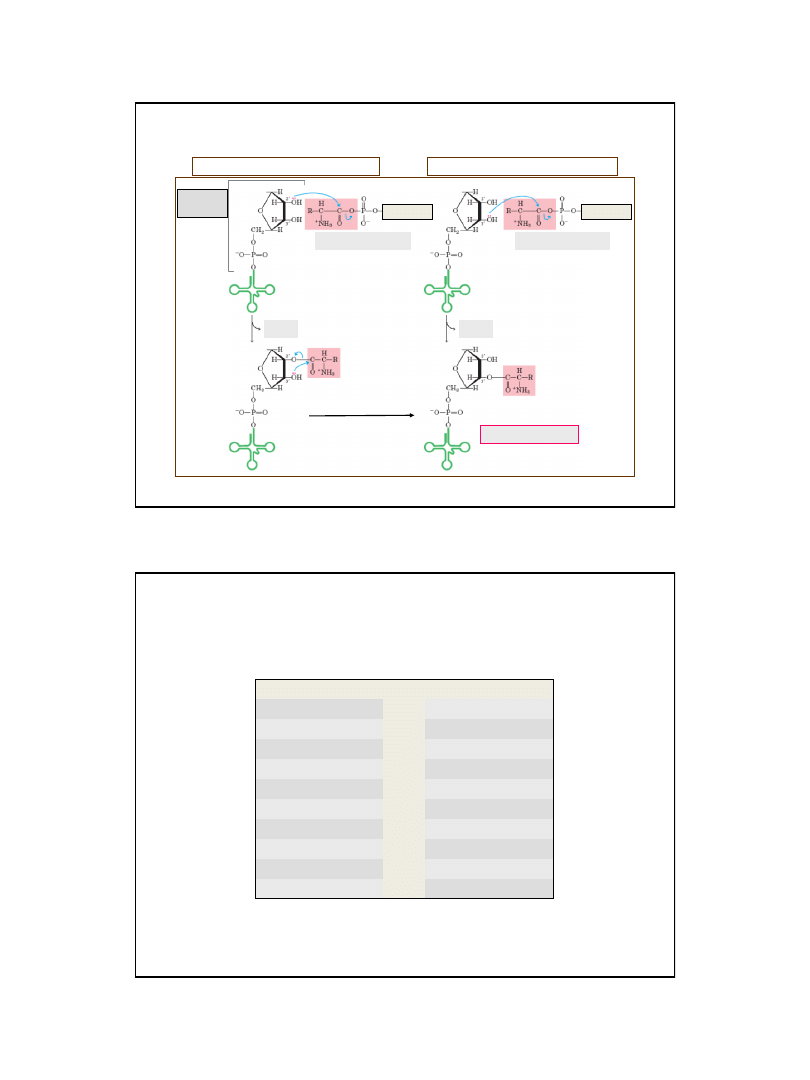
Reakcja aminoacylacji tRNA
etap I
– aktywacja aminokwasu
aminokwas
ATP
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
aminoacylo-AMP
47
Reakcja aminoacylacji tRNA
etap II
– przeniesienie reszty aminoacylowej na 2’ lub 3’ -OH tRNA
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
adenina
aminoacylo-AMP
adenozyna
adenozyna
adenina
AMP
koniec 3’
tRNA
aminoacylo-tRNA
AMP
tRNA
tRNA
transestryfikacja
syntetazy aminoacylo- tRNA klasy I
syntetazy aminoacylo- tRNA klasy II
aminoacylo-AMP
Klasa I
Klasa II
Arg
α
Ala
α
4
Cys
α
Asn
α
2
Gln
α
Asp
α
2
Glu
α
Gly
α
2
β
2
Ile
α
His
α
2
Leu
α
Lys
α
2
Met
α
Phe
α
2
β
2
Trp
α
2
Ser
α
2
Tyr
α
2
Pro
α
2
Val
α
Thr
α
2
Klasyfikacja i struktura podjednostkowa
syntetaz aminoacylo-tRNA z Escherichia coli
48
ramię dodatkowe
ramię akceptorowe
(aminoacylowe)
ramię TψC
ramię DHU
ramię
antykodonowe
antykodon
pozycje nukleotydów istotnych dla specyficzności rozpoznania tRNA przez wszystkie syntetazy aminoacylo-tRNA
pozycje nukleotydów istotnych dla specyficzności rozpoznania tRNA przez jedną syntetazę aminoacylo-tRNA
pozycje nukleotydów istotnych dla specyficzności rozpoznania tRNA przez kilka syntetaz aminoacylo-tRNA
Elementy struktury tRNA ważne dla specyficzności oddziaływania
z syntetazami aminoacylo-tRNA
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Syntetazy aminoacylo-tRNA w kompleksie z rozpoznawanymi
przez siebie specyficznymi cząsteczkami tRNA
Syntetaza glutaminowa (Gln) z E. coli
typowa monomeryczna syntetaza
klasy I
Syntetaza asparaginianowa (Asp) z drożdży
typowa dimeryczna syntetaza
klasy II

49
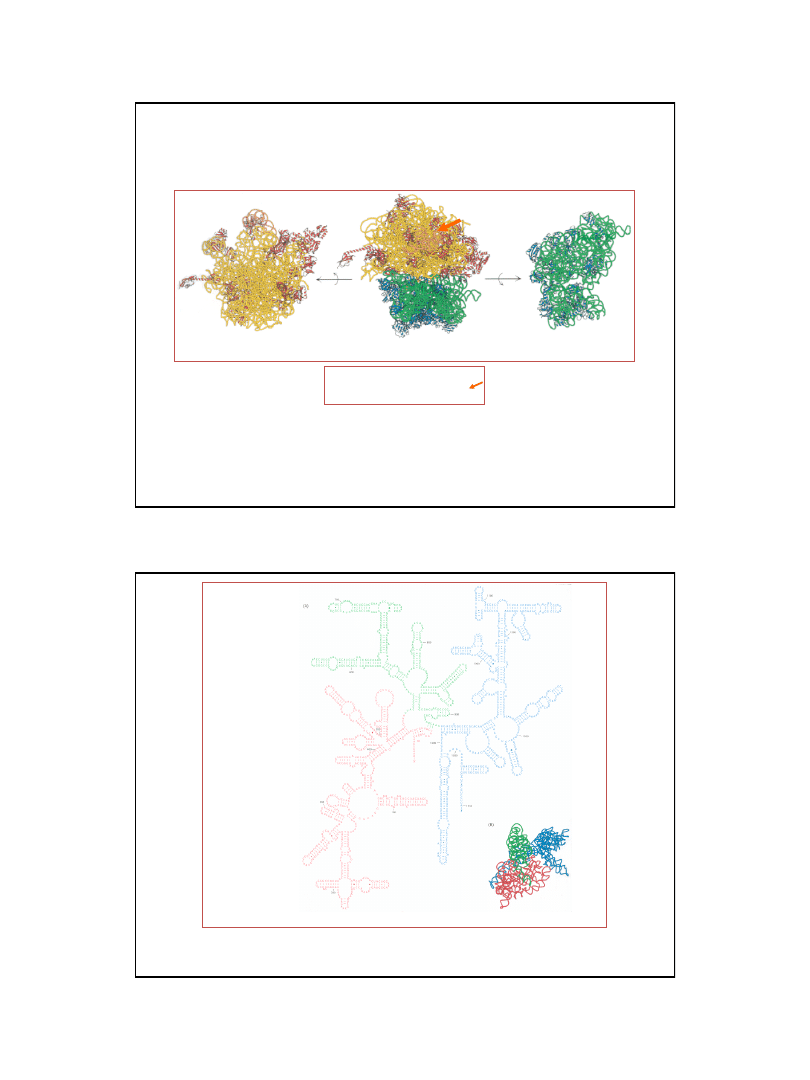
Rybosom prokariotyczny
70S
M. cz. 2,7 x 10
6
Rybosom eukariotyczny
80S
M. cz. 4,2 x 10
6
M. cz. 1,8 x 10
6
5S rRNA (120 nt.)
23S rRNA (3 200 nt.)
36 białek
M. cz. 2,8 x 10
6
5S rRNA (120 nt.)
28S rRNA (4 700 nt.)
5,8S rRNA (160 nt.)
ok. 49 białek
M. cz. 1,4 x 10
6
18S rRNA (1 900 nt.)
ok. 33 białka
M. cz. 0,9 x 10
6
16S rRNA (1 540 nt.)
21 białek
50S
40S
30S
60S
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Porównanie struktury rybosomów organizmów prokariotycznych i eukariotycznych
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
fot. James Lake
Mikrofotografia elektronowa
(A) podjednostek 30S (B) podjednostek 50S (C) rybosomów 70S
Polirybosomy (polisomy) w komórce Escherichia coli
z: Miller Jr. O.L., Hamkalo B.A., Thomas Jr. C.A.,
(1970), Science 169: 392.
50
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
rybosom 70S
podjednostka 50S
podjednostka 30S
Model struktury rybosomu prokariotycznego opracowany na podstawie
analizy krystalograficznej (analiza dyfrakcji promieniowania X)
kolor żółty – 23S rRNA
kolor pomarańczowy – 5S rRNA
kolor zielony
– 16S rRNA
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Struktura drugorzędowa
Struktura trzeciorzędowa
16S rRNA

51
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
miejsce syntezy wiązania peptydowego
zawiera wyłącznie RNA (kolor żółty)
Struktura fragmentu rybosomu obejmującego region transferazy peptydylowej
w promieniu 20
Ǻ od tego miejsca nie ma białek
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
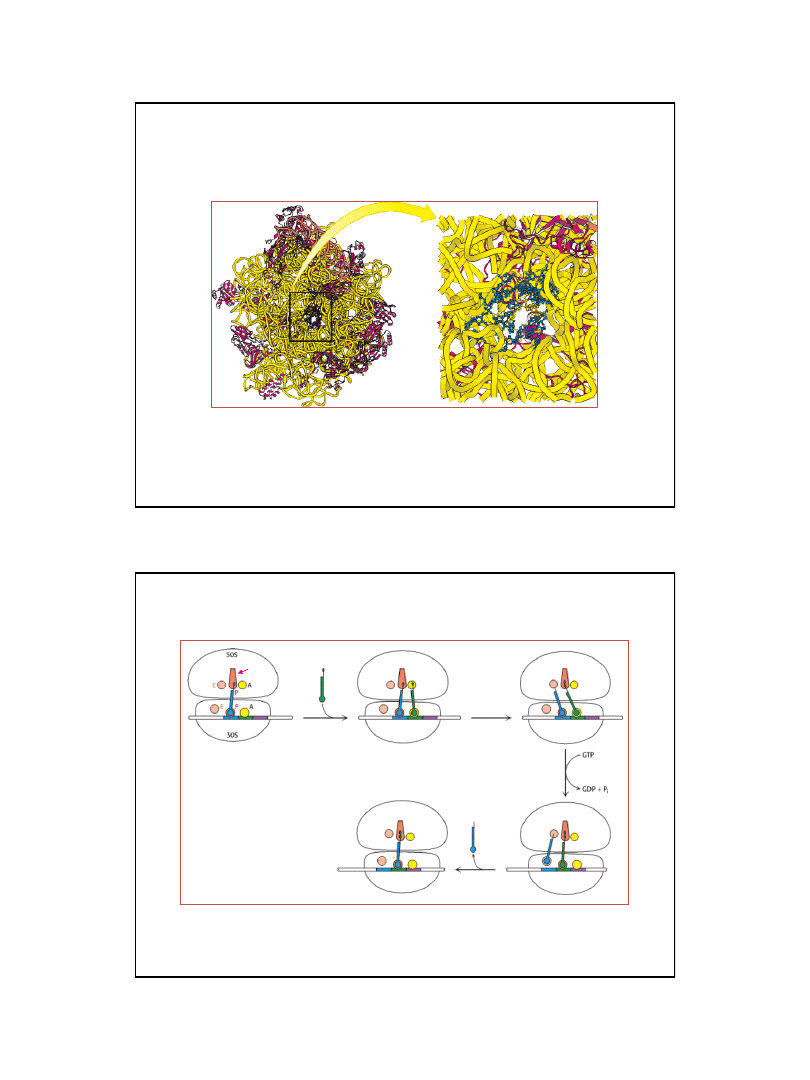
Biochemistry 5ed., W.H. Freeman and Co., New York
mRNA
miejsce
A
Aminoacylowe
Miejsce
P
Peptydylowe
Miejsce
E
Wyjścia (Exit)
E
A
P
Miejsca wiązania tRNA na rybosomie prokariotycznym
antykodony aminoacylo-tRNA w miejscu A oraz peptydylo-tRNA w miejscu P
są połączone komplementarnie z właściwymi sobie kodonami mRNA
52
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Tunel przechodzący przez podjednostkę 50S
rozpoczynający się w miejscu tworzenia wiązania peptydowego
Powstający łańcych polipeptydowy opuszcza rybosom przechodząc przez ten tunel
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Tunel
Wiązanie
aminoacylo-tRNA
Tworzenie
wiązania peptydowego
dysocjacja tRNA
Translokacja
EF-G
Przebieg translacji w komórkach prokariotycznych
Inicjacja i pierwsza reakcja elongacji
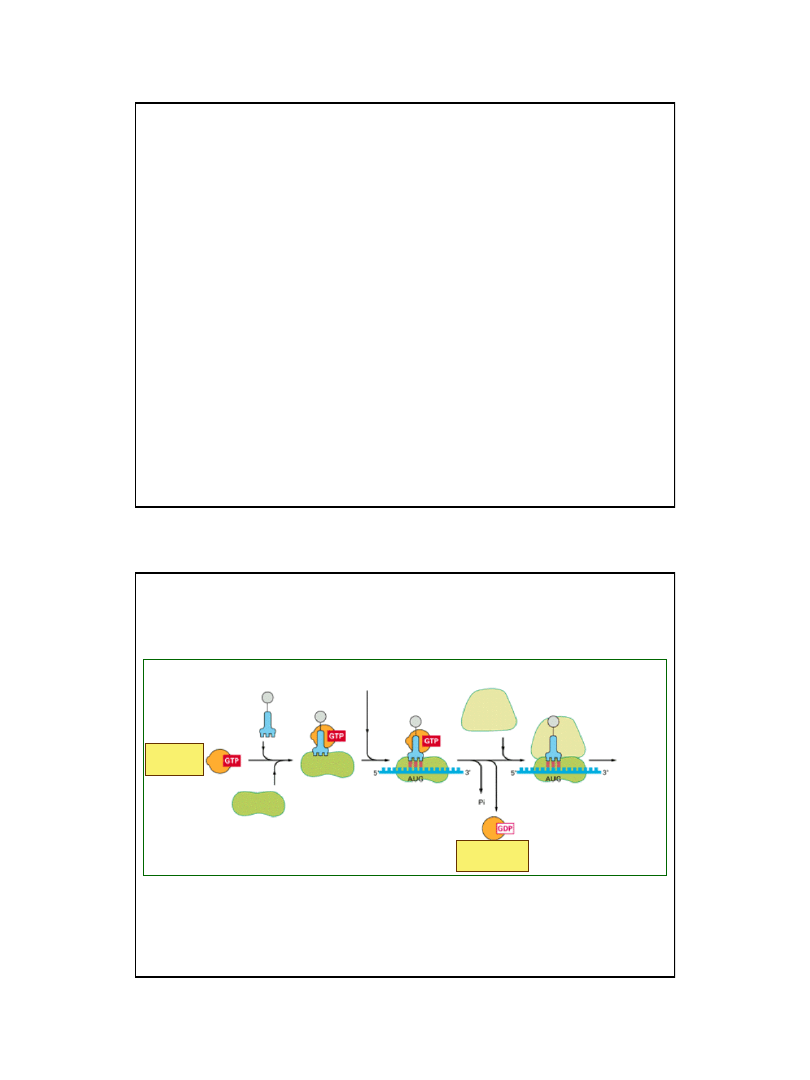
53
Podjednostka
30S
podjednostka
50S
mRNA
N-formylometionylo-tRNA
wiązanie czynników
inicjujących
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
Inicjacja procesu translacji w komórkach prokariotycznych
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Region oddziaływań komplementarnych
z sekwencjami Shine-Dalgarno
bakteryjnych mRNA
54
bakteriofag
λ białko cro
5’
Sekwencje Shine-Dalgarno
E. coli trpA
E.coli araB
E. coli lacI
bakteriofag
ΦX174 białko A
mRNA
5’
koniec 5’ mRNA
3’
koniec 3’ 16S rRNA
kodon
inicjatorowy
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
Miejsce wiązania rybosomu (RBS)
Rozpoznanie kodonu inicjatorowego w komórkach prokariotycznych
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
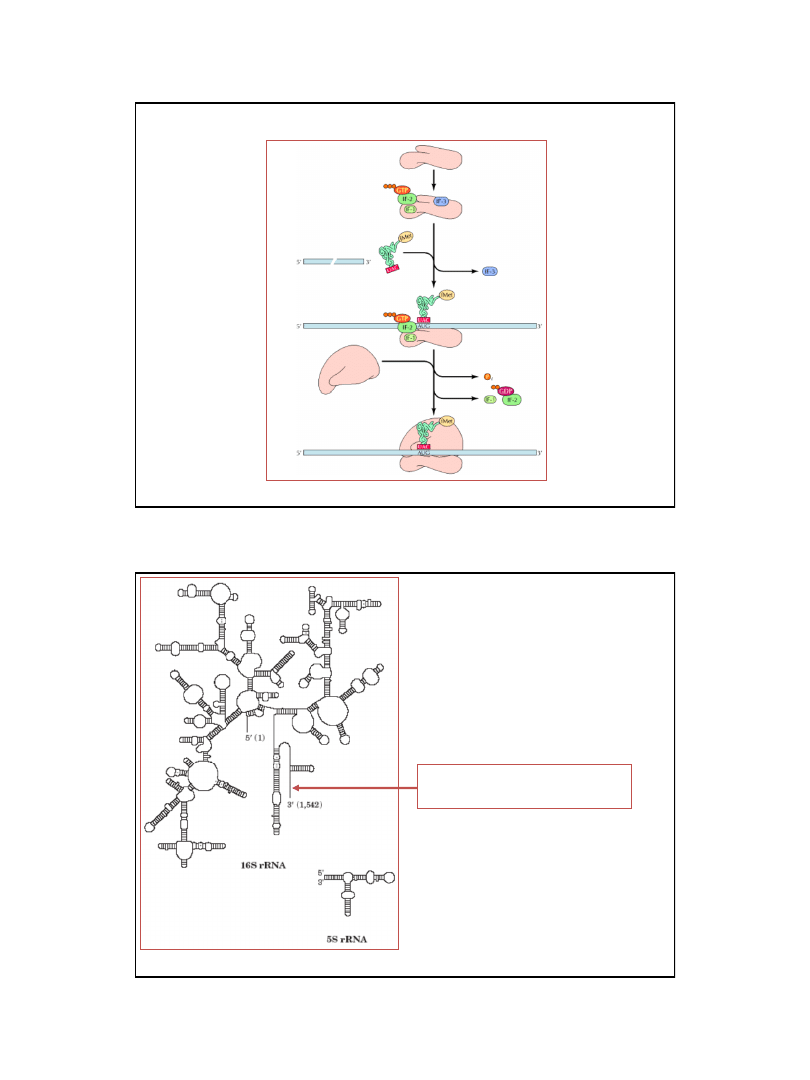
Inicjacja procesu translacji w komórkach eukariotycznych
Podjednostka
40S
podjednostka
60S
mRNA
kompleks
metionylo-tRNA
– eIF2-GTP
wiązanie czynników
inicjujących
Skanowanie
55
Bakteryjne
IF-1
-
zapobiega przedwczesnemu wiązaniu się tRNA w miejscu A,
IF-2
-
niezbędny do wiązania fMet-tRNA
fMet
z podjednostka 30S,
IF-3
-
wiążąc się z podjednostką 30S zapobiega przedwczesnemu dołączeniu sie podjednostki 50S;
zwiększa powinowactwo miejsca P do fMet-tRNA
fMet
.
Eukariotyczne
eIF-2
-
niezbędny do wiązania inicjatorowego Met-tRNA
Met
z podjednostka 40S,
eIF2B
,
eiF3
-
czynniki wiążące się, jako pierwsze z podjednostką 40S; warunek niezbędny
dla zajścia dalszych etapów inicjacji translacji,
eIF4A
- helikaza RNA
– usuwa struktury drugorzędowe mRNA umożliwiając jego wiązanie się
z podjednostką 40S; jest częścią kompleksu eIF4F,
eIF4B
-
wiąże się z mRNA, odpowiada za „skanowanie” mRNA do momentu zlokalizowania
pierwszego kodonu AUG,
eIF4E
-
wiąże się ze strukturą „cap” na końcu 5’ mRNA; jest częścią kompleksu eIF4F,
eIF4G
-
wiąże się z eIF4E oraz białkiem PABP (polyA binding protein);
jest częścią kompleksu eIF4F,
eIF5
-
odpowiada za odłączenie wcześniej związanych czynników inicjatorowych
od podjednostki 40S umożliwiając dołączenie podjednostki 60S i powstanie kompleksu
inicjującego 80S,
eIF6
-
umożliwia dysocjację nieaktywnego rybosomu 80S na podjednostki 40S i 60S.
Funkcje czynników inicjatorowych w translacji w komórkach
bakteryjnych i eukariotycznych
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
inicjatorowy
metionylo-tRNA
Aktywny
eIF2
podjednostka rybosomalna
40S
podjednostka rybosomalna
60S
mRNA
Nieaktywny
eIF2
Biosynteza
białka
Rola czynnika inicjującego eIF2 w inicjacji translacji w komórkach eukariotycznych
56
Ryc. z: Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D., 1994,
Molecular Biology of the Cell, 3rd Ed. Garland Publishing Inc.
eIF2B
białko uwalniające
nukleotyd guanylowy
Kinaza białkowa
fosforyluje eIF2
eIF2B
Aktywny
eIF2
Nieaktywny
eIF2
Nieaktywny
eIF2
ufosforylowany eIF2 wiąże
całą pulę eIF2B tworząc
nieaktywny kompleks
W nieobecności aktywnego
eIF2B większość eIF2
pozostaje w formie
nieaktywnego kompleksu
eIF2-GDP
Biosynteza białka zostaje
spowolniona dramatycznie
Udział eIF2 w regulacji translacji w organizmach eukariotycznych
Cykl eIF2
Fosforylacja eIF2 kontroluje
wydajność translacji
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
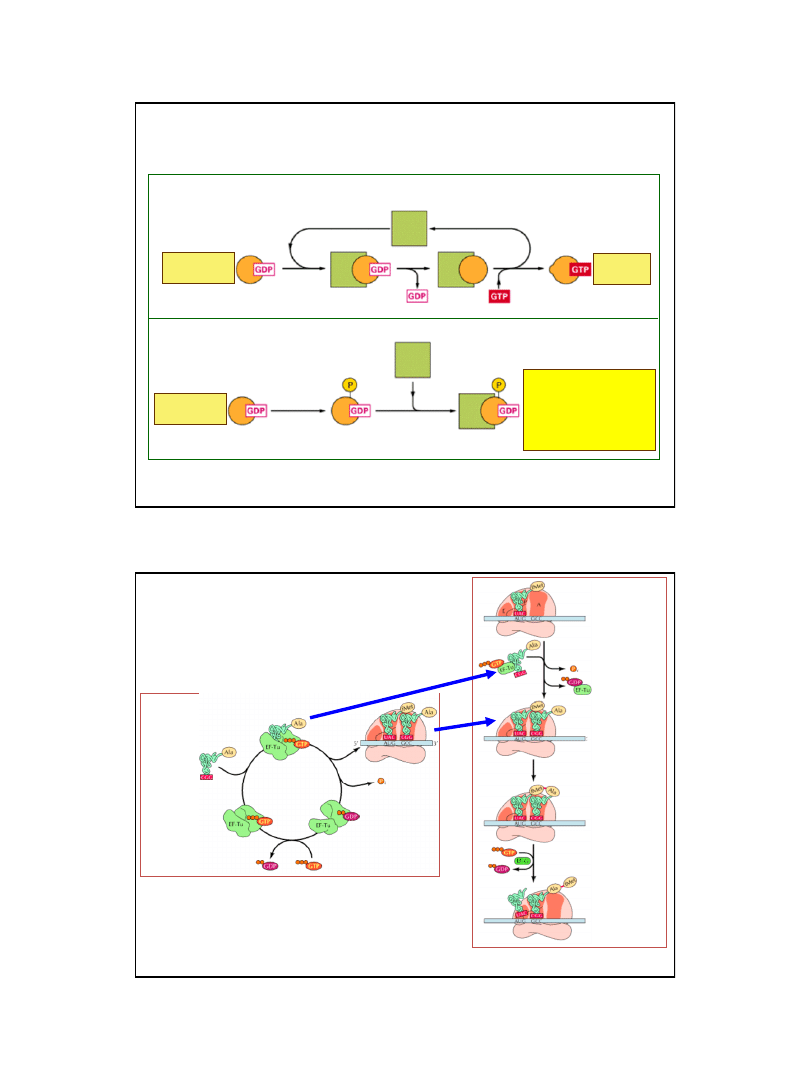
Translokacja
Powstawanie
wiązania peptydowego
aminoacylo-tRNA
EF-TS
nieaktywny
aktywny
5’
5’
5’
5’
Elongacja
Prokaryota
57
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Aminoacylo-tRNA
EF-Tu
GTP
Struktura kompleksu czynnika elongacyjnego Tu (EF-Tu) z aminoacylo-tRNA
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Intermediat
(tetraedralny)
miejsce P
miejsce A
Powstawanie wiązania peptydowego

58
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Mechanizm translokacji
W postaci kompleksu z GTP EF-
G ma zdolność wiązania się do miejsca wiązanie EF-Tu na podjednostce 50S.
Związanie z rybosomem powoduje hydrolizę GTP i w konsekwencji zmianę konformacji EF-G.
Zmiana konformacji związanego EF-G wprowadza jego domenę przypominającą tRNA w miejsce A wymuszając
translokację
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
GTP
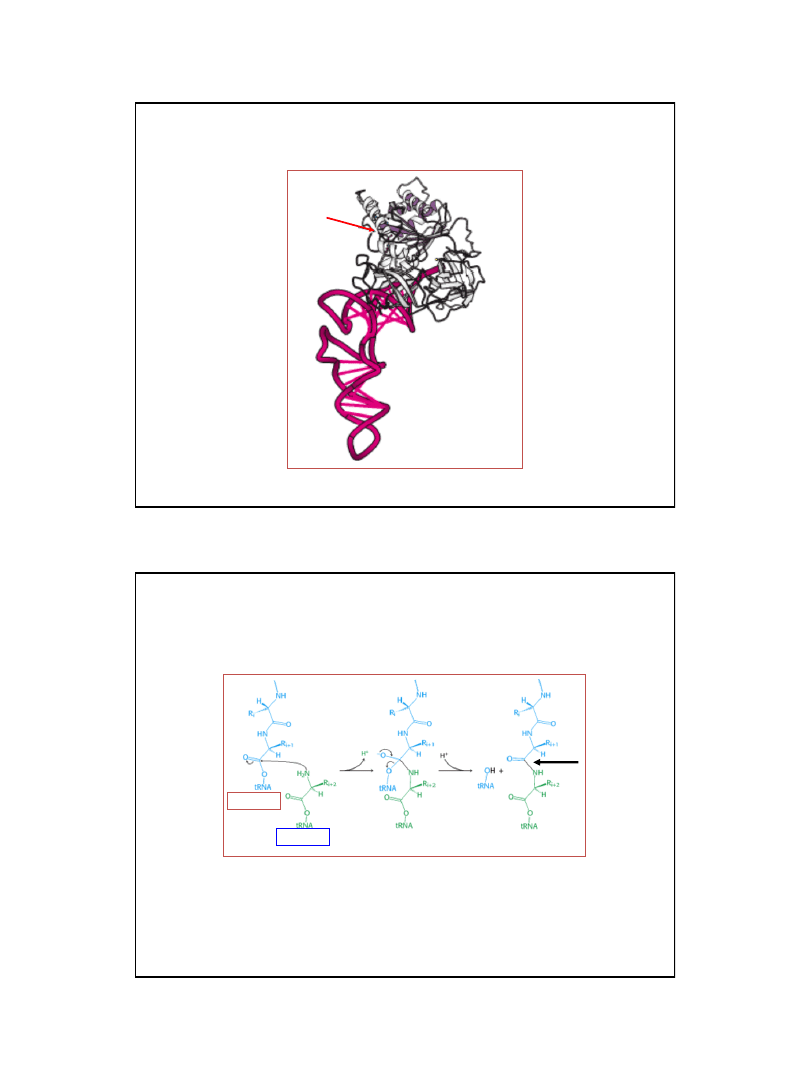
Struktura czynnika elongacyjnego G (EF-G)
59
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
GTP
EF-G
Aminoacylo-tRNA
EF-Tu
GTP
EF-Tu
– aa-tRNA
Ryc. z: Nelson D. L., Cox M.M., 2004, Lehninger Principles of Biochemistry.
4th Ed. W.H. Freeman & Co.; New York
tRNA
GDP
Kompleks EFTu
– tRNA
EF-G (w kompleksie z GDP)
60
Ryc. z: Cooper G. M., 2000, The Cell - A Molecular Approach.
2nd ed. Sunderland (MA)
Terminacja translacji
5’
5’
5’
5’
3’
3’
3’
3’
Czynnik RF
Prokariotyczne czynniki terminujące:
RF-1
rozpoznaje UAG i UAA
RF-2
rozpoznaje UGA i UAA
RF-3
?
(odłączenie podjednostek?)
W komórkach eukariotycznych:
jeden czynnik
eRF
rozpoznaje wszystkie kodony stop
Ryc. z: Berg J.M., Tymoczko J.L., Stryer L., (2002),
Biochemistry 5ed., W.H. Freeman and Co., New York
Struktura eukariotycznego czynnika RF
(Release Factor)
Kształt cząsteczki czynnika RF przypomina tRNA. Domena odpowiadająca ramieniu akceptorowemu
może wiązać cząsteczkę wody i przenosić ją do wnętrza centrum transferazy peptydylowej
Wyszukiwarka
Podobne podstrony:
BKNB 1213 03
Wykład 04
04 22 PAROTITE EPIDEMICA
04 Zabezpieczenia silnikówid 5252 ppt
Wyklad 04
Wyklad 04 2014 2015
04 WdK
04) Kod genetyczny i białka (wykład 4)
2009 04 08 POZ 06id 26791 ppt
2Ca 29 04 2015 WYCENA GARAŻU W KOSZTOWEJ
04 LOG M Informatyzacja log
04 Liczby ujemne i ułamki w systemie binarnym
UE i ochrona srodowiska 3 04 2011
04 QueryByExample Access
więcej podobnych podstron