WYKŁAD VII
WYKŁAD VII
EKSPRESJA INFORMACJI
EKSPRESJA INFORMACJI
GENETYCZNEJ
GENETYCZNEJ
czyli synteza RNA i białek
czyli synteza RNA i białek

Po poznaniu struktury DNA (1953) stało
Po poznaniu struktury DNA (1953) stało
się jasne, że informacja genetyczna w
się jasne, że informacja genetyczna w
DNA zawiera
DNA zawiera
KOD
KOD
, w którym
, w którym
zaszyfrowana jest budowa wszystkich
zaszyfrowana jest budowa wszystkich
niezbędnych komórce białek.
niezbędnych komórce białek.
W jaki sposób komórka przekształca
W jaki sposób komórka przekształca
informację genetyczną zawartą w DNA
informację genetyczną zawartą w DNA
w sekwencje aminokwasów w białku,
w sekwencje aminokwasów w białku,
czyli w jaki sposób dochodzi do
czyli w jaki sposób dochodzi do
EKSPRESJI INFORMACJI
EKSPRESJI INFORMACJI
GENETYCZNEJ?
GENETYCZNEJ?

Ekspresja genów odbywa
Ekspresja genów odbywa
się zasadniczo w 2 etapach:
się zasadniczo w 2 etapach:
1. przepisanie sekwencji nukl. w
1. przepisanie sekwencji nukl. w
DNA na RNA – TRANSKRYPCJA (w
DNA na RNA – TRANSKRYPCJA (w
jądrze)
jądrze)
2. przepisanie sekwencji w RNA na
2. przepisanie sekwencji w RNA na
sekwencję aminokwasów –
sekwencję aminokwasów –
TRANSLACJA (w cytoplaźmie na
TRANSLACJA (w cytoplaźmie na
rybosomach)
rybosomach)
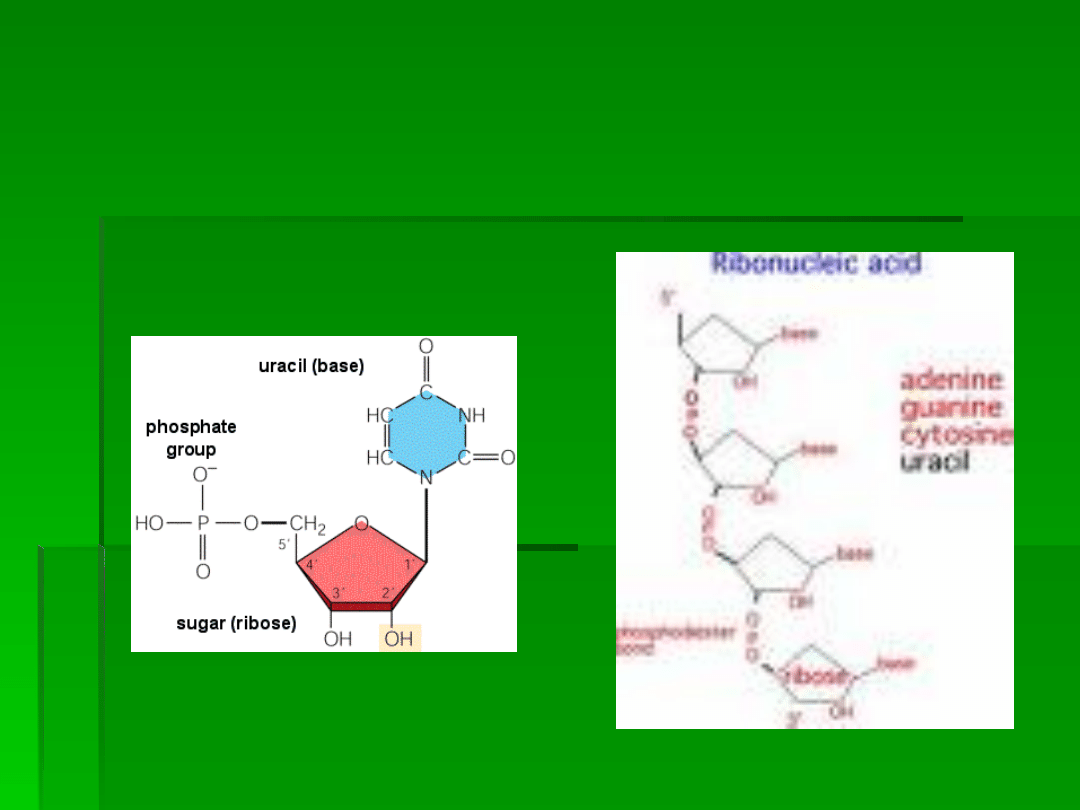
Pośrednikiem między DNA i
Pośrednikiem między DNA i
białkiem jest kwas
białkiem jest kwas
rybonukleinowy mRNA (iRNA)
rybonukleinowy mRNA (iRNA)
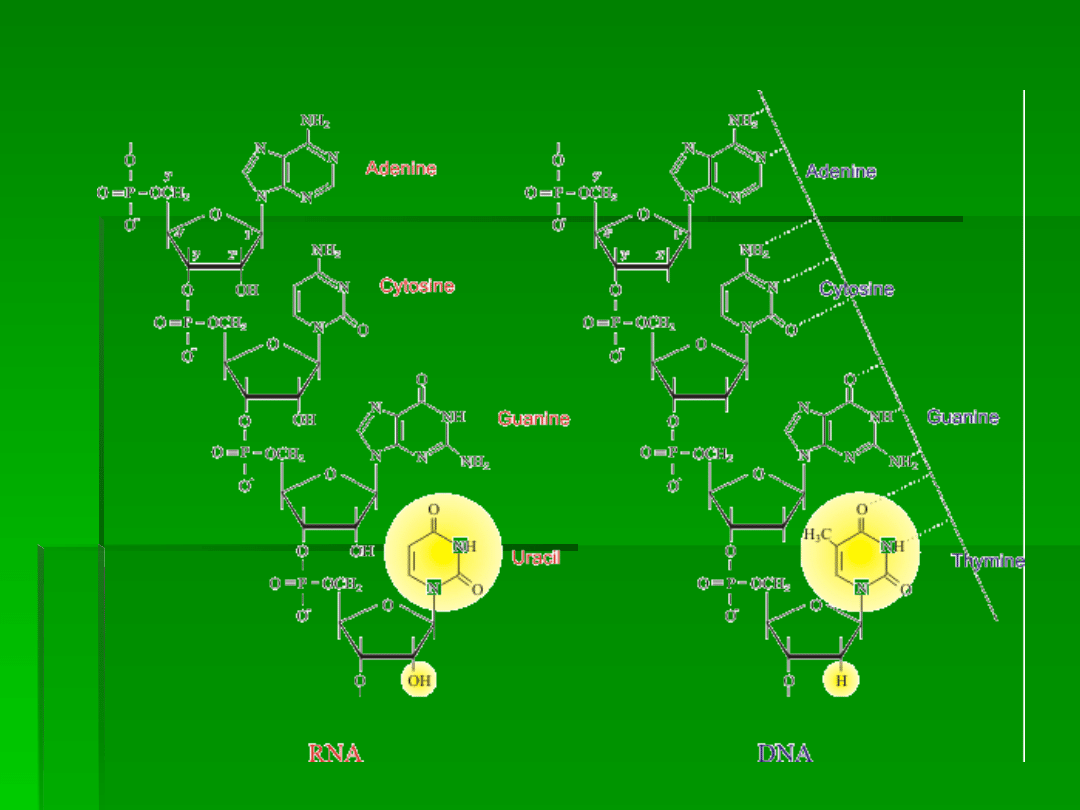
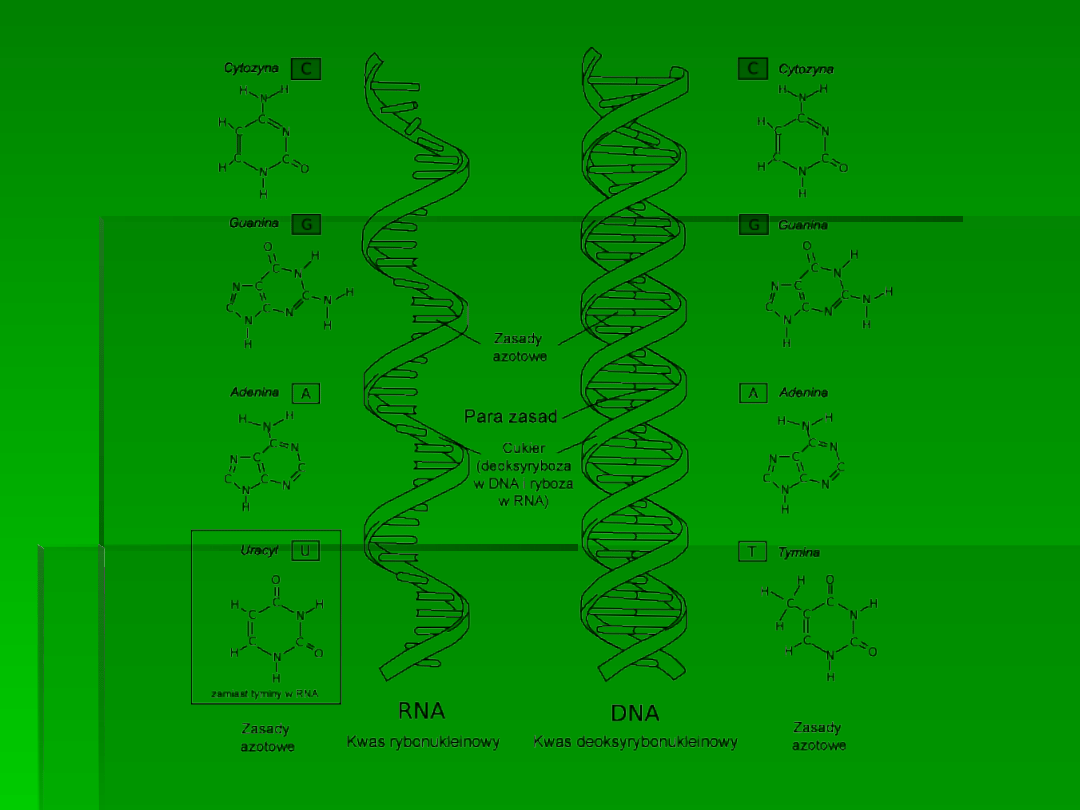

Rodzaje RNA
Rodzaje RNA
(przekazywanie informacji, określającej
(przekazywanie informacji, określającej
kolejność łączenia aminokwasow w
kolejność łączenia aminokwasow w
biosyntezie białka),
biosyntezie białka),
(cząsteczki przenoszące aminokwasy w
(cząsteczki przenoszące aminokwasy w
procesie biosyntezy białek i innych szlakach
procesie biosyntezy białek i innych szlakach
anabolicznych),
anabolicznych),
(cząsteczki biorące udział w budowie
(cząsteczki biorące udział w budowie
rybosomu),
rybosomu),

Rodzaje RNA
Rodzaje RNA
(cząsteczki
(cząsteczki
pełniące szereg funkcji w komórce,
pełniące szereg funkcji w komórce,
związanych z ekspresją materiału
związanych z ekspresją materiału
genetycznego).
genetycznego).
antysensowny RNA albo interferencyjny RNA
antysensowny RNA albo interferencyjny RNA
(RNAi)- produkowany w celu precyzyjnej
(RNAi)- produkowany w celu precyzyjnej
regulacji
.
.
mały jądrowy (
mały jądrowy (
) pełniący funkcje
) pełniący funkcje
przy wycinaniu
przy wycinaniu
z
transkryptów
transkryptów

Matrycowy, czyli informacyjny
Matrycowy, czyli informacyjny
RNA
RNA
Trójki nukleotydów, czyli kodony,
Trójki nukleotydów, czyli kodony,
rozmieszczone w jego łańcuchu
rozmieszczone w jego łańcuchu
wyznaczają kolejność aminokwasów
wyznaczają kolejność aminokwasów
syntetyzowanego białka.
syntetyzowanego białka.
W procesie transkrypcji u
W procesie transkrypcji u
eukariontów powstaje najpierw pre-
eukariontów powstaje najpierw pre-
mRNA, jako składnik frakcji
mRNA, jako składnik frakcji
heterogennego jądrowego hnRNA.
heterogennego jądrowego hnRNA.

mRNA u bakterii jest policistronowy
mRNA u bakterii jest policistronowy
Cząsteczka bakteryjnego mRNA
Cząsteczka bakteryjnego mRNA
może zawierać kod dla całego
może zawierać kod dla całego
zespołu białek
zespołu białek
Oprócz kodonów łańcuch mRNA
Oprócz kodonów łańcuch mRNA
zawiera tzw. trójki nonsensowne,
zawiera tzw. trójki nonsensowne,
które są znakami przestankowymi,
które są znakami przestankowymi,
umożliwiającymi syntezę wielu
umożliwiającymi syntezę wielu
białek.
białek.

U eukariontów mRNA jest
U eukariontów mRNA jest
monocistronowy,
monocistronowy,
zawiera informację tylko dla
zawiera informację tylko dla
jednego łańcucha polipeptydowego.
jednego łańcucha polipeptydowego.
Jest pojedynczym łańcuchem
Jest pojedynczym łańcuchem
skręconym w postaci spirali,
skręconym w postaci spirali,
chronionym białkami
chronionym białkami
informomerowymi.
informomerowymi.
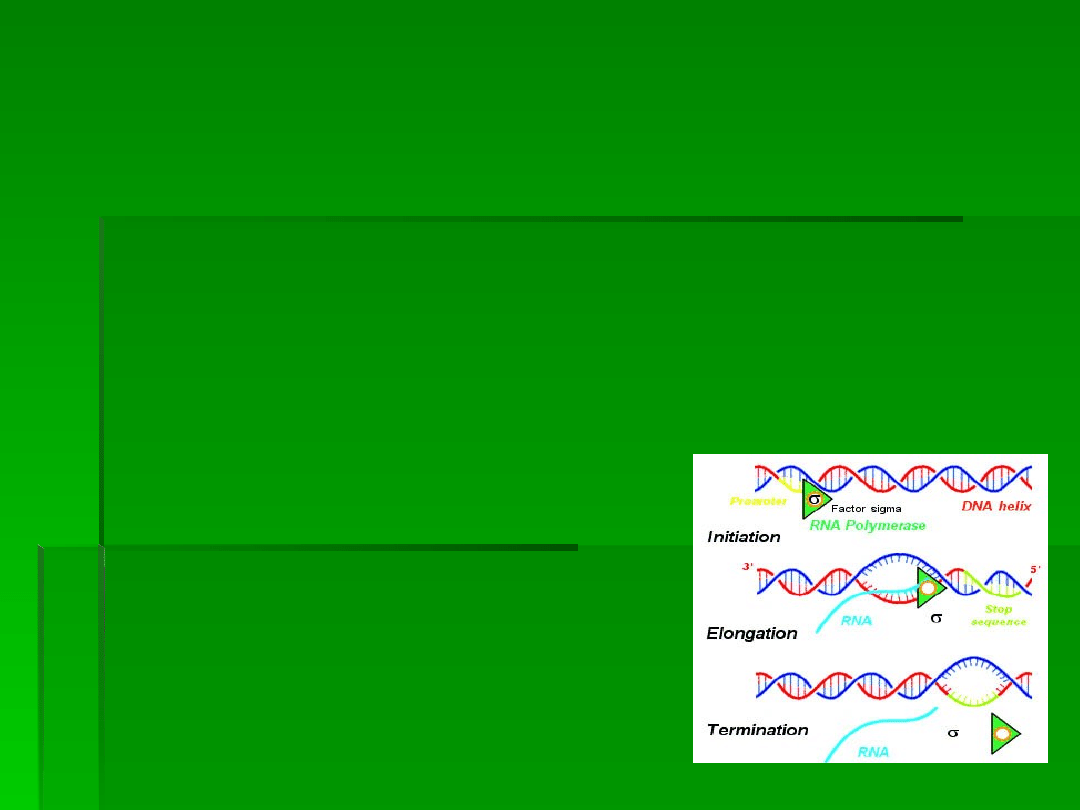
TRANSKRYPCJA
TRANSKRYPCJA
Transkrypcja to proces, w którym informacja
Transkrypcja to proces, w którym informacja
zawarta w DNA - zapisana w formie sekwencji
zawarta w DNA - zapisana w formie sekwencji
deoksynukleotydów - przepisana zostaje na
deoksynukleotydów - przepisana zostaje na
sekwencje rybonukleotydów w pre-mRNA
sekwencje rybonukleotydów w pre-mRNA
podczas reakcji katalizowanej przez
podczas reakcji katalizowanej przez
polimerazę RNA.
polimerazę RNA.
.
.
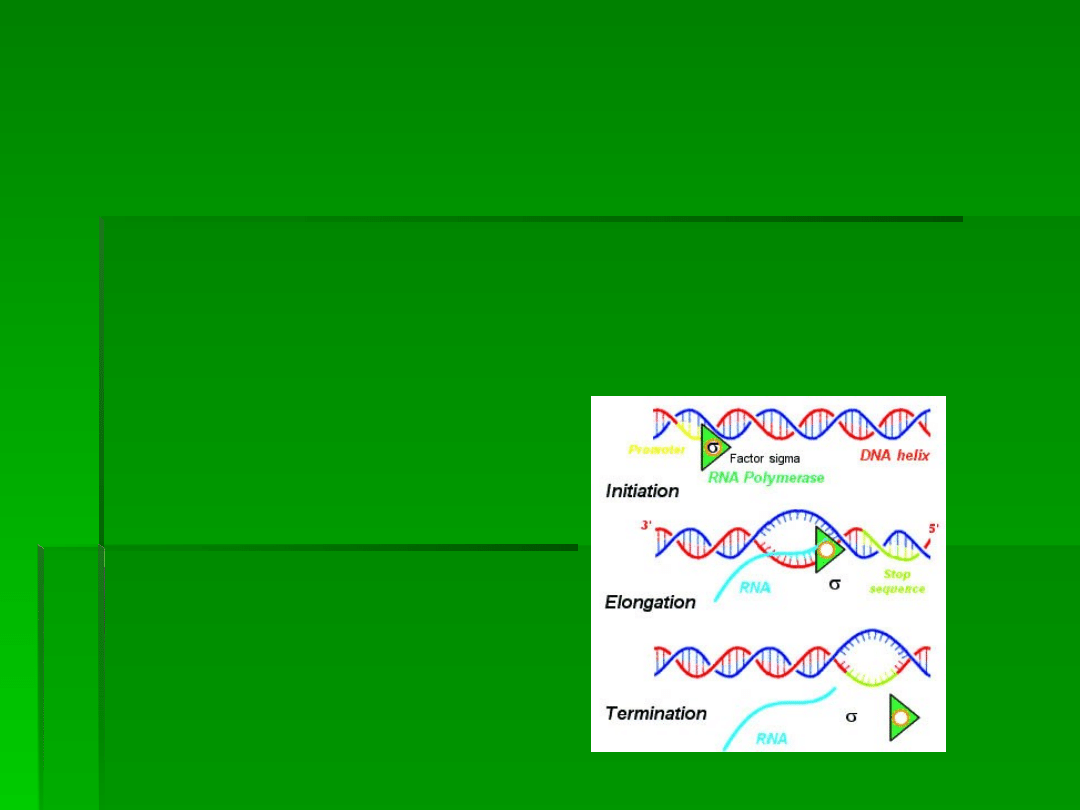
Dzieli się ją na trzy następujące po
Dzieli się ją na trzy następujące po
sobie procesy:
sobie procesy:
inicjację transkrypcji,
inicjację transkrypcji,
elongację łańcucha pre-mRNA
elongację łańcucha pre-mRNA
terminację
terminację

Inicjacja transkrypcji jest głównym
Inicjacja transkrypcji jest głównym
punktem kontrolnym ekspresji
punktem kontrolnym ekspresji
genu.
genu.
Polimeraza II RNA wymaga do
Polimeraza II RNA wymaga do
zapoczątkowania reakcji obecności
zapoczątkowania reakcji obecności
białek - czynników transkrypcyjnych
białek - czynników transkrypcyjnych
tzw. TF-ów (ang. transcription
tzw. TF-ów (ang. transcription
factors).
factors).
Białka te w określonym porządku
Białka te w określonym porządku
wiążą się do DNA w obrębie
wiążą się do DNA w obrębie
promotora, enhancerów bądź
promotora, enhancerów bądź
silencerów.
silencerów.

Polimeraza RNA jest DNA zależna.
Polimeraza RNA jest DNA zależna.
Została odkryta przez Weissa w
Została odkryta przez Weissa w
1959r, dlatego bywa nazywana
1959r, dlatego bywa nazywana
polimerazą Weissa
polimerazą Weissa
Polimeraza RNA
Polimeraza RNA
(nukleotydylotransferaza
(nukleotydylotransferaza
nukleozydotrifosforanów) zbudowana
nukleozydotrifosforanów) zbudowana
jest z 5 podjednostek:
jest z 5 podjednostek:
2 alfa, beta, beta prim i sigma.
2 alfa, beta, beta prim i sigma.

Wyróżnia się 3 polimerazy RNA:
Wyróżnia się 3 polimerazy RNA:
Polimeraza I – znajduje się w
Polimeraza I – znajduje się w
jąderku, uczestniczy w tworzeniu
jąderku, uczestniczy w tworzeniu
rybosomalnego kwasu
rybosomalnego kwasu
rybonukleinowego rRNA
rybonukleinowego rRNA

Polimeraza II – transkrybuje geny
Polimeraza II – transkrybuje geny
struktury. Występuje w chromatynie
struktury. Występuje w chromatynie
jądra i w cytoplazmie.
jądra i w cytoplazmie.
Polimeraza III – występuje w
Polimeraza III – występuje w
chromatynie jądra i w cytoplazmie,
chromatynie jądra i w cytoplazmie,
uczestniczy w tworzeniu tRNA i
uczestniczy w tworzeniu tRNA i
rRNA.
rRNA.

Rozsunięcie nici DNA na odcinku
Rozsunięcie nici DNA na odcinku
kilkunastu nukleotydów umożliwia
kilkunastu nukleotydów umożliwia
wstawianie (włączenie) kolejnych,
wstawianie (włączenie) kolejnych,
odpowiednich nukleotydów
odpowiednich nukleotydów
(elongacja transkrypcji).
(elongacja transkrypcji).
Substratami są trifosforany
Substratami są trifosforany
rybonukleozydów (
rybonukleozydów (
i
).
Polimeraza RNA przesuwa się
Polimeraza RNA przesuwa się
systematycznie wzdłuż
systematycznie wzdłuż
wydłuża łańcuch RNA, przy czym
wydłuża łańcuch RNA, przy czym
nukleotydy włączane są zgodnie z
nukleotydy włączane są zgodnie z

Powyżej aktualnego miejsca syntezy
Powyżej aktualnego miejsca syntezy
powstający hybrydowy kompleks DNA -
powstający hybrydowy kompleks DNA -
RNA ulega rozpadowi,
RNA ulega rozpadowi,
DNA powraca do swojej pierwotnej
DNA powraca do swojej pierwotnej
dwuniciowej struktury,
dwuniciowej struktury,
a łańcuch
a łańcuch
powstającego
powstającego
mRNA oddziela się.
mRNA oddziela się.
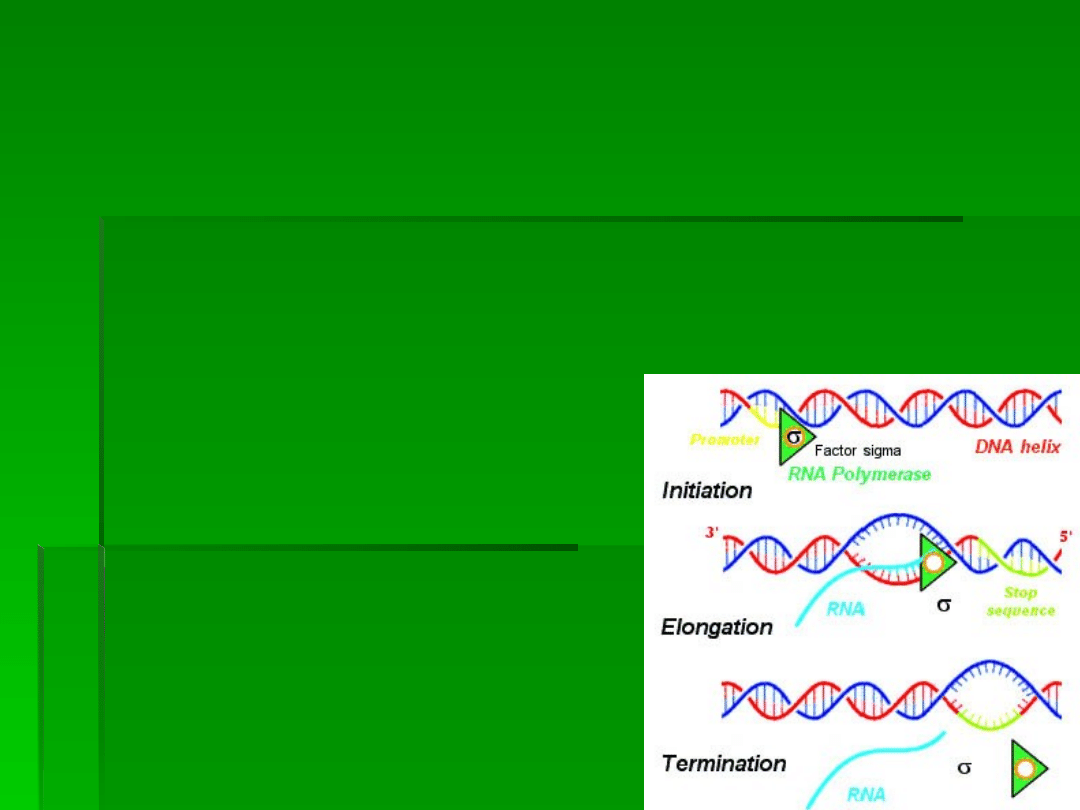
Etap elongacji (wydłużania RNA)
Etap elongacji (wydłużania RNA)
kończy się, gdy polimeraza RNA
kończy się, gdy polimeraza RNA
dotrze do sekwencji kończącej
dotrze do sekwencji kończącej
wyznaczającej miejsce terminacji
wyznaczającej miejsce terminacji
(zakończenia) transkrypcji.
(zakończenia) transkrypcji.
Nowo powstały produkt
Nowo powstały produkt
nazywamy pierwotnym
nazywamy pierwotnym
transkryptem
transkryptem
lub pre-mRNA
lub pre-mRNA

Pre-mRNA
Pre-mRNA
(hnRNA, heterogenny
(hnRNA, heterogenny
jądrowy RNA) - bezpośredni
jądrowy RNA) - bezpośredni
produkt transkrypcji
produkt transkrypcji
Po dołączeniu guanylowej „czapki” na
Po dołączeniu guanylowej „czapki” na
końcu 5' łańcucha
końcu 5' łańcucha
i sekwencji poli-A na końcu 3' łańcucha
i sekwencji poli-A na końcu 3' łańcucha
oraz po wycięciu niekodujących sekwencji,
oraz po wycięciu niekodujących sekwencji,
powstanie właściwy mRNA biorący udział
powstanie właściwy mRNA biorący udział
w translacji.
w translacji.
Modyfikacje hnRNA prowadzące do
Modyfikacje hnRNA prowadzące do
powstania mRNA nazywa się
powstania mRNA nazywa się
obróbką
obróbką
potranskrypcyjną
potranskrypcyjną
.
.

Guanylacja końca 5’=blokowanie końca
Guanylacja końca 5’=blokowanie końca
5’
5’
Polega na przyłączeniu do pierwszego
Polega na przyłączeniu do pierwszego
nukleotydu pre mRNA w pozycji 5’
nukleotydu pre mRNA w pozycji 5’
„czapeczki” czyli 7-metyloguanozyny.
„czapeczki” czyli 7-metyloguanozyny.
która łączy się z wszystkimi trzema
która łączy się z wszystkimi trzema
resztami fosforanowymi pierwszego
resztami fosforanowymi pierwszego
nukleotydu
nukleotydu
Ułatwia przyłączanie rybosomu do mRNA w
Ułatwia przyłączanie rybosomu do mRNA w
czasie translacji
czasie translacji
Ma znaczenie stabilizujące, ochronne przed
Ma znaczenie stabilizujące, ochronne przed
egzonukleazami.
egzonukleazami.
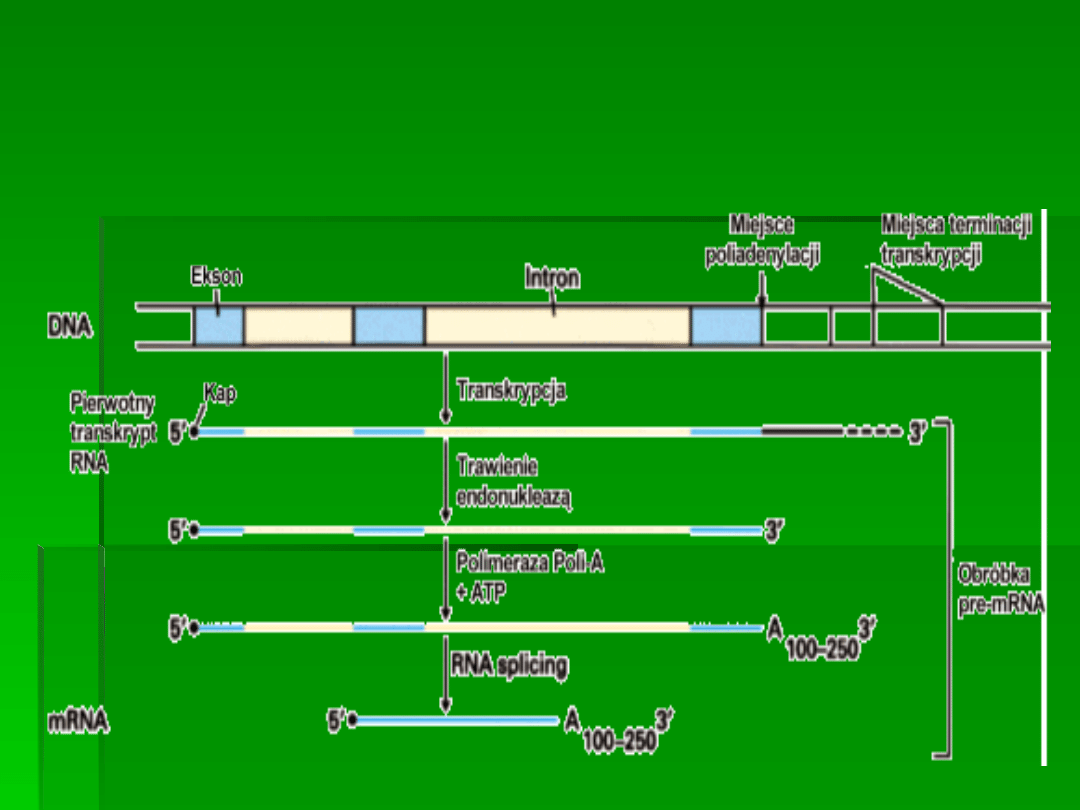
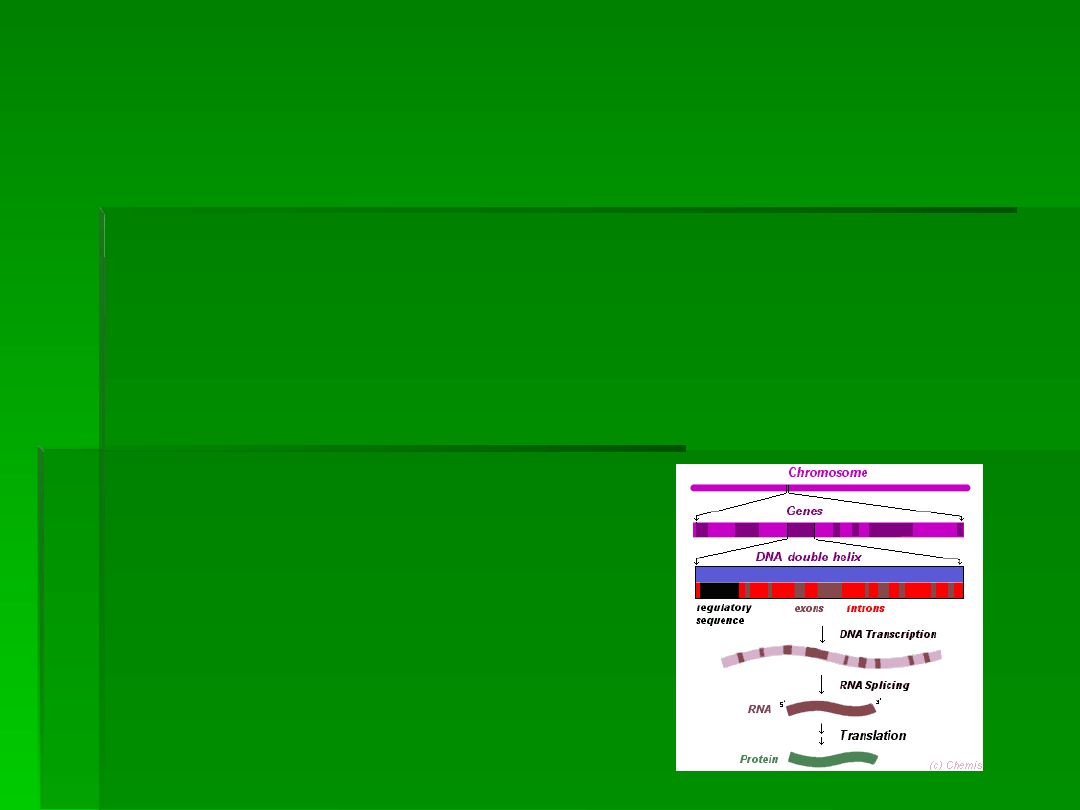
Schemat obrazujący procesy
Schemat obrazujący procesy
zachodzące na drodze od DNA do mRNA
zachodzące na drodze od DNA do mRNA

Poliadenylacja końca 3’
Poliadenylacja końca 3’
Polimeraza poli A dodaje na końcu 3'
Polimeraza poli A dodaje na końcu 3'
pierwotnego transkryptu nukleotydy
pierwotnego transkryptu nukleotydy
adeninowe (od 70 do 250)
adeninowe (od 70 do 250)
jednak dopiero po zadziałaniu
jednak dopiero po zadziałaniu
specyficznej nukleazy tj. enzymu
specyficznej nukleazy tj. enzymu
tnącego kwas nukleinowy w obrębie
tnącego kwas nukleinowy w obrębie
jego cząsteczki.
jego cząsteczki.

Okazało się, że ogon poli A nie jest
Okazało się, że ogon poli A nie jest
dołączany do ostatniego nukleotydu
dołączany do ostatniego nukleotydu
wbudowanego na drodze
wbudowanego na drodze
transkrypcji,
transkrypcji,
a do tego, który stał się ostatnim po
a do tego, który stał się ostatnim po
rozcięciu nici pre-mRNA.
rozcięciu nici pre-mRNA.

Miejsce atakowane przez nukleazę
Miejsce atakowane przez nukleazę
wyznacza sekwencja (sygnał
wyznacza sekwencja (sygnał
poliadenylacji): AAUAAA.
poliadenylacji): AAUAAA.
Istnieją geny zawierające więcej niż
Istnieją geny zawierające więcej niż
jeden sygnał poliadenylacji.
jeden sygnał poliadenylacji.
Oznacza to, że na ich matrycy
Oznacza to, że na ich matrycy
powstanie kilka pierwotnych
powstanie kilka pierwotnych
transkryptów różniących się
transkryptów różniących się
długością , a co za tym idzie kilka
długością , a co za tym idzie kilka
różnych mRNA.
różnych mRNA.
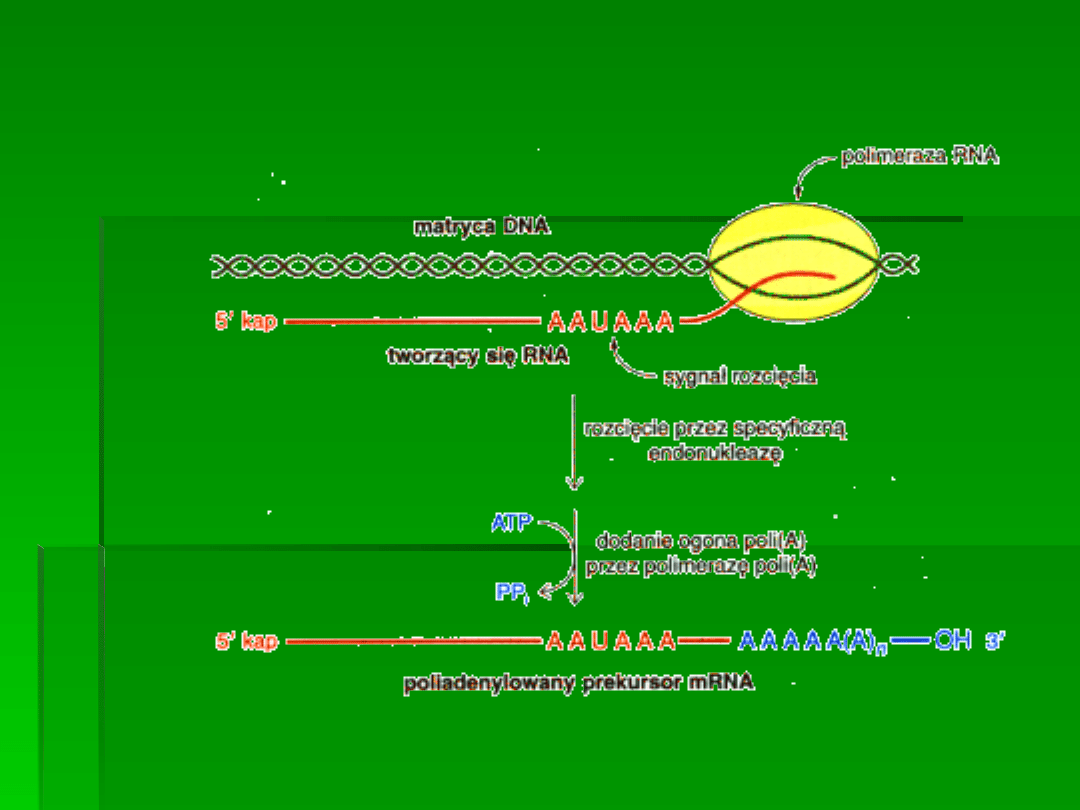
Mechanizm poliadenylacji
Mechanizm poliadenylacji
pre-mRNA
pre-mRNA

fragment poli A,
fragment poli A,
podobnie jak struktura czapeczki
podobnie jak struktura czapeczki
chroni cząsteczkę pierwotnego
chroni cząsteczkę pierwotnego
transkryptu przed działaniem nukleaz.
transkryptu przed działaniem nukleaz.
Może on mieć również znaczenie w
Może on mieć również znaczenie w
translacji,
translacji,
gdyż transkrypt pozbawiony poli A
gdyż transkrypt pozbawiony poli A
jest mniej wydajną matrycą przy
jest mniej wydajną matrycą przy
syntezie białka.
syntezie białka.

Obróbka potranskrypcyjna
Obróbka potranskrypcyjna
- s
- s
plicing
plicing
(czyt.
(czyt.
splaising), cięcie i
splaising), cięcie i
składanie genu
składanie genu
.
.
W intronach wielu genów kodujących białka
W intronach wielu genów kodujących białka
u Eucar. są trzy sekwencje sygnałowe dla
u Eucar. są trzy sekwencje sygnałowe dla
splicingu
splicingu
:
:
*sekwencje
*sekwencje
GU na końcu 5’
GU na końcu 5’
*AG na końcu 3’- (
*AG na końcu 3’- (
są częścią
są częścią
dłuższych sekwencji sygnałowych)
dłuższych sekwencji sygnałowych)
*wewnątrz intronu, w miejscu bliskim
*wewnątrz intronu, w miejscu bliskim
końca 3’znajduje się
końca 3’znajduje się
sekwencja
sekwencja
rozgałęziająca
rozgałęziająca
, u wyższych Euc.:
, u wyższych Euc.:
UACUAAC
UACUAAC
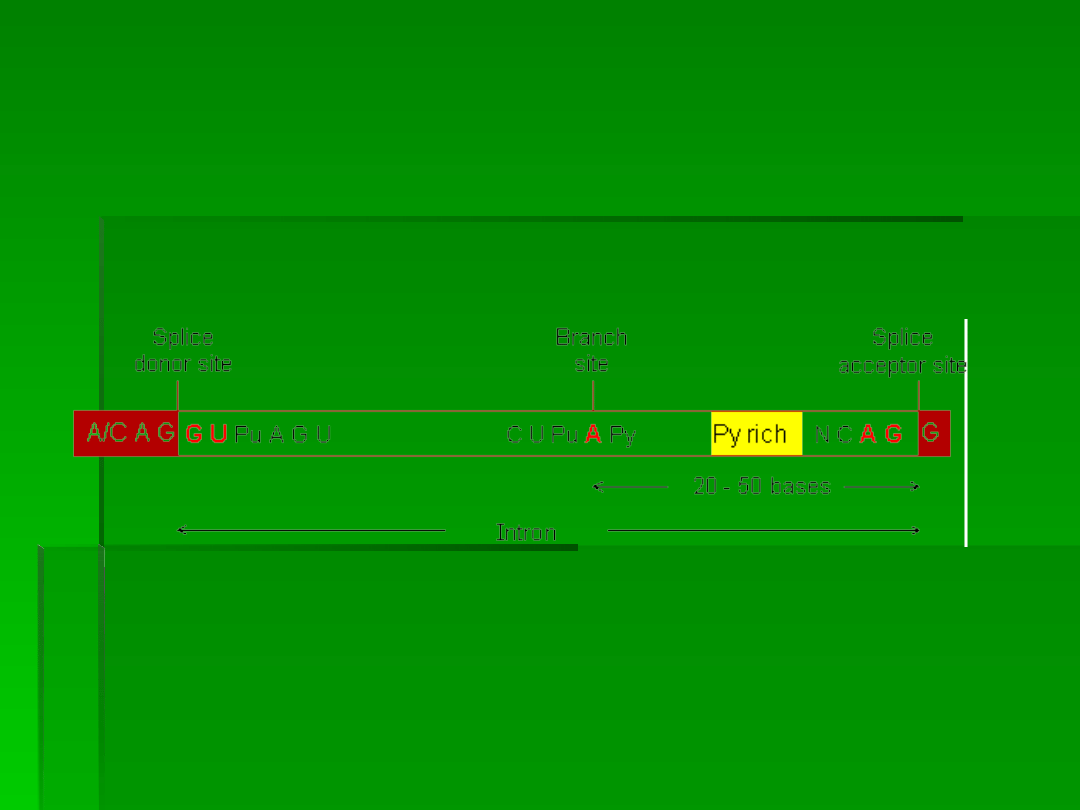
trzy sekwencje sygnałowe dla splicingu W
trzy sekwencje sygnałowe dla splicingu W
intronie:
intronie:
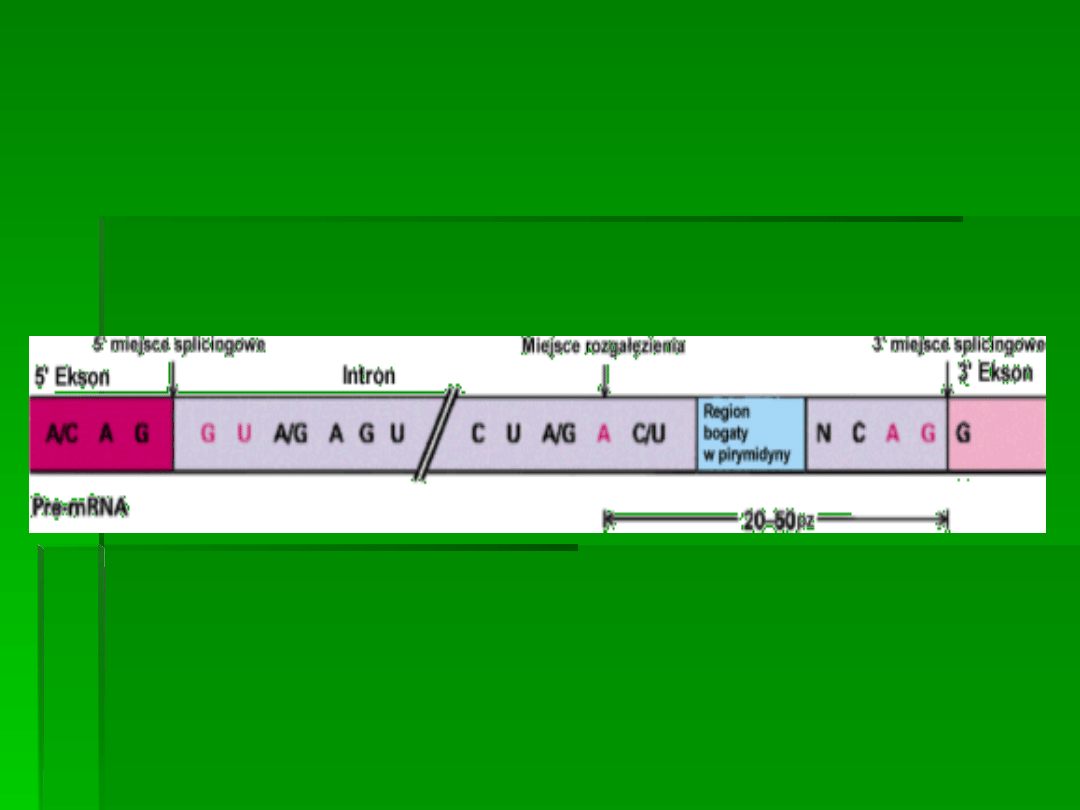
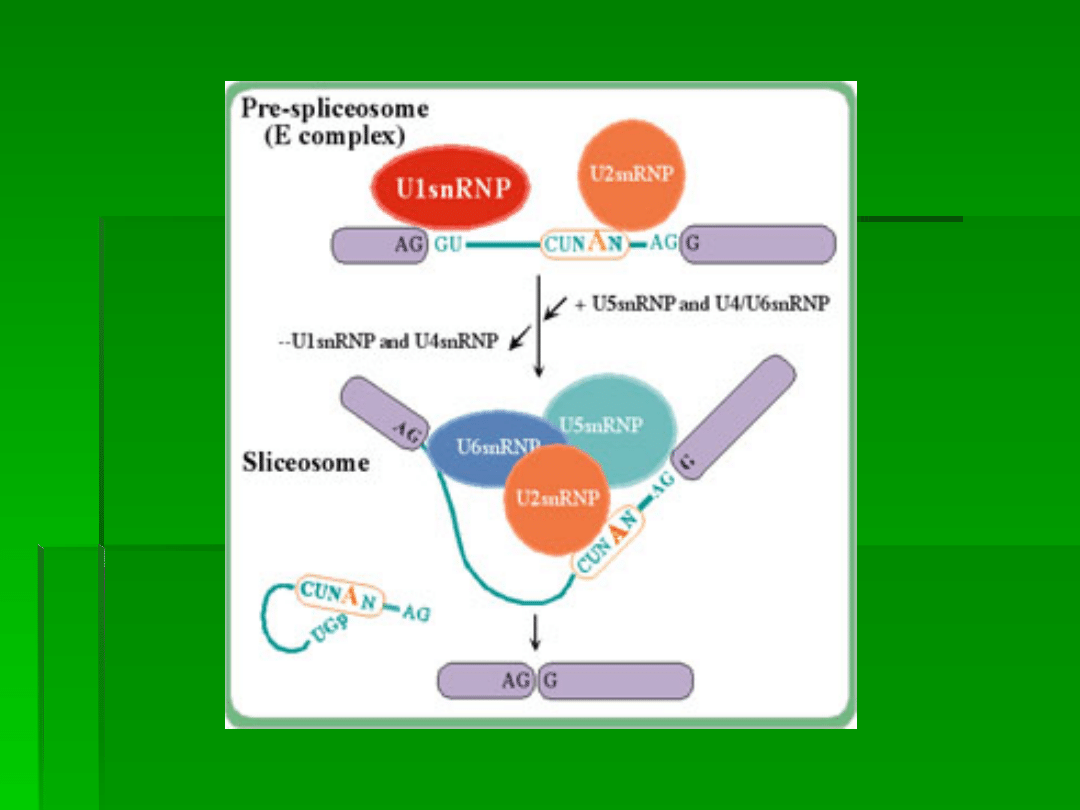
Sygnały splicingu
Sygnały splicingu

Wycinanie intronów
Wycinanie intronów
z pre-mRNA
z pre-mRNA
I-szy etap
I-szy etap
- uwolnienie końca 5’ intronu i
- uwolnienie końca 5’ intronu i
połączeniu go z miejscem rozgałęzienia –
połączeniu go z miejscem rozgałęzienia –
tworzy się struktura zwana lassem
tworzy się struktura zwana lassem
II-gi etap
II-gi etap
– rozszczepeinie transkryptu
– rozszczepeinie transkryptu
na końcu 3’ intronu
na końcu 3’ intronu

III etap – zbliżenie i kowalencyjne
III etap – zbliżenie i kowalencyjne
połączenie eksonów –
połączenie eksonów –
i jest to
i jest to
splicing czyli składanie
splicing czyli składanie
eksonów
eksonów
,
,
który zapewnia ciągłość sekwencji
który zapewnia ciągłość sekwencji
kodujących będących matrycą do
kodujących będących matrycą do
syntezy białka.
syntezy białka.
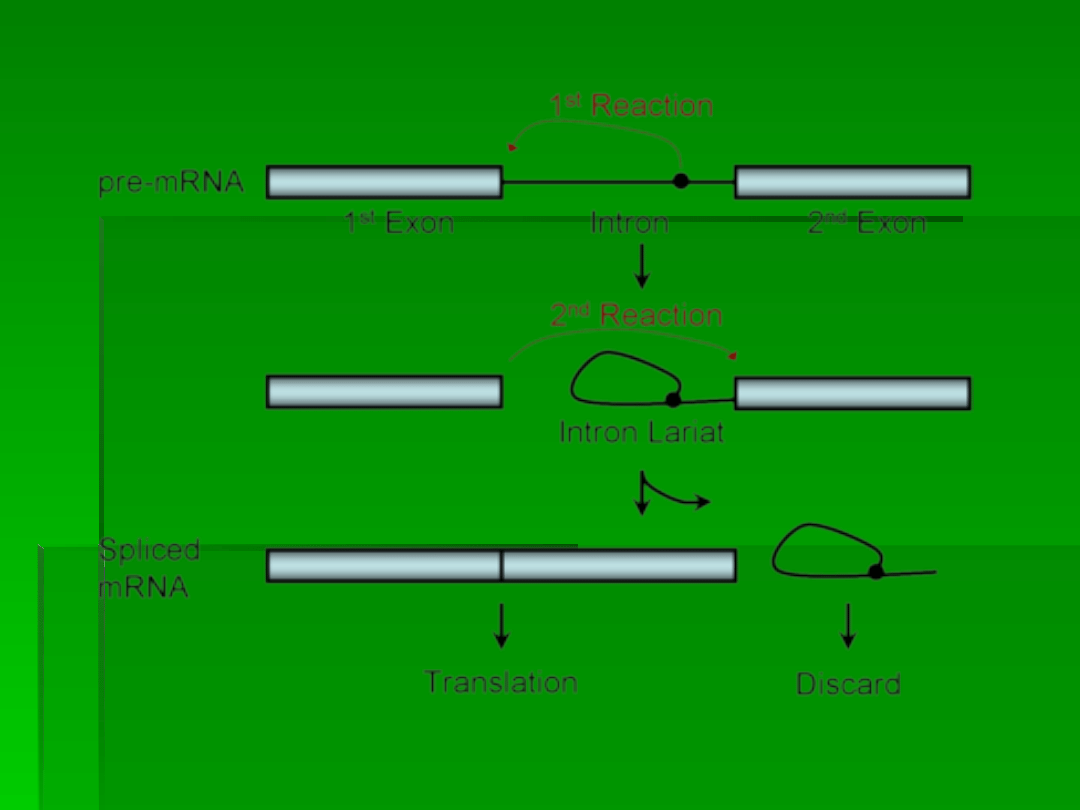

Splicing
Splicing
jest katalizowany przez grupę
jest katalizowany przez grupę
niskocząsteczkowych białek
niskocząsteczkowych białek
zwanych
zwanych
małymi jądrowymi
małymi jądrowymi
rybonukleoproteinami
rybonukleoproteinami
snoRNP=snRNP
snoRNP=snRNP
(
(
small nuclear
small nuclear
ribonucleoproteins
ribonucleoproteins
).
).

snoRNP
snoRNP
Składają się one z małych jądrowych
Składają się one z małych jądrowych
RNA (snRNA) bogatych w U
RNA (snRNA) bogatych w U
połączonych z białkami.
połączonych z białkami.
Są różne grupy snRNP: U1, U2, U4,
Są różne grupy snRNP: U1, U2, U4,
U5, U6.
U5, U6.
Różnią się m. in. miejscami
Różnią się m. in. miejscami
wiązania, tzn. albo z sygnałem na 5’
wiązania, tzn. albo z sygnałem na 5’
albo z sekwencją rozgałęziającą itd.
albo z sekwencją rozgałęziającą itd.

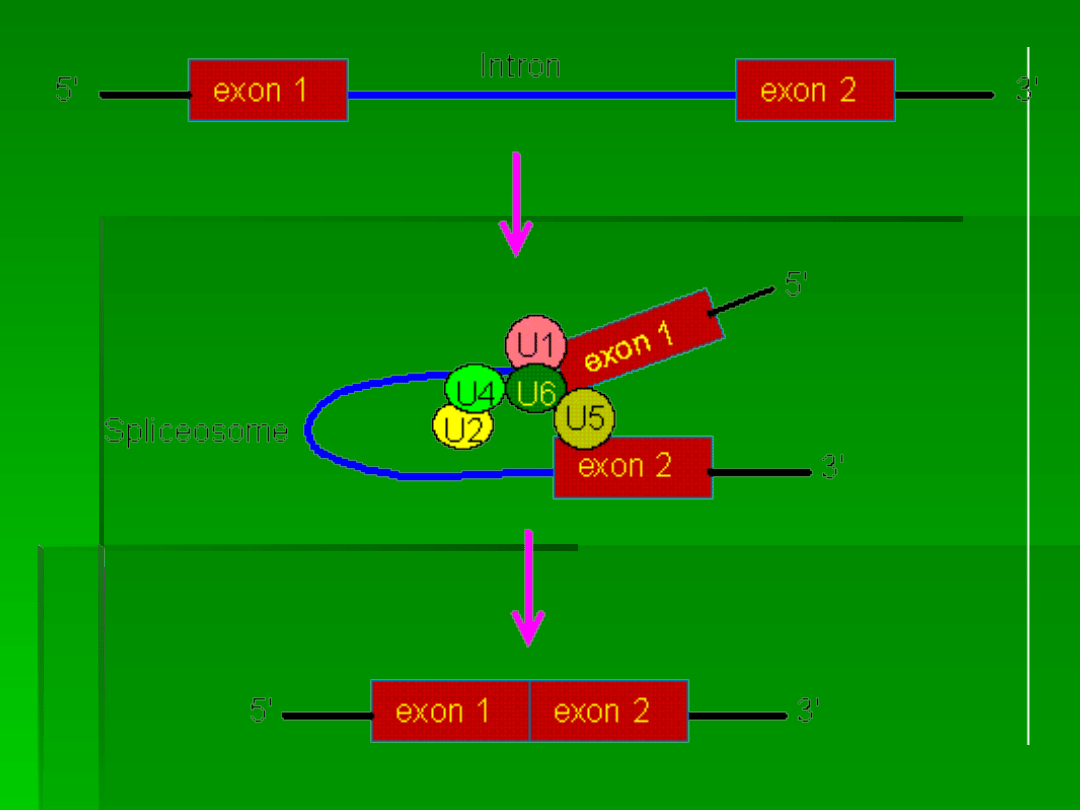
Kompleks pre-mRNA z snRNP
Kompleks pre-mRNA z snRNP
nazywany jest
nazywany jest
splicesomem
splicesomem
(spajsosomem
(spajsosomem
).
).
Kompleks ten przeprowadza
Kompleks ten przeprowadza
sfałdowanie i przyjęcie przez pre-
sfałdowanie i przyjęcie przez pre-
mRNA konformacji umożliwiającej
mRNA konformacji umożliwiającej
zajście splicingu,
zajście splicingu,
ponadto katalizuje reakcje rozcięcia
ponadto katalizuje reakcje rozcięcia
i ligacji prowadzące do wycięcia
i ligacji prowadzące do wycięcia
intronu i połączenia eksonów.
intronu i połączenia eksonów.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
Wyszukiwarka
Podobne podstrony:
7 WYKŁAD VII Ekspresja informacji genetycznej
Ekspresja informacji genetycznej-transkrypcja i translacja, NAUKA
REGULACJA EKSPRESJI INFORMACJI GENETYCZNEJ
Biotechnologia -W, Markery, Inżynieria genetyczna - zespół technik pozwalających na badanie procesów
ekspresja informacji genetycznej Eukaryota
Ekspresja informacji genetycznej-transkrypcja i translacja, NAUKA
Wykład VII, politechnika infa 2 st, Projektowanie Systemów Informatycznych
Wykład VII, politechnika infa 2 st, Projektowanie Systemów Informatycznych
kataster wykłady VII sem, „Ewidencja gruntów i budynków jest to jednolity dla kraju, systemat
Wykład VII, politechnika infa 2 st, Projektowanie Systemów Informatycznych
7 Wyklad VII WSAIB Narzędzia informatyczne
WYKŁAD VII
Wykład VII hazard, realizacja na NAND i NOR
Microsoft PowerPoint Wyklad 1 Wstep do informatyki i
Microsoft PowerPoint Wyklad 2 Wstep do informatyki i
Technologie informacyjne - wykład 2-4, AM SZCZECIN, Informatyka - Wykłady
wykład VII, LEŚNICTWO SGGW, MATERIAŁY LEŚNICTWO SGGW, Transport, TRANSPORT
Wykład VII, Studia Biologia, Mikrobiologia, wykłady z ogólnej
więcej podobnych podstron