
KLONOWANIE DNA

Technika klonowania
Polega na wprowadzeniu do komórek biorcy ściśle
określonego odcinka DNA dawcy zawierającego
jeden lub kilka genów w celu zmienienia
właściwości biorcy. Fragment DNA, który ma być
wprowadzony do komórek biorcy musi zostać
połączony odpowiednim wektorem (nośnikiem). W
efekcie otrzymuje się klony, czyli zbiór
identycznych organizmów. Narzędzia
wykorzystywane w klonowaniu to:
szczepy bakteryjne,
wektory,
enzymy.

Pierwszym etapem pracy z materiałem genetycznym
jest izolacja DNA, która polega na oddzieleniu DNA od
innych struktur komórkowych oraz od cząsteczek RNA i
białek (histonów). DNA można uzyskać z prawie
każdego materiału biologicznego (np. z krwi, nasienia,
plwociny, kału, bakterii). Do niedawna w celu izolacji
DNA wykorzystywano metodę z użyciem fenolu i
chloroformu, a obecnie coraz częściej korzysta się z
komercyjnych zestawów. Wyizolowany, totalny kwas
nukleinowy powinien być albo hydrolizowany
odpowiednimi enzymami restrykcyjnymi albo
bezpośrednio namnożony za pomocą PCR. Dalszym
procesem, w zależności od założeń, jest klonowanie w
wektorach i transformacja komórek gospodarza.
Badanie powinno zakończyć się analizą i/lub ekspresją
odpowiednich genów/fragmentów DNA w komórkach
docelowych.

Do klonowania genów potrzebne są :
Fragment DNA, który zamierzamy klonować. Najczęściej jest to
krótki odcinek DNA powstały po trawieniu odpowiednimi
enzymami restrykcyjnymi (posiada lepkie lub tępe końce) lub
mechanicznym fragmentowaniu (tępe końce lub lepkie
niespecyficzne) bądź produkt PCR.
Wektor, zapewniający powielenie obcego DNA w komórkach
gospodarza. Stosuje się generalnie dwa rodzaje wektorów:
plazmidowe i pochodzenia wirusowego. Wśród tych drugich dużą
popularność zyskały prokariotyczne fagi Escherichia coli
dwuniciowe (bakteriofag λ) i jednoniciowe (bakteriofagi M13 i f1)
oraz eukariotyczny wirus SV40.
Gospodarz, zwykle jest to szczep bakteryjny, drożdżowy lub
eukariotyczna linia komórkowa o odpowiednio zmodyfikowanym
dla celów inżynierii genetycznej i ściśle określonym genotypie.

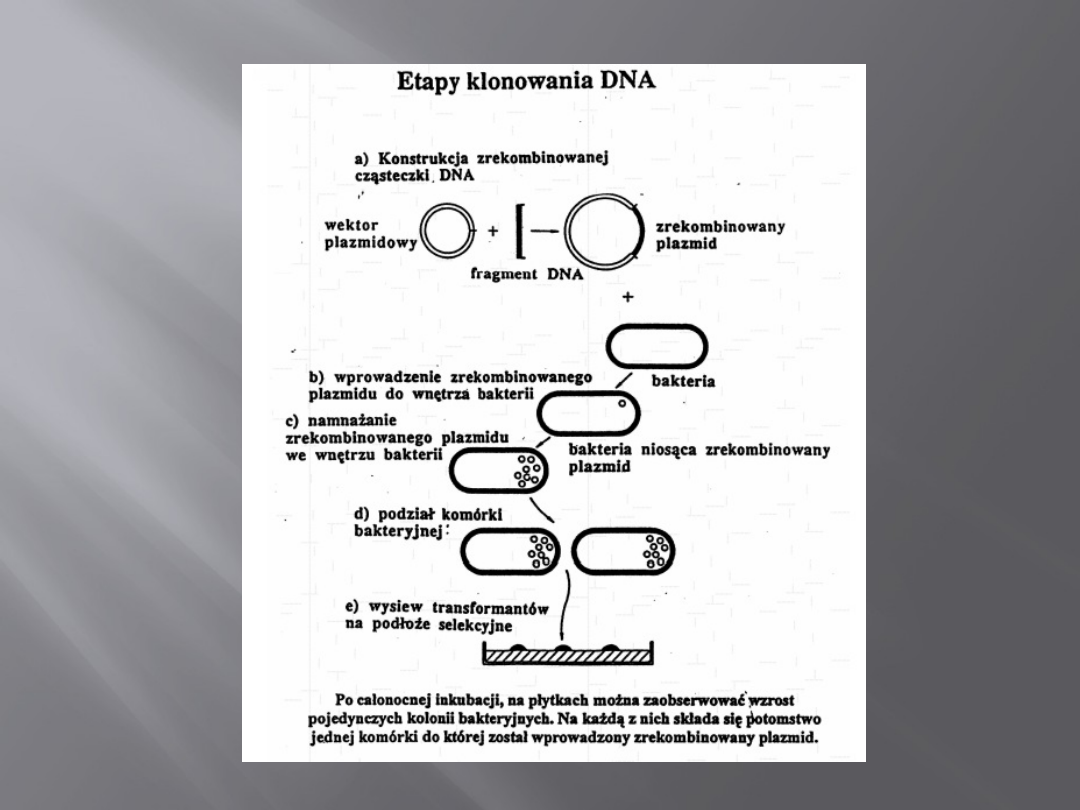
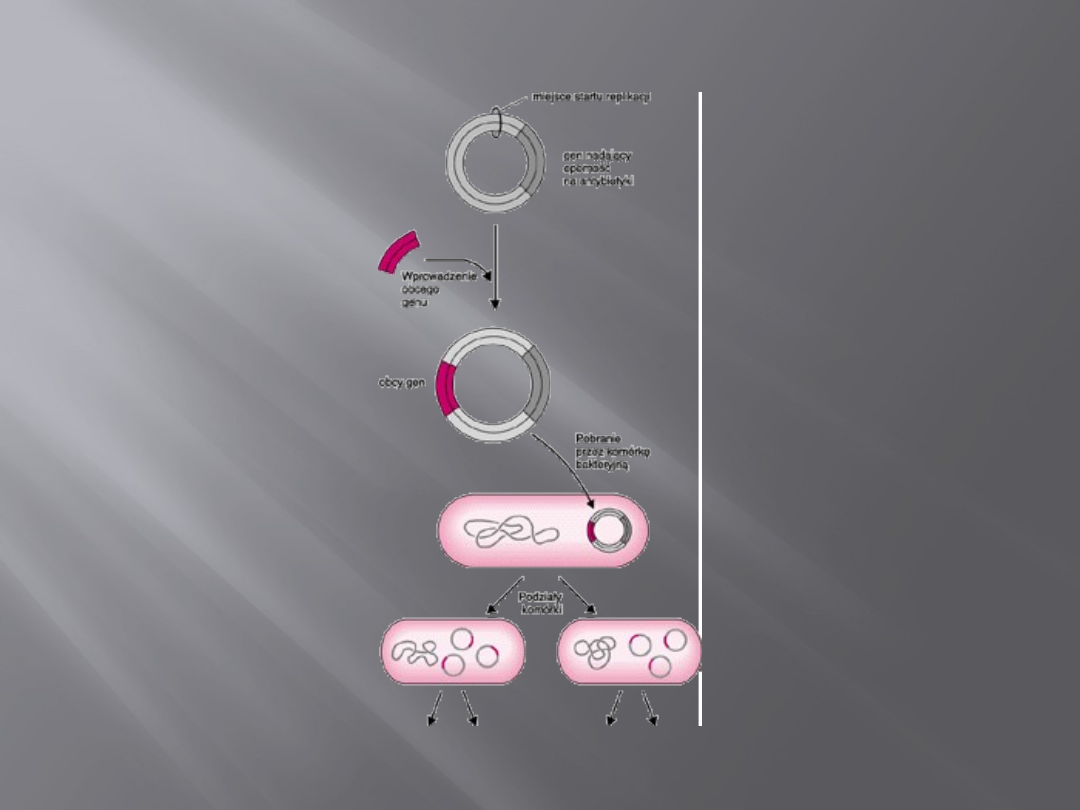
Schemat klonowania
DNA przeznaczony do klonowania może być wyizolowany
z komórek dawcy lub zsyntetyzowany chemicznie (PCR).
Następnie oba końce klonowanego fragmentu DNA
zostają pocięte przy użyciu enzymów restrykcyjnych.
Otrzymane fragmenty miesza się następnie in vitro w
odpowiednim stosunku z wektorem uciętym tymi samymi
enzymami restrykcyjnymi. Użycie odpowiednich
enzymów restrykcyjnych powoduje, że klonowany
fragment DNA i ucięty wektor posiadają komplementarne
do siebie końce (końce lepkie lub tępe ).

Wektor i fragment DNA zostają połączone przy
udziale ligazy DNA faga T4.
Uzyskane w ten sposób hybrydowe cząsteczki DNA
wprowadza się metodą transformacji do
odpowiednio przygotowanego biorcy. Biorca
powinien zapewnić ekspresję klonowanego DNA,
jak również zwiększyć liczbę kopii
zrekombinowanego wektora.
Uzyskane klony selekcjonuje się na odpowiednich
podłożach (zwykle zawierającymi antybiotyk).
Najczęściej stosowanym gospodarzem dla obcego
DNA są komórki bakteryjne.

Klonowanie umożliwia wyizolowanie z heterogennej
mieszaniny DNA fragment DNA będący
przedmiotem naszego zainteresowania i jego
powielenie, tzn. amplifikację w np. komórce
mikroorganizmu. Użyty do klonowania DNA nie
musi być pochodzenia bakteryjnego, z kolei musi
zostać połączony z wektorem, który ma zdolność do
autonomicznej replikacji w mikroorganizmie. Wybór
eukariotycznego gospodarza wiąże się z użyciem
wektora zdolnego do replikacji w tym organizmie.
Hodowanie bakterii niosących zrekombinowane
DNA nie stanowi większego problemu, umożliwia to
wyizolowanie dużej ilości zarówno specyficznego
DNA jak i produktu białkowego, które można
poddać dalszym badaniom.

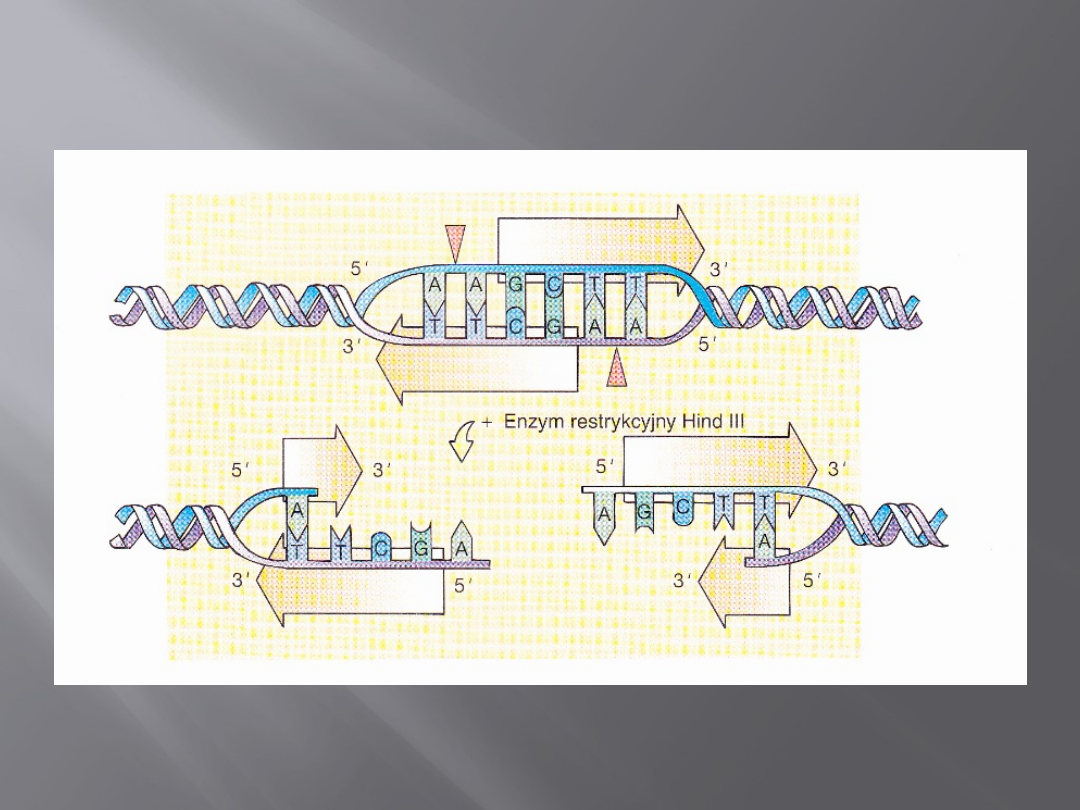
Enzymy restrykcyjne
Enzymy restrykcyjne (endonukleazy restrykcyjne, restryktazy) to
enzymy bakteryjne rozpoznające i hydrolizujące DNA w określonych
miejscach. Restryktazy są zaangażowane w obronę komórki
bakteryjnej przed wirusami. Znanych jest kilka klas restryktaz.
Największe znaczenie maja enzymy II klasy. Dotychczas
wyizolowano kilkaset enzymów restrykcyjnych, których nazwy
pochodzą od gatunku bakterii, np. Eco RI wyizolowano z Escherichia
coli ze szczepu RY 13, a Sau3A z Staphylococcus aureus. Oprócz
restryktaz, istnieje szereg innych enzymów wykorzystywanych w
inżynierii genetycznej. Należą do nich:
ligazy pozwalające na łączenie fragmentów DNA,
egzonukleazy umożliwiające odpowiednią "obróbką" końców w
modyfikowanych fragmentach DNA,
polimerazy, które są odpowiedzialne za powielanie
(amplifikacje) odpowiednich odcinków DNA (PCR - reakcja
łańcuchowej polimerazy).

Enzymy restrykcyjne przecinają DNA tylko w tych
obszarach , w których występują specyficzne
sekwencje zasad. Jeden enzym restrykcyjny może
rozpoznawać i przecinać cząsteczkę w rejonie
sekwencji 5`-AAGCTT-3`, podczas gdy inny
przecina ją w rejonie sekwencji 5’-GATC-3’.
Większość z nich rozpoznaje sekwencje
palindromowe sześcio- (NdeI, BamHI, PstI) lub
czteronukleotydowe (AluI). Fragmenty DNA
powstające w wyniku działania takich enzymów
wyposażone są w jednoniciowe końce, które są w
stosunku do siebie komplementarne. Nazywa się je
lepkimi końcami, ponieważ mogą łączyć się
wiązaniami wodorowymi z komplementarnymi
jednoniciowymi końcami innych fragmentów
cząsteczki DNA przeciętej przez ten sam enzym.

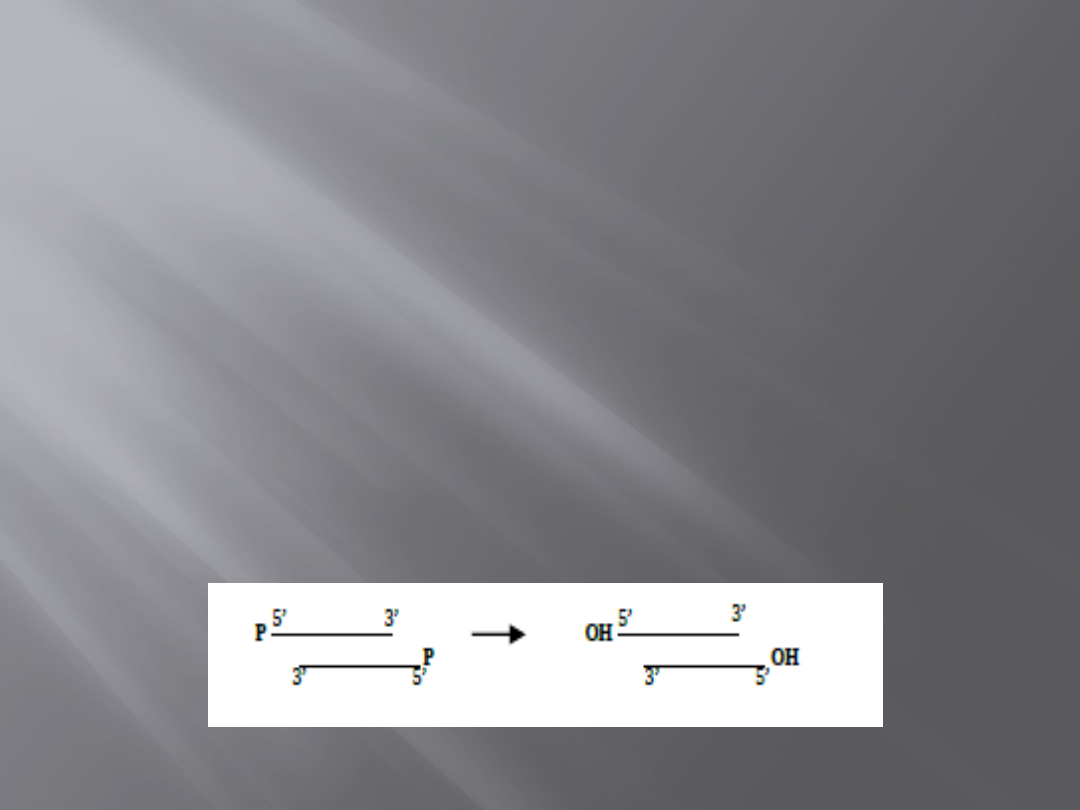
Fosfataza alkaiczna
Jest to enzym modyfikujący końce 5’ DNA.
Usuwa grupę fosforanową z nici DNA i RNA
z deoksyrybonukleotydotrójfosforanów i
rybonukleotydytrójfosforanów w celu
uniemożliwienia autoligacji wektora użytego
do klonowania oraz do znakowania końców
5’ za pomocą kinazy polinukleotydowej T4.
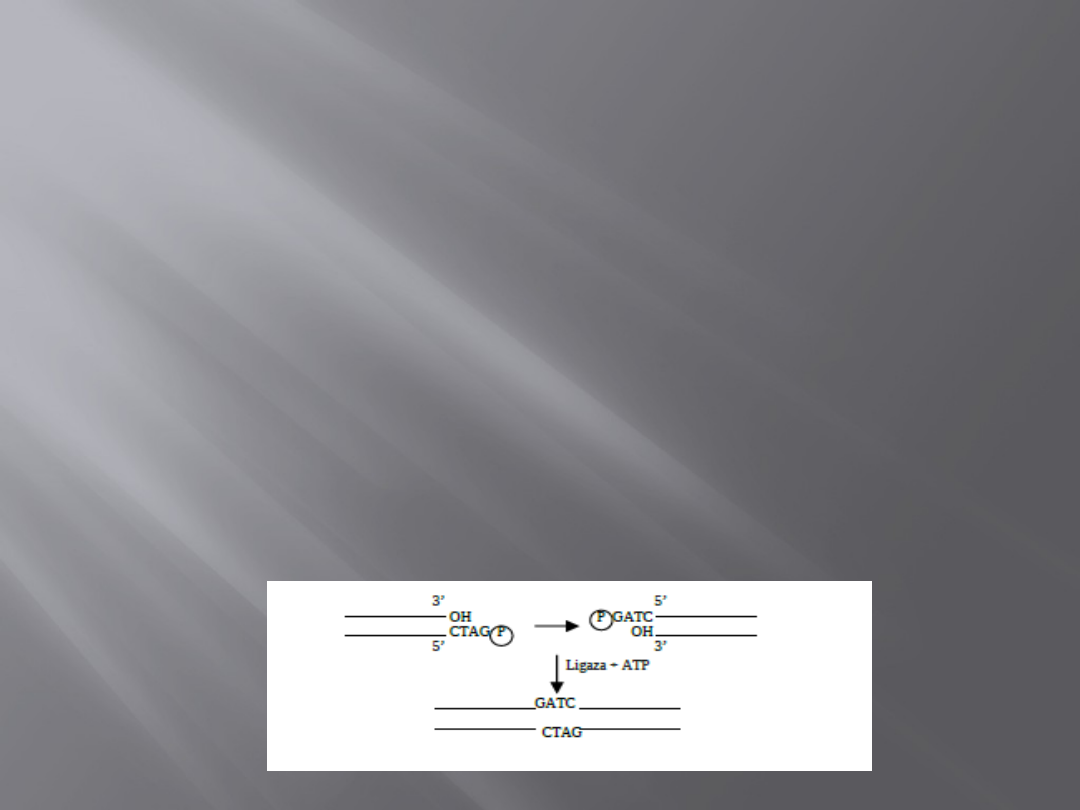
Ligaza DNA
Katalizuje odtwarzanie fosfodiestrowego
pomiędzy grupą fosforanową znajdującą się
na końcu 5’ fragmentu restrykcyjnego a
grupą hydroksylową końca 3’ (wcześniej te
wiązania były niszczone przez enzymy
restrykcyjne). Reakcja ta jest zależna od
ATP. In vitro enzym ten jest niezbędny w
procesie replikacji (łączenie fragmentów
Okazaki) oraz naprawie DNA.

Polimeraza DNA
Enzym ten katalizuje syntezę DNA podczas replikacji lub
naprawy DNA. Synteza ta polega
na polimeryzacji deoksyrybonukleotydów przez
wytwarzanie wiązań fosfodiestrowych między
nimi. Wymaga matrycy, w formie jednoniciowego DNA
lub RNA, z krótkim obszarem dwuniciowym. Odcinek
dwuniciowy powstaje przez przyłączenie się do
jednoniciowej matrycy krótkiego komplementarnego do
matrycy odcinka DNA lub RNA, zwanego primerem lub
starterem (zwykle ma długość od kilku do ok. 20
nukleotydów). Działanie polega właściwe na wydłużaniu
startera przez dobudowywanie nukleotydów
komplementarnych do matrycy na końcu 3' startera, a
następnie dobudowywaniu kolejnych nukleotydów.

Wektory
Wektor to cząsteczka DNA (wirus, plazmid,
kosmid, lub sztuczny chromosom), która służy do
wprowadzenia obcego materiału genetycznego
do innego gospodarza. Wektor, w który
umieszcza się stosunkowo krótki fragment DNA,
zawierający badany gen lub sekwencje,
nazywany jest rekombinowanym DNA.
Wektory stosuje się do klonowania i amplifikacji
sekwencji DNA, badania mechanizmów ekspresji
DNA, wprowadzania genów do komórek
zwierzęcych (transfekcja) i bakteryjnych
(transformacja).

Jednym z ważniejszych
problemów w biotechnologii
jest pojemność wektora,
która decyduje o wielkości
wprowadzanego DNA
(wielkości klonowanego
insertu). Plazmidy, które są
najczęściej stosowane jako
wektory, maja najmniejsza
pojemność; w przypadku
klonowania
dłuższych insertów stosowane
są bakteriofagi, kosmidy,
sztuczne chromosomy
bakteryjne
(BAC) E. coli lub sztuczne
chromosomy drożdżowe (YAC)
Saccharomyces cerevisiae

Wektory plazmidowe
Plazmid to pozachromosomowa, z reguły kolista cząsteczka
DNA zdolna do samodzielnej replikacji, która występuje u
prokariota i niektórych organizmów eukariotycznych. Plazmidy
pełnią w komórkach funkcje pomocniczych chromosomów i są
przenoszone w trakcie procesu koniugacji miedzy komórkami
bakteryjnymi. Jednym z najlepiej poznanych plazmidów jest
plazmid F z E. coli. Jest to plazmid typu koniugacyjnego,
ponieważ jest transferowany z komórki donora do akceptora.
Szczepy bakterii zawierają różna liczbę plazmidów.
Plazmidy ulegają replikacji niezalenie od macierzystego
genomu bakterii, także w komórkach gospodarzy należacych
do innych gatunków. Wśród najistotniejszych cech fenotypowych
kodowanych przez plazmidowe DNA wymienić można: oporność
na antybiotyki, jony metali ciężkich, produkcje antybiotyków,
kolicyn, prostych węglowodanów, niektórych toksyn itp..

Wektory oparte na plazmidach są stosowane jako narządzia
do poznawania genomów komórek roślin i zwierząt. Wektor
plazmidowy musi zawierać region początku replikacji (ori),
który pozwala na niezalene namnażanie się w komórkach.
Należy jednak podkreślić, że proces niezależnego namnażania
wektora zachodzi z udziałem polimerazy i innych składników
cytofizjologii komórki gospodarza. Drugim ważnym miejscem
w wektorze plazmidowym jest wielokrotne miejsce klonowania
(wiele miejsc rozpoznawanych przez różne enzymy
restrykcyjne, MCS). Jest to miejsce, do którego wprowadza się
badany fragment DNA, czyli insert. Wektor plazmidowy musi
posiadać promotor. Niektóre wektory plazmidowe wymagają
sekwencji wiążącej rybosom (RBS) i kodonu inicjacji translacji,
a inne - sekwencji umożliwiającej łatwiejsze oczyszczenie na
drodze chromatografii (metka HisTag). Wektory, które słżą
do ekspresji klonowanych fragmentów DNA w komórkach,
nazywamy plazmidowymi wektorami ekspresyjnymi.
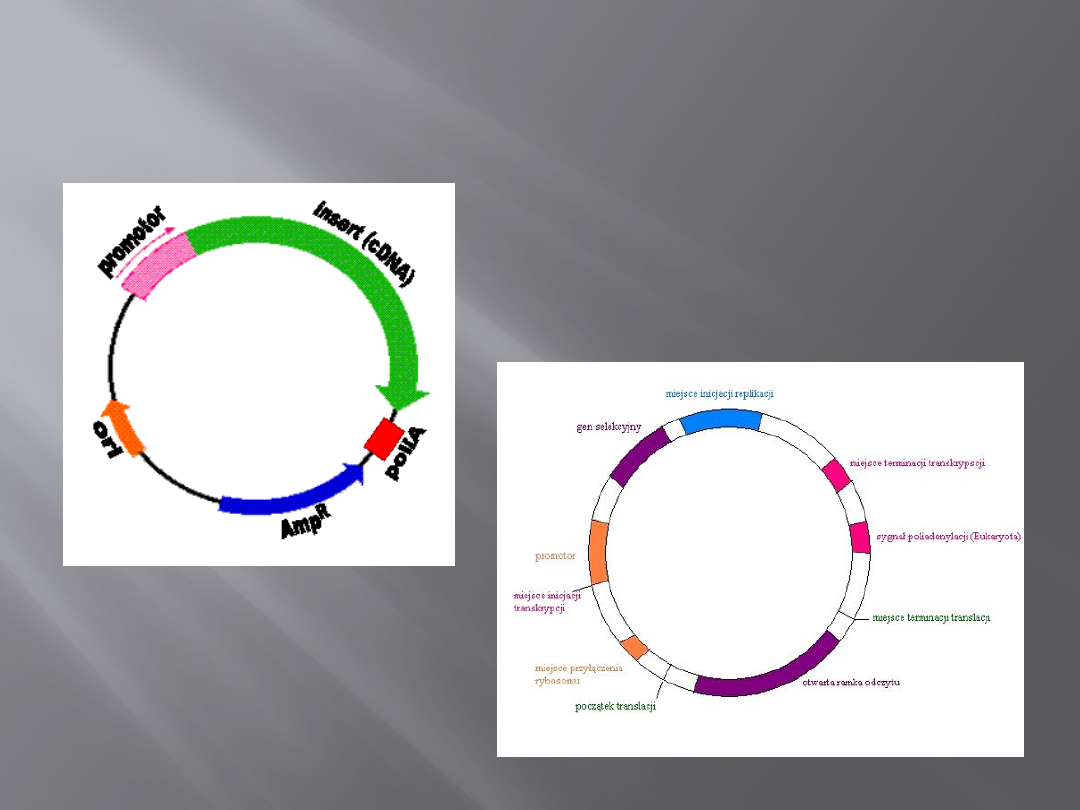
Plazmidowy wektor
ekspresyjny

Cechy dobrego wektora
Obecność jednego lub kilku genów
markerowych, służących do wyróżniania i
selekcji transformantów, czyli komórek, które
pobrały zrekombinowany wektor (głównie są to
geny odporności na antybiotyk np. ampicylinę,
tetracyklinę, chloramfenikol, kanamycynę).
Powinien być dobrze scharakteryzowaną pod
wzglądem fizycznym i chemicznym cząsteczką,
łatwa do oczyszczenia.
Nie powinien zawierać genów, których
rozprzestrzenianie się może stanowić
zagrożenie dla życia lub zdrowia ludzi, zwierząt
czy roślin.

Modyfikacje wektorów
plazmidowych
Redukcja masy cząsteczkowej. Im mniejszy wektor, tym
większa jego pojemność oraz prościej nim manipulować (np.
tworzyć mapę restrykcyjną klonowanego DNA)
Usunięcie genów warunkujących możliwość transferu
zrekombinowanego plazmidu do innych bakterii –
przekazywanie wektora będzie odbywać się tylko w pionie
( z komórek macierzystych do potomnych)
Włączenie w miejscu, gdzie ma być wklonowane obce DNA
syntetycznego oligonukleotydu z sekwencjami
rozpoznawanymi przez wiele enzymów restrykcyjnych –
miejsce wielokrotnego klonowania (ang. Multiple Cloning
Sites – MCS)
Wprowadzenie silnego promotora przed miejscem MCS. Jego
obecność zapewnia wydajną ekspresję klonowanego genu.

MCS
Multiple Cloning Sites umożliwia bezpośrednie
wstawienie fragmentów klonowanego DNA, które
powstały przez trawienie jednym z wielu, lub
(najczęściej) dwoma różnymi enzymami. Oznacza to,
że wektor, który zostanie wybrany do klonowania
musi w miejscu MCS posiadać miejsca restrykcyjne
dla takich samych enzymów, jakimi zostanie
potraktowany fragment obcego DNA, który ma być
do niego wprowadzony. Dzięki niemu łączone za
pomocą ligazy będą komplementarne. Klonowanie
tylko w miejscu MCS gwarantuje, że przy ekspresji
klonowanego genu będzie zachowana ramka
odczytu.
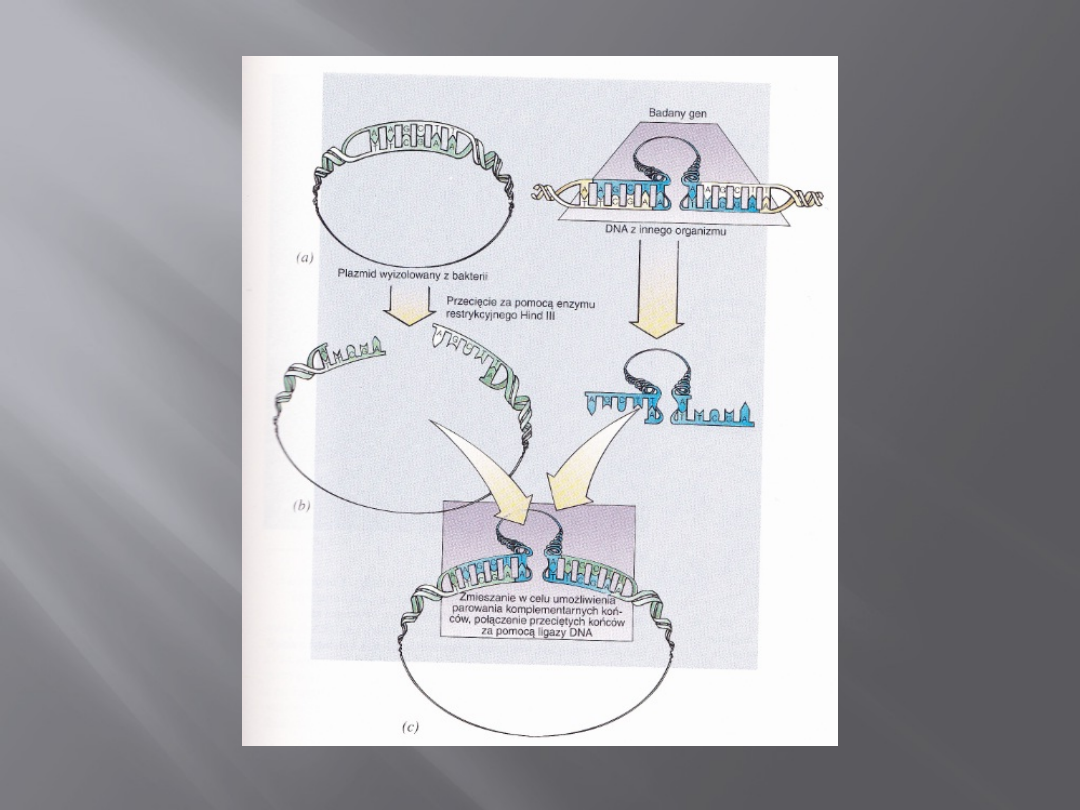

Wektory bakteriofagowe
Wektory bakteriofagowe pozwalają na wklonowanie większego
insertu ni wektory plazmidowe; np. do cząsteczki faga można
wprowadzać insert o wielkości 25 kpz. Na podstawie faga
opracowano szereg innych wektorów, zwanych wektorami
wymiennymi (np. EMBL3 lub DASH). Jako wektory w pracach z E.
coli używa się takie fagi pałeczkowate, np. M13. Cząsteczki tego
wirusa zawierają kolisty, jednoniciowy DNA. Po wniknięciu faga
M13 do komórki bakteryjnej syntetyzowana jest komplementarna
nic i fagowy DNA powiela się już jako dwuniciowy kolisty DNA; ta
forma replikacyjna (RF) występuje w około 100 kopiach na
komórkę. Ponadto, oprócz formy RF, jednocześnie powstają
opakowywane kapsydem cząsteczki jednoniciowego DNA, które są
uwalniane z komórek w kolejnych podziałach bakterii. Użyteczną
cecha wektora M13 jest to, że formę RF mona oczyszczać i
stosować tak jak plazmid, a jednocześnie można izolować DNA
wektora z pożywki w formie jednoniciowej.
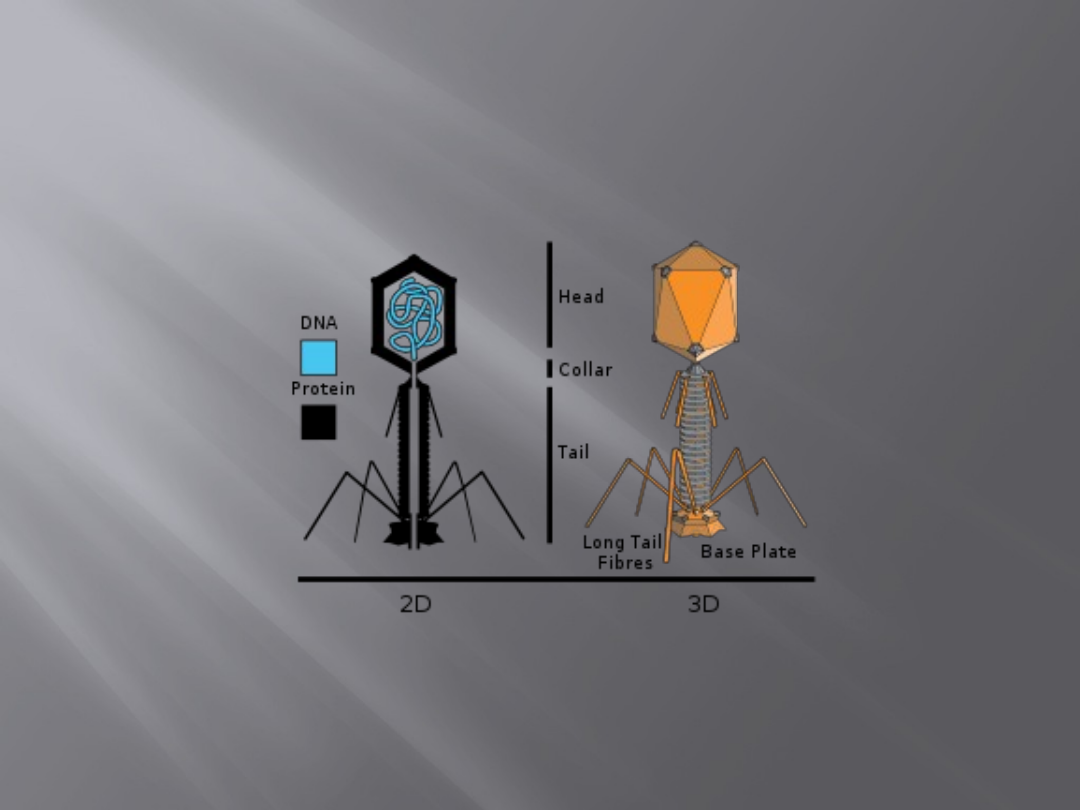
Bakteriofag λ

Kosmidy
Kosmidy są sztucznie stworzonymi wektorami, powstałymi z
połączenia plazmidu i sekwencji cos faga (sekwencja
odpowiedzialna za cyrkulizację DNA). Kosmidy z
wprowadzonym insertem są pakowane w kapsydy i
wykorzystywane do zakażenia komórek bakteryjnych. W
przeciwieństwie do faga , kosmidy nie niszczą zainfekowanych
komórek. Najprostszym wektorem kosmidowym jest typowy
plazmid z miejscem ori i markerem selekcyjnym, zawierającym
dodatkowo sekwencje cos i odpowiednie miejsce restrykcyjne
do klonowania. Po trawieniu (cieciu) wektora odpowiednim
enzymem restrykcyjnym i zligowaniu z docelowym insertem,
kosmid pakowany jest do cząsteczek fagowych. Po
wprowadzeniu większego insertu do kosmidu wydłuża się czas
jego replikacji oraz zmniejsza się liczba jego kopii. Kosmidy
umożliwiaja klonowanie długich fragmentów DNA (ok. 45 kpz).


Sztuczne chromosomy
drożdżowe (YAC)
Wektor YAC jest wektorem bifunkcjonalnym, ponieważ jest zdolny
do powielania w komórkach bakterii i drożdży. S. cerevisiae to
organizm dobrze scharakteryzowany metodami fizjologicznymi i
biochemicznymi, który jest hodowany w warunkach in vitro na dużą
skalę, m.in. dla potrzeb przetwórstwa żywności, ponieważ nie
wytwarza toksyn, a hodowle nie ulegają zakażeniom wirusowym.
Sekwencje „drożdżowe” centromeru (CEN 4), telomeru (TEL) i
miejsca początku replikacji (ARS) zostały wyizolowane i połączone z
plazmidami skonstruowanymi dla E. coli. Sekwencja TEL stanowi
część DNA, która w komórkach drożdży wydłużana jest przez enzym
telomerazę. Sekwencja CEN 4 funkcjonuje prawidłowo podczas
segregacji chromosomów S. cerevisiae w procesie mitozy.
Sekwencja ARS pełni role miejsca początku replikacji, podobnie jak
sekwencja ori u bakterii. Chociaż w wektorach YAC można klonować
bardzo długie odcinki DNA, często okazuje się, że klonowane inserty
zawierają nieciągłe sekwencje, przez co są niestabilne.

Sztuczne chromosomy
bakteryjne (BAC)
Wektory BAC skonstruowano, aby uniknąć
problemów związanych ze stosowaniem wektorów
YAC. Do wektorów BAC mona wprowadzić insert o
długości około 100-500 kpz. W porównaniu do
wektorów YAC, wektory BAC są bardziej stabilne,
łatwiej się nimi transformuje komórki E. coli oraz
łatwiej je namnażac i izolować z komórek
bakteryjnych. Wektory BAC zbudowane są na
bazie plazmida F; zawierają geny istotne dla
replikacji i utrzymywania się w komórce bakterii
E. coli. Wektory BAC są obecnie używane w
projektach mapowania genów.

Przygotowanie i transformacja
komórek kompetentnych
Wprowadzenie zrekombinowanych cząsteczek do wnętrza
komórek gospodarza stanowi jeden z najważniejszych etapów
klonowania DNA. Wprowadzone do wnętrza komórki DNA ulega
często degradacji, ale może się zdarzyć, że nie zostanie
zniszczone. Dotyczy to głównie plazmidów, które posiadają
miejsce startu replikacji (ang. origin), mogą autonomicznie
utrzymywać się we wnętrzu komórki bakteryjnej. Pobranie
plazmidu wiąże się z nabyciem przez komórkę nowych cech.
Wiele bakterii (np. Bacillus czy Hemophilus) posiada naturalną
zdolność do pobierania cząsteczek DNA ze środowiska, w
którym żyją. Często używana w biologii bakteria Escherichia
coli może być w ten stan wprowadzona na drodze indukcji
chemicznej (np. przez traktowanie komórek chlorkiem wapnia
tzw. metoda chlorkowa).
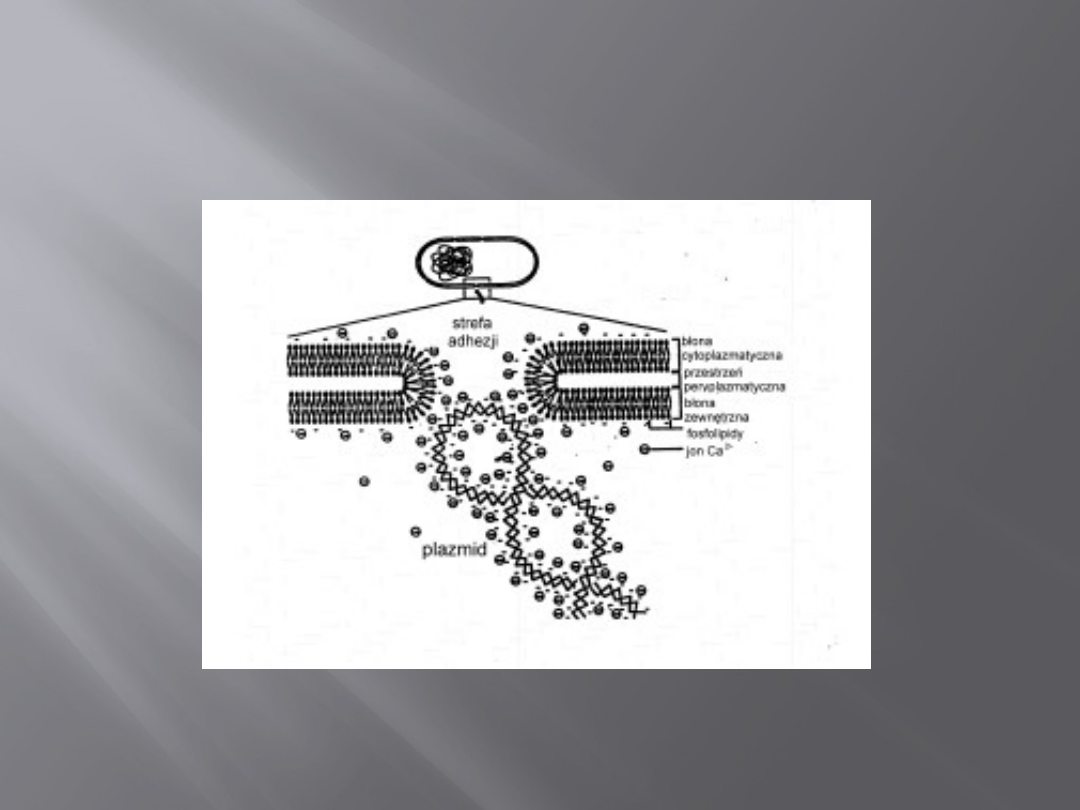

Inne metody wprowadzania DNA
do komórki
Liposomy to zamknięte pęcherzyki lipidowe, w których umieszcza się
zrekombinowany DNA; zapewnia to ochronę przed nukleazami (enzymy
hydrolizujące kwasy nukleinowe). Liposomy mogą być użyte do przenoszenia
DNA przez błonę komórkowa. Niestety wydajność tej metody jest niska.
Natomiast zaleta tej metody jest fakt braku odpowiedzi immunologicznej w
komórkach zwierzęcych. Jednakże jest to metoda pracochłonna i duże
fragmenty DNA nie zawsze udaje się właściwie zapakować do liposomów bez
uszkodzeń.
Elektroporacja to proces zachodzący w błonie komórkowej pod wpływem
wyładowań pól elektrycznych (milisekundy), który powoduje odwracalne
zmiany w błonie komórkowej.
Mikrowstrzeliwanie polega na wprowadzeniu DNA opłaszczonego na
mikroskopijnych kulkach złota lub wolframu za pomocą urządzenia zwanego
„armatka genowa”.
Mikroiniekcja stosowana jest do wprowadzania obcego DNA do komórek
zwierząt; w tym celu trzeba wyizolować zygotę lub wczesny embrion i przy
pomocy szklanej mikropipety wprowadzić obcy DNA do jadra komórki. W celu
dalszego rozwoju ztransformowaną komórkę umieszcza się w zastępczej
matce. Zaletą tej metody jest wysoka wydajność, lecz wadą - pracochłonność.

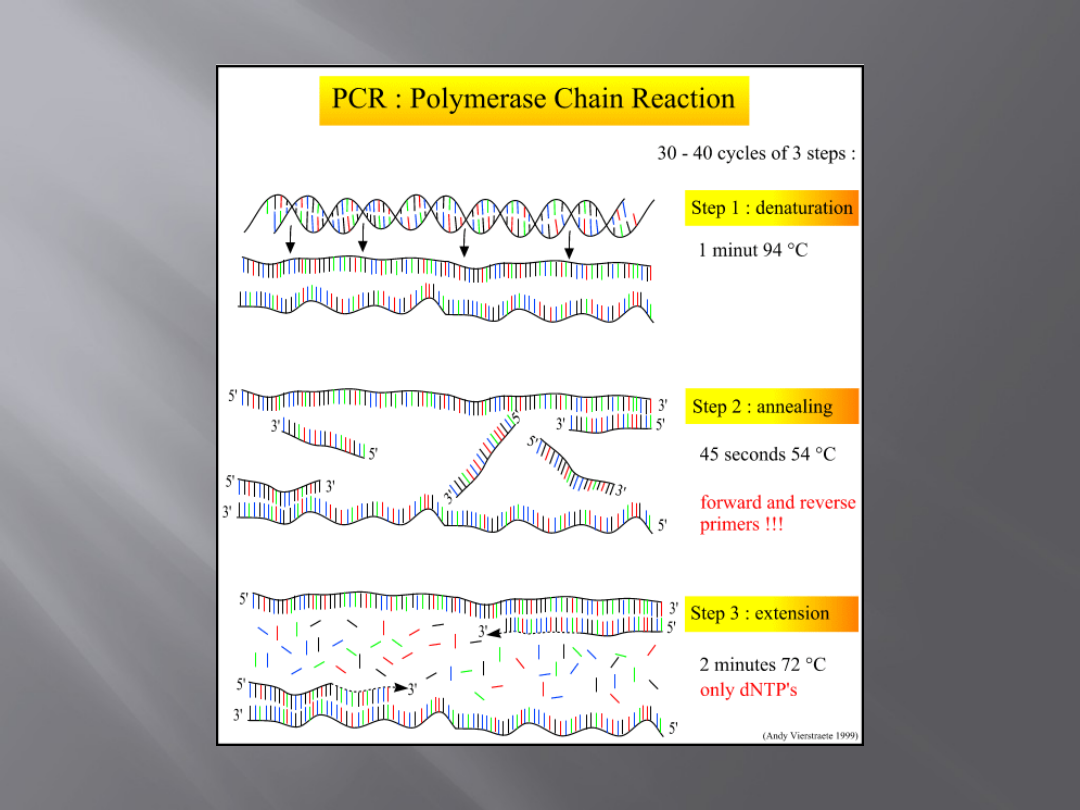
Amplifikacja DNA in vitro metodą
łańcuchowej reakcji polimerazy
(PRC)
Metoda ta służy do amplifikacji segmentu
DNA położonego pomiędzy regionami o
znanej sekwencji. W reakcji tej używane jako
primery (startery) są dwa oligonukleotydy,
komplementarne do sekwencji położonych
na przeciwległych niciach matrycowego
DNA, flankujące rejon DNA, który ma
podlegać amplifikacji. DNA jest
syntetyzowane w serii reakcji
katalizowanych przez polimerazę DNA.

Etapy reakcji
Denaturacja matrycowego DNA w temperaturze
94-95
0
C, w obecności czterech rodzajów dNTP
oraz dużego nadmiaru primerów.
Przyłączenie primerów do sekwencji
homologicznych w matrycowym DNA, co
następuje po ochłodzeniu do temperatury 45-
65
0
C,
Wydłużanie primerów przez polimerazę DNA w
temperaturze 72
0
C, co w rezultacie pozwala na
syntezę fragmentu DNA.

Zastosowanie PRC
Amplifikacja fragmentu DNA – uzyskanie
dużej ilości określonego fragmentu DNA np.
do klonowania
Diagnostyka molekularna – wykrywanie
patogenów w próbkach klinicznych,
identyfikacja próbek w medycynie sądowej
(możliwa z bardzo niewielkiej próbki, np.
pojedynczego włosa), analiza mutacji w
chorobach nowotworowych
Synteza specyficznych sond
Tworzenie bibliotek genomowych
Document Outline
- Slide 1
- Technika klonowania
- Slide 3
- Slide 4
- Schemat klonowania
- Slide 6
- Slide 7
- Slide 8
- Enzymy restrykcyjne
- Slide 10
- Slide 11
- Slide 12
- Fosfataza alkaiczna
- Ligaza DNA
- Polimeraza DNA
- Wektory
- Slide 17
- Wektory plazmidowe
- Slide 19
- Slide 20
- Plazmidowy wektor ekspresyjny
- Cechy dobrego wektora
- Modyfikacje wektorów plazmidowych
- MCS
- Slide 25
- Wektory bakteriofagowe
- Bakteriofag λ
- Kosmidy
- Slide 29
- Sztuczne chromosomy drożdżowe (YAC)
- Sztuczne chromosomy bakteryjne (BAC)
- Przygotowanie i transformacja komórek kompetentnych
- Slide 33
- Inne metody wprowadzania DNA do komórki
- Slide 35
- Etapy reakcji
- Slide 37
- Zastosowanie PRC
Wyszukiwarka
Podobne podstrony:
6?chy dobrego wektora Klonowanie DNA
Konstrukcja wektora plazmidowaego DNA do klonowania genów i do sekrecji w bakteriach mlekowych
Podstawowe enzymy modyfikujące DNA przydatne do klonowania
Replikacja DNA i choroby związane
Elektroforeza DNA komórkowego BioAut1, BioAut2 i Ch1
DNA Eng2
3 ogolny schemat replikacji i onkogeza DNA wirusowa
klonowanie1bw
Materiał genetyczny, mutacje, systemy naprawy DNA, test Amesa
osteoporoza i dna
Izolacja DNA z komórek prokariotycznych i eukariotycznych
Met. izol. oczysz.DNA dla studentów, Biologia molekularna
1-Kefir chroni przed mutacjami w DNA, ZDROWIE-Medycyna naturalna, Poczta Zdrowie
dna, INNE KIERUNKI, biologia
DNA, Biologia
12 Elektroforeza agarozowa wyizolowanego DNA ?łkowitego oraz produktów PCR
więcej podobnych podstron