lastik” – wyszepta∏
przyjaciel rodziny
do ucha granemu
przez Dustina Hoff-
mana bohaterowi
filmu Absolwent z
1967 roku, stajàc si´ or´downikiem nie
tylko nowoczesnej kariery, ale tak˝e ca∏-
kowicie innego stylu ˝ycia. Gdyby obraz
ten nakr´cono dzisiaj, w epoce rozszy-
frowania ludzkiego genomu, owo ma-
giczne s∏owo brzmia∏oby z pewnoÊcià
„bioinformatyka”.
Naukowcy pracujàcy dla rzàdu oraz
ró˝nych firm komercyjnych przetworzy-
li ju˝ 3 GB danych opisujàcych odczy-
tane sekwencje par zasad A, C, T i G, któ-
re tworzà zapis ludzkiego genomu. Taka
iloÊç danych mog∏aby zape∏niç ponad
2 tys. standardowych dyskietek kompu-
terowych, a przecie˝ to zaledwie wàski
strumyczek z wielkiej rzeki informacji za-
szyfrowanych w ludzkim genomie. Ba-
dacze tworzà obecnie gigantyczne bazy
danych dotyczàce miejsca i czasu akty-
wacji poszczególnych genów, struktury
kodowanych przez nie bia∏ek, ich wza-
jemnego oddzia∏ywania oraz roli, jakà
owe interakcje pe∏nià w ró˝nego rodzaju
chorobach. Dodajmy jeszcze do tego ma-
sowo nap∏ywajàce informacje o geno-
mach tzw. organizmów modelowych, jak
wywil˝na kar∏ówka, zwana muszkà owo-
cowà (Drosophila melanogaster) czy mysz
[ramka na stronie 61], a otrzymamy to, co
Gene Myers, Jr., wiceprezes ds. badaƒ in-
formatycznych w Celera Genomics
w Rockville w stanie Maryland – okreÊla
mianem „tsunami informacyjnego”.
Z maria˝u biologii i informatyki narodzi-
∏a si´ nowa dyscyplina – bioinformatyka
– z zadaniem uporzàdkowania tego cha-
osu danych. Osiàgni´cia w tej dziedzinie
zmieniajà oblicze biomedycyny.
„W ciàgu nast´pnych dwóch lub
trzech lat liczba informacji osiàgnie nie-
prawdopodobny poziom, a ka˝dy mo-
˝e zostaç nimi przyt∏oczony – twierdzi
Myers. – Rozpocznie si´ wtedy wyÊcig
o to, kto najlepiej je wykorzysta. To b´-
dà nieprzebrane bogactwa.”
Mnóstwo firm komercyjnych rywali-
zuje ze sobà, by mieç udzia∏ w tej ˝yle
z∏ota. Jason Reed z bankowej firmy in-
westycyjnej Oscar Gruss & Son z Nowe-
go Jorku oszacowa∏, ˝e w ciàgu pi´ciu
lat wartoÊç obrotów na bioinformatycz-
nym rynku mo˝e wynieÊç oko∏o 2 mld
dolarów. Zebra∏ on informacje o ponad
50 prywatnych i paƒstwowych przed-
si´biorstwach, które oferujà produkty
i us∏ugi bioinformatyczne. Firmy te sta-
rajà si´ dzia∏aç na ró˝nych p∏aszczy-
znach: gromadzà i przechowujà infor-
macje, interpretujà je oraz przeszukujà
bazy danych. Wi´kszoÊç z nich sprzeda-
je dost´p do posiadanych informacji
spó∏kom farmaceutycznym i biotechno-
logicznym, a op∏aty subskrypcyjne wy-
noszà niekiedy miliony dolarów.
Firmy farmaceutyczne tak ch´tnie
ustawiajà si´ w kolejce i p∏acà za tego
typu us∏ugi – albo tworzà w∏asne, rów-
nie kosztowne bazy danych – g∏ównie
dlatego, ˝e bioinformatyka daje mo˝li-
woÊç du˝o szybszego znalezienia bio-
chemicznych obiektów oddzia∏ywania
nowego leku na znacznie wczeÊniej-
szym etapie jego tworzenia, w odró˝-
nieniu od stosowanych dotàd metod.
To zaÊ mo˝e doprowadziç do zmniej-
szenia liczby wymaganych testów kli-
nicznych i znacznie ograniczyç ca∏ko-
wite koszty jego produkcji. Powinno
tak˝e przynieÊç firmom farmaceutycz-
nym dodatkowe zyski, bowiem gdy
tworzenie nowego medykamentu trwa
krócej, jest on wprowadzany na rynek
wczeÊniej i przez d∏u˝szy czas pozosta-
je obj´ty ochronà patentowà.
„Przyjmijmy, ˝e jestem koncernem
farmaceutycznym i potrafi´ wprowa-
dziç [mój] lek na rynek rok wczeÊniej –
wyjaÊnia Stelios Papadopoulos, dyrek-
tor naczelny Wydzia∏u Opieki Zdrowot-
nej w nowojorskiej bankowej firmie in-
westycyjnej SG Cowen. – Mo˝e to ozna-
czaç, ˝e zyskam dodatkowo na jego
sprzeda˝y jakieÊ pó∏ miliarda dolarów.”
Zanim jednak nastàpi nieoczekiwany
przyp∏yw gotówki, firmy bioinformatycz-
ne muszà uporaç si´ z nawa∏em nowych
danych o genomie i ciàgle udoskonalaç
technik´, metodyk´ badawczà i polityk´
ekonomicznà. Firmy bioinformatyczne
powinny si´ jednoczeÊnie skoncentrowaç
na praktycznych mo˝liwoÊciach, czyli na
tym, jak posk∏adaç w ca∏oÊç wszystkie in-
formacje, znaleêç powiàzania mi´dzy ni-
mi i na tej podstawie stworzyç ogólny ob-
raz w tej ga∏´zi nauki.
Metodyka jest ju˝ tak zaawansowa-
na, ˝e da si´ wytwarzaç ogromne iloÊci
danych – uwa˝a Michael R. Fannon, wi-
ceprezes i kierownik dzia∏u informacyj-
nego w Human Genome Sciences, ma-
jàcym równie˝ siedzib´ w Rockville. –
Nie wiemy jednak, jakà wag´ majà
wszystkie te informacje.”
Ustalenie ich znaczenia to w∏aÊnie za-
danie bioinformatyki. Dziedzina ta na-
rodzi∏a si´ na poczàtku lat osiemdziesià-
tych, wraz z utworzeniem w USA bazy
danych nazwanej GenBankiem. Amery-
kaƒski Departament Energii opracowa∏
ten program, aby zbieraç i przechowy-
waç informacje o sekwencjach krótkich
odcinków DNA, które naukowcy zacz´-
li w∏aÊnie zdobywaç, badajàc ró˝ne orga-
nizmy. Na poczàtku istnienia GenBanku
rzesze techników siedzia∏y nad klawiatu-
rami sk∏adajàcymi si´ z czterech klawiszy
A, C, T oraz G i ˝mudnie wstukiwa∏y do
komputera ciàgi liter oznaczajàcych opu-
blikowane w czasopismach naukowych
sekwencje DNA. Z czasem opracowano
protoko∏y przekazywania danych. Bada-
cze mogli teraz zadzwoniç do GenBanku
i bezpoÊrednio wprowadziç do kompu-
tera dane o sekwencji kolejnych odcin-
ków genomu. Administracj´ GenBanku
58 Â
WIAT
N
AUKI
Paêdziernik 2000
BIOINFORMATYCZNA
Przekszta∏canie wst´pnych danych
o genomie w wiedz´ potrzebnà
do tworzenia nowych leków zrodzi∏o
przemys∏ wartoÊci 300 mln dolarów
SCIENCE, VOL. 287, NR 5461; 24 III 2000
(powy˝ej i
na sàsiedniej stronie)
Ken Howard
P

G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C K O T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G K O T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A T
A T C G G C T A T A T C G G C
T A C G A T T A C A C G T A T
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
G A T A T C G G C T A C G A T
A T C G G C T A C G A T A T C
G G C T A C G A T A T C G G C
T A C G A T A T C G G C T A C
przeniesiono do Paƒstwowego Centrum In-
formacji Biotechnologicznej (NCBI –
National Center for Biotechnology Infor-
mation) w National Institutes of Health.
Z pojawieniem si´ us∏ugi WWW w Interne-
cie naukowcy z ca∏ego Êwiata uzyskali bez-
p∏atny dost´p do danych zgromadzonych
w GenBanku.
Kiedy w 1990 roku ruszy∏ oficjalnie Pro-
jekt Poznania Ludzkiego Genomu, liczba
informacji o zapisie genetycznym nap∏y-
wajàcych do GenBanku zacz´∏a rosnàç
w post´pie wyk∏adniczym. Po wprowadze-
niu w latach dziewi´çdziesiàtych metod
wysoko wydajnego sekwencjonowania
z wykorzystaniem robotyki, automatycz-
Â
WIAT
N
AUKI
Paêdziernik 2000 59
nych sekwenatorów i komputerów proces
ten nabra∏ wr´cz zawrotnego tempa. Do
dnia, w którym oddano do druku lipcowy
numer Scientific American, GenBank zdà˝y∏
zgromadziç dane o kolejnoÊci zapisu po-
nad 7 mld jednostek DNA
1
.
Mniej wi´cej w tym samym czasie, gdy
rozpocz´to realizacj´ Projektu Poznania
Ludzkiego Genomu, podobnymi badania-
mi zaj´∏y si´ firmy prywatne i stworzy∏y
w∏asne obszerne bazy danych. Dzisiaj kor-
poracje takie jak Incyte Genomics z Palo
Alto w Kalifornii sà w stanie odczytaç
w ciàgu jednego dnia zapis genetyczny li-
czàcy oko∏o 20 mln par zasad DNA.
A przedstawiciele prawdziwej fabryki in-
formacji genetycznej, czyli firmy Celera Ge-
nomics – którzy oznajmili w kwietniu te-
go roku, ˝e uzyskali wst´pnà wersj´ pe∏nej
sekwencji genomu ludzkiego [patrz: „Geno-
mowa goràczka z∏ota”, strona 50] – twier-
dzà, ˝e ich firma zgromadzi∏a na ten temat
50 TB danych. Taka iloÊç informacji zaj´∏a-
by 80 tys. p∏yt kompaktowych. Gdyby usta-
wiç je obok siebie, zamkni´te w plastiko-
wych pude∏kach na pó∏ce, to musia∏aby ona
mieç d∏ugoÊç
3
/
4
km!
GenBank i wymienione powy˝ej firmy
sà jednak zaledwie cz´Êcià bioinformatycz-
nej uk∏adanki. Istniejà jeszcze inne publicz-
ne i prywatne bazy danych zawierajàce in-
formacje o ekspresji genów (czyli o tym,
kiedy i gdzie geny sà w∏àczane), tzw. po-
limorfizmie sekwencji nukleotydowych
(czyli drobnych ró˝nicach genetycznych
mi´dzy poszczególnymi osobnikami), bu-
Rozpocznie si´ wyÊcig o najlepsze wykorzystanie
tego nieprzebranego bogactwa wiedzy.
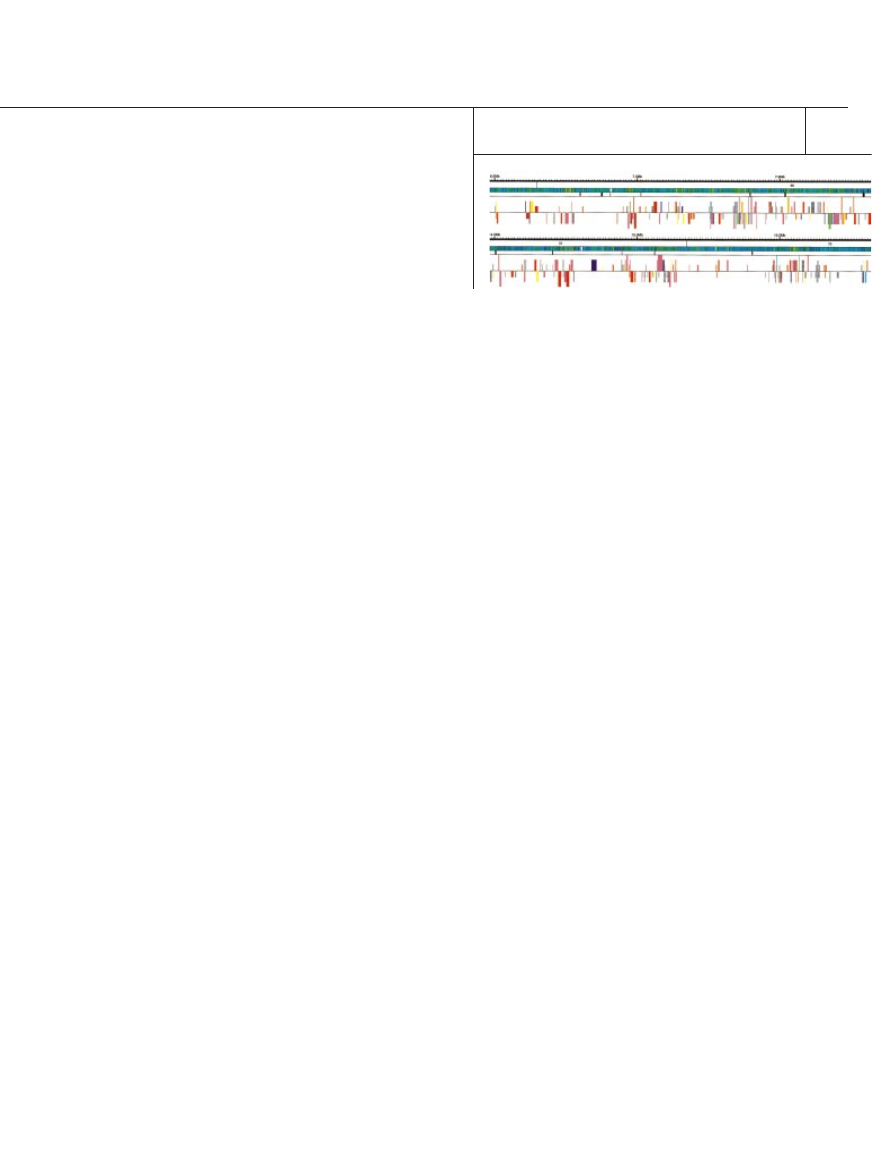
DANE GENETYCZNE sà surowcem bioinformatyki. Dzia∏ania w tej dziedzinie mo˝na jednak po-
równaç do szukania ig∏y w stogu siana. Na zabawnym rysunku z lewej ig∏à jest s∏owo KOT,
ukryte poÊród tysi´cy sekwencji zasad A, C, T i G – czterech jednostek budulcowych
4
kwasu de-
oksyrybonukleinowego. Bioinformatyka porównuje tak˝e geny ró˝nych organizmów. Inne ilu-
stracje na stronach tej i poprzedniej ukazujà fragment mapy genomu muszki owocowej. Kolorowe
s∏upki, przypominajàce kod kreskowy, oznaczajà sekwencje kodujàce wykazujàce podobieƒstwo
do genów ssaków, nicienia
Caenorhabditis elegans i dro˝d˝y piekarniczych.
SLIM FILMS
dowie ró˝nych bia∏ek, oraz schematy ich
wzajemnych oddzia∏ywaƒ [patrz: „Co da-
lej z ludzkim genomem”, strona 64].
Mieszanie i dobieranie
Jednym z podstawowych zadaƒ w bioin-
formatyce jest poszukiwanie podobieƒstw
(homologii) mi´dzy nowo odczytanymi od-
cinkami DNA a zsekwencjonowanymi
wczeÊniej fragmentami nici tego kwasu in-
nych organizmów. Odnalezienie takiego
„pokrewieƒstwa” daje badaczom mo˝li-
woÊç przewidywania, jaki rodzaj bia∏ka
mo˝e byç kodowany przez dopiero co roz-
szyfrowanà sekwencj´. Pozwala to nie tyl-
ko na wczesny wybór obiektu oddzia∏ywa-
nia potencjalnych nowych leków, ale tak-
˝e na wyeliminowanie wielu fa∏szywych
tropów prowadzàcych donikàd.
Popularnym pakietem programów s∏u-
˝àcych do porównywania zapisu genetycz-
nego jest BLAST (Basic Local Alignment
Search Tool), który pojawi∏ si´ w 1990 ro-
ku. To jedno z ca∏ej grupy narz´dzi stoso-
wanych do badaƒ sekwencji DNA oraz bia-
∏ek, dostarczanych w ró˝nych wersjach
przez komercyjnych us∏ugodawców i do-
st´pnych tak˝e bezpoÊrednio w NCBI. Pla-
cówka ta oferuje równie˝ Entrez, czyli tzw.
metawyszukiwark´, która wspó∏pracuje
z wi´kszoÊcià znajdujàcych si´ tam baz da-
nych, ∏àcznie z zawierajàcymi opisy trójwy-
miarowych modeli bia∏ek, pe∏nà sekwencj´
genomów organizmów, takich jak dro˝d˝e,
a tak˝e wykaz piÊmiennictwa – êród∏a zgro-
madzonych dotychczas informacji.
Jednym z pierwszych przyk∏adów przy-
datnoÊci bioinformatyki jest historia katep-
syny K, enzymu mogàcego okazaç si´ wa˝-
nym obiektem oddzia∏ywania w leczeniu
osteoporozy – choroby, która powoduje pro-
wadzàcà do kalectwa ∏amliwoÊç koÊci.
W 1993 roku badacze z filadelfijskiej firmy
Smith-Kline Beecham poprosili naukowców
BONANZA

Lion Bioscience
www.lionbioscience.com
W∏asnoÊç prywatna
Siedziba:
Heidelberg, Niemcy
Zarzàdzajàcy:
Friedrich von Bohlen,
dyrektor generalny
G∏ówni klienci/partnerzy:
Bayer, Aven-
tis, Pharmacia
Strategia:
Dostarczanie bioinforma-
tycznych systemów i us∏ug dost´p-
nych dla szerokiej grupy przedsi´-
biorstw.
Tegoroczny bud˝et:
Brak danych
Podstawowy cel:
Rozwijanie wspó∏-
pracy z du˝ymi i Êrednimi koncernami
biotechnologicznymi i farmaceutyczny-
mi; powtórzenie sukcesu, jakim by∏o
zawarcie umowy z firmà Bayer.
Przewaga nad konkurencjà:
Opiewa-
jàcy na 100 mln kontrakt z koncernem
Bayer stwarza ogromne perspektywy
i jest dla firmy dêwignià finansowà.
NetGenics
www.netgenics.com
W∏asnoÊç prywatna
Siedziba:
Cleveland, Ohio (USA)
Zarzàdzajàcy:
Manuel J. Glynias, pre-
zes i dyrektor generalny
G∏ówni klienci/partnerzy:
Abbott La-
boratories, Aventis, IBM
Strategia:
Dostarczanie systemów
i us∏ug bioinformatycznych wielkiej
grupie przedsi´biorstw.
Tegoroczny bud˝et:
21.3 mln dolarów
Podstawowy cel:
Rozwijanie wspó∏pra-
cy z du˝ymi i Êrednimi koncernami bio-
technologicznymi i farmaceutycznymi.
Przewaga nad konkurencjà:
Dobre
zaplecze finansowe i powiàzania z du-
˝ymi koncernami farmaceutycznymi.
DoubleTwist
www.doubletwist.com
W∏asnoÊç prywatna
Siedziba:
Oakland, Kalifornia
Zarzàdzajàcy:
John Couch, prezes
i dyrektor generalny
G∏ówni klienci/partnerzy:
Derwent In-
formation, Clontech Laboratories, My-
riad Genetics, AlphaGene, Universi-
ty of Pennsylvania
Strategia:
Umo˝liwianie dost´pu do
ró˝nych bioinformatycznych narz´dzi
i baz danych.
Tegoroczny bud˝et:
37 mln dolarów
Podstawowy cel:
Dostarczanie uni-
kalnych, firmowych produktów i przy-
ciàganie wystarczajàcej liczby klien-
tów, aby utrzymaç si´ na rynku us∏ug
internetowych.
Przewaga nad konkurencjà:
Szerokie
perspektywy i potencjalnie du˝y ry-
nek zbytu.
Compugen
www.cgen.com
W∏asnoÊç prywatna
Siedziba:
Tel Awiw, Izrael
Zarzàdzajàcy:
Mor Amitai, dyrektor
generalny
G∏ówni klienci/partnerzy:
Merck, Incy-
te Genomics, Amgen, Millennium Phar-
maceuticals, Bayer, Human Genome
Sciences, Janssen Pharmaceutica
Strategia:
Produkcja sprz´tu i opro-
gramowania komputerowego, które
przyÊpieszà prac´ algorytmów bioin-
formatycznych; zaanga˝owanie w od-
czytywanie genów oraz opracowanie
nowych leków; udost´pnianie narz´-
dzi bioinformatycznych przez Internet.
Tegoroczny bud˝et:
Brak danych
Podstawowy cel:
Specjalizacja w od-
krywaniu nowych leków; rozszerze-
nie oferty firmy; rozwój dzia∏alnoÊci
na rynku internetowym.
Przewaga nad konkurencjà:
Jedna
z pierwszych firm, które opracowa∏y
wyspecjalizowane narz´dzia bioinfor-
matyczne, co da∏o jej pozycj´ eksper-
ta w dziedzinie wyszukiwania infor-
macji. Ma solidnà, prawnie zastrze-
˝onà baz´ danych biologicznych, z
której korzysta podczas wst´pnego
opracowania nowych leków.
Oxford Molecular
Group
www.oxmol.co.uk
Symbol akcji:
OMG (Londyn)
Siedziba:
Oksford, Anglia
Zarzàdzajàcy:
N. Douglas Brown, pre-
zes
G∏ówni klienci/partnerzy:
Novartis, Gla-
xo Wellcome, Merck, Pfizer, Smith-
Kline Beecham, Abbott Laboratories
Strategia:
Dostarczanie wielu us∏ug
i oprogramowania zwiàzanego z opra-
cowaniem nowych leków.
Tegoroczny bud˝et:
Brak danych
Podstawowy cel:
Rozszerzenie dzia-
∏alnoÊci przez stworzenie wi´kszej
liczby produktów i us∏ug dost´pnych
szerokiej grupie przedsi´biorstw.
Przewaga nad konkurencjà:
Jest w∏a-
Êcicielem firmy Genetics Computer
Group, której sztandarowy produkt,
oprogramowanie Wisconsin Package,
jest uwa˝any w tym sektorze przemy-
s∏u za standard do analizy sekwencji
genowych.
InforMax
www.informaxinc.com
W∏asnoÊç prywatna
Siedziba:
Bethesda, Maryland (USA)
Zarzàdzajàcy:
Alex Titomirov, dyrek-
tor generalny
G∏ówni klienci/partnerzy:
Produkty fir-
my sà wykorzystywane przez 19 pod-
miotów prawnych
Strategia:
Dostarczanie komputero-
wych narz´dzi bioinformatycznych do-
st´pnych dla szerokiej grupy przed-
si´biorstw.
Tegoroczny bud˝et:
Brak danych
Podstawowy cel:
Udost´pnienie pro-
duktów firmy wi´kszej grupie odbior-
ców.
Przewaga nad konkurencjà:
Znaczà-
ca pozycja na rynku komputerowych
narz´dzi bioinformatycznych.
SLIM FILMS; èRÓD¸A: WYMIENIONE FIRMY; JASON REED
Oscar Gruss & Son
;
ADRIENNE BURKE
BioInform newsletter
G¸ÓWNI GRACZE
z Human Genome Sciences o pomoc
w przeanalizowaniu materia∏u genetycz-
nego, wyizolowanego z osteoklastów lu-
dzi cierpiàcych na raka koÊci. (Osteokla-
sty, czyli komórki koÊciogubne sà odpo-
wiedzialne za absorpcj´ i niszczenie ko-
Êci w prawid∏owym procesie jej przebu-
dowy; uwa˝a si´, ˝e u osób chorych na
osteoporoz´ sà one nadaktywne.)
Naukowcy z HGS zsekwencjonowa-
li DNA zawarty w powierzonej im prób-
ce i korzystajàc z bazy danych, zacz´li
szukaç homologii, które mia∏y im wska-
zaç bia∏ka kodowane przez wykryte
w ten sposób geny. Gdy odnaleêli ho-
mologiczne sekwencje, prowadzili ba-
dania dalej, a˝ odkryli, ˝e jedna z nich
wyst´puje w osteoklastach w bardzo
wielu kopiach. Ów fragment genomu
by∏ podobny do tych, o których ju˝ wie-
dziano, ˝e koduje wczeÊniej wykrytà
klas´ czàsteczek – katepsyny.
Dla SmithKline Beecham ta bioinfor-
matyczna „wprawka” zaowocowa∏a
w ciàgu zaledwie tygodni ustaleniem
obiecujàcego obiektu oddzia∏ywania no-
wego leku. Za pomocà klasycznych me-
tod doÊwiadczalnych taki wynik da∏oby
si´ uzyskaç dopiero po kilku latach. Te-
raz badacze zatrudnieni w firmie próbu-
jà znaleêç Êrodek blokujàcy aktywnoÊç
katepsyny K. Poszukiwania substancji,
które oddzia∏ujà na konkretny obiekt
i wywo∏ujà po˝àdany efekt, ciàgle jeszcze
odbywajà si´ g∏ównie w tradycyjnych la-
boratoriach biochemicznych, gdzie oce-
na aktywnoÊci zwiàzków, ich toksycz-
noÊci i wch∏aniania cz´sto trwa latami.
Ale gdy ma si´ do dyspozycji nowe na-
rz´dzia bioinformatyczne i dost´p do
wcià˝ rosnàcej liczby danych o struktu-
rze bia∏ek i molekularnych szlakach ich
dzia∏ania, wkrótce i ten etap tworzenia
nowych leków b´dzie mo˝na przenieÊç
do komputera – twierdzà niektórzy ba-
dacze. Ta nowa dziedzina aktywnoÊci
naukowej zyska wówczas nazw´ biolo-
gii „in silico”
2
[ramka na nast´pnej stronie].
Wszystko to dobrze wró˝y bioinfor-
matyce, która jak twierdzi wielu, zawie-
ra prawdziwe spe∏nienie genomiki. „Ge-
nomika bez bioinformatyki nie b´dzie
mia∏a zbyt wiele do zaoferowania” –
twierdzi Roland Somogyi, by∏y dyrektor
dzia∏u neurobiologii w Incyte Genomics,
obecnie pracujàcy w Molecular Mining
w Kingston w stanie Ontario.
Michael N. Liebman, szef dzia∏u bio-
logii obliczeniowej w Roche Bioscience
w Palo Alto, zgadza si´ z tym poglà-
dem. „Zmiana paradygmatów nie tkwi
w genomice, lecz w zrozumieniu, jak jà
wykorzystaç. – zapewnia. – Bioinforma-
tyka to dopiero poczàtek rewolucji.”
Uczestnicy gry prezentujà ró˝ne stra-
tegie. Niektóre firmy bioinformatyczne
Â
WIAT
N
AUKI
Paêdziernik 2000 61
G
. . .
. . .
A G A A C T G T T T A G A T G C A A
E
. . .
. . .
N C L
D
A K S T S
A A T C C A C A A G T
E
E
E
E
N
N
N
N
S
S
S
S
L
L
I
L
D
D
D
D
A
A
A
A
Q
G
N
G
S
A
A
A
T
T
T
T
H
E
N
C
L
D
A
K
S
T
S
E
M
R
. . .
. . .
. . .
. . .
. . .
. . .
. . .
. . .
. . .
. . .
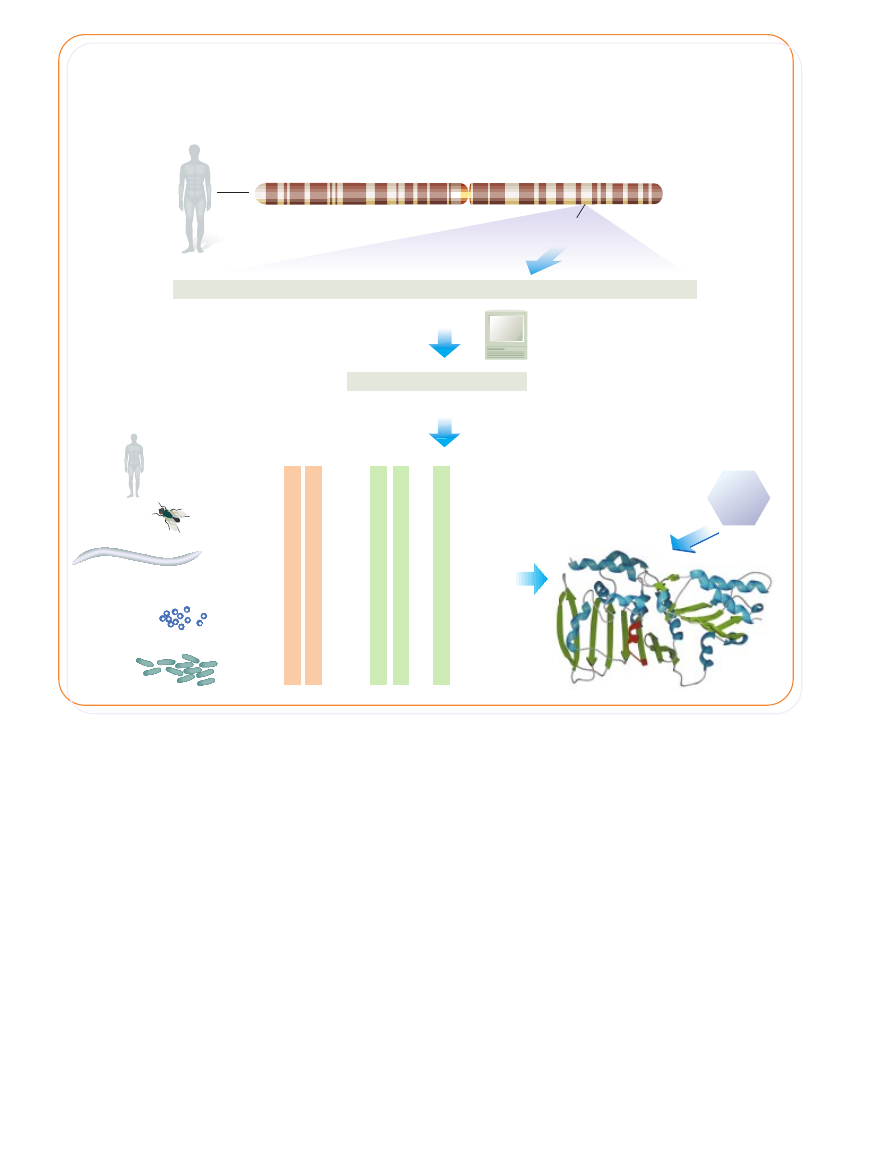
MODEL LUDZKIEGO BIA¸KA
STWORZONY W OPARCIU
O ZNANÑ STRUKTUR¢
PODOBNEJ PROTEINY
WYST¢PUJÑCEJ W ORGANIZMIE
MODELOWYM
(czerwonobràzowy
obszar jest kodowany przez
ukazanà tu sekwencj´)
ODNALEZIENIE ZWIÑZKU,
KTÓRY PRZY¸ÑCZA SI¢
DO SKONSTRUOWANEGO
WIRTUALNIE BIA¸KA
TEORETYCZNY
LEK
TRANSLACJA SEKWENCJI ZASAD W DNA
NA SEKWENCJ¢ AMINOKWASÓW
(jednostek budulcowych bia∏ka)
Z ZASTOSOWANIEM
PROGRAMU KOMPUTEROWEGO
MUSZKA
OWOCOWA
(Drosophila melanogaster)
SEKWENCJA AMINOKWASÓW CZ¸OWIEKA
NICIE¡
(Caenorhabditis elegans)
CZ¸OWIEK
DRO˚D˚E
PIEKARNICZE
(Saccharomyces cerevisiae)
BAKTERIA
(Escherichia coli
)
LUDZKI CHROMOSOM 3
GEN MLH1
(na prà˝ku 21.3)
q (d∏ugie rami´)
p (krótkie rami´)
4
5
2
WYIZOLOWANIE SEKWENCJI LUDZKIEGO DNA
1
POSZUKIWANIE SEKWENCJI HOMOLOGICZNYCH
W BAZACH DANYCH, ZAWIERAJÑCYCH
INFORMACJ¢ O BIA¸KACH ORGANIZMÓW
MODELOWYCH
(zielony oznacza du˝e ro˝nice,
pomaraƒczowy – mniejsze)
3
Wykorzystanie bioinformatyki do tworzenia leków
LAURIE GRACE, Z
POMOCÑ MARKA GERSTEINA I
PATA FLEMINGA
Yale University
ORAZ DAVIDA WHEELERA I
JENNIFER VYSKOCIL
NCBI
W organizmach modelowych badacze poszukujà sekwencji, które sà podobne do konkretnego genu ludzkiego. Mo˝na w ten
sposób poznaç struktur´ kodowanego przez niego bia∏ka, a nast´pnie znaleêç Êrodek, który zablokuje aktywnoÊç tego genu. Przy-
k∏adem mo˝e byç gen
MLH1
, którego aktywnoÊç jest u ludzi zwiàzana z nowotworem okr´˝nicy.

zaspokajajà potrzeby pot´˝nych klien-
tów, oferujàc swe us∏ugi konsultacyjne
i oprogramowanie koncernom ge-
nomicznym, biotechnologicznym i far-
maceutycznym. Korporacja Lion Bio-
science z siedzibà w Heidelbergu w
Niemczech odnios∏a szczególny sukces
w sprzeda˝y narz´dzi i us∏ug bioin-
formatycznych szerokiej grupie odbior-
ców. Zawartà przez nià opiewajàcà na
100 mln dolarów umow´ z firmà Bayer
na stworzenie dost´pnego dla wszyst-
kich jej oddzia∏ów bioinformatycznego
zaplecza i zarzàdzanie nim okrzykni´to
w prasie najwi´kszà tego typu transak-
cjà handlowà.
Inne firmy nastawiajà si´ na ma∏ych
odbiorców lub placówki naukowe. Spó∏-
ki dzia∏ajàce w Sieci, jak Double Twist
z Oakland czy eBioinformatics z g∏ów-
nà siedzibà w Pleasanton (oba miasta
znajdujà si´ w Kalifornii), oferujà zaku-
py przez Internet. Stworzone przez nie
witryny pozwalajà u˝ytkownikom
dotrzeç za op∏atà do ró˝nego rodzaju
baz danych oraz skorzystaç z oprogra-
mowania, aby przetwarzaç uzyskane
informacje.
W maju naukowcy z DoubleTwist
og∏osili, ˝e za pomocà w∏asnych tech-
nik ustalili liczb´ genów ludzkiego ge-
nomu na 105 tys., chocia˝ ostateczny
wynik to zapewne 100 tys
3
.
Tym zaÊ, którzy wolà u˝ywaç w∏asne-
go oprogramowania, bezpiecznie ukry-
tego przed innymi poszukiwaczami, fir-
my takie jak Informax z Rockville czy
Oxford Molecular Group z Wielkiej Bry-
tanii, oferujà oryginalne produkty
w zabezpieczonych opakowaniach.
Tworzenie po∏àczeƒ
Wielkie koncerny farmaceutyczne po-
szukiwa∏y równie˝ sposobu zwi´ksze-
nia skutecznoÊci dzia∏aƒ w genomice
i rozwija∏y w∏asne s∏u˝by bioinforma-
tyczne. W licznych stworzono osobne
wydzia∏y, w których miano ujednoliciç
oprogramowanie i jego obs∏ug´ oraz
u∏atwiç dost´p do baz danych wielu
podjednostkom zajmujàcym si´ tworze-
niem nowego leku – opracowaniem je-
go formu∏y, toksykologià czy testami
klinicznymi. W starej metodzie opraco-
wania lekarstw zwykle oddzielano te
etapy, a przez to informacje, które mo-
g∏yby zostaç spo˝ytkowane przez in-
nych badaczy, pozostawa∏y uwi´zione
w naukowych „gettach”. Dzi´ki bioin-
formatyce naukowcy pracujàcy w tej sa-
mej firmie majà dost´p do wszystkich
danych, które mogà indywidualnie
przetwarzaç.
Taka wewnàtrzzak∏adowa wymiana
informacji nie tylko zwi´ksza wydajnoÊç
tworzenia nowych leków, ale umo˝liwia
tak˝e zaoszcz´dzenie sporej iloÊci pie-
ni´dzy przeznaczonych na zakup opro-
gramowania. Firma Glaxo Wellcome
z Research Triangle Park w Karolinie Pó∏-
nocnej zastàpi∏a zaÊ indywidualne pa-
kiety, umo˝liwiajàce ró˝nym badaczom
i wydzia∏om dost´p do baz danych
i przetwarzanie zawartych tam informa-
cji, jednà platformà programowà. Robin
M. DeMent, amerykaƒski dyrektor dzia-
∏u bioinformatyki w Glaxo Wellcome,
szacuje, ˝e dzi´ki temu posuni´ciu jego
koncern w ciàgu 3–5 lat zaoszcz´dzi oko-
∏o 800 tys. dolarów.
Aby zintegrowaç bioinformatyk´
w swych firmach, giganci rynku farma-
ceutycznego zawierajà strategiczne so-
jusze i umowy licencyjne oraz wykupu-
jà mniejsze spó∏ki biotechnologiczne.
Partnerzy i handlowcy nie tylko rozsze-
rzajà zakres bioinformatycznych mo˝li-
woÊci „rekinów farmacji”, ale mobilizu-
jà ich równie˝ do korzystania z poja-
wiajàcych si´ na rynku nowych techno-
logii, zamiast nieustannego moderni-
zowania w∏asnych systemów. „JeÊli firma
62 Â
WIAT
N
AUKI
Paêdziernik 2000
Zapomnij o in vitro –
teraz dzia∏amy „in silico”
G
enom ludzki zosta∏ w zasadzie zsekwencjonowany. Fu-
turyÊci przewidujà, ˝e wkrótce naukowcy b´dà w stanie
za pomocà bioinformatyki stworzyç model wr´cz astrono-
micznej liczby reakcji biochemicznych, które sk∏adajà si´ na
ludzkie ˝ycie. Ken Howard rozmawia o mo˝liwoÊciach „kompu-
terowej biologii” z ekspertem w tej dziedzinie Stuartem A. Kauff-
manem, profesorem z Santa Fe Institute w Nowym Meksyku,
a tak˝e za∏o˝ycielem i g∏ównym kierownikiem naukowym Bios
Group w Santa Fe.
Howard:
Jakie obietnice daje bioinformatyka i biologia „in silico”?
Kauffman:
Mo˝emy uwa˝aç 100 tys. genów zapisanych w DNA
ludzkich komórek za pewien rodzaj stosujàcego przetwarza-
nie równoczesne chemicznego komputera, w którym geny nie-
ustannie w∏àczajà si´ i wy∏àczajà w ramach niezmiernie skom-
plikowanej sieci wzajemnych oddzia∏ywaƒ. Drogi przekazywania
sygna∏ów w komórkach sà powiàzane z genetycznymi mecha-
nizmami regulacyjnymi w sposób, który dopiero zaczynamy
poznawaç. Najwi´kszym zadaniem bioinformatycznym, przed
którym stoimy, jest rozszyfrowanie uk∏adu sterujàcego rozwo-
jem organizmu od zap∏odnienia komórki jajowej do osiàgni´-
cia dojrza∏oÊci.
Howard:
Co dzi´ki temu zyskamy?
Kauffman:
B´dziemy wiedzieli, dzia∏anie których genów lub ich
fragmentów – i w jakiej kolejnoÊci – trzeba zak∏óciç, aby zmu-
siç komórk´ nowotworowà do niez∏oÊliwego zachowania lub
apoptozy (zaprogramowanej Êmierci). Znajdziemy sposób na
pokierowanie regeneracjà konkretnej tkanki i kiedy zdarzy si´,
˝e straci pan po∏ow´ trzustki, my spowodujemy, ˝e si´ zregene-
ruje. Byç mo˝e uda si´ nam równie˝ doprowadziç do odtwo-
rzenia komórek ß u ludzi chorych na cukrzyc´.
Howard:
Co trzeba zrobiç, aby osiàgnàç ten cel?
Kauffman:
Na pewno nie jest to wy∏àcznie sprawa bioinforma-
tyki. Musimy doprowadziç do maria˝u ró˝nych nowoczesnych na-
rz´dzi matematycznych. Wska˝à nam one alternatyw´ praw-
dopodobnych po∏àczeƒ fragmentów komórkowej sieci
regulacyjnej. Nast´pnie zamierzamy powiàzaç to z nowymi me-
todami doÊwiadczalnymi, aby dowiedzieç si´, jakie te uk∏ady
kontrolne sà naprawd´. Bioinformatyk´ nale˝y poszerzyç o me-
tody doÊwiadczalne. Musimy za∏o˝yç, ˝e ka˝dy z fragmentów bio-
informatycznej szarady to hipoteza, którà nale˝y sprawdziç.
Howard:
Przed jakimi wyzwaniami stoimy?
Kauffman:
PrzypuÊçmy, ˝e wybieram 10 genów, o których wiem,
˝e wzajemnie regulujà swojà aktywnoÊç i próbuj´ opracowaç
model ich funkcjonowania. To cudowne i powinniÊmy to robiç.
Jednak istnieje druga strona medalu: na te 10 genów oddzia∏u-
jà tak˝e inne, które pozostajà poza badanym przez nas uk∏a-
dem. Tak wi´c ostatecznie poznajemy zaledwie jeden ma∏y try-
bik wielkiego mechanizmu z∏o˝onego z tysi´cy genów. Staramy
si´ ustaliç, w jaki sposób funkcjonuje wybrany przez nas uk∏ad,
nie wiedzàc, jak oddzia∏ujà naƒ pozosta∏e geny. NieznajomoÊç
wszystkich tych interakcji bardzo utrudnia rozwiàzywanie za-
gadnienia. Od lat wiemy wszystko o ka˝dej komórce nerwowej
w zwojach brzusznych homara (sà to skupiska nerwów po∏à-
czone z uk∏adem pokarmowym zwierz´cia) – znamy znajdujà-
ce si´ w nich po∏àczenia synaptyczne i neuroprzekaêniki. Taki
zwój sk∏ada si´ zaledwie z 13 lub 20 neuronów, a jednak wcià˝
farmaceutyczna ma wystarczajàco du˝y
bud˝et badawczy, mo˝e zrobiç wszystko
sama – twierdzi Somogyi z Celera Ge-
nomics. – Jest to jednak równie˝ kwestia
podejÊcia. Bioinformatyka przynosi zy-
ski, poniewa˝ dzi´ki niej ró˝ne firmy sà
w stanie zajmowaç si´ wieloma zagad-
nieniami, ale z mo˝liwoÊcià pokrywania
si´ ich obszarów dzia∏aƒ.”
Human Genome Sciences, Celera czy
Incyte funkcjonujà w niektórych z nich –
w dziedzinie zasobów, produktów i ryn-
ku kapita∏owego. Koncerny te okupujà te-
reny pomi´dzy „wielkà farmacjà” a firma-
mi zajmujàcymi si´ uzyskiwaniem i
przetwarzaniem informacji. Bardzo szyb-
ko zaadaptowa∏y tak˝e automatyzacj´,
którà bioinformatyka wnios∏a do biologii.
Ale ca∏e to zró˝nicowanie stwarza nie-
bezpieczeƒstwo powstania nieporozu-
mieƒ i zafa∏szowaƒ. Ró˝ne bazy danych
potrafià ju˝ dogadywaç si´ ze sobà (na-
zywamy to interoperatywnoÊcià), co jest
coraz wa˝niejsze dla u˝ytkowników,
którzy poszukujàc potrzebnych wiado-
moÊci, szperajà w zbiorach. Dobrym roz-
wiàzaniem wydawa∏o si´ przypisanie
ró˝nym informacjom „metek”, czyli
identyfikatorów, które by∏yby krzy˝o-
wo wykorzystywane przez wszystkie
bazy danych i rejestry systemowe. Taka
metoda sprawdza∏a si´ do pewnego
stopnia. „Uda∏o si´ nam po∏àczyç bazy
danych dzi´ki odsy∏aczom: bazy A do
bazy B, B do C, C do D – wyjaÊnia Lieb-
man z Roche Bioscience. – Jednak odsy-
∏acz w bazie A mo˝e si´ zmieniç i wtedy
gdy dotrzemy do D, trafimy na nie zmie-
nione odnoÊniki, zw∏aszcza przy ciàg∏ym
nap∏ywie tak du˝ej iloÊci danych.” Uwa-
˝a, ˝e problem stanie si´ powa˝ny, gdy
wiedza biologiczna i mo˝liwoÊci prze-
twarzania informacji znajdà si´ na wy˝-
szym poziomie. „Dopiero zaczynamy
rozumieç, jak skomplikowane sà te za-
gadnienia, a sposób przechowywania
danych to klucz do uzyskania odpowie-
dzi na nasze pytania” – twierdzi.
Wed∏ug Davida J. Lipmana, dyrekto-
ra NCBI, systematyczne wprowadzanie
ulepszeƒ na pewno oka˝e si´ pomocne.
Jednak post´p – a co za tym idzie, rów-
nie˝ zysk – wcià˝ jeszcze zale˝y od po-
mys∏owoÊci u˝ytkownika. „To kwestia
oprogramowania mózgu – przekonuje –
a nie sprz´tu czy programów kompu-
terowych.”
T∏umaczy∏a
Joanna Grabarek
Przypisy t∏umaczki i redakcji:
1
Czyli o kolejnoÊci u∏o˝enia w nici DNA par zasad
azotowych – jedna para zasad jest jednostkà d∏u-
goÊci odcinka kwasu nukleinowego.
2
Jest to doÊç zabawna dla biologów gra s∏ów. „In vi-
tro” oznacza „w szkle”, czyli w probówce, poza or-
ganizmem. W tym rozumieniu „in silico” oznacza-
∏oby prowadzenie badaƒ biologicznych „w
krzemie”, czyli za pomocà komputera.
3
Sà sugestie, ˝e jeszcze znacznie mniej.
4
Jednostkà budulcowà jest nukleotyd, czyli zasada
azotowa + cukier + kwas fosforowy. Zapis gene-
tyczny natomiast rzeczywiÊcie zale˝y od kolejnoÊci
u∏o˝enia zasad azotowych w nici DNA.
Â
WIAT
N
AUKI
Paêdziernik 2000 63
nie potrafimy ustaliç, jak dzia∏a. ˚adnemu matematykowi nawet
przez myÊl nie przyjdzie, ˝e zrozumienie systemu z 13 zmien-
nymi to prosta sprawa. A my chcemy to zrobiç z uk∏adem ze
100 tys. zmiennych (chodzi o ludzki genom). Pozwol´ sobie zde-
finiowaç status wzajemnych powiàzaƒ wszystkich genów tylko
wed∏ug tego, czy sà w∏àczone, czy te˝ nie. Ile mamy takich sta-
nów? Dwa dla ka˝dego genu, co daje 2
100 000
kombinacji, czyli
oko∏o 10
30 000
. Zatem nawet je˝eli traktujemy geny tylko jako ele-
menty, który sà aktywne lub nie – co jest oczywistym uproszcze-
niem, poniewa˝ notujemy wiele poziomów ich aktywnoÊci – ju˝
daje to wiele mo˝liwych stanów. Taki wynik po prostu nie mieÊci
si´ w g∏owie, zwa˝ywszy, ˝e liczba czàsteczek w znanym nam
WszechÊwiecie wynosi 10
80
.
Howard:
Jak d∏ugo b´dziemy si´ borykali z tym problemem?
Kauffman:
JesteÊmy dopiero na samym poczàtku. KiedyÊ jed-
nak nadejdzie dzieƒ, w którym choremu na raka postawimy
w∏aÊciwà diagnoz´ nie tylko na podstawie morfologii komó-
rek nowotworowych, ale tak˝e dzi´ki analizie szczegó∏owych
wzorów ekspresji genów i aktywnoÊci wiàzania bia∏ek w tych
komórkach.
Howard:
Kiedy to nastàpi? Za rok czy raczej za 200 lat?
Kauffman:
W ciàgu nast´pnych 10–12 lat u˝ywane do tych
celów narz´dzia osiàgnà taki poziom doskona∏oÊci, ˝e naprawd´
zaczniemy robiç post´py. Poznamy funkcjonowanie obszernych
fragmentów genomu i zrozumiemy, jak to wszystko dzia∏a. My-
Êl´, ˝e za 30–40 lat rozwià˝emy jego najwa˝niejsze zagadki.
Dost´p do pe∏nego zapisu rozmowy Kena Howarda ze Stuartem A.
Kauffmanem znajduje si´ na stronie internetowej
Scientific Ameri-
can: www.sciam.com/interview/2000/060500/kauffman.
RAIMOND L. WINSLOW I
DAVID SCOLLAN
Johns Hopkins University
I
PHYSIOME SCIENCES, PRINCETON, N. J.
Literatura uzupe∏niajàca
TRENDS IN COMMERCIAL BIOINFORMATICS.
Raport Jasona Reeda z firmy Oscar Gruss & Son
z 13 III 2000. Bezp∏atnà kopi´ mo˝na znaleêç w Internecie pod adresem: www.oscar-
gruss.com/reports.htm.
USING BIOINFORMATICS IN GENE AND DRUG DISCOVERY.
D. B. Searls; Drug Discovery Today,
vol. 5, nr 4, s. 135-143, IV/2000.
Dost´p do dwutygodnika nowoÊci w dziedzinie bioinformatyki BioInform mo˝na uzy-
skaç pod adresem: www.bioinform.com.
Dost´p do bioinformatycznych baz danych Paƒstwowego Centrum Informacji Bio-
technologicznej (NCBI): www.ncbi.nlm.nih.gov.
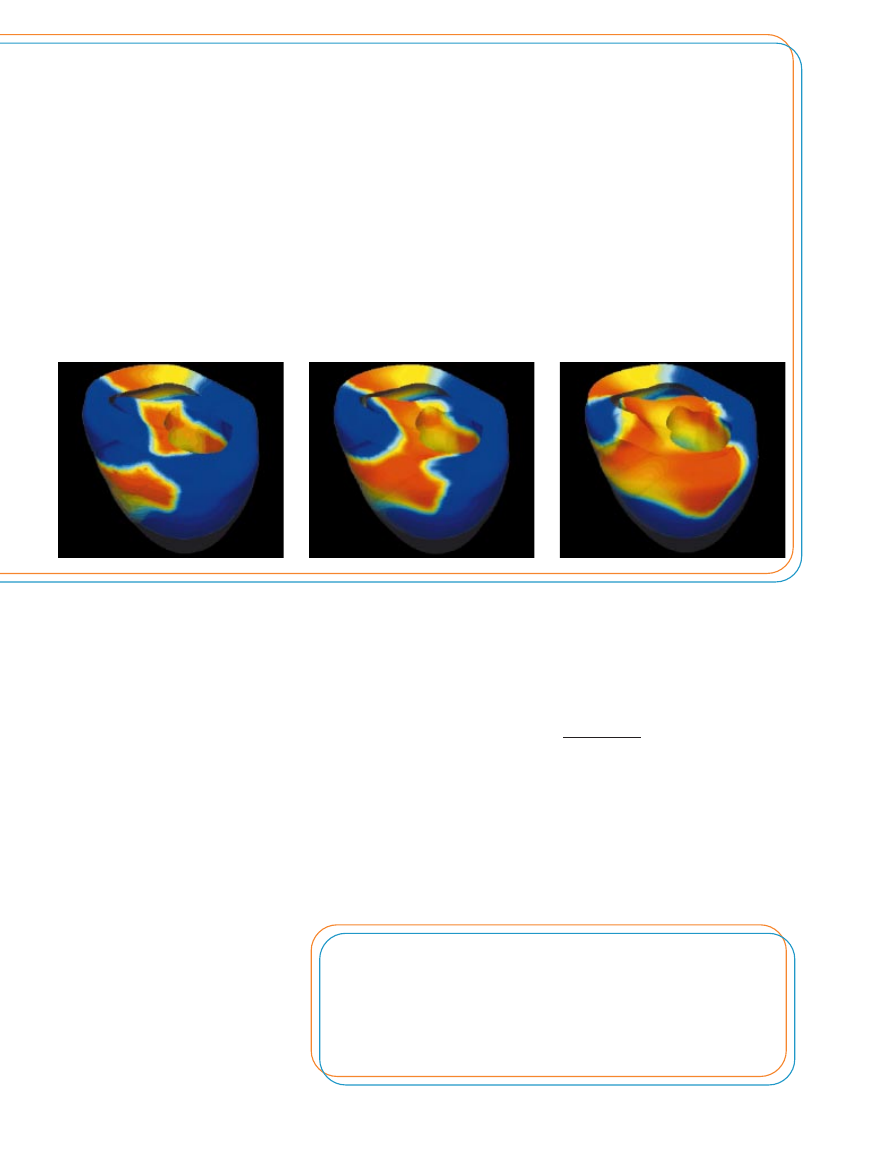
NA KOMPUTEROWYM MODELU SKURCZU SERCA widaç
przep∏ywajàce przez nie fale nieskoordynowanej aktywnoÊci elek-
trycznej. Model stworzono na podstawie zmian w ekspresji czte-
rech genów, których funkcjonowanie w przypadku przewlek∏ej
niewydolnoÊci mi´Ênia sercowego ulega zmianie.
Wyszukiwarka
Podobne podstrony:
Bioinformatyka6
bioinformatyczneBD lab1
Bioinformatyka4
sss teoria, Biotech, BIOTECHNOLOGIA, Semestr V, Spec. Bioinf, SSS, Egzamin
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
cw1 Zadania, Biotech, BIOTECHNOLOGIA, Semestr V, Spec. Bioinf, SSS, LAB, Lab 2
bioinformatyka w9 2008 web
Bonanza
elementy bioinformatyki wyklad2
bioinformatyka Bioinf8
bioinformatyka w6 2008 web
bioinformatyka, Bioinf11, 1
Bioinformatyka wykład 1
16 bioinformatryka
bioinfo-pyt odp-1, BIOINFORMATYKA
Bioinformatyka wykład 3
bioinformatyka w11 2008 web
BIOINFORMATYKA, Nauka - różności, Fizyka medyczna, Biofizyka
więcej podobnych podstron