DRUGA I TRZECIA GENERACJA SEKWENCJONOWANIA
Druga generacja
- wyizolować DNA z dowolnej próbki - eDNA - environmental DNA, DNA wyizolowane np. z wody, gleby, z wszystkich obecnych tam bakterii
- zaopatrzyć pofragmentowane DNA (kilkaset nt) w dodatkowe fragmenty DNA za pomocą ligazy - dołączanie krótkich oligonukleotydów które mogą być starterami do PCR ale nie muszą, ligacja musi być bardzo wydajna
- nie ma etapu wklonowywania do wektorów - stosując tradycyjne metody nie udawało się uzyskiwać reprezentatywnej biblioteki, bo fragmentów DNA w przypadku eDNA jest bardzo dużo - ważne, bo każdy odcinek każdego genomu każdego mikroorganizmu obecnego w próbce musi być dostępny
- zalety: szybciej niż pierwszej generacji, bo zautomatyzowane, nie ma etapu klonowania
Metoda 454 sequencing - pirosekwencjonowanie 454
- Life Sciences
- shotgun sequencing
- etapy:
- DNA fragmentujemy dołączając oligonukleotydy do reakcji PCR oraz związek który można swoiście przyczepić do powierzchni kulek magnetycznych opłaszczonych ligandem np. biotyna-streptawidyna, najczęściej oligonukleotydy znakuje się biotyną, bo wiązanie biotna-streptawidyna jest bardzo silne, roztwór musi być odpowiednio rozcieńczony żeby teoretycznie każda kulka magnetyczna miała jeden fragment DNA
- emPCR - emulsyjny PCR, PCR robi się w kropelce wody zmieszanej z olejem, w każdej kropelce jest taka mała kulka, do której przyczepiony jest DNA
- denaturacja DNA, przemywanie, otrzymujemy kulkę z przymocowanym jednoniciowym DNA, taki jeżyk
- kropelki trafiają do mikrodołków i prowadzi się sekwencjonowanie - jedna płytka może zawierać kilkaset tysięcy mikrodołków - możemy jednocześnie prowadzić kilkaset tysięcy reakcji sekwencjonowania na raz - high throughput
Sekwencjonowanie - 3 enzymy (polimeraza, ATP-sulfurylaza, lucyferaza)
(Matryca)n + nukleotyd polimeraza (Matryca)n+1 + PPi
PPi + APS (adenozyno - 5`-fosfosiarczan) ATP-sulfurylaza ATP + SO42-
ATP + lucyferyna + O2 lucyferaza AMP + PPi + oxylucyferyna + CO2 + światło
Mamy kuleczki w mikrodołkach
Dodajemy jeden nukleotyd
Jeśli jest komplementarny mamy światło i kamera to rejestruje
Przemywamy dołki - apyraza - degraduje niewłączone nukleotydy, w tym ATP
Dodajemy kolejny nukleotyd
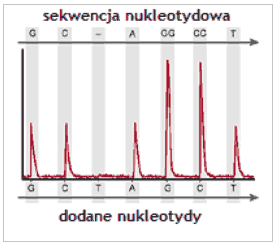
Tak mogą wyglądać wyniki. Jak pik jest wysoki znaczy, że były dwa po sobie np. G.
Sekwencjonowanie drugiej i trzeciej generacji -wyzwanie dla bioinformatyki żeby było możliwe przeanalizowanie tych ogromnych ilości danych.
Zastosowanie:
- możemy sekwencjonować eDNA
- możemy wyizolować całkowite RNA, przepisać na DNA i znowu sekwencjonować
-zastosowanie w badaniach populacji np. wykrywaniu mutacji pomiędzy osobnikami
-zastosowanie w badaniach nad zastosowaniem leków w przyszłości - jak podanie jakiegoś leku wpływa na zmianę ekspresji jakiś genów u człowieka
Trzecia generacja
- pojedynczą nić można odczytywać całą, obojętnie jak jest długa
- SMS - single molecule sequencing - nie trzeba amplifikować DNA, którego sekwencję chcemy poznać
- problem - polimeraza dodaje ok. 1000 nt/s trzeba ją więc trochę „zepsuć”, bo nie mamy na tyle wydajnych urządzeń które nadążyłyby z detekcją
-stosuje się zmodyfikowane nukleotydy np. z fluorofortami
2 główne strategie:
1. dołączamy nukleotydy i każdy z nich jest inaczej oznaczony np. fuloroforami albo
2. mamy nić gdzie na matrycy są nukleotydy zaznakowane fluoroforami i odcinamy nukleotyd po nukleotydzie kawałki matrycy i maszyna odczytuje jaki to był nukleotyd
Metoda VisiGen
-Stosuje FRET
-Polimeraza z donorem
-Akceptorem jest fluorescencyjny nukleotyd
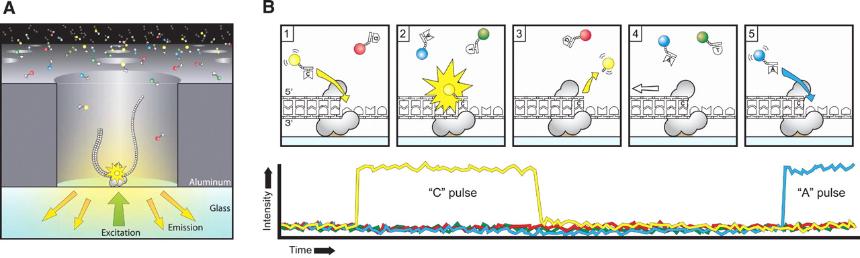
-Jeśli nukleotyd jest przyłączany do matrycy to następuje jego zbliżenie do polimerazy i zachodzi FRET i emisja światła, które jest sczytywane przez komputer
Metoda GridION system - Nanopore Technologies
- zastosowanie α-hemolizyny
- mikromaszynka wciąga DNA, odcinająca nukleotyd po nukleotydzie, a kolejna maszynka rejestruje jaki to nukleotyd
Link do filmiku, który był na wykładzie, opisującego jak to działa:
Pacific Biosciences - SMRT sequencing
-reakcja polimeryzacji
-10-21 l studzienki, w których dokonuje się rejestracji dołączanych nukleotydów już nawet bez FRET tak czułe są urządzenia
Link
Ion Torrent
- metoda pomiaru pH
Link
16s rRNA jest molekularnym markerem
Mamy gen 16S rRNA i on ma jakieś regiony zmienne, które są oflankowane sekwencjami silnie konserwatywnymi, amplifikujemy sobie te regiony zmienne żeby zanalizować ich sekwencje używając starterów komplementarnych do regionów konserwatywnych.
Jak to się robi?
-izolujemy albo DNA albo RNA i przepisujemy na DNA
-sekwencjonujemy ścieżką metagenomiczną wszystko na raz -stosując sekwencjonowanie 2 generacji możemy sprawdzić ile różnych regionów zmiennych mamy w danej populacji - im więcej tym więcej organizmów w tej populacji jest -umiemy hodować i tak tylko ułamek procenta tych mikroorganizmów
-w obrębie danego gatunku zróżnicowanie jest małe 1-2 nt, pomiędzy gatunkami już więcej - można szacunkowo określić ile grup organizmów mamy w próbce -w 1 g ziemi jest około 10 000 gatunków organizmów
-w ciele człowieka jest 10 razy więcej komórek bakteryjnych niż naszych własnych - kim w zasadzie jesteśmy skoro tych komórek bakteryjnych jest tak dużo?
-człowiek ok. 30 000 genów, reszta to sekwencje „milczące”
-organizm ludzki powinno się rozpatrywać całościowo razem z naszymi mikrobraćmi
Wyszukiwarka
Podobne podstrony:
Pytania z PCR 2, Studia Biotechnologia, Semestr 7 - II stopnia - mikrobiologia molekularna, Metody P
Zasady projektowania starterow, Studia Biotechnologia, Semestr 7 - II stopnia - mikrobiologia moleku
Dla wektorów z miejscem polilinkerowym, Studia Biotechnologia, Semestr 7 - II stopnia - mikrobiologi
Temat 10, Studia, I rok, I rok, II semestr, Mikrobiologia
14, studia, Budownctwo, Semestr II, fizyka, fizyka, Fizyka Sprawozdania, Nowy folder (2)
socjologia wykłady budrewicz I semestr II stopnia studnia, Studia, Rok 2, Socjologia, socjologia
Kwas cytrynowy, Studia, II semestr II rok, Mikrobiologia przemysłowa
Mikrobiologia ogólna- Pytania opracowane, Studia Biotechnologia, Semestr 3, Mikrobiologia ogólna
petrologia2 14.10, Studia (Geologia,GZMIW UAM), II rok, Petrologia, Wykłady, Wykłady
Pytania z mikrobiologii - pierwszy termin, Studia Biotechnologia, Semestr 4, Mikrobiologia przemysło
SPRAWOZDANIE MA 10.06.2012, Studia, AiR, SEMESTR II, MA
m4, Studia, II semestr II rok, Mikrobiologia przemysłowa
met1, Studia - Socjologia - Semestr II, metodologia
Cwiczenie zabawowe, STUDIA, Polibuda - semestr II, Hydraulika i hydrologia, laborki z hydro
linia cisnien, STUDIA, Polibuda - semestr II, Hydraulika i hydrologia, laborki z hydro, laborki
Kopia Opis techniczny B, Skrypty, UR - materiały ze studiów, studia, studia, 4 STASZEK, Semestr II,
odpowiedzi, Studia, Stopień 2 Semestr II, Zespolona, Analiza zespolona (aivliska), Analiza zespolona
więcej podobnych podstron