1
Metylacja
Metylacja
DNA oraz ekspansja
DNA oraz ekspansja
tr
tr
ó
ó
jnukleotydowa
jnukleotydowa
dr hab. Ewa Balcerczak
dr hab. Ewa Balcerczak
W biochemii
W biochemii metylacja
odnosi si
odnosi si
ę
ę
do zamiany
do zamiany
atomu wodoru na grup
atomu wodoru na grup
ę
ę
metylow
metylow
ą
ą
.
.
Metylacja
Metylacja
DNA jest typem modyfikacji DNA,
DNA jest typem modyfikacji DNA,
kt
kt
ó
ó
ra mo
ra mo
ż
ż
e zosta
e zosta
ć
ć
odziedziczona i p
odziedziczona i p
ó
ó
ź
ź
niej
niej
usuni
usuni
ę
ę
ta bez zmiany oryginalnej sekwencji DNA.
ta bez zmiany oryginalnej sekwencji DNA.
Zosta
Zosta
ł
ł
o udowodnione,
o udowodnione,
ż
ż
e
e
metylacja
metylacja
DNA
DNA
wyst
wyst
ę
ę
puje w wielu istotnych biologicznych
puje w wielu istotnych biologicznych
procesach, takich jak regulacja
procesach, takich jak regulacja
imprintingu
imprintingu
rodzicielskiego,
rodzicielskiego,
unieczynnienie
unieczynnienie
chromosomu X
chromosomu X
czy rozw
czy rozw
ó
ó
j nowotwor
j nowotwor
ó
ó
w.
w.
Metylacja
Metylacja
jest
jest
poreplikacyjn
poreplikacyjn
ą
ą
enzymatyczn
enzymatyczn
ą
ą
modyfikacj
modyfikacj
ą
ą
DNA.
DNA.
Proces ten nale
Proces ten nale
ż
ż
y do zmian epigenetycznych,
y do zmian epigenetycznych,
kt
kt
ó
ó
re w odr
re w odr
ó
ó
ż
ż
nieniu od genetycznych nie
nieniu od genetycznych nie
zale
zale
żą
żą
od sekwencji DNA i polegaj
od sekwencji DNA i polegaj
ą
ą
na zmianie
na zmianie
funkcji lub ekspresji genu.
funkcji lub ekspresji genu.
S
S
ą
ą
to zmiany odwracalne. Opr
to zmiany odwracalne. Opr
ó
ó
cz
cz
metylacji
metylacji
do
do
tych zmian nale
tych zmian nale
ż
ż
y zmiana struktury
y zmiana struktury
chromatyny spowodowana modyfikacj
chromatyny spowodowana modyfikacj
ą
ą
bia
bia
ł
ł
ek
ek
histonowych
histonowych
.
.
Metylacja
Metylacja
to proces kowalencyjnego przy
to proces kowalencyjnego przy
łą
łą
czania grup
czania grup
metylowych do zasad azotowych nukleotyd
metylowych do zasad azotowych nukleotyd
ó
ó
w.
w.
W procesie tym uczestnicz
W procesie tym uczestnicz
ą
ą
enzymy przenosz
enzymy przenosz
ą
ą
ce
ce
grupy metylowe (
grupy metylowe (
metylotransferazy
metylotransferazy
).
).
Najcz
Najcz
ęś
ęś
ciej produktami
ciej produktami
metylacji
metylacji
s
s
ą
ą
:
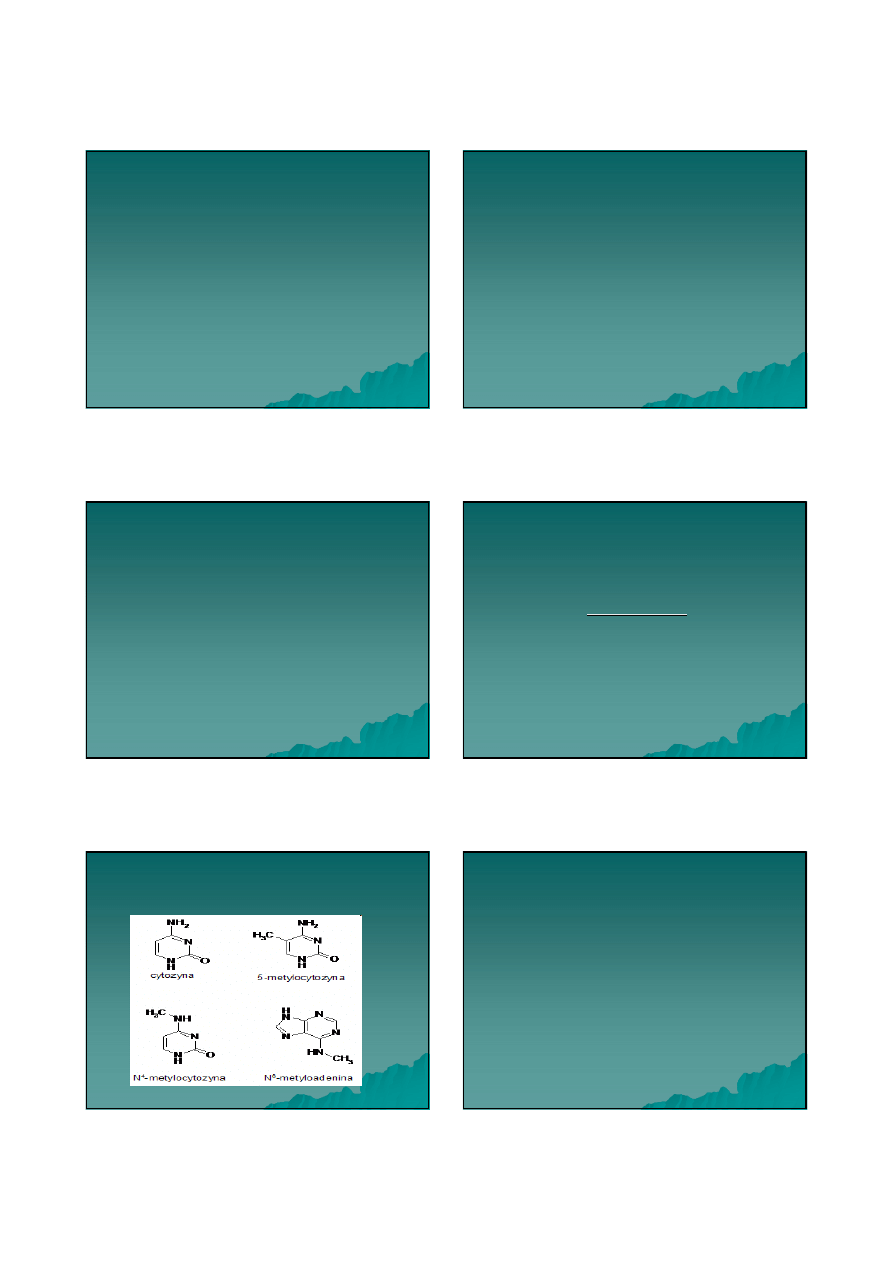
:
C5
C5
-
-
metylocytozyna
metylocytozyna
(m5C), N6
(m5C), N6
-
-
metyloadenina
metyloadenina
(m6A) i
(m6A) i
N4
N4
-
-
metylocytozyna
metylocytozyna
(m4C)
(m4C)
m4C wyst
m4C wyst
ę
ę
puje w obr
puje w obr
ę
ę
bie
bie
Procaryota
Procaryota
,
,
m5C u
m5C u
Procaryota
Procaryota
i
i
Eucaryota
Eucaryota
,
,
m6A u
m6A u
Procaryota
Procaryota
i zasadniczo u ni
i zasadniczo u ni
ż
ż
szych
szych
Eucaroyt
Eucaroyt
ó
ó
w
w
(bezkr
(bezkr
ę
ę
gowc
gowc
ó
ó
w)
w)
ROLA
ROLA
DNA
DNA
-
-
METYLOTRANSFERAZ
METYLOTRANSFERAZ
(DNMT) W PROCESIE METYLACJI
(DNMT) W PROCESIE METYLACJI
Enzymy te katalizuj
Enzymy te katalizuj
ą
ą
przy
przy
łą
łą
czenie si
czenie si
ę
ę
grup metylowych pochodz
grup metylowych pochodz
ą
ą
cych od donora
cych od donora
S
S
-
-
adenozylo
adenozylo
-
-
L
L
-
-
metioniny
metioniny
(
(
AdoMet
AdoMet
) do w
) do w
ę
ę
gla C5 w obr
gla C5 w obr
ę
ę
bie pier
bie pier
ś
ś
cienia
cienia
pirymidynowego cytozyny lub do grupy aminowej adeniny (N6) l ub c
pirymidynowego cytozyny lub do grupy aminowej adeniny (N6) l ub c
ytozyny (N4).
ytozyny (N4).
Produktami tych reakcji s
Produktami tych reakcji s
ą
ą
:
:
zmetylowany
zmetylowany
DNA i
DNA i
S
S
-
-
adenozylo
adenozylo
-
-
L
L
-
-
homocysteina
homocysteina
(
(
AdoHcy
AdoHcy
) .
) .
DNA
DNA
-
-
metylotransferazy
metylotransferazy
to rodzina enzym
to rodzina enzym
ó
ó
w, do kt
w, do kt
ó
ó
rych zalicza si
rych zalicza si
ę
ę
DNMT1,
DNMT1,
DNMT2, DNMT3A i DNMT3B oraz DN MT3L .
DNMT2, DNMT3A i DNMT3B oraz DN MT3L .
DNMT1 katalizuje 97
DNMT1 katalizuje 97
-
-
99,9% procesu
99,9% procesu
metylacji
metylacji
zachodz
zachodz
ą
ą
cego podczas mitozy.
cego podczas mitozy.
Enzym ten jest odpowiedzialny za przekazanie sta
Enzym ten jest odpowiedzialny za przekazanie sta
ł
ł
ego profilu
ego profilu
metylacji
metylacji
.
.
Rozpoznaje
Rozpoznaje
hemimetylowane
hemimetylowane
dinukleotydy
dinukleotydy
CpG
CpG
na matczynej i potomnej nici DNA
na matczynej i potomnej nici DNA
i przenosi grupy metylowe z
i przenosi grupy metylowe z
AdoMet
AdoMet
do
do
cytydyny
cytydyny
zlokalizowanej na
zlokalizowanej na
niezmetylowanej
niezmetylowanej
nici potomnej.
nici potomnej.
DNMT3A i 3B s
DNMT3A i 3B s
ą
ą
odpowiedzialne za
odpowiedzialne za
metylacj
metylacj
ę
ę
de
de
novo
novo
. Enzymy te s
. Enzymy te s
ą
ą
szczeg
szczeg
ó
ó
lnie
lnie
wa
wa
ż
ż
ne w rozwoju embrionalnym, kie dy to ustala si
ne w rozwoju embrionalnym, kie dy to ustala si
ę
ę
wz
wz
ó
ó
r
r
metylacji
metylacji
. Ich rola w
. Ich rola w
dojrza
dojrza
ł
ł
ych kom
ych kom
ó
ó
rkach jest nie do ko
rkach jest nie do ko
ń
ń
ca wyja
ca wyja
ś
ś
niona. Podejrzewa si
niona. Podejrzewa si
ę
ę
,
,
ż
ż
e DMNT3B
e DMNT3B
podtrzymuje
podtrzymuje
metylacj
metylacj
ę
ę
pericentrycznej
pericentrycznej
heterochromatyny. DNMT3L po
heterochromatyny. DNMT3L po
ś
ś
rednio
rednio
uczestniczy w procesie
uczestniczy w procesie
metylacji
metylacji
DNA. Nie ma w
DNA. Nie ma w
ł
ł
a
a
ś
ś
ciwo
ciwo
ś
ś
ci katalitycznych, ale
ci katalitycznych, ale
stymuluje aktywno
stymuluje aktywno
ść
ść
metylotransferaz
metylotransferaz
de
de
novo
novo
i zwi
i zwi
ę
ę
ksza ich powinowactwo do
ksza ich powinowactwo do
DNA.
DNA.

2
DINUKLEOTYDY I WYSPY CPG
DINUKLEOTYDY I WYSPY CPG
W genomie wyst
W genomie wyst
ę
ę
puj
puj
ą
ą
kr
kr
ó
ó
tkie (1000
tkie (1000
-
-
1500
1500
pz
pz
) odcinki DNA
) odcinki DNA
niezwykle bogate w
niezwykle bogate w
dinukleotydy
dinukleotydy
CpG
CpG
zwane wyspami
zwane wyspami
CpG
CpG
W genomie oko
W genomie oko
ł
ł
o 70
o 70
-
-
80%
80%
dinukleotyd
dinukleotyd
ó
ó
w
w
CpG
CpG
ma grup
ma grup
ę
ę
metylow
metylow
ą
ą
przy
przy
łą
łą
czon
czon
ą
ą
do cytozyny co sprawia, ze kodowane przez te
do cytozyny co sprawia, ze kodowane przez te
sekwencje geny
sekwencje geny
s
s
ą
ą
niedost
niedost
ę
ę
pne dla kom
pne dla kom
ó
ó
rkowych uk
rkowych uk
ł
ł
ad
ad
ó
ó
w
w
transkrypcyjnych
transkrypcyjnych
.
.
Chromatyna zawieraj
Chromatyna zawieraj
ą
ą
ca wyspy
ca wyspy
CpG
CpG
jest silnie
jest silnie
zacetylowana
zacetylowana
.
.
Tak
Tak
ą
ą
struktur
struktur
ę
ę
chromatyny nazywa si
chromatyny nazywa si
ę
ę
otwart
otwart
ą
ą
i mo
i mo
ż
ż
e by
e by
ć
ć
ona
ona
konsekwencj
konsekwencj
ą
ą
interakcji czynnik
interakcji czynnik
ó
ó
w
w
transkrypcyjnych
transkrypcyjnych
z genami
z genami
promotorowymi
promotorowymi
.
.
Oko
Oko
ł
ł
o po
o po
ł
ł
owa wszystkich ludzkich gen
owa wszystkich ludzkich gen
ó
ó
w zawiera
w zawiera
wyspy
wyspy
CpG
CpG
: s
: s
ą
ą
to tzw.
to tzw.
housekeeping
housekeeping
genes
genes
(niezb
(niezb
ę
ę
dne
dne
do funkcjonowania kom
do funkcjonowania kom
ó
ó
rki) i specyficzne tkankowe
rki) i specyficzne tkankowe
geny (ok. 40% wszystkich tkankowych gen
geny (ok. 40% wszystkich tkankowych gen
ó
ó
w)
w)
Wyspy
Wyspy
CpG
CpG
znajduj
znajduj
ą
ą
si
si
ę
ę
na ko
na ko
ń
ń
cu 5
cu 5
’
’
w regionach
w regionach
promotorowych
promotorowych
gen
gen
ó
ó
w maj
w maj
ą
ą
cych zasadnicze znaczenie
cych zasadnicze znaczenie
dla czynno
dla czynno
ś
ś
ci kom
ci kom
ó
ó
rki. Z regu
rki. Z regu
ł
ł
y s
y s
ą
ą
to geny
to geny
regulatorowe i nie ulegaj
regulatorowe i nie ulegaj
ą
ą
metylacji
metylacji
.
.
Brak
Brak
metylacji
metylacji
w regionach
w regionach
promotorowych
promotorowych
bywa
bywa
warunkiem wst
warunkiem wst
ę
ę
pnym dla kontrolowanej, aktywnej
pnym dla kontrolowanej, aktywnej
transkrypcji gen
transkrypcji gen
ó
ó
w.
w.
Metylacja
Metylacja
wysp
wysp
CpG
CpG
mo
mo
ż
ż
e skutkowa
e skutkowa
ć
ć
brakiem lub
brakiem lub
zaburzon
zaburzon
ą
ą
syntez
syntez
ą
ą
produkt
produkt
ó
ó
w tych gen
w tych gen
ó
ó
w.
w.
DINUKLEOTYDY I WYSPY C
DINUKLEOTYDY I WYSPY C
p
p
G
G
METYLACJA W TRAKCIE ROZWOJU
METYLACJA W TRAKCIE ROZWOJU
ORGANIZMU
ORGANIZMU
Najwi
Najwi
ę
ę
ksza cz
ksza cz
ęść
ęść
metylacji
metylacji
DNA zachodzi w
DNA zachodzi w
fazie S cyklu kom
fazie S cyklu kom
ó
ó
rkowego.
rkowego.
Wz
Wz
ó
ó
r
r
metylacji
metylacji
DNA jest charakterystyczny
DNA jest charakterystyczny
dla typu kom
dla typu kom
ó
ó
rki i podlega silnym zmianom
rki i podlega silnym zmianom
podczas rozwoju embrionalnego.
podczas rozwoju embrionalnego.
Metylacja
Metylacja
odgrywa znacz
odgrywa znacz
ą
ą
c
c
ą
ą
rol
rol
ę
ę
w regulacji
w regulacji
ekspresji gen
ekspresji gen
ó
ó
w podczas embriogenezy u
w podczas embriogenezy u
ssak
ssak
ó
ó
w i dalej podczas r
w i dalej podczas r
ó
ó
ż
ż
nicowania kom
nicowania kom
ó
ó
rek
rek
(myszy pozbawione genu
(myszy pozbawione genu
DNA
DNA
-
-
metylotransferazy
metylotransferazy
umieraj
umieraj
ą
ą
w ci
w ci
ą
ą
gu o
gu o
ś
ś
miu dni
miu dni
od zap
od zap
ł
ł
odnienia)
odnienia)
Metylacja
Metylacja
jest procesem dynamicznym: w zygocie niezw
jest procesem dynamicznym: w zygocie niezw
ł
ł
ocznie
ocznie
po zap
po zap
ł
ł
odnieniu nast
odnieniu nast
ę
ę
puj
puj
ą
ą
silne zmiany obejmuj
silne zmiany obejmuj
ą
ą
ce
ce
acetylacj
acetylacj
ę
ę
histon
histon
ó
ó
w i
w i
demetylacj
demetylacj
ę
ę
DNA. W stadium wczesnego
DNA. W stadium wczesnego
embrionu
embrionu
nast
nast
ę
ę
puje dramatyczna redukcja poziomu
puje dramatyczna redukcja poziomu
metylacji
metylacji
(tzw.
(tzw.
globalna
globalna
demetylacja
demetylacja
). Po implantacji nast
). Po implantacji nast
ę
ę
puje fala
puje fala
metylacji
metylacji
de
de
novo
novo
wi
wi
ę
ę
kszo
kszo
ś
ś
ci
ci
CpG
CpG
Tak dzieje si
Tak dzieje si
ę
ę
w przypadku genomu m
w przypadku genomu m
ę
ę
skiego, w genomie
skiego, w genomie
ż
ż
e
e
ń
ń
skim w tym czasie zachodzi pasywna
skim w tym czasie zachodzi pasywna
demetylacja
demetylacja
.
.
Podczas gastrulacji geny specyficzne tkankowo s
Podczas gastrulacji geny specyficzne tkankowo s
ą
ą
w
w
odpowiednich tkankach
odpowiednich tkankach
demetylowane
demetylowane
i ulegaj
i ulegaj
ą
ą
ekspresji, ale
ekspresji, ale
nale
nale
ż
ż
y podkre
y podkre
ś
ś
li
li
ć
ć
, ze wi
, ze wi
ę
ę
kszo
kszo
ść
ść
genomu pozostaje nadal
genomu pozostaje nadal
zmetylowana
zmetylowana
.
.
Kolejny etap intensywnej
Kolejny etap intensywnej
metylacji
metylacji
de
de
novo
novo
zale
zale
ż
ż
y od p
y od p
ł
ł
ci i
ci i
zachodzi podczas
zachodzi podczas
gametogenezy
gametogenezy
. Niewielki spadek
. Niewielki spadek
metylacji
metylacji
zaobserwowano w stadium
zaobserwowano w stadium
postembrionalnym
postembrionalnym
podobnie jak
podobnie jak
in
in
vitro
vitro
w starzej
w starzej
ą
ą
cych si
cych si
ę
ę
kom
kom
ó
ó
rkach
rkach
METYLACJA W TRAKCIE ROZWOJU
METYLACJA W TRAKCIE ROZWOJU
ORGANIZMU
ORGANIZMU
WP
WP
Ł
Ł
YW WIEKU NA POZIOM
YW WIEKU NA POZIOM
METYLACJI
METYLACJI
metylacja
metylacja
jest gatunkowo
jest gatunkowo
-
-
, tkankowo
, tkankowo
-
-
,
,
organellospecyficzna
organellospecyficzna
, lecz obserwuje si
, lecz obserwuje si
ę
ę
tak
tak
ż
ż
e zale
e zale
ż
ż
no
no
ść
ść
metylacji
metylacji
od wieku
od wieku
niekt
niekt
ó
ó
rzy naukowcy traktuj
rzy naukowcy traktuj
ą
ą
poziom
poziom
metylacji
metylacji
jako wska
jako wska
ź
ź
nik wieku lub narz
nik wieku lub narz
ę
ę
dzie do
dzie do
przewidywania d
przewidywania d
ł
ł
ugo
ugo
ś
ś
ci
ci
ż
ż
ycia. Nieprawid
ycia. Nieprawid
ł
ł
owa
owa
metylacja
metylacja
mo
mo
ż
ż
e prowadzi
e prowadzi
ć
ć
do przedwczesnego
do przedwczesnego
starzenia
starzenia
ZNACZENIE METYLACJI
ZNACZENIE METYLACJI
Metylacja
Metylacja
jest zwi
jest zwi
ą
ą
zana z nast
zana z nast
ę
ę
puj
puj
ą
ą
cymi
cymi
procesami:
procesami:
Imprinting
Imprinting
rodzicielski
rodzicielski
Inaktywacja chromosomu X w kom
Inaktywacja chromosomu X w kom
ó
ó
rkach
rkach
samic ssak
samic ssak
ó
ó
w
w
ł
ł
o
o
ż
ż
yskowych
yskowych
Regulacja ekspresji genu
Regulacja ekspresji genu
Modulacja struktury chromatyny
Modulacja struktury chromatyny
Zaburzenia metylacji są przyczyną chorób dziedzicznych, np.
zespołów Angelmana,Pradera- Williego, Beckwitha-Wiedemana,
a także transformacji nowotworowych ,
ICF (facial anomalies syndrome - zespół niedoboru odporności,
niestabilności centrometrów i anomalii twarzy),
syndromu Retta i zespołu łamliwego chromosomu X

3
Imprinting
Imprinting
rodzicielski
rodzicielski
Imprinting
Imprinting
rodzicielski, inaczej pi
rodzicielski, inaczej pi
ę
ę
tno
tno
genomowe
genomowe
lub pi
lub pi
ę
ę
tno
tno
rodzicielskie prowadzi do r
rodzicielskie prowadzi do r
ó
ó
ż
ż
nicowej ekspresji alleli niekt
nicowej ekspresji alleli niekt
ó
ó
rych
rych
gen
gen
ó
ó
w pochodz
w pochodz
ą
ą
cych od matki i od ojca.
cych od matki i od ojca.
Istnieje wiele gen
Istnieje wiele gen
ó
ó
w, kt
w, kt
ó
ó
rych ekspresja zale
rych ekspresja zale
ż
ż
y od tego, na
y od tego, na
kt
kt
ó
ó
rym
rym
allelu
allelu
–
–
pochodz
pochodz
ą
ą
cym od matki czy od ojca
cym od matki czy od ojca
-
-
si
si
ę
ę
znajduj
znajduj
ą
ą
.
.
Dwa allele
Dwa allele
imprintingowanych
imprintingowanych
gen
gen
ó
ó
w r
w r
ó
ó
ż
ż
ni
ni
ą
ą
si
si
ę
ę
stopniem
stopniem
metylacji
metylacji
DNA, struktur
DNA, struktur
ą
ą
chromatyny (modyfikacja histon
chromatyny (modyfikacja histon
ó
ó
w,
w,
nadwra
nadwra
ż
ż
liwo
liwo
ść
ść
na
na
nukleaz
nukleaz
ę
ę
) i czasem replikacji.
) i czasem replikacji.
Cech
Cech
ą
ą
imprintingu
imprintingu
jest dziedziczno
jest dziedziczno
ść
ść
podczas podzia
podczas podzia
ł
ł
ó
ó
w
w
kom
kom
ó
ó
rkowych i odwracalno
rkowych i odwracalno
ść
ść
podczas
podczas
gametogenezy
gametogenezy
.
.
Obecnie znanych jest ok. 30 ge n
Obecnie znanych jest ok. 30 ge n
ó
ó
w wyst
w wyst
ę
ę
puj
puj
ą
ą
cych u ludzi i u
cych u ludzi i u
myszy podlegaj
myszy podlegaj
ą
ą
cych
cych
imprintingowi
imprintingowi
Jednym z najlepszych modeli u
Jednym z najlepszych modeli u
ż
ż
ywanych do badania
ywanych do badania
procesu
procesu
imprintingu
imprintingu
jest system gen
jest system gen
ó
ó
w H19/IGF2.
w H19/IGF2.
H19 i IGF2 to 2 geny zlokalizowane w odleg
H19 i IGF2 to 2 geny zlokalizowane w odleg
ł
ł
o
o
ś
ś
ci oko
ci oko
ł
ł
o
o
90 par zasad. Gen H19 ulega transkrypcji tylko gdy
90 par zasad. Gen H19 ulega transkrypcji tylko gdy
jest zlokalizowany na chromosomie matczynym, za to
jest zlokalizowany na chromosomie matczynym, za to
IGF2 odwrotnie, tylko gdy znajduje si
IGF2 odwrotnie, tylko gdy znajduje si
ę
ę
na
na
chromosomie ojcowskim.
chromosomie ojcowskim.
Kluczow
Kluczow
ą
ą
rol
rol
ę
ę
w regulacji tych gen
w regulacji tych gen
ó
ó
w odgrywa odcinek
w odgrywa odcinek
z
z
ł
ł
o
o
ż
ż
ony z 2 tysi
ony z 2 tysi
ę
ę
cy par zasad DNA zlokalizowany tu
cy par zasad DNA zlokalizowany tu
ż
ż
przed regionem
przed regionem
promotorowym
promotorowym
genu H19.
genu H19.
Ten fragment nazywany jest
Ten fragment nazywany jest
DMR (
DMR (
Differentially
Differentially
Methylated
Methylated
Region),
Region),
poniewa
poniewa
ż
ż
jest silnie
jest silnie
zmetylowany
zmetylowany
na ojcowskim chromosomie, a
na ojcowskim chromosomie, a
niezmetylowany
niezmetylowany
na matczynym.
na matczynym.
Imprinting
Imprinting
rodzicielski
rodzicielski
Zgodnie z zasad
Zgodnie z zasad
ą
ą
dziedziczenia geny podlegaj
dziedziczenia geny podlegaj
ą
ą
ce
ce
ekspresji z ojcowskiego
ekspresji z ojcowskiego
allelu
allelu
stymuluj
stymuluj
ą
ą
wzrost i
wzrost i
r
r
ó
ó
ż
ż
nicowanie kom
nicowanie kom
ó
ó
rek, a geny z matczynego
rek, a geny z matczynego
allelu
allelu
maj
maj
ą
ą
dzia
dzia
ł
ł
anie przeciwne. Na przyk
anie przeciwne. Na przyk
ł
ł
ad: usuni
ad: usuni
ę
ę
cie
cie
gen
gen
ó
ó
w PEG1/MEST lub PEG3 z ojcowskiego
w PEG1/MEST lub PEG3 z ojcowskiego
chromosomu prowadzi do zaburzenia wzrostu.
chromosomu prowadzi do zaburzenia wzrostu.
Usuni
Usuni
ę
ę
cie genu H19 powoduje nadmierny rozrost
cie genu H19 powoduje nadmierny rozrost
p
p
ł
ł
odu.
odu.
Prawid
Prawid
ł
ł
owa ekspresja gen
owa ekspresja gen
ó
ó
w podlegaj
w podlegaj
ą
ą
cych
cych
imprintingowi
imprintingowi
jest bardzo wa
jest bardzo wa
ż
ż
na w ontogenezie. Ka
na w ontogenezie. Ka
ż
ż
da
da
nieprawid
nieprawid
ł
ł
owo
owo
ść
ść
mo
mo
ż
ż
e powodowa
e powodowa
ć
ć
nowotwory lub takie
nowotwory lub takie
genetyczne zaburzenia, jak zespo
genetyczne zaburzenia, jak zespo
ł
ł
y
y
Prader
Prader
-
-
Willy
Willy
’
’
ego
ego
,
,
Angelmana
Angelmana
i
i
Bechwith
Bechwith
-
-
Wiedemann
Wiedemann
’
’
a
a
Imprinting
Imprinting
rodzicielski
rodzicielski
Inaktywacja chromosomu X w
Inaktywacja chromosomu X w
kom
kom
ó
ó
rkach samic ssak
rkach samic ssak
ó
ó
w
w
ł
ł
o
o
ż
ż
yskowych
yskowych
Samice ssak
Samice ssak
ó
ó
w
w
ł
ł
o
o
ż
ż
yskowych posiadaj
yskowych posiadaj
ą
ą
dwie kopie
dwie kopie
chromosomu X.
chromosomu X.
Inaktywacja chromosomu X jest mechanizmem
Inaktywacja chromosomu X jest mechanizmem
kompensacyjnym, wyr
kompensacyjnym, wyr
ó
ó
wnuj
wnuj
ą
ą
cym poziom ekspresji
cym poziom ekspresji
gen
gen
ó
ó
w zlokalizowanych na chromosomach p
w zlokalizowanych na chromosomach p
ł
ł
ciowych.
ciowych.
Oba chromosomy s
Oba chromosomy s
ą
ą
aktywne w stadium wczesnego
aktywne w stadium wczesnego
zarodka. P
zarodka. P
ó
ó
ź
ź
niej w kom
niej w kom
ó
ó
rkach p
rkach p
ł
ł
odu dochodzi do
odu dochodzi do
losowej inaktywacji jednego chromosomu X.
losowej inaktywacji jednego chromosomu X.
Zmiany te s
Zmiany te s
ą
ą
przekazywane podczas podzia
przekazywane podczas podzia
ł
ł
ó
ó
w
w
mitotycznych
mitotycznych
W odr
W odr
ó
ó
ż
ż
nieniu od
nieniu od
imprintingu
imprintingu
inaktywacja chromosomu X obejm uje ca
inaktywacja chromosomu X obejm uje ca
ł
ł
y
y
chromosom.
chromosom.
Proces ten jest kierowany przez
Proces ten jest kierowany przez
Xic
Xic
(
(
X
X
-
-
inactivation
inactivation
center).
center).
Xic
Xic
zawiera 2 wa
zawiera 2 wa
ż
ż
ne
ne
geny: XIST i TSIX koduj
geny: XIST i TSIX koduj
ą
ą
ce
ce
nieulegaj
nieulegaj
ą
ą
ce
ce
translacji
translacji
mRNA
mRNA
. TSIX jest
. TSIX jest
zlokalizowany w regionie 3
zlokalizowany w regionie 3
’
’
XIST i jego
XIST i jego
mRNA
mRNA
jest
jest
antysensowym
antysensowym
transkryptem
transkryptem
dla XIST
dla XIST
mRNA
mRNA
.
.
Pocz
Pocz
ą
ą
tkowo XIST i TSIX ulegaj
tkowo XIST i TSIX ulegaj
ą
ą
ekspresji r
ekspresji r
ó
ó
wnolegle z obu chromosom
wnolegle z obu chromosom
ó
ó
w X, ale
w X, ale
represja transkrypcji TSIX na jednym z chromosom
represja transkrypcji TSIX na jednym z chromosom
ó
ó
w X prowadzi do
w X prowadzi do
zwi
zwi
ę
ę
kszenia poziomu ekspresji XIST i dalej do tworzenia si
kszenia poziomu ekspresji XIST i dalej do tworzenia si
ę
ę
XIST
XIST
mRNA
mRNA
w
w
chromosomie X, substytucji histonu H2A poprzez macro
chromosomie X, substytucji histonu H2A poprzez macro
-
-
H2A i
H2A i
metylacji
metylacji
lizyny w
lizyny w
pozycji 9. i 27. histonu H3.
pozycji 9. i 27. histonu H3.
Inaktywacja gen
Inaktywacja gen
ó
ó
w zlokalizowanych na chromosom ie X nast
w zlokalizowanych na chromosom ie X nast
ę
ę
puje podczas
puje podczas
rozprzestrzeniania si
rozprzestrzeniania si
ę
ę
nieaktywnej struktury chromaty ny wzd
nieaktywnej struktury chromaty ny wzd
ł
ł
u
u
ż
ż
chromosomu.
chromosomu.
Jest to proces, kt
Jest to proces, kt
ó
ó
ry obejmuje dzia
ry obejmuje dzia
ł
ł
anie r
anie r
ó
ó
ż
ż
nych czynnik
nych czynnik
ó
ó
w, w tym modyfikacj
w, w tym modyfikacj
ę
ę
histon
histon
ó
ó
w i cz
w i cz
ęś
ęś
ciow
ciow
ą
ą
metylacj
metylacj
ę
ę
wysp
wysp
CpG
CpG
powi
powi
ą
ą
zanych z promotorami tych gen
zanych z promotorami tych gen
ó
ó
w
w
.
.
Nieaktywny chromosom ulega akt ywacji w profazie I podzia
Nieaktywny chromosom ulega akt ywacji w profazie I podzia
ł
ł
u
u
mejotycznego
mejotycznego
w
w
kom
kom
ó
ó
rkach linii p
rkach linii p
ł
ł
ciowej
ciowej
Inaktywacja chromosomu X w
Inaktywacja chromosomu X w
kom
kom
ó
ó
rkach samic ssak
rkach samic ssak
ó
ó
w
w
ł
ł
o
o
ż
ż
yskowych
yskowych
Regulacja ekspresji gen
Regulacja ekspresji gen
ó
ó
w i
w i
modulacja struktury chromatyny
modulacja struktury chromatyny
Metylacja
Metylacja
DNA powoduje na og
DNA powoduje na og
ó
ó
ł
ł
wyciszenie ekspresji
wyciszenie ekspresji
gen
gen
ó
ó
w.
w.
Wi
Wi
ąż
ąż
e si
e si
ę
ę
to ze zmian
to ze zmian
ą
ą
struktury chromatyny na
struktury chromatyny na
nieaktywn
nieaktywn
ą
ą
, skondensowan
, skondensowan
ą
ą
(heterochromatyna).
(heterochromatyna).
Bia
Bia
ł
ł
ka
ka
MeCPs
MeCPs
(
(
Methyl
Methyl
-
-
CpG
CpG
-
-
Binding
Binding
Proteins
Proteins
) wi
) wi
ążą
ążą
si
si
ę
ę
ze
ze
zmetylowanym
zmetylowanym
DNA poprzez domeny wi
DNA poprzez domeny wi
ążą
ążą
ce
ce
fragment
fragment
zmetylowany
zmetylowany
(
(
MBD
MBD
-
-
Methyl
Methyl
Binding
Binding
Domain
Domain
).
).
MeCPs
MeCPs
oddzia
oddzia
ł
ł
uj
uj
ą
ą
z
z
deacetylaz
deacetylaz
ą
ą
histon
histon
ó
ó
w oraz
w oraz
kompleksami
kompleksami
remodeluj
remodeluj
ą
ą
cymi
cymi
chromatyn
chromatyn
ę
ę
. Dok
. Dok
ł
ł
adny
adny
mechanizm tego procesu nie jest poznany.
mechanizm tego procesu nie jest poznany.
Skutkiem powy
Skutkiem powy
ż
ż
szych zmian jest
szych zmian jest
deacetylacja
deacetylacja
histon
histon
ó
ó
w i
w i
metylacja
metylacja
lizyny 9. histonu H3.
lizyny 9. histonu H3.

4
Wp
Wp
ł
ł
yw
yw
metylacji
metylacji
na rozw
na rozw
ó
ó
j
j
nowotworu
nowotworu
podczas powstawania nowotworu
podczas powstawania nowotworu
epigenotyp
epigenotyp
kom
kom
ó
ó
rki znacz
rki znacz
ą
ą
co si
co si
ę
ę
zmienia.
zmienia.
Mo
Mo
ż
ż
liwe s
liwe s
ą
ą
nast
nast
ę
ę
puj
puj
ą
ą
ce zmiany w
ce zmiany w
metylacji
metylacji
DNA:
DNA:
Hipermetylacja
Hipermetylacja
wysp
wysp
CpG
CpG
Hipometylacja
Hipometylacja
gen
gen
ó
ó
w normalnie
w normalnie
zmetylowanych
zmetylowanych
(globalna
(globalna
demetylacja
demetylacja
gen
gen
ó
ó
w i lokalna w genach
w i lokalna w genach
promotorowych
promotorowych
)
)
Tranzycja
Tranzycja
: 5
: 5
-
-
metylocytozyna do tyminy i
metylocytozyna do tyminy i
metylacja
metylacja
„
„
non
non
-
-
CpG
CpG
”
”
w
w
kom
kom
ó
ó
rkach nowotworowych
rkach nowotworowych
Indukcja niestabilno
Indukcja niestabilno
ś
ś
ci chromosom
ci chromosom
ó
ó
w
w
-
-
poprzez nieprawid
poprzez nieprawid
ł
ł
owy poziom
owy poziom
metylacji
metylacji
dochodzi do zmiennego poziomu ekspresji gen
dochodzi do zmiennego poziomu ekspresji gen
ó
ó
w, co objawia
w, co objawia
si
si
ę
ę
niestabilno
niestabilno
ś
ś
ci
ci
ą
ą
chromosom
chromosom
ó
ó
w i stymuluje tym samym rozw
w i stymuluje tym samym rozw
ó
ó
j
j
nowotworu .
nowotworu .
Hipometylacja
Hipometylacja
Wp
Wp
ł
ł
yw
yw
hipometylacji
hipometylacji
na
na
onkogenez
onkogenez
ę
ę
wyra
wyra
ż
ż
a si
a si
ę
ę
nadekspresj
nadekspresj
ą
ą
onkogen
onkogen
ó
ó
w spowodowan
w spowodowan
ą
ą
demetylacj
demetylacj
ą
ą
region
region
ó
ó
w
w
promotorowych
promotorowych
tych onkogen
tych onkogen
ó
ó
w. W ten spos
w. W ten spos
ó
ó
b dochodzi do nadmiernej
b dochodzi do nadmiernej
stymulacji proliferacji kom
stymulacji proliferacji kom
ó
ó
rkowej.
rkowej.
W raku jelita grubego stwierdzono redukcj
W raku jelita grubego stwierdzono redukcj
ę
ę
og
og
ó
ó
lnej
lnej
metylacji
metylacji
o
o
10
10
-
-
30%, a w
30%, a w
przednowotworowych
przednowotworowych
stadiach gruczolaka
stadiach gruczolaka
zaobserwowano znacz
zaobserwowano znacz
ą
ą
ce zmniejszenie ilo
ce zmniejszenie ilo
ś
ś
ci 5
ci 5
-
-
metylocytozyny.
metylocytozyny.
Hipometylacja
Hipometylacja
powy
powy
ż
ż
ej 50% wyst
ej 50% wyst
ę
ę
puje w nowotworach klatki
puje w nowotworach klatki
piersiowej. Przy nowotworach krwi
piersiowej. Przy nowotworach krwi
hipometylacja
hipometylacja
wyst
wyst
ę
ę
puje w
puje w
przewlek
przewlek
ł
ł
ej bia
ej bia
ł
ł
aczce limfatycznej (CLL), podczas gdy w
aczce limfatycznej (CLL), podczas gdy w
przewlek
przewlek
ł
ł
ej (CML) i ostrej bia
ej (CML) i ostrej bia
ł
ł
aczce szpikowej (AML) oraz w
aczce szpikowej (AML) oraz w
szpiczaku mnogim wyst
szpiczaku mnogim wyst
ę
ę
puje tylko niewielka zmiana w schemacie
puje tylko niewielka zmiana w schemacie
metylacji
metylacji
DNA.
DNA.
Globalna
Globalna
demetylacja
demetylacja
wyst
wyst
ę
ę
puje we wczesnych stadiach
puje we wczesnych stadiach
nowotwor
nowotwor
ó
ó
w klatki piersiowej, jelita grubego i CLL.
w klatki piersiowej, jelita grubego i CLL.
W raku jelita grubego ponadto
W raku jelita grubego ponadto
hipometylacja
hipometylacja
wyst
wyst
ę
ę
puje w
puje w
zdrowych tkankach przylegaj
zdrowych tkankach przylegaj
ą
ą
cych do guza. W innych
cych do guza. W innych
nowotworach
nowotworach
np
np
. raku
. raku
w
w
ą
ą
trobowokom
trobowokom
ó
ó
rkowym
rkowym
hipometylacja
hipometylacja
wzrasta wraz z zaawansowaniem i stadium histologicznym guza.
wzrasta wraz z zaawansowaniem i stadium histologicznym guza.
Hipometylacja
Hipometylacja
specyficznych gen
specyficznych gen
ó
ó
w wyst
w wyst
ę
ę
puje w nowotworach
puje w nowotworach
jelita okr
jelita okr
ęż
ęż
nicy, trzustki, klatki piersiowej,
nicy, trzustki, klatki piersiowej,
ż
ż
o
o
łą
łą
dka, prostaty i w
dka, prostaty i w
bia
bia
ł
ł
aczce. Z regu
aczce. Z regu
ł
ł
y s
y s
ą
ą
to geny reguluj
to geny reguluj
ą
ą
ce wzrost, koduj
ce wzrost, koduj
ą
ą
ce
ce
enzymy, wa
enzymy, wa
ż
ż
ne dla rozwoju organizmu, geny
ne dla rozwoju organizmu, geny
tkankowospecyficzne
tkankowospecyficzne
i onkogeny .
i onkogeny .
Hipometylacja
Hipometylacja
Hipermetylacja
Hipermetylacja
Najcz
Najcz
ę
ę
stszymi miejscami podlegaj
stszymi miejscami podlegaj
ą
ą
cymi
cymi
hipermetylacji
hipermetylacji
w
w
r
r
ó
ó
ż
ż
nych rodzajach nowotwor
nych rodzajach nowotwor
ó
ó
w s
w s
ą
ą
chromosomy 3p, 11p i 17p .
chromosomy 3p, 11p i 17p .
Zjawisko to zachodzi w obr
Zjawisko to zachodzi w obr
ę
ę
bie wysp
bie wysp
CpG
CpG
, kt
, kt
ó
ó
re normalnie w
re normalnie w
genomie pozostaj
genomie pozostaj
ą
ą
niezmetylowane
niezmetylowane
. Najwa
. Najwa
ż
ż
niejsz
niejsz
ą
ą
tego
tego
konsekwencj
konsekwencj
ą
ą
jest wyciszenie funkcji gen
jest wyciszenie funkcji gen
ó
ó
w
w
supresorowych
supresorowych
.
.
Przyk
Przyk
ł
ł
adem mo
adem mo
ż
ż
e by
e by
ć
ć
hipermetylacja
hipermetylacja
promotora genu p16
promotora genu p16
(INK4A), kt
(INK4A), kt
ó
ó
ra wyst
ra wyst
ę
ę
puje w wielu nowotworach. p16 jest
puje w wielu nowotworach. p16 jest
inhibitorem kinazy
inhibitorem kinazy
cyklinozale
cyklinozale
ż
ż
nej
nej
, kt
, kt
ó
ó
ry negatywnie reguluje
ry negatywnie reguluje
przej
przej
ś
ś
cie kom
cie kom
ó
ó
rki z fazy G1 do S.
rki z fazy G1 do S.
Zatem nieprawid
Zatem nieprawid
ł
ł
owa ekspresja prowadzi do zaburze
owa ekspresja prowadzi do zaburze
ń
ń
cyklu
cyklu
kom
kom
ó
ó
rkowego i utrat
rkowego i utrat
ą
ą
kontroli nad nim, co stymuluje proliferacj
kontroli nad nim, co stymuluje proliferacj
ę
ę
i wp
i wp
ł
ł
ywa na rozw
ywa na rozw
ó
ó
j nowotworu . Zjawisko to zachodzi w
j nowotworu . Zjawisko to zachodzi w
nowotworach: p
nowotworach: p
ę
ę
cherza moczowego, nosa i gard
cherza moczowego, nosa i gard
ł
ł
a, trzustki, jelita
a, trzustki, jelita
grubego, p
grubego, p
ł
ł
uc, czerniaku, glejaku i bia
uc, czerniaku, glejaku i bia
ł
ł
aczce. W kancerogenezie
aczce. W kancerogenezie
raka gruczo
raka gruczo
ł
ł
owego prze
owego prze
ł
ł
yku
yku
metylacja
metylacja
promotora genu p16 mo
promotora genu p16 mo
ż
ż
e
e
pojawia
pojawia
ć
ć
si
si
ę
ę
ju
ju
ż
ż
w stadium metaplazji .
w stadium metaplazji .
Przeprowadzono badanie profilu
Przeprowadzono badanie profilu
hipermetylacji
hipermetylacji
15 nowotwor
15 nowotwor
ó
ó
w: okr
w: okr
ęż
ęż
nicy,
nicy,
ż
ż
o
o
łą
łą
dka,
dka,
trzustki, w
trzustki, w
ą
ą
troby, nerki, p
troby, nerki, p
ł
ł
uca, g
uca, g
ł
ł
owy, szyi, piersi, jajnik
owy, szyi, piersi, jajnik
ó
ó
w, p
w, p
ę
ę
cherza moczowego,
cherza moczowego,
endometrium
endometrium
, m
, m
ó
ó
zgu,
zgu,
ch
ch
ł
ł
oniaka
oniaka
i bia
i bia
ł
ł
aczki. Analiza obejmowa
aczki. Analiza obejmowa
ł
ł
a 3 grupy gen
a 3 grupy gen
ó
ó
w:
w:
·
·
Geny supresorowe: p16, p15, p14, p73, APC (gen polipowato
Geny supresorowe: p16, p15, p14, p73, APC (gen polipowato
ś
ś
ci jelita grubego) i
ci jelita grubego) i
BRCA1
BRCA1
Geny odpowiedzialne za napraw
Geny odpowiedzialne za napraw
ę
ę
DNA lub metabolizm
DNA lub metabolizm
ksenobiotyk
ksenobiotyk
ó
ó
w
w
: hMLH1,
: hMLH1,
GSTP1 (gen
GSTP1 (gen
transferazy
transferazy
S
S
-
-
glutationu
glutationu
klasy p), MGMT
klasy p), MGMT
Geny odpowiedzialne za inwazyjno
Geny odpowiedzialne za inwazyjno
ść
ść
i przerzuty: CDH1, TIMP3, DAPK
i przerzuty: CDH1, TIMP3, DAPK
Metylacja
Metylacja
w przynajmniej jednym genie by
w przynajmniej jednym genie by
ł
ł
a obecna w ka
a obecna w ka
ż
ż
dym typie nowotworu.
dym typie nowotworu.
Profile
Profile
metylacji
metylacji
by
by
ł
ł
y zale
y zale
ż
ż
ne zar
ne zar
ó
ó
wno od genu jak i od nowotworu . Niekt
wno od genu jak i od nowotworu . Niekt
ó
ó
re
re
geny,
geny,
np
np
. p16, MGMT, DAPK by
. p16, MGMT, DAPK by
ł
ł
y
y
zmetylowane
zmetylowane
w r
w r
ó
ó
ż
ż
nych typach nowotwor
nych typach nowotwor
ó
ó
w
w
(okr
(okr
ęż
ęż
nicy, p
nicy, p
ł
ł
uca, g
uca, g
ł
ł
owy, szyi, jajnik
owy, szyi, jajnik
ó
ó
w, p
w, p
ę
ę
cherza moczowego,
cherza moczowego,
ch
ch
ł
ł
oniaka
oniaka
i
i
bia
bia
ł
ł
aczki).
aczki).
Hipermetylacja
Hipermetylacja
p14, APC, p16, MGMT, hMLH1 wy st
p14, APC, p16, MGMT, hMLH1 wy st
ę
ę
powa
powa
ł
ł
a w
a w
nowotworach przewodu pokarmowego (okr
nowotworach przewodu pokarmowego (okr
ęż
ęż
nica,
nica,
ż
ż
o
o
łą
łą
dek), a GSTP1 w
dek), a GSTP1 w
steroidozale
steroidozale
ż
ż
nych
nych
nowotworach (piersi, w
nowotworach (piersi, w
ą
ą
troby, prostaty).
troby, prostaty).
Hipermetylacja
Hipermetylacja
Metylacja
Metylacja
„
„
non
non
-
-
CpG
CpG
”
”
w kom
w kom
ó
ó
rkach
rkach
nowotworowych
nowotworowych
Sugeruje si
Sugeruje si
ę
ę
,
,
ż
ż
e
e
metylacja
metylacja
„
„
non
non
-
-
CpG
CpG
”
”
katalizowana przez DNMT3 mo
katalizowana przez DNMT3 mo
ż
ż
e wyst
e wyst
ę
ę
powa
powa
ć
ć
w ludzkim genie p53.
w ludzkim genie p53.
To
zjawisko
wyst
To
zjawisko
wyst
ę
ę
puje
w
tkankach
puje
w
tkankach
przylegaj
przylegaj
ą
ą
cych do nowotworu p
cych do nowotworu p
ł
ł
uca, co
uca, co
wskazuje,
wskazuje,
ż
ż
e
e
metylacja
metylacja
„
„
non
non
-
-
CpG
CpG
”
”
mo
mo
ż
ż
e
e
wyst
wyst
ę
ę
powa
powa
ć
ć
we
wczesnym
stadium
we
wczesnym
stadium
kancerogenezy i s
kancerogenezy i s
ł
ł
u
u
ż
ż
y
y
ć
ć
jako wska
jako wska
ź
ź
nik dla
nik dla
wczesnego
rozpoznania
procesu
wczesnego
rozpoznania
procesu
nowotworowego
nowotworowego

5
Zesp
Zesp
ó
ó
ł
ł
Wernera
Wernera
(WS)
(WS)
Jest to
Jest to
autosomalna
autosomalna
recesywna choroba dziedziczna
recesywna choroba dziedziczna
charakteryzuj
charakteryzuj
ą
ą
ca si
ca si
ę
ę
przedwczesnym starzeniem i cz
przedwczesnym starzeniem i cz
ę
ę
stym
stym
wyst
wyst
ę
ę
powaniem nowotwor
powaniem nowotwor
ó
ó
w. Choroba jest spowodowana
w. Choroba jest spowodowana
mutacjami genu WRN, nale
mutacjami genu WRN, nale
żą
żą
cego do rodziny
cego do rodziny
RecQ
RecQ
i
i
posiadaj
posiadaj
ą
ą
cego aktywno
cego aktywno
ść
ść
enzymatyczn
enzymatyczn
ą
ą
helikazy
helikazy
i
i
egzonukleazy
egzonukleazy
.
.
Helikaza
Helikaza
WRN odgrywa znacz
WRN odgrywa znacz
ą
ą
c
c
ą
ą
rol
rol
ę
ę
w stabilizacji funkcji
w stabilizacji funkcji
telomeru . W kom
telomeru . W kom
ó
ó
rkach nowotworowych funkcja genu WRN jest
rkach nowotworowych funkcja genu WRN jest
zniesiona poprzez wyciszenie na drodze
zniesiona poprzez wyciszenie na drodze
hipermetylacji
hipermetylacji
wysp
wysp
CpG
CpG
w obr
w obr
ę
ę
bie promotora.
bie promotora.
Epigenetyczna inaktywacja WRN na poziomie biochemicznym i
Epigenetyczna inaktywacja WRN na poziomie biochemicznym i
kom
kom
ó
ó
rkowym prowadzi do straty
rkowym prowadzi do straty
egzonukleazowej
egzonukleazowej
aktywno
aktywno
ś
ś
ci
ci
WRN, zwi
WRN, zwi
ę
ę
kszonej niestabilno
kszonej niestabilno
ś
ś
ci chromosom
ci chromosom
ó
ó
w i
w i
apoptozy
apoptozy
indukowanej przez inhibitory
indukowanej przez inhibitory
topoizomerazy
topoizomerazy
.
.
Za rozw
Za rozw
ó
ó
j zespo
j zespo
ł
ł
u
u
Retta
Retta
odpowiadaj
odpowiadaj
ą
ą
powtarzaj
powtarzaj
ą
ą
ce si
ce si
ę
ę
nonsensowne mutacje w regionie MBD (
nonsensowne mutacje w regionie MBD (
Methyl
Methyl
binding
binding
domain
domain
)
)
MeCP2, proteinie wi
MeCP2, proteinie wi
ążą
ążą
cej
cej
zmetylowany
zmetylowany
DNA .
DNA .
W schizofrenii i chorobie dwubiegunowej zidentyfikowano
W schizofrenii i chorobie dwubiegunowej zidentyfikowano
niemal 100 pozycji ze zmienion
niemal 100 pozycji ze zmienion
ą
ą
metylacj
metylacj
ą
ą
, wi
, wi
ę
ę
kszo
kszo
ść
ść
nich by
nich by
ł
ł
a
a
specyficzna dla p
specyficzna dla p
ł
ł
ci .
ci .
Zesp
Zesp
ó
ó
ł
ł
ł
ł
amliwego chromosomu X jest zwi
amliwego chromosomu X jest zwi
ą
ą
zany z wielokrotnymi
zany z wielokrotnymi
powt
powt
ó
ó
rzeniami
rzeniami
trinukleotyd
trinukleotyd
ó
ó
w
w
CGG w obr
CGG w obr
ę
ę
bie genu FMR1, te
bie genu FMR1, te
powt
powt
ó
ó
rzenia id
rzenia id
ą
ą
w parze z anormaln
w parze z anormaln
ą
ą
metylacj
metylacj
ą
ą
tych region
tych region
ó
ó
w i w
w i w
konsekwencji wywo
konsekwencji wywo
ł
ł
uj
uj
ą
ą
wyciszenie genu FMR1 .
wyciszenie genu FMR1 .
Chroniczne nara
Chroniczne nara
ż
ż
enie na nikiel (II), chrom (VI) lub arsen
enie na nikiel (II), chrom (VI) lub arsen
nieorganiczny mo
nieorganiczny mo
ż
ż
e tak
e tak
ż
ż
e wywo
e wywo
ł
ł
ywa
ywa
ć
ć
modyfiakacj
modyfiakacj
ę
ę
bia
bia
ł
ł
ek
ek
histonowych
histonowych
i zmian
i zmian
ę
ę
profilu
profilu
metylacji
metylacji
DNA
DNA
Analiza
Analiza
metylacji
metylacji
MS
MS
-
-
PCR
PCR
Methylation
Methylation
spec
spec
i
i
fic
fic
PCR
PCR
RE
RE
-
-
PCR
PCR
(REP)
(REP)
Restriction enzyme PCR
Restriction enzyme PCR
B SSCP
B SSCP
Bisulfite
Bisulfite
single
single
-
-
strand
strand
conformation polymorphism
conformation polymorphism
BGS
BGS
-
-
bisulfite
bisulfite
genomic
genomic
sequencing
sequencing
MS
MS
nested
nested
PCR
PCR
real
real
-
-
time
time
PCR
PCR
MS
MS
-
-
AP
AP
-
-
PCR
PCR
(
(
Methylation
Methylation
Sensitive
Sensitive
Arbitrarily
Arbitrarily
Primed
Primed
PCR)
PCR)
PCR
PCR
Elektroforeza
Elektroforeza
Po PCR u
Po PCR u
ż
ż
ywa si
ywa si
ę
ę
enzym
enzym
ó
ó
w restrykcyjnych
w restrykcyjnych
HpaII
HpaII
kt
kt
ó
ó
ry rozcina w miejscach
ry rozcina w miejscach
metylacji
metylacji
oraz
oraz
MspI
MspI
kt
kt
ó
ó
ry
rozcina
miejsca
ry
rozcina
miejsca
niemetylowane
niemetylowane
. Dodatkowo stosuje si
. Dodatkowo stosuje si
ę
ę
enzym
enzym
usuwaj
usuwaj
ą
ą
cy artefakty powstaj
cy artefakty powstaj
ą
ą
ce w
ce w
PCR
PCR
-
-
RsaI
RsaI
Je
Je
ż
ż
eli po trawieniu
eli po trawieniu
HpaII
HpaII
i
i
RsaI
RsaI
widoczny
widoczny
jest
jest
produkt ci
produkt ci
ę
ę
cia to
cia to
ś
ś
wiadczy to o
wiadczy to o
metylacji
metylacji
Je
Je
ż
ż
eli po trawieniu
eli po trawieniu
MSpI
MSpI
i
i
RsaI
RsaI
nie
nie
widoczny
widoczny
jest produkt ci
jest produkt ci
ę
ę
cia to r
cia to r
ó
ó
wnie
wnie
ż
ż
ś
ś
wiadczy to o
wiadczy to o
metylacji
metylacji
Nu
Nu
kleotydy
kleotydy
niebieskie
niebieskie
to
to
niemetylowane
niemetylowane
cytozyny
cytozyny
kt
kt
ó
ó
re ulegaj
re ulegaj
ą
ą
przekszta
przekszta
ł
ł
caniu
caniu
w
w
urac
urac
yle
yle
pod wp
pod wp
ł
ł
ywem wodorosiarczynu
ywem wodorosiarczynu
,
,
podczas gdy
podczas gdy
nukleotydy zaznaczone na
nukleotydy zaznaczone na
czerwono
czerwono
s
s
ą
ą
to 5
to 5
’
’
metylocytozyny oporne na
metylocytozyny oporne na
przekszta
przekszta
ł
ł
cenie
cenie
Allel metylowany
Allel niemetylowany
traktowanie
wodorosiarczynem sodu
Methylation
Methylation
-
-
specific PCR
specific PCR
jest czu
jest czu
łą
łą
metod
metod
ą
ą
pozwalaj
pozwalaj
ą
ą
c
c
ą
ą
na
na
namna
namna
ż
ż
anie
anie
i
i
wykrywanie
wykrywanie
obszar
obszar
ó
ó
w
w
metylowanych
metylowanych
przy u
przy u
ż
ż
yciu specyficznych
yciu specyficznych
starter
starter
ó
ó
w.
w.
Startery przy
Startery przy
łą
łą
cz
cz
ą
ą
si
si
ę
ę
tyko
tyko
do sekwencji
do sekwencji
metylowanych
metylowanych
zawieraj
zawieraj
ą
ą
cych
cych
5
5
’
’
metylocytozyn
metylocytozyn
ę
ę
kt
kt
ó
ó
r
r
ą
ą
nie uleg
nie uleg
ł
ł
a przekszta
a przekszta
ł
ł
ceniu w uracyl pod wp
ceniu w uracyl pod wp
ł
ł
ywem
ywem
wodorosiarczynu
wodorosiarczynu
.
.
Eeeeee
Eeeee

6
Analiza
Analiza
metylacji
metylacji
DNA
DNA
oparta na
oparta na
non
non
methylation
methylation
-
-
specific PCR
specific PCR
.
.
Po
Po
traktowaniu wodorosiarczynem
traktowaniu wodorosiarczynem
genomowe
genomowe
DNA
DNA
jest
jest
namna
namna
ż
ż
ane
ane
w reakcji
w reakcji
PCR
PCR
kt
kt
ó
ó
ra nie rozr
ra nie rozr
ó
ó
ż
ż
nia
nia
sekwencji
sekwencji
metylowanych
metylowanych
i
i
niemetylowanych
niemetylowanych
.
.
Po namno
Po namno
ż
ż
eniu
eniu
DNA w reakcji PCR dost
DNA w reakcji PCR dost
ę
ę
pnych jest wiele metod kt
pnych jest wiele metod kt
ó
ó
re pozwalaj
re pozwalaj
ą
ą
na
na
r
r
ó
ó
ż
ż
nicowanie sekwencji
nicowanie sekwencji
metylowanych
metylowanych
na bazie dokonanych
na bazie dokonanych
wczesniej
wczesniej
zmian
zmian
(efekt traktowania
(efekt traktowania
wodorosirczynem
wodorosirczynem
)
)
Methylation-sensitive single-strand conformation analysis (MS-SSCA)
Methylation-sensitive single-nucleotide primer extension (MS-SnuPE)
High resolution melting analysis (HRM)
Ekspansja tr
Ekspansja tr
ó
ó
jnukleotydowa
jnukleotydowa
Genom człowieka w ponad połowie składa się z sekwencji
powtarzających się.
Należą do nich między innymi tandemowe powtórzenia, które
stanowią 10%całkowitej sekwencji DNA. Ta grupa obejmuje
satelity, minisatelity oraz mikrosatelity różniące się wielkością
powtarzającego się motywu sekwencji.
Znacznie mniejszą część genomu, bo tylko 1% zajmują sekwencje
kodujące około 22 tysięcy ludzkich genów i drugie tyle stanowią
ich sekwencje regulatorowe.
Sekwencje powtarzające się występują nie tylko w częściach
międzygenowych lecz także w genach, nawet w ich częściach
kodujących i mogą brać udział w regulacji ich ekspresji.
Genom człowieka
Geny i sekwencje
Pokrewne 25%
Regiony międzygenowe 75%
Regiony
kodujące
i regulatorowe
2%
Introny
promotory
Pseudogeny 23%
Powtarzające się 55%
Unikatowe 20%
Powtórzenia tandemowe 10%
Powtórzenia rozproszone 45%
Satelity
Minisatelity
Mikrosatelity
Mikrosatelity znane również jako krótkie tandemowe
powtórzenia (ang. Short Tandem Repeats (STR) albo
powtórzenia prostych sekwencji (ang. Simple
Sequence Repeats (SSR) składają się z
powtarzających się 1-6 nukleotydowych motywów.
Liczba powtórzeń takich sekwencji zwykle nie
przekracza 10, a jeśli jest wyższa to często taki ciąg
charakteryzuje się polimorfizmem długości w
populacji. W rzadkich przypadkach, najczęściej
patologicznych, liczba powtórzeń może osiągać setki a
nawet tysiące.
Wszystkie sekwencje typu STR obejmują łącznie 3%
ludzkiego genomu. Zarówno zawartość jak i
rozmieszczenie różnych powtórzeń tego typu w
genomie są silnie zróżnicowane.
Większość sekwencji STR występuje znacznie
częściej w regionach niekodujących.
Wyjątkiem są tutaj ciągi powtórzeń trój i
sześcionukleotydowych, które są niemal dwukrotnie
częstsze w eksonach niż w intronach i regionach
międzygenowych.
Ich pozytywna selekcja w eksonach sugeruje, że mogą
one mieć znaczenie funkcjonalne.
Wysoka podatność ciągów prostych powtórzeń
sekwencji na mutacje i wynikający z niej częsty
polimorfizm długości czynią z sekwencji
powtarzających się bogate źródło zmienności
fenotypowej.
Powtórzenia trójnukleotydowe naleza do grupy powtórzen
mikrosatelitarnych, tworzacych proste, tandemowe
powtórzenia złożone z jednego do szesciu nukleotydów.
Charakteryzuja sie one wysokim polimorfizmem i wystepują
równomiernie w genomie co 6–10 kpz.
Sekwencje mikrosatelitarne zlokalizowane sa nie tylko w
intronach i w
sekwencjach flankujacych, ale również w
sekwencjach kodujacych czyli eksonach.
W obrebie sekwencji powtórzen trójnukleotydowych moe dojsć
do zjawiska zwielokrotnienia motywu trójnukleotydowego, czyli
do ekspansji powtórzen.
Mechanizm ekspansji trójnukleotydowych powtórzen zwiazany jest
z błedami w czasie replikacji DNA.

7
Powt
Powt
ó
ó
rzenia w
rzenia w
mRNA
mRNA
Najczęściej występującymi motywami
powtórzonymi są: CAG, CGG, CCG,CUG, AGG
i ACC,
natomiast
ACG,AUC, CUU, AGU, CGU i ACU
należą da najrzadszych w transkryptomie.
Większość ciągów powtórzeń występuje w
ORF (ang. Open Reading Frame) (67%), mniej
w 5’UTR (ang. Untranslated Region) (24%) i
najmniej w 3’UTR (9%).
Powtórzenia bogate w GC przeważają w
5’UTR, a bogate w AU są częstsze w 3’UTR.
Wzrost liczby powtórzeń ponad normę
(określoną dla każdego genu) zwany jest
mutacją dynamiczna.
Mutacje dynamiczne dotyczą najczęsciej
zwielokrotnienia nawet 300-krotnego
trójnukleotydowej sekwencji powtórzonej, co
w efekcie prowadzi do wystąpienia choroby
genetycznej
Pl
Pl
ą
ą
sawica Huntingtona normalna liczba
sawica Huntingtona normalna liczba
powt
powt
ó
ó
rze
rze
ń
ń
6
6
-
-
36;zmutowanan 36
36;zmutowanan 36
-
-
121
121
Rdzeniowy Zanik miesni typu Kennedyego
normalna liczba powt
normalna liczba powt
ó
ó
rze
rze
ń
ń
11
11
-
-
33;zmutowana
33;zmutowana
38
38
-
-
66
66
Zespół łamliwego chromosomu X
normalna
normalna
liczba powt
liczba powt
ó
ó
rze
rze
ń
ń
6
6
-
-
52;zmutowanan >52
52;zmutowanan >52
Ekspansja niestabilnych ciągów
powtórzeń w pojedyńczych genach jest
przyczyną ponad 20 różnych chorób
dziedzicznych znanych pod wspólną
angielską nazwą Triplet Repeat
Expansion Diseases (TREDs).
Należą do nich:
- zespół łamliwego chromosomu X (FXS),
- dystrofia miotoniczna (DM),
- choroba Huntingtona (HD)
- szereg ataksji rdzeniowomóżdżkowych (SCAs).
Ciągi powtórzeń ulegające patogenicznym
ekspansjom występują we wszystkich
regionach funkcjonalnych genów, zarówno w
częściach ulegających translacji, jak i w
regionach niekodujących.
Choroby zwiazane z ekspansja powtórzen
trójnukleotydowych można podzielić ze względu na
miejsce wystepowania powtórzeń:
1. Trójnukleotydowe powtórzenia zlokalizowane w
regionie ulegajacym translacji.
Do tej grupy chorób zalicza sie:
plasawice Huntingtona – HD, ataksje rdzeniowomó
-dkowa typu pierwszego – SCA1, ataksje rdzeniowo-
módkowa typu drugiego – SCA2, ataksje rdzeniowo-
módkowa typu trzeciego – SCA3, ataksje rdzeniowo-
módkowa typu szóstego – SCA6, ataksje
rdzeniowo-módkowa typu siódmego – SCA7, ataksje
rdzeniowo-módkowa typu siedemnastego – SCA17,
zwyrodnienie zebato-czerwienne DRPLA, rdzeniowoopuszkowy
zanik miesni typu Kennedy`ego – SBMA,
dystrofie miesniowa oczno-gardzielowa – OPMD,
achondroplazje rzekoma – COMP.

8
2. Trójnukleotydowe powtórzenia zlokalizowane w
regionie nie ulegajacym translacji oraz w intronie.
Do tej grupy chorób zalicza sie:
zespół łamliwego chromosomu X – FRAXA,
uposledzenie
umysłowe zwiazane z FRAXE, dystrofie miotoniczna
– DM, ataksje rdzeniowo-módkowa typu
ósmego – SCA8, ataksje módkowo-rdzeniowa typu
dwunastego – SCA12, ataksje Friedreicha – FRDA.
Większość genów związanych z TREDs
zawiera powtórzenia CAG w regionie
kodującym i wywołująca chorobę liczba
powtórzeń mieści się najczęściej w zakresie
od około 40 do 100.
Ekspansje powtórzeń w regionach
niekodujących są większe i bardziej zmienne.
Powszechnie uważa się, że powtórzenia
występujące w regionach ulegających
translacji wywołują patogenezę poprzez
zmiany funkcji kodowanego białka
Większość chorób wywoływanych ekspansją
trójnukleotydowych powtórzeń jest warunkowana w
sposób autosomalny dominująy czyli wywołuje je
mutacja w jednym z dwóch wariantów genu.
Produkt zmutowanego genu, transkrypt lub białko,
albo obydwa są toksyczne dla komórki. Zatem
usunięcie zmutowanego transkryptu lub białka powinno
przeciwdziałać chorobie.
Terapia TREDs jest dużym wyzwaniem ze względu na
miejsce gdzie rozwijają się główne objawy choroby
czyli centralny układ nerwowy oraz konieczność
allelospecyficznej eliminacji produktów zmutowanych
genów.
Terapia
Terapia
Celem
allelospecyficznego
wyciszenia
ekspresji genu może być albo sam region
powtórzeń, który
różni
się
długością
i
strukturą
RNA
między
normalnym
a
zmutowanym wariantem mRNA, albo jak się
wydaje bardziej odpowiednie do zastosowania
iRNA
miejsca
występowania
polimorfizmu
pojedynczych nukleotydów SNP.
Wyszukiwarka
Podobne podstrony:
metylacja DNA
Lukasik metylacja DNA
12 Elektroforeza agarozowa wyizolowanego DNA ?łkowitego oraz produktów PCR
Ćwiczenie 1 Izolacja, oczyszczanie oraz detekcja genomowego DNA w żywności wer 2012
49 Zdefiniuj pojęcie „ruchome elementy DNA”, wymień je oraz podaj, co jest ich wspólną właściwością
12 Elektroforeza agarozowa wyizolowanego DNA ?łkowitego oraz produktów PCR
Polityka dynastycznych Jagiellonów , Ekspansja turecka oraz Gospodarka średniowiecznej Europy
w8 VLAN oraz IP w sieciach LAN
Układy Napędowe oraz algorytmy sterowania w bioprotezach
Replikacja DNA i choroby związane
(33) Leki stosowane w niedokrwistościach megaloblastycznych oraz aplastycznych
Elektroforeza DNA komórkowego BioAut1, BioAut2 i Ch1
DNA Eng2
Postawy ciała oraz wady postawy
Statyny oraz fibraty w leczeniu dyslipidemii
Krwawienie z przewodu pokarmowego lub zagrażające powikłania oraz dyskomfort pacjenta w zakresie hig
więcej podobnych podstron