
REPLIKACJA
(rekombinacja, naprawa DNA)
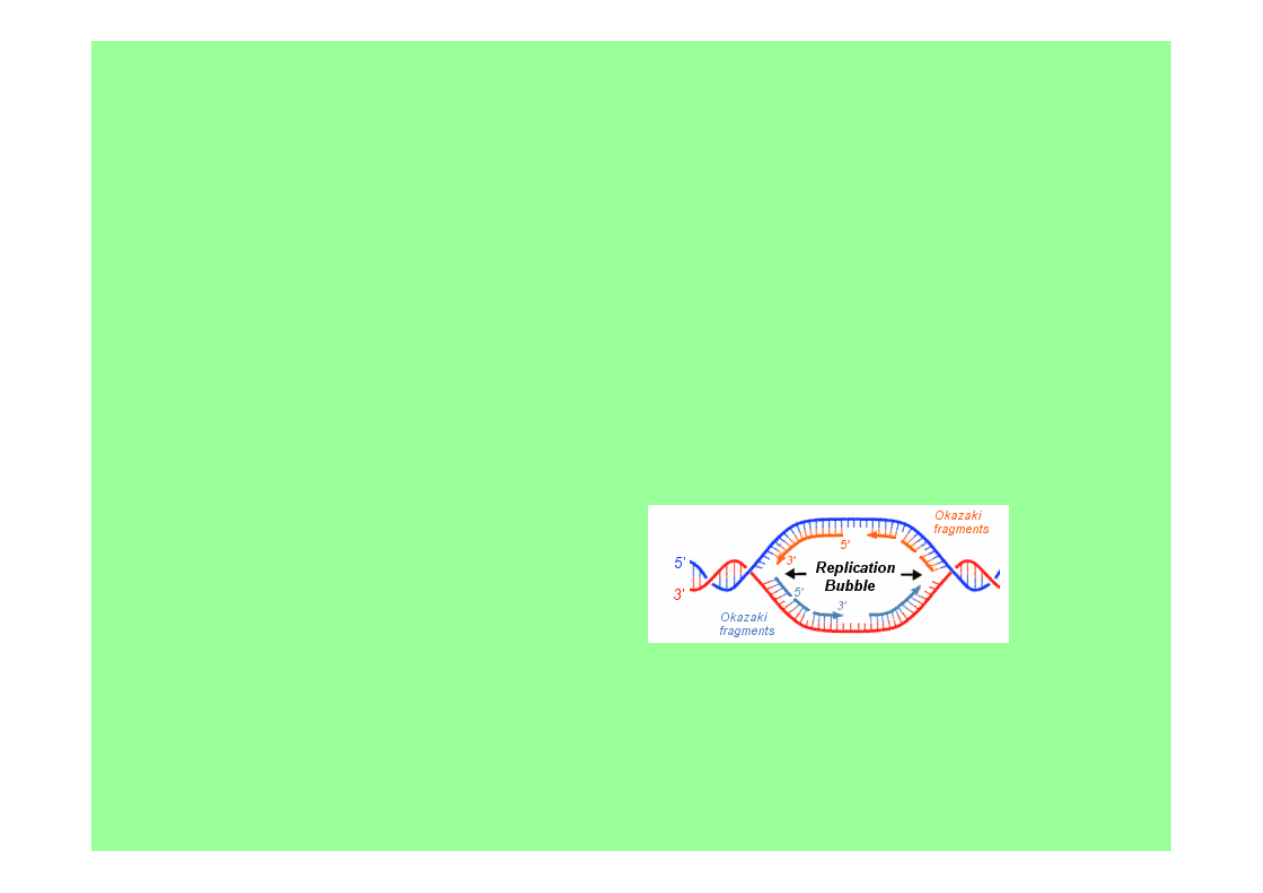
Zasady, które stosują się do wszystkich lub niemal wszystkich
replikacji DNA:
* 2-niciowe DNA replikuje w sposób semikonserwatywny
(gdy nici rodzicielskie rozdzielają się, każda służy jako
matryca do utworzenia nowej komplementarnej nici)
* replikacja 2-niciowego DNA jest połowicznie nieciągła
(jedna nić – wiodąca - jest replikowana w sposób ciągły w
kierunku posuwania się widełek replikacyjnych,
druga – opóźniona - replikuje w sposób nieciągły w postaci 1-2
kpz fragmentów Okazaki tworzonych w kierunku przeciwnym
do ruchu widełek replikacyjnych; dzięki temu obie nici są
replikowane w kierunku 5’→3’)
* inicjacja replikacji DNA wymaga primera (pojedynczego na
nici wiodącej, osobnego dla każdego fragmentu Okazaki nici
opóźnionej; u
E.coli
primerami są 10-12 nt RNA)
* większość pro- i eukariotycznych DNA replikuje
dwukierunkowo, plazmid colE1 replikuje jednokierunkowo.
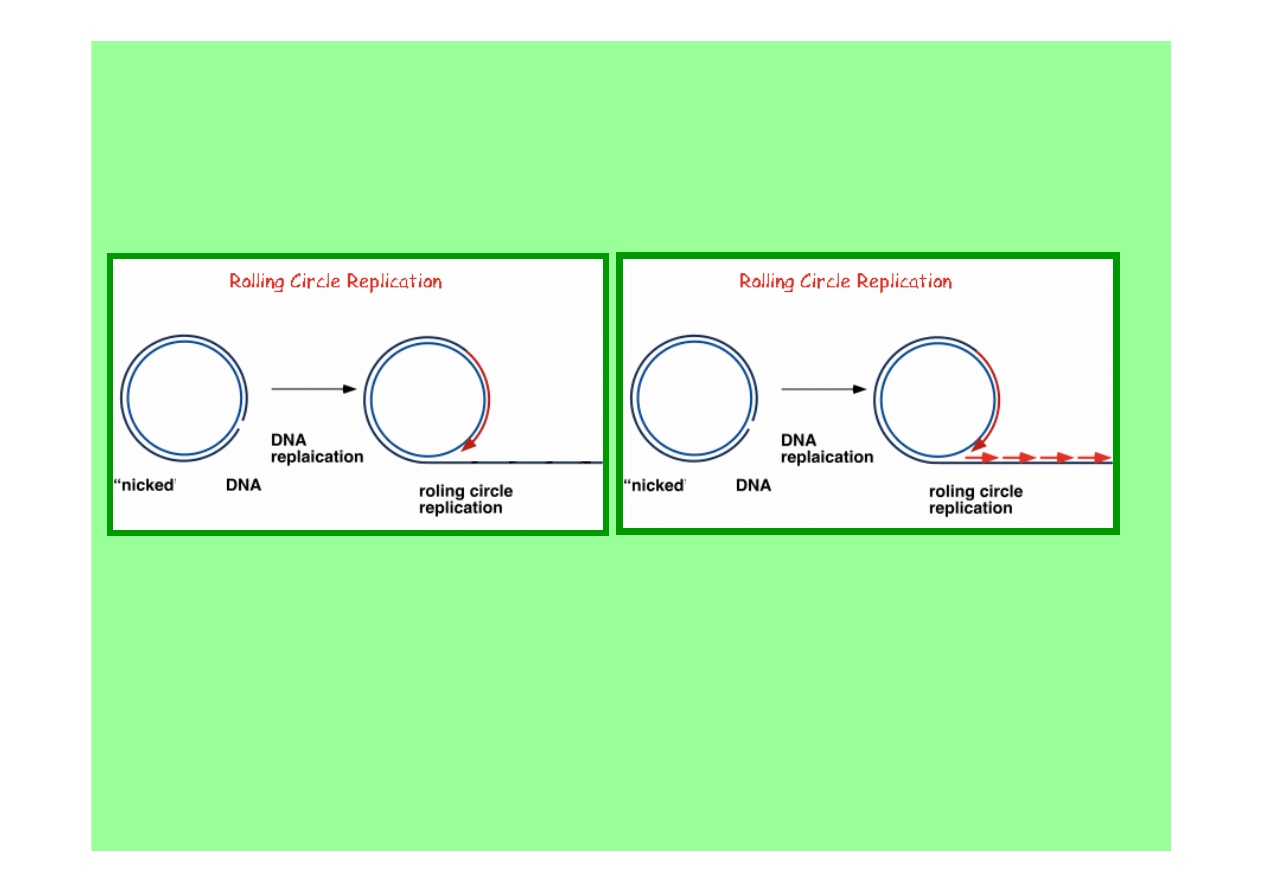
koliste DNA może replikować zgodnie z mechanizmem
toczącego się kółka – jedna nić 2-niciowego DNA jest
nacinana i koniec 3’ ulega wydłużaniu na matrycy nici
nienaruszonej, co oddziela od niej koniec 5’
• w replikacji φX174 – odseparowany od nici ciągłej koniec 5’
jest wiązany przez fagowe białko gpA, które wypętla go z
widełek replikacyjnych, po zakończeniu pełnej rundy replika-
cyjnej uwalniane jest pełnej długości 1-niciowe koliste DNA,
• u faga λ odseparowana nić służy jako matryca do nieciągłej
syntezy nici opóźnionej
3’
5’
3’
5’

* M13
używa
pol RNA gospodarza
jako
prymazy
* G4
używa
bakteryjnej
prymazy DnaG
* φX174
używa prymosomu złożonego z
DnaG
oraz
bakteryjnych białek
PriA, PriB, PriC
,
DnaB, DnaC i DnaT
– prymosom jest mobilny i może syntetyzować primery w
kilku miejscach kolistej 1-niciowej cząsteczki DNA
strategie inicjacji replikacji DNA kolifagów o 1-niciowym DNA:
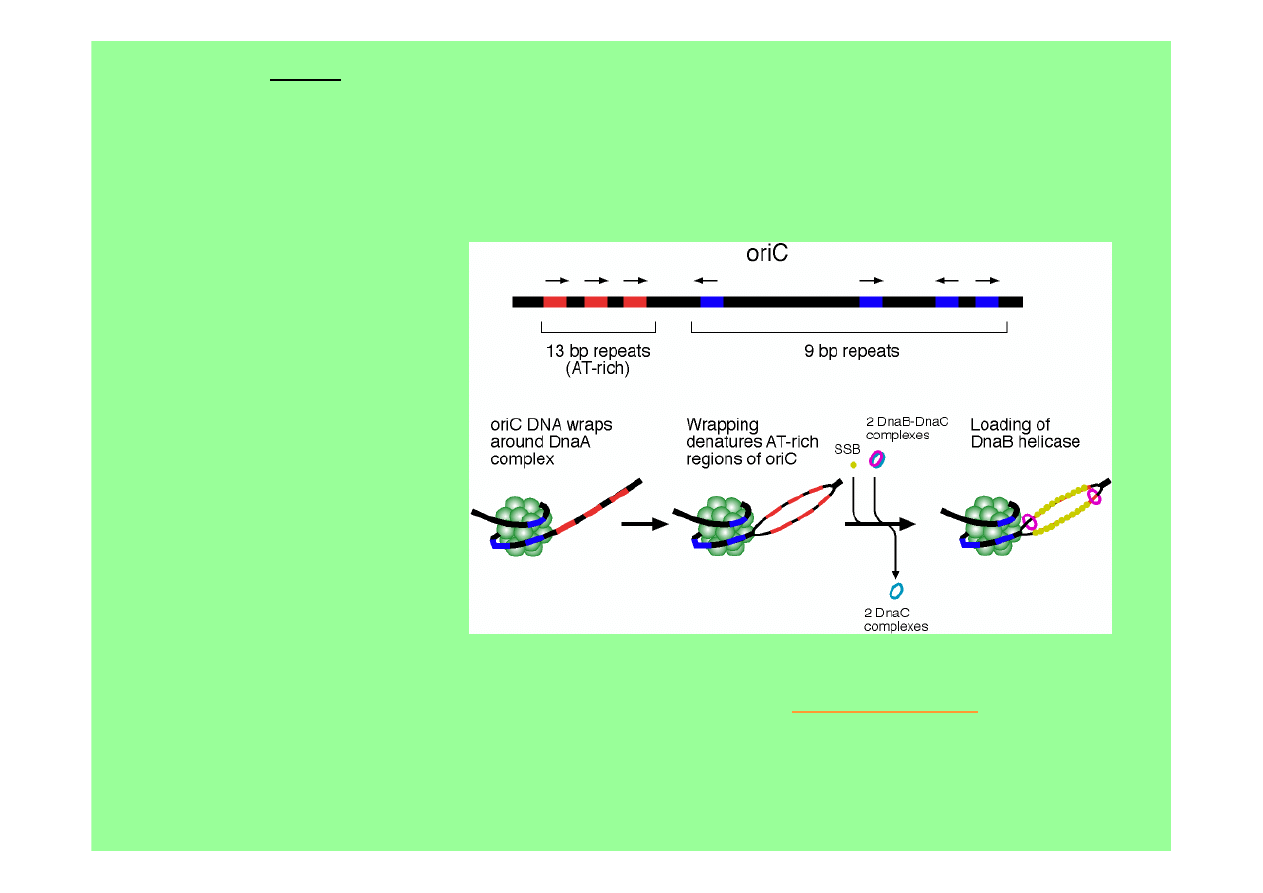
prymosom
E.coli
tworzy się w miejscu startu replikacji
oriC
białko
DnaA
(zastępuje kompleks [PriA, PriB, PriC, DnaT] inicjujący
tworzenie prymosomu φX174) wiąże się do
oriC
w miejscach zw.
kasetami dnaA
i kooperuje z pol RNA i białkiem HU w rozpleceniu
bogatego w AT regionu
DNA przyległego do najbardziej na lewo
położonej
kasety dnaA
do tak utworzonych widełek replikacyjnych dołączają się
SSB
i helikaza
DnaB
związana z
DnaC
i ułatwiają przyłączenie
prymazy DnaG
co
kompletuje prymosom
prymosom pozostaje w replisomie i w powtarzalny sposób inicjuje syntezę
fragmentów Okazaki na nici opóźnionej

SSB (single strand binding protein)
pomagają helikazie
przez silne i kooperatywne wiązanie nowo utworzonego
1-niciowego DNA uniemożliwiając odtworzenie struktury
2-niciowej (i chroniąc 1-niciowe DNA przed degradacją)
oraz stymulując homologiczne polimerazy DNA
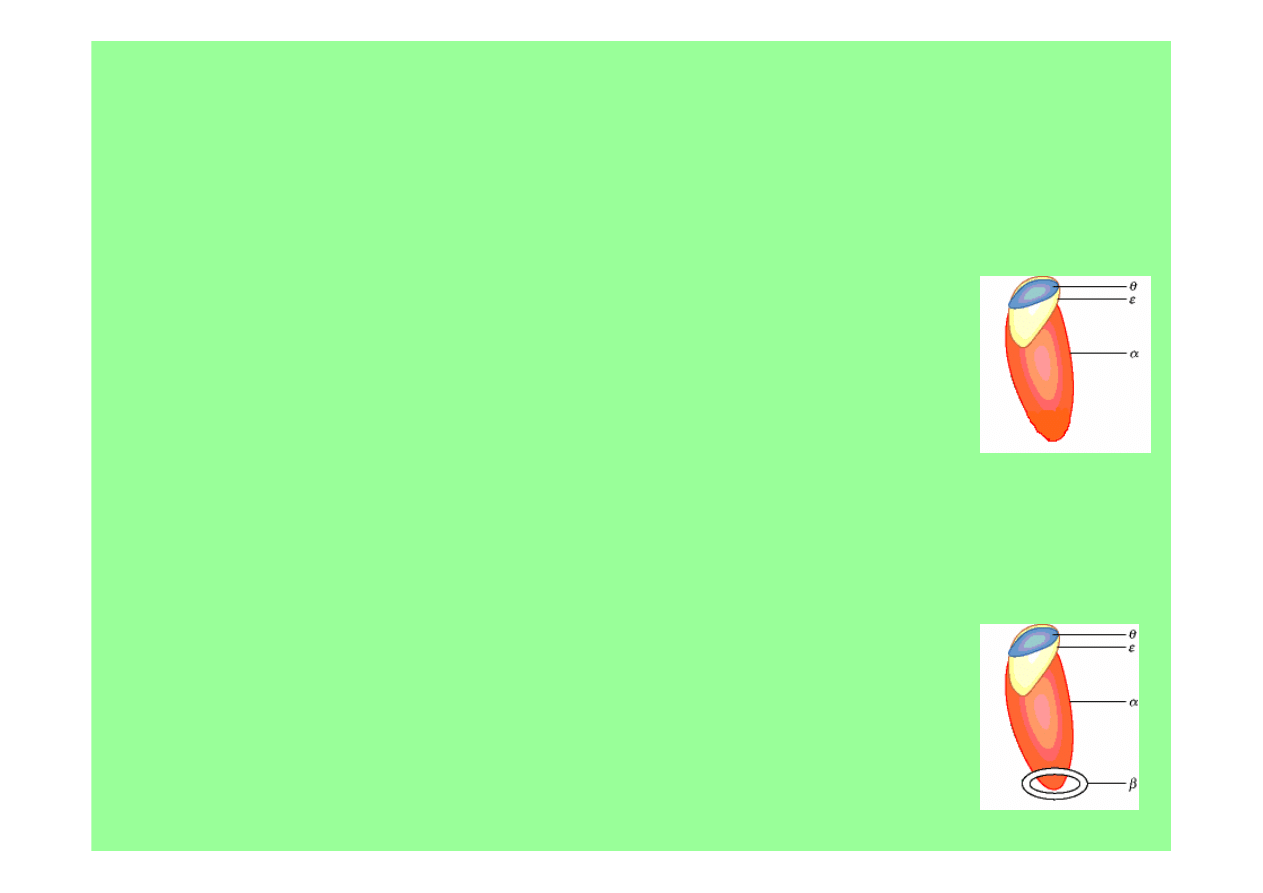
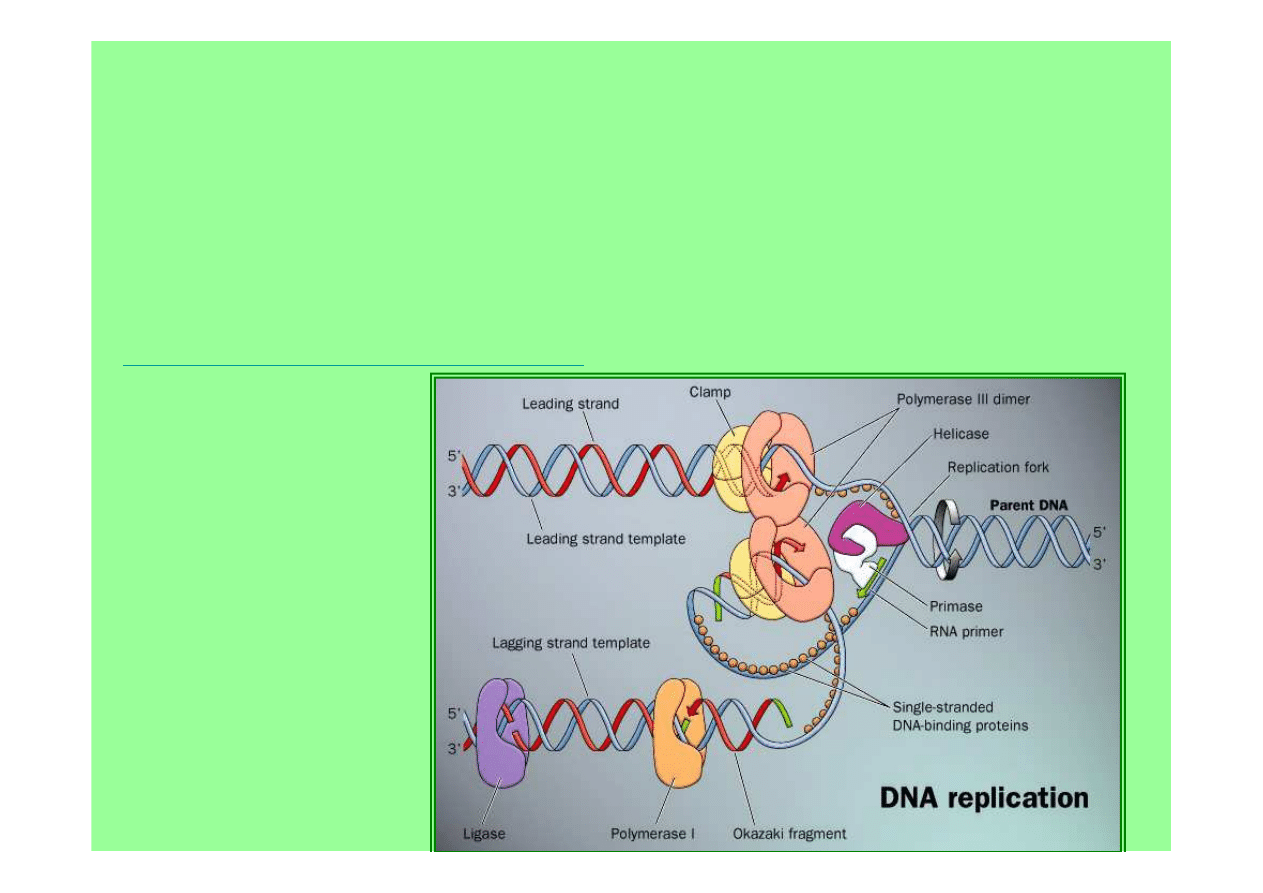
• z 3 pol DNA
E.coli
tylko
pol III
jest niezbędna do replikacji
DNA
• holoenzym pol III
zawiera
10 podjednostek
• rdzeń
pol III DNA składa się z podjednostek:
α
– o aktywności polimerazy
ε
– o aktywności korygującej egzonukleazy 3’→5’
θ
- ?
[pol I jest pojedynczym polipeptydem o aktywności polimerazy
i egzonukleazy 5’→3’ (obie w dużej domenie - fragmencie
Klenowa) oraz egzonukleazy 3’→5’ (w małej oddzielnej
domenie)]
• sam rdzeń pol III DNA może replikować jedynie
krótkie ciągi DNA i odpada od matrycy;
rdzeń +
podjedn. β
syntetyzują w sposób ciągły
(500 pz/s)

podjednostkę β
tworzy dimer w kształcie pierścienia opasującego DNA
i oddziałującego z
podjedn. α
rdzenia sprzęgając razem całą polimerazę
i matrycę; [eukariotyczny czynnik PCNA tworzy trimer o podobnym
pierścieniowatym kształcie]
holoenzym jest „dwugłowy”
z dwoma rdzeniami przyłączonymi
poprzez
dimer podjedn. τ
do
kompleksu γ
jeden rdzeń odpowiada za ciągłą
syntezę nici wiodącej, drugi za
nieciągłą nici opóźnionej
kompleks γ
ładuje
klamrę β
na
matrycę z primerem
http://www.youtube.com/watch?v=teV62zrm2P0
http://www.youtube.com/watch?v=4jtmOZaIvS0&NR=1
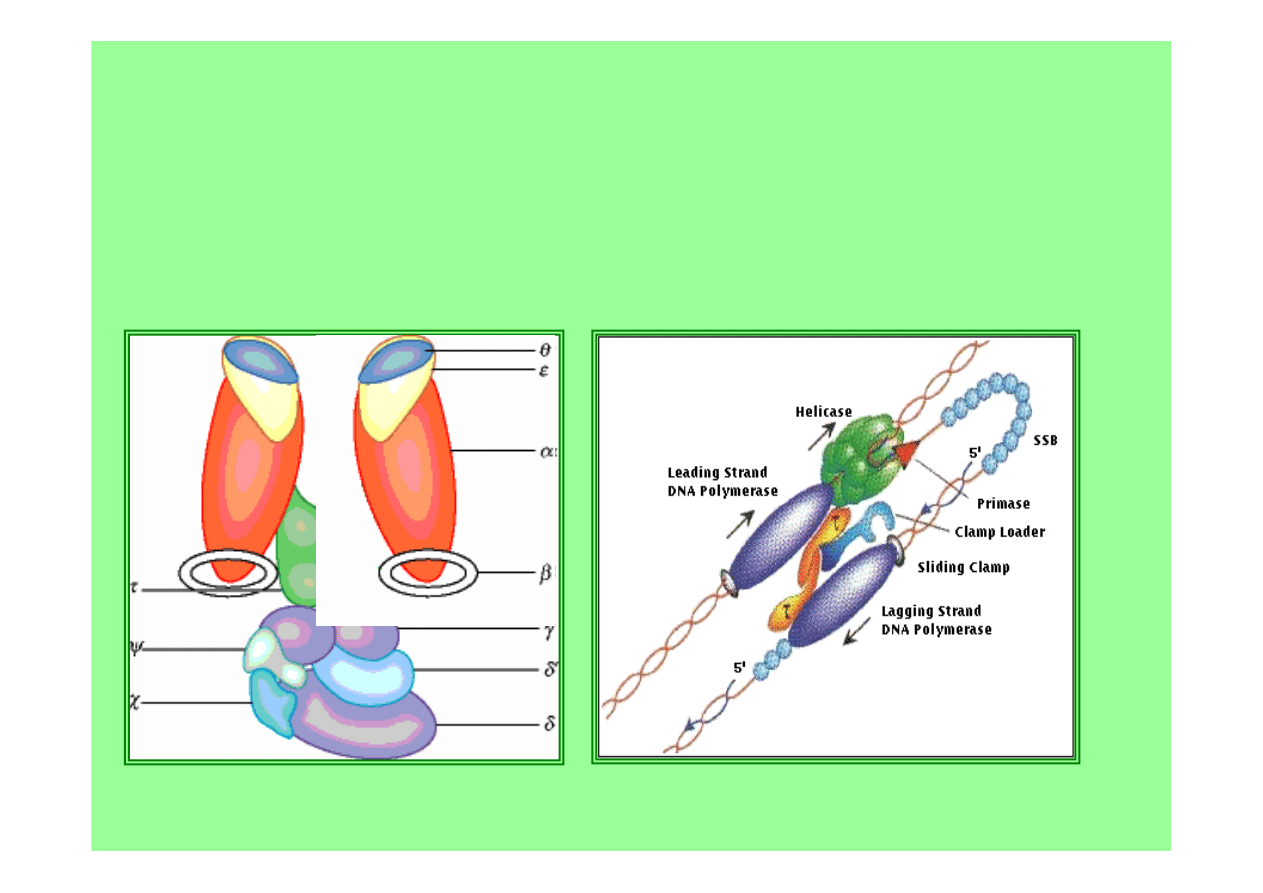
podjedn. β
tworzy dimer w kształcie pierścienia opasującego
DNA i oddziałującego z
podjedn. α
rdzenia sprzęgając
razem całą polimerazę i matrycę; [eukariotyczny czynnik
PCNA tworzy trimer o podobnym pierścieniowatym
kształcie]
holoenzym jest „dwugłowy”
z dwoma rdzeniami przyłączonymi
poprzez
dimer podjedn. τ
do
kompleksu γ
;
jeden rdzeń odpowiada za ciągłą
syntezę nici wiodącej, drugi za
nieciągłą nici opóźnionej;
kompleks γ
ładuje
klamrę β
na
matrycę z primerem;
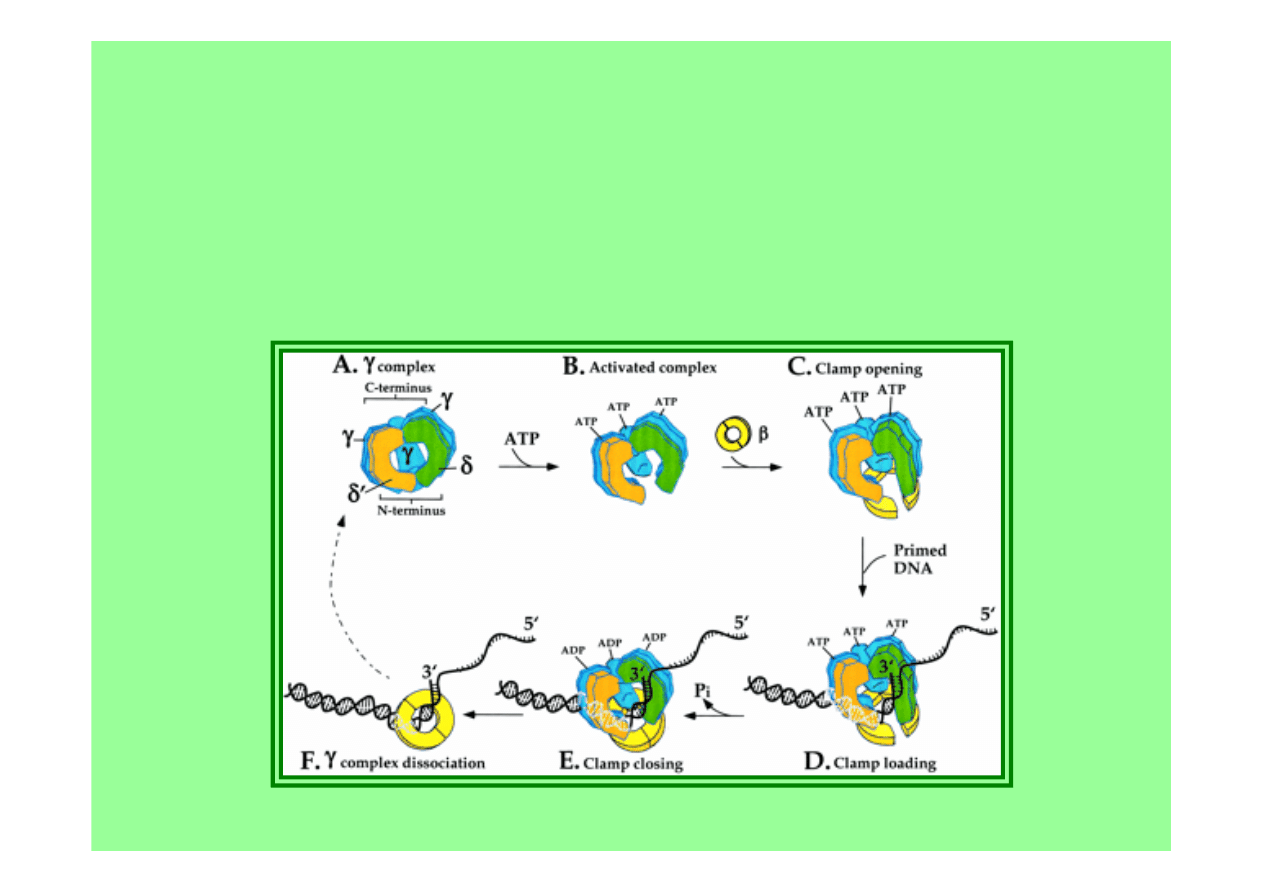
podjedn. β
(
clamp
) by objąć DNA klamrą wymaga pomocy
kompleksu γ
(
clamp loader:
γ, δ, δ’, χ, ψ)
kompleks γ
działa katalitycznie tworząc postępujący kompleks
rdzeń+
β
,
ale nie pozostaje z nim związany podczas replikacji
ładowanie
β
na DNA wymaga ATP
raz załadowana na DNA
klamra β
traci powinowactwo do
kompleksu
γ
i asocjuje z
rdzeniem
pomagając mu w postępującej syntezie
fragmentu Okazaki
gdy fragment Okazaki jest ukończony,
klamra β
traci
powinowactwo do rdzenia i asocjuje z
kompleksem γ
, który ją
„odpina” od DNA
proces powtarza się na następnym primerze
http://www.youtube.com/watch?v=-mtLXpgjHL0&NR=1

w ssaczych komórkach istnieje 5 różnych polimeraz:
α
i
δ
- uczestniczą w
replikacji DNA
:
α
rozpoczyna, a
δ
wydłuża syntezę każdej z nici
β
i
ε
- biorą udział w
naprawie DNA
γ
- prawdopodobnie replikuje
mitochondrialne DNA
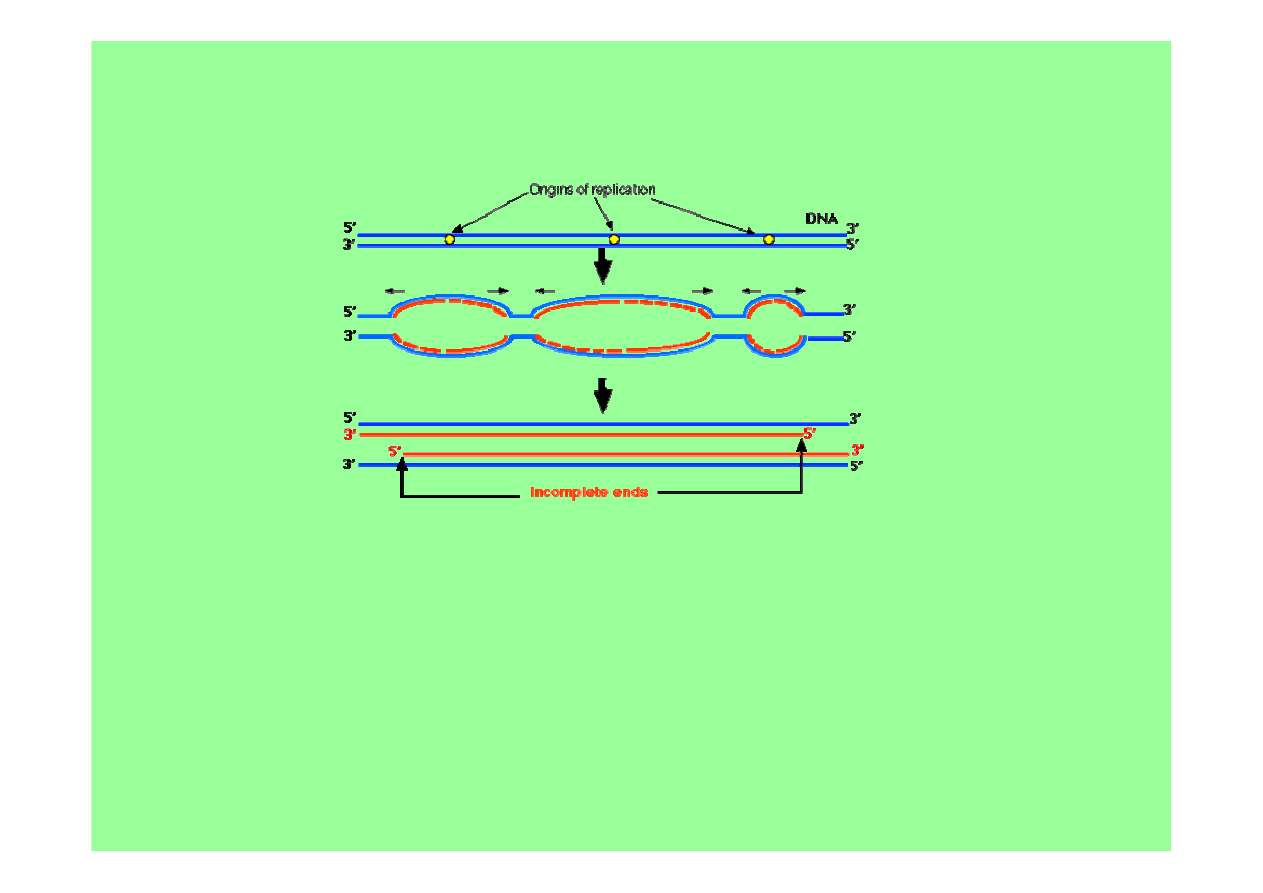
replikacja jądrowego eukariotycznego DNA (50 pz/s)
rozpoczyna się z wielu miejsc startu replikacji
jednocześnie - istnieją dwa mechanizmy, pozytywny
i negatywny, kontrolujące, które z nich już zostały
zreplikowane, a które oczekują na replikację
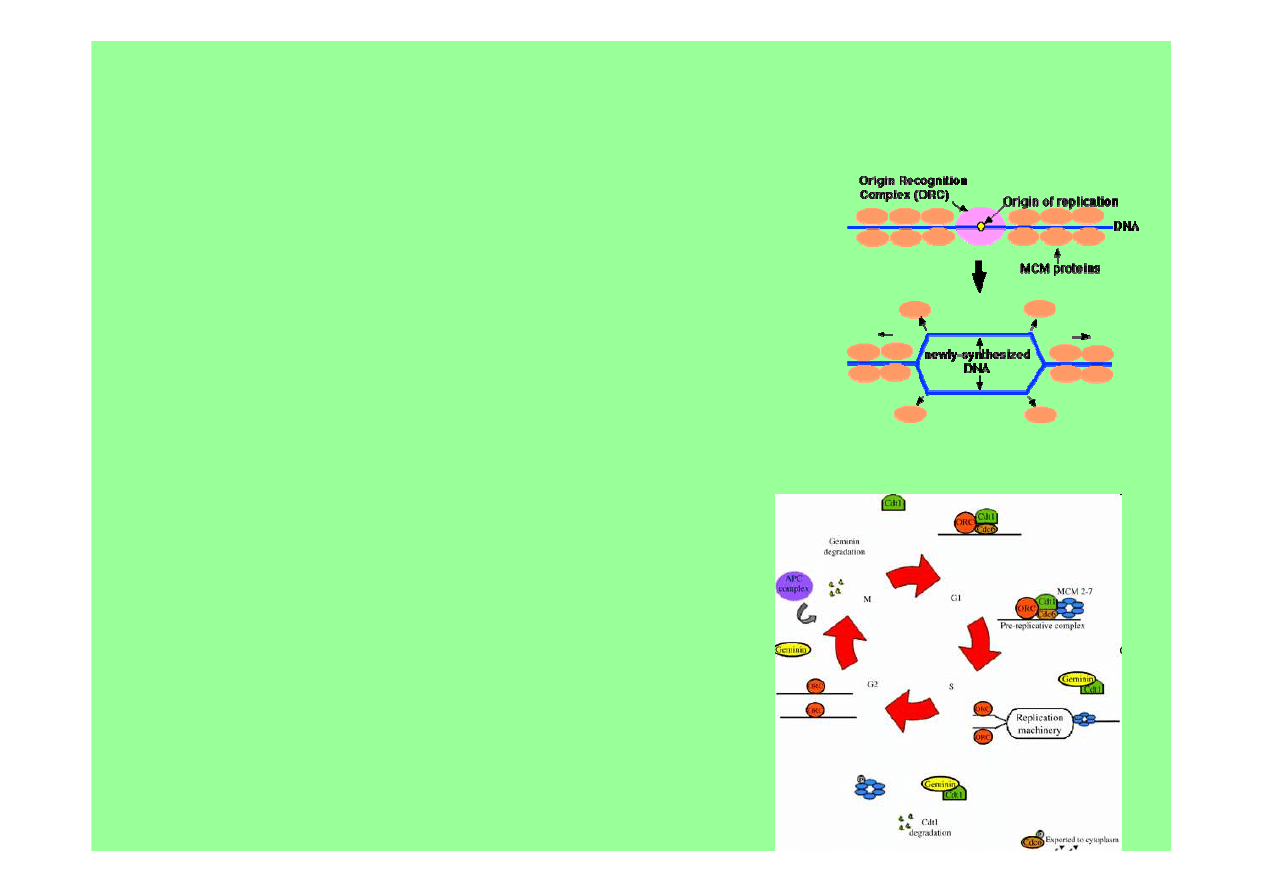
mechanizm pozytywny – licencjonowanie
* do każdego m-sca startu replikacji jest stale przyłączony
funkcjonalny analog DnaA –
kompleks białek ORC
(
O
rigin
R
ecognition
C
omplex of proteins)
* w fazie G
1
w jądrze kumulują się białka
Cdc-6
i
Cdt-1
zw.
czynnikami licencyjnymi
, które
wiążą się do
ORC
i są
istotne dla opłaszczenia DNA
białkami MCM2-7
o aktyw-
ności helikazy
* tylko DNA pokryte
białkami MCM
może zostać zreplikowane
* gdy replikacja się rozpoczyna
Cdc-6
i
Cdt-1
opuszczają
ORC
(
Cdt-1
ulega ubikwitynacji i rozpadowi w proteosomie),
zaś
białka MCM
rozpraszają się tuż przed postępującymi
widełkami replikacyjnymi
mechanizm negatywny –
jądra G
2
zawierają przynajmniej
jedno białko zw.
gemininą
, które uniemożliwia
(prawdopodobnie przez blokowanie działania
Cdt-1
)
wiązanie się
białek MCM
do świeżo zreplikowanego DNA –
- gdy komórka kończy proces mitozy
geminina
jest
degradowana i obie komórki potomne są zdolne do reakcji
na
czynniki licencyjne
i do replikacji swojego DNA w
następnej fazie S
Montanari i in. 2006 Cell Death and Differentiation 13, 1052–1056
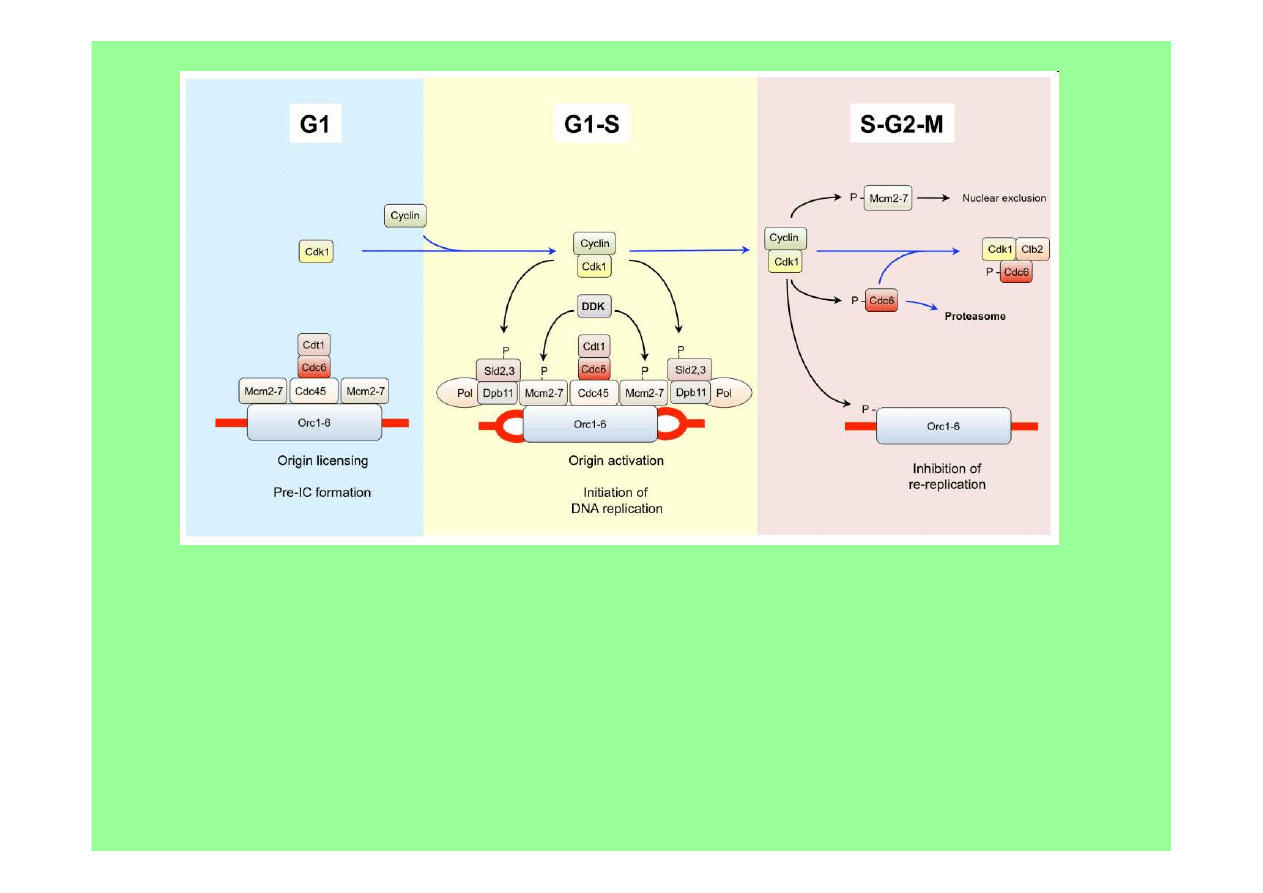
Podczas G1, gdy Cdk1 jest nieaktywne, na miejscach startu replikacji budują się kompleksy
pre-RC. Pod koniec G1 Cdk1 staje się aktywne i fosforyluje niektóre składniki kompleksu,
inicjacje replikacji wyzwala zwłaszcza fosforylacja Sld2 and Sld3. Po inicjacji z danego
miejsca ORI oddysocjowują niektóre składniki i nie mogą być ponownie przyłączone do ORI
zdolnych do replikacji dopóki nie zostaną zdefosforylowane, zaś Cdk1 zostaje w G1
nieaktywne, co stanowi mechanizm zabezpieczający przed re-replikacją.
Enserink & Kolodner 2010
Cell Division
5:11
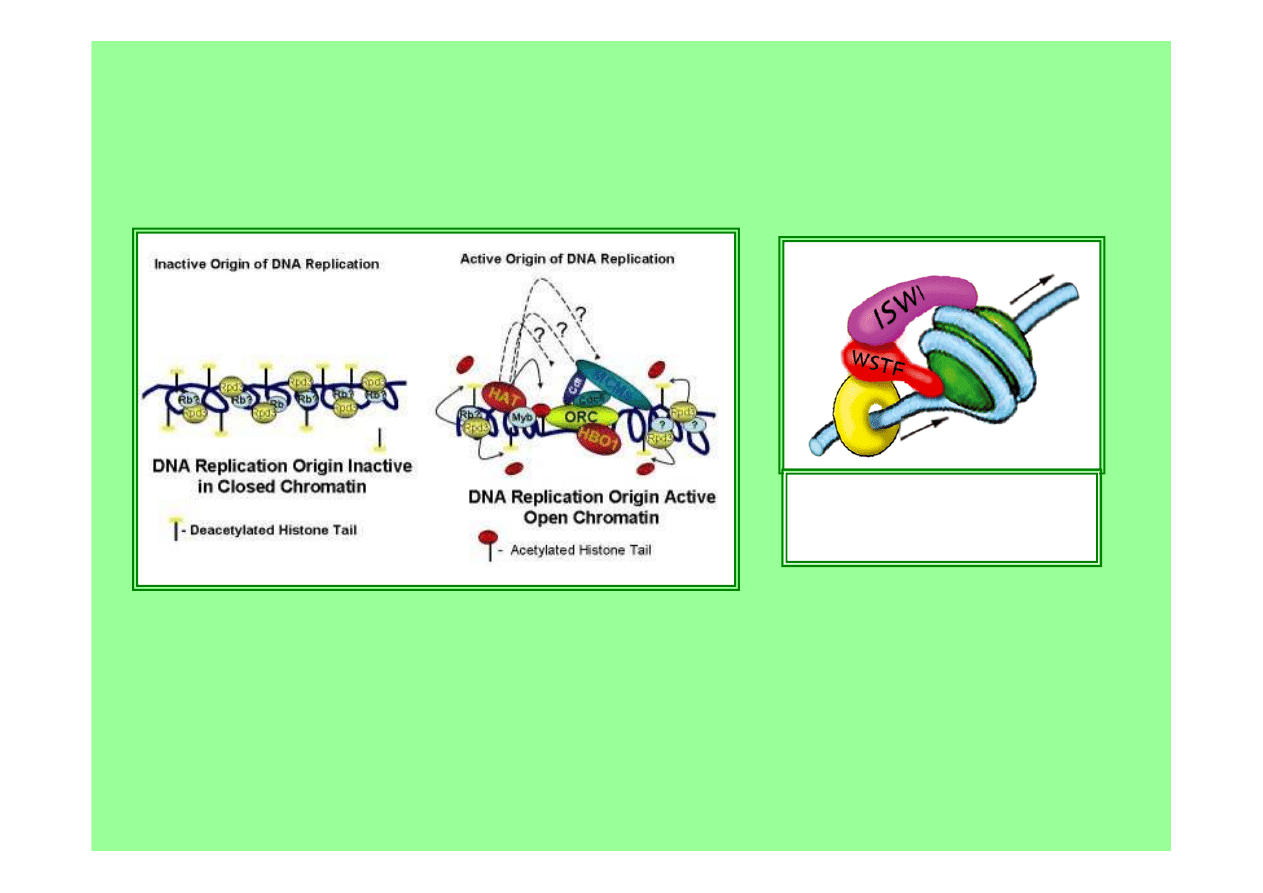
rola
chromatyny
w replikacji DNA - czas replikacji danego ori koreluje
z typem jego chromatyny i jest regulowany przez
deacetylazę
histonów HDAC2
oraz
kompleks remodelujący chromatynę WICH
specyficznie zlokalizowany na późno replikujących ori
replikacja jest mechanizmem ustanowienia i utrzymywania struktury
chromatyny – iniekcja egzogennego DNA do komórek we wczesnej
fazie S powoduje ustanowienie na nim transkrypcyjnie aktywnej,
hiperacetylowanej chromatyny, iniekcja DNA do komórek w późnej
fazie S ustanawia na nim transkrypcyjnie nieaktywną, hypoacetylowaną
chromatynę i raz ustanowiony profil replikacji jest zachowywany
poprzez podziały komórkowe
kompleks WICH (
WSTF
-
ISWI
chromatin remodelling
complex) oddziałuje z
PCNA
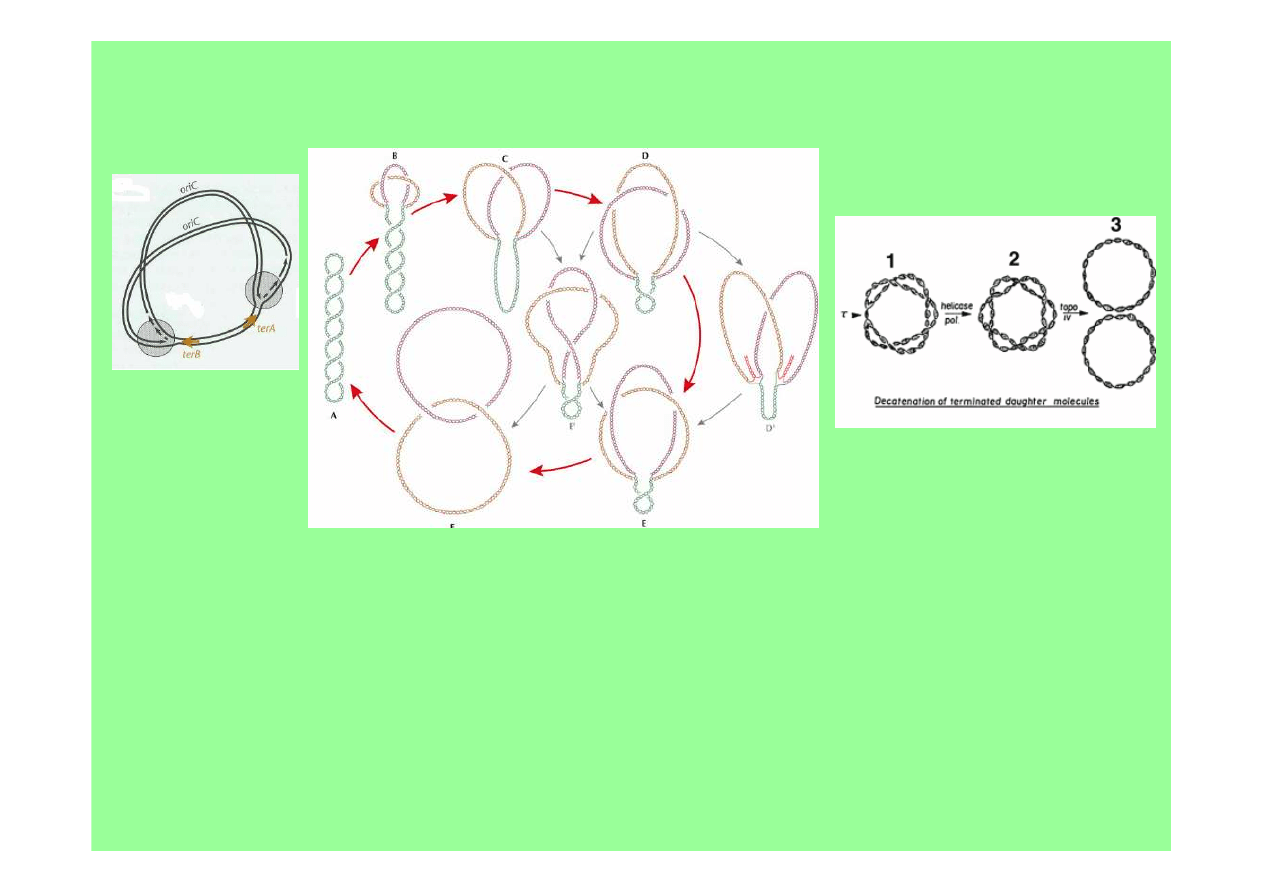
terminacja
replikacji kolistego DNA
terA i terB:
terminatory
replikacji
na koniec replikacji koliste DNA bakteryjne tworzy katenany, które są
odplątywane w 2 etapach - 1
°
denaturacja pozostałych niedoreplikowanych 2-
helisowych skrętów sprzęgających obie nici, 2
°
wypełnienie luk przez system
naprawczy; pozostawia to prawoskrętny równoległy pierścieniowaty katenan z
parzysta liczbą węzłów, które są rozwiązywane przez topoizomerazę IV
eukariotyczne chromosomy mają na końcach chromosomów
struktury zw. telomerami – jedna nić telomeru jest
złożona z wielu tandemowych powtórzeń krótkiego,
bogatego w G regionu (którego sekwencja różni się
pomiędzy gatunkami)
terminacja
replikacji liniowego DNA
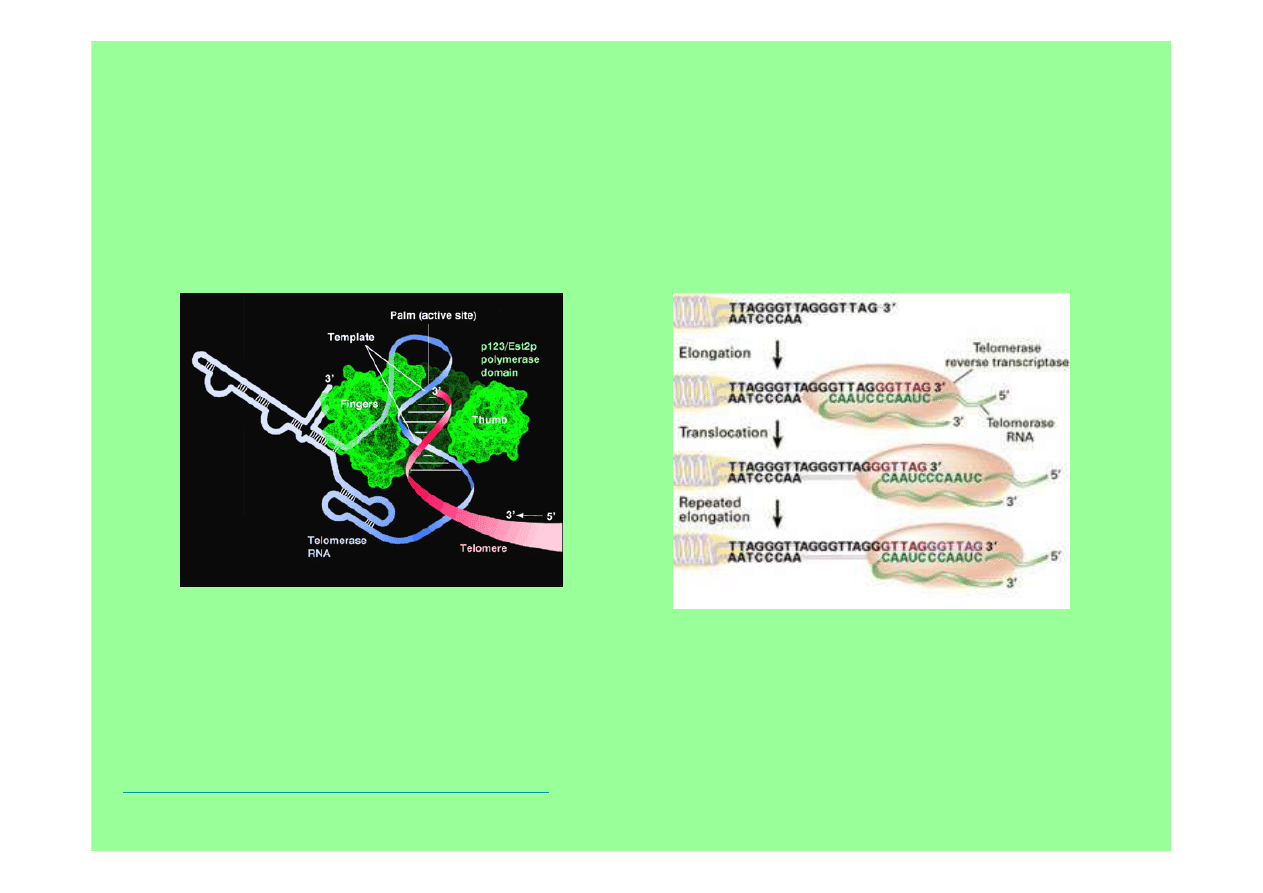
nić bogata w G jest syntetyzowana przez telomerazę – odwrotną
transkryptazę zawierającą krótki RNA służący jako matryca w syntezie
telomeru
nić bogata w C jest syntetyzowana przez zwykłą syntezę DNA z primera
RNA, tak jak nić opóźniona konwencjonalnej replikacji; mechanizm ten
zapewnia odbudowywanie telomerów
http://www.youtube.com/watch?v=AJNoTmWsE0s
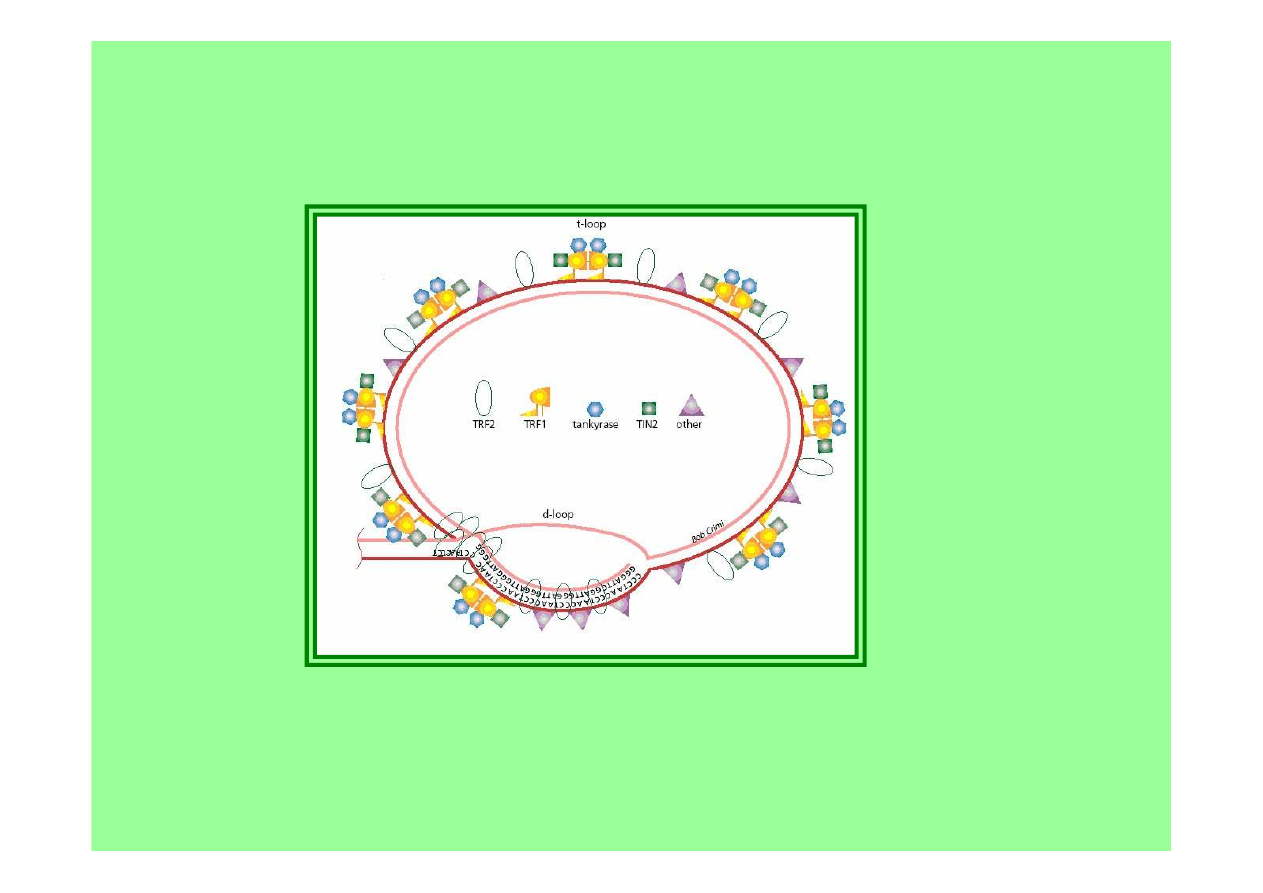
białka TRF1 i 2 pomagają telomerom utworzyć pętlę t,
w której 1-niciowy koniec 3’ dokonuje inwazji do wyżej
położonego odcinka 2-niciowego
niemniej na końcach chromosomów zawsze pozostaje
wystający 1-niciowy 3’ koniec – przed degradacją chroni
go białkowa struktura różna od nukleosomów
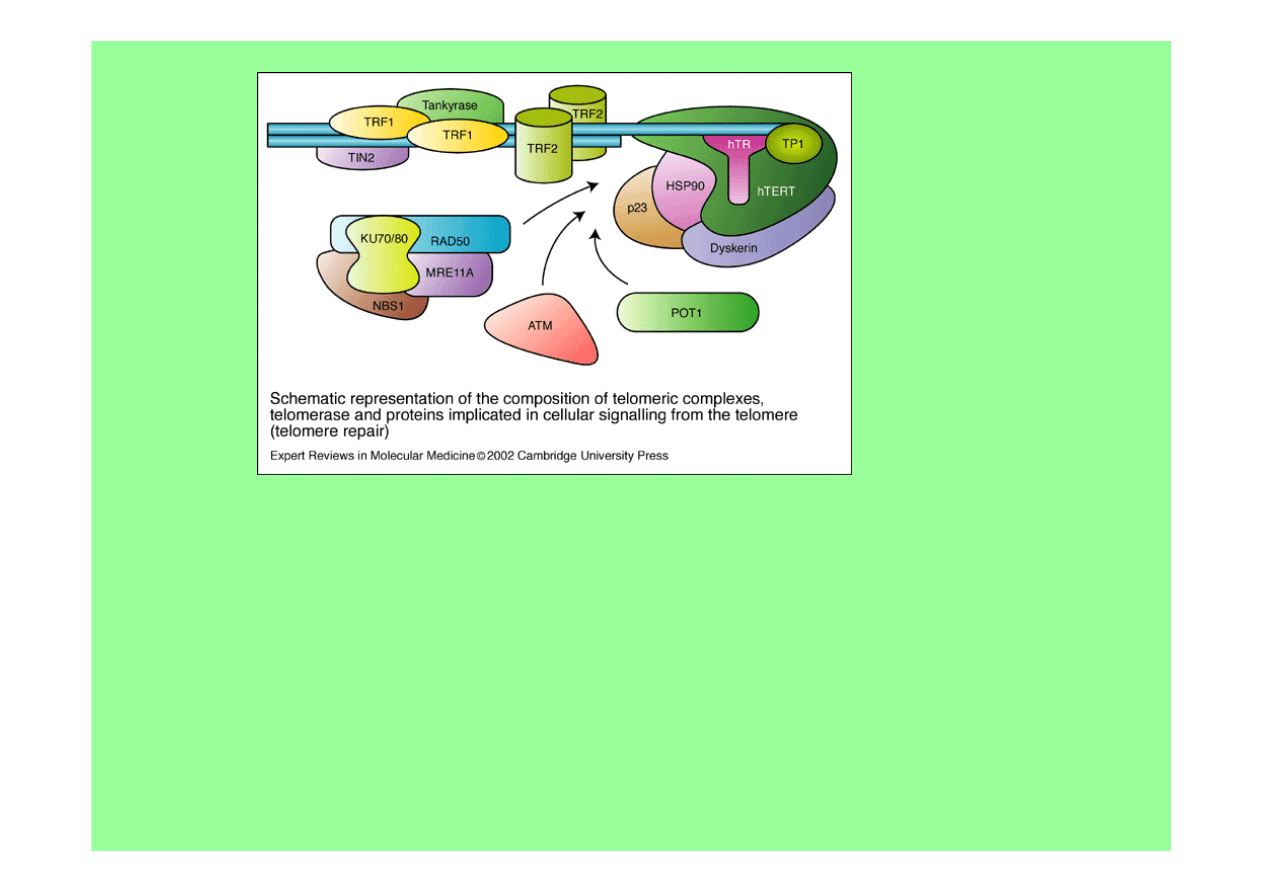
Schemat kompleksów telomerowch, telomerazy i białek uczestniczących w
komórkowym sygnałowaniu z telomerów (naprawie telomerów). Telomeraza
składa się z matrycowego RNA hTR i katalitycznego białka hTERT. Wydaje się,
że w regulacji
in vivo
aktywności hTR–hTERT i utrzymaniu struktury telomerów
biorą udział dodatkowe elementy. Np. białka TRF1, TRF2, tankyrase, TIN2, RAP1
(niepokazane) i POT1 oddziałują z telomerami i mogą regulować otwieranie i
zamykanie wolnych końców telomerów i ich dostępność dla innych kompleksów
białkowych jak telomeraza. Szereg białek i RNP, w tym HSP90, DKC1, L22, P23 i
GAR1 (niepokazane), może uczestniczyć w składaniu telomerazy i ułatwiać jej
interakcje z telomerami. Inne białka, jak MRE11A, NBS1, KU70, KU80, DNAPK
(niepokazane) i ATM mogą funkcjonować w szlaku wykrywania krótkich
telomerów i wyzwalać szlaki odpowiedzi na uszkodzenia DNA lub naprawę
sekwencji telomerowych.

The Nobel Prize in Physiology or Medicine 2009
"for the discovery of how chromosomes are protected by
telomeres and the enzyme telomerase"
b. 1952
(in London, United Kingdom)
b. 1961
(in San Diego, CA USA)
b. 1948
(in Hobart, Tasmania, Australia)
Harvard Medical School;
Massachusetts General Hospital
Boston, MA, USA;
Howard Hughes Medical Institute
Johns Hopkins University
School of Medicine
Baltimore, MD, USA
University of California
San Francisco, CA, USA
USA
USA
USA
1/3 of the prize
1/3 of the prize
1/3 of the prize
Jack W. Szostak
Carol W. Greider
Elizabeth H. Blackburn
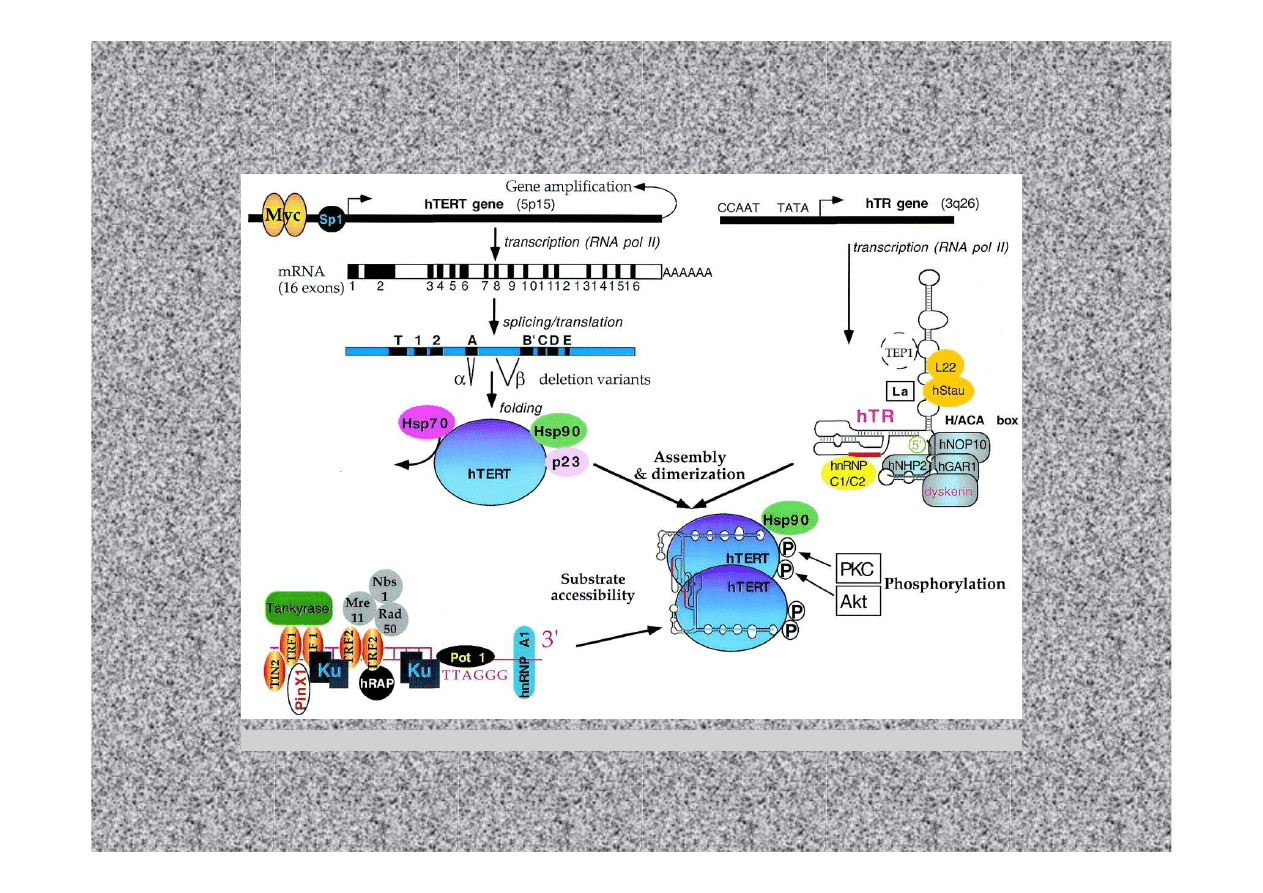
Mergny et al. 2002 Nucl. Acids Res. 30:839-865; doi:10.1093/nar/30.4.839
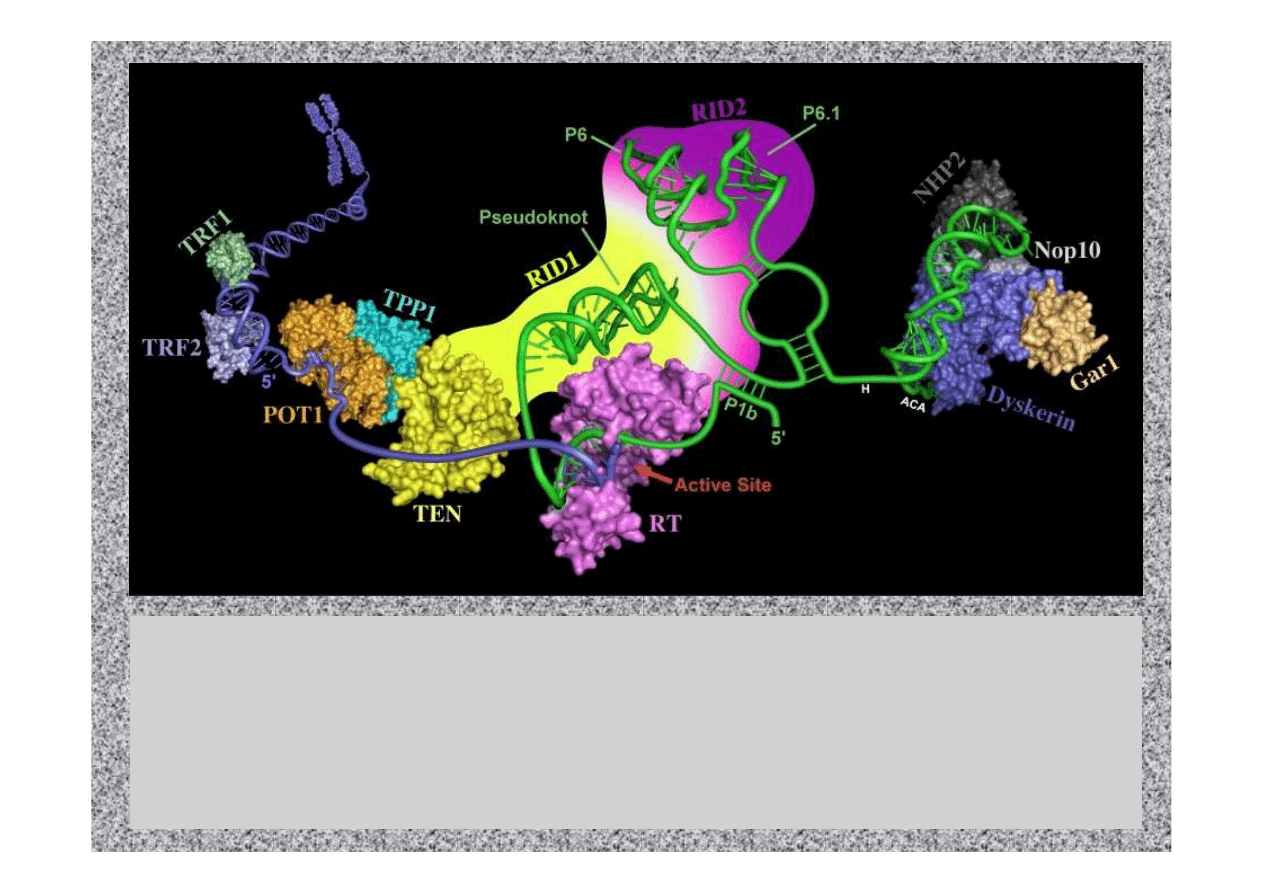
Illustration of the vertebrate telomerase ribonucleoprotein complexed with its substrate, telomeric DNA (blue).
Telomerase consists of a reverse transcriptase (TERT, in yellow and pink), and RNA component (TR, in green), and the
dyskerin protein complex. The TERT protein contains the N-terminal domain (TEN), RNA-interacting domains 1 and 2
(RID1 and RID2), and the reverse transcriptase domain (RT). The RNA component contains base-pairing region P1b
that defines the template boundary, the template that encodes the telomeric sequences, the pseudoknot, conserved
regions 4 and 5 (CR4-CR5) that include P6 and P6.1, and the 3’-box H/ACA stem-loop bound by the H/ACA
ribonucleoprotein complex (dyskerin, Nop10, NHP2, and Gar1). RID1 is bound to pseudoknot-template region and RID2
to CR4-CR5. There six proteins that associate with telomeres to form the shelterin complex (TRF1, TRF2, POT1,
directly bound telomeric DNA and TIN2, TPP1, and Rap1 associated with the telomeric DNA binding proteins). TRF1
and TRF2 are bound to double-stranded telomeric DNA, while POT1 is bound to the single-stranded region.
http://telomerase.asu.edu/
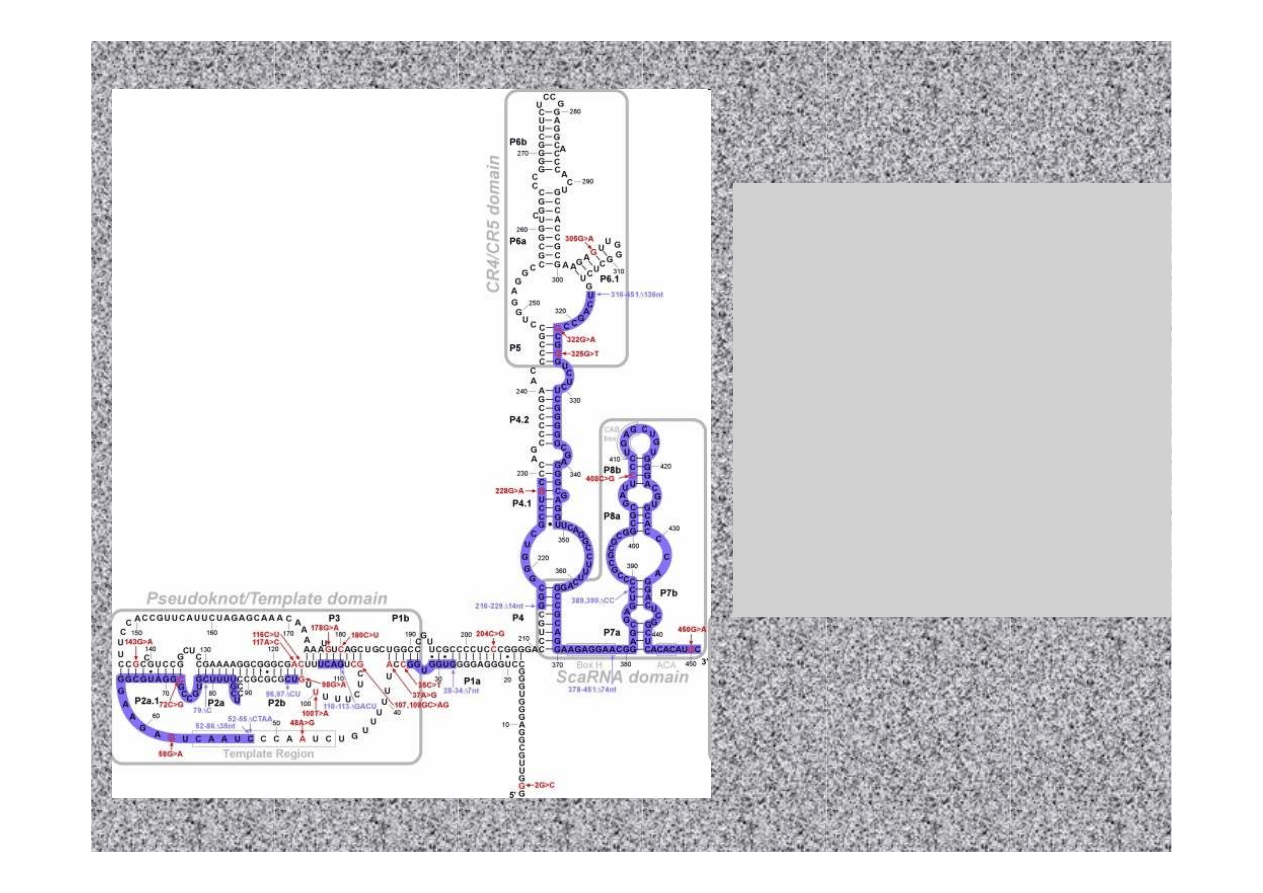
Numerous mutations within TR, the 451 nt
RNA component of the telomerase RNP,
have been connected with human
diseases.
Three domains characterize TR,
pseudoknot that includes the template,
conserved regions 4 and 5 (CR4-CR5),
and ScaRNA domain for nuclear
recruitment.
The primary nucleotide sequence is in
black,
point mutations are colored red,
while
deletion mutations are shaded blue
,
and both are labeled.
http://telomerase.asu.edu/diseases.html

Numerous mutations causing amino acid substitution, additions, deletions, and frame shifts within
TERT, the essential protein component of the telomerase, have been connected with human
diseases.
TERT is composed of three domains, N-terminal extension (NTE) that contains RNA-interaction
domains 1 and 2 (RID1 and RID2), reverse transcription domain (RT) where nucleotide transfer
occurs, and a C-terminal extension (CTE) for processivity and localization.
The black line represents the mRNA sequence of 4015 nt with the untranslated regions (UTR)
labeled and the grey box corresponds to the protein sequence. The individual motifs are labeled
and
NTE is denoted by green
,
CTE by orange
, and the
central RT domain by blue
boxes.
The mutated residues are colored red
and the change in amino acids is labeled.
gene sequence 41881 bp
TERT protein sequence 1132 AA
Human diseases can be also caused by mutations in genes coding for telomerase-associated
proteins: DKC1 (dyskerin), Nola3 (Nop10), Nola2 (NHP2), TINF2 (TIN2 – one of the six proteins
of shelterin complex).
http://telomerase.asu.edu/diseases.html

• replikacja ludzkiego DNA (3x10
9
pz) – 20 min
polimeryzacja jest wolniejsza niż u Procaryota,
ale istnieje wiele ori
i wiele cząsteczek pol DNA
• błędy replikacji: 5-7 nieprawidłowych
podstawień nukleotydowych na 1 kopię genomu
Wyszukiwarka
Podobne podstrony:
BiolMol 10 id 87436 Nieznany
BiolMol 12 id 87437 Nieznany (2)
Abolicja podatkowa id 50334 Nieznany (2)
4 LIDER MENEDZER id 37733 Nieznany (2)
katechezy MB id 233498 Nieznany
metro sciaga id 296943 Nieznany
perf id 354744 Nieznany
interbase id 92028 Nieznany
Mbaku id 289860 Nieznany
Probiotyki antybiotyki id 66316 Nieznany
miedziowanie cz 2 id 113259 Nieznany
LTC1729 id 273494 Nieznany
D11B7AOver0400 id 130434 Nieznany
analiza ryzyka bio id 61320 Nieznany
pedagogika ogolna id 353595 Nieznany
Misc3 id 302777 Nieznany
cw med 5 id 122239 Nieznany
więcej podobnych podstron