
Biosynteza białka
Janusz Szemraj
Zakład Biochemii Medycznej
Uniwersytet Medyczny

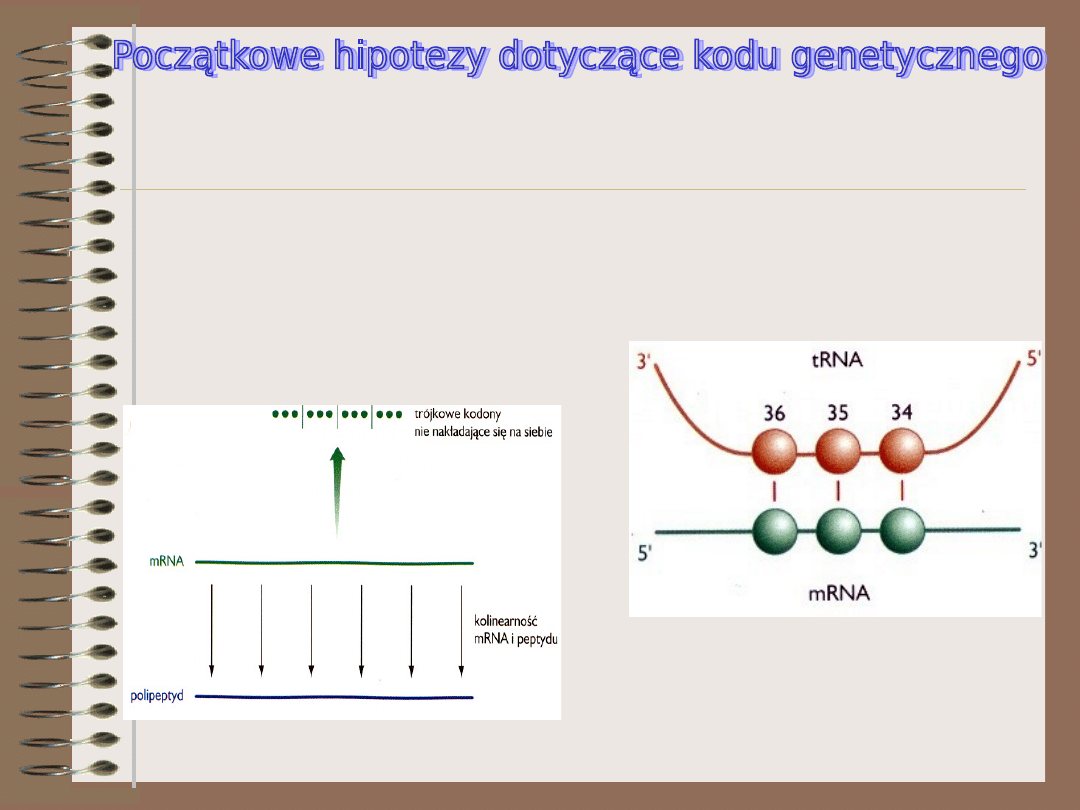
W latach 50. ubiegłego wieku zorientowano się, że kod genetyczny musi być
kodem trójkowym aby mógł zakodować informację o wszystkich 20 aminokwasach
wchodzących w skład białek. Jako hipotezę roboczą przyjęto też założenie, że
mRNA zawiera serie nie nakładających się na siebie kodonów kolinearnych z
kodowanym przez to mRNA polipeptydem. W późniejszych latach odkryto procesy,
które zmusiły do uzupełnienia omawianych założeń. Należy do nich na przykład
brak kolinearności między wieloma genami eukariotycznymi a kodowanymi przez
nie polipeptydami, wynikający z obecności intronów (introny odkryto dopiero w
1977r.)
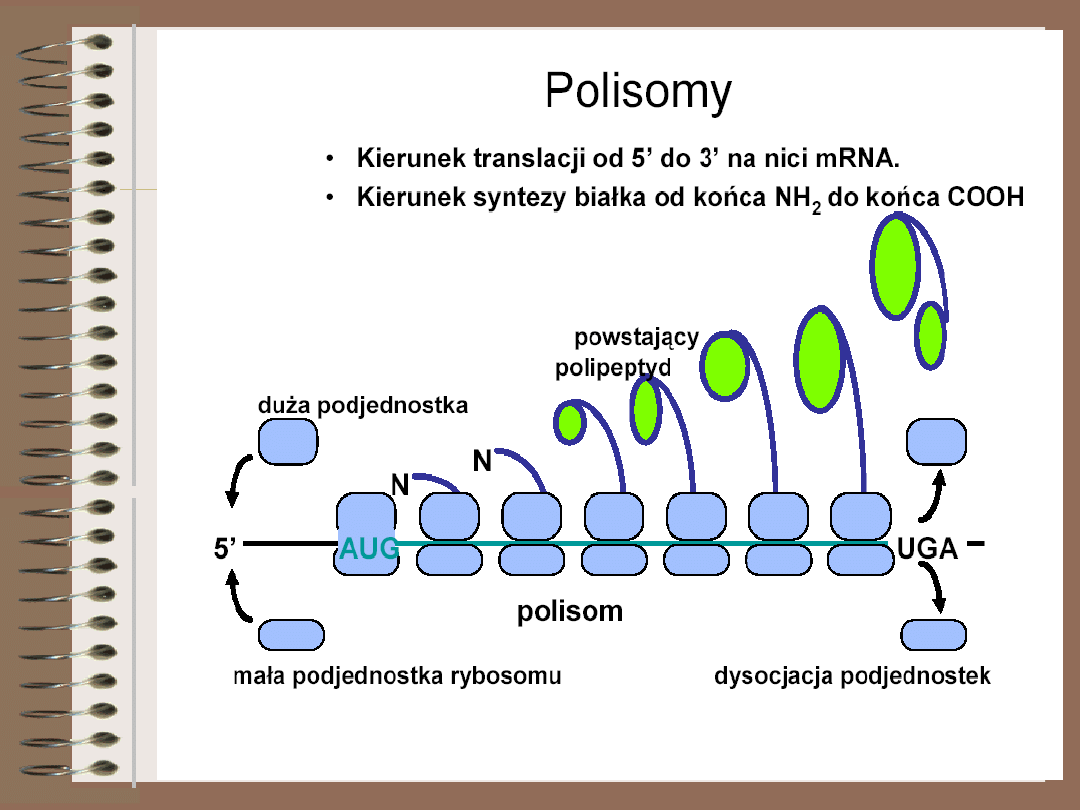
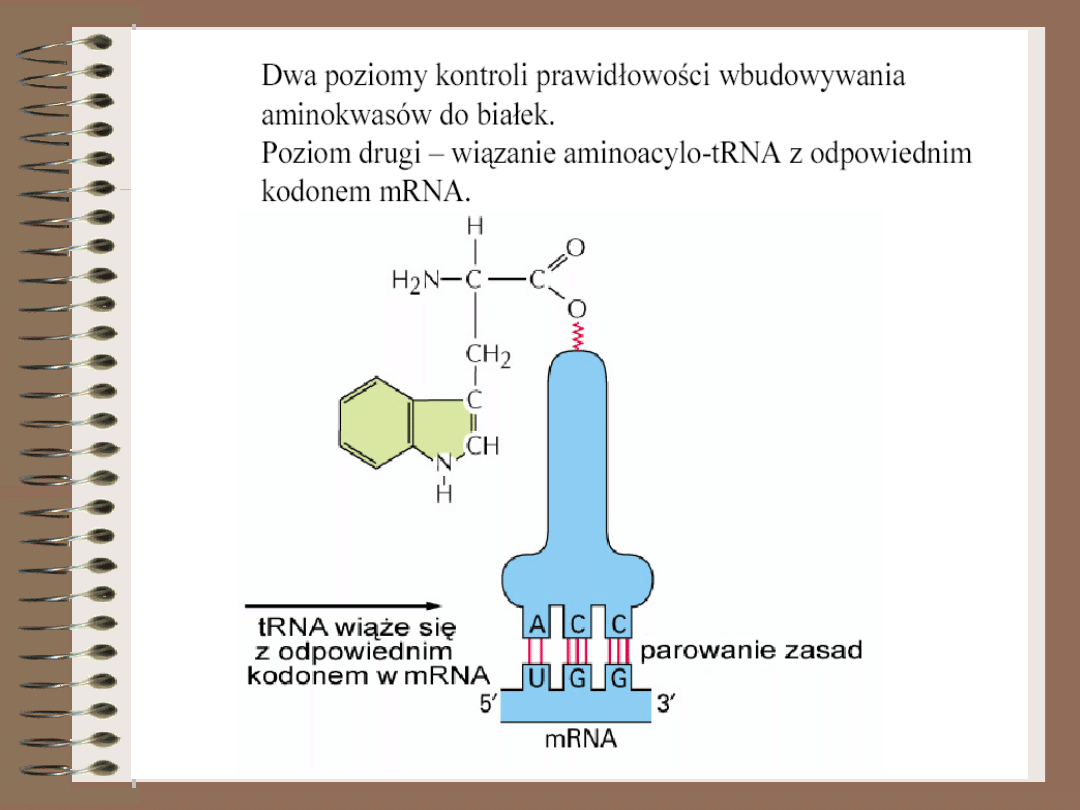
Oddziaływanie między kodonem i
antykodonem jest prostym procesem
opierającym się na łączeniu się w pary
zasad wchodzących e skład antykodonu i
kodonu. W rzeczywistości proces
rozpoznawania kodonu jest bardziej
skomplikowany z powodu dodatkowych
możliwości wynikających z zasady
tolerancji


Do syntezy białek potrzeba 20 aminokwasów, dlatego musi być
przynajmniej 20 oddzielnych kodonów, Kodony, z których każdy
składał by się z 2 nukleotydów mogłyby kodować tylko 16
aminokwasów (4
2
= 16) podczas gdy kodony o trzech nukleotydach –
64 (4
3
=64)
Wiadomo obecnie, że każdy kodon składa się z tripletu nukleotydów.
Trzy spośród kodonów nie kodują swoistego aminokwasu. Nazwano je
kodonami nonsensownymi. Przynajmniej dwa z nich używane są jako
sygnały terminacji.

Kod genetyczny jest:
Zdegenerowany,
Zdegenerowany,
ponieważ 20 aminokwasów kodowanych jest przez aż 61 kodonów.
Niektóre aminokwasy są kodowane przez kilka kodonów, np. serynę
koduje 6 różnych kodonów. Inne aminokwasy – metionina i tryptofan mają
pojedynczy kodon. Na ogół trzeci nukleotyd kodonu jest mniej ważny niż
dwa pierwsze w określaniu swoistego aminokwasu jaki ma być
wbudowany do białka.
Jednoznaczny,
ponieważ dla każdego swoistego kodonu można wskazać tylko jeden
aminokwas (z rzadkimi wyjątkami). Swoisty kodon przypisany jest
pojedynczemu aminokwasowi.
Nienakładający się
ponieważ odczytywanie kodu genetycznego podczas biosyntezy białek
nie obejmuje nakładających się kodonów (można tu znaleźć nieliczne
odstępstwa np. u wirusów))
Bezprzestankowy
ponieważ od momentu, od którego zacznie się odczytywanie od
swoistego kodonu nie ma żadnych znaków przestankowych.
Uniwersalny
Do niedawna sądzono, że kod genetyczny jest w pełni uniwersalny.
Obecnie wykazano, że między innymi grupa cząsteczek tRNA w
mitochondriach u niższych i wyższych eukariontów z człowiekiem
włącznie odczytuje kilka kodonów w inny sposób niż cząsteczki tRNA w
cytoplazmie nawet tych samych gatunków
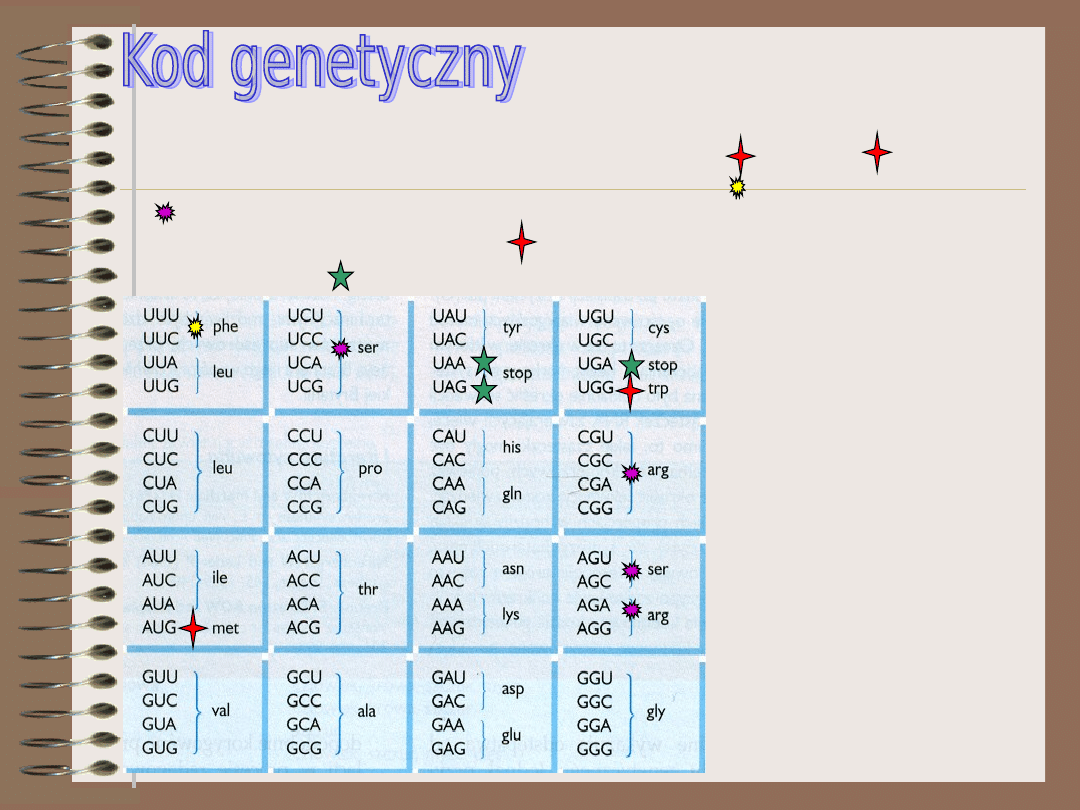
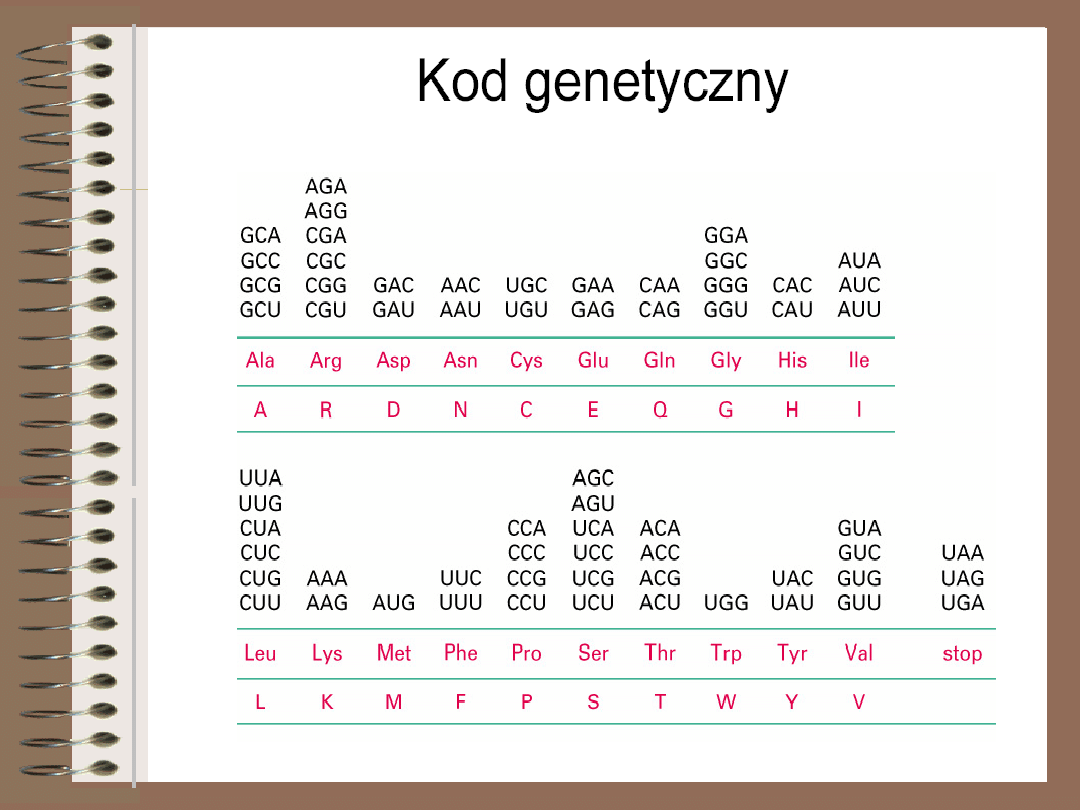
64 kodony tworzące kod genetyczny można podzielić na grupy. Kodony
64 kodony tworzące kod genetyczny można podzielić na grupy. Kodony
wchodzące w skład jednej grupy kodują jeden aminokwas. Tylko tryptofan
wchodzące w skład jednej grupy kodują jeden aminokwas. Tylko tryptofan
i metionina są kodowane przez jeden kodon. Pozostałe przez dwa (np.
i metionina są kodowane przez jeden kodon. Pozostałe przez dwa (np.
phe ) do sześciu kodonów (ser, arg ). Dlatego mówimy, że kod
phe ) do sześciu kodonów (ser, arg ). Dlatego mówimy, że kod
genetyczny jest zdegenerowany. Kod ma cztery kodony przestankowe.
genetyczny jest zdegenerowany. Kod ma cztery kodony przestankowe.
Kodon inicjacyjny AUG , który koduje również metioninę oraz trzy kodony
Kodon inicjacyjny AUG , który koduje również metioninę oraz trzy kodony
terminacyjne
terminacyjne
.
.

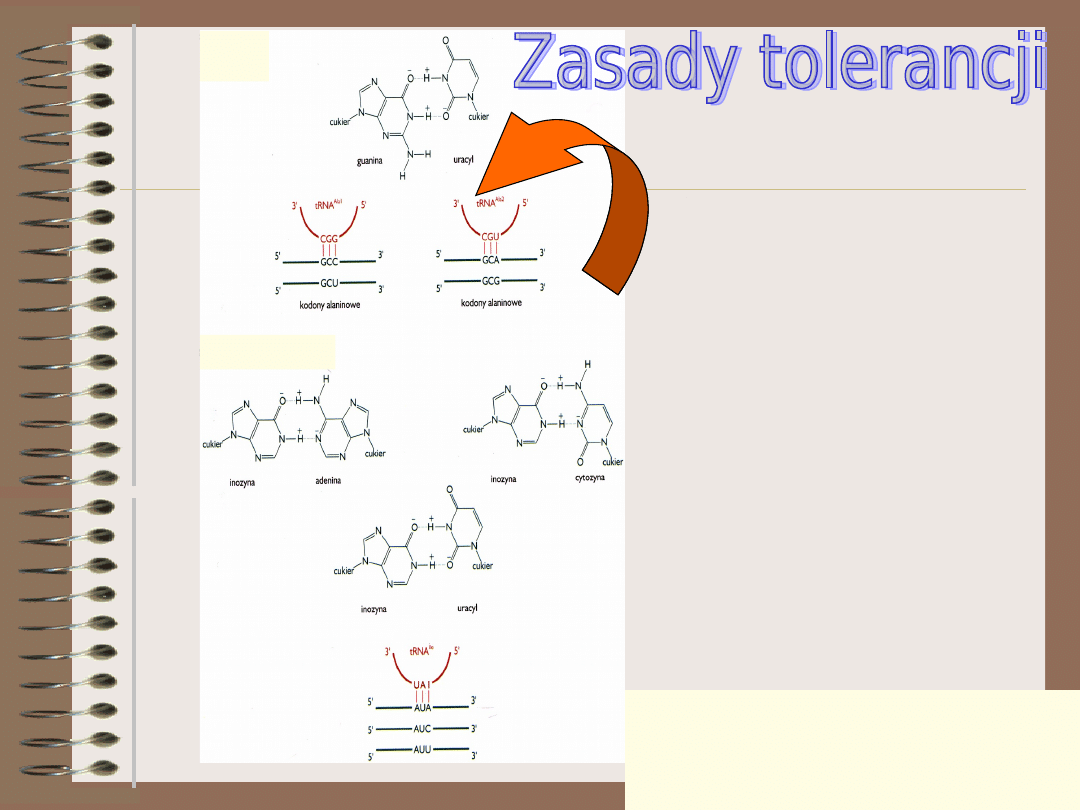
Kodony izoleucynowe
To podstawowe prawo ekspresji
genów zostało zaproponowane
przez Cricka i okazało się
prawdziwe.
Ponieważ antykodon znajduje się
w pętli cząsteczki tRNA, trójka
nukleotydów jest lekko wygięta i
nie może być na całej długości
tak samo ułożona wzdłuż
kodonu. W rezultacie możliwe
jest tworzenie nietypowych par
zasad między TRZECIM
NUKLEOTYDEM KODONU A
PIERWSZYM NUKLEOTYDEM
ANTYKODONU
Możliwe jest
powstawanie
różnych par
zasad, w
szczególności,
gdy nukleotyd w
pozycji 34 jest
zmodyfikowany.
Na rys. A i B
przedstawiono
dwie najczęstsze
możliwości
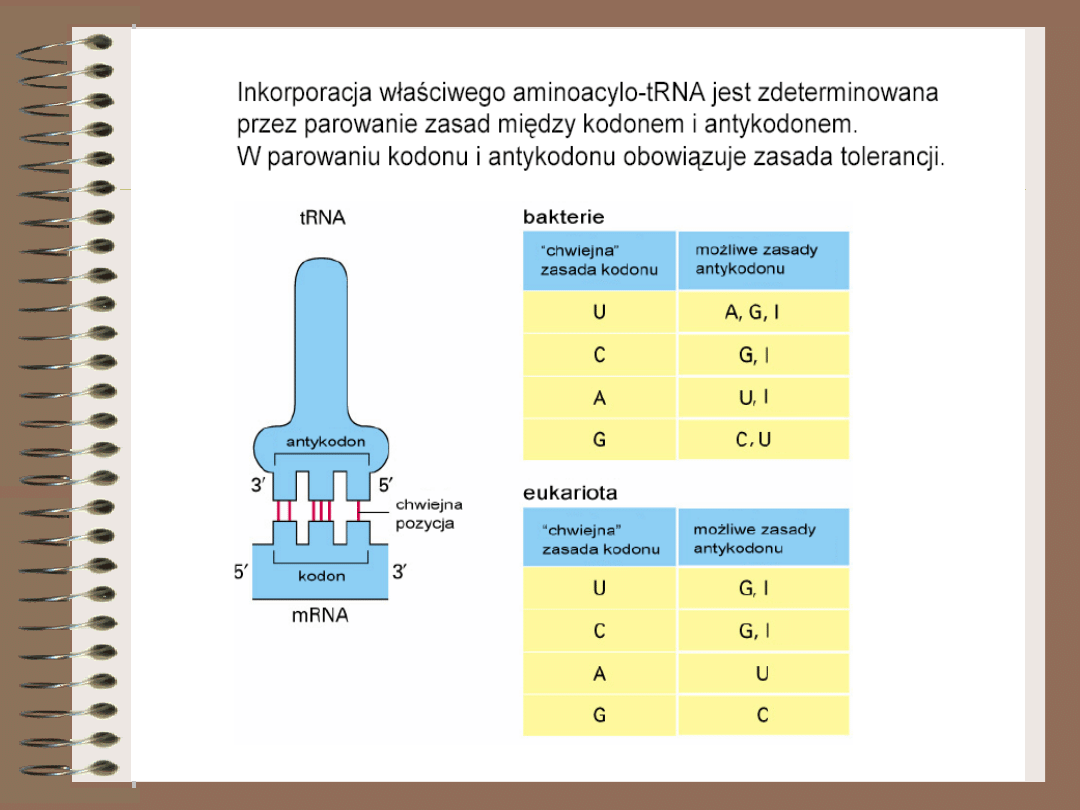
Zasada tolerancji redukuje
ilość
niezbędnych w komórce
cząsteczek tRNA, umożliwiając
jednej cząsteczce
odczytywanie dwóch a nawet
trzech kodonów
Inozyna występuje
tylko w tRNA.
mRNA nie jest
modyfikowany w
ten sposób
(A) G łączy się w parę z
U
(B) Inozyna łączy się w parę z A, C lub U
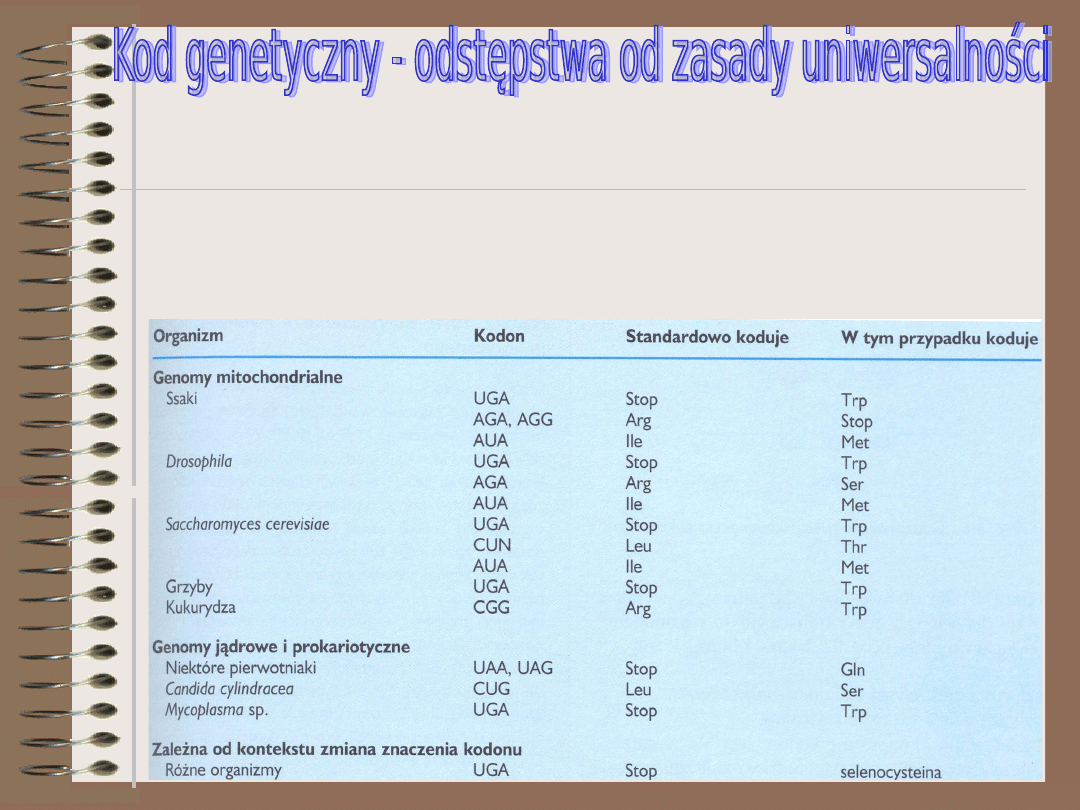
Ważną cechą kodu genetycznego jest jego uniwersalność, jednak odkryto trzy rodzaje
odstępstw od tej zasady (w mitochondrialnym kodzie genetycznym, w jądrowym kodzie
genetyczntm niższych eukaryota oraz zależne od kontekstu zmiany znaczenia kodonu). Ze
zmianą znaczenia kodonu zależną od kontekstu mamy so czynienia, gdy syntetyzowane
białko zawiera selenocysteinę. Dotyczy to wielu organizmów pro- i eukariotycznych łącznie
z człowiekiem, ponieważ białka zawierające selenocysteinę są ość rozpowszechnione
(należy do nich np. peroksydaza glutationowa). Selenocysteina kodowana jest przez kodon
UGA. MA on więc dwa znaczenia- jest także wykorzystywany jako kodon terminacyjny. W
procesie aminoacylacji tRNA
SeCys
początkowo przyłączana jest do tej cząsteczki
seryna, która
następnie przekształcana jest w selenocysteinę. Kodony UGA kodujące selenocysteinę,są
odróżnialne od prawdziwych kodonów STOP dzięki obecniści na mRNA struktury szpilki do
włosów.
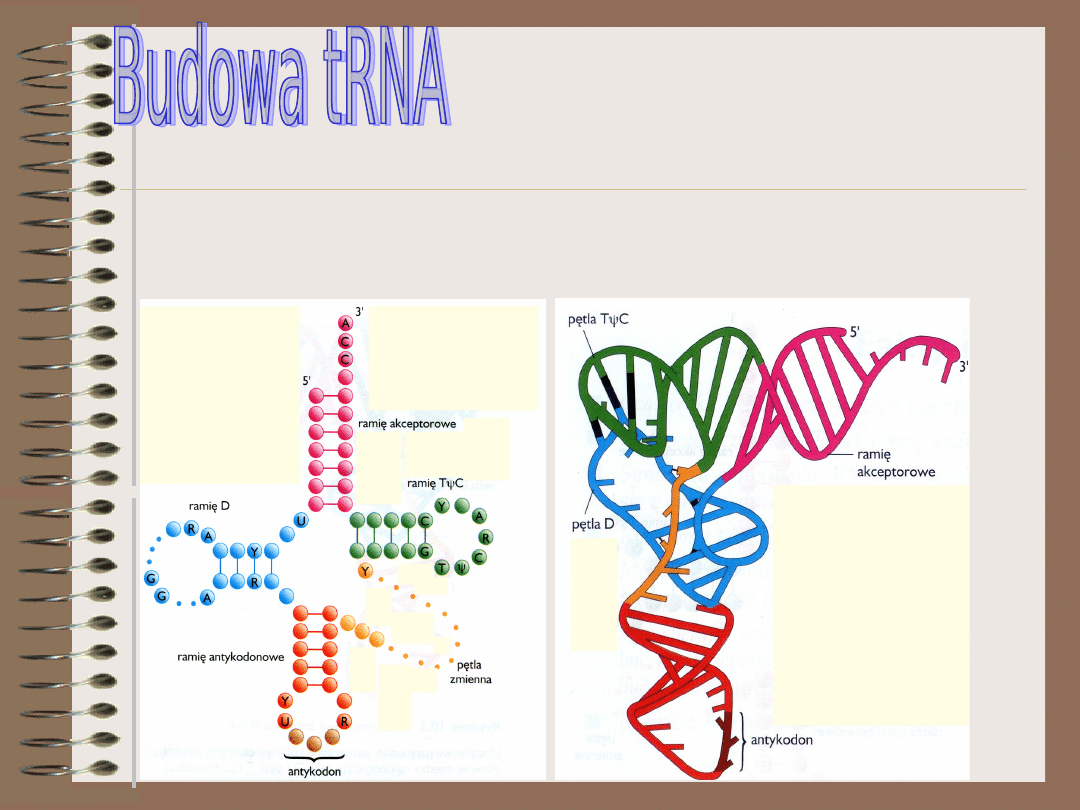
Cząsteczki tRNA składają się z około 75 nukleotydów.
Pomimo różnic w sekwencji mają wiele wspólnych
cech. Wewnątrzcząsteczkowa komplementarność
zasad pozwala wytworzyć drugorzędową strukturę
przypominającą liść koniczyny (A)
A
A
B
B
Wszystkie tRNA zawierają cztery główne ramiona (A). Ramię akceptorowe składa się z
szypuły utworzonej ze sparowanych zasad, która kończy się sekwencją CCA (koniec 3’).
Aminokwas przyłącza się do końcowej adenozyny. Ramię antykodonowe rozpoznaje
triplet nukleotydów , czyli kodon na matrycy mRNA. Ramię dodatkowe jest najbardziej
zmiennym regionem tRNA. Na rys B przedstawiono model obrazujący trójwymiarową
strukturę tRNA. Cząsteczka kształtem przypomina literę L
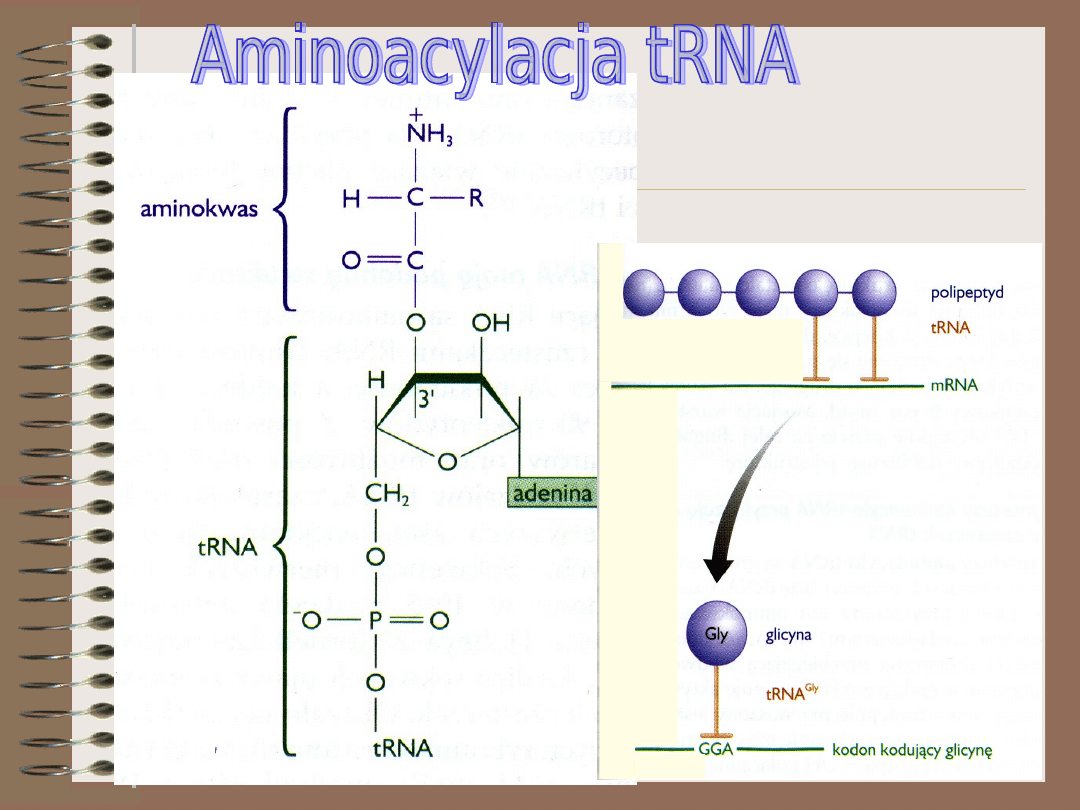
Pomiędzy grupą –OH znajdującą się
Pomiędzy grupą –OH znajdującą się
przy węglu 3’ adenozyny (na 3’ końcu
przy węglu 3’ adenozyny (na 3’ końcu
tRNA) a aminokwasem wytworzone
tRNA) a aminokwasem wytworzone
zostaje wiązanie estrowe
zostaje wiązanie estrowe
tRNA pełni w
procesie translacji
rolę cząsteczki
adaptorowej

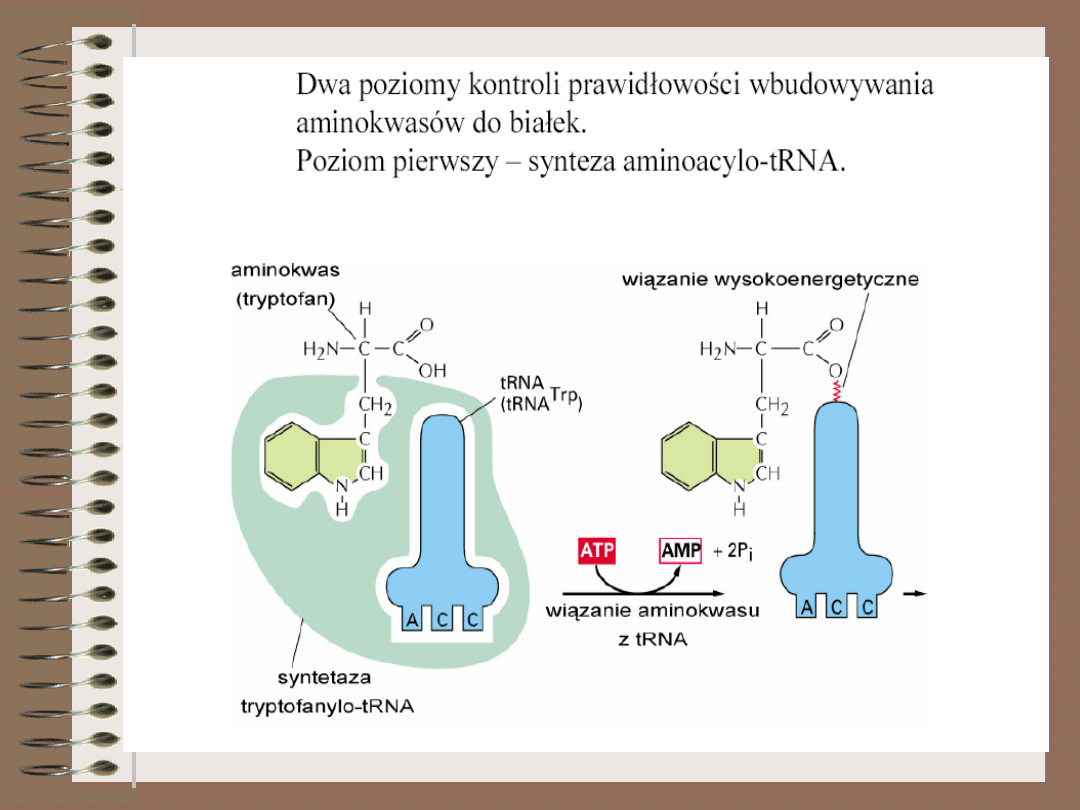
Właściwy proces biosyntezy białka poprzedzony jest
Właściwy proces biosyntezy białka poprzedzony jest
aktywacją aminokwasów i ich połączeniem z tRNA. Dla
aktywacją aminokwasów i ich połączeniem z tRNA. Dla
każdego z 20 aminokwasów istnieje przynajmniej jeden
każdego z 20 aminokwasów istnieje przynajmniej jeden
swoisty tRNA. Enzymami uczestniczącymi w
swoisty tRNA. Enzymami uczestniczącymi w
rozpoznawaniu właściwych par Aminokwas-tRNA i ich
rozpoznawaniu właściwych par Aminokwas-tRNA i ich
łączeniu są SYNTETAZY AMINOACYLO-tRNA. Zatem
łączeniu są SYNTETAZY AMINOACYLO-tRNA. Zatem
komórka musi dysponować przynajmniej 20 rodzajami
komórka musi dysponować przynajmniej 20 rodzajami
takich enzymów
takich enzymów
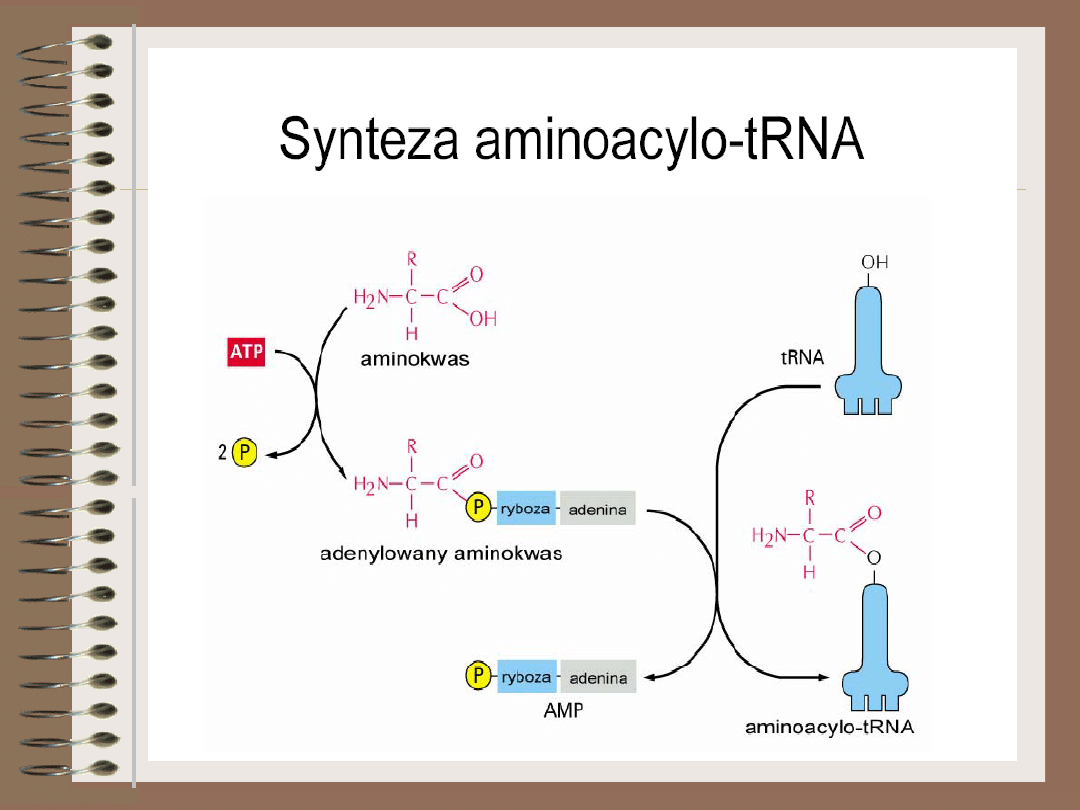
Reakcja aminoacylacji tRNA przebiega dwuetapowo
Aminokwas + ATP Aminoacylo~AMP + PP
i
Aminoacylo~AMP + tRNA Aminoacylo-tRNA +
AMP
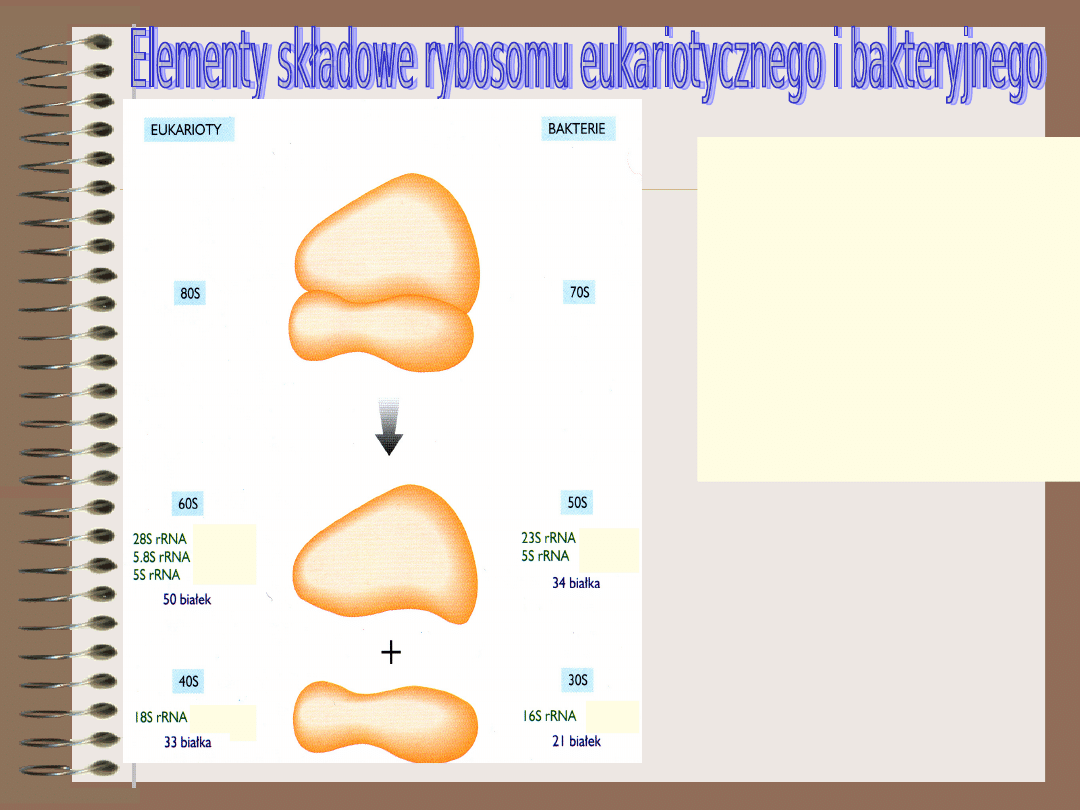
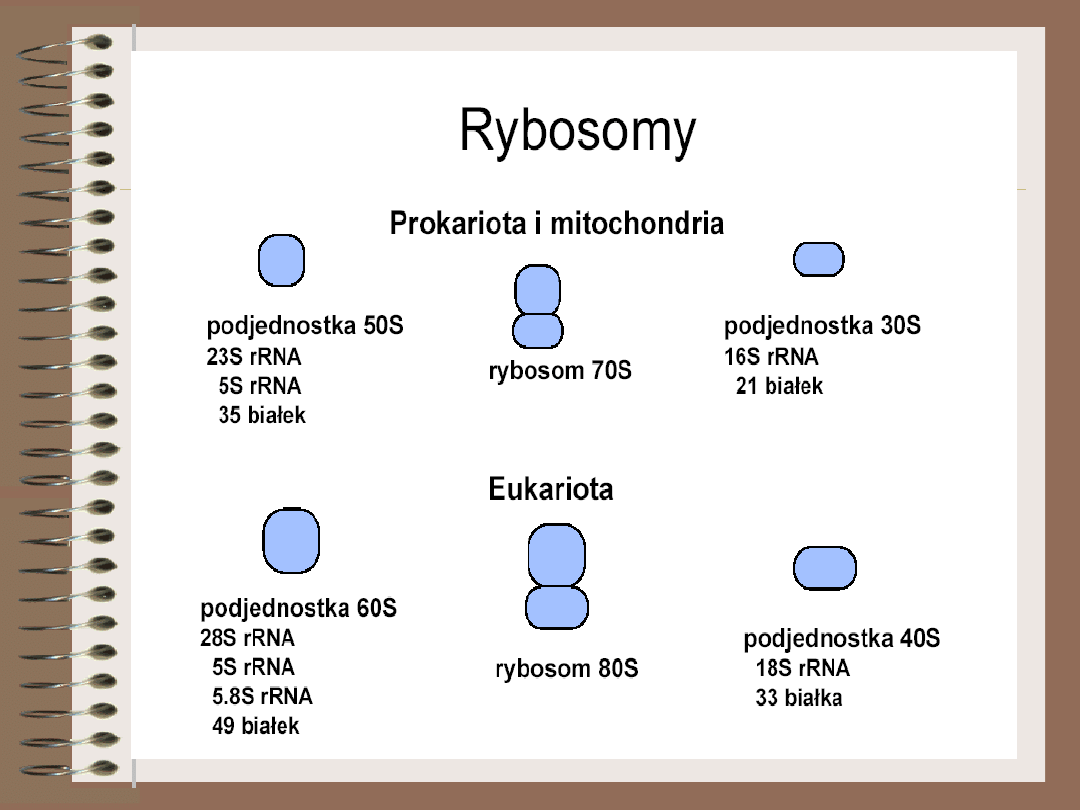
W komórce E.coli znajduje
się około 20 000
rybosomów; typowa
komórka ludzka zawiera
ich prawdopodobnie
więcej.
W skład obydwu
podjednostek poza rRNA
wchodzą rózne białka
rybosomowe
Duża
podjednostk
a
Mała
podjednostk
a

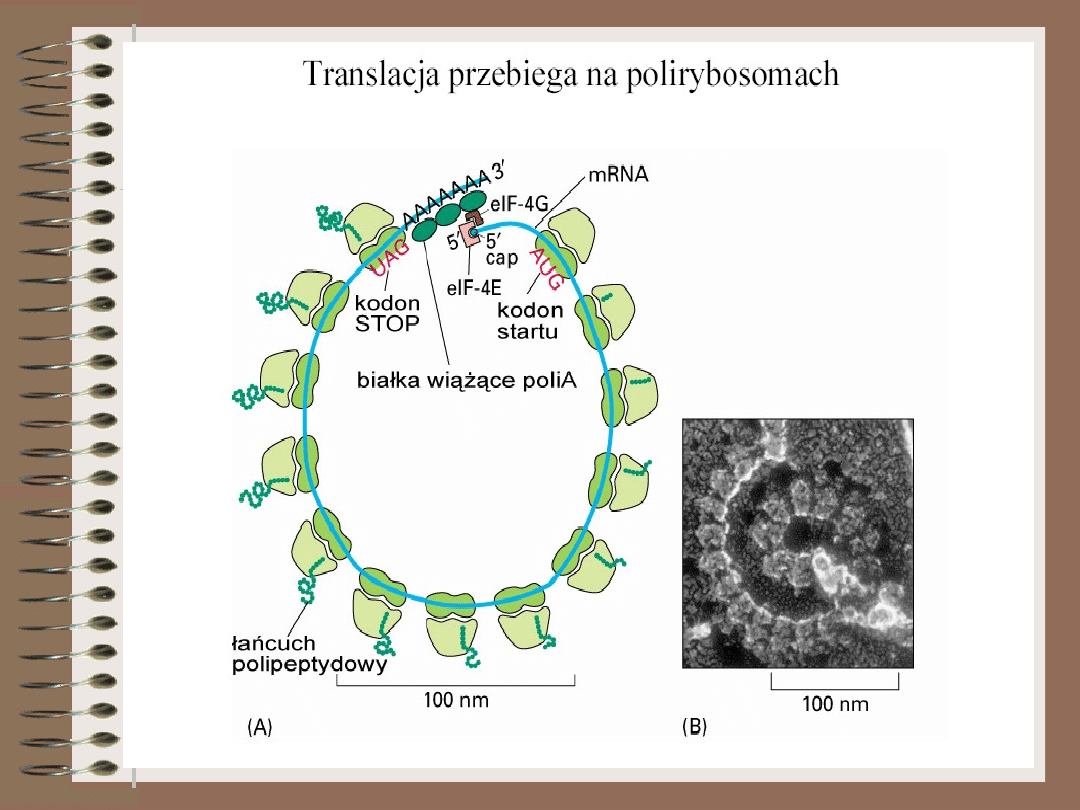
Rybosomy, które nie biorą aktywnego udziału w procesie syntezy
białka, rozdysocjowują na podjednostki pozostające w cytoplazmie, aż
do rozpoczęcia nowej rundy translacji.
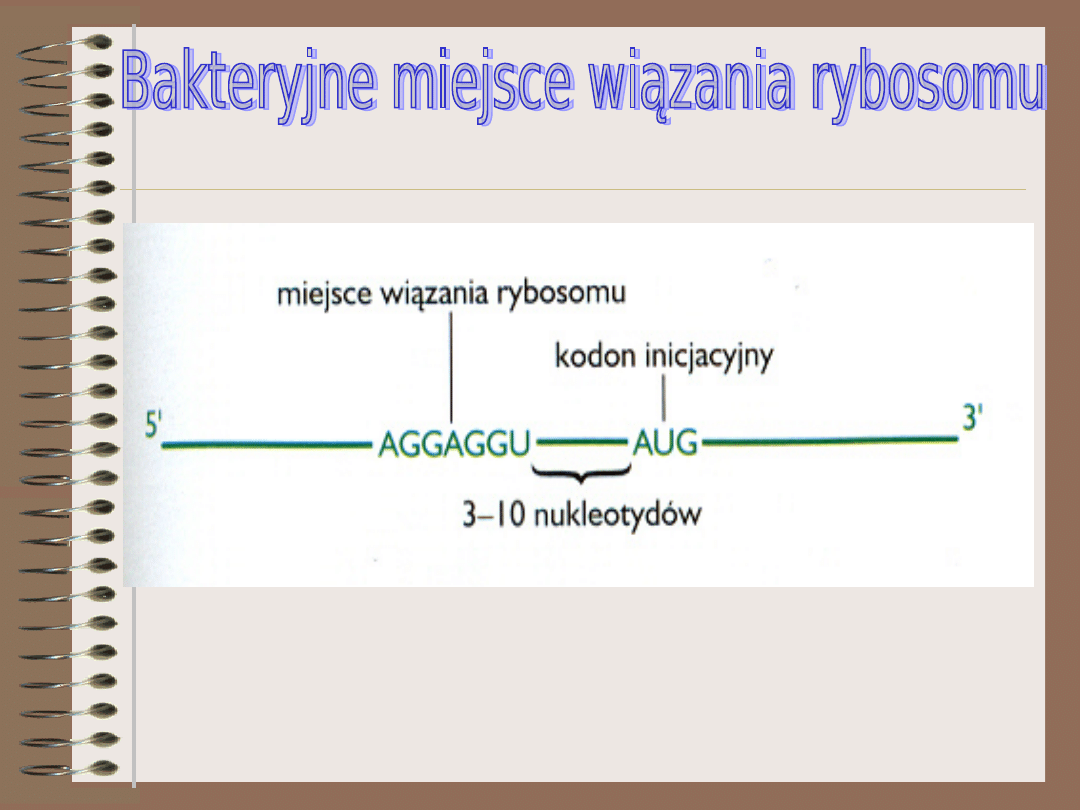
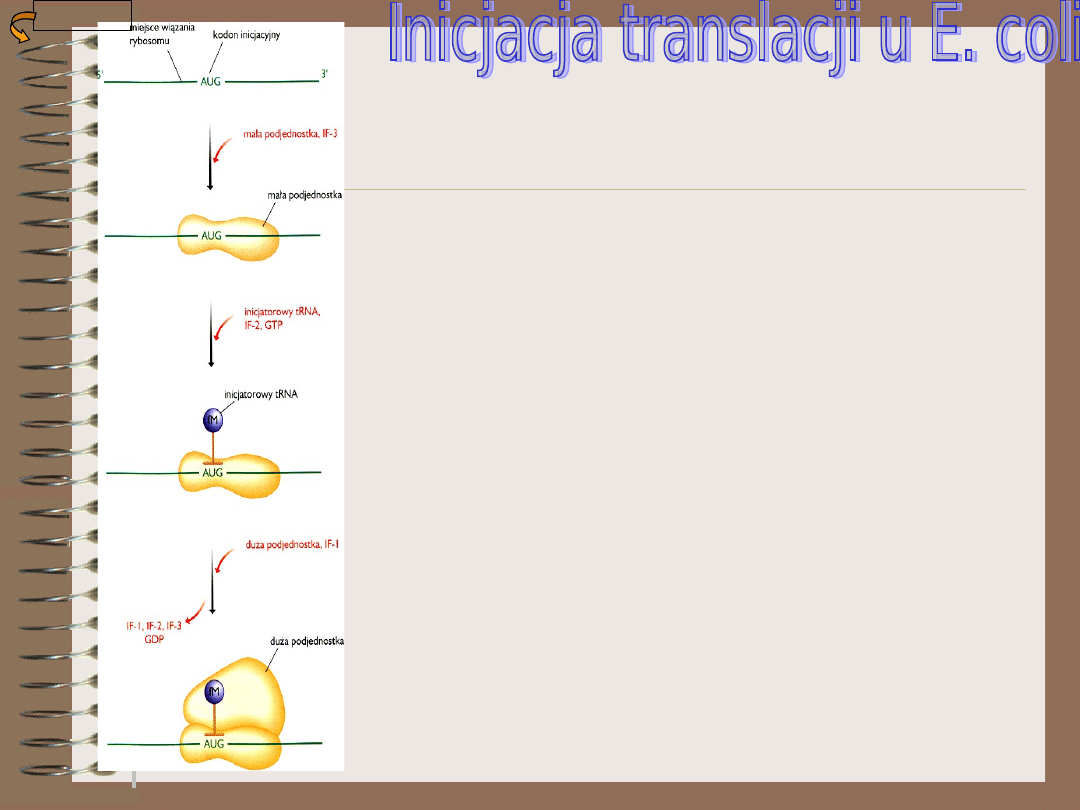
1. U bakterii proces ten rozpoczyna się w momencie
gdy mała podjednostka połączona z translacyjnym
czynnikiem inicjacyjnym IF-3 przyłącza się do
MIEJSCA WIĄZANIA RYBOSOMU NA mRNA.
2. Przyłączenie małej podjednostki rybosomu
umiejscawia ją w rejonie obejmującym kodon
inicjacyjny (AUG)
3. Dołączenie inicjatorowego tRNA przy udziale
czynnika inicjacyjnego IF-2 połączonego z
cząsteczką GTP, która jest źródłem energii w
ostatnim etapie inicjacji translacji. Inicjatorowy
tRNA połączony jest z metioniną która jest
modyfikowana do N-formylometioniny
(przyłączenie
grupy formylowej –COH do grupy aminowej; wolna jest tylko grupa
karboksylowa inicjatorowej metioniny i tylko ona może
uczestniczyć w tworzeniu wiązania peptydowego. Dzięki temu
synteza peptydu zachodzi wyłącznie od końca aminowego do
karboksylowego)
4. Etap inicjacji kończy się związaniem czynnika IF-
1, który stabilizuje kompleks inicjacyjny,
umożliwiając przyłączenie dużej podnednostki.Jej
przyłączenie wymaga energii, która jest
uwalniana w czasie hydrolizy związanej cząsteczki
GTP. Proces ten prowadzi do uwolnienia
czynników inicjacyjnych
czyli sekwencja
Shine Dalgarno
komplementarna
do fragmentu na
3’ końcu 16S
rRNA – składnika
małej
podjednostki
rybosomu
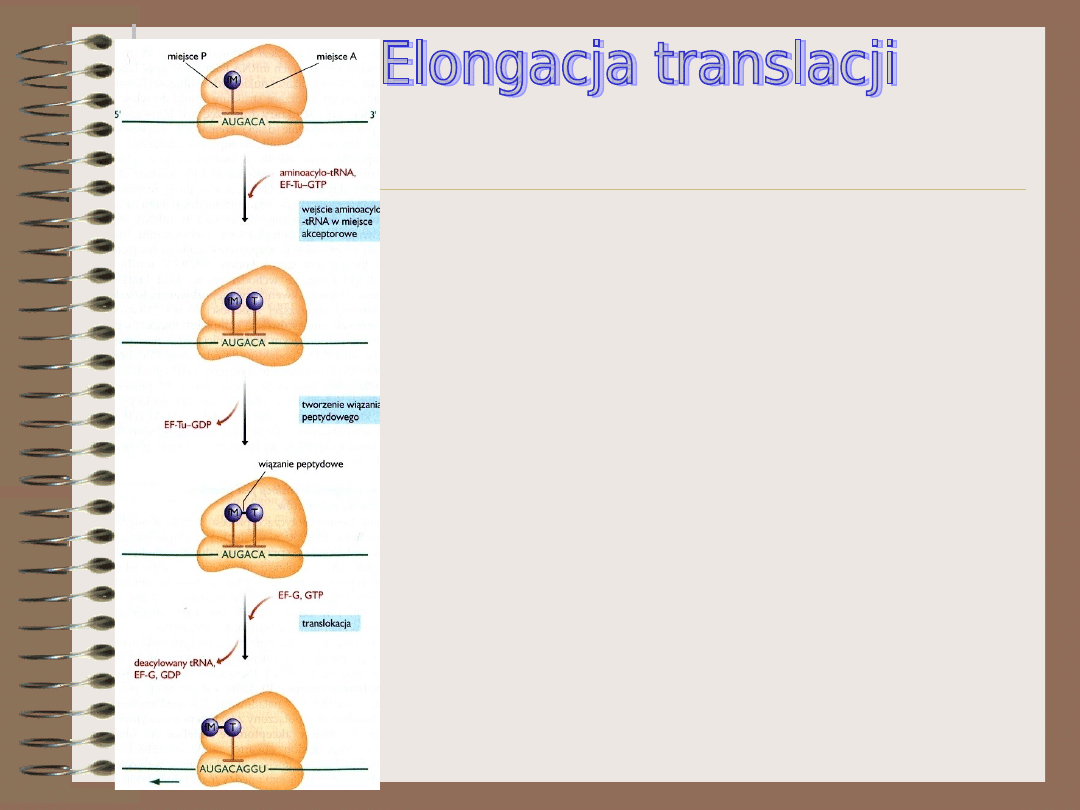
Główne różnice między bakteriami i eukariontami w przebiegu
procesu translacji występują na etapie inicjacji. Etapy
elongacji przebiegają podobnie u pro-i eukaryota.
1.
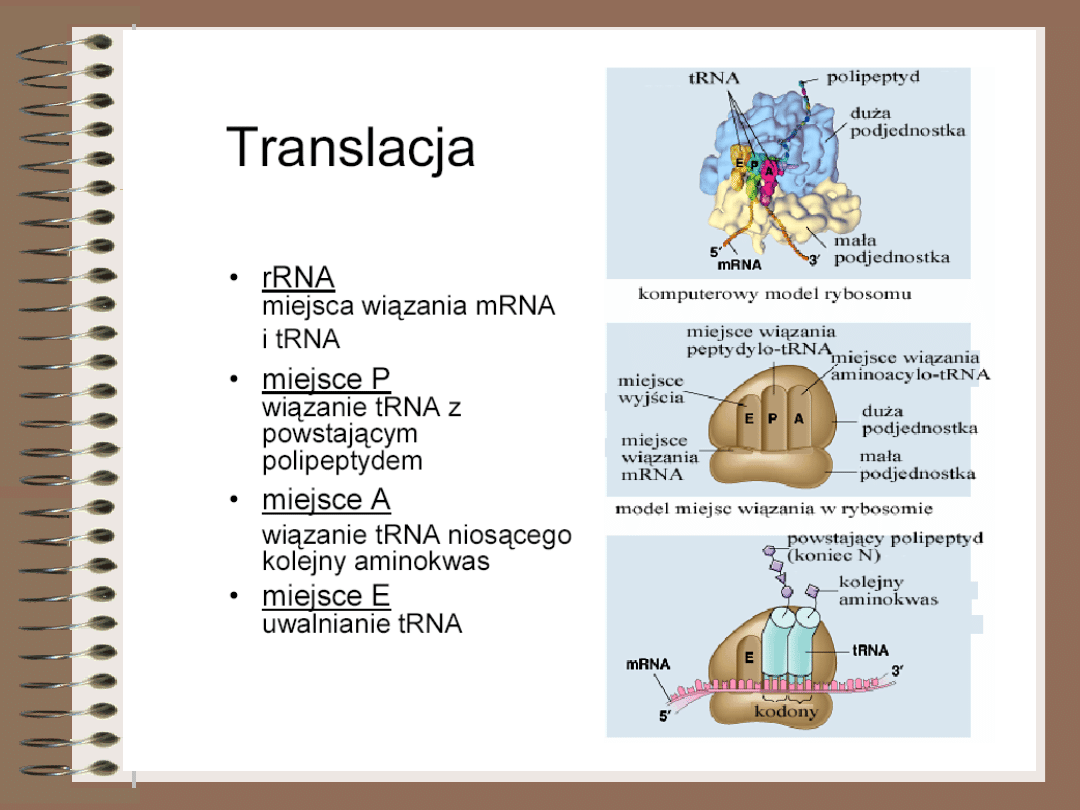
Po dołączeniu dużej podjednostki rybosomu powstają dwa
miejsca, z którymi może się wiązać aminoacylo-tRNA.
Pierwsze z nich – MIEJSCE PEPTYDYLOWE (P) jest już zajęte
przez inicjatorowy tRNA (z formylometioiną) połączony z
kodonem inicjatorowym. Drugie to MIEJSCE AKCEPTOROWE
(A), obejmujące drugi kodon otwartej ramki odczytu.
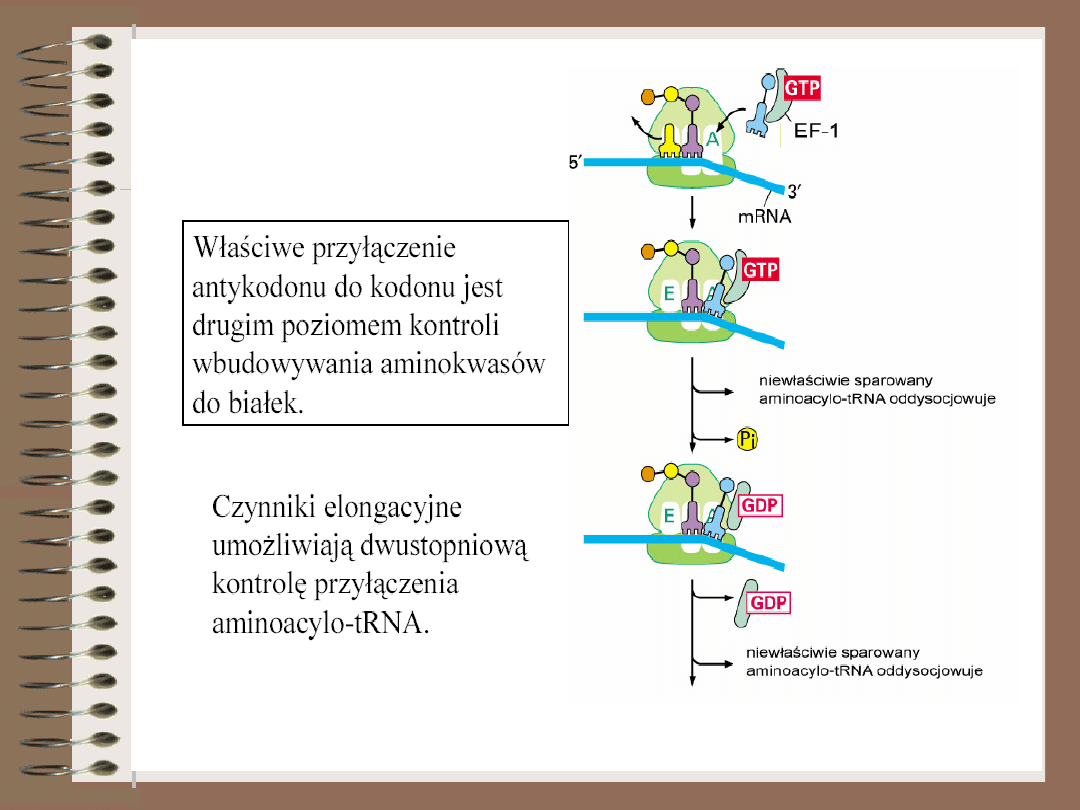
2.
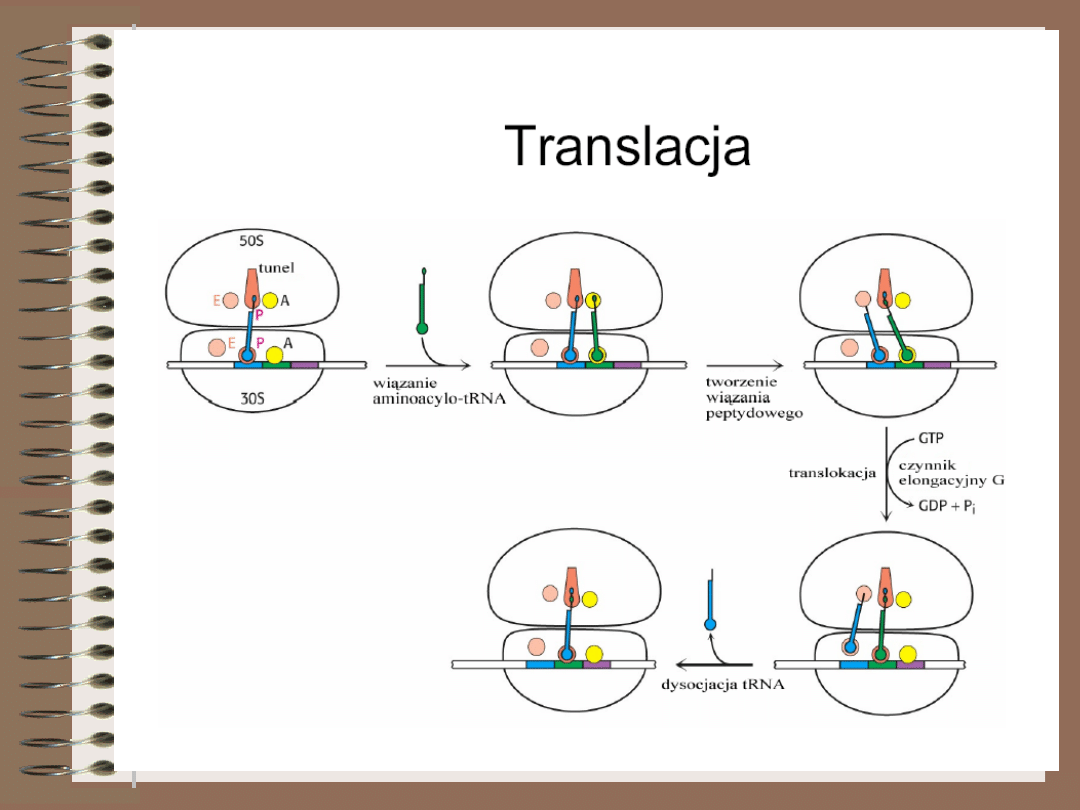
Przy udziale czynnika elongacyjnego EF-Tu miejsce A
zajmuje odpowiednie aminoacylo-tRNA. EF-Tu należy do
grupy białek G, co oznacza, że wiąże on cząsteczkę GTP.
3.
Po wejściu aminoacylo-tRNA w miejsce A następuje
połączenie dwóch aminokwasów za pomocą wiązania
peptydowego. Reakcję katalizuje peptydylotransferaza
(enzym odłącza aminokwas od inicjatorowego tRNA i
tworzy wiązanie peptydowe między tym aminokwasem a
aminokwasem połączonym z drugim tRNA). Reakcja
wymaga energii dostarczonej w wyniku hydrolizy GTP
związanego z czynnikiem EF-Tu. Prowadzi to do inaktywacji
EF-Tu i jego usunięcia z rybosomu. Po czym jest on
regenerowany przez kolejny czynnik EF-Ts. Dipeptyd
odpowiadający pierwszym dwóm kodonom otwartej ramki
odczytu jest teraz połączony z tRNA znajdującym się w w
miejscu A.
4.
Następny etap to translokacja podczas której;
- rybosom przesuwa się o trzy nukleotydy, tak, że następny
kodon wchodzi w miejsce A
- dipeptydylo- tRNA przesuwa się z miejsca A na miejsce P
- deacylowany tRNA jest usuwany z rybosomu
Translokacja wymaga hydrolizy GTP (pośredniczy czynnik EF-G).
Prowadzi ona do opróżnienia miejsca A, które zajmuje
kolejny aminoacylo-tRNA
U E coli
aktywność
peptydylotrans
ferazy
związana jest z
23S rRNA.
Enzym ten jest
więc
przykładem
rybozymu.
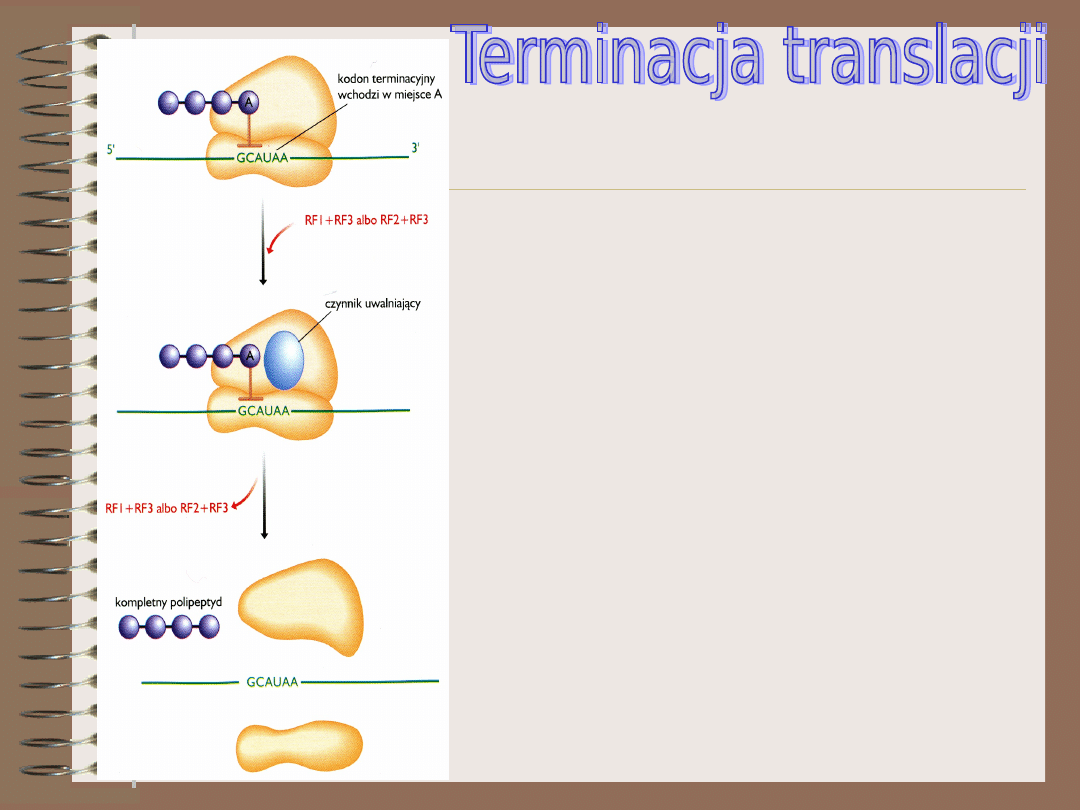
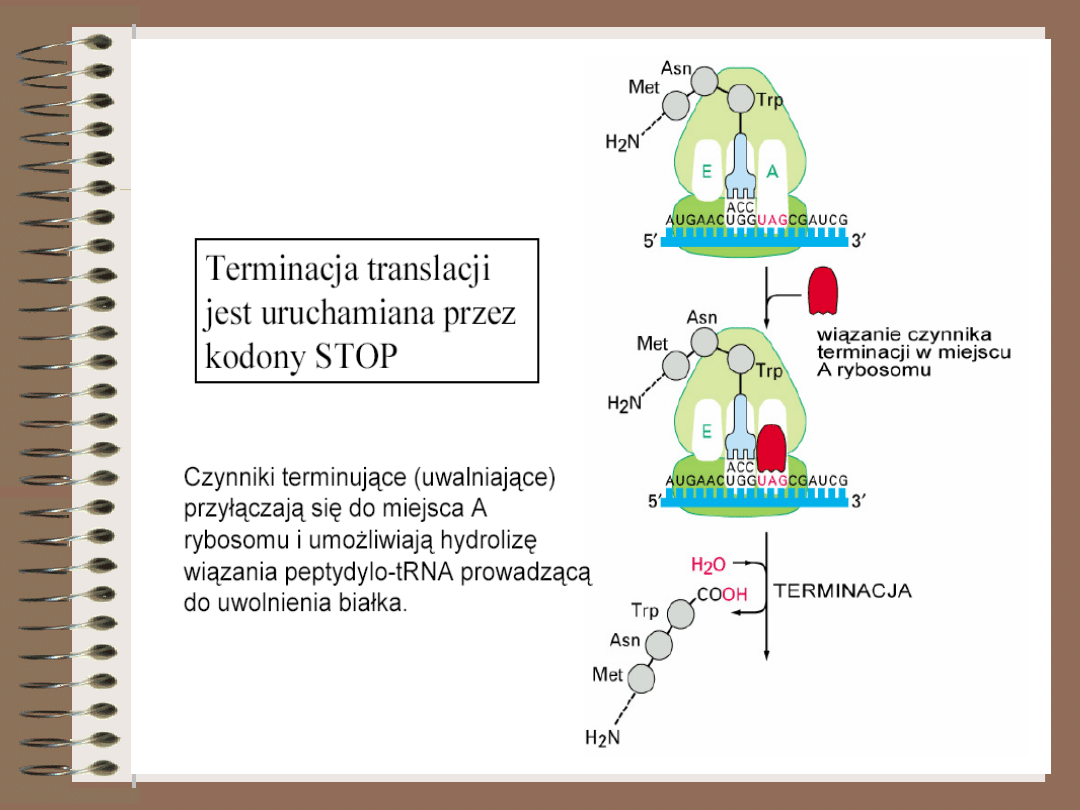
Synteza białka kończy się w momencie
napotkania jednego z trzech kodonów
terminacyjnych. W miejsce A wchodzi teraz
nie cząsteczka tRNA niosąca aminokwas ale
białkowy czynnik uwalniający. Bakterie mają
trzy takie czynniki:
RF-1 – rozpoznający kodony terminacyjne
UAA i UAG
RF-2 – rozpoznający kodony terminacyjne
UAA i UGA
RF-3 – działający jako czynnik pomocniczy
U eukaryota jest tylko jeden czynnik
uwalniający eRF.
U bakterii proces terminacji nie wymaga
energii, natomiast u eukariontów potrzebna
jest energia uwalniana podczas hydrolizy
GTP.
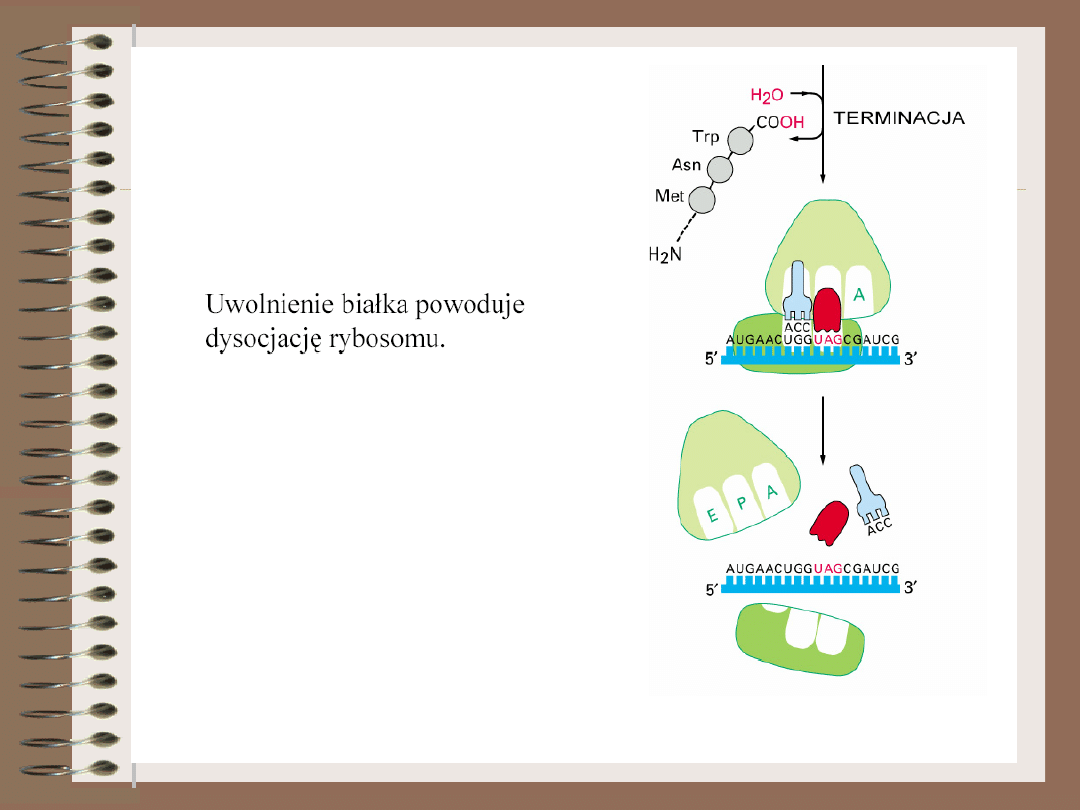
W wyniku terminacji następuje odłączenie
od tRNA kompletnego polipeptydu,
znajdującego się w miejscu P, oraz
rozdysocjowanie kompleksu translacyjnego.
Podjednostki rybosomu stają się częścią puli
cytoplazmatycznej.

Fałdowanie białek- polipeptyd jest nieaktywny, dopóki nie zostanie
sfałdowany w prawidłową strukturę trzeciorzędową
Proteolityczne rozszczepianie białka – cięcie usuwa fragmenty z jednego lub
obu końców. Możliwe jest również cięcie na kilka fragmentów, z których
każdy jest aktywny
Modyfikacje chemiczne
Wycinanie intelin czyli sekwencji „przerywających” w białkach (podobnie jak
intronów w mRNA). Po nim następuje łączenie ekstelin.






Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
Wyszukiwarka
Podobne podstrony:
Biologia - Budowa i synteza białka, szkola, Biologia
06 Podstawy syntezy polimerówid 6357 ppt
Białka wiążące wapń
11 Ch organiczna AMINOKWASY I BIAŁKAid 12388 ppt
BM1 Białka
04) Kod genetyczny i białka (wykład 4)
8a Syntezy prostych aminokwasów
Wykład VIII Synteza układów sekwencyjnych
Wyklad 03 Białka3
BIOSYNTEZA BIAŁKA
Białka 08 06 05
Białka
Białka
02 Metody syntezy organicznej VI s1id 3675
więcej podobnych podstron