
Zastosowanie
spektroskopii NMR
w chemii i biologii

Początki
•
Pierwszym biologicznym zastosowaniem metody NMR
były badania żywych komórek oraz wycinków tkanek
zwierzęcych przeprowadzone przez szwedzkich
uczonych Erika Odeblada i Gunnara Lindströma
•
W następnych latach przeprowadzono wiele badań
relaksacji, dyfuzji i chemicznej wymiany wody w
różnych komórkach i tkankach, spostrzegając duże
zróżnicowanie tych parametrów).
•
Zauważono między innymi interesujące różnice
czasów relaksacji między wycinkami normalnej i
nowotworowej tkanki ludzkiej

Spektroskopia NMR służy do:
• określania i potwierdzania postulowanej
struktury
chemicznej
nieznanych
cząsteczek oraz oznaczania struktury nowo
syntetyzowanych związków.
• dostarcza
informacji
o
przestrzennym
ułożeniu atomów i ich ruchu w ważnych
biologicznie cząsteczkach oraz kompleksach
molekularnych.
• Umożliwia wyjaśnienie mechanizmu działania
enzymów oraz śledzenie przebiegu reakcji
chemicznych.

Zaletą tej metody jest jej
nieinwazyjność!
/Stosowane są impulsy o wysokiej
częstości, lecz niskiej energii rzędu 4x10
-
25
J - znacznie niższej od energii koniecznej
do rozerwania wiązania kowalencyjnego,
która wynosi 10
-17
J. Nie jest to zatem
metoda inwazyjna w przeciwieństwie do
metod wykorzystujących promieniowanie
jonizujące/

Zastosowanie spektroskopii NMR w
biochemii dla małych cząsteczek:
analiza chemiczna
określenie nieznanej struktury chemicznej
nieinwazyjne oznaczanie stężenia
badanie mechanizmów i produktów reakcji
oznaczanie stałych wiązania
oznaczanie stałych szybkości reakcji
określanie konformacji substratów w centrum
aktywnym enzymu

Dla dużych cząsteczek:
badanie zmian konformacyjnych wywołanych
czynnikami zewnętrznymi (pH, temperatura, siła
jonowa, ciśnienie)
badanie zmian konformacyjnych wywołanych
przyłączeniem substratu
badanie struktury centrum aktywnego enzymu,
przestrzennego ułożenia łańcuchów bocznych w
stosunku do substratu, jonizacji grup funkcyjnych
oznaczanie stałych czasowych (czasów korelacji) i
rodzajów ruchu w przypadku zmian konformacyjnych
analiza oddziaływań między cząsteczkami: enzym-
substrat, białko-białko, białko – kw. nukleinowy
oznaczanie struktury drugo- i trzeciorzędowej w
roztworze

Widma
1
H NMR
Wzorzec: tetrametylosilan (TMS)
(CH
3
)
4
Si
Znakowanie izotopowe jest tu niepotrzebne, ze
względu na bardzo dużą czułość detekcji i
wysoką naturalną abundancję jąder
1
H
Z drugiej strony mały zakres przesunięć
chemicznych, obejmujący częstości
rezonansowe cząsteczek biologicznych
utrudnia rozdzielenie poszczególnych
rezonansów

Intensywny sygnał od wody zakłóca obserwowanie
sygnałów od interesujących nas metabolitów.
Na widmie NMR in vivo linie rezonansowe silnie się
nakładają. Aby uzyskać potrzebną informację
można celowo uprościć widmo (edycja
spektralna)
Sekwencja echa spinowego powoduje zmianę
sygnałów w zależności od ich multipletowości
(widma echa zawierają sygnały dodatnie i
składowe ujemne).
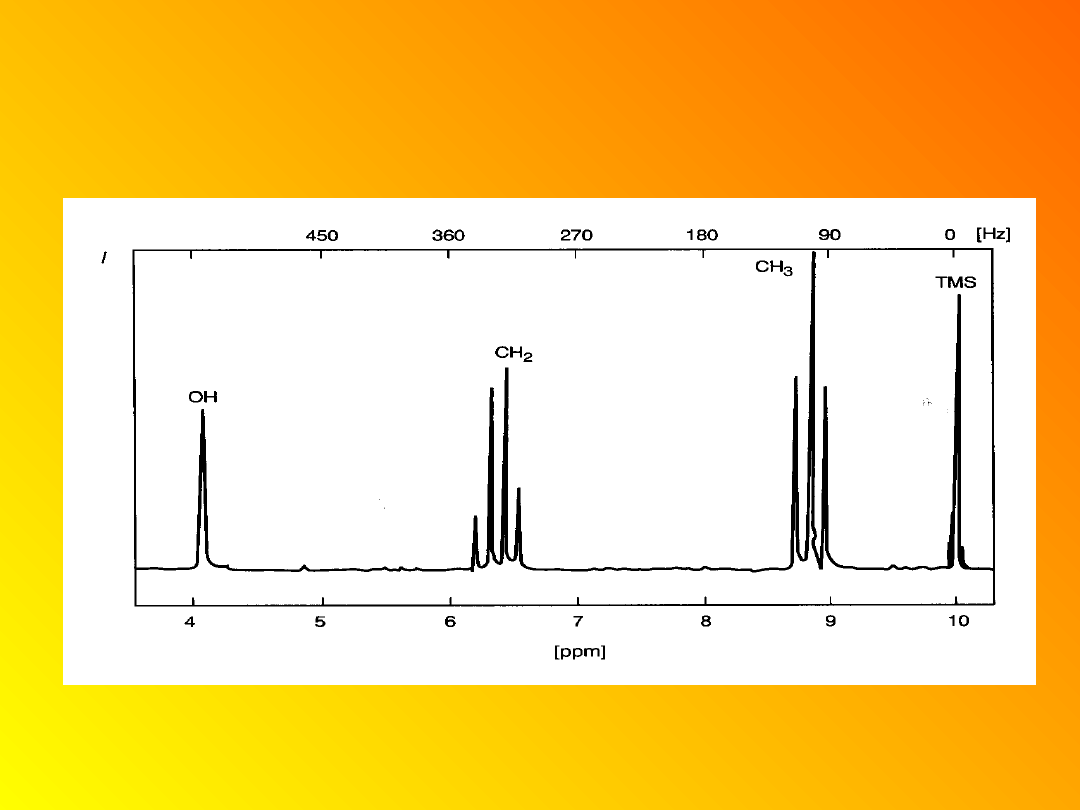
Widmo
1
H etanolu

Rodzaj protonu
Przesuniecie
• Cyklopropanu
0,2
• Pierwszorzędowy RCH3
0,9
• Drugorzędowy
R2CH2 1,3
• Trzeciorzędowy R3CH
1,5
• Winylowy C-C-H
4,6-5,9
• Aromatyczny Ar-H
6-8,5
• Benzylowy
Ar-C -H 2,2-3
• Allilowy
C=C-CH3
1,7
• Fluorków HC-F
4-4,5
• Chlorków HC-Cl
3-4
• Bromków H-Br
2,5-4
• Jodków HC-I 2-4
• Alkoholi HC-OH
3,4-4
• Eterów HC-OR
3,3-4
• Estrów RCOO-CH
3,7-4,1
• Estrów HC-COOR
2-2,2
• Kwasów HC-COOH 2-2,6
• Związków karbonylowych HC-C=O 2-2,7
• Aldehydowy RCHO
9-10
• Wodorotlenowy ROH1-5,5
• Fenolowy
ArOH
4-12
• Enolowy
C=C-OH15-17
• Karboksylowy
RCOOH 10,5-12
• Aminowy
RNH~
1-5
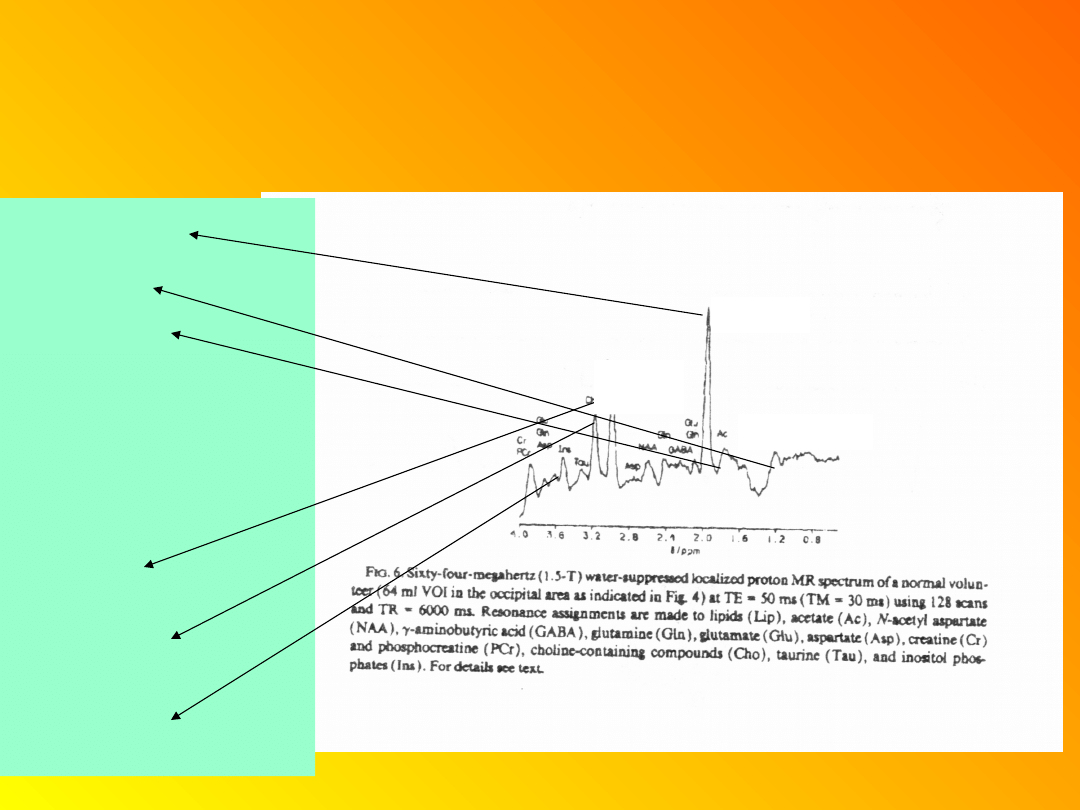
Widmo
1
H mózgu in vivo
NAA N-acetylo-
asparaginian
Lip lipidy
Lac mleczan
GABA kwas
aminomasłowy
GLN glutaminian
Glu glutamina
Asp asparaginian
Cr kreatyna
PCr fosfokreatyna
Cho cholina
Tau tauryna
Ins inozytol
Lip
NAA
Cr
+PCr
Interpretacja widm
1
H NMR
mózgu
Linia
Choroby
NAA
2.02 ppm
Neurony, aksony,
dendryty
„Marker neuronów”
udar, nowotwory,
stwardnienie rozsiane
(MS), choroba
Alzheimera,
stany niedotlenienia
Odwracalne zmiany
natężenia linii NAA:
MS, AIDS, epilepsja
Cholina
3.24 ppm
sygnał grupy
N(CH
3
)
3
Błony komórkowe
(tworzenie i
degradacja),
mielinizacja,
komórki rakowate
Nowotwory,
aktywna demielinizacja
dieta może zmieniać
poziom choliny
Kreatyna,
fosfokreatyna
3.02 ppm
Metabolizm
energetyczny
(ATP)
Choroby wątroby,
nowotwory
Suma PCr+Cr=const
w różnych obszarach
mózgu co daje
możliwość
„ilościowej
standaryzacji linii
NMR”
Mleczan
1.33 ppm
Zaburzony
metabolizm
energetyczny
udar, nowotwory,
choroby
mitochondrialne,
choroba Parkinksona.
Nie obserwowany w
zdrowym mózgu
Mioinozytol
3.56 ppm
Funkcja nieznana
Nowotwory (marker
komórek glejaka),
choroba Alzheimera,
demielinizacja
Glutamina,
glutaminian
Aminokwasy
Choroby wątroby,
syndromy Retta i Reye
Trudno rozseparować
obie linie

Rezonans fluorowy
19
F
Wzorzec CFCl
3
(C
6
F
6
)
Szeroki zakres przesunięć chemicznych od 100 do
–300 ppm
Selektywna metoda badania wielu leków takich,
jak: cytostatyki (5FU), leki antydepresyjne (Prozac),
nowoczesne leki antybakteryjne fluorochinolony
(Ciprinol), anestetyki (sevofluran, halotan),
pomocnicze:
- tomografia PET (fluorodezoksyglukoza)
- w badaniach pO2 -ciśnienia parcjalnego tlenu
- w tkankach- perfluorowęglan czyli sztuczna krew

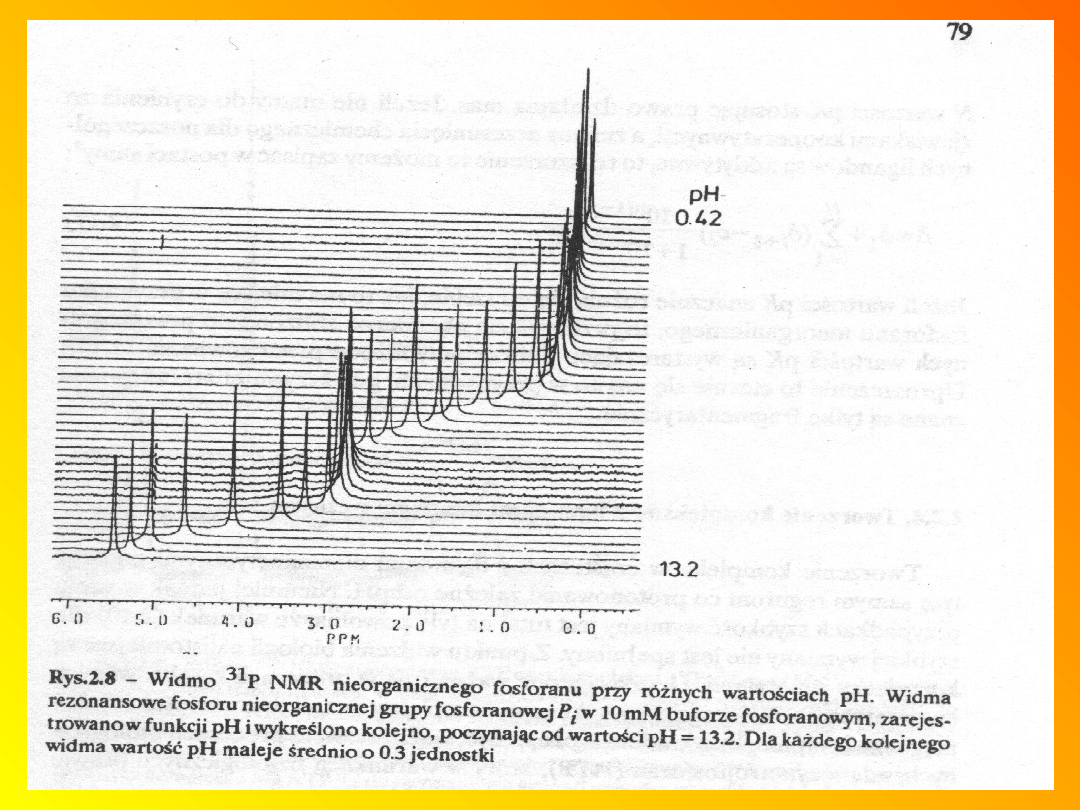
Rezonans fosforowy
31
P
• Wzorzec H
3
PO
4
,
• zakres przesunięć chemicznych od 230 do –
200 ppm,
• Stosunkowo małe problemy z nakładaniem
się linii rezonansowych, gdyż w większości
badań występuje jednocześnie jedynie kilka
cząsteczek zawierających fosfor.
• charakterystyczne widmo z liniami: ATP
(ADP), fosfokreatyny (PCr), fosforu
nieorganicznego (P
i
), fosfoestrów (PME i PDE)
oraz glicerylofosfocholiny (GPC) i
glicerylofosfoetanolaminy (GPE),
• pomiar pH in vivo,
• ocena wewnątrzkomórkowego stężenia
poziomu jonów magnezu.

Rezonans węgla
13
C
• Wzorzec TMS,
• zakres przesunięć chemicznych od 240 do
–10 ppm
• obserwacja sygnałów: glukozy,
13
C –
glikogenu, glutamatu, tłuszczów,
mleczanów, mioinozytolu.
• choroby: nowotwory prostaty, choroba
Alzheimera, cukrzyca.

Rezonans azotu
15
N
• Naturalna abundancja wynosi 0.37%
• Wyniki w postaci dobrze rozdzielonych
widm
• Badanie wbudowywania się atomów azotu
do komórek bakterii na pożywce NH
4
Cl (po
pewnym czasie na zewnątrz komórek
można zarejestrować Ala znakowaną
15
N,
która została wydalona przez bakterie w
przeciwieństwie do innych aminokwasów).
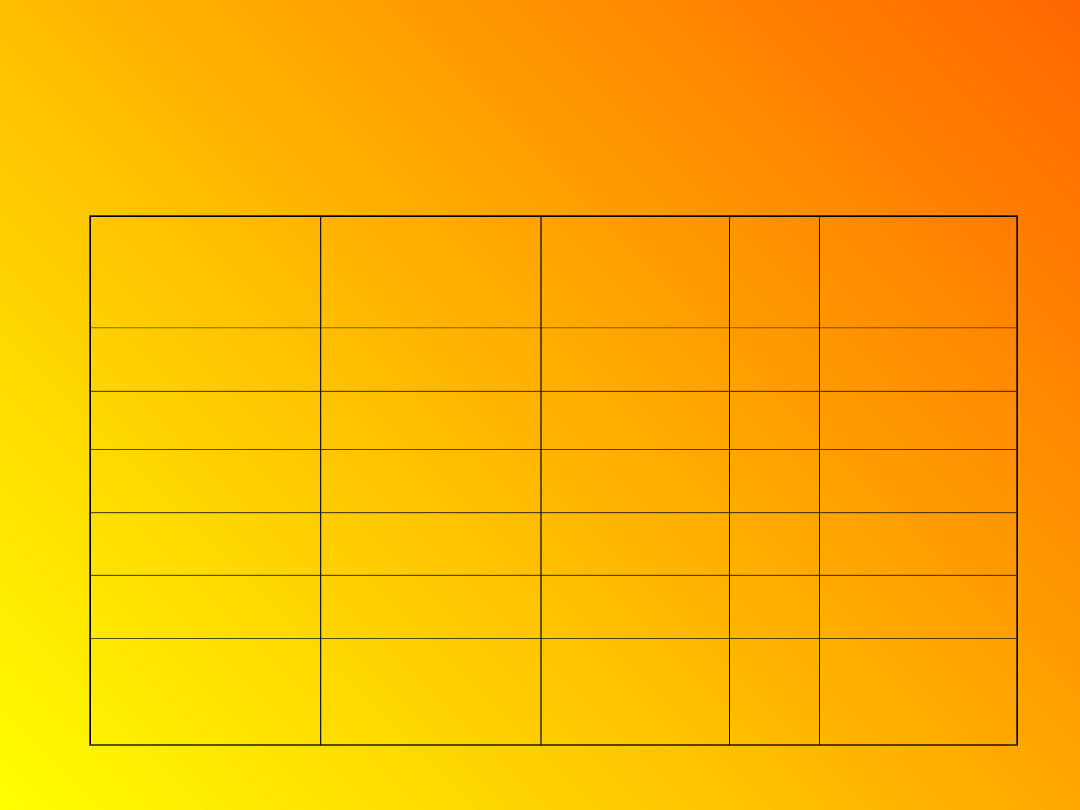
Pierwiastki rezonansowe
identifikowane
w układach biologicznych in vivo
Pierwiastek
Częstość
rezonansu (MHz)
w polu 1.41 T
Czułość
względem
1
H
Spin
jądra
Naturalna
abundancja
%
1
H
60
1
1/2
99.98
31
P
24,34
6.63.10
-2
1/2
100
19
F
56,45
0.83
1/2
100
13
C
15,09
1.59.10
-2
1/2
1.10
7
Li
23,33
0.29
3/2
92.58
15
N
23
Na
6,08
15,6
1.04.10
-3
9.25.10
-2
1/2
3/2
0.37
100

Spektroskopia NMR białek
Za pomocą widma
1
H NMR może być
śledzone
przejście
białka
od
konformacji natywnej do całkowicie
zdenaturowanej.
•
Gdy białko znajduje się w stanie
pofałdowania widmo jest silnie rozbudowane
i
nie
ma
możliwości
precyzyjnego
przewidzenia kształtu białka, nawet gdy
znana jest struktura przestrzenna cząsteczki.
• W stanie denaturacji widmo NMR jest w
przybliżeniu odbiciem składu aminokwasów.
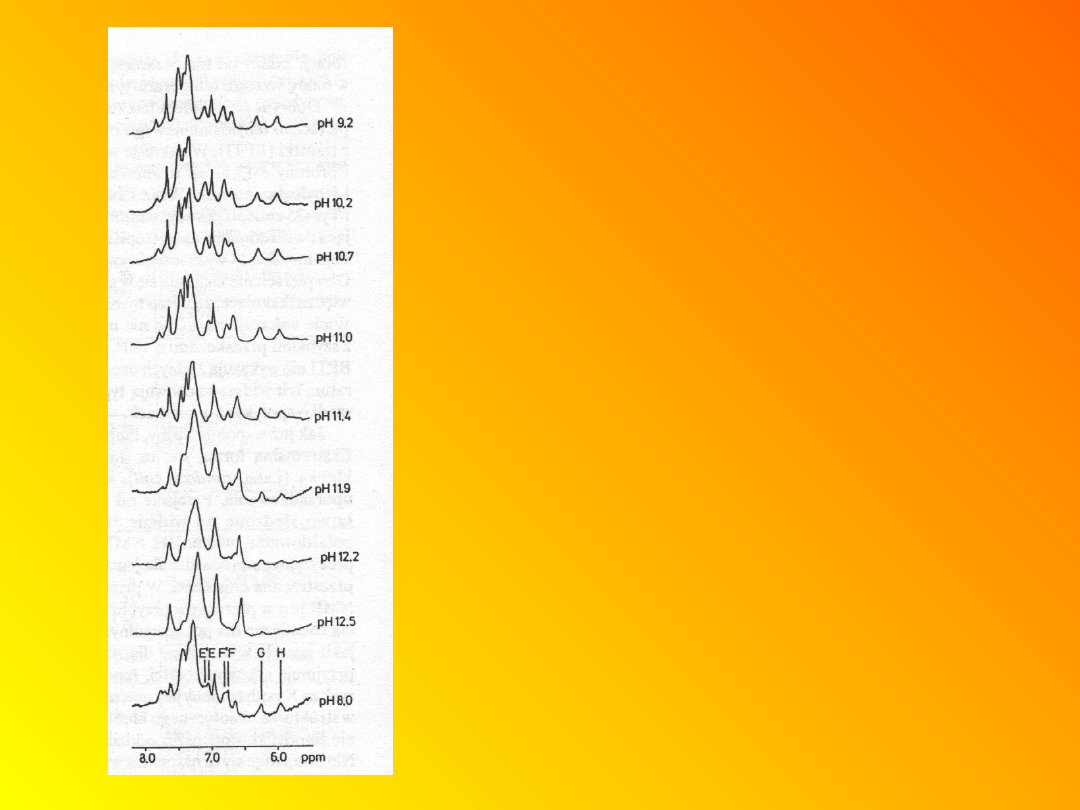
Denaturacja czynnika
III lac wraz ze
wzrostem pH. E, E’,
F, F’ to linie
rezonansowe Tyr w
cząsteczce
natywnej i
częściowo
zdenaturowanej.
Proces
denatuturacji jest
szczególnie
widoczny dla dwóch
linii rezonansowych
G i H.

Za pomocą spektroskopii NMR można
śledzić zmiany konformacyjne enzymów
po przyłączeniu substratu lub efektora
allosterycznego.
/np. obserwowane są znaczne zmiany widma NMR
ludzkiej kinazy adenylowej po przyłączeniu do niej
analogu substratu./
Aminokwasem, który bierze udział w
wielu procesach katalitycznych, a jego
sygnały są łatwe do rozróżnienia nawet
dla dużych białek, jest His. Fosforylacja
His prowadzi do typowych zmian jej
częstości rezonansowych.
Jeżeli do badanej próbki zostanie
wprowadzona np. deuterowana Leu, to
jądra deuteru nie dadzą sygnałów na
widmie
1
H NMR. W związku z tym
wszystkie linie rezonansowe, których
brak
w
porównaniu
z
widmem
początkowym, pochodzą od jąder
wodoru Leu. W podobny sposób można
uzyskać informacje porównując dwa
białka o zbliżonej sekwencji. Brakujące
linie
rezonansowe
pochodzą
od
zamienionych aminokwasów.
Spektroskopia NMR kwasów
nukleinowych
Opiera się w zasadzie na tych samych regułach co NMR
białek.
Określenie struktury przestrzennej jest tu łatwiejsze,
ponieważ parowanie zasad redukuje znacznie liczbę
możliwych struktur przestrzennych.
Problemem może być stosunkowo silne, zależne od
konformacji przesunięcie chemiczne wynikające z faktu,
że części zasadowe nukleotydów są pierścieniami
aromatycznymi, co prowadzi do silnego wzbogacenia
struktury widma NMR.
Główny łańcuch DNA i RNA składa się z diestrów kw.
fosforowego, dlatego spektroskopia
31
P NMR okazała
się równie przydatna w ich badaniu jak spektroskopia
1
H
NMR.

Spektroskopia NMR
polisacharydów
Za pomocą NMR można określić
strukturę monosacharydu, np. w
przypadku heksozy: krzesełko lub
łódeczka.
Dwuwymiarowa
spektroskopia
NMR
umożliwia
określenie
czy
poszczególne
monosacharydy połączone są ze
sobą
wiązaniem
α
czy
β-
glikozydowym.

Spektroskop
NMR
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
Wyszukiwarka
Podobne podstrony:
Biomedyczne zastosowanie spektroskopii NMR
Biomedyczne zastosowanie spektroskopii NMR
9 Zastosowanie norm żywienia i wyżywienia w pracy dietetyka
Zastosowanie SEM
Spektroskopia NMR
Metodologia SPSS Zastosowanie komputerów Golański Standaryzacja
Metodologia SPSS Zastosowanie komputerów Golański Anowa założenia
Metodologia SPSS Zastosowanie komputerów Brzezicka Rotkiewicz Podstawy statystyki
Metodologia SPSS Zastosowanie komputerów Brzezicka Rotkiewicz Testy zależne
Zastosowanie metody problemowej w nauczaniu
Metodologia SPSS Zastosowanie komputerów Golański Statystyki
Laser i jego zastosowanie
Metodologia SPSS Zastosowanie komputerów Brzezicka Rotkiewicz Regresja
A A Praktyczne zastosowane myślenia logistycznego
Zastosowanie światła w medycynie i kosmetologii
Widmo NMR
02 czujniki, systematyka, zastosowania
więcej podobnych podstron