
Metody
wykrywania
mutacji
Izolacja DNA
Izolacja DNA polega na oddzieleniu
DNA od innych struktur komórkowych
oraz od cząsteczek RNA i białek
związanych z DNA.
Istnieje kilka metod izolacji DNA. O ich
wyborze decyduje rodzaj materiału
biologicznego oraz przeznaczenie
uzyskanego DNA.
Izolacja DNA - etapy
Liza komórek
Denaturacja i hydroliza białek
Usunięcie białek
Zagęszczenie DNA
Izolacja RNA - metody
Izolacja specyficznego RNA w wyniku
frakcjonowania całkowitego RNA
komórki
Bezpośrednia izolacja specyficznego
RNA ograniczona do kom syntezujących
dany RNA w zwiększonej ilości
Izolacja poli(A)RNA
Izolacja RNA z polirybosomów
Izolacja całkowitego RNA komórki, a
następnie uzyskanie określonego RNA w
formie DNA w procesie odwrotnej
transkrypcji
Izolacja RNA - różnice
RNA jest bardziej narażony na
degradację niż DNA
Konieczność usunięcia DNA podczas
izolacji
rRNA, tRNA oraz RNA
niskocząsteczkowy w komórkach
eukariotycznych stanowią łącznie ok.
80-98% RNA
Elektroforeza
Elektroforeza preparatywna –
umożliwia izolację z żelu tej części
preparatu, która jest niezbędna do
dalszych prac
Elektroforeza analityczna – służy do
charakterystyki badanego preparatu
Elektroforeza kapilarna – stosowana
podczas analizy DNA celem ustalenia
sekwencji pojedynczego łańcucha DNA
DGGE/TGGE – elektroforeza na
żelach z gradientem
czynnika
denaturującego/temperatury
Metoda polega na elektroforezie
dwuniciowego DNA w żelu o
wzrastającym stężeniu czynnika
denaturującego. Pod wpływem
denaturacji fragmenty DNA ulegają
dysocjacji na pojedyncze nici. W
momencie rozdzielenia na pojedyncze
nici przemieszczanie DNA w żelu zostaje
gwałtownie przyhamowane. Moment
denaturacji DNA zmutowanego jest inny
niż DNA prawidłowego.
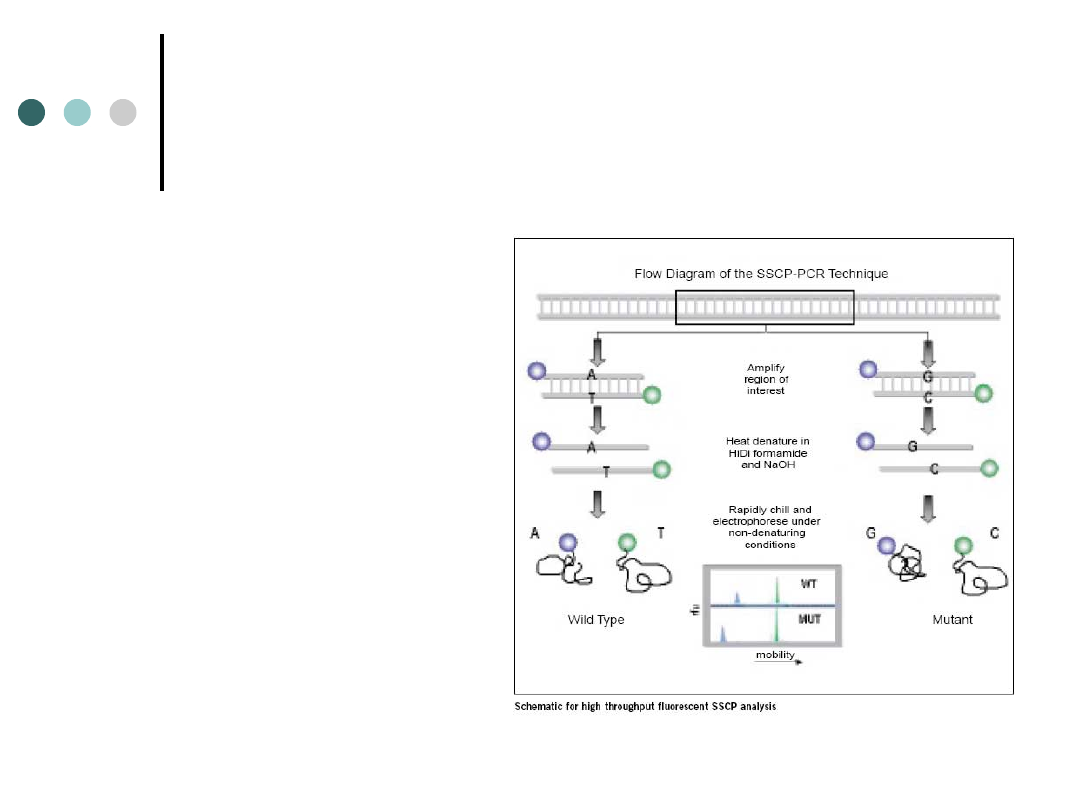
SSCP – single strand
conformation
polymorphism
Metoda służąca do
badania polimorfizmu
konformacyjnego
jednoniciowych
cząsteczek DNA, czyli
różnic powstałych w
wyniku tworzenia
odmiennych struktur
trójwymiarowych przez
jednoniciowe cząsteczki
DNA, normalne i
zmutowane tego
samego fragmentu
łańcucha
polinukleotydowego –
dzięki wykorzystaniu
elektroforezy.
Techniki hybrydyzacyjne
analizy kwasów
nukleinowych
Hybrydyzacja – tworzenie podwójnej
helisy między komplementarnymi
nićmi DNA lub RNA pochodzących z
różnych źródeł
Sondy molekularne i
znakowanie
SONDY
Fragmenty
jednoniciowego DNA
i RNA poszukiwanego
genu sklonowanego
wcześniej
Syntetyczny
oligonukleotyd o
sekwencji
komplementarnej do
odcinka DNA
Sonda utworzona
dzięki amplifikacji
metodą PCR
ZNAKOWANIE
Radioaktywne
Nieradioaktywne (np
metodami
fluorescencyjnymi)
Immunochemiczne,
cytochemiczne
enzymatyczne
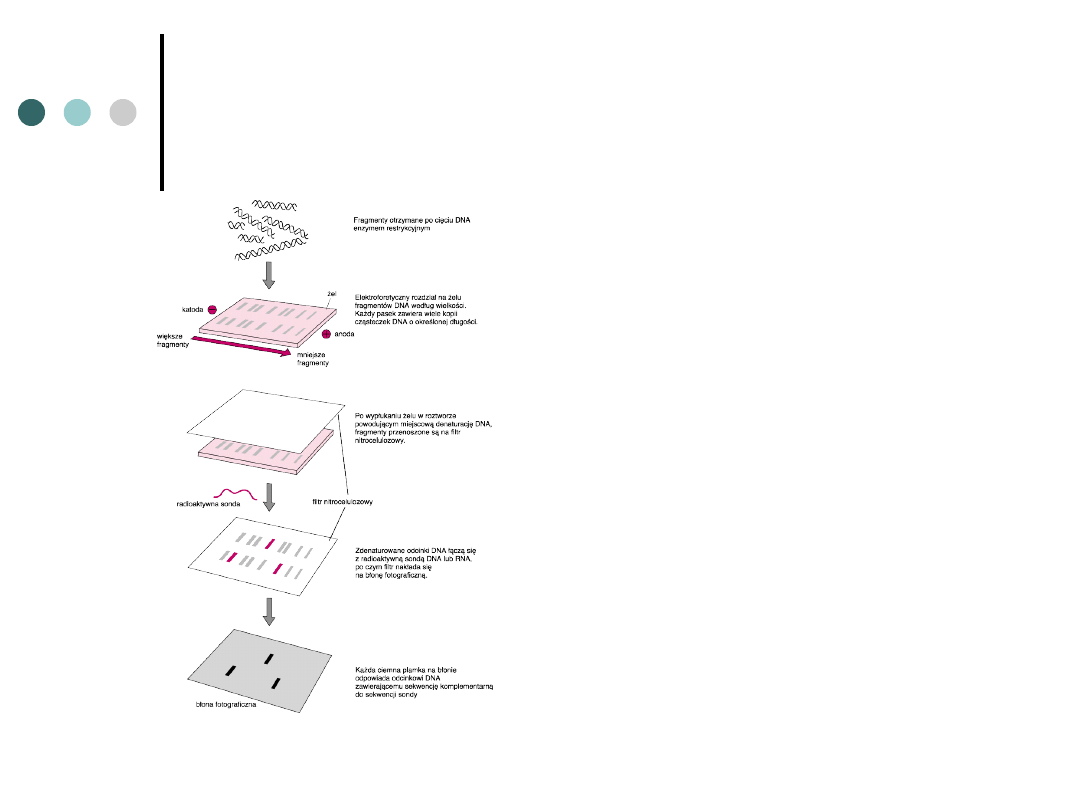
Metoda Southerna –
Southern blot
Umożliwia
zidentyfikowanie
fragmentów
restrykcyjnych
poprzez użycie
elektroforezy
oraz sond
molekularnych
Metoda Southerna –
zastosowanie i
ograniczenia
Metoda b. często stosowana w
diagnostyce DNA, użyteczna w
wykrywaniu mutacji punktowych
Konieczna jest znaczna ilość DNA
Wynik można uzyskać po 7-14 dniach
Ekspozycja na znaczniki radioaktywne
Hybrydyzacja typu
northern
Wykorzystywana do analizy RNA i
procesu transkrypcji poszczególnych
genów. Dzięki niej można ustalić, które
geny ulegają transkrypcji w badanych
tkankach lub na danym etapie rozwoju
organizmu. Ponadto umożliwia
poznanie długości RNA badanych
genów a także intensywność, z jaką są
one transkrybowane w różnych
układach biologicznych.
Hybrydyzacja typu
western
Rozdziela zdenaturowane białka według
ich mas oraz niezdenaturowane według
ich struktury przestrzennej, za pomocą
elektroforezy w żelu
poliakrylamidowym. Później białka są
przenoszone na membranę (zwykle
nitrocelulozową). Obecność
odpowiednich białek jest sprawdzana za
pomocą barwników takich jak Ponceau
S, czerń amidowa czy Coomassie
Brilliant Blue lub za pomocą przeciwciał
przyłączających się do ich epitopów.
RFLP - Restriction
Fragments Length
Polymorphism
Tą techniką wykrywa się różnice
pomiędzy rozmiarami fragmentów
DNA pociętych restryktazami. Różnice
długości fragmentów DNA powstają w
wyniku mutacji, które tworzą lub
eliminują miejsca rozpoznawane przez
te enzymy. Sekwencje polimorficzne
RFLP, które wykrywa się tą metodą, są
używane jako znaczniki zarówno w
mapach fizycznych jak i genetycznych.
ASO - Hybrydyzacja
Oligonukleotydu
Specyficznego względem
allelu
Stosuje się sondy molekularne – jedną
komplementarną do sekwencji
prawidłowej, drugą do zmutowanej.
pomiędzy sondą a badanym DNA
dochodzi do hybrydyzacji. Obecność
sygnału po hybrydyzacji z sondą
komplementarną do sekwencji
prawidłowej oznacza brak mutacji, sygnał
otrzymany po hybrydyzacji z sondą
komplementarną do odcinka
zmutowanego oznacza obecność mutacji.
Hybrydyzacja z użyciem
mikromacierzy
Naniesienie na odpowiedni nośnik w sposób
zaplanowany i uporządkowany licznych
wzorcowych sekwencji, będących
podłożem do hybrydyzacji materiału
badanego.
Analiza transkryptominczna (RNA)
Badanie DNA
Badanie sekwencji genów
Analiza jednonukleotydowych
polimorfizmów DNA typu SNP
Badanie oddziaływań DNA z białkami
Analiza heterodupleksów
Polega na wychwyceniu powstającego w
wyniku mutacji braku parowania
pomiędzy pojedynczymi nukleotydami
w komplementarnych (dwuniciowych)
fragmentach kwasów nukleinowych.
Struktury wytwarzane w zależności od
typu mutacji: „pęcherzyki”
(substytucje), zgięcia nici DNA,
wybrzuszenia jednostronne (insercje
lub delecje).

FISH- fluorescent in situ
hybridization
Stosuje się fluorescencyjnie
wyznakowane sondy DNA,
metoda ta pozwala na
wykrycie mutacji i jej
lokalizację.
Można stosować
jednocześnie wiele sond
wyznakowanych różnymi
związkami
fluorescencyjnymi.
Zastosowanie do
wykrywania aneuploidów,
mikrodelecji, duplikacji,
utraty heterozygotyczności
(nowotwory).
Enzymatyczna - metoda
Sangera
Na jednoniciowej matrycy syntetyzuje się in
vitro DNA. Synteza przebiega od
radioaktywnego, oligonukleotydowego startera
komplementarnego do 3` końca matrycy.
Reakcję wykonuje się równolegle w czterech
probówkach, w których oprócz kompletu
deoksytrifosforanów nukleotydów (dATP , dCTP ,
dTTP , dGTP) dodana jest niewielka ilość jednego
z nich w postaci dideoksytrifosforanu
nukleotydu, co uniemożliwia kontynuowanie
reakcji w momencie włączenia takiego
nukleotydu w syntetyzowany łańcuch, ponieważ
brakuje wtedy grupy hydroksylowej na 3` koncu.
Chemiczna - metoda
Maxama-Gilberta
Polega na degradacji wyznakowanych na 5` końcu
cząsteczek DNA związkami chemicznymi
przecinającymi specyficznie wiązania
fosfodiestrowe za nukleotydem odpowiadającym
określonej zasadzie azotowej. Wynikiem reakcji jest
zbiór fragmentów DNA o różnej długości co
spowodowane jest takim doborem warunków , że w
poszczególnych cząsteczkach przecinane jest tylko
jedno lub dwa wiązania. Fragmenty z czterech
niezależnych reakcji rozdziela się elektroforetycznie
po czym poddaje autoradiografii. Prążki na
autoradiogramie odpowiadają fragmentom DNA
posiadającym na końcu 5` znakowany fosfor a na
końcu 3` określoną zasadę
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
Wyszukiwarka
Podobne podstrony:
Metody wykrywania mutacji
GMO metody wykrywania 2
hodowlane i niehodowlane metody wykrywania drobnoustrojów
Metody wykrywania antygenu D
Metody wykrywania zagrozenia przedsiebiorstwa upadkiem w2
DSC Metody wykrywania zafałszowań
Główne metody wykrywania włamań są następujące
metody wykrywania włamań są następujące
Metody wykrywania i identyfikacji oraz oceny lekowrazliwści drobnoustrojów
Metody Wykrywania Skażeń Promieniotwórczych, szkoła
WYKRYWANIE MUTACJI 2
Metody wykrywania mechanizmów oporności bakterii na antybiotyki nowa (1)
Instrumentalne metody wykrywania skazy korkowej w winach
GMO metody wykrywania 2
hodowlane i niehodowlane metody wykrywania drobnoustrojów
więcej podobnych podstron