
T E C H N I KA O D C Z Y T Y WA N I A S E K W E N C J I ,
C Z Y L I K O L E J N O Ś C I I R O D Z A J U PA R
N U K L E O T Y D Ó W W D N A
N A P O D S TAW I E S E K W E N C J I
N U K L E O T Y D Ó W M O Ż L I W E J E S T
P R Z E W I D Z E N I E K O L E J N O Ś C I
A M I N O K WA S Ó W W B I A Ł K U, K T Ó R E J E S T
K O D O WA N E P R Z E Z D A N Y G E N
Sekwencjonowanie DNA
Miłosz Lorek gr. 14
Metody sekwencjonowania DNA
Metoda
terminacji
łańcucha
Sangera
Metoda
degradacji
chemicznej
Maxama-
Gilberta
Metody
rozwojowe
Pirosekwencjonowa
nie

Metoda Sangera (terminacji łańcucha)
Jest oparta na zdolności do rozdziału jednoniciowych
cząsteczek DNA, różniących się długością zaledwie o jeden
nukleotyd, przez elektroforeze w żelu poliakryloamidowym
Matrycą jest jednoniciowe DNA. Do niej dołącza się starter
(tzw. primer), syntetycznie wyprodukowany. Jego koniec 5' jest
oznakowany radioaktywnie, przez co oprócz zapoczątkowania
syntezy wyznacza na matrycy sekwencjonowany region
Reakcje syntezy nici katalizuje polimeraza DNA i wymaga, jako
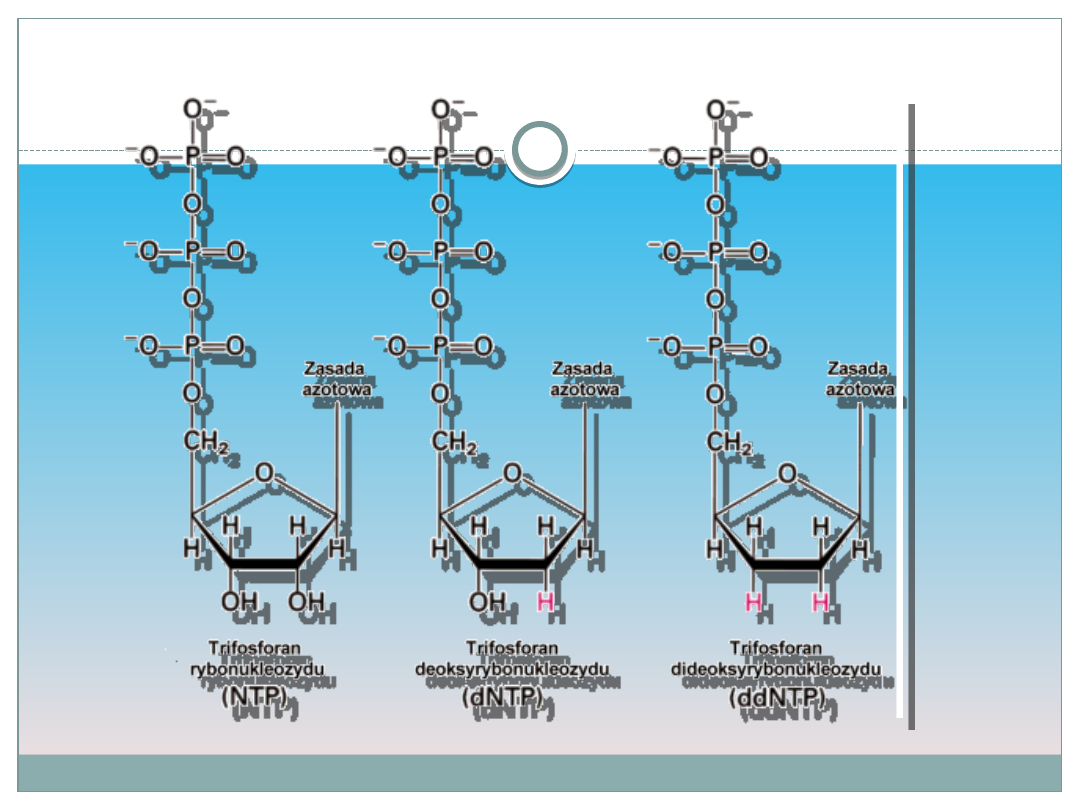
substratów, czterech trifosforanów deoksyrybonukleozydów
(dATP, dCTP, dGTP, dTTP).
Mała ilość jednego z czterech trifosforanów
dideoksyrybonukleozydu (ddNTP, ddATP, ddCTP, ddGTP,
ddTTP). Każdy z tych dideoksynukleozydów jest wyznakowany
innym znacznikiem fluorescencyjnym.
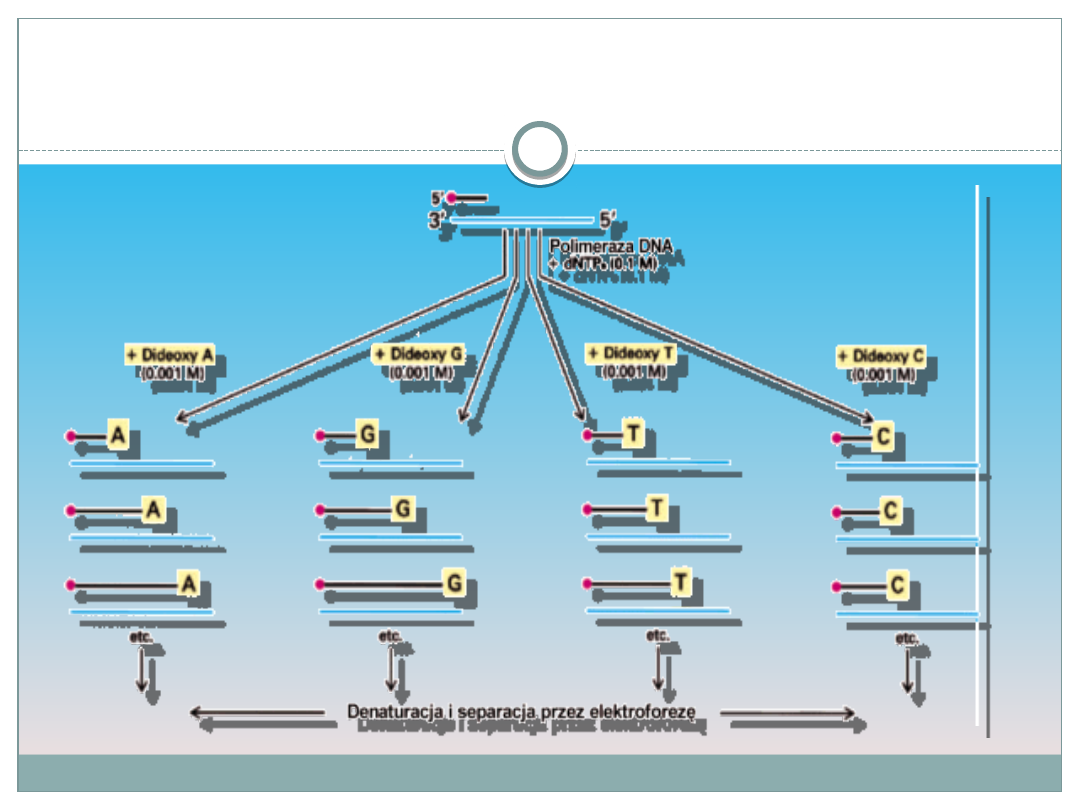
Synteza zostaje zahamowana losowo
Powstaje mieszania fragmentów o różnej
długości
Następnie należy zidentyfikować dideoksynukleotyd z końca każdej
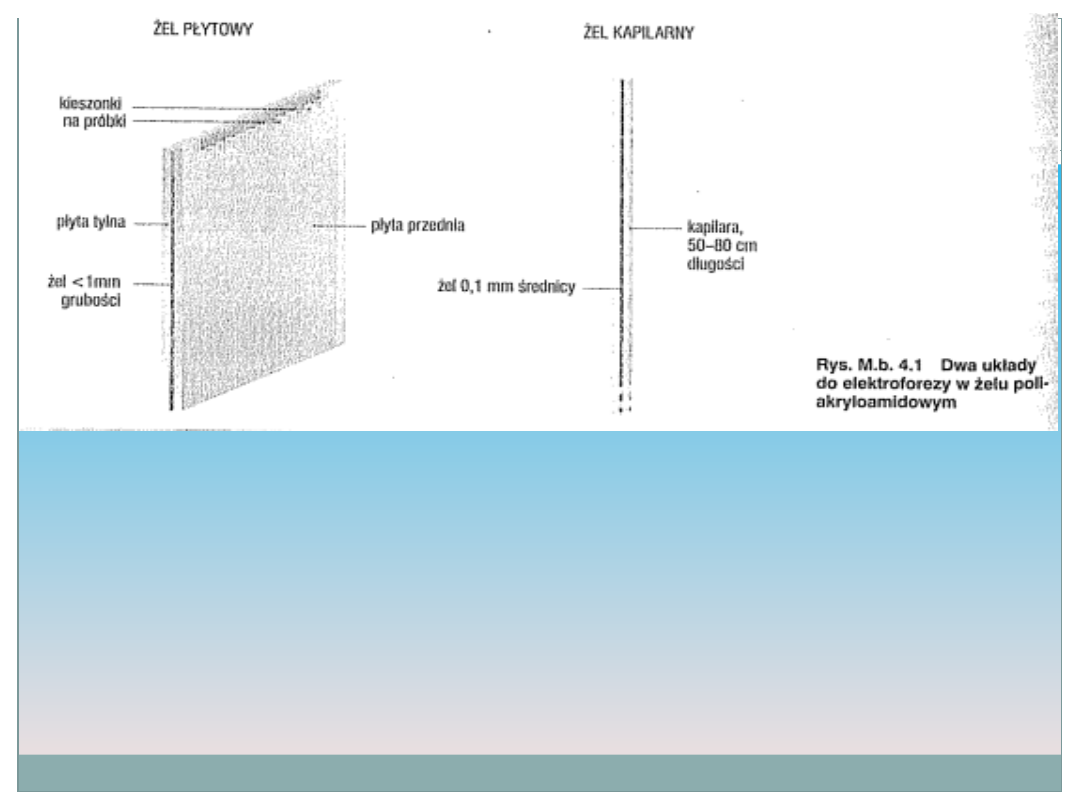
cząsteczki. Do tego właśnie służy żel poliakryloamidowy. W celu
rozdzielenia cząsteczek ze względu na ich długość mieszaninę DNA
nanosi się do kieszonki płytowej żelu poliakryloamidowego albo do
przewodu systemu żelu kapilarnego i prowadzi się elektroforezę. Po
rozdziale cząsteczki przesuwają się przed detektorem fluorescencji,
który rozróżnia znaczniki dołączone do dideoksynukleotydów.

Matryca w postaci jednoniciowego
DNA
DNA sklonowany na wektorze plazmidowym –
Jednoniciowy DNA uzyskuje się przez
denaturacje alkaliczną lub termiczną.
DNA sklonowany na wektorze M13 – Genom
bakteriofaga M13 to jednoniciowa cząsteczka
DNA, która po infekcji E. Coli zamieniana jest na
formę replikacyjną. Po osiągnięciu optymalnej
liczby komórki bakterii wydzielają nowe cząstki
faga M13. Wydzielane cząsteczki DNA składają
się z wektora i włączonego do niego DNA.
DNA sklonowany na fagmidzie.

Enzymy stosowane w sekwencjonowaniu:
Duża procesywność – polimeraza używana w
sekwencjonowaniu musi się charakteryzować
dużą procesywnością, aby nie uległa
oddysocjowaniu przed włączeniem
dideoksyrybonukleotydu.
Brak aktywności egzonukleazy 5’->3’
Brak aktywności egzonukleazy 3’->5’
Te z kolei mogą prowadzić do skrócenia
sekwencji, uniemożliwiając odczytanie
prawidłowej sekwencji.

Enzymy sztucznie zmodyfikowane
Fragment Klenowa – pochodna polimerazy
DNA I E. Coli z usuniętą aktywnością
egzonukleazy 5’->3’. Charakteryzuje się
jednak stosunkowo małą procesywnością
Sekwenaza – zmodyfikowana wersja
polimerazy DNA kodowanej przez
bakteriofaga T7. Charakteryzuje się dużą
procesywnością i brakiem aktywności
egzonukleazy
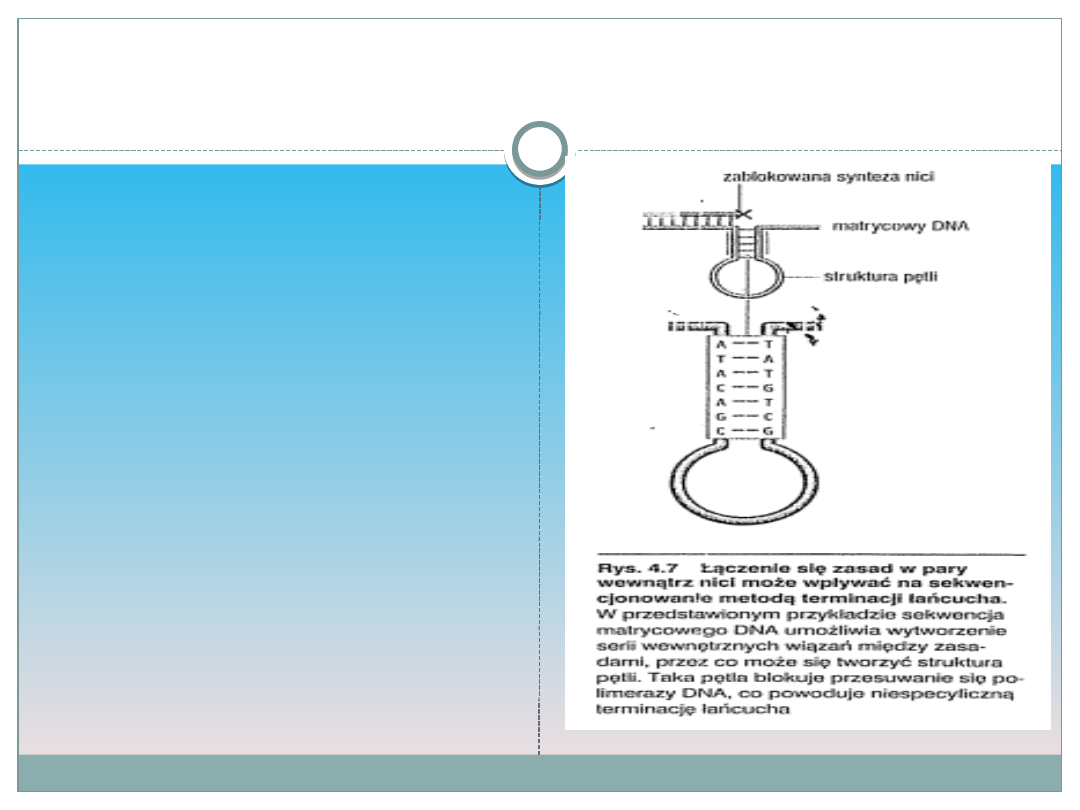
Wewnętrzne parowanie zasad
Jednym z ograniczeń
sekwencjonowania
metodą terminacji
łańcucha są trudności
w uzyskaniu
dokładnej sekwencji,
jeżeli matrycowy DNA
jest zdolny do
wewnętrznego
parowania zasad.

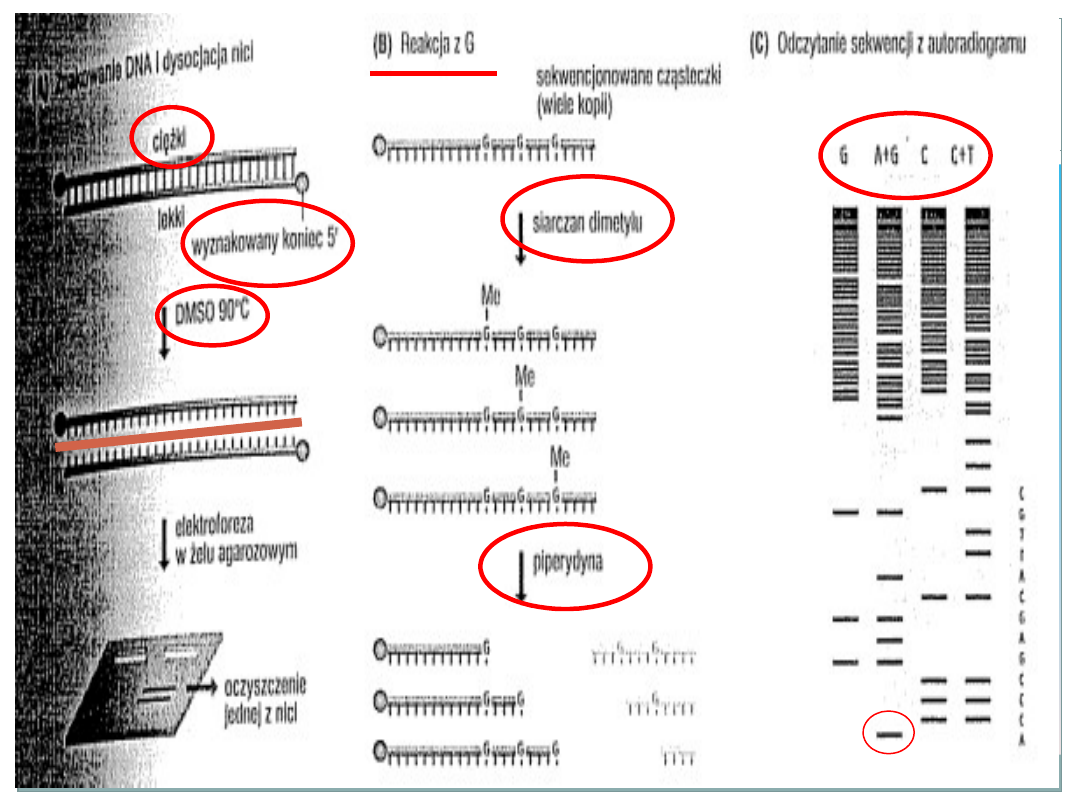
Metoda Maxama- Gilberta (degradacji chemicznej)
Alternatywna metoda, gdy pojawią się problemy
związane z wewnętrznym parowaniem zasad.
Sekwencje ustala się na podstawie długości
cząsteczek, w których znany jest ostatni nukleotyd.
Cząsteczki w metodzie degradacji chemicznej są
wytwarzane przez działanie odczynnikami, które
przecinają specyficznie w miejscu określonego
nukleotydu. Oznacza to, że muszą być
prowadzone, co najmniej cztery oddzielne reakcje
sekwencjonowania, po jednej dla każdego
nukleotydu.

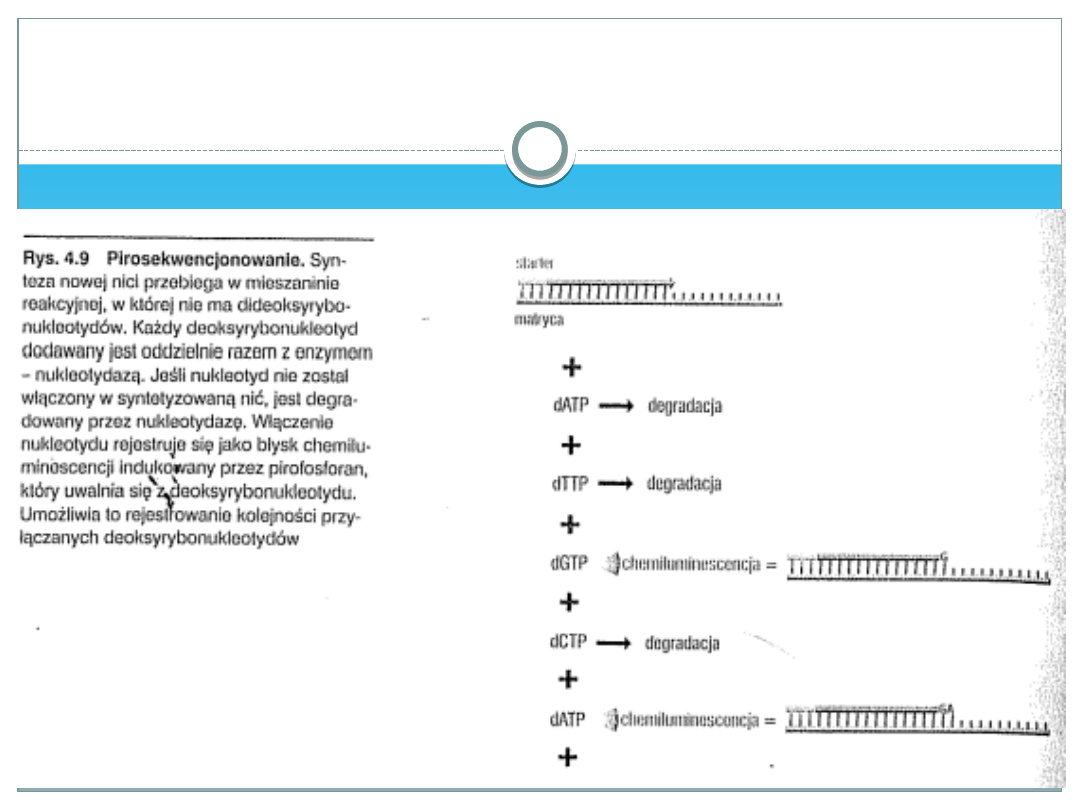
Pirosekwencjonowanie
Nie wymaga elektroforezy ani innego rozdzielania
fragmentów, przez co jest dużo szybsza zarówno od
sekwencjonowania metodą terminacji łańcucha jak i
degradacji chemicznej.
W jednym eksperymencie udaje się odczytać
zaledwie kilkadziesiąt par zasad
Matryca jest kopiowana w prosty sposób bez
dodawania dideoksynukleotydów do mieszaniny
reakcyjnej
Rejestrowana jest kolejność wbudowywanych
nukleotydów, co pozwala bezpośrednio odczytać
sekwencje powstającej nici.

Pirosekwencjonowanie
Dołączeniu nowego nukleotydu do końca
syntetyzowanej nici towarzyszy uwolnienie
cząsteczki pirofosforanu, której przekształcenie za
pomocą sulfurylazy (adenylilotransferazy
siarczanowej) daje błysk będący wynikiem
chemiluminescencji. Do mieszaniny reakcyjnej każdy
nukleotyd dodawany jest osobno jeden po drugim.
Ponadto w mieszaninie reakcyjnej znajduje się
nukleotydaza, która degraduje dany dNTP, jeśli nie
zostanie on wbudowany w łańcuch polinukleotydów.
Tępo uzyskiwania sekwencji jest 100 razy szybsze
niż z zastosowaniem metody terminacji łańcucha.
Pirosekwencjonowanie

Praktyczne wykorzystanie
Metody sekwencjonowania pozwoliły na szybki postęp w
dziedzinie sekwencjonowania całych genomów. W 1995 poznano
pierwsze genomy bakterii (Haemophilus influenzae i Mycoplasma
genitalium), w 1997 genom pierwszego eukarionta - drożdży
piekarskich (Saccharomyces cerevisiae), w 1998 genom
pierwszego organizmu wielokomórkowego nicienia
Caenorhabditis elegans, w 2000 genom muszki owocowej
(Drosophila melanogaster), w 2000 genom rzodkiewnika
(Arabidopsis thaliana).
W 1999 poznano kolejność nukleotydów w ludzkim chromosomie
22, w 2000 w chromosomie 21. W lutym 2001 niezależnie Human
Genome Project i Celera Genomics opublikowali sekwencję ok. 3
miliardów nukleotydów genomu ludzkiego (tj. ok. 90% genomu
ludzkiego).

Dziękuje za uwagę.
Document Outline
- Slide 1
- Metody sekwencjonowania DNA
- Metoda Sangera (terminacji łańcucha)
- Slide 4
- Slide 5
- Slide 6
- Matryca w postaci jednoniciowego DNA
- Enzymy stosowane w sekwencjonowaniu:
- Enzymy sztucznie zmodyfikowane
- Wewnętrzne parowanie zasad
- Metoda Maxama- Gilberta (degradacji chemicznej)
- Slide 12
- Pirosekwencjonowanie
- Pirosekwencjonowanie
- Pirosekwencjonowanie
- Praktyczne wykorzystanie
- Slide 17
Wyszukiwarka
Podobne podstrony:
wiczenie Nr Sekwencjonowanie DNA USTALENIE SEKWENCJI NUKLEOTYDO
Lekcja 6 sekwencjonowanie DNA sekwencje genomów
Sekwencjonowanie DNA prezentacja
BIOTECHNOLOGIA 10d sekwencjonowanie DNA
W4 Metody powielania sekwencji DNA
Sekwencjonowanie DNA
SEKWENCJONOWANIE DNA 2
Sekwencjonowanie DNA
Nowosad K, Stępień P, Musiał P Sekwencjonowanie DNA Przegląd metod [2015]
Wykład 10x, Genom- obejmuje sekwencje kodujące i niekodujące DNA
Replikacja DNA i choroby związane
W5 sII PCR i sekwencjonowanie cz 2
Elektroforeza DNA komórkowego BioAut1, BioAut2 i Ch1
DNA Eng2
Metoda symultaniczno sekwencyjna
Wykład VIII Synteza układów sekwencyjnych
więcej podobnych podstron