
SEKWENCJONOWANIE
DNA
Mateusz Lorenc gr.8a

•
Sekwencjonowanie DNA jest to technika
odczytywania sekwencji par
nukleotydowych w cząsteczce DNA.
Cele sekwencjonowania:
•
dostarcza wielu cennych informacji o
strukturze i funkcji genów,
•
znajomość pełnej sekwencji DNA
badanych organizmów umożliwia
zrozumienie molekularnych mechanizmów
ich funkcjonowania i ewolucji
•
umożliwia szukanie mutacji
Metody sekwencjonowania
DNA:
•
Metoda Maxama i Gilberta (1977)
•
Metoda Sangera - metoda terminacji wydłużania
łańcucha tzw. metoda dideoksy (1981)
•
Automatyczne sekwencjonowanie z
wykorzystaniem dideoksyrybonukleotydów
znakowanych fluorescencyjnie(1987)
•
Pirosekwencjonowania – sekwencjonowanie w
czasie rzeczywistym
•
Stosowanie polimerazy
•
Składanie z wielu sekwencji wyjściowej sekwencji
chromosomów.
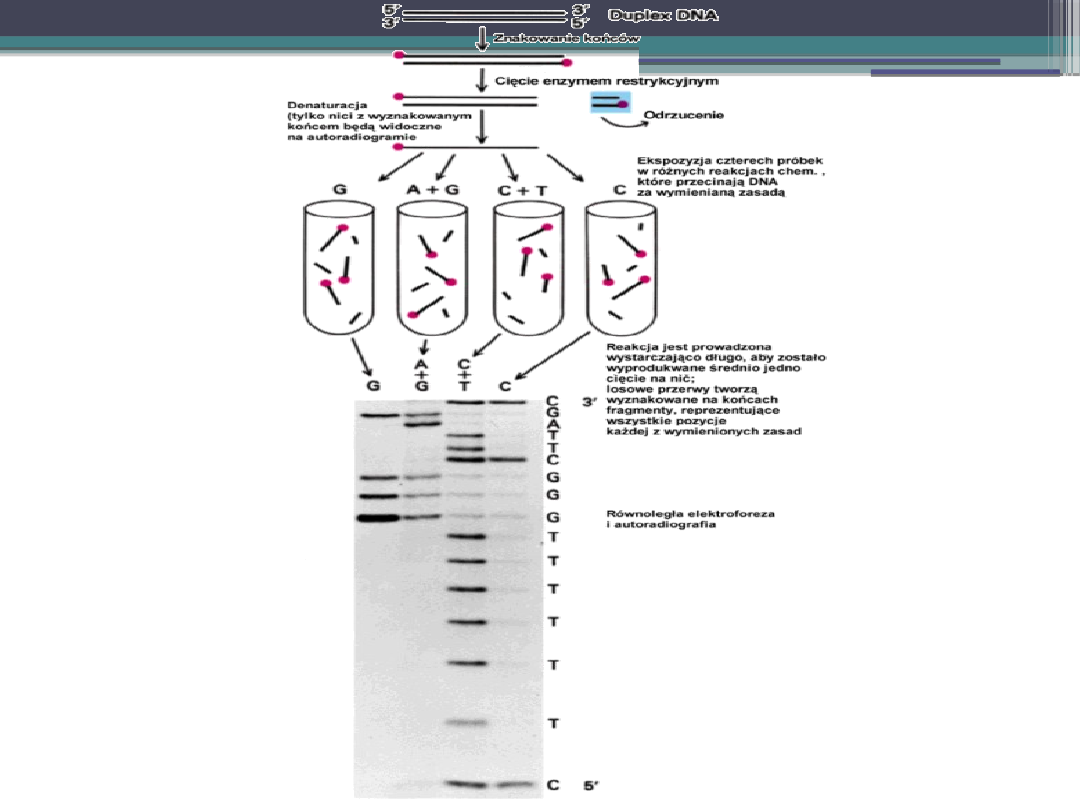
Metoda Maxama i Gilberta - metoda
chemicznego sekwencjonowania
Polega na użyciu związków chemicznych do
specyficznego rozczepienia DNA.
1.
Fragment DNA znakuje się na jednym końcu:
- Radioaktywnym fosforanem na końcu 5`
-Radioaktywnym nukleotydem na końcu 3`
2.
Znakowany DNA poddawany jest reakcjom
zrywania w miejscach występowania
określonych zasad
3.
Uzyskane fragmenty rozdziela się przy
użyciu elektroforezy, a następnie poddaje
autoradiografii.
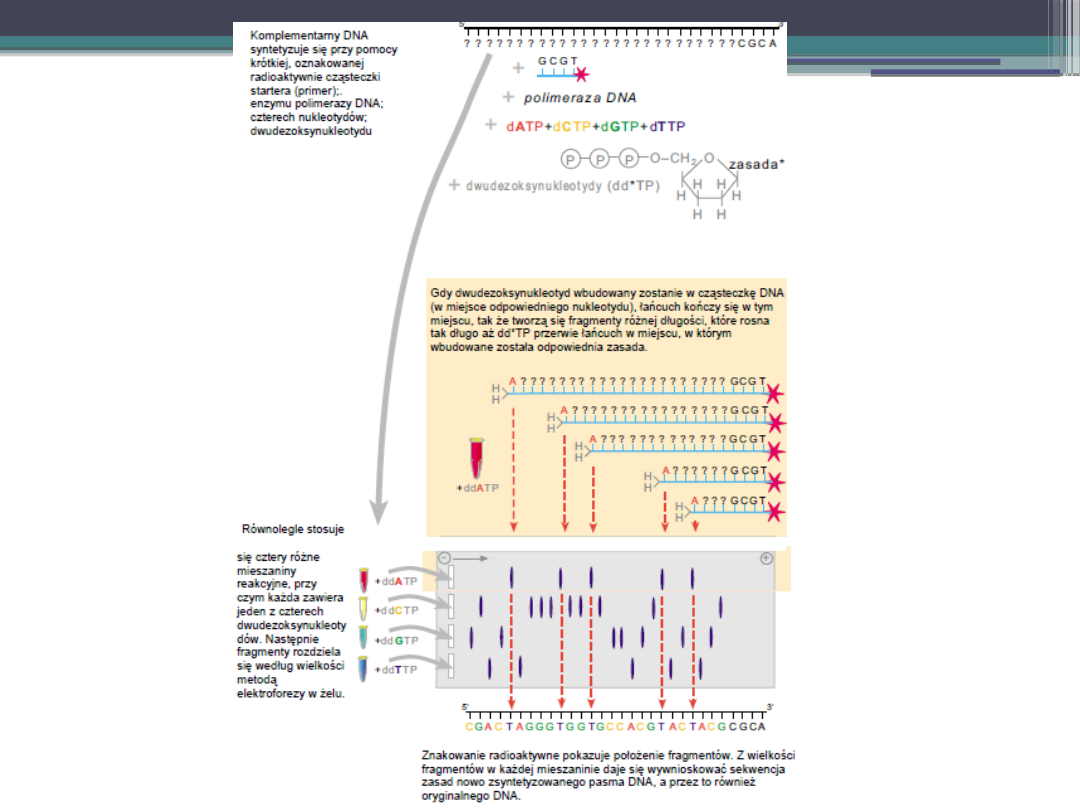
Metoda Sangera
Polega na przedwczesnej terminacji syntezy
DNA wynikającej z przyłączenia
dideoksynukleotydem.
Przebeg:
- Przygotowanie 4 mieszanin reakcyjnych
zawierających: kopie jednoniciowego DNA
do sekwencjonowania, polimeraze DNA,
radioaktywnie znakowane startery, 4 rodzaje
deoksyrybonukleoidów( dATP, dCTP, dGTP,
dTTP)
w każdej z mieszanin znajduje się również
jeden z dideoksyrybonukleoidów(ddATP,
ddCTP, ddGTP, ddTTP) .
Metoda Sangera
Przebieg w mieszaninie z ddATP:
-
W każdym miejscy gdzie występuje adenina,
rosnąca nić przypadkowo wbudowuje ddATP, co
hamuje jej wydłużanie.
-
W mieszaninie powstaje wiele fragmętów DNA
różnej wielkości. W każdym fragmęcie w
miejscu gdzie znajduje się ddATP znajdowałaby
się adenina – w normalnie syntetyzowanej nici.
-
Radioaktywne fragmęty każdej z reakcji
denaturuje się, następnie dokonuje się
elektroferezy powstałych cząsteczek, gdzie
każda mieszanina tworzy osobny prążek w żelu.
Automatyczne sekwencjonowanie z
wykorzystaniem
dideoksyrybonukleotydów
znakowanych fluorescencyjnie.
W systemie tym stosuje się nukleotydy
dideoxy znakowane czterema różnymi
znacznikami fluorescencyjnymi. Detektor
fluorescencji w trakcie ekektroferezy
ustala zakończenia cząsteczek ( czy
kończy się ona na A, T, C czy G)
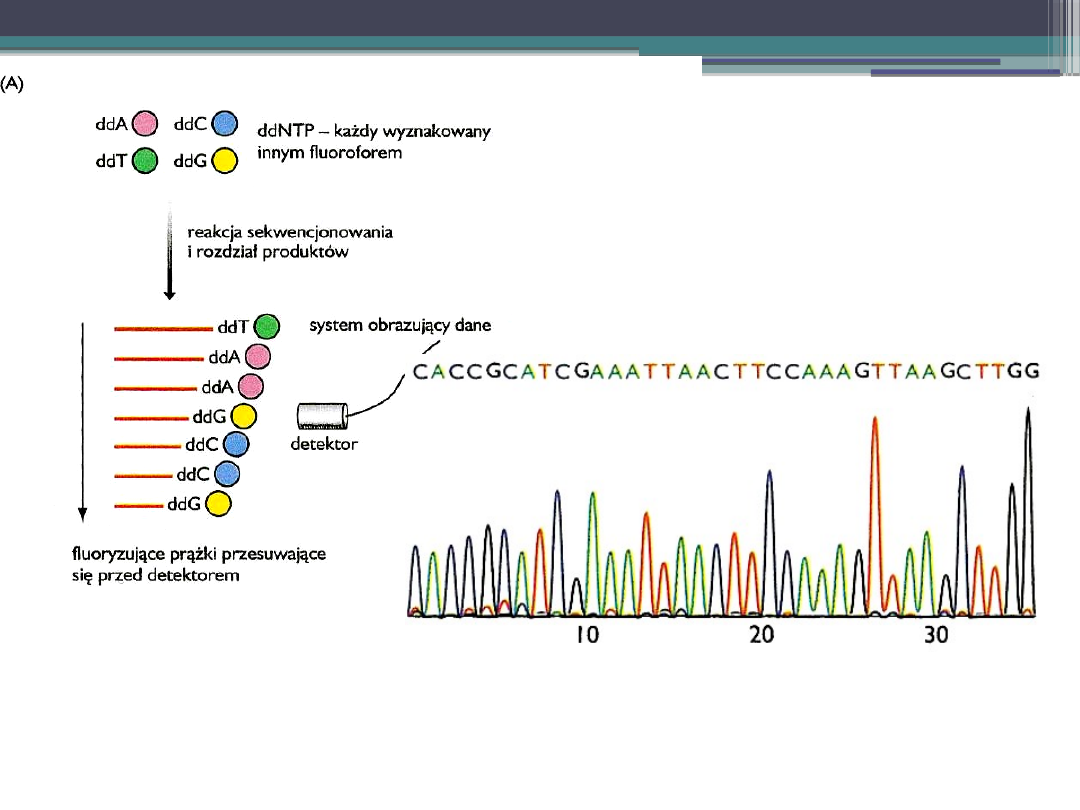
Wydruk z sekwenatora, sekwencja w
postaci serii szczytów, z których
każdy
odpowiada innemu nukleotydowi.
W przedstawionym przykładzie A-
zielony, C-niebieski, G- czarny, T-
czerwony.
Pirosekwencjonowanie – w czasie
rzeczywistym
Metoda ta jest dużo szybsza od metody terminacji łańcucha ze
względu na to, że nie wymaga elektroferezy ani innego
rozdzielania fragmętów.
Przebieg:
-
Matryca jest kopiowana w prosty sposób bez dodawania
dideoksynukleotydów do mieszaniny.
-
Dołączeniu nowego nukleotydu towarzyszy uwolnienie
cząsteczki pirofosforanu, której przekształcenie za pomocą
fosfatazy daje błysk będący wynikiem chemiluminescencji.
-
Każdy nukleotyd dodawany jest osobno, a jeśli dany nukleotyd
nie zostanie wbudowany to jest degenerowany przez
nukloetydaze.
-
W czasie syntezy rejstruje się kojeność wbudowywanych
nukleotydów co pozwala na bezpośredni odczyt.

Pirosekwencjonowanie
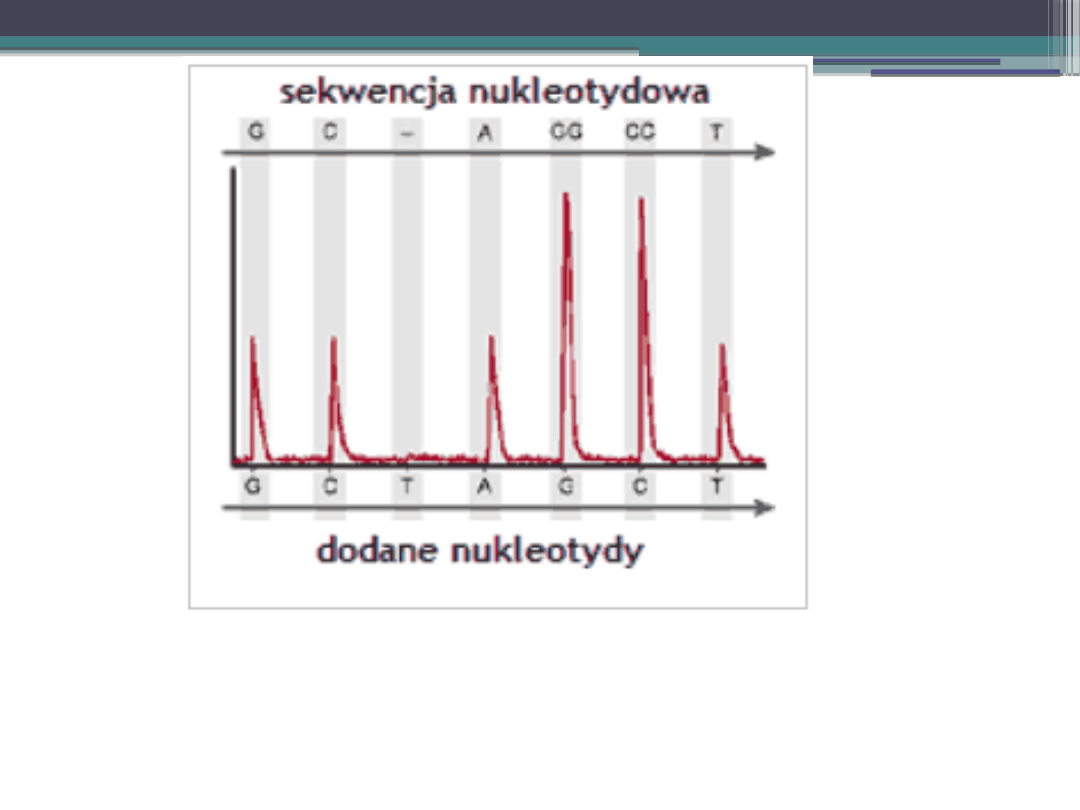
Im wyższy jest pik świetlny, tym większa ilość
nukleotydów została wbudowana kolejno po sobie. Brak
sygnału świetlnego świadczy o tym, że nukleotyd nie
został wbudowanym z powodu niekomplementarności.
Składanie sekwencji
chromosomu
•
Strategia „shotgun” – dotyczy krótkich genomów
prokariotycznych. Polega na znalezienie za
pomocą komputera zachodzących na siebie
końców w celu złożenia sekwencji wyjściowej.
Strategie tą zastosowana po raz pierwszy do
zsekwencjonowania genomu Haemophilus
influenzae.
•
Ukierunkowana strategia „shotgun” – dotyczy
wiekszych genów eukariotycznych. Wykorzystuje
się informacje zawarte w mapie genomowej.
Document Outline
- Slide 1
- Slide 2
- Cele sekwencjonowania:
- Metody sekwencjonowania DNA:
- Metoda Maxama i Gilberta - metoda chemicznego sekwencjonowania
- Slide 6
- Metoda Sangera
- Metoda Sangera
- Slide 9
- Slide 10
- Slide 11
- Pirosekwencjonowanie – w czasie rzeczywistym
- Pirosekwencjonowanie
- Slide 14
- Składanie sekwencji chromosomu
Wyszukiwarka
Podobne podstrony:
wiczenie Nr Sekwencjonowanie DNA USTALENIE SEKWENCJI NUKLEOTYDO
Lekcja 6 sekwencjonowanie DNA sekwencje genomów
Sekwencjonowanie DNA prezentacja
BIOTECHNOLOGIA 10d sekwencjonowanie DNA
W4 Metody powielania sekwencji DNA
Sekwencjonowanie DNA
Sekwencjonowanie DNA
Nowosad K, Stępień P, Musiał P Sekwencjonowanie DNA Przegląd metod [2015]
Sekwencjonowanie DNA
Wykład 10x, Genom- obejmuje sekwencje kodujące i niekodujące DNA
Replikacja DNA i choroby związane
W5 sII PCR i sekwencjonowanie cz 2
Elektroforeza DNA komórkowego BioAut1, BioAut2 i Ch1
DNA Eng2
Metoda symultaniczno sekwencyjna
Wykład VIII Synteza układów sekwencyjnych
więcej podobnych podstron