
Klonowanie genów
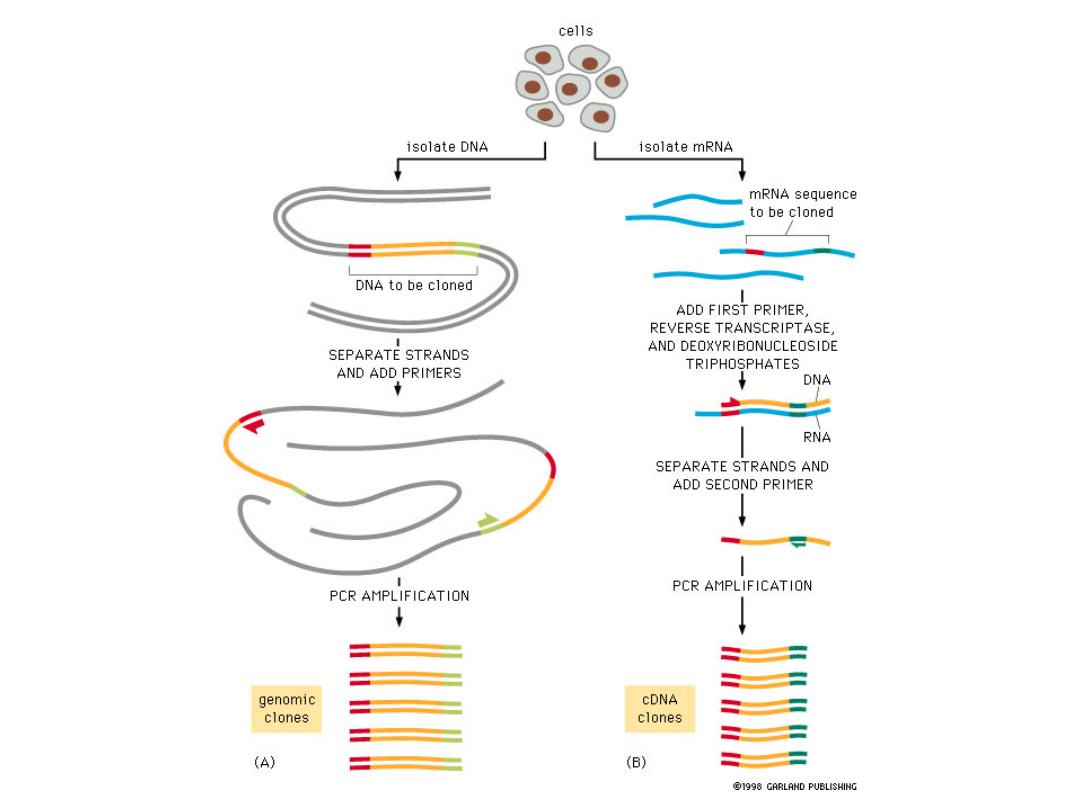
Biblioteki genomowe
Reakcja PCR
Metody powielania
sekwencji DNA
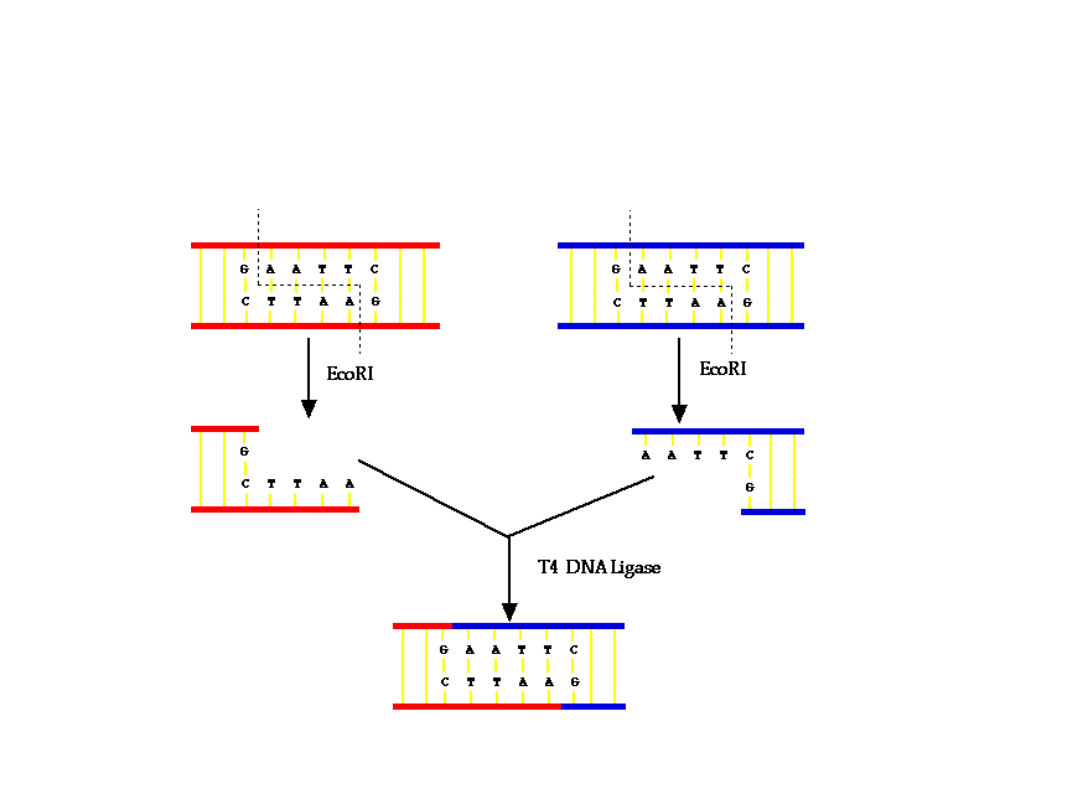
Łączenie fragmentów DNA ciętych
enzymami restrykcyjnymi

Klonowanie
•Trawienie wektora enzymem restrykcyjnym
•Trawienie interesującego nas fragmentu DNA tym
samym enzymem
•Łączenie – fragmentu z wektorem (ligacja)
•Wprowadzenie plazmidu do bakterii
(transformacja)
•Wysiewanie bakterii na płytkę z odpowiednim
antybiotykiem
•Minipreparatyka DNA z pojedynczych kolonii
•Trawienie enzymem restrykcyjnym (wycinanie
„wstawki”)
•Elektroforeza
•Namnażanie bakterii z kolonii zawierających
wstawkę
•Bankowanie
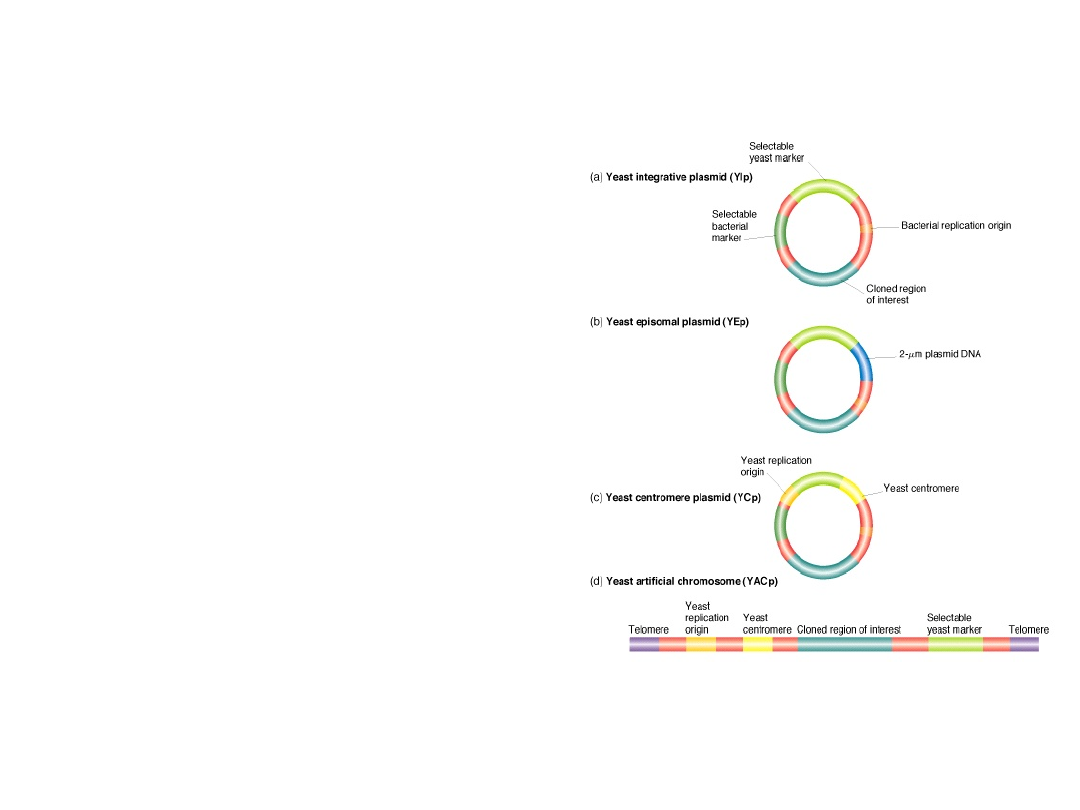
Wektory
•Plazmidowe – najmniejsze,
nadają się do klonowania
fragmentów do kilku tysięcy
pz
•Fagowe – większe,
klonowanie fragmentów do
kilkudziesięciu tysięcy pz
•Drożdżowe – największe,
klonowanie fragmentów
kilkuset tysięcy pz i więcej
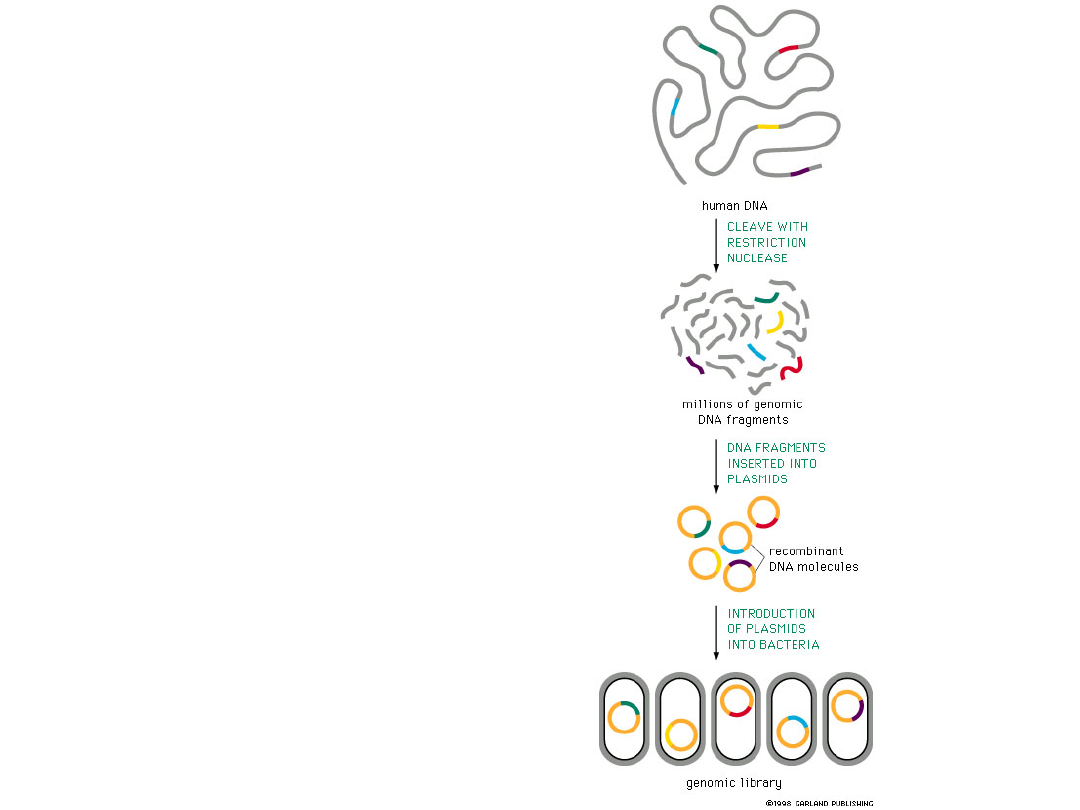
Biblioteki genomowe
Biblioteka genomowa
jest zbiorem różnych
sekwencji
sklonowanych w
wektorach
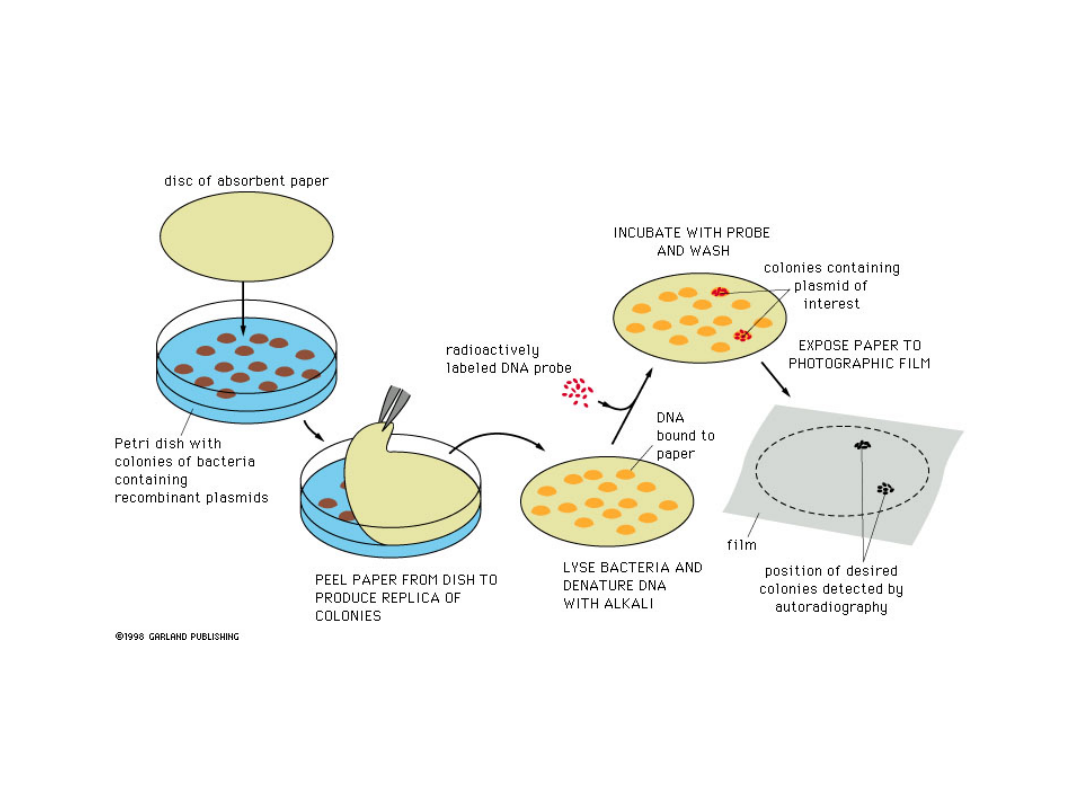
Przeszukiwanie biblioteki
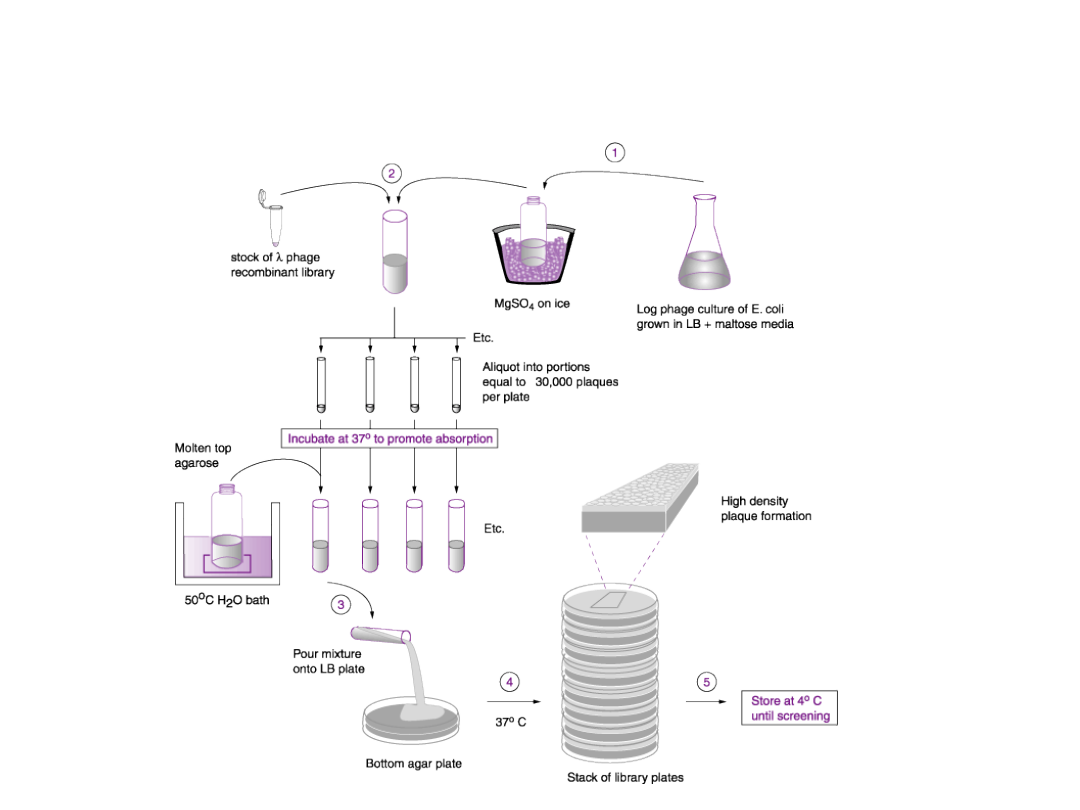
Biblioteki fagowe (fag Lambda)
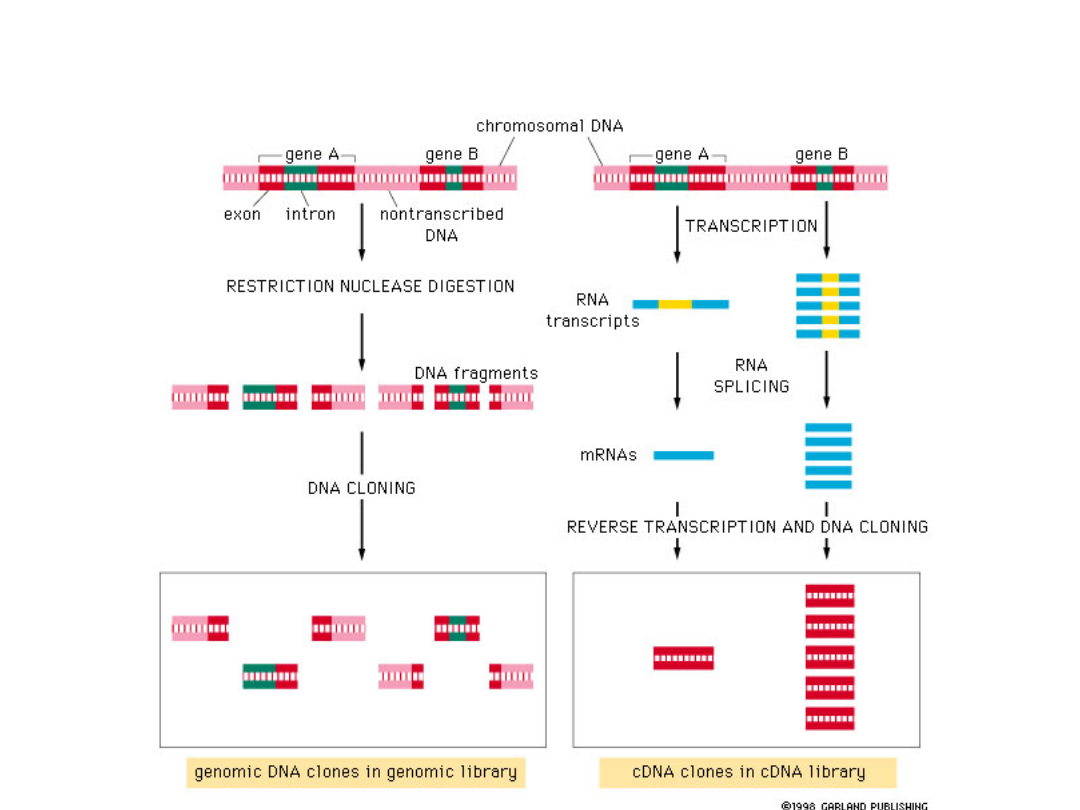
Biblioteka genomowa
Biblioteka cDNA

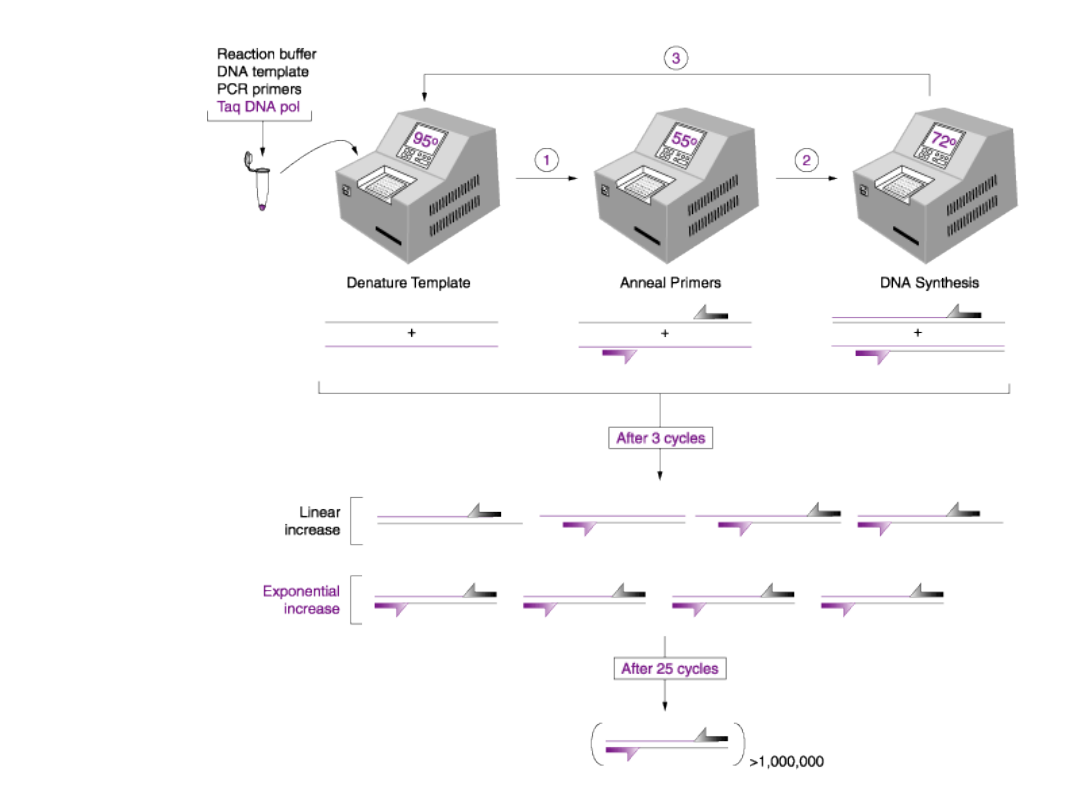
Reakcja PCR (Polymerase Chain Reaction)
•Najczulsza metoda wykrywania i
powielania fragmentów DNA o
określonej sekwencji
•Pozwala na wykrycie i amplifikację
pojedynczej kopii interesującej
sekwencji

Główne etapy reakcji PCR
•Denaturacja wyjściowych cząsteczek DNA
•Wiązanie primerów (annealing)
•Wydłużanie łańcucha (extension)
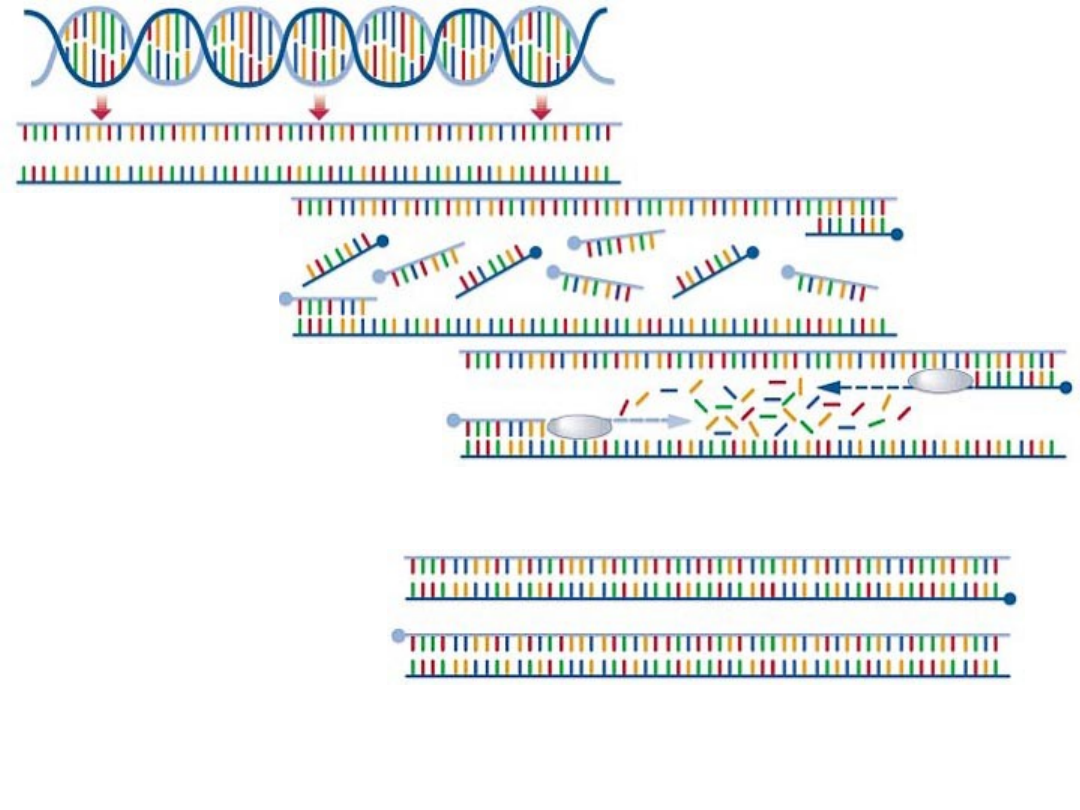
Denaturacja
Annealing
(wiązanie
primerów
)
Extension
(wydłużanie)
Powielone cząsteczki
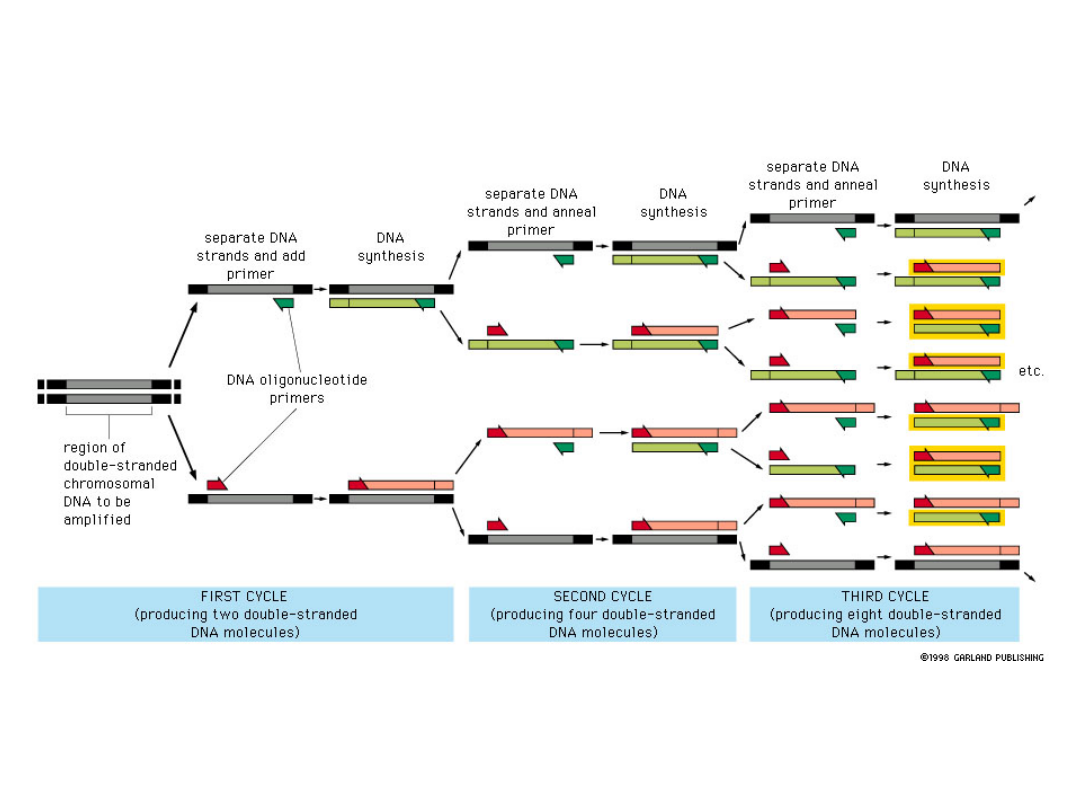
PCR

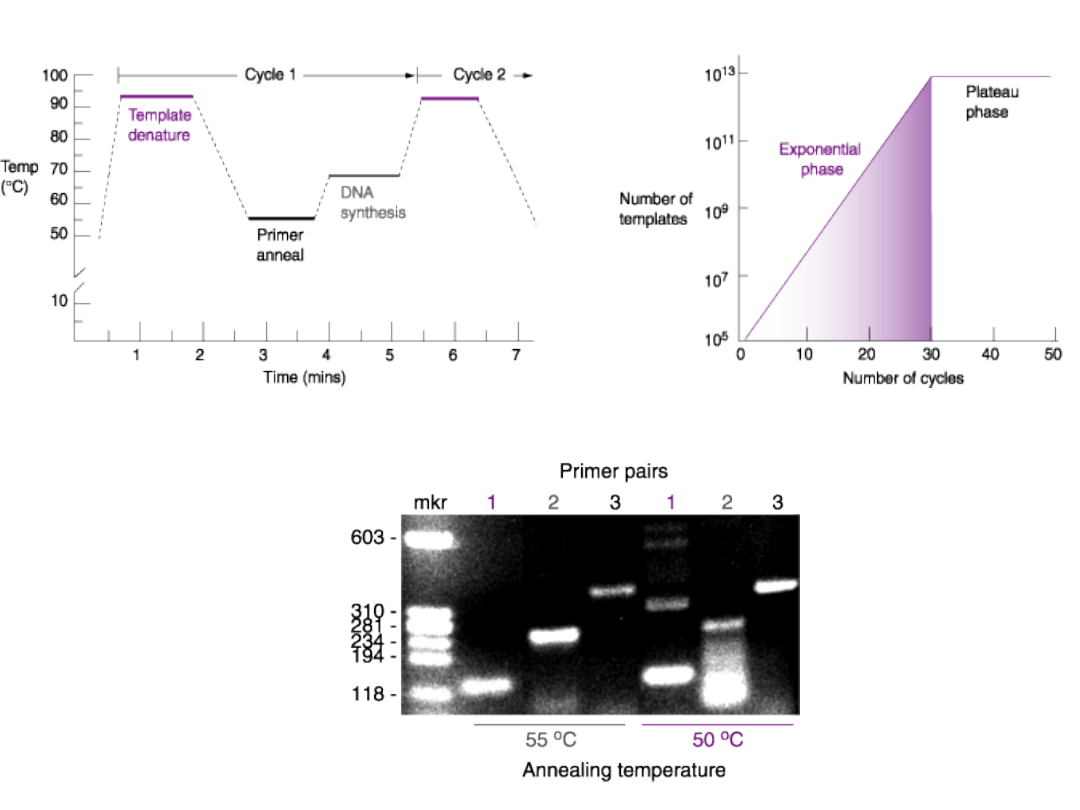
Temperatura jest najważniejszym
czynnikiem wpływającym na wynik
w reakcji PCR

PCR
Primery (startery)
1. długość 18-25 nukleotydów
2. skład 40-60% GC
3. nie powinny zawierać odwróconych powtórzeń
4. temperatura topnienia podwójnoniciowej cząsteczki
tworzonego przez
oba primery z matrycą powinna być
podobna
5. na końcu 3’ powinny mieć G lub C, ale nie więcej niż
jeden nukleotyd

Denaturacja
•Temperatura zależy od składu zasad fragmentu który chcemy
powielić
•Przy fragmentach bogatych w GC używać polimeraz bardziej
opornych na wysoką temperaturę
•Najpopularniejsza polimeraza używana standartowo w PCR – Taq
wytrzymuje temperaturę 94-95 stopni C
Annealing
Temperatura za wysoka – słaba wydajność,
za niska niespecyficzne powielanie
Stosuje się temperaturę około 3-5 stopni poniżej oszacowanej
temperatury topnienia
[Praktyczny wzór na temperaturę topnienia dla primera o 20
nukleotydach:
T=2x(A+T) + 4(G+C)]
Extension
Temperatura tej części cyklu zależy od właściwości polimerazy.
Dla polimerazy Taq optymalna jest temperatura 72-78 stopni
Szybkość reakcji w optymalnych warunkach około 2000
nukleotydów/min
PCR
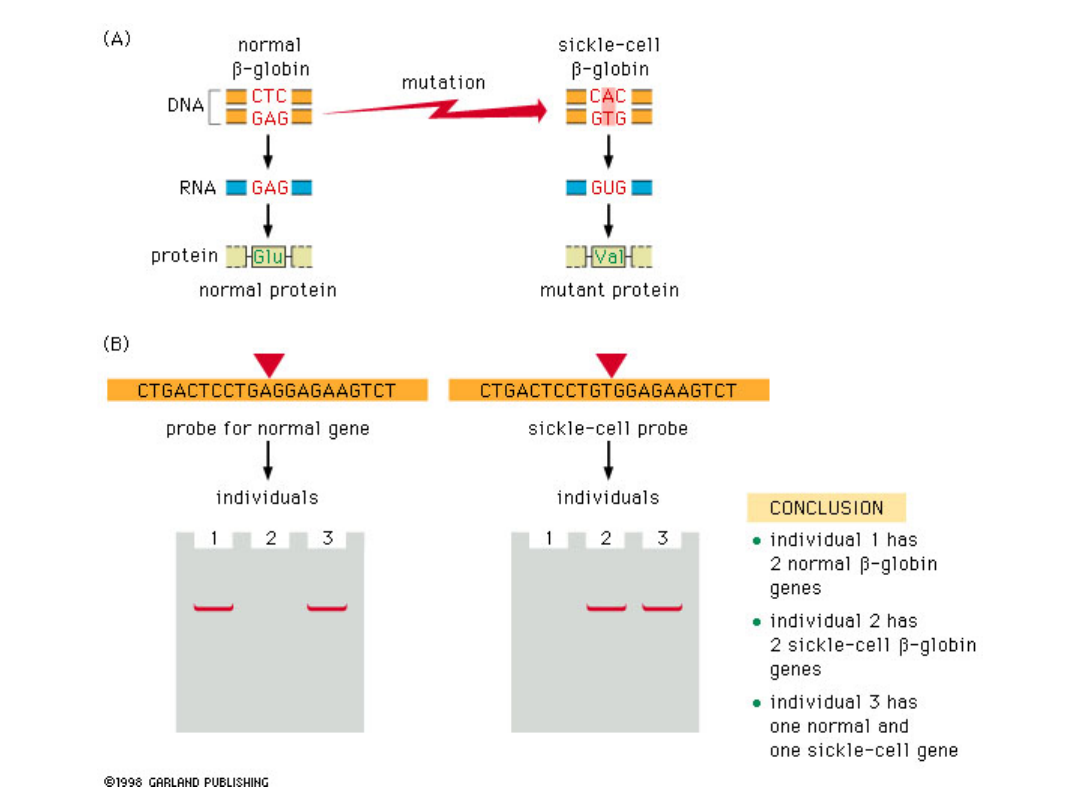
Anemia sierpowata
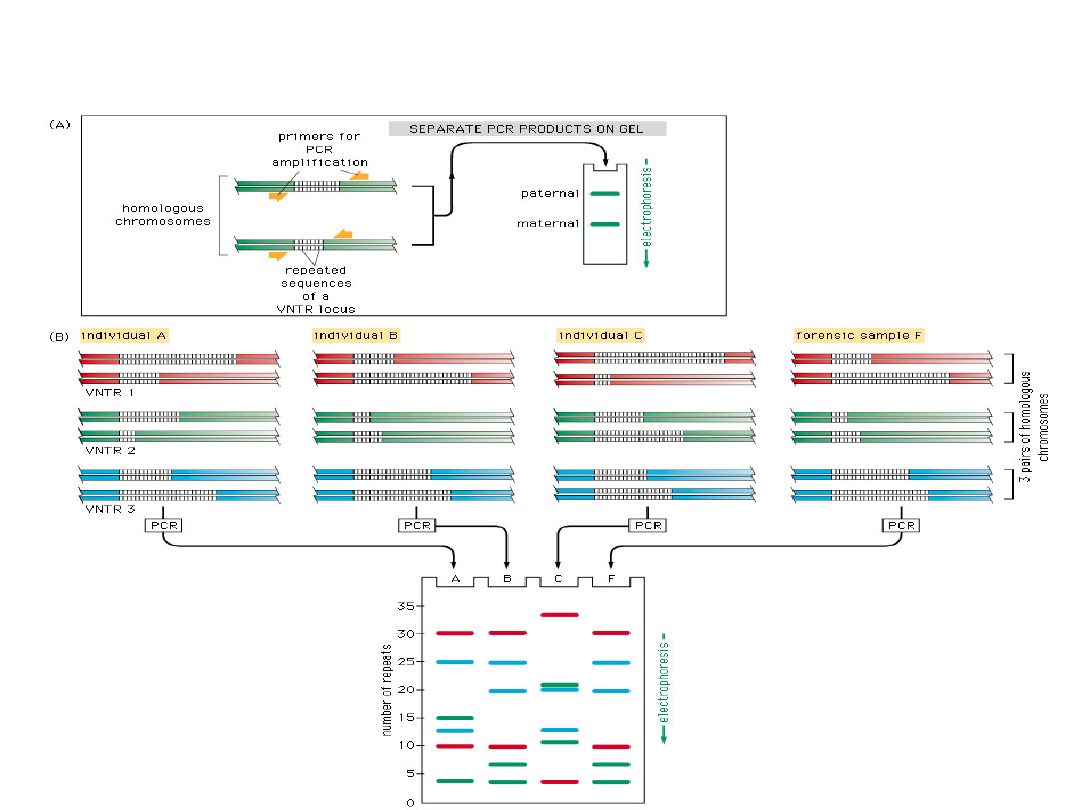
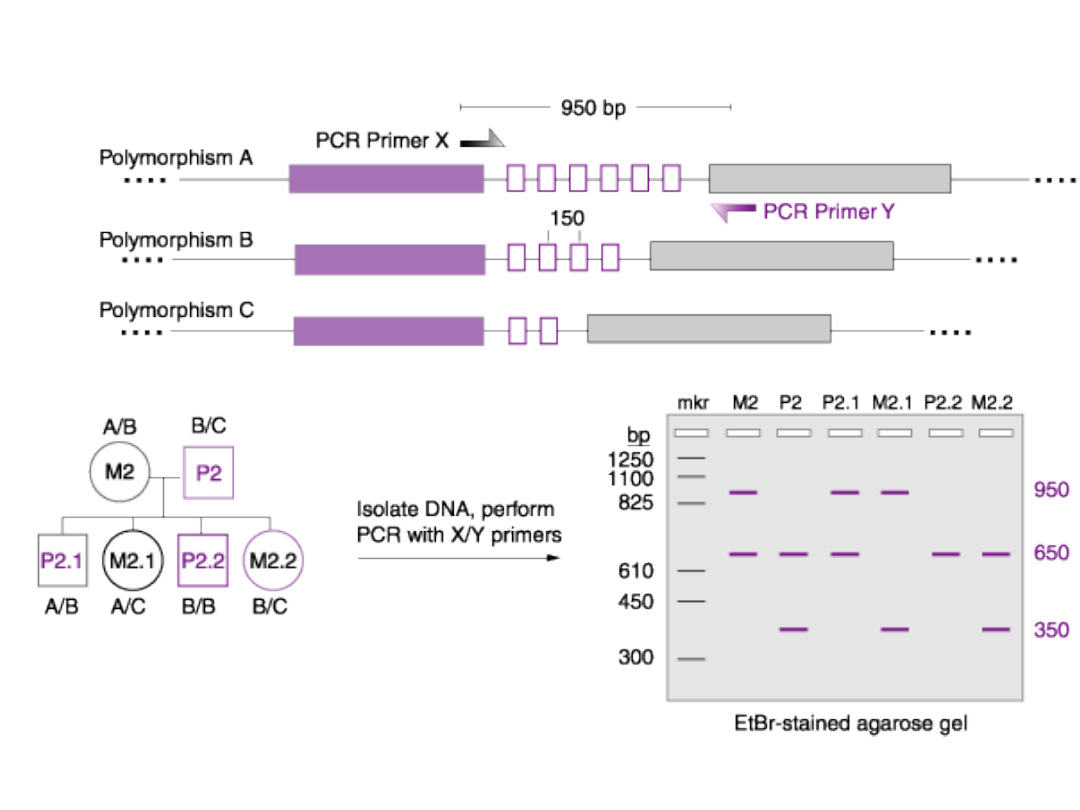
RFLP PCR – polimorfizm długości fragmentów
restrykcyjnych
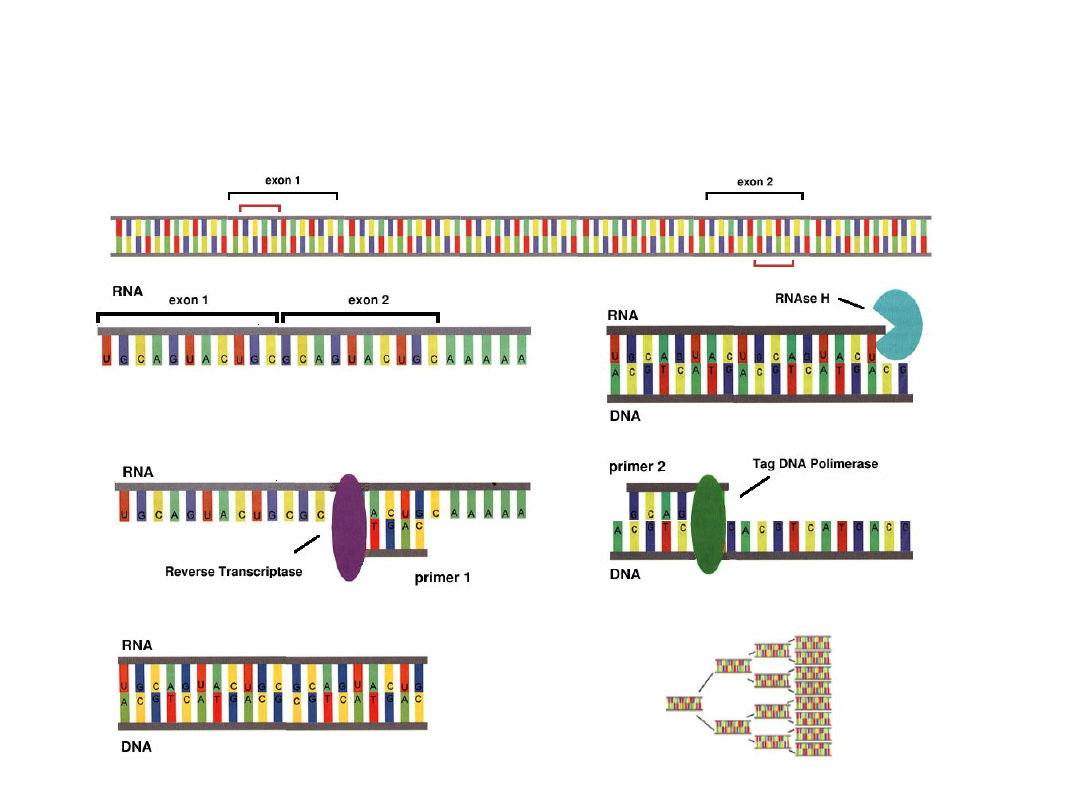
Reverse Transcription PCR
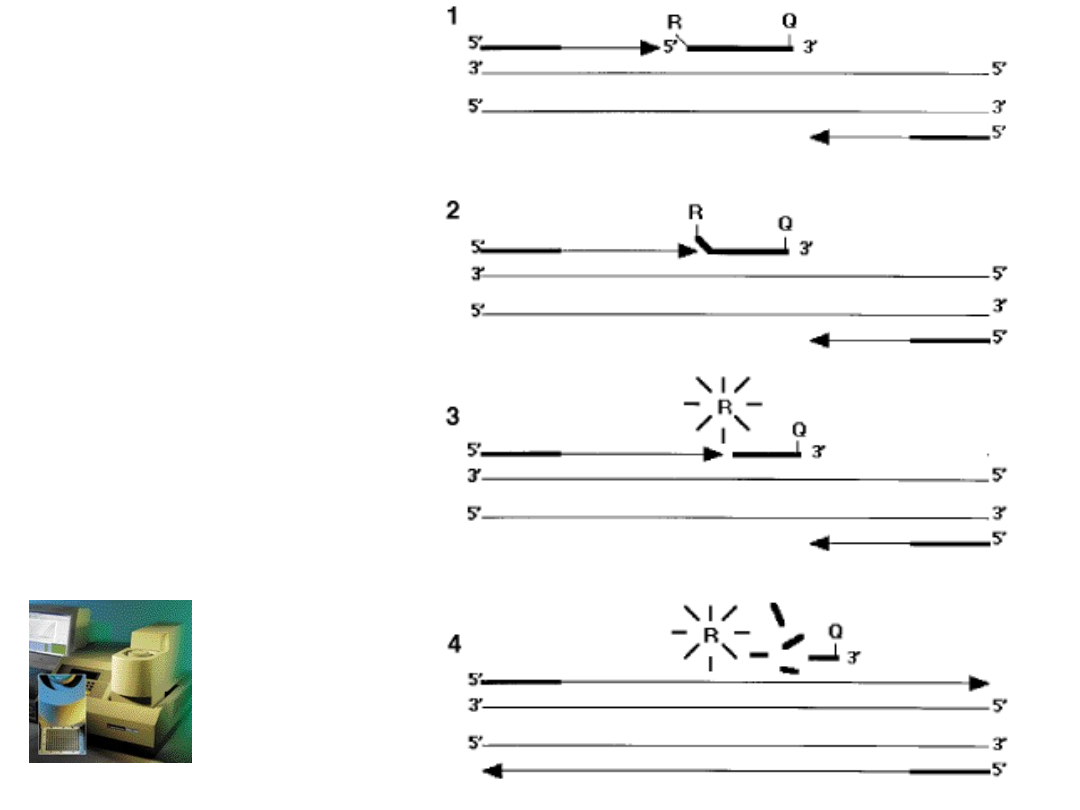
Real Time PCR
Pozwala na śledzenie
akumulacji produktów w
trakcie przebiegu
reakcji PCR
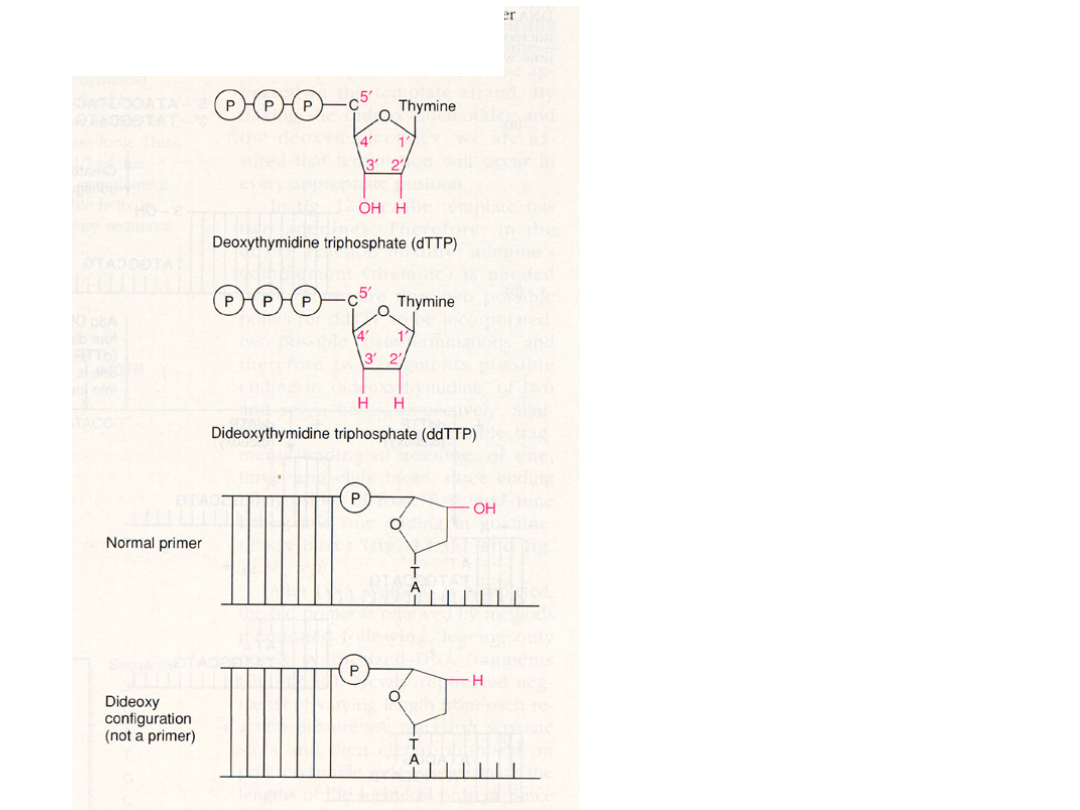
Sekwencjonowani
e DNA
(metoda
dideoxy-)
Nukleotydy dideoxy- powodują
zakończenie replikacji DNA
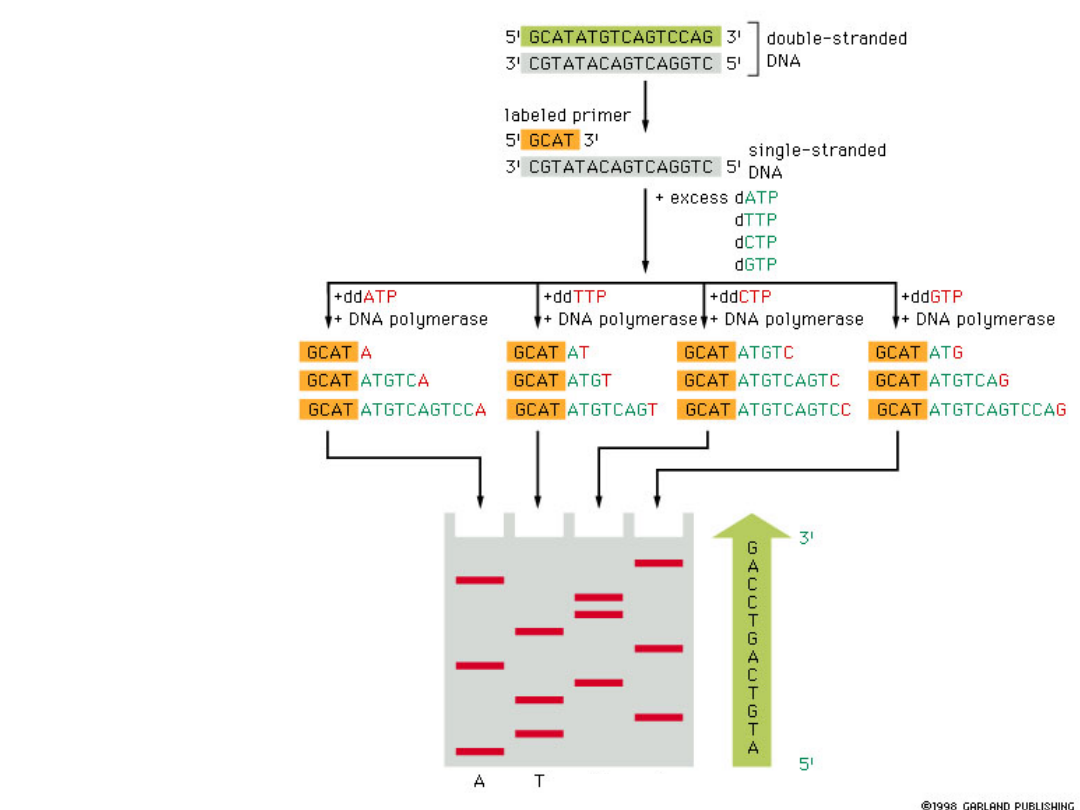
Sekwencjonowanie
DNA
C
G
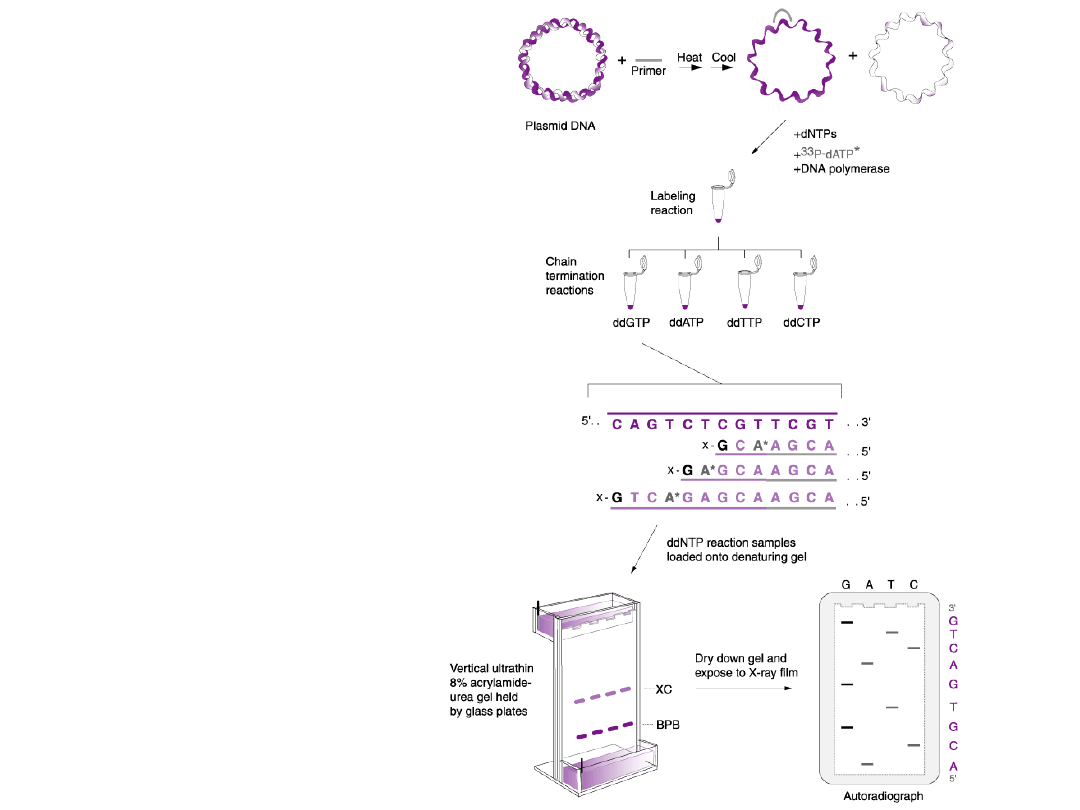
Znakowanie
sekwencjonowane
go
fragmentu
pojedynczym
nukleotydem

Znakowanie końcowego
nukleotydu (dideoxy)

Odczytywanie sekwencji DNA
Metoda chemiczna sekwencjonowania polega na
wykorzystaniu specyficznych wobec
poszczególnych zasad reakcji chemicznych, co
pozwala na identyfikację miejsca występowania
danej zasady
Zasada
Specyficzna modyfikacja
G
Metylacja N7 dimetylosiarczanem w pH 8.0
sprawia, że wiązanie C8-C9 staje się specyficznie
wrażliwe na rozkład przy pomocy zasady.
A+G
Mrówczan piperydyny w pH 2.0 osłabia wiązania
glikozydowe puryn, co powoduje depurynację
C+T
Hydrazyna otwiera pierścień pirymidynowy, który
następnie recyklizuje w formę podatną na
usunięcie
C
W obecności 1.5 M NaCl jedynie cytozyna reaguje
specyficznie z hydrazyną
A>C
1.2 N NaOH w temp. 90
0
C powoduje częste
przecinanie reszt A i rzadsze - C
Piperydyna przecina łańcuch fosfocukrowy w miejscu modyfikacji zasady
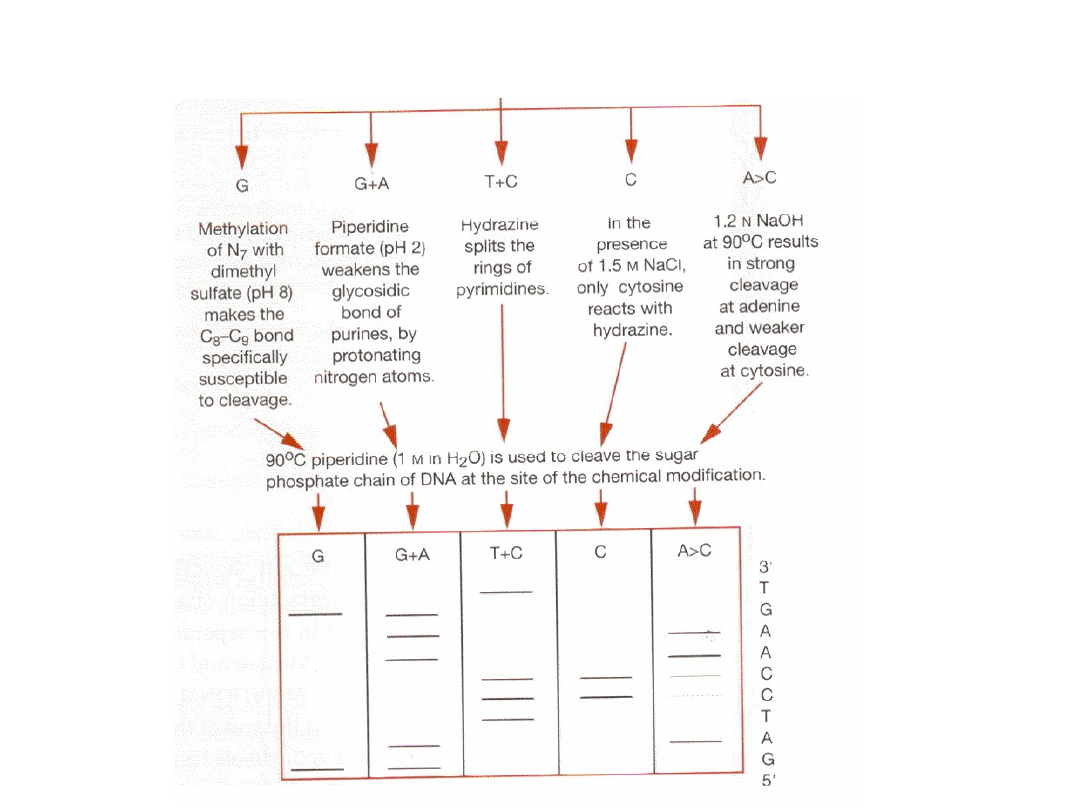
Odczytywanie sekwencji
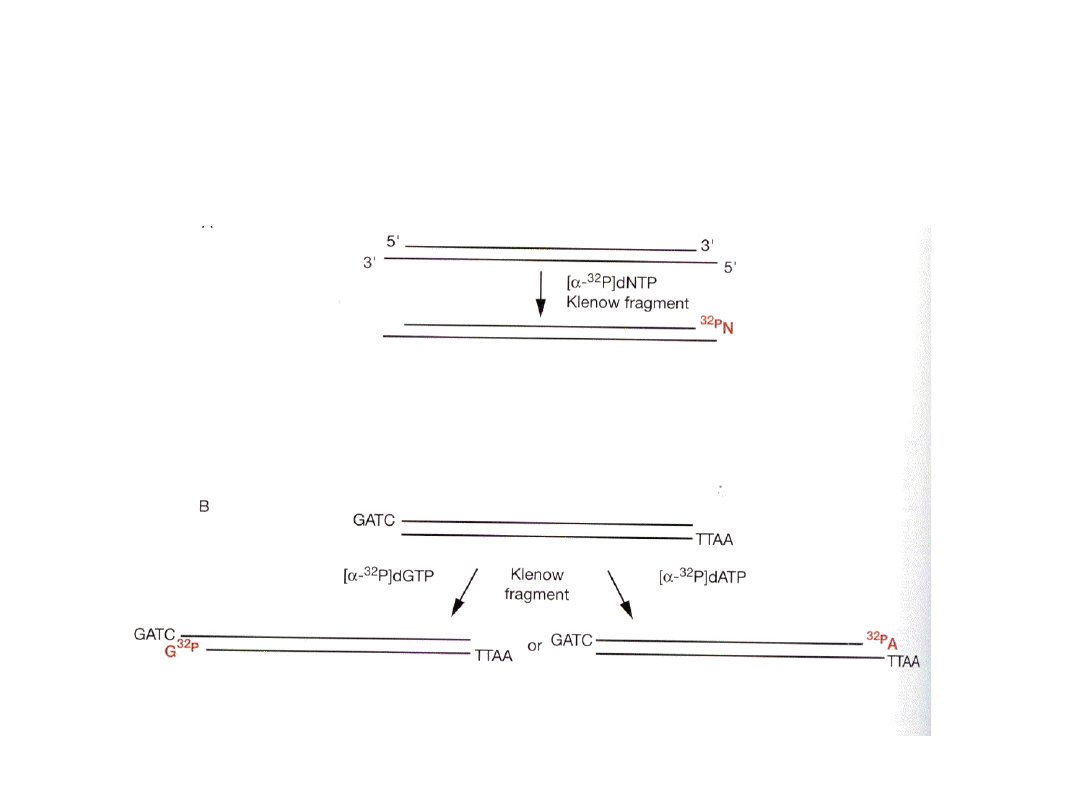
Specyficzne znakowanie jednej
nici DNA przy użyciu fragmentu
Klenowa polimerazy I
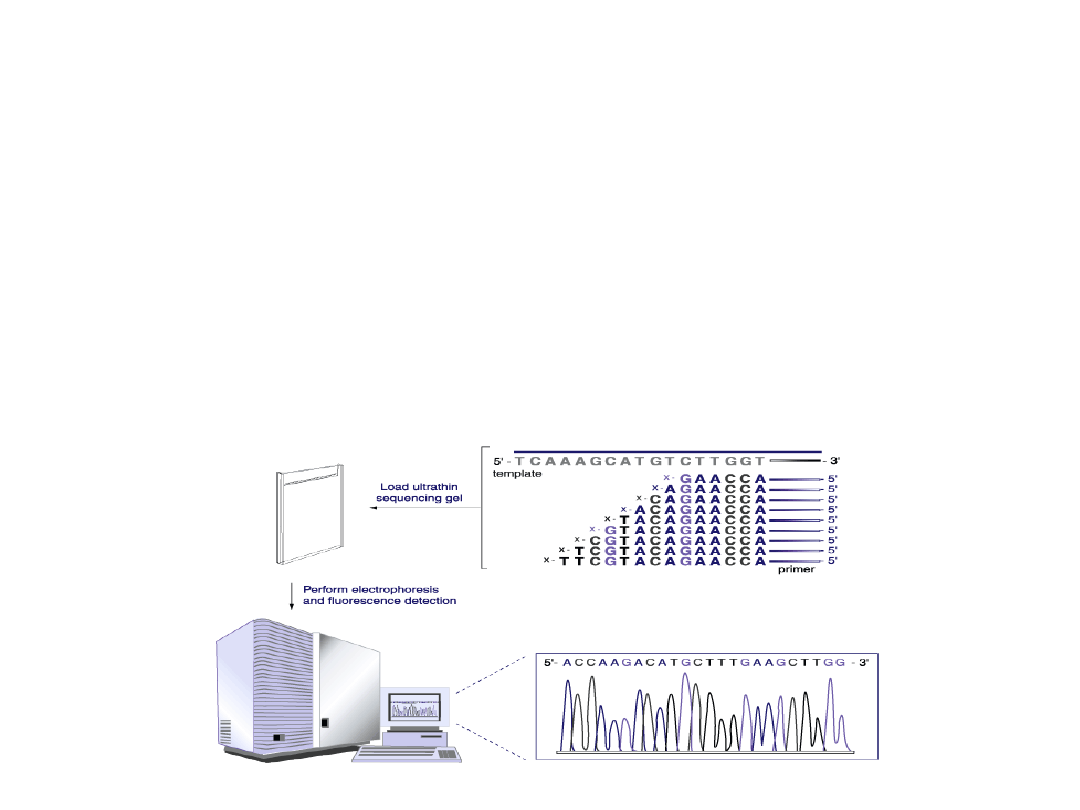
Sekwencjonowanie automatyczne
W systemie tym stosuje się nukleotydy dideoxy lub startery
znakowane czterema różnymi znacznikami fluorescencyjnymi.
Można stosować sekwencjonowanie rozpoczynające się od jednego
startera
i zatrzymujące się na znakowanych nukleotydach dideoxy.
Wszystkie cztery znakowane nukleotydy dideoxy występują wtedy
w jednej mieszaninie.
Inną możliwością jest stosowanie znakowanych starterów,
prowadzenie czterech niezależnych reakcji, a następnie rozdział na
jednej ścieżce żelu. Po reakcji mieszanina nanoszona jest na żel
poliakrylamidowy,
a elektroforeza prowadzona jest w aparacie wyposażonym w
detektor promieniowania.
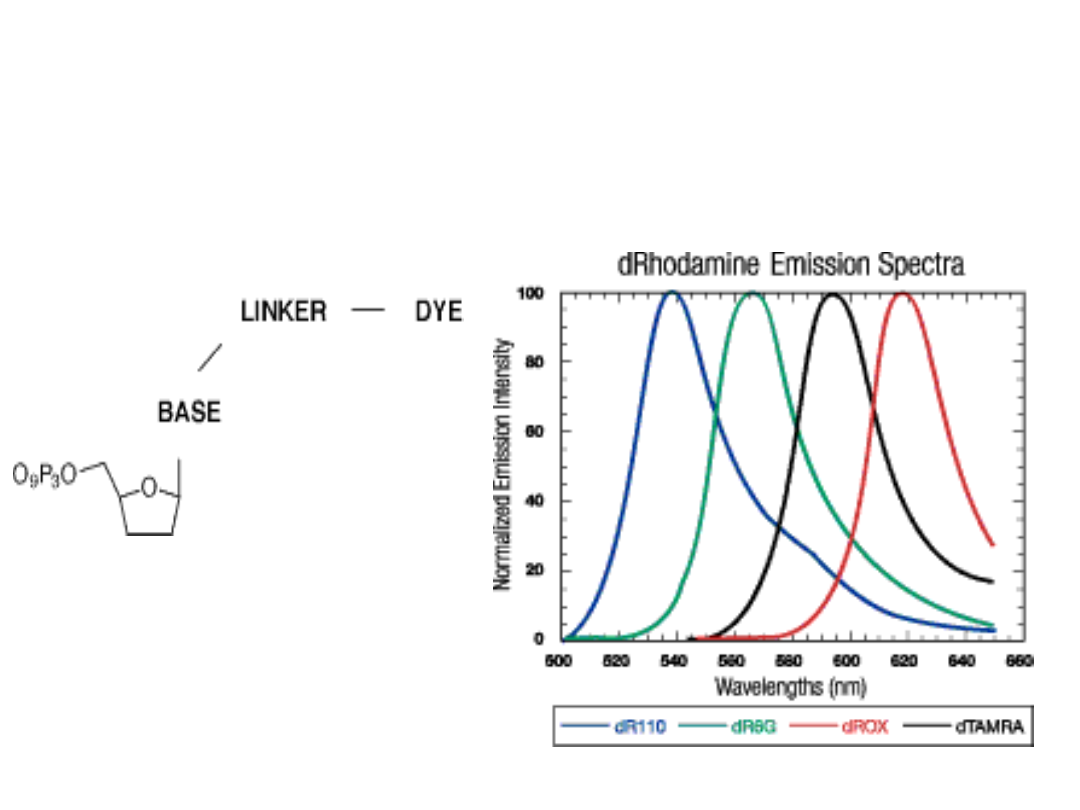
System
ABI
(Applied
Biosystems
Incorporated)
używa
czterech
znakowanych
fluorescencyjnymi
barwnikami
ddNTPs
i polimerazy Taq
(AmpliTaq).
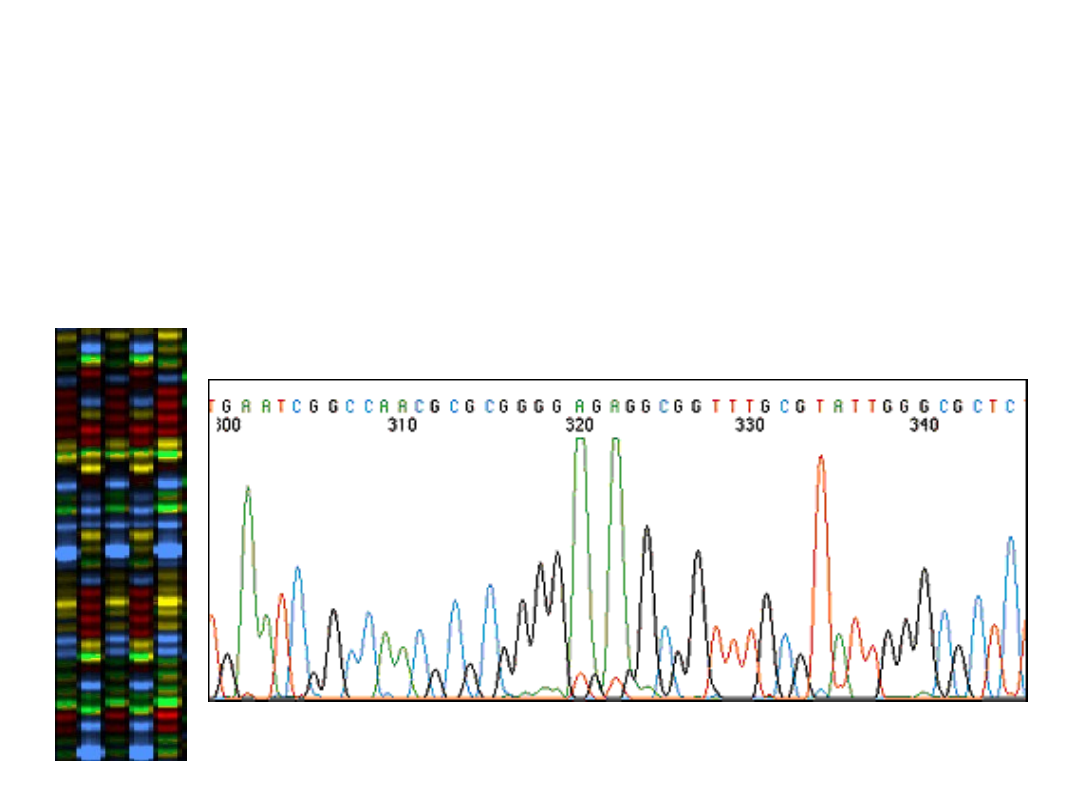
Wzbudzanie fluorochromów i detekcja emisji
mają miejsce w określonej pozycji w pobliżu
dolnej części żelu. Dane są odczytywane na
podstawie spektrum emisyjnego kolejnych pasm
mijających
detektor
w czasie elektroforezy. Dane przekazywane są
do komputera podłączonego do urządzenia
.
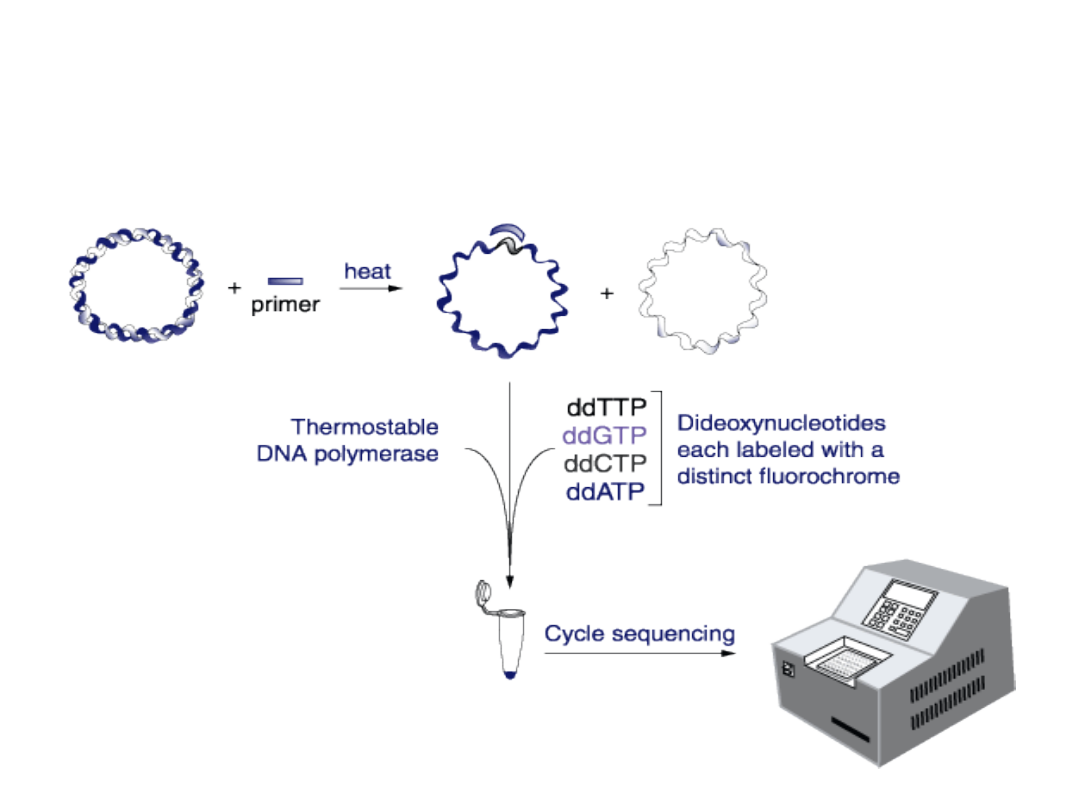
Sekwencjonowanie cykliczne
(połączenie metody PCR i
sekwencjonowania) pozwala
na liniową amplifikację sygnału
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
Wyszukiwarka
Podobne podstrony:
wiczenie Nr Sekwencjonowanie DNA USTALENIE SEKWENCJI NUKLEOTYDO
Lekcja 6 sekwencjonowanie DNA sekwencje genomów
Sekwencjonowanie DNA prezentacja
BIOTECHNOLOGIA 10d sekwencjonowanie DNA
Sekwencjonowanie DNA
SEKWENCJONOWANIE DNA 2
Sekwencjonowanie DNA
Nowosad K, Stępień P, Musiał P Sekwencjonowanie DNA Przegląd metod [2015]
Sekwencjonowanie DNA
Metody badania polimorfizmu DNA
metody numeryczne w4
Wykład 10x, Genom- obejmuje sekwencje kodujące i niekodujące DNA
W4 PCR, Sekwencjonowanie
metody wprow DNA do kom
więcej podobnych podstron