
Zastosowanie
markerów
molekularnych w
ogrodnictwie
Anna Olejnik
Ogrodnictwo II
Kształtowanie Terenów Zieleni

Markery molekularne -
podział

• Markery DNA
to prążki na
żelach lub membranach
odpowiadające odcinkom
DNA o różnej długości,
będące końcowym efektem
reakcji enzymatycznych lub
hybrydyzacji.
• Wzory prążkowe są
specyficzne dla
poszczególnych roślin.
Dziedziczą się zgodnie z
genetyką mendlowską nie
podlegając wpływowi
środowiska

Selekcja przy użyciu markerów
molekularnych
Długotrwały i skomplikowany cykl hodowlany
można znacznie skrócić prowadząc selekcję z
wykorzystaniem markerów DNA,
w tym celu wykorzystuje się istnienie
bliskiego sprzężenia pomiędzy markerem a
locus odpowiedzialnym za dziedziczenie cechy
użytkowej,
metoda ta często jest określana skrótem MAS
(Marker Assisted Selection), gdzie ma
szerokie zastosowanie w hodowli roślin
drzewiastych i hodowli odpornościowej.

Wykorzystanie markerów
molekularnych do selekcji
rodów ziemniaka odpornych na
Globodera rostochiensis
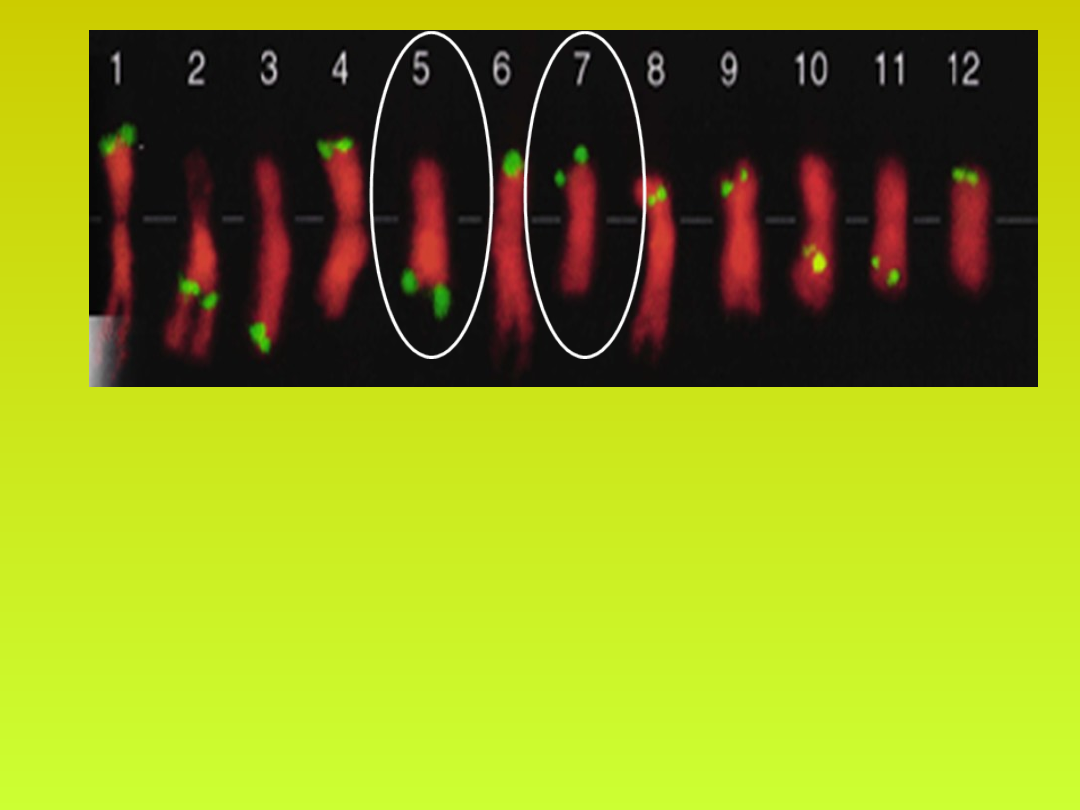
Na zaznaczonych chromosomach
znajdują się geny sprzężone z
opornością ziemniaka na
Globodera rostochiensis

Materiał i metody
• Materiał użyty do badań stanowiły liście
utrwalonych w ciekłym azocie odmian i rodów
ziemniaka, pochodzących z pośród roślin
wysadzonych w kwietniu 2002/2004 na polu
doświadczalnym w RZD - Swojec.
• W pracy wykorzystano metodę izolacji
genomowego DNA, która stanowiła
modyfikację metody izolacji roślinnego DNA
opracowanej przez H. Junghans i M. Metzlaff,
opublikowanej w czasopiśmie Bio Techniques
1990.

ZASTOSOWANE
STARTERY
• Primery BCH (marker kontrolny dla przebiegu
reakcji PCR)
• Primer GT698 (oczekujemy produktu długości 141
bp, który pochodzi z V chromosomu i jest sprzężony z
genem odporności na Globodera rostochiensis R01)
• Primer F,R (oczekujemy produkt wielkośći 800 bp),
który pochodzi z chromosomu V i jest sprzężony z
genem odporności na Globodera rostochiensis R01)

Wyniki
• Pierwszy marker molekularny (800 bp) nie
nadaje się do selekcji genotypów
ziemniaka odpornych na Globodera
rostochiensis.
• Zastosowany marker (141 bp) wydaje się
być odpowiedni do przeprowadzania
selekcji na mątwika ziemniaczanego na
dowolnym etapie hodowli ziemniaka
• Dla badanych rodów ziemniaka należy
dopracować reakcję PCR.

Wykorzystanie markerów
SSR do molekularnej
charakterystyki zasobów
genowych jabłoni

Celem pracy była ocena możliwości
wykorzystania wybranych
markerów SSR w zarządzaniu
kolekcją jabłoni oraz molekularna
charakterystyka zasobów
genowych części kolekcji z Pola
Doświadczalnego SGGW w
Wilanowie

Materiał i metody
• Izolację DNA metodą CTAB z niewielkimi
modyfikacjami przeprowadzono z liści klonu U 211
oraz 44 odmian jabłoni z kolekcji zlokalizowanej na
Polu Doświadczalnym SGGW w Wilanowie.
• Do powielenia DNA wykorzystano osiem par
starterów SSR: NZ28f4, NZ23g4, NZ04h11,
NZ02b1, NZ01a6, CH02D12, CH01C06 i CH01H10.
• Produkty amplifikacji były rozdzielane w 6-
procentowym poliakrylamidowym żelu
denaturującym i barwione azotanem srebra.
• Analizę statystyczną przeprowadzono przy użyciu
programu POPGENE 1.32.

Wyniki
• Na podstawie odległości genetycznych, przy użyciu metody UPGMA w
programie POPGENE, sporządzony został dendrogram będący
graficznym przedstawieniem odległości genetycznych pomiędzy 44
badanymi odmianami i klonem U 211.
• W otrzymanym dendrogramie można wyodrębnić kilka wyraźnych
grup. Do pierwszej należą odmiany takie, jak: ‘Freedom’, ‘Ligol’,
‘Šampion’, ‘Gala’ i ‘Elstar’, posiadające w rodowodzie ‘Golden
Delicious’. Kolejną stanowiły te wywodzące się od ‘Cox’s Orange
Pippin’ (‘Alkmene’, ‘Delikates’, ‘James Grieve’, ‘Fiesta’). ‘Fiesta’
pochodzi z krzyżowania ‘Cox’s Orange Pippin’ i ‘Idared’, co może
tłumaczyć obecność odmiany Idared w grupie. Blisko siebie znalazły
się również odmiany spokrewnione z odmianą McIntosh. Dendrogramy
• Przy użyciu markerów mikrosatelitarnych odmiany jabłoni mogą być
jednoznacznie identyfikowane. Na podstawie porównania długości
alleli udało się zidentyfikować odmianę mylnie w kolekcji oznaczoną
jako ‘Kovelit’. W osobniku opisanym jako ‘Kovelit II’ stwierdzono inne
długości alleli w większości analizowanych loci SSR niż w odmianie
Kovelit

Monitorowanie zjawiska
odporności przędziorka
chmielowca na METI-
akarycydy w gospodarstwach
ogrodniczych przy użyciu
markerów SCAR

Celem pracy było opracowanie
markerów molekularnych,
umożliwiające analizę zmienności
genetycznej przędziorka
chmielowca pod kątem wykrywania
mutacji skutkujących
wystąpieniem odporności
szkodnika na akarycydy.

Charakterystyka opracowanych
markerów
• Opracowane markery są sprzężone z genami kodującymi
monoksygenazę cytochromozależną (P450-00830) oraz S-
transferazę glutationu (GSt-O_03900 i GSt-D_00220).
Zmienność genetyczna między rasami obejmowała długość
amplikonów oraz zmiany nukleotydowe w obrębie produktów
PCR.
• Polimorficzne sekwencje SCAR zostały zidentyfikowane w
genomach rasy referencyjnej GSS (rasa wrażliwa na
akarycydy) oraz rasy AKITA (zmutowana rasa odporna),
udostępnionych przez firmę Bayer CropScience. Skuteczność
wykrywania osobników przędziorka odpornych na METI-
akarycydy z użyciem markerów zweryfikowano w populacjach
szkodnika pochodzących z sadów eksperymentalnych i sadów
produkcyjnych, w których stosowano intensywną ochronę.

Próba znalezienia markerów
genetycznych odporności na
mączniaka rzekomego u ogórka
techniką RAPD

Charakterystyka
•
próba znalezienia markerów genetycznych odporności na
mączniaka rzekomego u ogórka techniką RAPD (w doświadczeniu
przy użyciu 6 wyselekcjonowanych starterów uzyskano
amplifikowane fragmenty DNA polimorficzne dla osobników
badanych populacji.
•
Analiza wzorów fragmentów DNA otrzymanych przy użyciu tych
starterów wykazała występowanie produktów PCR
charakterystycznych dla osobników o wysokim stopniu
odporności oraz dla osobników wrażliwych.
•
Aby jednak marker można było uznać za „informatywny”
(sprzężony z cechą), musi on znajdować się w odległości
mniejszej niż 15 cM od genu odpowiedzialnego za daną cechę,
dlatego uzyskane wyniki potraktowano jako badanie wstępne,
którego kontynuacją będzie zidentyfikowanie znalezionych
odcinków DNA jako markerów RAPD oraz ich przyporządkowanie
cesze odporności na mączniaka rzekomego).

Wykorzystanie markera QTL w
hodowli jabłoni odpornej na
mączniaka (Podosphaera
leucotricha (Ellis et Ev.) Salm.)

Celem przeprowadzonych badań było
sprawdzenie możliwości szerokiego
zastosowania markera NZ28f04 w
programach hodowlanych
wykorzystujących jako źródło
odporności na mączniaka klon U 211.

Materiał i metody
• Ekstrakcję DNA z liści 50 drzew z potomstwa Granny Smith × U
211 przeprowadzono przy użyciu metody CTAB z modyfikacjami.
DNA roślin rodzicielskich oraz każdego z osobników z
potomstwa amplifikowano w reakcji PCR przy użyciu starterów i
procedury dla markera NZ28f04.
• Produkty amplifikacji były rozdzielane w 6% poliakrylamidowym
żelu denaturującym i barwione azotanem srebra.
• Równocześnie rośliny były oceniane w latach 2000–2001 w
szkółce, a 22 z nich posadzono w sadzie w 2001 roku i
obserwowano w następnym sezonie wegetacyjnym. Reakcję na
mączniaka jabłoni oceniano w sześciostopniowej skali od 0 do 5
(0 — nieporażone, 5 — bardzo silnie porażone).
• Klon U 211 zaliczany jest do klasy 0, natomiast Granny Smith,
jako odmiana wrażliwa na mączniaka, do klasy 4.
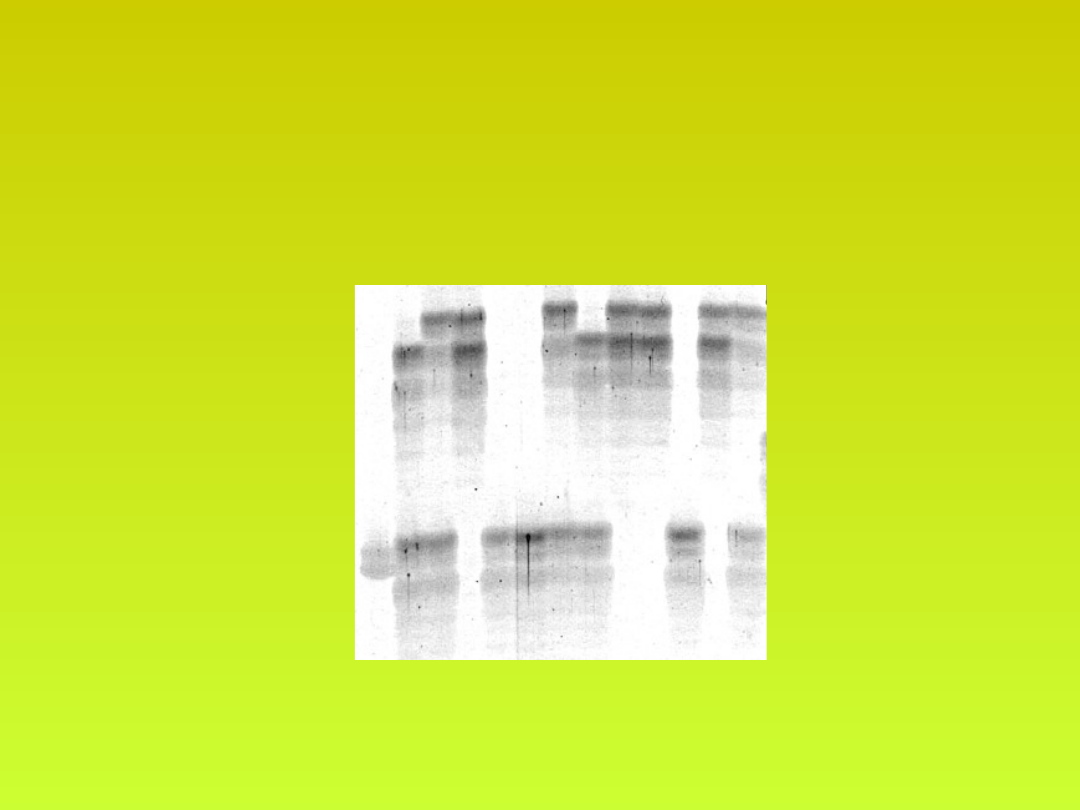
Wyniki i wnioski
• Wynik amplifikacji markera NZ28f04 przy użyciu DNA roślin
rodzicielskich oraz części osobników z potomstwa Granny Smith ×
U 211 przedstawia rysunek 1. Allel związany ze wzrostem
odporności na mączniaka jest obecny u klonu U 211 oraz u części
potomstwa, brak go natomiast u odmiany Granny Smith
M 1 2 3 4 5 6 7 8 9 10 11 12
r→
100 bp →
Rys. 1. Elektroforogram prezentujący produkty amplifikacji markera NZ28f04. M
— marker wielkości, 1 — U 211, 2 — Granny Smith, 3 — 12: osobniki z potomstwa
Granny Smith × U 211, r — allel markera związany ze wzrostem odporności na
mączniaka jabłoni

• Selekcja prowadzona przy użyciu markera mikrosatelitarnego
NZ28f04 pozwoliłaby na usunięcie 8 roślin średnio podatnych
zaliczonych do klasy 3, jeśli wzięto by pod uwagę wyniki oceny
polowej ze szkółki z roku 2000, co stanowi 72% wszystkich roślin w
klasie 3 w tym roku. W roku 2001 do klasy 3 zakwalifikowano 9 roślin,
z których 3, czyli 33% nie wykazało obecności allela NZ28f04
związanego ze wzrostem odporności na mączniaka. W potomstwie
Granny Smith × U 211 w ciągu 2 lat obserwacji w szkółce żadnego z
osobników nie zakwalifikowano do klas 4 i 5.
• Gdyby wzięto pod uwagę wyniki oceny fenotypowej z sadu (rok 2002)
oraz wynik analizy z markerem, wyeliminowane zostałyby 2 rośliny, co
stanowi 100% osobników zaliczonych do klasy 4 oraz 2 rośliny z klasy
3, czyli 66%. Rośliny charakteryzujące się odpornością lub małą
podatnością na mączniaka jabłoni (klasy 0–2) nie wymagają
prowadzenia ochrony w sadzie, tak więc uzasadnione jest
pozostawienie tylko takich roślin do dalszej ich oceny pod względem
innych cech użytkowych.
WNIOSKI
1. Wyniki analizy przeprowadzonej z markerem NZ28f04 na potomstwie
Granny Smith × U 211 potwierdzają stabilność QTL oznaczonego
jako U7, zidentyfikowanego w potomstwie Idared × U 211.
2. Marker NZ28f04 może znaleźć zastosowanie w programach
hodowlanych wykorzystujących jako źródło odporności na mączniaka
klon U 211.

Poszukiwanie markerow
RAPD sprzężonych z
odpornością na nicienie u
pomidora

Charakterystyka
•
Poszukiwano też markerów RAPD sprzężonych z
odpornością na nicienie u pomidora. Jeden z
polimorficznych produktów amplifikacji przekształcono
następnie w marker typu SCAR, różnicujący rośliny
wrażliwe i odporne. Wprawdzie specyficzny starter
powodował namnożenie odcinka DNA we wszystkich
roślinach, ale po trawieniu produktu reakcji enzymem
restrykcyjnym Taq I, wzór prążkowy roślin odpornych
był inny niż roślin wrażliwych.
•
Marker ten okazał się znacznie silniej sprzężony z
odpornością na nicienie niż używany do tej pory
marker izoenzymatyczny.

Inne
• Metody identyfikacji leśnego materiału rozmnożeniowego w oparciu o
markery molekularne DNA (przykłady praktycznych zastosowań
markerów molekularnych w badaniach identyfikacyjnych):
identyfikacja gatunków dębów w oparciu o wybrane jądrowe markery
mikrosatelitarne
identyfikacja pochodzenia świerka pospolitego w oparciu o markery
cytoplazmatyczne
możliwości weryfikacji pochodzenia nasion z drzewostanów dębowych w
oparciu o markery cytoplazmatyczne
możliwości wykorzystania jądrowych sekwencji mikrosatelitarnych do
weryfikacji poprawności zbioru nasion z pojedynczych drzew matecznych
dębów
wykorzystanie chloroplastowych sekwencji mikrosatelitarnych w celu
weryfikacji poprawności zbioru nasion przechowywanych w zasobach LBG,
pozyskanych z drzew matecznych sosny zwyczajnej
wykorzystanie chloroplastowych sekwencji mikrosatelitarnych do weryfikacji
przynależności szczepów do poszczególnych klonów na przykładzie klonalnej
plantacji nasiennej sosny zwyczajnej
• Znając marker sprzężony z genem warunkującym purpurową barwę
owoców czereśni, można prowadzić selekcję w tym kierunku, na kilka
lat przed wejściem drzewa w okres owocowania

• Zastosowanie markerów RFLP do opracowania
dendrogramu obrazującego pokrewieństwa między 38
genotypami z rodzaju Brassica:
Udowodniono między innymi istnienie bliskiego
pokrewieństwa pomiędzy genomami brokuła i kalafiora oraz
kapusty głowiastej i jarmużu. Wykazano, że wraz z ewolucją
rodzaju Brassica, zwiększyła się liczba chromosomów w jego
genomie
• Użycie markerów RFLP generowanych u pomidora do
badań podobieństwa uszeregowania oraz liczby genów u
trzech rodzajów Solanaceae: papryki (Capsicum annuum
L.), ziemniaka (Solanum tuberosum L.) i pomidora:
We wszystkich trzech gatunkach mapy sprzężeń konstruowano
przy użyciu wielu tych samych markerów. Grupy sprzężeń
pomidora i ziemniaka wykazały wysokie podobieństwo,
dziewięć chromosomów odznaczało się homologią sekwencji.
Genom papryki wyraźnie różnił się od poprzednich znaczną
liczbą przegrupowań interchromosomowych i w konsekwencji
rozmieszczeniem genów markerowych.

Dziękuję za uwagę!
Document Outline
- Slide 1
- Markery molekularne - podział
- Slide 3
- Selekcja przy użyciu markerów molekularnych
- Slide 5
- Slide 6
- Materiał i metody
- ZASTOSOWANE STARTERY
- Wyniki
- Slide 10
- Slide 11
- Materiał i metody
- Wyniki
- Slide 14
- Slide 15
- Charakterystyka opracowanych markerów
- Slide 17
- Charakterystyka
- Slide 19
- Slide 20
- Materiał i metody
- Wyniki i wnioski
- Slide 23
- Slide 24
- Charakterystyka
- Inne
- Slide 27
- Slide 28
Wyszukiwarka
Podobne podstrony:
CECHY MARKERÓW MOLEKULARNYCH
Kosmos zastosowanie markerów
Markery molekularne
markery 42, Semestr VI, Markery molekularne
DDRT-PCR, Semestr VI, Markery molekularne
Zestawy pytań z ubiegłego roku z Markerów Molekularnych, Semestr VI, Markery molekularne
CECHY MARKERÓW MOLEKULARNYCH
Kosmos zastosowanie markerów
biolo molo yolo zebrane + odp, OGRODNICTWO SGGW, biologia molekularna
Materiały i sprzęt użyte w doświadczeniu, Studia, Ogrodnictwo, Biologia molekularna, Materiały różne
diagnostyka molekularna kandydozy markery
Markery kolagenu i elastyny â zastosowanie w diagnostyce
9 Zastosowanie norm żywienia i wyżywienia w pracy dietetyka
Zastosowanie SEM
Wybrane markery chorb nowotworowych
więcej podobnych podstron