7
mógłby'po eksporcie działać regulująco na genom jądrowy. Zarazem, wbrew poprzednim sugestiom, żadne RNA powstałe z transkrypcji genomu jądrowego nie wnika do mitochondrium poza jedynym znanym wyjątkiem, jakim jest RNA odpowiadające 135 p.z., a stanowiące istotny składnik endonukleazy czynnej w metabolizmie primerowego RNA przy replikacji mtDNA. Natomiast wszystkie syntetazy aminoacylo-tRNA i polimerazy mtDNA i mtRN A są importowane Ho mitochondrium z cytoplazmy.
Tabela 11.5. Produkty genomu mitochondrialnego w różnych typach organizmów
|
Zwierzęta |
Drożdże |
Grzyby |
Rośliny | |
|
Wielkość genomu (kpz) |
14-18 |
78 |
19-108 |
200-2400 |
|
Wielkość genomu (pm) |
5 |
12 |
30 | |
|
rRNA duża podjedn. |
16s |
21s |
21s |
26$ |
|
mała podjedn. |
12s |
15s |
15s |
18s |
|
5s RNA |
— |
— |
— |
+ |
|
tRNA (liczba rodzajów) |
22 |
23-25 |
23-25 |
30 |
|
Rybosomowe białko (var-l) |
— |
+ |
+ |
? |
|
Oksydaza cyt. c, podjedn. 1, 2, 3 |
+ |
+ |
+ |
+ |
|
Oksydored. CoQ-cyt. c (apocytochrom b) |
+ |
+ |
+ |
+ |
|
FoATPaza podjedn. 6, 8 |
+ |
+ |
+ |
+ |
|
podjedn. 9 |
— |
+ |
— |
+ |
|
FiATPaza podjedn. a |
— |
— |
— |
+ |
|
Oksydored. NADH-CoQ (liczba kodowanych podjedn.) |
7 |
Ch- |
6 |
9 |
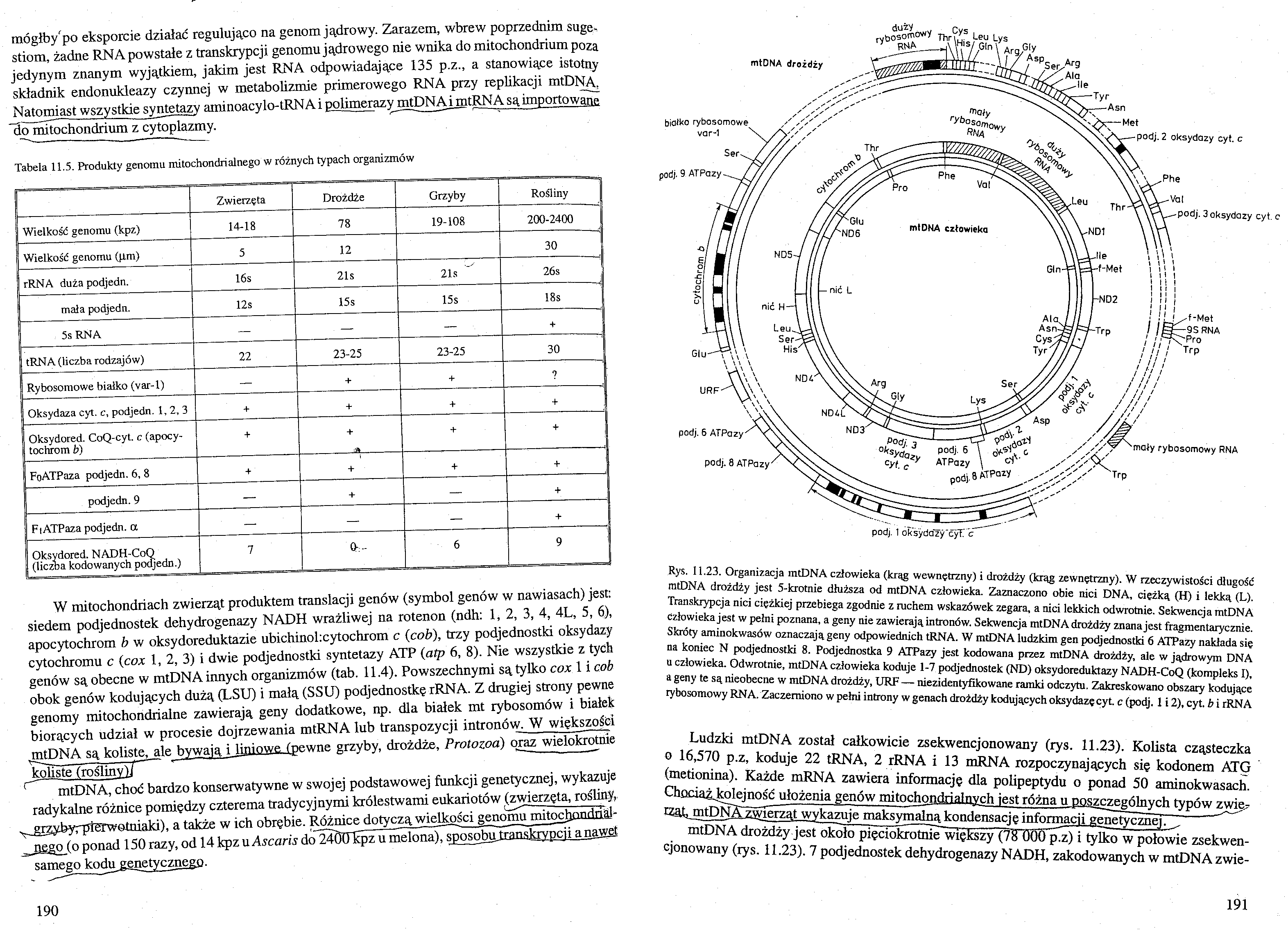
W mitochondriach zwierząt produktem translacji genów (symbol genów w nawiasach) jest: siedem podjednostek dehydrogenazy NADH wrażliwej na rotenon (ndh: 1, 2, 3, 4, 4L, 5, 6), apocytochrom b w oksydoreduktazie ubichinokcytochrom c (cob), trzy podjednostłri oksydazy cytochromu c (cox 1, 2, 3) i dwie podjednostki syntetazy ATP (atp 6, 8). Nie wszystkie z tych genów są obecne w mtDNA innych organizmów (tab. 11.4). Powszechnymi są tylko cox 1 i cob obok genów kodujących dużą (LSU) i małą (SSU) podjednostkę rRNA. Z drugiej strony pewne genomy mitochondrialne zawierają geny dodatkowe, np. dla białek mt rybosomów i białek biorących udział w procesie dojrzewania mtRNA lub transpozycji intronów. W większości .mtDNA są koliste, ale bywają i liniowe (pewne grzyby, drożdże, Protozoa) oraz wielokrotnie koliste (rośliny)J
r mtDNA, choć bardzo konserwatywne w swojej podstawowej funkcji genetycznej, wykazuje radykalne różnice pomiędzy czterema tradycyjnymi królestwami eukariotów (zwierzęta, rośliny,, -^grgybyrpierwetniaki), a także w ich obrębie. Różnice dotyczą wielkości genomu mitochondri&l-
_negojo ponad 150 razy, od 14 kpz u Ascańs do~24(Knćpz u melonahsgosobu transkrypcji a natyst
samego kodu genetycznego.
mtDNA drożdży
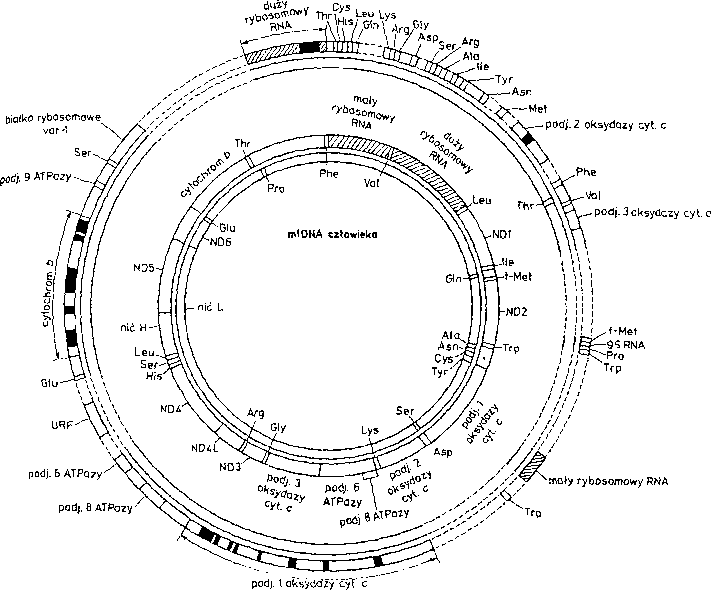
Rys. 11.23. Organizacja mtDNA człowieka (krąg wewnętrzny) i drożdży (krąg zewnętrzny). W rzeczywistości długość mtDNA drożdży jest 5-krotnie dłuższa od mtDNA człowieka. Zaznaczono obie nici DNA, ciężką (H) i lekką (L), Transkrypcja nici ciężkiej przebiega zgodnie z ruchem wskazówek zegara, a nici lekkich odwrotnie. Sekwencja mtDNA człowieka jest w pełni poznana, a geny nie zawierają intronów. Sekwencja mtDNA drożdży znana jest fragmentarycznie. Skróty aminokwasów oznaczają geny odpowiednich tRNA. W mtDNA ludzkim gen podjednostki 6 ATPazy nakłada się na koniec N podjednostki 8. Podjednostka 9 ATPazy jest kodowana przez mtDNA drożdży, ale w jądrowym DNA u człowieka. Odwrotnie, mtDNA człowieka koduje 1-7 podjednostek (ND) oksydoreduktazy NADH-CoQ (kompleks I), a geny te są nieobecne w mtDNA drożdży, URF — niezidentyfikowane ramki odczytu. Zakreskowano obszary kodujące rybosomowy RNA. Zaczerniono w pełni introny w genach drożdży kodujących oksydazę cyt. c (podj. 1 i 2), cyt. b i rRNA
Ludzki mtDNA został całkowicie zsekwencjonowany (rys, 11.23). Kolista cząsteczka o 16,570 p.z, koduje 22 tRNA, 2 rRNA i 13 mRNA rozpoczynających się kodonem ATG (metionina). Każde mRNA zawiera informację dla połipeptydu o ponad 50 aminokwasach. Chodażjcolejność ułożenia genów mitochondrialnych jest różna u poszczególnych typów zwier tZŁt, mtDNSlzffterzat wykazuje maksymalną kondensację informacji genetyczne!. „
mtDNA drożdży jest około pięciokrotnie^ większy (7TO0O^z) i tylko w połowie zsekwencjonowany (rys. 11.23). 7 podjednostek dehydrogenazy NADH, zakodowanych w mtDNA zwie-
191
Wyszukiwarka
Podobne podstrony:
mógłby po eksporcie działać regulująco na genom jądrowy. Zarazem, wbrew poprzednim sugestiom, żadne
Eksport pośredni Eksport pośredni
SKMBT?5007122709250�07 n CZęŚĆ II • DZIAŁANIE ona na sporządzenie długiej listy, gdzie po lewej stro
SKMBT?5007122709250�07 CZĘSC II • DZIAŁANIE ona na sporządzenie długiej listy, gdzie po lewej stroni
noóoi polskiego lotnictwa komunikacyjnego na najbliższy okres" po zakończeniu działań wojennych
85 (153) 85 ich działania polega na tym, że promień padający na reflektor pryzmatyczny po odbiciu wy
Bezpośrednio po zakończeniu działań wojennych we wrześniu i październiku 1939 roku Niemcy nałożyli n
Po rozpoczęciu działania na giełdzie członek giełdy obowiązany jest do wnoszenia opłat przewidzianyc
1 (1) Dlaczego 20 godzin wykładów poświęcamy na 3 godziny po „transporcie”PODSTAWĄ DZIAŁAŃ RATOWNICZ
lastscan6 Instytucja kmlyhmn-dt-po/ytowii jest definiowana jako przedsiębiorstwo, którego działalnoś
wyposażeniem rzeczowym i osobowym, oraz prawo, które reguluje jej działalność, mająca na celu dobro
Ekologia (8) W celu oceny skuteczności działania metronidazolu na Trichomonas sp. umieszczono po 250
CCF20130305�037 39 by»ają ® zajezdni, po wyjeździe z zajez- 39 _;F-:=nowy proces:eks-3 działa obsług
więcej podobnych podstron