
˝ycia
i nanotechnologii
N A G R A N I C Y

W
ubieg∏ym roku Êwi´towaliÊmy 50 rocznic´ odkrycia helikalnej struktury
DNA. Osiàgni´cie Jamesa D. Watsona i Francisa H. Cricka sprowadzi∏o
genetyk´ do chemii i ukierunkowa∏o rozwój biologii na ca∏e pó∏wiecze. DziÊ
tysiàce naukowców wytrwale pracujà nad poznaniem niezliczonych spo-
sobów, na jakie geny kontrolujà rozwój i funkcjonowanie organizmów. Wszystkie te ge-
ny zapisane sà w szczególnym noÊniku danych – DNA.
Zastosowania tej niezwyk∏ej czàsteczki wykraczajà jednak poza ramy biochemii.
Naukowcy, wytwarzajàc i dowolnie zestawiajàc d∏ugie ∏aƒcuchy DNA, wkraczajà na
Êcie˝ki, którymi przyroda nie podà˝a∏a podczas ewolucji ˝ycia. Na przyk∏ad w roku 1994
Leonard M. Adleman z University of Southern California pokaza∏, jak u˝yç DNA do wy-
konywania obliczeƒ [patrz: Leonard M. Adleman „DNA – komputerem”; Âwiat Na-
uki, paêdziernik 1998]. Ja opisz´ inne niebiologiczne zastosowania DNA, takie jak
konstruowanie struktur i urzàdzeƒ, których zasadnicze cz´Êci i mechanizmy majà
rozmiary od jednego do stu nanometrów. Opowiem o nanotechnologii.
Struktury takie majà wiele potencjalnych zastosowaƒ. W sieci krystalicznej wykona-
nej z DNA mo˝na by umieszczaç du˝e czàsteczki biologiczne i metodami krystalogra-
fii rentgenowskiej badaç ich struktur´, co u∏atwi∏oby tzw. racjonalne projektowanie
nowych leków. Sieci DNA mog∏yby te˝ pos∏u˝yç za rusztowania dla elementów nano-
elektronicznych – jako gotowe urzàdzenia lub pomoc w ich produkcji. Pojawià si´ rów-
nie˝ prawdopodobnie nowe tworzywa wykonane albo z DNA, albo przez DNA, któ-
rych struktury dok∏adnie da si´ zaprojektowaç ju˝ na poziomie czàsteczkowym. W
przysz∏oÊci zapewne powstanà z DNA równie˝ maszyny – nanomechaniczne czujniki,
prze∏àczniki i p´sety, a nawet du˝o bardziej z∏o˝one mechanizmy.
Wieloramienny DNA
NANOSKALA
to skala czàsteczek. Typowe wiàzanie mi´dzy dwoma atomami ma oko∏o
0.15 nm d∏ugoÊci. Helisa DNA ma Êrednic´ oko∏o 2 nm, jej skok wynosi 3.5 nm – na
tym odcinku zazwyczaj znajduje si´ 10 par zasad azotowych, które tworzà „szczeble”
drabiny [górna ilustracja na stronie 41]. Krótki fragment nici DNA, zale˝nie od se-
kwencji par zasad, wchodzi w specyficzne interakcje z innymi substancjami chemicz-
nymi. Nici te mo˝na wykorzystaç do rozpoznawania okreÊlonych czàsteczek albo kon-
troli sk∏adu substancji chemicznych przez katalizowanie nimi okreÊlonych reakcji.
Biolodzy od wielu ju˝ lat wykorzystujà zdolnoÊç DNA do rozpoznawania si´ oraz przy-
LIPIEC 2004 ÂWIAT NAUKI
39
DNA
jest wi´cej ni˝ istotà ˝ycia.
To tak˝e uniwersalny materia∏,
z którego mo˝na budowaç maszyny
o rozmiarach nanometrów
Nadrian C. Seeman
DNA SAMORZUTNIE ORGANIZUJE SI¢ w skomplikowane struktury, gdy sekwencje
zasad zaprojektuje si´ tak, by nici ∏àczy∏y si´ w okreÊlony sposób. Ilustracja przedstawia
sztywny szkielet Êci´tego oÊmioÊcianu, który ma osiem szeÊciokàtnych Êcian i szeÊç
kwadratowych. Kraw´dzie majà d∏ugoÊç oko∏o 20 nm. Z ka˝dego wierzcho∏ka wystaje
„spinka” DNA. Spinki mo˝na tak zmodyfikowaç, by po∏àczy∏y Êci´te oÊmioÊciany
i uformowa∏y regularne trójwymiarowe rusztowanie.
KEN EW
ARD
BioGrafx
(na gór
ze
); ALICE Y
. CHEN (
na dole
)

∏àczania fragmentów z odpowiadajàcymi sobie „lepkimi koƒ-
cami”. Lepki koniec powstaje, gdy jedna niç helisy jest d∏u˝-
sza o kilka niesparowanych zasad od drugiej [dolna ilustracja
na stronie 41]. LepkoÊç to sk∏onnoÊç takiego wystajàcego od-
cinka DNA do wiàzania si´ z nicià, która ma ustawione we
w∏aÊciwym szyku komplementarne zasady: adenina jednej
nici wià˝e si´ z tyminà przeciwleg∏ej, cytozyna wià˝e si´ z
guaninà [patrz: Stephen H. Friend i Roland B. Stoughton
„Magiczne mikromacierze”; Âwiat Nauki, kwiecieƒ 2002].
Na pierwszy rzut oka nie wydaje si´, by DNA móg∏ uk∏adaç
si´ w interesujàce struktury. Spotykany w przyrodzie formu-
je liniowe ∏aƒcuchy podobne do d∏ugich kawa∏ków szpagatu.
Wszystko wi´c, co mo˝na sobie wyobraziç, to odcinki, okr´gi
lub co najwy˝ej splàtane k∏´bki nici. Ale DNA przybiera nie
tylko form´ liniowà. Podczas pewnych procesów komórko-
wych przez krótkà chwil´ ma kszta∏t rozga∏´zionej czàsteczki.
Dzieje si´ tak w czasie replikacji DNA (w fazie przygotowaƒ do
podzia∏u komórki) oraz rekombinacji (kiedy materia∏ gene-
tyczny jest wymieniany mi´dzy parami chromosomów, np. w
trakcie produkcji plemników i komórek jajowych).
Rozga∏´zienia powstajà, gdy podwójna helisa cz´Êciowo
si´ rozplàtuje. Podczas replikacji z ka˝dej nici tworzy si´
nowa podwójna helisa – na ca∏ej d∏ugoÊci nici do∏àczane sà
komplementarne nukleotydy (nukleotyd to zasada azotowa i
odpowiadajàcy jej fragment szkieletu helisy). Bardziej inte-
resujàcy jest jednak proces crossing-over, który zachodzi
podczas rekombinacji, gdy dwa fragmenty DNA sà rozrywa-
ne i cz´Êciowo rozplàtywane, a powsta∏e w ten sposób czte-
ry nici ∏àczà si´, tworzàc struktur´ podobnà do skrzy˝owa-
nia dwóch autostrad.
W czasie rekombinacji DNA rozga∏´zienie pojawia si´ w
miejscu, w którym nast´puje wymiana nici mi´dzy ∏aƒcu-
chami. Skrzy˝owanie to przemieszcza si´, poniewa˝ uk∏ad
otaczajàcych je sekwencji zasad azotowych jest symetryczny
(z∏o˝enie dwóch symetrii osiowych o osiach przecinajàcych
si´). Symetria sprawia, ˝e ka˝da z nici pierwszej czàsteczki
DNA mo˝e po∏àczyç si´ z jednà z dwóch nici drugiej czàstecz-
ki. Kiedy w roku 1979 wraz Bruce’em H. Robinsonem, obec-
nie w University of Washington, pracowa∏em nad opisem isto-
ty tego ruchu, dostrzeg∏em, ˝e syntetyczne czàsteczki DNA, któ-
rym brak tej symetrii, mogà formowaç czàsteczki o nierucho-
mych punktach rozga∏´zienia. Budowa takiego w´z∏a wymaga
stworzenia czterech nici DNA. Sekwencja jednej po∏owy
ka˝dej z nici powinna odpowiadaç sekwencji po∏owy drugiej
nici, a pozosta∏a sekwencja pierwszej nici powinna byç kom-
plementarna do sekwencji po∏owy trzeciej nici [dolna ilustra-
cja na stronie obok].
W przyrodzie DNA najch´tniej uk∏ada si´ w konwencjo-
nalnà podwójnà helis´, t´ opisanà przez Watsona i Cricka.
Czynnikiem rozstrzygajàcym, która ze struktur powstanie,
jest wielkoÊç zwana energià swobodnà. Na ogó∏ energia ta
determinuje kierunek przebiegu reakcji chemicznej. Decydu-
je te˝ o konformacji – zagi´ciach i po∏àczeniach – du˝ych czà-
steczek, takich jak DNA, RNA i bia∏ka. Uk∏ad chemiczny za-
wsze dà˝y do stanu o najni˝szej energii swobodnej. Dwie
komplementarne nici nukleotydów majà najmniejszà ener-
gi´, gdy sparujà si´ w podwójnà helis´.
Cztery pojedyncze nici naszego nieruchomego skrzy˝owa-
nia utworzà najd∏u˝szà konwencjonalnà podwójnà helis´ DNA
tylko wtedy, gdy po∏àczà si´ w rozga∏´zione moleku∏y. Zazwy-
czaj rozga∏´zienia nie przynoszà korzyÊci czàsteczce, zwi´k-
szajà bowiem jej energi´ swobodnà. W rozwa˝anym przypad-
ku wzrost ten jest jednak nieznaczny w porównaniu z
oszcz´dnoÊcià energii, jaka wynika z uformowania czterech
ramion podwójnej helisy DNA. Wspó∏czeÊnie mo˝na ∏atwo syn-
tetyzowaç nici o dowolnych sekwencjach zasad i realizowaç
idee stabilnych rozga∏´zionych moleku∏ DNA. W roku 1979 by-
∏a to jednak chemia najwy˝szych lotów, a ja by∏em krystalo-
grafem, nie chemikiem organicznym, tak wi´c o sieciach kry-
stalicznych DNA mog∏em sobie jedynie rozmyÊlaç (samodzielnie
wytwarzaç DNA nauczy∏em si´ dopiero w roku 1982).
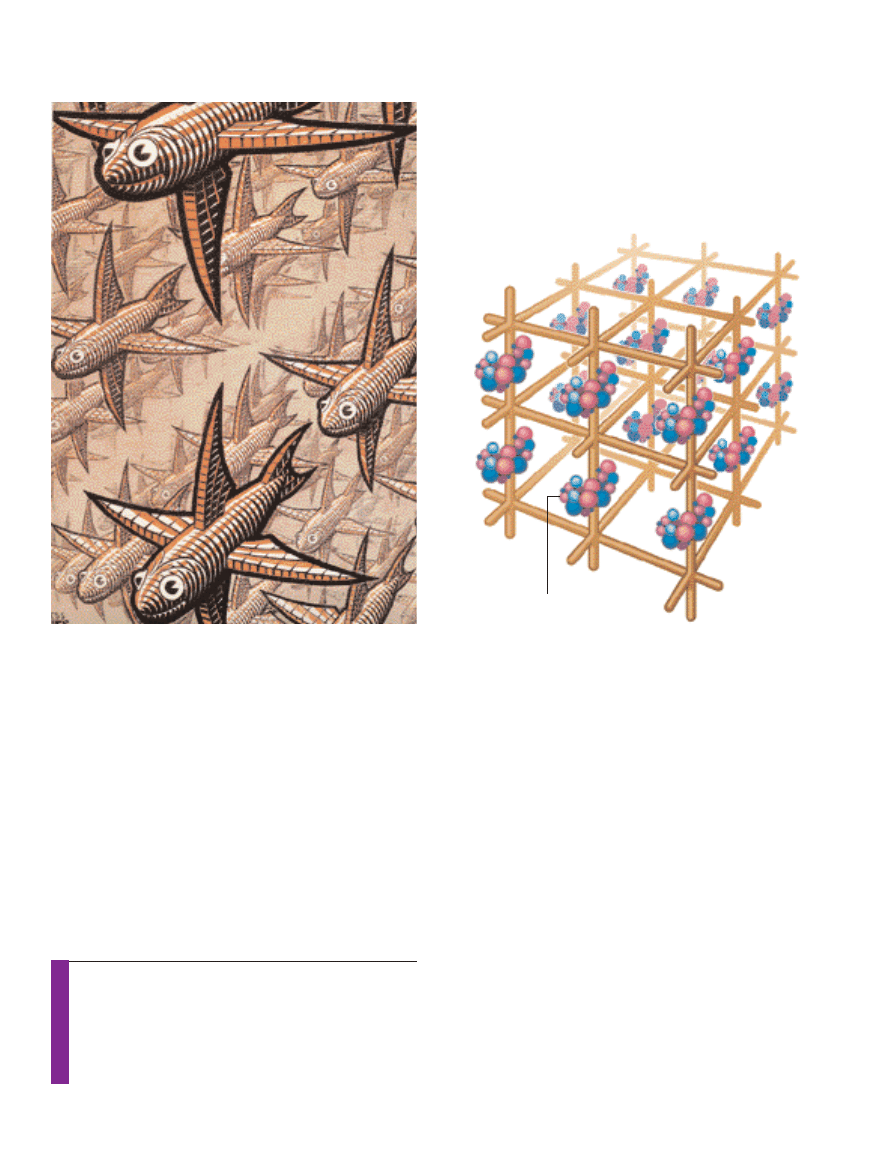
Inspiracja Escherem
DOSZED
¸EM DO WNIOSKU
, ˝e mo˝liwe powinno byç utworzenie
rozga∏´zionego DNA z wieloma ramionami, nie tylko z czte-
rema. Pewnego jesiennego dnia 1980 roku wybra∏em si´ do
pubu w miasteczku uniwersyteckim. Z jakiegoÊ powodu my-
Êla∏em o drzeworycie holenderskiego artysty M. C. Eschera
G∏´bia [ilustracja na stronie 42]. UÊwiadomi∏em sobie, ˝e Êro-
dek cia∏a ka˝dej z ryb na obrazie przypomina wyidealizowa-
ny punkt skrzy˝owania szeÊciu ramion DNA. Z korpusu ryby
symetrycznie wyrastajà cztery p∏etwy: grzbietowa, brzuszna,
lewa i prawa. Ryby u∏o˝one sà jak czàsteczki w krysztale –
ten sam wzór powtarza si´ we wszystkich kierunkach. Pomy-
Êla∏em, ˝e gdybym spoi∏ rozga∏´zienia za pomocà lepkich koƒ-
ców, móg∏bym uporzàdkowaç materi´ w skali nanometrów
podobnie jak Escher swojà ∏awic´ ryb w wyobraêni.
Jest kilka powodów, dla których warto budowaç takie struk-
tury. Po pierwsze, chcemy wytwarzaç makroskopowe frag-
menty tworzyw sk∏adajàcych si´ z czàsteczek uprzednio zapro-
jektowanych i osadzonych w strukturze z nanometrowà
dok∏adnoÊcià. Ta technika mo˝e zaowocowaç tworzywami o
40
ÂWIAT NAUKI LIPIEC 2004
n
DNA doskonale nadaje si´ do budowania struktur w nanoskali.
Nici DNA mo˝na zaprogramowaç tak, by same organizowa∏y si´
w z∏o˝one uk∏ady. Komplementarne zasady azotowe dwóch nici
samorzutnie i przewidywalnie ∏àczà si´ ze sobà, tworzàc
podwójne helisy.
n
W rusztowaniach z DNA planuje si´ umieszczaç inne czàsteczki
i za pomocà krystalografii badaç ich struktur´. Mo˝na do nich
te˝ wk∏adaç urzàdzenia elektroniczne o rozmiarach moleku∏
albo wykorzystaç je do konstruowania tworzyw o okreÊlonych
uk∏adach czàsteczek.
n
Dzia∏anie nanomaszyn opiera si´ na zmieniajàcych konformacje
elementach z DNA. Ruch ten mo˝na kontrolowaç za pomocà
czynników chemicznych lub specjalnych nici DNA.
Przeglàd /
Nanotechnologia DNA
¸A¡CUCHY DNA
wchodzà w niezwykle przewidywalne interakcje,
a ich ró˝norodnoÊç daje swobod´
W PROJEKTOWANIU CZÑSTECZEK
.
nowych w∏aÊciwoÊciach lub nowymi zestawieniami w∏aÊci-
woÊci. Na przyk∏ad materia∏y o wybranych i zaprojektowa-
nych cechach optycznych, takie jak kryszta∏y fotoniczne, mo-
g∏yby powstawaç na bazie precyzyjnie zdefiniowanych matryc
[patrz: Eli Yablonovitch „Kryszta∏y fotoniczne – pó∏przewod-
niki Êwiat∏a”; Âwiat Nauki, luty 2002].
Drugim celem jest zastosowanie DNA jako rusztowania pod-
trzymujàcego inne czàsteczki, a wÊród nich te, które nie two-
rzà samodzielnie struktury krystalicznej. W eksperymentach
krystalograficznych przydatne mogà byç sieci konstruowane
z „klatek” z DNA, wewnàtrz których mieÊci∏yby si´ du˝e czà-
steczki biologiczne, na przyk∏ad bia∏ka [ilustracja na nast´pnej
stronie]. Klatki umo˝liwi∏yby krystalografom ustalenie trójwy-
miarowej struktury unieruchomionych w nich moleku∏. Tym
samym u∏atwi∏yby proces wytwarzania leków, które przy∏àcza-
jà si´ do okreÊlonych fragmentów czàsteczek i bezpoÊrednio
na nie oddzia∏ujà. (W∏aÊnie mo˝liwoÊç zastosowania DNA w
krystalografii najbardziej motywowa∏a mnie do dalszej pracy
LIPIEC 2004 ÂWIAT NAUKI
41
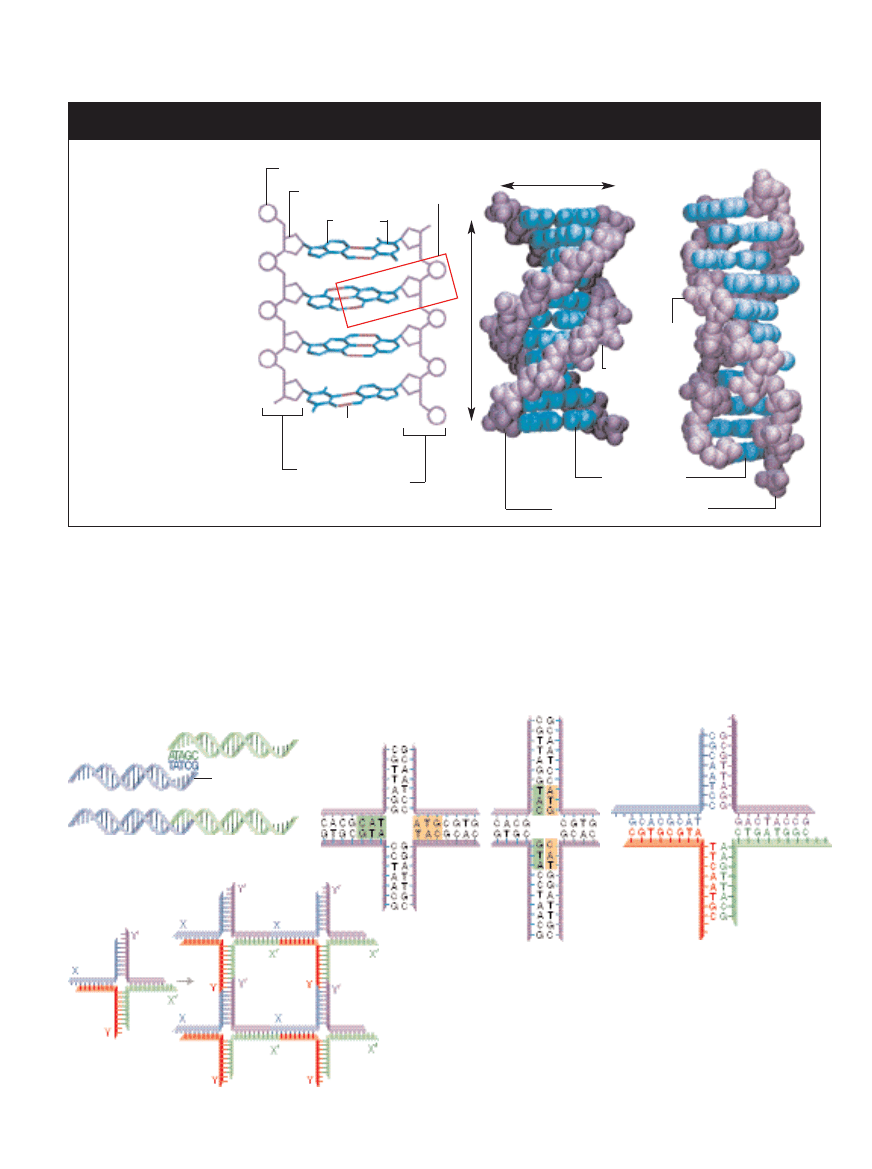
CZÑSTECZKA DNA ma
nanometrowe rozmiary.
Sk∏ada si´ z dwóch
szkieletów cukrowo-
-fosforanowych, pomi´dzy
którymi znajdujà si´ pary
zasad komplementarnych
(A i T oraz C i G),
po∏àczonych wiàzaniami
wodorowymi (z lewej).
Najcz´Êciej w przyrodzie
wyst´puje konformacja
B-DNA (poÊrodku), która
zwija si´ w podwójnà
prawoskr´tnà helis´
o Êrednicy 2 nm. Jeden
skok helisy to oko∏o 3.5 nm
(czyli 10–10.5 par zasad).
W szczególnych
okolicznoÊciach DNA
mo˝e utworzyç lewoskr´tnà
podwójnà helis´ zwanà
Z-DNA (z prawej).
2.0 nm
3.5 nm
B-DNA
Nukleotyd
Zasady
azotowe
Grupa fosforanowa
Deoksyryboza
Wiàzania
wodorowe
mi´dzy zasadami
Z-DNA
STRUKTURA DNA
Zasady azotowe
Helisa
prawoskr´tna
Helisa
lewoskr´tna
Szkielet cukrowo-fosforanowy
Szkielet
cukrowo-fosforanowy
a
b
c
d
e
Lepki koniec
SK¸ONNOÂå do wiàzania si´ nici o komplementarnych sekwencjach zasad
umo˝liwia samoorganizacj´ DNA w z∏o˝one struktury. Do ∏àczenia poszczególnych
segmentów wykorzystuje si´ tzw. lepkie koƒce (a), krótkie odcinki niesparowa-
nych nici wystajàce z koƒców czàsteczki DNA. Drugim bardzo wa˝nym elemen-
tem konstrukcyjnym jest rozga∏´ziony DNA (b), który w punkcie rozwidlenia spa-
ja co najmniej trzy helisy. W naturalnie rozga∏´zionym DNA punkt rozwidlenia mo˝e
si´ przemieszczaç (c), poniewa˝ sekwencje zasad na czterech ramionach sà sy-
metryczne. Pozbawiony tej symetrii syntetyczny DNA ma nieruchomy punkt roz-
widlenia (d). Kopie rozga∏´zionego DNA wyposa˝one w komplementarne lepkie
koƒce (e) samodzielnie uk∏adajà si´ w struktur´ sieci krystalicznej.
KEN EW
ARD
BioGrafx
(na gór
ze
); ALICE Y
. CHEN (
na dole
)
w tej dziedzinie). Na razie wielu czàsteczek receptorowych,
doskona∏ych punktów uchwytu dla leków, nie da si´ badaç me-
todami tradycyjnej krystalografii. W sieci DNA mo˝na by∏oby
równie˝ umieszczaç elementy nanoelektroniczne, otrzymujàc
bardzo ma∏e uk∏ady pami´ci, co zasugerowa∏em z Robinso-
nem w roku 1987. Co prawda, mój zespó∏ nie u˝y∏ jeszcze DNA
jako rusztowania, ale odnieÊliÊmy wiele sukcesów, które przy-
bli˝ajà nas do tego celu.
Dlaczego chcemy to wszystko osiàgnàç za pomocà DNA?
Przede wszystkim dlatego, ˝e jego nici oddzia∏ujà ze sobà w
wyjàtkowo dobrze programowalny i przewidywalny sposób.
W lepkim koƒcu d∏ugoÊci N zasad zasady te sà u∏o˝one na je-
den z 4
N
mo˝liwych sposobów. Niezwyk∏a ró˝norodnoÊç oraz
sk∏onnoÊç koƒców do wiàzania si´ wy∏àcznie z odpowiadajà-
cymi im sekwencjami zasad pozwalajà na swobodne projek-
towanie czàsteczek, które b´dà z∏o˝one z wielu nici DNA w pe∏-
ni zaplanowany sposób. Na dodatek wiemy, ˝e gdy dwa lepkie
koƒce zwià˝à si´ ze sobà, uformujà klasycznà struktur´ po-
dwójnej helisy DNA i b´dzie one relatywnie sztywna. Mo˝e-
my zatem przewidzieç nie tylko, które fragmenty nici przylgnà
do siebie, ale tak˝e dok∏adny kszta∏t powsta∏ych segmentów.
Tak dok∏adnymi informacjami nie dysponujemy w przypadku
bia∏ek i przeciwcia∏, innych kandydatów na elementy kon-
strukcyjne w nanoin˝ynierii, poniewa˝ za ka˝dym razem trze-
ba ustalaç, jak dwa bia∏ka lub przeciwcia∏a si´ po∏àczà albo
jaki te pierwsze przyjmà kszta∏t.
DNA u˝ywamy równie˝ ze wzgl´du na prostot´ jego synte-
zy za pomocà narz´dzi przemys∏u biotechnologicznego. Mo-
˝emy nim manipulowaç, u˝ywajàc wielu enzymów, takich jak
enzymy restrykcyjne (które przecinajà nici w okreÊlonych
miejscach) albo ligazy (katalizujàce proces ∏àczenia dwóch
czàsteczek wiàzaniem kowalencyjnym – silnym wiàzaniem
chemicznym, które uwspólnia elektrony dwóch atomów).
Enzymy te wykorzystuje si´ przy tworzeniu (i póêniejszych
manipulacjach) zarówno konwencjonalnego DNA, jak i jego
egzotycznych pochodnych, które zawierajà inne ni˝ cztery
podstawowe zasady azotowe lub do których (po bokach dra-
biny DNA) do∏àczone sà dodatkowe czàsteczki. Naukowcy
majàcy nadziej´ na zastosowanie kwasów nukleinowych (DNA
i RNA) w medycynie stworzyli wiele takich czàsteczek. DNA
wyjàtkowo dobrze nadaje si´ do tego typu modyfikacji, po-
niewa˝ w ka˝dym nukleotydzie helisy znajdujà si´ miejsca,
do których mogà zostaç przymocowane inne czàsteczki.
Warto wreszcie wspomnieç, ˝e – jak zobaczymy dalej –
DNA mo˝na sk∏oniç do utworzenia struktury innej ni˝ stan-
42
ÂWIAT NAUKI LIPIEC 2004
G¸¢BIA
, M.C. ESCHER
. © 2004 THE M. ESCHER COMP
ANY
. WSZYSTKIE PRA
W
A
ZASTRZE˚ONE (
z lewej
); JEN CHRISTIANSEN (
z prawej
)
DRZEWORYT ESCHERA G∏´bia zainspirowa∏ autora artyku∏u do zbudo-
wania sieci z szeÊcioramiennych rozga∏´zieƒ tworzàcych trójwymiarowy
kryszta∏ (poni˝ej). Ârodek ka˝dej ryby przypomina szeÊcioramienne z∏à-
cze. Zamiast ramion z punktu centralnego wyrastajà g∏owa i ogon oraz
cztery p∏etwy: grzbietowa i brzuszna, lewa i prawa. Rusztowanie mo˝e
utrzymywaç w regularnej sieci inne czàsteczki. Na przyk∏ad klatki DNA
zawierajàce chemicznie zorientowane makroczàsteczki biologiczne mo-
gà byç u˝ywane w doÊwiadczeniach krystalograficznych. W podobny
sposób elementy uk∏adów nanoelektronicznych da si´ grupowaç w bar-
dzo ma∏e uk∏ady pami´ci.
Makroczàsteczka
NADRIAN C. „NED” SEEMAN jest krystalografem. Zniech´cony
nieudanymi próbami krystalizacji makroczàsteczkowej, wpad∏ na
pomys∏ wykorzystania rozga∏´zieƒ DNA i przystàpi∏ do jego realiza-
cji. Seeman pracuje od 16 lat na Wydziale Chemii w New York Uni-
versity. Kiedy w po∏owie lat osiemdziesiàtych powiedziano mu, ˝e
to, czym si´ zajmuje, nazywa si´ nanotechnologià, poczu∏ si´ tak jak
Jourdain, g∏ówny bohater Mieszczanina szlachcicem Moliera, gdy si´
dowiedzia∏, ˝e przez ca∏e ˝ycie mówi∏ prozà.
O
AUTORZE
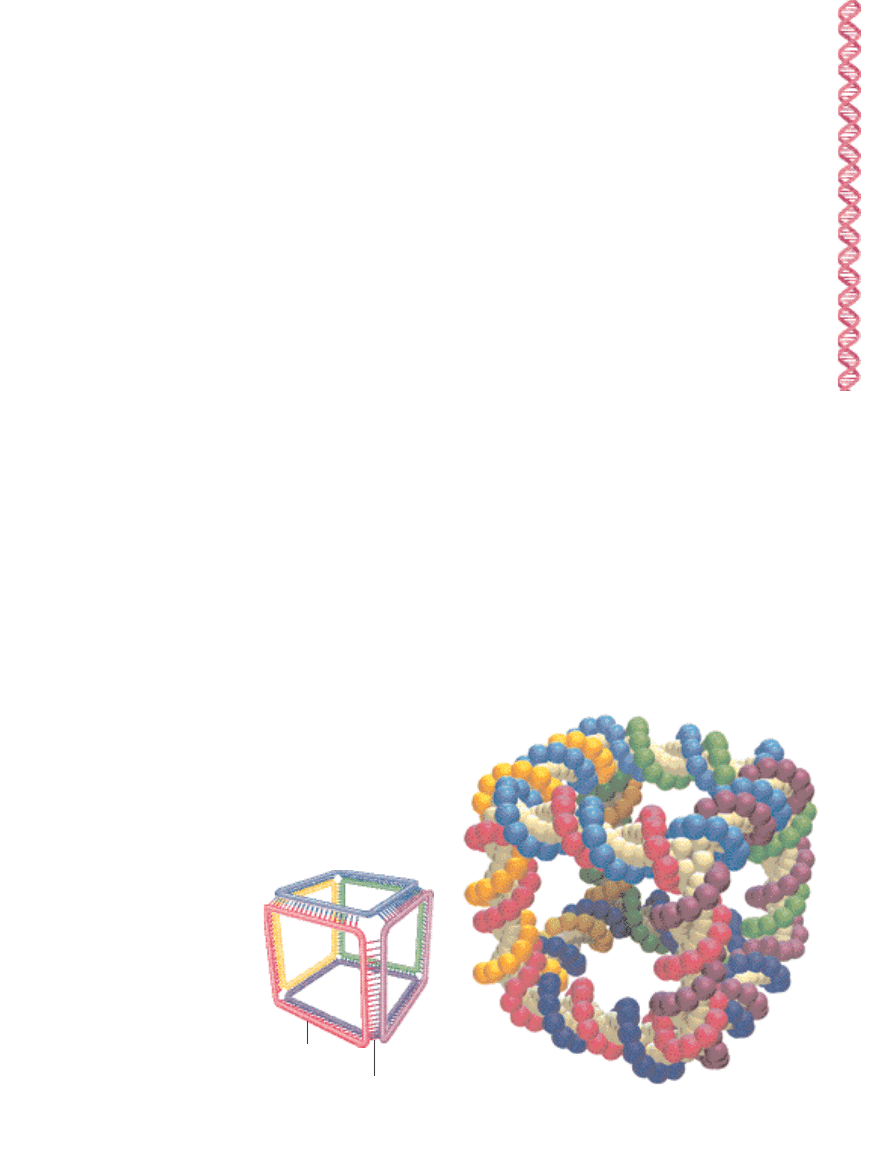
dardowa podwójna helisa. Udaje si´ nawet budowaç urzà-
dzenia nanomechaniczne, których cz´Êci si´ poruszajà (takie
jak zaciskajàce si´ p´sety lub obracajàce wa∏ki), gdy nast´-
puje przejÊcie z jednej konformacji DNA do drugiej. Jedna
z wad takich uk∏adów polega na tym, ˝e trzeba je konstru-
owaç w roztworach wodnych. Osuszenie gotowych struktur
(np. na p∏ytce z miki) nie sprawia nam jednak k∏opotu, tak
jak wtedy gdy chcemy otrzymaç mikroskopowe obrazy wy-
ników naszych badaƒ.
Sztywne modele
PIERWSZYM ETAPEM
ka˝dego naukowego projektu badawcze-
go jest ustalenie jego wykonalnoÊci. Uda∏o si´ to zrobiç w ro-
ku 1991, gdy wraz z Junghueiem Chenem, obecnie w Uni-
versity of Delaware, zbudowaliÊmy czàsteczk´ DNA w kszta∏cie
szkieletu szeÊcianu [ilustracja poni˝ej]. Kraw´dzie szeÊcianu
sà utworzone przez odcinki podwójnej helisy DNA, a wierz-
cho∏ki – przez trójramienne rozga∏´zienia. Ka˝dy wierzcho-
∏ek po∏àczony jest z trzema innymi – mówi si´, ˝e zdolnoÊç
przy∏àczeniowa szeÊcianu wynosi trzy. In˝ynierowie gene-
tyczni stworzyli wiele liniowych konstrukcji DNA, lecz nasza
kostka jest pierwszà czàsteczkà DNA majàcà zdolnoÊç przy-
∏àczeniowà wi´kszà od dwóch. SzeÊcian montuje si´ samo-
rzutnie z odcinków DNA zaprojektowanych tak, by do siebie
przywar∏y, lecz ich koƒce pozosta∏y swobodne. Mogà je spo-
iç ligazy, dajàc szeÊç zamkni´tych p´tli, po jednej na Êcian´.
Poniewa˝ DNA jest helisà, ka˝da p´tla oplata si´ wokó∏ p´tli
do niej przylegajàcej. SzeÊcian nie mo˝e si´ zatem rozpaÊç
nawet wtedy, gdy wszystkie wiàzania mi´dzy zasadami azo-
towymi zostanà rozerwane.
Razem z Yuwenem Zhangiem, dziÊ pracownikiem Baxter
Healthcare, skonstruowa∏em obiekt w kszta∏cie szkieletu Êci´-
tego oÊmioÊcianu, podobnego nieco do szeÊcianu, lecz bar-
dziej skomplikowanego [ilustracja na stronie 38]. Chocia˝
trójramienne rozga∏´zienia wystarczy∏yby do stworzenia po-
jedynczego Êci´tego oÊmioÊcianu, zbudowaliÊmy go, u˝ywa-
jàc rozga∏´zieƒ czteroramiennych. ChcieliÊmy, by dodatko-
we ramiona wystajàce z ka˝dego wierzcho∏ka pos∏u˝y∏y do
po∏àczenia oÊmioÊcianów w wi´kszà struktur´, ale w koƒcu
nie poszliÊmy w tym kierunku. StworzyliÊmy niewielkà licz-
b´ Êci´tych oÊmioÊcianów – wystarczajàcà do opisania ich
struktury, lecz zbyt ma∏à, by podjàç prób´ ich po∏àczenia. Na-
wet ta niewielka próbka okaza∏a si´ kresem naszych mo˝liwo-
Êci. Nie osiàgn´libyÊmy wi´cej bez znacznego ulepszenia na-
szych metod badawczych (np. automatyzacji powtarzalnych
czynnoÊci). ZabraliÊmy si´ wi´c za prostsze podzespo∏y.
Kierunek badaƒ zmieniliÊmy równie˝ dlatego, ˝e nasze wie-
loÊciany nie by∏y sztywne. Samo DNA jest sztywnà czàstecz-
kà: odcinek d∏ugoÊci dwóch lub trzech skoków helisy (z ta-
kich w∏aÊnie konstruowaliÊmy kraw´dzie wieloÊcianów)
odchyla si´ od pionu nie bardziej ni˝ niç ugotowanego spaghet-
ti d∏ugoÊci 2–3 mm. To zapewnia∏o sztywnoÊç kraw´dziom
naszych wieloÊcianów, zaobserwowaliÊmy jednak niestabil-
noÊç kàtów przy wierzcho∏kach. Skonstruowane przez nas
wieloÊciany przypomina∏y budowle z wetkni´tych w ˝elki
wyka∏aczek. Takie struktury prawdopodobnie znalaz∏yby za-
stosowania, ale nie do budowy regularnych sieci. Du˝o ∏a-
twiej z∏o˝yç coÊ na kszta∏t kryszta∏u z elementów bardziej
przypominajàcych cegie∏ki ni˝ ˝elki.
Aby rozwiàzaç ten problem, moja grupa zaj´∏a si´ innym,
znanym z rekombinacji uk∏adem z rozga∏´zieniem – podwójnie
przeplecionà czàsteczkà DNA (DX – double-crossover DNA).
Czàsteczka DX sk∏ada si´ z dwóch u∏o˝onych obok siebie od-
cinków podwójnych helis, których nici krzy˝ujà si´ mi´dzy so-
bà, ∏àczàc obie helisy [ilustracja w ramce na nast´pnej stronie].
Okaza∏o si´, ˝e czàsteczka ta jest sztywna. DowiedliÊmy te˝,
˝e bardzo sztywna jest czàsteczka DX, która dodatkowo zawie-
ra jeden ma∏y fragment podwójnej helisy (nazwany DX + J). Ten
LIPIEC 2004 ÂWIAT NAUKI
43
KEN EW
ARD
BioGrafx
(z lewej
); ALICE Y
. CHEN (
z prawej
)
Skonstruowane przez nas
WIELOÂCIANY
przypomina∏y
raczej
BUDOWLE Z WYKA¸ACZEK
wetkni´tych w ˝elki.
SZTYWNY SZEÂCIAN (z prawej)
wykonany z szeÊciu p´tli DNA jest
dowodem na to, ˝e z DNA mo˝na
zbudowaç struktur´ trójwymiaro-
wà. Na rysunku szkielet ka˝dej ni-
ci reprezentujà kolorowe kule (ka˝-
dej nici przypisano inny kolor),
zasady przedstawiono jako kule bia-
∏e. Wszystkie kraw´dzie szeÊcien-
nej kostki sk∏adajà si´ z 20 par nu-
kleotydów, odpowiadajàcych mniej
wi´cej dwóm skokom podwójnej
helisy. W wierzcho∏kach kostki sà
trójramienne rozga∏´zienia. Sposób
po∏àczenia nici DNA bez uwzgl´d-
nienia skr´tów helisy przedstawia
uproszczony schemat (z lewej).
Para zasad
Szkielet DNA
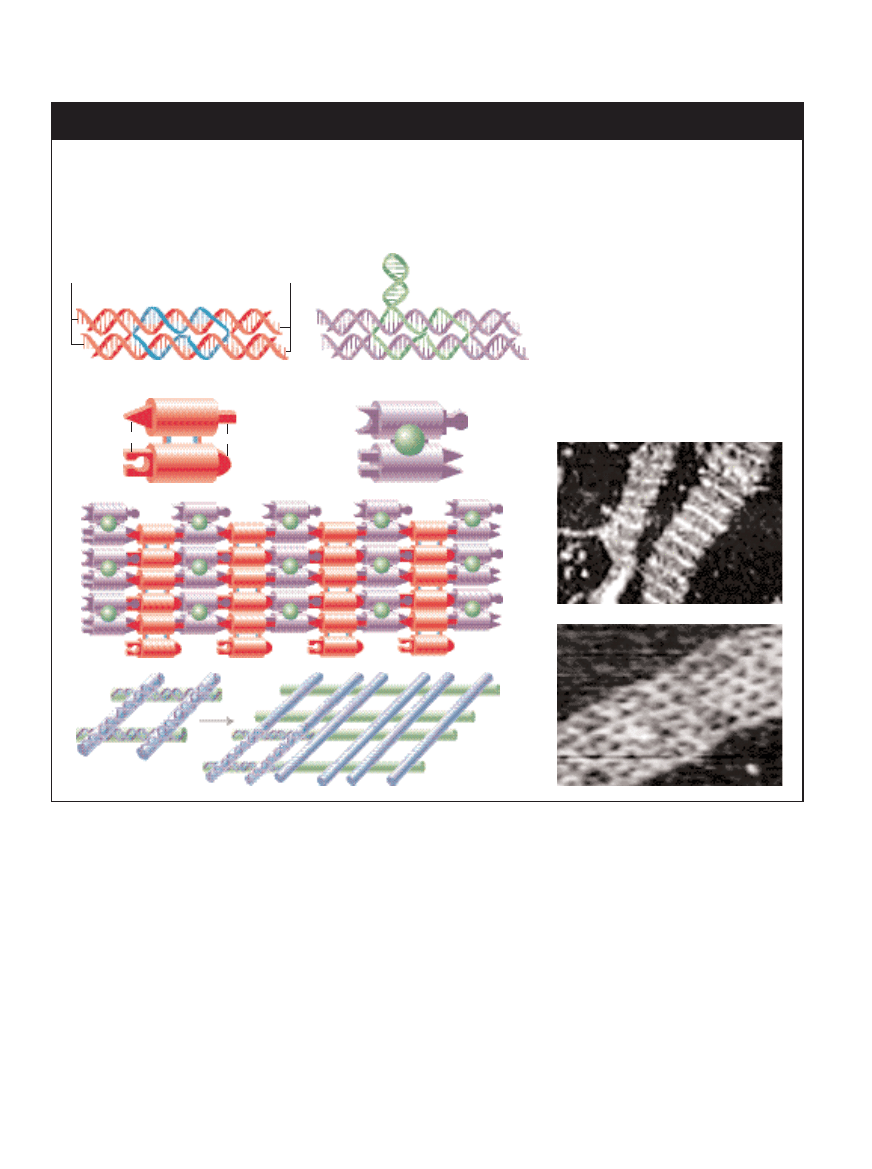
krótki podwójnie helikalny odcinek tworzy wybrzuszenie na
powierzchni czàsteczki DX i s∏u˝y jako marker.
Erik Winfree z California Institute of Technology wraz z
Furongiem Liu i Lisà A. Wenzler z mojej grupy z New York
University u˝yli zestawów czàsteczek DX i DX + J jako ka-
felków do tworzenia dwuwymiarowych kryszta∏ów o zdefi-
niowanych wczeÊniej wzorach. Kafelki sà po∏àczone za po-
mocà lepkich koƒców ka˝dej z helis. W jednym z ustawieƒ, w
którym kolumny kafelków DX le˝à na przemian z kolumna-
mi DX + J, otrzymaliÊmy paski oddalone od siebie o oko∏o
32 nm. By sprawdziç, ˝e struktury majà w∏aÊciwe wymiary,
umieÊciliÊmy je na mice i zbadaliÊmy za pomocà mikroskopu
si∏ atomowych. Wytwarzajàc drugi kryszta∏, tym razem ze
zmodyfikowanych kafelków, w którym trzy kolumny DX przy-
pada∏y na jednà kolumn´ DX + J, przekonaliÊmy si´, ˝e wzór
ten nie by∏ dzie∏em przypadku. W ten sposób otrzymaliÊmy
paski rozmieszczone dwa razy rzadziej. Zespó∏ Johna H. Reifa
z Duke University zademonstrowa∏ niedawno uzyskane w po-
dobny sposób „kody kreskowe DNA”. W mozaikach tych u∏o-
˝enie pasków zosta∏o zaprogramowane tak, by przedstawia-
∏o wzór „01101” (z czàsteczkami analogicznymi do DX i
DX + J odpowiadajàcymi zerom i jedynkom). Wzór zosta∏ za-
programowany przy u˝yciu nici DNA, której sekwencja ko-
dowa∏a ciàg 01101. Odpowiedniki kafelków DX i DX + J u∏o-
˝y∏y si´ samorzutnie wzd∏u˝ odcinka nici DNA, przylegajàc do
zakodowanych na niej zer i jedynek. Nast´pnie wiele takich
pi´ciokafelkowych segmentów po∏àczy∏o si´ równolegle, two-
rzàc z pasków wzór 01101. Paski oddalone by∏y od siebie
o oko∏o 15 nm. Pod mikroskopem si∏ atomowych odczytuje
si´, korzystajàc z kodu paskowego, zakodowane na nici DNA
dane wejÊciowe. Ta wizualna metoda rozpoznawania sekwen-
cji DNA mo˝e znacznie przyÊpieszyç faz´ odczytu w oblicze-
44
ÂWIAT NAUKI LIPIEC 2004
ALICE Y
. CHEN (
ilustracje
); NADRIAN C. SEEMAN (
mikr
ofotografia
)
KRYSZTA¸Y DWUWYMIAROWE mo˝na budo-
waç ze sztywnych „kafelków” DNA (a). Sà to
modu∏y podwójnie przeplecione (DX) oraz mo-
du∏y podwójnie przeplecione ze skrzy˝owa-
niem (DX + J). Takie modu∏y, w przeciwieƒ-
stwie do wieloramiennych, nie wyginajà si´ w
punktach z∏àczeƒ. Aby kafelki mog∏y si´ ze
sobà ∏àczyç, zosta∏y wyposa˝one w zestawy
czterech ró˝nych lepkich koƒców. Wyd∏u˝ona
zielona niç czàsteczki DX + J wystaje ponad
powierzchni´. Ka˝dy z modu∏ów ma wymia-
ry oko∏o 4 na 16 nm. Dla uproszczenia DX i
DX + J zosta∏y przedstawione schematycz-
nie. Geometryczne koƒcówki symbolizujà lep-
kie koƒce (b). Gdy czàsteczki sà zanurzone
w roztworze, zlepiajà si´ i uk∏adajà samo-
rzutnie w dwuwymiarowy wzór (c). Z obra-
zu wykonanego mikroskopem si∏ atomowych
wy∏ania si´ paskowany deseƒ (d) (na po-
trzeby mikroskopii kryszta∏ zosta∏ umieszczo-
ny na p∏ytce z miki). Jasne, oddalone o oko-
∏o 32 nm paski zosta∏y utworzone przez DNA
wystajàcy z modu∏ów DX + J. Równie˝ rów-
noleg∏oboki DNA uk∏ada∏y si´ samoczynnie
w dwuwymiarowe wzory (e, f).
a
b
c
e
d
f
SZTYWNE SIECI DNA
Lepkie koƒce
Lepkie koƒce
Lepkie koƒce
Modu∏ podwójnie przepleciony
ze skrzy˝owaniem
Modu∏ podwójnie przepleciony
Lepkie koƒce

niach czàsteczkowych z wykorzystaniem DNA. Mo˝e te˝ byç
zastosowana do mapowania mutacji.
Wraz z Chengde Mao, który teraz jest zatrudniony w Pur-
due University, wykonaliÊmy dwuwymiarowe siatki z równo-
leg∏oboków DNA podobnych do naszych szkieletów wielo-
Êcianów. Dajà si´ one po∏àczyç w kryszta∏, który przypomina
wafel. WielkoÊç „oczek” tej struktury mo˝na regulowaç, zmie-
niajàc rozmiary równoleg∏oboków. Pojedyncze rozga∏´zienia
sà wiotkie, ale uk∏adajàc po cztery z nich w ka˝dym wierz-
cho∏ku równoleg∏oboku, uzyskaliÊmy dobrze sprawujàcy si´
element sieci.
Nanomaszyny
KLUCZOWE ZNACZENIE
w nanotechnologii majà nanomaszyny.
DNA okaza∏ si´ niezwykle przydatny w konstruowaniu takich
urzàdzeƒ. ZbudowaliÊmy kilka maszyn z DNA, lecz w arty-
kule skupimy si´ tylko na dwóch, które majà dobrze zdefi-
niowanà struktur´. W obu przypadkach mechanizm ich dzia-
∏ania opiera si´ na przekszta∏ceniach czàsteczek DNA
polegajàcych na zmianie z jednej konformacji (np. zwyk∏ej
podwójnej helisy) na innà.
Konwencjonalny DNA jest helisà prawoskr´tnà. Wyobraê-
my sobie, ˝e schodzimy po spiralnych schodach z prawà d∏o-
nià na por´czy wewn´trznej, a lewà na zewn´trznej. Takie scho-
dy to helisa prawoskr´tna. Zwyk∏a prawoskr´tna helisa DNA jest
okreÊlana symbolem B-DNA i jest najbardziej korzystnà ener-
getycznie strukturà w Êrodowisku wodnym.
Podwójna helisa DNA mo˝e równie˝ przyjmowaç ró˝ne in-
ne formy, zale˝nie od sekwencji zasad i od sk∏adu chemiczne-
go roztworu, w którym jest zanurzona. Jednà z nich jest
Z-DNA, opisana po raz pierwszy w 1979 roku przez Alexan-
dra Richa i jego wspó∏pracowników z Massachusetts Insti-
tute of Technology [górna ilustracja na stronie 41]. Z-DNA jest
helisà lewoskr´tnà.
By przygotowaç Z-DNA, zwykle potrzebne sà nici, w których
cytozyna wyst´puje na przemian z guaninà. Szkielet DNA za-
wiera na∏adowane ujemnie grupy fosforanowe, które w struk-
turze Z-DNA le˝à blisko siebie. Ta struktura jest faworyzo-
wana tylko wówczas, gdy ∏adunki fosforanów sà od siebie
ekranowane. Dzieje si´ tak w roztworach soli o wysokim st´˝e-
niu lub w roztworach specjalnych „efektorowych” substancji
chemicznych, takich jak heksaaminakobalt(III), Co(NH
3
)
6
+++
,
które wykonujà t´ samà robot´ przy ni˝szym st´˝eniu. Na-
przemienne u∏o˝enie cytozyny i guaniny wyznacza miejsce, w
którym w czàsteczce DNA zachodzi przejÊcie typu B–Z (a
wi´c to, co robi nasza maszyna), a czynniki Êrodowiskowe
pozwalajà ustaliç, kiedy nastàpi transformacja (a wi´c i czyn-
noÊç wykonana przez maszyn´).
Razem ze wspó∏pracownikami z New York University
– Weiqiongiem Sunem, Zhiyongiem Shenem i Mao – zbudo-
wa∏em urzàdzenie sk∏adajàce si´ z dwóch czàsteczek DX po∏à-
czonych „wa∏kiem” podwójnej helisy DNA [ilustracja obok].
Centralnà cz´Êç wa∏ka stanowi sekwencja 20 par zasad, która
w odpowiednich warunkach mo˝e uk∏adaç si´ w struktur´ ty-
pu Z. Zazwyczaj ka˝dy z elementów urzàdzenia ma postaç
B-DNA, a obie czàsteczki DX znajdujà si´ po tej samej stronie
osi wa∏ka. Kiedy do roztworu dodajemy heksaaminakobalt(III),
Êrodkowy odcinek wa∏ka przekszta∏ca si´ w Z-DNA i jedna z czà-
steczek DX obraca si´ oko∏o 3.5 razy w stosunku do drugiej
(nieparzysta po∏owa obrotu oznacza, ˝e moleku∏y DX znajdà si´
po przeciwleg∏ych stronach osi wa∏ka). Usuni´cie heksaami-
nakobaltu(III) przywraca stan poczàtkowy urzàdzenia. Ruch
uk∏adu zaobserwowaliÊmy metodami spektroskopii, do∏àczajàc
do czàsteczek DX znaczniki w dwóch kolorach.
Nasze urzàdzenie jest doÊç solidne, lecz ma pewnà u∏om-
noÊç. Gdyby wi´cej maszyn typu B-Z w∏àczyç w wi´kszà su-
perstruktur´ (np. w jednà z omawianych wczeÊniej dwu-
wymiarowych sieci krystalicznych), ca∏y uk∏ad móg∏by
przyjmowaç tylko dwa stany: wszystkie maszyny jednocze-
Ênie znajdowa∏yby si´ w stanie B lub Z. Indywidualna kontro-
la urzàdzeƒ wymaga zastosowania elementów z niezale˝ny-
mi mechanizmami wyzwalajàcymi. W przypadku DNA istnieje
oczywiÊcie naturalne rozwiàzanie tego problemu: jako wy-
zwalaczy poszczególnych urzàdzeƒ mo˝na u˝yç nici DNA o
ró˝nych sekwencjach zasad.
By zrealizowaç taki plan, Hao Yan, dziÊ pracownik Duke
University, Xiaoping Zhang z New York University, Shen oraz
ja obmyÊliliÊmy uk∏ad, który zmienia kszta∏t, gdy przy∏àcza-
jà si´ doƒ ró˝ne nici. Sk∏ada si´ on z dwóch równoleg∏ych
podwójnych helis DNA, które na Êrodkowym odcinku sà zre-
dukowane do pojedynczych, krzy˝ujàcych si´ nici. Odcinek ten
mo˝e przybieraç dwa stany, w zale˝noÊci od tego, jakie nici –
wià˝àce si´ z odcinkami jednoniciowymi urzàdzenia – zo-
stanà dodane do roztworu [ramka na nast´pnej stronie]. Dwa
stany urzàdzenia okreÊlane sà symbolami PX (paranemic
LIPIEC 2004 ÂWIAT NAUKI
45
KEN EW
ARD
BioGrafx
NAJWA˚NIEJSZYM CELEM
nanotechnologii opartej
na DNA jest
PRZEJÂCIE Z DWÓCH WYMIARÓW DO TRZECH
.
NANOMECHANIZM TYPU B-Z sk∏ada si´ z dwóch czàsteczek DX (niebie-
ski i pomaraƒczowy) po∏àczonych „wa∏kiem” z 20 par zasad (fioletowy).
Dwie kolorowe czàsteczki barwnika (srebrna i z∏ota kula) pokazujà po∏o-
˝enie moleku∏ DX. W stanie B (na górze) wa∏ek jest zwyczajnym prawo-
skr´tnym B-DNA, a dwie czàsteczki DX znajdujà si´ po tej samej stronie
osi urzàdzenia. Kiedy do roztworu dodany zostaje heksaaminakobalt(III),
wa∏ek ulega konwersji do lewoskr´tnego Z-DNA (ilustracja na stronie 41
na górze) i modu∏y DX wykonujà wzgl´dem siebie 3.5 obrotu.
Czàsteczka barwnika
Z-DNA
B-DNA
Heksaaminakobalt(III)
dodany
Heksaaminakobalt(III)
usuni´ty
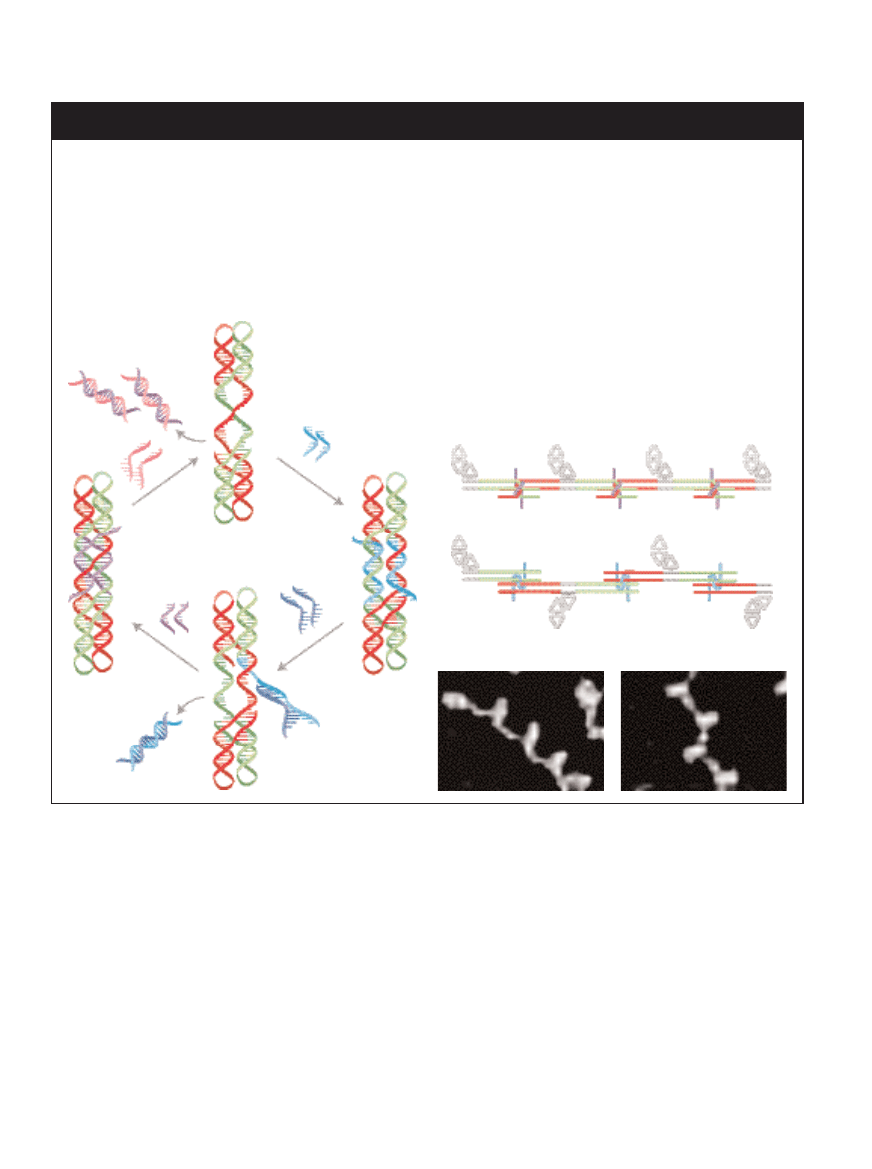
crossover – z∏àcze splecione) i JX (juxtaposed crossover – z∏à-
cze równoleg∏e). Kiedy urzàdzenie znajduje si´ w stanie PX,
dwie helisy po jednej stronie centralnego skrzy˝owania sà
przekr´cone o pó∏ obrotu w stosunku do swojego po∏o˝enia w
stanie JX.
Dodanie do roztworu specjalnej pary nici (zwanych niçmi
konfiguracyjnymi) przeprowadza urzàdzenie w stan JX – ni-
ci te wià˝à si´ ze Êrodkowà cz´Êcià urzàdzenia, nie krzy˝ujàc
si´. Aby prze∏àczyç uk∏ad w stan PX, musimy najpierw usunàç
nici konfiguracyjne. W roku 2000 Bernard Yurke i jego wspó∏-
pracownicy z Lucent Technologies wykazali, ˝e z DNA mo˝-
na usunàç fragment nici za pomocà nici do nich komplemen-
tarnych. Procedur´ t´ zrealizowaliÊmy, wyposa˝ajàc nasze
nici konfiguracyjne w krótkie lepkie koƒce, które nie parujà
si´ z nanourzàdzeniem. Gdy do roztworu dodamy nici kom-
plementarne do nici konfiguracyjnych, po∏àczà si´ one z nie-
sparowanymi przed∏u˝eniami, a nast´pnie „zedrà” reszt´ ni-
ci z mechanizmu.
Kiedy pierwsze nici konfiguracyjne zostajà usuni´te z urzà-
dzenia, mo˝emy dodaç inne nici, które tym razem po∏àczà
si´ krzy˝owo z jego Êrodkowà, jednoniciowà cz´Êcià. Wiàza-
nie to obraca dwiema podwójnymi helisami, prze∏àczajàc
urzàdzenie w stan PX. Proces mo˝na odwróciç przez usuni´-
cie tej drugiej pary nici konfiguracyjnych i dodanie pierw-
szej. W ten sposób podwójne helisy sà obracane w t´ i z po-
wrotem na zawo∏anie. Ró˝ne urzàdzenia typu PX-JX mogà
wi´c byç sterowane niezale˝nie przez dodawanie i usuwanie
nici konfiguracyjnych zaprojektowanych z myÊlà o obsza-
rach, z którymi majà si´ wiàzaç.
Ruchy naszych konstrukcji zaobserwowaliÊmy pod mikro-
skopem si∏ atomowych. Urzàdzenia zestawiliÊmy w d∏ugie
∏aƒcuchy, a do boku ka˝dego z nich do∏àczyliÊmy modu∏ DNA
46
ÂWIAT NAUKI LIPIEC 2004
ALICE Y
. CHEN (
ilustracja
); NADRIAN C. SEEMAN (
mikr
ofotografia
)
PRZE¸ÑCZNIK Z DNA
INDYWIDUALNIE KONTROLOWANE urzàdzenie DNA mo˝na prze∏à-
czaç mi´dzy dwoma stanami (a, 1–8) przez dodawanie lub usuwanie
odcinków DNA zwanych niçmi konfiguracyjnymi. Gotowe do dzia∏ania
urzàdzenie sk∏ada si´ z czterech podwójnych helis po∏àczonych dwie-
ma niesparowanymi niçmi DNA (1). Gdy dodamy do uk∏adu nici kon-
figuracyjne (jasnoniebieski) (2), zwià˝à si´ one z niçmi niesparowa-
nymi, wymuszajàc przejÊcie urzàdzenia w stan „podwójnie równoleg∏y”
(JX – doubly juxtaposed) (3). W tym stanie górne i dolne helisy zazna-
czone na rysunku na czerwono znajdujà si´ po lewej stronie, a heli-
sy zielone – po prawej. Do usuni´cia nici konfiguracyjnych (jasnonie-
bieski) s∏u˝à nici do nich komplementarne (ciemnoniebieski) (4). Ta-
ki uk∏ad ponownie jest w stanie gotowoÊci (5). Teraz dodane zostajà
drugie nici konfiguracyjne (fioletowy) (6), które w inny sposób wià˝à
si´ z odcinkami jednoniciowymi, ustawiajàc urzàdzenie w konfigura-
cji (7) zwanej z∏àczem splecionym (PX – paranemic crossover). Wów-
czas dolna cz´Êç urzàdzenia obraca si´, a dolne czerwone i zielone he-
lisy zamieniajà si´ miejscami. Usuni´cie drugich nici konfiguracyjnych
(fioletowy) z maszyny zamyka jej cykl pracy (8).
By przetestowaç dzia∏anie prze∏àcznika, zestawiono jego kopie w
∏aƒcuch i do∏àczono markery w kszta∏cie du˝ych trapezoidalnych frag-
mentów DNA. Kiedy wszystkie urzàdzenia w ∏aƒcuchu znajdowa∏y
si´ w stanie PX (b), wszystkie trapezy u∏o˝y∏y si´ po tej samej stronie
osi ∏aƒcucha. Gdy wszystkie urzàdzenia sà w stanie JX (c), co drugi
trapez zmienia po∏o˝enie. Obrazy wykonane mikroskopem si∏ atomo-
wych potwierdzajà ten schemat dzia∏ania (d, e).
d
b
a
1
2
PX
Nici konfiguracyjne
Stan JX
Stan PX
Nici
komplementarne
JX
JX
JX
PX
PX
3
4
5
6
7
8
c
e

w kszta∏cie trapezu. Kiedy wszystkie urzàdzenia by∏y w sta-
nie PX, trapezy uk∏ada∏y si´ po jednej stronie ∏aƒcucha. Gdy
wszystkie by∏y w stanie JX, co drugi trapez zmienia∏ po∏o˝e-
nie, tworzàc zygzakowaty wzór.
W roku 2000 Yurke i jego wspó∏pracownicy zademonstro-
wali nanometrowej wielkoÊci p´sety wykonane z trzech nici
DNA. Nici konfiguracyjne, które Yurke nazywa niçmi nap´-
dzajàcymi, otwiera∏y i zamyka∏y p´set´. Inni naukowcy podob-
ne metody stosowali do aktywowania rybozymów – enzymów
RNA. W 1998 roku Michael P. Robinson i Andrew D. Elling-
ton z University of Texas w Austin pokazali, jak wzmocniç
aktywnoÊç rybozymów 10 tys. razy. Dodawana przez nich od-
powiednia niç konfiguracyjna wiàza∏a si´ z rybozymem, zmie-
niajàc jego konformacj´.
Chemiczne linie monta˝owe
W NANOTECHNOLOGII OPARTEJ NA DNA
najistotniejsze teraz jest
uzyskanie w trzech wymiarach tego, co uda∏o si´ zrobiç w
dwóch. Gdy si´ to uda, nab´dziemy umiej´tnoÊci projekto-
wania tworzyw krystalicznych przez definiowanie sekwencji
odcinków DNA, a nast´pnie ∏àczenie ich w ca∏oÊç. JeÊli uk∏a-
dy te b´dà dostatecznie uporzàdkowane, wykonalne stanà si´
wspomniane wy˝ej doÊwiadczenia krystalograficzne z czà-
steczkami usadowionymi w regularnej sieci.
Innym zamierzeniem jest w∏àczenie urzàdzeƒ zbudowa-
nych z DNA w ramy wi´kszych struktur roboczych. B´dzie
to pierwszy krok w kierunku nanorobotyki, która pozwoli na
zbudowanie chemicznych linii monta˝owych. Stosujàc urzà-
dzenia podobne do opisanych w artykule, nowe tworzywa
mo˝na by∏oby montowaç z du˝à precyzjà. Wspólnie z Jame-
sem W. Canary’m i Philipem S. Lukemanem z New York Uni-
versity oraz Lei Zhu, który obecnie pracuje w University of
Texas w Austin, niedawno zamontowa∏em na szkielecie z kwa-
su nukleinowego ma∏y p∏atek nylonu. Spodziewamy si´, ˝e
pewnego dnia b´dziemy w stanie produkowaç nowe polime-
ry o konkretnych w∏aÊciwoÊciach i topologiach (np. krzywi-
znach szkieletów).
Zrealizowanie tych planów wià˝e si´ z u˝yciem DNA jako
programowalnej cz´Êci, ale ani krystalografia, ani nanoelek-
tronika nie mo˝e polegaç wy∏àcznie na DNA. Na przyk∏ad
elementy nanoelektroniczne, takie jak nanoczàstki metalicz-
ne lub w´glowe nanorurki, b´dà musia∏y byç spajane z czà-
steczkami DNA w uk∏adach i roztworach wodnych kompa-
tybilnymi zarówno z DNA, jak i innymi sk∏adnikami. Ze
wzgl´du na ró˝norodnoÊç tych moleku∏ ten cel nie∏atwo osià-
gnàç. W dodatku nawet jeÊli elementy nanoelektroniczne po-
wstawa∏yby przy udziale samoorganizujàcego si´ DNA, na-
nourzàdzenia ostatecznie muszà wchodziç w interakcje ze
Êwiatem makroskopowym metodami du˝o bardziej wyrafi-
nowanymi ni˝ przez dodawanie i ujmowanie nici z roztworu.
Realizacja tych planów jest ogromnym wyzwaniem.
Nanotechnologiczna maszyna przysz∏oÊci umie si´ powielaç.
Jednak w przeciwieƒstwie do liniowego rozga∏´ziony DNA
nie najlepiej nadaje si´ do autoreplikacji. Mimo to William M.
Shih, Joel D. Quispe i Gerald F. Joyce ze Scripps Research In-
stitute w La Jolla w Kalifornii mogà poszczyciç si´ pierwszy-
mi, fascynujàcymi osiàgni´ciami w dziedzinie samopowiela-
jàcych si´ obiektów DNA. Z jednej d∏ugiej nici (oko∏o 1700
zasad) zbudowali szkielet oÊmioÊcianu, wykaƒczajàc konstruk-
cj´ pi´cioma krótkimi niçmi „pomocniczymi” [ilustracja po-
wy˝ej]. Ka˝da z kraw´dzi oÊmioÊcianu wykonana jest z dwóch
splecionych podwójnych helis DNA – ∏aƒcucha czàsteczek ty-
pu DX oraz PX. Kraw´dzie majà d∏ugoÊç oko∏o 14 nm, która
odpowiada mniej wi´cej czterem skokom podwójnej helisy.
Gotowy oÊmioÊcian nie mo˝e si´ reprodukowaç, ale w stanie
niez∏o˝onym mo˝e byç klonowany w milionach egzemplarzy.
D∏uga niç bez trudu daje si´ bowiem kopiowaç standardowà
metodà zwanà ∏aƒcuchowà reakcjà polimerazy (PCR – poly-
merase chain reaction). Proces ten jest dalece mniej efektyw-
ny ni˝ replikacja, która zachodzi w ka˝dym ˝ywym orga-
nizmie, ale do czasu setnej rocznicy odkrycia Watsona i Cricka
powinniÊmy ju˝ otrzymaç maszyny DNA, które nie b´dà pod
tym wzgl´dem ust´powaç naturze.
n
LIPIEC 2004 ÂWIAT NAUKI
47
MIKE PIQUE
Scripps R
esear
ch Institute
OÂMIOÂCIAN Z DNA powsta∏ z jednej d∏ugiej nici DNA i pi´ciu krótkich
nici pomocniczych. Ka˝da kraw´dê sk∏ada si´ z dwóch równoleg∏ych,
splecionych ze sobà podwójnych helis. Dane, na których podstawie wy-
generowano ilustracj´, pochodzà z wykonanych mikroskopem elektrono-
wym obrazów ponad 600 oÊmioÊcianów. Skala kolorów odpowiada
wzgl´dnemu zag´szczeniu elektronów (czerwony to zag´szczenie wy-
sokie, niebieski – niskie).
A 1.7-Kilobase Single-Stranded DNA That Folds into a Nanoscale
Octahedron. William M. Shih, Joel D. Quispe i Gerald F. Joyce;
Nature, tom 427, s. 618-621; 12 II 2004.
DNA as an Engineering Material. Andrew Turberfield; Physics World,
tom 16, nr 3, s. 43-46; III/2003.
DNA in a Material World. Nadrian C. Seeman; Nature, tom 421,
s. 427-431; 23 I 2003.
A Robust DNA Mechanical Device Controlled by Hybridization Topology.
Hao Yan, Xiaoping Zhang, Zhiyong Shen i Nadrian C. Seeman; Nature,
tom 415, s. 62-65; 3 I 2002.
Logical Computation Using Algorithmic Self-Assembly of DNA Triple
Crossover Molecules. Chengde Mao, Thomas H. LaBean, John H. Reif
i Nadrian C. Seeman; Nature, tom 407, s. 493-496; 28 IX 2000.
(Erratum: Nature, tom 408, s. 750; 7 XII 2000.)
A DNA-Fuelled Molecular Machine Made of DNA. Bernard Yurke, Andrew
J. Turberfield, Allen P. Mills, jr., Friedrich C. Simmel i Jennifer L. Neumann;
Nature, tom 406, s. 605-608; 10 VIII 2000.
Strona laboratorium Nadriana C. Seemana:
http://seemanlab4.chem.nyu.edu/
JEÂLI CHCESZ WIEDZIEå WI¢CEJ
Wyszukiwarka
Podobne podstrony:
200407 3694
200407 3692
200407 3689
200407 3706
ZPORR zalacznik8 kontrolerealizacjiprojektow 200407
200407 3696
3702
200407 3705
200407 3712
ZPORR zalacznik1 Stanrealizacjiprojektowwramachpriorytetow dzialan 200407
200407 3687
200407 3704
200407 3711
200407 3707
więcej podobnych podstron