
W1
DNA: izolacja i
analiza
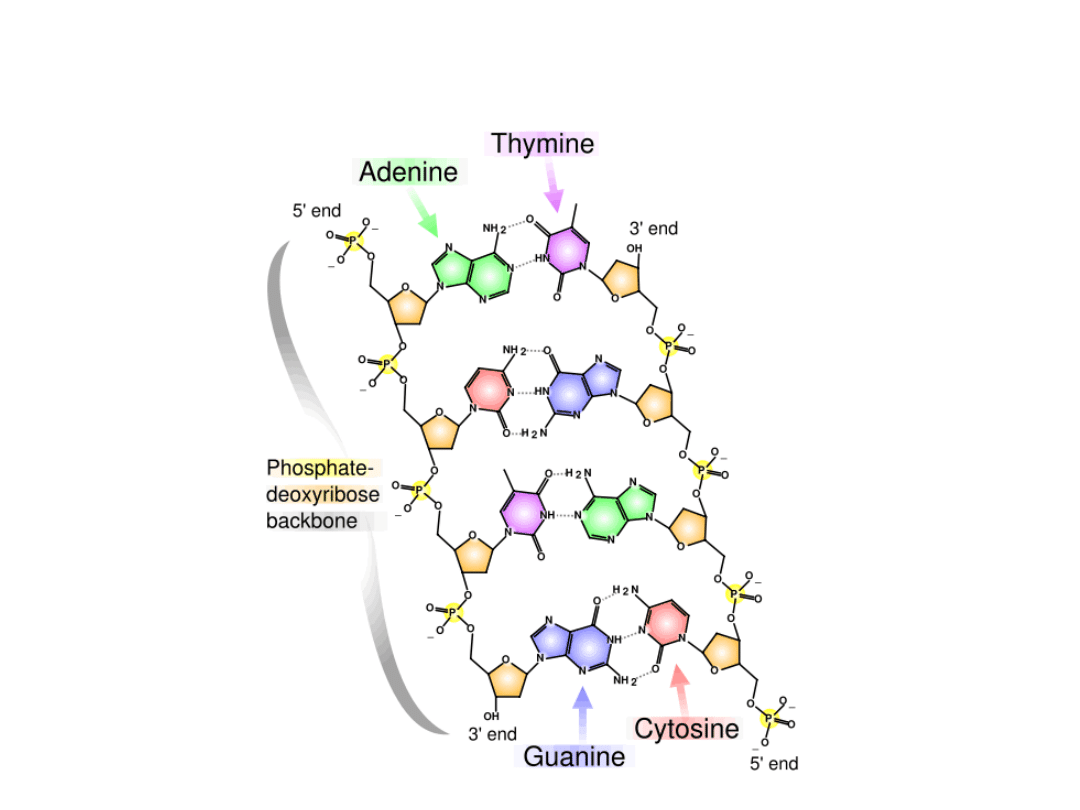
Budowa DNA
DNA jest materiałem
genetycznym komórki

Analiza DNA
• Dzięki
swojej
regularnej
budowie
i
niewielkiej ilości elementów niosących
informację
(4 zasady azotowe) DNA jest wdzięcznym
materiałem do badań.
• Posiadamy szereg narzędzi pozwalających
na izolację i analizę DNA.
• Fragmenty DNA o identycznej sekwencji
mogą być łatwo rozpoznawane.

Izolacja DNA
• Źródło: bakterie, komórki, tkanki
• Liza komórek
• Wytrawienie białek
• Odbiałczenie
• Wytrącenie DNA
• Odwirowanie osadu i rozpuszczenie DNA
• Oznaczenie stężenia

•
Źródło: bakterie, komórki, tkanki
•
Liza komórek [3M NaCl, 0.4 M Tris*HCl pH 7.8,
20 mM EDTA (bufor rozbijający wiązania jonowe)
+ SDS (rozbija wiązania hydrofobowe)]
•
Wytrawienie białek (proteinaza K: 0.1 mg/ml)
•
Odbiałczenie przez wytrząsanie z równą
objętością mieszaniny fenolu i chloroformu (pH
8.0) i wirowanie (tworzą się 2 fazy: DNA pozostaje
w fazie wodnej, wytrącone białka na granicy faz,
po wirowaniu zbiera się fazę wodną i odbiałcza
ponownie)
•
Wytrącenie DNA z odbiałczonego roztworu
(dodanie octanu amonu do stężenia ok.0.3 M i 2
objętości zimnego /-20
0
C/ etanolu)
•
Odwirowanie precypitatu i rozpuszczenie osadu
DNA
•
Oznaczenie stężenia: spektrofotometryczne
przy długości fali 260nm
SDS
Sodium dodecyl sulfate
Siarczan dodecylu sodu
wpływa na rozpad wiązań hydrofobowych
Obecność jonów jednowartościowych (3M
NaCl) wpływa na rozpad wiązań jonowych

Izolacja plazmidowego DNA oparta
jest
przede wszystkim na różnicy w
renaturacji
DNA komórkowego i DNA plazmidu
•Najpowszechniej stosowana jest metoda lizy
w warunkach alkalicznych lub przez gotowanie.
W tych warunkach następuje denaturacja DNA
chromosomowego i większości białek. Koliste
cząsteczki plazmidu mimo denaturacji nie mogą
rozdzielić nici, co ułatwia późniejszą renaturację.
•Uwaga!
Przedłużone
działanie
zasad
lub
wysokiej temperatury powoduje nieodwracalną
denaturację
plazmidu.
Powstający
nieuporządkowany kłębek DNA nie poddaje się
obróbce enzymatycznej.
•Duże plazmidy (powyżej 15 kpz) wymagają
ostrożnego traktowania, co zapobiega ich
rozerwaniu.
Oznaczanie DNA metodą
spektrofotometryczną
Dla kwasów nukleinowych lambda
max
=260 nm
Dla białek (aminokwasy aromatyczne) lambda
max
=280 nm
Stosunek A
260
/A
280
powinien wynosić dla czystego DNA 1.8,
wartości wyższe świadczą o zanieczyszczeniu RNA, niższe o
zanieczyszczeniu białkami lub fenolem.
Wiązania peptydowe absorbują światło o długości fali 220-230 nm
A
b
so
rp
c
j
a
Długość fali (nm)
220
260
300

Inne metody oznaczania DNA
1. Metoda
fluorometryczna
przy
użyciu
barwnika Hoechst 33258. Próbę badaną
porównuje
się
z krzywą wzorcową (w zakresie 10-250
ng/ml) o tym samym składzie zasad.
Stosowane
tylko
do
DNA
wysokocząsteczkowego.
2. Test kroplowy z bromkiem etydyny. Na
płytkę agarozową zawierającą 0.5 g/ml
bromku etydyny nakrapia się wzorzec oraz
badane DNA. Rezultaty można zapisywać w
formie fotografii.

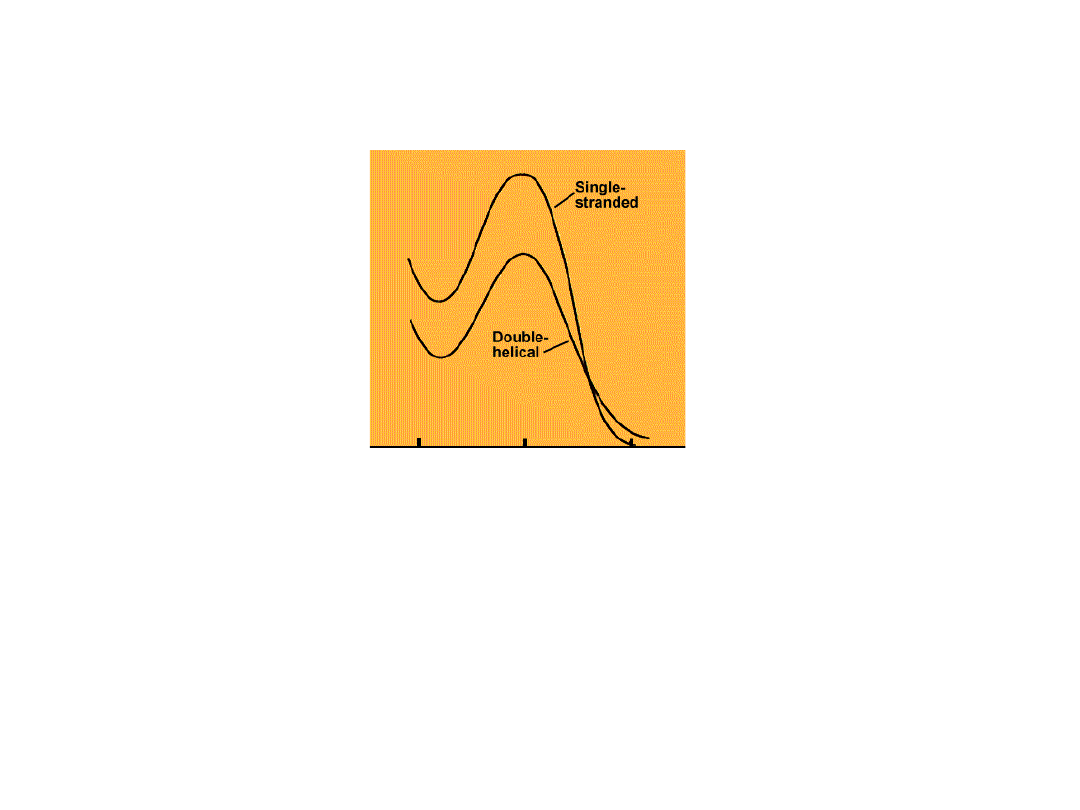
Temperatura topnienia
DNA
Ze względu na różnice w absorbancji
A
260
pomiędzy DNA dwuniciowym,
a jednoniciowym, można określić
temperaturę,
w
której
połowa
cząsteczek DNA występuje w formie
jednoniciowej (zdenaturowanej).
Tę
temperaturę
nazywamy
temperaturą topnienia = Tm
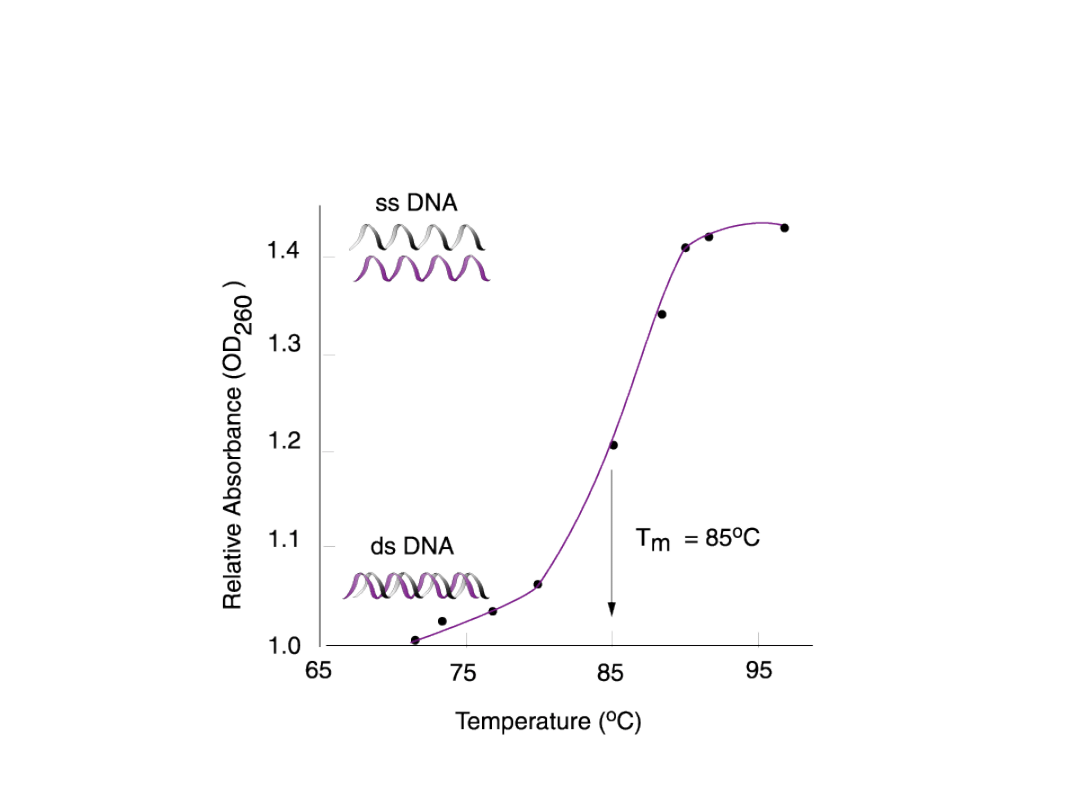
Wyznaczanie temperatury topnienia DNA
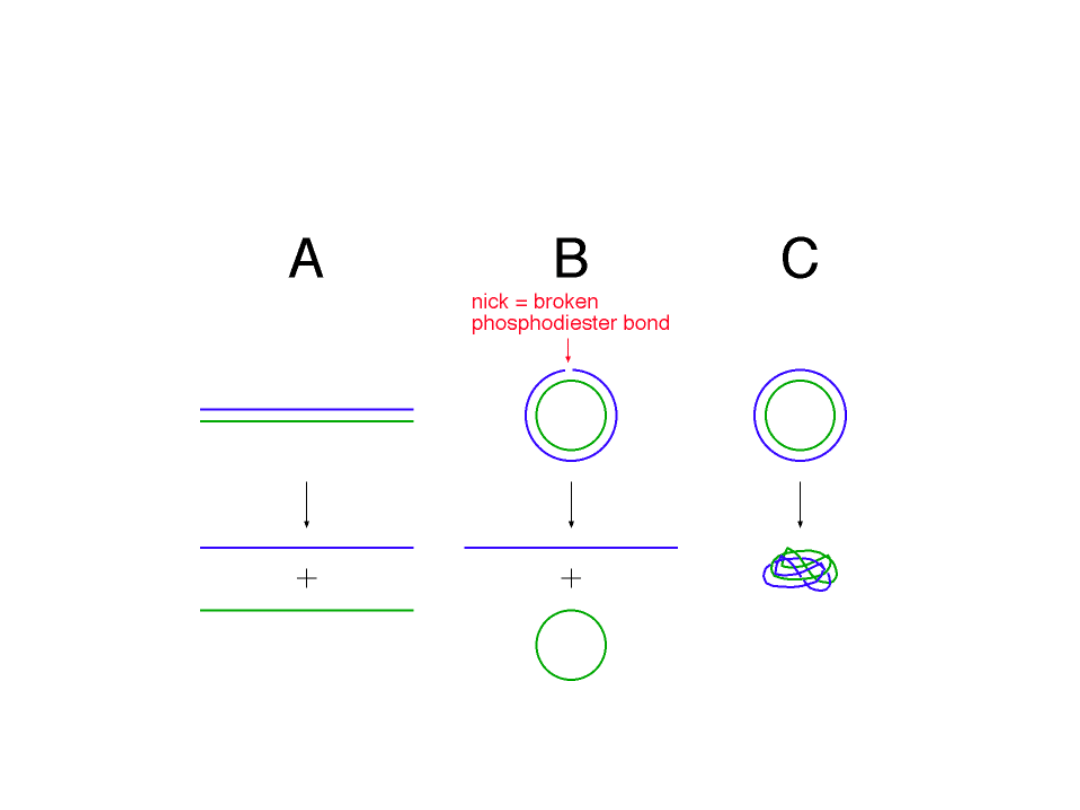
Denaturacja różnych
cząsteczek DNA
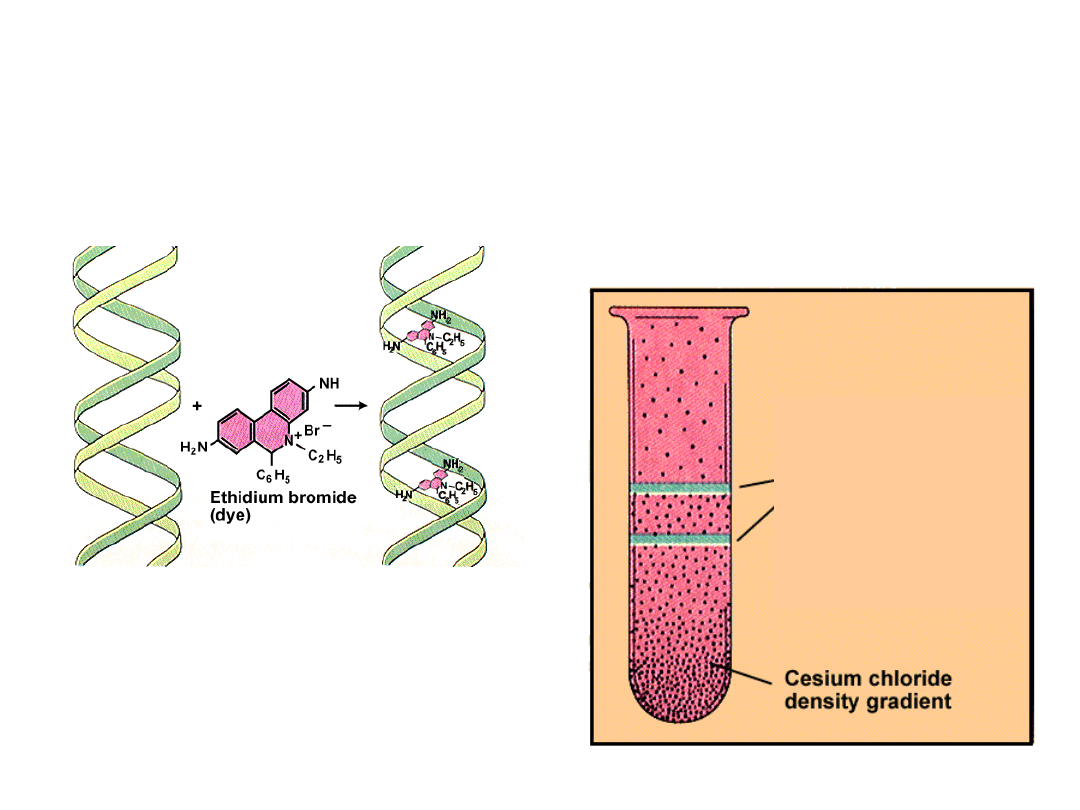
Oczyszczanie plazmidowego DNA
przez wirowanie w gradiencie
chlorku cezu
Chromosomowe
i plazmidowe DNA
różnią się gęstością
(odpowiednio 1.54 i
1.59 g/cm
3
) i
tworzą dwa
niezależne pasma
w gradiencie CsCl
Wiązanie EtBr do DNA (w
warunkach nasycenia średnio 1
cząsteczka na 2.5 pz) obniża
jego gęstość
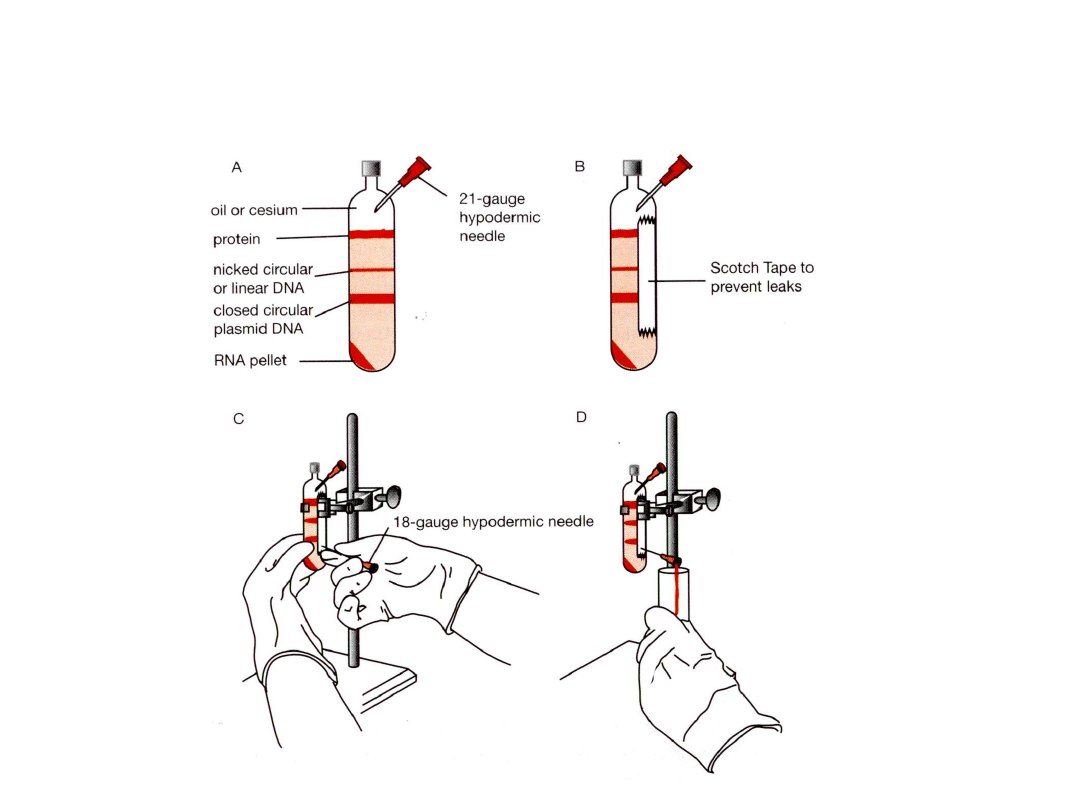
Zbieranie plazmidowego DNA z
gradientu CsCl zawierającego bromek
etydyny

Cięcie cząsteczek DNA
na fragmenty
Rozbijanie ultradźwiękami (ultrasonikacja)
Rozrywanie w czasie przepływu cieczy
(nebulizacja)
Trawienie nukleazami
Dezoksyrybonukleazy (DNazy):
Egzonukleazy
Endonukleazy:
•
niespecyficzne względem sekwencji
(nukleaza
mikrokokalna, DNaza I,
egzonukleaza III)
•
specyficzne względem sekwencji (enzymy
restrykcyjne)

Typy enzymów
restrykcyjnych
• Typ I
– złożone, zawierające wiele podjednostek,
przecinają DNA w przypadkowym miejscu z dala
od sekwencji rozpoznawanej. Często zawierają
kombinację enzymów restrykcji-modyfikacji.
• Typ II
– przecinają DNA w określonych miejscach,
blisko lub w samej sekwencji rozpoznawanej.
• Typ IIs
– enzymy, które przecinają DNA w
określonej (kilka, kilkanaście nukleotydów)
odległości od sekwencji rozpoznawanej, zawsze
po jednej stronie.
• Typ III
podobne do typu I, jednak do
rozpoznawania sekwencji wymagają dwóch
takich
sekwencji
o naprzeciwległej orientacji na tej samej
cząsteczce DNA.
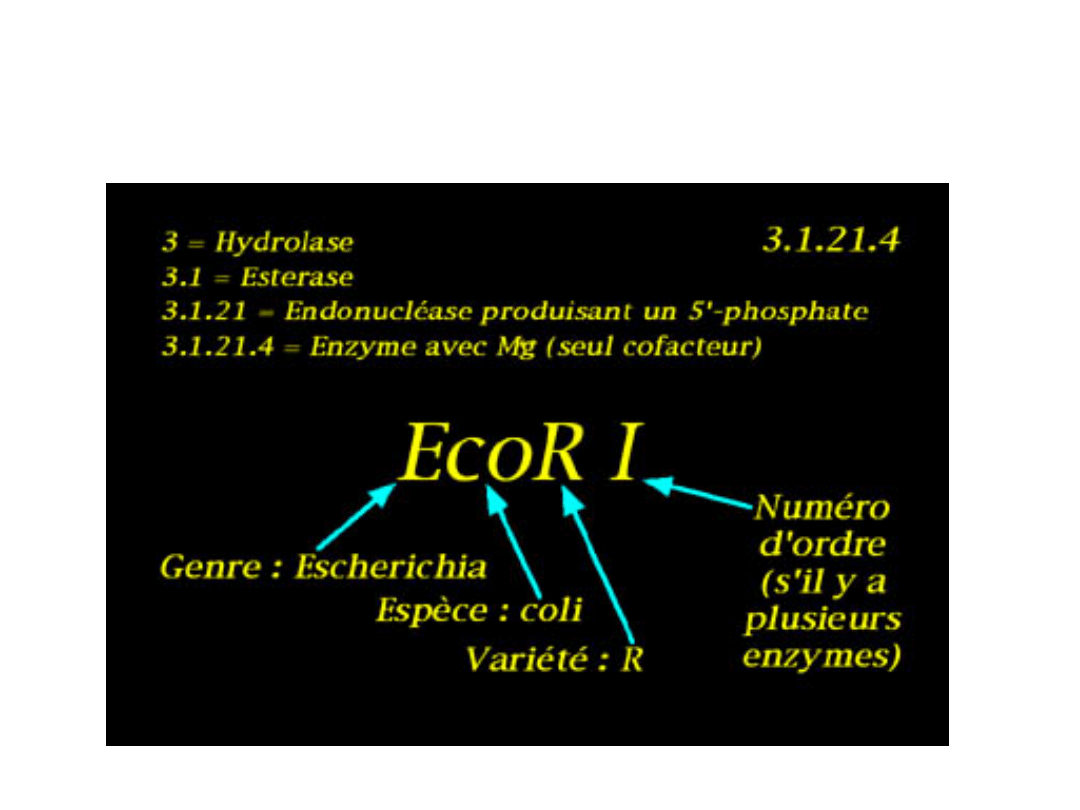
Nazewnictwo enzymów
restrykcyjnych

Sekwencje rozpoznawane
przez enzymy restrykcyjne
Enzymy
restrykcyjne
często
rozpoznają
sekwencje
palindromiczne,
o
różnych
długościach,
od
czterech
do ośmiu i więcej zasad.
W genomie człowieka i wyższych eukariontów:
•enzymy „czwórkowe” generują fragmenty o
średniej długości 260 pz.
•enzymy „szóstkowe”
trawią DNA na
fragmenty
o średniej długości 4100 pz.
•enzym „szóstkowy” swoisty dla sekwencji
zawierającej GC daje fragmenty o średniej
długości 65000 pz.
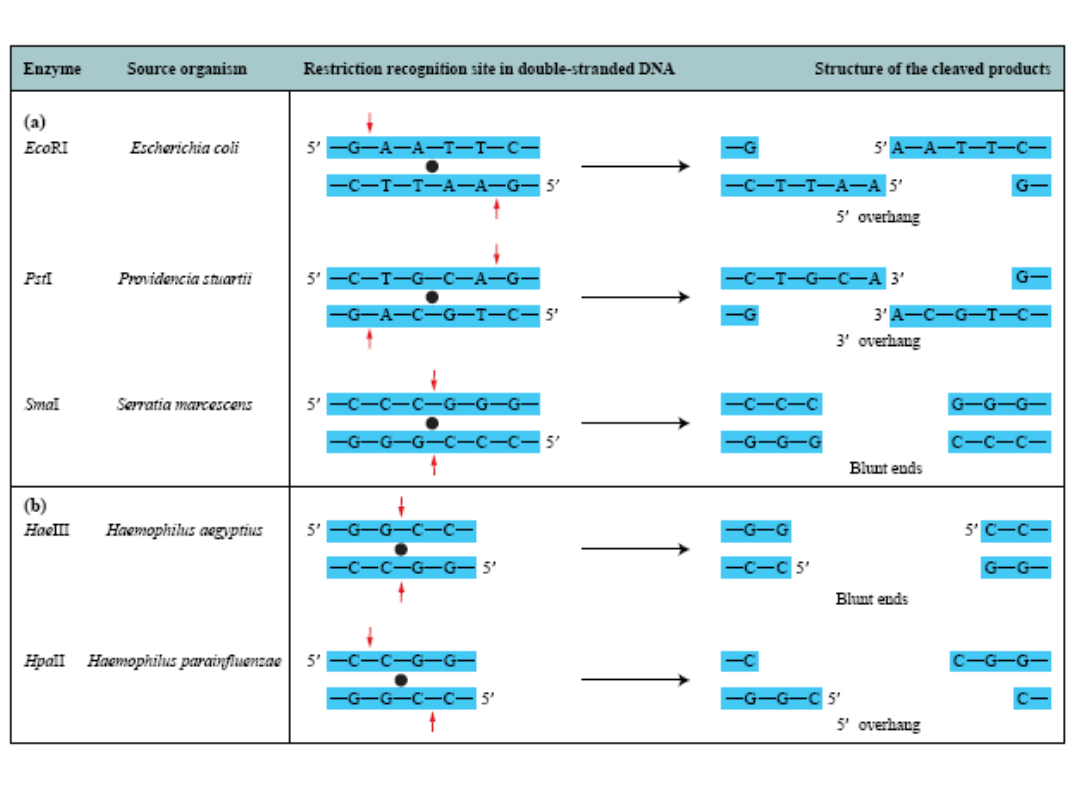

Specyficzność enzymów
restrykcyjnych
• Specyficzne rozpoznawanie
sekwencji
• Specyficzne cięcie sekwencji DNA
Specyficzność cięcia DNA dotyczy
wyłącznie enzymów restrykcyjnych
typu II
Tylko endonukleazy restrykcyjne, które
przecinają DNA specyficznie względem
sekwencji, zapewniają powtarzalność
cięcia DNA na fragmenty
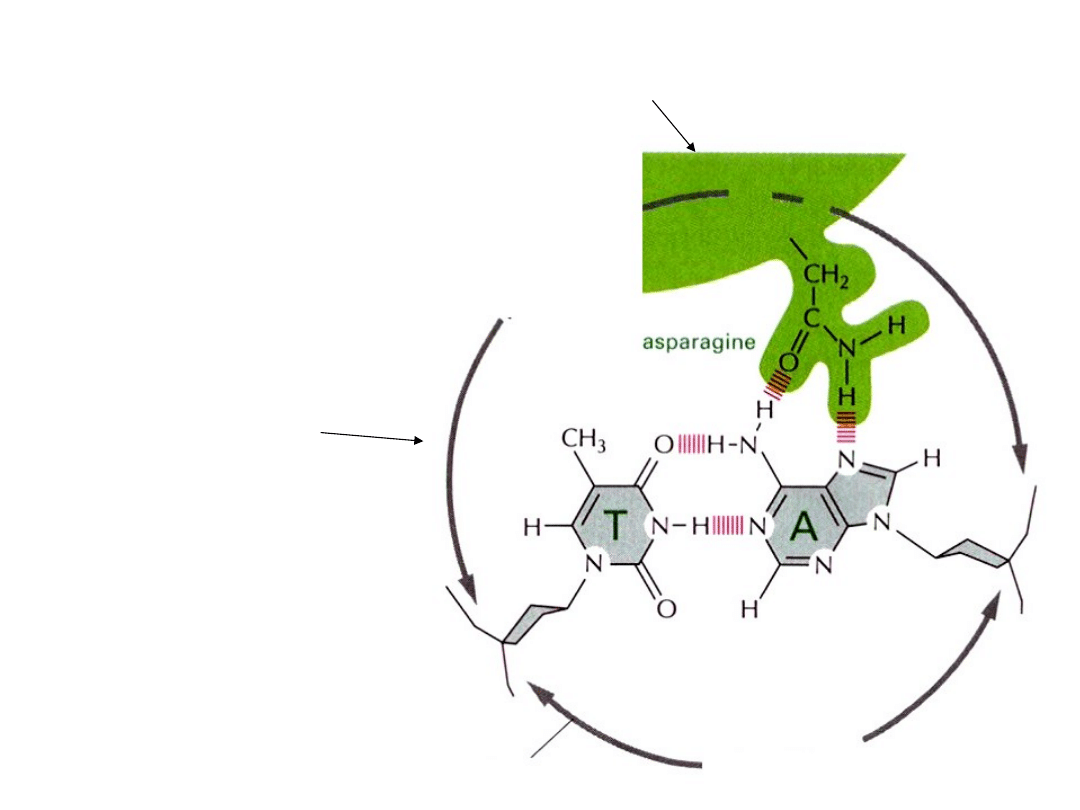
Rozpoznawanie
sekwencji
nukleotydowych
przez białka -
A
du
ży
ro
we
k
mały
row
ek
DNA
Białko
Specyficzne
rozpoznawanie
sekwencji
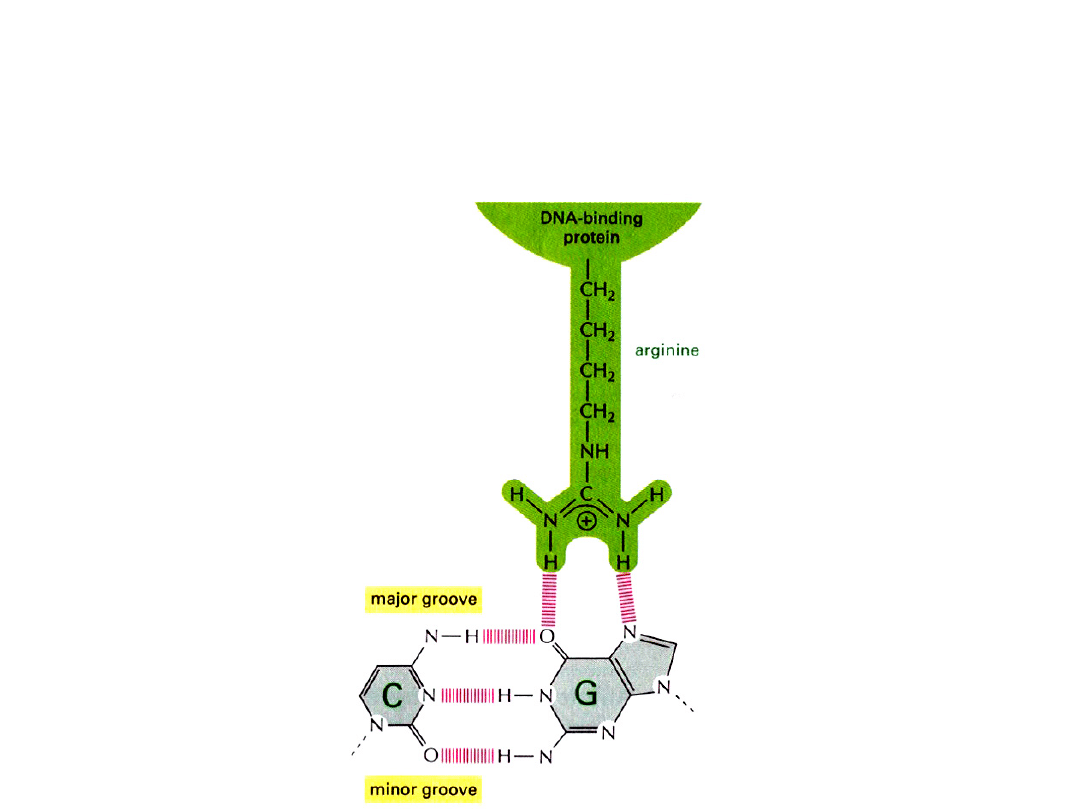
Rozpoznawanie sekwencji
nukleotydowych przez
białka -
G

BamHI przecina rozpoznawaną
sekwencję: G ' GATCC
Struktura dimeru BamHI
w sąsiedztwie sekwencji
niespecyficznej
Struktura dimeru BamHI
w miejscu
specyficznie
rozpoznawanej
sekwencji

Cięcie DNA na fragmenty -
końce
Enzymy restrykcyjne zawsze
zostawiają końce DNA:
• 3’ - OH
• 5’ – grupa fosforanowa
Takie końce są odpowiednie do
łączenia fragmentów przez ligazę
DNA

Rozdział cząsteczek różniących
się masą molekularną
Wytrącanie różnicowe
Elektroforeza w żelach
w stałym polu elektrycznym
w zmiennym polu elektrycznym
Wirowanie i ultrawirowanie
Chromatografia
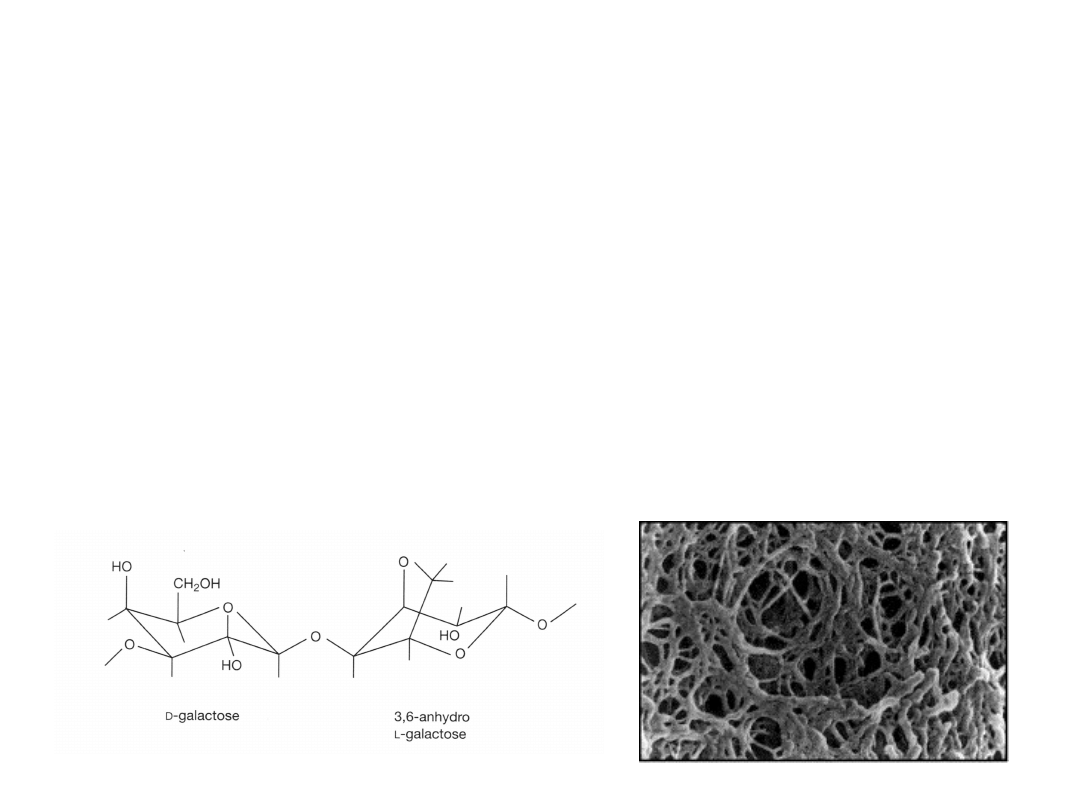
Agaroza
Polisacharyd otrzymany z glonów.
Rozpuszczona w wodzie tworzy żel.
Używana w stężeniu 0.5 – 4%
standard – temperatura topnienia około 90
o
C
niskotopliwa – temperatura topnienia 40-85
o
C
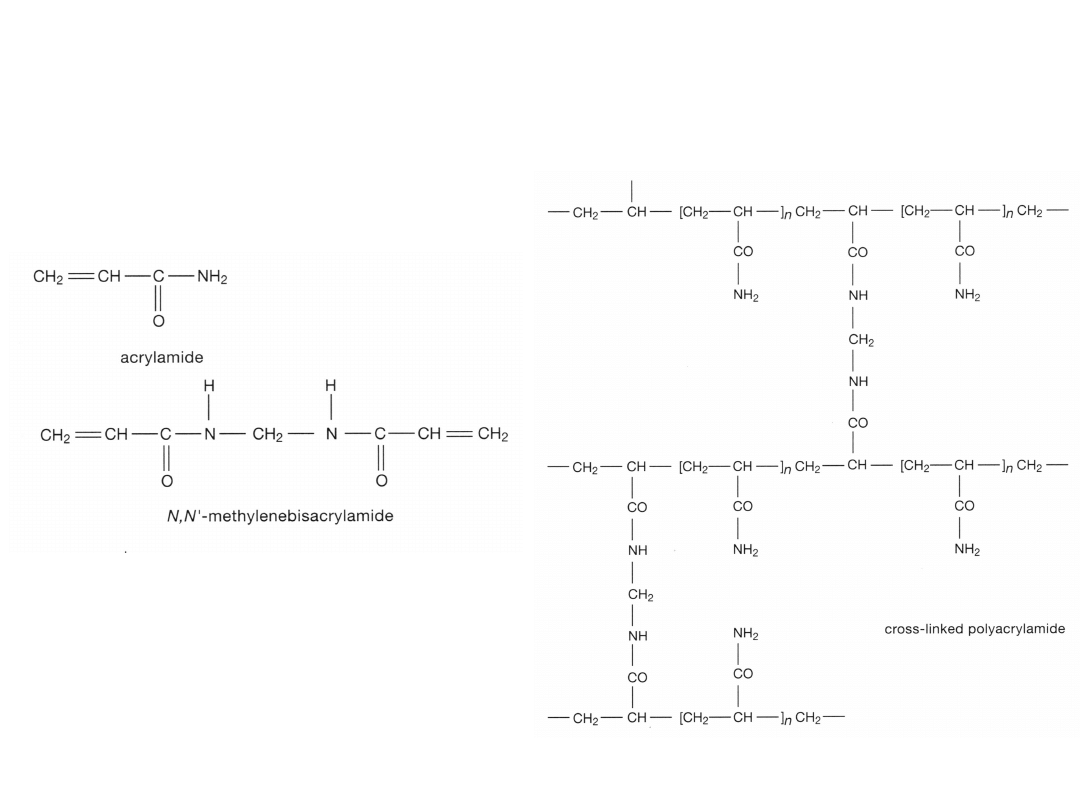
Akrylamid - struktura
Żele agarozowe
i poliakrylamidowe
Agaroza
1. Duża rozpiętość rozdziału,
przy stosunkowo małej
rozdzielczości.
2. Żele 4 - 0.5% pozwalają
na rozdział cząsteczek
o długościach 100-60 tys.
pz.
3. Technika Puls-Field zwiększa
zasięg rozdziału w granicach
50 tys. do 6-7 mln pz.
4. Nietoksyczna.
5. Łatwe przygotowanie żelu.
6. Żele horyzontalne
.
Poliakrylamid
1. Mała rozpiętość rozdziału,
ale wysoka rozdzielczość
rozdziału (do 1 pz).
2. Żele 3.5 - 20% pozwalają
na rozdział DNA o
długościach 10 pz do 2
tys pz.
3. Akrylamid jest związkiem
silnie toksycznym,
poliakrylamid może
zawierać niewielkie ilości
niespolimeryzowanego
akrylamidu.
4. Bardziej skomplikowane
przygotowanie żelu.
5. Żele wertykalne
(pomiędzy szybami).
Typy agarozy i ich
zastosowanie
1. Agaroza standard – temp. topnienia
90
-95
0
C;
temp.
krzepnięcia
35-45
0
C. Rozdziały fragmentów 100
- 25 tys. pz.
2. Agaroza
niskotopliwa
-
temp.
topnienia
40
-
85
0
C;
temp.
krzepnięcia 8 - 35
0
C. Rozdziały
fragmentów 10 - 10 tys. pz
(np. po reakcji PCR). Zatapianie
żywych komórek do elektroforezy w
zmiennym polu elektrycznym.
3. Elektroendoosmoza (EEO) pogarsza
możliwości
rozdziału
w
żelach
agarozowych.
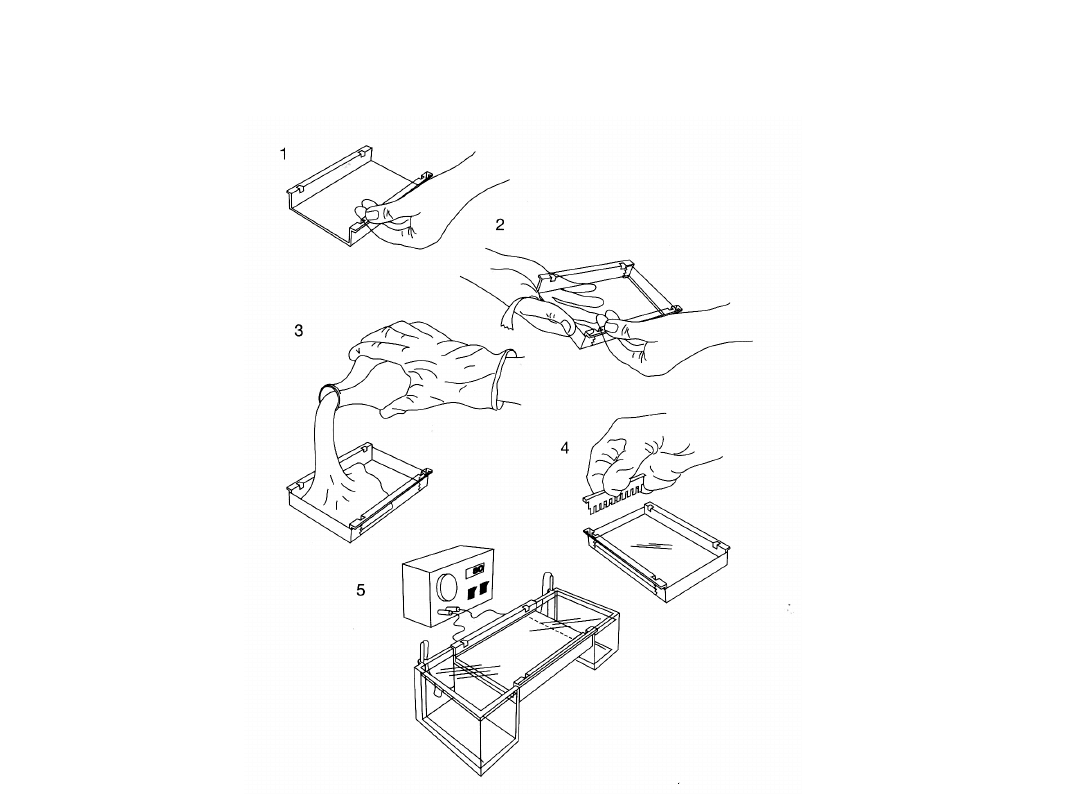
Wylewanie żelu
agarozowego
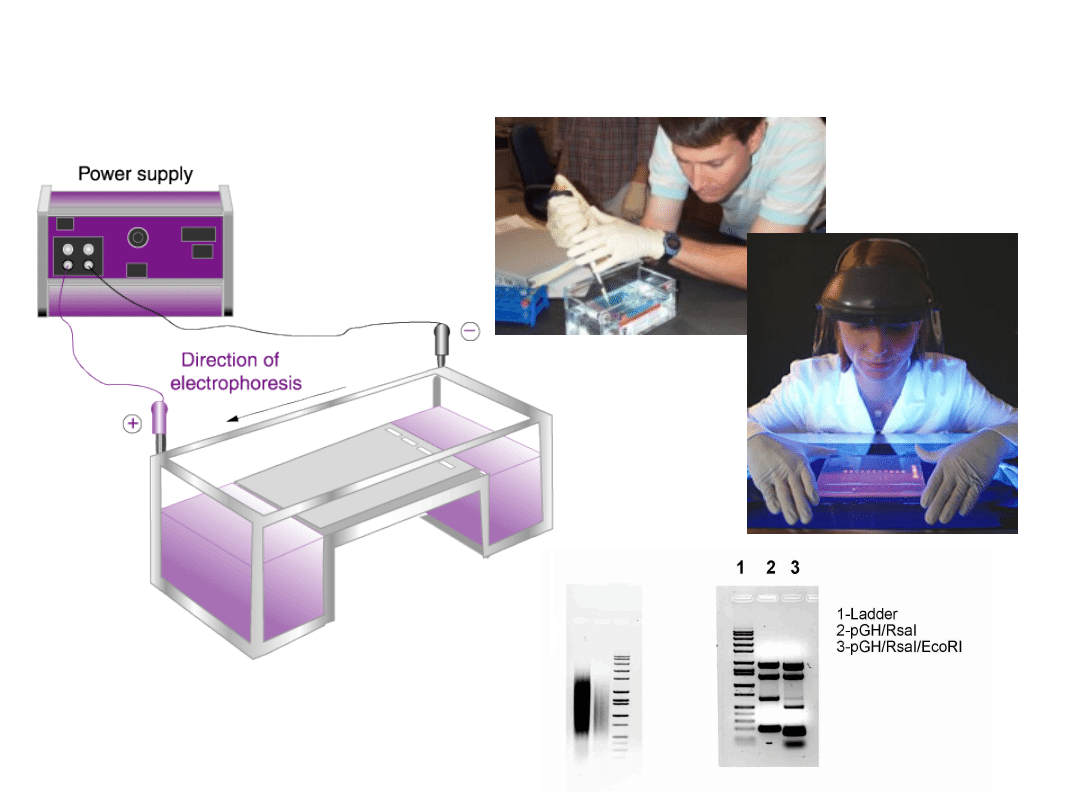
Nanoszenie próby, elektroforeza, obraz po
elektroforezie
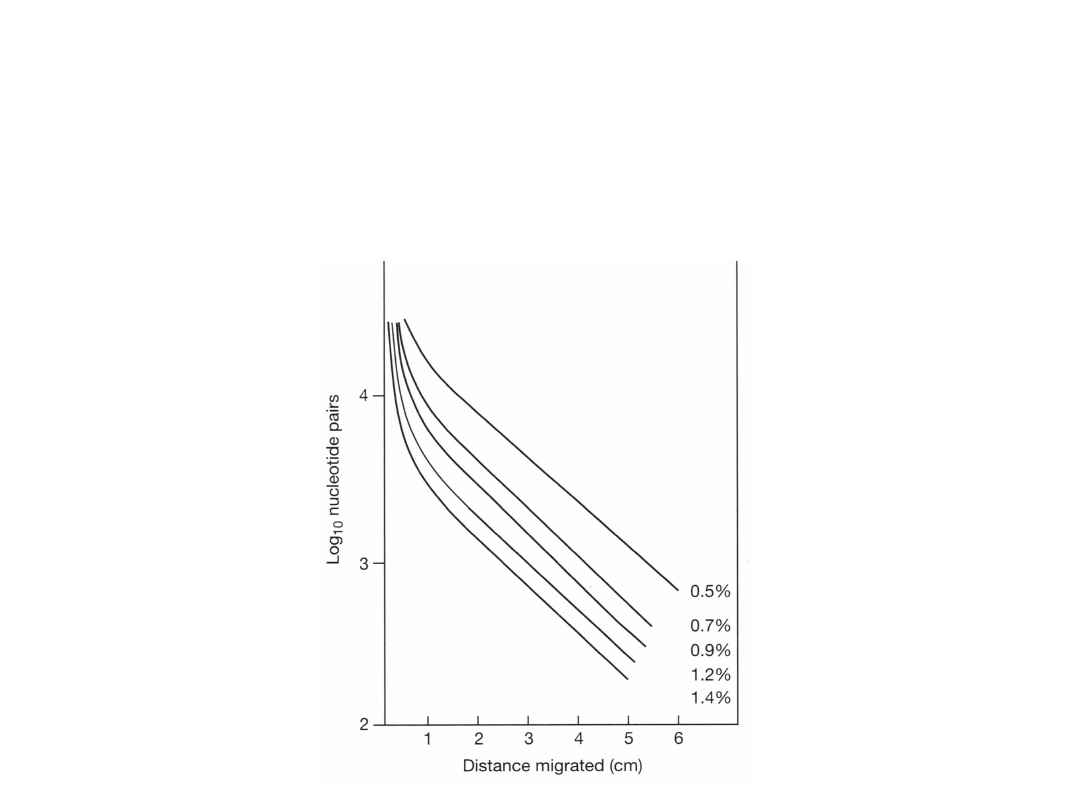
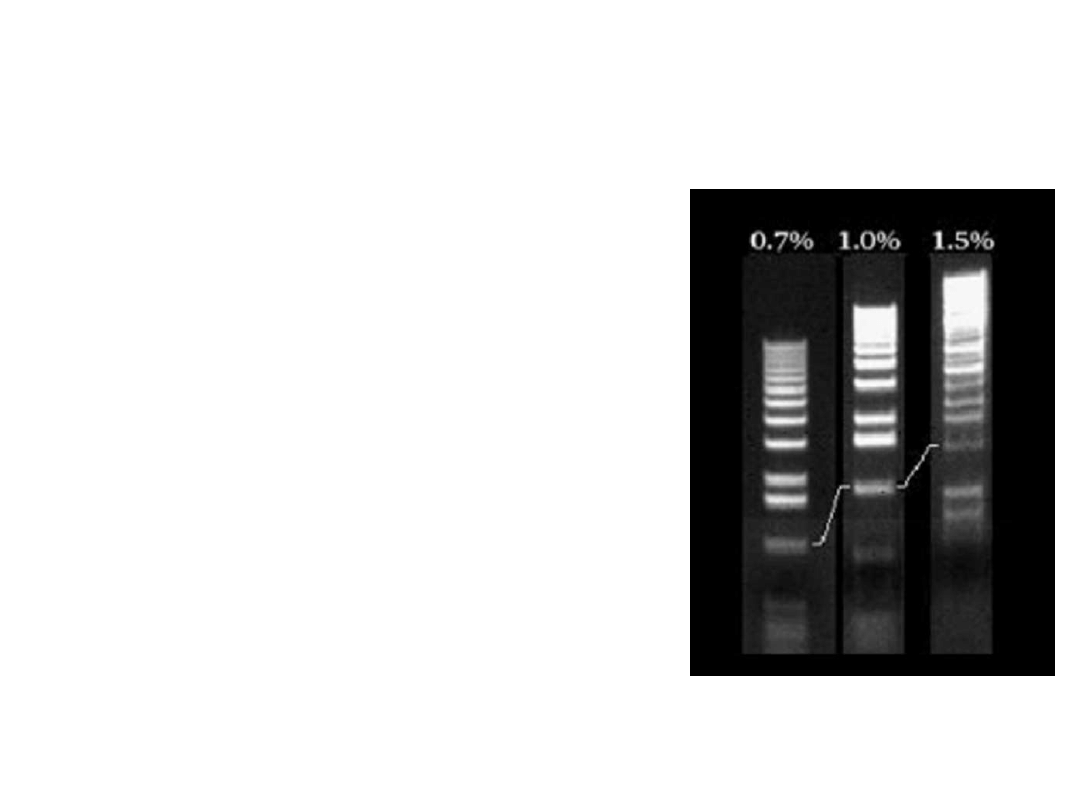
Elektroforeza w żelu agarozowym;
zależność szybkości migracji od
masy (długości) cząsteczek DNA i od
stężenia agarozy
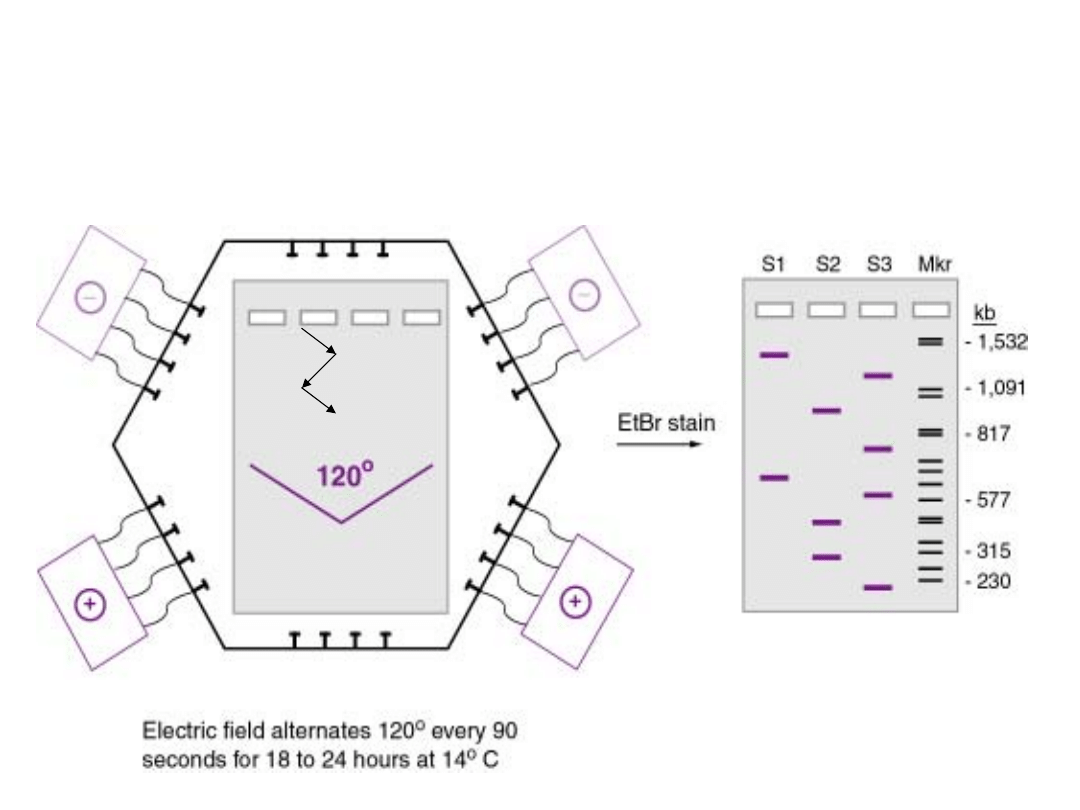
Elektroforeza w zmiennym
polu elektrycznym (PFGE)

Markery masy
cząsteczkowej
Fag lambda – 48502 pz, 40 genów
Hind III przecina go na 8 fragmentów
Eco RI na 6 fragmentów
Eco RV na 22 fragmenty
Lambda DNA/HindIII (pz)
plazmid pBR322 (4 361
pz) trawiony MspI
(fragmenty 67-622 pz)
23 130
9 416
6 557
4 361
2 322
2 027
564
125
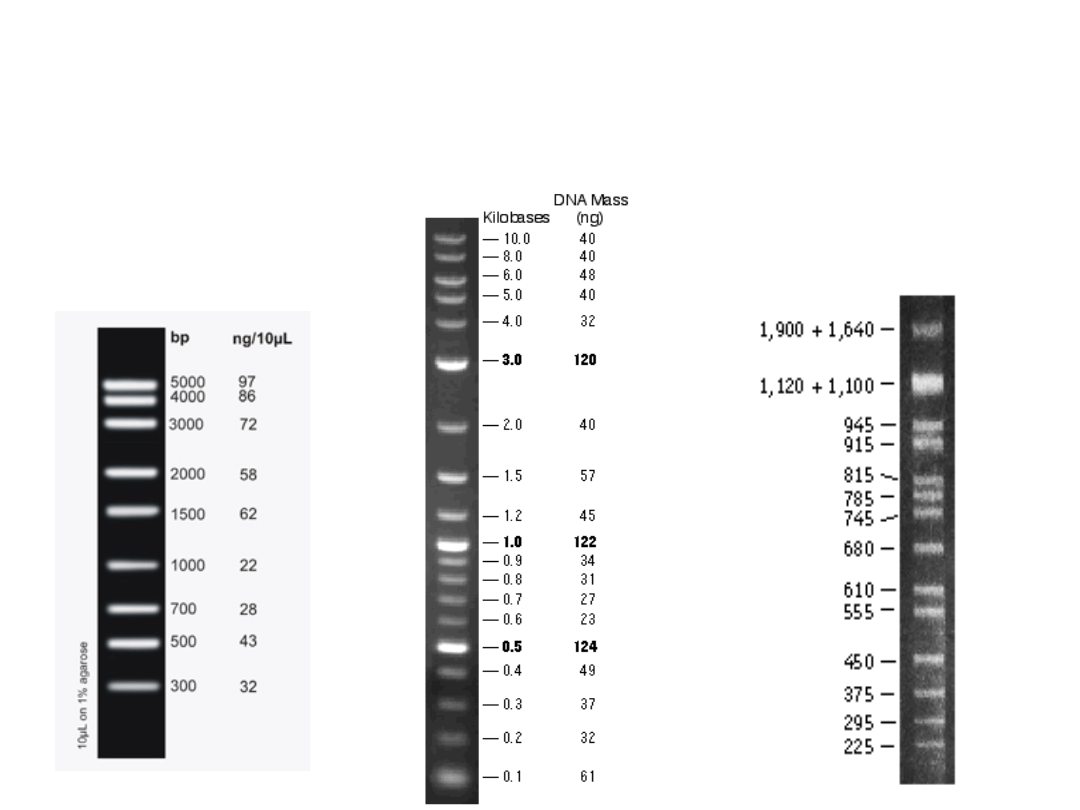
Markery masy cząsteczkowej
2-log ladder
(0.1-10 kb)
1 kb ladder
(0,3-5 kb)
Chromosomy drożdży
(225-1 900 kb)
Identyfikacja DNA po
elektroforezie
•Użycie radioaktywnego DNA do
elektroforezy
•Hybrydyzacja z sondą radioaktywną
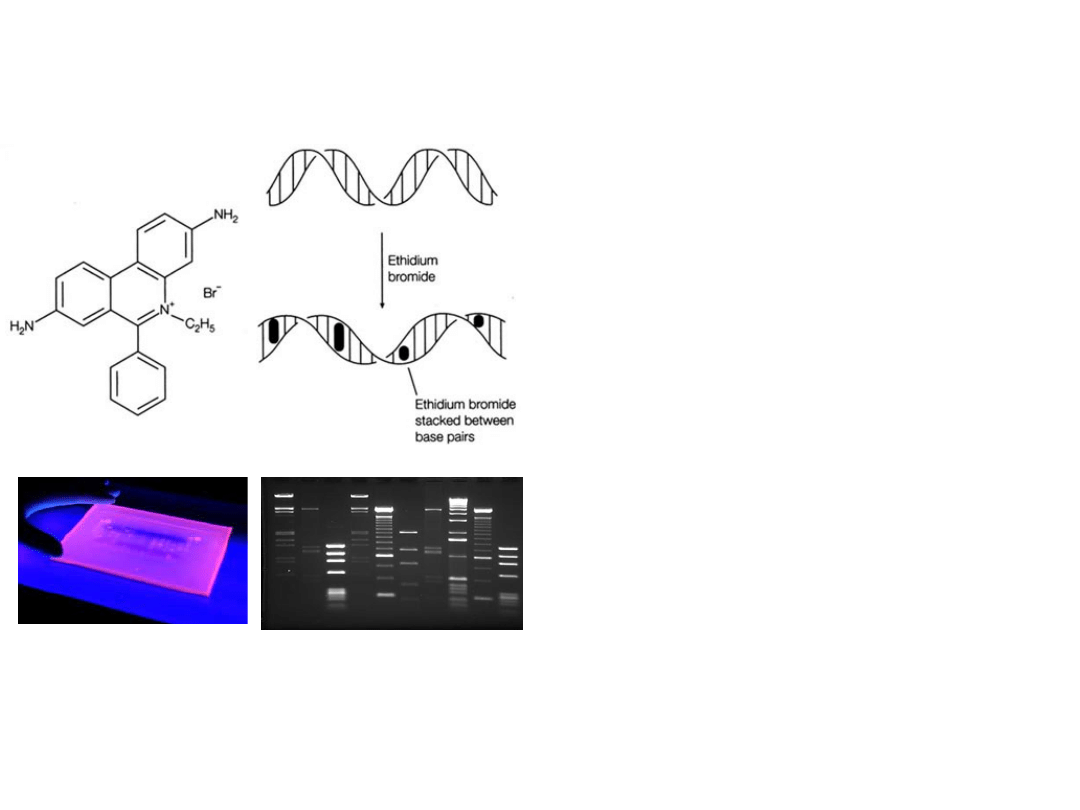
Interkalujący barwnik -
bromek etydyny (EtBr)
1. dodawanie do żelu –
obecny w trakcie
elektroforezy
2. płukanie żelu w
roztworze EtBr po
elektroforezie
Izolacja DNA z żelu
agarozowego
1. Elektroforeza do skrawków DEAE celulozy.
2. Elektroelucja do worka dializacyjnego
3. Odzyskiwanie DNA z niskotopliwej agarozy
4. Odzyskiwanie DNA z żelu przez adsorpcję na kulkach szklanych
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
Wyszukiwarka
Podobne podstrony:
BMiGO Wykład 5 Izolacja RNA, hybrydyzacja, RT PCR
wykład 2 cz.1, Teoria i analiza rynku- semestr V
Wykład 4 - Agencje ratingowe, Analiza i rating sektora finansowego
Modelowanie i analiza systemów - wykład III, Modelowanie i analiza systemów
Szeregi liczbowe mechatronika, wykłady i notatki, mechatronika, analiza ćwiczenia
Budownictwo Ogolne II wyklad 14 izolacje b
Wykład 9 Przybliżone metody analizy sieci
Modelowanie i analiza systemów - wykład II, Modelowanie i analiza systemów
BMiGO Wykład 4 PCR, Sekwencjonowanie
Wykłady DNA 12 (notatki)
3 Izolacja i analiza lipidów
Wyklad-02-AM1, Analiza matematyczna, Analiza matematyczna, Wykłady
Modelowanie i analiza systemów - wykład VI, Modelowanie i analiza systemów
Wykład 4 - 2 sem, 2 Semestr, Analiza matematyczna i algebra liniowa
Wykład 05 - NMR, Analiza instrumentalna
Wykład 3, Firmy farmaceutyczne – analiza 60 lat
Wykład 9 AR Metody analizy udziałów rynkowych i grup strategicznych
Międzynarodowe stosunki gospodarcze - wykłady, ekonomia, 2 rok, Analiza ekonomiczna, Miedzynarodowe-
więcej podobnych podstron