
Izolacja RNA,
hybrydyzacja, RT-
PCR
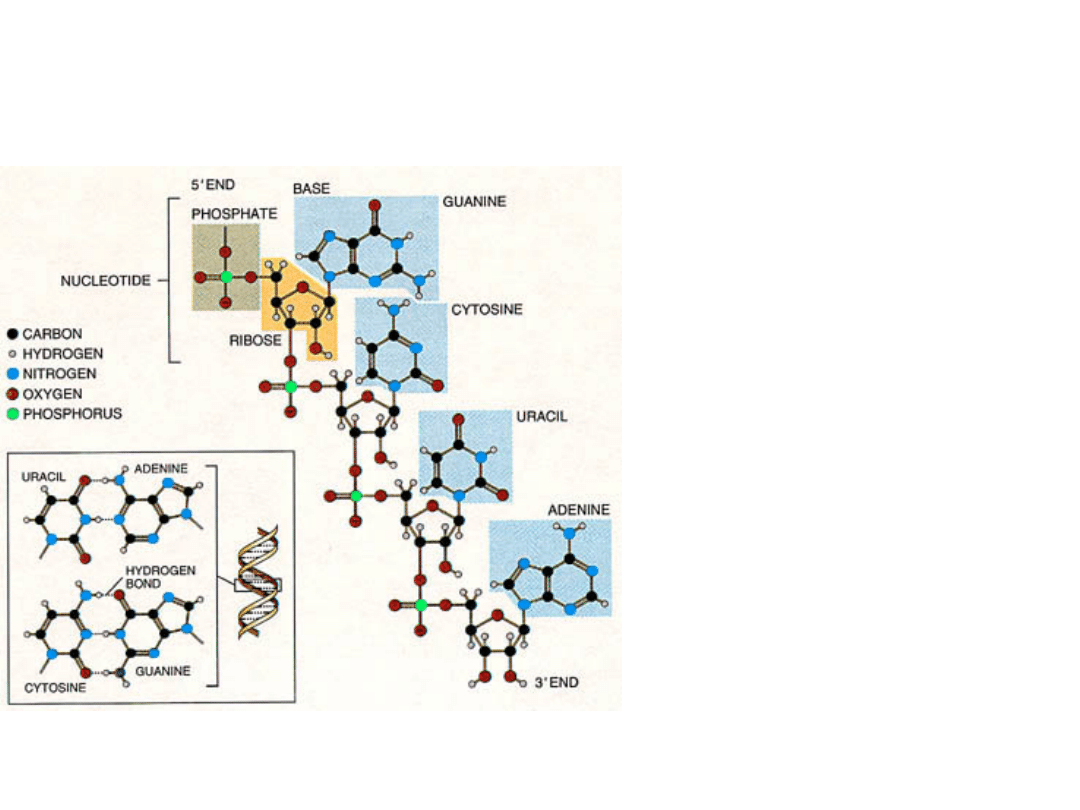
RNA
•Ryboza ma w RNA
grupę
hydroksylową
przy węglu 2’ przez co
cząsteczki
RNA
są
znacznie
bardziej
reaktywne
i mniej stabilne niż
cząsteczki DNA.
•Wszystkie
komórki
produkują
enzymy
degradujące RNA -
RNazy.
•RNazy są uwalniane
w trakcie lizy komórek
w związku z czym są
obecne na skórze i
przenoszone
przez
dotyk.
RNA
Typowa
komórka
ssacza
zawiera 10
-5
μg RNA, z tego
80 – 85% stanowi RNA
rybosomalny (28S, 18S, 5.8S
i 5S).
Większość pozostałej części
stanowią tzw. „małe RNA”
(tRNA, snRNA, RNAi).
Cząsteczki mRNA stanowią
od 1 do 5% populacji RNA.
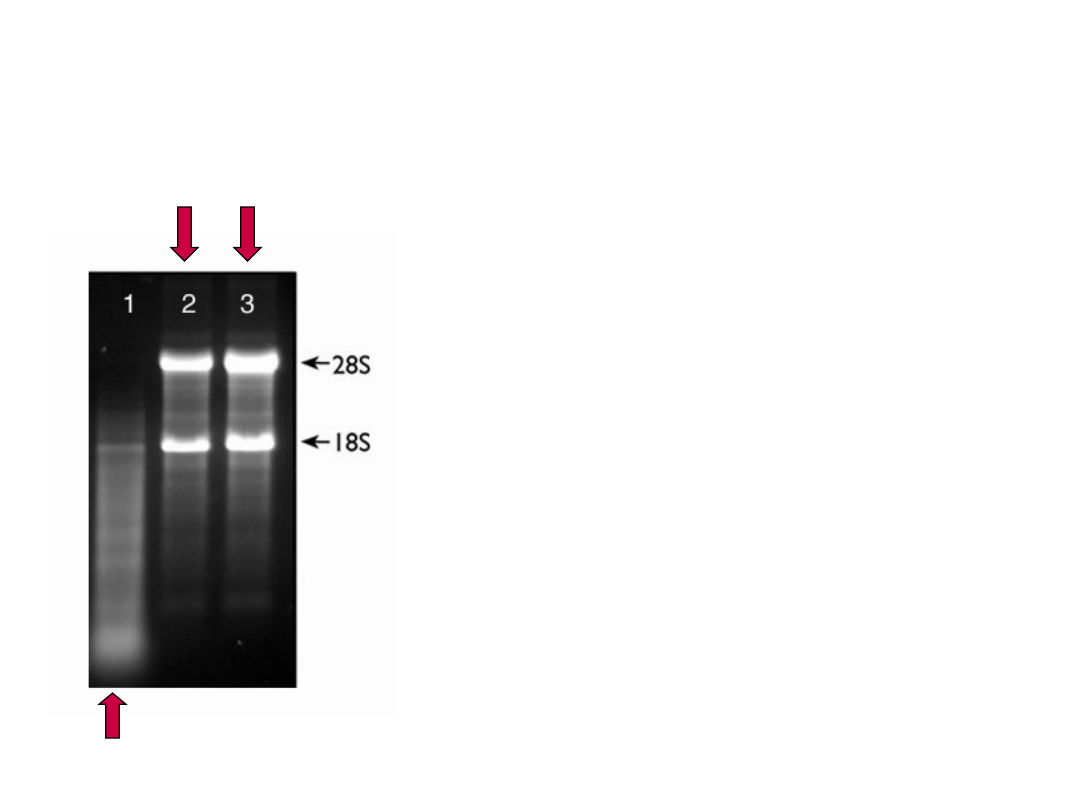
RNA całkowity, obraz prawidłowy
RNA zdegradowany
Elektroforeza

1. Sterylizacja szkła i plastików przed przystąpieniem
do pracy
2. Przygotowywanie roztworów w czystym szkle
i natychmiastowa sterylizacja w autoklawie
3. Roztwory używane do elektroforezy RNA powinny
być traktowane inhibitorem RNaz, np. DEPC
(Diethylpyrocarbonate) lub przygotowane na wodzie
traktowanej DEPC
4. Naczynie do elektroforezy powinno być również
płukane wodą traktowaną DEPC, najlepiej wydzielić
osobny
aparat
do elektroforezy dla pracy z RNA
5. Roztwory RNA należy trzymać w lodzie w czasie
pracy
a przechowywać w postaci wytrąconej pod etanolem
w temperaturze -70
o
C
6. W trakcie pracy z RNA należy zawsze używać
rękawiczek gumowych
Środki ostrożności przy pracy z RNA:
RNazy są enzymami stabilnymi, występują na powierzchni
skóry
i wszędzie gdzie obumierają komórki, dlatego
są na wszystkim czego dotykał człowiek lub zwierzę,
a także w wydychanym powietrzu
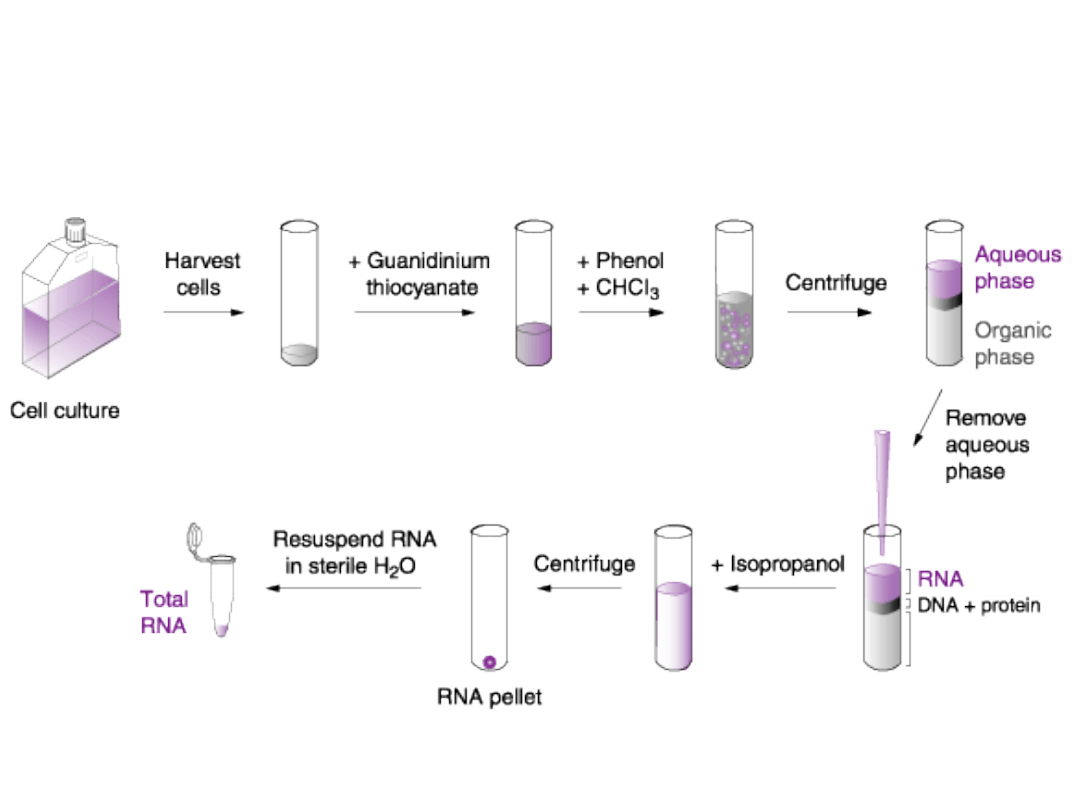
Przygotowanie całkowitego RNA
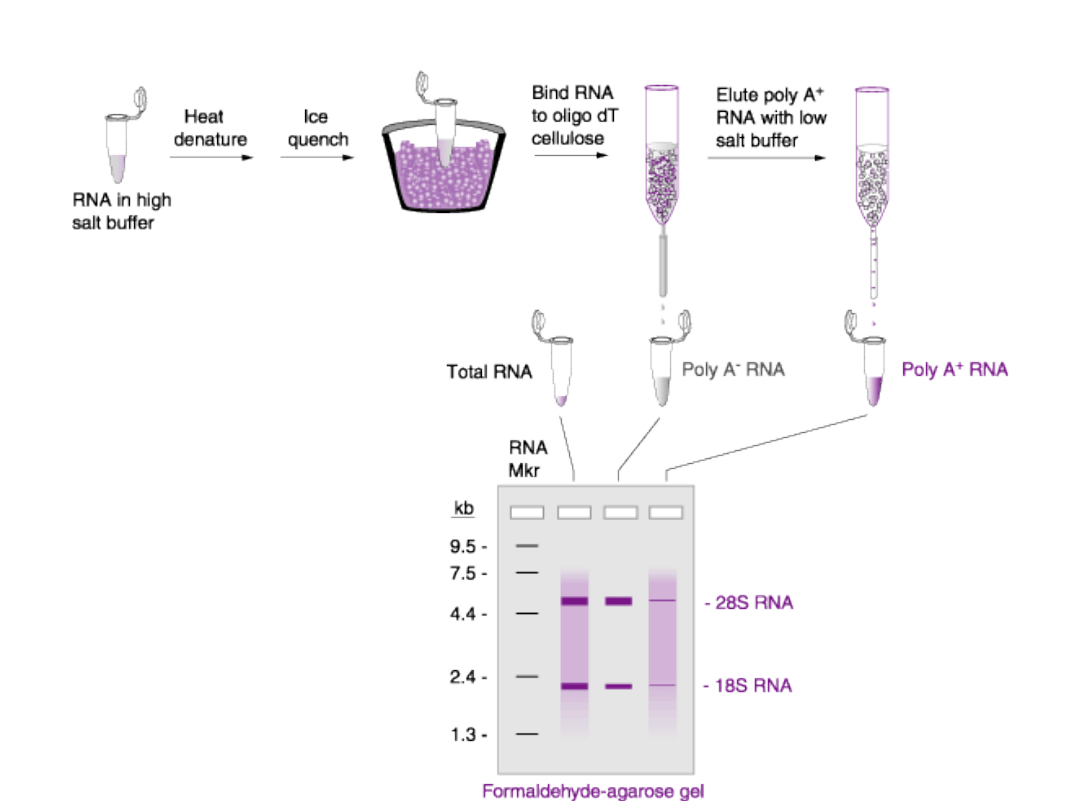
Przygotowanie poliA RNA – chromatografia powinowactwa

Elektroforeza RNA –
normalizacja, standardy
Normalizacja:
•Te same ilości RNA
•Te same ilości powszechnie występujących
transkryptów (tzw. housekeeping genes)
•Te same ilości poliA
+
RNA
(mierzone
metodami hybrydyzacyjnymi)
•Porównanie z syntetycznym pseudo-mRNA
Standardy masy
•Glioksalowane DNA
•Markery RNA syntetyzowane za pomocą
transkrypcji in vitro
•Rybosomalne RNA

Analiza sekwencji RNA lub DNA –
detekcja sekwencji
nukleotydowych
Masa molekularna
- elektroforeza w żelach
Metoda hybrydyzacji ze znakowana sondą
- znakowanie radioaktywnymi izotopami
-
znakowanie
biotyną
i
wykrywanie
przeciwciałami przeciw biotynie
• Elektroforeza jest metodą rozdzielania
zarówno cząsteczek DNA jak i RNA
• Detekcja
sekwencji
nukleotydowych
we fragmentach rozdzielonych drogą
elektroforezy
odbywa
się
poprzez
hybrydyzację ze znakowaną sondą

Metody detekcji sekwencji
nukleotydowych, hybrydyzacja
Ekstrakcja DNA
Cięcie enzymami restrykcyjnymi
Elektroforeza DNA w żelu i
przeniesienie rozdzielonych
fragmentów na membranę
nitrocelulozową
Reakcja hybrydyzacji z
radioaktywną sondą
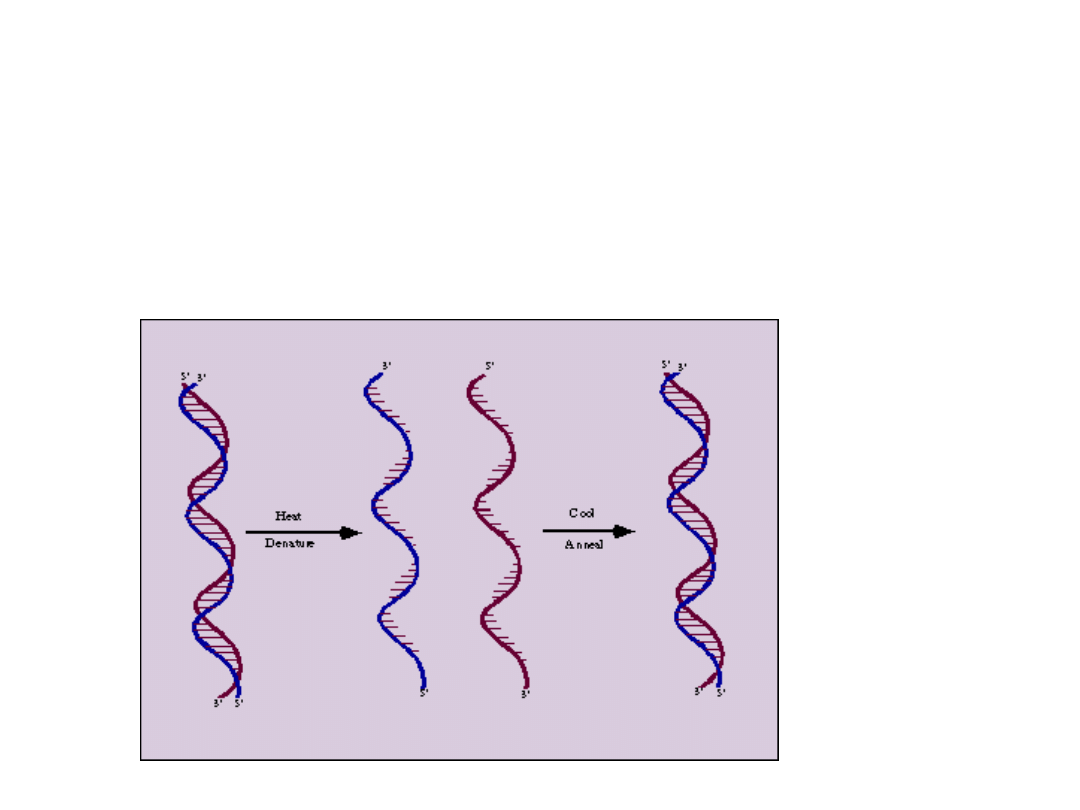
Metoda hybrydyzacji DNA (lub RNA):
denaturacja badanego DNA
denaturacja sondy
renaturacja DNA w obecności sondy i powstanie
cząsteczek hybrydowych
Elektroforeza w żelu
agarozowym
Bufor obciążający:
Błękit bromofenolowy
Cyjanian ksylenu
Glicerol
Bufory używane do elektroforezy:
TAE: Tris - octan-EDTA
TBE: Tris – boran-EDTA
Metodą hybrydyzacji można wykrywać
obecność określonych sekwencji
nukleotydowych w DNA i w RNA
• Elektroforeza DNA lub RNA w
żelu agarozowym.
• Przenoszenie na filtr nylonowy
lub nitrocelulozowy.
• Denaturacja cząsteczek.
• Renaturacja
w
obecności
dużego nadmiaru znakowanej
sondy
(krótkiego,
jednoniciowego fragmentu DNA
zawierającego interesującą nas
sekwencję nukleotydową).
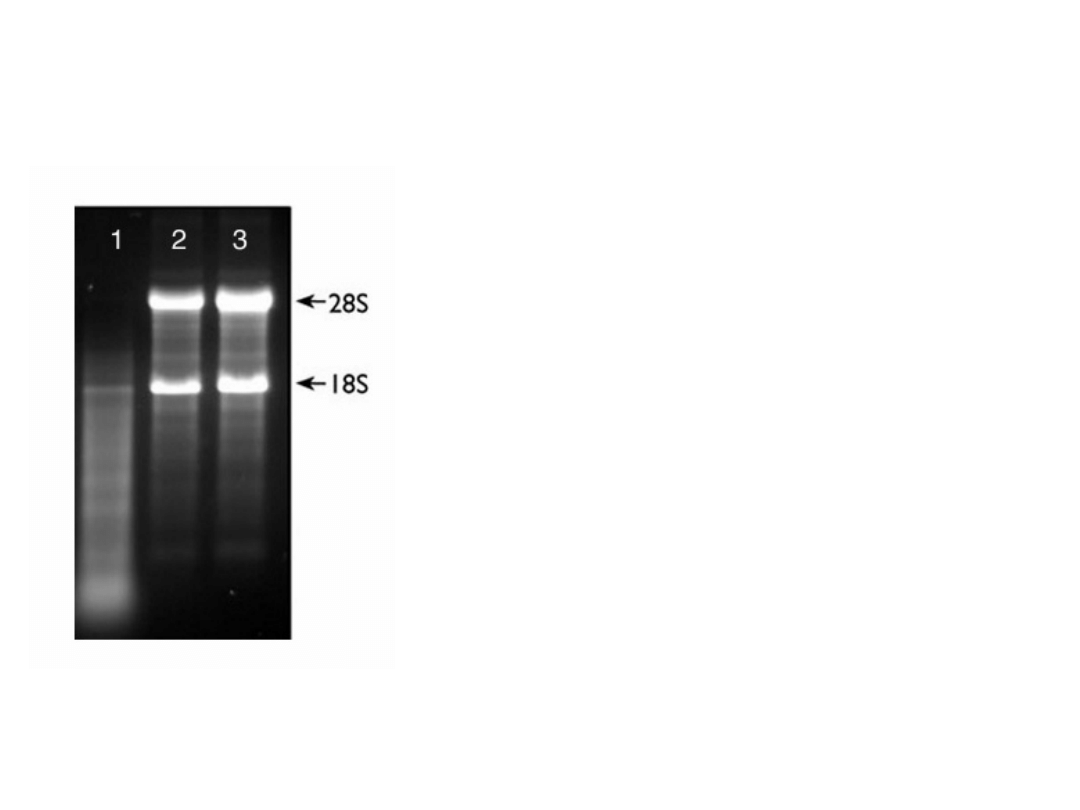
Wynik elektroforezy
całkowitego RNA
1-Preparat
zdegradowa
ny
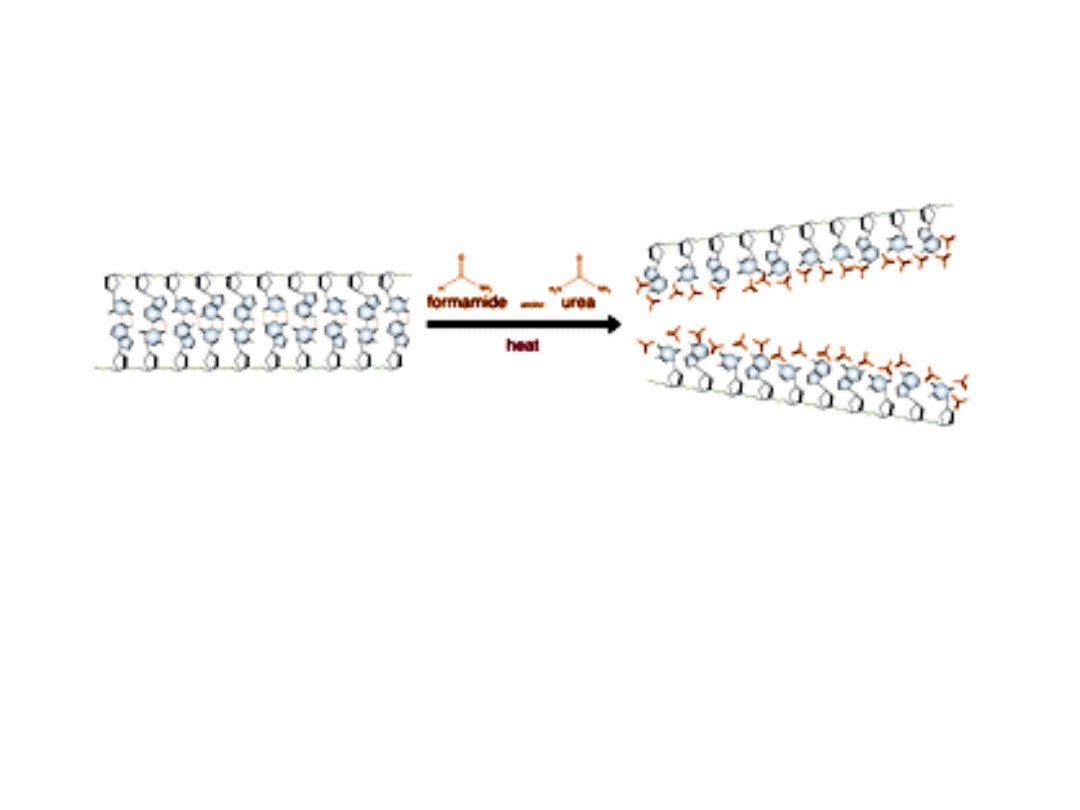
Denaturacja DNA zachodzi w podwyższonej
temperaturze lub w obecności wysokich stężeń
formamidu lub mocznika
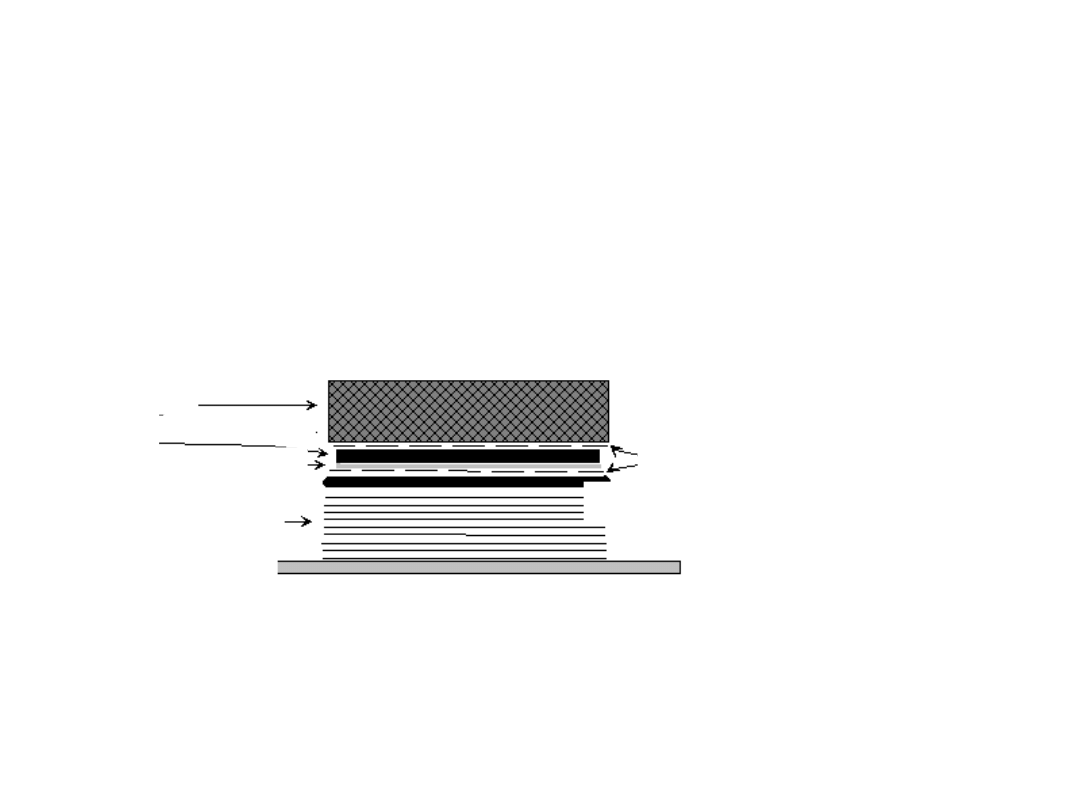
Przenoszenie materiału z żelu na filtr może
odbywać
się
z
wykorzystaniem
sił
kapilarnych
(blotting)
lub
poprzez
przyłożenie
pola
elektrycznego
(elektroblotting)
Bibuła Whatmann 3MM
Gruby papier lub bibuła Whatmann

Piec hybrydyzacyjny
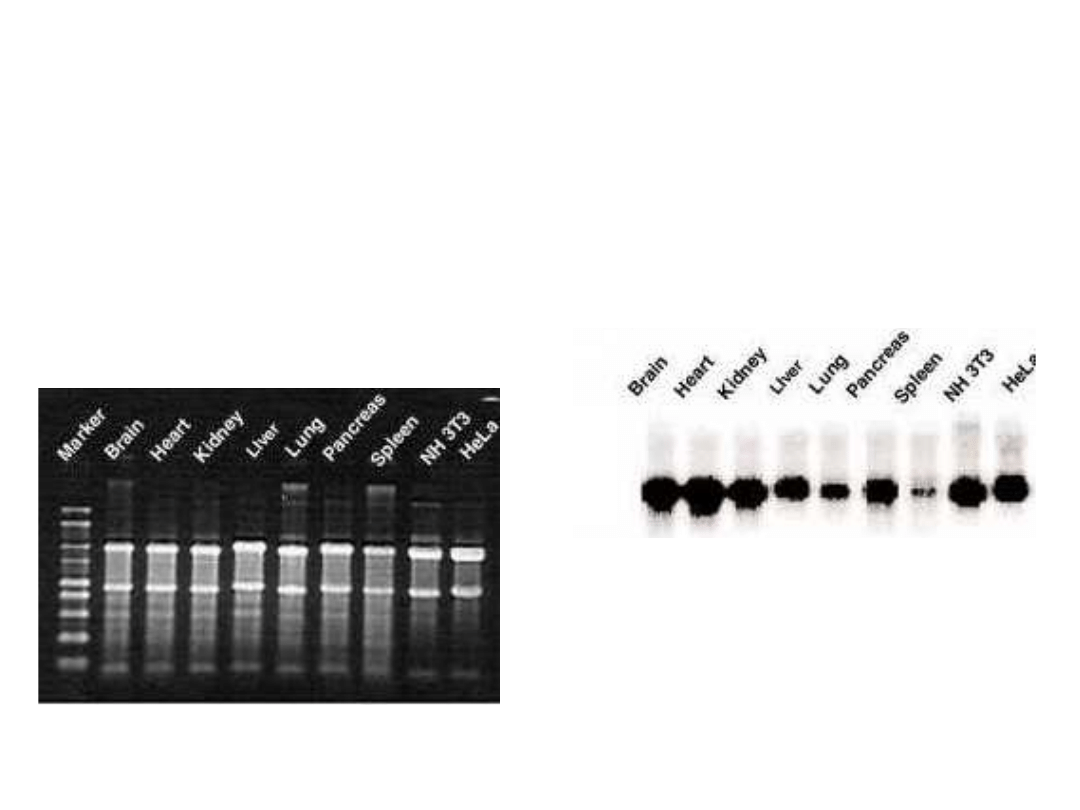
Wyniki hybrydyzacji
mRNA
z sondą GADPH
(dehydrogenaza 3-
fosforanu aldehydu
glicerolowego)
W
wyniku
hybrydyzacji
otrzymujemy
radioaktywny albo znakowany biotyną
prążek
w
miejscu,
w którym na żelu była obecna interesująca
nas sekwencja nukleotydowa
Elektroforeza
preparatów RNA z
różnych tkanek
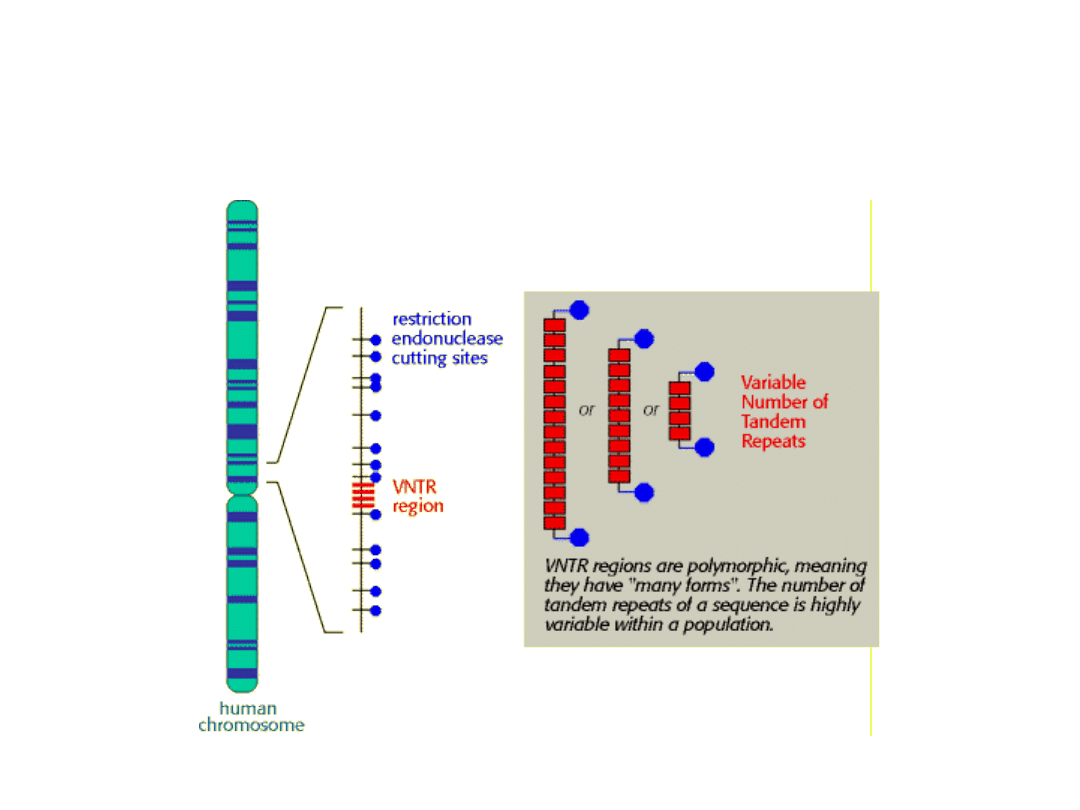
Regiony o zmiennnej liczbie powtarzających się
tandemowo sekwencji
(VNTR – variable number of tandem repeats)
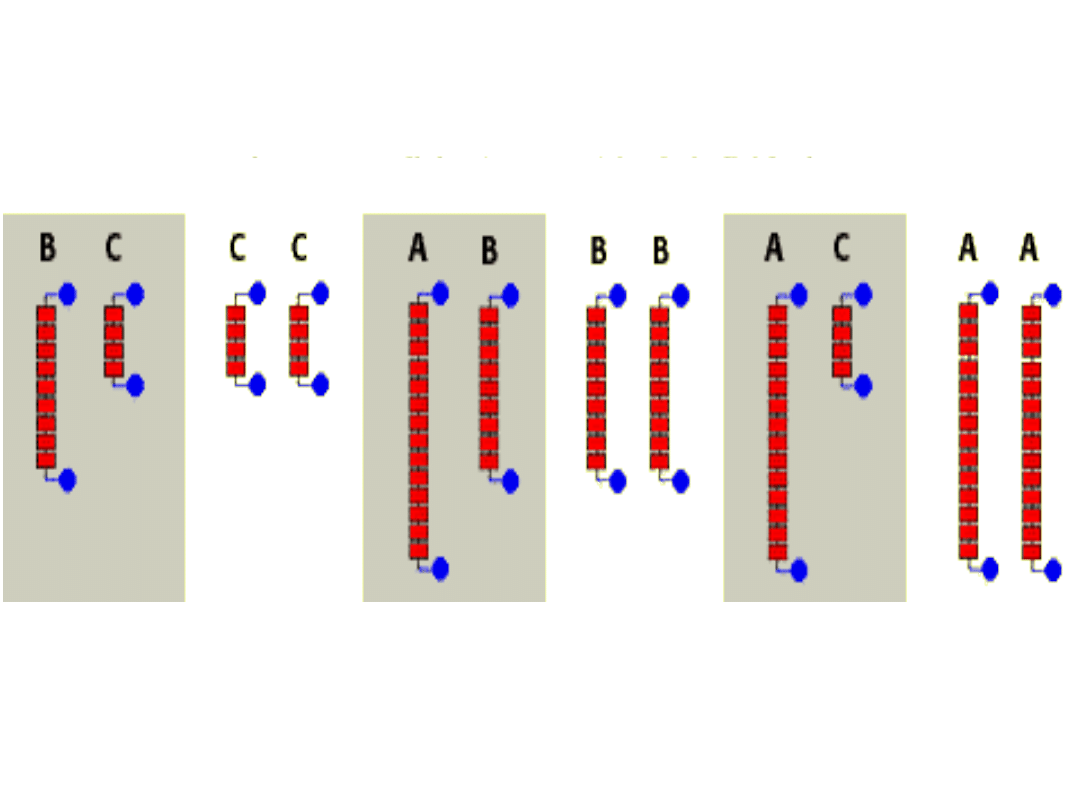
Trzy typy alleli VNTR występujące w różnych
kombinacjach u 6 osobników
Polimorfizm powtarzających się sekwencji

1 2 3 4 5 6
Wynik hybrydyzacji
Osobnik
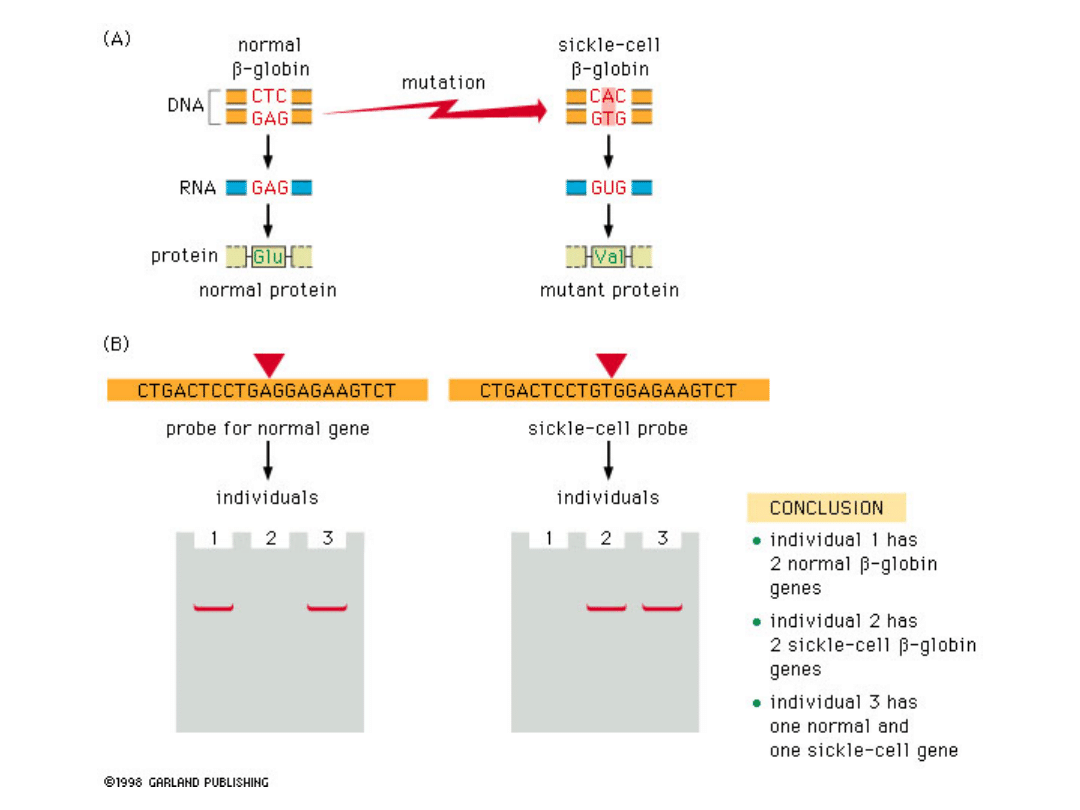
Anemia sierpowata

PCR rozpoczynający się od
wyizolowanych cząsteczek
RNA
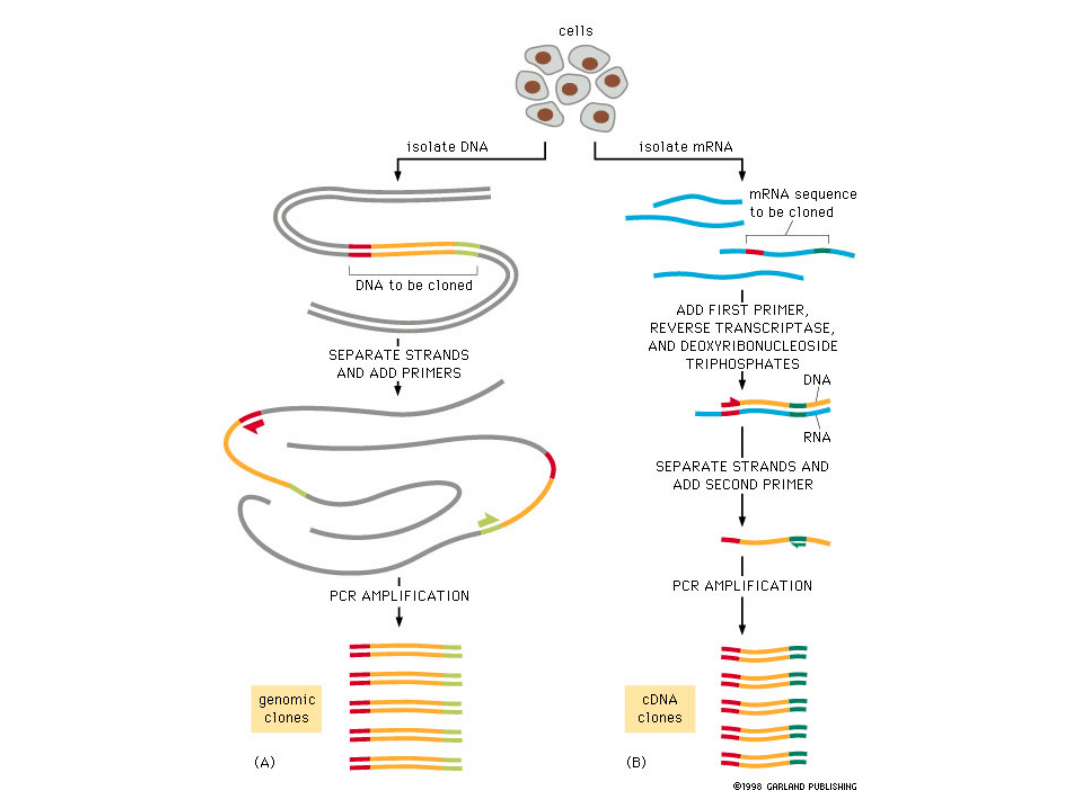

Reverse Transcriptase Mediated
PCR
(RT-PCR)
Polega
na
połączeniu
reakcji
odwrotnej transkrypcji z reakcją
klasycznego PCR
Służy
wykrywaniu
rzadkich
transkryptów
Stosuje się go także w aplikacjach:
1. RACE 5’: klonowanie końców 5'
transkryptów
2. RACE 3’: klonowanie końców 3’
transkryptów
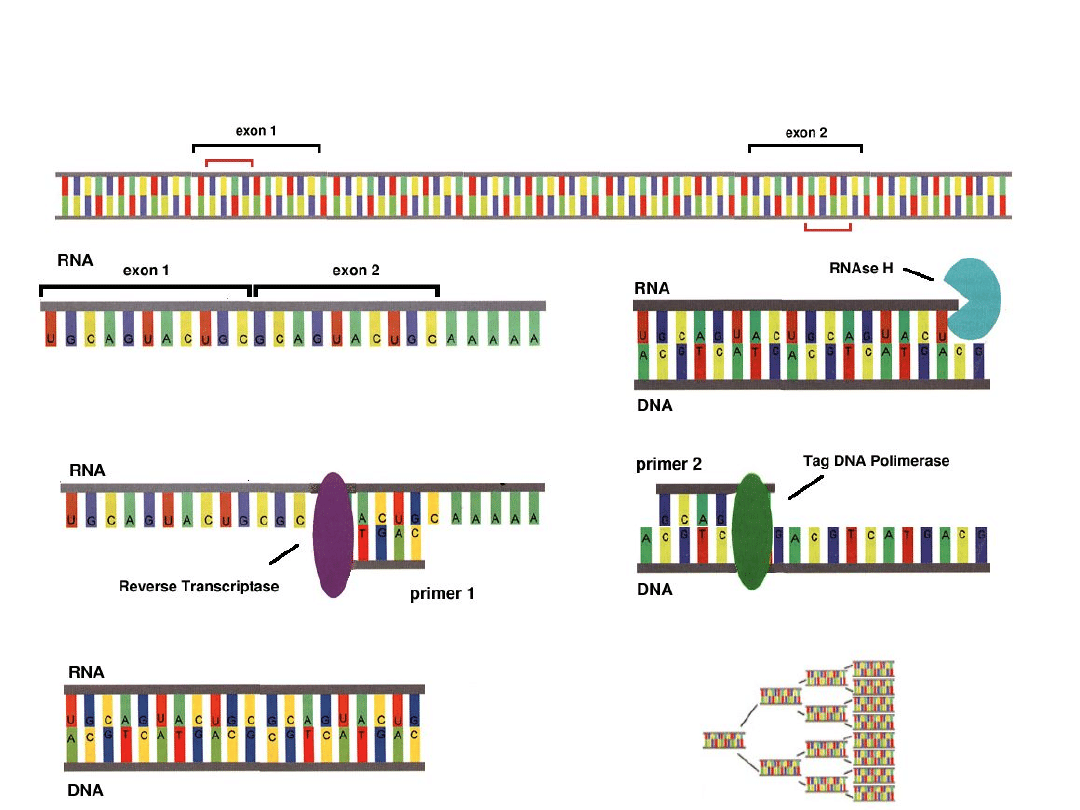
Reverse Transcriptase Mediated PCR
(RT-PCR)
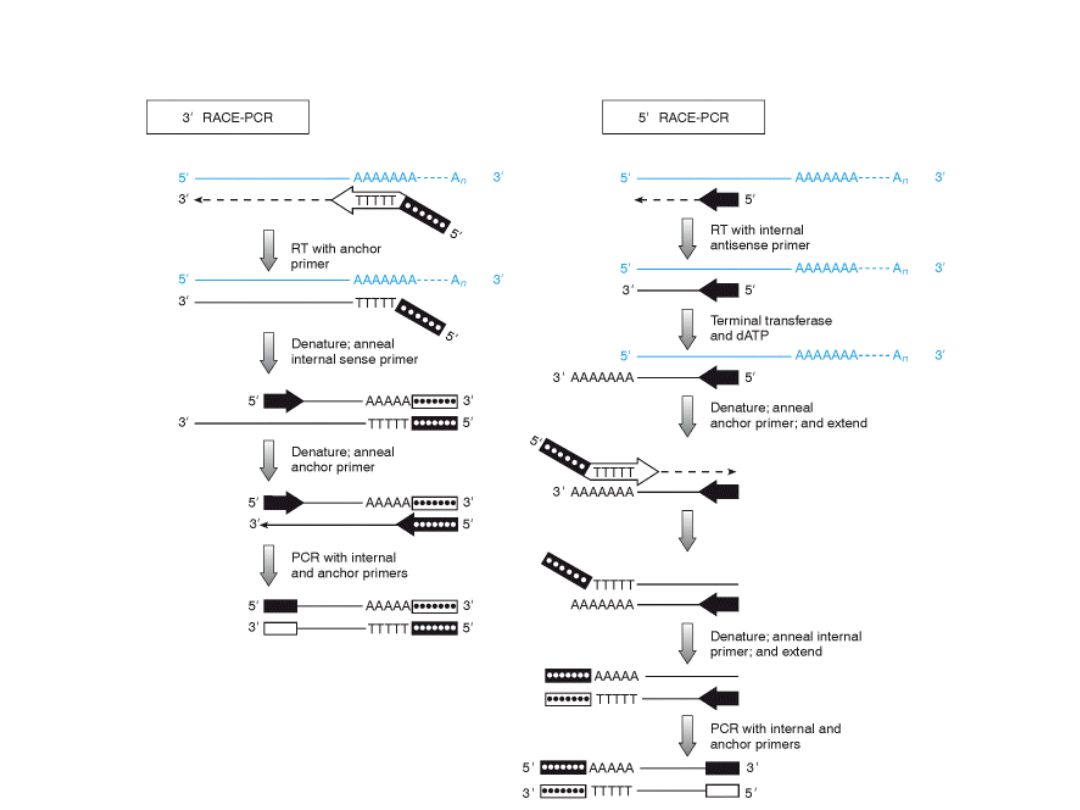
RACE (Rapid Amplification of cDNA Ends) to
metoda wzbogacania cDNA w końcowe fragmenty
sekwencji mRNA
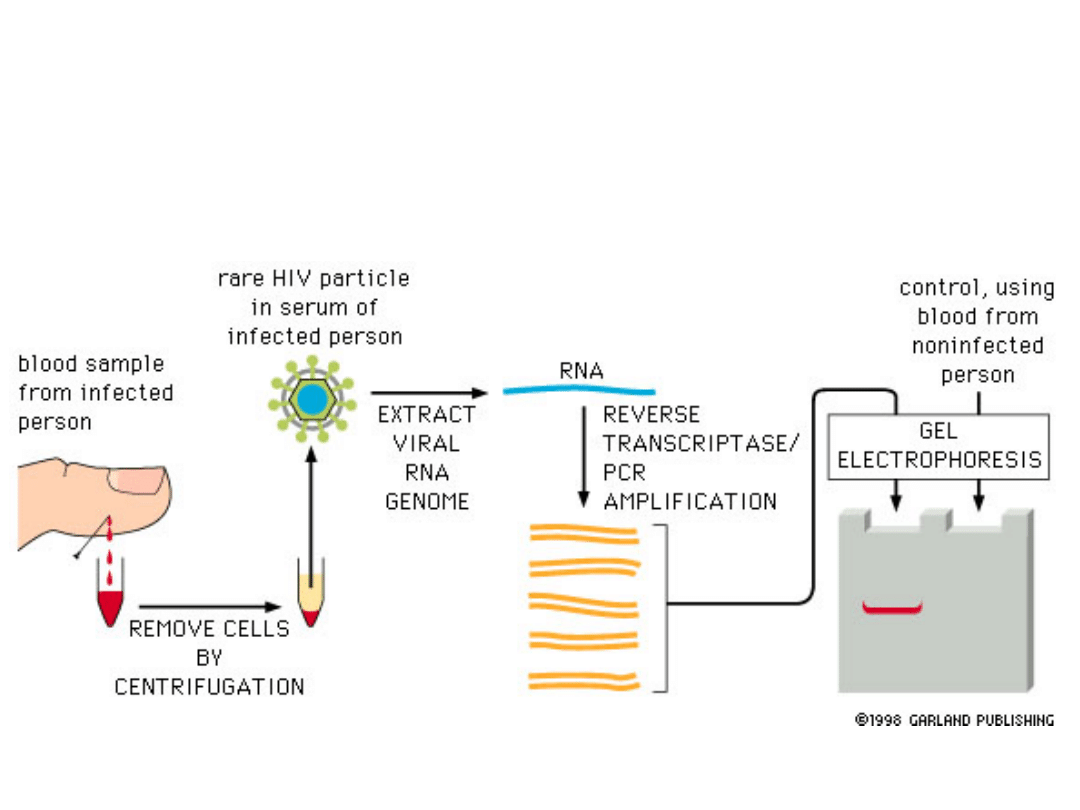
Wykrywanie wirusa HIV metodą RT-PCR

Ilościowy PCR (Q-PCR)
3.
Pomiar
ilości
produktu
powstającego
we
wczesnej
fazie logarytmicznego wzrostu
(Real-time PCR).
1. Pomiar końcowego produktu
PCR
przy
użyciu
standardów
zewnętrznych
lub
wewnętrznych.
2. Pomiar produktu w czasie
(materiał pobierany w trakcie
reakcji PCR).

Real-Time
PCR
Aparat do PCR
sprzężony jest ze
spektrofotometrem
i wyposażony
w oprogramowanie
konieczne do analizy
danych.

Najlepsze metody określania ilości powielanej sekwencji
opierają się na pomiarze przyrostu produktu PCR w czasie
rzeczywistym.
Istnieją dwa typy metod różniące się specyficznością sondy
fluorescencyjnej
Barwniki interkalujące do
DNA (bromek etydyny) lub
inne, których fluorescencja
wzrasta pod wpływem
związania do dwuniciowego
DNA.
Obecnie najczęściej
stosowanym barwnikiem jest
SYBR
®
Green I.
Zalety:
Sonda jednakowa dla każdej
sekwencji. Amplifikacja
sygnału.
Wady:
Wiązanie z niespecyficznymi
produktami PCR. Zależność
od długości produktu.
Specyficzne sondy
zawierające barwnik
fluorescencyjny oraz
wygaszacz, zasada
działania z reguły
związana ze
zjawiskiem FRET
Zalety:
Detekcja
specyficznego
produktu. Brak
zależności od długości
produktu.
Wady:
Konieczność syntezy
specyficznej sondy dla
każdego produktu.
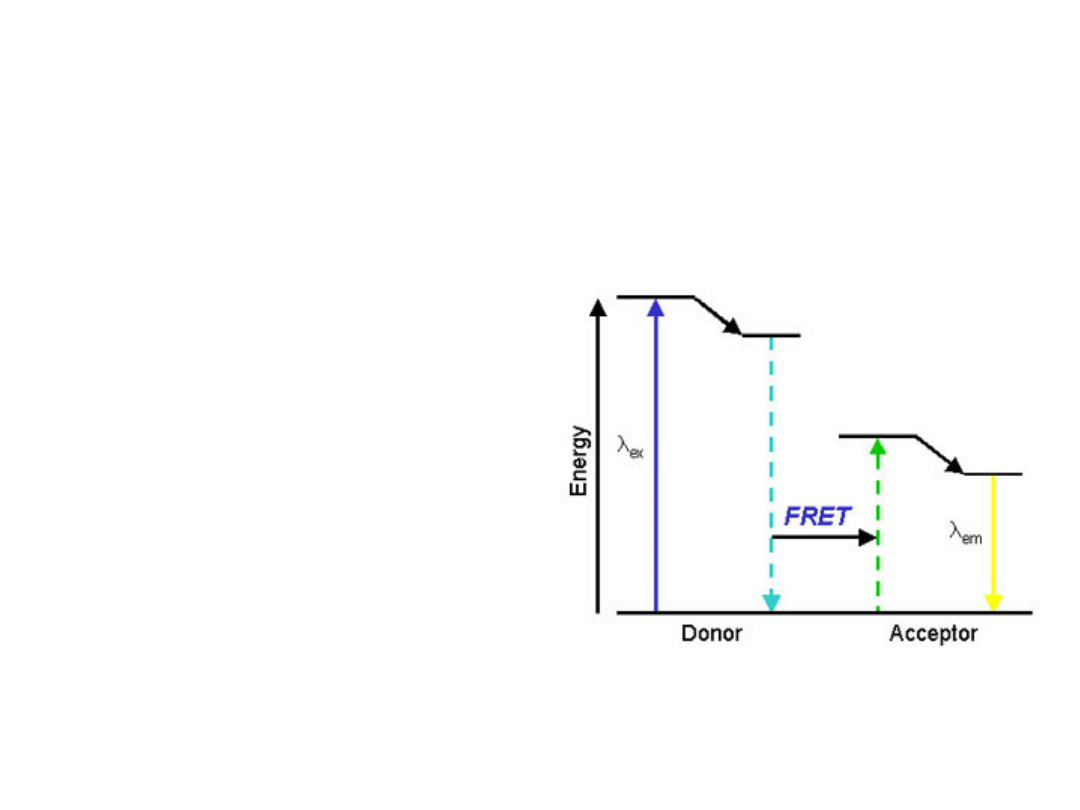
Zasady fizyczne FRET
(fluorescence resonance energy
transfer)
Transfer energii zachodzi
pomiędzy dwoma dipolami
o nakładających się
widmach emisji/wzbudzenia
i odpowiednim ustawieniu
względem siebie.
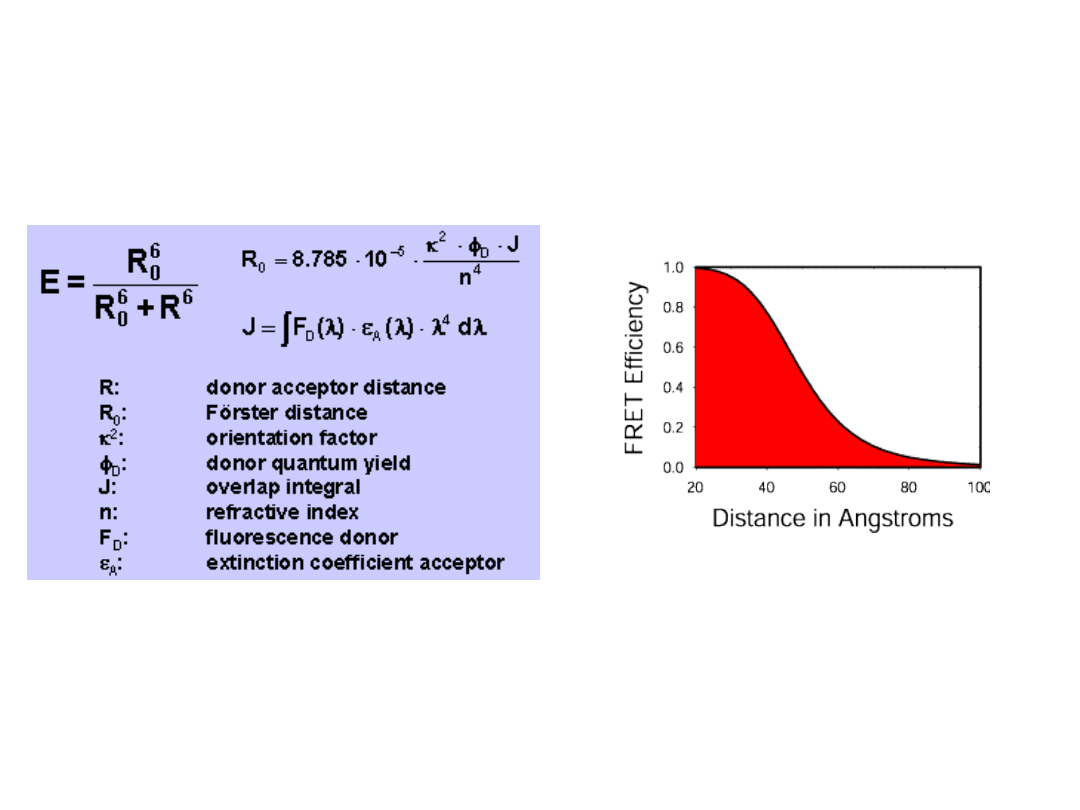
Efektywność transferu energii jest odwrotnie
proporcjonalna do r
6
, dlatego FRET występuje
pomiędzy cząsteczkami oddalonymi o 1-10 nm
(10-100 Å)
R
0
reprezentuje odległość, przy której
występuje 50% efektywność
przenoszenia energii
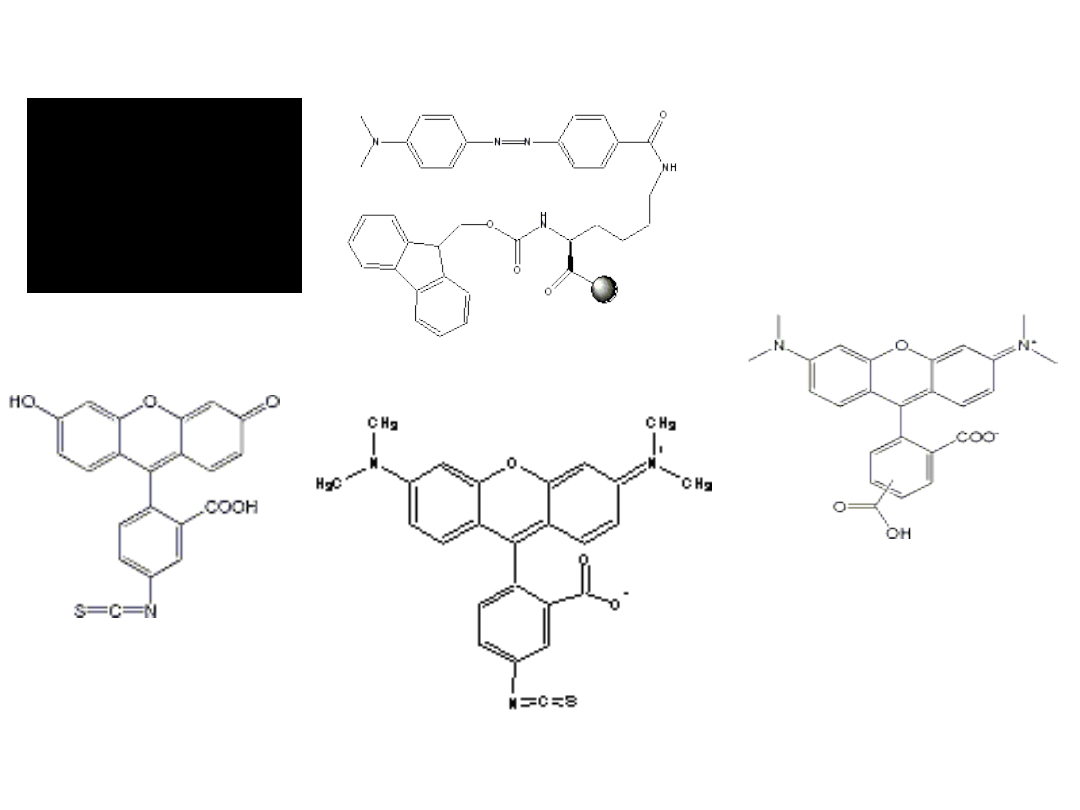
Przykładowe związki stosowane w technikach wykorzystujących FRET
fluoresceina
Izotiocjanian
fluoresceiny
(FITC)
Izotiocjanian
Tetrametylorodaminy (TRITC)
DABCYL, kwas [4-
((4-
(dimetyloamino)feny
lo)azo)benzoesowy]
5(6)TAMRA:
5-(and-6)-Karboksy
tetrametylorhodami
na
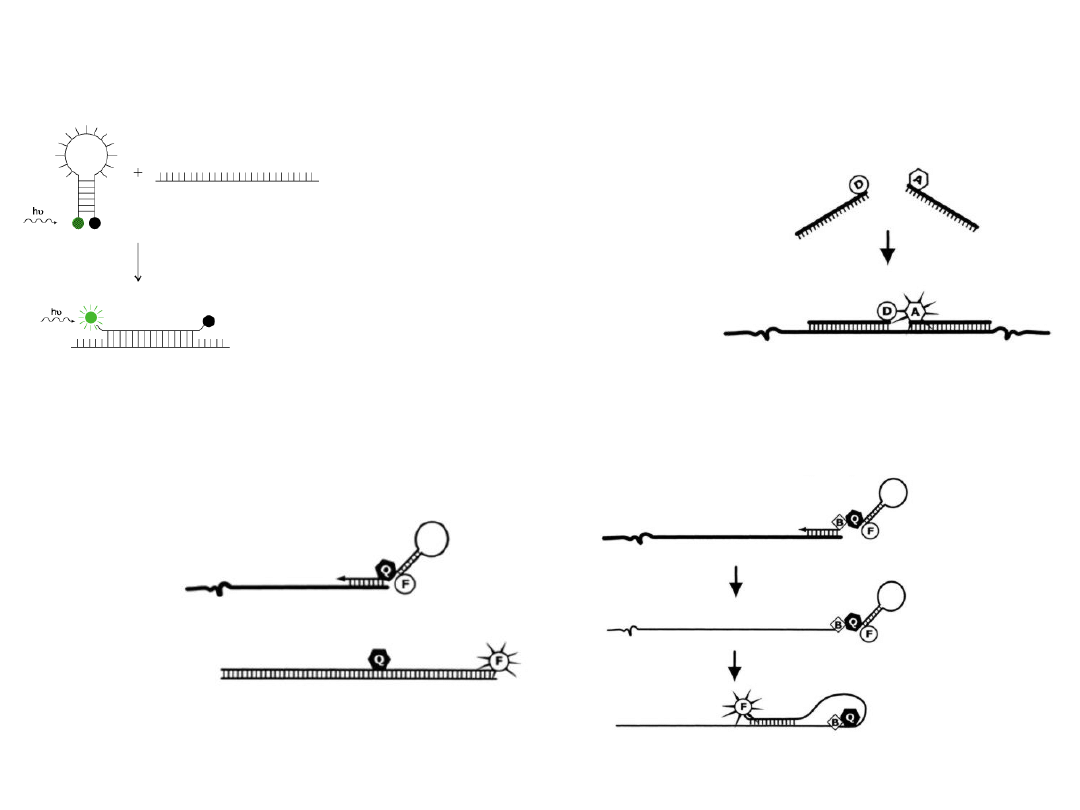
Sonda zawiera na końcach
komplementarne sekwencje tworzące
szpilkę do włosów
Sondy do RT-PCR
Dwuczęściowa sonda
wiążąca się do sąsiadujących
sekwencji
Dwa różne sposoby powiązania sondy ze starterem
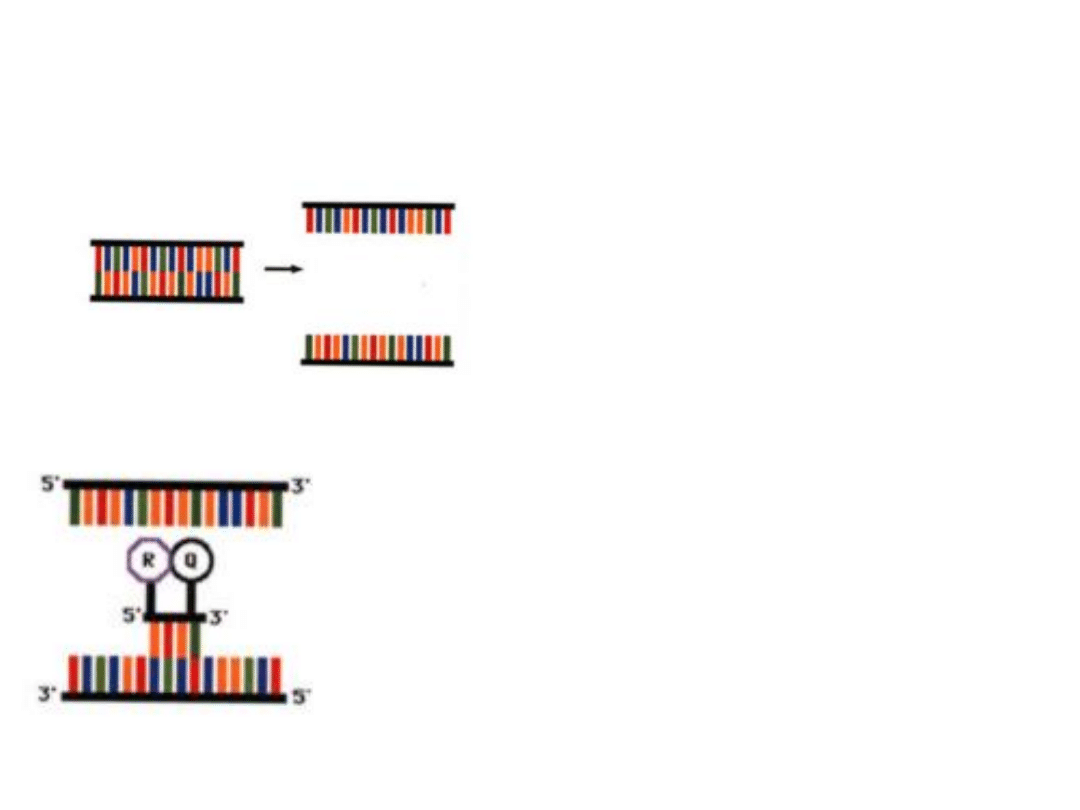
1.
Produkt
DNA,
zdenaturowany
na dwie pojedyncze nici.
2. Sonda DNA, nie ulegająca wydłużeniu
(zablokowane końce).
Sonda ma barwnik fluorescencyjny na
końcu 5’ (R) i wygaszacz (Q) na końcu 3’.
W czasie hybrydyzacji (annealing) sonda
wiąże się do matrycy DNA.
Temperatura renaturacji dla sondy jest o
5-10
0
C wyższa niż dla starterów.
Sondy 5’ nukleazowe – schemat działania
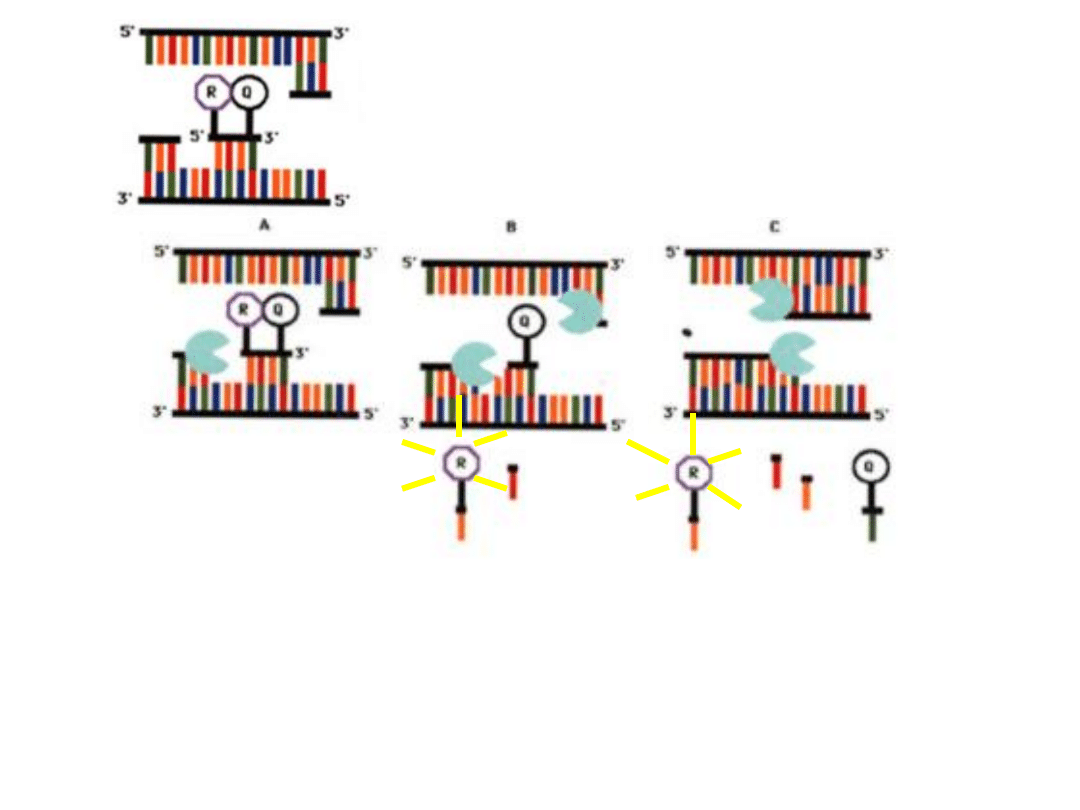
3. Przyłączanie starterów do DNA
zawierającego sondę jest możliwe
po obniżeniu temperatury
4. Polimeraza Taq syntetyzuje nowe fragmenty DNA
rozpoczynając
od starterów (A).
Natknąwszy się na sondę DNA polimeraza odcina
nukleotydy począwszy od końca 5’ (aktywność 5’3’
egzonukleazy) (B).
W trakcie degradacji wygaszacz jest oddzielany od barwnika
fluorescencyjnego co wyzwala emisje światła (C).
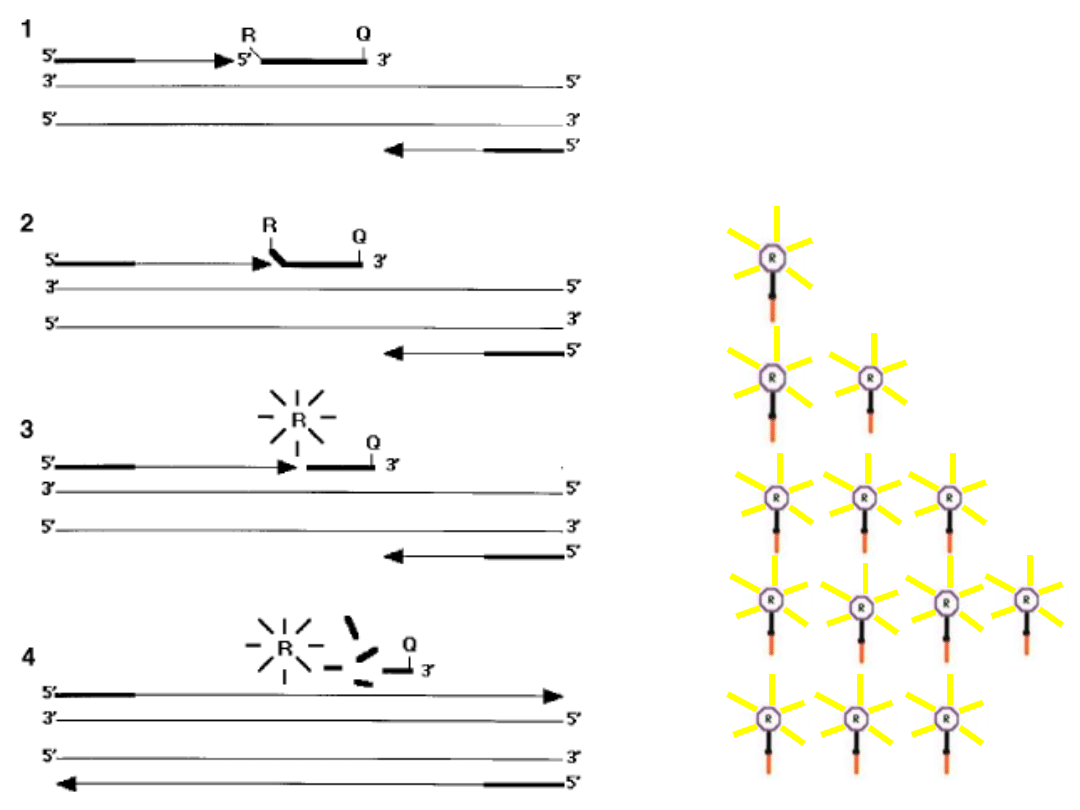
W miarę postępu
reakcji w mieszaninie
zwiększa się ilość
wolnego, świecącego
barwnika
fluorescencyjnego.
Cykl 1 –
Cykl 2 –
Cykl 10 –
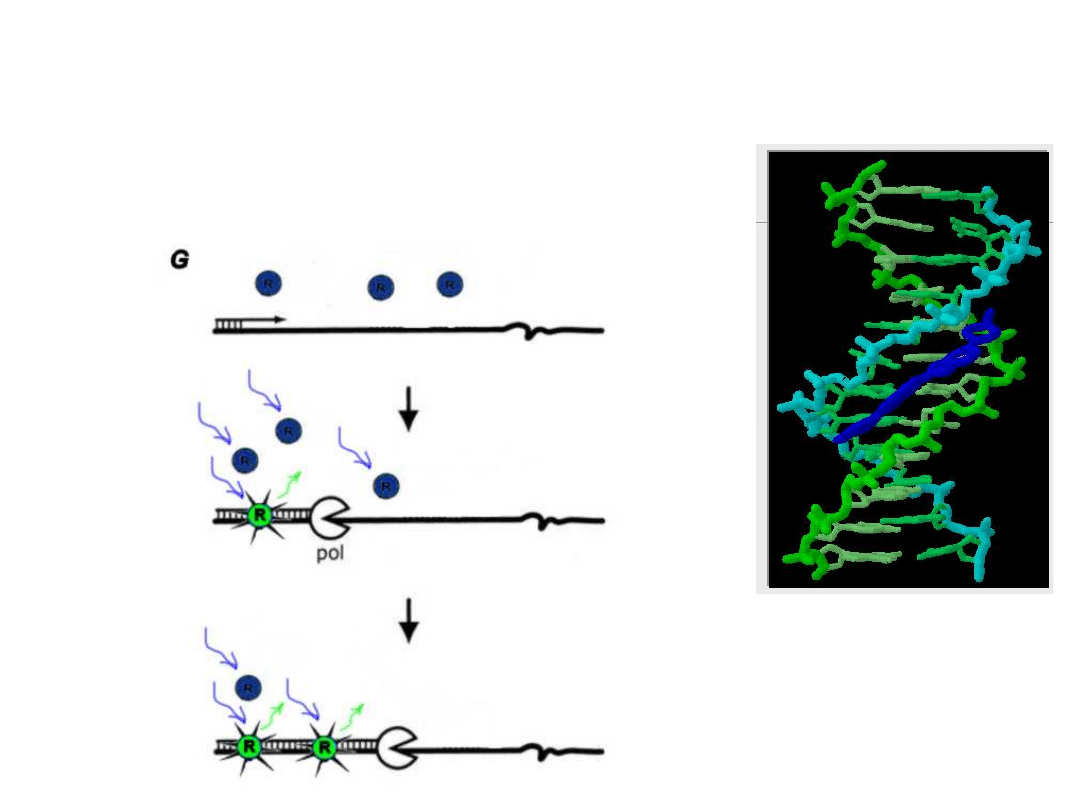
Detekcja przyrostu produktu
PCR
poprzez
przyłączanie
barwnika
Sybr®
Green
I.
Intensywność
świecenia
wzrasta po związaniu do ds
DNA i jest proporcjonalna do
ilości produktu PCR.
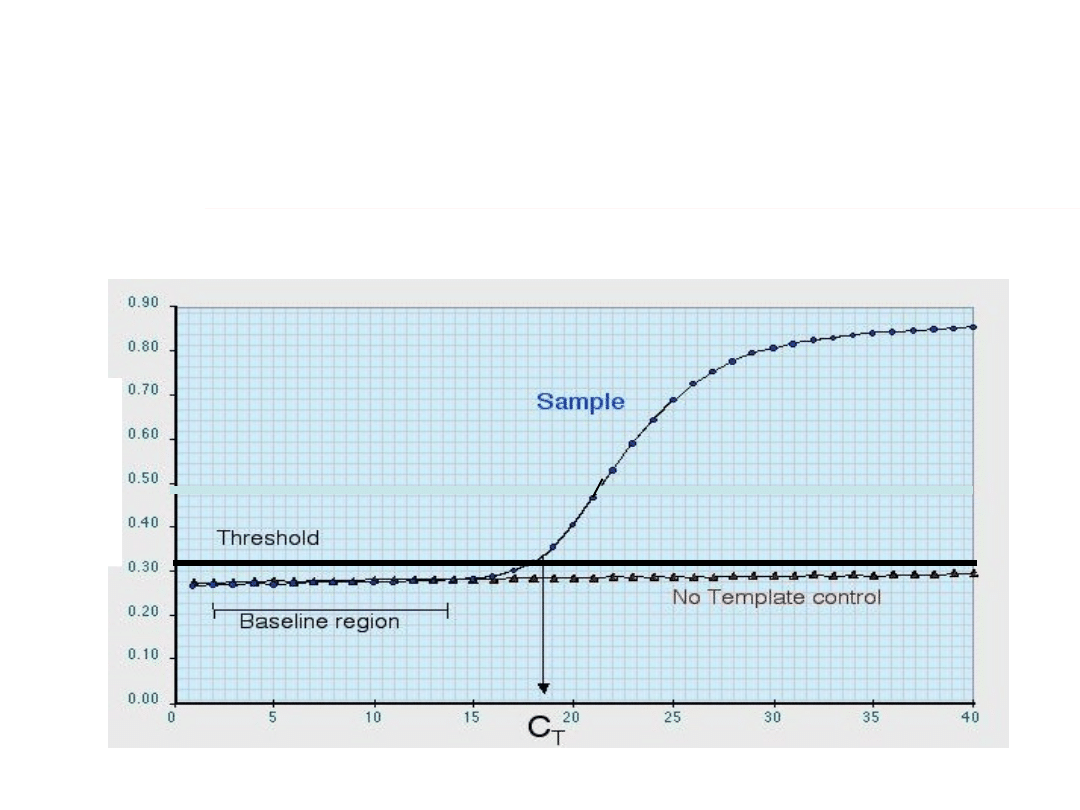
Podstawowe terminy analizy ilościowej PCR:
•
Obszar wartości podstawowej
-ilośċ cykli podczas których nie
obserwuje się zmiany sygnału fluorescencji
•
Ustalony próg (threshold)
wyznacza się zwykle jako
dziesięciokrotną wartość odchylenia standardowego mierzonego w
obszarze wartości podstawowej (zwykle w 5-15 cyklu)
•
Parametr C
T
(threshold cycle)
jest zdefiniowany jako numer cyklu
w którym wartości fluorescencji przewyższają ustalony próg
Numer kolejny cyklu
fl
u
o
re
s
c
e
n
c
ja
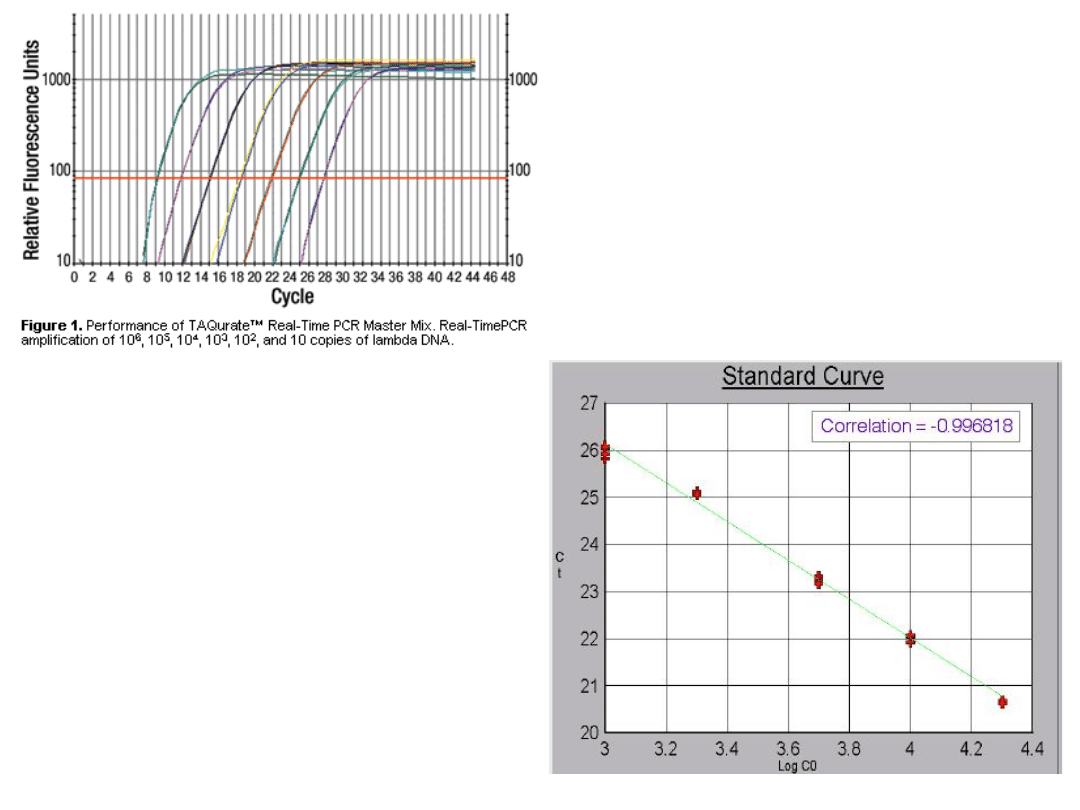
Krzywa zależności C
T
od
ilości próby (skala
logarytmiczna)
Wyznaczanie krzywej
standardowej przez
zastosowanie kolejnych
rozcieńczeń wzorcowego
DNA
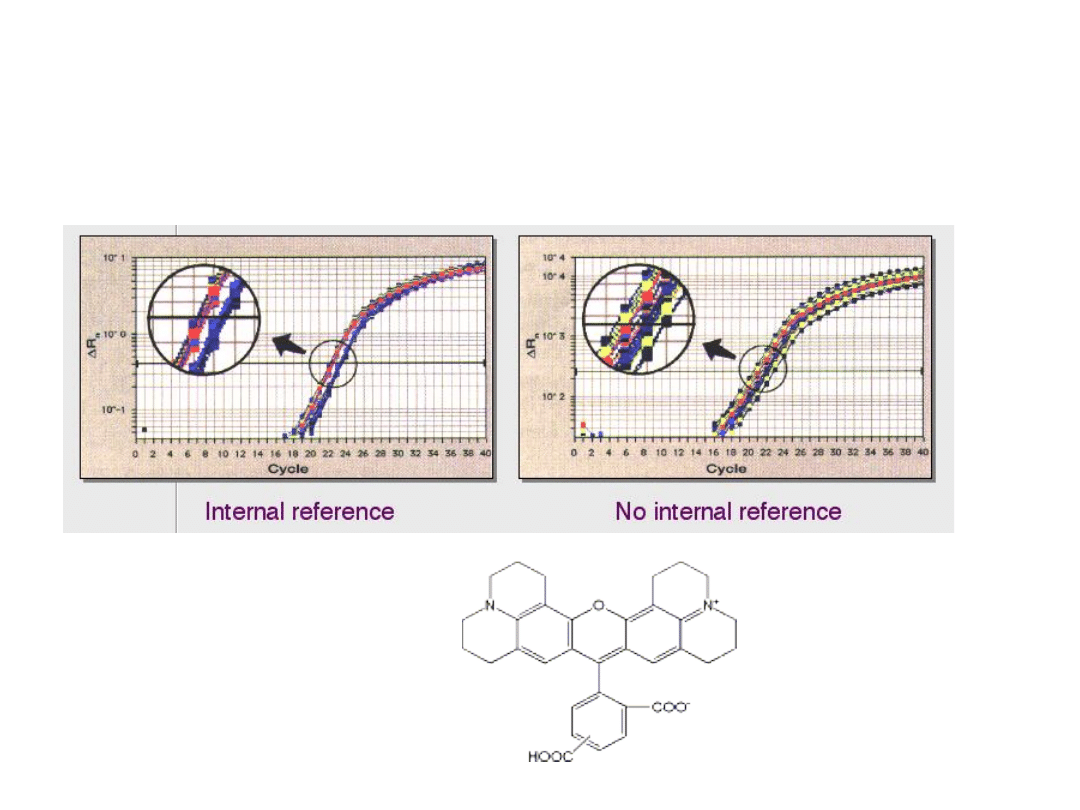
Obecność wewnętrznego standardu fluorescencji
(np. ROX), który pozwala wyeliminować fluktuacje
fluorescencji
niezwiązane
z
oddziaływaniem
sondy z DNA, zwiększa rozdzielczość metody
ROX

Ilościowy PCR można też
wykorzystać do badania
aneuploidalności, czy
duplikacji sekwencji DNA.

Przypominam o przygotowaniu na
kolokwia
• 27. III. BioAut 1
• 28. III. Ch 1, ISE 1
• 1. budowa komórek prokariotycznych
i eukariotycznych.
2. zadania z rozcieńczeń roztworów.
3. budowa DNA i zasad azotowych.
4. instrukcja do ćwiczeń – działanie
odczynników w izolacji DNA (wstęp
teoretyczny).

Odrabianie zajęć wymaga
powiadomienia stałego
opiekuna grupy, oraz (na
odrabianych ćwiczeniach) –
opiekuna grupy,
w której są odrabiane
ćwiczenia.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
Wyszukiwarka
Podobne podstrony:
W 5 Izolacja RNA, RT PCR, hybrydyzacja 1
BMiGO Wykład 4 PCR, Sekwencjonowanie
BMiGO Wykład 1 DNA izolacja i analiza
Metoda RT-PCR, 3. rok, genetyka kliniczna
Metoda RT PCR
Hybrydyzacja, sondy, PCR
BMiGO Wykład 2 Klonowanie
cw RT PCR
ODWROTNA TRANSKRYPCJA RT PCR
Metody izolacji RNA
Chemia wyklad I i II (konfiguracja wiÄ…zania Pauling hybrydyzacja wiazania pi i sigma)
Hybrydyzacja polega na tworzeniu podwojnej helisy miedzy komplementarnymi niciami DNA lub RNA
kw nukleinowe, izolacja, rozdział, PCR
Lista uzupełniona studentów zwonionych z zaliczenia wykładów RT
Budownictwo Ogolne II wyklad 14 izolacje b
Przenoszenie DNA i RNA na membrany hybrydyzacyjne
Izolacja całkowitego RNA - KONSPEKT, studia - biotechnologia, biologia molekularna
Biochemia Wykład VII 9 01 15 r Kwasy nukleinowe, DNA, RNA
więcej podobnych podstron