Porównywanie sekwencji białkowych
z wykorzystaniem metody
ewolucyjno-progresywnej
Paweł Kupis
Jacek Mańdziuk

Biologiczna geneza problemu
• białko (polipeptyd)
• polimer liniowy aminokwasowy
• monomery – aminokwasy
• 20 rodzajów aminokwasów
• pierwszorzędowa struktura protein
• sekwencja białkowa
• kolejność aminokwasów
• polaryzacja (kierunek czytania sekwencji)

Biologiczna geneza problemu
• przykład
• HBA_HUMAN (prefix ludzkiej hemoblobiny)
VLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPT
TKTYFPHFDLSHGSAQVKGHGKKVADALTNAVAHVDDM
PNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLP
AEFTPAVHASLDKFLASVSTVLTSKYR

Porównywanie sekwencji
• problem
• trudno wyznaczyć kryterium porównywania
• pomysł
• ilość identycznych pozycji w sekwencjach o
identycznej długości
• rozwiązanie
• uliniowienie sekwencji

Uliniowienie sekwencji
• uliniowienie sekwencji (ang. sequence alignment)
• warunki
• n-ty wiersza po usunięciu znaków ‘-‘ daje n-tą
sekwencję
• długość wszystkich wierszy uliniowienia jest jednakowa
• żadna kolumna uliniowienia nie zawiera tylko znaków
‘-‘
CA-GCUUAUCGCUUAG
AAUGCAU-UGACG--G

Uliniowienie wielu sekwencji
• MSA (ang. multiple sequence alignment)
• warunki
• n-ty wiersza po usunięciu znaków ‘-‘ daje n-tą sekwencję
• długość wszystkich wierszy uliniowienia jest jednakowa
• żadna kolumna uliniowienia nie zawiera tylko znaków ‘-‘
• takie same jak dla uliniwienia dwóch sekwencji
LGB2_LUPLU VPQ--NNPELQAHAGKVFKLVYEAAIQLQVTGVVVTDATLKNLGSVHVSK-GVADAHFPV
MYG_PHYCA EAEMKASEDLKKHGVTVLTALGAILKKKG--HHEAELKPLAQS---HATKHKIPIKYLEF
GLB5_PETMA ADQLKKSADVRWHAERIINAVNDAVASMD--DTEKMSMKLRDLSGKHAKSFQVDPQYFKV
HBB_HUMAN PDAVMGNPKVKAHGKKVLGAFSDGLAHLD--NLKGTFATLSEL---HCDKLHVDPENFRL
HBB_HORSE PGAVMGNPKVKAHGKKVLHSFGEGVHHLD--NLKGTFAALSEL---HCDKLHVDPENFRL
HBA_HUMAN -----GSAQVKGHGKKVADALTNAVAHVD--DMPNALSALSDL---HAHKLRVDPVNFKL
HBA_HORSE -----GSAQVKAHGKKVGDALTLAVGHLD--DLPGALSNLSDL---HAHKLRVDPVNFKL
. .:: *. : . * : * . : : .

Metoda ewolucyjno-
progresywna
• metoda 2-etapowa
• etap 1. - ewolucyjny
• dopasowywanie kolumn całkowicie identycznych
• znajdowanie optymalnego tzw. „wstępnego
uliniowienia”
• etap wykonywany rekurencyjnie
• etap 2. - progresywny
• uliniowienie obszarów między kolumnami
zidentyfikowanymi w etapie 1.
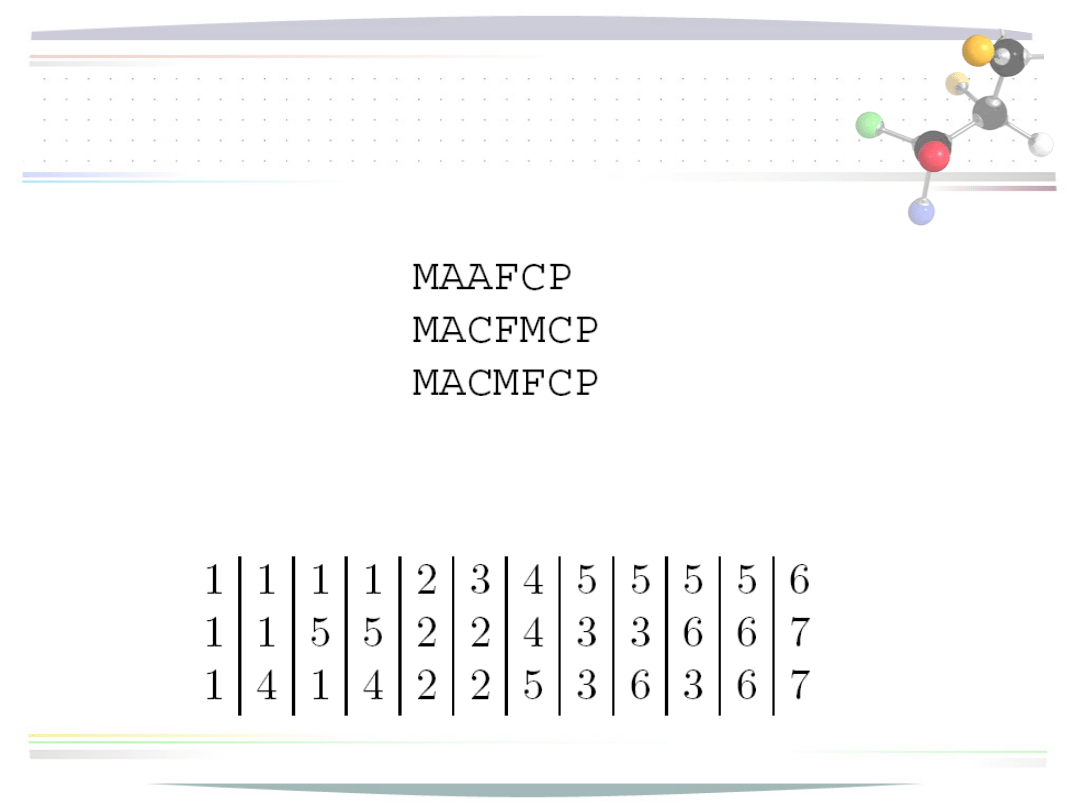
Etap ewolucyjny
dopasowywanie kolumn całkowicie identycznych, przykład:
wszystkie możliwe kolumny zgodne
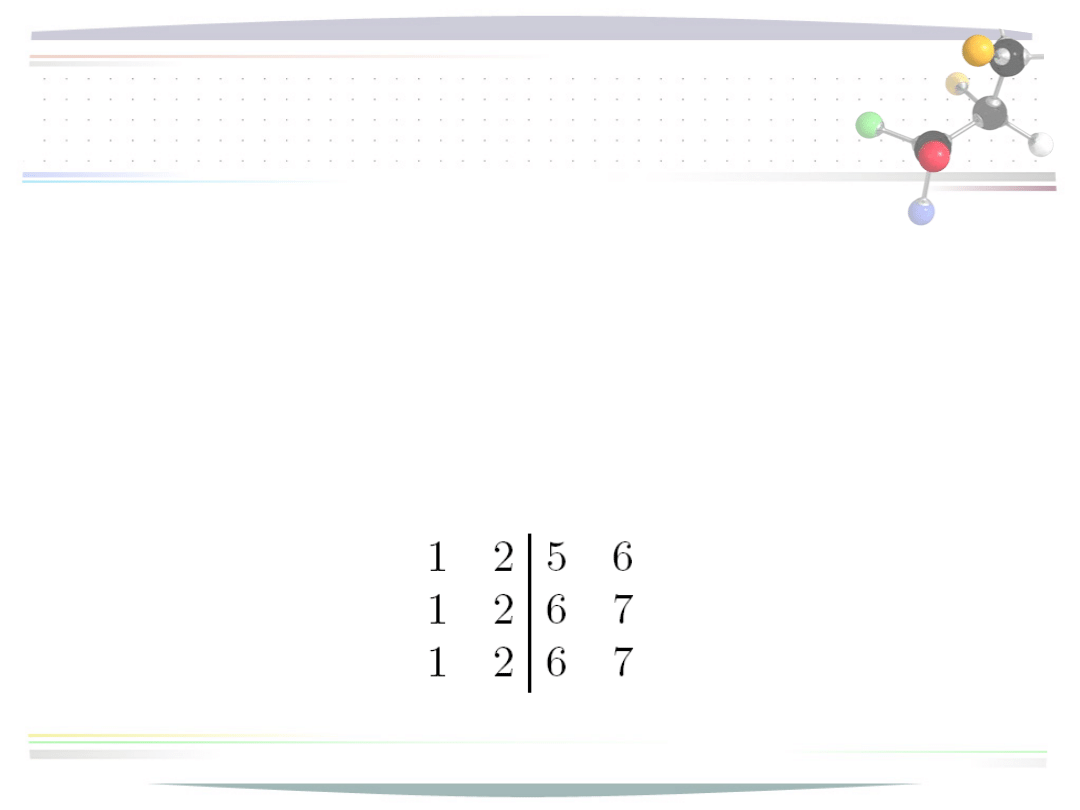
Etap ewolucyjny
blok kolumn identycznych
• kolumny tworzą blok jeśli we wszystkich wierszach
różnica w indeksach wynosi jeden (większy indeks
– mniejszy indeks)
• blok może mieć dowolną długość
• w szczególności pojedynczą kolumną również
można traktować jako blok
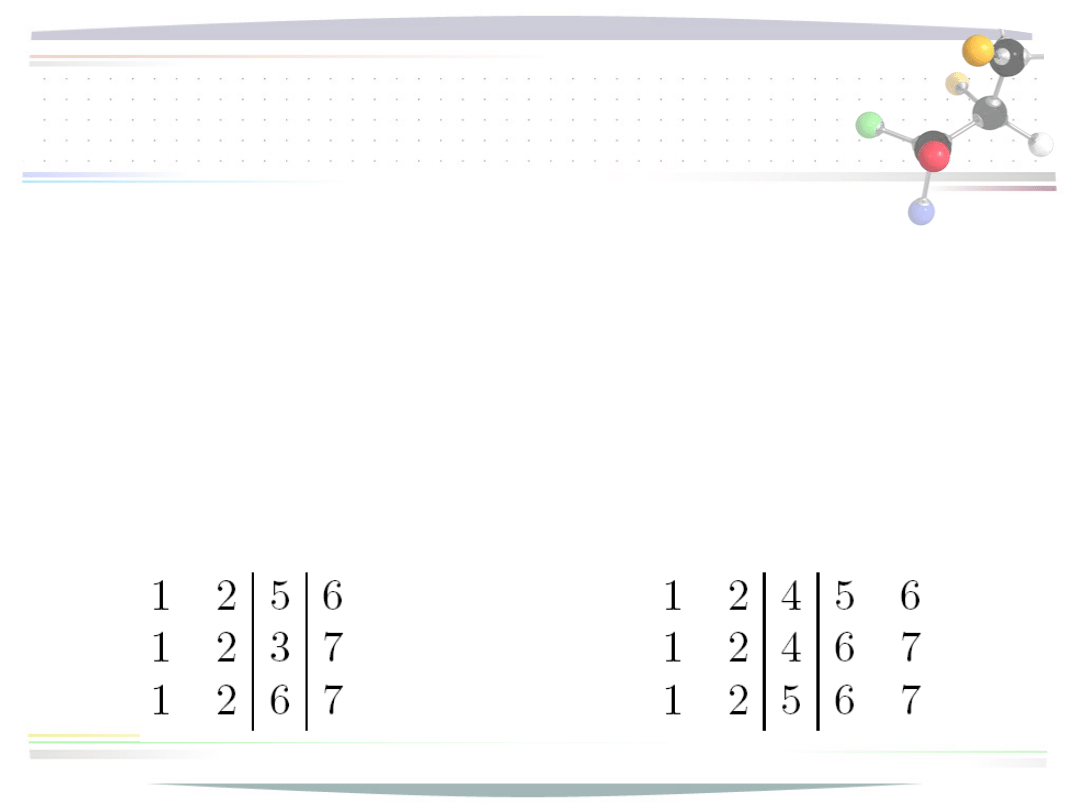
Etap ewolucyjny
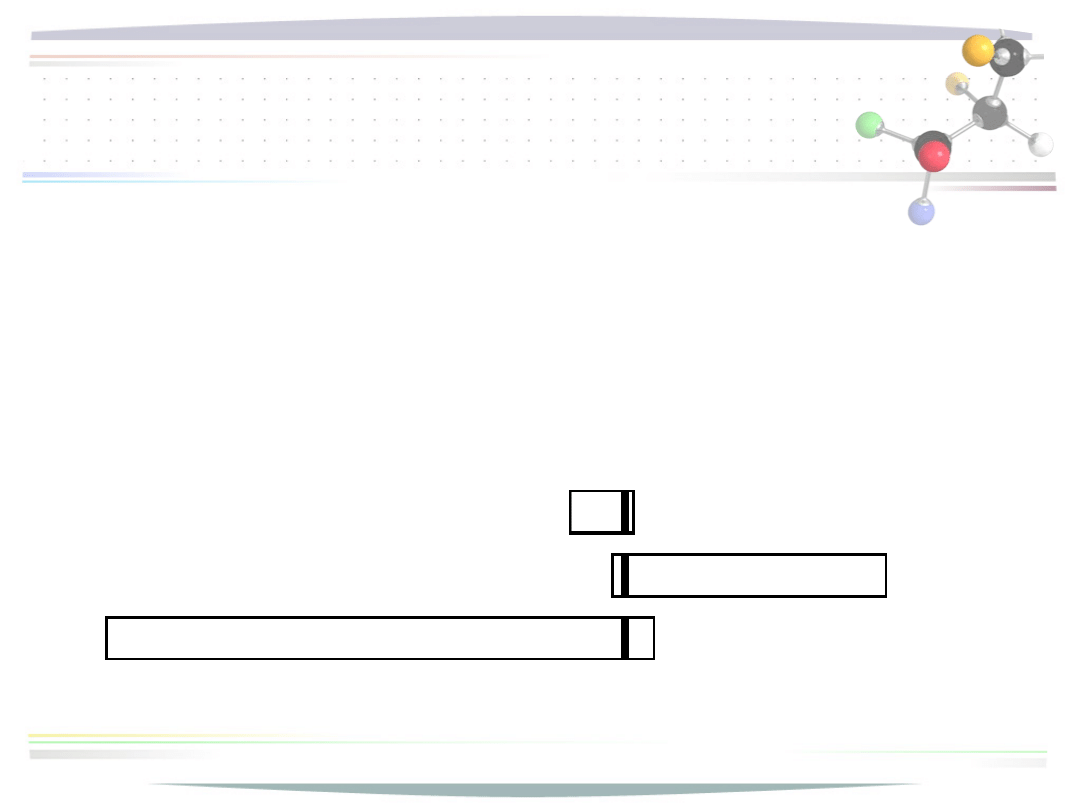
wstępne uliniowienie
• szereg bloków spełniający następujące warunki
• dowolny indeks może wystąpić w wierszu tylko
raz
• w każdym wierszu indeksy są w porządku
rosnącym
• powyższe warunki gwarantują, że na podstawie
wstępnego uliniowienia można zbudować pełne
uliniowienie
(zachowując ustalone kolumny identyczne)
Etap ewolucyjny
kolumny szkodliwe
• intuicyjnie możemy określić taką kolumnę jako
łączącą „zbyt” odległe części różnych sekwencji
• kolumna taka, uniemożliwia bardzo często
lepsze dopasowanie innych kolumn identycznych
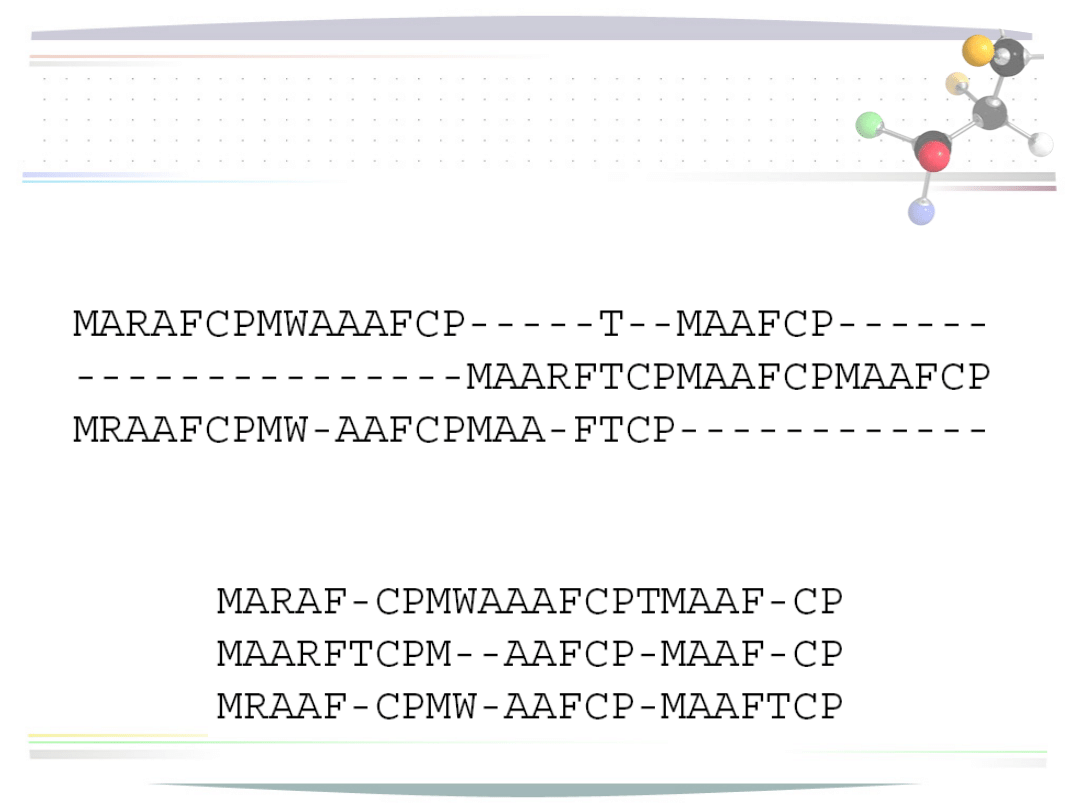
Etap ewolucyjny
• bliskie optymalnemu uliniowienie z wymuszeniem
uzgodnienia kolumny symboli T
• uliniowienie tych samych sekwencji bez
uzgadnianie symboli T

Etap ewolucyjny
• zadania algorytmu ewolucyjnego
• znalezienie optymalnego wstępnego uliniowienia
• budowa populacji startowej
• czas budowy musi być „kontrolowalny”
• wprowadzenie to populacji startowej
reprezentatywnego
podzbioru możliwych kolumn identycznych
• użycie wszystkich (z wszystkich części sekwencji)
symboli z sekwencji
• unikanie szkodliwych kolumn
• ew. późniejsza ich eliminacja
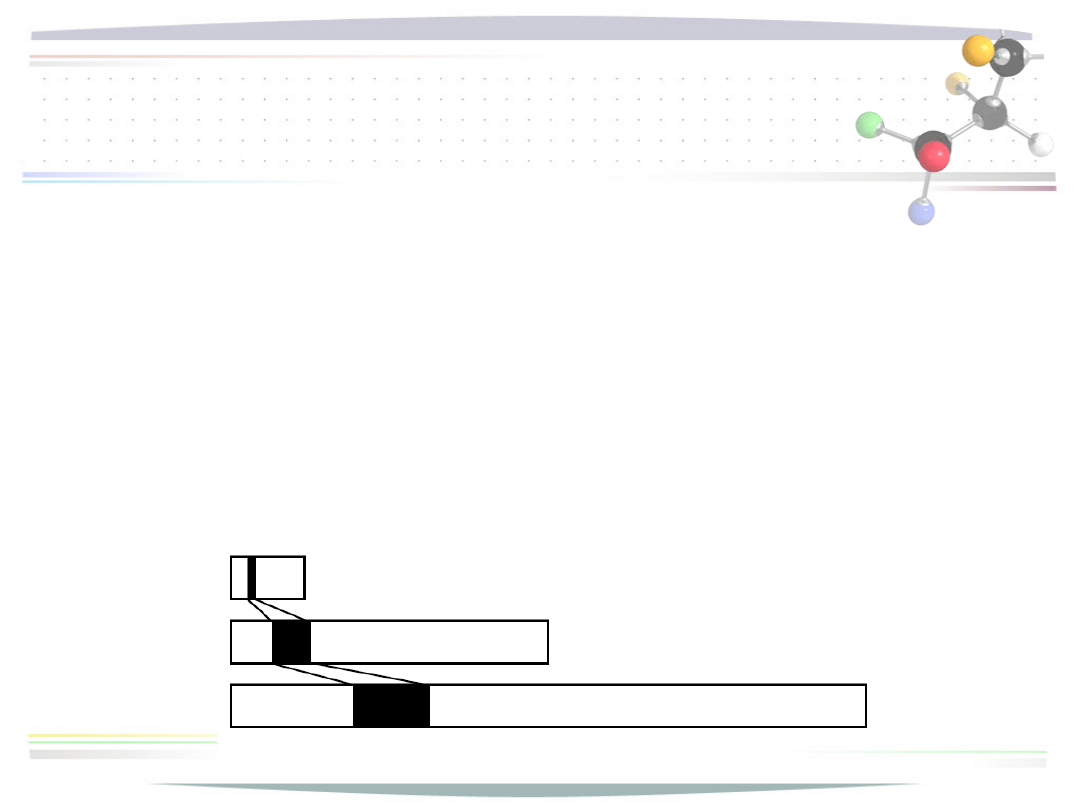
Budowa populacji startowej
• metodę charakteryzują dwa podstawowe parametry
• c
max
– górny limit (w przybliżeniu) ilości
zidentyfikowanych kolumn identycznych
• w
%
– szerokość tzw. „okna przeszukiwania”
• symbole tworzące kolumnę identyczną nie mogą
pochodzić z dowolnych części sekwencji
• każdy symbol pochodzi z aktywnego okna
przeszukiwania danej sekwencji

Budowa populacji startowej
• względna długość okna przeszukiwania (w stosunku do dł.
sekwencji) jest taka sama dla wszystkich sekwencji
• analogicznie względna pozycja środka okna (względem
początku sekwencji)
• z każdego okna, losowo, wybierany jest jeden symbol
• jeśli wszystkie symbole są identyczne, tworzona jest
kolumna identyczna
• nie jest sprawdzana unikalność kolumny
• czynność jest wykonywana razy dla każdego
symbolu
(okna szerokości jednego symbolu) wyróżnionej sekwencji
• gdzie m – dł. wyróżnionej sekwencji (np. najkrótszej)
m
c
max

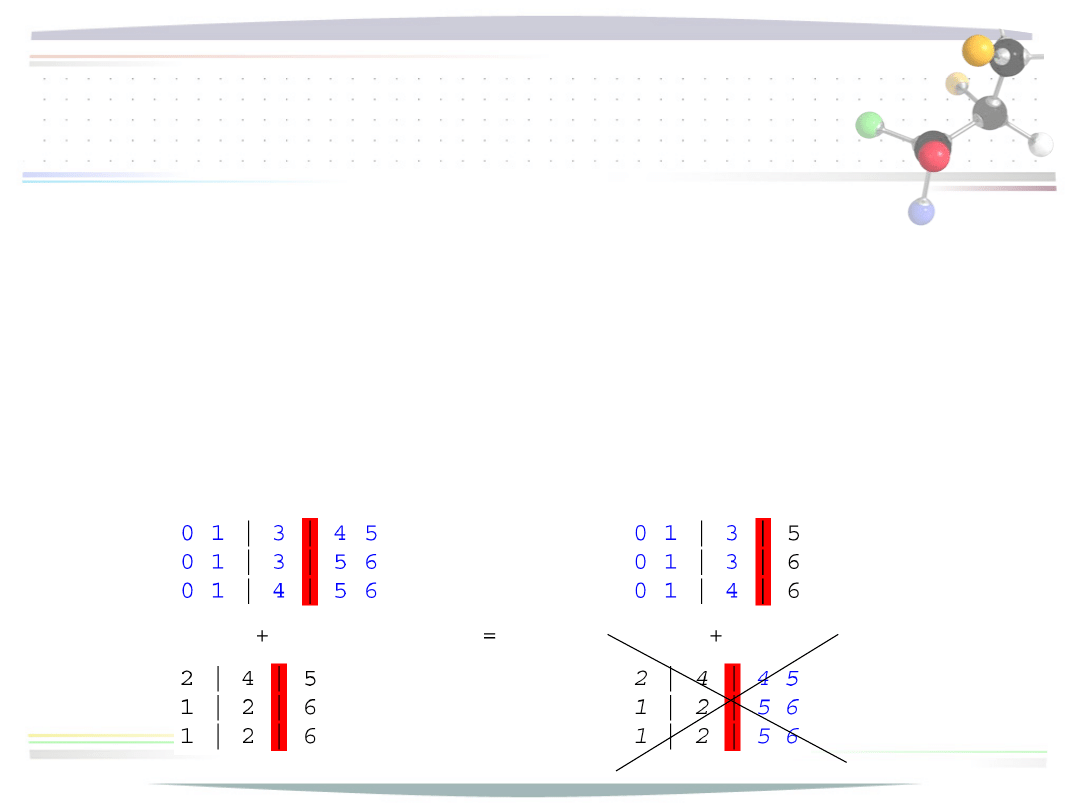
Budowa populacji startowej
• zbieranie informacji (tworzenie wstępnych uliniowień)
A – zbiór kolumn
identycznych
(porządek
odnajdywania)
P – populacja startowa,
początkowo pusta
c
p
– nominalny rozmiar
populacji startowej
Algorytm ewolucyjny
• populacja startowa
(c
max
=4000, w
%
=0.04)
• c
p
= (m
a
* n) / 10, m
a
– śr. dł. sekwencji, n –
ilość sekwencji
• c
p
>= 100 oraz c
p
<= 400
• tylko jeden operator genetyczny -
krzyżowanie

Algorytm ewolucyjny
• krzyżowanie
• jednopunktowe
• losowe punkty cięcia (możliwe przed pierwszym i
za ostatnim blokiem)
• punkt cięcia nigdy nie rozdziela bloku
• po wymianie informacji sprawdzana jest możliwość
złączenia bloków sąsiadujących z punktem cięcia
• „lepszy” z potomków musi być lepszy od obojga
rodziców
• domyślne prawdopodobieństwo krzyżowania = 0.4
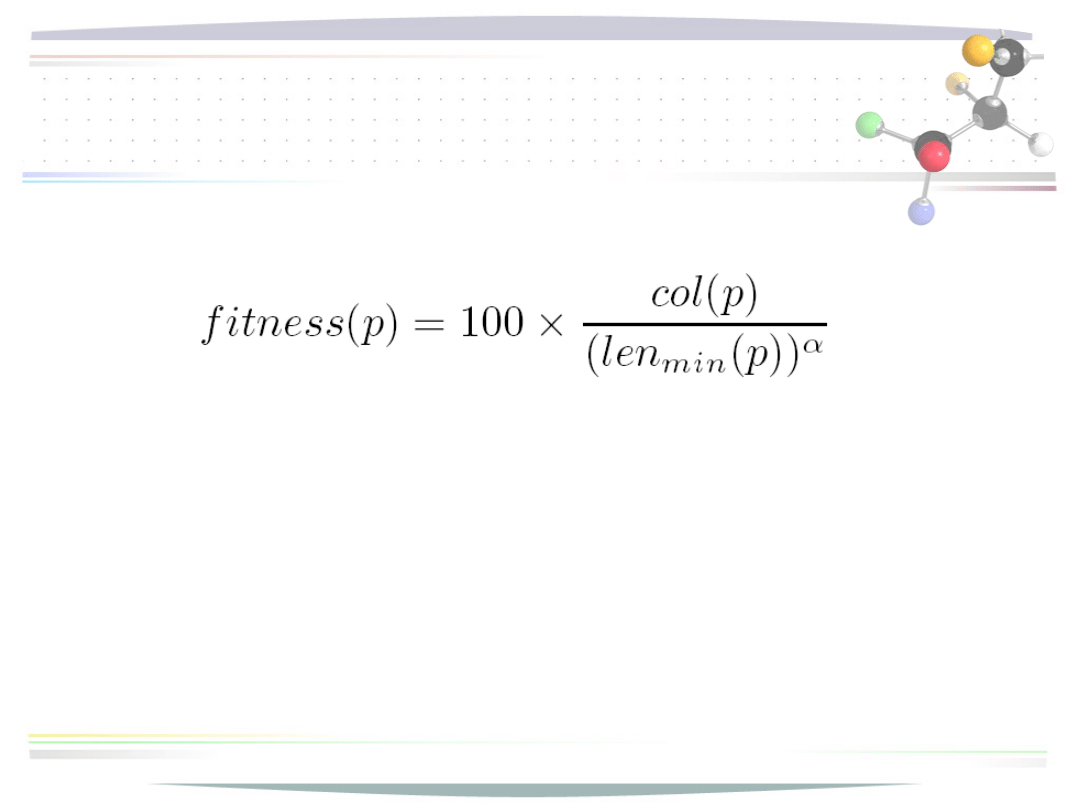
Algorytm ewolucyjny
• funkcja przystosowania
col(p) – ilość kolumn identycznych w osobniku p
len
min
(p) – minimalna długość uliniowienia powstałego
na
podstawie uliniowienia wstępnego
reprezentowanego
przez osobnika p
α – wykładnik określający istotność karania na
powstawanie
nadmiernie długich uliniowień
(=20)

Algorytm ewolucyjny
• jeśli i-ty blok wstępnego uliniowienia p oznaczymy
jako b
i
to funkcja len
min
(p) wyraża się wzorem

Algorytm ewolucyjny
• warunki stopu
• przystosowanie najlepszego osobnika nie zmieniło
się od 40 generacji
• osiągnięto limit 1000 generacji
• wywołania rekurencyjne dla obszarów między blokami
(w najlepszym z osobników)
• koniec rekurencji
• alg. ewolucyjny nie znalazł żadnej kolumny
identycznej
• minimalna odległość między danymi blokami jest
<= 20
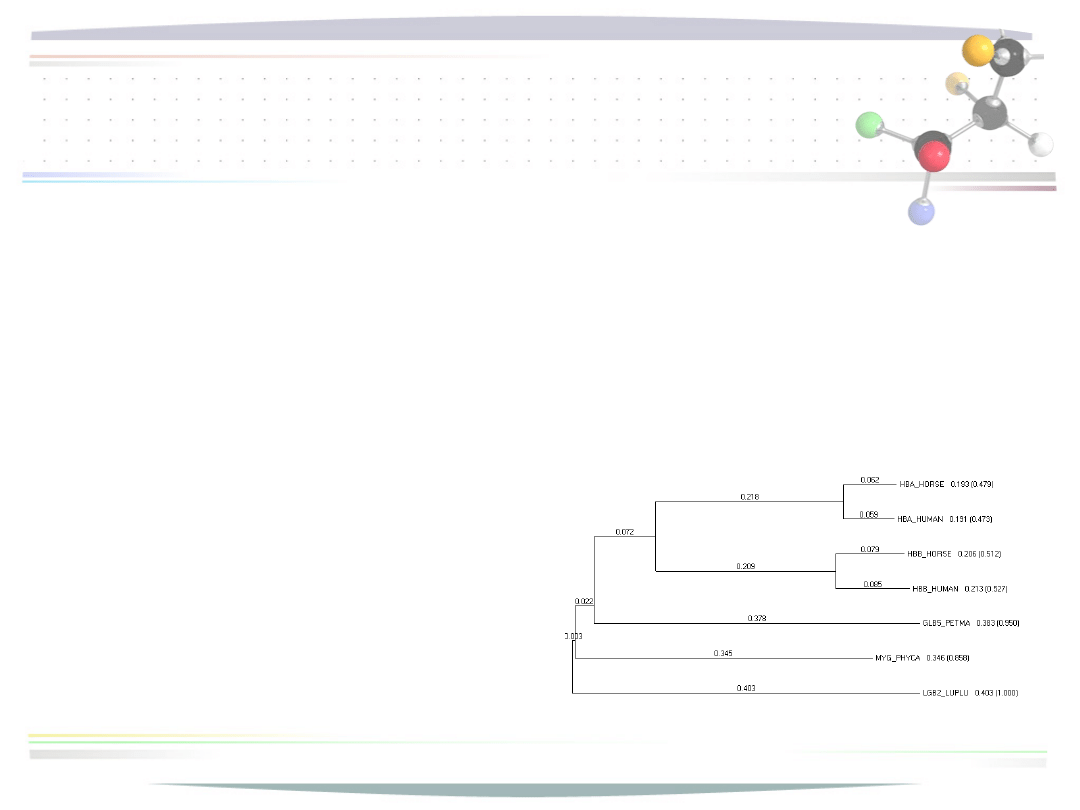
Algorytm progresywny
• uruchamiany dla obszarów między blokami
zidentyfikowanymi przez alg. ewolucyjny
• implementacja zbliżona do ClustalW
• drzewo filogenetyczne
budowane metodą
neighbor-joining
(z ukorzenianiem
metoda mid-point rooting)
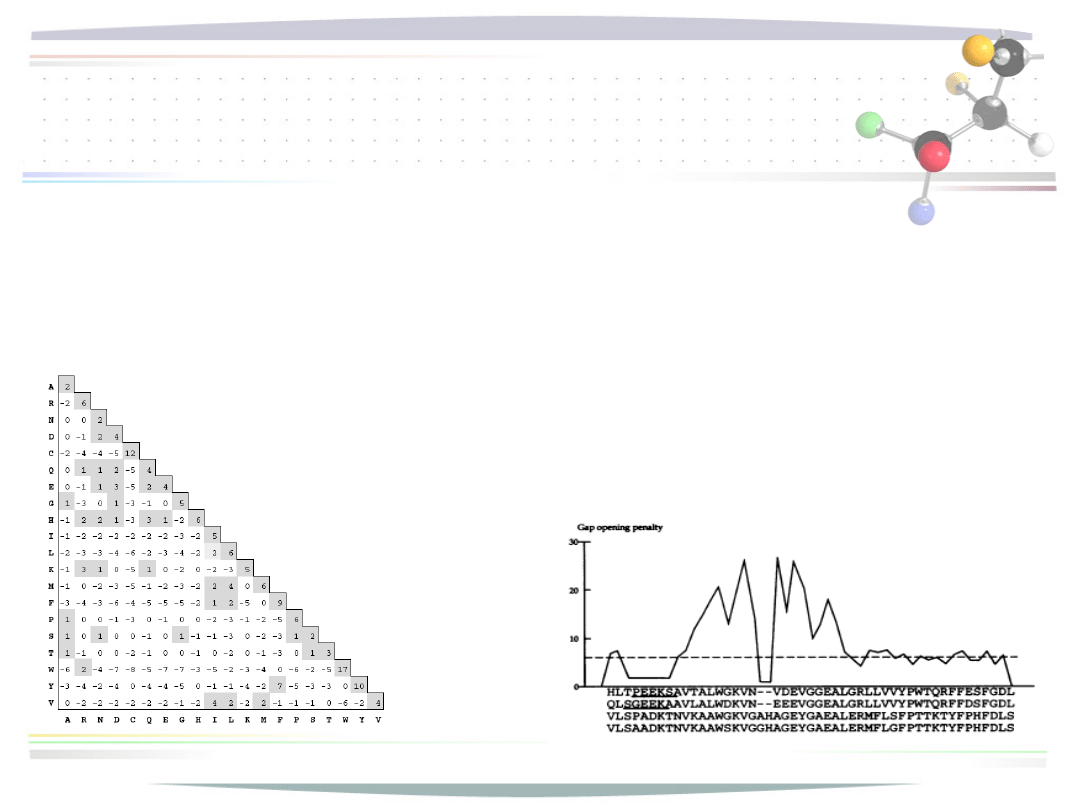
Algorytm progresywny
• uliniawianie par metodą Myersa-Millera
• przystosowanie do uliniawiania uliniowień
• przystosowanie do afinicznej kary za wprowadzane
przerwy
• kara k(w) = GOP + w*GEP, w – dł. Wprowadzonej
przerwy
• kary za wprowadzanie przerw zależne od pozycji w
sekwencji (funkcyjny opis parametrów kary afinicznej)
• stosowanie macierzy substytucji (automatyczny dobór w
zależności do odległości sekwencji w drzewie
filogenetycznym)

Testy
• Na podstawie referencyjnych baz BAliBASE
bazy udostępniają zarówno testowe zestawy
sekwencji, jak i gotowe uliniowienia tych
zestawów

Ocena uliniowienia
• miara SPS (Sum-of-Pair Score)
,
N - ilość sekwencji,
n - długość uliniowienia danej pary sekwencji,
m - ilość przerw w uliniowieniu pary sekwencji
• miara CS (Column Score)
• ilość kolumn identycznych w stosunku do dł. uliniowienia
• wszystkie wyniki podawane są jako średni stosunek miar w
odniesieniu do rezultatów dla uliniowień z bazy referencyjnej
N
j
i
j
i
S
S
sim
1
#
#
)
,
(
m
r
r
n
p
B
A
B
A
w
k
p
S
p
S
sub
S
S
sim
1
1
#
#
)
(
])
[
],
[
(
)
,
(
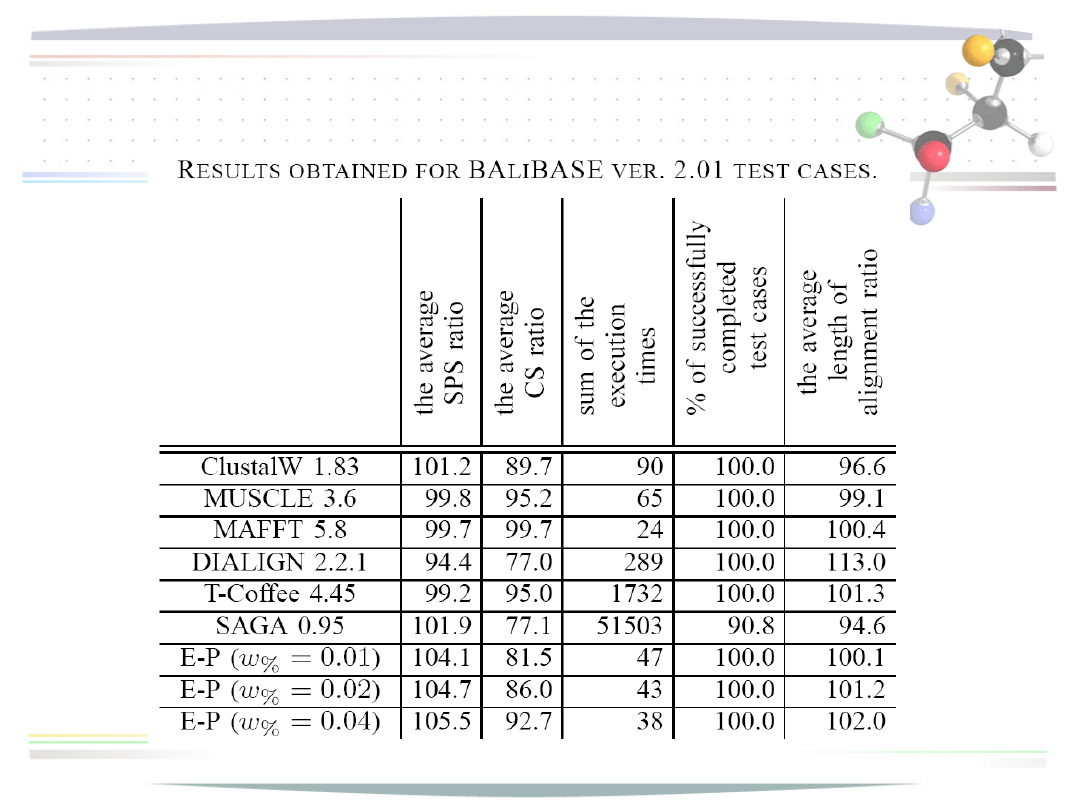
Wyniki
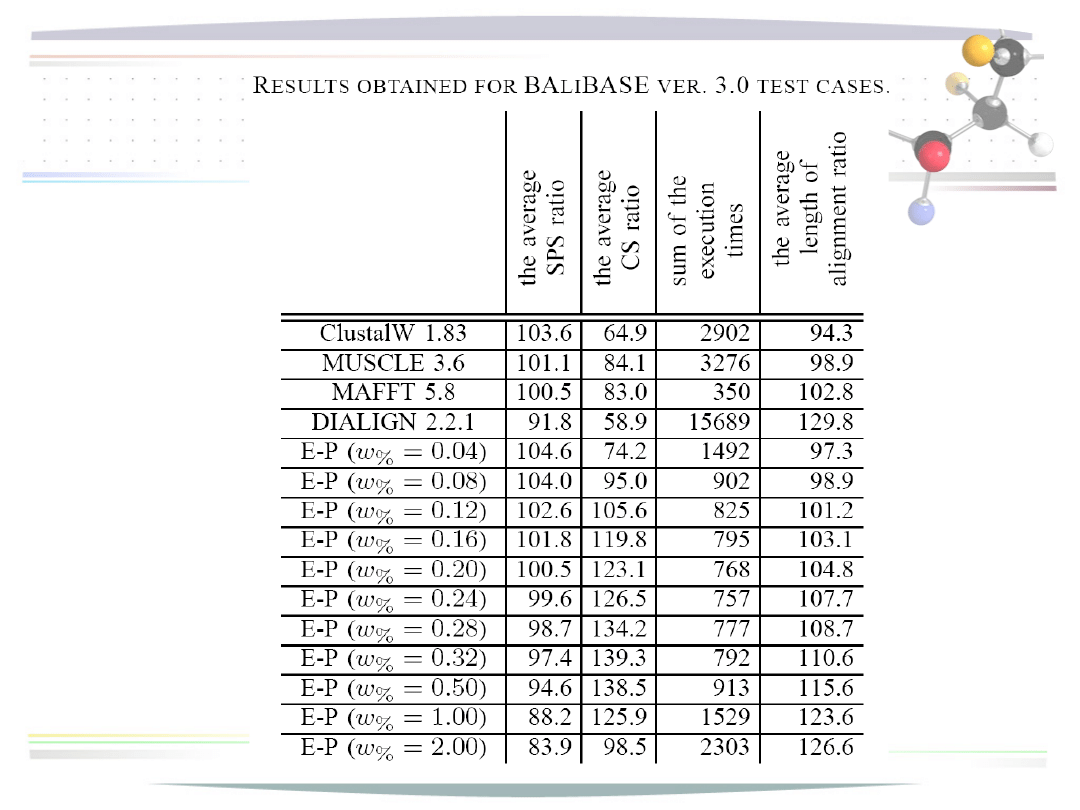
Wyniki

Koniec
• Pytania?
• Sugestie
Dziękuje za
uwagę
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
Wyszukiwarka
Podobne podstrony:
8b analiza sekwencyjna białek
8b analiza sekwencyjna białek
Sekwencje Sygnałowe białek
Przykłady roli biologicznej białek
W5 sII PCR i sekwencjonowanie cz 2
Właściwości fizykochemiczne białek
1 Przyswajanie białek przez organizmid 8658 ppt
właściwości białek mięśniowych
Metoda symultaniczno sekwencyjna
Wykład VIII Synteza układów sekwencyjnych
Budowa Chemiczna Białek
Funkcje białek, Biochemia
Zadania zajęcia 3 PSI - diagramy sekwencji i kooperacji, szkoła, PSI
19.Budowa białek, Notatki AWF, Biochemia
Amoniak można oznaczyć z krwi i moczu produkt rozkładu białek
Projektowanie nowych białek o zadanych właściwościach katalitycznych
sekwencyjne dlugopis
więcej podobnych podstron