
BADANIA
MOLEKULARNE
W
HEMATOONKOLOGI
I
KATEDRA I KLINIKA HEMATOONKOLOGII
I TRANSPLANTACJI SZPIKU
UNIWERSYTET MEDYCZNY W LUBLINIE

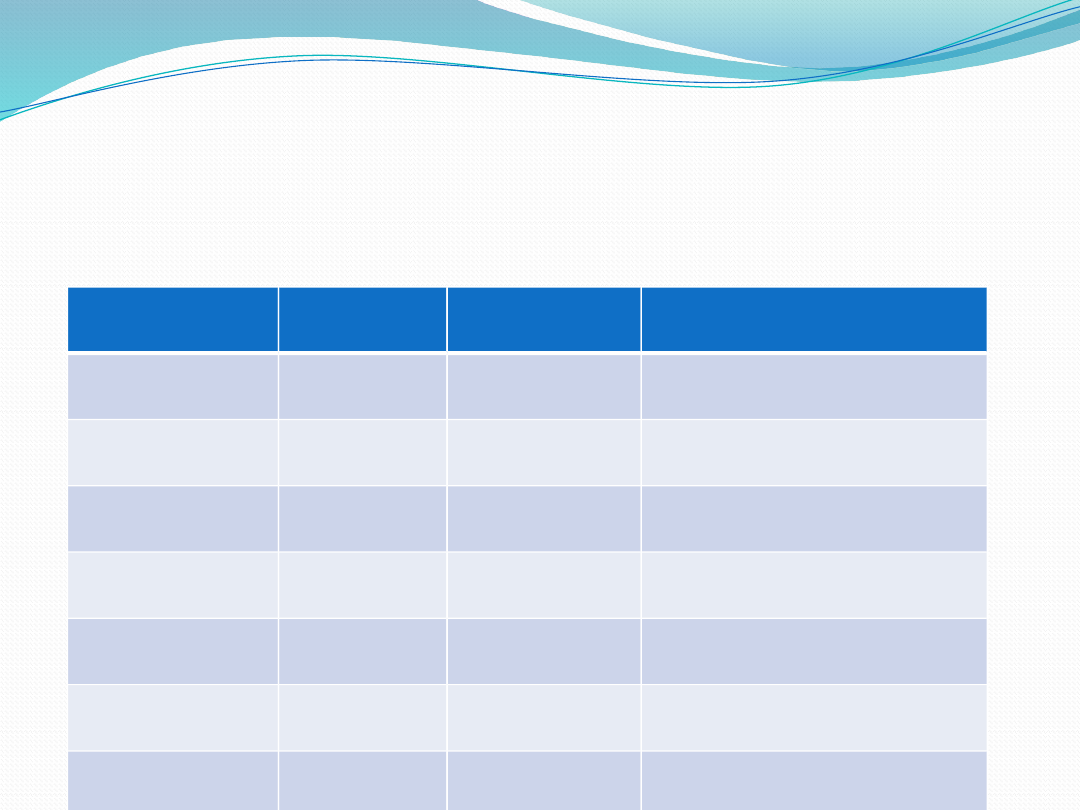
Tabela 1
Metody genetyczne w diagnostyce i monitorowaniu chorób rozrostowych układu
krwiotwórczego
Metoda
Materiał
diagnostyc
zny
Stadium
analizowan
ych
komórek
Liczba
analizowan
ych
komórek
Czuło
ść
Specyficz
ność
Możliwoś
ć oceny
ilościowe
j
Ukierunkowani
e – zakres
informacji
genetycznej
Cytogenet
yka
klasyczna
(GTG;
GTW)
szpik
metafaza
do 100
+
++
+ w
wąskim
zakresie
szeroki – metoda
nieukierunkowan
a
FISH
szpik
metafaza
interfaza
do 1000
++
+++
+
wąski – metoda
ukierunkowana
RT-PCR
szpik
bez
znaczenia
> 1 milion
+++
+++
–
wąski – metoda
ukierunkowana
RQ-PCR
krew
bez
znaczenia
> 1 milion
+++
+++
+
wąski – metoda
ukierunkowana

Cytogenetyka klasyczna przedstawia całościowy obraz zmian
chromosomowych w komórce. Umożliwia analizę od kilkunastu do stu komórek
metafazowych, przy czym miarodajna jest ocena co najmniej 20-25 metafaz.
Minusem metody jest konieczność prowadzenia hodowli komórkowej, która nie
zawsze jest skuteczna. Cytogenetyka klasyczna stanowi podstawową,
referencyjną metodę badań genetycznych. Znaczenie diagnostyczne i rokownicze
mają tylko aberracje klonalne, tj. monosomie chromosomów obecne w co
najmniej 3 komórkach oraz trisomie lub jednolite zmiany strukturalne
występujące w co najmniej 2 komórkach.
Fluorescencyjna hybrydyzacja in situ (FISH) jest metodą cytogenetyki
molekularnej, która uzupełnia cytogenetykę klasyczną. W przypadku zmian o
charakterze mikroaberracji, wielkości 1-10 Mpz, FISH funkcjonuje jako
samodzielna metoda referencyjna. Analiza FISH dotyczy zarówno komórek w
stadium metafazy, jak i interfazy i jest badaniem ukierunkowanym. W
hematoonkologii FISH służy analizie fuzji i innych rearanżacji genowych oraz
wyjaśnianiu charakteru złożonych aberracji chromosomowych.
RT-PCR (reverse-transcriptase PCR) to czuła, ukierunkowana metoda oceny
ekspresji genów na podstawie analizy ich transkryptów (mRNA). W
hematoonkologii służy głównie do oceny fuzji i innych rearanżacji lub mutacji
genowych. RT-PCR pozwala na uzyskanie dodatnich wyników już przy obecności
jednej komórki nieprawidłowej na milion prawidłowych, stąd jej rola w ocenie
choroby resztkowej (MRD; minimal residual disease).
RQ-PCR (reverse-transcriptase quantitative PCR) to ilościowa metoda RT-PCR,
która pozwala na ocenę zmian w ilości wybranego transkryptu genowego w
przebiegu choroby, co odpowiada zmianom liczby komórek nowotworowych. RQ-
PCR to czuła i miarodajna metoda oceny skuteczności leczenia.

Standardized RT-PCR analysis of fusion gene transcripts from
chromosome aberrations in acute leukemia for detection of minimal
residual disease.
Report of the BIOMED-1 concerted action: Investigation of minimal
residual disease in acute leukemia.
van Dongen JJM et el. Leukemia (1999) 13, 1901-28
Standardowy protokół RT-PCR
1. Reakcja odwrotnej transkrypcji (RT)
•
1 µg RNA (lub 0,1 µg mRNA) w 9,5 µg H
2
O
•
inkubacja w 70°C przez 10 min
•
inkubacja w lodzie i dodanie pozostałych
składników
do objętości całkowitej 20 µl:
bufor RT: 20 mM Tris-HCl, 50 mM KCl,
pH 8,3
MgCl
2
: 5 mM
DTT: 10 mM
heksamery: 5 µM
RNAsin: 20 U
enzym RT: 200 U/µl
dNTP: 1 mM
•
temperatura i czas inkubacji:
temperatura pokojowa przez 10 min
42°C przez 45 min
99°C przez 3 min
4°C do zakończenia etapu RT
2. PCR lub pierwsze runda nested PCR
•
objętość całkowita 50 µl
•
2-3 µl cDNA (i.e. 10-15% mieszaniny RT)
•
startery: stężenie końcowe 400 nM
•
dNTP: stężenie końcowe 200 µM
•
bufor PCR: 20 mM Tris-HCl, 50 mM KCl, pH
8,3
•
MgCl
2
: 2,5 mM
•
enzym Taq: 1 U/50 µl objętości
3. Temperatura i czas trwania etapów
PCR
•
denaturacja wstępna: 95°C przez 30 s
•
etapy PCR:
94°C przez 30 s (denaturacja)
65°C przez 60 s (przyłączanie
starterów)
72°C przez 60 s (synteza DNA)
•
liczba cykli: 35
4. Druga runda nested PCR
•
1 µl mieszaniny z pierwszej rundy PCR
•
taka sama objętość, odczynniki i warunki
reakcji jak w pierwszej rundzie PCR, z
użyciem starterów wewnętrznych
Przewlekłe choroby mieloproliferacyjne –
MPD
HSC
Mieloidalna
komórka
prekursorowa
Komórka
tuczna
Erytrocyt
y
Trombocyt
y
Eozynofil
e
Neutrofile
Monocyty
Mastocytoza
uogólniona (SM)
Czerwienica
prawdziwa (PV)
Nadpłytkowość
samoistna (ET)
Przewlekła
białaczka
eozynofilowa (CEL)
Przewlekła
białaczka szpikowa
(CML)
Przewlekła białaczka
mielomonocytowa
(CMML)
Samoistne zwłóknienie
szpiku (PMF)
MPD
Mutacja
KITD816V
FIP1L1-
PDGFRA
JAK2V617F
JAK2 Exon 12
JAK2V617F
MPLW515L/K
FIP1L1-
PDGFRA
BCR-ABL
JAK2V617F
MPLW515L/K
TEL-PDGFRB
BCR-PDGFRA
TEL-JAK2
Klasyfikacja i patogeneza MPD. W identyfikacji alleli, które przyczyniają się do
rozwoju danej choroby, wykorzystano różnorodne metody i stwierdzono, że
we wszystkich przypadkach allele te powodują zmianę aktywności kinazy
tyrozynowej (aktywacja konstytutywna).

Kinaza tyrozynowa
Pierwszym zidentyfikowanym allelem, charakterystycznym dla
MPD, jest
onkogen fuzyjny BCR-ABL
, który powstaje w wyniku
wzajemnej translokacji pomiędzy chromosomami 9 i 22, a jego
pojawienie się prowadzi do rozwoju
CML
.
Dalsze badania pozwoliły na identyfikację kolejnych genów fuzyjnych, w
tym
PDGFRB
(ang. platelet derived growth factor receptor-β),
charakterystycznego m.in. dla
CMML
. Dzięki obserwacji, że w
komórkach tucznych ma miejsce zwiększona ekspresja KIT,
zidentyfikowano alell
KITD816V
w
SM
. Z kolei na podstawie odpowiedzi
chorych na
CEL
i
SM
na leczenie imatinibem zidentyfikowano delecję,
która prowadzi do powstania genu fuzyjnego
FIP1L1-PDGFRA
.
Najnowsze badania pozwoliły stwierdzić, że u podstaw rozwoju PV,
ET
i PMF leży mutacja w obrębie genu kodującego Janus kinazę
2 (
JAK2
).
Wysoka aktywność kinazy tyrozynowej
powoduje utratę kontroli
nad podziałem komórki i prowadzi do rozwoju
nowotworu/choroby.
Przewlekła białaczka szpikowa – CML
W zależności od punktu złamania w obrębie genu
BCR
, gen
fuzyjny
BCR
/
ABL
może mieć różną długość
chromosom 9
BC
R
BCR
/
ABL
ABL
Przewlekła białaczka szpikowa – CML
Krótsze białko fuzyjne BCR/ABL jest związane z bardziej agresywnym
przebiegiem białaczki. Więcej białka BCR w białku fuzyjnym
BCR/ABL oznacza mniej agresywny przebieg choroby.
BCR
/
AB
L
p19
0
p21
0
p23
0
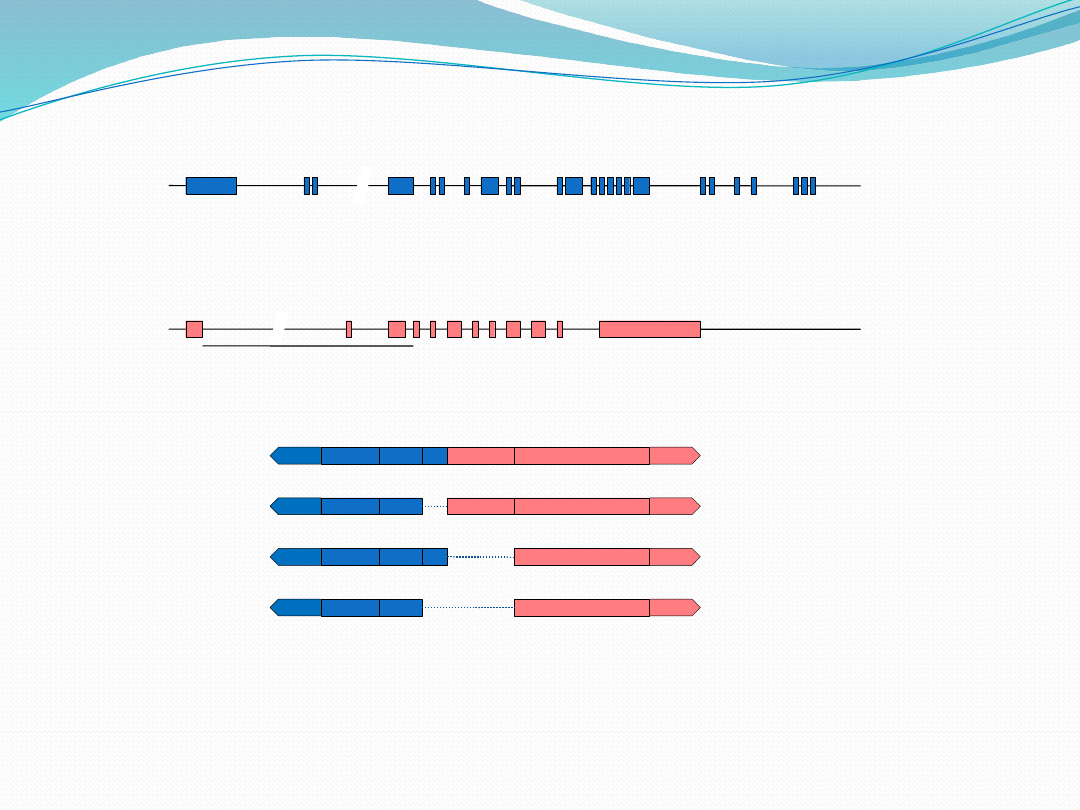
BCR (22q11)
ABL (9q34)
1 aternative 2 3 4 5 6 7 8 9 10 11-15 16 17 18 19 20 21-23
exons b2 b3 c3 c4
← cen M- bcr µ-bcr
~2.9 kb
→ tel
1b 1a 2 3 4 5 6 7 8 9 10
11
a2 a3
← cen breakpoint
region
~200 kb
→ tel
a
b
type
b3-a2
b2-a2
b3-a3
b2-a3
11
12
13
14
11
11
11
12
12
12
13
13
13
14
2
2
frequency
~55%
~40%
rare
rare
3
3
3
3
4
4
4
4
(a) Schematic diagram of the exon/intron structure of the BCR and ABL genes, involved in t(9;22)
(q34;q11) with focus on the major
breakpoint cluster region (M-bcr). The centromere (cen) and telomere (tel) orientation, exon numbering,
and relevant breakpoint
regions are indicated, including the micro breakpoint cluster region (µ-bcr). (b) Schematic diagram of the
BCR-ABL p210 transcripts.
The number under the fusion gene transcripts refer to the first (5’) nucleotide of the involved exon,
except when the last (3’) nucleotide of the upstream gene is indicated. The b3-a2 and b2-a2 transcripts
are found most frequently, but sporadic cases with b3-a3 and b2-a3 transcripts have been reported.
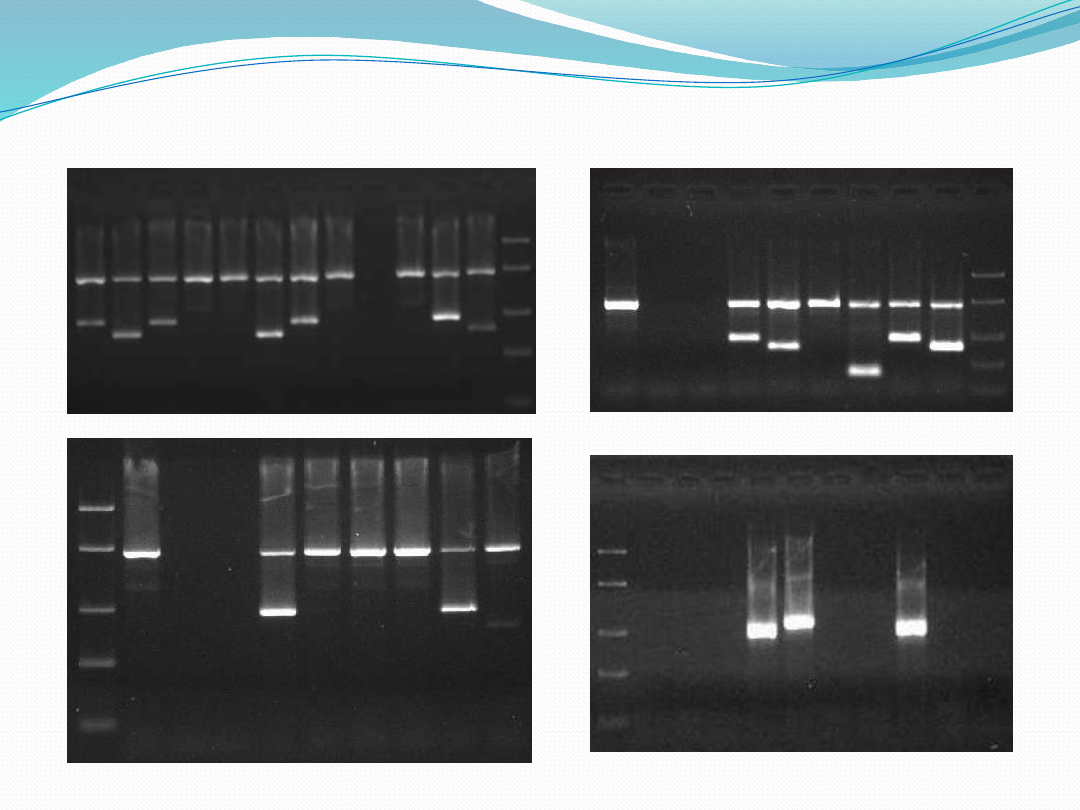
Przewlekła białaczka szpikowa – CML
Przewlekła białaczka szpikowa – CML
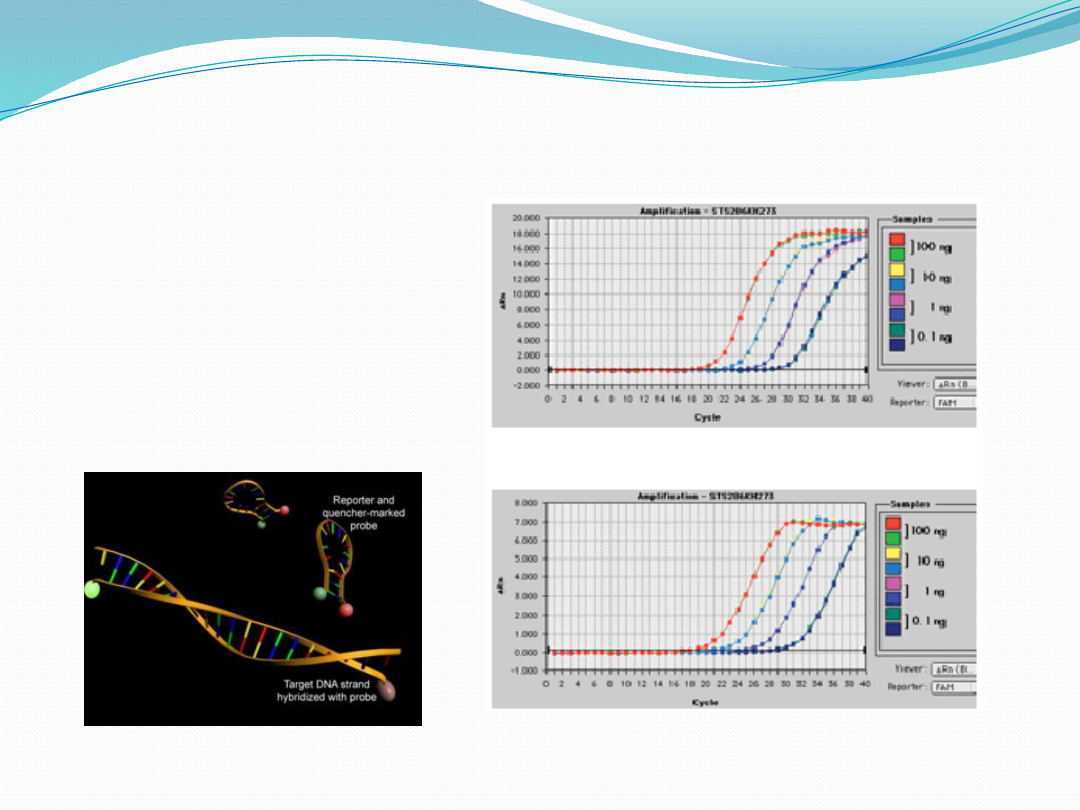
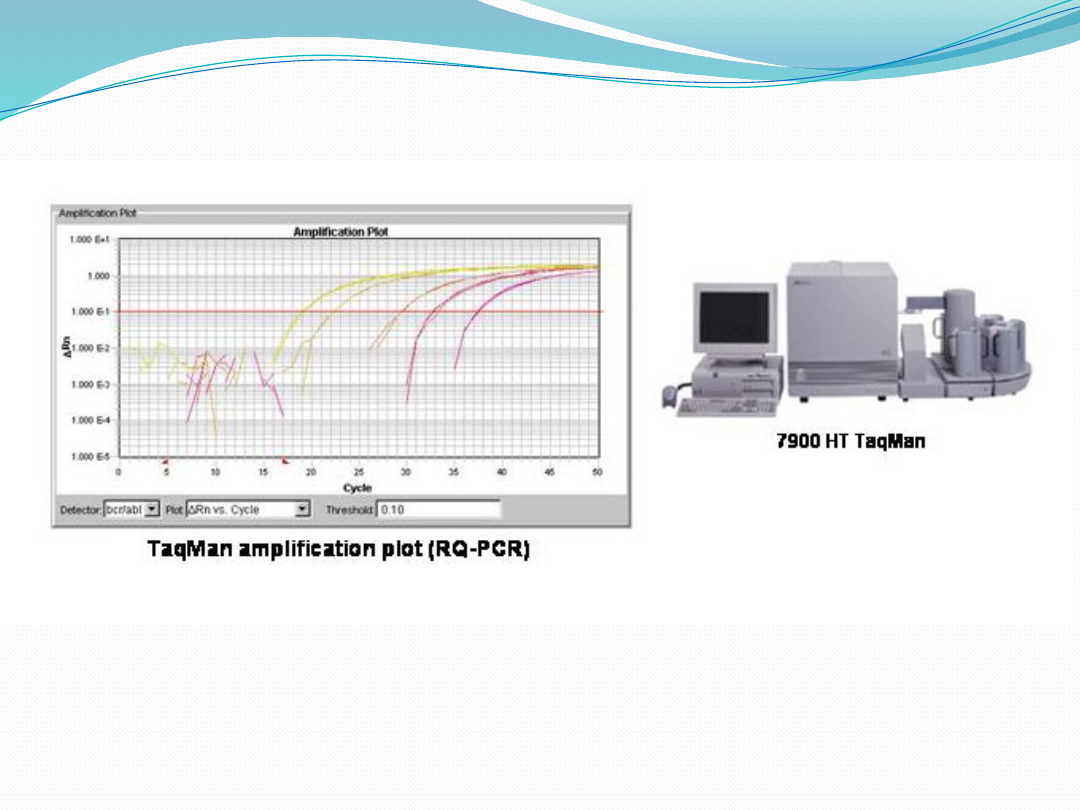
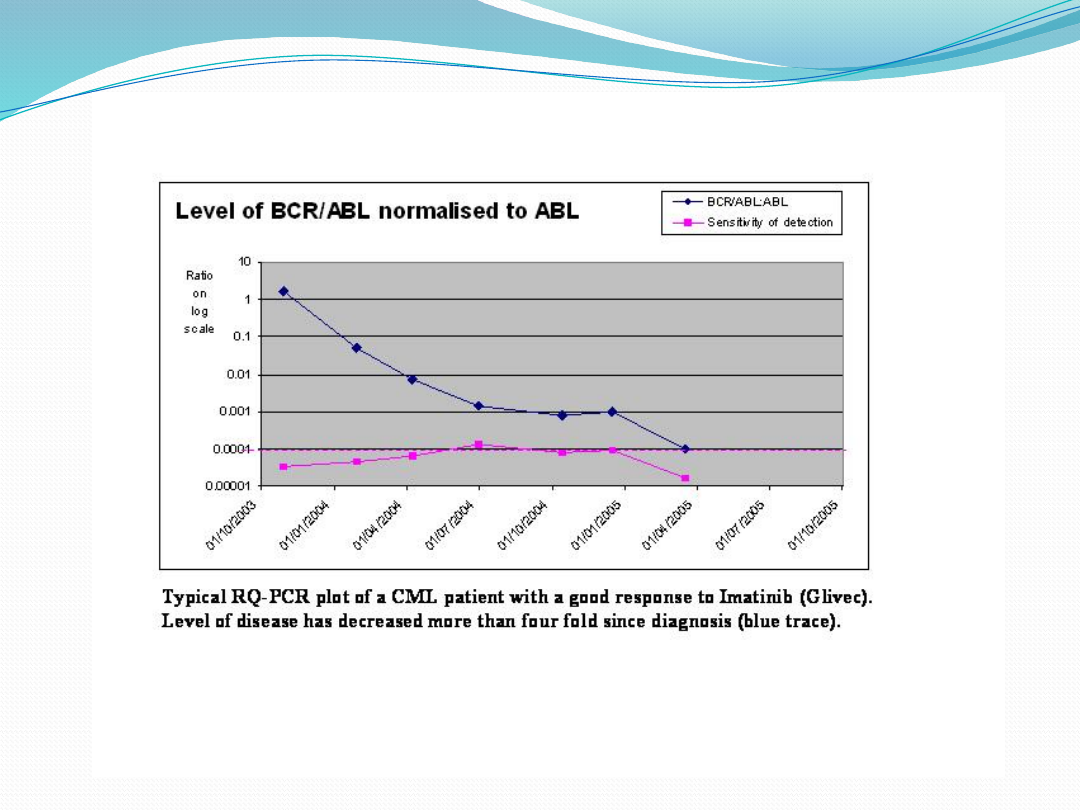
RQ-PCR
PCR w czasie rzeczywistym
(ang. real time PCR)
przebiega wg następujących etapów:
• izolacja RNA,
• reakcja odwrotnej transkrypcji (RT),
• amplifikacja cDNA (PCR),
• interpretacja wyniku.
GAPDH
BCR/ABL
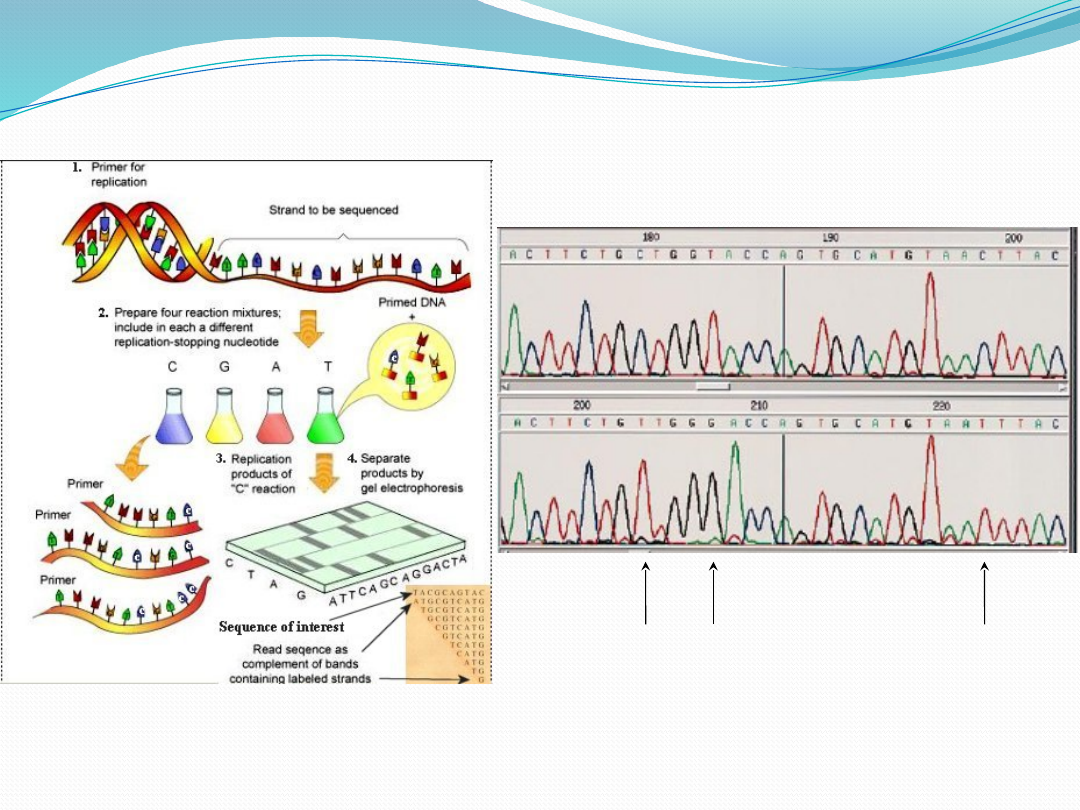
Sekwencjonowanie DNA

Przewlekła białaczka szpikowa – CML
Mutacje kinazy tyrozynowej BCR/ABL
upośledzają efekt inhibitorowy imatynibu
i innych kinaz tyrozynowych.
Wrażliwość poszczególnych form
mutacyjnych na określone inhibitory
cechuje się wysoką zmiennością, ale
inhibitory drugiej linii, tj. dasatynib,
nilotynib, bosutynib, są z reguły bardziej
skuteczne niż imatynib.
Wykrycie mutacji T315I kwalifikuje
pacjenta do alotransplantacji szpiku albo
włączenia do badań klinicznych nad
inhibitorami T315I.

Samoistna nadpłytkowość – ET
Analizę nabytej, somatycznej mutacji punktowej (Val617Phe) w
genie JAK2 (substytucja G → T) można prowadzić przy
zastosowaniu jednej z poniższych metod:
sekwencjonowanie genomowego DNA;
ASO-PCR
(ang. allele-specific oligonucleotide PCR)
;
PCR-RFLP
(ang. PCR followed by restriction fragment length
polymorphism)
;
RQ-PCR
(ang. quantitative real-time PCR)
;
ARMS-PCR / elektroforeza kapilarna
(ang. amplification
refractory mutation system PCR)
;
dHPLC
(ang. denaturing high-performance liquid chromatography)
.
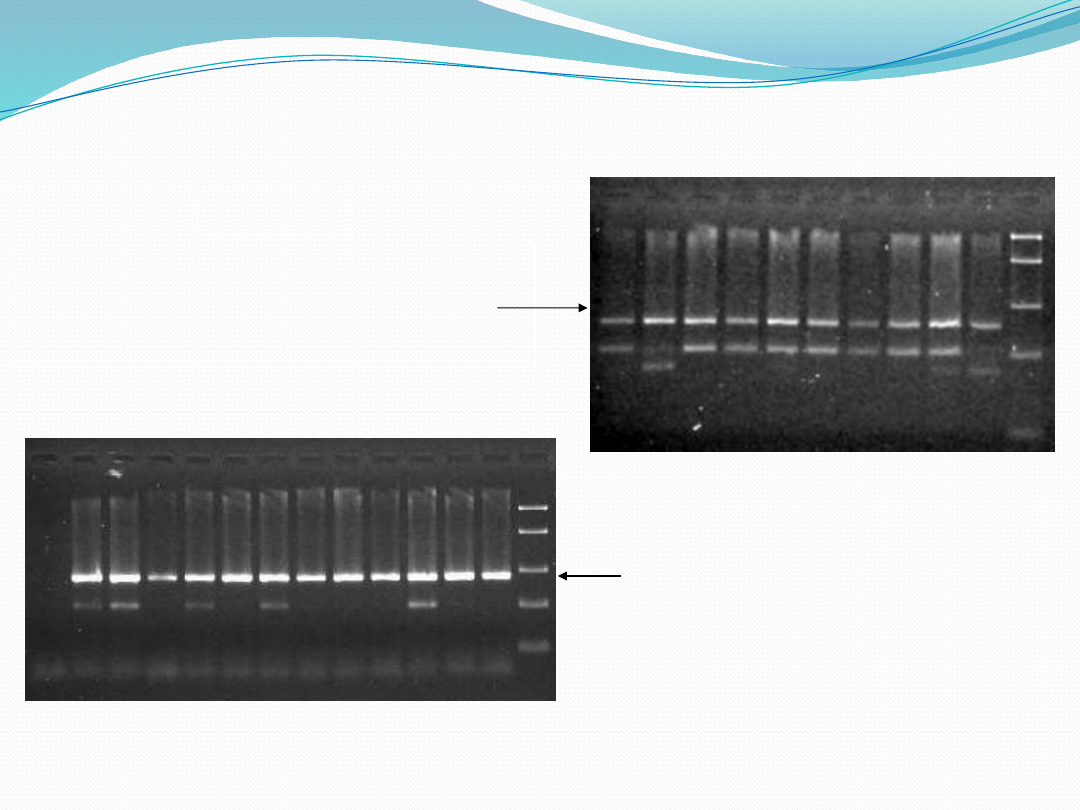
Samoistna nadpłytkowość – ET
JAK2F-C AAC TAT TTA TGG ACA ACA GTC AAA CAA C
JAK2R-C GAA TAG TCC TAC AGT GTT TTC AGT TTC A
JAK2F-V GCA TTT GGT TTT AAA TTA TGG AGT ATA TG
JAK2R-F GTT TTA CTT ACT CTC GTC TCC ACA AAA
ASO-PCR
PCR-RFLP
JAK2-R CTG AAT AGT CCT ACA GTG TTT TCA GTT
JAK2-F1 AGC ATT TGG TTT TAA ATT ATG GAG TAT
JAK2-F2 ATC TAT AGT CAT GCT GAA AGT AGG AGA
enzym restrykcyjny: BsaXI

Mutacja Val617Phe w genie JAK2
Obecność nabytej mutacji V617F w genie JAK2 stwierdza się u
prawie wszystkich chorych na czerwienicę prawdziwą (PV; 80%), a
także u istotnego odsetka pacjentów z nadpłytkowością samoistną
(ET; 40%) i samoistnym zwłóknieniem szpiku (PMF; 40%).
JAK2V617F jest kinazą tyrozynową o aktywności konstytutywnej,
która aktywuje przekaźniki sygnału komórkowego i aktywator
transkrypcji (Stat), aktywowaną mitogenem kinazę białkową
(MAPK) oraz kinazę fosfatydyloinozytolu 3 (PI3K), co prowadzi do
transformacji hematopoetycznych komórek prekursorowych.
Identyfikacja JAK2V617F pozwoliła na opracowanie swoistych
inhibitorów JAK2, które znajdują zastosowanie w leczeniu PV, ET i
PMF.
Źródło: Levine R.L. et al. 2007 Nature Rev Cancer
Standardized RT-PCR analysis of fusion gene transcripts from
chromosome aberrations in acute leukemia for detection of
minimal residual disease.
Report of the BIOMED-1 concerted action:
Investigation of minimal residual disease in acute leukemia.
van Dongen JJM et
el. Leukemia (1999) 13, 1901-28
Table 4 RT-PCR analysis of fusion gene transcripts in acute leukemia
Chromosome
aberration
RT-PCR
target
Responsible
laboratory
Collaborating
laboratory
t(1;19)
(q23;p13)
E2A-PBX1
A Rambaldi et
al.
A Biondi et al.
t(4;11)
(q21;q23)
MLL-AF4
F Griesinger et
al.
A Biondi et al. and EA
Macintyre et al.
t(9;21)
(q22;q22)
AML1-ETO
EA Macintyre et
al.
F Griesinger et al. and JA
Gabert et al.
t(9;22)
(q34;q11)
BCR-ABL
p190
G Saglio et al.
A Rambaldi et al.
t(9;22)
(q34;q11)
BCR-ABL
p210
JA Gabert et al.
M Gonzales et al.
t(12;21)
(p13;q22)
TEL-AML1
JA Gabert et al.
G Saglio et al. and A Biondi et
al.
t(15;17)
(q22;q21)
PML-RARA
A Biondi et al.
P Gameiro et al.
inv(16)
(p13;q22)
CBFB-
MYH11
EA Macintyre et
al.
JA Gabert et al.
del(1)(p32;p32) SIL-TAL1
EA Macintyre et
al.
JJM van Dongen et al.

U około 40-45% chorych na AML nie wykrywa się żadnych zmian w kariotypie,
ale w przypadkach tych można stwierdzić występowanie zmian submikroskopowych.
Najistotniejsze są zmiany typu rearanżacji, mutacji i hiperekspresji w obrębie genów.
Gen
Locus
Typ zmiany
Częstość
nawrotów
Całkowity
czas przeżycia
Zmiany o rokowaniu niekorzystnym
WT1
MLL
FLT3
c-KIT
BCL2
TP53
MDR1
EVI1
11p13
11q23
13q12
4q11-q12
18q21
17p13
7q21.1
3q26
hiperekspresja
częściowa tandemowa
duplikacja PTD
wewnętrzna tandemowa
duplikacja ITD
mutacja
hiperekspresja
mutacja
hiperekspresja
hiperekspresja
wysoka
wysoka
wysoka
wysoka
wysoka
bez znaczenia
wysoka
wysoka
krótki
bez znaczenia
krótki
krótki
krótki
krótki
krótki
krótki
Zmiany o rokowaniu korzystnym
NPM1
CEBPA
5q35
19q13.1
hiperekspresja
hiperekspresja
mutacja
niska przy braku
mutacji FLT3
niska
długi przy braku
mutacji FLT3
długi
Tabela 5
Nowe, molekularne markery o znaczeniu rokowniczym w ostrych białaczkach szpikowych

BADANIA
MOLEKULARNE
W
HEMATOONKOLOGI
I
KATEDRA I KLINIKA HEMATOONKOLOGII
I TRANSPLANTACJI SZPIKU
UNIWERSYTET MEDYCZNY W LUBLINIE
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
Wyszukiwarka
Podobne podstrony:
WYKŁADY GENETYKA W.Badanie molekularne 10.10.09r....
badanie srodoperacyjne, Analityka Medyczna UMB, III, Patomorfologia, Wykłady
Badania molekularne serotypów wirusa pryszczycy
Badanie natężenia czynników szkodliwych na stanowisku pracy-hałas, ANALITYCZNE METODY INSTRUMENTALNE
Paski Analityczne do badania moczu, testy ogólno dostępne w aptece 1
Metody badania kwasów nukleinowych - lekcja IV, egzamin biologia molekularna i parazytologia
badania analit
cwiczenie-5-Badanie-ogolne-moczu-2, Nauka, Analityka
Epidemiologia analityczna(metoda porównawcza) doraźne badania epidemio ppt
Badania analityczne
Badanie ogólne kału, Studia - materiały, Analityka ogólna i techniki pobierania materiału
Badanie ogólne moczu, Analityka
Badania kliniczne, serologiczne, molekularne
Paski Analityczne do badania moczu, testy ogólno dostępne w aptece 1
1 podejście indukcyjne jest metodą badania analitycznego
3 ćwiczenia BADANIE asfaltów
BADANIEepipelne
BADANIA 3
więcej podobnych podstron