
WYKRYWANIE
MUTACJI

RFLP
(Restriction
Fragment-
Lenght Polymorphism)
Polimorfizm Długości
Fragmentów Restrykcyjnych
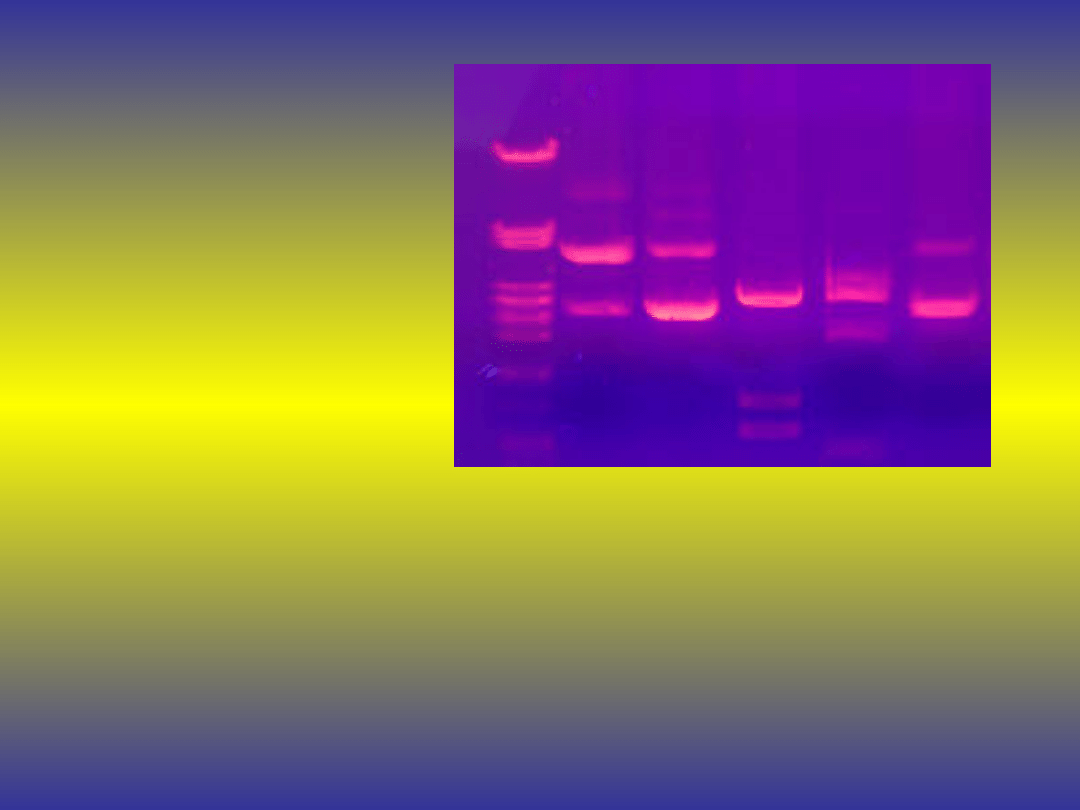
Wykrywanie mutacji
w produktach PCR
pociętych
restryktazami, a
następnie
poddanych
elektroforezie w
żelu agarozowym
lub
poliakrylamidowy
m

SSCP
(single strand
conformation
polimorphism )
Polimorfizm
konformacyjny
jednoniciowego DNA
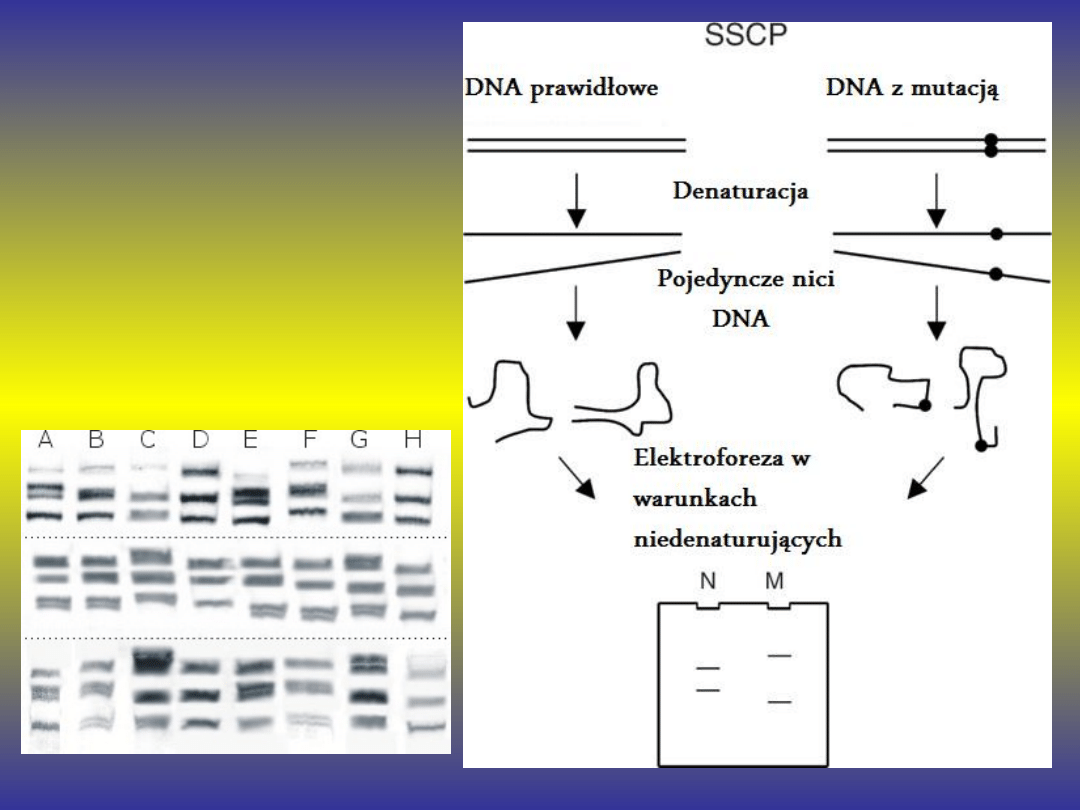
Metoda służąca do badania
polimorfizmu
konformacyjnego
jednoniciowych
cząsteczek DNA, (różnic
powstałych w wyniku
tworzenia odmiennych
struktur
trójwymiarowych przez
jednoniciowe cząsteczki
DNA, normalne i
zmutowane tego
samego fragmentu
łańcucha
polinukleotydowego)
dzięki wykorzystaniu
elektroforezy.
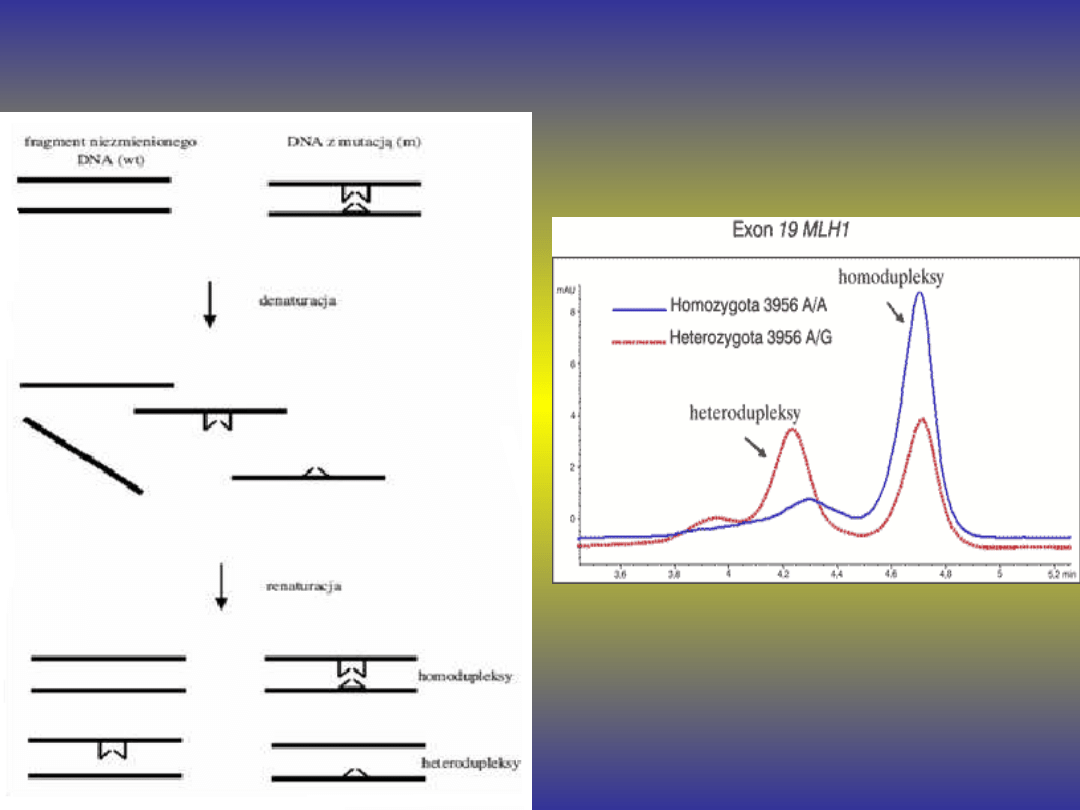
HET – analiza
heterodupleksów

DGGE
(Denaturing gradient gel
electrophoresis)
Elektroforeza na Żelach
z Gradientem Czynnika
Denaturującego
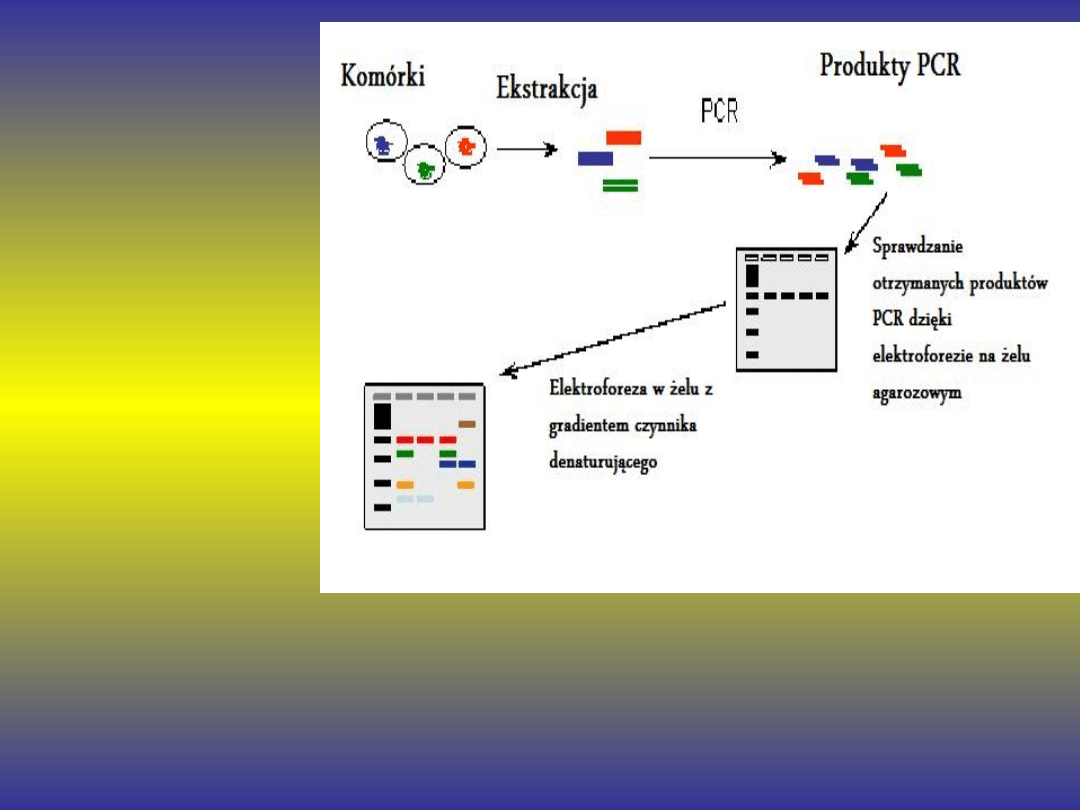
Metoda polegająca
na wykryciu
mutacji za
pomocą
elektroforezy na
żelu z
wzrastającym
stężeniem
czynnika
denaturującego,
np. formamidu.
Fragmenty DNA
zawierające
mutacje ulegają
denaturacji przy
innym stężeniu
formamidu niż
nić prawidłowa

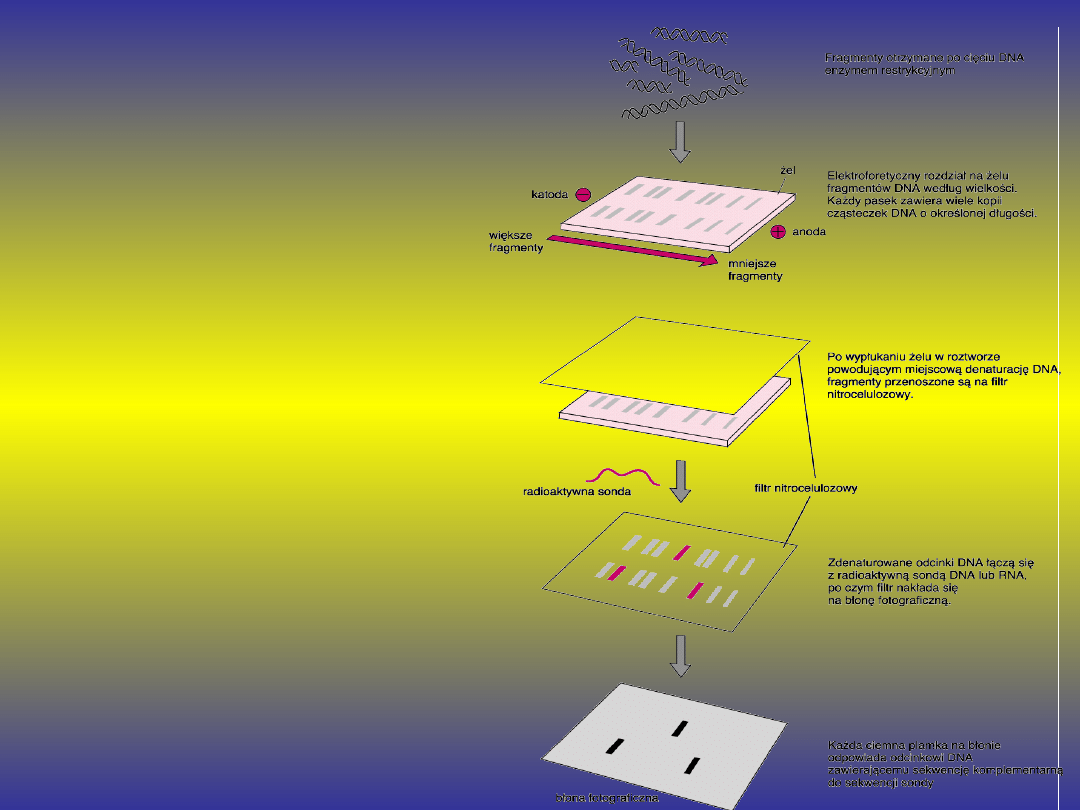
Metoda Southern`a
Metoda ta służy do wykrywania
dużych zmian w obrębie
genomu. Aby ją wykonać,
należy przeprowadzić
elektroforetyczne rozdzielenie
mieszaniny fragmentów DNA
(np. po cięciu enzymami
restrykcyjnymi), a następnie.
umieścić w alkalicznym
buforze, który wywoła
denaturację DNA. Następnie
całość przenosi się na błonę
nylonową. Po odpowiednim
spreparowaniu błony
nylonowej z DNA wykonuje się
hybrydyzację z sondą, a
następnie przy pomocy
odpowiedniej metody detekcji
uwidacznia się interesujące
nas fragmenty DNA.

ASO
(
Allele Specific
Oligonucleotide
hybridization)
Hybrydyzacja
Oligonukleotydu
Specyficznego względem
Allelu
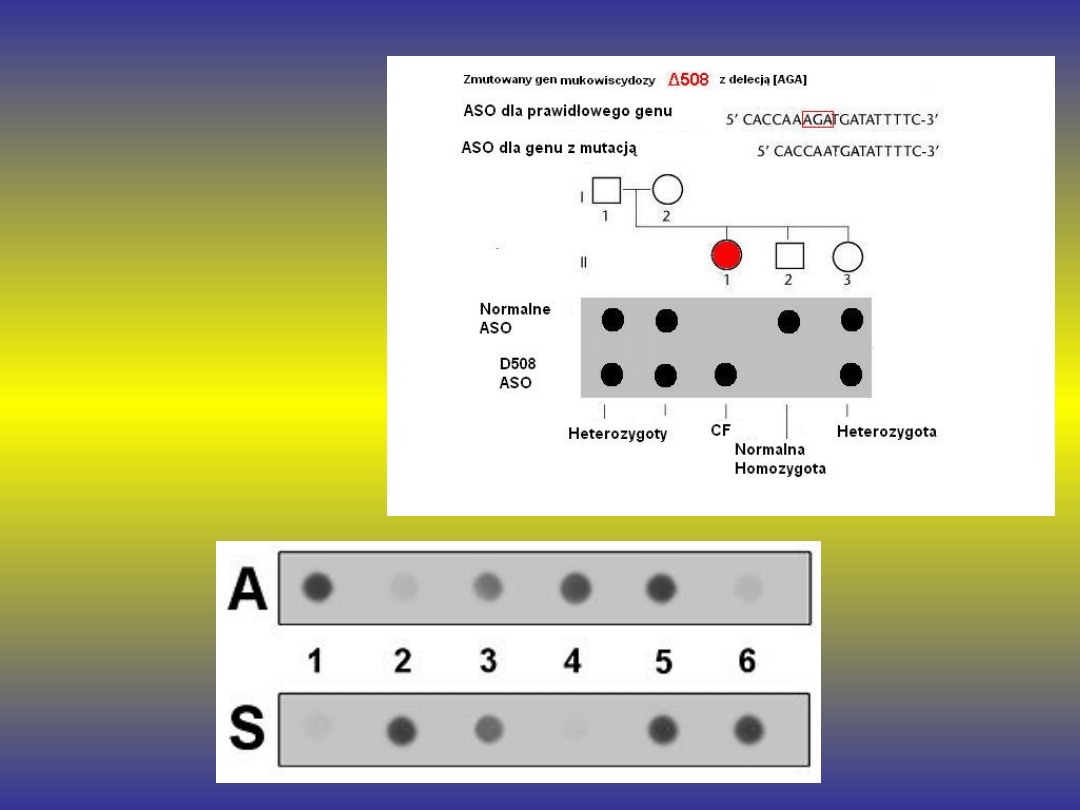
Metoda, w której
wykorzystuje się
oligonukleotydy, z
których jeden jest
komplementarny do
genu prawidłowego, a
drugi do fragmentu o
znanej mutacji tego
genu. Wykorzystuje
się ją do badań
przesiewowych
anemii
sierpowatokrwinkowej
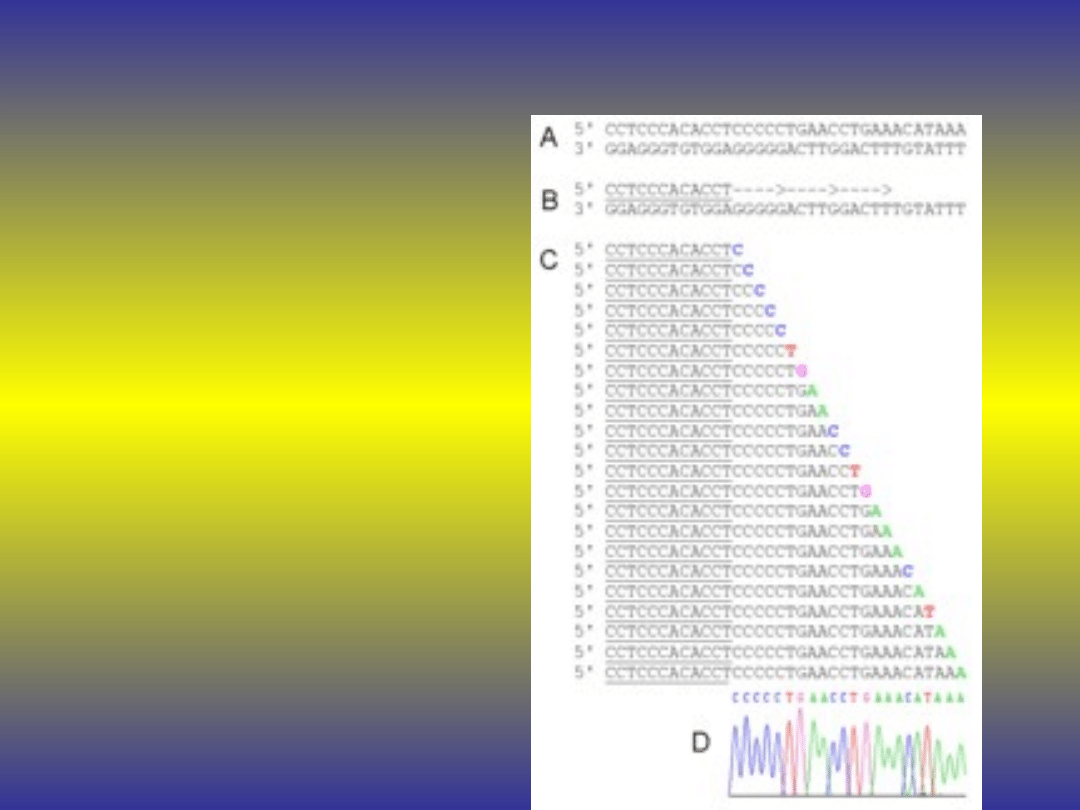
Sekwencjonowanie
Najbardziej czuła
metoda w
wykrywaniu
zmian w
materiale
genetycznym
umożliwiająca
pełną jego
charakterystykę
.
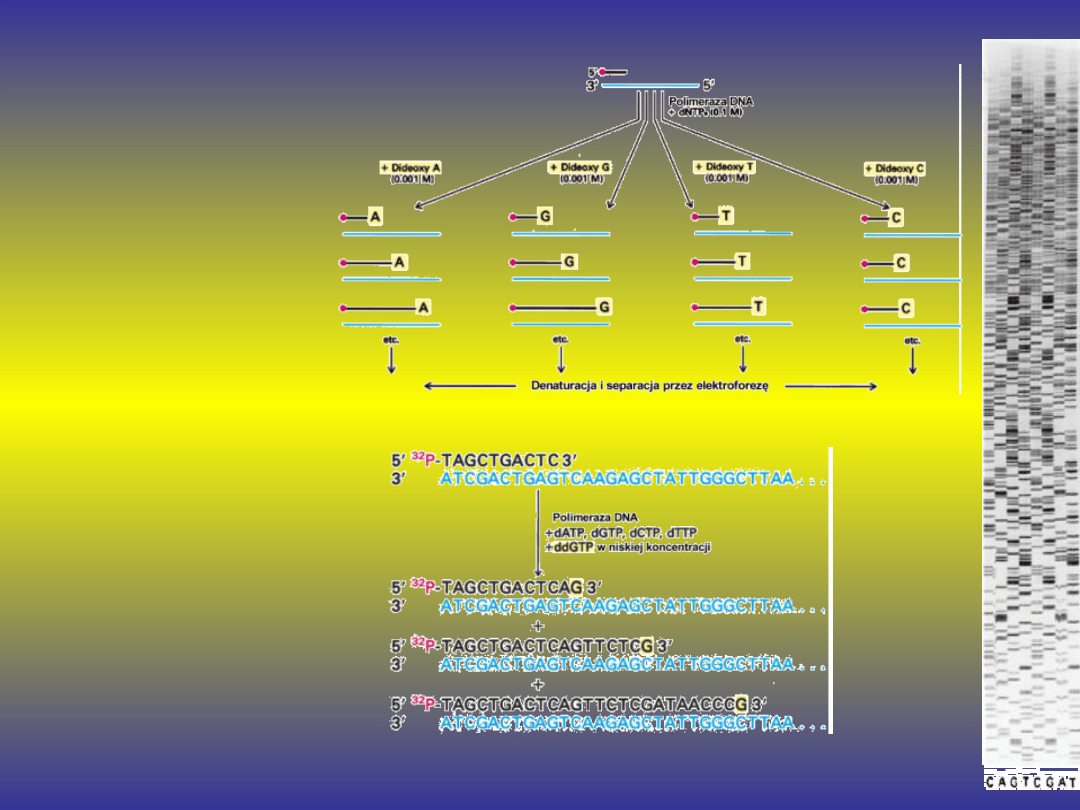
Enzymatyczna - metoda
Sangera
Na jednoniciowej matrycy
syntetyzuje się in vitro
DNA. Syteza przebiega
od radioaktywnego ,
oligonukleotydowego
startera
komplementarnego do
3` końca matrycy.
Reakcję wykonuje się
równolegle w czterech
probówkach , w których
oprócz kompletu
deoksytrifosforanów
nukleotydów (dATP ,
dCTP , dTTP , dGTP)
dodana jest niewielka
ilość jednego z nich w
postaci
dideoksytrifosforanu
nukleotydu , co
uniemożliwia
kontynuowanie reakcji w
momencie włączenia
takiego nukleotydu w
syntetyzowany łańcuch ,
ponieważ brakuje wtedy
grupy hydroksylowej na
3` końcu.
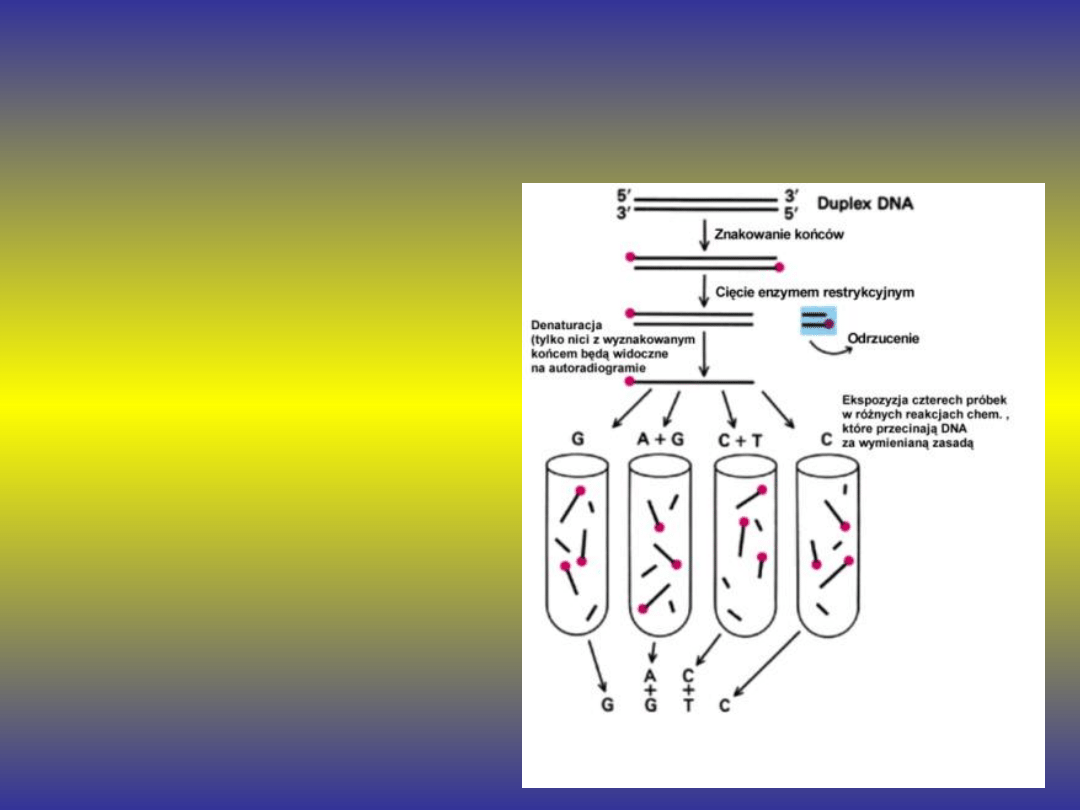
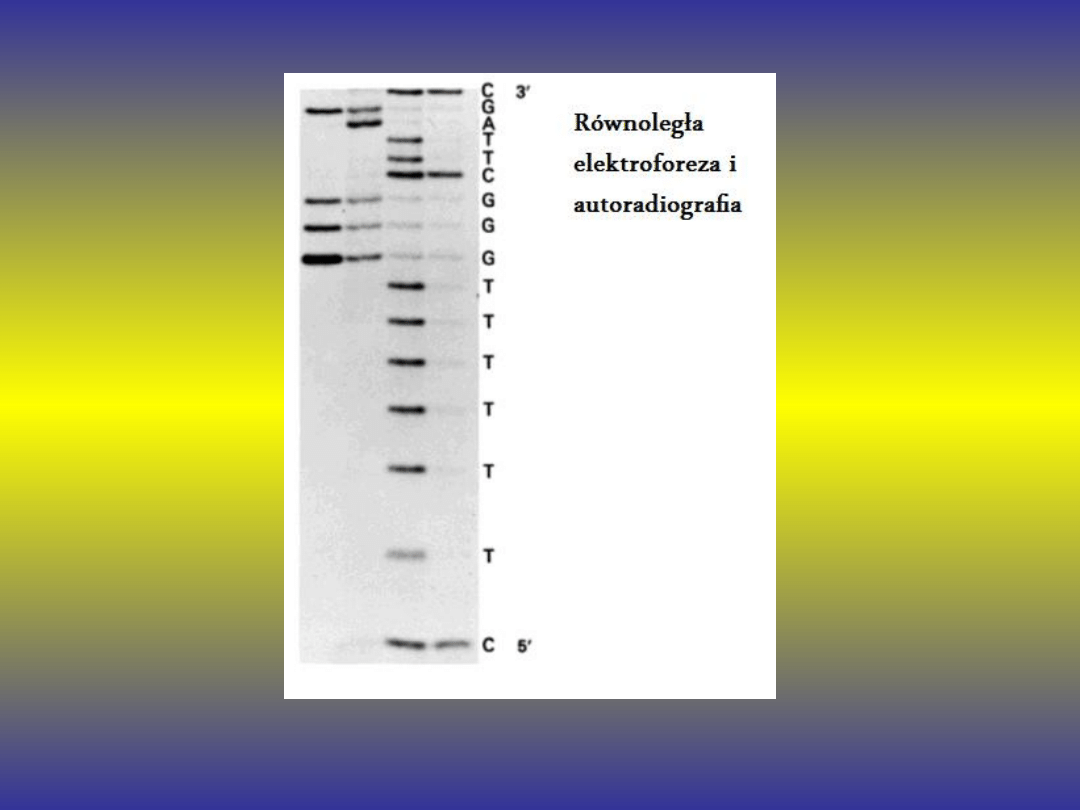
Chemiczna - metoda
Maxama-Gilberta
Polega na degradacji
wyznakowanych na 5` końcu
cząsteczek DNA związkami
chemicznymi przecinającymi
specyficznie wiązania
fosfodiestrowe za nukleotydem
odpowiadającym określonej
zasadzie azotowej. Wynikiem
reakcji jest zbiór fragmentów
DNA o różnej długości co
spowodowane jest takim
doborem warunków , że w
poszczególnych cząsteczkach
przecinane jest tylko jedno lub
dwa wiązania. Fragmenty z
czterech niezależnych reakcji
rozdziela się elektroforetycznie
po czym poddaje autoradiografii.
Prążki na autoradiogramie
odpowiadają fragmentom DNA
posiadającym na końcu 5`
znakowany fosfor a na końcu 3`
określoną zasadę.

FISH
(fluorescent in situ
hybridization)
Hybrydyzacja
Fluorescencyjna
in situ
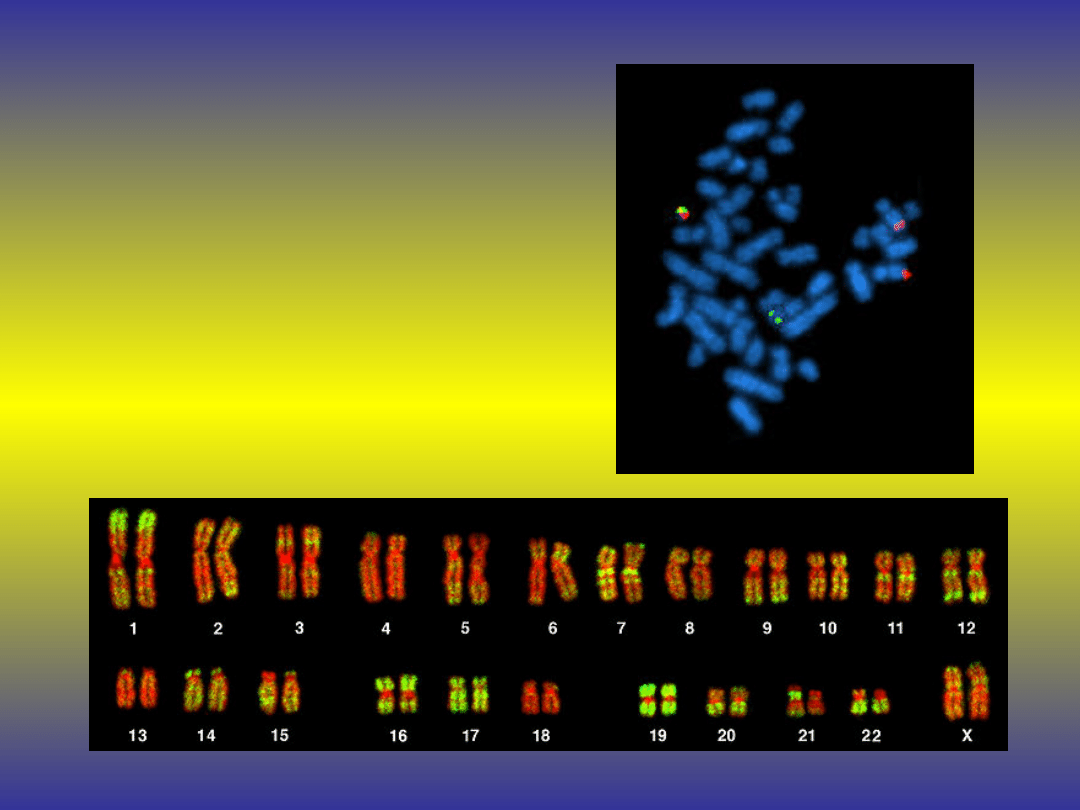
Metodę tą wykorzystuje
się do wykrywania
m.in. aberacji
chromosomowych z
wykorzystaniem sond
wyznakowanych
fluorescencyjnie
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
Wyszukiwarka
Podobne podstrony:
PREZ metody wykrywania mutacji
WYKRYWANIE MUTACJI 2
PREZ metody wykrywania mutacji
Metody wykrywania mutacji
GMO metody wykrywania 2
Mutacje chromosomowe strukturalne
Podmiotowe i przedmiotowe badanie lekarskie w wykrywaniu nowotworów
Materiał genetyczny, mutacje, systemy naprawy DNA, test Amesa
Etanol skażony formaldehydem w nielegalnym obrocie napojami alkoholowymi wykrywanie i oznaczanie
hodowlane i niehodowlane metody wykrywania drobnoustrojów
Domowy wykrywacz gazu
1-Kefir chroni przed mutacjami w DNA, ZDROWIE-Medycyna naturalna, Poczta Zdrowie
ZMIENNOSC I MUTACJE, fizjoterapia, biologia medyczna
Jak wykrywać i usuwać uszkodzenia sieci 8 sieci
Podział mutacji
IMiD Test przesiewowy do wykrywania zaburzen w rozwoju fizycznym u dzieci
200603avt745 avt 2788 WYKRYWACZ PLUSKIEW, BottomLayerNormal
więcej podobnych podstron