BADANIA MUTACJI W DIAGNSTYCE NOWOTWORÓW
Badania:
badania chromosomów
metody genetyki klasycznej (prążkowanie)
cytogenetyka molekularna (FISH, MC-FISH, CGH)
badania molekularne
poszukiwanie mutacji: PCR, SSCP...
sekwencjonowanie genu lub fragmentu genu
badanie szczególnych zdarzeń w genomie
MSI
LOH
SNP (single nucleotide polymorphism)
Mikroczipy (microarrays)
DNA
Białkowe
chromosom 17q21
100 k par zadad
24 eksony
duże białko 1863 aa
funkcje:
aktywator transkrypcji
rola w naprawie dwuniciowych pęknięć DNA (DBS)
uczestniczy w remodelowaniu chromatyny
mutacje de novo -bardzo rzadko(przyczyna ęfektu założyciela)
rodzaje mutacji(bardzo duże spektrum):
5382insC (najczestsza)
C61G
4153delA
185delAG
13,9% przypadkow klinicznych w Polsce ma mutację BRCA1
badania:
sekwencjonowanie
long PCR , Soutern-RFLP, SSCP
ACRS-PCR, ASA -PCR
test BRCA1 (mulitiplex PCR)-z krwi obwodowej ,szybki i skuteczny
wskazania do badań - stwierdzenie u krewnych I i II stopnia:
raka piersi/jajnika
zachorowania przed 50 r.ż na raka piersi lub w dowolnym wieku na raka jajnika
chromosom 13q12-13
26 eksonów
białko 3418 aa
brak „hot spots”(trzeba sekwencjonowac)
funkcje:
aktywator transkrypcji
wykazuje aktywność acylotransferazy histonów
sztuczne wytworzenie miejsca restrykcyjnego
sekwencje które chcemy badać byłyby palindromowe, gdyby nie 1 nukleotyd lub kilka
wykrywanie mutacji przy pomocy specyficznych nukleotydów
oprócz starterów flankujących(rewers i foward) stosuje się
starter w pełni komplementarny do allela z mutacją (foward)
starter w pełni komplementarny do allela niezmienionego(foward)
startery sa tak zlokalizowane ze powstaja różne produkty
2 h
geny naprawy DNA
mutacje w genie MSH2
transwersja A na T w intronie 5 (50%)
mutacje w genie MLH1
tranzycja A na G w intronie 18
90% mutacji dziedzicznych w HNPCC (3-6% nowotworów jelita grubego)
inne geny których mutacje są odpowiedzialne za HNPCC to :
MSH3,
MSH6,
PMS1,
PMS2
badania:
test nosicielstwa mutacji MSH2, MLH1
immunohistochemiczne metody (ekspresja bialka)
sekwencjonowanie
wskazania do wykonania:
podejrzenie o HNPCC
kryteria Amsterdamskie
wiąże się z utratą jednego z dwóch różnych alleli tego samego genu (heterozygota), co prowadzi do hemizygotyczności (obecność pojedynczego allelu) danego genu.
odgrywa ważną rolę w genetycznej etiologii nowotworów, gdyż
delecja jest jednym z głównych mechanizmów odpowiedzialnych za utratę funkcji genów supresorowych i mutatorowych.
geny obu tych grup mają recesywny efekt działania na poziomie komórkowym, tzn. mutacje muszą dotyczyć obu alleli danego genu, aby doszło do jego inaktywacji.
odgrywa kluczową rolę zarówno w inicjacji procesu nowotworowego, jest też obserwowane we wczesnych i zaawansowanych stadiach karcinogenezy.
badanie LOH
polimorfizm długości markerów mikrosatelitarnych
wykorzystanie polimorficznych markerów mikrosatelitarnych
1-6 nukleotydów
równomiernie rozproszone w genomie, np. (CA)n,
zidentyfikowanych ponad 5000
allel matczyny i ojcowski - heterozygoty informatywne
markery informatywne, tzn. takie które występują w komórkach prawidlowych w postaci heterozygotycznej (posiadają różne allele w badanym locus).
DNA guza jest amplifikowane ze starterami (najczęściej znakowanymi fluorescencyjnie) swoistymi dla wybranych markerów mikrosatelitarnych.
otrzymane produkty są następnie
rozdzielane na żelu (najczęściej poliakrylamidowym) i uwidaczniane za pomocą barwienia bromkiem etydyny, srebrzeniem, audioradiografią
analizowane w sekwenatorach DNA z użyciem markerów fluorescencyjnych.
wynik badania:
w żelu poliakrylamidowym heterozygota rozpoznawana jest na podstawie obecności dwóch prążków
w automatycznym sekwenatorze na podstawie występowania dwóch krzywych dla badanego locus
analiza polegaja na porównaniu markerów mikrosatelitarnych pomiędzy DNA wyizolowanym z tkanki nowotworu a DNA tkanek prawidłowych (głównie z krwi obwodowej), pochodzących od tego samego pacjenta.
LOH rozpoznawana jest jako utrata jednego z prazkow w DNA guza w porównaniu z DNA komórek prawidłowych.
analiza LOH w sekwenatorze polega na porównaniu pola pod krzywymi odpowiadającymi allelom badanego markera mikrosatelitarnego.
[N1/N2]/[T1/T2] - częściowa redukcja (co najmniej 20%) ilości jednego z alleli tkanki nowotworowej w stosunku do tkanki referencyjnej
całkowity LOH - rzadko
są to krótkie sekwencje DNA,
składają się z tandemowo ułożonych, powtarzających się motywów
motywy te sa 1-6 nukleotydwe (mono, di, tri, tetra, penta, sexta - nukleotydowe).
są równomiernie rozmieszczone w genomie, zarówno w obszarach kodujacych, jak i niekodujących
zmienne
zmiana długości (wielkości) alleli na skutek zwiększenia lub zmniejszenia liczby powtórzeń nukleotydowych
cecha fenotypu mutatorowego (Replication Error Phenotype; RER).
nowotwory RER+
niepolipowatym raku jelita grubego(92%)
sporadyczny rakiem jelita grubego (15%)
2-64% innych nowotworów zarówno dziedzicznych, jak i sporadycznych.
do oceny MSI
polimorfizm długości markerów mikrosatelitarnych
obecnie rekomendowane jest stosowanie 5 markerów mikrosatelitarnych:
2 mononukleotydowych
3 dinukleotydowych
rozpoznawana na podstawie każdej zmiany długości allelu, bedacej wyrazem zmiany liczby jednostek powtarzalnych w mikrosatelitach w komórkach guza w porównaniu z tkanką prawidłową.
nieswoista utrata heterozygotycznosci, wyrazajaca sie utrata jednego z dwoch alleli danego markera w mniej niz 20% badanych guzow, jest traktowana jako wyraz MSI.
analizę niestabilności mikrosatelitarnej w automatycznym sekwenatorze można przeprowadzać zarówno u konstytucjonalnych homozygot, jak i heterozygot w odniesieniu do badanego markera.
zaleta jest olbrzymia ilość informacji jaką dostarcza to badanie
pozwala na wykrycie naddatków lub ubytków materiału genetycznego w badanym DNA nowotworowym
BRCA1
BRCA2
ACRS-PCR (ang.artificial creating restriction site -PCR)
wt
naprawa
mut
enzym nie trawi
ASA-PCR (ang.allele-specific amplification -PCR)
wt
mut
MSH2, MLH1
LOH = utrata heterozygotyczności (ang.Loss of Heterozygosity)
Polimorfizm długości markerów mikrosatelitarnych w ocenie LOH
MS = mikrosatelity = sekwencje mikrosatelitarne
MSI = niestabilność mikrosatelitarna (ang. microsatellite instability)
Polimorfizm długości markerów mikrosatelitarnych w ocenie MSI
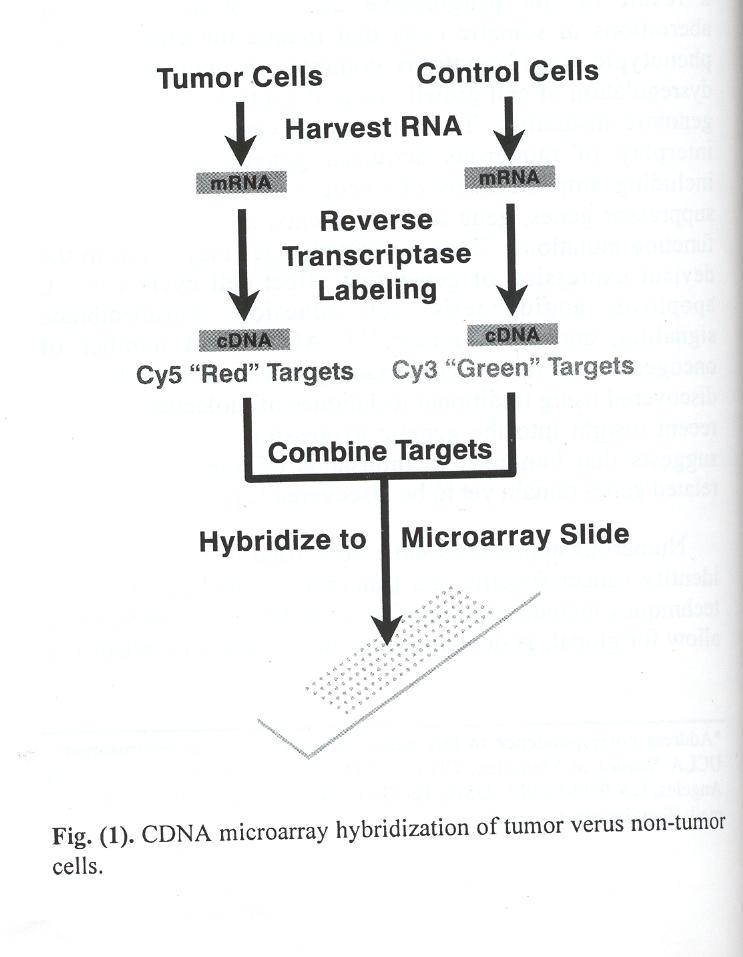
mikroczipy = mikromacierze (ang.microarrays)
DNA guza
DNA kontrolne
mRNA mRNA
odwrotna transkrypcja
znakowanie
cDNA cDNA
hybrydyzacja
(płytka mikroczipowa)
□□□□□□□□
□ czerwono -zielony - norma
□ nadmiar czerwonego -naddatek (amplifikacja) w komórkach guza
□ nadmiar zielonego- delecja w komorkach guza
□ brak barwy - brak transferuslepa próba
Wyszukiwarka
Podobne podstrony:
Podmiotowe i przedmiotowe badanie lekarskie w wykrywaniu nowotworów
Wczesna diagnostyka nowotworów
KARTA BADANIA MOWY, Diagnoza
Formułowanie problemu w badaniach naukowych i diagnozie psychologicznej
Metody diagnostyki nowotworow, Medycyna, Onkologia
diagnostyka nowotworów
8 Diagnostyka nowotworo¦üw j brzusznej
15 Diagnostyka nowotworow
8 Badania stosowane w diagnostyce chorób płuc, Medycyna, Interna, Pulmonologia
Podstawowe objawy i algorytmy diagnostyczne nowotworów III lek
Wczesna diagnostyka nowotworów
Badania dodatkowe w diagnostyce Nieznany
badanie nasienia, Diagnostyka laboratoryjna
Badania obrazowe w diagnostyce chorób serca
Badania obrazowe w diagnostyce chorób serca
więcej podobnych podstron