Tadeusz Trzmiel
Instytut Biochemii Technicznej
Biodegradacja aromatycznych związków nitrowych
Występowanie aromatycznych związków nitrowych w przyrodzie jest głównie wynikiem działalności człowieka. Związki te wytwarzane są przez przemysł na wielką skalę i stosowane m.in. jako pestycydy, farmaceutyki, barwniki i materiały wybuchowe [2,25-30]. Większość związków nitroaromatycznych jest silnie toksyczna dla ludzi i zwierząt ,a także dla mikroorganizmów, np. obecność tych ksenobiotyków w osadzie czynnym może destabilizować ciągły proces oczyszczania ścieków [2,31].
Związki aromatyczne mogą być degradowane przez różne drobnoustroje zarówno w tlenowych, jak i beztlenowych warunkach [2,30]. Znane są dwa główne sposoby bezpośredniej eliminacji grupy nitrowej tych ksenobiotyków przez mikroorganizmy: proces może zachodzić na drodze redukcji, bądź utleniania [26,30,32,33].
Redukcja różnych związków nitroaromatycznych przebiegająca przy udziale mikroorganizmów wydaje się mieć wspólny mechanizm zarówno w tlenowych jak i beztlenowych warunkach .Katalizatorami w tego typu reakcjach są nitroreduktazy, z których większość wykazuje szeroką specyficzność substratową [2]. Powszechnie spotykaną odmianą redukcyjnego szlaku degradacji jest przemiana nitroaromatycznych związków w pochodne aminowe (rys.17) [30].
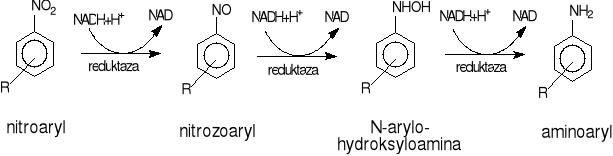
Rys.17. Redukcyjny szlak degradacji związków nitroaromatycznych katalizowany przez Pseudomonas R= -OH, -COOH, -Cl, -NO2
Utworzone aminy aromatyczne często ulegają transformacjom do trwałych związków azotowych [2]. Możliwa jest także dalsza przemiana pochodnych aniliny w reakcji acetylacji [25,36,38], która stanowi ważny mechanizm mikrobiologicznej detoksykacji [25].
Drugi typ redukcyjnego szlaku degradacji omawianych ksenobiotyków, sprowadzający się do tworzenia związków nitrozoaromatycznych i N-hydroksyloaminoaromatycznych zazwyczaj poprzedza oksydacyjną biotransformację i mineralizację tych związków [30].
Reakcje utlenienia związków nitroaromatycznych, którym towarzyszy eliminacja azotynu [2,26,35,39,40], katalizowane są przez oksygenazy - monooksygenazy lub dioksygenazy [2,26,35,37,39,42]. W procesach tych uczestniczy tlen cząsteczkowy. Mechanizm eliminacji grupy nitrowej drogą oksydacyjną pozwala uniknąć tworzenia się toksycznych pochodnych amin aromatycznych[35].
Nitrobenzeny
Nitrobenzen zaliczany do najbardziej toksycznych polutantów, wytwarzany jest w dużych ilościach przez przemysł chemiczny i szeroko stosowany, głównie do produkcji aniliny i pyroksyliny oraz mydeł i past do obuwia. Mimo, że nitrobenzen jest odporny na działanie większości enzymów drobnoustrojowych i toksyczny dla wielu mikroorganizmów, znane są szczepy zdolne do jego biodegradacji wyizolowane min. z osadu czynnego, ścieków komunalnych, gleby i wody gruntowej [2,43-45]. Istnieje kilka różnych dróg degradacji tego związku [44-46].
Neribeg i Nelde [5] udowodnili, że w obecności drożdży nitrobenzen może ulegać redukcji do aniliny. Opisano także całkowitą redukcję grupy nitrowej do aminowej tego ksenobiotyku, przebiegającą w warunkach beztlenowych przy udziale bakterii bytujących w ściekach [1,43,44].
Ralstonia eutropha JMP134 prowadzi częściową redukcję nitrobenzenu i następującą po niej izomeryzację N-hydroksyloaminobenzenu, w wyniku której powstają 2- i 4-aminofenol, które są końcowymi metabolitami tego szlaku (rys.18) [25]
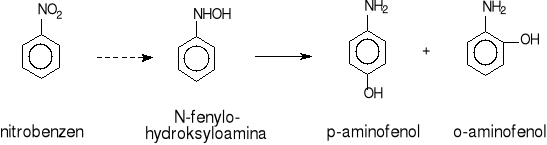
Rys.18. Przemiana nitrobenzenu do 2- i 4-aminofenolu
Badania nad zdolnością szczepu Pseudomonas pseudoalcaligenes JS45 do metabolizowania nitrobenzenu przyczyniły się do nakreślenia całkowitej drogi degradacji tego ksenobiotyku (rys.18) [44-46]. Szczep Pseudomonas pseudoalcaligenes JS45 może wykorzystywać nitrobenzen jako jedyne źródło węgla, azotu i energii.
W początkowych etapach tego szlaku nitrobenzen jest redukowany przez dwie kolejno działające nitroreduktazy sprzężone z NADPH odpowiednio do nitrozobenzenu i N-hydroksyloaminobenzenu, Ten ostatni ulega przekształceniu w 2-aminofenol, który dalej w wyniku rozszczepienia pierścienia aromatycznego (katalizowanego przez dioksygenazę) daje semialdehyd 2-aminomukonowy. W kolejnym etapie powstały semialdehyd jest utleniany w obecności NADH do 2-aminomukonianu. W procesie deaminacji hydrolitycznej z 2-aminomukonianu powstaje kwas 2-hydroksymukonowy, który może ulegać spontanicznej izomeryzacji (lub pod działaniem odpowiedniej mutazy).
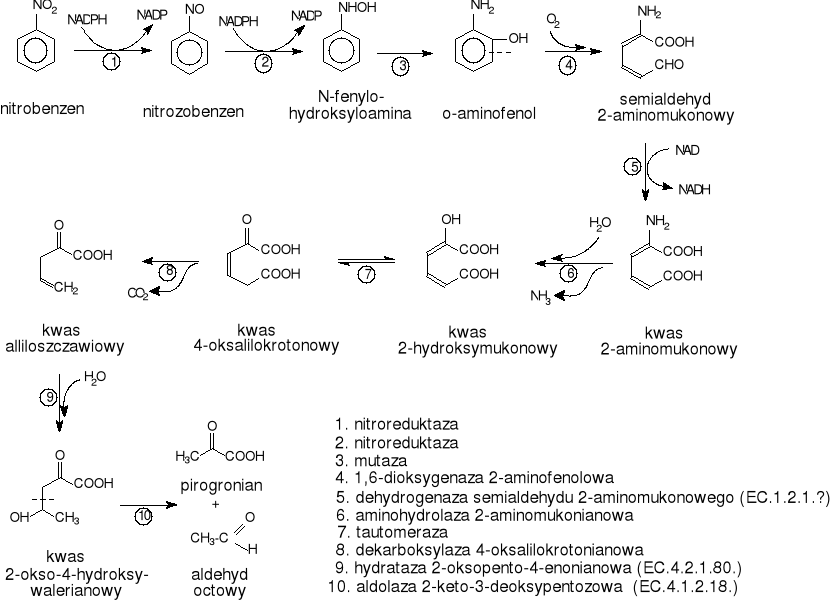
Rys.19. Droga biodegradacji nitrobenzenu u bakterii Ps.pseudoalcaligenes IS45
Pirogronian i aldehyd octowy są końcowymi metabolitami tego szlaku. Badania wykazały [44,45,47,48], że powyższa droga funkcjonuje także w przypadku drobnoustrojów wyizolowanych z zanieczyszczonych wód gruntowych.
Katalizowany przez Comamonas sp. alternatywny szlak biodegradacji nitrobenzenu, rozpoczyna się dioksygenacją tego ksenobiotyku, w wyniku której powstaje nitrohydrochinon, który ulega następnie spontanicznym przekształceniom w katechol [44-46].
Pseudomonas putida F1 i Pseudomonas sp. 150 są także zdolne do degradacji nitrobenzenu drogą dioksygenacji. U innych mikroorganizmów działa z kolei monooksygenaza, która katalizuje wstępny atak na nitrobenzen [2,43].
Reakcja dioksygenacji, której towarzyszy eliminacja grupy nitrowej, zachodzi także w początkowych etapach szlaku degradacji 1,3-dinitrobenzenu przy udziale Rhodococcus sp. QJ—1 (rys.20) [2].
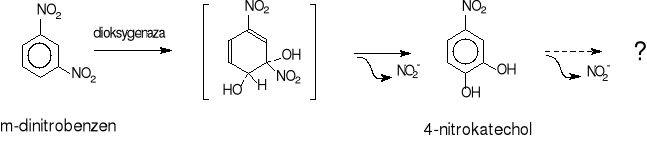
Rys.20. Degradacja 1,3-dinitrobenzenu przez Rhodococcus sp.
Nitrobenzoesany
W mikrobiologicznej degradacji kwasów nitrobenzoesowych, podobnie jak innych związków nitroaromatycznych, zasadniczą rolę spełniają reakcje redukcji i utleniania.
Redukcyjna droga biodegradacji kwasów nitrobenzoesowych, której końcowymi metabolitami są odpowiednie kwasy aminobenzoesowe (porównaj rys.17), była obserwowana m.in. w przypadku Pseudomonas sp. CBC3 [2], Pseudomonas fluorescens i Nocardia [45].
Niektóre mikroorganizmy są zdolne do przemiany kwasu 2-nitrobenzoesowego poprzez kolejne redukcje, w wyniku których powstaje kwas nitrozobenzoesowy i N-hydroksyloaminobenzoesowy [2,49-52].
Podczas degradacji kwasu 4-nitrobenzoesowego przez Comamonas acidovorans NB 210 ma miejsce utworzenie 4-nitrozobenzoesanu i 4-hydroksyloaminobenzoesanu. Kolejnym etapem tej drogi metabolicznej jest utlenienie 4hydroksyloaminobenzoesanudo kwasu protokatechowego (rys.21) [2].
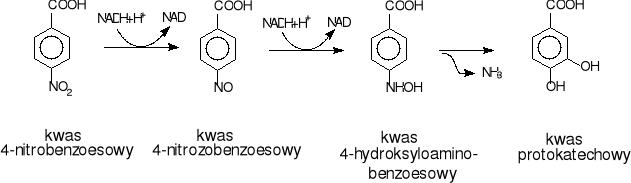
Rys.21. Szlak degradacji 4-nitrobenzoesanu przy udziale Comamonas acidovorans NB 210
Andreoni i wsp. [2] opisali oksydacyjną degradację 2-nitrobenzoesanu, która funkcjonuje u szczepu Achromobacter. Dioksygenaza produkowana przez ten mikroorganizm katalizuje utlenienie kwasu 2-nitrobenzoesowego, któremu towarzyszy eliminacja azotynu.
Z kolei szczep Pseudomonas JS51 w warunkach tlenowych prowadzi oksydacyjną degradację kwasu 3-nitrobenzoesowego. W początkowym etapie tego szlaku zachodzi utlenienie 3-nitrobenzoesanu do kwasu protokatechowego katalizowane przez 3,4-dioksygenazę. Dalej ma miejsce przemiana kwasu protokatechowego w semialdehyd 2-hydroksy-4-karboksymukonowy, w której bierze udział najprawdopodobniej 4,5-dioksygenaza protokatechowa (rys.22) [39,53].
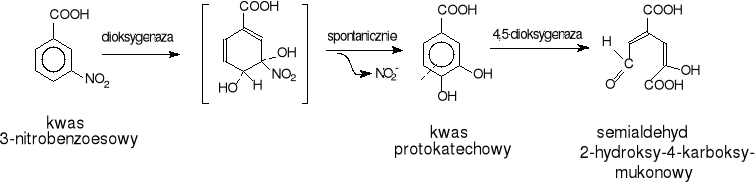
Rys.22. Degradacja kwasu 3-nitrobenzoesowego przez Pseudomonas IS51
W literaturze [16,49] można znaleźć wzmianki, że również pewien gatunek Nocardia przekształca 3-nitrobenzoesan oraz 4-nitrobenzoesan w kwas protokatechowy, a ten ostatni jest utleniany do kwasu 2-oksoadypinowego.
Nitrofenole
Nitrofenole zaliczane są do głównych substancji zanieczyszczających środowisko naturalne [2,54]. Znaleziono jednak mikroorganizmy, m.in. izolowane z gleby, które wykazują zdolność do degradacji tych ksenobiotyków [2,55].
4-Nitrofenol, uznawany za jeden z głównych polutantów obciążających środowisko naturalne, jest często obecny w ściekach przemysłowych, a także w glebie m.in. jako produkt hydrolizy insektycydów, takich jak „parathion” czy „methyl parathion” [2,25,53-58]. Jego biodegradacji poświęcono też najliczniejsze badania. Wiadomo już, że kilka tlenowych kultur bakteryjnych należących do różnych gatunków Flavobacterium, Moraxella, Nocardia, Arthrobacter i Bacillus może metabolizować ten związek, i w podobny sposób 2-nitrofenol, usuwając grupę nitrową w postaci azotynu [25,17,37,41,59]. Pomimo pewnych różnic w biochemicznych mechanizmach biodegradacji 4-nitofenolu u poszczególnych gatunków tych bakterii ogólnie wyróżnia się dwie alternatywne drogi. Pierwsza z nich (rys.23), wspólna dla gram-ujemnych bakterii, polega na eliminacji grupy nitrowej pod działaniem monooksygenazy z utworzeniem 1,4-benzochinonu, który jest redukowany do hydrochinonu, a ten dalej pod działaniem dioksygenazy przekształcany jest w semialdehyd 4-hydroksymukonowy z dalszą transformacją poprzez maleinooctan do kwasu 2-oksoadypinowego [25,17,41].
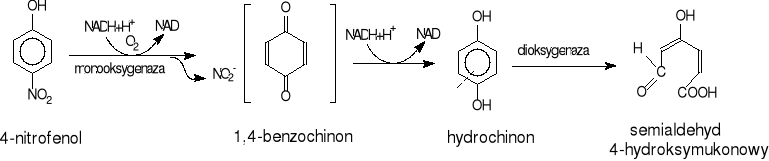
Rys. 23, Eliminacja grupy nitrowej z 4-nitrofenolu
Druga droga eliminacji grupy nitrowej z 4-nitrofenolu, funkcjonująca m.in. u Arthrobacter sp. lub u Bacillus sphaericus JS905, jest dwustopniowa: w pierwszym etapie, pod działaniem monooksygenazy, następuje hydroksylacja pierścienia z utworzeniem 4-nitrokatecholu i dopiero w drugim etapie usuwana jest grupa nitrowa z utworzeniem 1,2,4-trihydroksybenzenu [17,59].
Pseudomonas putida B2 [25,37] jest zdolny do degradacji 2-nitrofenolu drogą oksydacyjną. W pierwszym etapie katalizowanym przez monooksygenazę nitrofenolową sprzężoną z NADPH ma miejsce utlenienie 2-nitrofenolu do katecholu z jednoczesną eliminacją grupy nitrowej w postaci azotynu. Następnie pierścień aromatyczny katecholu ulega intradiolowemu rozszczepieniu pod działaniem 1,2-dioksygenazy katecholowej. Katalizowana przez monooksygenazę nitrofenolową przemiana 2-nitrofenolu, w wyniku której następuje eliminacja grupy nitrowej w postaci azotynu, przypomina początkowy etap poprzednio omawianej drogi degradacji 4-nitrofenolu [55]. Interesujący jest fakt, że ten sam szczep bakteryjny Pseudomonas putida B2 degraduje 3-nitrofenol drogą redukcyjną z wytworzeniem 1,2,4-trihydroksybenzenu jako produktu przejściowego[26].
Biodegradacja 3-nitrofenolu przez Ralstonia eutropha (Alcaligenes eutrophus) JMP 134 [25] rozpoczyna się jednoetapową redukcją tego ksenobiotyku do 3-hydroksyloaminofenolu (rys.24). Ta wstępna przemiana, przebiegająca zarówno w warunkach tlenowych, jak i beztlenowych, jest katalizowana przez nitroreduktazę 3-nitrofenolową sprzężoną z NADPH. Podobna reakcja redukcji nitrobenzenu przebiegająca u Pseudomonas pseudoalcaligenes JS45 jest dwuetapowa [46]. 3-Hydroksyloaminofenol jest dalej przekształcany w aminohydrochinon, z którego w warunkach beztlenowych po acetylacji powstaje N-acetyloaminohydrochinon. Podobnie przebiegającą redukcyjną drogę degradacji 3-nitrofenolu u Pseudomonas sp. opisali Schackmann i Müller [36]. Różnica polega na tym, że 3-nitrofenol u Pseudomonas sp. ulega pełnej redukcji do 3-aminofenolu, który jest dalej przekształcany przez acetylację grupy aminowej w 3-N-acetyloaminofenol. Obie N-acetylowe pochodne aminofenoli uważane są za metabolity końcowe biodegradacji 3-nitrofenolu w warunkach beztlenowych („ślepe uliczki metabolizmu”). Natomiast w warunkach tlenowych Ralstonia eutropha (Alcaligenes eutrophus) JMP 134 może wykorzystywać 3-nitrofenol jako podstawowe źródło energii, węgla i azotu [25,60], jakkolwiek biochemiczny mechanizm tej przemiany jeszcze nie został ustalony.
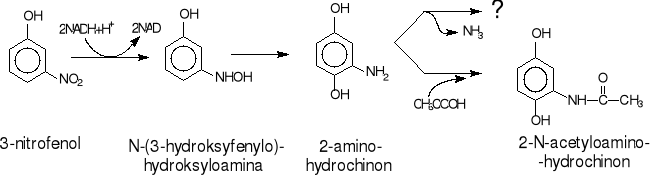
Rys.24. Początkowe etapy degradacji 3-nitrofenolu przez Ralstonia eutropha
Nitrotolueny
Opisano liczne przykłady biodegradacji mononitrotoluenów, 2,4-dinitrotoluenu i 2,4,6-trinitrotoluenu.
Niektóre szczepy Pseudomonas metabolizują 4-nitrotoluen poprzez wstępne utlenienie grupy metylowej do karboksylowej i następującą po nim redukcję grupy nitrowej do hydroksyloaminowej [26,40].
Na przykład Haigler i Spain [36] przedstawili drogę degradacji 4-nitrotoluenu dla szczepu Pseudomonas sp. 4NT (rys.25). W pierwszym etapie tej drogi (1) ma miejsce utlenienie grupy metylowej 4-nitrotoluenu (katalizowane najprawdopodobniej przez monooksygenazę toluenową), w wyniku czego powstaje alkohol 4-nitrobenzylowy. Następnie przekształcany jest on (2) do 4-nitrobenzaldehydu w obecności dehydrogenazy alkoholowej i dalej (3) przez odpowiednią dehydrogenazę (np. aldehydową) utleniany jest do kwasu 4-nitrobenzoesowego. Dalsza droga przemian tego związku u Pseudomonas sp. 4NT jest podobna do opisanej przy biodegradacji 4-nitrobenzoesanu u Comamonas acidovorans NB 210 (porównaj rys.21). Kolejne redukcje grupy nitrowej (4) i (5) prowadzą do 4-hydroksyloaminobenzoesanu, który ulega utlenieniu do kwasu protokatechowego pod wpływem 3,4-dioksygenazy (6). Końcowym etapem tego szlaku jest rozszczepienie pierścienia aromatycznego pod działaniem 4,5-dioksygenazy (7).
Rys.25. Droga degradacji 4-nitrotoluenu u Pseudomonas sp. 4NT
Utlenienie grupy metylowej 3- lub 4-nitrotoluenu prowadzące do wytworzenia odpowiedniego alkoholu nitrobenzylowego katalizowane jest przez monooksygenazę toluenową kodowaną w plazmidzie TOL [26,40,61]. Natomiast Pseudomonas putida F1 i Pseudomonas sp. JS150 wytwarzają dioksygenazę toluenową, która przekształca 4-nitrotoluen w 2-metylo-5-nitrofenol i 3-metylo-6-nitrokatechol, natomiast 2- i 3-nitrotoluen przekształcane są przez ten enzym do odpowiednich alkoholi benzylowych [62].
Alternatywna droga degradacji 4-nitrotoluenu zaproponowana przez Spiess`a i wsp. [33] dla Mycobacterium sp. (rys.26) rozpoczyna się redukcją grupy nitrowej do hydroksyloaminowej (1) bez wstępnego etapu utleniania grupy metylowej. N-Hydroksy-p-toluidyna (4-hydroksyloaminotoluen) powstała w wyniku tej reakcji jest dalej (2) przekształcana do 3-hydroksy-p-toluidyny. Rozszczepienie typu meta pierścienia aromatycznego (3) daje semialdehyd 2-amino-5-metylomukonowy. W reakcji utleniania tego ostatniego związku w obecności NAD (4) powstaje kwas 2-amino-5-metylomukonowy i w kolejnej przemianie (5) zostaje uwolniony amoniak (mechanizm tej reakcji nie został jeszcze poznany). Wykazano ponadto, że semialdehyd 2-amino-5-metylomukonowy może również ulec spontanicznej transformacji (6) do kwasu 5-metylopikolinowego. Ta odnoga uważana jest za ślepą uliczkę w omawianym szlaku biodegradacji 4-nitrotoluenu.
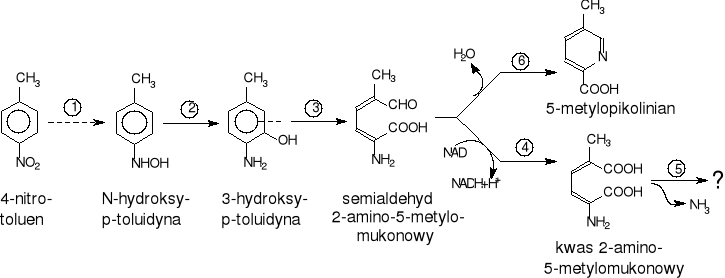
Rys.26. Droga degradacji 4-nitrotoluenu u Mycobacterium sp. 4NT
Oksydacyjna eliminacja azotynu z pojawieniem się 3-metylokatecholu jest początkowym etapem biodegradacji 2-nitrotoluenu u Pseudomonas sp. JS42 (rys.27) [40].
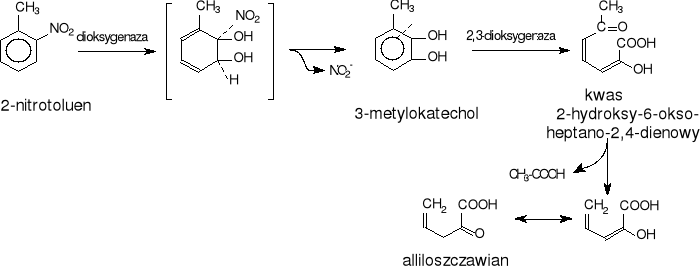
Rys.27. Droga degradacji 2-nitrotoluenu u Pseudomonas sp. IS 42
Haigler i wsp. [40] przypuszczają, że przekształcenie 2-nitrotoluenu w 3-metylokatechol z wydzieleniem azotynu katalizowane jest u tego szczepu Pseudomonas przez specyficzną dioksygenazę 2-nitrotoluenową. Powstały 3-metylokatechol jest dalej metabolizowany poprzez rozszczepienie pierścienia typu meta (ekstradiolowe). W ekstrakcie komórkowym Pseudomonas sp. JS42 stwierdzono także aktywność 2,3-dioksygenazy katecholowej oraz hydrolazy kwasu 2-hydroksy-6-oksoheptadienowego, które najprawdopodobniej uczestniczą w tym szlaku degradacji 2-nitrotoluenu.
2,4-Dinitrotoluen może być biodegradowany przez drobnoustroje z wykorzystaniem reakcji redukcji i rzadziej utlenienia. Wykazano, że mikroorganizmy osadu czynnego mają zdolność redukcji 2,4-dinitrotoluenu do 2-amino-4-nitrotoluenu i 4-amino-2-nitrotoluenu w obecności egzogennego źródła węgla zarówno w tlenowych jak i beztlenowych warunkach [2,63]. Związki te są także początkowymi metabolitami podczas mineralizacji 2,4-dinitrotoluenu przez Phanerochaete chrysosporium (rys.28) [30,64].
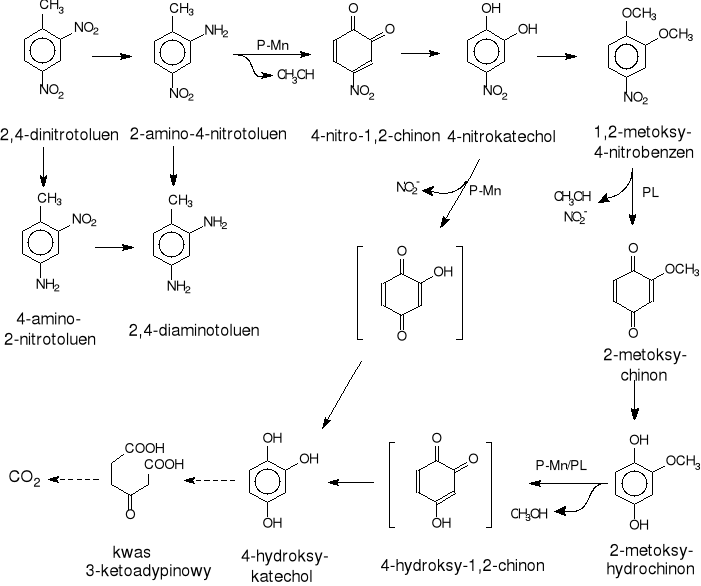
Rys.28. Droga degradacji 2,4-dinitrotoluenu u Phanerochaete chrysosporium P-Mn - peroksydaza manganozależna; PL - peroksydaza ligninowa
Noguera i Freedman [30] dowiedli, że szczep Pseudomonas aeruginosa jest zdolny do kometabolicznej redukcji obu grup nitrowych 2,4-dinitrotoluenu w warunkach tlenowych i beztlenowych. Głównymi produktami tego szlaku degradacji są 2-amino-4-nitrotoluen i 4-amino-2-nitrotoluen, ale mogą powstawać także niewielkie ilości 2,4-diaminotoluenu. Ponadto, utworzone aminy aromatyczne mogą ulegać dalej acetylacji (rys.29).
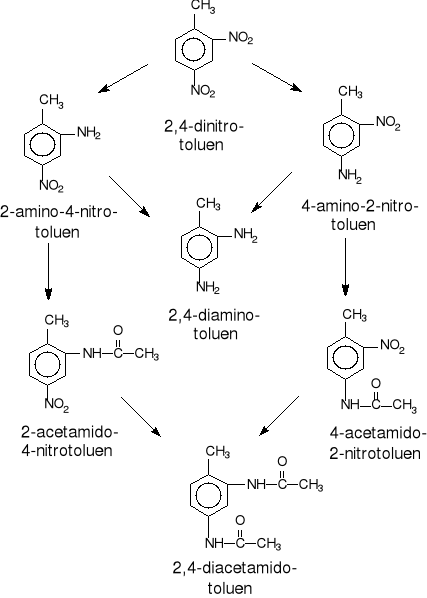
Rys.29. Drogi degradacji 2,4-dinitrotoluenu w warunkach beztlenowych
McCormic i wsp. [65,63] wykazali, że produktami biotransformacji dinitrotoluenów mogą być także związki azowe.
Natomiast Spanggord i wsp. [2] opisali początkowe etapy oksydacyjnej biodegradacji 2,4-dinitrotoluenu prowadzonej przy udziale Pseudomonas sp. (rys.30). Utlenienie 2,4-dinitrotoluenu do 4-metylo-5-nitrotoluenu katalizuje dioksygenaza.
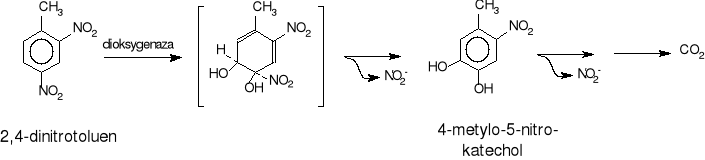
Rys.30. Wstępne etapy oksydacyjnej biodegradacji 2,4-dinitrotoluenu
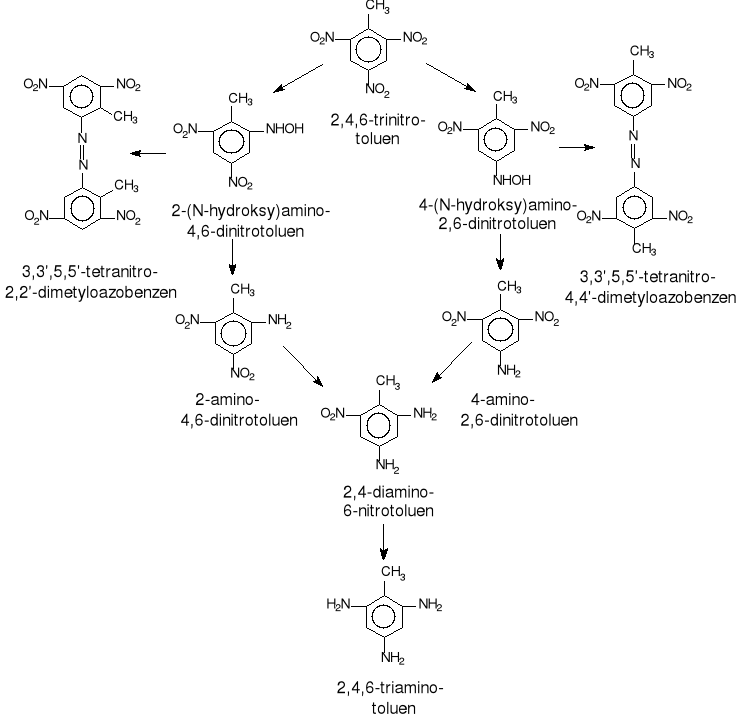
Rys. 31. Szlak mikrobiologicznej degradacji 2,4,6-trinitrotoluenu
Redukcja grup nitrowych do aminowych wydaje się być także dominującym mechanizmem degradacji 2,4,6-trinitrotoluenu zarówno u tlenowych jak i beztlenowych bakterii i grzybów [2]. Niespecyficzne reduktazy większości badanych bakterii preferują atak na grupę -NO2 w pozycji 4, co prowadzi do powstania w pierwszej kolejności 4-amino-2,6-dinitrotoluenu [27,66-68]. Pośrednimi metabolitami tej przemiany i ewentualnych kolejnych redukcji grup nitrowych są odpowiednie nitrozo- i hydroksyloamino-pochodne toluenu [27,28,66]. W warunkach tlenowych w wyniku redukcji powstają głównie 4-amino-2,6-dinitrotoluen oraz jego regioizomer 2-amino-4,6-dinitrotoluen, które uważane są za końcowe produkty metabolizmu (tzw. ślepa uliczka biodegradacji) [28,66] i akumulowane są w komórkach [66,69]. Całkowita redukcja trinitrotoluenu do triaminotoluenu wymaga natomiast warunków beztlenowych [27,66,68].
McCormic i wsp. [66] opisali szlak mikrobiologicznej degradacji 2,4,6-trinitrotoluenu, którego metabolitami są dinitro(N-hydroksylo)aminotolueny, dinitroaminotolueny, nitrodiaminotolueny i zawiązki azowe (rys.31). Identyczne metabolity zidentyfikowano w czasie degradacji trinitrotoluenu przez bakterie z rodzaju Pseudomonas [2].
Fiorella i Spain [27] wykazali, że Pseudomonas pseudoalcaligenes JS52 może degradować trinitrotoluen nie znaną dotąd drogą redukcyjną przebiegającą poprzez 2,4-(N-hydroksy) diamino-6-nitrotoluen i 2-(N-hydroksy) amino-4-amino-6-nitrotoluen (rys.32). Dwa pierwsze etapy tej drogi katalizuje nitroreduktaza nitrobenzenowa działając w obecności NADPH. Stwierdzono, że w warunkach tlenowych powstaje żółty polarny związek A i wydziela się azotyn. Mechanizmy odpowiedzialne za tworzenie związku X oraz jego tożsamość nie są znane. W warunkach beztlenowych nie powstaje ten metabolit ani nie wydziela się amoniak. Przypuszcza się, że związek X może być pośrednim produktem rozszczepienia pierścienia aromatycznego.
Ustalono, że zredukowane metabolity 2,4,6-trinitrotoluenu mogą ulegać dalszym przemianom na drodze mikrobiologicznej. Do tego typu transformacji należy zaliczyć utlenianie 4-amino-2,6-dinitrotoluenu do kwasu 4-amino-2,6-dinitrobenzoesowego, addycję grupy metoksy do 2,4-diamino-6-nitrotoluenu, w wyniku czego powstaje eter metylowo-2,4-diamino-6-nitrobenzylowy, acetylację 2,4-diamino-6-nitrotoluenu do 4-N-acetylo-2-amino-6-nitrotoluenu i inne [27,28,70].
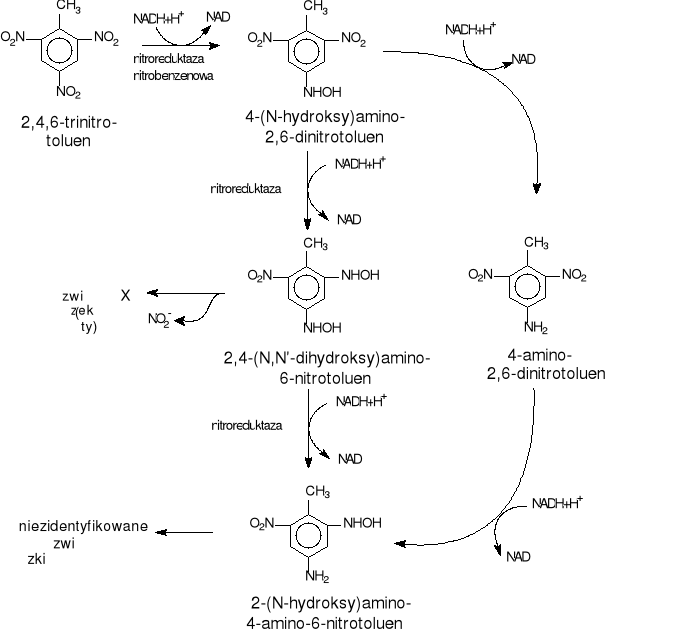
Rys.32. Transformacja 2,4,6-trinitrotoluenu przez Pseudomonas
pseudoalcaligenes JS52
Podsumowanie
Procesy biodegradacji obcych substancji nabrały obecnie szczególnie dużego znaczenia w związku z daleko posuniętym zanieczyszczeniem środowiska naturalnego. Ważnym zagadnieniem związanym z procesami biodegradacji jest fakt, że w wyniku rozkładu lub przemian niektórych ksenobiotyków powstają produkty znacznie trwalsze lub bardziej odporne. W związku z tym w wielu pracowniach naukowych prowadzi się badania nad zdolnością różnych mikroorganizmów do rozkładu ksenobiotyków i ewentualnym zastosowaniem aktywnych szczepów do detoksykacji tych związków.
Z danych literaturowych wynika, że najczęściej tego typu badania prowadzi się pod kątem biodegradacji jednego, z góry określonego ksenobiotyku. W tym celu prowadzi się skrining drobnoustrojów wykazujących zdolność do jego degradacji, następnie ustala się warunki hodowli wyselekcjonowanych szczepów, ustala się pośrednie produkty degradacji tego ksenobiotyku, enzymy uczestniczące w procesie i ich właściwości, mechanizm ich działania i na koniec, o ile jest to możliwe, proponuje się szlak biodegradacyjny.
Literatura
Russel S.: Biotechnologia, PWN, W-wa 1990 str. 355-361.
Progress in industrial microbiology vol. 32 1995 str. 1-35.
Soruvtseva E.G., Volnova A.J., Shatskaja T.Ye.: Degradacja monochlorozamieszciennych anilinow Alcaligenes faecalis. Mikrobiologiya (1980) 49, str. 351-354.
You I-S., Bartha R.: Stimulation of 3,4-dichloroaniline mineralization by aniline.Appl.Environ. Microbiol. Vol. 44 (1982) str. 678-681.
Advaances in Applied Microbiol . (1974) 75, str.75-114.
Libudzisz Z., Kowal K.: Mikrobiologia techniczna. T.2. Łódź 2000.str.151-152.
Schlegel H. G.: Mikrobiologia ogólna, PWN W-wa 1996. str. 534.
Ekologia. Jej związki z rżnymi dziedzinami wiedzy. Wybrane zagadnienia pod redakcją A Kurnatowskiej, PWN W-wa - Łódź 1997. str. 81.
Enzyme nomnclature. Academic Pres New York, San Francisco, London 1984.
Streyer L.: Biochemia, PWN W-wa 1997.
Filipowicz B., Więckowski W.: Biochemia, Tom 2, PWN W-wa-Łódź 1986.
Biochemia Harpera, PZWL W-wa 1994.
Kączkowski J.: Podstawy biochemii, WNT W-wa 1999. str.210-213.
Biochemistry of the Amino Acid, Academic Press New York-London 1965.
Voet D., Voet J.B.: Biochemistry, John Wiley and Sons 1995.
Kim Ch-H., Hollocher T.C.: N isotope studies on the pathway of ammonia asimilation in Bacills meganterium and Escherichia coli. J. Bacteriol. (1982) 151, str.358-366.
Erwin V.G., Hellerman L.:Mitochondrial monoamine oxidas. J. Biol. Chem. (1967) 242, str.242-249.
Eady R.R., Large P.J.: Microbial oxidation of amea. Biochem. J. (1971) 123, str. 757-771
Iwaki., Shimizu M., Takuyama T., Hasegawa Y.: Biodegradation of cyclohexylamine by Brevibacterim oxydans ICH-35A. Appl. Environ.Microbiol. (1999) 65, str.232-2234.
Decker R.H., Kang H.A., Leach F.R., Henderson l.M.: Purification and properties of 3-hydroxyanthranilic acid oxidase. J. Biol. Chem. (1961) 236, str.3076-3082.
Kastivela E., Wray V., Pieper D.H., Wittich R-M.: Initial reactions in the biodegradation of 1-chloro-4-nitrobenzene by newly isolated bacterium, strain LW 1. Appl. Environ. Microbiol. (1999) 65, str.1405-1412.
Wallnofer P., Engelhardt G.: Biotechnology vol. 6a, Biotrnsformations (1984), str.306.
Bordeleau L.M., Bartha R.: Biochemical transformation of herbicide-derived anilines: requirements of molecular configuration. Can. J. Microbiol. (1972) 18, str.1873-1882.
Hawari J., Halasz A., Paquet L., Zhou e., Spencer B., Ampleman G., Thibourt S.:Characterization of metabolites in the biotransformation of 2,4,6-trinitrotoluene with an aerobic sludge: role of triaminotoluene. Appl. Environ. Microbiol. (1998) 64, str.2200-2206.
Schenzle a., Lenke H., Fischer P., Williams P.A., Kmackmus H-J.: Catabolism of 3-nitrophenol by Ralstonia eutropa JMP 134. appl. Environ. Microbiol. (1997) 63, str.1421-1427.
Higler B.E., Spaain J.C.: Biodegradation of 4-nitroluene by Pseudomonas sp.strain 4NT. Appl. Environ. Microbiol. (1993) 59, str.2239-2243.
Fiorella P.D., Spain J.C.: Transformation of 2,4,6-trinitrotoluene by Pseudomonas pseudoalcaligenes JS 52. Apll. Environ. Microbol. (1997) 63, str.2007-2015.
Vorbeck c., Lenke H.,Fischer P., Spain J.C.,Knackmuss H-J.: Initial reductive reactions in microbial metabolism of 2,4,6-trinitrotoluene. Appl. Environ. Microbiol. (1998) 64, str.246-252.
French C.E., Nicklin S., Bruce N.C.: Aerobic degradation of 2,4,6-trinitrotoluene by Entherobacer cloacae PB 2 and by pentaerythritol tetrnitrate reductase. Appl. Environ. Microbiol. (1998) 64, str.2864-286).
Noguera D.R., Freedman D.L.:Reduction and acetylation of 2,4-dinitrotolue by a Pseudomonas aeruginosa strain. Appl. Environ. Microbiol. (1996) 62, str.2257-2263.
Kinbuchi T., Ohnishi Y.: Purification and characterization of 1-nitrpyrene nitroreductases from Bacteroides fragilis.Appl. Environ. Microbiol. (1983) 46, str.596-604.
Lenke H.,Knackmuss H-J.: Initial hydrogenaion during catabolism of picric acid by Rhodococcus erythropolis HL24-2. Appl. Environ. Microbiol. (1992) 58, str.2933-2937
Spiess T., Desiere F., Fischer P.,Spain J.C., Knackmuss H.J., Lenke H.: A new 4-nitrotoluene degradation pathway in a Mycobacterium strain. Appl. Environ.Microbiol. (1998) 64, str.446-452.
Brunh C., Lenke H., Knackmus H-J.:Nitrosubstituted aromatic compounds as nitrogen source for baceria. Appl. Environ. Microbial. (1987) 53, str.208-210.
Spanggord R.J., Spain J.C., Nishino S.F., Mortelmans K.E.: Biodegradation of 2,4-dinitotoluene by a Pseudomonas sp. Appl. Environ. Microbiol. (1991) 57, str.3200-3205.
Schackman A., Muller R.: Reduction of nitroaromatic compounds by different Pseudomonas species under aerobic cunditions. Appl. Microbiol. Biotechnol. (1991) 34, str.809-813.
Zeyer J., Kocher H.P.: Purification an characterization of a bacteriae nitrophenol oxygenase converts ortho-nitrophenol to ctechol and nitrite. J. Bacteriol. (1988) 170, str.1789-1794.
Gilcrease P.C., Murphy V.G.: Bioconversion of 2,4-diamino-6-nitrotoluene to a novel metabolite under anoxic and aerobic conditions. Appl. Environ. Microbiol. (1995) 61, str.4209-4214.
Nadeau L.J., Spain J.C.:Bacterial degradation of m-nitrobenzoic acid. Appl. Environ. Microbiol. (1995) 61, str.840-843.
Haigler B.E., Wallace W.H.: Biodegradation of 2-nitrotoluene by Pseudomonas sp. Strain JS 42. Appl. Environ. Microbiol. (1994) 60, str.3664-3469.
Spain J.C., Gibson D.T.:Patway for biodegradation of p-nitrophenol in Moraxella sp. Appl. Environ. Microbiol. (1991) 57, str.812-819.
Sander P., Wittich R.-M., Fortnagel P., wilkes h., France W.: Degradation of 1,2,4-trichloro and 1,2,4,5-tetrachlorobenzene by Pseudomonas strains. Appl. Environ. Microbiol. (1991) 57, str.1430-1440.
Haigler B., Spai J.C.: Biotransformtion of nitrobenzene by bacteria containing toluene degradaive pathways. Appl. Environ. Microbiol. (1991) 57, str.3156-3162.
He Z., Spain J.C.: Studies of catabolic pathway of degradation of nitrobenzene by Pseudomonas pseudoalcaligenes JS 45; removal of the amino group from 2-aminomuconic emialdehyde. Appl. Environ. Microbiol. (1997) 63, str.4839-4843.
Nishino S.F., Spain J.C.: Degradation of nitrobenzene by Pseudomonas pseudoalcaligenes. Appl. Environ. Microbiol. (1993) 59, str.2520-2525.
Nishino S.F., Spain J.C.: Oxidative pathway for the biodegradation of nitrobenzene by Comamonas sp. Srain JS 765. Appl. Environ. Microbiol. (1995) 61, str.2308-2313.
Harayama s., Rekik M., Ngai K.-L., Ornstos L.N.:Physically associated enzymes produce and metabolize 2-hydroxy-2,4-dienate, a chemicaly unstable intermediate formed in catechol metabolism via meta clevage in Pseudomonas putida. J. Bacteriol. (1989) 171, str.6251-6258.
Sala-Trepat J.M., Evans W.Ch.: The meta clevage of atechol by Azotobacter species 4-oxalocrotonate pathway .Eur. J. Biochem. (1971) 20 str.400-413.
Progrss in industrial microbiology vol.4 (1963), str.18-21.
Cain R.B.:Induction of antranilane oxidation system during the metabolism of ortho-nitrobenzoate certain bacteria. J.Gen. Microbiol. (1966),42 str.197-217.
Cain R.B.:Utilization of antranilic acid and nitrobezoic acid by Nocardia opaca and Flavobactrium.J. Gen. Microbiol. (966) 42, str.219-235.
Ke Y-H., Gee Z.L., Durham M.N.:Mechanism involved in the metabolism of nitrophenylcarboxylic acid compounds by microorganism. J. Bacteriol. (1959) 77, str.593-598.
Munnecke D.M., Hsich D.P.H.:Microbial decontamination of parathion and p-nitrophenol in aquenos media. Appl. Microbiol. (1974) 28, str.212-217.
Kadiyala V., Spain J.C.: a two-component mnooxygenase catalyzes both the ydroxylation of p-nitrophenol and the oxidative release of nitrite from 4-nitrocatechol in Bacillus sphaericus JS 906. Appl. Environ. Microbiol. (1998) 64, str.2479-2484.
Blasco R., Castillo T.:Light-dependent degradation of nitrphenols by the phototrophic bacterium Rhodobacter capsulatus E1F1. Appl. Environ. Microbiol. (1992) 58, str.690-695.
Sharmila M., Ramenand K., Sethunathan N.:Effect of yeas extract on the degradatin of organophosphorius by soil enrichment and bacterial cultures. Can.J.Micribiol. (1989) 35, str.1105-1110.
Siddaramappa R., Rajara K.P., Sethunathan N.:Degradation of parathion by bacterie isolated from Flooded soil. Appl. Microbiol. (1973) 26 str.846-849.
Munnecke D.M.:Enzymatic hydrolysis of organophosphate insecticides a possible pesticide disposal method. Appl. Environ. Microbiol. (1976) 32, str.7-13.
Jain R.K., Dreishach J.H., Spain J.C.:Biodegradation of 4-nitropenol via 1,2,4-benzenotriol by an Arthrobacter sp. Appl. Environ. Microbiol. (1994) 60, str.3030-3032.
Bruhn C., bayly R.C., Knackmuss H.-J.: The in vivo construction of 4-chloro-2-nitrophenol assimilatory bacterie. Arch. Microbiol. (1988) 150, str.171-177.
Delgado A., Wubbolts m.G., Abril M.-A., Ramos J.L.:Nitroaromatics are substrates for the TOL plasmid upper pathway enzymes. Appl. Environ. Microbiol.(1992) 58, str.415-417.
Robertson J.B., Spain J.C., Haddock J.D.,Gibson D.T.:Oxidation of nitrotoluenes by toluene dioxygaenase: evidence for monooxygenase reaction. Appl. Environ. Microbiol. (1992) 58, str.2643-2648.
Liu D., Thomson K.,Anderson.:Identification of nitroso compounds from biotransformation of 2,4-dinitrotolene. Apl. Environ. Microbiol. (1984) 47, str.1295-1298).
Valli K., Brock B.J. Joshi D.K., Gold M.H.;Degrading of 2,4 dinitrotolene by lignin-degrading fungus Phanerochate chrysosporium. Appl. Environ. Microbiol. (1992) 58, str.221-228.
Mc Cormic N.G., Cornell J.H., Kaplan A.M.:Identification of biotransformation products from 2,4-dinitrotoluene. Appl. Environ. Microbiol. (1978) 35, str. 945-948.
Mc Cormic N.G., Focherry F.E., Levinson H.S.:Microbial transformation of 2,4,6-trinirotoluene and other nitroaromatic compunds. Appl. Environ. Microbiol. (1976) 31, str.949-958.
Kaplan D.L., Kaplan A.M.:Thermophilic biotransformation of 2,4,6-trinitrotoluene under smulated composting conditions. Appl. Environ. Microbiol. (1982) 44, str.757-760.
Boopathy R., Kupla C.F., Wilson M.:Metabolism of 2,4,6-trinitrotoluene (TNT) by Desulfovibrio sp. (B.strain). Appl. Microbiol. Biotechnol. (1993) 39, str.270-275.
Kamiński L., Demsa D., Low E.: Biodegradation of 2,4,6-trinitrotoluene-contaminaed solis by two different aerated compost systems. Appl. Microbiol. Biotechnol. (1996) 44, str.795-800.
Vanderberg L.A., Perry J.J., Unkefer P.J.: Catabolism of 2,4,6-trinitrotoluene by Mycobacterium vaccae. Appl. Microbiol. Biotechnol. (1995) 43, str.937-945.
Anson J.G.,Mackinnon G.: Novel Pseudomonas plasmid involved in aniline degradation. Appl. Environ. Microbiol. (1984) 48, str.868-869.
Latorre J., Reineke W., Knackmus H.-J.:Microbial metabolism of chloroanilines:enhanced evolution by natural genetic exchange. Arch. Microbiol. (1984) 140, str.159-165.
Reber H., Hel V., Karanth N.G.K.: Comparative studies on the metabolism of aniline and chloroanilines by Pseudomonas multivorans strain An 1.Eur. J. Appl. Microbiol. (1979) 7, str.181-189.
Sandermann H., Werner Heller.,Hertkorn.,Hoque E.,Dietmar Pieper .,Winkler R.: A nev intermediate in the mineralization of 3,4-dichloroaniline by the white rot fungus Phanerochaete chrysosporium .Appl. Environ. Microbiol.(1998) 64 str.3305-3312.
21
Wyszukiwarka
Podobne podstrony:
Biodegradacja heterocyklicznych, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Biodegradacja halogenopochodnych, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Biodegradacja węglowodorów aromatycznych, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Główne szlaki biodegradacji związków aromatycznych, Naukowe PL, Biotechnologia, Enzymologia, Genetyk
związki aromatycznych, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Bakteriocyny i L-antybiityki, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Rośliny transgeniczne, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Lipazy, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
notatek pl , Biotechnologie, BAKTERIE LAB
Witaminy, Naukowe PL, Witaminy
Biotechnologia i inżynieria genetyczna
IZOL.DNA Z ŻELI, Biotechnologia notatki, Genetyka - biologia molekularna
fermentacja beztlenowa 2014-2015, Biotechnologia PŁ, Biotechnologia środowiska
Enzym 3 kolo, Szkoła Rolnictwo studia, Szkoła, Materiały studia, materialy - biotechnologia, Enzymol
wykład - Polimeraza DNA, Biotechnologia notatki, Genetyka - biologia molekularna
więcej podobnych podstron