
Analiza filogenetyczna
Jacek Dabert
Zakład Morfologii Zwierząt
dabert@amu.edu.pl

Z czego się uczyć
• Wiley i in. (1991). The Compleat Cladist.
• Lipscomb (1998). Basics of Cladistic Analysis.
• Felsenstein (2004). Inferring phylogeny.
• Hall (2008).
Łatwe drzewa filogenetyczne.
•
Wykłady ....

Tematyka wykładów
1. Drzewa filogenetyczne - podstawowe terminy stosowane w rekonstrukcji
filogenii
2. Algorytmy do konstrukcji drzew filogenetycznych
a.
metody dystansowe
b.
maksymalna parsymonia
c.
metody wiarygodnościowe (maximum likelihood, wnioskowanie
Bayesowskie)
3. Poszczególne etapy komputerowej analizy filogenetycznej danych
molekularnych
a.
przygotowanie matrycy danych
b.
przyrównanie (alignment)
c.
matryca danych
d.
parametry wejściowe (np. modele ewolucji sekwencji i cech
morfologicznych)
e.
analiza statystyczna rezultatów
f.
prezentacja wyników
4. Procedura total evidence
5. Współbieżne zdarzenia ewolucyjne (rekoncyliacja, parsymonia Brooksa,
jungle)
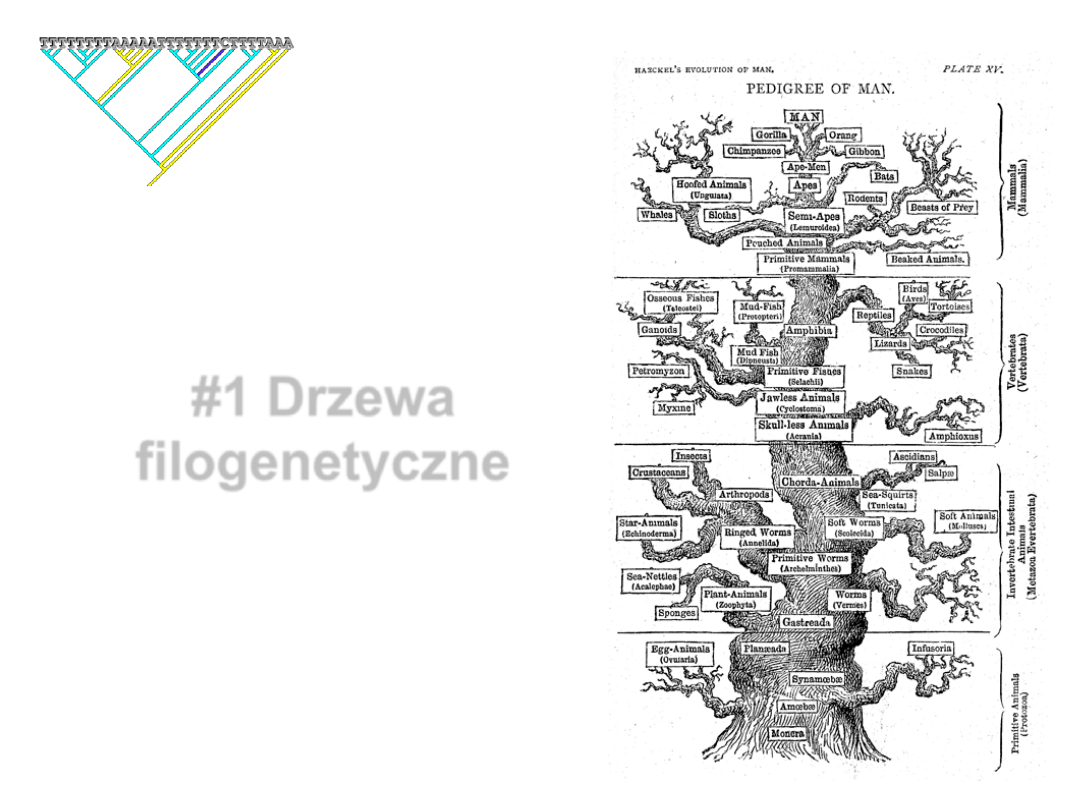
TTTTTTTT
AAAAA
TTTTTTT
C
TTTT
AAA
#1 Drzewa
filogenetyczne

Podstawowe pojęcia
Filogeneza (filogenia)
Powiązania historyczno-genealogiczne pomiędzy taksonami
Filogenetyka
(grecki phylon -
gałąź +genesis – pochodzenie)
Dziedzina wiedzy zajmująca się badaniem powiązań
ewolucyjnych pomiędzy gatunkami i rekonstruująca historię
ich dywersyfikacji od momentu powstania do chwili obecnej
Drzewo filogenetyczne
Graficzny obraz filogenezy badanych organizmów, genów i
innych OTU (operational taxonomic unit)

Liniowa koncepcja „filogenezy”
Arystoteles
(IV w. p.n.e)
Scala Naturae
(drabina jestestw naturalnych)
Jehovah
Christus
Angeli
Beati (święci)
Genius (duchy)
Quadrupes
Aves
Insecta
Vermes („robaki”)
Phytozoa (np. hydra)
Sensitiva (np.mimoza
Herbae
Gramina
Filices
Mucus
Litophyta (np.korale)
Praetiosa
Terra
wg Charles Bonnet, 1780 (skrócone)
wzrastająca
doskonałość

Teoria ewolucji Darwina
Tezy teorii Darwina
1.
Wszystkie gatunki
pochodzą od jednego lub
kilku wspólnych
przodków.
2.
Zasadniczym
mechanizmem ewolucji
jest dobór naturalny.
24.11.1859
„O powstawaniu gatunków w
drodze doboru naturalnego
albo o utrzymywaniu si
ę
doskonalszych ras w walce
o byt”
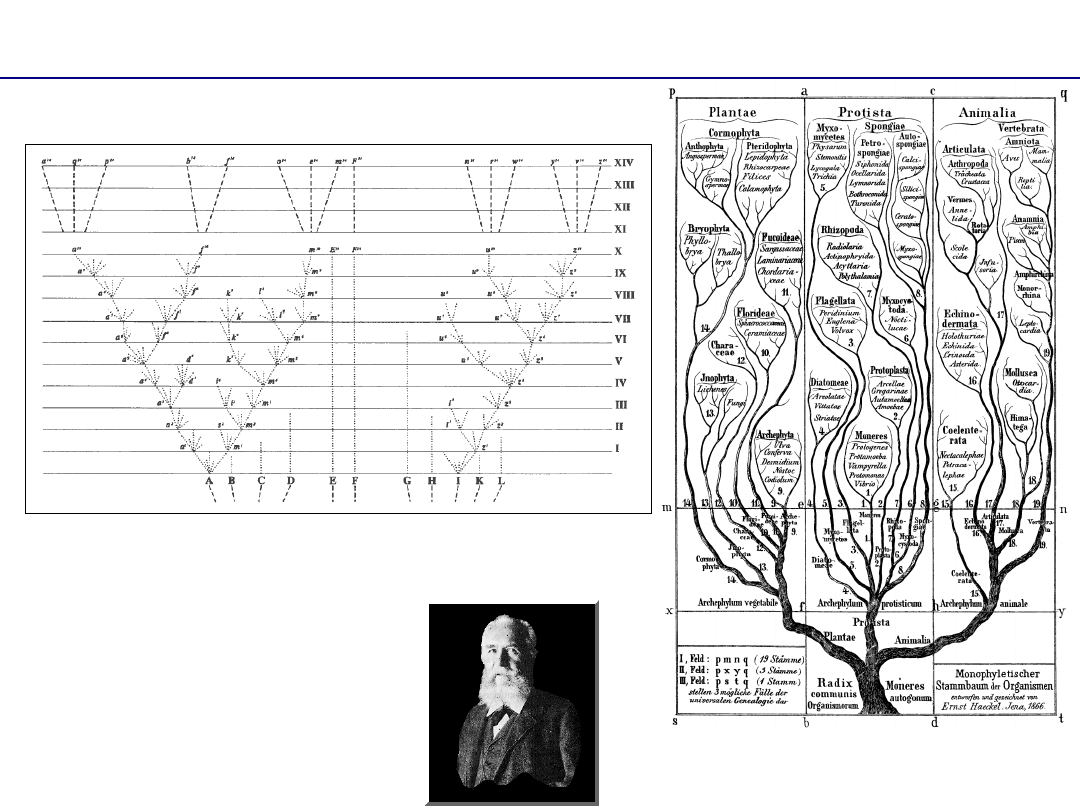
Współczesna koncepcja przebiegu filogenezy
Drzewo filogenetyczne
jest
hipotezą
Ernst Haeckel - Tree of life (1891)
Karol Darwin – Origin of Species (1859)

Historia filogenetyki (najkrótsza z możliwych)
Rozwój algorytmów
filogenetycznych
Robert Sokal (z prawej) i Peter Sneath
-
pierwsze podejście algorytmiczne –
taksonomia numeryczna (fenetyka)
bazująca na podobieństwie
morfologicznym
Luca Cavalli-
Sforza (powyżej) i Anthony Edwards
– ich studia filogenetyczne nad populacjami
ludzkimi bazujące na frekwencji alleli krwi
wprowadziły maksymalną parsymonię, metody
dystansowe i wiarygodnościowe

•
W aplikacjach biomedycznych np. epidemiologii i medycynie
sądowej
•
W badaniach molekularnych nad organizacj
ą genomu i
struktur
ą genów.
•
W badaniach nad powstawaniem nowych alleli i szczepów
laboratoryjnych
•
W studiach porównawczych w ekologii i etologii
•
W badaniu procesów fizjologicznych
•
Generalnie na wszystkich polach, w których dokonuje si
ę
porówna
ń między obiektami lub procesami.
•
Do rekonstrukcji historycznych zwi
ązków filogenetycznych
pomi
ędzy taksonami.
Gdzie stosuje się metodykę analizy filogenetycznej?
Zastosowania analizy filogenetycznej
tree house
baum haus
treow hus
tre hus
strom domovni
arbol casa
albero casa
arbore casa
arbre maison
arbor domus casa
dom na drzewie
Angielski
Niemiecki
Anglosaski
Norweski
Czeski
Polski
Hiszpański
Włoski
Rumuński
Francuski
Łacina
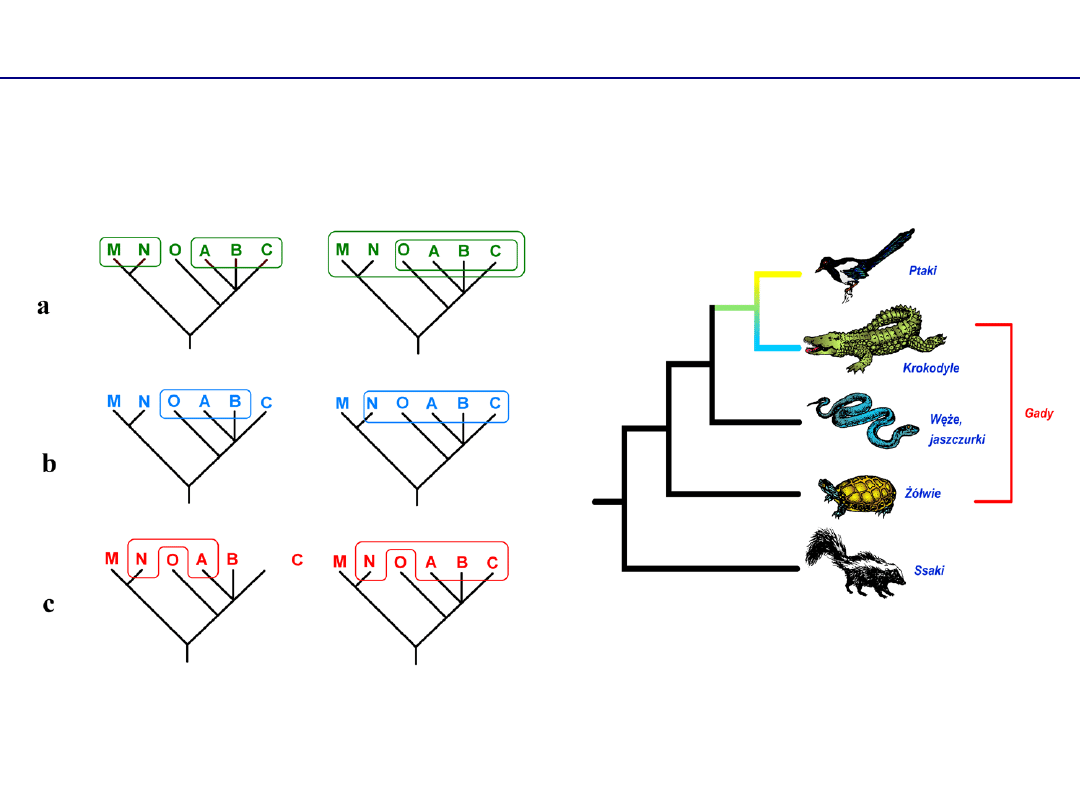
Taksony
naturalne
i
sztuczne
a.
grupa monofiletyczna
, b.
grupa parafiletyczna
, c.
grupa polifiletyczna
Systematyka filogenetyczna
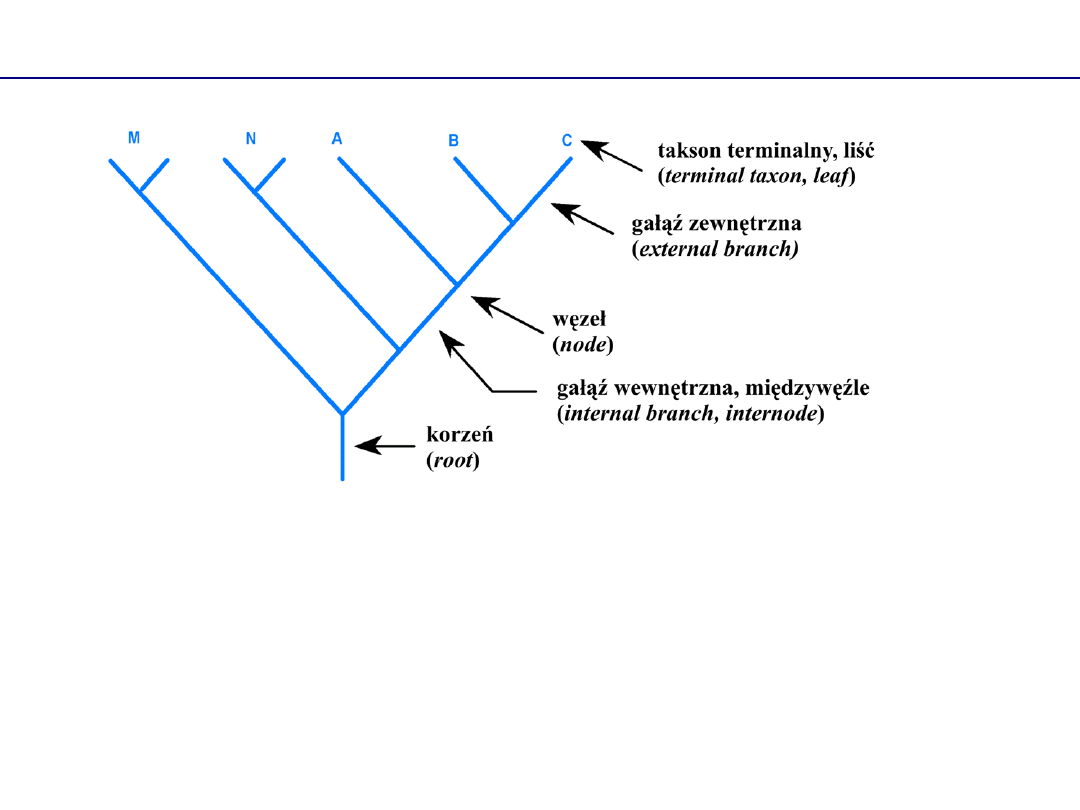
Terminologia dotycząca drzew filogenetycznych
Topologia drzewa
–
kształt drzewa, porządek rozgałęziania się pomiędzy
węzłami
Taksony terminalne
reprezentują współcześnie żyjące organizmy lub
analizowane fosylia,
węzły
natomiast reprezentują: (1) hipotetycznych
wymarłych przodków, (2) zjawiska specjacji, (3) klastry gatunków w danym
poddrzewie (kladzie, subtree)
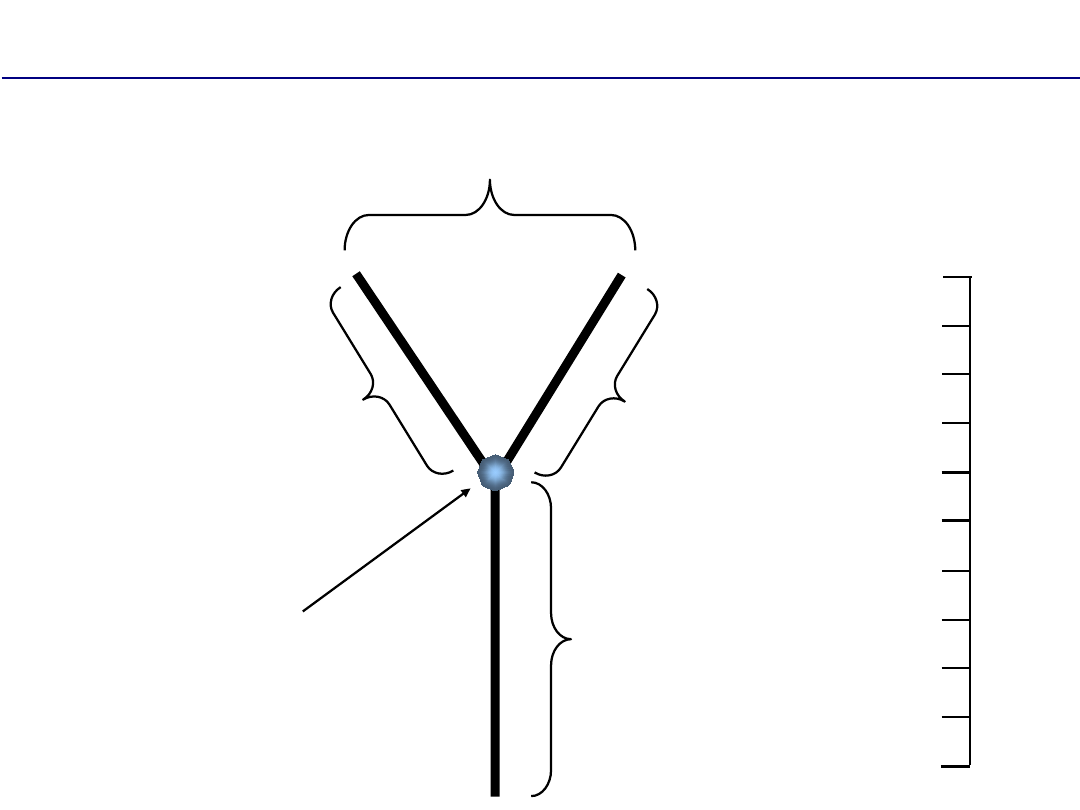
Ewolucyjna interpretacja drzewa filogenetycznego
kladogeneza
anageneza
gatunku A
anageneza
gatunku A’’
anageneza
gatunku A’
specjacja
czas
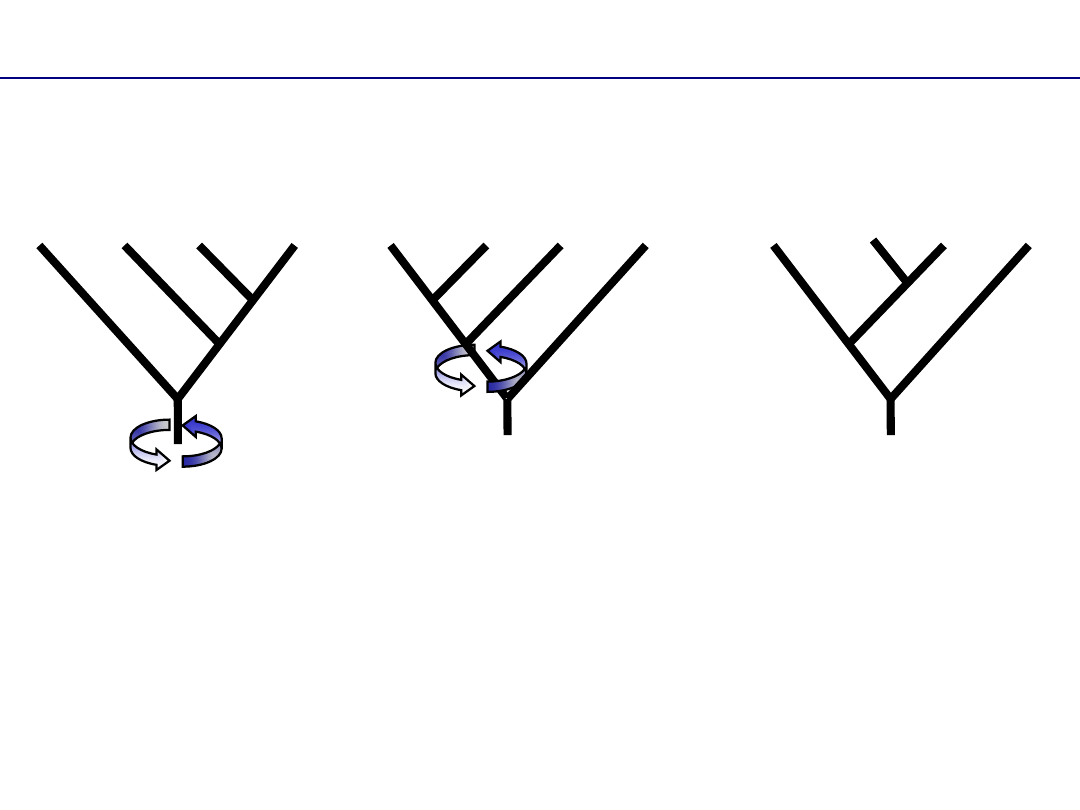
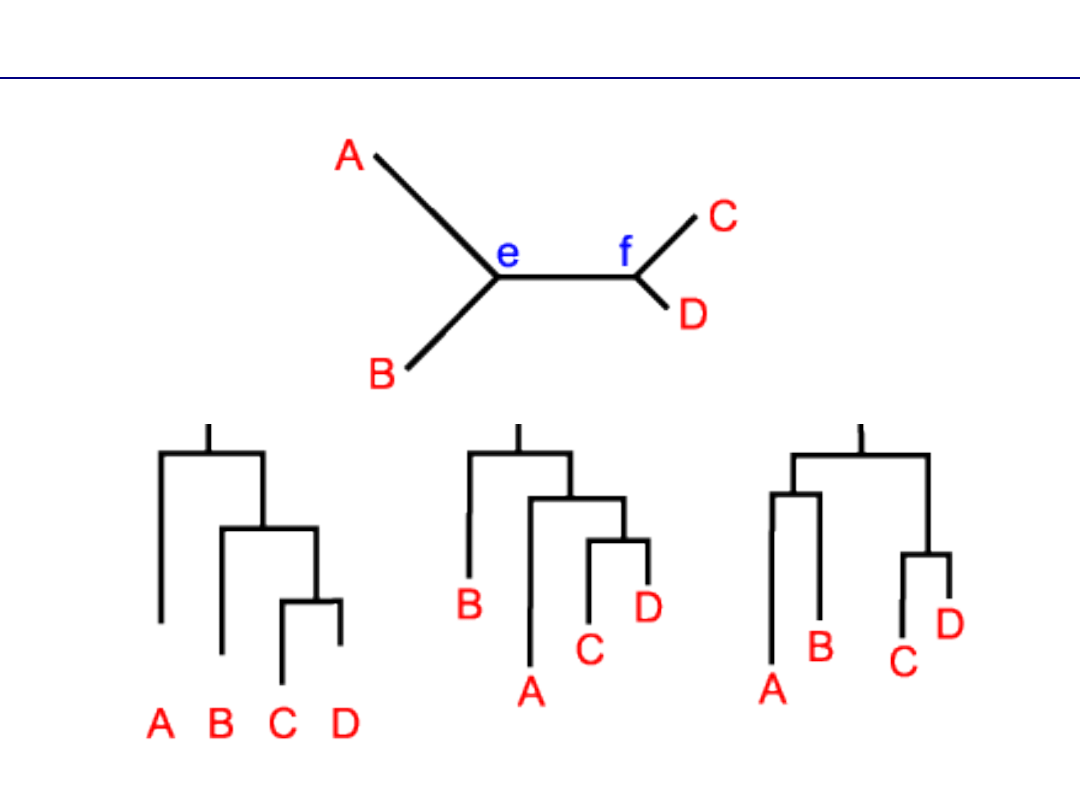
Układ gałęzi
Kolejność taksonów terminalnych nie ma
znaczenia –
każdą gałąź można „obracać”
A B C
D
A
B
C
D
=
A
B
C D
=
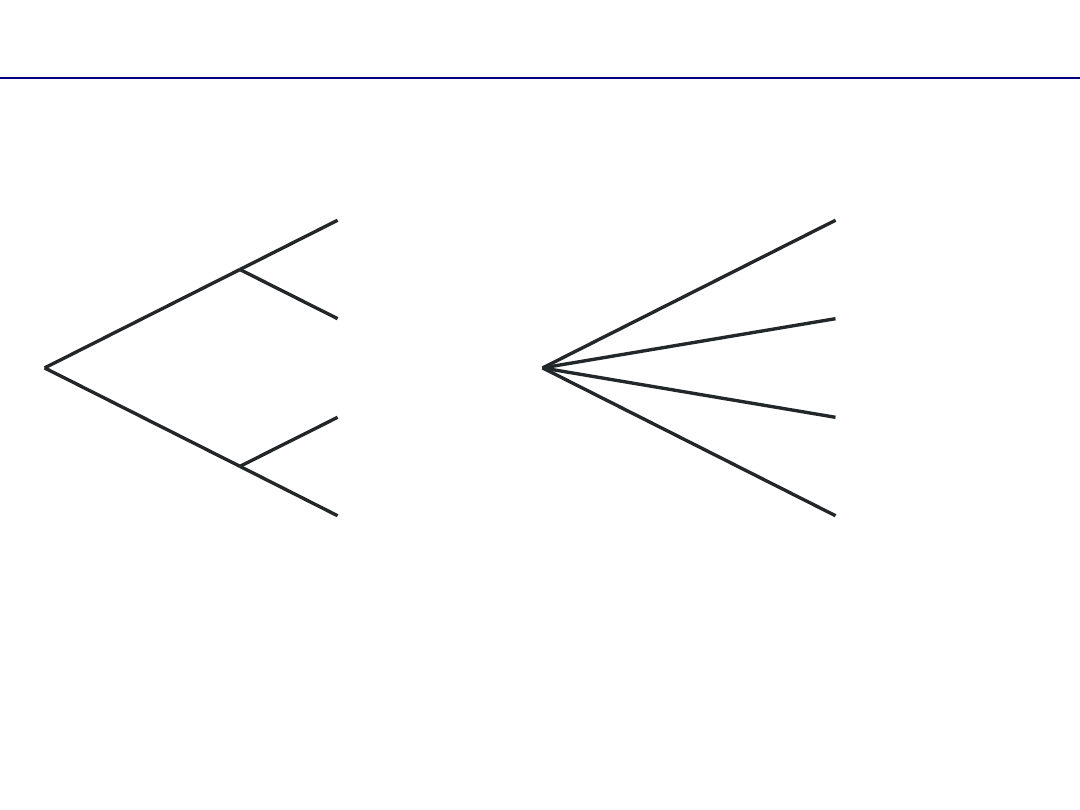
Politomia
Miękka politomia (soft polytomy)
–
niepewność co do powiązań
filogenetycznych lub kolejności dywergencji = brak dowodów powiązań, często
w wyniku niewystarczających danych
Twarda politomia (hard polytomy)
– jednoczesna „eksplozywna” dywergencja =
wszystkie potomne gatunki wyewoluowały w tym samym czasie (radiacja)
Jeden
Dwa
Trzy
Cztery
Jeden
Dwa
Trzy
Cztery
Drzewo dychotomiczne
–
w pełni rozwiązane
Drzewo politomiczne
–
brak rozwiązania

Politomia
Jeden
Dwa
Trzy
Cztery
Drzewo politomiczne
–
brak rozwiązania
Ile jest drzew dla n taksonów?
Dla dichotomicznych ukorzenionych drzew:
)
3
2
(
...
11
9
7
5
3
−
×
×
×
×
×
×
n
)!
2
(
2
)!
3
2
(
2
−
−
−
n
n
n
[1]
[2]
Dla dichotomicznych nieukorzenionych drzew:
)
5
2
(
...
11
9
7
5
3
−
×
×
×
×
×
×
n
Jest to ta sama formuła jak [2] tylko dla n-1
[3]
10 395 drzew
n=8
n=7
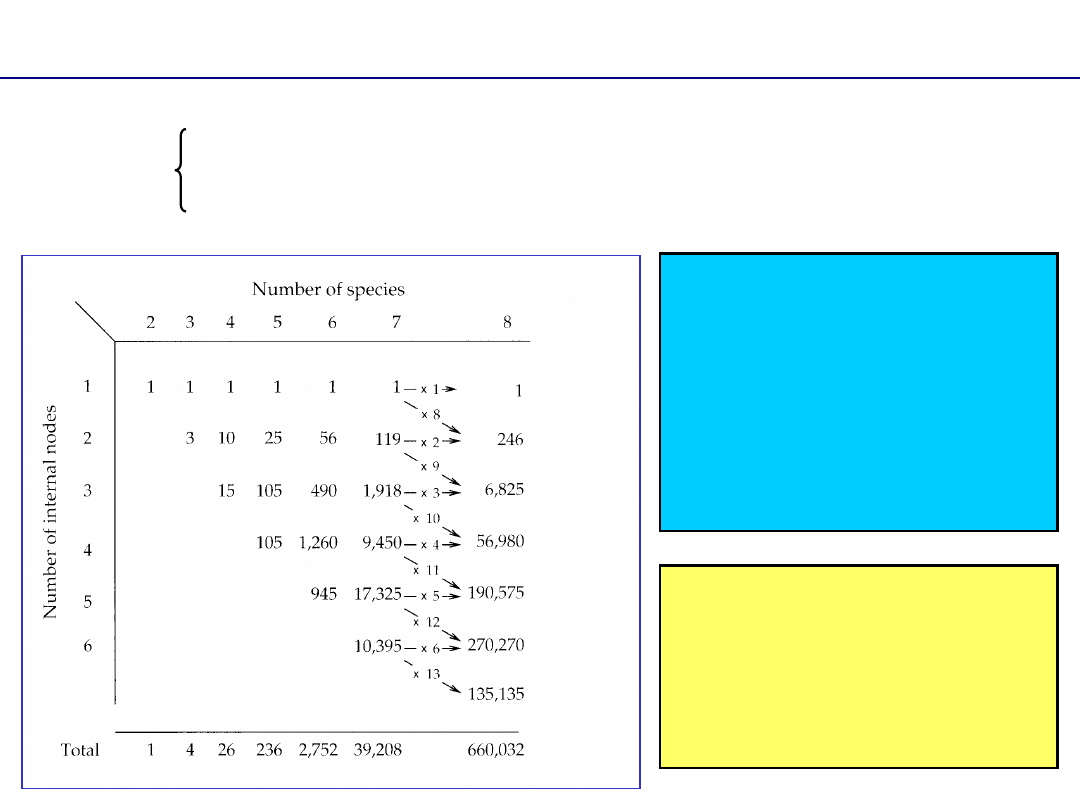
Ile jest drzew dla n taksonów?
Dla drzew, w których
dopuszczamy możliwość
politomii musimy jeszcze
uwzględnić liczbę węzłów
(m). Dla drzew
ukorzenionych posługujemy
się formułą Felsensteina.
Analogicznie jak dla
dichotomicznych
nieukorzenionych drzew
posługujemy się tą formułą
dla n-1
T
n,m
=
(n+m-2)T
n-1,m-1
+ mT
n-1,m
dla m>1
T
n-1,m
dla m=1
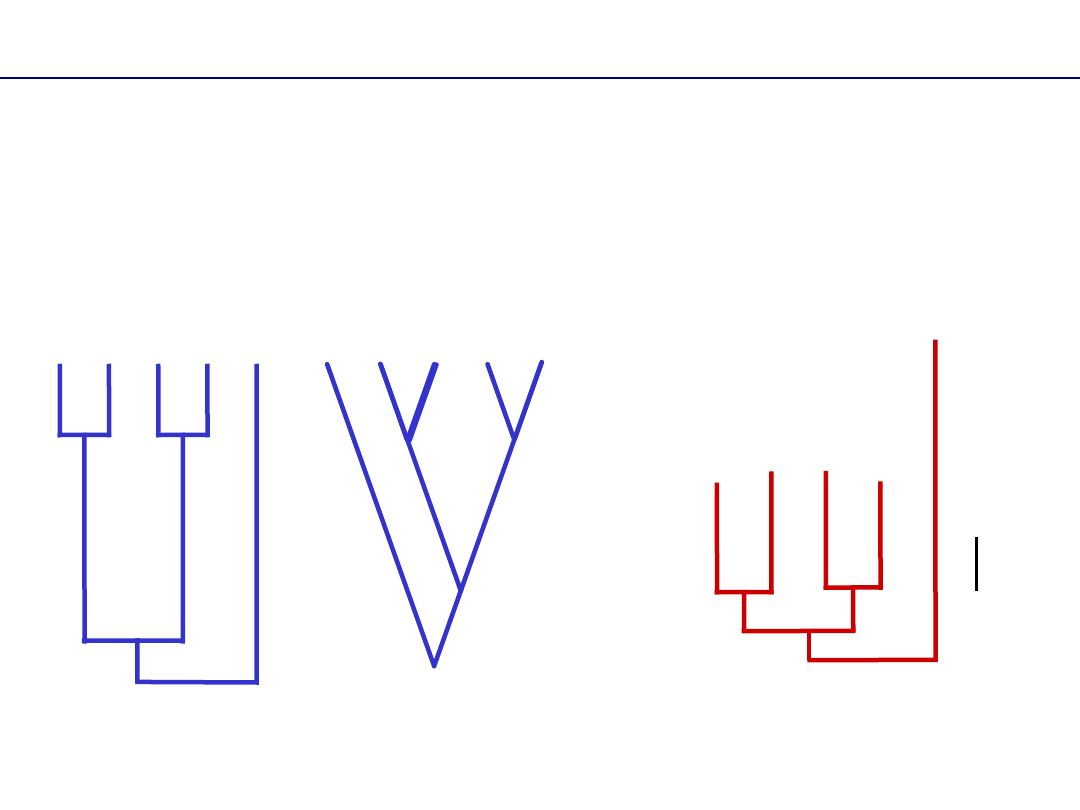
Rodzaje drzew
kladogram
(=topologia) –
długość gałęzi równa, nie jest
proporcjonalna do dystansu
ewolucyjnego = brak informacji o
tempie ewolucji
k. prostokątny k. ukośny
0.01
skala –
np. liczba substytucji/pozycję
filogram
–
długość gałęzi różna,
proporcjonalna do dystansu
ewolucyjnego = jest informacja
o tempie ewolucji
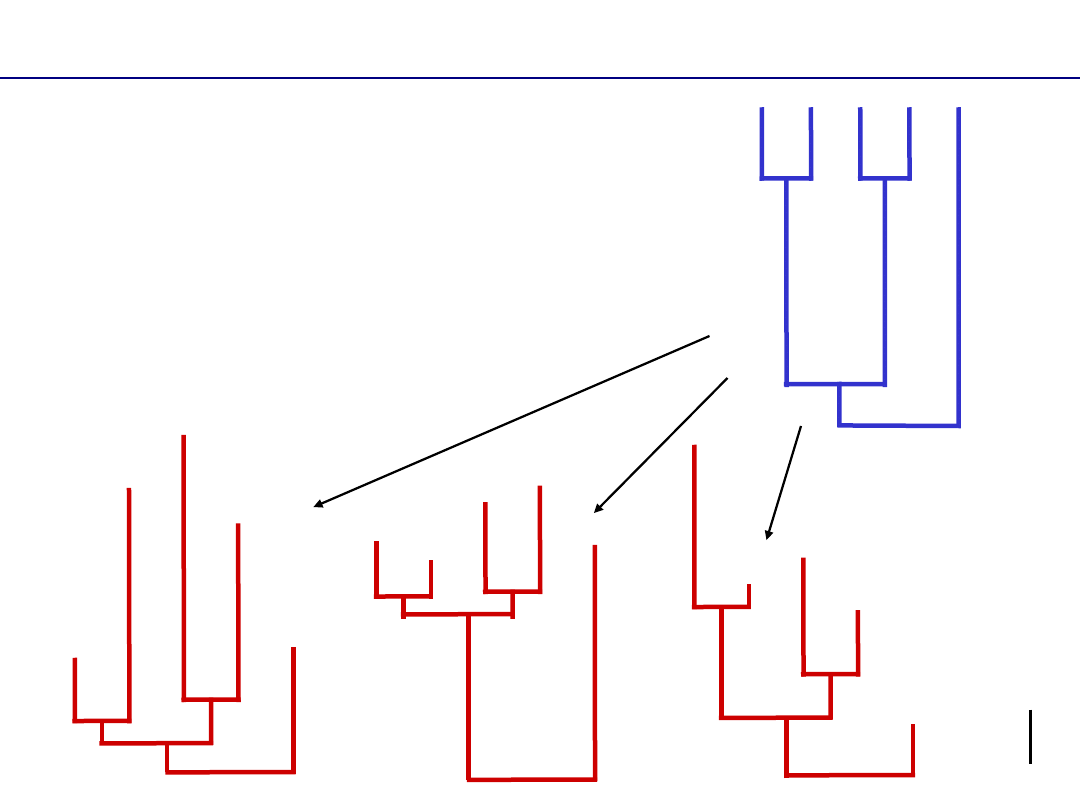
Rodzaje drzew
Ten sam kladogram może
reprezentować bardzo różne filogramy.
Filogramy te mają taką samą topologię,
ale różną wagę.
0.01
Rodzaje drzew
C
o
llo
h
N
o
th
ru
H
e
rma
n
N
e
h
y
p
o
M
al
a
co
P
lat
y
n
T
rh
yp
o
A
rc
heg
G
ym
no
d
Ere
ma
e
Ac
ro
ga
Sc
uto
v
Tec
toc
Xenill
Liodes
Adoris
Euzete
Nanher
Allon
o
Steg
a1
Ste
ga2
Ac
ar
us
R
hiz
og
C
ho
rto
C
ar
p
o
g
B
o
n
o
m
o
H
is
tio
A
n
a
lg
e
P
te
ro
n
O
to
d
e
c
C
h
o
ri
o
D
e
rma
t
Kn
e
m
id
A
v
en
z
o
T
ro
u
e
s
P
ro
ct
o
M
o
n
o
ju
G
eh
yp
o
Hy
po
c1
Hy
po
c2
Lo
hm
an
Me
rist
Micr
oc
Rhagid
Anysti
Torotr
Neuman
Megal
u
Labid
o
Tetr
an
Lin
op
o
Alic
or
C
ry
pto
C
h
an
b
r
E
u
si
m
o
E
u
k
o
e
n
O
lig
o
l
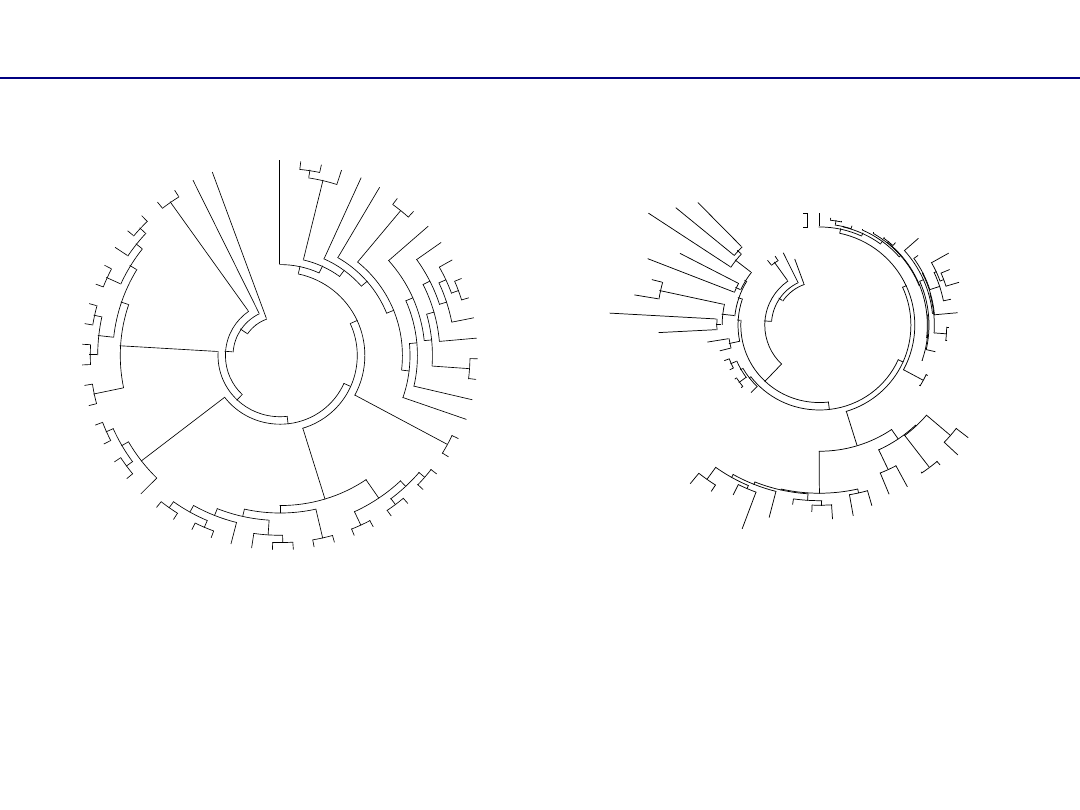
kladogram cyrkularny filogram cyrkularny
C
o
llo
h
N
o
th
ru
H
e
rma
n
N
e
h
y
p
o
M
al
a
co
P
lat
y
n
T
rh
yp
o
A
rc
heg
G
ym
no
d
Ere
ma
e
Ac
ro
ga
Sc
uto
v
Tec
toc
Xenill
Liodes
Adoris
Euzete
Nanher
Allon
o
Steg
a1
Ste
ga2
Ac
ar
us
R
hiz
og
C
ho
rto
C
ar
p
o
g
B
o
n
o
m
o
H
is
tio
A
n
a
lg
e
P
te
ro
n
O
to
d
e
c
C
h
o
ri
o
D
e
rma
t
Kn
e
m
id
A
v
en
zo
T
ro
u
e
s
P
ro
ct
o
M
o
n
o
ju
G
eh
yp
o
Hy
po
c1
Hy
po
c2
Lo
hm
an
Me
rist
Micr
oc
Rhagid
Anysti
Torotr
Neuman
Megal
u
Labid
o
Tetr
an
Lin
op
o
Alic
or
C
ry
pto
C
h
an
b
r
E
u
si
m
o
E
u
k
o
e
n
O
lig
o
l
0.01
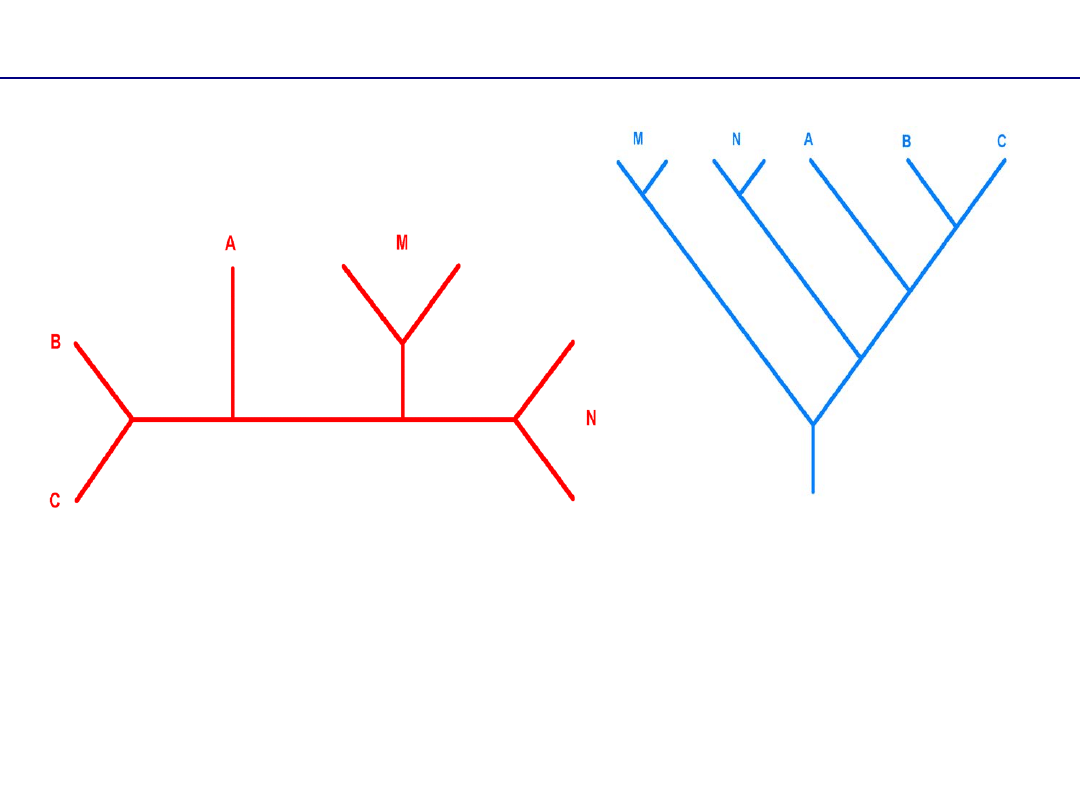
Drzewa ukorzenione vs. nieukorzenione
drzewo ukorzenione
- jest informacja o kierunku
ewolucji
Zarówno kladogramy jak i filogramy mogą być ukorzenione lub nie.
Przypadek ukorzenionego filogramu, w którym każdy takson terminalny
ma taką samą odległość od korzenia nazywamy
dendrogramem
drzewo nieukorzenione
- brak informacji o kierunku ewolucji
Ukorzenianie drzew
Ukorzenianie drzew
1. ukorzenianie za pomocą grupy zewnętrznej
Włączenie do analizy grupy znanej a priori jako grupa zewnętrzna do
badanych taksonów powoduje, że korzeń siłą rzeczy stanowi gałąź
łącząca grupę zewnętrzną z grupą wewnętrzną.
korzeń

Ukorzenianie drzew
2. ukorzenianie za pomocą zegara molekularnego
•
Koncepcja zegara molekularnego (Zuckerlandl i Pauling,
1965) postuluje równe tempo substytucji we wszystkich
liniach ewolucyjnych. Dzięki danym fosylnym możliwe jest
kalibrowanie zegara i określanie bezwzględnego czasu
dywergencji.
13
2
×
x
człowiek 13 mln lat orangutan
Jeżeli wszystkie linie ewolucyjne wykazują taką samą prędkość zmian
ewolucyjnych od czasu dywergencji od ich wspólnego przodka, to korzeń
znajduje się w punkcie równo odległym od wszystkich taksonów
terminalnych (liści)

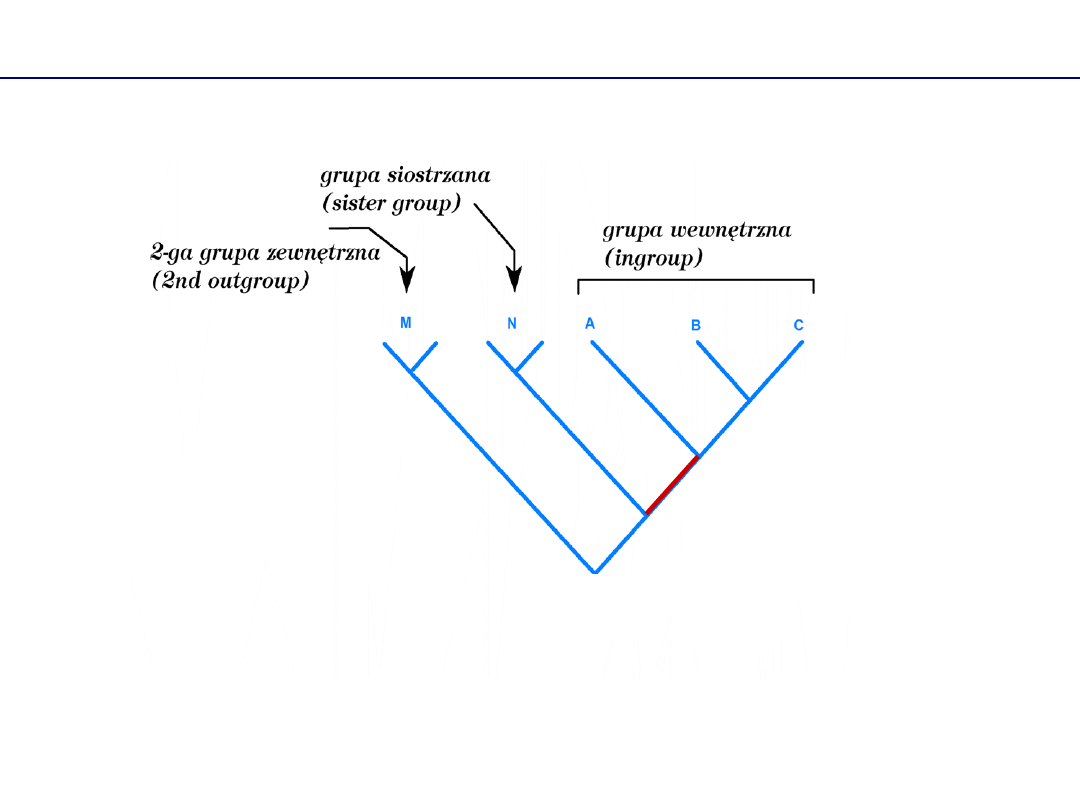
Określanie kierunku zmian ewolucyjnych - definiowanie cech ancestralnych
Grupa zewnętrzna
Grupa zewnętrzna (lub lepiej kilka grup zewnętrznych) z definicji muszą łączyć
się z naszą grupą wewnętrzną u jej nasady poniżej wspólnego przodka. Cechy,
które definiują grupę wewnętrzna i są odmienne od grupy zewnętrznej są
cechami nowymi (wg. kladystów apomorfiami). Cechy takie same jak u grupy
wewnętrznej są cechami ancestralnymi (plezjomorficznymi). Jest to obecnie
podstawowa metoda ustalania cech apomorficznych i plezjomorficznych dla
sekwencji DNA.
Fosylia
Jeśli mamy dobrze udokumentowane i zachowane dane fosylne (kopalne), co do
których jesteśmy pewni, że są ancestralne w stosunku do grupy wewnętrznej, to
rozumujemy analogicznie jak w przypadku grupy zewnętrznej.
Ontogeneza
Wczesne etapy rozwoju embrionalnego zdają się być podobne dla szerokiego
spektrum organizmów. Bardziej progresywne cechy, które definiują różne
taksony pojawiają się później w ontogenezie. Jeżeli jest to prawdą (nie zawsze),
to cechy ancestralne naszej grupy wewnętrznej powinny być tymi, które
pojawiają się na wczesnych etapach ontogenezy.
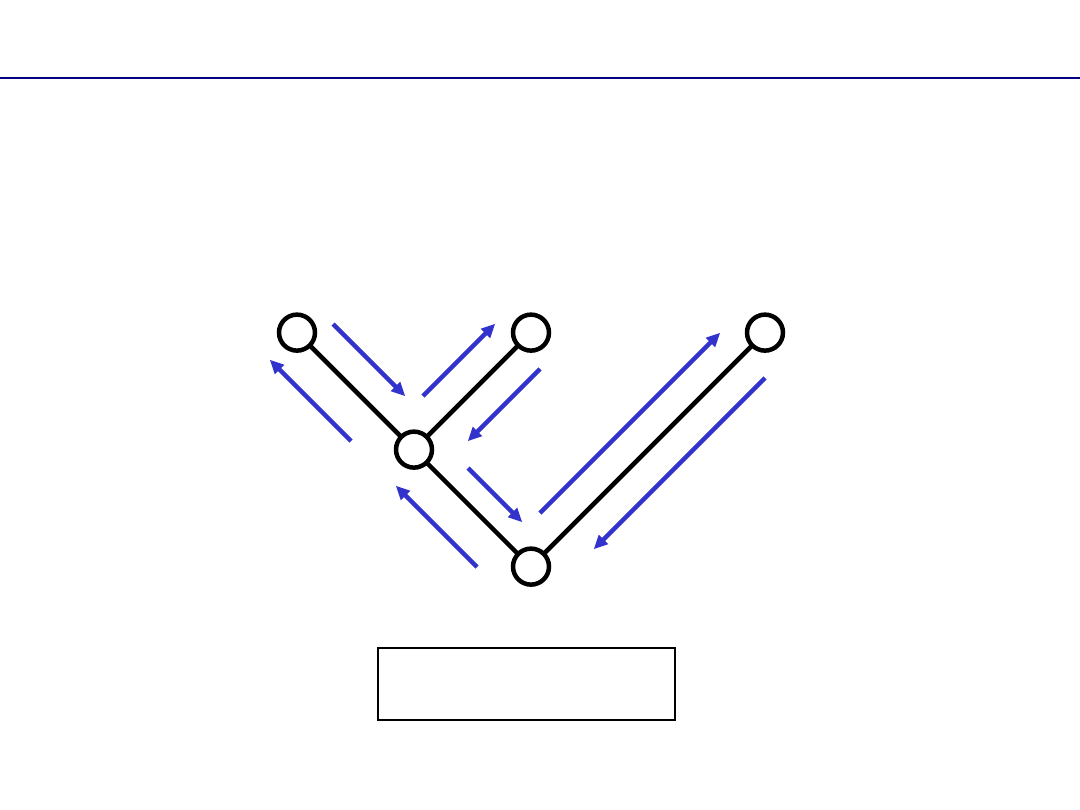
Konotacja tekstowa drzewa
,
)
(
,
)
(
( A
( , B
)
) , C
A
C
B
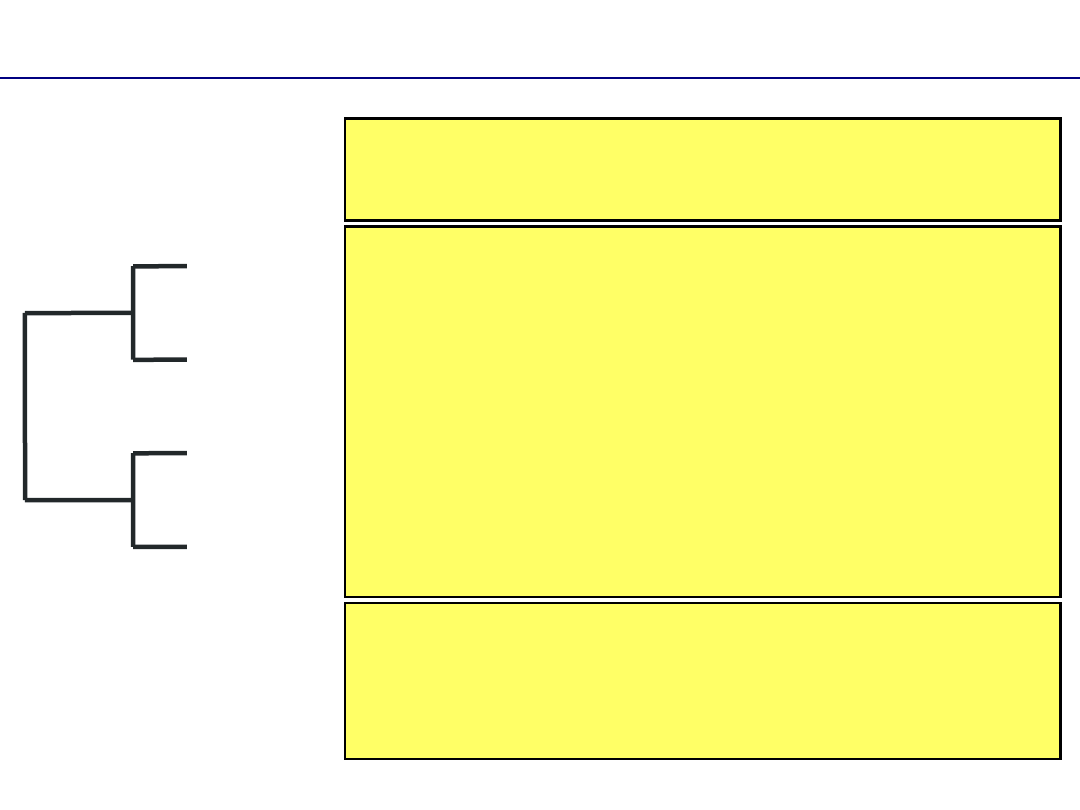
Formaty zapisu
Takson 1
Takson 2
Takson 3
Takson 4
Phylip/Newick
((Takson_1,Takson_2),(Takson_3,Takson_4));
Nexus
#NEXUS
BEGIN TREES;
TRANSLATE
1
Takson_1,
2
Takson_2,
3
Takson_3,
4
Takson_4
;
UTREE * PHYLIP_1= ((1,2),(3,4));
ENDBLOCK;
Hennig 86
tread
((0 1 )(2 3 ));
proc/;
kladogram
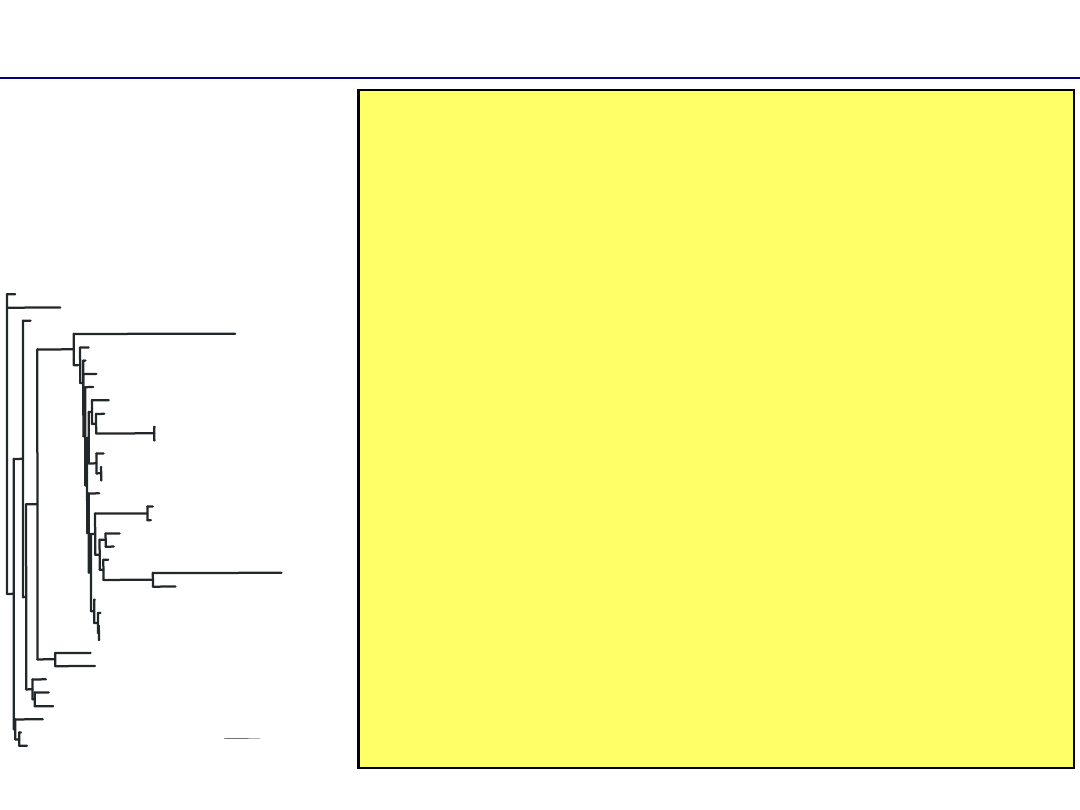
Formaty zapisu
((((((((((((((Hyd14_Lebe:0.022672,(Hyd11_Torr:0.
000518,Torrentico:0.001431):0.162006):0.010563,H
yd5_Front:0.046552):0.009222,(Hyd12_Limn:0.02006
4,(Hyd13_Limn:1.883e-008, Hyd6_Limne:0.001526)
:0.012873):0.021439):0.005219,(Hyd9_Mideo:0.0286
24,(((Hyd7_Hydra:0.016235,Hydrachna_:0.010107):0
.144794,((Hyd1_Neuma:0.039715,Neumania_d:0.02296
1):0.015615,((Hyd2_Piona:0.355877,Hyd15_Pion:0.0
61876):0.138478,Tiphys_sp:0.014139):0.008958):0.
013667):0.010696,(Horreolanu:0.001928,((Arrenuru
s_:0.001521,Hyd4_Arren:0.000746):0.003149,Hyd3_A
rren:0.008264):0.009890):0.008199):0.006716):0.0
05096):0.005425,Sperchon_s:0.023146):0.004681,Ch
imerathy:0.037079):0.000347,Thyas_barb:0.006470)
:0.008888,Hydryphant:0.024106):0.016748,Hyd6_Hyd
ro:0.446658):0.100423,(Limnochare:0.098468,Eylai
s_sp:0.111426):0.048096):0.032619,((Platythrom:0
.039664,Microtromb:0.051667):0.005902,Valgoperuv
:0.037742):0.018412):0.008891,Chyzeria_s:0.02251
4):0.024864,(Balaustium:0.076184,(Lasioeryth:0.0
04966,Erythraeus:0.021553):0.011638):0.004029):0
.018571,Sphaerotar:0.022422,Anystis_sp:0.147512)
;
0.1
Sphaerotar
Anystis sp
Chyzeria s
Hyd6 Hydro
Hydryphant
Thyas barb
Chimerathy
Sperchon s
Hyd5 Front
Hyd14 Lebe
Hyd11 Torr
Torrentico
Hyd12 Limn
Hyd13 Limn
Hyd6 Limne
Hyd9 Mideo
Hyd7 Hydra
Hydrachna
Hyd1 Neuma
Neumania d
Tiphys sp
Hyd2
Piona
Hyd15 Pion
Horreolanu
Hyd3 Arren
Arrenurus
Hyd4 Arren
Limnochare
Eylais sp
Valgoperuv
Platythrom
Microtromb
Balaustium
Lasioeryth
Erythraeus
filogram
((A:v
1
, B:v
1
):v
2
, C:v
4
, D:v
5
)
v –
długość gałęzi
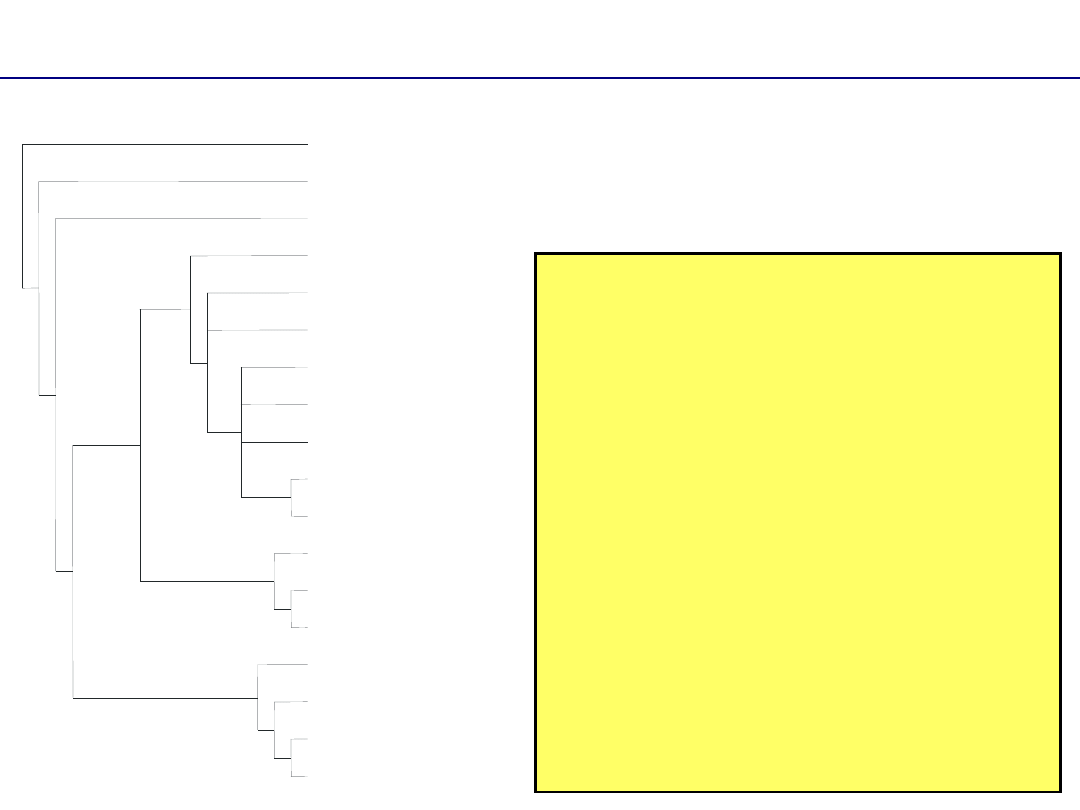
Formaty zapisu
Dodatkowe informacje
–
opis węzłów
(Sturnotrogus_subtrincatus,
(Scutulanyssus_dasyritidis,
(Pteronyssoides_holoplax,
((((M._glossifer, M._hordacei,
(M._plocei, M._capensis,
(M._puylaerti, M._anaplecti)
'd= 2', M._gaudi) 'd= 3') 'd=
1', M._bubalornis) 'd= 1',
(M._lonchurae, (M._angolensis,
M._amadinae) 'd= 1') 'd= 2')
'd= 1', ((M._plocepasseri,
(M._anoplonotus, M._daberti)
'd= 1') 'd= 1',
M._pseudonigritae) 'd= 1') 'd=
10') 'd= 1'));
PHYLIP_1
Sturnotrogus subtrincatus
Scutulanyssus dasyritidis
Pteronyssoides holoplax
M. bubalornis
M. glossifer
M. hordacei
M. plocei
M. capensis
M. gaudi
M. puylaerti
M. anaplecti
d= 2
d= 3
d= 1
d= 1
M. lonchurae
M. angolensis
M. amadinae
d= 1
d= 2
d= 1
M. pseudonigritae
M. plocepasseri
M. anoplonotus
M. daberti
d= 1
d= 1
d= 1
d= 10
d= 1
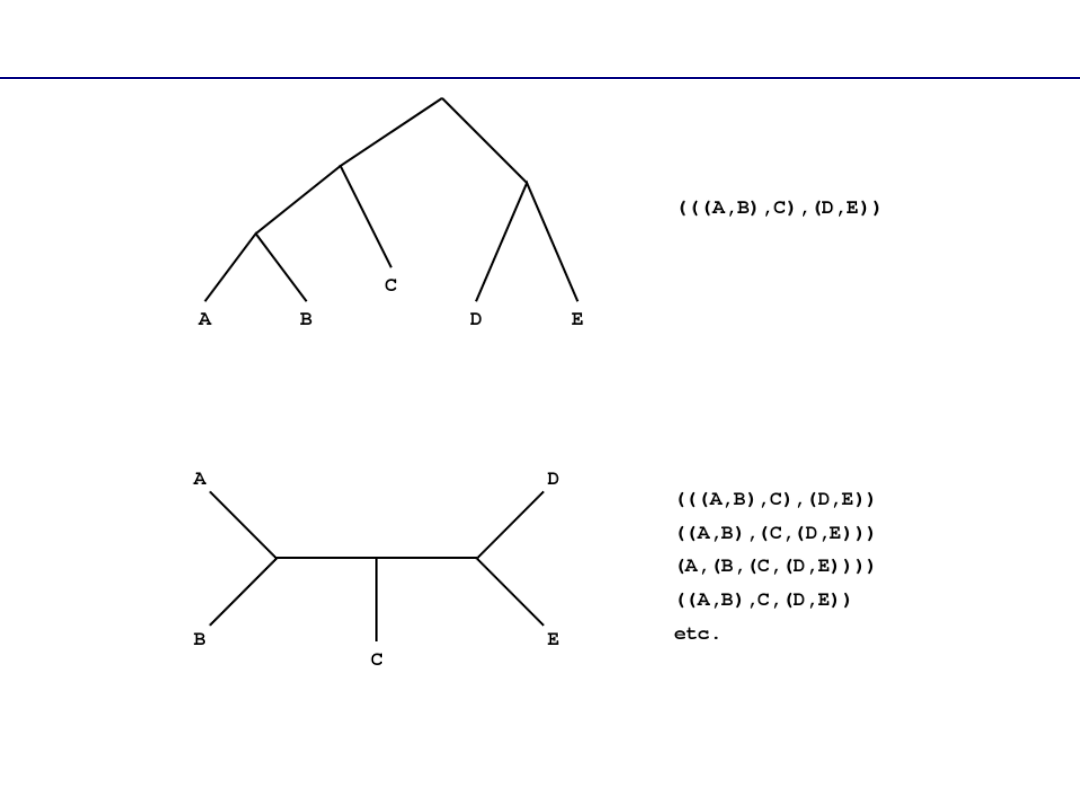
Formaty zapisu
Dla drzew nieukorzenionego dozwolone są różne zapisy tej samej
topologii
Zapis tekstowy zakłada drzewo ukorzenione (korzeń reprezentowany
przez skrajne nawiasy).

Kiedy drzewo filogenetyczne nie jest dobrym modelem przebiegu ewolucji?
Genealogia osobników w ramach gatunku
Dla gatunków rozmnażających się płciowo materiał genetyczny pochodzi po
połowie od każdego z rodziców, a nie od pojedynczego osobnika („wspólnego
przodka”
). Dla gatunków bezpłciowych drzewo jest odpowiednie dla analizy
genealogii, choć również tu może niekiedy zachodzić wymiana materiału
genetycznego (np. koniugacja) i drzewo jest nieodpowiednie.
Filogenia blisko spokrewnionych gatunków
W wyniku sporadycznych krzyżówek między osobnikami należącymi do ściśle
spokrewnionych gatunków może dochodzić do włączenia do ich puli genowej
materiału genetycznego obcego gatunku.
Gatunki hybrydowe (specjacja retikularna)
Szczególnie wśród roślin powstawanie nowych gatunków przez hybrydyzację
może być częstą drogą ewolucji.
Dalekie związki filogenetyczne
Istnieje wiele możliwości transferu materiału genetycznego pomiędzy daleko
spokrewnionymi lub obcymi gatunkami np. między bakteriami lub wirusami a
innymi organizmami.
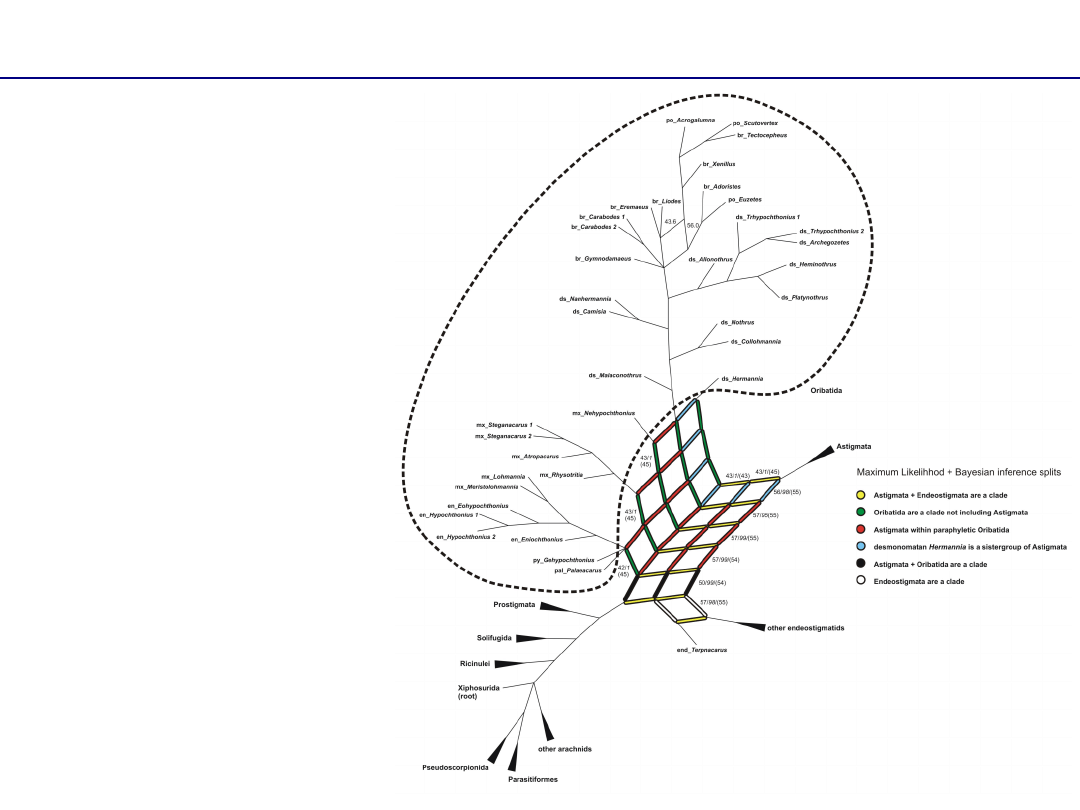
Alternatywy dla drzewa filogenetycznego
Sieć (network)
–
wygenerowana za pomocą
programu SplitsTree obrazuje
alternatywne hipotezy
filogenetyczne na jednym grafie
23
17
Alpha1
16
Alpha2
Alpha3
20
19
Beta3
18
Beta1
Beta2
22
Gamma1
21
Gamma2
Gamma3
25
24
Cyanob1
Cyanob2
26
Gonyaulax
27
Cyanophora
28
Red_brown
Green
ROOT
1
2
3
4
5
6
7
8
9
10
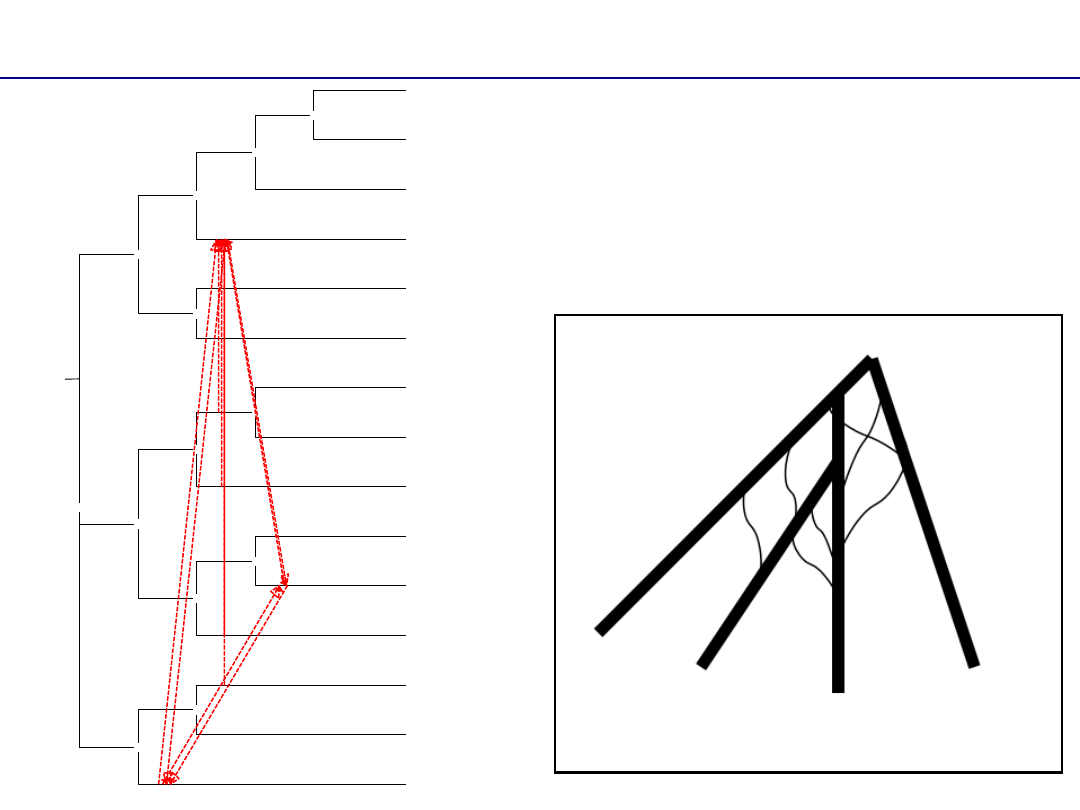
Alternatywy dla drzewa filogenetycznego
Retikulogram
– wygenerowany za
pomocą programu T-REX obrazuje
(czerwone strzałki) zjawiska
specjacji retikularnej (hybrydyzacja)
lub transferu horyzontalnego genów
Hipotetyczne inicjalne stadium specjacji
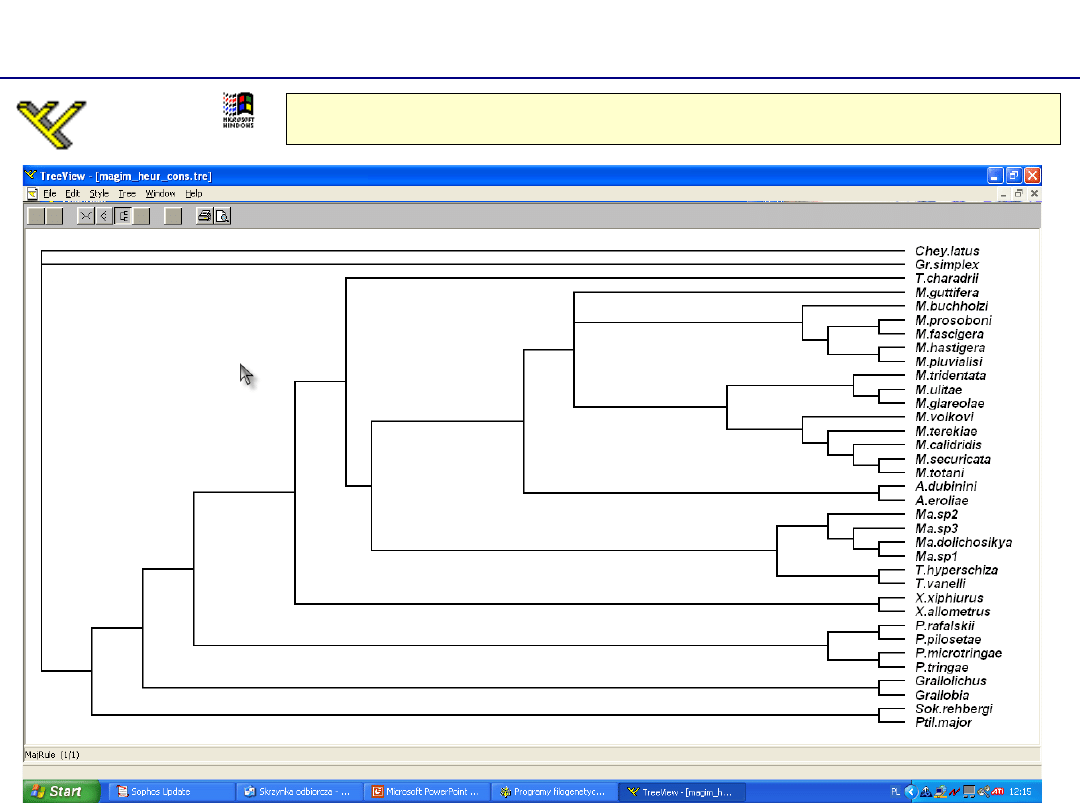
Graficzna prezentacja
TreeView
http://taxonomy.zoology.gla.ac.uk/rod/treeview.html
Graficzna prezentacja
WinClada
Graficzna prezentacja
TreeMe!
Graficzna prezentacja
Macclade
Document Outline
- Slajd numer 1
- Z czego się uczyć
- Tematyka wykładów
- Slajd numer 4
- Podstawowe pojęcia
- Liniowa koncepcja „filogenezy”
- Teoria ewolucji Darwina
- Współczesna koncepcja przebiegu filogenezy
- Historia filogenetyki (najkrótsza z możliwych)
- Slajd numer 10
- Zastosowania analizy filogenetycznej
- Taksony naturalne i sztuczne
- Terminologia dotycząca drzew filogenetycznych
- Ewolucyjna interpretacja drzewa filogenetycznego
- Układ gałęzi
- Politomia
- Politomia
- Ile jest drzew dla n taksonów?
- Ile jest drzew dla n taksonów?
- Rodzaje drzew
- Rodzaje drzew
- Rodzaje drzew
- Drzewa ukorzenione vs. nieukorzenione
- Ukorzenianie drzew
- Ukorzenianie drzew
- Ukorzenianie drzew
- Określanie kierunku zmian ewolucyjnych - definiowanie cech ancestralnych
- Konotacja tekstowa drzewa
- Formaty zapisu
- Formaty zapisu
- Formaty zapisu
- Formaty zapisu
- Kiedy drzewo filogenetyczne nie jest dobrym modelem przebiegu ewolucji?
- Alternatywy dla drzewa filogenetycznego
- Alternatywy dla drzewa filogenetycznego
- Graficzna prezentacja
- Graficzna prezentacja
- Graficzna prezentacja
- Graficzna prezentacja
Wyszukiwarka
Podobne podstrony:
Niven Larry Opowieści ze znanego kosmosu 01 Całkowe drzewa
01 Szczepanowska drzewa w mieście
TD 01
Ubytki,niepr,poch poł(16 01 2008)
01 E CELE PODSTAWYid 3061 ppt
01 Podstawy i technika
01 Pomoc i wsparcie rodziny patologicznej polski system pomocy ofiarom przemocy w rodzinieid 2637 p
zapotrzebowanie ustroju na skladniki odzywcze 12 01 2009 kurs dla pielegniarek (2)
01 Badania neurologicz 1id 2599 ppt
01 AiPP Wstep
ANALIZA 01
01 WPROWADZENIA
01 piątek
choroby trzustki i watroby 2008 2009 (01 12 2008)
syst tr 1 (2)TM 01 03)13
więcej podobnych podstron