
Wydział Chemiczny Politechniki Gdańskiej
Katedra Technologii Leków i Biochemii
Kultury tkankowe i komórkowe roślin i zwierząt
IZOLACJA DNA Z TYTONIU
Wstęp.
Niemal cała ilość informacji dotyczącej budowy organizmu roślinnego, jego
rozwoju i funkcjonowania zawarta jest w jądrze komórkowym i zakodowana w DNA.
Tylko niewielka część tej informacji znajduje się również w DNA organelli
(mitochondrialnym, a u roślin zielonych – także w chloroplastowym). Informacja ta
jest przekazywana do nowo powstających komórek w wyniku replikacji DNA w czasie
podziału komórek merystematycznych, a podczas rozmnażania roślin – komórkom
generatywnym i następnym pokoleniom.
Biorąc pod uwagę najmniejszą ilość DNA znajdującego się w komórce rośliny
kwiatowej (Arabidopsis thaliana – 0,14 pg, co odpowiada 125 x 10
6
par zasad),
przeciętną masę cząsteczkową polipeptydu (50 kDa) i zakładając, że cały DNA pełni
funkcję kodującą, wyliczono, że mógłby on zawierać ponad 18 000 genów. Ilość DNA
w komórkach innych roślin wielokrotnie przekracza zawartość DNA u Arabidopsis,
toteż teoretycznie liczba genów jest dla nich znacznie większa. W rzeczywistości
liczba kodowanych (i ulegających ekspresji) genów nawet w roślinach o genomie
100-krotnie większym niż genom Arabidopsis nie przekracza 20 000. Jest to
skutkiem występowania u roślin 1) licznych genów w wielokrotnych powtórzeniach
(kopiach), 2) długich tzw. powtarzalnych (ang. repetitive) sekwencji DNA, które nie
mają zdolności kodowania, a w dużych genomach roślinnych stanowią do 60%
całkowitego DNA, oraz 3) intronów (sekwencji niekodujących) w strukturze genu.
Ponadto u wielu roślin występuje zjawisko poliploidalności, polegające na
zwielokrotnieniu liczby chromosomów, a tym samym – ilości DNA, bez powiększania
asortymentu kodowanych białek.
1
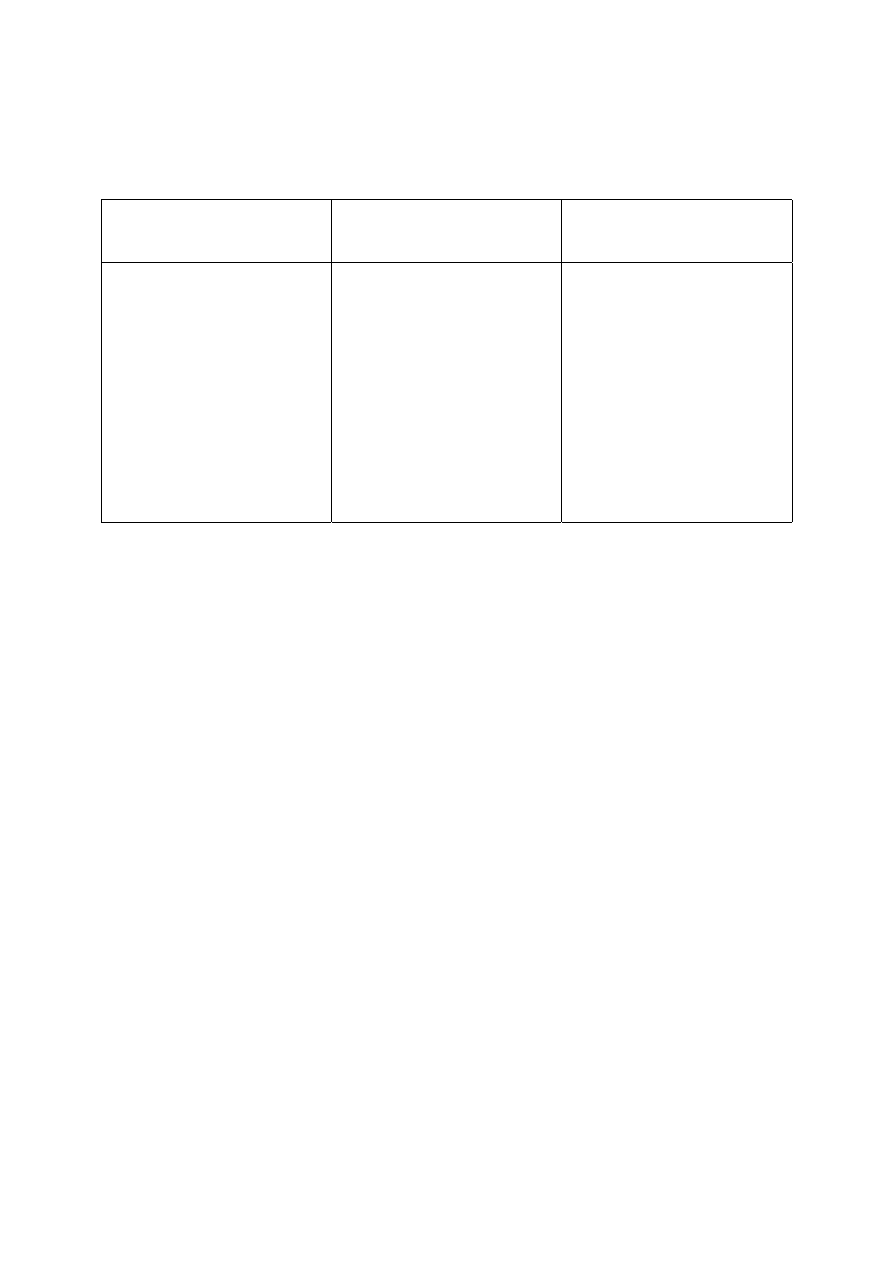
Tabela1. Wielkość genomu niektórych organizmów modelowych i roślin użytkowych,
które są przedmiotem badań molekularnych. W celu porównania zamieszczono
również wielkość genomu ludzkiego. Dane z początku 2001 roku.
Organizm
Przybliżona wielkość
genomu
(w milionach par zasad)
Zaawansowanie
sekwencjonowania
genomu
Wirusy
Bakterie
Drożdże – S. Cerevisiae
Nicień – C. Elegans
Arabidopsis thaliana
Drosophila melanogaster
Ryż
Kukurydza
Homo sapiens
Pszenica
0,3
4
12
100
125
200
700
2500
5000
15000
wiele zakończone
wiele zakończone
zakończone
zakończone
niemal zakończone
zakończone
częściowe
częściowe
zakończone
częściowe
Organizm modelowy to taki, który badany jest przez wielu naukowców ze względu
na jego cechy, które czynią go łatwym obiektem badań oraz jest on na tyle podobny
do innych organizmów, że wnioski wyciągnięte z jego badań mogą być szeroko
zastosowane. Kilka gatunków roślin było intensywnie badanych i z czasem stało się
układami modelowymi; najważniejszym jest ostatnio Arabidopsis thaliana, a inne
szeroko badane to kukurydza i ryż.
Arabidopsis thaliana (rzodkiewnik pospolity, arabidopsis) należy do rodziny
kapustowatych (Brassicaceae), jest małym i raczej niezbyt ważnym chwastem
rolnicznym. Ma wiele istotnych cech, które czynią go odpowiednim organizmem
modelowym do badania roślin:
• Małe rozmiary genomu, 120 000 kpz (w porównaniu z ryżem – 450 000 kpz,
czy z haploidalnym genomem kukurydzy – 4 500 000 kpz), umożliwiły
praktycznie całkowite zsekwencjonowanie genomu (Arabidipsis Genome
Project – AGP).
• Niewielkie rozmiary. Dużą liczbę roślin można łatwo hodować w komorach
hodowlanych lub szklarniach.
• Krótki cykl życiowy. Od wykiełkowania do wytworzenia nowych nasion mija
6 – 8 tygodni.
2

• Małe nasiona, ważące 20 µg każde, około 20 000 z każdej rośliny. Nasiona
można łatwo przechowywać i łatwo też uzyskiwać dużą liczbę roślin.
• Łatwo jest indukować mutacje (poddawać mutagenezie) za pomocą
czynników chemicznych lub promieniowania jonizującego.
• Łatwy obiekt do stosowania technik biologii molekularnej.
Totipotencja – w żywej komórce roślinnej somatycznej rośliny zawarta jest całość
informacji genetycznej. Jednak różne komórki i tkanki zawierają różne białka. Inaczej
mówiąc, różnice między komórkami (i tkankami) polegają na tym, że ulegają w nich
ekspresji różne zespoły genów. Dotyczy to jednak niewielkiej części materiału
genetycznego, genów kodujących białka organospecyficzne. Większość białek jest
identyczna we wszystkich organach rośliny. Na kilkanaście tysięcy różnych białek
syntetyzowanych w komórce roślinnej tylko kilkaset występuje wyłącznie w
komórkach wyspecjalizowanych organów i tkanek. Pełnią tam one rolę strukturalną,
katalityczną i regulacyjną, właściwą tylko dla tych organów i tkanek.
Genom chloroplastowy.
Plastydy mają pewien stopień autonomii genetycznej związanej z obecnością DNA w
tych organellach. DNA chloroplastowy wykazuje wiele cech wspólnych z materiałem
genetycznym organizmów prokariotycznych. Jego cząsteczka ma kształt kolisty i nie
zawiera białek histonowych. Pojedynczy chloroplast zawiera 200 – 300 identycznych
kopii DNA, które występują w grupach liczących od 5 do 20 cząsteczek, zazwyczaj w
połączeniu z wewnętrzną błoną osłonki lub błonami tylakoidów. Poznano całkowitą
sekwencję nukleotydów kolistego DNA chloroplastowego niektórych gatunków roślin
(tytoń, ryż) i wyjaśniono funkcję większości występujących tam genów. Oprócz
genów niektórych białek chloroplastowych, DNA plastydowy zawiera także geny
kodujące wszystkie rodzaje RNA chloroplastowego (mRNA, tRNA, rRNA)
uczestniczące w procesie translacji. Pojemność kodująca chloroplastowego DNA jest
stosunkowo niewielka. Waha się ona u roślin lądowych w zakresie 120 – 160 tys. par
zasad, ale w niektórych przypadkach liczba ta może przekraczać 200 tys. par zasad.
DNA chloroplastowy zawiera prawie zawsze obszar liczący ok. 22 – 26 tys. par
zasad, w którym część genów jest powtórzona (ang. inverted repeat), jednak
większość genów w cząsteczce występuje pojedynczo. Szacuje się, że genom
3

plastydowy zawiera geny dla około 120 białek, co stanowi niewielką część wszystkich
białek występujących w chloroplastach, zaś pozostałe białka kodowane są przez
genom jądrowy. Wiele białek chloroplastowych, jak np. karboksylaza
1,5-bisfosforybulozy, kluczowy enzym w procesie asymilacji CO
2
, powstaje w wyniku
współdziałania genomu plastydowego i jądrowego.
Genom mitochondrialny.
W macierzy mitochondrialnej roślin występuje wiele kopii kolistego DNA oraz
rybosomów charakterystycznych dla organizmów prokariotycznych (o stałej
sedymentacji 70S). Mitochondrialny DNA (mtDNA) koduje u roślin rRNA dla obu
podjednostek tych rybosomów, wszystkie tRNA biorące udział w biosyntezie białek
mitochondrialnych oraz niektóre białka mitochondrialne.
Wykonanie ćwiczenia.
Celem ćwiczenia jest izolacja DNA z liści tytoniu pochodzącego z hodowli in vitro.
Materiały i sprzęt
Zestaw do izolacji DNA z roślin Genomic Mini AX Plant ( A&A Biotechnology,
Gdańsk).
Przygotować na 1 podgrupę:
liście
tytoniu
50
mg
ciekły azot
10 ml
70% EtOH
2 ml
bufor TE (10mM Tris-zasada, 1mM EDTA, pH 8)
100
µl
probówki eppendorfa
probówki 15 ml z kolumną
pipety automatyczne
moździerz
łopatka metalowa
wirówka eppendorfa 12 – 14 000 obr./min.
łaźnia wodna na 50
o
C
worteks
4

Odczynniki do elektroforezy agarozowej:
1%
agaroza
0.35
g
agarozy/35
ml
TBE
1x
bufor TBEx5 :
(0,045 M Tris-zasada, 0,045 M kwas borowy, 0,001 M EDTA, pH 8)
50 ml
bromek etydyny 0,5
µg/ml
bufor obciążający LB (50% glicerol, 0,25% błękit bromofenolowy, 1 mM EDTA, pH 8)
marker wielkości DNA
bufor TE
cylinder miarowy 0,5 l i 100 ml
kolba Erlenmeyer’a 25 ml
aparat do elekroforezy poziomej
zasilacz do elektroforezy
naczynie do wybarwienia żelu
transiluminator UV
Izolacja DNA.
1. Około 50 mg świeżej masy roślinnej (liści tytoniu) umieścić w probówce
eppendorfa i wstawić probówkę do lodu.
2. Następnie tkankę roślinną przenieść do moździerza i ucierać energicznie i
szybko z 10 ml ciekłego azotu.
3. Przenieść utartą masę roślinną z powrotem do eppendorfa i dodać 0.9 ml
zawiesiny lizującej LS oraz 20 µl roztworu Proteazy.
4. Całość wymieszać i inkubować w 50
o
C przez 30 minut. Próbkę należy od
czasu do czasu mieszać.
5. Po inkubacji próbkę intensywnie worteksować przez 15 sek. A następnie
wirować 5 minut przy 14 000 obr./min.
6. Po wirowaniu pobrać supernatant i nanieść go na kolumnę umieszczoną w
probówce 15 ml. Poczekać aż lizat przejdzie przez kolumnę.
7. Następnie dwukrotnie przepłukać kolumnę poprzez dodanie 1.5 ml roztworu
płuczącego K2. Za każdym razem poczekać aż roztwór wypłynie z kolumny.
8. Dodać do kolumny 0.25 ml roztworu elucyjnego K3 i poczekać aż wypłynie z
kolumny.
9. Przenieść kolumnę do nowej probówki eppendorfa 2 ml, która posiada
odpowiednie żeberka pozwalające na łatwe umieszczenie w probówce i
eluować DNA przez dodanie do kolumny 1 ml roztworu elucyjnego K3.
10. Do eluatu zawierającego DNA dodać 0.8 ml mieszaniny precypitacyjnej PM.
Całość wymieszać i wirować 10 minut przy 12 000 obr./min.
11.
Po odwirowaniu zlać supernatant (na dnie powinien być widoczny
jasnoniebieski osad DNA) o dodać do probówki 0.5 ml 70% EtOH. Próbkę
wymieszać i wirować 3 minuty przy 12 000 obr./min.
12. Po wirowaniu zlać supernatant i odessać za pomocą pipety resztki etanolu.
Następnie próbkę suszyć pod wyciągiem około 10 minut.
13. Osad zawiesić w 20
µl buforu TE, inkubować przez 5 minut w 50
o
C, schłodzić
i przechowywać w zamrażarce.
5

Rozdział elektroforetyczny wyizolowanego DNA.
1. Próbkę z DNA wyjąć z zamrażarki i pozostawić w temperaturze pokojowej.
2. 1% żel agarozowy umieścić w aparacie do elektroforezy pionowej i wlać
odpowiednią ilość buforu TBEx1. W tym celu bufor TBEx5 rozcieńczyć: 100 ml
TBEx5 i 400 ml wody redestylowanej.
3. Sprawdzić czy studzienki w żelu są prawidłowo utworzone.
4. Do próbek z DNA dodać 5 µl buforu obciążającego LB i próbki zwirować przez
1 minutę przy 5 tys. obr./min.
5. Nanieść po 25 µl zawartości każdej próbki do studzienki oraz po 25 µl
markera wielkości DNA.
6. Rozdział DNA przeprowadzić przy stałym napięciu 50 V przez 3 godziny.
7. Żel wybarwić bromkiem etydyny (100
µl/200 ml wody redestylowanej) przez ½
godziny, przepłukać dwukrotnie wodą destylowaną i oglądać na
transiluminatorze UV.
Przygotowanie sprawozdania.
1. Opisać procedurę izolacji DNA z komórki roślinnej i wyjaśnić cel każdego
etapu i rolę każdego czynnika.
2. Porównać genom mitochondrialny komórki roślinnej i zwierzęcej.
3. Podać przykłady wielkości genomów mitochondrialnych i chloroplastowych dla
różnych organizmów.
Literatura uzupełniająca:
1. Fizjologia roślin. J. Kopcewicz, S. Lewak, PWN 2002.
2. Biologia roślin. Krótkie wykłady. A.J. Lack, D.E. Evans, PWN 2003.
3. Genomy. T.A. Brown, PWN 2001.
6
Wyszukiwarka
Podobne podstrony:
Izolacja DNA z komórek prokariotycznych i eukariotycznych
cwiczenie 8 izolacja DNA
Izolacja DNA z komorek zwierzecych
Izolacja DNA plazmidy Genetyka Nieznany
Etapy izolacji DNA
Izolacja DNA
Protokó- izolacji DNA na -wiczenia (1), analiza DNA
Izolacja DNA
Spr Izolacja DNA
Izolacja DNA
IZOLACJA DNA ROŚLINNEGO METODĄ MIKRO C, Biotechnologia notatki, Genetyka - biologia molekularna
Izolacja DNA, Biologia molekularna
2 Izolacja DNA
Izolacja DNA z kom zwierzecej
Ćw 2 Izolacja DNA plazmidy Genetyka
ćw 3 Protokół izolacji DNA genomowego
VI-Izolacja DNA, Genetyka
Laboratorium 3 - Instrukcja - Izolacja DNA z hodowanych komórek eukariotycznych, Semestr II, biologi
więcej podobnych podstron