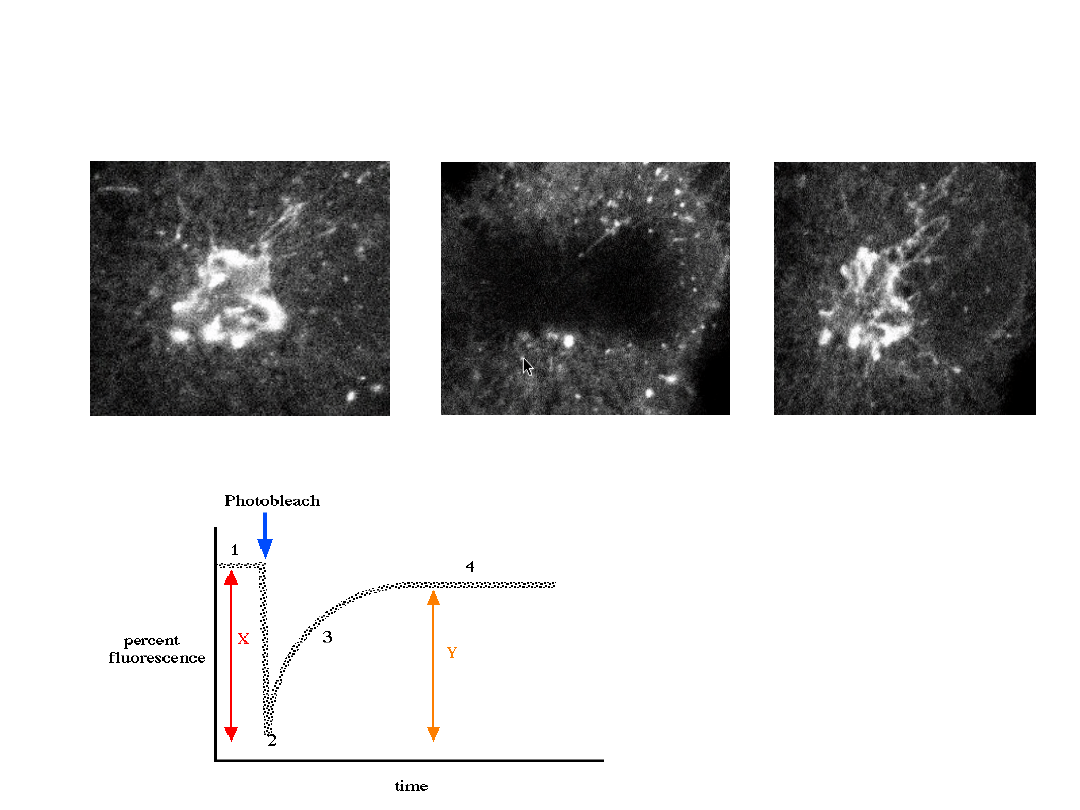
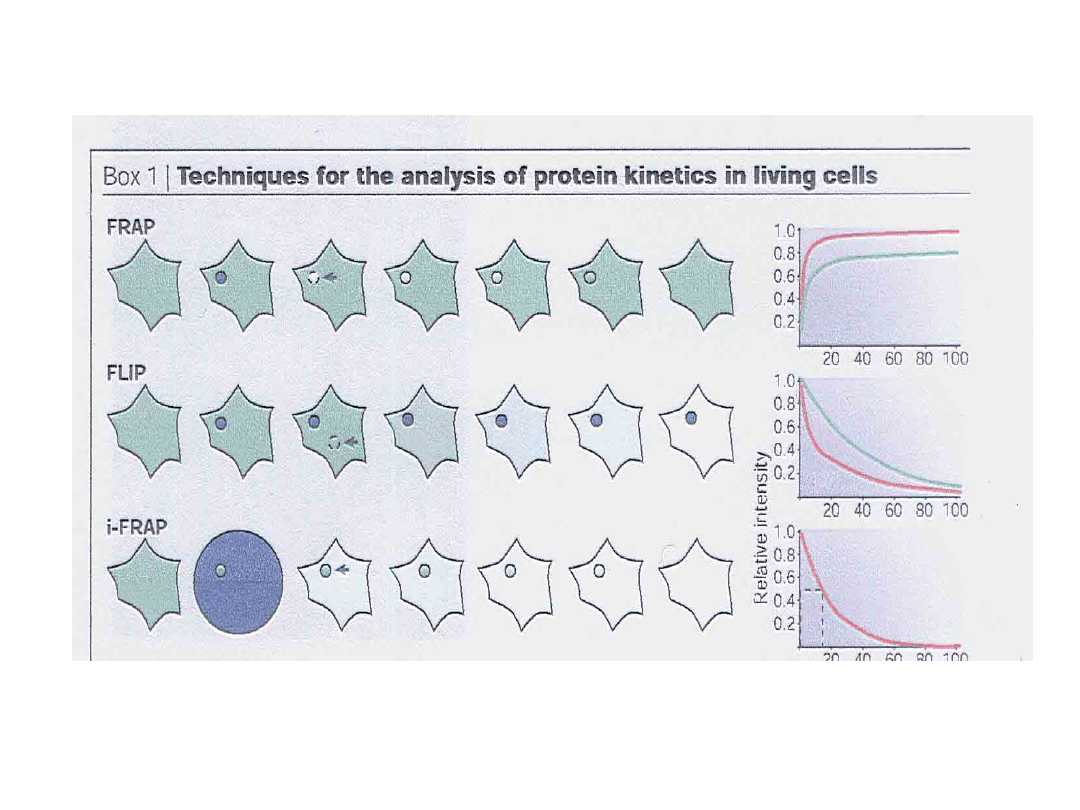
Przykład metody FRAP= Fluorescence Recovery After
Photobleaching :
Figure 1. Monitoring the
fluorescence before photobleaching
Figure 2. Monitoring the
fluorescence after photobleaching
Figure 3. Monitoring the recovery
of fluorescence after
photobleaching
Figure 4. Graphical presentation of data collected
during a FRAP experiment. A baseline of
fluorescence is collected (1) before the
photobleaching occurs (arrow) so that the amount of
fluorescence is reduced significantly (2). Over time,
the amount of fluorescence in the photobleached
area increases as unbleached molecules diffuse into
this area (3). Later, there is a stabilization of the
amount of fluorescence recovery (4) and a flat line is
obtained. The percent recovery uses the formula: (Y/
X) x 100 = % recovery. In the diagram, the
percentage of fluorescence lost due to
photobleaching is X and the amount of fluorescence
that returned to the bleached area is Y. In practice,
the percent recovery almost never reaches 100%.
The lateral mobility is determined by the slope of the
curve (3). The steeper the curve, the faster the
recovery and therefore, the more mobile the
molecules.
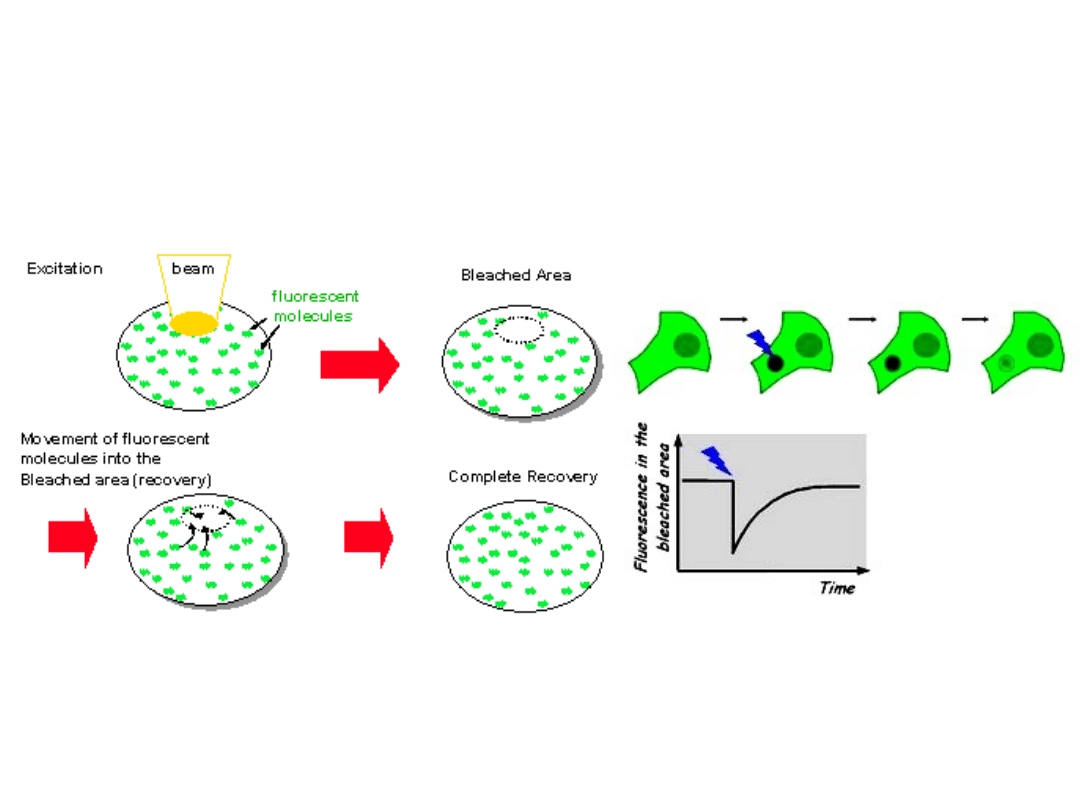
Metoda FRAP
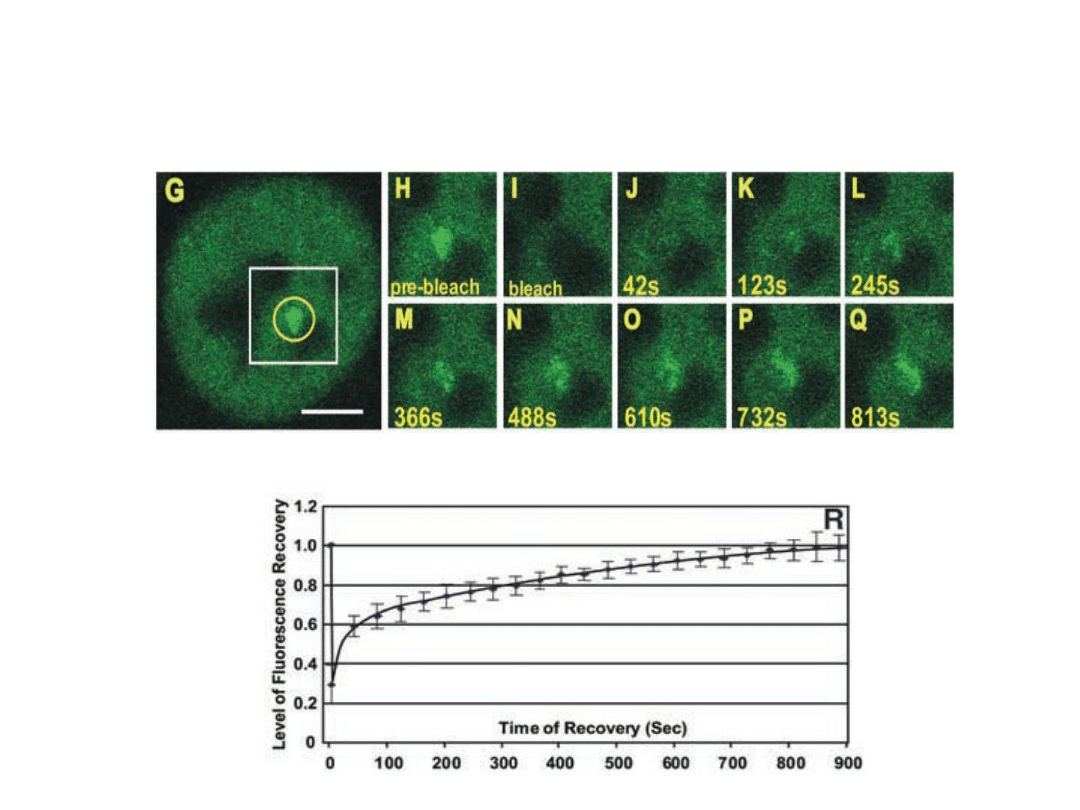
Wyniki analizy dynamiki pol II metodą FRAP :
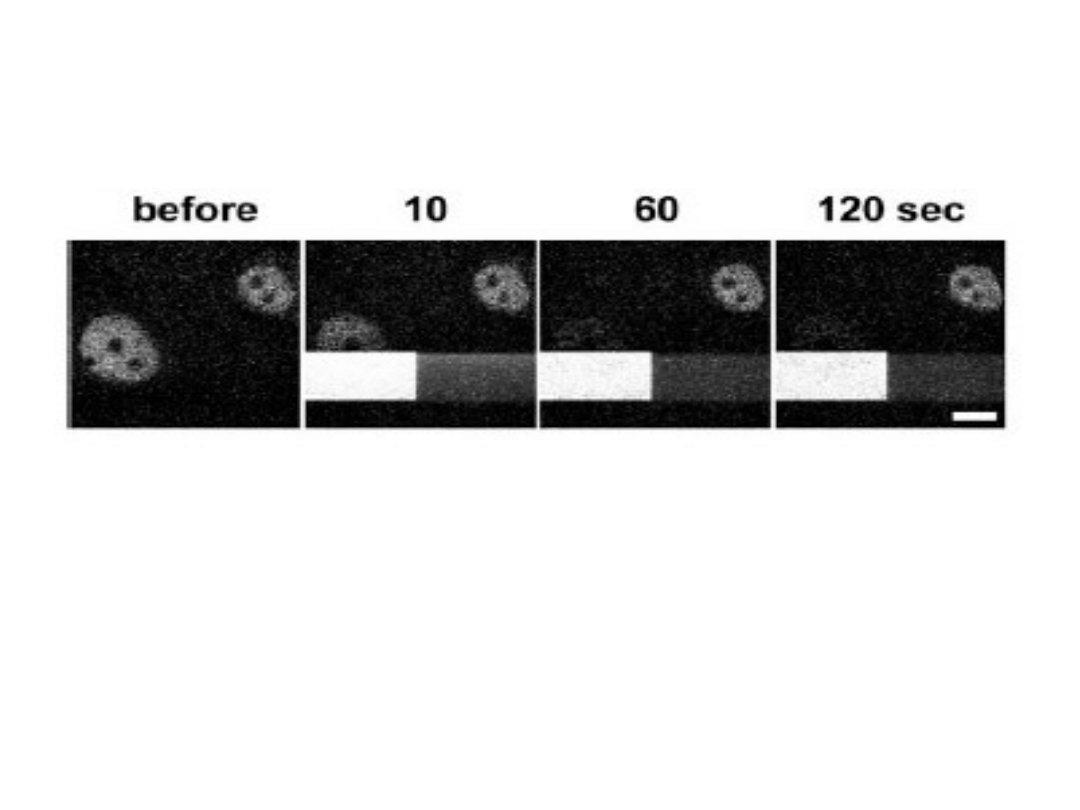
Kinetyka pol – GFP
metoda FLIP
:
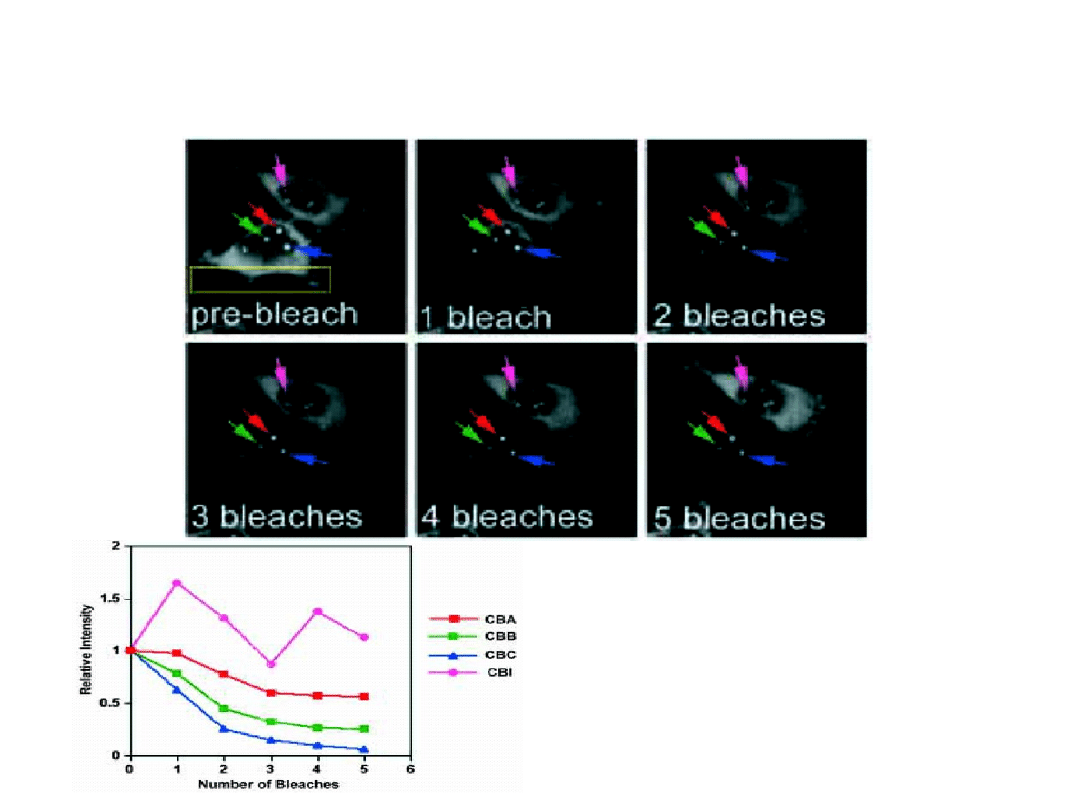
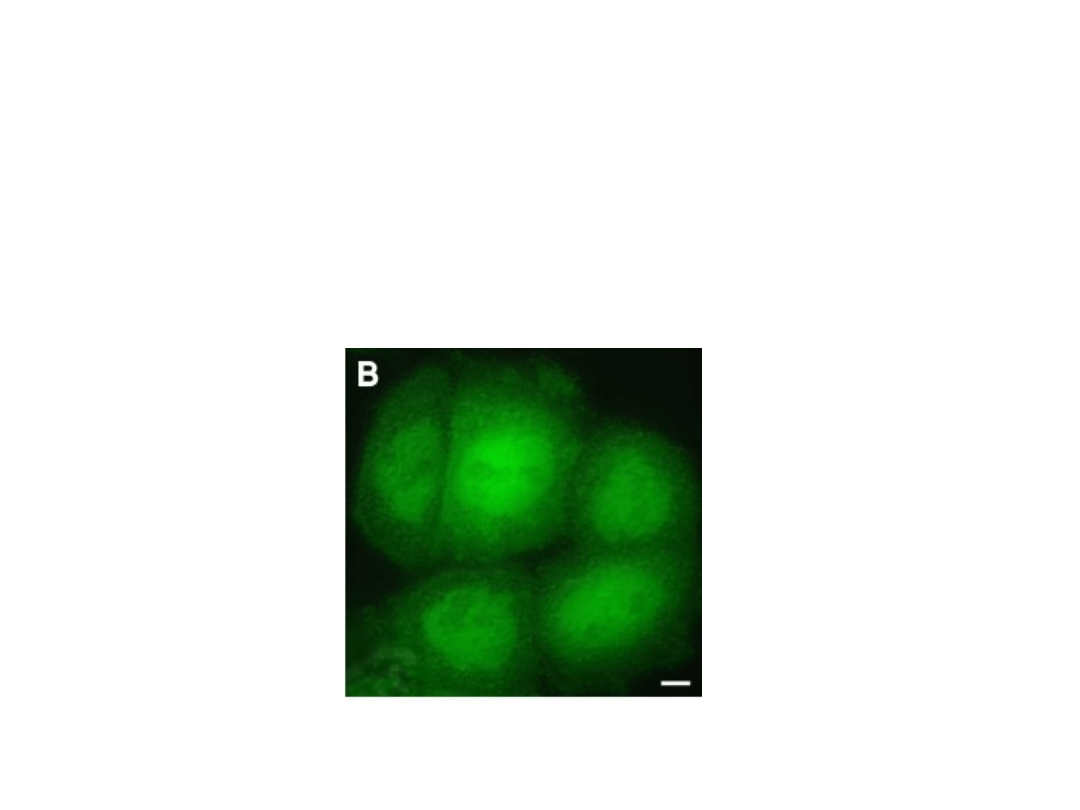
Przemieszczanie białek GFP-SMN między jądrem
a cytoplazmą
Zaobserwowano
spadek intensywności
sygnału GFP-SMN w
jądrze, w ciałach Cajala
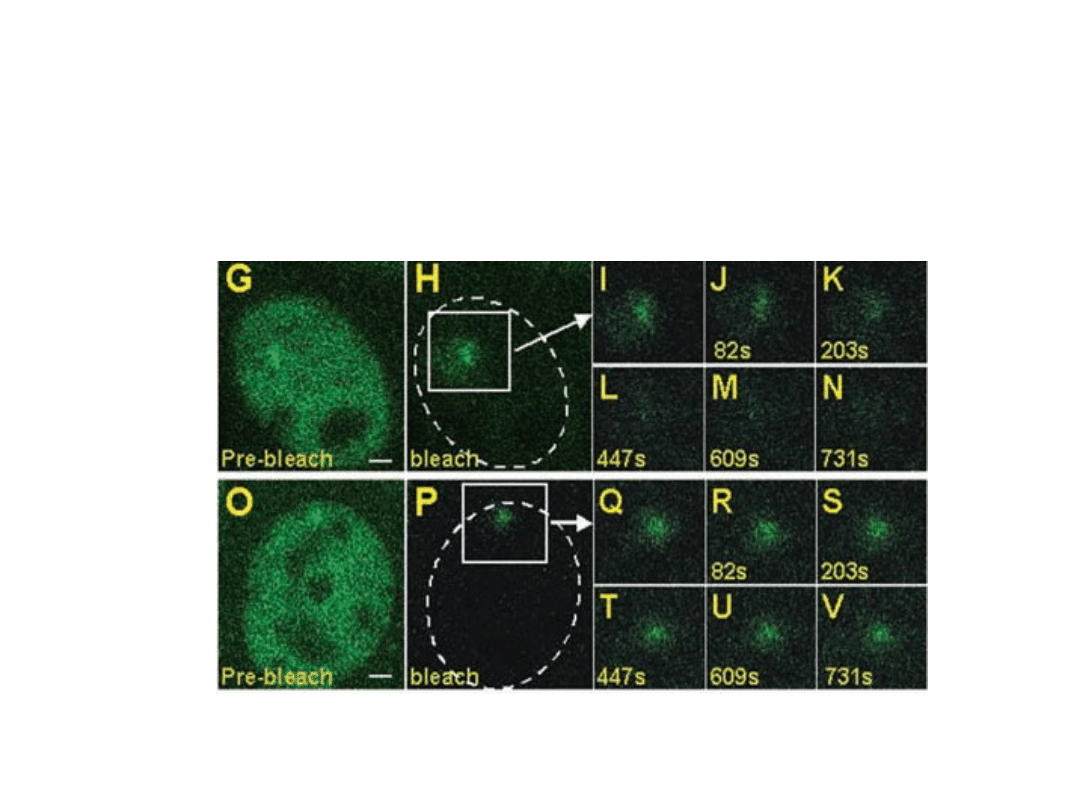
Badanie dynamiki GFP-pol II metoodą iFRAP :
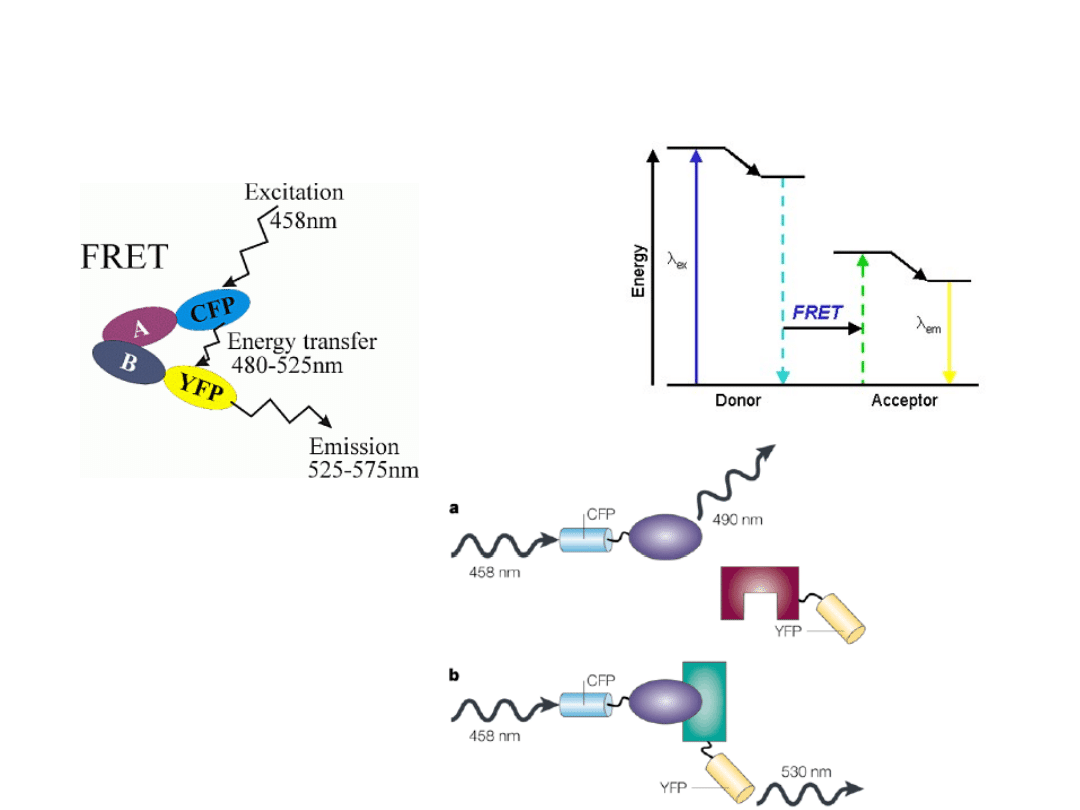
FRET –
FRET – F
F
luorescencyjny
luorescencyjny E
E
nergii
nergii T
T
ransfer
ransfer

Potrafimy już wykrywać białka w żywej komórce.
Jak obserwować DNA czy RNA w żywej komórce?
Metody pośrednie:
np. histonowe białka fuzyjne, fuzyjne białka SSB
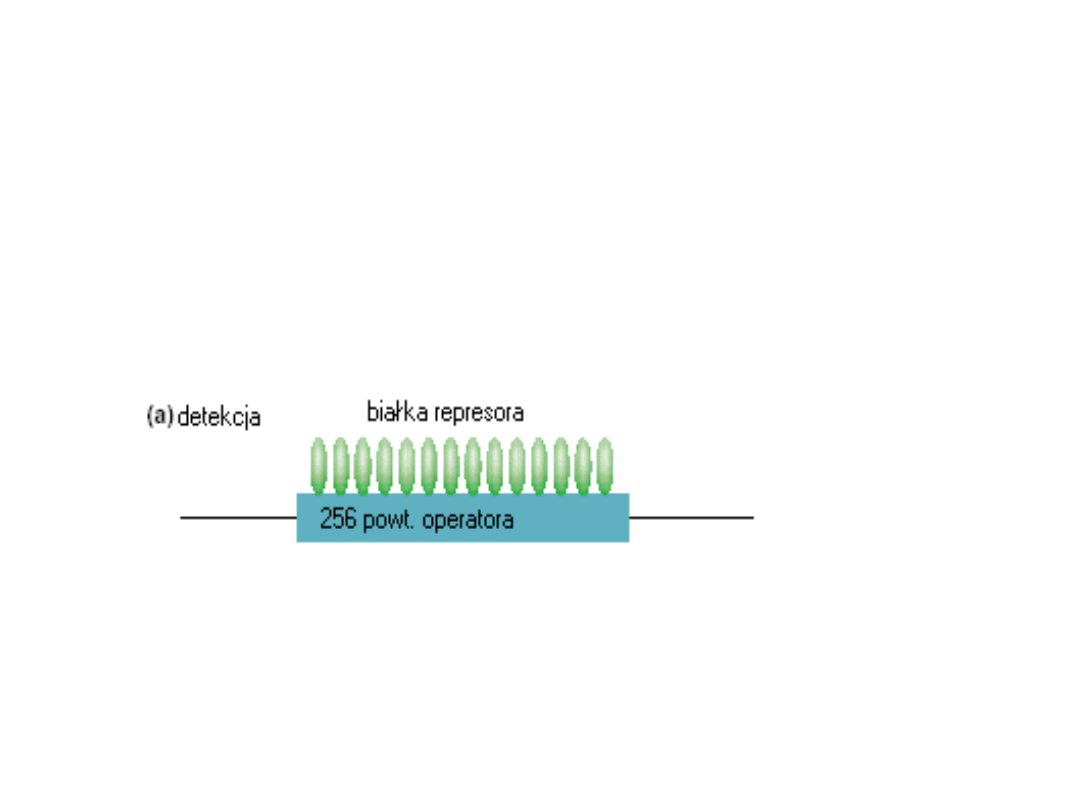
Wklonowywanie fragmentów markerowych DNA
w określone miejsca w genomie
przykład: represor-laktozowy

Obecnie dostępne technologie do detekcji genów w
komórkach,
takie jak in situ hybrydyzacja czy in situ PCR
nie pozwalają na detekcje in vivo
ponieważ linearne sondy używane w tych próbach
muszą być odpłukane po hybrydyzacji
aby zmiejszyć różnice miedzy sygnałem a tłem.
FISH: od in situ do in vivo - Nowe perspektywy

Aby można to zrobić sonda musi rozpoznawać cel
(szukany gen lub transkrypt) z dużą wydajnością,
a następnie po hybrydyzacji stać się wykrywalna,
wykazywać dużą różnicę miedzy tłem a sygnałem
pozwalając rozróżnić pozytywne i fałszywie pozytywne
wyniki.
Próba musi również wnikać do komórek z dużą
wydajnością.

Pomiędzy obecnie dostępnymi technologiami do
detekcji
kwasów nukleinowych w żywch komórkach
najbardziej
obiecująca wydaje się technologia
Molecular
Beacons.

Lighting
the WA
Y


•MBs są nowymi sondami o kształcie główki od
szpilki
które emitują fluorescencje kiedy zhybrydyzują z
komplementarnym do nich DNA/RNA.
•Molecular beacons zostały niedawno odkryte
przez Tyagi i Kramera z Public Health Research
Institute,
New York, USA
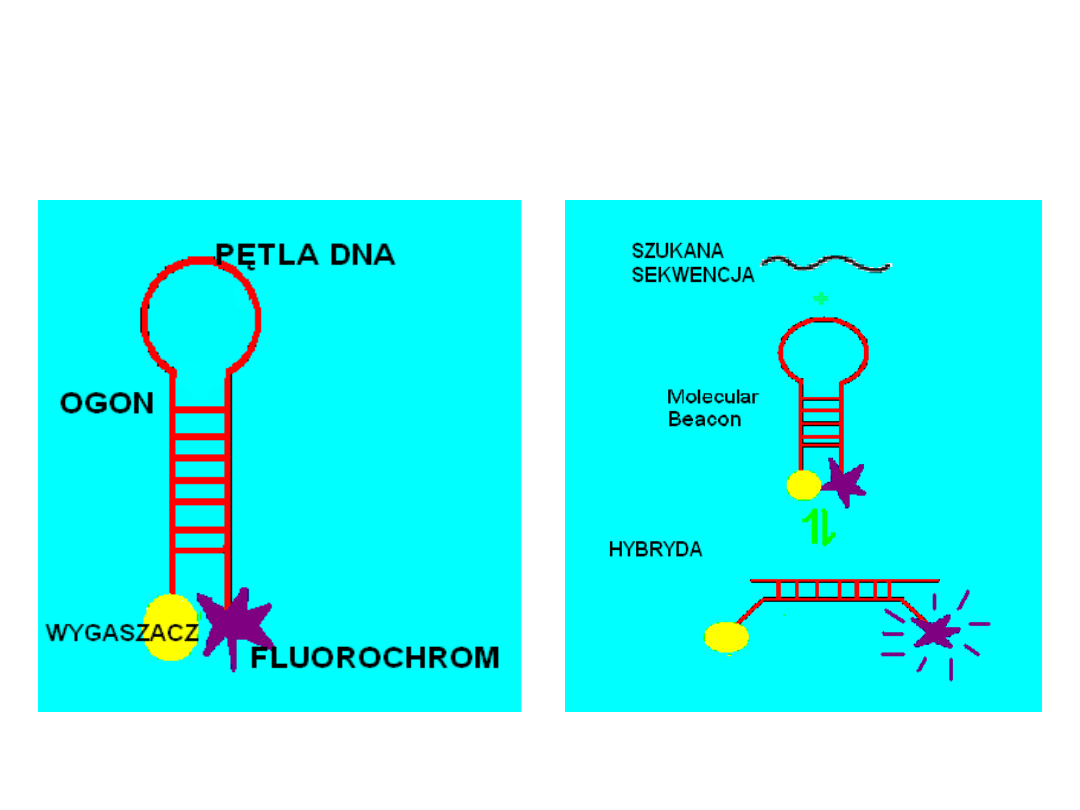
Budowa Molecular Beacon
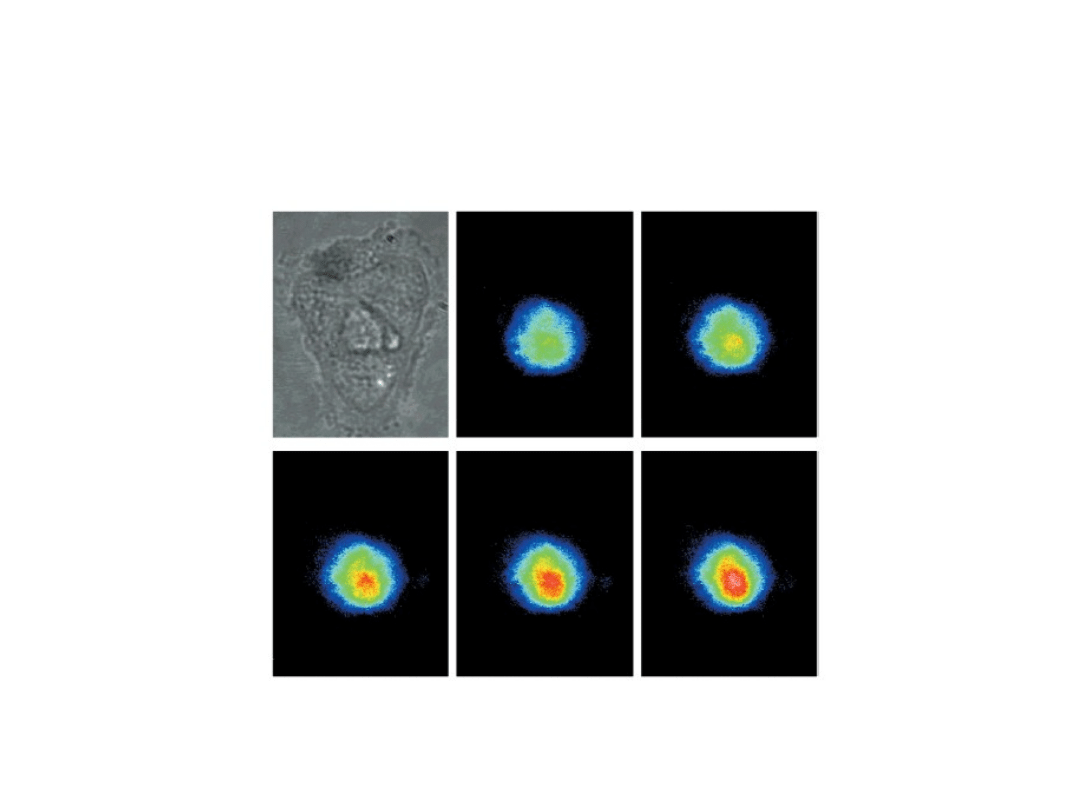
Perlette andTan 2001
specyficzna dlamRNA -actyny,
fluorescencyjne obrazy co 3-min
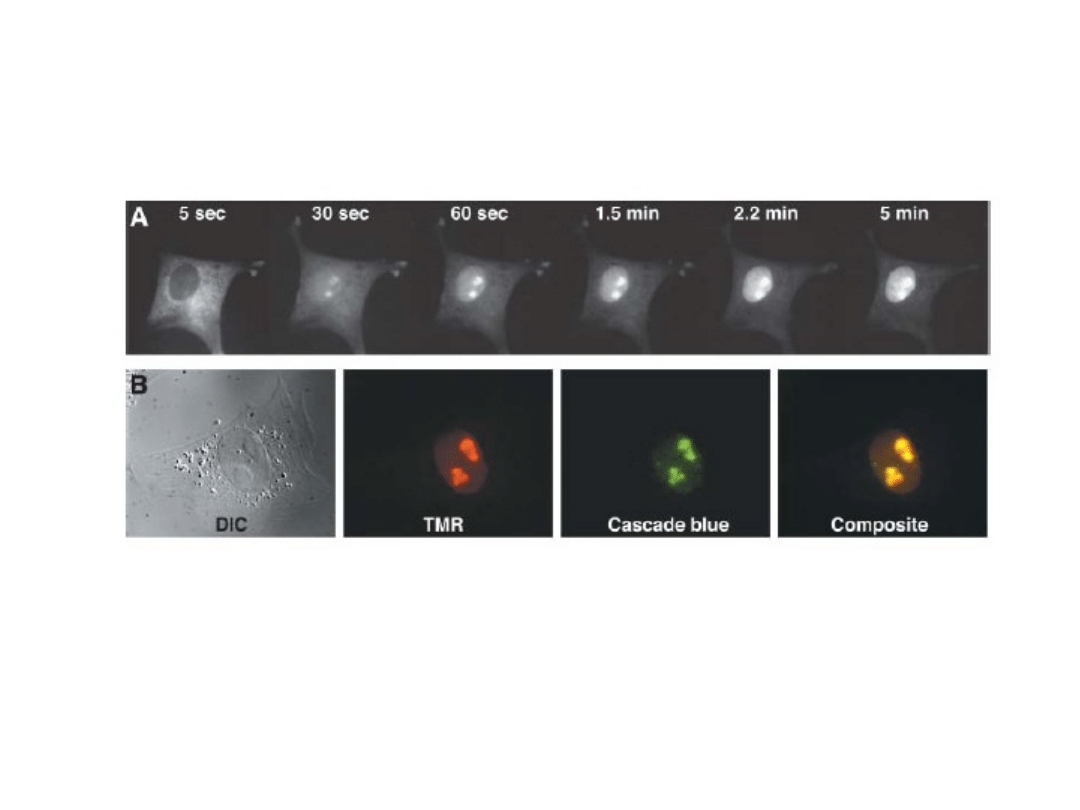
Actin mRNA
Tyagi and Alsmadi 2004

Tyagi and Alsmadi 2004
Nie do każdego fragmentu szukanego RNA należy konstruować sondę
typu MB
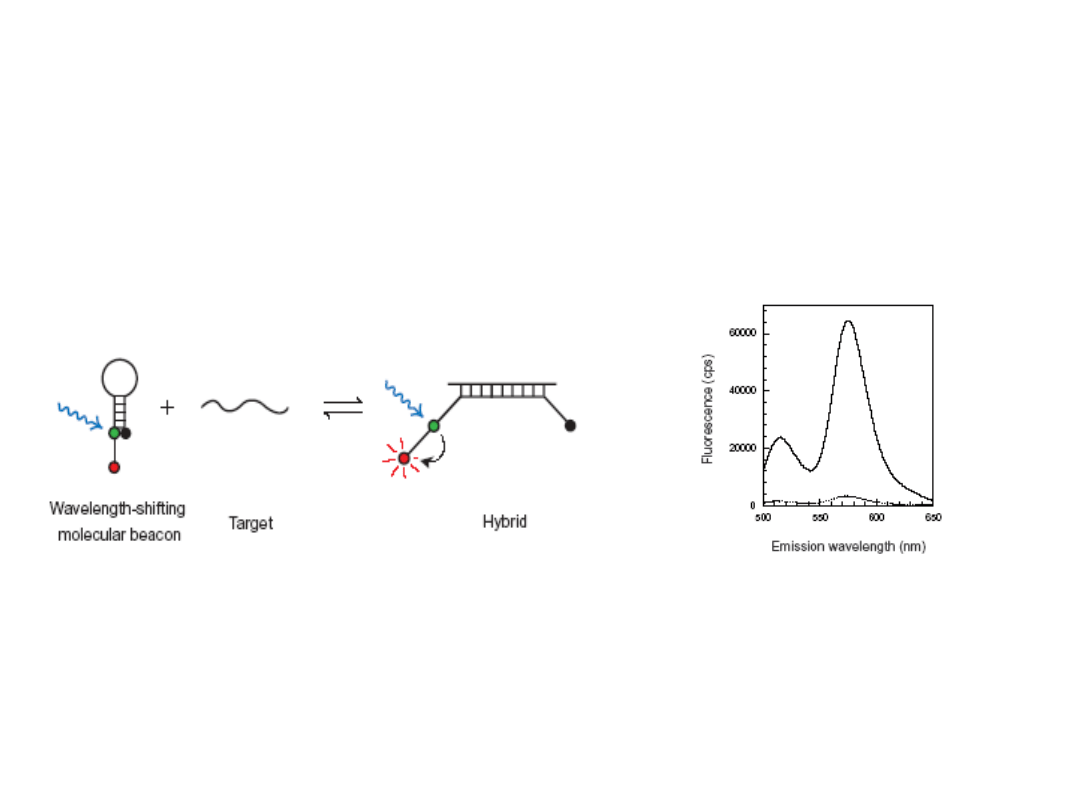
Wavelength-shifting molecular beacons
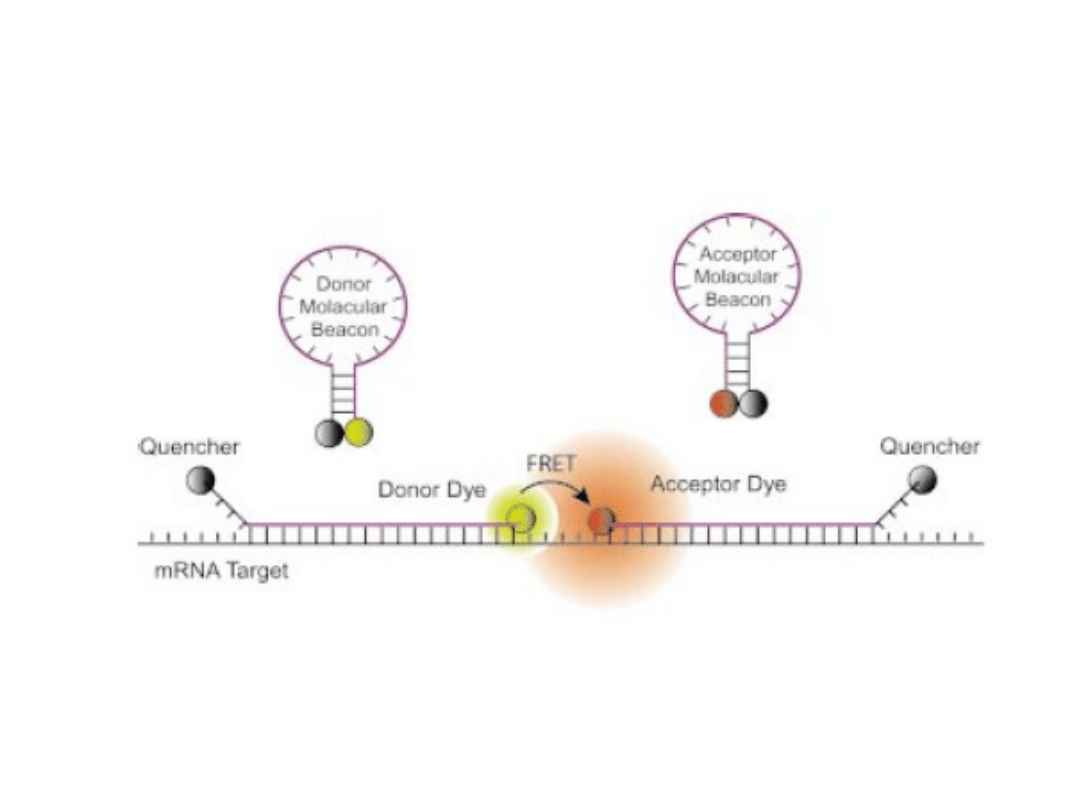
System 2x Molecular Beacon
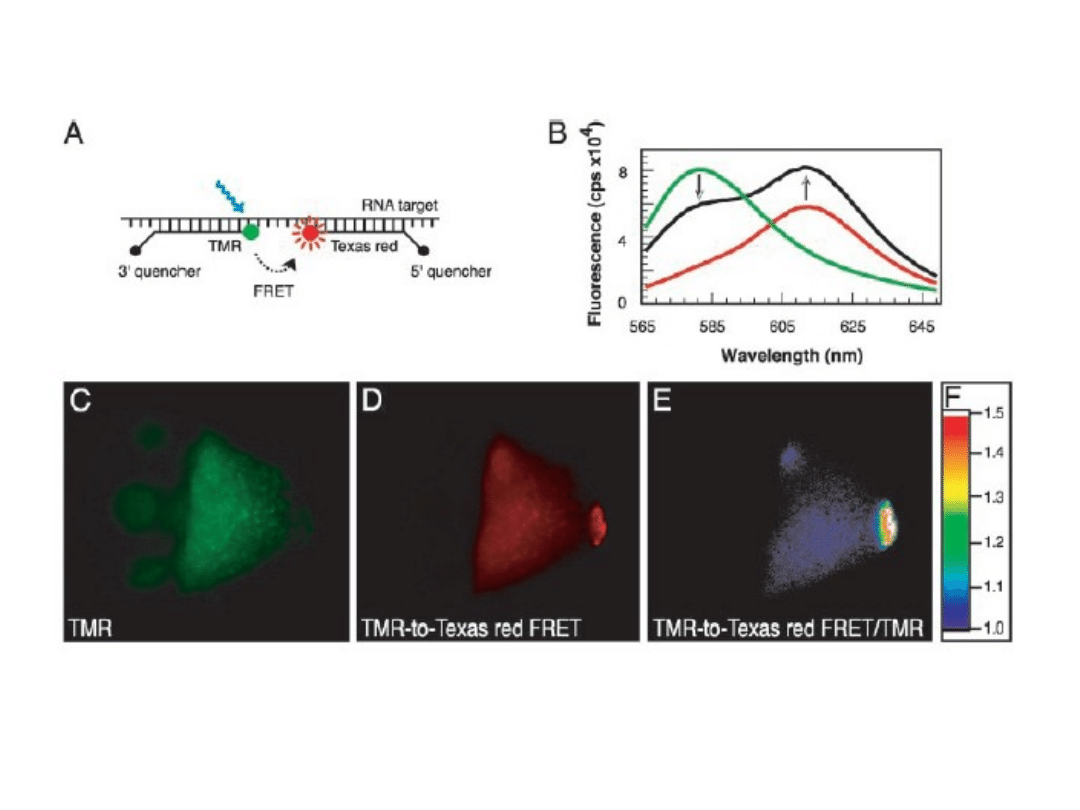
Santangelo et al. 2004

Jak wprowadza się sondy typu Molecular beacon
do wnętrza komórki ?
1. Mikroiniekcja
2. Hybrydowe sondy MB
Ostatnie badania pozwoliły zidentyfikować
małe
regiony
w
niektórych
białkach
(9±16
aminokwasów)
nazwanych protein transduction domains
(PTDs) lub
cell penetrating peptides (CPPs) które które
powodują, że białka je posiadające mają
zdolność do
łatwego przenikania przez memnrany
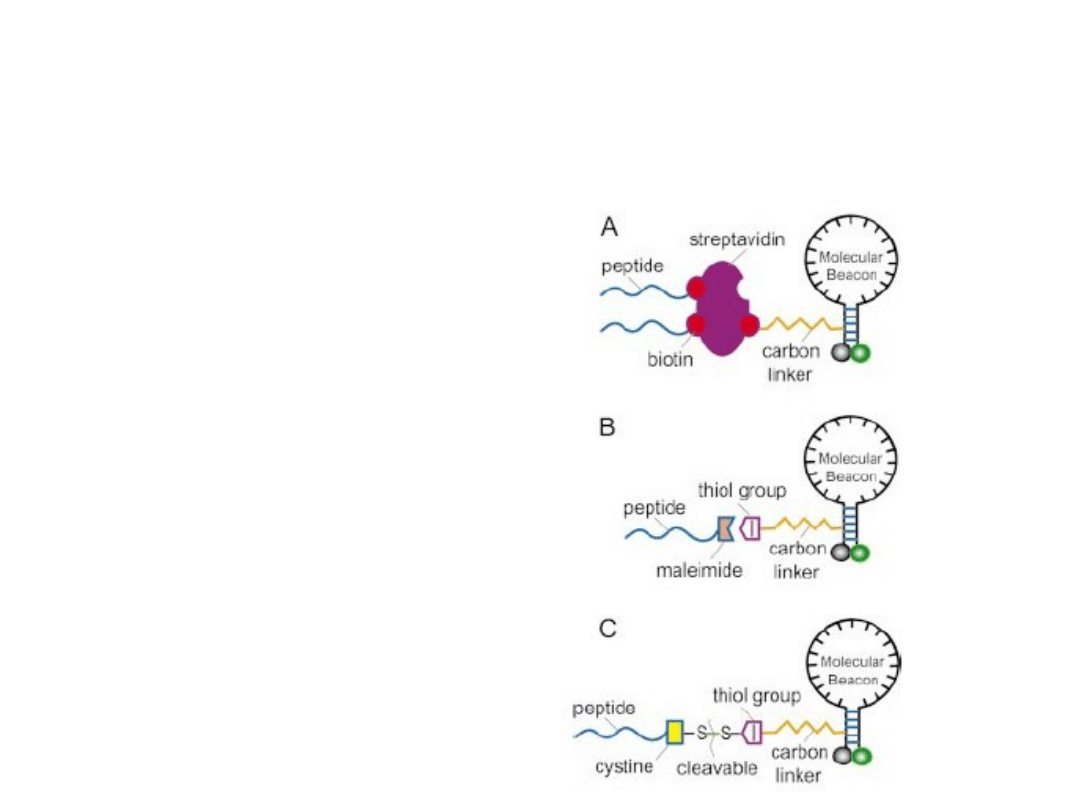
Sondy typu PNA – Peptide Nucleic Acid
Trzy rodzaje sond typu PNA
posadających
różny
sposób
przyłączenia
peptydu do sondy typu
molecular beacon.
Konstructy PNA molecular beacon wnikają do wnętrza żywej
komórki w ciągu 30 min z prawie 100% wydajnością.
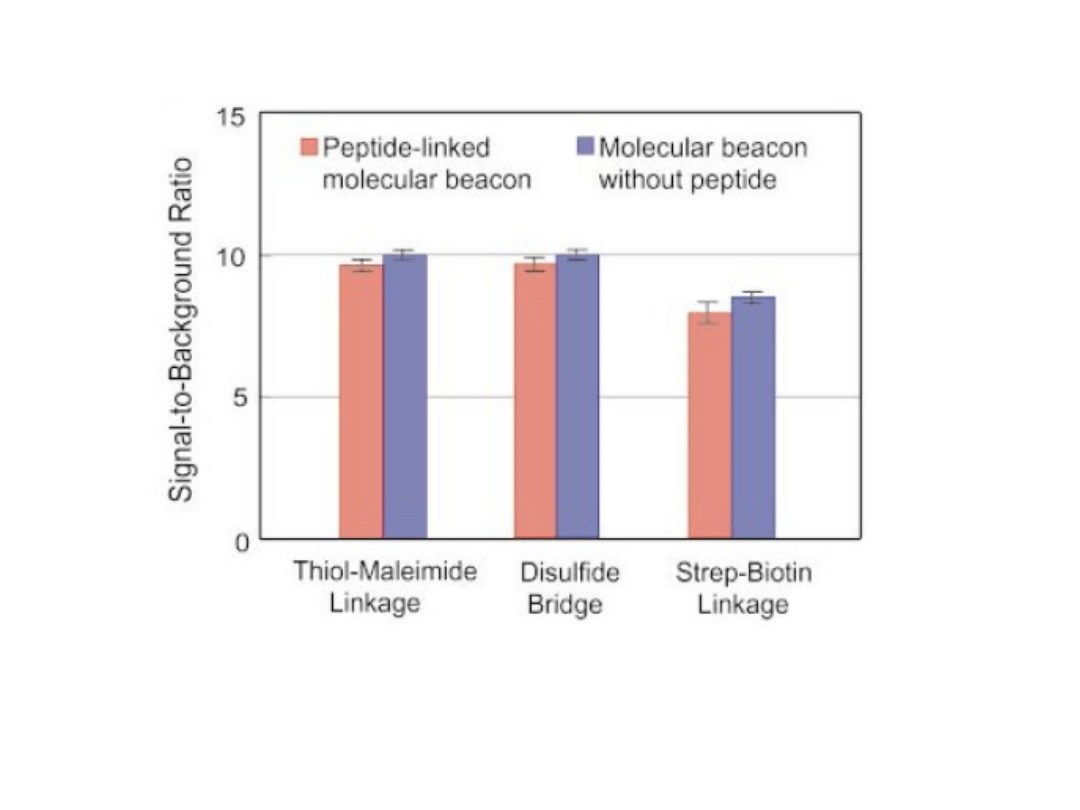
Nitin N. at al. 2004

Problem 1: hybrydy RNA/DNA
są potencjalnym celem dla
komórkowych RNAz
Rozwiązanie :
Sondy MB typu 2`-o-methyl RNA
Jakieś inne problemy?
Fluorescencyjnie znakowane 2`-o-methyl
RNA
Molenaar et al. 2001
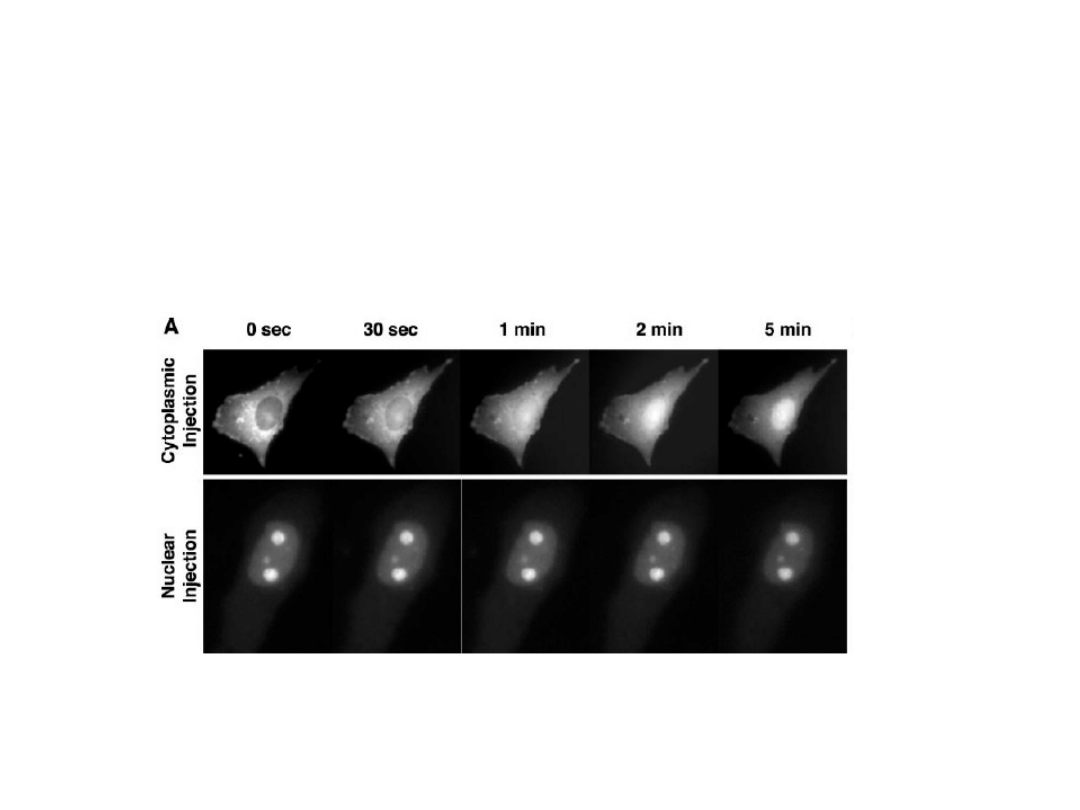
Kiedy oligonukleotydowe sondy są wstrzykiwane do komórek
żeby pokazać rozkład RNA, są one szybko transportowane
i zatrzymywane na terenie jądra.
Rezultat trudno wykrywać mRNA w cytoplazmie żywych komórek.
Rozwiązanie – e.g. tRNA molecular beacons
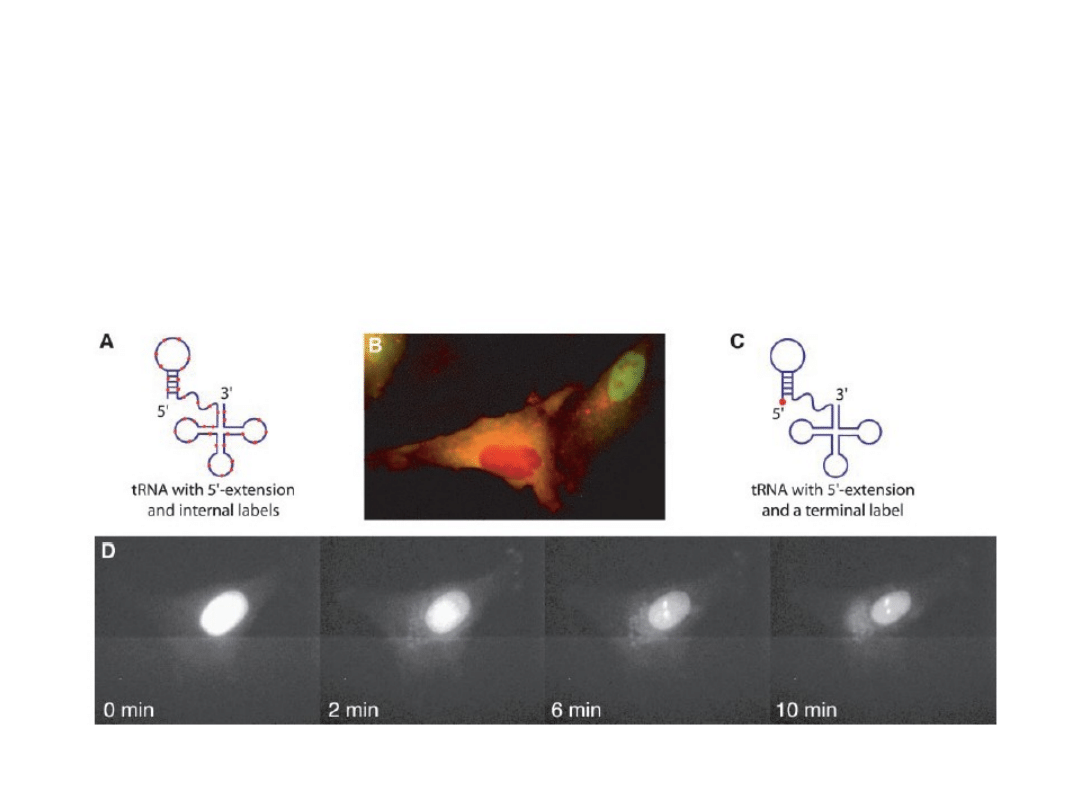
Znakowane tRNAs, po wniknięciu do jądra
są następnie eksportowane do cytoplazmy.
Mhlanga M. et al. 2005
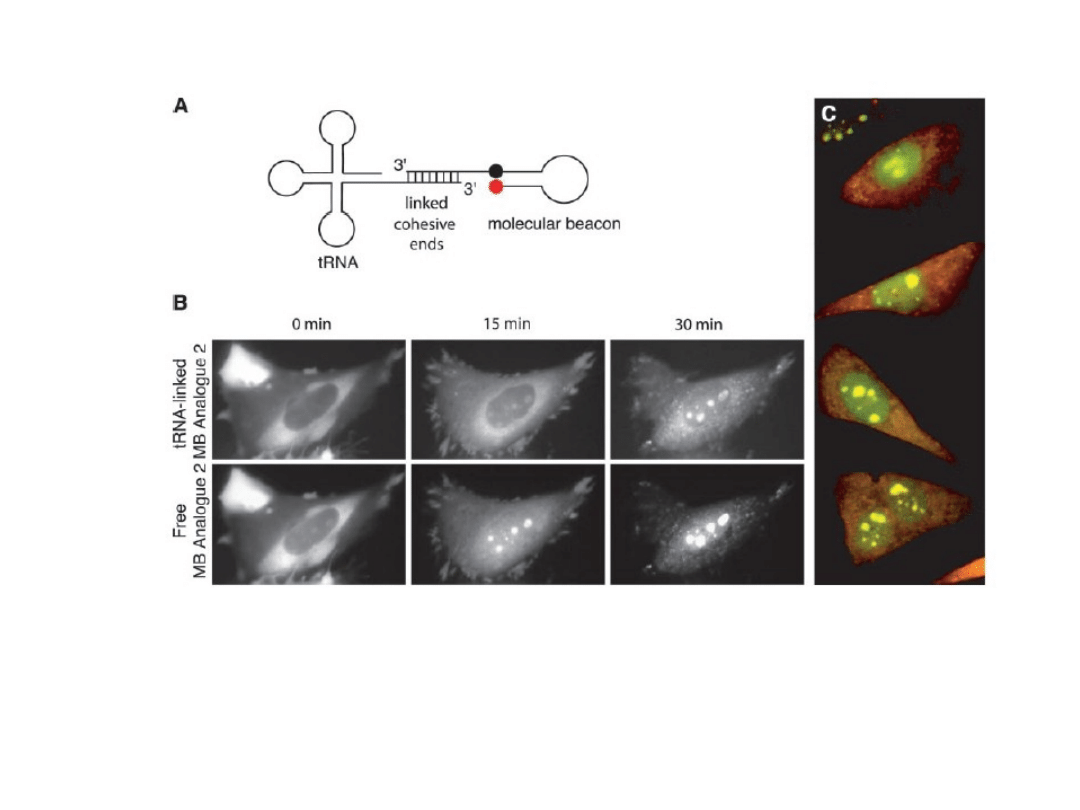
Fig c – obraz złożony pokazujący komórki po 30 min
of inkubacji. Czerwona fluorescencja - tRNA-MB
zielona fluorescencja zwykłych MB.
Mhlanga M. et al. 2006

Jak hybrydyzować do DNA w żywej komórce?
Główny problem denaturacja dwunicowego DNA
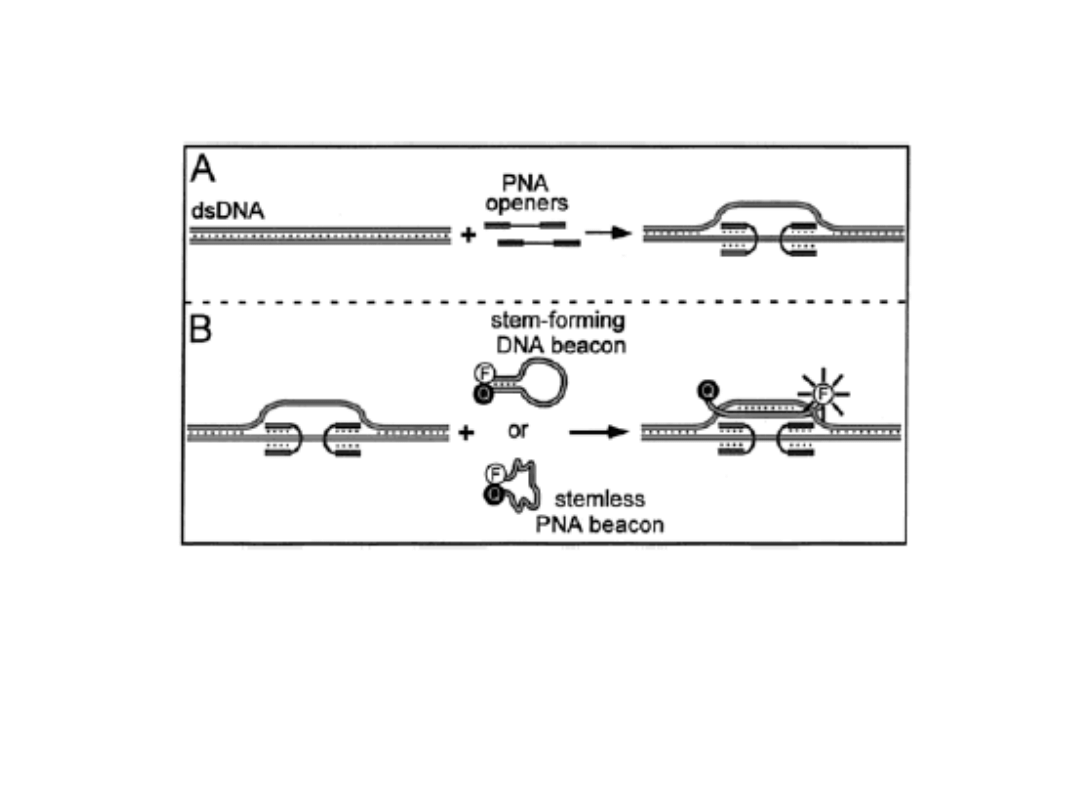
Hybrydizacja DNA i PNA Molecular Beacon do
jedno i dwuniciowego DNA
1. PNA otwieracze tworzą tripletowe struktury na
flankach szukanej sekwencji
2. Kiedy dsDNA jest otwarty sondy typu MB mogą
hybrydyzować do drugiej nici.
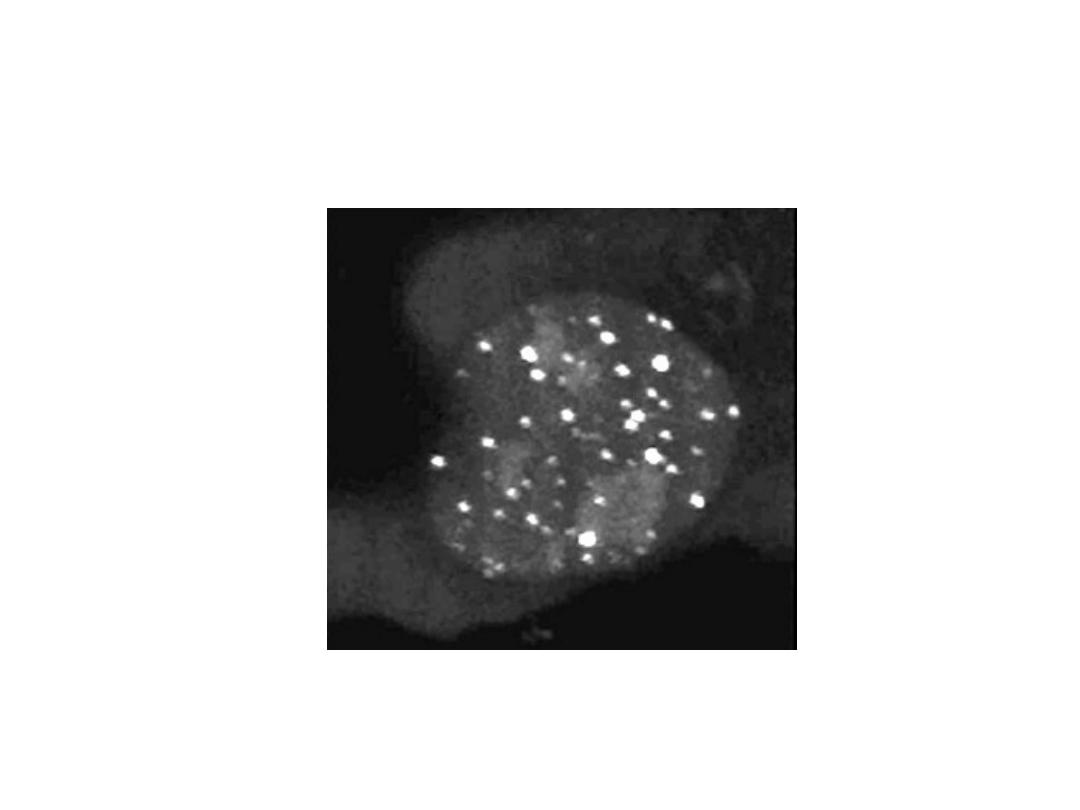
PNA denaturacja i Molecular Beacon`s in vivo fluorescencenccja
hybrydizacja do chromosomalnych telomerowych powtórzonych
sekwencji w żywej komórce
Tanke et al. 2006
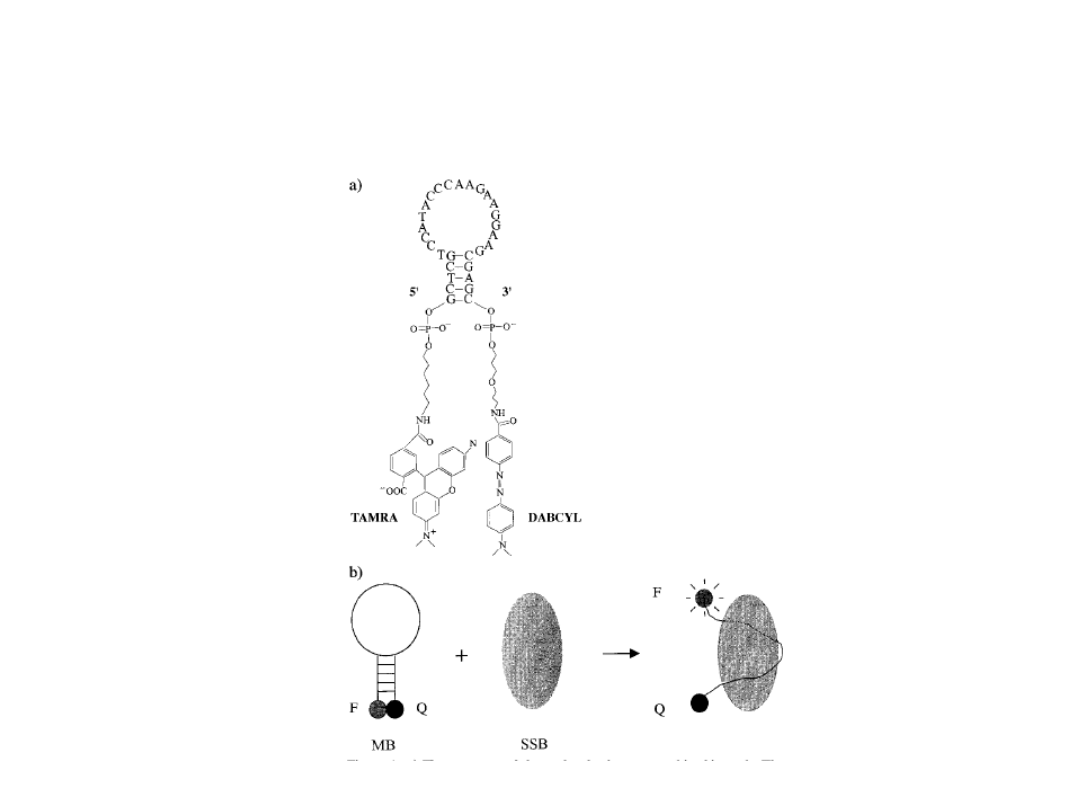
Molecular Beacon:
nowe zastosowanie do detekcji białek w żywych
komórkach
Sekwencje przyłączające
się do białek SSB

Nowe spojrzenie na regulacje
ekspresji genetycznej
Reinicjacja transkrypcji

Transkrypcja jest procesem powtarzalnym, podczas którego
następuje synteza wielu identycznych kopii RNA z tego
samego genu
W ujęciu klasycznym w skrócie proces ten odbywa się w czterech etapach:
* rozpoznanie promotora genu, etapy *inicjacji, *elongacji i *terminacji transkrypcji
Pytania:
1. Kiedy gen zostanie już raz uruchomiony, czy następne cykle
transkrypcyjne przebiegają w ten sam sposób czy następuje
„przetarcie szlaku”?
2. Czy istnieją mechanizmy zatrzymujące przy genie maszynerię
transkrypcyjną po każdym cyklu?
3. Czy sam gen i jego otoczenie ulegają w takim przypadku
„imprintingowi transkrypcyjnemu”
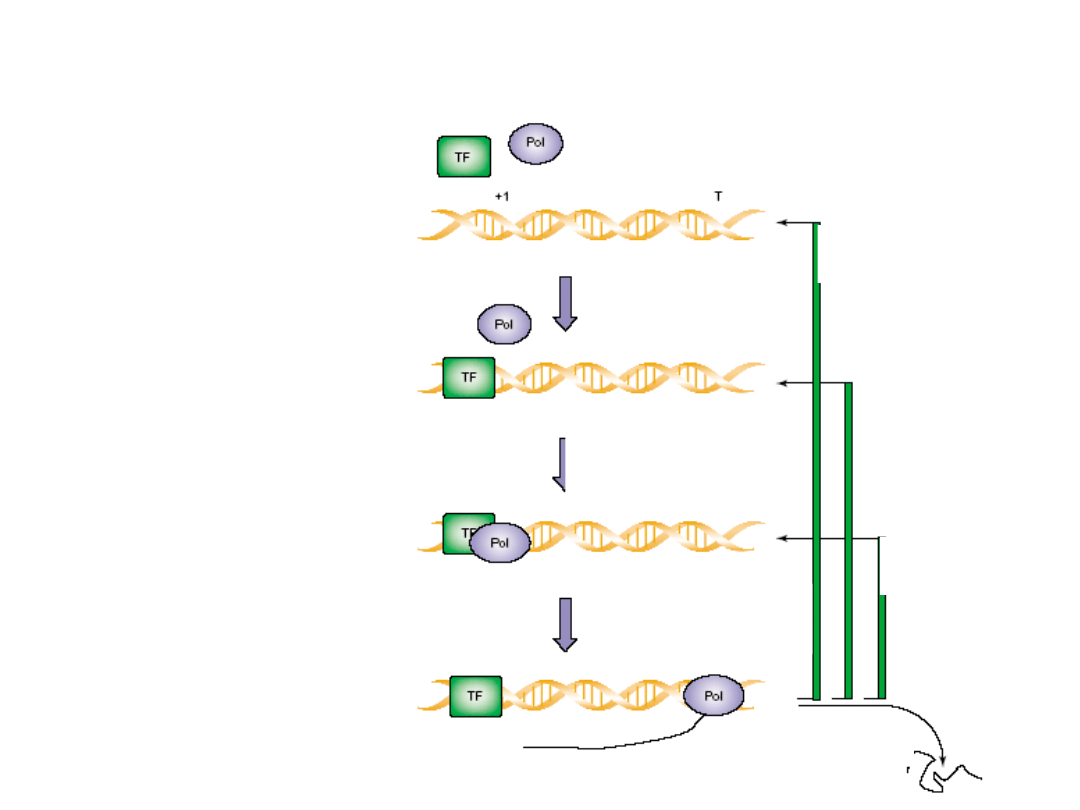
Tworzenie PIK*
Mechanizm zależnej od matrycy reinicjacji transkrypcji
*PIK -
P
renic
j
acyjny
k
ompleks
Przyłączenie
polimerazy
RNA
Inicjacja, elongacja
i terminacja syntezy
RNA
(a)
Reinicjacja podstawowa
(b)
Reinicjacja typu PIK
(c)
Szybka reinicjacja
Oddysocjowanie
RNA
(b) W PIK reinicjacji podczas pierwszego
cyklu matryca jest modyfikowana.
Powstaje stabilny kompleks DNA-białka.
Zawiera on wszystkie lub tylko niektóre
czynniki transkrypcyjne (TF) niezbędne
do następnego cyklu transkrypcji.
Charakterystyczna dla wszystkich trzech
polimeraz eukariotycznych
(a)
Podstawowy
szlak
reinicjacji
transkrypcji. Wszystkie procesy i etapy
które odbywają się w pierwszej rundzie
transkrypcyjnej
muszą
zostać
powtórzone w każdym kolejnym cyklu.
Charakterystyczny
dla
polimeraz
bakteryjnych.
(c) W szybkiej reinicjacji polimeraza
może
prowadzić
wiele
cykli
transkrypcyjnych na tym samym genie
bez oddysocjowania przy każdym cyklu.
Występuje tylko dla polimerazy III
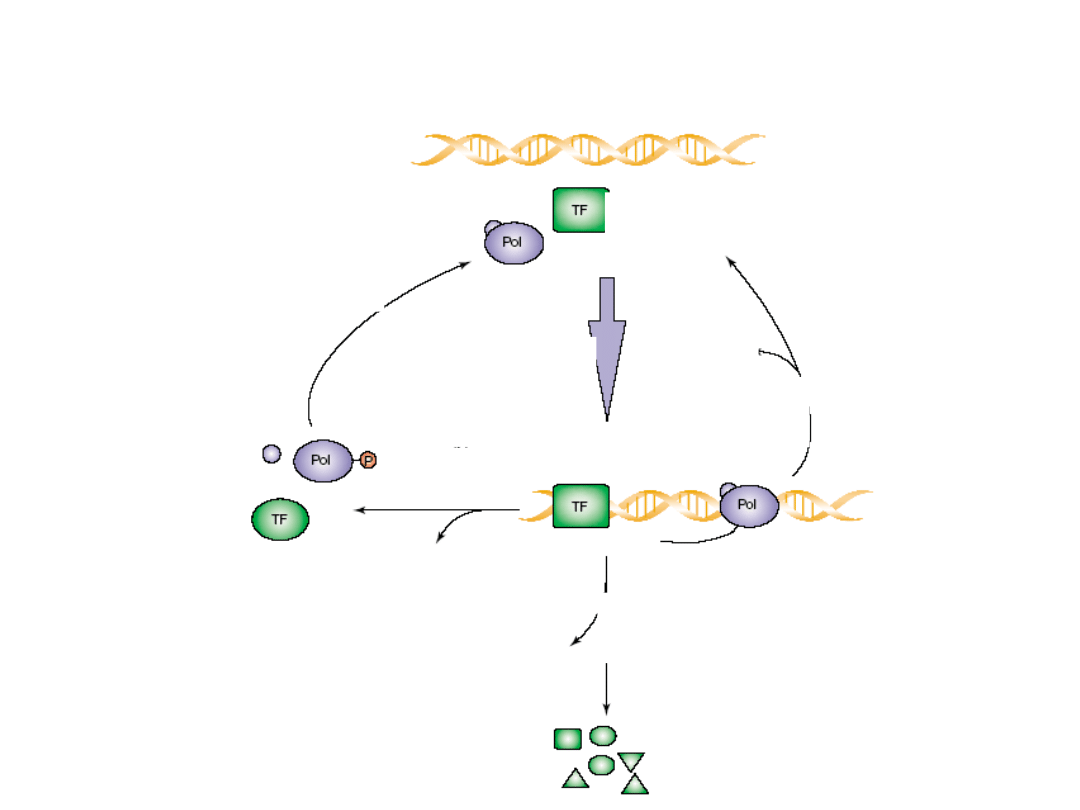
Mechanizm zależnej od białek reinicjacji transkrypcji
(model uproszczony nie zawiera czynników elongacyjnych i terminacyjnych)
Matryca DNA
Białkowe składniki
transkrypcyjne
Regeneracja
Transkrypcja
DNA, RNA
DNA, RNA
DNA, RNA
Modyfikacje
odwracalne
Inaktywacja
nieodwracalna
Bezpośrednia
reaktywacja

100 aktywnych genów 5S rDNA produkuje ~ 2000 kopii 5S
rRNA w ciągu minuty. Jeden cykl zachodzi co 3 sekundy.
Wydajność transkrypcji ~ 40 nukleotydów/sekunde.
Długość 5S rRNA ~ 120 nukleotydów.
Czas na reinicjacje pozostaje mniej niż pół sekundy!!!
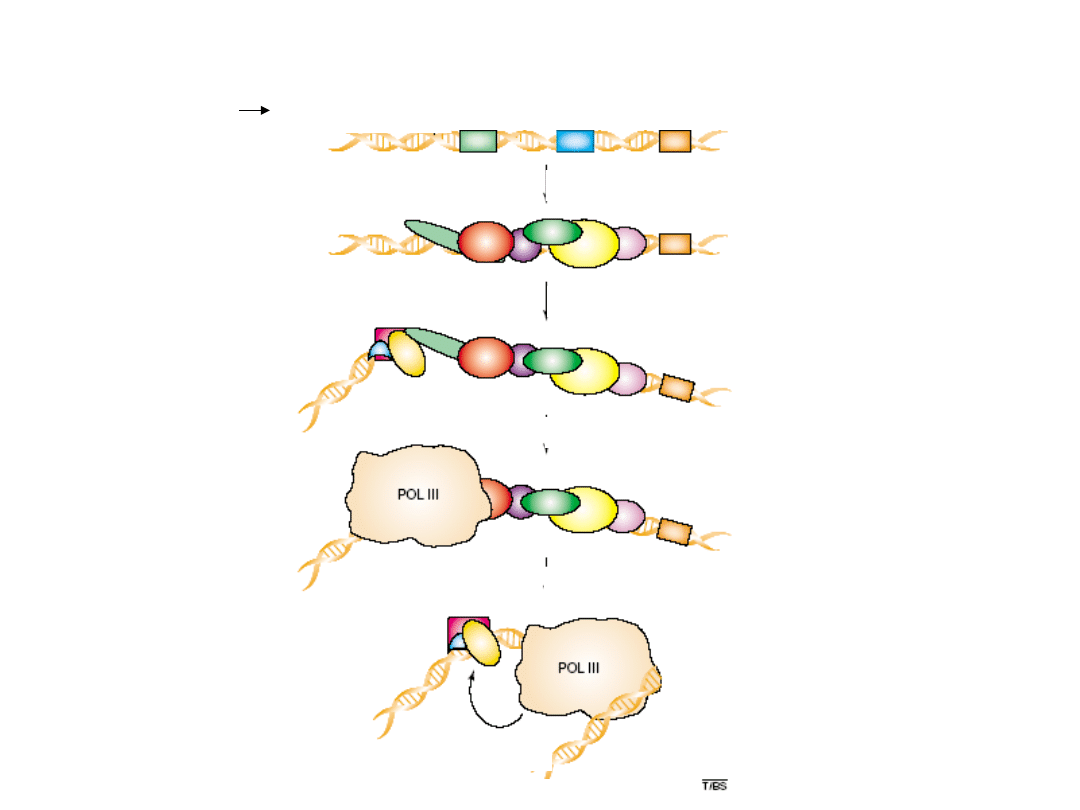
5’ sekwencje flankujące A-blok B-blok Terminacja
Pol III
TFIIIC
TFIIIB
Transkrypcja
Szybka reinicjacja
Eukariotyczna polimeraza III
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
Wyszukiwarka
Podobne podstrony:
Biol kom cz 1
Biol kom cz 4
Biol kom cz 6
Biol kom cz੠2
Biol kom cz 2
Biol kom cz 1
Biol mol kom cz 3
Sp asp proc kom cz VII 2010
Test biol kom, biologia komórki(3)
BIOL.KOM pytania chyba Witaliński, biologia uj, biologia II, biologia komorki, egz
Biol KOM zaliczenie
Organella Biol Kom
Kult uwar kom cz VIII 2010
Kult uwar kom cz II 2010
biol kom wyklad 2103, Chemia środków bioaktywnych (umcs), BIOLOGIA KOMÓRKI
Kultura a kom cz I 2010
Biol KOM zaliczenie id 87300
Pare pytań z egzaminu na biol kom
biol kom pyt 11
więcej podobnych podstron