Regulacja ekspresji
genów
u organizmów
eukariotycznych. I.
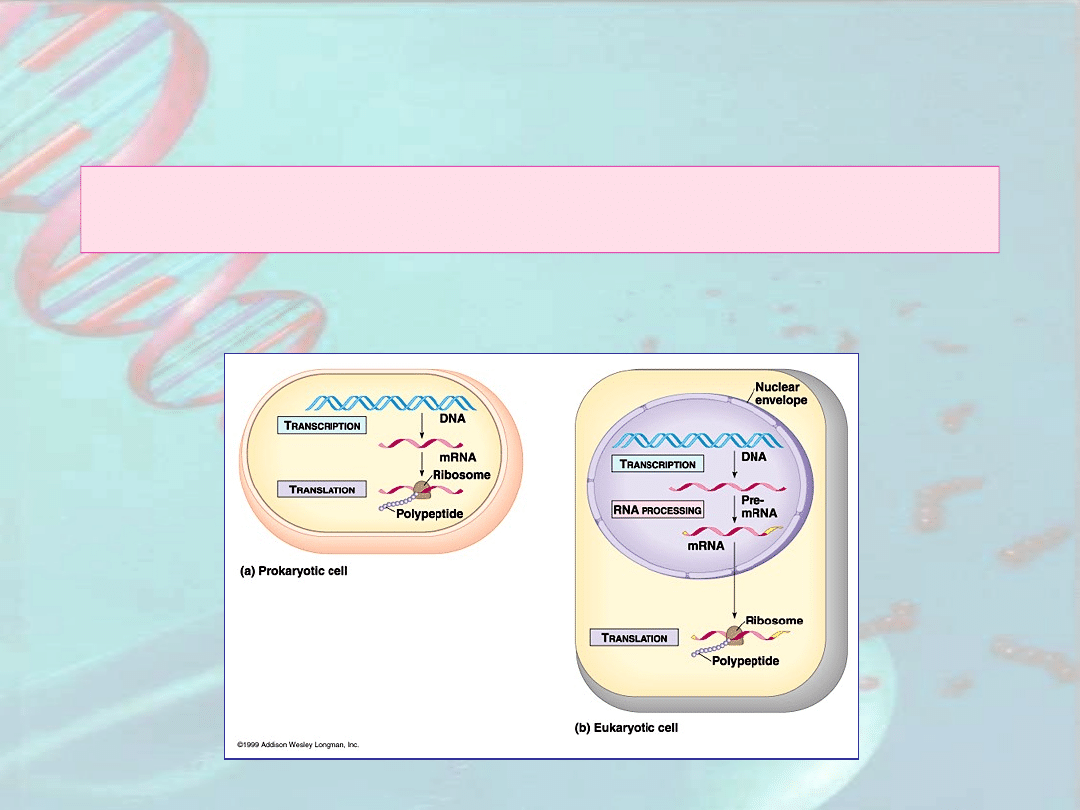
Zasadnicze różnice między organizmami
prokariotycznymi i eukariotycznymi
Regulacja ekspresji genów u Eukaryota
Organizmy eukariotyczne posiadają jądro, a ich
geny są nieciągłe
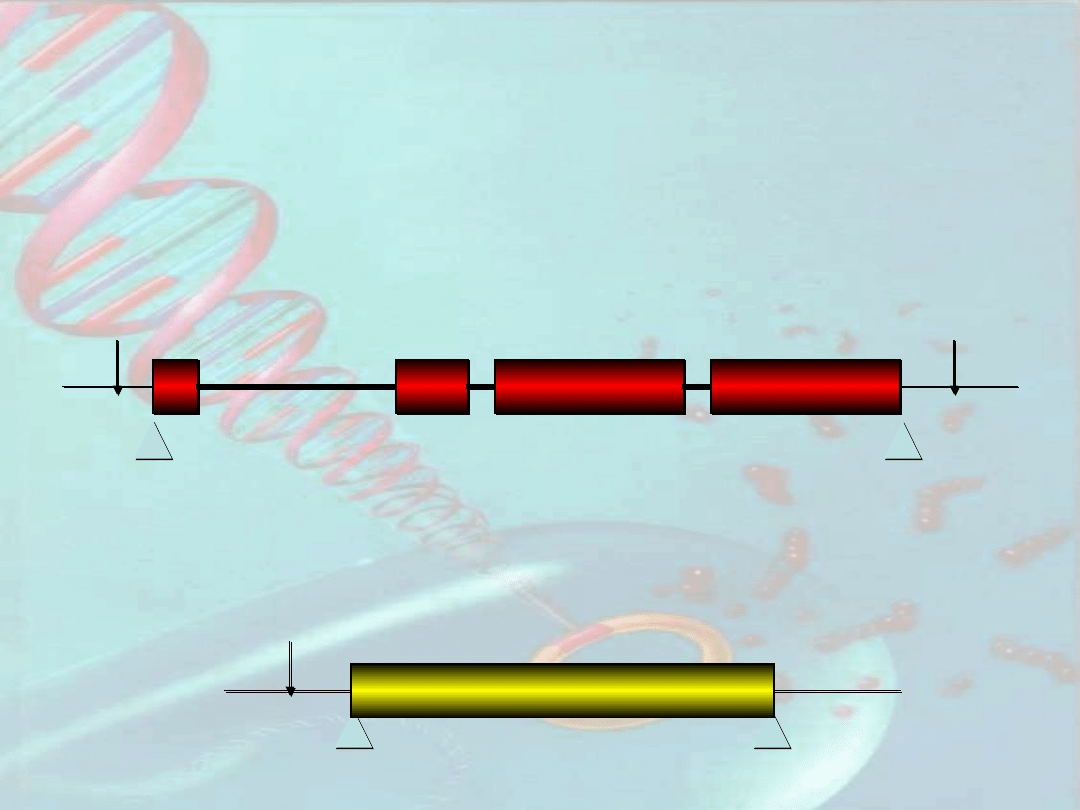
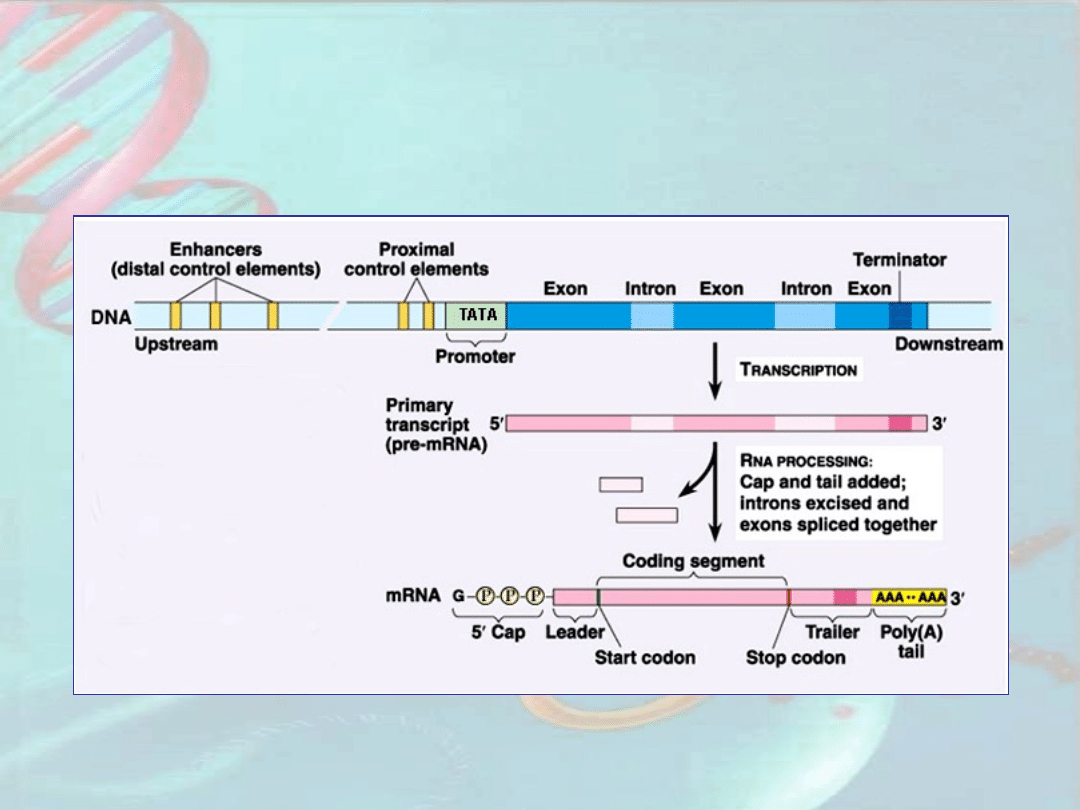
Struktura genu kodującego
dehydrogenazę alkoholową – Adh u
Eukaryota i Prokaryota
Promoter region
Promoter region
•
TATA box
TATA box
•
CAAT box (in mammals)
CAAT box (in mammals)
•
GC box (GGGCGGG)
GC box (GGGCGGG)
Initiation codon
Initiation codon
Stop codon
Stop codon
Polyadenylation
Polyadenylation
signal
signal
AATAA
AATAA
Exon 1
Exon 1
Exon 2
Exon 2
Exon 3
Exon 3
Exon 4
Exon 4
Intron 1
Intron 1
Intron 2
Intron 2
Intron 3
Intron 3
5’
5’
3’
3’
Eukaryot
Eukaryot
a
a
Initiation codon
Initiation codon
Stop codon
Stop codon
Promoter region
Promoter region
•
Shine-Dalgarno box (AGGAGG)
Shine-Dalgarno box (AGGAGG)
•
Pribnow box (TATAAT)
Pribnow box (TATAAT)
•
-35 site (TTGACA)
-35 site (TTGACA)
Prokaryot
Prokaryot
a
a
5’
5’
3’
3’
Jim Provan
Poziomy regulacji ekspresji genów
eukariotycznych
Transkrypcja
Po transkrypcji
Translacja
Po translacji
Regulacja ekspresji genów
eukariotycznych na poziomie
transkrypcji
Elementy systemu regulacji:
Polimerazy RNA
polimeraza I: synteza 18S, 5.8S, 26S rRNA
polimeraza II: większość białek, wszystkie mają
czapeczkę (GTP 5’-5’)
polimeraza III: synteza małych cząsteczek RNA (tRNA,
5S rRNA,
snRNA)
Czynniki transkrypcyjne
Promotory (sekwencje TATA i CAAT)
Sekwencje wzmacniające (enhancery)
Sekwencje osłabiające (silencery)
Regulacja ekspresji niektórych genów
eukariotycznych jest pozornie podobna do
prokariontów.
A. Regulon fosforanowy u Neurospora crassa
Geny regulatorowe
Geny struktury
PHO-2
Brak PO
4
-2
NUC-2 PREG NUC-1
PHO-3
PVOG
- -
+
+
-
Pho-2 – reprymowalna alkaliczna fosfataza
Pho-3 – kwaśna fosfataza


http://www.pubmedcentral.nih.gov/pagerender.fcgi?artid=361368&pageindex=1

http://www.pubmedcentral.nih.gov/pagerender.fcgi?artid=359321&pageindex=1

Regulacja ekspresji niektórych genów
eukariotycznych jest pozornie podobna do
prokariontów.
B. Biosynteza aminokwasów aromatycznych u Neurospora
crassa
Arom1 – reduktaza kwasu dehydroszikimowego (1)
Arom9 – dehydrogenaza kwasu dehydroszikimowego (2)
Arom5 – kinaza kwasu szikimowego (3)
Arom4 – syntetaza kwasu enolopirogronylofosfoszikimowego
(4)
Arom2 – syntetaza kwasu dehydrohinowego
Kwas fosfoenolopirogronowy + erytrozo-4-fosforan kwas 3-
deoksyarabinozoheptulozofosforanowy
5 2 1
3
Kwas dehydrohinowy kwas dehydroszikimowy
kwas szikimowy
4
Kwas fosfoszikimowy kwas 3-enolopirogronylofosfoszikimowy
Kwas choryzmowy
*)
*)
prekursor aminokwasów aromatycznych
Arom1 Arom9 Arom5 Arom4
Arom2
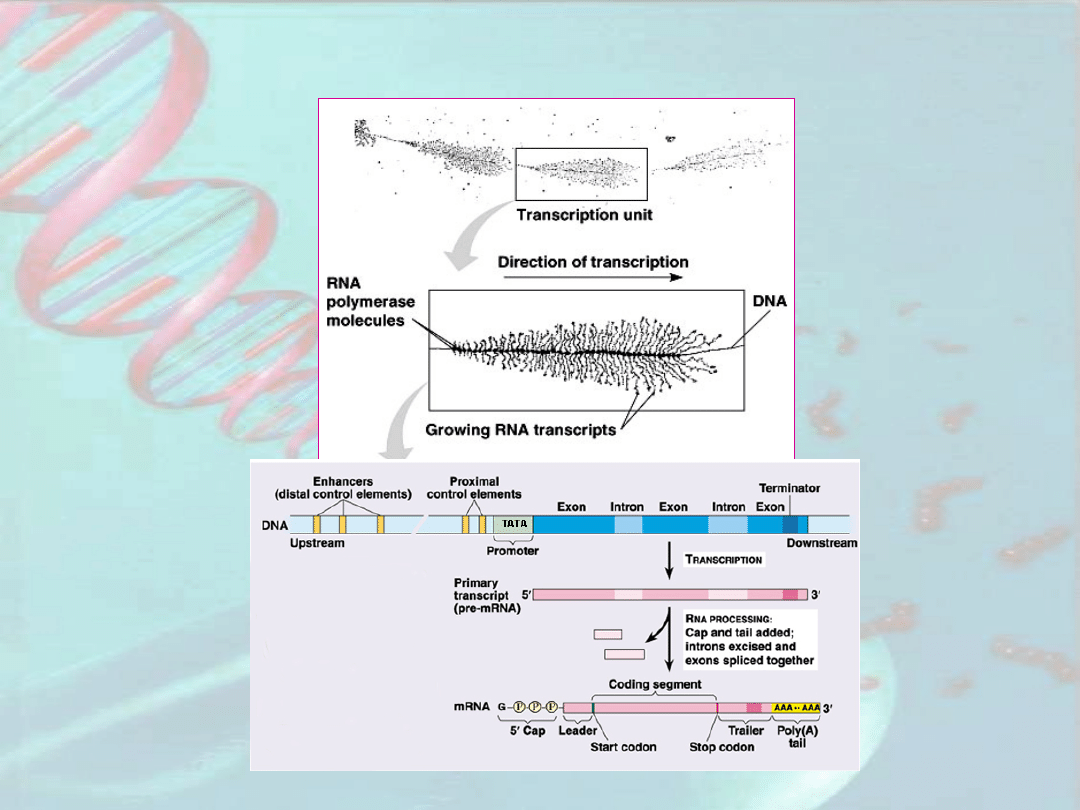
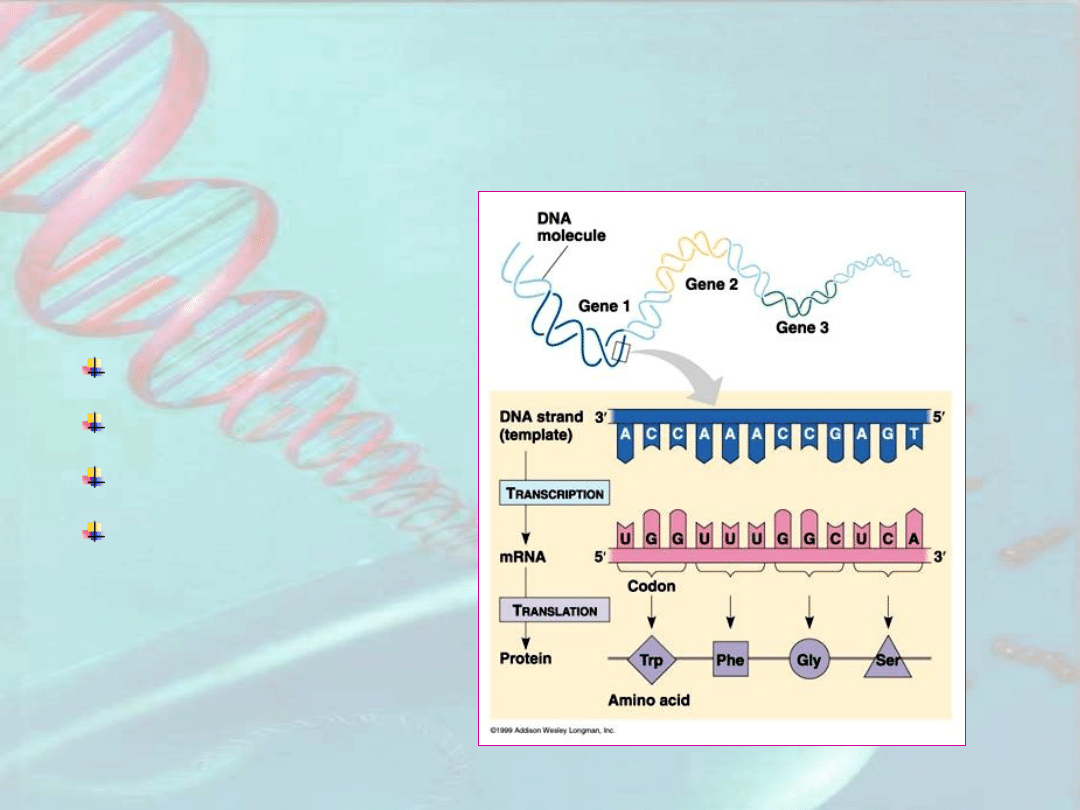
Transkrypcyja genów
eukariotycznych
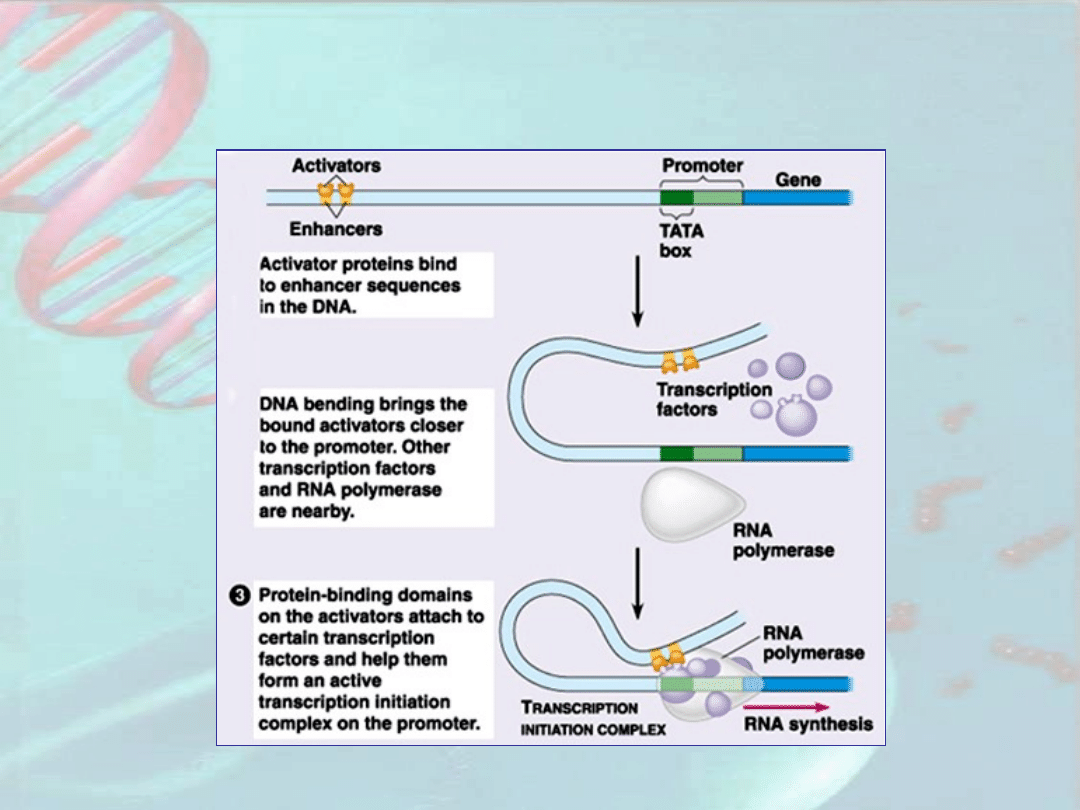
Transkrypcyja genów eukariotycznych
- utworzenie kompleksu
transkrypcyjnego
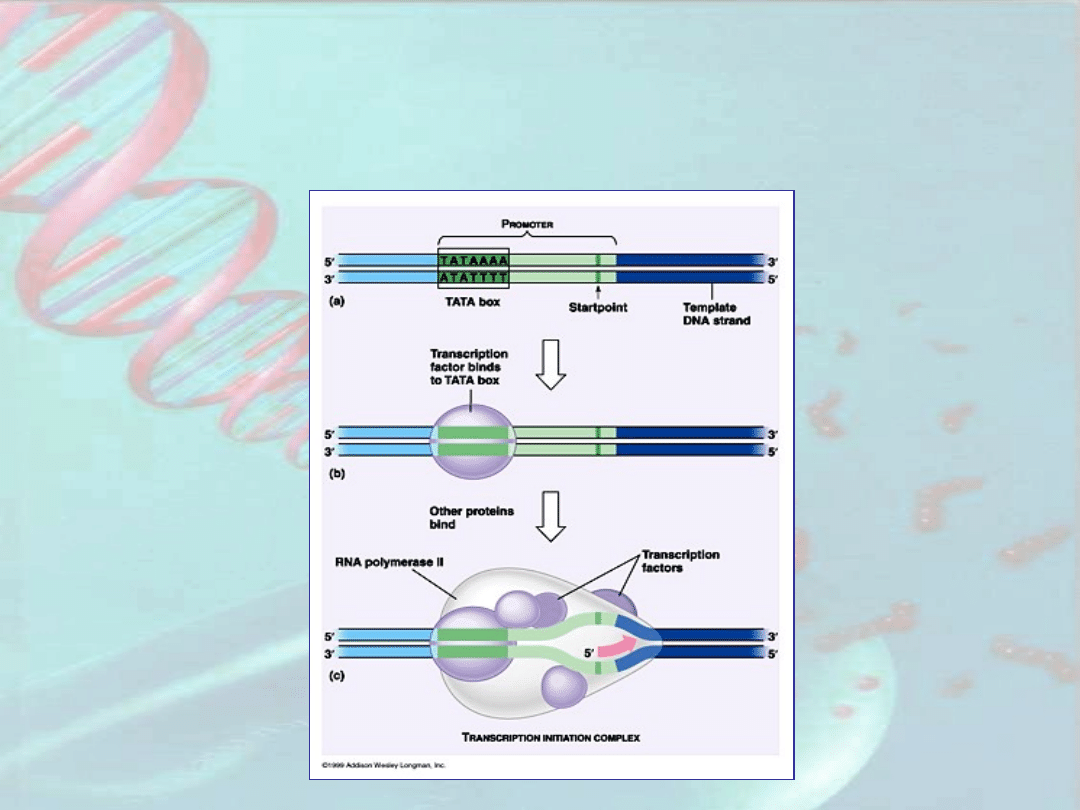
Czynniki transkrypcyjne
(„TF-y” od ang. transcriptinal
factors)
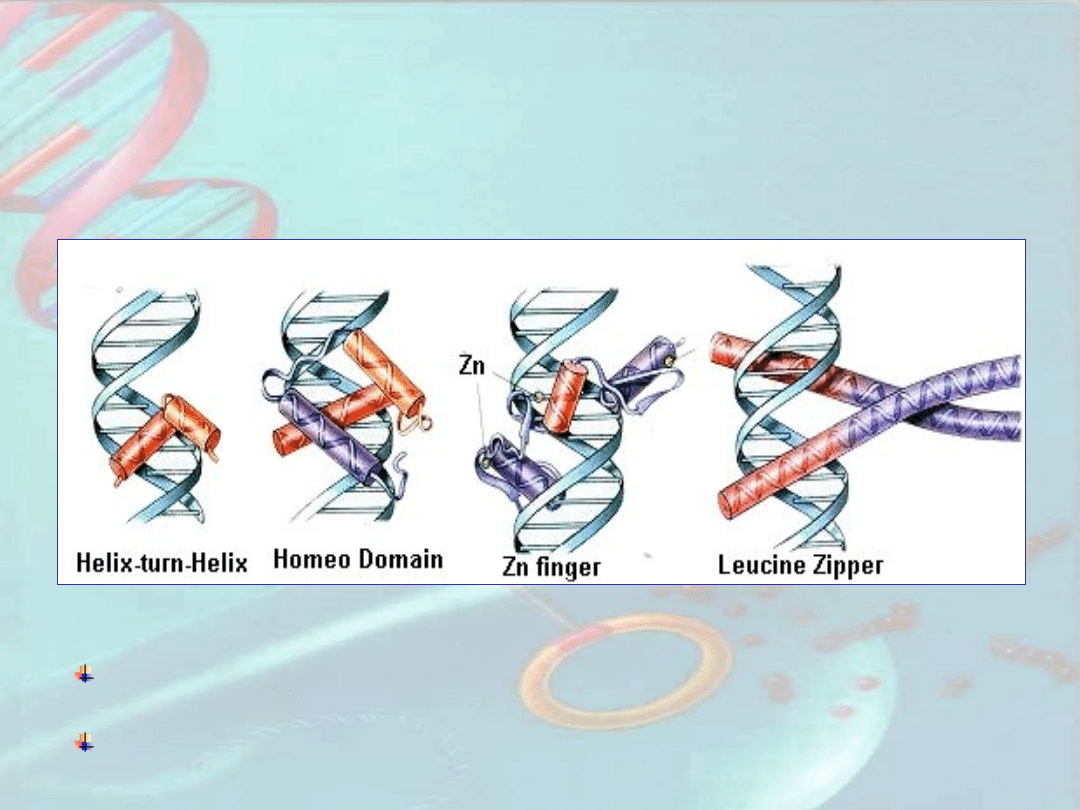
Budowa czynników
transkrypcyjnych
Domeny wiążace DNA: helisa-zwrot-helisa, palce cynkowe,
helisa-pętla-helisa
Domeny odpowiedzialne za dimeryzację: suwak leucynowy,
helisa-pętla-helisa
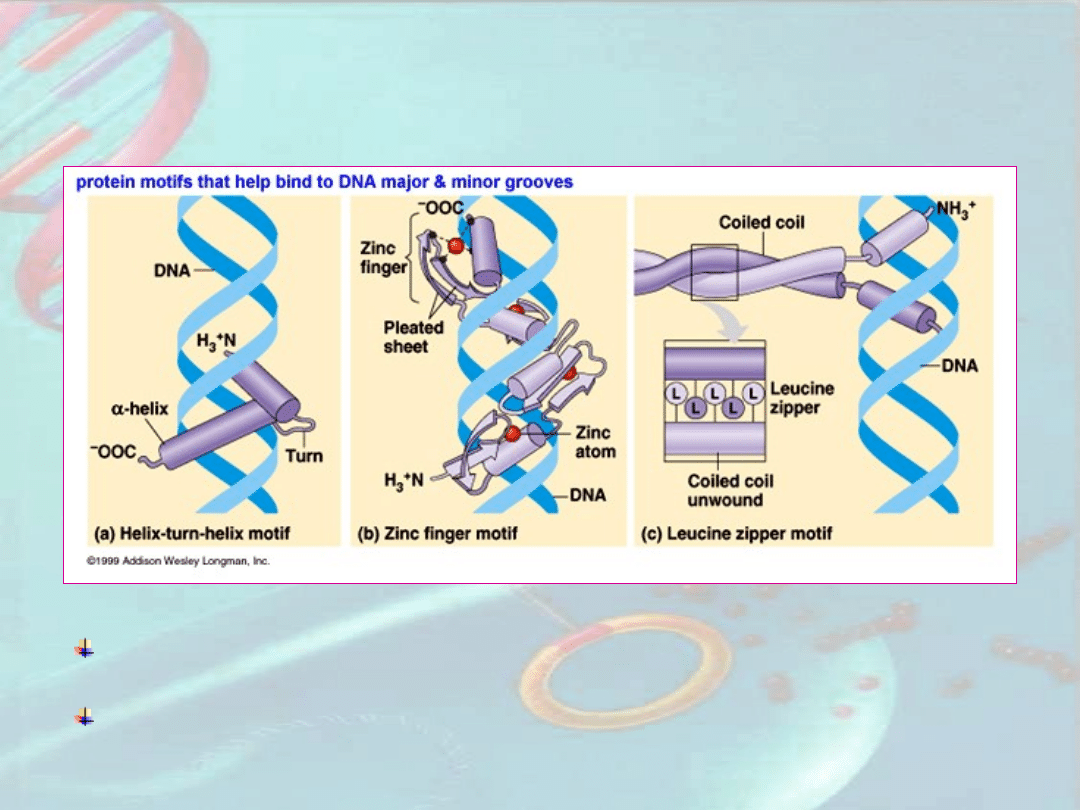
Budowa czynników
transkrypcyjnych
Domeny wiążace DNA: helisa-zwrot-helisa, palce cynkowe,
helisa-pętla-helisa
Domeny odpowiedzialne za dimeryzację: suwak leucynowy,
helisa-pętla-helisa
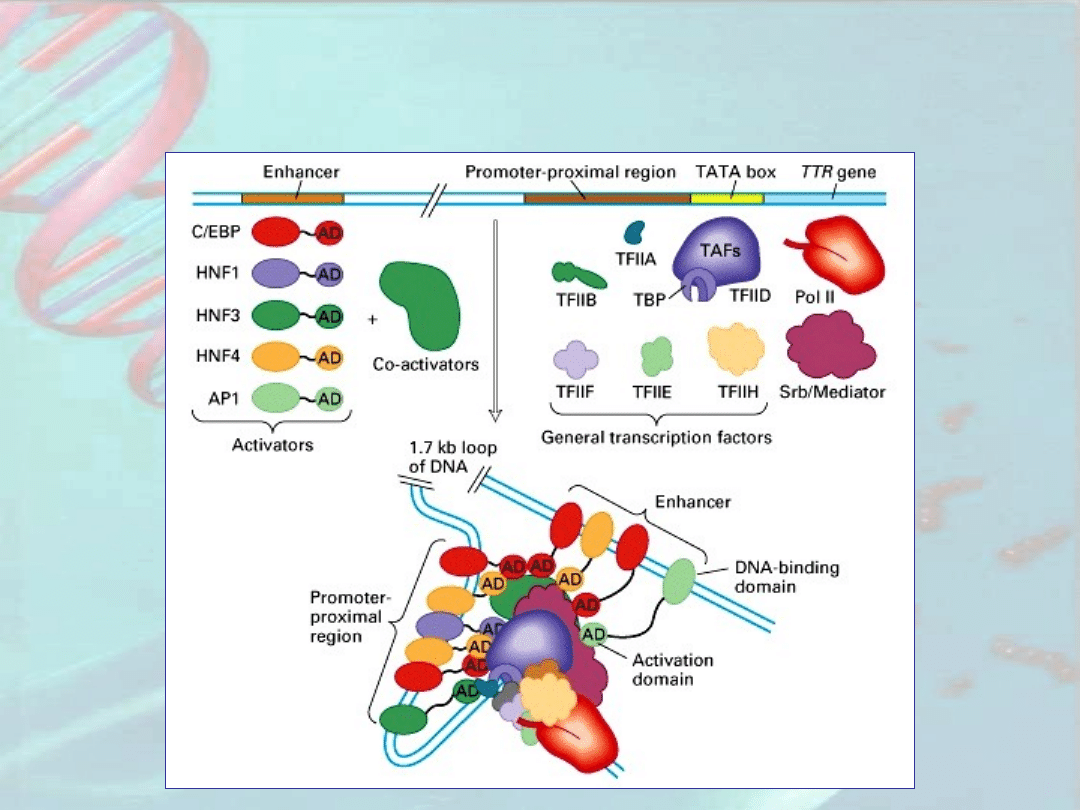
Udział sekwencji wzmacniających
w regulacji
ekspresji genów eukariotycznych
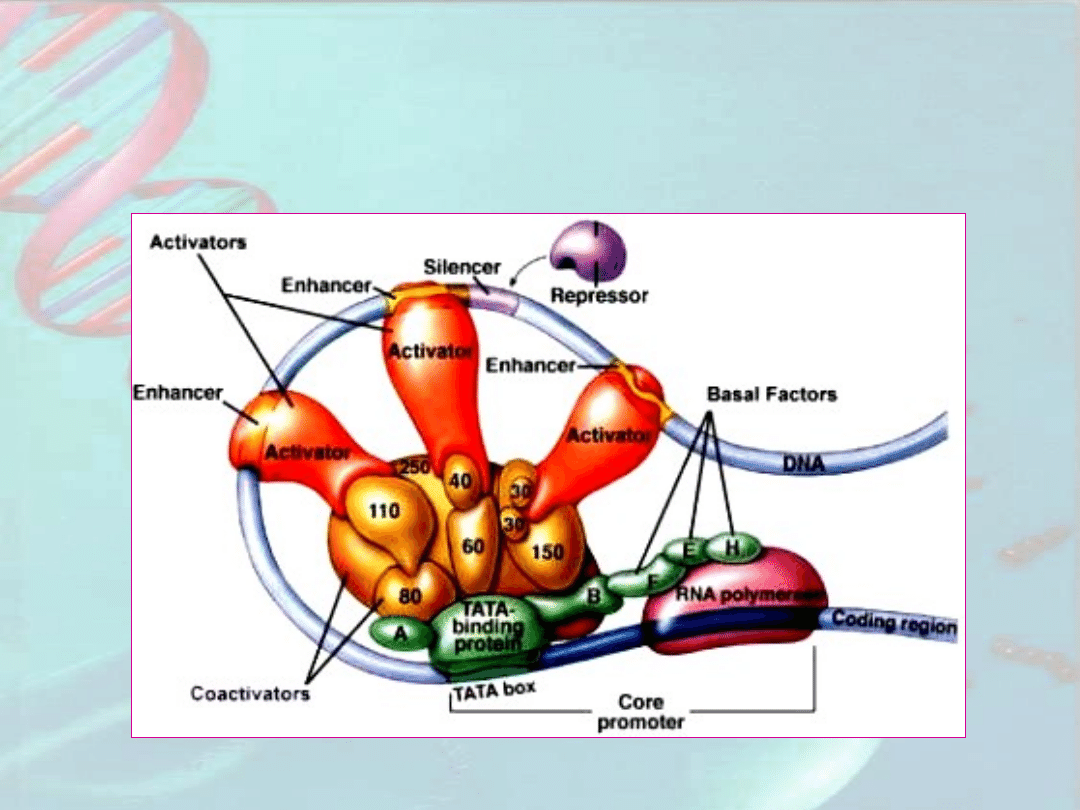
Udział sekwencji wzmacniających i
osłabiających
w regulacji ekspresji genów
eukariotycznych
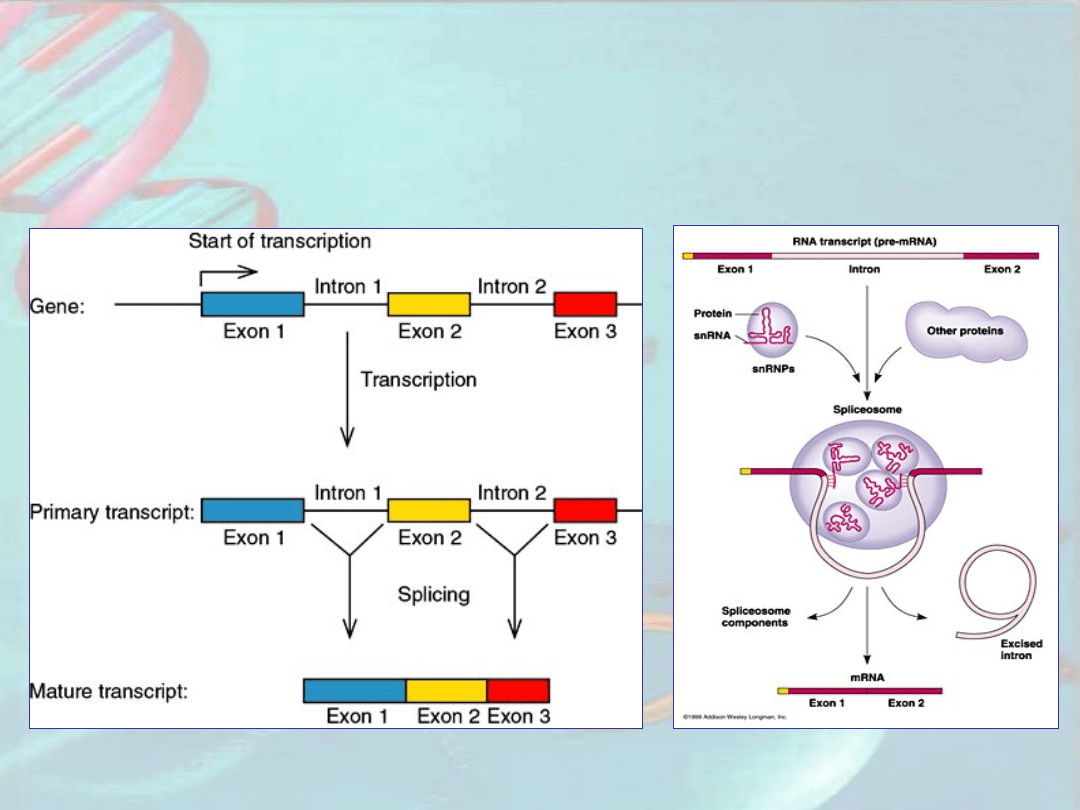
Regulacja ekspresji genów
eukariotycznych
po transkrypcji
A. Dojrzewanie (składanie) transkryptu
Regulacja ekspresji genów
eukariotycznych
po transkrypcji
A. Dojrzewanie (składanie) transkryptu

Regulacja ekspresji genów
eukariotycznych
po transkrypcji
A. Dojrzewanie (składanie) transkryptu
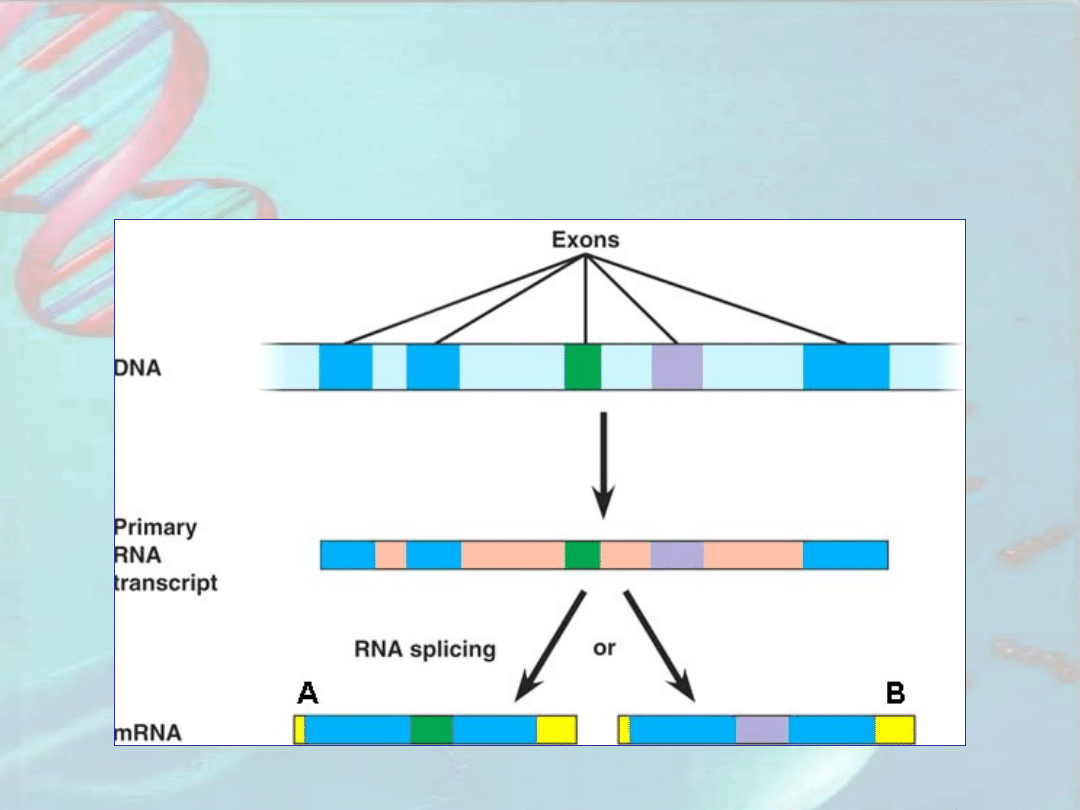
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
B. Alternatywne składanie transkryptu
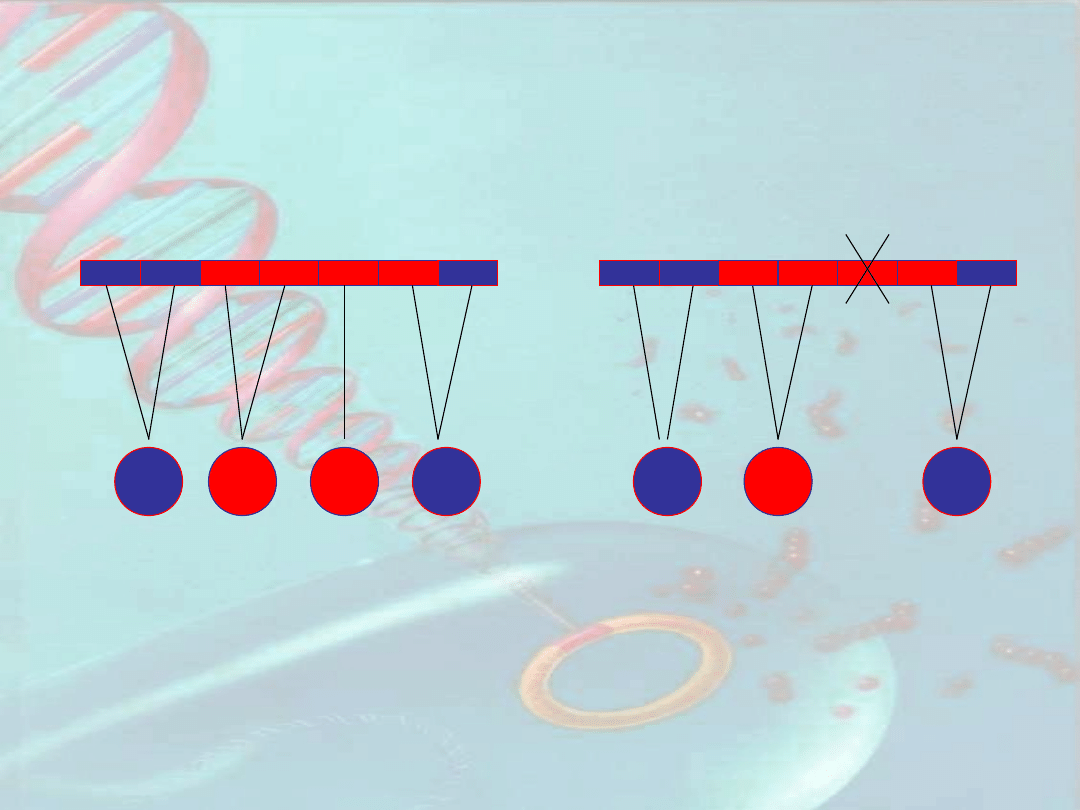
1 2 3 4 5 6 7 1 2 3
4 5 6 7
Fibronektyna powierzchni
komórki
Fibronektyna
osoczowa
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
B. Alternatywne składanie transkryptu
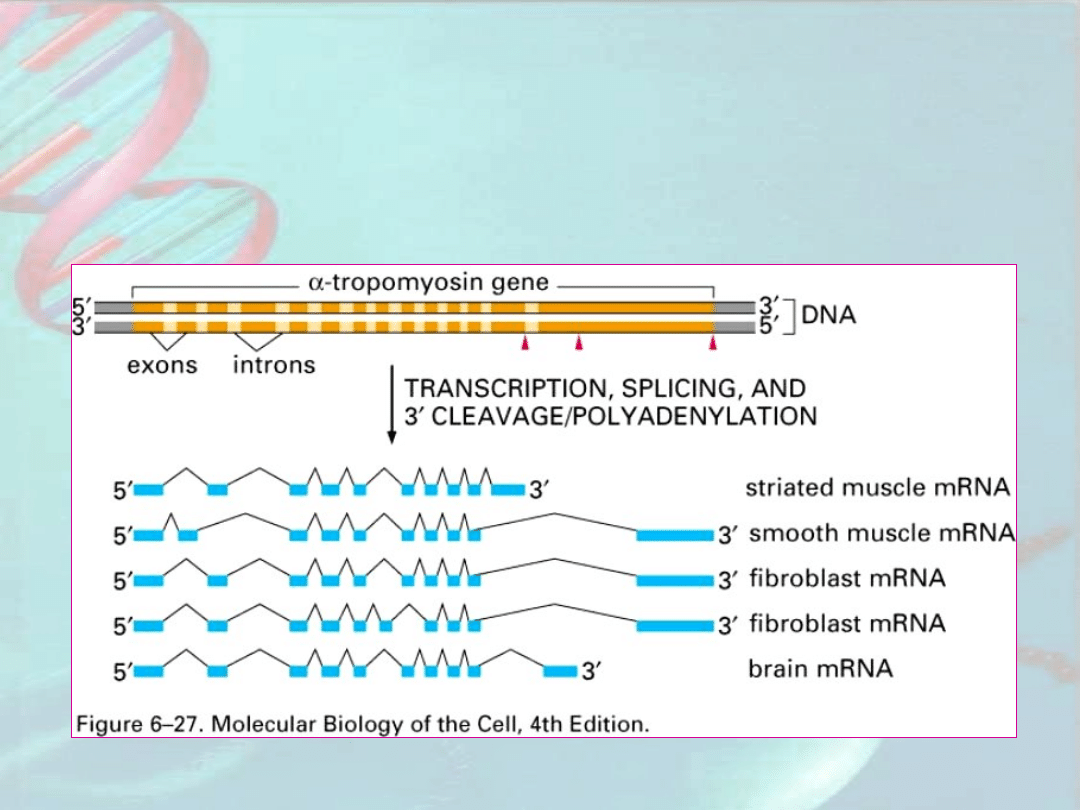
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
B. Alternatywne składanie transkryptu
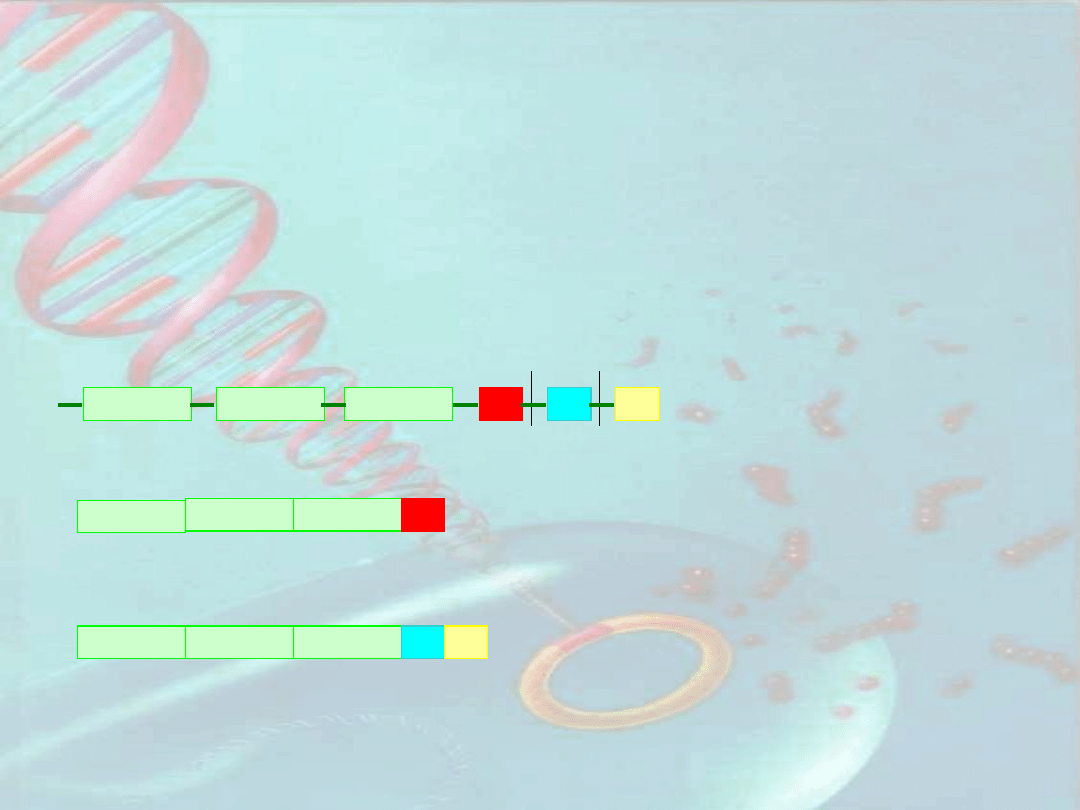
E1
E2
E3
I1
I2
I3
E1
E1
E3
E2
E2
E3
I1
I2
I3
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
B. Wybór miejsca terminacji transkrypcji (jednego z
kilku możliwych
miejsc poliadenylacji)
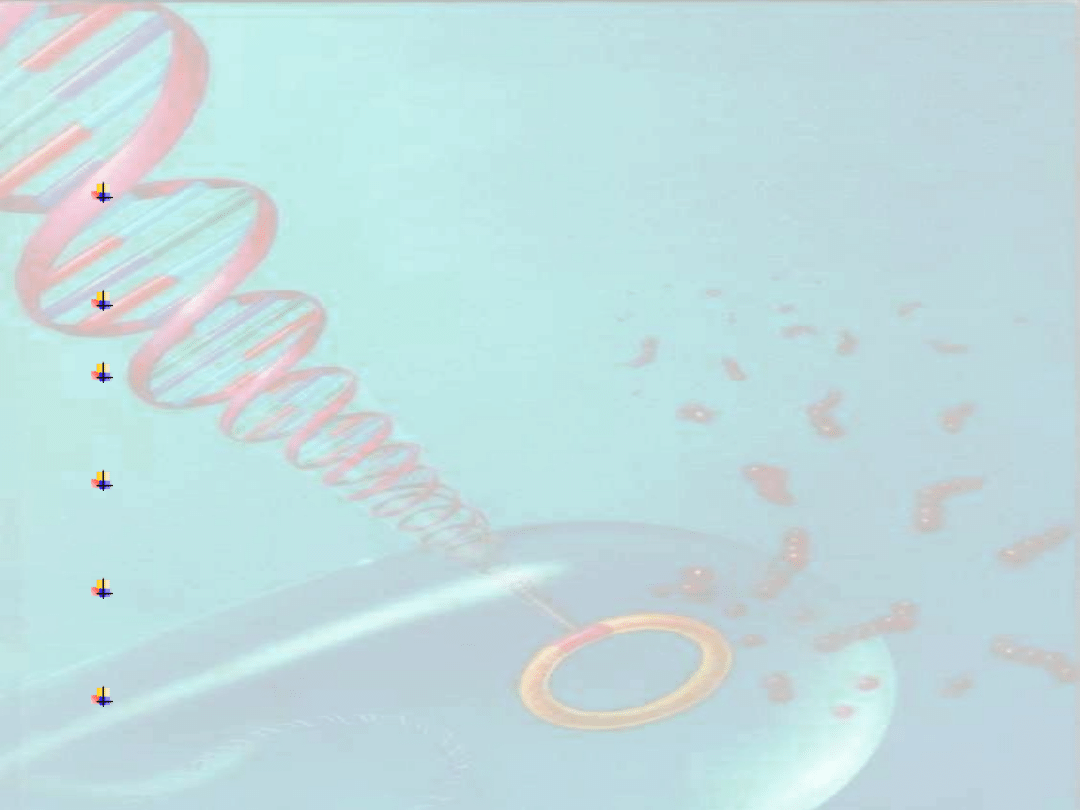
Redagowanie występuje głównie w mt mRNA i ct
mRNA i ma
charakter losowy
Redagowanie może dotyczyć od 0.8% do 5.8%
nukleotydów
Dotychczas odnotowano ok. 300 przypadków
redagowania
transkryptów
Redagowanie najczęściej dotyczy kodonów dla
aminokwasów
Pro - Leu, Ser - Leu, Ser – Phe i Trp - Arg.
Inne zmiany: start kodon - treonina
(
AUG - ACG i
odwrotnie); stop
kodony mogą powstać z kodonów CAG, CAA i CGA.
Nie odnotowano przypadków redagowania
strukturalnego RNA,
np. tRNA czy rRNA
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
C. Zmiany sekwencji transkryptu (redagowanie,
editing)
Typ redagowania
Występowanie
Insercja i delecja jednego
lub więcej U
Zamiana C na U
Zamiana A na G
Dodanie dodatkowych C
Dodanie dodatkowych G
mt mRNA u pierwotniaków
Trypanosoma
i Leihmania
mRNA apolipoproteiny B u ssaków,
mt mRNA
u roślin
mRNA receptora kwasu
glutaminowego w mózgu
ssaków
mRNA mitochondrialnego genu
kodującego
podjednostkę syntazy ATP u
Physarium
Niektóre geny paramyksowirusów
Potranskrypcyjna regulacja ekspresji
genów eukariotycznych
C. Zmiany sekwencji transkryptu - przykłady
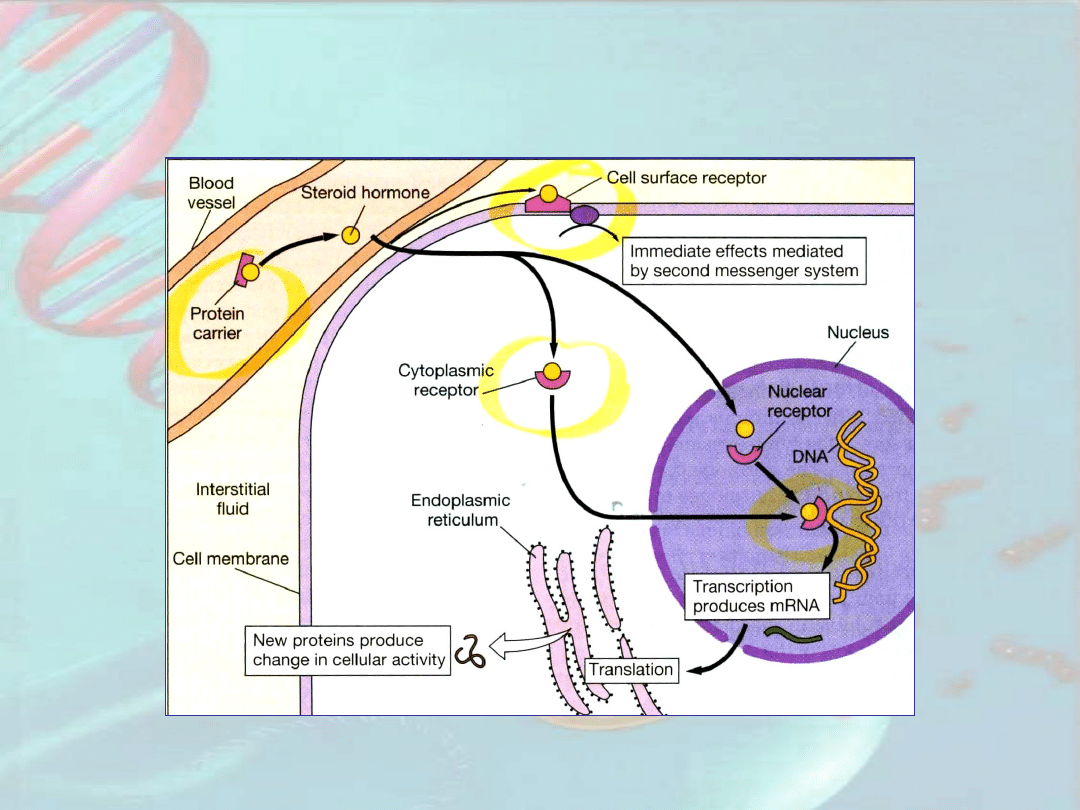
Regulacja ekspresji genów z udziałem
hormonów
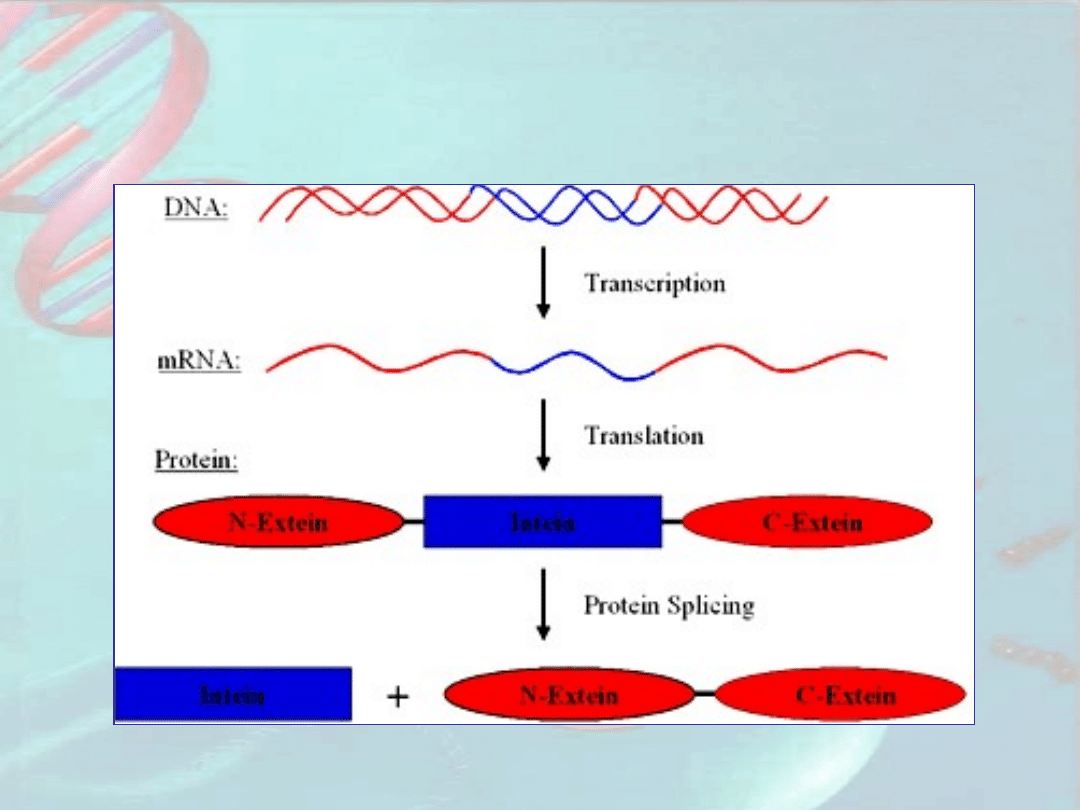
Regulacja ekspresji genów eukariotycznych
po translacji.
A. Składanie pierwotnych produktów translacji
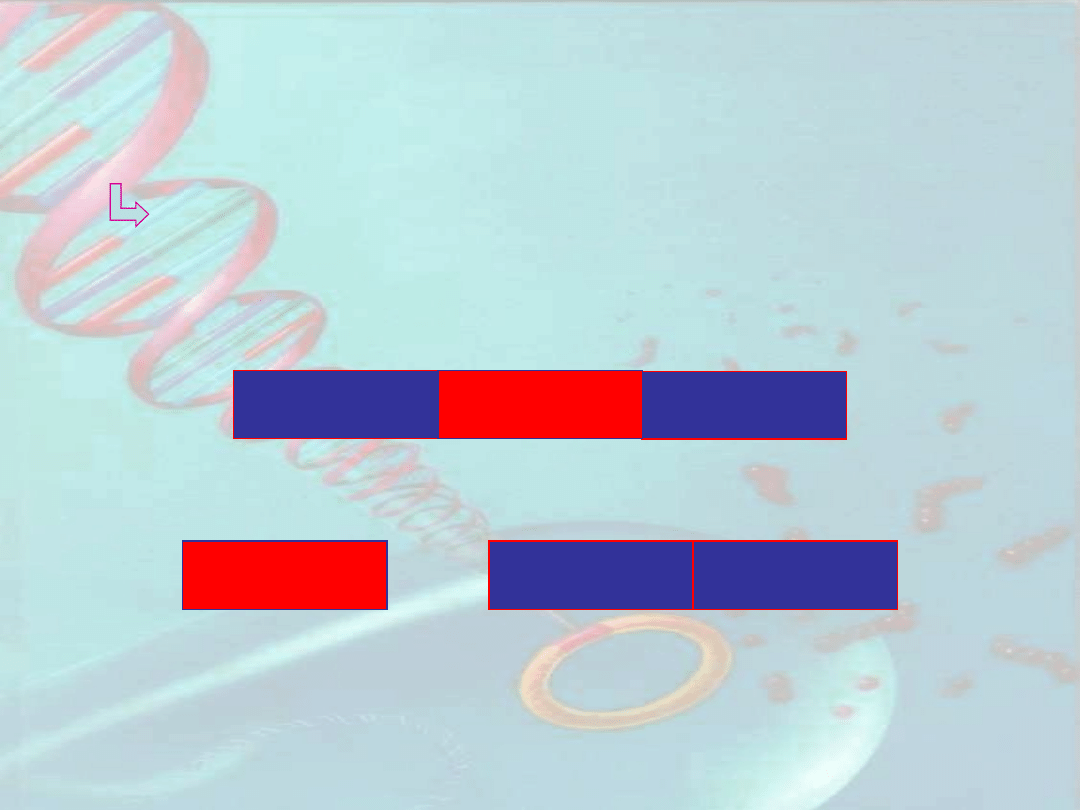
Regulacja ekspresji genów eukariotycznych po
translacji.
A. Składanie pierwotnych produktów translacji
Prekursor białkowy
119 kDa
N-eksteina
inteina
C-eksteina
Endonukleaza 50
kDa
Podjednostka ATPazy
69 kDa
U drożdży dwa różne białka powstają z prekursora
białkowego kodowanego przez gen TFP1
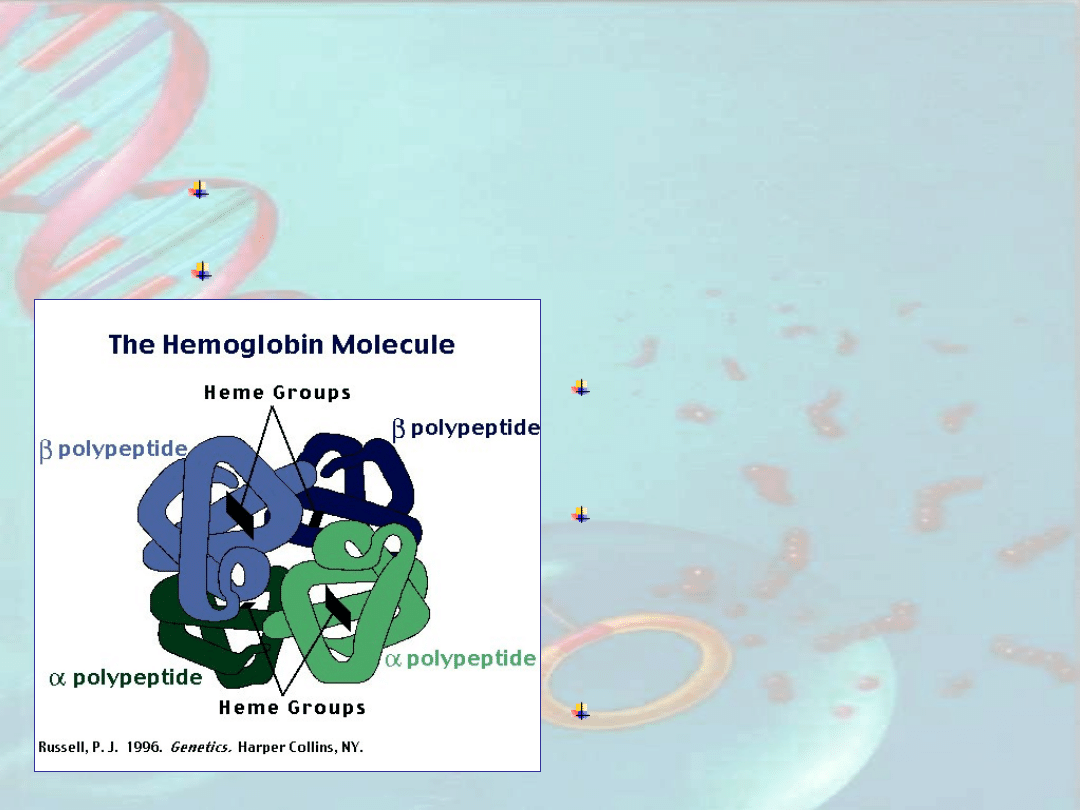
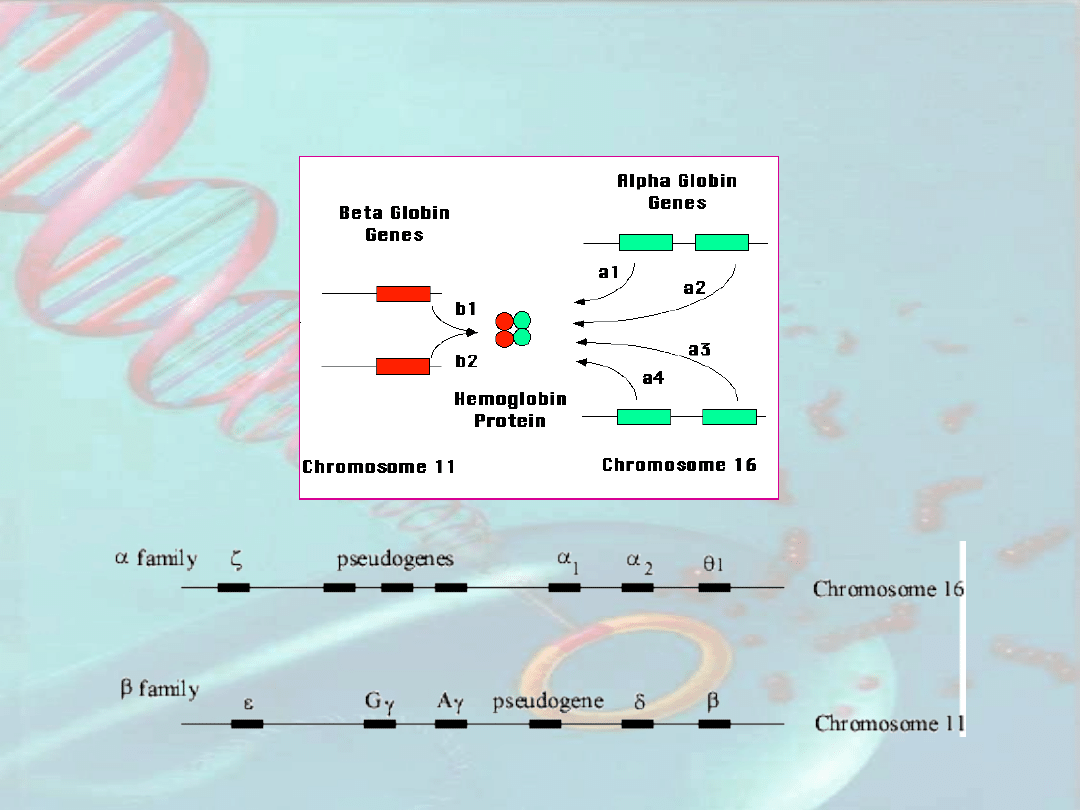
Regulacja ekspresji genów
globinowych
Globiny są apoenzymami, które po połączeniu
z cząsteczką hemu wytwarzają hemohlobinę
Hemoglobiny embrionalne i postembrionalne
różnią się powinowactwem do O
2
Hemoglobiny postembrionalne:
Hemoglobina A (HbA) = 2
łańcuchy
-globiny + 2 łańcuchy -globiny
+ hem
Hemoglobina A2 (HbA2) = 2
łańcuchy
-globiny + 2 łańcuchy -globiny
+ hem
Hemoglobiny embrionalne:
Hemoglobina E (HbE) - łańcuch
zamiast
(wczesnoembrionalna; do 6-tego
tyg. ciąży)
Hemoglobina F (HbF) – łańcuch
zamiast
(późnoembrionalna; od 6-tego
tyg. ciąży)
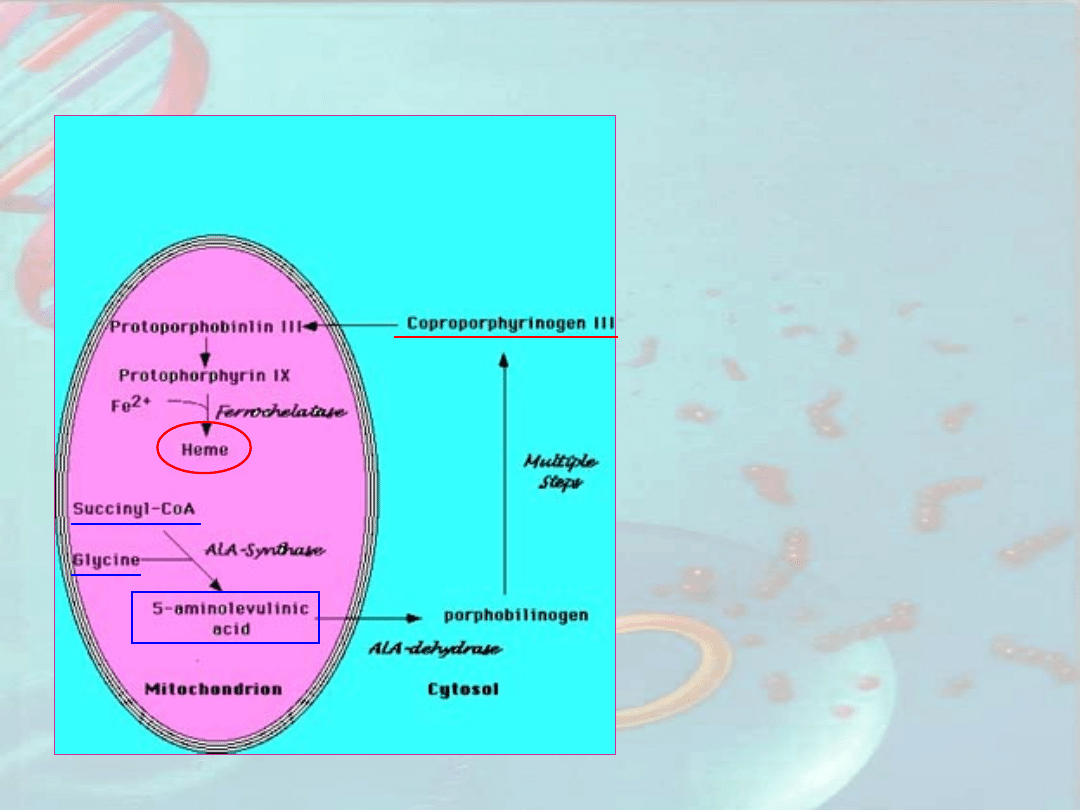
LCR
LCR
Synteza hemu jest
złożonym, wieloetapowym
procesem. Rozpoczyna się
on w mitochondrium, gdzie
następuje kondensacja
sukcynylo-CoA
i glicyny - powstaje kwas 5-
aminolewulinowy.
Następnie
w cytoplaźmie produkowany
jest koproporfyrynogen III,
który jest transportowany
do mitochondrium.
Ostatnim etapem jest
wytworzenie hemu
Biosynteza
hemu
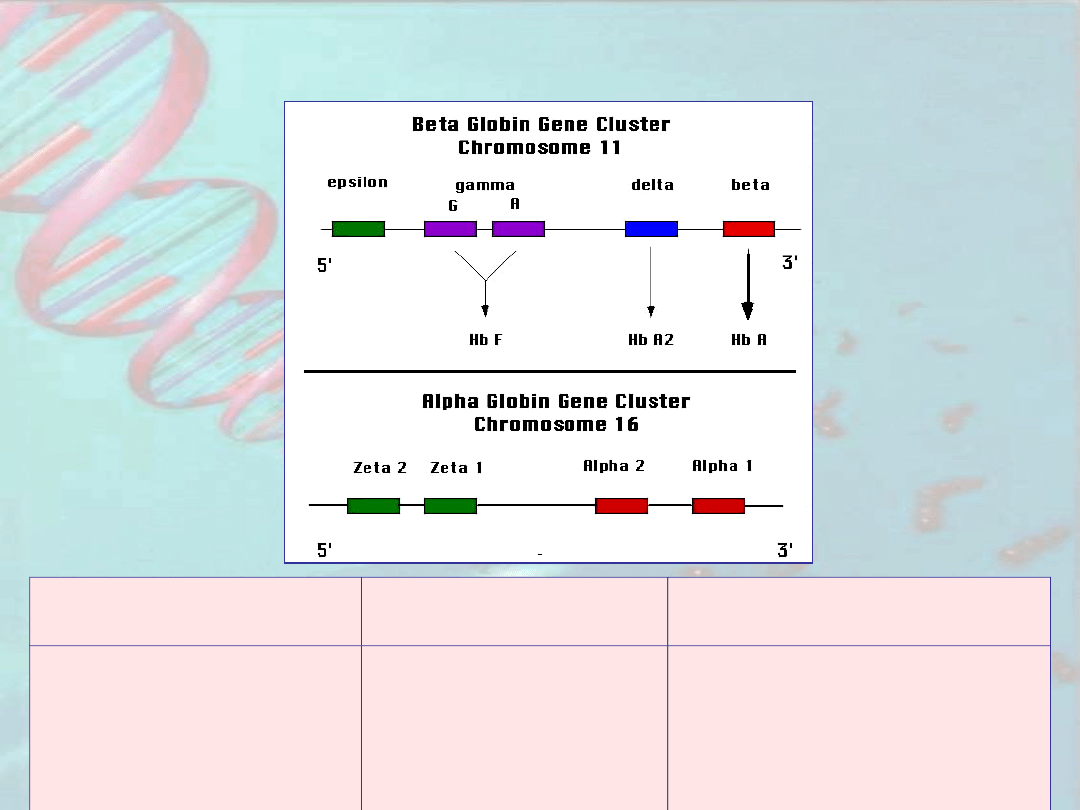
Hemoglobina
wczesnozarodkowa
Hemoglobina
późnozarodkowa
Hemoglobina dorosłego
człowieka
gower 1- zeta(2),
epsilon(2)
gower 2- alpha(2),
epsilon (2)
Portland- zeta(2),
gamma (2)
hemoglobina F-
alpha(2), gamma(2)
hemoglobina A- alpha(2),
beta(2)
hemoglobina A2- alpha(2),
delta(2) – u 3% populacji
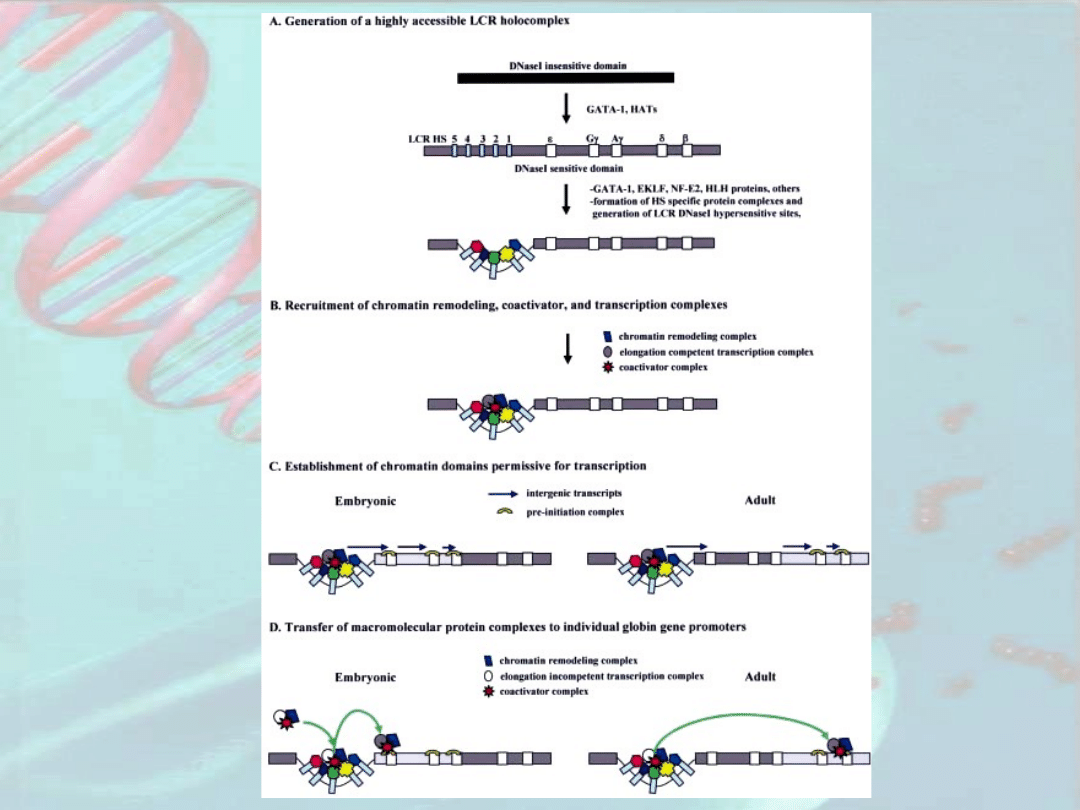
Rozwojowa regulacja ekspresji genów
globinowych
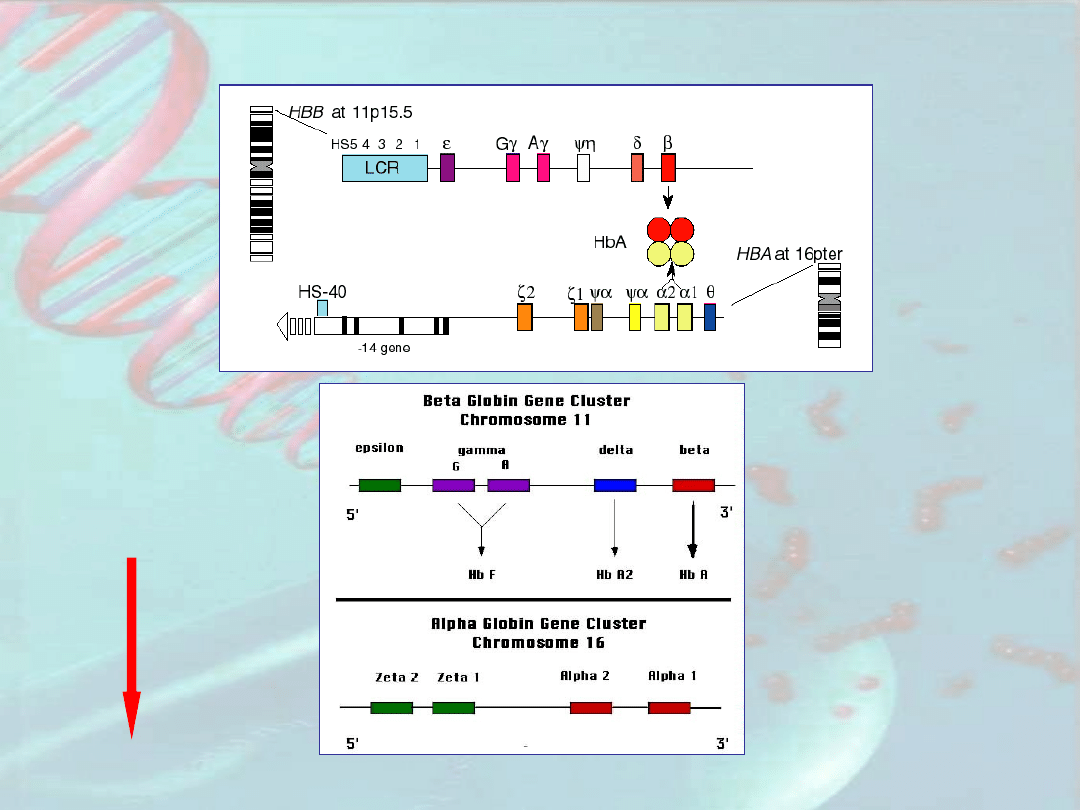
Rozwojowa regulacja ekspresji genów
globinowych
http://www.oup.com/genesvii
http://batzerlab.lsu.edu/Hum_Mol_Gen_Lectures/Levings_and_Bungert_2002_-_Bglobin_LCR_function.pdf
LCR – locus control
region
HS – hypersensitive
site
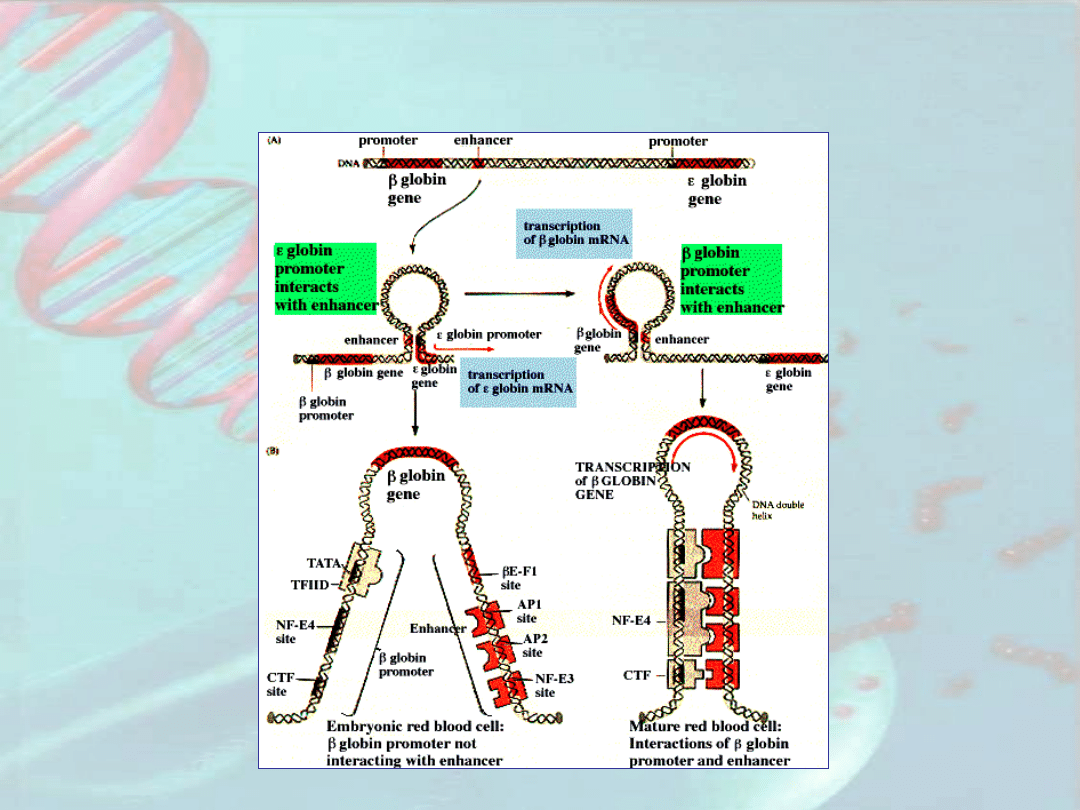
Rozwojowa regulacja ekspresji genów
globinowych
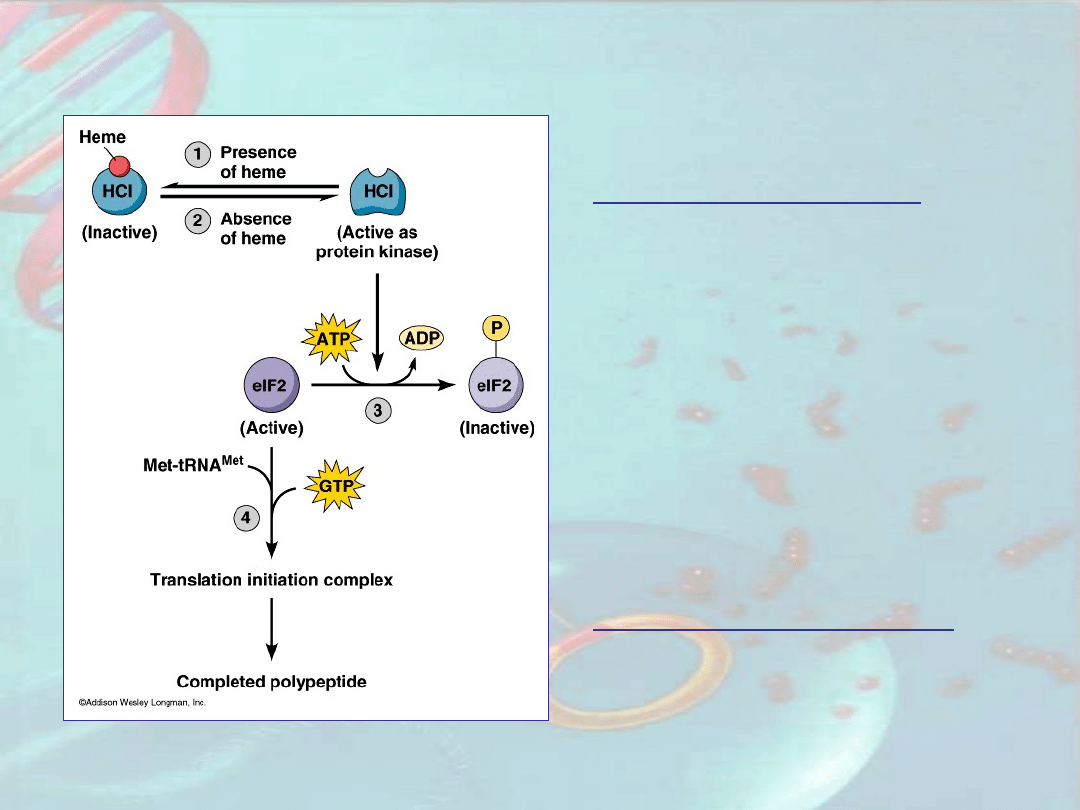
Regulacja ekspresji genów globinowych zależna od
poziomu hemu
Brak hemu (heminy*) -
przy niedoborach żelaza
powoduje wzrost syntezy
kinazy białkowej (2) i
fosforylację mniejszej
podjednostki białka eIF2
(3), białka koniecznego do
zainicjowania kompleksu
translacyjnego mRNA
genów globinowych – brak
translacji
Normalny poziom hemu
(heminy) – defosforylacja
eIF2 (4) – translacja genów
globinowych
*) hemina = hematyna – pochodna hemu zawierająca
Fe
+3
zamiast Fe
+2

Choroby wywołane substytucją
amonokwasów w łańcuchu i globiny
Substytucja
Pozycja
Nazwa Hb
Łańcuch
Ala/Asp
Gli/Asp
Liz/Glu
Gli/Asp
Glu/Arg
His/Tyr
5
15
16
22
54
87
J Toronto
J Oxford
I
J Mendluu
Shimonoseki
M Iwate
Łańcuch
Glu/Liz
Glu/Wal
Glu/Liz
Glu/Gli
His/Tyr
Hus/Arg
Liz/Glu
6
6
7
7
63
63
95
C
S
Siriraj
San Jose
M Saskatoon
Zurich
N
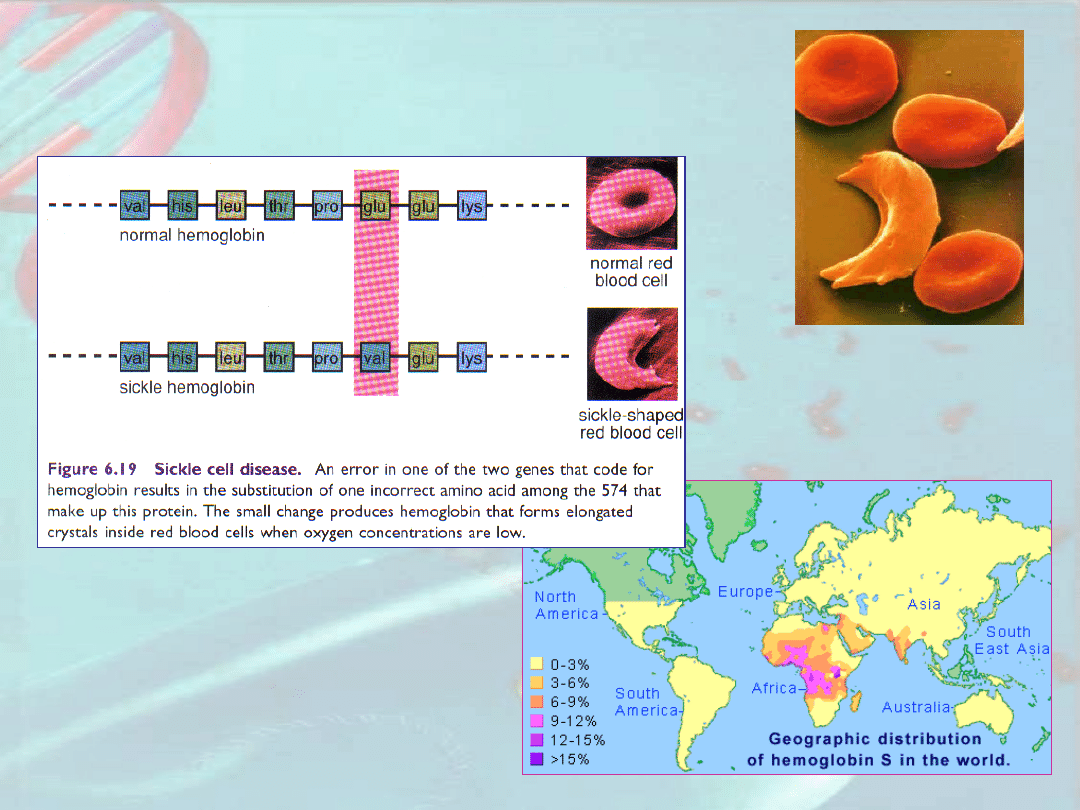
Anemia
sierpowata
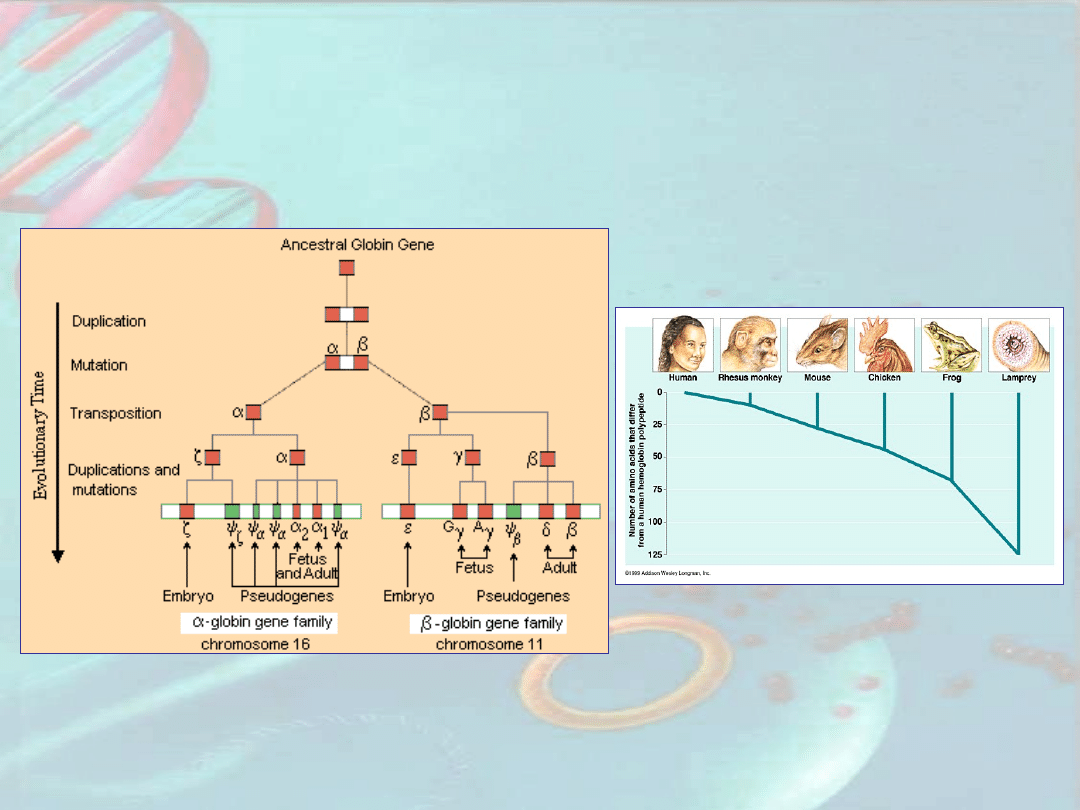
Ewolucja genów
globinowych
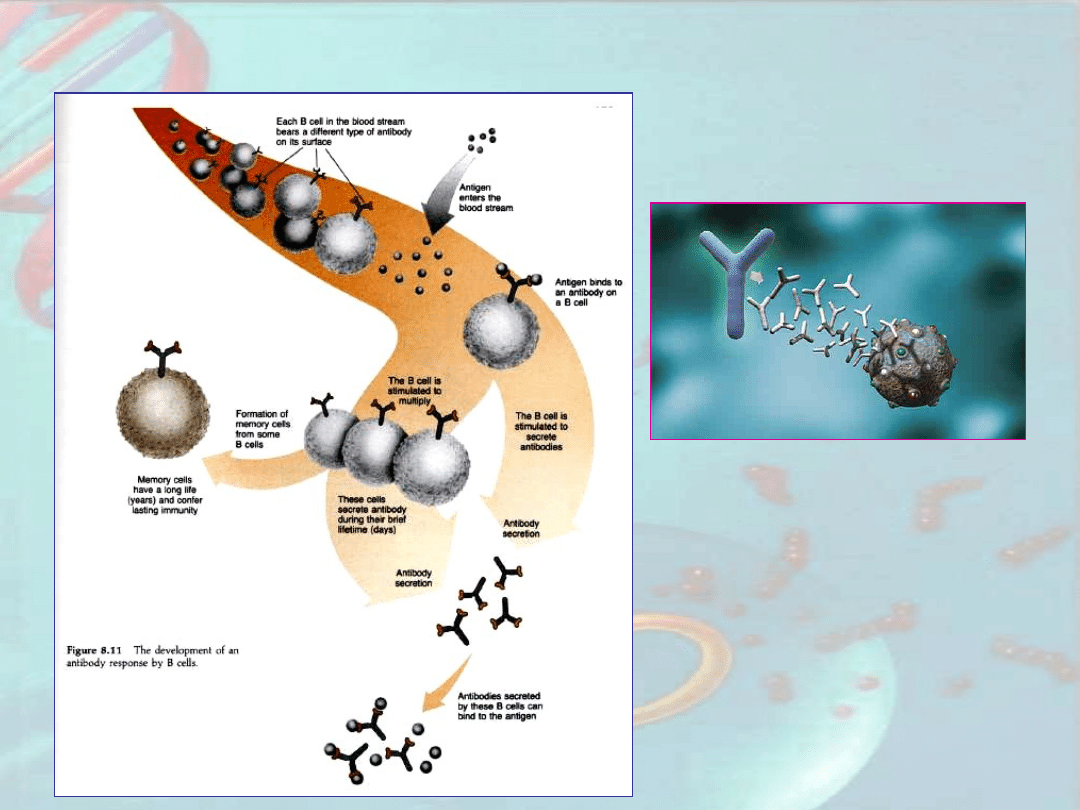
Rozwój odpowiedzi immunologicznej
limfocytów B
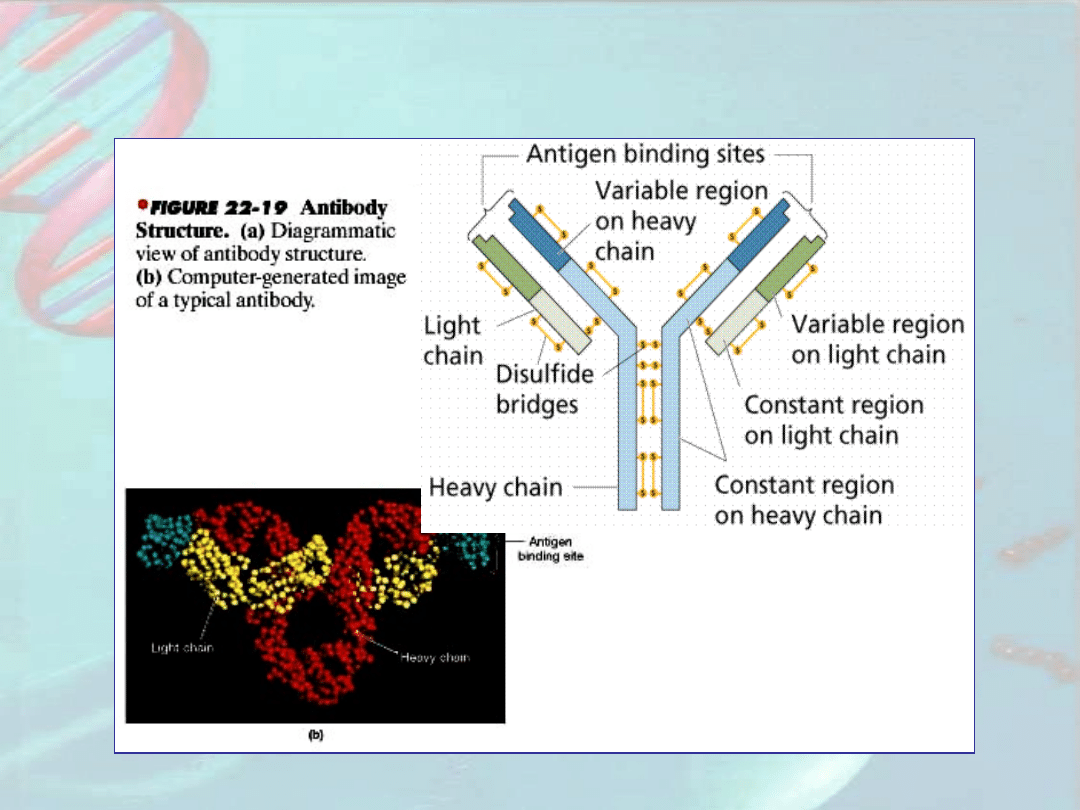
Schemat budowy
przeciwciała
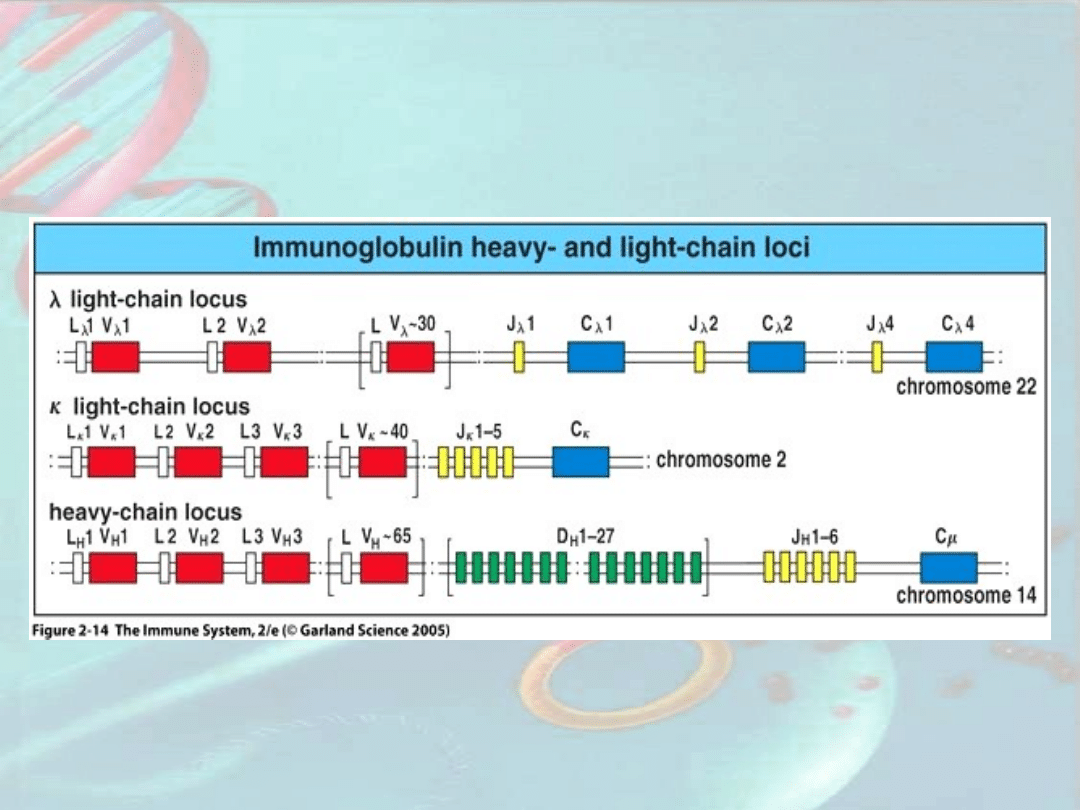
Struktura loci przeciwciał u człowieka
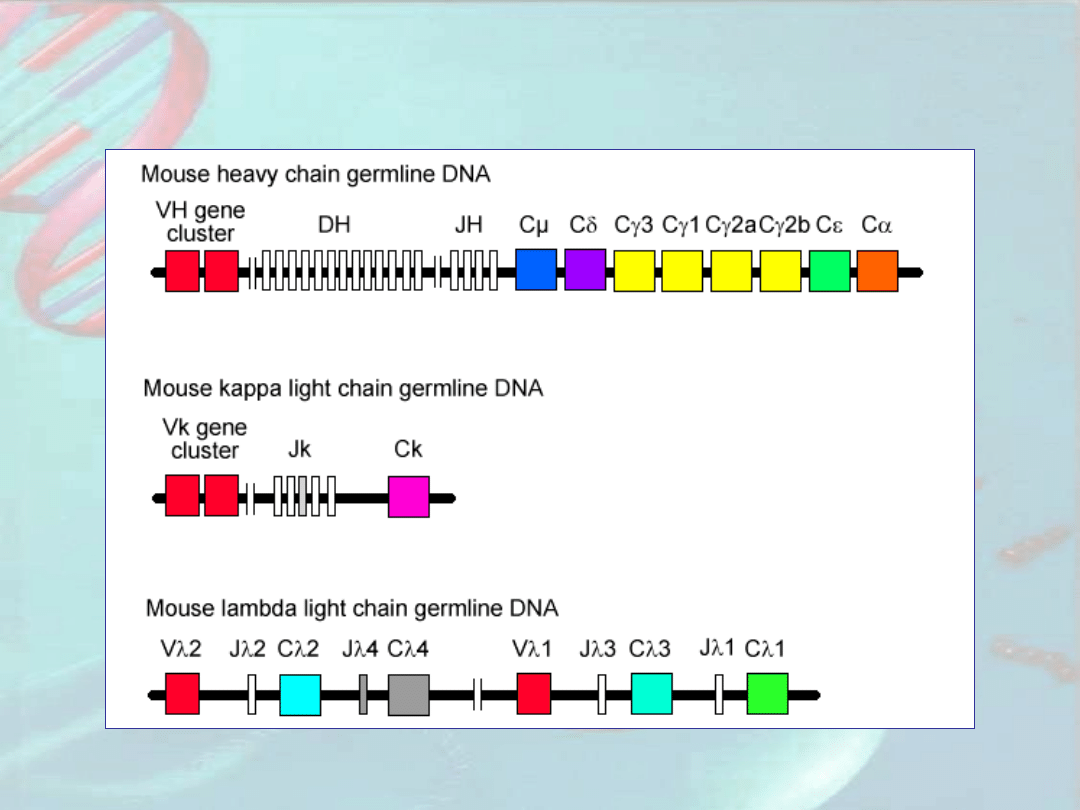
Struktura loci przeciwciał u myszy
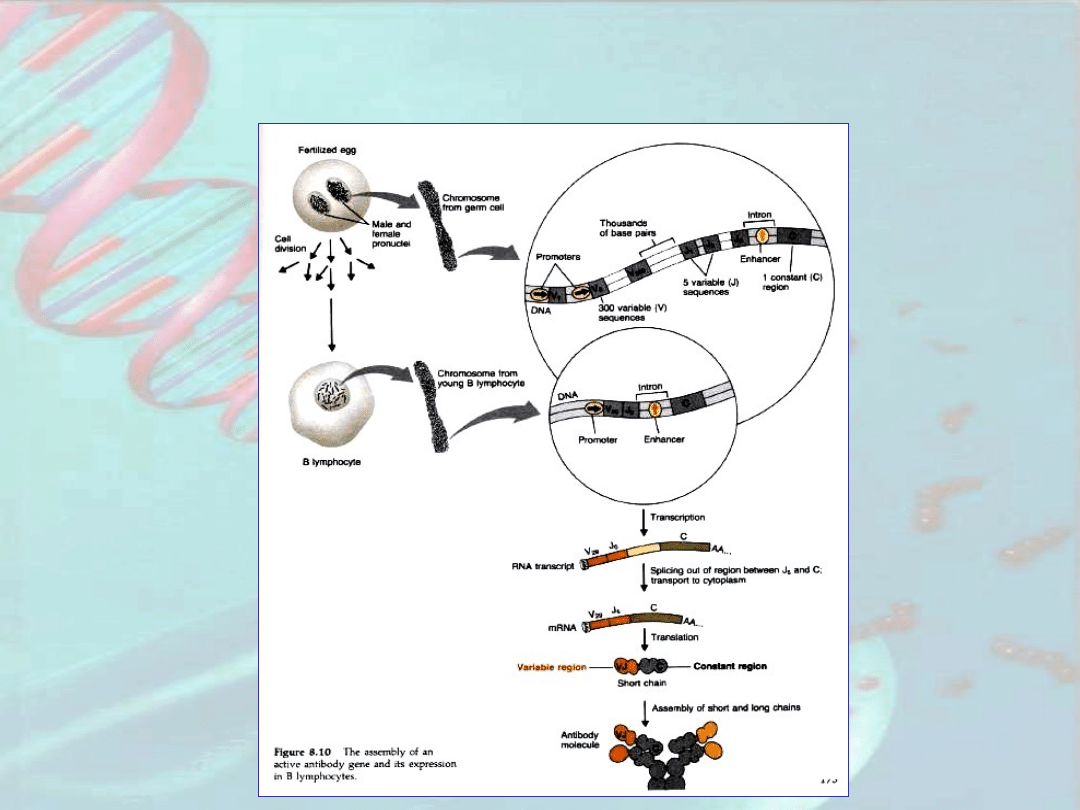
Składanie aktywnych genów
przeciwciał
i ich ekspresja w limfocytach B
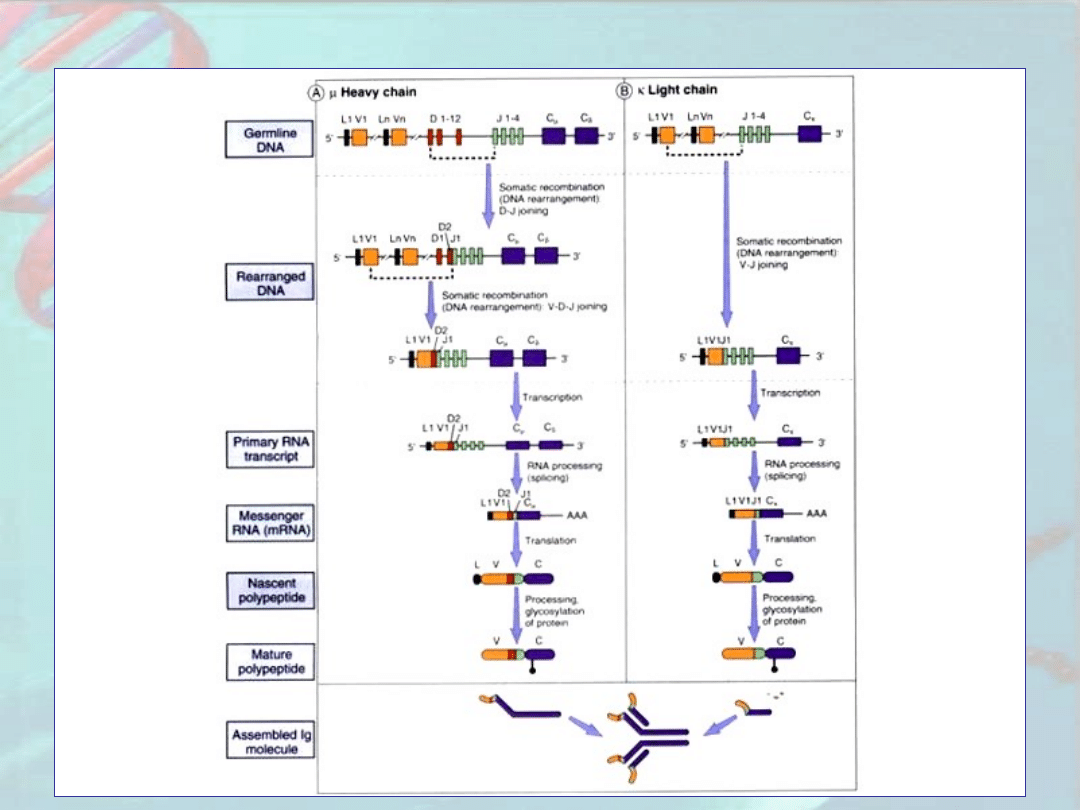
Składanie aktywnych genów przeciwciał
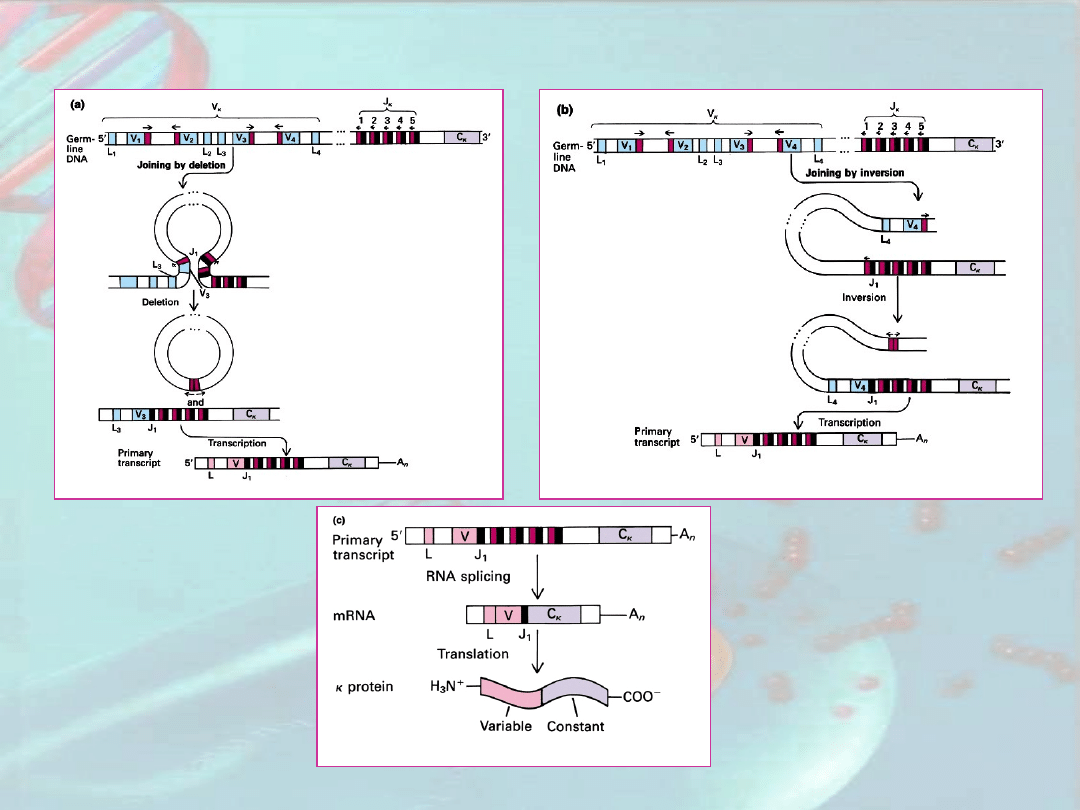
Składanie aktywnych genów
przeciwciał
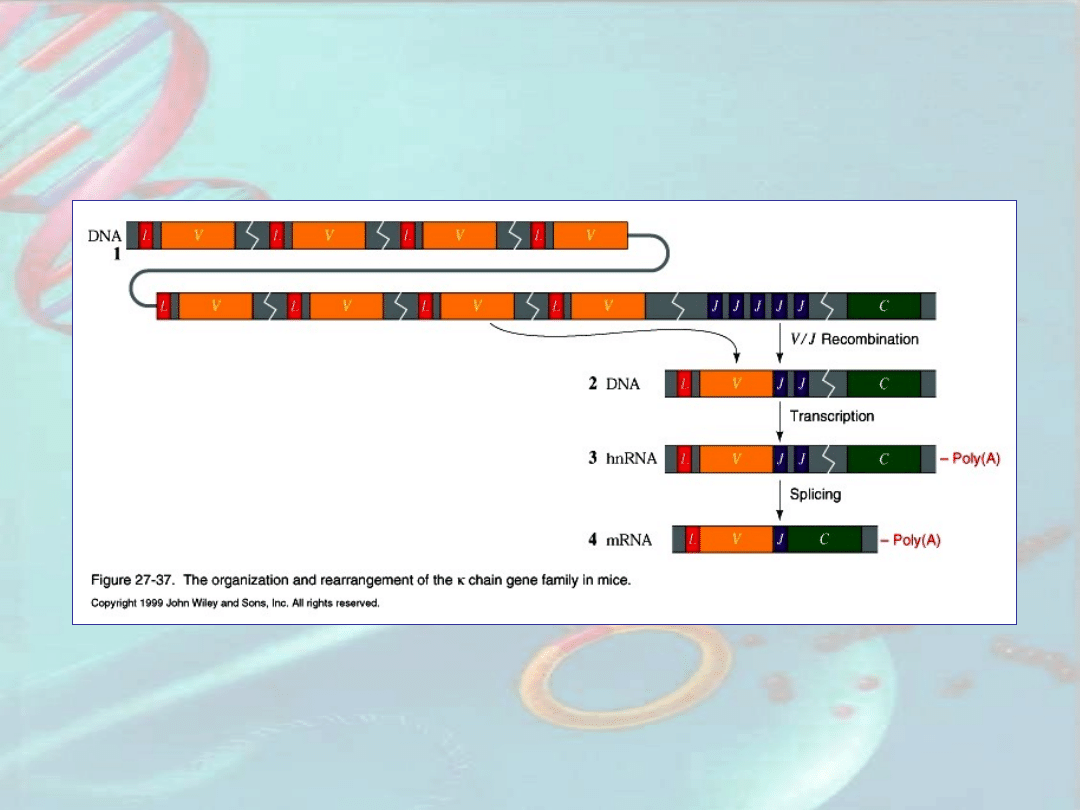
Składanie aktywnych genów przeciwciał i
ich ekspresja w limfocytach B myszy
http://www.mrc-cpe.cam.ac.uk/phage/images/germline.gif
Łańcuchy lekkie: 300 sekwencji V, 5
sekwencji J, do 10 możliwości składania V- J –
15.000 kombinacji
Łańcuchy ciężkie: 80 sekwencji V, 50
sekwencji D,
6 sekwencji J, do 100 możliwości składania V-
D i D-J –
2.4 mln. kombinacji
15.000 x 2.4 mln. -
36 bilionów
różnych przeciwciał
Regulacja ekspresji genów
eukariotycznych
z udziałem zjawiska RNAi
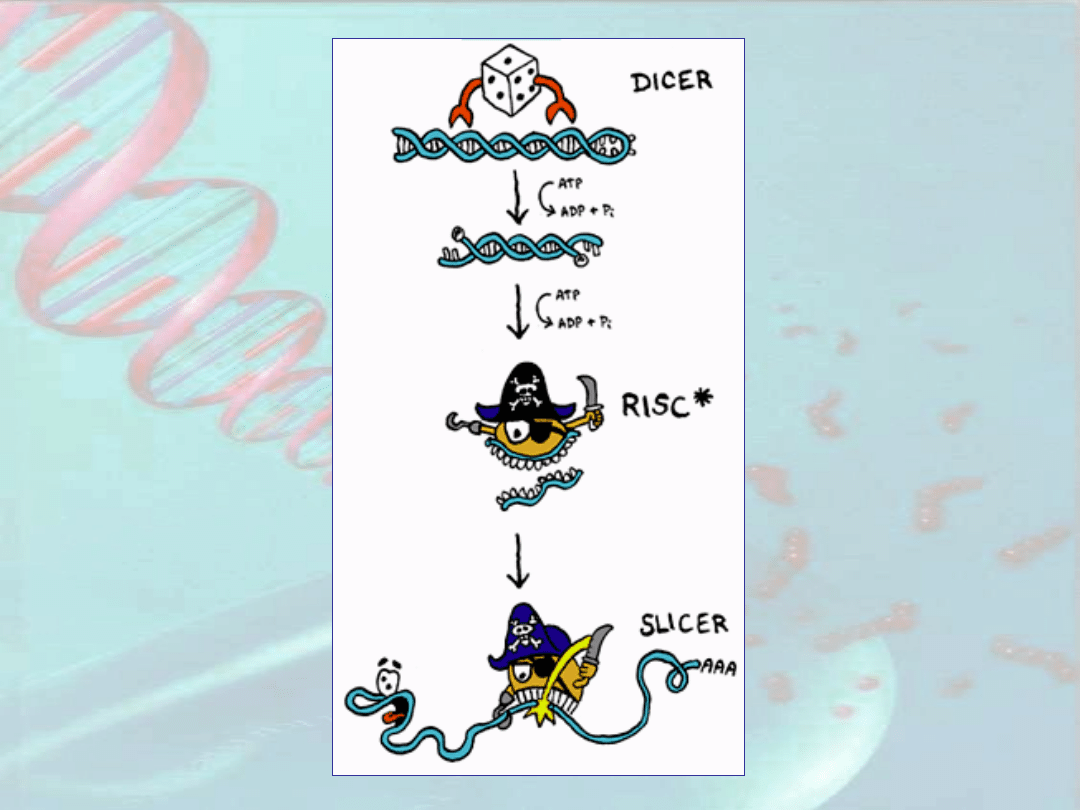
1.Charakterystyka zjawiska
Proces mający na celu wykrycie i inaktywację kwasów
nukleinowych, których aktywność może w jakiś sposób
zagrozić komórce
Cząsteczka dsRNA powstaje dzięki obecności polimerazy
RNA zależnej od RNA (RdRp – RNA dependent RNA
polymerase) lub można ją wytworzyć sztucznie
dsRNA są rozpoznawane i rozcinane przez specyficzną
endonukleazę „Dicer” na małe dwuniciowe fragmenty 21-25
nukleotydów – siRNA (lub miRNA)
Dwuniciowe siRNA/miRNA wiąże się z kompleksem
rybonukleaz tworząc indukowany przez RNA kompleks
wyciszający (RISC – RNA-induced silencing complex)
Aktywacja kompleksu RISC wymaga rozwinięcia siRNA
Aktywny RISC „odnajduje” komplementarny mRNA i
przecina go w odległości 12 nukleotydów od końca 3’ siRNA
Pierwotne siRNA/miRNA mogą być starterami do
amplifikacji dsRNA przez RdRp (jako matryca służy
wyciszany mRNA lub wprowadzony sztucznie dsRNA), co
prowadzi do powstania wtórnych siRNA i rozprzestrzenienia
się sygnału

Regulacja ekspresji genów
eukariotycznych
z udziałem zjawiska RNAi
2. Mechanizmy wyciszania genów
Wyciszanie potranskrypcyjne (PTGS) – indukcja degradacji
mRNA, blokowanie translacji
Wyciszanie transkrypcyjne (TGS) – indukcja zmian w
strukturze chromatyny
3. Znaczenie biologiczne
Reakcje odporności na wirusy
Regulacja procesów wzrostu i rozwoju
Zaprogramowana eliminacja DNA
Represja/depresja genów w obszarze heterochromatyny
4. Wykorzystanie
Analiza funkcjonalna genomów przez wyciszanie ekspresji
wybranych genów
Intensyfikacja ekspresji wybranych genów przez
wykorzystanie supresorów RNAi
Jeszcze kilkanaście lat temu nikt nie przypuszczał,
że w komórce istnieje mechanizm specyficznego
hamowania pracy genów
za pomocą cząsteczek RNA. Dziś interferencja RNA
rewolucjonizuje wiele dziedzin nauki.
Stary ewolucyjnie i szeroko rozpowszechniony wśród
organizmów system regulacji pracy genów, pełniący także
rolę strażnika genomu zwany interferencją RNA (RNAi)
przez długi czas umykał uwadze naukowców. Jeszcze
kilkanaście lat temu nikt nie przypuszczał, że aby
„wyłączyć” gen, wystarczy „zabić” jego posłańca, czyli
zniszczyć cząsteczkę mRNA (messenger RNA), która
pośredniczy w przepływie informacji genetycznej między
genem a jego produktem. Obecnie interferencja RNA,
która polega właśnie na „wyłączaniu” określonych genów
na poziomie ich mRNA, stanowi coraz powszechniej
stosowane narzędzie do ich badania. Co więcej, niesie
nadzieję na wytworzenie skutecznych leków przeciwko
nowotworom, AIDS i innym chorobom. W zasadzie
wszędzie tam, gdzie trzeba wyłączyć jakiś gen, można
wykorzystać RNAi. „Science” okrzyknęło interferencję
RNA przełomem roku 2002, od tamtej pory jej znaczenie
ciągle wzrasta.
A w jaki sposób udało się wpaść na trop tego zjawiska? Po
prostu w pewnym doświadczeniu uzyskano wyniki
odwrotne do zamierzonych.

Za odkrycie zjawiska interferencji RNA
amerykańcy naukowcy
Fire i
Mello otrzymali w
Nagrodę Nobla w dziedzinie medycyny i fizj
ologii
. Dzięki badaniom i odkryciom noblistów
poznano fundamentalny mechanizm
kontroli przepływu informacji genetycznej,
które może mieć zastosowanie w
. Już udało się wyciszyć gen
odpowiedzialny za podwyższony poziom
cholesterolu u zwierząt. W przyszłości
będzie można opracować skuteczne metody
leczenia chorób genetycznych czy
nowotworów, a także nowe odmiany
zwierząt i roślin hodowlanych.
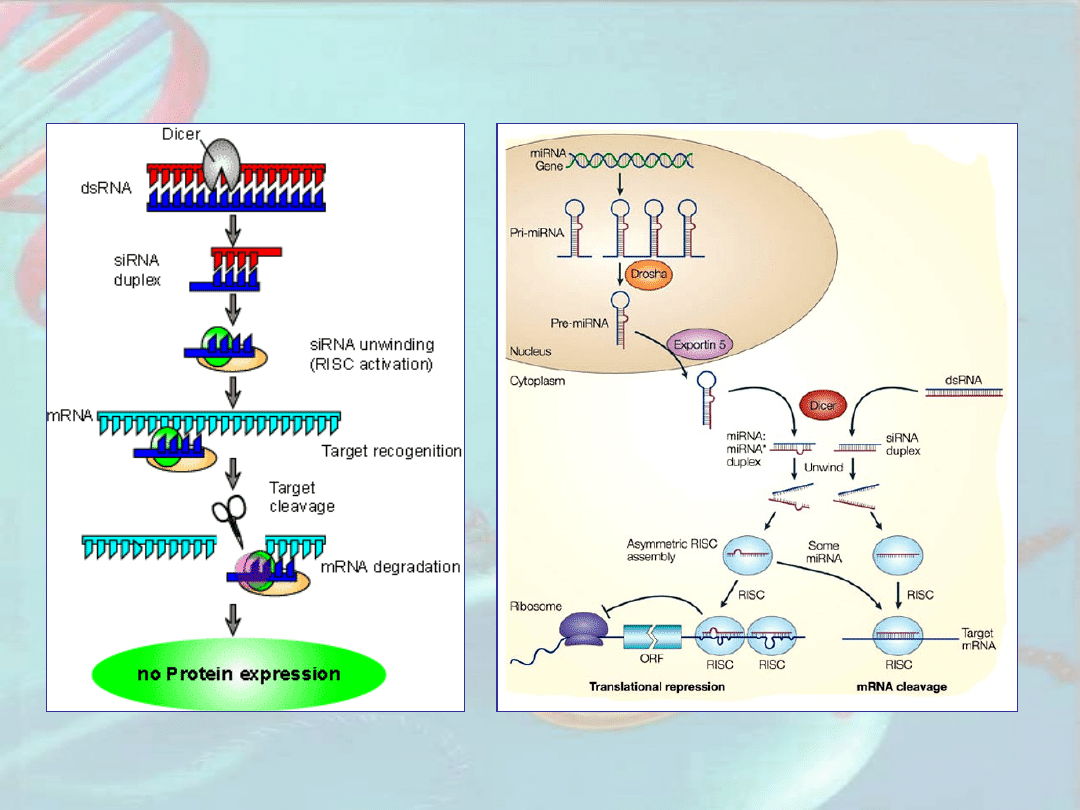
Regulacja ekspresji genów eukariotycznych z
udziałem RNAi
siRNA
miRN
A

http://www.blc.arizona.edu/
http://www.ndsu.nodak.edu
http://www.genomicobject.net
http://www-biology.ucsd.edu/
http://www.ndsu.nodak.edu/
http://opbs.okstate.edu/
http://home.houston.rr.com/
http://www.utexas.edu/courses/
http://www.sicklecellinfo.net/
http://www.unc.edu/~lviscrst/
http://bio.winona.edu/bates/Bio241/
http://www.people.virginia.edu/
http://www.cbs.dtu.dk/staff/dave/roanoke/
http://departments.oxy.edu/biology/
http://sickle.bwh.harvard.edu/
http://www.rnai.dk/
http://home.houston.rr.com/
http://www.utexas.edu/courses/
http://www.sicklecellinfo.net/
http://www.unc.edu/~lviscrst/
http://bio.winona.edu/bates/Bio241/
http://www.people.virginia.edu/
http://www.cbs.dtu.dk/staff/dave/roanoke/
http://departments.oxy.edu/biology/
http://sickle.bwh.harvard.edu/
http://www.rnai.dk/
Rozwojowa regulacja ekspresji genów
globinowych
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
- Slide 51
- Slide 52
- Slide 53
- Slide 54
- Slide 55
- Slide 56
- Slide 57
Wyszukiwarka
Podobne podstrony:
wykład5 reg ekspr I proc
MIKRO ŚCIĄGI Z WYKŁADU, studia, studia II rok, mikrobiologia, mikro egz, Ściągi RAZY 2
Położnictwo wykład 4, Pielęgniarstwo, rok II, położnictwo, wykłady
Wyklad GENY, Koło II GENY
Rola panstwa w gospodarce, Finanse i rachunkowość UMK notatki wykłady pytania egzaminy, II część, Ma
pieniadze i banki, Finanse i rachunkowość UMK notatki wykłady pytania egzaminy, II część, Makroekono
Wykład III cz II moder kot
ZARZĄDZANIE STRATEGICZNE - WYKŁADY, UE Katowice, II stopień sem2, ZARZĄDZANIE STRATEGICZNE
POLITYKA GOSPODARCZA WYKŁADY, FIR UMCS II°, Polityka gospodarcza
logistyka wykłady, UE Katowice, II stopień sem2, LOGISTYKA
Geologia... - wykład 2, Polibuda, Semestr II, Geologia, Wykłady
Geologia... - wykład 2, Polibuda, Semestr II, Geologia, Wykłady
Makroekonomia wykład 1, zarządzanie, Makroekonomia II
wyklad 1 stany nieustalone II rzedu cz1
Polityka wzrostu i stabilnoci (S M), Finanse i rachunkowość UMK notatki wykłady pytania egzaminy, II
procesy poznawcze wyklad 13, Psychologia, II Semestr, Procesy Poznawcze
Wykład I socjologia, Studia II stopnia, SOCJOLOGIA notatki
więcej podobnych podstron